아연손가락
Zinc finger
아연 핑거는 접힌 부분을 안정시키기 위해 하나 이상의 아연 이온(Zn2+)의 배위를 특징으로 하는 작은 단백질 구조 모티브이다.이것은 원래 아프리카발톱개구리(Xenopus laevis) 전사인자 IIIA로부터 가설된 구조의 손가락 같은 모습을 묘사하기 위해 만들어졌다.그러나, 그것은 진핵 [1]세포에서 다양한 단백질 구조를 포함하는 것으로 밝혀졌다.Xenopus laevis TFIIIA는 1983년에 아연을 포함하고 기능하기 위해 금속을 필요로 하는 것으로 원래 입증되었으며, 유전자 조절[2][3] 단백질에 대한 아연 요구 사항이 보고된 후 드로소필라의 [4]크뤼펠 인자가 곧 뒤따랐다.그것은 종종 다중 도메인 [3]단백질에서 금속 결합 도메인으로 나타난다.
아연 핑거(아연 핑거 단백질)를 포함하는 단백질은 몇 가지 다른 구조 계열로 분류됩니다.그리스 열쇠나 β 머리핀과 같이 명확하게 정의된 많은 초 이차 구조와는 달리, 아연 핑거에는 여러 가지 종류가 있으며, 각각 독특한 3차원 구조를 가지고 있다.특정 아연 핑거 단백질의 클래스는 이 3차원 구조에 의해 결정되지만, 단백질의 1차 구조 또는 아연 이온을 배위하는 리간드의 동일성에 근거해 인식될 수도 있다.그러나, 이러한 단백질의 큰 다양성에도 불구하고, 대부분의 단백질은 전형적으로 DNA, RNA, 단백질 또는 다른 작고 유용한 분자들과 결합하는 상호작용 모듈로서 기능하며, 구조의 변화는 주로 특정 단백질의 결합 특이성을 변화시키는 역할을 한다.
이들의 최초 발견과 구조의 해명 이후, 이러한 상호작용 모듈은 생물학 세계 어디에서나 볼 수 있는 것으로 입증되었으며 인간 [5]게놈의 유전자 중 3%에서 발견될 수 있다.게다가, 아연 손가락은 다양한 치료 및 연구 능력에 매우 유용하게 쓰이게 되었다.특정 배열에 대한 친화성을 갖는 아연 핑거는 활발한 연구 영역이며, 아연 핑거 핵산 분해효소 및 아연 핑거 전사 인자는 지금까지 실현된 가장 중요한 응용 중 두 가지이다.
역사
아연 손가락은 Aaron Klug의 연구실에서 아프리카 발톱이 있는 개구리 Xenopus laevis의 전사 연구에서 처음 확인되었습니다.특정 RNA 배열의 전사에 대한 연구는 작은 전사인자(전사인자 IIIA; TFIIIA)[6]의 결합 강도가 손가락과 같은 구조의 존재에 기인한다는 것을 밝혀냈다.TFIIA의 아미노산 배열 분석 결과 시스테인과 히스티딘 잔기의 불변 쌍 2개를 포함한 30개 아미노산의 9개 연속 배열이 밝혀졌다.확장된 X선 흡수 미세 구조는 아연 배위자의 정체를 확인하였다: 2개의 시스테인과 2개의 히스티딘.[5]아연에 의한 이들 배위자의 배위에 의해 형성된 DNA 결합 루프는 손가락과 비슷하다고 생각되어 이름이 [1]붙여졌다.그 직후 1986년 [4]슈 팀에 의해 드로소필라에서 크뤼펠 인자가 발견되었다.다양한 유기체의 단백질 특성화에 대한 보다 최근의 연구는 폴리펩타이드 [7][8]안정화에 있어 아연 이온의 중요성을 밝혀냈다.
1991년과 1993년에 해결된 아연 핑거-DNA 복합체의 결정 구조는 [9][10]아연 핑거와 DNA의 상호작용의 표준 패턴을 밝혀냈다.아연 집게의 결합은 이중 나선의 2배 대칭을 통해 DNA를 결합하는 많은 다른 DNA 결합 단백질과 구별되는 것으로 발견되었으며, 대신 아연 집게는 길이가 [5]다양한 핵산 염기서열을 결합하기 위해 선형으로 연결되어 있다.아연 손가락은 종종 GC [11]박스로 알려진 일련의 DNA에 결합합니다.아연 핑거 모티브의 모듈러 특성은 많은 DNA와 RNA 배열의 조합이 높은 친화력과 특이성으로 결합될 수 있도록 하며, 따라서 특정 DNA 배열을 목표로 하고 결합할 수 있는 엔지니어링 단백질에 이상적입니다.1994년, 인공적으로 구성된 세 손가락 단백질이 마우스 세포주에서의 종양 유전자의 발현을 차단할 수 있는 것으로 나타났다.아연 핑거가 다양한 다른 이펙터 도메인으로 융합되었으며, 일부는 치료적 의미가 있다.[5]
도메인
아연 핑거(Znf) 도메인은 표적 분자와 탠덤 접촉하는 여러 손가락 모양의 돌기를 포함하는 비교적 작은 단백질 모티브이다.이러한 도메인 중 일부는 아연을 결합하지만, 많은 도메인은 아연을 결합하지 않고, 대신 철과 같은 다른 금속을 결합하거나 금속을 결합하지 않습니다.예를 들어, 어떤 가족 구성원들은 손가락 같은 주름을 안정시키기 위해 소금 다리를 형성한다.이들은 처음에는 Xenopus laevis(아프리카 발톱 개구리)의 전사 인자 TFIIIA에서 DNA 결합 모티브로 확인되었지만, 현재는 DNA, RNA, 단백질 및/또는 지질 [12][13][14][15][16]기질에 결합하는 것으로 인식되고 있다.이들의 결합 특성은 손가락 영역의 아미노산 배열과 손가락 사이의 링커, 그리고 고차 구조와 손가락 수에 따라 달라집니다.Znf 도메인은 종종 클러스터에서 발견되며 핑거가 서로 다른 바인딩 사양을 가질 수 있습니다.Znf 모티브는 순서와 구조에서 모두 다른 여러 관련 없는 단백질 수퍼패밀리에서 발생한다.이들은 같은 등급의 구성원(예: 일부 결합 DNA, 다른 단백질) 사이에서도 결합 모드에서 상당한 다용도를 나타내며, Znf 모티브가 전문화된 기능을 진화시킨 안정적인 발판임을 시사한다.예를 들어 Znf 함유 단백질은 유전자 전사, 번역, mRNA 밀매, 세포골격 조직, 상피 발달, 세포 접착, 단백질 접힘, 염색질 리모델링, 아연 감지 등에서 기능한다.[17]아연 결합 모티브는 안정적인 구조이며, 목표물을 결합할 때 구조 변화를 거의 겪지 않는다.
반
처음에 아연 핑거라는 용어는 Xenopus laevis에서 발견된 DNA 결합 모티브를 설명하는 데만 사용되었지만, 현재는 아연 이온의 배위에 의해 관련된 모든 구조들을 지칭하는 데 사용됩니다.일반적으로 아연 핑거는 아연 이온을 시스테인과 히스티딘 잔기의 조합으로 배위시킨다.원래 이러한 잔류물의 수와 순서는 다양한 유형의 아연 핑거(예: CysHis22, Cys4 및 Cys6)를 분류하는 데 사용되었습니다.보다 최근에는 아연 손가락 단백질을 분류하기 위해 보다 체계적인 방법이 사용되었습니다.이 방법은 아연 손가락 단백질을 접힌 영역의 단백질 골격의 전체적인 모양에 따라 "접힌 그룹"으로 분류합니다.아연 핑거의 가장 일반적인 "접이식 그룹"은 CysHis-like22 ("고전 아연 핑거"), 고음 음표 및 아연 [18]리본입니다.
다음[18] 표에서는 다양한 구조와 주요 기능을 보여 줍니다.
| 그룹 접기 | 대표구조 | 배위자 배치 |
|---|---|---|
| Cys2His2 |  | 관절에서 2개의 리간드와 나선의 종단에서 2개의 리간드가 더 있습니다. |
| 개그너클 |  | 손가락 관절에서 2개의 리간드와 짧은 나선 또는 루프에서 2개의 리간드가 더 있습니다. |
| 고음 기호 | 관절에서 2개의 리간드와 나선의 N 말단에서 2개의 리간드가 더 있습니다. | |
| 아연 리본 | 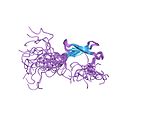 | 손가락 관절 두 개에서 각각 두 개의 리간드. |
| Zn2/Cys6 | 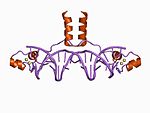 | 2개의 리간드는 나선의 N단으로부터, 2개의 리간드는 루프로부터 각각 송신됩니다. |
| TAZ2 도메인과 같은 | 두 개의 나선형의 흰개미에서 나온 두 개의 배위자. |
Cys2His2
| 아연 핑거, C2H2 타입 | |||||||||
|---|---|---|---|---|---|---|---|---|---|
| 식별자 | |||||||||
| 기호. | zf-C2H2 | ||||||||
| 팜 | PF00096 | ||||||||
| 빠맘 클랜 | CL0361 | ||||||||
| 인터프로 | IPR007087 | ||||||||
| 프로 사이트 | PS00028 | ||||||||
| |||||||||
CysHis-like22 fold group(C2H2)은 지금까지 가장 잘 특징지어진 아연 핑거 클래스이며 포유류의 전사 인자에서 일반적입니다.이러한 도메인은 단순한 ββα 접힘을 채택하고 아미노산 배열 [19]모티브를 가진다.
- X2-Cys-X2,4-Cys-X12-His-X3,4,5-His
아연 손가락의 이 등급은 RNA 결합과 단백질-단백질 상호작용을 매개하는 것과 같은 다양한 기능을 가질 수 있지만, Zif268과 같은 배열 특이적 DNA 결합 단백질에서의 역할로 가장 잘 알려져 있다.이러한 단백질에서 개별 아연 핑거 도메인은 일반적으로 단백질의 DNA 결합 도메인을 구성하는 2개 또는 3개 이상의 핑거와 연속적으로 반복되면서 발생한다.이러한 탠덤 배열은 DNA의 주요 홈에 결합할 수 있으며 일반적으로 3bp 간격으로 배치됩니다.각 도메인의 α-나선(종 "인식 나선"이라고 함)은 DNA 염기에 배열 특이적으로 접촉할 수 있다. 단일 인식 나선에서 잔류물은 인접한 아연 핑거와 겹치는 접촉 패턴을 생성하기 위해 4개 이상의 염기와 접촉할 수 있다.
개그너클
| 아연 너클 | |||||||||
|---|---|---|---|---|---|---|---|---|---|
| 식별자 | |||||||||
| 기호. | zf-CCHC | ||||||||
| 팜 | PF00098 | ||||||||
| 인터프로 | IPR001878 | ||||||||
| 스마트 | SM00343 | ||||||||
| 프로 사이트 | PS50158 | ||||||||
| |||||||||
이 접힘 그룹은 회전(아연 너클)에 이어 짧은 나선 또는 루프로 연결된 두 개의 짧은 β-스트랜드로 정의되며, 나선의 많은 부분과 β-헤어핀이 잘린 고전적인 CysHis22 모티브와 유사하다.
HIV 및 다른 관련 레트로바이러스로부터의 레트로바이러스 뉴클레오캡시드(NC) 단백질은 이러한 모티브를 가진 단백질의 예이다.HIV NC 단백질에 있는 개그너클 아연 손가락은 아연 손가락 억제제로 알려진 약물의 대상이다.
고음 음소음
트레블 클리프 모티브는 N 말단의 β-헤어핀과 C 말단의 α-헬릭스 각각에 아연 결합을 위한 두 개의 배위자를 기여하는 C 말단의 β-헬릭스로 구성되지만, 길이와 구성이 다양한 루프와 두 번째 β-헤어핀이 N 말단 β-헤어핀과 C 말단 α-헬릭스 사이에 존재할 수 있다.이 손가락들은 종종 서로 배열이나 기능적 유사성을 공유하지 않는 다양한 단백질 그룹에 존재한다.고음-고음-아연 손가락을 포함하는 가장 특징적인 단백질은 핵호르몬 수용체이다.
아연 리본
| TFIIB 아연 결합 | |||||||||
|---|---|---|---|---|---|---|---|---|---|
| 식별자 | |||||||||
| 기호. | TF_Zn_리본 | ||||||||
| 팜 | PF08271 | ||||||||
| 빠맘 클랜 | Zn_Beta_Ribbon | ||||||||
| 인터프로 | IPR013137 | ||||||||
| 프로 사이트 | PS51134 | ||||||||
| |||||||||
아연 리본 접힘은 구조적으로 유사한 두 개의 아연 결합 서브사이트를 형성하는 두 개의 베타 헤어핀으로 특징지어진다.
Zn2/Cys6
| 곰팡이 Zn(2)-Cys(6) 2핵 클러스터 도메인 | |||||||||
|---|---|---|---|---|---|---|---|---|---|
| 식별자 | |||||||||
| 기호. | Zn_clus | ||||||||
| 팜 | PF00172 | ||||||||
| 인터프로 | IPR001138 | ||||||||
| 스마트 | GAL4 | ||||||||
| 프로 사이트 | PS00463 | ||||||||
| CDD | cd00067 | ||||||||
| |||||||||
이 세분류의 표준 구성원은 2개의 아연 이온이 6개의 시스테인 잔기에 의해 결합되는 2핵 아연 클러스터를 포함한다.이 아연 집게는 효모 Gal4 단백질을 포함한 여러 전사인자에서 찾을 수 있습니다.
| zf-C2HC | |||||||||
|---|---|---|---|---|---|---|---|---|---|
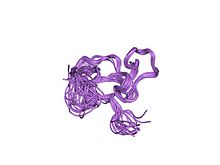 신경 아연 핑거 인자-1의 cchhc 도메인 용액 구조 | |||||||||
| 식별자 | |||||||||
| 기호. | zf-C2HC | ||||||||
| 팜 | PF01530 | ||||||||
| 인터프로 | IPR002515 | ||||||||
| |||||||||
| zf-C2HC5 | |||||||||
|---|---|---|---|---|---|---|---|---|---|
| 식별자 | |||||||||
| 기호. | zf-C2HC5 | ||||||||
| 팜 | PF06221 | ||||||||
| 인터프로 | IPR009349 | ||||||||
| |||||||||
여러가지 종류의
아연 핑거 항바이러스 단백질(ZAP)은 CpG 부위에 결합합니다.그것은 항바이러스 [20][21]방어용으로 포유류에 사용된다.
적용들
아연[19] 핑거의 DNA 결합특성을 변화시키기 위해 다양한 단백질 공학적 기술을 사용할 수 있으며, 이러한 공학적 아연 핑거의 탠덤 반복을 사용하여 원하는 게놈 DNA [22]배열을 목표로 할 수 있다.특정 유전자의 프로모터 근방에 결합하는 공학적 아연 핑거 배열에 전사활성제 또는 억제제 등의 제2단백질 도메인을 융합함으로써 해당 [22]유전자의 전사를 변경할 수 있다.DNA를 절단하거나 다른 방법으로 수정하는 아연 핑거 어레이와 단백질 도메인 사이의 융합은 또한 그러한 활동을 원하는 게놈 궤적으로 표적화하는 데 사용될 수 있다.[22]엔지니어링된 아연 핑거 어레이의 가장 일반적인 적용에는 아연 핑거 전사 인자와 아연 핑거 핵산 분해 효소가 포함되지만, 다른 적용도 설명되었습니다.전형적인 공학적 아연 핑거 어레이는 3개에서 6개 사이의 개별 아연 핑거 모티브를 가지며 길이가 9개에서 18개의 베이스 페어에 이르는 결합 대상 부위가 있다.6개의 아연 핑거 모티브를 가진 배열은 [23]포유류의 게놈에서 특이할 가능성이 충분히 있는 표적 부위와 결합하기 때문에 특히 매력적입니다.
아연핑거핵산가수분해효소
공학적 아연 핑거 어레이는 종종 DNA 분할 영역(일반적으로 FokI의 분할 영역)에 융합되어 아연 핑거 핵산 분해 효소를 생성한다.이러한 아연 손가락-FokI 융합은 드로소필라 멜라노가스터, 케노하브디티스 엘레강스, 담배, 옥수수,[24] 제브라피쉬,[25] 다양한 종류의 포유동물 세포,[26] [27]쥐를 포함한 많은 고등 유기체의 게놈을 조작하는 데 유용한 시약이 되었다.원하는 게놈 궤적에 대한 이중사슬 절단을 목표로 하는 것은 비호몰로지 DNA 수복 경로의 오류 발생 가능성이 높은 성질 때문에 유전자의 코드 배열에 프레임 시프트 돌연변이를 도입하기 위해 사용될 수 있다.상동 DNA "공여 배열"도 사용될 경우, 유전체 궤적은 상동성 유도 복구 경로를 통해 정의된 배열로 변환될 수 있다.현재 진행 중인 임상시험은 인간 CD4+ T세포의 CCR5 유전자를 교란시키는 아연 핑거 핵산가수분해효소를 HIV/[28]AIDS의 잠재적 치료제로 평가하고 있다.
아연 핑거 어레이 엔지니어링 방법
일부 그룹은 인간 전사인자 SP1에 기초한 아연 핑거 어레이를 사용했지만 대부분의 공학적 아연 핑거 어레이는 뮤린 전사인자 Zif268의 아연 핑거 도메인에 기초하고 있다. Zif268은 9bp 시퀀스를 높은 [29]친화성으로 집합적으로 결합하는 3개의 개별 아연 핑거 모티브를 가지고 있다.DNA에 결합된 이 단백질의 구조는 1991년에[9] 해결되었고 공학적 아연 핑거 어레이에 대한 많은 연구를 자극했습니다.1994년과 1995년에 많은 그룹이 파지 디스플레이를 사용하여 [30][31][32][33]Zif268의 단일 아연 핑거의 특이성을 변경했다.현재 공학적 아연 핑거 어레이를 생성하기 위해 사용되는 두 가지 주요 방법, 모듈러 어셈블리 및 세균 선택 시스템이 있으며, 대부분의 용도에 [34][35]가장 적합한 방법에 대한 논란이 있습니다.
새로운 아연 핑거 어레이를 생성하는 가장 간단한 방법은 알려진 특이성의 작은 아연 핑거 "모듈"을 결합하는 것입니다.파블레티치와 파보가 1991년 발표한 DNA에 결합된 아연 핑거 단백질 Zif268의 구조는 이 연구의 많은 부분을 차지했으며 64개의 가능한 염기쌍 세쌍 각각에 대해 손가락을 얻은 다음 원하는 [9]배열 특이성을 가진 단백질을 설계하기 위해 이들 손가락을 혼합하고 맞추는 개념을 설명한다.가장 일반적인 모듈러 조립 프로세스에서는 각각 3베이스쌍의 DNA 시퀀스를 인식할 수 있는 개별 아연 핑거를 조합하여 9베이스쌍에서 18베이스쌍까지의 타깃사이트를 인식하는 3핑거, 4핑거, 5핑거 또는 6핑거 어레이를 생성합니다.또 다른 방법은 2개의 핑거 모듈을 사용하여 최대 6개의 개별 아연 [24]핑거로 아연 핑거 어레이를 생성합니다.스크립스 연구소의 Barbas Laboratory는 파지 디스플레이를 사용하여 대부분의 DNA 세쌍의[36][37][38] 염기서열을 인식하는 아연 핑거 도메인을 개발하고 특징짓는 한편, 다른 그룹은 인간 [39]게놈에서 개별 손가락을 분리하고 특징짓습니다.일반적으로 모듈러 조립의 잠재적인 단점은 개별 아연 핑거의 특이성이 겹칠 수 있고 주변의 아연 핑거와 DNA의 맥락에 따라 달라질 수 있다는 것입니다.최근 연구에 따르면 모듈러 조립에 의해 생성된 3개 손가락 아연 핑거 어레이의 높은 비율이 세균 2-하이브리드 분석에서 의도한 표적을 충분한 친화력으로 결합하지 못하고 아연 핑거 핵산 분해효소로서 기능하지 못했지만 GNNGNN 형태의 부위가 [40]표적화되었을 때 성공률이 다소 높았다.
후속 연구에서는 모듈러 어셈블리를 사용하여 3핑거 어레이와 4핑거 어레이 모두에서 아연 핑거 핵산가수분해효소를 생성했으며, [41]4핑거 어레이에서 훨씬 높은 성공률을 보였습니다.인접한 손가락의 맥락을 고려하는 모듈러 어셈블리의 변형도 보고되었으며, 이 방법은 표준 모듈러 [42]어셈블리에 비해 개선된 성능을 가진 단백질을 생산하는 경향이 있다.
원하는 시퀀스를 타겟팅할 수 있는 아연 핑거 어레이를 생성하기 위해 수많은 선택 방법이 사용되었습니다.초기 선택 노력은 부분적으로 무작위화된 아연 핑거 어레이의 큰 풀에서 주어진 DNA 표적을 결합하는 단백질을 선택하기 위해 페이지 디스플레이를 이용했다.이 기술은 한 번에 한 개 이상의 아연 핑거에 사용하기 어렵기 때문에 한 번에 한 개의 아연 핑거를 추가하고 최적화함으로써 완전히 최적화된 3개 핑거 어레이를 생성하는 다단계 공정이 [43]개발되었습니다.보다 최근의 노력들은 효모 1-하이브리드 시스템, 박테리아 1-하이브리드 및 2-하이브리드 시스템, 그리고 포유동물 세포를 이용했다.새로운 3 손가락 아연 핑거 어레이를 선택하는 유망한 새로운 방법은 박테리아 2 하이브리드 시스템을 사용하여 [44]개발자들에 의해 "OPEN"으로 불리고 있습니다.이 시스템은 미리 선택된 개별 아연 핑거 풀을 조합하여 각각 특정 트리플렛을 결합하고 두 번째 선택 라운드를 사용하여 원하는 9-bp 시퀀스를 결합할 수 있는 3개의 핑거 어레이를 얻습니다.이 시스템은 아연 핑거 컨소시엄에 의해 엔지니어링된 아연 핑거 어레이의 상업적 공급원에 대한 대안으로 개발되었습니다.OPEN법에 의해 생성된 단백질의 특이성 프로파일이 보고된 적이 없기 때문에 이 방법으로 생성된 단백질과 모듈러 조립에 의해 생성된 단백질의 결합 특성을 직접 비교하는 것은 다소 어렵다.
예
이 항목은 진핵생물에서 발견되는 CysCysHisCys(C2HC) 유형의 아연 핑거 도메인을 나타냅니다.이러한 도메인을 포함하는 단백질은 다음과 같습니다.
「 」를 참조해 주세요.
레퍼런스
- ^ a b Klug A, Rhodes D (1987). "Zinc fingers: a novel protein fold for nucleic acid recognition". Cold Spring Harbor Symposia on Quantitative Biology. 52: 473–82. doi:10.1101/sqb.1987.052.01.054. PMID 3135979.
- ^ Hanas JS, Hazuda DJ, Bogenhagen DF, Wu FY, Wu CW (December 1983). "Xenopus transcription factor A requires zinc for binding to the 5 S RNA gene". The Journal of Biological Chemistry. 258 (23): 14120–5. doi:10.1016/S0021-9258(17)43831-2. PMID 6196359.
- ^ a b Berg JM (April 1990). "Zinc fingers and other metal-binding domains. Elements for interactions between macromolecules". The Journal of Biological Chemistry. 265 (12): 6513–6. doi:10.1016/S0021-9258(19)39172-0. PMID 2108957.
- ^ a b Bruno M, Mahgoub M, Macfarlan TS (December 2019). "The Arms Race Between KRAB-Zinc Finger Proteins and Endogenous Retroelements and Its Impact on Mammals". Annual Review of Genetics. Annual Reviews. 53 (1): 393–416. doi:10.1146/annurev-genet-112618-043717. PMID 31518518. S2CID 202572327.
- ^ a b c d Klug A (2010). "The discovery of zinc fingers and their applications in gene regulation and genome manipulation". Annual Review of Biochemistry. 79: 213–31. doi:10.1146/annurev-biochem-010909-095056. PMID 20192761. – 연차 리뷰 (서브스크립션 필요)
- ^ Miller J, McLachlan AD, Klug A (June 1985). "Repetitive zinc-binding domains in the protein transcription factor IIIA from Xenopus oocytes". The EMBO Journal. 4 (6): 1609–14. doi:10.1002/j.1460-2075.1985.tb03825.x. PMC 554390. PMID 4040853.
- ^ Miller Y, Ma B, Nussinov R (May 2010). "Zinc ions promote Alzheimer Abeta aggregation via population shift of polymorphic states". Proceedings of the National Academy of Sciences of the United States of America. 107 (21): 9490–5. Bibcode:2010PNAS..107.9490M. doi:10.1073/pnas.0913114107. PMC 2906839. PMID 20448202.
- ^ Low LY, Hernández H, Robinson CV, O'Brien R, Grossmann JG, Ladbury JE, Luisi B (May 2002). "Metal-dependent folding and stability of nuclear hormone receptor DNA-binding domains". Journal of Molecular Biology. 319 (1): 87–106. doi:10.1016/S0022-2836(02)00236-X. PMID 12051939.
- ^ a b c Pavletich NP, Pabo CO (May 1991). "Zinc finger-DNA recognition: crystal structure of a Zif268-DNA complex at 2.1 A". Science. 252 (5007): 809–17. Bibcode:1991Sci...252..809P. doi:10.1126/science.2028256. PMID 2028256. S2CID 38000717.
- ^ Fairall L, Schwabe JW, Chapman L, Finch JT, Rhodes D (December 1993). "The crystal structure of a two zinc-finger peptide reveals an extension to the rules for zinc-finger/DNA recognition". Nature. 366 (6454): 483–7. Bibcode:1993Natur.366..483F. doi:10.1038/366483a0. PMID 8247159. S2CID 4371511.
- ^ Lundin M, Nehlin JO, Ronne H (March 1994). "Importance of a flanking AT-rich region in target site recognition by the GC box-binding zinc finger protein MIG1". Molecular and Cellular Biology. 14 (3): 1979–85. doi:10.1128/MCB.14.3.1979. PMC 358557. PMID 8114729.
- ^ Klug A (October 1999). "Zinc finger peptides for the regulation of gene expression". Journal of Molecular Biology. 293 (2): 215–8. doi:10.1006/jmbi.1999.3007. PMID 10529348.
- ^ Hall TM (June 2005). "Multiple modes of RNA recognition by zinc finger proteins". Current Opinion in Structural Biology. 15 (3): 367–73. doi:10.1016/j.sbi.2005.04.004. PMID 15963892.
- ^ Brown RS (February 2005). "Zinc finger proteins: getting a grip on RNA". Current Opinion in Structural Biology. 15 (1): 94–8. doi:10.1016/j.sbi.2005.01.006. PMID 15718139.
- ^ Gamsjaeger R, Liew CK, Loughlin FE, Crossley M, Mackay JP (February 2007). "Sticky fingers: zinc-fingers as protein-recognition motifs". Trends in Biochemical Sciences. 32 (2): 63–70. doi:10.1016/j.tibs.2006.12.007. PMID 17210253.
- ^ Matthews JM, Sunde M (December 2002). "Zinc fingers--folds for many occasions". IUBMB Life. 54 (6): 351–5. doi:10.1080/15216540216035. PMID 12665246. S2CID 22109146.
- ^ Laity JH, Lee BM, Wright PE (February 2001). "Zinc finger proteins: new insights into structural and functional diversity". Current Opinion in Structural Biology. 11 (1): 39–46. doi:10.1016/S0959-440X(00)00167-6. PMID 11179890.
- ^ a b Krishna SS, Majumdar I, Grishin NV (January 2003). "Structural classification of zinc fingers: survey and summary". Nucleic Acids Research. 31 (2): 532–50. doi:10.1093/nar/gkg161. PMC 140525. PMID 12527760.
- ^ a b Pabo CO, Peisach E, Grant RA (2001). "Design and selection of novel Cys2His2 zinc finger proteins". Annual Review of Biochemistry. 70: 313–40. doi:10.1146/annurev.biochem.70.1.313. PMID 11395410.
- ^ 쉬화샤: 사스-CoV-2의 유전체 CpG 결핍과 숙주 항바이러스 방어 회피.인: 분자생물학과 진화학회지, 2020년 4월 14일, doi:10.1093/molbev/msaa094
- ^ 유기견이 COVID-19 대유행의 원인일 수 있다는 증거.온: SciTech Daily, 2020년 4월 14일.출처:오타와 대학교
- ^ a b c Jamieson AC, Miller JC, Pabo CO (May 2003). "Drug discovery with engineered zinc-finger proteins". Nature Reviews. Drug Discovery. 2 (5): 361–8. doi:10.1038/nrd1087. PMID 12750739. S2CID 6417869.
- ^ Liu Q, Segal DJ, Ghiara JB, Barbas CF (May 1997). "Design of polydactyl zinc-finger proteins for unique addressing within complex genomes". Proceedings of the National Academy of Sciences of the United States of America. 94 (11): 5525–30. Bibcode:1997PNAS...94.5525L. doi:10.1073/pnas.94.11.5525. PMC 20811. PMID 9159105.
- ^ a b Shukla VK, Doyon Y, Miller JC, DeKelver RC, Moehle EA, Worden SE, Mitchell JC, Arnold NL, Gopalan S, Meng X, Choi VM, Rock JM, Wu YY, Katibah GE, Zhifang G, McCaskill D, Simpson MA, Blakeslee B, Greenwalt SA, Butler HJ, Hinkley SJ, Zhang L, Rebar EJ, Gregory PD, Urnov FD (May 2009). "Precise genome modification in the crop species Zea mays using zinc-finger nucleases". Nature. 459 (7245): 437–41. Bibcode:2009Natur.459..437S. doi:10.1038/nature07992. PMID 19404259. S2CID 4323298.
- ^ Reynolds IJ, Miller RJ (December 1988). "[3H]MK801 binding to the N-methyl-D-aspartate receptor reveals drug interactions with the zinc and magnesium binding sites". The Journal of Pharmacology and Experimental Therapeutics. 247 (3): 1025–31. PMID 2849655.
- ^ Carroll D (November 2008). "Progress and prospects: zinc-finger nucleases as gene therapy agents". Gene Therapy. 15 (22): 1463–8. doi:10.1038/gt.2008.145. PMC 2747807. PMID 18784746.
- ^ Geurts AM, Cost GJ, Freyvert Y, Zeitler B, Miller JC, Choi VM, Jenkins SS, Wood A, Cui X, Meng X, Vincent A, Lam S, Michalkiewicz M, Schilling R, Foeckler J, Kalloway S, Weiler H, Ménoret S, Anegon I, Davis GD, Zhang L, Rebar EJ, Gregory PD, Urnov FD, Jacob HJ, Buelow R (July 2009). "Knockout rats via embryo microinjection of zinc-finger nucleases". Science. 325 (5939): 433. Bibcode:2009Sci...325..433G. doi:10.1126/science.1172447. PMC 2831805. PMID 19628861.
- ^ Tebas P, Stein D (2009). "Autologous T-Cells Genetically Modified at the CCR5 Gene by Zinc Finger Nucleases SB-728 for HIV". ClinicalTrials.gov.
{{cite journal}}:Cite 저널 요구 사항journal=(도움말) - ^ Christy B, Nathans D (November 1989). "DNA binding site of the growth factor-inducible protein Zif268". Proceedings of the National Academy of Sciences of the United States of America. 86 (22): 8737–41. Bibcode:1989PNAS...86.8737C. doi:10.1073/pnas.86.22.8737. PMC 298363. PMID 2510170.
- ^ Rebar EJ, Pabo CO (February 1994). "Zinc finger phage: affinity selection of fingers with new DNA-binding specificities". Science. 263 (5147): 671–3. Bibcode:1994Sci...263..671R. doi:10.1126/science.8303274. PMID 8303274.
- ^ Jamieson AC, Kim SH, Wells JA (May 1994). "In vitro selection of zinc fingers with altered DNA-binding specificity". Biochemistry. 33 (19): 5689–95. doi:10.1021/bi00185a004. PMID 8180194.
- ^ Choo Y, Klug A (November 1994). "Toward a code for the interactions of zinc fingers with DNA: selection of randomized fingers displayed on phage". Proceedings of the National Academy of Sciences of the United States of America. 91 (23): 11163–7. Bibcode:1994PNAS...9111163C. doi:10.1073/pnas.91.23.11163. PMC 45187. PMID 7972027.
- ^ Wu H, Yang WP, Barbas CF (January 1995). "Building zinc fingers by selection: toward a therapeutic application". Proceedings of the National Academy of Sciences of the United States of America. 92 (2): 344–8. Bibcode:1995PNAS...92..344W. doi:10.1073/pnas.92.2.344. PMC 42736. PMID 7831288.
- ^ Kim JS, Lee HJ, Carroll D (February 2010). "Genome editing with modularly assembled zinc-finger nucleases". Nature Methods. 7 (2): 91, author reply 91–2. doi:10.1038/nmeth0210-91a. PMC 2987589. PMID 20111032.
- ^ Joung JK, Voytas DF, Cathomen T (February 2010). "Reply to "Genome editing with modularly assembled zinc-finger nucleases"". Nat. Methods. 7 (2): 91–2. doi:10.1038/nmeth0210-91b. PMC 2987589.
- ^ Segal DJ, Dreier B, Beerli RR, Barbas CF (March 1999). "Toward controlling gene expression at will: selection and design of zinc finger domains recognizing each of the 5'-GNN-3' DNA target sequences". Proceedings of the National Academy of Sciences of the United States of America. 96 (6): 2758–63. Bibcode:1999PNAS...96.2758S. doi:10.1073/pnas.96.6.2758. PMC 15842. PMID 10077584.
- ^ Dreier B, Fuller RP, Segal DJ, Lund CV, Blancafort P, Huber A, Koksch B, Barbas CF (October 2005). "Development of zinc finger domains for recognition of the 5'-CNN-3' family DNA sequences and their use in the construction of artificial transcription factors". The Journal of Biological Chemistry. 280 (42): 35588–97. doi:10.1074/jbc.M506654200. PMID 16107335.
- ^ Dreier B, Beerli RR, Segal DJ, Flippin JD, Barbas CF (August 2001). "Development of zinc finger domains for recognition of the 5'-ANN-3' family of DNA sequences and their use in the construction of artificial transcription factors". The Journal of Biological Chemistry. 276 (31): 29466–78. doi:10.1074/jbc.M102604200. PMID 11340073.
- ^ Bae KH, Kwon YD, Shin HC, Hwang MS, Ryu EH, Park KS, Yang HY, Lee DK, Lee Y, Park J, Kwon HS, Kim HW, Yeh BI, Lee HW, Sohn SH, Yoon J, Seol W, Kim JS (March 2003). "Human zinc fingers as building blocks in the construction of artificial transcription factors". Nature Biotechnology. 21 (3): 275–80. doi:10.1038/nbt796. PMID 12592413. S2CID 29575555.
- ^ Ramirez CL, Foley JE, Wright DA, Müller-Lerch F, Rahman SH, Cornu TI, Winfrey RJ, Sander JD, Fu F, Townsend JA, Cathomen T, Voytas DF, Joung JK (May 2008). "Unexpected failure rates for modular assembly of engineered zinc fingers". Nature Methods. 5 (5): 374–5. doi:10.1038/nmeth0508-374. PMC 7880305. PMID 18446154. S2CID 30677821.
- ^ Kim HJ, Lee HJ, Kim H, Cho SW, Kim JS (July 2009). "Targeted genome editing in human cells with zinc finger nucleases constructed via modular assembly". Genome Research. 19 (7): 1279–88. doi:10.1101/gr.089417.108. PMC 2704428. PMID 19470664.
- ^ Sander JD, Dahlborg EJ, Goodwin MJ, Cade L, Zhang F, Cifuentes D, Curtin SJ, Blackburn JS, Thibodeau-Beganny S, Qi Y, Pierick CJ, Hoffman E, Maeder ML, Khayter C, Reyon D, Dobbs D, Langenau DM, Stupar RM, Giraldez AJ, Voytas DF, Peterson RT, Yeh JR, Joung JK (January 2011). "Selection-free zinc-finger-nuclease engineering by context-dependent assembly (CoDA)". Nature Methods. 8 (1): 67–9. doi:10.1038/nmeth.1542. PMC 3018472. PMID 21151135.
- ^ Greisman HA, Pabo CO (January 1997). "A general strategy for selecting high-affinity zinc finger proteins for diverse DNA target sites". Science. 275 (5300): 657–61. doi:10.1126/science.275.5300.657. PMID 9005850. S2CID 46237752.
- ^ Maeder ML, Thibodeau-Beganny S, Osiak A, Wright DA, Anthony RM, Eichtinger M, Jiang T, Foley JE, Winfrey RJ, Townsend JA, Unger-Wallace E, Sander JD, Müller-Lerch F, Fu F, Pearlberg J, Göbel C, Dassie JP, Pruett-Miller SM, Porteus MH, Sgroi DC, Iafrate AJ, Dobbs D, McCray PB, Cathomen T, Voytas DF, Joung JK (July 2008). "Rapid "open-source" engineering of customized zinc-finger nucleases for highly efficient gene modification". Molecular Cell. 31 (2): 294–301. doi:10.1016/j.molcel.2008.06.016. PMC 2535758. PMID 18657511.
- ^ Smith AT, Tucker-Samaras SD, Fairlamb AH, Sullivan WJ (December 2005). "MYST family histone acetyltransferases in the protozoan parasite Toxoplasma gondii". Eukaryotic Cell. 4 (12): 2057–65. doi:10.1128/EC.4.12.2057-2065.2005. PMC 1317489. PMID 16339723.
- ^ Akhtar A, Becker PB (February 2001). "The histone H4 acetyltransferase MOF uses a C2HC zinc finger for substrate recognition". EMBO Reports. 2 (2): 113–8. doi:10.1093/embo-reports/kve022. PMC 1083818. PMID 11258702.
- ^ Kim JG, Armstrong RC, v Agoston D, Robinsky A, Wiese C, Nagle J, Hudson LD (October 1997). "Myelin transcription factor 1 (Myt1) of the oligodendrocyte lineage, along with a closely related CCHC zinc finger, is expressed in developing neurons in the mammalian central nervous system". Journal of Neuroscience Research. 50 (2): 272–90. doi:10.1002/(SICI)1097-4547(19971015)50:2<272::AID-JNR16>3.0.CO;2-A. PMID 9373037. S2CID 37120295.
- ^ Jandrig B, Seitz S, Hinzmann B, Arnold W, Micheel B, Koelble K, Siebert R, Schwartz A, Ruecker K, Schlag PM, Scherneck S, Rosenthal A (December 2004). "ST18 is a breast cancer tumor suppressor gene at human chromosome 8q11.2". Oncogene. 23 (57): 9295–302. doi:10.1038/sj.onc.1208131. PMID 15489893.
외부 링크
- C2H2 패밀리 2016-02-15 공장 웨이백 머신에 보관TFDB: 플랜트 트랜스크립션 팩터 데이터베이스
- McDowall J. "Protein of the Month: Zinc Fingers". European Molecular Biology Laboratory - European Bioinformatics Institute (EMBL-EBI). Retrieved 2008-01-13.
- Goodsell DS. "Molecule of the Month: Zinc Fingers". Research Collaboratory for Structural Bioinformatics (RCSB) Protein Data Bank (PDB). Archived from the original on 2008-05-31. Retrieved 2008-01-13.
- 아연손가락 사이의 이중나선은
- 아연 핑거 툴 설계 및 정보 사이트
- Wayback Machine에 보관된 인간 KZNF 유전자 카탈로그 2012-03-16
- PROSITE의 아연 핑거 C2H2 타입 도메인
- SMART 데이터베이스의 아연 핑거 클래스 C2H2 엔트리
- 아연 핑거 컨소시엄
- ZiFiT - 아연 핑거 디자인 도구
- Addgene의 아연 핑거 컨소시엄 재료
- C2H2 아연 핑거 단백질의 DNA 결합 특이성 예측



