시그마 인자
Sigma factor시그마인자(γ인자 또는 특이성 인자)는 박테리아에서 [1][2]전사를 시작하기 위해 필요한 단백질이다.그것은 유전자 촉진제에 RNA 중합효소(RNAP)의 특이적 결합을 가능하게 하는 세균 전사 개시 인자이다.그것은 고고학적 전사 인자 B와 진핵생물 인자 TFIIB와 [3]상동한다.특정 유전자의 전사를 시작하는 데 사용되는 특정 시그마 인자는 유전자와 그 유전자의 전사를 시작하는 데 필요한 환경 신호에 따라 달라집니다.RNA 중합효소에 의한 프로모터의 선택은 RNA [4]중합효소와 관련된 시그마 인자에 따라 달라집니다.그것들은 또한 식물 엽록체에서 박테리아와 같은 플라스티드 부호화 중합효소(PEP)[5]의 일부로 발견됩니다.
시그마 인자는 RNA 중합효소와 함께 RNA 중합효소 홀로엔자임으로 알려져 있다.RNA 중합효소 홀로엔자임의 모든 분자는 정확히 하나의 시그마 인자 서브유닛을 포함하고 있으며, 모델 박테리아에서 대장균은 아래에 열거된 것 중 하나이다.시그마 인자의 수는 박테리아 [1][6]종에 따라 다르다.대장균에는 7개의 시그마 인자가 있습니다.시그마 인자는 그 특징적인 분자량에 의해 구별된다.예를 들어 δ는70 분자량이 70kDa인 시그마 인자이다.
RNA 중합효소 홀로엔자임 복합체의 시그마 인자는 전사의 시작을 위해 필요하지만, 일단 단계가 끝나면 복합체로부터 분리되고 RNAP는 스스로 신장을 지속한다.
특수 시그마 인자
다른 시그마 계수는 다른 환경 조건 하에서 사용됩니다.이러한 특수 시그마 인자는 환경 조건에 적합한 유전자의 프로모터를 결합시켜 유전자의 전사를 증가시킨다.
대장균의 시그마 인자:
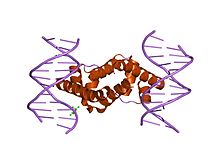
- γ70(RpoD) – βA – "하우스키핑" 시그마 인자 또는 1차 시그마 인자(그룹 1)라고도 불리는 대부분의 유전자를 성장 세포에 전사시킨다.모든 세포는 필수적인 유전자와 경로를 [1]작동시키는 "하우스키핑" 시그마 인자를 가지고 있습니다.대장균 및 기타 그램 음성 막대형 세균의 경우 하우스키핑 시그마 인자는 [1]δ이다70.§에70 의해 인식된 유전자는 모두 두 [1]부분으로 구성된 유사한 프로모터 컨센서스 서열을 포함한다.RNA 전사의 개시에 대응하는 DNA 베이스에 대해서, 컨센서스 프로모터 배열은 전사가 개시되기 전의 10 및 35 뉴클레오티드에 특징적으로 집중된다(-10 및 -35).
- γ(FecI) – 구연산철 시그마인자로 철분수송 및 신진대사를 위한 펙유전자를 조절한다19.
- γ24(RpoE) – 극도의 열응력 반응 및 세포외 단백질 시그마 인자
- γ28 (RpoF/FliA) – 편모 합성 및 화학축성 시그마 인자
- δ32 (RpoH) – 열충격 시그마계수로 세균이 열에 노출되면 켜진다.높은 발현으로 인해 인자는 중합효소 코어 효소에 높은 확률로 결합합니다.그렇게 함으로써, 다른 열충격 단백질이 발현되어 세포가 더 높은 온도에서 살아남을 수 있게 한다.γ의32 활성화로 발현되는 효소로는 샤페론, 단백질분해효소, DNA수복효소 등이 있다.
- § (RpoS)– 기아38/정지상 시그마 계수
- γ54 (RpoN) – 질소제한 시그마 계수
시그마 인자의 기능을 저해하는 안티시그마 인자와 시그마 인자의 기능을 회복시키는 안티시그마 인자도 있다.
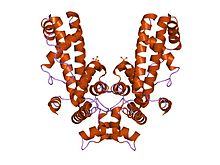
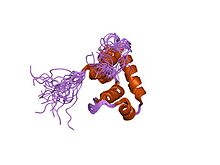
구조.
시퀀스 유사성에 의해 대부분의 시그마 인자는 δ70 유사합니다(InterPro: IPR000943).일반적으로 보존되는 4개의 주요 영역(도메인)이 있습니다.
N-terminus ----------------------------------- C-terminus 1.1 2 3 4
지역들은 더욱 세분화된다.예를 들어 영역 2에는 1.2와 2.1~2.4가 포함됩니다.
도메인 1.1은 "프라이머리 시그마 계수"(RpoD, E.coli의 RpoS, "그룹 1")에만 있습니다.시그마 인자가 RNA 중합효소와 [7]복합될 때만 프로모터와 결합하도록 하는 데 관여합니다.도메인 2-4는 각각 특정 프로모터 요소 및 RNAP와 상호 작용합니다.영역 2.4는 프로모터 -10 요소("Pribnow box")를 인식하고 결합합니다.영역 4.2는 프로모터 -35 [7]요소를 인식하고 결합한다.
family70 패밀리의 모든 시그마 팩터가 모든 도메인을 포함하는 것은 아닙니다.RpoS를 포함한 그룹2는 그룹1과 매우 비슷하지만 도메인1이 없습니다.그룹3에는 도메인1이 없고, 그룹4에는28 11.1과 group3이 모두 없습니다.RpoE가 [7]멤버입니다.
기타 알려진 시그마 인자는 γ54/RpoN(InterPro: IPR000394) 유형입니다.그것들은 기능적인 시그마 인자들이지만, 그들은 유의하게 다른 1차 아미노산 [8]서열을 가지고 있다.
|
|
|
|
|
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
전사 연장 시 유지
핵심 RNA 중합효소(2α, 베타(β), 베타(β), 베타(β'), 오메가(β) 서브유닛 1개로 구성)는 시그마 인자와 결합하여 RNA 중합효소 홀로엔자임이라고 불리는 복합체를 형성합니다.이전에는 RNA 중합효소 홀로엔자임이 전사를 시작하는 반면, 핵심 RNA 중합효소만이 RNA를 합성하는 것으로 여겨졌다.따라서 시그마 인자는 전사 시작에서 전사 신장으로의 전환 시 분리되어야 한다는 것이 통설이었다(이 전환을 "촉진제 탈출"이라고 한다).이러한 관점은 시작 및 신장 시 정지된 RNA 중합효소 정제 복합체 분석에 기초하였다.마지막으로, RNA 중합효소 복합체의 구조 모델은 성장하는 RNA 생성물이 최대 15개의 뉴클레오티드보다 길어짐에 따라 RNA와 시그마 도메인 사이에 입체적 충돌이 있기 때문에 시그마는 홀로엔자임에서 "밀어내야" 한다고 예측했습니다.그러나 γ는70 초기 신장[9] 및 때로는 신장 [10]내내 코어 RNA 중합효소와의 복합체 결합 상태를 유지할 수 있다.실제로 프로모터-근접 일시정지 현상은 시그마가 초기 신장 중에 역할을 한다는 것을 나타낸다.모든 연구는 프로모터 탈출이 시작 시 매우 긴 시그마 코어 상호작용의 수명을 감소시킨다는 가정과 일치한다(일반 생화학적 실험에서 측정하기에는 너무 긴 시간).
시그마 사이클
시그마 인자는 전사를 시작하면 필수적으로 핵심 효소를 떠나 다른 핵심 효소와 연결되고 다른 부위에서 전사를 시작할 수 있다고 오랫동안 생각되어 왔다.따라서 시그마 인자는 한 코어에서 다른 코어로 순환합니다.그러나 형광 공명 에너지 전달은 시그마 인자가 의무적으로 [9]코어를 떠나지 않는다는 것을 보여주기 위해 사용되었습니다.대신 시작 및 신장 중에 코어와의 결합을 변화시킨다.따라서 시그마 인자는 시작 시 강한 결합 상태와 신장 시 약한 결합 상태를 순환한다.
시그마 인자 경쟁
박테리아 세포(예: 대장균)의 RNAP 수는 시그마 인자 수보다 적은 것으로 나타났다.결과적으로 특정 시그마 인자가 과도하게 발현되면 프로모터가 시그마 인자를 선호하는 유전자의 발현 수준이 높아질 뿐만 아니라 프로모터가 다른 시그마 [11][12][13][14]인자를 선호하는 유전자가 존재할 확률도 감소한다.
한편, 전사 개시에는, 클로즈드형성과 오픈 복합형성의 2개의 주요한 레이트 제한 스텝이 있습니다.그러나 첫 번째 단계의 역학만 시그마 인자의 농도에 따라 달라집니다.흥미롭게도 개방 복합체 형성에 비해 닫힌 복합체 형성이 가장 빠를수록 시그마 인자의 농도 변화에 대한 반응성이 떨어진다(이 현상의 모델 및 경험적 데이터에 대해서는 참조).
이중 시그마 인자 선호 유전자
대장균의 대부분의 유전자는 하나의 시그마 인자(예를 들어 시그마 70)를 가진 RNAP에 의해 인식될 수 있지만, 소수의 유전자(약 5%)는 소위 "이중 시그마 인자 선호도"[15]라고 불리는 즉, RegulonDB에 [16]보고된 바와 같이 두 개의 다른 시그마 인자에 반응할 수 있다.가장 일반적인 것은 시그마 70과 시그마 38에 반응할 수 있는 촉진제입니다.이들 유전자의 역학 연구는 세포가 정지 성장에 들어갔을 때 38파운드를 선호하는 유전자와 거의 같은 수준으로 유도된다는 것을 보여줍니다.이 유도 수준은 프로모터 [15]시퀀스에서 예측 가능한 것으로 나타났다.그림에는 동역학 모델이 나와 있습니다.미래에는 이러한 프로모터가 대장균의 합성 유전자 구조에 유용한 도구가 될 수 있다.

「 」를 참조해 주세요.
레퍼런스
- ^ a b c d e Gruber TM, Gross CA (2003). "Multiple sigma subunits and the partitioning of bacterial transcription space". Annual Review of Microbiology. 57: 441–66. doi:10.1146/annurev.micro.57.030502.090913. PMID 14527287.
- ^ Kang JG, Hahn MY, Ishihama A, Roe JH (July 1997). "Identification of sigma factors for growth phase-related promoter selectivity of RNA polymerases from Streptomyces coelicolor A3(2)". Nucleic Acids Research. 25 (13): 2566–73. doi:10.1093/nar/25.13.2566. PMC 146787. PMID 9185565.
- ^ Burton SP, Burton ZF (6 November 2014). "The σ enigma: bacterial σ factors, archaeal TFB and eukaryotic TFIIB are homologs". Transcription. 5 (4): e967599. doi:10.4161/21541264.2014.967599. PMC 4581349. PMID 25483602.
- ^ Ho TD, Ellermeier CD (April 2012). "Extra cytoplasmic function σ factor activation". Current Opinion in Microbiology. 15 (2): 182–8. doi:10.1016/j.mib.2012.01.001. PMC 3320685. PMID 22381678.
- ^ Schweer J, Türkeri H, Kolpack A, Link G (December 2010). "Role and regulation of plastid sigma factors and their functional interactors during chloroplast transcription - recent lessons from Arabidopsis thaliana". European Journal of Cell Biology. 89 (12): 940–6. doi:10.1016/j.ejcb.2010.06.016. PMID 20701995.
- ^ Sharma UK, Chatterji D (September 2010). "Transcriptional switching in Escherichia coli during stress and starvation by modulation of sigma activity". FEMS Microbiology Reviews. 34 (5): 646–57. doi:10.1111/j.1574-6976.2010.00223.x. PMID 20491934.
- ^ a b c Paget MS (June 2015). "Bacterial Sigma Factors and Anti-Sigma Factors: Structure, Function and Distribution". Biomolecules. 5 (3): 1245–65. doi:10.3390/biom5031245. PMC 4598750. PMID 26131973.
- ^ Merrick MJ (December 1993). "In a class of its own--the RNA polymerase sigma factor sigma 54 (sigma N)". Molecular Microbiology. 10 (5): 903–9. doi:10.1111/j.1365-2958.1993.tb00961.x. PMID 7934866. S2CID 84789281.
- ^ a b Kapanidis AN, Margeat E, Laurence TA, Doose S, Ho SO, Mukhopadhyay J, Kortkhonjia E, Mekler V, Ebright RH, Weiss S (November 2005). "Retention of transcription initiation factor sigma70 in transcription elongation: single-molecule analysis". Molecular Cell. 20 (3): 347–56. doi:10.1016/j.molcel.2005.10.012. PMID 16285917.
- ^ Harden TT, Wells CD, Friedman LJ, Landick R, Hochschild A, Kondev J, Gelles J (January 2016). "Bacterial RNA polymerase can retain σ70 throughout transcription". Proc Natl Acad Sci U S A. 113 (3): 602–7. Bibcode:2016PNAS..113..602H. doi:10.1073/pnas.1513899113. PMC 4725480. PMID 26733675.
- ^ Jishage, M; Ishihama, A (December 1995). "Regulation of RNA polymerase sigma subunit synthesis in Escherichia coli: intracellular levels of sigma 70 and sigma 38". Journal of Bacteriology. 177 (23): 6832–6835. doi:10.1128/jb.177.23.6832-6835.1995. ISSN 0021-9193. PMC 177550. PMID 7592475.
- ^ Jishage, M; Iwata, A; Ueda, S; Ishihama, A (September 1996). "Regulation of RNA polymerase sigma subunit synthesis in Escherichia coli: intracellular levels of four species of sigma subunit under various growth conditions". Journal of Bacteriology. 178 (18): 5447–5451. doi:10.1128/jb.178.18.5447-5451.1996. ISSN 0021-9193. PMC 178365. PMID 8808934.
- ^ Grigorova, Irina L.; Phleger, Naum J.; Mutalik, Vivek K.; Gross, Carol A. (2006-04-04). "Insights into transcriptional regulation and σ competition from an equilibrium model of RNA polymerase binding to DNA". Proceedings of the National Academy of Sciences. 103 (14): 5332–5337. Bibcode:2006PNAS..103.5332G. doi:10.1073/pnas.0600828103. ISSN 0027-8424. PMC 1459355. PMID 16567622.
- ^ a b Kandavalli, Vinodh K.; Tran, Huy; Ribeiro, Andre S. (October 2016). "Effects of σ factor competition are promoter initiation kinetics dependent". Biochimica et Biophysica Acta (BBA) - Gene Regulatory Mechanisms. 1859 (10): 1281–1288. doi:10.1016/j.bbagrm.2016.07.011. PMID 27452766.
- ^ a b Baptista, Ines S.C.; Kandavalli, Vinodh; Chauhan, Vatsala; Bahrudeen, Mohamed N.M.; Almeida, Bilena L.B.; Palma, Cristina S.D.; Dash, Suchintak; Ribeiro, Andre S. (April 2022). "Sequence-dependent model of genes with dual σ factor preference". Biochimica et Biophysica Acta (BBA) - Gene Regulatory Mechanisms. 1865 (3): 194812. doi:10.1016/j.bbagrm.2022.194812. PMID 35338024. S2CID 247636833.
- ^ Santos-Zavaleta, Alberto; Salgado, Heladia; Gama-Castro, Socorro; Sánchez-Pérez, Mishael; Gómez-Romero, Laura; Ledezma-Tejeida, Daniela; García-Sotelo, Jair Santiago; Alquicira-Hernández, Kevin; Muñiz-Rascado, Luis José; Peña-Loredo, Pablo; Ishida-Gutiérrez, Cecilia (2019-01-08). "RegulonDB v 10.5: tackling challenges to unify classic and high throughput knowledge of gene regulation in E. coli K-12". Nucleic Acids Research. 47 (D1): D212–D220. doi:10.1093/nar/gky1077. ISSN 0305-1048. PMC 6324031. PMID 30395280.