RNA
RNA
| 다음에 대한 시리즈 일부 |
| 유전학 |
|---|
 |
리보핵산(RNA)은 유전자의 부호화, 해독, 조절, 발현에서 다양한 생물학적 역할에 필수적인 고분자 분자다.RNA와 디옥시리보핵산(DNA)은 핵산이다.지질, 단백질, 탄수화물과 함께 핵산은 알려진 모든 형태의 생명체에 필수적인 4대 고분자 중 하나를 구성한다.DNA와 마찬가지로 RNA도 뉴클레오티드의 사슬로서 조립되지만, DNA와 달리 RNA는 쌍쌍의 이중 가닥이 아니라 스스로 접힌 하나의 가닥으로 자연에서 발견된다.세포 유기체는 메신저 RNA(mRNA)를 사용하여 유전 정보(구아닌, 우라실, 아데닌, 시토신의 질소 염기 사용, 특정 단백질의 합성을 지시하는 문자 G, U, A, C)를 전달한다.많은 바이러스들이 RNA 게놈을 사용하여 그들의 유전 정보를 암호화한다.
일부 RNA 분자는 생물학적 반응을 촉진하거나 유전자 발현을 조절하거나 세포 신호에 대한 반응을 감지하고 전달함으로써 세포 내에서 능동적인 역할을 한다.이러한 활성 과정 중 하나는 단백질 합성이며, RNA 분자가 리보솜에 단백질의 합성을 지시하는 보편적 기능이다.이 과정은 전이 RNA(transfer RNA) 분자를 사용하여 아미노산을 리보솜에 전달하는데, 리보솜 RNA(rRNA)는 아미노산을 서로 연결하여 코딩 단백질을 형성한다.
DNA와의 비교
DNA와 마찬가지로 mRNA, tRNA, rRNA, snRNA, 기타 비코딩 RNA 등 대부분의 생물학적으로 활성 RNA는 RNA의 일부를 접고[5] 자체와 짝을 지어 이중 나선형을 형성할 수 있는 자기 보완 시퀀스를 포함하고 있다.이들 RNA를 분석한 결과 구조성이 높은 것으로 밝혀졌다.DNA와 달리, 그들의 구조는 긴 이중 나선형이 아니라 단백질과 유사한 구조로 함께 포장된 짧은 나선형의 집합체로 이루어져 있다.
이런 방식으로 RNA는 화학적 촉매(효소처럼)를 얻을 수 있다.[6]예를 들어, 펩타이드 결합 형성을 촉진하는 RNA 단백질 복합체인 리보솜의 구조 결정은 리보솜의 활성 부위가 전적으로 RNA로 구성되어 있음을 입증했다.[7]
구조
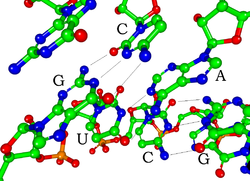
RNA의 각 뉴클레오티드는 리보스 당분을 함유하고 있으며, 탄소는 1'에서 5'까지 번호가 매겨져 있다.기지는 일반적으로 아데닌(A), 시토신(C), 구아닌(G) 또는 우라실(U) 위치에 부착된다.아데닌과 구아닌은 청진이고, 시토신과 우라실은 피리미딘이다.인산염 그룹은 한 리보스의 3' 위치와 다음 리보스의 5' 위치에 부착된다.인산염 그룹은 각각 음전하를 가지며, RNA를 전하 분자(폴리야니온)로 만든다.염기들은 시토신과 구아닌사이에, 아데닌과 우라실사이에, 구아닌과 우라실사이에 수소 결합을 형성한다.[8]그러나 아데닌 염기군이 서로 불룩하게 결합하거나 [9]구아닌-아데닌 염기쌍을 가진 GNRA 4중첩과 같은 다른 상호작용이 가능하다.[8]
DNA와 구별되는 RNA의 중요한 구조적 구성요소는 리보스 설탕의 2' 위치에 히드록실 그룹이 존재하는 것이다.비록 단일 가닥 디뉴클레오티드 맥락에서 RNA는 DNA에서 가장 일반적으로 관찰되는 B 형태를 거의 채택할 수 없지만,[10] 이 기능 그룹의 존재는 나선형이 A-폼 형상을 대부분 차지하게 한다.[11]A형 기하학은 매우 깊고 좁은 큰 홈과 얕고 넓은 작은 홈을 이룬다.[12]2'-히드록실 그룹의 존재에 따른 두 번째 결과는 RNA 분자의 순응적으로 유연한 영역(이중 나선 형성에 관여하지 않음)에서 인접 인광체 결합을 화학적으로 공격하여 등뼈를 절단할 수 있다는 것이다.[13]
RNA는 4개의 염기(아데닌, 사이토신, 구아닌, 우라실)[14]만 가지고 전사하지만, 이러한 염기와 부착된 당은 RNA가 성숙함에 따라 수많은 방법으로 변형될 수 있다.요람과 리보스의 연계가 C-N 결합에서 C-C 결합으로 바뀌는 유사우리딘(TR)과 리보시미딘(T)은 여러 곳에서 발견된다(tRNA의 THRC 루프에 있는 것이 가장 눈에 띈다).[15]또 다른 주목할 만한 변형 베이스는 탈염 아데닌 베이스인 저산산산틴으로, 핵산은 이노신(I)이라고 불린다.이노신(inosine)은 유전법칙의 흔들리는 가설에서 핵심적인 역할을 한다.[16]
다른 자연적으로 발생하는 변형 뉴클레오시드가 100개 이상 있다.[17]수정의 가장 큰 구조적 다양성은 tRNA에서 찾을 수 있는 반면,[18] 2'-O-메틸리보스를 가진 유사수리딘과 뉴클레오시드가 rRNA에서 가장 흔하다.[19]RNA에서 이러한 많은 수정의 구체적인 역할은 완전히 이해되지 않는다.단, 리보솜 RNA에서는 펩티딜전달효소 중심, 서브유닛 인터페이스 등 기능성이 높은 부위에서 많은 후전송변형이 발생하여 정상기능에 중요하다는 것을 시사하는 점이 눈에 띈다.[21]
단백질과 마찬가지로 단일 가닥 RNA 분자의 기능적 형태는 특정한 3차 구조를 필요로 하는 경우가 많다.이 구조의 비계는 분자 내의 수소 결합인 이차 구조 요소에 의해 제공된다.이것은 헤어핀 루프, 돌출부 및 내부 루프와 같은 2차 구조의 몇 가지 인식 가능한 "도메인"으로 이어진다.[22]설계, 즉 주어진 이차 구조에 대한 RNA를 생성하기 위해서는 2, 3개의 염기로는 충분하지 않지만, 4개의 염기로는 충분하다.[23]이것이 자연이 4개의 기본 알파벳을 "초센"으로 하는 이유일 것이다: 4개 미만의 알파벳은 모든 구조를 만들 수 없고, 4개 이상의 기본은 필요하지 않다.RNA가 충전되기 때문에 많은 2차, 3차 구조를 안정화시키기 위해서는 Mg와2+ 같은 금속 이온이 필요하다.[24]
RNA의 자연적으로 발생하는 항산화 물질은 D-리보뉴클레오티드로 구성된 D-RNA이다.모든 chirality 센터는 D-리보스에 위치한다.L-리보스 또는 오히려 L-리보뉴클레오티드를 사용하여 L-RNA를 합성할 수 있다.L-RNA는 RNAE에 의한 분해에 대해 훨씬 안정적이다.[25]
단백질과 같은 다른 구조화된 생물 고분자처럼 접힌 RNA 분자의 위상을 정의할 수 있다.이것은 종종 회로 위상이라고 불리는 접힌 RNA 내의 체인 내부 접점의 배열을 기반으로 한다.
합성
RNA의 합성은 보통 효소에 의해 촉매된다.RNA 중합효소—DNA를 템플릿으로 사용하여 전사라고 알려진 프로세스.전사의 시작은 DNA에서 촉진자 수열에 효소가 결합하는 것으로 시작된다(일반적으로 유전자의 "상류"가 발견됨).DNA 이중나선은 효소의 헬리코아제 활성에 의해 분해된다.그 후 효소는 3~5' 방향으로 템플릿 가닥을 따라 진행되며, 5'~3' 방향으로 연장이 일어나는 보완 RNA 분자를 합성한다.DNA 서열은 또한 RNA 합성의 종단이 발생할 장소를 지시한다.[26]
일차대신 RNA는 전사 후 효소에 의해 변형되는 경우가 많다.예를 들어, 진핵 전 mRNA에 폴리(A) 꼬리와 5'의 캡을 추가하고 인트론은 스플라이소솜에 의해 제거된다.
RNA의 새로운 가닥 RNA의 합성을 위한 그들의 템플릿으로 RNA를 사용하는 RNA 의존적 RNA 중합체도 다수 존재한다.예를 들어, 많은 RNA 바이러스(예: 소아마비 바이러스)는 그들의 유전 물질을 복제하기 위해 이러한 종류의 효소를 사용한다.[27]또한, RNA 의존 RNA 중합효소는 많은 유기체에서 RNA 간섭 경로의 일부분이다.[28]
RNA의 종류
개요

MRNA(Messenger RNA)는 DNA에서 세포 내 단백질 합성(번역) 부위인 리보솜으로 정보를 전달하는 RNA이다.mRNA는 DNA의 복제품이다.mRNA의 코딩 순서는 생성되는 단백질에서 아미노산 순서를 결정한다.[29]그러나 많은 RNA는 단백질에 대해 코딩하지 않는다(전사 출력의 약 97%는 진핵생물에[30][31][32][33] 있어서 비단백질 코딩이다).
이러한 소위 비코딩 RNA("ncRNA")는 그들 자신의 유전자(RNA 유전자)에 의해 인코딩될 수 있지만 mRNA 인트론에서도 파생될 수 있다.[34]비코딩 RNA의 가장 두드러진 예는 전이 RNA(transfer RNA)와 리보솜 RNA(rRNA)로, 둘 다 번역 과정에 관여한다.[4]유전자 조절, RNA 처리 및 기타 역할과 관련된 비코딩 RNA도 있다.특정 RNA는 다른 RNA 분자를 자르고 묶는 것과 [35]같은 화학반응을 촉진할 수 있고, 리보솜에서 펩타이드 결합 형성의 촉매작용이 가능하다; 이것들은 [7]리보솜이라고 알려져 있다.
길이
RNA 체인의 길이에 따라 RNA는 작은 RNA와 긴 RNA를 포함한다.[36] 보통 작은 RNA는 길이가 200nt보다 짧고 긴 RNA는 200nt보다 길다.[37]롱 RNAs, 또한, 주로non-coding RNA(lncRNA)과 mRNA를 포함한다. 중소 RNAs 주로 5.8S 리보솜 RNA(rRNA), 5SrRNA을 전송하며, RNA(tRNA), microRNA(miRNA), 작은 간섭 RNA(siRNA), 작은 인의 RNA(snoRNAs), Piwi-interacting RNA(piRNA), tRNA-derived 작은 RNA(tsRNA)[38]고 작은rDNA-derived RNA(srRNA을 포함한다 큰 RNAs 요구했다.).[39]삽입이 있는 할로코쿠스(아카이아)의 성분의 5S rRNA의 경우와 같이 일정한 예외가 있어 크기가 커진다.[40][41][42]
번역하면
메신저 RNA(mRNA)는 세포 내 단백질 합성 공장인 리보솜에 단백질 서열에 대한 정보를 전달한다.세 개의 뉴클레오티드(코돈)가 한 개의 아미노산에 해당하도록 코딩된다.진핵 세포에서는 일단 전구 mRNA(pre-mRNA)가 DNA에서 옮겨지면 성숙한 mRNA로 가공된다.이렇게 하면 사전 mRNA의 비코딩 섹션인 인트론이 제거된다.그런 다음 mRNA는 핵에서 세포질로 수출되는데, 여기서 리보솜에 결합되어 tRNA의 도움을 받아 그에 상응하는 단백질 형태로 번역된다.핵, 세포질 구획이 없는 원핵세포에서 mRNA는 리보솜이 DNA에서 옮겨지는 동안 결합할 수 있다.일정 시간이 지나면 리보핵산의 도움으로 메시지는 성분 뉴클레오티드로 분해된다.[29]
전이 RNA(transfer RNA, tRNA)는 약 80개의 뉴클레오티드로 이루어진 작은 RNA 체인으로, 번역 중에 단백질 합성 리보솜 부위의 성장하는 폴리펩타이드 체인으로 특정 아미노산을 전달한다.수소 본딩을 통해 메신저 RNA 체인의 특정 시퀀스에 바인딩되는 아미노산 부착 부위와 코돈 인식용 안티코돈 부위가 있다.[34]
리보솜 RNA(rRNA)는 리보솜의 촉매 성분이다.rRNA는 번역을 진행하는 리보솜의 구성요소다.진핵 리보솜은 18S, 5.8S, 28S, 5S rRNA의 네 가지 다른 rRNA 분자를 포함한다.rRNA 분자 중 3개는 뉴클레오루스에서 합성되며, 1개는 다른 곳에서 합성된다.세포질에서는 리보솜 RNA와 단백질이 결합하여 리보솜이라고 불리는 뉴클레오프로테인을 형성한다.리보솜은 mRNA를 결합하고 단백질 합성을 수행한다.몇 개의 리보솜이 언제든지 단일 mRNA에 부착될 수 있다.[29]전형적인 진핵 세포에서 발견되는 거의 모든 RNA는 rRNA이다.
전달-메신저 RNA는 많은 박테리아와 플라스티드에서 발견자 RNA)는 많은 박테리아와 플라스티드에서 발견된다.mRNA로 인코딩된 단백질에 분해 정지 코돈이 부족한 단백질을 태그하고 리보솜이 멈추는 것을 막는다.[43]
규제 RNA
가장 초기에 알려진 유전자 발현 조절기는 억제제와 활성제로 알려진 단백질로, 조절할 유전자 근처에 있는 강한 물질 영역 내에 특정한 짧은 결합 부위가 있는 조절기였다.[44]이후 연구에서는 RNA도 유전자를 조절한다는 것이 밝혀졌다.진핵생물에는 유전자 발현을 조절하는 여러 종류의 RNA 의존적 과정이 있는데, 이를테면 RNAi는 전치 후 유전자를 억제하고, 긴 비코딩 RNA는 후생유전적으로 염색질 블록을 폐쇄하며, 진한제 RNA는 유전자 발현을 증가시킨다.[45]박테리아와 고고학도 박테리아 소형 RNA와 CRISPR과 같은 규제 RNA 시스템을 사용하는 것으로 나타났다.[46]파이어와 멜로는 mRNA로 베이스 페어링할 수 있는 특정한 짧은 RNA 분자인 miRNA(microRNAs)를 발견한 공로로 2006년 노벨 생리의학상을 받았다.[47]
miRNA에 의한 RNA 간섭
많은 유전자의 변환 후 발현 수준은 RNA 간섭에 의해 조절될 수 있는데, RNA 간섭에서는 mRNA 영역과 특정한 짧은 RNA 분자가 짝을 지어 그들을 분해 대상으로 삼는다.[48]이 항균 기반 공정은 RNA가 표적 mRNA의 영역과 베이스 페어링할 수 있도록 RNA를 먼저 처리하는 단계를 포함한다.일단 염기쌍이 일어나면, 다른 단백질들은 mRNA가 핵에 의해 파괴되도록 지시한다.[45]
긴 비코딩 RNA
다음으로 규제와 연계된 것은 Xist와 X염색체 불활성화와 관련된 다른 긴 비코딩 RNA들이었다.처음에는 신비로운 그들의 역할은 지니 T에 의해 보여졌다. 이씨 등은 폴리콤 콤플렉스 채용을 통해 염색질 블록을 음소거하여 메신저 RNA가 이들로부터 전자가 되지 않도록 해야 한다.[49]현재 코딩 잠재력이 없는 것으로 보이는 200개 이상의 염기쌍의 RNA로 정의되고 있는 추가적인 lncRNA는 줄기세포 플뤼리포텐도 및 세포분열 조절과 관련된 것이 발견되었다.[50][50]
엔한서 RNA
세 번째 주요 규제 RNA 집단은 엔한서 RNA라고 불린다.[50]그것들이 다양한 길이의 RNA의 고유한 범주인지, 아니면 lncRNA의 뚜렷한 부분집합을 구성하는지는 현재로서는 명확하지 않다.어떤 경우든, 그것들은 그들이 조절하는 유전자 근처에 있는 DNA에 있는 규제 사이트로 알려진 엔핸서로부터 옮겨진다.[50][51]그들은 자신이 전사되는 진약제의 통제 하에 유전자의 전사를 상향 조절한다.[50][52]
원핵생물의 규제 RNA
처음에, 규제 RNA는 진핵 현상으로 생각되었는데, 왜 그렇게 높은 유기체에서 왜 그렇게 많은 전사가 예측되었던 것보다 더 많이 관찰되었는지에 대한 설명의 한 부분이다.그러나 연구원들이 박테리아에서 가능한 RNA 조절기를 찾기 시작하자마자, 그들은 작은 RNA(small RNA)라고 불리는 그 곳에서도 나타났다.[53][46]현재 유전자 RNA 규제의 시스템 유비쿼터스 성격이 RNA 월드 이론의 지원으로서 논의되고 있다.[45][54]박테리아 소형 RNA는 일반적으로 안정성에 영향을 미치거나 시스 바인딩 능력에 영향을 미침으로써 mRNA와 항이센스 페어링을 통해 그 변환을 하향 조절하는 작용을 한다.[45]리보스 와인도 발견되었다.그것들은 알로스테리릭하게 작용하는 시스 작용 규제 RNA 시퀀스다.그들은 대사물을 묶을 때 모양을 바꿔서 유전자의 발현을 조절하기 위해 염색체를 묶는 능력을 얻거나 잃는다.[55][56]
고세아는 또한 규제 RNA 시스템을 가지고 있다.[57]최근 현장에서 DNA를 편집하는 데 사용되고 있는 크리스퍼 시스템은 바이러스 침입자에 대한 보호를 제공하기 위해 고대의 규제 RNA와 박테리아를 통해 작용한다.[45][58]
RNA 처리 중
많은 RNA들이 다른 RNA들을 수정하는데 관여한다.인트론은 여러 개의 작은 핵 RNA(snRNA)를 포함하고 있는 스플라이소솜에 의해 사전 mRNA에서 분할되거나,[4] 인트론은 스스로 분열하는 리보이가 될 수 있다.[59]RNA는 뉴클레오티드 뉴클레오티드에는 A, C, G, 미국 eukaryotes안에서 제외하기 위하여 수정됨으로써 변경할 수 있, RNA뉴클레오티드의 수정을 작은 핵소체 RNAs(snoRNA, 60–300 nt)[34]고cajal 인 몸에서 발견된 대개의 경우에 효소와 함께 snoRNAs원은 RNA에 한 지점 tha에 basepairing에 의해 그들을 지도하고 있다.R 있어NA. 이 효소들은 뉴클레오티드 수정을 수행한다. rRNA와 tRNA는 광범위하게 수정되지만 snRNA와 mRNA도 염기변조의 대상이 될 수 있다.[60][61]RNA는 메틸화 될 수도 있다.[62][63]
RNA 게놈
DNA처럼, RNA는 유전 정보를 운반할 수 있다.RNA 바이러스는 많은 단백질을 암호화하는 RNA로 구성된 게놈을 가지고 있다.바이러스 게놈은 일부 단백질에 의해 복제되는 반면, 다른 단백질은 바이러스 입자가 새로운 숙주 세포로 이동하면서 게놈을 보호한다.비로이드는 또 다른 병원체 집단이지만, 그것들은 RNA로만 구성되어 있고, 단백질을 인코딩하지 않으며, 숙주 식물 세포의 중합효소에 의해 복제된다.[64]
역전사
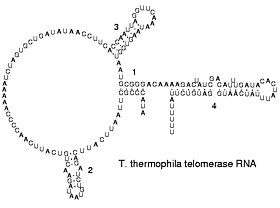
역전사 바이러스는 그들의 RNA로부터 DNA 복사본을 역전사함으로써 그들의 게놈을 복제한다; 이 DNA 복사본들은 새로운 RNA로 옮겨진다. 역전사 또한 서로로부터 DNA와 RNA를 복제함으로써 퍼지고,[65] 텔로머레이즈는 진핵 염색체의 끝을 형성하는 데 사용되는 RNA를 포함한다.[66]
이중 가닥 RNA
이중 가닥 RNA(dsRNA)는 두 개의 보완 가닥을 가진 RNA로, 모든 세포에서 발견되는 DNA와 유사하지만 우라실(yuracil)에 의한 티민 대체와 하나의 산소 원자의 첨가. dsRNA는 일부 바이러스(이중 가닥 RNA 바이러스)의 유전 물질을 형성한다.바이러스성 RNA나 siRNA와 같은 이중 가닥 RNA는 진핵생물에서 RNA 간섭을 유발할 수 있으며 척추동물에서 인터페론 반응도 유발할 수 있다.[67][68][69][70]
원형 RNA
1970년대 후반에, 동식물 왕국 전체에 걸쳐 표현된 RNA의 원형 형태, 즉 공동 폐쇄된 단일 좌초된 것이 보였다(circRNA 참조).[71]circRNA는 스플라이소솜이 업스트림 3의 수용자와 다운스트림 5의 기증자 스플라이스 부지에 결합하는 "백-스플라이스" 반응을 통해 발생하는 것으로 생각된다.비록 몇 가지 사례에 대해 마이크로RNA 스폰지 활동이 증명되었지만, 지금까지 circRNA의 기능은 대체로 알려져 있지 않다.
RNA생물학의 주요 발견
RNA에 대한 연구는 많은 중요한 생물학적 발견과 수많은 노벨상으로 이어졌다.핵산은 1868년 프리드리히 미에셔에 의해 발견되었는데, 그는 이 물질을 핵에서 발견되었기 때문에 '핵인'이라고 불렀다.[72]핵이 없는 원핵세포에도 핵산이 들어 있다는 사실이 뒤늦게 밝혀졌다.단백질 합성에 있어서 RNA의 역할은 이미 1939년에 의심되었다.[73]세베로 오초아는 연구실에서 RNA를 합성할 수 있는 효소를 발견해 1959년 노벨 의학상(아서 코른베르크와 공동상)을 수상했다.[74]그러나 오초아(폴리뉴클레오티드인산화효소)가 발견한 효소는 나중에 RNA 합성이 아닌 RNA 분해의 원인이 되는 것으로 밝혀졌다.1956년 알렉스 리치와 데이비드 데이비스는 분리된 두 가닥의 RNA를 혼합하여 X선 결정학에 의해 구조를 결정할 수 있는 RNA의 첫 번째 결정체를 형성했다.[75]
효모 tRNA의 77 뉴클레오티드의 순서는 로버트 W에 의해 발견되었다. 홀리는 1965년 홀리를 1968년 노벨 의학상(하 고빈드 코라나, 마샬 니렌버그와 공동 수상)으로 수상하였다.[76]
1970년대 초 복고현상과 역분해효소가 발견돼 효소가 RNA를 DNA로 복제할 수 있다는 사실을 처음으로 보여줬다(유전자 정보의 일반적인 전달 경로와는 반대).이 작품으로 데이비드 볼티모어, 레나토 둘베코, 하워드 테민 등이 1975년 노벨상을 받았다.1976년, 월터 피어스와 그의 팀은 RNA 바이러스 게놈의 완전한 뉴클레오티드 염기서열인 박테리오파지 MS2를 결정했다.[77]
1977년 포유류 바이러스와 세포 유전자 모두에서 인트론과 RNA 스플라이싱이 발견되어 필립 샤프와 리처드 로버츠에게 1993년 노벨상을 안겨주었다.촉매 RNA 분자(리보이즈)는 1980년대 초에 발견되어 토마스 체흐와 시드니 알트먼에게 1989년 노벨상을 수여하게 되었다.1990년 페투니아에서 소개된 유전자가 식물 자체의 유사한 유전자를 잠재울 수 있다는 것이 발견되었는데, 현재는 RNA 간섭의 결과로 알려져 있다.[78][79]
이와 거의 동시에 현재 마이크로RNA라고 불리는 22nt의 긴 RNA가 C. 선충의 발달에 역할을 하는 것으로 밝혀졌다.[80]RNA 간섭에 관한 연구는 2006년 앤드류 파이어와 크레이그 멜로에게 노벨상을 수여했고, 같은 해 로저 코른버그에게 RNA를 전사한 연구로 또 다른 노벨상을 받았다.유전자 규제 RNA의 발견은 유전자를 침묵시키기 위해 siRNA와 같은 RNA로 만들어진 약물을 개발하려는 시도로 이어졌다.[81]2009년 RNA 연구로 수여된 노벨상에 추가하여, 리보솜의 원자 구조를 벤키 라마크리쉬난, 토마스 A에게 해명하여 수여되었다. 스티츠, 그리고 에이다 요나스.
생물학적 이전의 화학 및 자연발생과의 관련성
1968년 칼 워즈는 RNA가 촉매일지도 모른다는 가설을 세우고, 가장 초기 형태의 생명체(자기복제 분자)가 유전자 정보를 전달하고 생화학적 반응인 RNA 세계를 촉매하기 위해 RNA에 의존할 수 있었을 것이라고 제안했다.[82][83]
2015년 3월 요석, 시토신, 티민 등 복합 DNA와 RNA 뉴클레오티드가 우주 조건에서 운석에서 흔히 발견되는 유기 화합물인 피리미딘과 같은 스타터 화학물질을 이용해 실험실에서 형성된 것으로 알려졌다.피리미딘은 다순환 방향족 탄화수소(PAHs)와 마찬가지로 우주에서 가장 탄소가 풍부한 화합물 중 하나로 적색 거성 또는 성간 먼지와 가스 구름에서 형성되었을 수 있다.[84]
참고 항목
참조
- ^ "RNA: The Versatile Molecule". University of Utah. 2015.
- ^ "Nucleotides and Nucleic Acids" (PDF). University of California, Los Angeles. Archived from the original (PDF) on 2015-09-23. Retrieved 2015-08-26.
- ^ Shukla RN (2014). Analysis of Chromosomes. ISBN 978-93-84568-17-7.
- ^ a b c Berg JM, Tymoczko JL, Stryer L (2002). Biochemistry (5th ed.). WH Freeman and Company. pp. 118–19, 781–808. ISBN 978-0-7167-4684-3. OCLC 179705944.
- ^ Tinoco I, Bustamante C (October 1999). "How RNA folds". Journal of Molecular Biology. 293 (2): 271–81. doi:10.1006/jmbi.1999.3001. PMID 10550208.
- ^ Higgs PG (August 2000). "RNA secondary structure: physical and computational aspects". Quarterly Reviews of Biophysics. 33 (3): 199–253. doi:10.1017/S0033583500003620. PMID 11191843. S2CID 37230785.
- ^ a b Nissen P, Hansen J, Ban N, Moore PB, Steitz TA (August 2000). "The structural basis of ribosome activity in peptide bond synthesis". Science. 289 (5481): 920–30. Bibcode:2000Sci...289..920N. doi:10.1126/science.289.5481.920. PMID 10937990.
- ^ a b Lee JC, Gutell RR (December 2004). "Diversity of base-pair conformations and their occurrence in rRNA structure and RNA structural motifs". Journal of Molecular Biology. 344 (5): 1225–49. doi:10.1016/j.jmb.2004.09.072. PMID 15561141.
- ^ Barciszewski J, Frederic B, Clark C (1999). RNA biochemistry and biotechnology. Springer. pp. 73–87. ISBN 978-0-7923-5862-6. OCLC 52403776.
- ^ Salazar M, Fedoroff OY, Miller JM, Ribeiro NS, Reid BR (April 1993). "The DNA strand in DNA.RNA hybrid duplexes is neither B-form nor A-form in solution". Biochemistry. 32 (16): 4207–15. doi:10.1021/bi00067a007. PMID 7682844.
- ^ Sedova A, Banavali NK (February 2016). "RNA approaches the B-form in stacked single strand dinucleotide contexts". Biopolymers. 105 (2): 65–82. doi:10.1002/bip.22750. PMID 26443416. S2CID 35949700.
- ^ Hermann T, Patel DJ (March 2000). "RNA bulges as architectural and recognition motifs". Structure. 8 (3): R47–54. doi:10.1016/S0969-2126(00)00110-6. PMID 10745015.
- ^ Mikkola S, Stenman E, Nurmi K, Yousefi-Salakdeh E, Strömberg R, Lönnberg H (1999). "The mechanism of the metal ion promoted cleavage of RNA phosphodiester bonds involves a general acid catalysis by the metal aquo ion on the departure of the leaving group". Journal of the Chemical Society, Perkin Transactions 2 (8): 1619–26. doi:10.1039/a903691a.
- ^ Jankowski JA, Polak JM (1996). Clinical gene analysis and manipulation: Tools, techniques and troubleshooting. Cambridge University Press. p. 14. ISBN 978-0-521-47896-0. OCLC 33838261.
- ^ Yu Q, Morrow CD (May 2001). "Identification of critical elements in the tRNA acceptor stem and T(Psi)C loop necessary for human immunodeficiency virus type 1 infectivity". Journal of Virology. 75 (10): 4902–6. doi:10.1128/JVI.75.10.4902-4906.2001. PMC 114245. PMID 11312362.
- ^ Elliott MS, Trewyn RW (February 1984). "Inosine biosynthesis in transfer RNA by an enzymatic insertion of hypoxanthine". The Journal of Biological Chemistry. 259 (4): 2407–10. doi:10.1016/S0021-9258(17)43367-9. PMID 6365911.
- ^ Cantara WA, Crain PF, Rozenski J, McCloskey JA, Harris KA, Zhang X, Vendeix FA, Fabris D, Agris PF (January 2011). "The RNA Modification Database, RNAMDB: 2011 update". Nucleic Acids Research. 39 (Database issue): D195-201. doi:10.1093/nar/gkq1028. PMC 3013656. PMID 21071406.
- ^ Söll D, RajBhandary U (1995). TRNA: Structure, biosynthesis, and function. ASM Press. p. 165. ISBN 978-1-55581-073-3. OCLC 183036381.
- ^ Kiss T (July 2001). "Small nucleolar RNA-guided post-transcriptional modification of cellular RNAs". The EMBO Journal. 20 (14): 3617–22. doi:10.1093/emboj/20.14.3617. PMC 125535. PMID 11447102.
- ^ Tirumalai MR, Rivas M, Tran Q, Fox GE (November 2021). "The Peptidyl Transferase Center: a Window to the Past". Microbiol Mol Biol Rev. 85 (4): e0010421. doi:10.1128/MMBR.00104-21. PMC 8579967. PMID 34756086.
- ^ King TH, Liu B, McCully RR, Fournier MJ (February 2003). "Ribosome structure and activity are altered in cells lacking snoRNPs that form pseudouridines in the peptidyl transferase center". Molecular Cell. 11 (2): 425–35. doi:10.1016/S1097-2765(03)00040-6. PMID 12620230.
- ^ Mathews DH, Disney MD, Childs JL, Schroeder SJ, Zuker M, Turner DH (May 2004). "Incorporating chemical modification constraints into a dynamic programming algorithm for prediction of RNA secondary structure". Proceedings of the National Academy of Sciences of the United States of America. 101 (19): 7287–92. Bibcode:2004PNAS..101.7287M. doi:10.1073/pnas.0401799101. PMC 409911. PMID 15123812.
- ^ Burghardt B, Hartmann AK (February 2007). "RNA secondary structure design". Physical Review E. 75 (2): 021920. arXiv:physics/0609135. Bibcode:2007PhRvE..75b1920B. doi:10.1103/PhysRevE.75.021920. PMID 17358380. S2CID 17574854.
- ^ Tan ZJ, Chen SJ (July 2008). "Salt dependence of nucleic acid hairpin stability". Biophysical Journal. 95 (2): 738–52. Bibcode:2008BpJ....95..738T. doi:10.1529/biophysj.108.131524. PMC 2440479. PMID 18424500.
- ^ Vater A, Klussmann S (January 2015). "Turning mirror-image oligonucleotides into drugs: the evolution of Spiegelmer(®) therapeutics". Drug Discovery Today. 20 (1): 147–55. doi:10.1016/j.drudis.2014.09.004. PMID 25236655.
- ^ Nudler E, Gottesman ME (August 2002). "Transcription termination and anti-termination in E. coli". Genes to Cells. 7 (8): 755–68. doi:10.1046/j.1365-2443.2002.00563.x. PMID 12167155. S2CID 23191624.
- ^ Hansen JL, Long AM, Schultz SC (August 1997). "Structure of the RNA-dependent RNA polymerase of poliovirus". Structure. 5 (8): 1109–22. doi:10.1016/S0969-2126(97)00261-X. PMID 9309225.
- ^ Ahlquist P (May 2002). "RNA-dependent RNA polymerases, viruses, and RNA silencing". Science. 296 (5571): 1270–73. Bibcode:2002Sci...296.1270A. doi:10.1126/science.1069132. PMID 12016304. S2CID 42526536.
- ^ a b c Cooper GC, Hausman RE (2004). The Cell: A Molecular Approach (3rd ed.). Sinauer. pp. 261–76, 297, 339–44. ISBN 978-0-87893-214-6. OCLC 174924833.
- ^ Mattick JS, Gagen MJ (September 2001). "The evolution of controlled multitasked gene networks: the role of introns and other noncoding RNAs in the development of complex organisms". Molecular Biology and Evolution. 18 (9): 1611–30. doi:10.1093/oxfordjournals.molbev.a003951. PMID 11504843.
- ^ Mattick JS (November 2001). "Non-coding RNAs: the architects of eukaryotic complexity". EMBO Reports. 2 (11): 986–91. doi:10.1093/embo-reports/kve230. PMC 1084129. PMID 11713189.
- ^ Mattick JS (October 2003). "Challenging the dogma: the hidden layer of non-protein-coding RNAs in complex organisms" (PDF). BioEssays. 25 (10): 930–39. CiteSeerX 10.1.1.476.7561. doi:10.1002/bies.10332. PMID 14505360. Archived from the original (PDF) on 2009-03-06.
- ^ Mattick JS (October 2004). "The hidden genetic program of complex organisms". Scientific American. 291 (4): 60–67. Bibcode:2004SciAm.291d..60M. doi:10.1038/scientificamerican1004-60. PMID 15487671.[데드링크]
- ^ a b c Wirta W (2006). Mining the transcriptome – methods and applications. Stockholm: School of Biotechnology, Royal Institute of Technology. ISBN 978-91-7178-436-0. OCLC 185406288.
- ^ Rossi JJ (July 2004). "Ribozyme diagnostics comes of age". Chemistry & Biology. 11 (7): 894–95. doi:10.1016/j.chembiol.2004.07.002. PMID 15271347.
- ^ Storz G (May 2002). "An expanding universe of noncoding RNAs". Science. 296 (5571): 1260–63. Bibcode:2002Sci...296.1260S. doi:10.1126/science.1072249. PMID 12016301. S2CID 35295924.
- ^ Fatica A, Bozzoni I (January 2014). "Long non-coding RNAs: new players in cell differentiation and development". Nature Reviews Genetics. 15 (1): 7–21. doi:10.1038/nrg3606. PMID 24296535. S2CID 12295847.[영구적 데드링크]
- ^ Chen Q, Yan M, Cao Z, Li X, Zhang Y, Shi J, et al. (January 2016). "Sperm tsRNAs contribute to intergenerational inheritance of an acquired metabolic disorder" (PDF). Science. 351 (6271): 397–400. Bibcode:2016Sci...351..397C. doi:10.1126/science.aad7977. PMID 26721680. S2CID 21738301.
- ^ Wei H, Zhou B, Zhang F, Tu Y, Hu Y, Zhang B, Zhai Q (2013). "Profiling and identification of small rDNA-derived RNAs and their potential biological functions". PLOS ONE. 8 (2): e56842. Bibcode:2013PLoSO...856842W. doi:10.1371/journal.pone.0056842. PMC 3572043. PMID 23418607.
- ^ Luehrsen KR, Nicholson DE, Eubanks DC, Fox GE (1981). "An archaebacterial 5S rRNA contains a long insertion sequence". Nature. 293 (5835): 755–756. Bibcode:1981Natur.293..755L. doi:10.1038/293755a0. PMID 6169998. S2CID 4341755.
- ^ Stan-Lotter H, McGenity TJ, Legat A, Denner EB, Glaser K, Stetter KO, Wanner G (1999). "Very similar strains of Halococcus salifodinae are found in geographically separated permo-triassic salt deposits". Microbiology. 145 (Pt 12): 3565–3574. doi:10.1099/00221287-145-12-3565. PMID 10627054.
- ^ Tirumalai MR, Kaelber JT, Park DR, Tran Q, Fox GE (August 2020). "Cryo-Electron Microscopy Visualization of a Large Insertion in the 5S ribosomal RNA of the Extremely Halophilic Archaeon Halococcus morrhuae". FEBS Open Bio. 10 (10): 1938–1946. doi:10.1002/2211-5463.12962. PMC 7530397. PMID 32865340.
- ^ Gueneau de Novoa P, Williams KP (January 2004). "The tmRNA website: reductive evolution of tmRNA in plastids and other endosymbionts". Nucleic Acids Research. 32 (Database issue): D104–08. doi:10.1093/nar/gkh102. PMC 308836. PMID 14681369.
- ^ Jacob F, Monod J (1961). "Genetic Regulatory Mechanisms in the Synthesis of Proteins". Journal of Molecular Biology. 3 (3): 318–56. doi:10.1016/s0022-2836(61)80072-7. PMID 13718526.
- ^ a b c d e Morris K, Mattick J (2014). "The rise of regulatory RNA". Nature Reviews Genetics. 15 (6): 423–37. doi:10.1038/nrg3722. PMC 4314111. PMID 24776770.
- ^ a b Gottesman S (2005). "Micros for microbes: non-coding regulatory RNAs in bacteria". Trends in Genetics. 21 (7): 399–404. doi:10.1016/j.tig.2005.05.008. PMID 15913835.
- ^ "2006년 노벨 생리의학상"Nobelprize.org.노벨 미디어 AB 2014.웹. 2018년 8월 6일.http://www.nobelprize.org/nobel_prizes/medicine/laureates/2006
- ^ Fire, et al. (1998). "Potent and Specific Genetic Interference by double stranded RNA in Ceanorhabditis elegans". Nature. 391 (6669): 806–11. Bibcode:1998Natur.391..806F. doi:10.1038/35888. PMID 9486653. S2CID 4355692.
- ^ Zhao J, Sun BK, Erwin JA, Song JJ, Lee JT (2008). "Polycomb proteins targeted by a short repeat RNA to the mouse X chromosome". Science. 322 (5902): 750–56. Bibcode:2008Sci...322..750Z. doi:10.1126/science.1163045. PMC 2748911. PMID 18974356.
- ^ a b c d e Rinn JL, Chang HY (2012). "Genome regulation by long noncoding RNAs". Annu. Rev. Biochem. 81: 1–25. doi:10.1146/annurev-biochem-051410-092902. PMC 3858397. PMID 22663078.
- ^ Taft RJ, Kaplan CD, Simons C, Mattick JS (2009). "Evolution, biogenesis and function of promoter- associated RNAs". Cell Cycle. 8 (15): 2332–38. doi:10.4161/cc.8.15.9154. PMID 19597344.
- ^ Orom UA, Derrien T, Beringer M, Gumireddy K, Gardini A, et al. (2010). "'Long noncoding RNAs with enhancer-like function in human cells". Cell. 143 (1): 46–58. doi:10.1016/j.cell.2010.09.001. PMC 4108080. PMID 20887892.
- ^ EGH 바그너, P Romby.(2015)."세균과 고고학의 작은 RNA: 그들이 누구인지, 그들이 무엇을 하는지, 그리고 그것을 어떻게 하는지."유전학의 진보 (Vol. 90, 페이지 133–208)
- ^ J.W. 넬슨, R.R. 브레이커 (2017) "RNA 세계의 잃어버린 언어."Sci. Signal.10, eaam8812 1–11.
- ^ Winklef WC (2005). "Riboswitches and the role of noncoding RNAs in bacterial metabolic control". Curr. Opin. Chem. Biol. 9 (6): 594–602. doi:10.1016/j.cbpa.2005.09.016. PMID 16226486.
- ^ Tucker BJ, Breaker RR (2005). "Riboswitches as versatile gene control elements". Curr. Opin. Struct. Biol. 15 (3): 342–48. doi:10.1016/j.sbi.2005.05.003. PMID 15919195.
- ^ Mojica FJ, Diez-Villasenor C, Soria E, Juez G (2000). "" "Biological significance of a family of regularly spaced repeats in the genomes of archaea, bacteria and mitochondria". Mol. Microbiol. 36 (1): 244–46. doi:10.1046/j.1365-2958.2000.01838.x. PMID 10760181. S2CID 22216574.
- ^ Brouns S, Jore MM, Lundgren M, Westra E, Slijkhuis R, Snijders A, Dickman M, Makarova K, Koonin E, Der Oost JV (2008). "Small CRISPR RNAs guide antiviral defense in prokaryotes". Science. 321 (5891): 960–64. Bibcode:2008Sci...321..960B. doi:10.1126/science.1159689. PMC 5898235. PMID 18703739.
- ^ Steitz TA, Steitz JA (July 1993). "A general two-metal-ion mechanism for catalytic RNA". Proceedings of the National Academy of Sciences of the United States of America. 90 (14): 6498–502. Bibcode:1993PNAS...90.6498S. doi:10.1073/pnas.90.14.6498. PMC 46959. PMID 8341661.
- ^ Xie J, Zhang M, Zhou T, Hua X, Tang L, Wu W (January 2007). "Sno/scaRNAbase: a curated database for small nucleolar RNAs and cajal body-specific RNAs". Nucleic Acids Research. 35 (Database issue): D183–87. doi:10.1093/nar/gkl873. PMC 1669756. PMID 17099227.
- ^ Omer AD, Ziesche S, Decatur WA, Fournier MJ, Dennis PP (May 2003). "RNA-modifying machines in archaea". Molecular Microbiology. 48 (3): 617–29. doi:10.1046/j.1365-2958.2003.03483.x. PMID 12694609. S2CID 20326977.
- ^ Cavaillé J, Nicoloso M, Bachellerie JP (October 1996). "Targeted ribose methylation of RNA in vivo directed by tailored antisense RNA guides". Nature. 383 (6602): 732–35. Bibcode:1996Natur.383..732C. doi:10.1038/383732a0. PMID 8878486. S2CID 4334683.
- ^ Kiss-László Z, Henry Y, Bachellerie JP, Caizergues-Ferrer M, Kiss T (June 1996). "Site-specific ribose methylation of preribosomal RNA: a novel function for small nucleolar RNAs". Cell. 85 (7): 1077–88. doi:10.1016/S0092-8674(00)81308-2. PMID 8674114. S2CID 10418885.
- ^ Daròs JA, Elena SF, Flores R (June 2006). "Viroids: an Ariadne's thread into the RNA labyrinth". EMBO Reports. 7 (6): 593–98. doi:10.1038/sj.embor.7400706. PMC 1479586. PMID 16741503.
- ^ Kalendar R, Vicient CM, Peleg O, Anamthawat-Jonsson K, Bolshoy A, Schulman AH (March 2004). "Large retrotransposon derivatives: abundant, conserved but nonautonomous retroelements of barley and related genomes". Genetics. 166 (3): 1437–50. doi:10.1534/genetics.166.3.1437. PMC 1470764. PMID 15082561.
- ^ Podlevsky JD, Bley CJ, Omana RV, Qi X, Chen JJ (January 2008). "The telomerase database". Nucleic Acids Research. 36 (Database issue): D339–43. doi:10.1093/nar/gkm700. PMC 2238860. PMID 18073191.
- ^ Blevins T, Rajeswaran R, Shivaprasad PV, Beknazariants D, Si-Ammour A, Park HS, Vazquez F, Robertson D, Meins F, Hohn T, Pooggin MM (2006). "Four plant Dicers mediate viral small RNA biogenesis and DNA virus induced silencing". Nucleic Acids Research. 34 (21): 6233–46. doi:10.1093/nar/gkl886. PMC 1669714. PMID 17090584.
- ^ Jana S, Chakraborty C, Nandi S, Deb JK (November 2004). "RNA interference: potential therapeutic targets". Applied Microbiology and Biotechnology. 65 (6): 649–57. doi:10.1007/s00253-004-1732-1. PMID 15372214. S2CID 20963666.
- ^ Schultz U, Kaspers B, Staeheli P (May 2004). "The interferon system of non-mammalian vertebrates". Developmental and Comparative Immunology. 28 (5): 499–508. doi:10.1016/j.dci.2003.09.009. PMID 15062646.
- ^ Whitehead KA, Dahlman JE, Langer RS, Anderson DG (2011). "Silencing or stimulation? siRNA delivery and the immune system". Annual Review of Chemical and Biomolecular Engineering. 2: 77–96. doi:10.1146/annurev-chembioeng-061010-114133. PMID 22432611.
- ^ Hsu MT, Coca-Prados M (July 1979). "Electron microscopic evidence for the circular form of RNA in the cytoplasm of eukaryotic cells". Nature. 280 (5720): 339–40. Bibcode:1979Natur.280..339H. doi:10.1038/280339a0. PMID 460409. S2CID 19968869.
- ^ Dahm R (February 2005). "Friedrich Miescher and the discovery of DNA". Developmental Biology. 278 (2): 274–88. doi:10.1016/j.ydbio.2004.11.028. PMID 15680349.
- ^ Caspersson T, Schultz J (1939). "Pentose nucleotides in the cytoplasm of growing tissues". Nature. 143 (3623): 602–03. Bibcode:1939Natur.143..602C. doi:10.1038/143602c0. S2CID 4140563.
- ^ Ochoa S (1959). "Enzymatic synthesis of ribonucleic acid" (PDF). Nobel Lecture.
- ^ Rich A, Davies D (1956). "A New Two-Stranded Helical Structure: Polyadenylic Acid and Polyuridylic Acid". Journal of the American Chemical Society. 78 (14): 3548–49. doi:10.1021/ja01595a086.
- ^ Holley RW, et al. (March 1965). "Structure of a ribonucleic acid". Science. 147 (3664): 1462–65. Bibcode:1965Sci...147.1462H. doi:10.1126/science.147.3664.1462. PMID 14263761. S2CID 40989800.
- ^ Fiers W, et al. (April 1976). "Complete nucleotide sequence of bacteriophage MS2 RNA: primary and secondary structure of the replicase gene". Nature. 260 (5551): 500–07. Bibcode:1976Natur.260..500F. doi:10.1038/260500a0. PMID 1264203. S2CID 4289674.
- ^ Napoli C, Lemieux C, Jorgensen R (April 1990). "Introduction of a Chimeric Chalcone Synthase Gene into Petunia Results in Reversible Co-Suppression of Homologous Genes in trans". The Plant Cell. 2 (4): 279–89. doi:10.1105/tpc.2.4.279. PMC 159885. PMID 12354959.
- ^ Dafny-Yelin M, Chung SM, Frankman EL, Tzfira T (December 2007). "pSAT RNA interference vectors: a modular series for multiple gene down-regulation in plants". Plant Physiology. 145 (4): 1272–81. doi:10.1104/pp.107.106062. PMC 2151715. PMID 17766396.
- ^ Ruvkun G (October 2001). "Molecular biology. Glimpses of a tiny RNA world". Science. 294 (5543): 797–99. doi:10.1126/science.1066315. PMID 11679654. S2CID 83506718.
- ^ Fichou Y, Férec C (December 2006). "The potential of oligonucleotides for therapeutic applications". Trends in Biotechnology. 24 (12): 563–70. doi:10.1016/j.tibtech.2006.10.003. PMID 17045686.
- ^ Siebert S (2006). "Common sequence structure properties and stable regions in RNA secondary structures" (PDF). Dissertation, Albert-Ludwigs-Universität, Freiburg im Breisgau. p. 1. Archived from the original (PDF) on March 9, 2012.
- ^ Szathmáry E (June 1999). "The origin of the genetic code: amino acids as cofactors in an RNA world". Trends in Genetics. 15 (6): 223–29. doi:10.1016/S0168-9525(99)01730-8. PMID 10354582.
- ^ Marlaire R (3 March 2015). "NASA Ames Reproduces the Building Blocks of Life in Laboratory". NASA. Retrieved 5 March 2015.
외부 링크
| Wikiquote는 RNA와 관련된 인용구를 가지고 있다. |
| 위키미디어 커먼즈에는 RNA와 관련된 미디어가 있다. |
- RNA World 웹 사이트 링크 모음(구조, 시퀀스, 도구, 저널)
- DNA, RNA 및 복합체의 핵산 데이터베이스 이미지
- Anna Marie Pyle의 세미나: RNA 구조