DNA
DNA| 다음에 대한 시리즈 일부 |
| 유전학 |
|---|
 |
디옥시리보핵산(/diːˈɒksksɪrararabobonjujukliː-/ -ˌkle--/![]() (듣기);[1] DNA)은 두 개의 폴리뉴클레오티드 사슬로 구성된 중합체로, 서로 감겨 모든 알려진 유기체와 많은 바이러스의 개발, 기능, 성장 및 번식을 위한 유전적 지시사항을 담고 있는 이중나선을 형성한다.DNA와 리보핵산은 핵산이다.단백질, 지질, 복합탄수화물(폴리사당체)과 함께 핵산은 알려진 모든 형태의 생명체에 필수적인 4가지 주요 형태의 고분자 중 하나이다.
(듣기);[1] DNA)은 두 개의 폴리뉴클레오티드 사슬로 구성된 중합체로, 서로 감겨 모든 알려진 유기체와 많은 바이러스의 개발, 기능, 성장 및 번식을 위한 유전적 지시사항을 담고 있는 이중나선을 형성한다.DNA와 리보핵산은 핵산이다.단백질, 지질, 복합탄수화물(폴리사당체)과 함께 핵산은 알려진 모든 형태의 생명체에 필수적인 4가지 주요 형태의 고분자 중 하나이다.
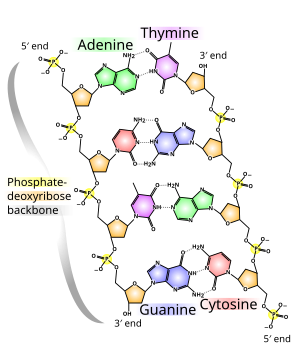
두 개의 DNA 가닥은 뉴클레오티드라고 불리는 더 단순한 단핵단위로 구성되어 있기 때문에 폴리뉴클레오티드라고 알려져 있다.[2][3]각 뉴클레오티드는 질소를 함유한 네 가지 뉴클레오베이스(시토신[C], 구아닌[G], 아데닌[A] 또는 티민[T]), 디옥시리보스라고 불리는 설탕, 인산염 그룹 중 하나로 구성되어 있다.뉴클레오티드는 한 뉴클레오티드의 당분과 다음 뉴클레오티드의 인산염 사이의 공밸런트 결합(인산-디젤 연결로 알려져 있음)에 의해 체인으로 서로 결합되어, 그 결과 설탕-인산염 등뼈가 교대로 형성된다.두 개의 분리된 폴리뉴클레오티드 가닥의 질소 베이스는 베이스 페어링 규칙(A는 T, C는 G)에 따라 서로 묶여 있으며, 수소 결합은 이중 가닥 DNA를 만든다.보완적 질소 베이스는 피리미딘과 청록의 두 그룹으로 나뉜다.DNA에서 피리미딘은 티민과 시토신이며, 청개는 아데닌과 구아닌이다.
두 가닥의 이중 가닥 DNA는 동일한 생물학적 정보를 저장한다.이 정보는 두 가닥이 분리될 때 복제된다.DNA의 큰 부분(인간의 경우 98% 이상)은 비코딩인데, 이는 이 부분들이 단백질 배열을 위한 패턴으로 작용하지 않는다는 것을 의미한다.두 가닥의 DNA는 서로 반대 방향으로 흐르기 때문에 반감각이다.각 설탕에 부착된 것은 네 가지 종류의 뉴클레오바제(또는 염기) 중 하나이다.유전 정보를 암호화하는 것은 등뼈를 따라 이 네 개의 뉴클레오바제의 순서다.RNA 가닥은 전사라고 불리는 과정에서 DNA 가닥을 템플릿으로 삼아 생성되는데, 여기서 RNA가 우라실(U)을 대체하는 티민(T)의 경우를 제외하고 DNA 베이스가 해당 베이스와 교환된다.[4]유전자 코드에 따르면, 이 RNA 가닥들은 번역이라고 불리는 과정에서 단백질 내의 아미노산의 순서를 명시한다.
진핵 세포 안에서 DNA는 염색체라고 불리는 긴 구조로 조직된다.전형적인 세포분열이 일어나기 전에 이들 염색체들은 DNA 복제 과정에서 복제되어 각 딸 세포에 완전한 염색체 세트를 제공한다.진핵생물(동물, 식물, 곰팡이, 양성자)은 대부분의 DNA를 세포핵 안에 핵 DNA로 저장하고, 일부는 미토콘드리아 DNA로 미토콘드리아에 저장하거나 엽록체 DNA로 엽록체 속에 저장한다.[5]이와는 대조적으로, 원핵생물(박테리아와 고고학)은 세포질, 원형 염색체에만 DNA를 저장한다.진핵 염색체 내에서는 히스톤과 같은 염색질 단백질이 DNA를 압축하고 조직한다.이러한 압축 구조는 DNA와 다른 단백질 사이의 상호작용을 유도하여 DNA의 어느 부분이 전사되는지를 통제하는 데 도움을 준다.
특성.
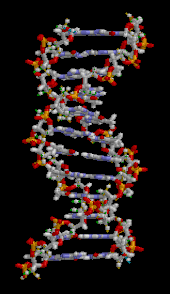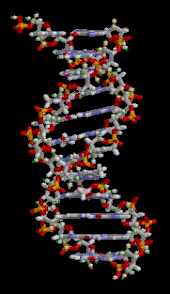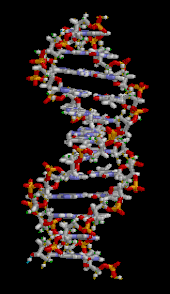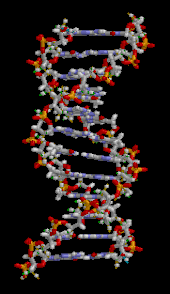
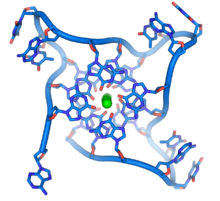
DNA는 뉴클레오티데스라고 불리는 반복적인 단위로 만들어진 긴 중합체로, 보통은 하나의 문자로 상징된다.A, T, C 또는 G.[6][7]DNA의 구조는 그 길이에 따라 동적으로 작용하며, 타이트한 루프와 다른 모양으로 감을 수 있다.[8]모든 종에서 그것은 수소 결합에 의해 서로 묶여 있는 두 개의 나선 사슬로 구성되어 있다.두 체인 모두 같은 축을 중심으로 코일링되며, 34 å스트룀(3.4 nm)의 동일한 피치를 가진다.체인 쌍의 반지름은 10㎛(1.0nm)이다.[9]다른 연구에 따르면, 다른 용액에서 측정했을 때, DNA 체인은 22–26 å(2.2–2.6 nm)의 폭을 측정했고, 하나의 뉴클레오티드 단위는 3.3 å(0.33 nm)의 길이를 측정했다.[10]비록 각각의 개별 뉴클레오티드는 매우 작지만, DNA 중합체는 매우 길 수 있고 1번 염색체와 같이 수억 개의 뉴클레오티드를 포함할 수 있다.염색체 1은 약 2억 2천만 개의 염기쌍을 가진 가장 큰 인간 염색체로, 곧게 펴면 길이가 85mm가 된다.[11]
DNA는 보통 한 가닥으로 존재하는 것이 아니라 한 쌍의 가닥으로 단단하게 고정되어 있다.[9][12]이 두 개의 긴 가닥은 이중나선 모양으로 서로 감겨 있다.뉴클레오티드는 분자의 백본의 한 부분(사슬을 서로 붙들고 있는 것)과 뉴클레오바아제(나선의 다른 DNA 가닥과 상호작용하는 것)를 모두 포함한다.설탕과 연결된 뉴클레오바제를 뉴클레오사이드라고 하며, 설탕과 하나 이상의 인산염 그룹에 연결된 밑동을 뉴클레오타이드라고 한다.다중 연계 뉴클레오티드(DNA와 같이)로 구성된 생물폴리머를 폴리뉴클레오티드라고 한다.[13]
DNA 가닥의 등뼈는 인산염과 설탕 그룹이 번갈아 가며 만들어진다.[14]DNA의 설탕은 2데옥시리보스인데, 이것은 펜토오스(5탄소)의 설탕이다.이 당분들은 인접한 설탕 고리의 세 번째와 다섯 번째 탄소 원자들 사이에 인광 결합을 형성하는 인산염 그룹에 의해 결합된다.이것들은 3′-end (3 prime-end) 탄소와 5end-end (5 prime-end) 탄소로 알려져 있는데, 이것은 이 탄소 원자와 디옥시리보스가 글리코시드 결합을 형성하는 기초 원자를 구별하기 위해 사용되고 있다.따라서 어떤 DNA 가닥도 일반적으로 리보스의 5㎛ 탄소(5㎛ 인광)에 인산염 그룹이 붙어 있는 한쪽 끝과 리보스의 3㎛ 탄소(3㎛ hydroxyl)에 자유로운 히드록실 그룹이 붙어 있는 다른 쪽 끝을 가지고 있다.설탕-인산염 등뼈를 따라 3′과 5′ 탄소의 방향은 각 DNA 가닥에 방향성(극성이라고도 함)을 혼동한다.핵산 이중나선에서는 한 가닥에 있는 뉴클레오티드의 방향이 다른 가닥에 있는 방향과 반대인데, 그 가닥은 항타렐이다.DNA 가닥의 비대칭 끝은 방향성이 5 prime end (5㎛ )와 3 prime end (3㎛)라고 하며, 5 perme end는 말단 인산염 그룹을, 3 permend는 말단 히드록실 그룹을 갖는다고 한다.DNA와 RNA의 한 가지 주요한 차이점은 설탕인데, DNA의 2데옥시리보스가 RNA의 관련 펜토오스 당 리보오스로 대체된다.[12]
DNA 이중나선은 주로 뉴클레오티드 사이의 수소 결합과 방향성 뉴클레오바제들 사이의 염기 쌓기 상호작용의 두 가지 힘에 의해 안정된다.[16]DNA에서 발견된 네 가지 염기는 아데닌(A), 시토신(C), 구아닌(G), 티민(T)이다.이 네 가지 염기들은 아데노신 단인산염에 대해 알 수 있듯이 완전한 뉴클레오티드를 형성하기 위해 당인산염에 부착되어 있다.아데닌은 티민, 구아닌 쌍과 시토신 쌍을 이루며 A-T와 G-C 염기쌍을 형성한다.[17][18]
뉴클레오바아제 분류
뉴클레오바제는 5mb와 6mb가 융합된 이형성분인 청개류 A와 G와 6mb의 고리인 피리미딘, C와 T의 두 종류로 분류된다.[12]제5의 피리미딘 뉴클레오바아제인 우라실(U)은 보통 RNA에서 티민 대신 쓰이며 링에 메틸군이 없어 티민과는 다르다.RNA와 DNA 외에도 핵산의 성질을 연구하거나 생명공학에 사용하기 위해 인공 핵산 아날로그가 많이 만들어졌다.[19]
비수평적 근거
변형된 염기는 DNA에서 발생한다.이 중 첫 번째로 인정된 것은 5메틸시토신이었는데, 1925년 마이코박테리움 결핵의 게놈에서 발견되었다.[20]박테리아 바이러스(박테리오파지)에 이러한 비카노니컬 베이스가 존재하는 이유는 박테리아에 존재하는 제한 효소를 피하기 위해서입니다.이 효소 체계는 적어도 부분적으로 바이러스에 의한 감염으로부터 박테리아를 보호하는 분자 면역 체계로서 작용한다.[21]염기서열과 DNA 염기서열인 아데닌의 수정은 식물과 동물의 유전자 발현 후생유전학적 제어에 중요한 역할을 한다.[22]
DNA에서 발견된 비수평적 기초 목록
DNA에서 많은 비수체적 기초가 발생하는 것으로 알려져 있다.[23]이 중 대부분은 성기 베이스와 요람의 변형이다.
- 수정아데노신
- N6-카르바모일-메틸아데닌
- N6-메틸아데닌
- 수정구아닌
- 7-다자와닌
- 7-메틸과닌
- 수정사이토신
- N4-메틸시토신
- 5-카르복실시토신
- 5-포르밀시토신
- 5-글리코실하이드록시메틸시토신
- 5-히드록시토신
- 5-메틸시토신
- 수정 티미딘
- α-글루타미시미딘
- α-푸르트레시닐시민
- 우라실 및 수정
- 베이스 J
- 우라실
- 5-디히드록시펜타우라실
- 5-히드록시메틸데옥시우라실
- 다른이들
- 데옥시아케오신
- 2,6-다이아미노푸린(2-아미노아데닌)

그루브
두 개의 나선 가닥이 DNA 등뼈를 형성한다.가닥 사이의 공간 또는 홈을 추적하는 또 다른 이중나선이 발견될 수 있다.이러한 공극은 기본 쌍에 인접하며 바인딩 사이트를 제공할 수 있다.가닥이 서로 대칭적으로 위치하지 않기 때문에 홈의 크기가 서로 다르다.주 홈은 폭 22 22스트룀(2.2 nm)이고, 부 홈은 폭 12 å(1.2 nm)이다.[24]주 그루브의 폭이 넓어 단부 그루브보다 주 그루브에서 베이스의 가장자리의 접근성이 좋다.그 결과 이중 가닥 DNA에서 특정 염기서열과 결합할 수 있는 전사인자와 같은 단백질은 대개 주요 홈에 노출된 염기서열과 접촉하게 된다.[25]이러한 상황은 세포 내에서 DNA의 비정상적인 순응에 따라 달라지지만(아래 참조), 주형 및 경형 홈은 항상 DNA가 다시 일반적인 B형식으로 꼬일 경우 볼 수 있는 폭의 차이를 반영하여 명명된다.
베이스 페어링
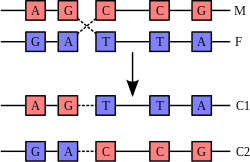
DNA 이중나선에서는 각 유형의 뉴클레오바제가 한 가닥의 결합에 다른 가닥에는 하나의 뉴클레오바아제만 있다.이것을 보완 베이스 페어링이라고 한다.청진은 피리미딘에 수소 결합을 형성하며, 아데닌은 두 개의 수소 결합에서 티민에만 결합하고, 시토신은 세 개의 수소 결합에서 구아닌에만 결합한다.이 두 개의 뉴클레오티드가 이중 나선(6탄소 링에서 6탄소 링까지)을 가로질러 함께 결합하는 배열을 왓슨 크릭 베이스 쌍이라고 한다.GC 함량이 높은 DNA는 GC 함량이 낮은 DNA보다 안정적이다.후그스틴 베이스 페어(6-탄소 링과 5-탄소 링을 결합하는 수소)는 드물게 베이스 페어링의 변형이다.[26]수소결합은 공밸런스가 아니기 때문에 비교적 쉽게 깨지고 재결합할 수 있다.따라서 이중나선 안에 있는 두 가닥의 DNA는 기계적 힘이나 고온에 의해 지퍼처럼 떼어낼 수 있다.[27]이 염기쌍보완성의 결과, DNA 나선의 이중 가닥 배열 순서에 있는 모든 정보가 각 가닥에 중복되어 DNA 복제에 필수적이다.상호보완적인 염기쌍들 사이의 이러한 가역적 그리고 특정한 상호작용은 유기체의 DNA의 모든 기능에 매우 중요하다.[7]
ssDNA 대 dsDNA
위에서 언급했듯이, 대부분의 DNA 분자는 실제로 두 개의 중합체 가닥으로, 비협착 결합에 의해 나선형으로 결합된다; 이 이중 가닥(dsDNA) 구조는 주로 G,C 스택에 가장 강한 주내 염기 쌓기 상호작용에 의해 유지된다.이 두 가닥은 두 개의 단일 가닥 DNA(ssDNA) 분자를 형성하기 위해 용해라고 알려진 과정으로 분리될 수 있다.용해는 고온, 저염, 고 pH에서 발생한다(저 pH도 DNA를 녹이지만 산성 감퇴로 DNA가 불안정하기 때문에 저 pH는 거의 사용되지 않는다).
dsDNA 형태의 안정성은 GC-함량(% G,C basepares)뿐만 아니라 시퀀스(쌓기가 시퀀스마다 다르기 때문에)와 길이(더 긴 분자는 더 안정적이다)에 따라 달라진다.안정성은 다양한 방법으로 측정할 수 있다; 일반적인 방법은 이중 가닥 분자의 50%가 단일 가닥 분자로 변환되는 온도인 용융 온도(Tm 값이라고도 한다)이다; 용융 온도는 이온 강도와 DNA 농도에 의존한다.결과적으로, DNA의 두 가닥 사이의 연관성의 강도를 결정하는 것은 GC 염기쌍의 비율과 DNA 이중나선의 전체 길이 둘 다이다.GC 함량이 높은 긴 DNA 나선은 교호작용 강도가 높은 반면 AT 함량이 높은 짧은 나선은 교호작용 강도가 더 약하다.[28]생물학에서는 일부 프로모터의 TATAAT Pribnow 상자처럼 쉽게 분리해야 하는 DNA 이중나선의 일부분은 AT 함량이 높은 경향이 있어 가닥이 쉽게 분리된다.[29]
실험실에서는 수소 결합의 절반을 깨는 데 필요한 용해 온도 T를m 찾아 이 상호작용의 강도를 측정할 수 있다.DNA 이중나선의 모든 염기쌍이 녹으면 가닥은 분리되어 완전히 독립적인 두 개의 분자로 용액에 존재한다.이 단일 가닥의 DNA 분자들은 하나의 공통 형태를 가지고 있지 않지만, 어떤 순응은 다른 것들보다 더 안정적이다.[30]
감각과 반감
DNA 염기서열은 단백질로 번역되는 메신저 RNA 카피와 동일하면 '센스' 염기서열이라고 한다.[31]반대쪽 가닥의 순서를 "항체" 순서라고 한다.감각 및 항이센스 시퀀스는 모두 동일한 DNA 가닥의 서로 다른 부분에 존재할 수 있다(즉, 양쪽 가닥 모두 감지 및 항이센스 시퀀스를 포함할 수 있다).원핵생물과 진핵생물의 경우 모두 항산화 RNA 염기서열이 생성되지만 이들 RNA의 기능이 완전히 명확한 것은 아니다.[32]한 가지 제안은 항이센스 RNA가 RNA-RNA 베이스 페어링을 통해 유전자 발현을 조절하는 데 관여한다는 것이다.[33]
원핵생물과 진핵생물의 DNA 서열 몇 개, 플라스미드와 바이러스의 DNA 서열은 유전자가 겹쳐서 감각과 항이센스 가닥의 구분을 흐리게 한다.[34]이 경우 어떤 DNA 시퀀스는 이중 듀티(double duty)를 하며, 한 가닥을 따라 읽으면 한 단백질을, 다른 가닥을 따라 반대 방향으로 읽으면 두 번째 단백질을 인코딩한다.박테리아에서는 이러한 중첩이 유전자 전사의 규제에 관여할 수 있는 반면 바이러스에서는 중첩 유전자가 작은 바이러스 게놈 안에서 암호화할 수 있는 정보의 양을 증가시킨다.[35][36]
슈퍼코일링
DNA는 DNA 슈퍼코일링이라 불리는 과정에서 밧줄처럼 꼬일 수 있다.DNA가 "완화"된 상태에서 가닥은 보통 10.4 염기쌍마다 한 번씩 이중나선의 축을 돌지만 DNA가 꼬이면 가닥이 더 단단하게 또는 느슨하게 감겨진다.[37]나선의 방향으로 DNA가 꼬이면 이것은 양의 초코일링이고, 밑부분은 더욱 단단하게 고정되어 있다.반대로 꼬이면 음의 슈퍼코일링이고, 베이스가 더 쉽게 분리된다.자연에서 대부분의 DNA는 topoisomerase라고 불리는 효소에 의해 유입되는 약간 부정적인 슈퍼코일링을 가지고 있다.[38]이러한 효소들은 또한 전사나 DNA 복제와 같은 과정에서 DNA 가닥에 유입되는 비틀림 스트레스를 완화시키기 위해 필요하다.[39]
대체 DNA 구조
DNA는 A-DNA, B-DNA, Z-DNA 형태를 포함하는 많은 가능한 순응에 존재하지만, B-DNA와 Z-DNA만이 기능 유기체에서 직접 관찰되었다.[14]DNA가 채택하는 순응은 수화 수준, DNA 염기서열, 슈퍼코일링의 양과 방향, 염기의 화학적 변형, 금속 이온의 종류와 농도, 용액 내 폴리아민의 유무에 따라 달라진다.[40]
A-DNA X선 회절 패턴과 B-DNA에 대한 첫 번째 보고서는 패터슨 기능에 기반한 분석을 사용했으며, 이는 DNA의 방향 섬유에 대한 구조 정보를 제한적으로 제공했을 뿐이다.[41][42]대체 분석은 1953년 윌킨스 외 연구진에 의해 베셀 함수의 사각형 측면에서 고수화 DNA 섬유의 생체내 B-DNA X선 회절-스캐터링 패턴에 대해 제안되었다.[43]같은 저널에서 제임스 왓슨과 프란시스 크릭은 DNA X선 회절 패턴의 분자 모델링 분석을 발표하여 그 구조가 이중 나선형임을 시사했다.[9]
B-DNA 형태는 세포에서 발견되는 조건 하에서 가장 흔하지만,[44] 세포에 존재하는 높은 수화 수준에서 발생하는 것은 잘 정의된 순응이 아니라 관련 DNA 순응의[45] 계열이다.그들의 상응하는 X선 회절과 산란 패턴은 상당한 수준의 장애를 가진 분자 파라크리스탈의 특징이다.[46][47]
A-DNA 형태는 B-DNA에 비해 더 넓은 오른손 나선형으로 얕고 넓은 마이너 그루브와 좁고 깊은 메인 그루브가 있다.A 형태는 부분적으로 탈수된 DNA 샘플에서 비생리학적 조건에서 발생하는 반면, 세포에서는 DNA와 RNA 가닥의 혼합 쌍과 효소-DNA 복합체에서 생성될 수 있다.[48][49]염기들이 메틸화에 의해 화학적으로 변형된 DNA의 부분들은 순응에서 더 큰 변화를 겪을 수 있고 Z 형태를 채택할 수 있다.여기서 가닥은 보다 일반적인 B형과는 정반대인 왼손 나선형에서 나선축을 중심으로 돈다.[50]이러한 특이한 구조는 특정 Z-DNA 결합 단백질에 의해 인식될 수 있으며 전사 규제에 관여할 수 있다.[51]
대체 DNA 화학
수년 동안, 외계 생물학자들은 현재 알려진 생명체와는 근본적으로 다른 생화학 및 분자 과정을 사용하는 지구의 가정된 미생물 생물권인 그림자 생물권의 존재를 제안해 왔다.제안 중 하나는 DNA에 인 대신 비소를 사용하는 생명체의 존재였다.2010년 GFAJ-1 박테리아에 대한 연구 결과는 [52][53]논쟁의 여지가 있었지만 발표되었고,[53][54] 증거는 박테리아가 비소가 DNA 백본과 다른 생체 분자에 통합되는 것을 적극적으로 막았다는 것을 암시한다.[55]
쿼드플렉스 구조물
선형 염색체의 끝에는 텔로미어라고 불리는 DNA의 전문 영역이 있다.정상적으로 DNA를 복제하는 효소는 염색체의 극한 3′ 끝부분을 복제할 수 없기 때문에 이들 부위의 주요 기능은 세포가 텔로머레이즈 효소를 사용하여 염색체 끝을 복제할 수 있도록 하는 것이다.[56]이러한 전문 염색체 캡은 또한 DNA 끝을 보호하는데 도움을 주며, 세포의 DNA 수리 시스템이 그것들을 교정할 손상으로 취급하는 것을 막는다.[57]인간의 세포에서 텔로미어는 보통 간단한 TTAGGG 수열의 수천 번 반복을 포함하는 단일 가닥 DNA의 길이다.[58]
이러한 구아닌이 풍부한 염기서열은 다른 DNA 분자에서 발견되는 일반적인 염기쌍이 아니라 4 염기단위의 쌓인 세트의 구조를 형성함으로써 염색체의 끝을 안정시킬 수 있다.여기서 구아닌 테트라드로 알려진 구아닌 4기지가 평판을 이루고 있다.그런 다음 이 평평한 4 베이스 유닛들이 서로 위로 쌓아 올려 안정적인 G-quadruplex 구조를 형성한다.[60]이러한 구조물은 베이스의 가장자리 사이의 수소 결합과 각 4 베이스 유닛의 중심에 있는 금속 이온의 킬레이트화에 의해 안정화된다.[61]다른 구조물도 형성될 수 있는데, 4개의 베이스의 중심 세트는 베이스를 중심으로 접힌 단일 가닥 또는 여러 개의 서로 다른 평행 가닥 중 하나로 각각 중앙 구조에 하나의 베이스를 기여한다.
이러한 적층 구조 외에도, 텔로미어는 텔로미어 루프 또는 T-루프라고 불리는 큰 루프 구조를 형성한다.여기서 단발성 DNA는 텔로미레 결합 단백질에 의해 안정화된 긴 원형으로 둥글게 휘어진다.[62]T-루프의 맨 끝에서, 단일 가닥 텔로미어 DNA는 텔로미어 가닥이 이중 나선 DNA와 두 가닥 중 하나에 염기쌍을 교란시킴으로써 이중 가닥 DNA의 영역에 고정된다.이 세 가닥 구조물을 변위 루프 또는 D-루프라고 한다.[60]
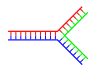 |  |
| 단일 분기 | 여러 가지 가지 |
갈라진 DNA
DNA에서 마찰은 DNA의 다른 보완적 이중 가닥의 끝에 비완성 영역이 존재할 때 발생한다.그러나, 만약 세 번째 가닥의 DNA가 도입되고 기존 이중 가닥의 닳은 부분과 교배할 수 있는 인접 영역을 포함한다면, 분기된 DNA는 발생할 수 있다.가장 간단한 DNA 갈림길의 예는 DNA의 세 가닥만 포함하지만, 추가 가닥과 여러 가지 가지를 포함하는 복합체도 가능하다.[63]분기된 DNA는 나노기술에서 기하학적 형상을 만들기 위해 사용될 수 있다. 아래 기술에서의 사용에 관한 섹션을 참조하라.
인공 베이스
여러 개의 인공 뉴클레오바제가 합성되어, 하치모지 DNA라는 8 베이스 DNA 아날로그에 성공적으로 접목되었다.S, B, P, Z로 명명된 이들 인공 베이스는 예측 가능한 방식으로 서로 결합할 수 있으며(S-B와 P-Z), DNA의 이중나선 구조를 유지하며 RNA에 전사될 수 있다.그들의 존재는 지구상에서 진화한 네 가지 자연 핵물질에 대해 특별한 것이 없다는 것을 암시하는 것으로 볼 수 있다.[64][65]한편, DNA는 DNA의 성적증명서로서의 역할뿐만 아니라 세포 내 여러 가지 임무를 수행하는 RNA와 밀접한 관련이 있다.이 목적을 위해 그것은 하나의 구조로 접혀져야 한다.가능한 모든 구조를 만들 수 있도록 하기 위해서는 해당 RNA에 최소한 4개의 베이스가 필요한 반면,[66] 높은 숫자도 가능하지만 이는 최소한의 노력의 자연 원리에 반하는 것으로 나타났다.
화학적 수정 및 변경된 DNA 포장
베이스 수정 및 DNA 포장
유전자의 표현은 염색체라고 불리는 구조에서 DNA가 염색체로 포장되는 방법에 의해 영향을 받는다.염기변형은 포장에 관여할 수 있으며, 유전자 발현이 낮거나 없는 부위는 대개 시토신 염기의 높은 메틸화를 포함한다.DNA 포장과 유전자 발현에 미치는 영향은 또한 DNA가 염색질 구조로 포장된 히스톤 단백질 코어의 공동가변형이나 염색질 리모델링 복합체에 의해 수행되는 리모델링에 의해 발생할 수 있다(크롬질 리모델링 참조).더 나아가 DNA 메틸화와 히스톤 수정 사이에는 교차점이 있으므로, 그들은 염색질과 유전자 발현에 조화롭게 영향을 줄 수 있다.[67]
예를 들어, 시토신 메틸화는 5-메틸시토신을 생성하는데, 이것은 염색체의 X-비활성화에 중요하다.[68]평균 메틸화 수준은 유기체마다 다르다. 즉, 기생충인 선충은 시토신 메틸화가 없는 반면, 척추동물은 DNA의 최대 1%가 5-메틸시토신을 함유하고 있어 더 높은 수준을 가지고 있다.[69]5메틸시토신의 중요성에도 불구하고 티민 기지를 남기는 것은 탈아민화 할 수 있기 때문에 메틸화된 시토신은 특히 변이되기 쉽다.[70]다른 염기변형으로는 박테리아에 아데닌 메틸화, 뇌에 5-하이드록시메틸시토신 존재,[71] 키네토플라스틱에 'J-베이스'를 생산하기 위한 우라실 글리코실화 등이 있다.[72][73]
피해
DNA는 많은 종류의 돌연변이들에 의해 손상될 수 있는데, 이것은 DNA 서열을 바꾼다.무타겐에는 산화제, 알킬링제, 자외선, X선 등 고에너지 전자기 방사선도 포함된다.생산되는 DNA 손상의 유형은 돌연변이 물질의 종류에 따라 다르다.예를 들어, UV 빛은 피리미딘 염기들 사이의 교차 링크인 티민 다이머를 생성함으로써 DNA를 손상시킬 수 있다.[75]반면 활성산소나 과산화수소 같은 산화물은 특히 구아노신을 비롯한 염기변형, 이중스트란드가 파손되는 등 여러 형태의 피해를 발생시킨다.[76]대표적인 인간 세포에는 산화 손상을 입은 약 15만 개의 염기가 들어 있다.[77]이러한 산화성 병변 중 가장 위험한 병변은 이중 가닥 파손인데, 이는 수리가 어렵고 점 변이, 삽입, DNA 염기서열에서 삭제, 염색체 변환 등이 발생할 수 있기 때문이다.[78]이러한 돌연변이는 암을 유발할 수 있다.DNA 수리 메커니즘에 내재된 한계 때문에 인간이 충분히 오래 산다면 결국 모두 암에 걸릴 것이다.[79][80]반응성 산소종을 생성하는 정상적인 세포 과정으로 인해 자연적으로 발생하는 DNA 손상, 세포수 등의 수력학적 활동도 빈번하게 발생한다.이러한 손상의 대부분은 수리되지만, 어떤 세포에서도 수리 과정의 작용에도 불구하고 일부 DNA 손상은 남아 있을 수 있다.이 남아있는 DNA 손상은 나이가 들면서 포유류 사후 조직에 축적된다.이러한 축적은 노화의 중요한 근본 원인으로 보인다.[81][82][83]
많은 돌연변이들이 인접한 두 염기쌍 사이의 공간에 들어맞는데, 이를 중간합계라고 한다.대부분의 인터칼레이터는 방향성 및 평면 분자로, 예로는 에티듐브로마이드, 아세리딘, 다우노마이신, 독소루비신 등이 있다.염기쌍 사이에 인터칼레이터가 맞으려면 염기들이 분리되어 이중나선을 풀어서 DNA 가닥을 왜곡해야 한다.이것은 전사와 DNA 복제를 억제하여 독성과 돌연변이를 일으킨다.[84]그 결과, DNA 인터칼레이터는 발암물질일 수 있으며, 탈리도마이드의 경우 테라토겐이 될 수 있다.[85]벤조[아]피렌 디올 에폭시드와 아플라톡신 같은 다른 것들은 복제의 오류를 유발하는 DNA 유도체를 형성한다.[86]그럼에도 불구하고, DNA 전사와 복제를 억제하는 능력 때문에, 다른 유사한 독소들이 빠르게 성장하는 암세포를 억제하기 위해 화학 요법에도 사용된다.[87]
생물학적 함수
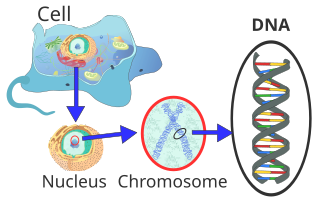
DNA는 보통 진핵생물에서는 선형 염색체로, 원핵생물에서는 원형 염색체로 발생한다.세포의 염색체 세트는 그것의 게놈을 구성한다; 인간의 게놈은 46개의 염색체로 배열된 대략 30억개의 염기쌍의 DNA를 가지고 있다.[88]DNA에 의해 운반되는 정보는 유전자라고 불리는 DNA 조각들의 순서에 따라 보관된다.유전자의 유전정보 전달은 보완적 염기쌍을 통해 이루어진다.예를 들어 전사에서는 세포가 유전자의 정보를 사용할 때 DNA와 정확한 RNA 뉴클레오티드 사이의 흡인력을 통해 DNA 서열을 보완 RNA 서열로 복사한다.보통, 이 RNA 복사는 RNA 뉴클레오티드 사이의 동일한 상호작용에 의존하는 번역이라고 불리는 과정에서 일치하는 단백질 서열을 만드는 데 사용된다.대체적인 방법으로, 세포는 DNA 복제라고 불리는 과정으로 단순히 그것의 유전 정보를 복사할 수 있다.이러한 기능의 세부사항은 다른 기사에서 다루어진다; 여기서 초점은 게놈의 기능을 중재하는 DNA와 다른 분자 사이의 상호작용에 있다.
유전자와 게놈
유전체 DNA는 DNA 응축이라고 불리는 과정에서 세포의 작은 부피에 맞도록 단단하고 질서 있게 포장되어 있다.진핵생물에서 DNA는 세포핵에 위치하며, 미토콘드리아와 엽록체에는 소량이 있다.원핵생물에서, DNA는 핵체라고 불리는 세포질에서 불규칙하게 생긴 몸체 안에 들어 있다.[89]게놈의 유전 정보는 유전자 안에 들어 있으며, 유기체의 이 정보의 완전한 집합을 유전자형이라고 부른다.유전자는 유전의 단위로서 유기체의 특정 특성에 영향을 미치는 DNA의 한 부분이다.유전자는 전사할 수 있는 개방형 독서 프레임과 개방형 독서 프레임의 전사를 제어하는 프로모터, 엔핸서 등의 규제 시퀀스를 포함하고 있다.
많은 종에서 게놈의 총 염기서열 중 극히 일부만이 단백질을 부호화한다.예를 들어 인간 게놈의 약 1.5%만이 단백질 코딩 엑손으로 구성되며, 50% 이상의 인간 DNA가 비코딩 반복 시퀀스로 구성된다.[90]진핵 게놈에 그렇게 많은 비코딩 DNA가 존재하는 이유와 종들 간의 게놈 크기, 즉 C-값의 엄청난 차이는 "C-값 수수께끼"[91]라고 알려진 오랜 퍼즐을 나타낸다.그러나 단백질을 코드화하지 않는 일부 DNA 염기서열은 유전자 발현 규제에 관여하는 기능적인 비코딩 RNA 분자를 여전히 암호화할 수 있다.[92]
일부 비코딩 DNA 염기서열은 염색체에서 구조적 역할을 한다.텔로미어와 센트롬은 일반적으로 유전자가 거의 없지만 염색체의 기능과 안정성을 위해 중요하다.[57][94]인간에게 부호화되지 않은 DNA의 풍부한 형태는 돌연변이에 의해 장애가 된 유전자의 복제품인 유사 유전자다.[95]이러한 염기서열은 유전자 복제와 분리의 과정을 통해 때때로 새로운 유전자를 생성하기 위한 원유전자 재료로 작용할 수 있지만, 대개는 분자화석에 지나지 않는다.[96]
전사 및 번역
유전자는 유전 정보를 포함하고 유기체의 표현형에 영향을 줄 수 있는 DNA의 배열이다.유전자 내에서, DNA 가닥을 따라 염기서열은 메신저 RNA 염기서열을 정의하고, 그 다음 하나 이상의 단백질 염기서열을 정의한다.유전자의 뉴클레오티드 염기서열과 단백질의 아미노산 염기서열 사이의 관계는 유전코드라고 총칭되는 번역 규칙에 의해 결정된다.유전자 코드는 세 개의 뉴클레오티드(예: ACT, CAG, TTT)의 시퀀스에서 형성된 코돈이라 불리는 세 글자의 '단어'로 구성된다.
전사에서는 유전자의 코돈들이 RNA 중합효소에 의해 메신저 RNA로 복사된다.이 RNA 복사는 리보솜에 의해 해독되는데, 리보솜은 아미노산을 운반하는 RNA를 전달하기 위해 메신저 RNA를 베이스 페어링하여 RNA 염기서열을 판독한다.3글자 조합은 4개의 베이스가 있으므로 64개의 코돈(4개의3 조합)이 가능하다.이것들은 20개의 표준 아미노산을 암호화하여, 대부분의 아미노산을 1개 이상의 가능한 코돈으로 제공한다.또한 코딩 영역의 끝을 나타내는 세 개의 '정지' 또는 '말도 안 되는' 코돈들이 있다; 이것들은 TAG, TAA, TGA 코돈이다. (mRNA의 UAG, UAA, UGA)
복제
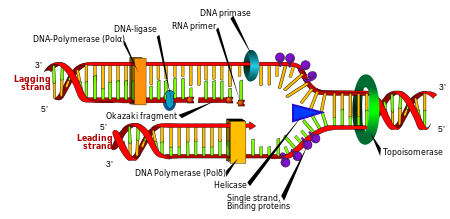
세포분열은 유기체가 성장하기 위해 필수적이지만 세포가 분열할 때 게놈의 DNA를 복제해야 두 딸세포가 모체와 동일한 유전정보를 가질 수 있다.DNA의 이중 가닥 구조는 DNA 복제를 위한 간단한 메커니즘을 제공한다.여기서 두 가닥이 분리되어 각 가닥의 보완 DNA 염기서열은 DNA 중합효소라는 효소에 의해 재생된다.이 효소는 상호보완적인 염기쌍을 통해 정확한 염기쌍을 찾아 원래의 가닥에 결합시켜 보완 가닥을 만든다.DNA 중합체는 DNA 가닥을 5㎛에서 3㎛ 방향으로만 연장할 수 있기 때문에 이중나선의 항타렐 가닥을 복사하는 데 서로 다른 메커니즘이 사용된다.[97]이렇게 해서 낡은 가닥의 기지는 새로운 가닥에 어떤 기지가 나타나는지 지시하고, 세포는 결국 그 DNA의 완벽한 사본으로 끝난다.
세포외 핵산
세포사멸에 의해 방출되는 알몸 세포외 DNA(eDNA)는 거의 어디서나 볼 수 있다.토양에서의 농도는 2μg/L 정도로 높을 수 있으며, 자연 수생 환경에서의 농도는 88μg/L로 높을 수 있다.[98]다양한 가능한 기능이 eDNA에 제안되었다: 그것은 수평적인 유전자 전달에 관여할 수 있고,[99] 영양분을 제공할 수 있으며,[100] 그리고 그것은 이온이나 항생제를 모집하거나 적정화하는 완충제 역할을 할 수 있다.[101]세포외 DNA는 여러 박테리아 종의 생물필름에서 기능성 세포외 매트릭스 성분으로 작용한다.그것은 바이오필름의 특정 세포 유형의 부착과 분산을 규제하는 인식 요인으로 작용할 수 있고,[102] 그것은 바이오필름 형성에 기여할 수 있으며,[103] 바이오필름의 체력과 생물학적 스트레스에 대한 내성에 기여할 수 있다.[104]
세포가 없는 태아의 DNA는 산모의 혈액에서 발견되며, 성장 중인 태아에 대한 많은 정보를 결정하기 위해 배열될 수 있다.[105]
환경 DNA eDNA라는 이름 아래, 자연과학에서 생태를 위한 조사 도구로서, 물, 공기 또는 육지에서 종의 이동과 존재를 감시하고, 한 지역의 생물 다양성을 평가하는 것이 증가하였다.[106][107]
중성미자외 트랩
중성미자 세포외 트랩(NET)은 주로 백혈구의 일종인 중성미자가 숙주세포의 손상을 최소화하면서 세포외 병원체를 죽일 수 있도록 하는 DNA로 구성된 세포외 섬유망이다.
단백질과의 상호작용
DNA의 모든 기능은 단백질과의 상호작용에 의존한다.이러한 단백질 상호작용은 특정되지 않을 수도 있고, 단백질이 단일 DNA 서열에 특별히 결합될 수도 있다.효소는 또한 DNA와 결합할 수 있으며, 이 중 전사 및 DNA 복제에서 DNA 염기서열을 복사하는 중합체는 특히 중요하다.
DNA 결합 단백질

DNA를 결합하는 구조 단백질은 특정되지 않은 DNA-단백질 상호작용의 잘 알려진 예들이다.염색체 내에서는 DNA가 구조적 단백질을 가진 복합체에서 유지된다.이 단백질들은 DNA를 염색질이라고 불리는 콤팩트한 구조로 구성한다.진핵생물에서, 이 구조는 히스톤이라고 불리는 작은 기본 단백질의 복합체에 DNA 결합을 수반하는 반면, 원핵생물에서는 여러 종류의 단백질이 관여한다.[108][109]히스톤은 뉴클레오솜이라고 불리는 원반 모양의 콤플렉스를 형성하는데, 이 콤플렉스는 표면에 두 개의 이중 가닥 DNA를 완전히 감싼 것을 포함하고 있다.이러한 비특이적 상호작용은 히스톤의 기본적인 잔류물을 통해 형성되어 DNA의 산성 당인산염 등뼈에 이온 결합을 이루며, 따라서 염기서열과는 크게 독립적이다.[110]이러한 기본 아미노산 잔류물의 화학적 수정에는 메틸화, 인산화, 아세틸화가 포함된다.[111]이러한 화학적 변화들은 DNA와 히스톤 사이의 상호작용의 강도를 변화시켜, DNA가 전사 인자에 다소 접근 가능하게 만들고 전사 속도를 변화시킨다.[112]다른 특정되지 않은 염색질의 DNA 결합 단백질은 굽거나 왜곡된 DNA에 결합하는 고이동성 집단 단백질을 포함한다.[113]이 단백질들은 핵소체의 배열을 구부리고 염색체를 구성하는 더 큰 구조로 배열하는데 중요하다.[114]
DNA를 결합하는 단백질의 구별되는 집단은 단일 가닥의 DNA를 특별히 결합하는 DNA 결합 단백질이다.인간에서 복제 단백질 A는 이 계열에서 가장 잘 이해되는 구성원으로 DNA 복제, 재조합, DNA 수리 등 이중나선이 분리되는 과정에 사용된다.[115]이 결합 단백질들은 단일 가닥의 DNA를 안정시키고 줄기-루프가 형성되거나 핵에 의해 분해되는 것을 막아주는 것 같다.
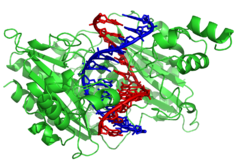
대조적으로, 다른 단백질들은 특정한 DNA 배열에 결합하도록 진화해왔다.이들 중 가장 집중적으로 연구된 것은 다양한 전사인자로, 전사를 조절하는 단백질이다.각 전사 인자는 하나의 특정한 DNA 시퀀스 집합에 결합되어 촉진자와 가까운 이러한 시퀀스를 가진 유전자의 전사를 활성화하거나 억제한다.전사 인자는 두 가지 방법으로 이것을 한다.첫째로, 그들은 직접 또는 다른 중재자 단백질을 통해 전사를 담당하는 RNA 중합효소를 결합시킬 수 있다; 이것은 중합효소를 촉진자에 위치시키고 전사를 시작할 수 있게 한다.[117]대신, 전사 인자는 촉진자의 히스톤을 수정하는 효소를 결합시킬 수 있다.이것은 DNA 템플릿의 중합효소에 대한 접근성을 변화시킨다.[118]
이러한 DNA 표적은 유기체의 게놈 전체에 걸쳐 발생할 수 있기 때문에, 한 종류의 전사 인자의 활동 변화가 수천 개의 유전자에 영향을 미칠 수 있다.[119]결과적으로, 이러한 단백질들은 종종 환경 변화나 세포 분화와 발달에 대한 반응을 조절하는 신호 전달 과정의 대상이 된다.이러한 전사 요인의 DNA와의 상호작용의 특수성은 단백질들이 DNA 염기들의 가장자리와 여러 접점을 만들어 DNA 염기서열을 "읽을" 수 있게 하는 것에서 나온다.이러한 베이스 인터랙션의 대부분은 베이스가 가장 접근하기 쉬운 메인 홈에서 만들어진다.[25]
DNA수정효소
핵 및 묶음
핵은 인산염 결합의 가수분해를 촉진하여 DNA 가닥을 자르는 효소다.DNA 가닥의 끝에서 뉴클레오티드를 가수분해하는 핵들을 엑소뉴클레아제라고 부르는 반면 엔도뉴클레아제는 가닥 안에서 잘라진다.분자생물학에서 가장 많이 사용되는 핵은 제한적 내핵으로, 특정 염기서열에서 DNA를 절단한다.예를 들어 왼쪽에 보이는 EcoRV 효소는 6 염기서열 5′-GATATC-3′을 인식하고 수평선에서 절단한다.자연에서 이러한 효소는 세균이 박테리아 세포에 들어갔을 때 페이지 DNA를 소화시켜 세균을 페이지 감염으로부터 보호하는데, 이는 제한 수정 시스템의 일부로서 작용한다.[121]기술에서, 이러한 시퀀스별 핵들은 분자 복제와 DNA 지문 채취에 사용된다.
DNA 리깅스라고 불리는 효소는 잘리거나 부러진 DNA 가닥에 다시 결합할 수 있다.[122]리가스는 복제 포크에서 생성된 DNA의 짧은 부분을 DNA 템플릿의 완전한 복사본으로 결합하기 때문에, 후행 가닥 DNA 복제에 특히 중요하다.그것들은 또한 DNA 복구와 유전적 재조합에도 사용된다.[122]
토포아세머레이즈 및 헬리코아제
Topoisomerase는 누클리스와 리가아제 활성을 모두 가진 효소다.이 단백질들은 DNA의 슈퍼코일링의 양을 변화시킨다.이 효소들 중 일부는 DNA 나선형을 자르고 한 부분이 회전할 수 있게 함으로써 슈퍼코일링의 수준을 낮추고, 효소는 DNA 파단을 봉인한다.[38]이러한 효소의 다른 종류들은 나선에 다시 합류하기 전에 한 개의 DNA 나선을 자른 다음 두 번째 DNA 가닥을 이 파단 사이로 통과시킬 수 있다.[123]DNA 복제와 전사 등 DNA와 관련된 많은 과정에 토포아세머레이스가 필요하다.[39]
헬리케아스는 분자 모터의 일종인 단백질이다.그들은 아데노신 트리인산염(ATP)이 대부분인 뉴클레오사이드 3인산염의 화학적 에너지를 사용하여 염기 사이의 수소 결합을 깨뜨리고 DNA 이중나선을 한 가닥으로 풀어낸다.[124]이 효소들은 효소가 DNA 베이스에 접근해야 하는 대부분의 과정에 필수적이다.
중합체
폴리머아제는 뉴클레오사이드 3인산염으로부터 폴리뉴클레오티드 체인을 합성하는 효소다.그들의 제품 순서는 기존의 폴리뉴클레오티드 체인을 기반으로 만들어지는데, 이를 템플릿이라고 한다.이러한 효소는 성장하는 폴리뉴클레오티드 체인의 끝에서 3 at 히드록실 그룹에 뉴클레오티드를 반복적으로 첨가하여 기능한다.그 결과, 모든 중합체는 5㎛에서 3㎛ 방향으로 작용한다.[125]이러한 효소의 활성 부위에서, 들어오는 뉴클레오사이드 3인산염 베이스 페어는 템플릿으로: 이것은 중합체가 템플릿의 보완 가닥을 정확하게 합성할 수 있게 한다.중합체는 사용하는 템플릿의 종류에 따라 분류된다.
DNA 복제에서, DNA에 의존하는 DNA 중합체는 DNA 폴리뉴클레오티드 체인의 복사본을 만든다.생물학적 정보를 보존하기 위해서는 각 사본의 염기서열이 템플릿 스트랜드의 염기서열과 정밀하게 상호보완되는 것이 필수적이다.많은 DNA 중합체는 교정 활동을 한다.여기서 중합효소는 일치하지 않는 뉴클레오티드 사이의 염기쌍의 부족으로 인해 합성반응에서 가끔 발생하는 실수를 인식한다.불일치가 감지되면 3 3~5′ exonuclease 활동이 활성화되고 잘못된 base가 제거된다.[126]대부분의 유기체에서, DNA 중합체는 DNA 클램프나 헬리케이드와 같은 여러 부속물 하위 단위를 포함하는 환지체라고 불리는 큰 복합체에서 기능한다.[127]
RNA 의존형 DNA 중합체는 RNA 가닥의 순서를 DNA로 복제하는 전문 중합체종이다.레트로바이러스에 의한 세포의 감염에 관여하는 바이러스 효소인 역성분해효소, 텔로미어 복제에 필요한 텔로머레이즈 등이 그것이다.[56][128]예를 들어, HIV 역분해효소는 에이즈 바이러스 복제를 위한 효소다.[128]텔로머레이즈는 구조물의 일부로 자체 RNA 템플릿을 포함하고 있기 때문에 특이한 중합효소다.염색체 끝에 있는 텔로미어를 합성한다.텔로미어는 인접한 염색체 끝의 융합을 방지하고 염색체 끝을 손상으로부터 보호한다.[57]
전사는 DNA에 의존하는 RNA 중합효소에 의해 수행되며, DNA 가닥의 순서를 RNA로 복사한다.유전자 변형을 시작하기 위해 RNA 중합효소는 촉진제라 불리는 DNA의 순서에 결합하여 DNA 가닥을 분리한다.그런 다음 유전자 서열을 메신저 RNA 대본으로 복사해 종단기라 불리는 DNA 영역에 도달할 때까지 거기서 DNA로부터 정지하고 분리한다.인간의 DNA에 의존하는 DNA 중합효소와 마찬가지로 인간 게놈에서 대부분의 유전자를 변형하는 효소인 RNA 중합효소 II는 여러 가지 규제와 부속품 서브유닛을 가진 큰 단백질 복합체의 일부로 작용한다.[129]
유전적 재조합
DNA 나선은 보통 DNA의 다른 부분과 상호작용하지 않으며, 인간 세포에서는 서로 다른 염색체들이 "염색체 영역"이라고 불리는 핵의 별도 영역을 차지하기도 한다.[131]염색체가 상호작용하는 몇 안 되는 시간 중 하나가 유전적 재조합이 발생할 때 발생하는 염색체 교차점에 있기 때문에, 서로 다른 염색체의 물리적 분리는 DNA가 정보의 안정적인 저장소로 기능하는 데 중요하다.염색체 교차란 두 개의 DNA 나선선이 부러졌을 때 단면을 교환한 다음 다시 결합하는 것이다.
재조합은 염색체가 유전자 정보를 교환할 수 있게 하고 새로운 유전자 조합을 만들어 내는데, 이는 자연선택의 효율성을 높이고 새로운 단백질의 빠른 진화에 중요할 수 있다.[132]유전적 재조합은 DNA 복구에도 관여할 수 있으며, 특히 이중 가닥 파손에 대한 세포의 반응에도 관여할 수 있다.[133]
염색체 교차에서 가장 흔한 형태는 동음이의 재조합인데, 관련된 두 염색체가 매우 유사한 염기서열을 공유한다.비호몰로 재조합은 염색체 변환과 유전적 이상을 일으킬 수 있기 때문에 세포에 손상을 줄 수 있다.재결합 반응은 RAD51과 같이 재조합이라고 알려진 효소에 의해 촉매된다.[134]재조합의 첫 번째 단계는 내과나 DNA 손상에 의해 야기되는 이중 가닥의 단절이다.[135]재조합에 의해 부분적으로 촉매된 일련의 단계는 각 나선의 단일 가닥의 세그먼트가 다른 나선의 보완적 가닥에 맞춰져 있는 적어도 하나의 Hollyday 분기점에 의해 두 개의 나선형 접합으로 이어진다.홀리데이 교차로(Hollyday junction)는 염색체 쌍을 따라 이동할 수 있는 사면 접합 구조로, 한 가닥을 다른 가닥으로 교환한다.그런 다음 접합부의 갈라짐과 방출된 DNA의 재결합에 의해 재결합 반응이 중단된다.[136]같은 극성의 가닥만이 재조합 중에 DNA를 교환한다.갈라짐의 종류는 동서 갈라짐과 남북 갈라짐 두 가지가 있다.남북 갈라짐은 두 가닥의 DNA를 흠집내고, 동서 갈라짐은 한 가닥의 DNA가 온전하게 남아 있다.재조합 시 홀리데이 접합부가 형성되면 유전적 다양성, 염색체에서 유전자가 교환할 수 있으며 야생형 바이러스 게놈의 발현이 가능하다.
진화
DNA에는 모든 형태의 생명체가 기능하고 자라고 번식할 수 있는 유전 정보가 들어 있다.그러나 생명체의 초기 형태들이 RNA를 그들의 유전 물질로 사용했을지도 모른다고 제안되어 왔기 때문에 40억 년의 생명체 DNA가 얼마나 오랫동안 이 기능을 수행해 왔는지는 불분명하다.[137][138]RNA는 유전자 정보를 전달하고 리보핵종의 일부로 촉매작용을 수행할 수 있기 때문에 초기 세포대사의 중심부 역할을 했을 수 있다.[139]핵산이 촉매와 유전학 둘 다에 사용되었을 이 고대 RNA 세계는 네 개의 뉴클레오티드 베이스에 기초한 현재의 유전자 코드의 진화에 영향을 미쳤을지도 모른다.이러한 유기체에서 서로 다른 염기들의 수는 복제 정확도를 높이는 소수의 염기들과 리보자의 촉매 효율을 증가시키는 다수의 염기들 사이의 절충이기 때문에 이러한 현상이 발생할 수 있다.[140]그러나 대부분의 화석으로부터 DNA의 복구가 불가능하기 때문에, DNA가 100만 년도 채 되지 않는 환경 속에서 생존하고, 용해에서 서서히 단편적인 조각으로 분해되기 때문에, 고대 유전체계에 대한 직접적인 증거는 없다.[141]가장 두드러진 것은 2억 5천만 년 된 소금 결정에서 살아있는 박테리아를 분리했다는 보고서인 오래된 DNA에 대한 주장이 제기되었지만,[142] 이러한 주장은 논란이 되고 있다.[143][144]
DNA의 구성 요소(아데닌, 구아닌 및 관련 유기 분자)는 우주 공간에서 치외적으로 형성되었을 수 있다.[145][146][147]우라실, 시토신, 티민 등 생명의 복잡한 DNA와 RNA 유기화합물도 운석에서 발견되는 피리미딘과 같은 시작 화학물질을 사용하여 우주에서 발견되는 것을 모방한 조건에서 실험실에서 형성되어 왔다.피리미딘은 다순환 방향족 탄화수소(PAHs)와 마찬가지로 우주에서 발견되는 탄소가 가장 풍부한 화학 물질로 붉은 거성이나 성간 우주 먼지와 가스 구름에서 형성되었을지도 모른다.[148]
2021년 2월, 과학자들은 처음으로 동물에서 나온 DNA의 염기서열 분석을 보고했는데, 이 경우 매머드는 100만년 이상 된 것으로 현재까지 가장 오래된 DNA 서열이다.[149][150]
기술에서의 사용
유전공학
페놀-클로로폼 추출과 같은 유기체로부터 DNA를 정화하고, 제한소화효소, 중합효소 연쇄반응 등 실험실에서 조작하는 방법이 개발됐다.현대 생물학과 생화학은 DNA 재조합 기술에서 이러한 기법을 집중적으로 이용한다.재조합 DNA는 인간이 만든 DNA 서열로 다른 DNA 서열에서 조립한 것이다.그것들은 바이러스 벡터를 사용하여 플라스미드의 형태나 적절한 형태로 유기체로 변형될 수 있다.[151]유전자 변형 유기체는 재조합 단백질과 같은 제품을 생산하거나 의학 연구에 사용되거나 [152]농업에서 재배될 수 있다.[153][154]
DNA 프로파일링
법의학자들은 범죄현장에서 발견된 혈액, 정액, 피부, 침, 머리카락에 DNA를 사용하여 가해자와 같은 개인의 DNA와 일치하는 것을 확인할 수 있다.[155]이 과정은 공식적으로 DNA 프로파일링이라고 불리며, DNA 지문 채취라고도 불린다.DNA 프로파일링에서, 짧은 탠덤 반복과 미니사이트와 같은 반복 DNA의 가변 섹션의 길이를 사람들 간에 비교한다.이 방법은 일반적으로 일치하는 DNA를 식별하는 매우 신뢰할 수 있는 기술이다.[156]그러나 여러 사람의 DNA로 오염된 장면일 경우 신원 확인이 복잡해질 수 있다.[157]DNA 프로파일링은 1984년 영국의 유전학자 알렉 제프리스 경에 의해 개발되었으며,[158] 1988년 엔더비 살인사건에서 콜린 피치포크를 유죄로 판결하기 위해 법의학에 처음 사용되었다.[159]
법의학의 발달과 혈액, 피부, 침, 머리카락의 미세한 샘플에 대한 유전적 매칭을 얻을 수 있는 능력은 많은 사례들을 재조사하는 것으로 이어졌다.이제 원래 시험 당시 과학적으로 불가능했던 증거가 밝혀질 수 있다.이는 일부 지역에서 이중위험법을 폐지하는 것과 함께, 이전 재판이 배심원들을 납득시킬 충분한 증거를 제시하지 못한 경우 사건을 재개할 수 있다.중범죄로 기소된 사람들은 일치하는 목적을 위해 DNA 샘플을 제공해야 할 것이다.법의학적으로 입수한 DNA 일치에 대한 가장 명백한 방어는 증거의 교차 오염이 발생했다고 주장하는 것이다.이로 인해 중범죄가 새로 발생하는 등 치밀한 처리 절차가 까다로워졌다.
DNA 프로파일링은 또한 가족 구성원과의 매칭을 통해 대규모 사상자,[160] 중대 사고 시 신체 또는 신체 부위, 대량 전쟁 묘지에 있는 개별 희생자를 긍정적으로 식별하는 데 성공적으로 사용된다.
DNA 프로파일링은 또한 DNA 친자확인 검사에서 누군가가 친부모인지 또는 양육 확률을 가진 아이의 조부모인지를 결정하기 위해 사용된다. 이는 일반적으로 부모가 생물학적으로 아이와 관련이 있을 때 99.99%이다.정상적인 DNA 염기서열 분석법은 출생 후 발생하지만 산모가 아직 임신 중인 상태에서 친자확인 검사하는 새로운 방법이 있다.[161]
DNA 효소 또는 촉매 DNA
DNAZymes 또는 촉매 DNA라고도 불리는 디옥시리보이메스는 1994년에 처음 발견되었다.[162]그것들은 대부분 체외 선택 또는 지수 농축에 의한 리간드의 체계적 진화라고 불리는 결합적 접근법을 통해 많은 양의 무작위 DNA 서열에서 격리된 단일 좌초된 DNA 서열이다.DNA자임체는 RNA-DNA 갈라짐, RNA-DNA 레인지, 아미노산 인산화-탈인산화, 탄소-탄소 결합 형성 등을 포함한 다양한 화학반응을 촉진한다.DNA자연은 분석되지 않은 반응에 비해 화학 반응의 촉매 속도를 최대 1억 배까지 높일 수 있다.[163]DNAZymes의 가장 광범위하게 연구된 종류는 다른 금속 이온을 검출하고 치료제를 설계하는데 사용된 RNA-cleaving 유형이다.GR-5 DNAzyme(납부 고유),[162] CA1-3 DNAzymes(코퍼 고유),[164] 39E DNAzyme(우라닐 고유), NaA43 DNAzyme(소듐 고유)를 포함한 여러 금속 특이 DNAzymes가 보고되었다.[165]다른 금속 이온보다 나트륨에 대해 1만배 이상 선택성이 있다고 보고된 NaA43 DNAzyme는 세포 내 실시간 나트륨 센서를 만드는 데 이용됐다.
생물정보학
생물정보학은 DNA 핵산 염기서열 데이터를 포함한 생물학적 데이터를 저장, 데이터 마이닝, 검색 및 조작하는 기법의 개발을 포함한다.이러한 것들은 컴퓨터 과학, 특히 끈 검색 알고리즘, 기계 학습, 데이터베이스 이론에 널리 응용된 진보로 이어졌다.[166]더 큰 문자 순서 안에서 일련의 문자열이 발생하는 것을 찾아내는 문자열 검색이나 일치 알고리즘이 개발되어 뉴클레오티드의 특정 순서를 탐색하였다.[167]DNA 시퀀스는 동질적 시퀀스를 식별하고 구별되는 특정 돌연변이를 찾기 위해 다른 DNA 시퀀스와 정렬될 수 있다.이러한 기술, 특히 다중 시퀀스 정렬은 계통 발생적 관계와 단백질 기능을 연구하는 데 사용된다.[168]인간 게놈 프로젝트에 의해 생산된 것과 같은 게놈의 전체 DNA 염기서열을 나타내는 데이터 세트는 각 염색체에서 유전자의 위치와 규제 요소의 위치를 식별하는 주석이 없으면 사용하기 어렵다.단백질이나 RNA 코딩 유전자와 관련된 특성 패턴을 가진 DNA 염기서열의 영역은 유전자 발견 알고리즘으로 식별할 수 있으며, 이를 통해 연구자들은 실험적으로 격리되기도 전에 특정 유전자 생산물의 존재와 유기체 내에서의 가능한 기능을 예측할 수 있다.[169]또한 전체 게놈을 비교할 수도 있는데, 이는 특정 생물의 진화 역사를 조명할 수 있고 복잡한 진화 사건의 조사를 허용할 수 있다.
DNA 나노기술
DNA 나노기술은 DNA와 다른 핵산의 고유한 분자 인식 특성을 이용하여 유용한 성질을 가진 자가조립 브랜딩 DNA 콤플렉스를 만든다.[171]따라서 DNA는 생물학적 정보의 매개체로서가 아니라 구조적인 물질로 사용된다.이로 인해 2차원 주기 격자(타일 기반 및 DNA 종이접기 방법 사용)와 다면체 모양의 3차원 구조물이 생성되었다.[172]나노기계장치와 알고리즘 자가조립도 입증되었으며,[173] 이러한 DNA 구조는 금 나노입자와 스트렙타비딘 단백질과 같은 다른 분자의 배열을 형상화하는 데 사용되었다.[174]
역사와 인류학
DNA는 시간이 지남에 따라 유전되는 돌연변이를 수집하기 때문에 역사적 정보를 담고 있으며, DNA 서열을 비교함으로써 유전학자들이 유기체의 진화적 역사, 그들의 계통생성을 유추할 수 있다.[175]이 유전학 분야는 진화 생물학에서 강력한 도구다.한 종 내의 DNA 서열을 비교한다면, 인구 유전학자는 특정 집단의 역사를 배울 수 있다.이것은 생태유전학에서 인류학에 이르는 연구에 사용될 수 있다.
정보창고
정보 저장장치로서의 DNA는 전자장치에 비해 저장밀도가 훨씬 높아 잠재력이 엄청나다.그러나 높은 비용, 느린 읽기 및 쓰기 시간(메모리 지연 시간), 불충분한 신뢰성으로 인해 실용화가 불가능해졌다.[176][177]
역사
DNA는 1869년 버려진 외과용 붕대의 고름에서 미세한 물질을 발견한 스위스 의사 프리드리히 미에셔에 의해 처음 격리되었다.그것이 세포의 핵 속에 살고 있었기 때문에, 그는 그것을 "뉴클레인"[178][179]이라고 불렀다.1878년 알브레히트 코셀은 "핵산"의 비단백질 성분을 분리시켰고, 이후 5개의 1차 핵물질을 분리시켰다.[180][181]
1909년, Phoebus Levene은 RNA의 염기, 설탕, 인산염 뉴클레오티드 단위를 식별했다(그 후 "야스트 핵산"으로 명명됨).[182][183][184]1929년에 레베네는 "티머스 핵산"(DNA)에서 디옥시리보오스 당분을 확인했다.[185]레베네는 DNA가 인산염 그룹("테트라뉴클레오티드 가설")을 통해 서로 연결된 네 개의 뉴클레오티드 단위의 끈으로 구성되어 있다고 제안했다.레베네는 체인이 짧고 베이스가 일정한 순서로 반복된다고 생각했다.1927년 니콜라이 콜트소프는 "각 가닥을 템플릿으로 삼아 반보수적으로 복제할 수 있는 두 개의 거울 가닥"으로 구성된 "거대한 유전분자"를 통해 유전 형질을 계승할 것을 제안했다.[186][187]1928년 프레드릭 그리피스는 실험에서 폐렴구균의 "매끄러운" 형태의 특성이 죽은 "매끄러운" 박테리아와 살아있는 "매끄러운" 박테리아를 혼합함으로써 같은 박테리아의 "매끄러운" 형태로 전이될 수 있다는 것을 발견했다.[188][189]이 시스템은 DNA가 유전 정보를 운반한다는 첫 번째 분명한 암시를 제공했다.
1933년, 처녀 성게 난자를 연구하던 중 장 브라체트는 세포핵에서 DNA가 발견되고 RNA가 세포질에만 존재한다고 제안했다.당시 '여스트 핵산'(RNA)은 식물에서만, '시머스 핵산'(DNA)은 동물에서만 발생하는 것으로 생각되었다.후자는 세포 pH를 버퍼링하는 기능을 가진 테트라머로 생각되었다.[190][191]
1937년 윌리엄 애스트베리는 DNA가 규칙적인 구조를 가지고 있다는 것을 보여주는 최초의 X선 회절 패턴을 만들어냈다.[192]
1943년 오스왈드 에이버리는 동료인 콜린 맥리어드, 매클린 맥카티와 함께 DNA를 변환원리로 확인하여 그리피스의 제안(Avery–MacLeod-Mcarty 실험)[193]을 지지하였다.에르윈 샤르가프는 현재 샤르가프의 법칙으로 알려진 관찰을 개발하여 발표했는데, 어떤 유기체의 어떤 종에서든 DNA에서 구아닌의 양은 시토신과 같아야 하고 아데닌의 양은 티민과 같아야 한다고 명시하고 있다.[194][195]1951년 말, 프랜시스 크릭은 케임브리지 대학의 캐번디쉬 연구소에서 제임스 왓슨과 함께 일하기 시작했다.유전에서 DNA의 역할은 1952년 허쉬-체이스 실험에서 알프레드 허쉬와 마사 체이스가 DNA가 장내박테리아 페이지의 유전 물질이라는 것을 보여주면서 확인되었다.[196]
1952년 5월 로잘린드 프랭클린의 감독 아래 일하는 대학원생 레이먼드 고슬링은 DNA의 높은 수화 수준에서 '사진 51'[197]이라는 라벨이 붙은 X선 회절 사진을 찍었다.이 사진은 Maurice Wilkins에 의해 Watson과 Crick에게 주어졌고 그들이 DNA의 정확한 구조를 얻는 데 결정적인 역할을 했다.프랭클린은 크릭과 왓슨에게 등뼈가 바깥쪽에 있어야 한다고 말했다.그 전에, 리너스 폴링과 왓슨과 크릭은 체인이 안쪽에 있고 베이스가 바깥쪽을 가리키는 잘못된 모델을 가지고 있었다.프랭클린이 DNA 결정체를 위해 우주군을 확인한 결과 크릭에게 두 개의 DNA 가닥이 대위선이라는 것이 밝혀졌다.[198]
1953년 2월, 리너스 폴링과 로버트 코레이는 세 개의 서로 얽힌 사슬을 포함하고 있는 핵산 모델을 제안했는데, 축 근처에는 인산염, 바깥쪽에는 베이스가 있다.[199]왓슨과 크릭은 그들의 모델을 완성했는데, 이것은 현재 DNA의 이중나선의 첫 번째 정확한 모델로 받아들여지고 있다.1953년 2월 28일 크릭은 자신과 왓슨이 "삶의 비밀을 발견했다"[200]고 발표하기 위해 케임브리지의 이글 펍에서 고객들의 점심시간을 방해했다.
1953년 4월 25일자 네이처지는 왓슨과 크릭의 이중 헥스 구조 DNA와 이를 뒷받침하는 증거를 제시하는 5개 기사를 연재했다.[201]이 구조물은 '핵산의 분자구조 A DECTOR OF NEFIC A Structure OF DE옥시리보스 핵산을 위한 구조'라는 제목의 서한에서 보고되었는데, 그들은 "우리가 즉시 상정해 놓은 특정 쌍이 유전 물질에 대한 가능한 복사 메커니즘을 시사한다는 것은 우리의 주목을 벗어나지 못했다"[9]고 말했다.이 편지에 이어 프랭클린과 고슬링의 편지가 왔는데, 이 편지는 그들 자신의 X선 회절 데이터와 원래의 분석 방법의 첫 번째 간행물이었다.[42][202]그 후 윌킨스와 그의 동료 두 명이 보낸 편지에 이어, 이 편지에 생체내 B-DNA X선 패턴의 분석이 수록되어 있으며, 왓슨과 크릭 구조의 생체내 존재를 뒷받침하고 있다.[43]
프랭클린이 죽은 후인 1962년 왓슨, 크릭, 윌킨스는 공동으로 노벨 생리의학상을 받았다.[203]노벨상은 살아있는 수상자에게만 수여된다.이 발견에 대한 공로를 누가 받아야 하는지에 대한 논쟁이 계속되고 있다.[204]
1957년 영향력 있는 발표에서 크릭은 분자생물학의 중심 도그마를 내놓았는데, 이 도그마는 DNA, RNA, 단백질의 관계를 예언하고, '어댑터 가설'[205]을 명확히 했다.1958년 메셀슨-스탈 실험을 통해 이중헬릭 구조가 암시했던 복제 메커니즘의 최종 확인.[206]크릭과 동료들의 추가 연구는 유전자 코드가 코돈이라고 불리는 과대평가되지 않은 세 쌍의 염기들에 기초하고 있다는 것을 보여주었고, 하 고빈드 코라나, 로버트 W. 홀리와 [207]마샬 워렌 니렌버그가 유전 암호를 해독할 거야이 발견들은 분자생물학의 탄생을 나타낸다.[208]
참고 항목
- Autosome – 성별 염색체를 제외한 모든 염색체
- 결정학 – 결정 구조에 대한 과학적 연구
- DNA 데이 – 4월 25일 기념일
- DNA 마이크로 어레이 – 단단한 표면에 부착된 미세한 DNA 점의 집합
- DNA 염기서열 분석 – DNA 분자 내 뉴클레오티드의 순서를 결정하는 과정
- 유전적 장애 – 게놈의 하나 이상의 이상으로 인한 건강 문제
- 유전적 계보 – DNA 검사를 전통적인 계보 방법과 결합하여 개인 간의 관계를 유추하고 조상을 찾는 방법
- Happlotype – 한 부모로부터 온 유전자 그룹
- 감수분열 – 생식체를 생산하는 데 사용되는 성재생산 유기체의 세포분열 유형
- 핵산 표기법 – 4개의 DNA 뉴클레오티드를 호출하기 위해 로마자 A, C, G, T를 사용하는 보편적 표기법
- 핵산 순서 – 핵산 내 뉴클레오티드의 계승
- 리보솜 DNA
- Southern Blot – DNA 분석 기법
- X선 산란 기법
- 제노 핵산
참조
- ^ "deoxyribonucleic acid". Merriam-Webster Dictionary.
- ^ Alberts B, Johnson A, Lewis J, Raff M, Roberts K, Walter P (2014). Molecular Biology of the Cell (6th ed.). Garland. p. Chapter 4: DNA, Chromosomes and Genomes. ISBN 978-0-8153-4432-2. Archived from the original on 14 July 2014.
- ^ Purcell A. "DNA". Basic Biology. Archived from the original on 5 January 2017.
- ^ "Uracil". Genome.gov. Retrieved 21 November 2019.
- ^ Russell P (2001). iGenetics. New York: Benjamin Cummings. ISBN 0-8053-4553-1.
- ^ Saenger W (1984). Principles of Nucleic Acid Structure. New York: Springer-Verlag. ISBN 0-387-90762-9.
- ^ a b Alberts B, Johnson A, Lewis J, Raff M, Roberts K, Peter W (2002). Molecular Biology of the Cell (Fourth ed.). New York and London: Garland Science. ISBN 0-8153-3218-1. OCLC 145080076. Archived from the original on 1 November 2016.
- ^ Irobalieva RN, Fogg JM, Catanese DJ, Catanese DJ, Sutthibutpong T, Chen M, Barker AK, Ludtke SJ, Harris SA, Schmid MF, Chiu W, Zechiedrich L (October 2015). "Structural diversity of supercoiled DNA". Nature Communications. 6: 8440. Bibcode:2015NatCo...6.8440I. doi:10.1038/ncomms9440. ISSN 2041-1723. PMC 4608029. PMID 26455586.
- ^ a b c d Watson JD, Crick FH (April 1953). "Molecular structure of nucleic acids; a structure for deoxyribose nucleic acid" (PDF). Nature. 171 (4356): 737–38. Bibcode:1953Natur.171..737W. doi:10.1038/171737a0. ISSN 0028-0836. PMID 13054692. S2CID 4253007. Archived (PDF) from the original on 4 February 2007.
- ^ Mandelkern M, Elias JG, Eden D, Crothers DM (October 1981). "The dimensions of DNA in solution". Journal of Molecular Biology. 152 (1): 153–61. doi:10.1016/0022-2836(81)90099-1. ISSN 0022-2836. PMID 7338906.
- ^ Gregory SG, Barlow KF, McLay KE, Kaul R, Swarbreck D, Dunham A, et al. (May 2006). "The DNA sequence and biological annotation of human chromosome 1". Nature. 441 (7091): 315–21. Bibcode:2006Natur.441..315G. doi:10.1038/nature04727. PMID 16710414.
- ^ a b c Berg J, Tymoczko J, Stryer L (2002). Biochemistry. W.H. Freeman and Company. ISBN 0-7167-4955-6.
- ^ IUPAC-IUB Commission on Biochemical Nomenclature (CBN) (December 1970). "Abbreviations and Symbols for Nucleic Acids, Polynucleotides and their Constituents. Recommendations 1970". The Biochemical Journal. 120 (3): 449–54. doi:10.1042/bj1200449. ISSN 0306-3283. PMC 1179624. PMID 5499957. Archived from the original on 5 February 2007.
- ^ a b Ghosh A, Bansal M (April 2003). "A glossary of DNA structures from A to Z". Acta Crystallographica Section D. 59 (Pt 4): 620–26. doi:10.1107/S0907444903003251. ISSN 0907-4449. PMID 12657780.
- ^ PDB 1D65에서 생성됨
- ^ Yakovchuk P, Protozanova E, Frank-Kamenetskii MD (2006). "Base-stacking and base-pairing contributions into thermal stability of the DNA double helix". Nucleic Acids Research. 34 (2): 564–74. doi:10.1093/nar/gkj454. ISSN 0305-1048. PMC 1360284. PMID 16449200.
- ^ Tropp BE (2012). Molecular Biology (4th ed.). Sudbury, Mass.: Jones and Barlett Learning. ISBN 978-0-7637-8663-2.
- ^ Carr S (1953). "Watson-Crick Structure of DNA". Memorial University of Newfoundland. Archived from the original on 19 July 2016. Retrieved 13 July 2016.
- ^ Verma S, Eckstein F (1998). "Modified oligonucleotides: synthesis and strategy for users". Annual Review of Biochemistry. 67: 99–134. doi:10.1146/annurev.biochem.67.1.99. ISSN 0066-4154. PMID 9759484.
- ^ Johnson TB, Coghill RD (1925). "Pyrimidines. CIII. The discovery of 5-methylcytosine in tuberculinic acid, the nucleic acid of the tubercle bacillus". Journal of the American Chemical Society. 47: 2838–44. doi:10.1021/ja01688a030. ISSN 0002-7863.
- ^ Weigele P, Raleigh EA (October 2016). "Biosynthesis and Function of Modified Bases in Bacteria and Their Viruses". Chemical Reviews. 116 (20): 12655–12687. doi:10.1021/acs.chemrev.6b00114. ISSN 0009-2665. PMID 27319741.
- ^ Kumar S, Chinnusamy V, Mohapatra T (2018). "Epigenetics of Modified DNA Bases: 5-Methylcytosine and Beyond". Frontiers in Genetics. 9: 640. doi:10.3389/fgene.2018.00640. ISSN 1664-8021. PMC 6305559. PMID 30619465.
- ^ Carell T, Kurz MQ, Müller M, Rossa M, Spada F (April 2018). "Non-canonical Bases in the Genome: The Regulatory Information Layer in DNA". Angewandte Chemie. 57 (16): 4296–4312. doi:10.1002/anie.201708228. PMID 28941008.
- ^ Wing R, Drew H, Takano T, Broka C, Tanaka S, Itakura K, Dickerson RE (October 1980). "Crystal structure analysis of a complete turn of B-DNA". Nature. 287 (5784): 755–58. Bibcode:1980Natur.287..755W. doi:10.1038/287755a0. PMID 7432492. S2CID 4315465.
- ^ a b Pabo CO, Sauer RT (1984). "Protein-DNA recognition". Annual Review of Biochemistry. 53: 293–321. doi:10.1146/annurev.bi.53.070184.001453. PMID 6236744.
- ^ Nikolova EN, Zhou H, Gottardo FL, Alvey HS, Kimsey IJ, Al-Hashimi HM (2013). "A historical account of Hoogsteen base-pairs in duplex DNA". Biopolymers. 99 (12): 955–68. doi:10.1002/bip.22334. PMC 3844552. PMID 23818176.
- ^ Clausen-Schaumann H, Rief M, Tolksdorf C, Gaub HE (April 2000). "Mechanical stability of single DNA molecules". Biophysical Journal. 78 (4): 1997–2007. Bibcode:2000BpJ....78.1997C. doi:10.1016/S0006-3495(00)76747-6. PMC 1300792. PMID 10733978.
- ^ Chalikian TV, Völker J, Plum GE, Breslauer KJ (July 1999). "A more unified picture for the thermodynamics of nucleic acid duplex melting: a characterization by calorimetric and volumetric techniques". Proceedings of the National Academy of Sciences of the United States of America. 96 (14): 7853–58. Bibcode:1999PNAS...96.7853C. doi:10.1073/pnas.96.14.7853. PMC 22151. PMID 10393911.
- ^ deHaseth PL, Helmann JD (June 1995). "Open complex formation by Escherichia coli RNA polymerase: the mechanism of polymerase-induced strand separation of double helical DNA". Molecular Microbiology. 16 (5): 817–24. doi:10.1111/j.1365-2958.1995.tb02309.x. PMID 7476180. S2CID 24479358.
- ^ Isaksson J, Acharya S, Barman J, Cheruku P, Chattopadhyaya J (December 2004). "Single-stranded adenine-rich DNA and RNA retain structural characteristics of their respective double-stranded conformations and show directional differences in stacking pattern" (PDF). Biochemistry. 43 (51): 15996–6010. doi:10.1021/bi048221v. PMID 15609994. Archived (PDF) from the original on 10 June 2007.
- ^ Wayback Machine JCBN/NC-IUB 뉴스레터 1989에 보관된 두 가닥의 DNA 지정.2008년 5월 7일 검색됨
- ^ Hüttenhofer A, Schattner P, Polacek N (May 2005). "Non-coding RNAs: hope or hype?". Trends in Genetics. 21 (5): 289–97. doi:10.1016/j.tig.2005.03.007. PMID 15851066.
- ^ Munroe SH (November 2004). "Diversity of antisense regulation in eukaryotes: multiple mechanisms, emerging patterns". Journal of Cellular Biochemistry. 93 (4): 664–71. doi:10.1002/jcb.20252. PMID 15389973. S2CID 23748148.
- ^ Makalowska I, Lin CF, Makalowski W (February 2005). "Overlapping genes in vertebrate genomes". Computational Biology and Chemistry. 29 (1): 1–12. doi:10.1016/j.compbiolchem.2004.12.006. PMID 15680581.
- ^ Johnson ZI, Chisholm SW (November 2004). "Properties of overlapping genes are conserved across microbial genomes". Genome Research. 14 (11): 2268–72. doi:10.1101/gr.2433104. PMC 525685. PMID 15520290.
- ^ Lamb RA, Horvath CM (August 1991). "Diversity of coding strategies in influenza viruses". Trends in Genetics. 7 (8): 261–66. doi:10.1016/0168-9525(91)90326-L. PMC 7173306. PMID 1771674.
- ^ Benham CJ, Mielke SP (2005). "DNA mechanics" (PDF). Annual Review of Biomedical Engineering. 7: 21–53. doi:10.1146/annurev.bioeng.6.062403.132016. PMID 16004565. S2CID 1427671. Archived from the original (PDF) on 1 March 2019.
- ^ a b Champoux JJ (2001). "DNA topoisomerases: structure, function, and mechanism" (PDF). Annual Review of Biochemistry. 70: 369–413. doi:10.1146/annurev.biochem.70.1.369. PMID 11395412. S2CID 18144189.
- ^ a b Wang JC (June 2002). "Cellular roles of DNA topoisomerases: a molecular perspective". Nature Reviews Molecular Cell Biology. 3 (6): 430–40. doi:10.1038/nrm831. PMID 12042765. S2CID 205496065.
- ^ Basu HS, Feuerstein BG, Zarling DA, Shafer RH, Marton LJ (October 1988). "Recognition of Z-RNA and Z-DNA determinants by polyamines in solution: experimental and theoretical studies". Journal of Biomolecular Structure & Dynamics. 6 (2): 299–309. doi:10.1080/07391102.1988.10507714. PMID 2482766.
- ^ Franklin RE, Gosling RG (6 March 1953). "The Structure of Sodium Thymonucleate Fibres I. The Influence of Water Content" (PDF). Acta Crystallogr. 6 (8–9): 673–77. doi:10.1107/S0365110X53001939. Archived (PDF) from the original on 9 January 2016.
Franklin RE, Gosling RG (1953). "The structure of sodium thymonucleate fibres. II. The cylindrically symmetrical Patterson function" (PDF). Acta Crystallogr. 6 (8–9): 678–85. doi:10.1107/S0365110X53001940. - ^ a b Franklin RE, Gosling RG (April 1953). "Molecular configuration in sodium thymonucleate" (PDF). Nature. 171 (4356): 740–41. Bibcode:1953Natur.171..740F. doi:10.1038/171740a0. PMID 13054694. S2CID 4268222. Archived (PDF) from the original on 3 January 2011.
- ^ a b Wilkins MH, Stokes AR, Wilson HR (April 1953). "Molecular structure of deoxypentose nucleic acids" (PDF). Nature. 171 (4356): 738–40. Bibcode:1953Natur.171..738W. doi:10.1038/171738a0. PMID 13054693. S2CID 4280080. Archived (PDF) from the original on 13 May 2011.
- ^ Leslie AG, Arnott S, Chandrasekaran R, Ratliff RL (October 1980). "Polymorphism of DNA double helices". Journal of Molecular Biology. 143 (1): 49–72. doi:10.1016/0022-2836(80)90124-2. PMID 7441761.
- ^ Baianu IC (1980). "Structural Order and Partial Disorder in Biological systems". Bull. Math. Biol. 42 (4): 137–41. doi:10.1007/BF02462372. S2CID 189888972.
- ^ Hosemann R, Bagchi RN (1962). Direct analysis of diffraction by matter. Amsterdam – New York: North-Holland Publishers.
- ^ Baianu IC (1978). "X-ray scattering by partially disordered membrane systems" (PDF). Acta Crystallogr A. 34 (5): 751–53. Bibcode:1978AcCrA..34..751B. doi:10.1107/S0567739478001540.
- ^ Wahl MC, Sundaralingam M (1997). "Crystal structures of A-DNA duplexes". Biopolymers. 44 (1): 45–63. doi:10.1002/(SICI)1097-0282(1997)44:1<45::AID-BIP4>3.0.CO;2-#. PMID 9097733.
- ^ Lu XJ, Shakked Z, Olson WK (July 2000). "A-form conformational motifs in ligand-bound DNA structures". Journal of Molecular Biology. 300 (4): 819–40. doi:10.1006/jmbi.2000.3690. PMID 10891271.
- ^ Rothenburg S, Koch-Nolte F, Haag F (December 2001). "DNA methylation and Z-DNA formation as mediators of quantitative differences in the expression of alleles". Immunological Reviews. 184: 286–98. doi:10.1034/j.1600-065x.2001.1840125.x. PMID 12086319. S2CID 20589136.
- ^ Oh DB, Kim YG, Rich A (December 2002). "Z-DNA-binding proteins can act as potent effectors of gene expression in vivo". Proceedings of the National Academy of Sciences of the United States of America. 99 (26): 16666–71. Bibcode:2002PNAS...9916666O. doi:10.1073/pnas.262672699. PMC 139201. PMID 12486233.
- ^ Palmer J (2 December 2010). "Arsenic-loving bacteria may help in hunt for alien life". BBC News. Archived from the original on 3 December 2010. Retrieved 2 December 2010.
- ^ a b Bortman H (2 December 2010). "Arsenic-Eating Bacteria Opens New Possibilities for Alien Life". Space.com. Archived from the original on 4 December 2010. Retrieved 2 December 2010.
- ^ Katsnelson A (2 December 2010). "Arsenic-eating microbe may redefine chemistry of life". Nature News. doi:10.1038/news.2010.645. Archived from the original on 12 February 2012.
- ^ Cressey D (3 October 2012). "'Arsenic-life' Bacterium Prefers Phosphorus after all". Nature News. doi:10.1038/nature.2012.11520. S2CID 87341731.
- ^ a b Greider CW, Blackburn EH (December 1985). "Identification of a specific telomere terminal transferase activity in Tetrahymena extracts". Cell. 43 (2 Pt 1): 405–13. doi:10.1016/0092-8674(85)90170-9. PMID 3907856.
- ^ a b c Nugent CI, Lundblad V (April 1998). "The telomerase reverse transcriptase: components and regulation". Genes & Development. 12 (8): 1073–85. doi:10.1101/gad.12.8.1073. PMID 9553037.
- ^ Wright WE, Tesmer VM, Huffman KE, Levene SD, Shay JW (November 1997). "Normal human chromosomes have long G-rich telomeric overhangs at one end". Genes & Development. 11 (21): 2801–09. doi:10.1101/gad.11.21.2801. PMC 316649. PMID 9353250.
- ^ 2016년 10월 17일 웨이백 머신에 보관된 제품에서 생성
- ^ a b Burge S, Parkinson GN, Hazel P, Todd AK, Neidle S (2006). "Quadruplex DNA: sequence, topology and structure". Nucleic Acids Research. 34 (19): 5402–15. doi:10.1093/nar/gkl655. PMC 1636468. PMID 17012276.
- ^ Parkinson GN, Lee MP, Neidle S (June 2002). "Crystal structure of parallel quadruplexes from human telomeric DNA". Nature. 417 (6891): 876–80. Bibcode:2002Natur.417..876P. doi:10.1038/nature755. PMID 12050675. S2CID 4422211.
- ^ Griffith JD, Comeau L, Rosenfield S, Stansel RM, Bianchi A, Moss H, de Lange T (May 1999). "Mammalian telomeres end in a large duplex loop". Cell. 97 (4): 503–14. CiteSeerX 10.1.1.335.2649. doi:10.1016/S0092-8674(00)80760-6. PMID 10338214. S2CID 721901.
- ^ Seeman NC (November 2005). "DNA enables nanoscale control of the structure of matter". Quarterly Reviews of Biophysics. 38 (4): 363–71. doi:10.1017/S0033583505004087. PMC 3478329. PMID 16515737.
- ^ Warren M (21 February 2019). "Four new DNA letters double life's alphabet". Nature. 566 (7745): 436. Bibcode:2019Natur.566..436W. doi:10.1038/d41586-019-00650-8. PMID 30809059.
- ^ Hoshika S, Leal NA, Kim MJ, Kim MS, Karalkar NB, Kim HJ, et al. (22 February 2019). "Hachimoji DNA and RNA: A genetic system with eight building blocks (paywall)". Science. 363 (6429): 884–887. Bibcode:2019Sci...363..884H. doi:10.1126/science.aat0971. PMC 6413494. PMID 30792304.
- ^ Burghardt B, Hartmann AK (February 2007). "RNA secondary structure design". Physical Review E. 75 (2): 021920. arXiv:physics/0609135. Bibcode:2007PhRvE..75b1920B. doi:10.1103/PhysRevE.75.021920. PMID 17358380. S2CID 17574854.
- ^ Hu Q, Rosenfeld MG (2012). "Epigenetic regulation of human embryonic stem cells". Frontiers in Genetics. 3: 238. doi:10.3389/fgene.2012.00238. PMC 3488762. PMID 23133442.
- ^ Klose RJ, Bird AP (February 2006). "Genomic DNA methylation: the mark and its mediators". Trends in Biochemical Sciences. 31 (2): 89–97. doi:10.1016/j.tibs.2005.12.008. PMID 16403636.
- ^ Bird A (January 2002). "DNA methylation patterns and epigenetic memory". Genes & Development. 16 (1): 6–21. doi:10.1101/gad.947102. PMID 11782440.
- ^ Walsh CP, Xu GL (2006). "Cytosine methylation and DNA repair". Current Topics in Microbiology and Immunology. 301: 283–315. doi:10.1007/3-540-31390-7_11. ISBN 3-540-29114-8. PMID 16570853.
- ^ Kriaucionis S, Heintz N (May 2009). "The nuclear DNA base 5-hydroxymethylcytosine is present in Purkinje neurons and the brain". Science. 324 (5929): 929–30. Bibcode:2009Sci...324..929K. doi:10.1126/science.1169786. PMC 3263819. PMID 19372393.
- ^ Ratel D, Ravanat JL, Berger F, Wion D (March 2006). "N6-methyladenine: the other methylated base of DNA". BioEssays. 28 (3): 309–15. doi:10.1002/bies.20342. PMC 2754416. PMID 16479578.
- ^ Gommers-Ampt JH, Van Leeuwen F, de Beer AL, Vliegenthart JF, Dizdaroglu M, Kowalak JA, Crain PF, Borst P (December 1993). "beta-D-glucosyl-hydroxymethyluracil: a novel modified base present in the DNA of the parasitic protozoan T. brucei". Cell. 75 (6): 1129–36. doi:10.1016/0092-8674(93)90322-H. hdl:1874/5219. PMID 8261512. S2CID 24801094.
- ^ 2008년 9월 22일 웨이백 시스템에 보관된 PDB 1JDG에서 생성
- ^ Douki T, Reynaud-Angelin A, Cadet J, Sage E (August 2003). "Bipyrimidine photoproducts rather than oxidative lesions are the main type of DNA damage involved in the genotoxic effect of solar UVA radiation". Biochemistry. 42 (30): 9221–26. doi:10.1021/bi034593c. PMID 12885257.
- ^ Cadet J, Delatour T, Douki T, Gasparutto D, Pouget JP, Ravanat JL, Sauvaigo S (March 1999). "Hydroxyl radicals and DNA base damage". Mutation Research. 424 (1–2): 9–21. doi:10.1016/S0027-5107(99)00004-4. PMID 10064846.
- ^ Beckman KB, Ames BN (August 1997). "Oxidative decay of DNA". The Journal of Biological Chemistry. 272 (32): 19633–36. doi:10.1074/jbc.272.32.19633. PMID 9289489.
- ^ Valerie K, Povirk LF (September 2003). "Regulation and mechanisms of mammalian double-strand break repair". Oncogene. 22 (37): 5792–812. doi:10.1038/sj.onc.1206679. PMID 12947387.
- ^ Johnson G (28 December 2010). "Unearthing Prehistoric Tumors, and Debate". The New York Times. Archived from the original on 24 June 2017.
If we lived long enough, sooner or later we all would get cancer.
- ^ Alberts B, Johnson A, Lewis J, et al. (2002). "The Preventable Causes of Cancer". Molecular biology of the cell (4th ed.). New York: Garland Science. ISBN 0-8153-4072-9. Archived from the original on 2 January 2016.
A certain irreducible background incidence of cancer is to be expected regardless of circumstances: mutations can never be absolutely avoided, because they are an inescapable consequence of fundamental limitations on the accuracy of DNA replication, as discussed in Chapter 5. If a human could live long enough, it is inevitable that at least one of his or her cells would eventually accumulate a set of mutations sufficient for cancer to develop.
- ^ Bernstein H, Payne CM, Bernstein C, Garewal H, Dvorak K (2008). "Cancer and aging as consequences of un-repaired DNA damage". In Kimura H, Suzuki A (eds.). New Research on DNA Damage. New York: Nova Science Publishers. pp. 1–47. ISBN 978-1-60456-581-2. Archived from the original on 25 October 2014.
- ^ Hoeijmakers JH (October 2009). "DNA damage, aging, and cancer". The New England Journal of Medicine. 361 (15): 1475–85. doi:10.1056/NEJMra0804615. PMID 19812404.
- ^ Freitas AA, de Magalhães JP (2011). "A review and appraisal of the DNA damage theory of ageing". Mutation Research. 728 (1–2): 12–22. doi:10.1016/j.mrrev.2011.05.001. PMID 21600302.
- ^ Ferguson LR, Denny WA (September 1991). "The genetic toxicology of acridines". Mutation Research. 258 (2): 123–60. doi:10.1016/0165-1110(91)90006-H. PMID 1881402.
- ^ Stephens TD, Bunde CJ, Fillmore BJ (June 2000). "Mechanism of action in thalidomide teratogenesis". Biochemical Pharmacology. 59 (12): 1489–99. doi:10.1016/S0006-2952(99)00388-3. PMID 10799645.
- ^ Jeffrey AM (1985). "DNA modification by chemical carcinogens". Pharmacology & Therapeutics. 28 (2): 237–72. doi:10.1016/0163-7258(85)90013-0. PMID 3936066.
- ^ Braña MF, Cacho M, Gradillas A, de Pascual-Teresa B, Ramos A (November 2001). "Intercalators as anticancer drugs". Current Pharmaceutical Design. 7 (17): 1745–80. doi:10.2174/1381612013397113. PMID 11562309.
- ^ Venter JC, Adams MD, Myers EW, Li PW, Mural RJ, Sutton GG, et al. (February 2001). "The sequence of the human genome". Science. 291 (5507): 1304–51. Bibcode:2001Sci...291.1304V. doi:10.1126/science.1058040. PMID 11181995.
- ^ Thanbichler M, Wang SC, Shapiro L (October 2005). "The bacterial nucleoid: a highly organized and dynamic structure". Journal of Cellular Biochemistry. 96 (3): 506–21. doi:10.1002/jcb.20519. PMID 15988757.
- ^ Wolfsberg TG, McEntyre J, Schuler GD (February 2001). "Guide to the draft human genome". Nature. 409 (6822): 824–26. Bibcode:2001Natur.409..824W. doi:10.1038/35057000. PMID 11236998.
- ^ Gregory TR (January 2005). "The C-value enigma in plants and animals: a review of parallels and an appeal for partnership". Annals of Botany. 95 (1): 133–46. doi:10.1093/aob/mci009. PMC 4246714. PMID 15596463.
- ^ Birney E, Stamatoyannopoulos JA, Dutta A, Guigó R, Gingeras TR, Margulies EH, et al. (June 2007). "Identification and analysis of functional elements in 1% of the human genome by the ENCODE pilot project". Nature. 447 (7146): 799–816. Bibcode:2007Natur.447..799B. doi:10.1038/nature05874. PMC 2212820. PMID 17571346.
- ^ PDB 1MSW 2008년 1월 6일 웨이백 시스템에 아카이브된 PDB에서 생성
- ^ Pidoux AL, Allshire RC (March 2005). "The role of heterochromatin in centromere function". Philosophical Transactions of the Royal Society of London. Series B, Biological Sciences. 360 (1455): 569–79. doi:10.1098/rstb.2004.1611. PMC 1569473. PMID 15905142.
- ^ Harrison PM, Hegyi H, Balasubramanian S, Luscombe NM, Bertone P, Echols N, Johnson T, Gerstein M (February 2002). "Molecular fossils in the human genome: identification and analysis of the pseudogenes in chromosomes 21 and 22". Genome Research. 12 (2): 272–80. doi:10.1101/gr.207102. PMC 155275. PMID 11827946.
- ^ Harrison PM, Gerstein M (May 2002). "Studying genomes through the aeons: protein families, pseudogenes and proteome evolution". Journal of Molecular Biology. 318 (5): 1155–74. doi:10.1016/S0022-2836(02)00109-2. PMID 12083509.
- ^ Albà M (2001). "Replicative DNA polymerases". Genome Biology. 2 (1): REVIEWS3002. doi:10.1186/gb-2001-2-1-reviews3002. PMC 150442. PMID 11178285.
- ^ Tani K, Nasu M (2010). "Roles of Extracellular DNA in Bacterial Ecosystems". In Kikuchi Y, Rykova EY (eds.). Extracellular Nucleic Acids. Springer. pp. 25–38. ISBN 978-3-642-12616-1.
- ^ Vlassov VV, Laktionov PP, Rykova EY (July 2007). "Extracellular nucleic acids". BioEssays. 29 (7): 654–67. doi:10.1002/bies.20604. PMID 17563084. S2CID 32463239.
- ^ Finkel SE, Kolter R (November 2001). "DNA as a nutrient: novel role for bacterial competence gene homologs". Journal of Bacteriology. 183 (21): 6288–93. doi:10.1128/JB.183.21.6288-6293.2001. PMC 100116. PMID 11591672.
- ^ Mulcahy H, Charron-Mazenod L, Lewenza S (November 2008). "Extracellular DNA chelates cations and induces antibiotic resistance in Pseudomonas aeruginosa biofilms". PLOS Pathogens. 4 (11): e1000213. doi:10.1371/journal.ppat.1000213. PMC 2581603. PMID 19023416.
- ^ Berne C, Kysela DT, Brun YV (August 2010). "A bacterial extracellular DNA inhibits settling of motile progeny cells within a biofilm". Molecular Microbiology. 77 (4): 815–29. doi:10.1111/j.1365-2958.2010.07267.x. PMC 2962764. PMID 20598083.
- ^ Whitchurch CB, Tolker-Nielsen T, Ragas PC, Mattick JS (February 2002). "Extracellular DNA required for bacterial biofilm formation". Science. 295 (5559): 1487. doi:10.1126/science.295.5559.1487. PMID 11859186.
- ^ Hu W, Li L, Sharma S, Wang J, McHardy I, Lux R, Yang Z, He X, Gimzewski JK, Li Y, Shi W (2012). "DNA builds and strengthens the extracellular matrix in Myxococcus xanthus biofilms by interacting with exopolysaccharides". PLOS ONE. 7 (12): e51905. Bibcode:2012PLoSO...751905H. doi:10.1371/journal.pone.0051905. PMC 3530553. PMID 23300576.
- ^ Hui L, Bianchi DW (February 2013). "Recent advances in the prenatal interrogation of the human fetal genome". Trends in Genetics. 29 (2): 84–91. doi:10.1016/j.tig.2012.10.013. PMC 4378900. PMID 23158400.
- ^ Foote AD, Thomsen PF, Sveegaard S, Wahlberg M, Kielgast J, Kyhn LA, et al. (2012). "Investigating the potential use of environmental DNA (eDNA) for genetic monitoring of marine mammals". PLOS ONE. 7 (8): e41781. Bibcode:2012PLoSO...741781F. doi:10.1371/journal.pone.0041781. PMC 3430683. PMID 22952587.
- ^ "Researchers Detect Land Animals Using DNA in Nearby Water Bodies".
- ^ Sandman K, Pereira SL, Reeve JN (December 1998). "Diversity of prokaryotic chromosomal proteins and the origin of the nucleosome". Cellular and Molecular Life Sciences. 54 (12): 1350–64. doi:10.1007/s000180050259. PMID 9893710. S2CID 21101836.
- ^ Dame RT (May 2005). "The role of nucleoid-associated proteins in the organization and compaction of bacterial chromatin". Molecular Microbiology. 56 (4): 858–70. doi:10.1111/j.1365-2958.2005.04598.x. PMID 15853876. S2CID 26965112.
- ^ Luger K, Mäder AW, Richmond RK, Sargent DF, Richmond TJ (September 1997). "Crystal structure of the nucleosome core particle at 2.8 A resolution". Nature. 389 (6648): 251–60. Bibcode:1997Natur.389..251L. doi:10.1038/38444. PMID 9305837. S2CID 4328827.
- ^ Jenuwein T, Allis CD (August 2001). "Translating the histone code" (PDF). Science. 293 (5532): 1074–80. doi:10.1126/science.1063127. PMID 11498575. S2CID 1883924. Archived (PDF) from the original on 8 August 2017.
- ^ Ito T (2003). "Nucleosome assembly and remodeling". Current Topics in Microbiology and Immunology. 274: 1–22. doi:10.1007/978-3-642-55747-7_1. ISBN 978-3-540-44208-0. PMID 12596902.
- ^ Thomas JO (August 2001). "HMG1 and 2: architectural DNA-binding proteins". Biochemical Society Transactions. 29 (Pt 4): 395–401. doi:10.1042/BST0290395. PMID 11497996.
- ^ Grosschedl R, Giese K, Pagel J (March 1994). "HMG domain proteins: architectural elements in the assembly of nucleoprotein structures". Trends in Genetics. 10 (3): 94–100. doi:10.1016/0168-9525(94)90232-1. PMID 8178371.
- ^ Iftode C, Daniely Y, Borowiec JA (1999). "Replication protein A (RPA): the eukaryotic SSB". Critical Reviews in Biochemistry and Molecular Biology. 34 (3): 141–80. doi:10.1080/10409239991209255. PMID 10473346.
- ^ 2008년 1월 6일 웨이백 시스템에 보관된 PDB 1LMB에서 생성
- ^ Myers LC, Kornberg RD (2000). "Mediator of transcriptional regulation". Annual Review of Biochemistry. 69: 729–49. doi:10.1146/annurev.biochem.69.1.729. PMID 10966474.
- ^ Spiegelman BM, Heinrich R (October 2004). "Biological control through regulated transcriptional coactivators". Cell. 119 (2): 157–67. doi:10.1016/j.cell.2004.09.037. PMID 15479634.
- ^ Li Z, Van Calcar S, Qu C, Cavenee WK, Zhang MQ, Ren B (July 2003). "A global transcriptional regulatory role for c-Myc in Burkitt's lymphoma cells". Proceedings of the National Academy of Sciences of the United States of America. 100 (14): 8164–69. Bibcode:2003PNAS..100.8164L. doi:10.1073/pnas.1332764100. PMC 166200. PMID 12808131.
- ^ 2008년 1월 6일 웨이백 시스템에 PDB 1RVA 아카이브에서 생성
- ^ Bickle TA, Krüger DH (June 1993). "Biology of DNA restriction". Microbiological Reviews. 57 (2): 434–50. doi:10.1128/MMBR.57.2.434-450.1993. PMC 372918. PMID 8336674.
- ^ a b Doherty AJ, Suh SW (November 2000). "Structural and mechanistic conservation in DNA ligases". Nucleic Acids Research. 28 (21): 4051–58. doi:10.1093/nar/28.21.4051. PMC 113121. PMID 11058099.
- ^ Schoeffler AJ, Berger JM (December 2005). "Recent advances in understanding structure-function relationships in the type II topoisomerase mechanism". Biochemical Society Transactions. 33 (Pt 6): 1465–70. doi:10.1042/BST20051465. PMID 16246147.
- ^ Tuteja N, Tuteja R (May 2004). "Unraveling DNA helicases. Motif, structure, mechanism and function" (PDF). European Journal of Biochemistry. 271 (10): 1849–63. doi:10.1111/j.1432-1033.2004.04094.x. PMID 15128295.
- ^ Joyce CM, Steitz TA (November 1995). "Polymerase structures and function: variations on a theme?". Journal of Bacteriology. 177 (22): 6321–29. doi:10.1128/jb.177.22.6321-6329.1995. PMC 177480. PMID 7592405.
- ^ Hubscher U, Maga G, Spadari S (2002). "Eukaryotic DNA polymerases" (PDF). Annual Review of Biochemistry. 71: 133–63. doi:10.1146/annurev.biochem.71.090501.150041. PMID 12045093. S2CID 26171993. Archived from the original (PDF) on 26 January 2021.
- ^ Johnson A, O'Donnell M (2005). "Cellular DNA replicases: components and dynamics at the replication fork". Annual Review of Biochemistry. 74: 283–315. doi:10.1146/annurev.biochem.73.011303.073859. PMID 15952889.
- ^ a b Tarrago-Litvak L, Andréola ML, Nevinsky GA, Sarih-Cottin L, Litvak S (May 1994). "The reverse transcriptase of HIV-1: from enzymology to therapeutic intervention". FASEB Journal. 8 (8): 497–503. doi:10.1096/fasebj.8.8.7514143. PMID 7514143. S2CID 39614573.
- ^ Martinez E (December 2002). "Multi-protein complexes in eukaryotic gene transcription". Plant Molecular Biology. 50 (6): 925–47. doi:10.1023/A:1021258713850. PMID 12516863. S2CID 24946189.
- ^ PDB 1M6G Wayback Machine에 2010년 1월 10일 아카이브된 PDB에서 생성
- ^ Cremer T, Cremer C (April 2001). "Chromosome territories, nuclear architecture and gene regulation in mammalian cells". Nature Reviews Genetics. 2 (4): 292–301. doi:10.1038/35066075. PMID 11283701. S2CID 8547149.
- ^ Pál C, Papp B, Lercher MJ (May 2006). "An integrated view of protein evolution". Nature Reviews Genetics. 7 (5): 337–48. doi:10.1038/nrg1838. PMID 16619049. S2CID 23225873.
- ^ O'Driscoll M, Jeggo PA (January 2006). "The role of double-strand break repair – insights from human genetics". Nature Reviews Genetics. 7 (1): 45–54. doi:10.1038/nrg1746. PMID 16369571. S2CID 7779574.
- ^ Vispé S, Defais M (October 1997). "Mammalian Rad51 protein: a RecA homologue with pleiotropic functions". Biochimie. 79 (9–10): 587–92. doi:10.1016/S0300-9084(97)82007-X. PMID 9466696.
- ^ Neale MJ, Keeney S (July 2006). "Clarifying the mechanics of DNA strand exchange in meiotic recombination". Nature. 442 (7099): 153–58. Bibcode:2006Natur.442..153N. doi:10.1038/nature04885. PMC 5607947. PMID 16838012.
- ^ Dickman MJ, Ingleston SM, Sedelnikova SE, Rafferty JB, Lloyd RG, Grasby JA, Hornby DP (November 2002). "The RuvABC resolvasome". European Journal of Biochemistry. 269 (22): 5492–501. doi:10.1046/j.1432-1033.2002.03250.x. PMID 12423347. S2CID 39505263.
- ^ Joyce GF (July 2002). "The antiquity of RNA-based evolution". Nature. 418 (6894): 214–21. Bibcode:2002Natur.418..214J. doi:10.1038/418214a. PMID 12110897. S2CID 4331004.
- ^ Orgel LE (2004). "Prebiotic chemistry and the origin of the RNA world". Critical Reviews in Biochemistry and Molecular Biology. 39 (2): 99–123. CiteSeerX 10.1.1.537.7679. doi:10.1080/10409230490460765. PMID 15217990.
- ^ Davenport RJ (May 2001). "Ribozymes. Making copies in the RNA world". Science. 292 (5520): 1278a–1278. doi:10.1126/science.292.5520.1278a. PMID 11360970. S2CID 85976762.
- ^ Szathmáry E (April 1992). "What is the optimum size for the genetic alphabet?". Proceedings of the National Academy of Sciences of the United States of America. 89 (7): 2614–18. Bibcode:1992PNAS...89.2614S. doi:10.1073/pnas.89.7.2614. PMC 48712. PMID 1372984.
- ^ Lindahl T (April 1993). "Instability and decay of the primary structure of DNA". Nature. 362 (6422): 709–15. Bibcode:1993Natur.362..709L. doi:10.1038/362709a0. PMID 8469282. S2CID 4283694.
- ^ Vreeland RH, Rosenzweig WD, Powers DW (October 2000). "Isolation of a 250 million-year-old halotolerant bacterium from a primary salt crystal". Nature. 407 (6806): 897–900. Bibcode:2000Natur.407..897V. doi:10.1038/35038060. PMID 11057666. S2CID 9879073.
- ^ Hebsgaard MB, Phillips MJ, Willerslev E (May 2005). "Geologically ancient DNA: fact or artefact?". Trends in Microbiology. 13 (5): 212–20. doi:10.1016/j.tim.2005.03.010. PMID 15866038.
- ^ Nickle DC, Learn GH, Rain MW, Mullins JI, Mittler JE (January 2002). "Curiously modern DNA for a "250 million-year-old" bacterium". Journal of Molecular Evolution. 54 (1): 134–37. Bibcode:2002JMolE..54..134N. doi:10.1007/s00239-001-0025-x. PMID 11734907. S2CID 24740859.
- ^ Callahan MP, Smith KE, Cleaves HJ, Ruzicka J, Stern JC, Glavin DP, House CH, Dworkin JP (August 2011). "Carbonaceous meteorites contain a wide range of extraterrestrial nucleobases". Proceedings of the National Academy of Sciences of the United States of America. 108 (34): 13995–98. Bibcode:2011PNAS..10813995C. doi:10.1073/pnas.1106493108. PMC 3161613. PMID 21836052.
- ^ Steigerwald J (8 August 2011). "NASA Researchers: DNA Building Blocks Can Be Made in Space". NASA. Archived from the original on 23 June 2015. Retrieved 10 August 2011.
- ^ ScienceDaily Staff (9 August 2011). "DNA Building Blocks Can Be Made in Space, NASA Evidence Suggests". ScienceDaily. Archived from the original on 5 September 2011. Retrieved 9 August 2011.
- ^ Marlaire R (3 March 2015). "NASA Ames Reproduces the Building Blocks of Life in Laboratory". NASA. Archived from the original on 5 March 2015. Retrieved 5 March 2015.
- ^ Hunt, Katie (17 February 2021). "World's oldest DNA sequenced from a mammoth that lived more than a million years ago". CNN News. Retrieved 17 February 2021.
- ^ Callaway, Ewen (17 February 2021). "Million-year-old mammoth genomes shatter record for oldest ancient DNA - Permafrost-preserved teeth, up to 1.6 million years old, identify a new kind of mammoth in Siberia". Nature. 590 (7847): 537–538. doi:10.1038/d41586-021-00436-x. ISSN 0028-0836. PMID 33597786.
- ^ Goff SP, Berg P (December 1976). "Construction of hybrid viruses containing SV40 and lambda phage DNA segments and their propagation in cultured monkey cells". Cell. 9 (4 PT 2): 695–705. doi:10.1016/0092-8674(76)90133-1. PMID 189942. S2CID 41788896.
- ^ Houdebine LM (2007). "Transgenic animal models in biomedical research". Target Discovery and Validation Reviews and Protocols. Methods in Molecular Biology. Vol. 360. pp. 163–202. doi:10.1385/1-59745-165-7:163. ISBN 978-1-59745-165-9. PMID 17172731.
- ^ Daniell H, Dhingra A (April 2002). "Multigene engineering: dawn of an exciting new era in biotechnology". Current Opinion in Biotechnology. 13 (2): 136–41. doi:10.1016/S0958-1669(02)00297-5. PMC 3481857. PMID 11950565.
- ^ Job D (November 2002). "Plant biotechnology in agriculture". Biochimie. 84 (11): 1105–10. doi:10.1016/S0300-9084(02)00013-5. PMID 12595138.
- ^ Curtis C, Hereward J (29 August 2017). "From the crime scene to the courtroom: the journey of a DNA sample". The Conversation. Archived from the original on 22 October 2017. Retrieved 22 October 2017.
- ^ Collins A, Morton NE (June 1994). "Likelihood ratios for DNA identification". Proceedings of the National Academy of Sciences of the United States of America. 91 (13): 6007–11. Bibcode:1994PNAS...91.6007C. doi:10.1073/pnas.91.13.6007. PMC 44126. PMID 8016106.
- ^ Weir BS, Triggs CM, Starling L, Stowell LI, Walsh KA, Buckleton J (March 1997). "Interpreting DNA mixtures" (PDF). Journal of Forensic Sciences. 42 (2): 213–22. doi:10.1520/JFS14100J. PMID 9068179. S2CID 14511630.
- ^ Jeffreys AJ, Wilson V, Thein SL (1985). "Individual-specific 'fingerprints' of human DNA". Nature. 316 (6023): 76–79. Bibcode:1985Natur.316...76J. doi:10.1038/316076a0. PMID 2989708. S2CID 4229883.
- ^ 콜린 피치포크 – DNA 증거에 대한 첫 살인 유죄판결도 2006년 12월 23일에 접근한 주요 용의자의 무죄를 입증한다.
- ^ "DNA Identification in Mass Fatality Incidents". National Institute of Justice. September 2006. Archived from the original on 12 November 2006.
- ^ "2012년 6월 20일 뉴욕 타임즈 6월 24일 웨이백 머신에 보관된 "임신 초기에 효과가 있는 소아 혈액 검사"
- ^ a b Breaker RR, Joyce GF (December 1994). "A DNA enzyme that cleaves RNA". Chemistry & Biology. 1 (4): 223–29. doi:10.1016/1074-5521(94)90014-0. PMID 9383394.
- ^ Chandra M, Sachdeva A, Silverman SK (October 2009). "DNA-catalyzed sequence-specific hydrolysis of DNA". Nature Chemical Biology. 5 (10): 718–20. doi:10.1038/nchembio.201. PMC 2746877. PMID 19684594.
- ^ Carmi N, Shultz LA, Breaker RR (December 1996). "In vitro selection of self-cleaving DNAs". Chemistry & Biology. 3 (12): 1039–46. doi:10.1016/S1074-5521(96)90170-2. PMID 9000012.
- ^ Torabi SF, Wu P, McGhee CE, Chen L, Hwang K, Zheng N, Cheng J, Lu Y (May 2015). "In vitro selection of a sodium-specific DNAzyme and its application in intracellular sensing". Proceedings of the National Academy of Sciences of the United States of America. 112 (19): 5903–08. Bibcode:2015PNAS..112.5903T. doi:10.1073/pnas.1420361112. PMC 4434688. PMID 25918425.
- ^ Baldi P, Brunak S (2001). Bioinformatics: The Machine Learning Approach. MIT Press. ISBN 978-0-262-02506-5. OCLC 45951728.
- ^ Gusfield D (15 January 1997). Algorithms on Strings, Trees, and Sequences: Computer Science and Computational Biology. Cambridge University Press. ISBN 978-0-521-58519-4.
- ^ Sjölander K (January 2004). "Phylogenomic inference of protein molecular function: advances and challenges". Bioinformatics. 20 (2): 170–79. CiteSeerX 10.1.1.412.943. doi:10.1093/bioinformatics/bth021. PMID 14734307.
- ^ Mount DM (2004). Bioinformatics: Sequence and Genome Analysis (2nd ed.). Cold Spring Harbor, NY: Cold Spring Harbor Laboratory Press. ISBN 0-87969-712-1. OCLC 55106399.
- ^ Strong, Michael (2004). "Protein Nanomachines". PLOS Biology. 2 (3): e73. doi:10.1371/journal.pbio.0020073. PMC 368168. PMID 15024422. S2CID 13222080.
- ^ Rothemund PW (March 2006). "Folding DNA to create nanoscale shapes and patterns" (PDF). Nature. 440 (7082): 297–302. Bibcode:2006Natur.440..297R. doi:10.1038/nature04586. PMID 16541064. S2CID 4316391.
- ^ Andersen ES, Dong M, Nielsen MM, Jahn K, Subramani R, Mamdouh W, Golas MM, Sander B, Stark H, Oliveira CL, Pedersen JS, Birkedal V, Besenbacher F, Gothelf KV, Kjems J (May 2009). "Self-assembly of a nanoscale DNA box with a controllable lid". Nature. 459 (7243): 73–76. Bibcode:2009Natur.459...73A. doi:10.1038/nature07971. hdl:11858/00-001M-0000-0010-9362-B. PMID 19424153. S2CID 4430815.
- ^ Ishitsuka Y, Ha T (May 2009). "DNA nanotechnology: a nanomachine goes live". Nature Nanotechnology. 4 (5): 281–82. Bibcode:2009NatNa...4..281I. doi:10.1038/nnano.2009.101. PMID 19421208.
- ^ Aldaye FA, Palmer AL, Sleiman HF (September 2008). "Assembling materials with DNA as the guide". Science. 321 (5897): 1795–99. Bibcode:2008Sci...321.1795A. doi:10.1126/science.1154533. PMID 18818351. S2CID 2755388.
- ^ Wray GA (2002). "Dating branches on the tree of life using DNA". Genome Biology. 3 (1): REVIEWS0001. doi:10.1186/gb-2001-3-1-reviews0001. PMC 150454. PMID 11806830.
- ^ Panda D, Molla KA, Baig MJ, Swain A, Behera D, Dash M (May 2018). "DNA as a digital information storage device: hope or hype?". 3 Biotech. 8 (5): 239. doi:10.1007/s13205-018-1246-7. PMC 5935598. PMID 29744271.
- ^ Akram F, Haq IU, Ali H, Laghari AT (October 2018). "Trends to store digital data in DNA: an overview". Molecular Biology Reports. 45 (5): 1479–1490. doi:10.1007/s11033-018-4280-y. PMID 30073589. S2CID 51905843.
- ^ Miescher F (1871). "Ueber die chemische Zusammensetzung der Eiterzellen" [On the chemical composition of pus cells]. Medicinisch-chemische Untersuchungen (in German). 4: 441–60.
[p. 456] Ich habe mich daher später mit meinen Versuchen an die ganzen Kerne gehalten, die Trennung der Körper, die ich einstweilen ohne weiteres Präjudiz als lösliches und unlösliches Nuclein bezeichnen will, einem günstigeren Material überlassend. (Therefore, in my experiments I subsequently limited myself to the whole nucleus, leaving to a more favorable material the separation of the substances, that for the present, without further prejudice, I will designate as soluble and insoluble nuclear material ("Nuclein"))
- ^ Dahm R (January 2008). "Discovering DNA: Friedrich Miescher and the early years of nucleic acid research". Human Genetics. 122 (6): 565–81. doi:10.1007/s00439-007-0433-0. PMID 17901982. S2CID 915930.
- ^ 참조:
- Kossel A (1879). "Ueber Nucleïn der Hefe" [On nuclein in yeast]. Zeitschrift für physiologische Chemie (in German). 3: 284–91.
- Kossel A (1880). "Ueber Nucleïn der Hefe II" [On nuclein in yeast, Part 2]. Zeitschrift für physiologische Chemie (in German). 4: 290–95.
- Kossel A (1881). "Ueber die Verbreitung des Hypoxanthins im Thier- und Pflanzenreich" [On the distribution of hypoxanthins in the animal and plant kingdoms]. Zeitschrift für physiologische Chemie (in German). 5: 267–71.
- Kossel A (1881). Trübne KJ (ed.). Untersuchungen über die Nucleine und ihre Spaltungsprodukte [Investigations into nuclein and its cleavage products] (in German). Strassburg, Germany. p. 19.
- Kossel A (1882). "Ueber Xanthin und Hypoxanthin" [On xanthin and hypoxanthin]. Zeitschrift für physiologische Chemie. 6: 422–31.
- Albret Kosel (1883년) "Zur Chemie des Zellkerns" 2017년 11월 17일 웨이백머신 (세포핵의 화학에 관하여), Zeitschrift für Phr Chemie, 7 : 7–22에 보관.
- Kossel A (1886). "Weitere Beiträge zur Chemie des Zellkerns" [Further contributions to the chemistry of the cell nucleus]. Zeitschrift für Physiologische Chemie (in German). 10: 248–64.
On p. 264, Kossel remarked presciently: Der Erforschung der quantitativen Verhältnisse der vier stickstoffreichen Basen, der Abhängigkeit ihrer Menge von den physiologischen Zuständen der Zelle, verspricht wichtige Aufschlüsse über die elementaren physiologisch-chemischen Vorgänge. (The study of the quantitative relations of the four nitrogenous bases—[and] of the dependence of their quantity on the physiological states of the cell—promises important insights into the fundamental physiological-chemical processes.)
- ^ Jones ME (September 1953). "Albrecht Kossel, a biographical sketch". The Yale Journal of Biology and Medicine. 26 (1): 80–97. PMC 2599350. PMID 13103145.
- ^ Levene PA, Jacobs WA (1909). "Über Inosinsäure". Berichte der Deutschen Chemischen Gesellschaft (in German). 42: 1198–203. doi:10.1002/cber.190904201196.
- ^ Levene PA, Jacobs WA (1909). "Über die Hefe-Nucleinsäure". Berichte der Deutschen Chemischen Gesellschaft (in German). 42 (2): 2474–78. doi:10.1002/cber.190904202148.
- ^ Levene P (1919). "The structure of yeast nucleic acid". J Biol Chem. 40 (2): 415–24. doi:10.1016/S0021-9258(18)87254-4.
- ^ Cohen JS, Portugal FH (1974). "The search for the chemical structure of DNA" (PDF). Connecticut Medicine. 38 (10): 551–52, 554–57. PMID 4609088.
- ^ Koltsov는 세포의 유전 정보가 긴 아미노산 체인으로 암호화되어 있다고 제안했다.참조:
- Кольцов, Н. К. (12 December 1927). Физико-химические основы морфологии [The physical-chemical basis of morphology] (Speech). 3rd All-Union Meeting of Zoologist, Anatomists, and Histologists (in Russian). Leningrad, U.S.S.R.
- 다시 인쇄된 위치:
- 다음과 같이 독일어로 다시 인쇄됨:
- 1934년, 콜트소프는 세포의 유전 정보를 포함하는 단백질이 복제된다고 주장했다.:Koltzoff N(10월 1934년)을 참조하십시오."염색체의 염색체의 침샘의 구조".과학. 80(예를 들어 2075년):312–13.Bibcode:1934Sci....80..312K. doi:10.1126/science.80.2075.312.PMID 17769043.페이지 313.`에서 나는 염색체의 타액선[염색체의]의 크기 genonemes의 증가에 의해 결정된다 생각한다.이번 학기까지 나는;…은 정상적인 염색체에는은 보통 하나 genoneme다;cell-division기 전에 이 genoneme 두 strands."로 나뉘어 져화 되고 있는 염색체의 유전 학자들 유전자의 선형 조합을 찾은 축 방향 스레드를 지정한다.
- ^ Soyfer VN (September 2001). "The consequences of political dictatorship for Russian science". Nature Reviews Genetics. 2 (9): 723–29. doi:10.1038/35088598. PMID 11533721. S2CID 46277758.
- ^ Griffith F (January 1928). "The Significance of Pneumococcal Types". The Journal of Hygiene. 27 (2): 113–59. doi:10.1017/S0022172400031879. PMC 2167760. PMID 20474956.
- ^ Lorenz MG, Wackernagel W (September 1994). "Bacterial gene transfer by natural genetic transformation in the environment". Microbiological Reviews. 58 (3): 563–602. doi:10.1128/MMBR.58.3.563-602.1994. PMC 372978. PMID 7968924.
- ^ Brachet J (1933). "Recherches sur la synthese de l'acide thymonucleique pendant le developpement de l'oeuf d'Oursin". Archives de Biologie (in Italian). 44: 519–76.
- ^ Burian R (1994). "Jean Brachet's Cytochemical Embryology: Connections with the Renovation of Biology in France?" (PDF). In Debru C, Gayon J, Picard JF (eds.). Les sciences biologiques et médicales en France 1920–1950. Cahiers pour I'histoire de la recherche. Vol. 2. Paris: CNRS Editions. pp. 207–20.
- ^ 참조:
- Astbury WT, Bell FO (1938). "Some recent developments in the X-ray study of proteins and related structures" (PDF). Cold Spring Harbor Symposia on Quantitative Biology. 6: 109–21. doi:10.1101/sqb.1938.006.01.013. Archived from the original (PDF) on 14 July 2014.
- Astbury WT (1947). "X-ray studies of nucleic acids". Symposia of the Society for Experimental Biology (1): 66–76. PMID 20257017. Archived from the original on 5 July 2014.
- ^ Avery OT, Macleod CM, McCarty M (February 1944). "Studies on the Chemical Nature of the Substance Inducing Transformation of Pneumococcal Types: Induction of Transformation by a Desoxyribonucleic Acid Fraction Isolated from Pneumococcus Type III". The Journal of Experimental Medicine. 79 (2): 137–58. doi:10.1084/jem.79.2.137. PMC 2135445. PMID 19871359.
- ^ Chargaff, Erwin (June 1950). "Chemical specificity of nucleic acids and mechanism of their enzymatic degradation". Experientia. 6 (6): 201–209. doi:10.1007/BF02173653. PMID 15421335. S2CID 2522535.
- ^ Kresge, Nicole; Simoni, Robert D.; Hill, Robert L. (June 2005). "Chargaff's Rules: the Work of Erwin Chargaff". Journal of Biological Chemistry. 280 (24): 172–174. doi:10.1016/S0021-9258(20)61522-8.
- ^ Hershey AD, Chase M (May 1952). "Independent functions of viral protein and nucleic acid in growth of bacteriophage". The Journal of General Physiology. 36 (1): 39–56. doi:10.1085/jgp.36.1.39. PMC 2147348. PMID 12981234.
- ^ 이 링크된 이미지의 오른쪽에 있는 B-DNA X선 패턴 2012년 5월 25일 아카이브.오늘
- ^ Schwartz J (2008). In pursuit of the gene : from Darwin to DNA. Cambridge, Mass.: Harvard University Press. ISBN 9780674026704.
- ^ Pauling L, Corey RB (February 1953). "A Proposed Structure For The Nucleic Acids". Proceedings of the National Academy of Sciences of the United States of America. 39 (2): 84–97. Bibcode:1953PNAS...39...84P. doi:10.1073/pnas.39.2.84. PMC 1063734. PMID 16578429.
- ^ Regis E (2009). What Is Life?: investigating the nature of life in the age of synthetic biology. Oxford: Oxford University Press. p. 52. ISBN 978-0-19-538341-6.
- ^ "Double Helix of DNA: 50 Years". Nature Archives. Archived from the original on 5 April 2015.
- ^ "Original X-ray diffraction image". Oregon State Library. Archived from the original on 30 January 2009. Retrieved 6 February 2011.
- ^ "The Nobel Prize in Physiology or Medicine 1962". Nobelprize.org.
- ^ Maddox B (January 2003). "The double helix and the 'wronged heroine'" (PDF). Nature. 421 (6921): 407–08. Bibcode:2003Natur.421..407M. doi:10.1038/nature01399. PMID 12540909. S2CID 4428347. Archived (PDF) from the original on 17 October 2016.
- ^ Crick F (1955). A Note for the RNA Tie Club (PDF) (Speech). Cambridge, England. Archived from the original (PDF) on 1 October 2008.
- ^ Meselson M, Stahl FW (July 1958). "The Replication of DNA in Escherichia Coli". Proceedings of the National Academy of Sciences of the United States of America. 44 (7): 671–82. Bibcode:1958PNAS...44..671M. doi:10.1073/pnas.44.7.671. PMC 528642. PMID 16590258.
- ^ "The Nobel Prize in Physiology or Medicine 1968". Nobelprize.org.
- ^ Pray L (2008). "Discovery of DNA structure and function: Watson and Crick". Nature Education. 1 (1): 100.
추가 읽기
- Berry A, Watson J (2003). DNA: the secret of life. New York: Alfred A. Knopf. ISBN 0-375-41546-7.
- Calladine CR, Drew HR, Luisi BF, Travers AA (2003). Understanding DNA: the molecule & how it works. Amsterdam: Elsevier Academic Press. ISBN 0-12-155089-3.
- Carina D, Clayton J (2003). 50 years of DNA. Basingstoke: Palgrave Macmillan. ISBN 1-4039-1479-6.
- Judson HF (1979). The Eighth Day of Creation: Makers of the Revolution in Biology (2nd ed.). Cold Spring Harbor Laboratory Press. ISBN 0-671-22540-5.
- Olby RC (1994). The path to the double helix: the discovery of DNA. New York: Dover Publications. ISBN 0-486-68117-3. 1974년 10월 맥밀런에 의해 처음 출판되었고, 프란시스 크릭의 서문과 함께, 9페이지 분량의 추신으로 1994년에 개정되었다.
- Olby R (January 2003). "Quiet debut for the double helix". Nature. 421 (6921): 402–05. Bibcode:2003Natur.421..402O. doi:10.1038/nature01397. PMID 12540907.
- Olby RC (2009). Francis Crick: A Biography. Plainview, N.Y: Cold Spring Harbor Laboratory Press. ISBN 978-0-87969-798-3.
- Micklas D (2003). DNA Science: A First Course. Cold Spring Harbor Press. ISBN 978-0-87969-636-8.
- Ridley M (2006). Francis Crick: discoverer of the genetic code. Ashland, OH: Eminent Lives, Atlas Books. ISBN 0-06-082333-X.
- Rosenfeld I (2010). DNA: A Graphic Guide to the Molecule that Shook the World. Columbia University Press. ISBN 978-0-231-14271-7.
- Schultz M, Cannon Z (2009). The Stuff of Life: A Graphic Guide to Genetics and DNA. Hill and Wang. ISBN 978-0-8090-8947-5.
- Stent GS, Watson J (1980). The Double Helix: A Personal Account of the Discovery of the Structure of DNA. New York: Norton. ISBN 0-393-95075-1.
- Watson J (2004). DNA: The Secret of Life. Random House. ISBN 978-0-09-945184-6.
- Wilkins M (2003). The third man of the double helix the autobiography of Maurice Wilkins. Cambridge, England: University Press. ISBN 0-19-860665-6.
외부 링크
| 라이브러리 리소스 정보 DNA |
| Wikiquote는 DNA와 관련된 인용구를 가지고 있다. |
| 위키다양성은 DNA에 대한 학습자원을 가지고 있다. |
| 위키미디어 커먼즈에는 DNA와 관련된 미디어가 있다. |
- 컬리에서의 DNA
- 단백질에 대한 DNA 결합 부위 예측
- 공식 노벨상 웹사이트에서 나온 이중 나선 게임 DNA
- 전자현미경에 의한 DNA
- 돌란 DNA학습관
- 이중나선: 50년 DNA, 자연
- 프로테오피디아 DNA
- 프로테오피디아 Forms_of_DNA
- 인코드 스레드 탐색기 인코드 홈 페이지.자연
- Double Helix 1953-2003 미국 생명공학 교육 센터
- 교사를 위한 유전자 교육 모듈 – 기초 학습 가이드의 DNA
- 이달의 PDB 분자 DNA
- "유전의 화학적 결함을 발견했다"뉴욕 타임즈, 1953년 6월DNA 구조의 발견에 대한 첫 미국 신문 보도
- 시작부터 DNA 또 다른 DNA 학습 센터 사이트 멘델에서 인간 게놈 프로젝트에 이르는 DNA, 유전자, 유전자에 관한 사이트.
- 미국 샌디에이고 주 캘리포니아 대학교 맨더빌 특별 소장 도서관의 프랜시스 크릭 개인 논문 목록 1938 - 2007
- 크릭이 1953년 12살짜리 아들 마이클에게 보낸 7페이지 분량의 친필 편지에는 DNA 구조를 기술하고 있다.크릭의 메달이 2013년 4월 5일 네이처라는 망치 밑에 들어가는 것을 보라.