베타 시트
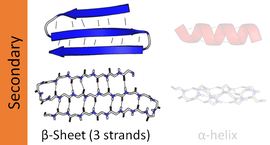
Beta sheet
베타 시트(β-시트)(β-완성 시트도)는 일반 단백질 2차 구조의 일반적인 모티브이다.베타 시트는 적어도 두세 개의 골격 수소 결합에 의해 측면으로 연결된 베타 가닥(β-스트랜드)으로 구성되며, 일반적으로 꼬인 주름 시트를 형성한다.β 가닥은 확장된 형태에서 골격을 가진 일반적으로 3~10개의 아미노산 길이의 폴리펩타이드 사슬이다.β-시트의 초분자 연관성은 아밀로이드증, 특히 알츠하이머병에서 관찰되는 섬유소와 단백질 응집체의 형성과 관련이 있다.
역사

최초의 β-시트 구조는 1930년대에 윌리엄 애스트베리에 의해 제안되었다.그는 평행 또는 역평행 확장 β-스트랜드 펩타이드 결합 사이의 수소 결합 아이디어를 제안했다.하지만, Astbury는 정확한 모델을 만들기 위해 아미노산의 결합 기하학에 대한 필요한 데이터를 가지고 있지 않았다. 왜냐하면 그는 그 때 펩타이드 결합이 평면이라는 것을 알지 못했기 때문이다.1951년 리누스 폴링과 로버트 코리에 의해 개량된 버전이 제안되었다.이들의 모델은 이전에 케토에놀 호변이성(tautomerization)의 결과로 설명했던 펩타이드 결합의 평면성을 통합했다.
구조 및 방향
기하학.
β-아세틸의 대부분은 다른 가닥에 인접해 배열되며, 인접한 이웃과 광범위한 수소 결합 네트워크를 형성하며, 한 가닥의 백본에 있는 N-H 그룹은 인접한 가닥의 백본에 있는 C=O 그룹과 수소 결합을 형성한다.전연장β스트랜드에서는 연속된 사이드체인이 직하방향으로 번갈아 패턴으로 포인트된다.β시트 내의 인접한 β스트랜드는 C원자가α 인접하고 측쇄가 같은 방향을 가리키도록 배열되어 있다.β-스트랜드의 "완벽한" 모양은 C 원자의α 사면체 화학 결합에서 발생한다. 예를 들어, 측면 사슬이 직선으로 위쪽을 가리키면 결합 각도가 약 109.5°이기 때문에 Cθ에 대한 결합은 약간 아래쪽을 가리켜야 한다.주름으로 인해 C와α
i + 2 C 사이의 거리는α
i 2개의 완전 확장 트랜스 펩타이드에서 예상되는 7.6 †(0.76 nm)가 아닌 약 6Ω(0.60 nm)가 된다.수소 결합 β-스트랜드에서 인접한α C 원자 사이의 "측면" 거리는 약 5Ω(0.50 nm)이다.
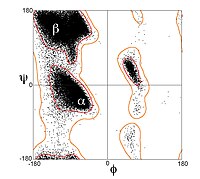
그러나, β-스트랜드는 거의 완벽하게 확장되지 않으며, 오히려 비틀림을 나타낸다.에너지적으로 선호하는 (θ, θ) 근처 이면각 = (-135°, 135°)(예: 라마찬드란 플롯의 왼쪽 상단 영역) 완전 확장 형상(예: θ, θ) = (–180°, 180°)[1]에서 상당히 다르다.비틀림은 종종 큰 시트의 개별 β-스트랜드가 흩어지는 것을 방지하기 위해 이면각의 교대로 변동하는 것과 관련이 있다.단백질 BPTI에서 강하게 꼬인 β헤어핀의 좋은 예를 볼 수 있다.
사이드 체인은 시트 평면에 거의 수직으로 주름의 바깥쪽을 가리키며, 연속적인 아미노산 잔류물은 시트의 번갈아 가면서 바깥쪽을 가리킵니다.
수소 결합 패턴
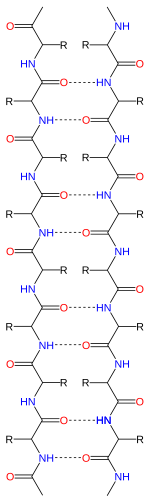
펩타이드 사슬은 N 말단 및 C 말단에 의해 부여된 방향성을 가지기 때문에 β 가닥도 방향성이 있다고 할 수 있다.그것들은 보통 단백질 토폴로지 다이어그램에서 C 말단을 가리키는 화살표로 나타납니다.인접한 β 가닥은 역평행, 평행 또는 혼합 배열로 수소 결합을 형성할 수 있다.
역평행 배열에서는 연속된 β-스트랜드가 하나의 스트랜드의 N-말단이 다음 스트랜드의 C-말단에 인접하도록 방향을 바꾼다.이것은 카르보닐과 아민 사이의 사슬간 수소 결합이 그들이 선호하는 방향인 평면이 되도록 하기 때문에 가장 강한 가닥간 안정성을 생성하는 배열이다.펩타이드 골격 이면각(θ, θ)은 약 (-140°, 135°)이다.역평행 시트로.이 경우, 두 개의 수소 결합 β-스트랜드에 두 개의 원자α
i C와α
j C가 인접해 있으면, 그들은 서로의 옆구리 펩타이드 그룹에 두 개의 상호 골격 수소 결합을 형성한다. 이것은 수소 결합의 근접 쌍으로 알려져 있다.
병렬 배열에서는 연속된 가닥의 모든 N-termini는 같은 방향으로 배향된다.이 배향은 가닥간 수소 결합 패턴에 비평행성을 도입하기 때문에 약간 덜 안정적일 수 있다.이면각(θ, θ)은 약(-120°, 115°)이다.평행하게.모티브에서 상호작용하는 평행 가닥이 5개 미만인 경우는 드물며, 이는 더 적은 수의 가닥이 불안정할 수 있음을 시사하지만, N과 C 흰자리가 정렬된 가닥은 반드시 순차적으로[citation needed] 매우 멀어야 하기 때문에 평행 β-시트가 형성되는 것도 근본적으로 더 어렵다.또한 작은 아밀로이드 생성 서열이 일반적으로 일차적으로 평행한 β 시트 가닥으로 구성된 β 시트 섬유로 집합하는 것으로 나타나기 때문에 평행 β 시트가 더 안정적일 수 있다는 증거가 있다.
병렬 β-시트 구조에서, 2개의 원자 C와 C가 2개의 수소 결합α
iα
j β-스트랜드에 인접해 있으면, 그들은 서로 수소 결합하지 않고, 오히려 하나의 잔류물이 다른 잔류물에 수소 결합을 형성한다(단, 그 반대는 아니다).예를 들어 잔류물 i는 잔류물 j - 1과 j + 1에 수소 결합을 형성할 수 있으며, 이는 수소 결합의 넓은 쌍으로 알려져 있다.반면 잔류물 j는 다른 잔류물에 모두 수소 결합하거나 전혀 결합하지 않을 수 있다.
병렬 베타 시트의 수소 결합 배열은 11개의 원자를 가진 아미드 고리 모티브의 수소 결합 배열과 유사하다.
마지막으로 개별 가닥은 한쪽은 평행 가닥, 다른 한쪽은 역평행 가닥을 가진 혼합 결합 패턴을 나타낼 수 있다.그러한 배열은 무작위로 분포된 배향보다 덜 일반적이며, 이는 이 패턴이 반평행 배열보다 덜 안정적이라는 것을 시사한다. 그러나 생물정보 분석은 항상 전체 단백질에 다른 수많은 구조적 특징이 존재하기 때문에 구조 열역학을 추출하는 데 항상 어려움을 겪는다.또한 단백질은 접이식 열역학뿐만 아니라 접이식 열역학에 의해 본질적으로 구속되기 때문에 생체정보 분석에서 안정성을 결론내릴 때 항상 주의해야 한다.
β-스트랜드의 수소 결합은 완벽할 필요는 없지만, β-bulge로 알려진 국소적인 교란을 보일 수 있다.
수소 결합은 시트 평면에 대략적으로 존재하며, 펩타이드 카르보닐기는 연속 잔류물과 번갈아 방향을 가리키며, 비교를 위해 연속 카르보닐기는 알파 나선에서 동일한 방향을 가리키고 있다.
아미노산성
β-시트의 중간에 있는 β-스트랜드에서 큰 방향족 잔류물(티로신, 페닐알라닌, 트립토판) 및 β-분기 아미노산(트레오닌, 발린, 이소류신)이 검출되는 것이 바람직하다.다른 유형의 잔류물(프롤린 등)이 β-시트의 가장자리 가닥에서 발견될 가능성이 높으며, 이는 응집과 아밀로이드 [2]형성을 초래할 수 있는 단백질 사이의 "엣지-투-엣지" 관련성을 피하기 위한 것으로 추정된다.
공통 구조 모티브

β-헤어핀 모티브
β-시트를 포함한 매우 단순한 구조 모티브는 β-헤어핀으로, 2개의 역평행 스트랜드가 2~5개의 잔기로 이루어진 짧은 루프로 연결되어 있으며, 그 중 1개는 글리신 또는 프롤린이며, 2개는 엄격한 턴 또는 β-벌지 루프에 필요한 이면각 구조를 가정할 수 있다.개별 가닥은 또한 α-헬리스를 포함할 수 있는 더 긴 루프로 더 정교한 방법으로 연결될 수 있다.
그리스어 키 모티브
그리스의 주요 모티브는 인접한 4개의 반평행 가닥과 그 연결 루프로 구성됩니다.그것은 머리핀으로 연결된 세 개의 역평행 가닥으로 구성되어 있고, 네 번째 가닥은 첫 번째 가닥과 인접하고 더 긴 루프로 세 번째 가닥과 연결되어 있습니다.이러한 구조는 단백질 접힘 과정에서 [3][4]쉽게 형성됩니다.그것은 그리스 장식 미술품에 공통적인 패턴에서 이름을 따왔다.
β-α-β 모티브
성분 아미노산의 키랄성으로 인해, 모든 가닥은 대부분의 고차 β 시트 구조에서 분명히 오른손으로 비틀림을 보인다.특히, 2개의 평행한 스트랜드간의 링크 루프는 거의 항상 우측 크로스오버 키랄리티를 가지며,[5] 이는 시트의 고유 트위스트에 의해 강하게 선호된다.이 연결 루프는 종종 나선형 영역을 포함하며, 이 경우 β-α-β 모티브라고 불립니다.β-α-β-α 모티브라고 불리는 밀접하게 관련된 모티브는 가장 일반적으로 관찰되는 단백질 3차 구조인 TIM 배럴의 기본 구성 요소를 형성합니다.


β-meander 모티브
헤어핀 [7][8]루프로 연결된 2개 이상의 연속된 반팔레알β스트랜드로 이루어진 간단한 초2차 단백질 위상.이 모티브는 β-시트에서 일반적이며 β-배럴과 β-프로펠러를 포함한 여러 구조 구조에서 찾을 수 있다.
단백질의 β-meander 영역의 대부분은 폴리펩타이드 사슬의 다른 모티브 또는 단면에 대해 채워져 있으며, 접힌 [9]구조의 형성을 규범적으로 추진하는 소수성 코어의 일부를 형성한다.단, 몇 가지 주목할 만한 예외로는 기존의 소수성 코어가 없을 때 단층 β-시트를 포함하는 외부 표면 단백질 A(OspA) 변종과 단층 β-시트 단백질(SLBP)[10]이[6] 있다.이러한 β가 풍부한 단백질은 주로 개별 가닥을 연결하는 턴 영역에 존재하는 β-스트랜드 간 상호작용과 소수성 상호작용을 통해 안정화되는 확장된 단층 β-meander β-시트를 특징으로 한다.
Psi-루프 모티브
psi-loop(δ-loop) 모티브는 수소 [11]결합에 의해 양쪽 모두에 연결된 하나의 가닥이 있는 두 개의 역평행 가닥으로 구성됩니다.싱글 Ω [12]루프에는 4개의 스트랜드토폴로지가 있어요이 모티브는 단백질이 접히는 동안 형성되는 과정이 일어날 가능성이 거의 없어 보이기 때문에 희귀합니다.δ-루프는 아스파르트 단백질 분해효소 계열에서 [12]처음 확인되었다.
β-시트를 가진 단백질의 구조
β-리보핵산은 전체 [13][14]β, α+β 및 α/β 도메인과 전체 구조가 제대로 정의되지 않은 많은 펩타이드 또는 작은 단백질에 존재한다.all-β 도메인은 β-배럴, β-샌드위치, β-프리즘, β-프로펠러 및 β-헬리스를 형성할 수 있다.
구조 토폴로지
β-시트의 위상은 골격을 따라 수소 결합 β-스트랜드의 순서를 설명한다.예를 들어 플라보독신 접힘은 위상 21345의 5가닥 병렬 β-시트를 가지며, 따라서 엣지 가닥은 골격을 따라 β-strand 2 및 β-strand 5이다.β스트랜드2는 β스트랜드1에 H결합되어 β스트랜드3에 H결합되어 β스트랜드4에 H결합되어 β스트랜드5에 H결합되어 있다.같은 시스템에서 위에서 설명한 그리스어 키 모티브는 4123 토폴로지를 가집니다.β-시트의 2차 구조는 가닥의 수, 위상, 수소 결합이 평행인지 반평행인지 여부를 제공함으로써 대략적으로 설명할 수 있다.
β-시트는 개방될 수 있으며, 즉 두 개의 가장자리 가닥(플라보독신 접힘 또는 면역글로불린 접힘)을 가지거나 닫힌 β-배럴(TIM 배럴 등)이 될 수 있다.β-배럴은 종종 비틀림 또는 전단(shear)으로 설명된다.일부 열린 β-시트는 매우 곡면이고 스스로 접히거나(SH3 도메인에서처럼) 말발굽 모양을 형성한다(리보핵산가수분해효소 억제제에서처럼).개방된 β-시트는 면대면(β-프로펠러 도메인 또는 면역글로불린 접힘 등) 또는 엣지-투-에지를 조립하여 하나의 큰 β-시트를 형성할 수 있다.
동적 기능
β-완성 시트 구조는 수소 결합에 의해 이웃하는 가닥과 연결된 확장된 β-결합 폴리펩타이드 사슬에서 만들어진다.이 확장된 백본 구성에 의해 β 시트는 스트레칭에 저항합니다.단백질의 β-시트는 라만 분광법에[15] 의해 관찰되고 준연속 [16]모델로 분석되는 저주파 아코디언 유사 운동을 수행할 수 있다.
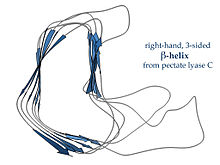
평행 β-헬리시스
짧은 루프로 연결된 2개 또는 3개의 짧은 β스트랜드로 이루어진 반복구조단위로부터 β헬릭스를 형성한다.이들 유닛은 나선형으로 서로 "스택"하여 동일한 가닥의 연속된 수소 결합이 평행한 방향으로 서로 결합합니다.자세한 내용은 β-helix 문서를 참조하십시오.
왼손잡이 β-나선은 스트랜드 자체가 상당히 직선이며 비틀리지 않으며, 그 결과 발생하는 나선 표면은 거의 평탄하며, 우측에 있는 1QRE 고탄산 무수분해효소처럼 정삼각형 프리즘 형상을 형성한다.다른 예로는 지질 A 합성 효소 LpxA와 [17]얼음의 구조를 모방하는 한 면에 규칙적인 Thr 사이드체인 배열이 있는 곤충 부동액 단백질이 있다.
왼쪽 또는 P22 phage tailspike 단백질에 나타나는 펙테이트 리아제 효소로 대표되는 β-헬리스는 단면이 덜 규칙적이고 길며 한쪽이 움푹 패인다. 3개의 링커 루프 중 하나는 일관되게 단지 2개의 잔류물이고 다른 하나는 가변적이며 종종 결합 또는 활성 [18]부위를 형성하기 위해 정교하게 형성한다.
GGXGXD 배열 [19]모티브의 골격 및 Asp 측쇄 산소를 사용하여 구조의 무결성을 유지하기 위해 2개의 루프가 각각 6개의 잔류물로 결합 안정화 칼슘 이온으로 이루어진 양면 β-나선(오른손잡이)이 일부 세균성 금속단백질화효소에서 발견된다.이 접힘을 SCOP 분류에서는 β-roll이라고 합니다.
병리학에서
아밀로이드 β(아밀로이드 플라크 참조)와 같이 단량체로서 무질서하거나 나선형인 일부 단백질은 병리학적 상태와 관련된 β-시트가 풍부한 올리고머 구조를 형성할 수 있다.아밀로이드 β 단백질의 올리고머 형태는 알츠하이머의 원인으로 관련된다.그것의 구조는 아직 완전히 결정되지 않았지만, 최근의 데이터는 이것이 특이한 두 가닥의 β-나선을 [20]닮았을 수도 있다는 것을 암시한다.
아미노산 잔류물은 β-sheet 구조에서 발견된 쪽은 체인점들은 구성할 수 있으면 시트를 boundar을 형성하는 유용할 수 있는 반면 서로 인접하게 시트의 대체 쪽에 많은 것이 또는 충전된 극지(친수성)[21]은 시트의 한쪽 면에 인접 sidechains의 많은, 소수성과 같은.y극지/극지 및 비극지/극지 환경.
「 」를 참조해 주세요.
레퍼런스
- ^ Voet D, Voet JG (2004). Biochemistry (3rd ed.). Hoboken, NJ: Wiley. pp. 227–231. ISBN 0-471-19350-X.
- ^ Richardson JS, Richardson DC (March 2002). "Natural beta-sheet proteins use negative design to avoid edge-to-edge aggregation". Proceedings of the National Academy of Sciences of the United States of America. 99 (5): 2754–9. Bibcode:2002PNAS...99.2754R. doi:10.1073/pnas.052706099. PMC 122420. PMID 11880627.
- ^ 제3단백질 구조 및 접힘: 섹션 4.3.2.1.단백질 구조, 비교 단백질 모델링 및 시각화 원리에서
- ^ Hutchinson EG, Thornton JM (April 1993). "The Greek key motif: extraction, classification and analysis". Protein Engineering. 6 (3): 233–45. doi:10.1093/protein/6.3.233. PMID 8506258.
- ^ 섹션 II B 및 III C, D를 참조하십시오.
- ^ a b Makabe K, McElheny D, Tereshko V, Hilyard A, Gawlak G, Yan S, et al. (November 2006). "Atomic structures of peptide self-assembly mimics". Proceedings of the National Academy of Sciences of the United States of America. 103 (47): 17753–8. Bibcode:2006PNAS..10317753M. doi:10.1073/pnas.0606690103. PMC 1693819. PMID 17093048.
- ^ "SCOP: Fold: WW domain-like". Archived from the original on 2012-02-04. Retrieved 2007-06-01.
- ^ PPS '96 – 슈퍼 세컨더리 구조
- ^ Biancalana M, Makabe K, Koide S (February 2010). "Minimalist design of water-soluble cross-beta architecture". Proceedings of the National Academy of Sciences of the United States of America. 107 (8): 3469–74. Bibcode:2010PNAS..107.3469B. doi:10.1073/pnas.0912654107. PMC 2840449. PMID 20133689.
- ^ Xu, Qingping; Biancalana, Matthew; Grant, Joanna C.; Chiu, Hsiu-Ju; Jaroszewski, Lukasz; Knuth, Mark W.; Lesley, Scott A.; Godzik, Adam; Elsliger, Marc-André; Deacon, Ashley M.; Wilson, Ian A. (September 2019). "Structures of single-layer β-sheet proteins evolved from β-hairpin repeats". Protein Science. 28 (9): 1676–1689. doi:10.1002/pro.3683. ISSN 1469-896X. PMC 6699103. PMID 31306512.
- ^ Hutchinson EG, Thornton JM (February 1996). "PROMOTIF--a program to identify and analyze structural motifs in proteins". Protein Science. 5 (2): 212–20. doi:10.1002/pro.5560050204. PMC 2143354. PMID 8745398.
- ^ a b Hutchinson EG, Thornton JM (1990). "HERA--a program to draw schematic diagrams of protein secondary structures". Proteins. 8 (3): 203–12. doi:10.1002/prot.340080303. PMID 2281084. S2CID 28921557.
- ^ Hubbard TJ, Murzin AG, Brenner SE, Chothia C (January 1997). "SCOP: a structural classification of proteins database". Nucleic Acids Research. 25 (1): 236–9. doi:10.1093/nar/25.1.236. PMC 146380. PMID 9016544.
- ^ Fox NK, Brenner SE, Chandonia JM (January 2014). "SCOPe: Structural Classification of Proteins--extended, integrating SCOP and ASTRAL data and classification of new structures". Nucleic Acids Research. 42 (Database issue): D304-9. doi:10.1093/nar/gkt1240. PMC 3965108. PMID 24304899.
- ^ Painter PC, Mosher LE, Rhoads C (July 1982). "Low-frequency modes in the Raman spectra of proteins". Biopolymers. 21 (7): 1469–72. doi:10.1002/bip.360210715. PMID 7115900.
- ^ Chou KC (August 1985). "Low-frequency motions in protein molecules. Beta-sheet and beta-barrel". Biophysical Journal. 48 (2): 289–97. Bibcode:1985BpJ....48..289C. doi:10.1016/S0006-3495(85)83782-6. PMC 1329320. PMID 4052563.
- ^ Liou YC, Tocilj A, Davies PL, Jia Z (July 2000). "Mimicry of ice structure by surface hydroxyls and water of a beta-helix antifreeze protein". Nature. 406 (6793): 322–4. Bibcode:2000Natur.406..322L. doi:10.1038/35018604. PMID 10917536. S2CID 4385352.
- ^ Branden C, Tooze J (1999). Introduction to Protein Structure. New York: Garland. pp. 20–32. ISBN 0-8153-2305-0.
- ^ Baumann U, Wu S, Flaherty KM, McKay DB (September 1993). "Three-dimensional structure of the alkaline protease of Pseudomonas aeruginosa: a two-domain protein with a calcium binding parallel beta roll motif". The EMBO Journal. 12 (9): 3357–64. doi:10.1002/j.1460-2075.1993.tb06009.x. PMC 413609. PMID 8253063.
- ^ Nelson R, Sawaya MR, Balbirnie M, Madsen AØ, Riekel C, Grothe R, Eisenberg D (June 2005). "Structure of the cross-beta pine of amyloid-like fibrils". Nature. 435 (7043): 773–8. Bibcode:2005Natur.435..773N. doi:10.1038/nature03680. PMC 1479801. PMID 15944695.
- ^ Zhang S, Holmes T, Lockshin C, Rich A (April 1993). "Spontaneous assembly of a self-complementary oligopeptide to form a stable macroscopic membrane". Proceedings of the National Academy of Sciences of the United States of America. 90 (8): 3334–8. Bibcode:1993PNAS...90.3334Z. doi:10.1073/pnas.90.8.3334. PMC 46294. PMID 7682699.
추가 정보
- Cooper J (31 May 1996). "Super Secondary Structure - Part II". Principles of Protein Structure Using the Internet. Retrieved 25 May 2007.
- "Open-sided Beta-meander". Structural Classification of Proteins (SCOP). 20 October 2006. Archived from the original on 4 February 2012. Retrieved 31 May 2007.
외부 링크
- 단백질 구조의 해부 및 분류 - 웨이백 머신에서 2019-03-16년 조사 아카이브
- NetSurfP - 세컨더리 구조 및 표면 접근성 프레딕터