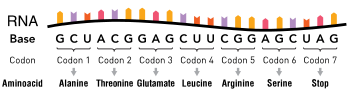진
Gene생물학에서, 유전자(그리스어: έςςςς, génos;[1] 생성, 출생[1] 또는 성별을 의미함[2])는 유전의 기본 단위이며 RNA 또는 [3][4][5]단백질의 유전자 생성물의 합성을 암호화하는 DNA의 일련의 뉴클레오티드이다.
유전자 발현 중에 DNA는 먼저 RNA로 복사된다.RNA는 직접 기능하거나 기능을 수행하는 단백질의 중간 템플릿이 될 수 있습니다.유전자가 유기체의 자손에게 전달되는 것은 표현형 특성의 유전의 기초이다.이 유전자들은 유전자형이라고 불리는 다른 DNA 서열을 구성한다.유전자형은 환경적, 발달적 요인들과 함께 표현형이 될 것을 결정한다.대부분의 생물학적 특성은 유전자-환경 상호작용뿐만 아니라 다유전자(많은 다른 유전자)의 영향을 받는다.눈 색깔이나 팔다리의 수와 같은 어떤 유전적 특징들은 즉시 눈에 띄고, 혈액형, 특정 질병의 위험, 또는 생명을 구성하는 수천 개의 기본적인 생화학적 과정과 같은 어떤 것들은 그렇지 않다.
유전자는 염기서열에서 돌연변이를 획득할 수 있고, 집단에서 대립 유전자로 알려진 다른 변종들을 초래할 수 있다.이 대립 유전자들은 단백질의 약간 다른 버전을 암호화하는데, 이것은 다른 표현형 특성을 일으킨다."유전자 보유"라는 용어의 사용은 일반적으로 동일한 공유 [6]유전자의 다른 대립 유전자를 포함하는 것을 의미한다.유전자는 적자의 자연선택/생존과 대립 유전자의 유전적 표류에 의해 진화한다.
유전자의 개념으로 새로운 현상 발견되고 있어 세분화될 계속되고 있다.[7]예를 들어 유전자의 규제 지역 멀리 떨어진 부호화 지역에서 및 코딩 지역 여러 구조 부위로 분할될 수 있어 제거할 수 있다.일부 바이러스는 RNA대신 DNA에 있고 일부 유전자 상품은 기능non-coding RNAs 그들의 게놈을 저장한다.는 기능성 제품이나 유전자 표현의 규제에 표현되는 것에 의해 한 유기체의 특질에 영향을 미치는 유전, 게놈 서열의 유전자의 넓은, 현대적인 일하는 정의는 어떤 분리된 활동 중심지.[8][9]
용어는 유전자 덴마크의 식물 학자, 식물 생리학자와 가 빌헬름 요한센 1909년에 의해 소개되었다.[10]그것은 고대 그리스어:과 생식 자식을 의미하 γόνος, gonos에 의해 영감을 받았다.
역사
상속된 개별 유닛의 검출
개별 상속 가능한 단위의 존재는 그레고르 멘델 (1822–1884)[11]에 의해 처음 제안되었다.1857년부터 1864년까지, 오스트리아 제국(오늘날의 체코 공화국)의 브르노에서, 그는 부모에서 자손까지 구별되는 특징을 추적하면서, 8000개의 일반적인 식용 완두콩 식물의 유전 패턴을 연구했습니다.그는 이것을 수학적으로 두 가지 조합으로n 설명했는데, 여기서 n은 원래 완두콩의 다른 특징의 수이다.그는 유전자라는 용어를 사용하지 않았지만 관찰 가능한 신체적 특성을 발생시키는 개별 유전 단위의 관점에서 결과를 설명했다.이 설명은 빌헬름 요한센의 유전자형과 표현형 사이의 구별을 미리 구성했다.멘델은 또한 독립적인 분류, 지배적 특성과 열성적 특성 사이의 차이, 헤테로 접합체와 호모 접합체 사이의 차이, 그리고 불연속 유전 현상을 최초로 입증했다.
멘델의 연구 이전에, 유전의 지배적인 이론은 혼합 [12]유전의 하나였는데, 이것은 각각의 부모가 수정 과정에 액체를 기여했고, 자손을 생산하기 위해 부모의 특성이 섞이고 섞였다는 것을 암시했다.찰스 다윈은 그리스 팬 ("전체, 전체")과 창세기 ("태생")/제노스 ("기원")[13][14]로부터 그가 판게네시스라고 부르는 유전 이론을 발전시켰다.다윈은 생식 중에 섞일 가상의 입자를 설명하기 위해 보석이라는 용어를 사용했다.
멘델의 작품은 1866년 첫 출판 이후 크게 주목을 받지 못했지만, 19세기 후반 휴고 드 브리스, 칼 코렌스, 그리고 에리히 폰 체르막에 의해 재발견되었는데, 그는 그들의 [15]연구에서 비슷한 결론에 도달했다고 주장했다.구체적으로, 1889년에 휴고 드 브리스는 그의 책 세포 내 판게네시스를 [16]출판했는데, 그는 다른 성격들이 개별 유전 매개체를 가지고 있고 유기체의 특정한 특징의 유전은 입자로 이루어진다고 가정했다.드 브리스는 다윈의 1868년 판게네시스 이론을 따 이 단위들을 "판게네스"라고 불렀습니다.
20년 후인 1909년, 빌헬름 요한센은 '유전'[10][16]: Translator's preface, viii 이라는 용어를 도입했고, 1906년에는 '유전학'[17][18]의 용어인 '윌리엄 베이트슨'이 '유전학'이라는 용어를 도입했고, 에두아르트 스트라스버거는 여전히 유전의 기본적인 물리적 기능적 단위를 '판젠'이라는 용어를 사용했다.
DNA발견
유전자에 대한 이해와 유전의 발전은 20세기 내내 계속되었다.디옥시리보핵산(DNA)은 1940~1950년대 [19][20]실험에 의해 유전자 정보의 분자 저장소로 밝혀졌다.DNA의 구조는 로잘린드 프랭클린과 모리스 윌킨스에 의해 X선 결정학을 이용하여 연구되었고, 제임스 D.를 이끌었다. 왓슨과 프란시스 크릭은 쌍으로 구성된 뉴클레오티드 염기가 유전자 [21][22]복제 메커니즘에 대한 설득력 있는 가설을 나타내는 이중 가닥 DNA 분자의 모델을 발표했다.
1950년대 초반에는 염색체 내 유전자가 재결합으로 분리되지 않고 끈에 구슬처럼 배열된 분리된 개체처럼 작용한다는 견해가 지배적이었다.Benzer가 박테리오파지 T4(1955–1959)의 rII 영역에서 결함이 있는 돌연변이를 사용한 실험은 개별 유전자가 단순한 선형 구조를 가지고 있고 DNA의 [23][24]선형 단면과 동등할 가능성이 있다는 것을 보여주었다.
총체적으로, 이 연구는 단백질이 DNA에서 전사된 RNA에서 번역된다는 분자생물학의 중심 교리를 확립했다.이 교의는 레트로바이러스의 역전사와 같은 예외를 가지고 있는 것으로 나타났다.DNA 수준에서 유전학에 대한 현대 연구는 분자 유전학으로 알려져 있다.
1972년, 월터 피어스와 그의 팀은 박테리오파지 MS2 [25]코팅 단백질이라는 유전자의 염기서열을 최초로 알아냈다.1977년 Frederick Sanger에 의한 연쇄 종단 DNA 염기서열 분석의 후속 개발은 염기서열 분석의 효율성을 향상시켰고 이를 일상적인 실험실 [26]도구로 만들었다.인간 게놈 프로젝트의 [27]초기 단계에서 Sanger 방법의 자동 버전이 사용되었습니다.
현대 종합과 그.
멘델 유전학과 다윈의 진화를 통합하기 위해 20세기 초에 개발된 이론들은 줄리안 헉슬리에 [28]의해 소개된 용어인 현대 합성이라고 불린다.
진화생물학자들은 조지 C와 같은 이 개념을 나중에 수정했다. 진화론에 대한 윌리엄스의 유전자 중심적 견해.그는 "상당히 [29]: 24 자주 분리되고 재결합하는 것"이라는 정의와 함께 자연선택의 단위로서 유전자의 진화적 개념을 제안했다.이 관점에서는 분자 유전자는 단위로서 전사하고, 진화 유전자는 단위로서 계승한다.진화에서 유전자의 중심성을 강조하는 관련 사상은 리처드 도킨스에 [30][31]의해 대중화되었다.
분자 기준
DNA
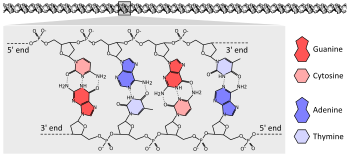
대부분의 유기체는 DNA의 긴 가닥으로 그들의 유전자를 암호화한다.DNA는 4가지 종류의 뉴클레오티드 소단위들로 이루어진 사슬로 구성되어 있으며, 각각은 5개의 탄소당(2-디옥시리보스), 인산기, 그리고 4개의 염기 중 하나인 아데닌, 시토신, 구아닌,[32]: 2.1 티민으로 구성되어 있다.
두 개의 DNA 사슬이 서로 꼬여 DNA 이중나선을 형성하고, 인산-당 골격은 바깥쪽으로 나선형으로 돌고, 염기는 티민에 아데닌 염기쌍으로 안쪽으로 향하고, 구아닌은 사이토신에 연결된다.염기쌍의 특이성은 아데닌과 티민이 두 개의 수소 결합을 형성하기 위해 정렬하는 반면, 시토신과 구아닌은 세 개의 수소 결합을 형성하기 때문에 발생합니다.따라서 이중 나선의 두 가닥은 한 가닥의 아데닌이 다른 가닥의 티마인과 쌍을 이루도록 일치하는 염기서열과 상호보완적이어야 합니다.[32]: 4.1
염기의 펜토오스 잔기의 화학적 조성에 의해 DNA 가닥은 방향성을 가진다.DNA 폴리머의 한쪽 끝은 디옥시리보스에 노출된 수산기를 포함하고 있으며, 이는 분자의 3' 끝이라고 알려져 있습니다.다른 쪽 끝에는 노출된 인산기가 포함되어 있으며, 이것이 5' 끝입니다.이중나선의 두 가닥은 반대 방향으로 뻗어 있다.노출된 3' 수산기를 [33]: 27.2 친핵체로 사용하는 탈수 반응을 통해 새로운 뉴클레오티드가 첨가되기 때문에 DNA 복제 및 전사를 포함한 핵산 합성이 5'→3' 방향으로 일어난다.
DNA로 암호화된 유전자의 발현은 DNA와 매우 유사하지만 디옥시리보스 대신 당 리보스를 함유하는 두 번째 유형의 핵산인 RNA로 유전자를 전사하는 것으로 시작합니다.RNA는 또한 티민 대신 염기성 우라실을 포함하고 있다.RNA 분자는 DNA보다 안정성이 떨어지고 전형적으로 외가닥이다.단백질을 코드하는 유전자는 코돈이라고 불리는 일련의 세 개의 뉴클레오티드 배열로 구성되어 있는데, 코돈은 유전적인 "언어"에서 "단어" 역할을 합니다.유전자 코드는 코돈과 아미노산 사이의 단백질 번역 중 대응 관계를 규정한다.유전자 코드는 알려진 모든 [32]: 4.1 유기체들과 거의 같다.
염색체
유기체나 세포에 있는 유전자의 총 보체는 게놈으로 알려져 있으며, 하나 이상의 염색체에 저장될 수 있다.염색체는 수천 개의 유전자가 [32]: 4.2 암호화되어 있는 하나의 매우 긴 DNA 나선으로 구성되어 있다.특정 유전자가 위치한 염색체의 영역을 유전자자리라고 한다.각각의 궤적은 하나의 유전자의 대립 유전자를 포함하고 있다; 하지만, 집단의 구성원들은 각각의 유전자의 배열이 약간 다른 궤적에서 다른 대립 유전자를 가질 수 있다.
대부분의 진핵생물 유전자는 일련의 크고 선형적인 염색체에 저장되어 있다.염색체들은 핵 안에 히스톤이라고 불리는 저장 단백질과 복합적으로 채워져 뉴클레오솜이라고 불리는 단위를 형성한다.이렇게 포장되고 응축된 DNA를 염색질이라고 [32]: 4.2 한다.히스톤 자체의 화학적 변형뿐만 아니라 DNA가 히스톤에 저장되는 방식은 DNA의 특정 영역이 유전자 발현에 접근할 수 있는지 여부를 조절합니다.유전자 외에도, 진핵세포 염색체는 DNA가 끝 영역의 분해 없이 복제되고 세포 분열 동안 딸 세포로 분류되는 것을 보장하는 것과 관련된 염기서열을 포함한다: 복제 기원, 텔로미어,[32]: 4.2 그리고 동원체.복제 원점은 염색체의 두 개의 복사본을 만들기 위해 DNA 복제가 시작되는 배열 영역입니다.텔로미어는 선형 염색체의 끝을 막고 DNA 복제 중 코딩과 조절 영역의 저하를 방지하는 긴 반복 배열입니다.텔로미어의 길이는 게놈이 복제되고 노화 [35]과정에 관여할 때마다 감소한다.동원체는 세포 [32]: 18.2 분열 중에 자매 염색체를 딸 세포로 분리하기 위해 방추 섬유와 결합하는 데 필요합니다.
원핵생물(박테리아와 고세균)은 전형적으로 하나의 크고 둥근 염색체에 그들의 게놈을 저장합니다.마찬가지로, 몇몇 진핵생물 세포들은 소수의 [32]: 14.4 유전자를 가진 남은 원형 염색체를 포함하고 있다.원핵생물들은 때때로 플라스미드라고 불리는 DNA의 작은 원들로 염색체를 보충하는데, 이것은 보통 몇 개의 유전자만 암호화하고 개인들 사이에서 옮겨질 수 있다.예를 들어, 항생제 내성을 위한 유전자는 보통 박테리아 플라스미드에 암호화되어 수평 유전자 전달을 통해 [36]개별 세포들, 심지어 다른 종의 세포들 사이에 전달될 수 있다.
원핵생물의 염색체는 상대적으로 유전자 밀도가 높은 반면, 진핵생물의 염색체는 종종 뚜렷한 기능을 하지 않는 DNA 영역을 포함한다.인간을 포함한 복잡한 다세포 유기체의 게놈은 확인된 [37]기능 없이 DNA의 절대 다수를 포함하고 있는 반면, 단순한 단세포 진핵 생물은 상대적으로 적은 양의 DNA를 가지고 있다.이 DNA는 종종 "정크 DNA"라고 언급되어 왔다.하지만, 더 최근의 분석들은 비록 단백질 코드 DNA가 인간 게놈의 2%를 차지하지만, 게놈의 약 80%가 발현될 수 있기 때문에, "정크 DNA"라는 용어는 잘못된 [9]명칭일 수 있다는 것을 암시한다.
구조와 기능
구조.
| |
유전자의 구조는 실제 단백질 코드 배열이 종종 작은 부분에 불과한 많은 요소들로 구성되어 있다.여기에는 RNA의 미번역 영역뿐만 아니라 전사되지 않은 DNA 영역도 포함됩니다.
열린 판독 프레임 옆에 있는 유전자는 발현에 필요한 조절 배열을 포함합니다.첫째, 유전자는 프로모터 배열을 필요로 한다.프로모터는 전사를 시작하기 위해 RNA [32]: 7.1 중합효소가 영역에 결합하고 결합하는 것을 돕는 전사 인자에 의해 인식되고 결합됩니다.인식은 일반적으로 TATA 박스와 같은 합의 시퀀스로 이루어진다.하나의 유전자는 하나 이상의 프로모터를 가질 수 있으며, 결과적으로 메신저 RNA(mRNA)가 5'[39] 말기까지 확장되는 정도에 차이가 난다.고도로 전사된 유전자는 전사 인자와 강한 연관성을 형성하는 "강력한" 프로모터 서열을 가지고 있으며, 따라서 빠른 속도로 전사를 시작합니다.다른 유전자들은 전사인자와 약한 연관성을 형성하고 전사를 덜 [32]: 7.2 자주 시작하는 "약한" 촉진제를 가지고 있다.진핵생물 프로모터 영역은 원핵생물 [32]: 7.3 프로모터보다 훨씬 복잡하고 식별하기 어렵다.
또한 유전자는 발현을 변화시키는 열린 판독 프레임의 상류 또는 하류에서 많은 킬로베이스 조절 영역을 가질 수 있다.이들은 DNA를 루프시키는 전사 인자에 결합함으로써 작용하여 조절 배열(및 결합된 전사 인자)이 RNA 중합효소 결합 [40]부위에 가까워집니다.예를 들어, 인핸서는 RNA 중합효소를 프로모터에 모집하는 것을 돕는 활성단백질을 결합함으로써 전사를 증가시키고, 반대로 소음기는 억제단백질을 결합시켜 RNA 중합효소에 [41]DNA를 덜 이용하게 한다.
전사된 사전 mRNA는 양쪽 끝에 리보솜, RNA 결합 단백질, miRNA 및 터미네이터, 시작 및 [42]중지 코돈의 결합 부위를 포함하는 미번역 영역을 포함합니다.또한, 대부분의 진핵생물 개방형 판독 프레임은 제거된 미번역 인트론과 RNA 스플라이싱으로 알려진 과정에서 함께 연결된 엑손들을 포함합니다.마지막으로 유전자 전사의 말단은 분할 및 폴리아데닐화(CPA) 부위에 의해 정의되며, 여기서 새로 생성된 사전 mRNA는 분할되고 3' 말미에 200개 이상의 아데노신 모노인산염이 첨가된다.폴리(A) 꼬리는 성숙한 mRNA를 열화로부터 보호하고 다른 기능을 가지고 있으며, 핵에서 전사체의 번역, 국재화 및 전송에 영향을 미칩니다.CPA에 이은 스플라이싱은 단백질 또는 RNA [43]생성물을 코드하는 최종 성숙한 mRNA를 생성한다.비록 인간 유전자의 위치를 정의하는 일반적인 메커니즘이 알려져 있지만, 이러한 세포 과정을 조절하는 정확한 요소의 확인은 활발한 연구 분야이다.예를 들어, 3'-UTR의 알려진 배열 특성은 모든 인간 유전자 [44]말단의 절반만 설명할 수 있다.
많은 원핵 유전자들은 하나의 단위로 [45][46]전사되는 여러 단백질 코드 배열과 함께 오퍼론으로 조직된다.오퍼론의 유전자는 폴리시스트론 mRNA라고 불리는 연속 전달 RNA로 전사됩니다.이 맥락에서 시스트론이라는 용어는 유전자와 같다.오퍼론의 mRNA의 전사는 종종 특정 대사물의 [47]존재에 따라 활성 또는 비활성 상태에서 발생할 수 있는 억제제에 의해 제어된다.활성 상태일 경우, 억제기는 오퍼론 시작 부분에서 DNA 염기서열(operon region)에 결합하고 오퍼론 전사를 억제합니다.억제기가 비활성 상태일 경우 오퍼론 전사가 발생할 수 있습니다(예: 참조).랙 오퍼론).오퍼론 유전자의 산물은 일반적으로 관련된 기능을 가지고 있으며 동일한 조절 [32]: 7.3 네트워크에 관여한다.
기능 정의
DNA 배열의 정확히 어떤 부분이 유전자로 구성되는지 정의하는 것은 어렵다.[7][48]유전자 및 그 조절 영역을 근접시키기 위해 개입하는 DNA가 루프아웃될 수 있기 때문에 강화제와 같은 유전자의 조절 영역은 반드시 선형 분자상의 코드 배열에 가까울 필요는 없다.비슷하게, 유전자의 인트론은 엑손보다 훨씬 클 수 있다.조절 영역은 완전히 다른 염색체 위에 있을 수 있고, 한 염색체의 조절 영역이 [49][50]다른 염색체의 표적 유전자와 접촉할 수 있도록 하기 위해 전이적으로 작동할 수 있습니다.
분자 유전학의 초기 연구는 하나의 유전자가 하나의 단백질을 만든다는 개념을 제시했다.이 개념은 1941년 조지 비들(George Beadle)과 에드워드 테이텀(Edward Tatum)이 뉴로스포라 크라사 [51]균의 돌연변이를 실험한 영향력 있는 논문에서 나왔습니다.Neurospora 연구의 초기 동료인 Norman Horowitz는 2004년에 "이 실험들은 비들과 테이텀이 생화학적 유전학이라고 부르는 것의 과학을 만들었다"고 회상했다.사실 그들은 분자 유전학이 된 것과 그에 따른 모든 발전의 시작점이라는 것이 입증되었다.[52]하나의 유전자 1 단백질 개념은 mRNA가 트랜스 [9][53][54]스플라이싱에 의해 연결된 게놈 전체에 걸쳐 짧은 섹션으로 분할된 코드 배열과 대체 스플라이싱에 의해 다중 단백질을 부호화할 수 있는 유전자의 발견 이후 정제되었다.
유전자가 잠재적으로 중복될 수 있는 기능성 [18]산물의 일관성 있는 세트를 코드하는 게놈 배열의 결합으로 정의되는 이러한 다양한 현상의 복잡성을 포함하기 위해 광범위한 연산 정의가 때때로 사용된다.이 정의는 유전자 관련 [18]영역으로 분류되는 조절 요소와 함께 특정 DNA 위치가 아닌 기능 산물(단백질 또는 RNA)에 따라 유전자를 분류합니다.
유전 인자들 사이의 Overlap
또한 유전자가 같은 DNA 염기서열과 겹치는 것이 가능하고 구별되지만 중복되는 [55]유전자로 간주될 수 있다.중복 유전자의 현재 정의는 진핵생물, 원핵생물, 바이러스마다 다르다.진핵생물에서 그들은 최근 "적어도 하나의 뉴클레오티드가 두 개 이상의 유전자의 1차 전사의 가장 바깥쪽 경계 사이에 공유될 때, 중복되는 지점에서 DNA 염기 돌연변이가 중복되는 모든 유전자의 전사에 영향을 미칠 것"으로 정의되었다.원핵생물과 바이러스에서 그것들은 최근 "두 유전자의 코드 배열이 동일하거나 반대되는 [55]가닥에서 뉴클레오티드를 공유할 때"로 정의되었다.
유전자 발현
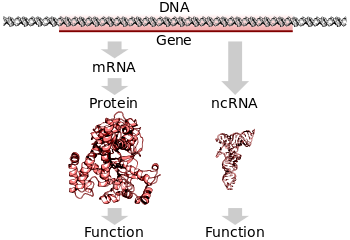
모든 유기체에서, 유전자의 DNA에 암호화된 정보를 읽고 그것이 지정하는 단백질을 생산하기 위해서는 두 단계가 필요하다.첫째, 유전자의 DNA는 메신저 RNA로 전사된다.[32]: 6.1 둘째, mRNA는 단백질로 [32]: 6.2 변환됩니다.RNA 코드화 유전자는 여전히 첫 단계를 거쳐야 하지만 [56]단백질로 변환되지는 않는다.RNA나 단백질의 생물학적으로 기능하는 분자를 만드는 과정을 유전자 발현이라고 하고, 그 결과 생기는 분자를 유전자 생성물이라고 한다.
코돈
유전자 DNA의 뉴클레오티드 배열은 유전자 코드를 통해 단백질의 아미노산 배열을 특정한다.코돈으로 알려진 세 개의 뉴클레오티드가 각각 특정 아미노산에 [32]: 6 해당합니다.1961년 박테리오파지 T4의[57] rIIB 유전자의 프레임시프트 돌연변이를 사용하여 각 아미노산에 대한 DNA 코드의 세 가지 염기서열 원리가 입증되었다(크릭, 브레너 등 참조). 실험)을 실시합니다.
또한 "시작 코돈"과 3개의 "정지 코돈"은 단백질 코딩 영역의 시작과 끝을 나타냅니다.64개의 가능한 코돈(3개의 위치에 각각 4개의 가능한 뉴클레오티드, 따라서 4개의3 가능한 코돈)과 20개의 표준 아미노산이 있습니다. 따라서 코드는 중복되고 여러 코돈은 동일한 아미노산을 지정할 수 있습니다.코돈과 아미노산 사이의 대응은 알려진 모든 [58]생물 중에서 거의 보편적이다.
문자 변환
전사는 메신저 RNA로 알려진 단일 가닥 RNA 분자를 생성하는데, 그 뉴클레오티드 배열은 [32]: 6.1 전사가 이루어진 DNA와 상호 보완적이다.mRNA는 DNA 유전자와 최종 단백질 생성물 사이의 중간체 역할을 한다.그 유전자의 DNA는 상보적인 mRNA를 생성하기 위한 템플릿으로 사용된다.mRNA는 템플릿 가닥의 보완체로 합성되기 때문에 유전자 코드 가닥의 염기서열과 일치한다.전사는 RNA 중합효소라는 효소에 의해 이루어지며, RNA 중합효소는 3'에서 5' 방향으로 템플릿 가닥을 읽고 5'에서 3'로 RNA를 합성한다.전사를 시작하기 위해 중합효소는 먼저 유전자의 프로모터 영역을 인식하고 결합한다.따라서 유전자 조절의 주요 메커니즘은 중합효소를 물리적으로 차단하는 억제제 분자에 의한 엄격한 결합이나 프로모터 영역에 접근할 [32]: 7 수 없도록 DNA를 구성함으로써 프로모터 영역을 차단하거나 격리하는 것이다.
원핵생물에서, 전사는 세포질에서 일어난다; 매우 긴 전사의 경우, 3' 말단이 여전히 전사되는 동안 RNA의 5' 말단에서 번역을 시작할 수 있다.진핵생물에서, 전사는 세포의 DNA가 저장되는 핵에서 일어난다.중합효소에 의해 생성된 RNA 분자는 1차 전사물로 알려져 있으며 번역을 위해 세포질로 내보내기 전에 전사 후 수정을 거친다.수행된 변형 중 하나는 단백질을 부호화하지 않는 전사 영역 내 배열인 인트론의 스플라이싱이다.대체 스플라이싱 메커니즘은 동일한 유전자의 성숙한 전사를 다른 염기서열을 가지며 따라서 다른 단백질을 코딩하는 결과를 가져올 수 있다.이것은 진핵 세포에서 조절의 주요 형태이고 또한 일부 원핵 [32]: 7.5 [59]생물에서도 발생합니다.
번역.
번역은 성숙한 mRNA 분자가 새로운 [32]: 6.2 단백질을 합성하기 위한 템플릿으로 사용되는 과정이다.펩타이드 결합의 형성에 의해 성장하는 폴리펩타이드 사슬에 새로운 아미노산을 첨가하는 화학반응을 담당하는 리보솜, RNA의 큰 복합체 및 단백질에 의해 번역된다.유전자 코드는 전달 RNA라고 불리는 특수 RNA 분자들과의 상호작용을 통해 코돈이라고 불리는 단위로 한 번에 세 개의 뉴클레오티드를 읽는다.각 tRNA에는 mRNA에서 판독되는 코돈을 보완하는 안티코돈으로 알려진 세 개의 비쌍 염기가 있습니다.또한 tRNA는 상보 코돈에 의해 특정된 아미노산에 공유 결합된다.tRNA가 mRNA 가닥의 상보적 코돈에 결합할 때, 리보솜은 아미노 말단에서 카르복실 말단으로 합성되는 새로운 폴리펩타이드 사슬에 아미노산 화물을 부착합니다.합성하는 동안과 후에, 대부분의 새로운 단백질은 세포 [32]: 3 기능을 수행하기 전에 활성 3차원 구조로 접혀야 한다.
규정
발현에는 한정된 [32]: 7 자원이 필요하기 때문에 유전자는 제품이 필요할 때만 발현되도록 조절된다.세포는 외부 환경(예: 이용 가능한 영양소, 온도 및 기타 스트레스), 내부 환경(예: 세포 분열 주기, 신진대사, 감염 상태) 및 다세포 유기체의 경우 특정 역할에 따라 유전자 발현을 조절한다.유전자 발현은 전사 개시부터 RNA 처리, 단백질의 번역 후 변형까지 모든 단계에서 조절될 수 있다.대장균(lac operon)에서 유당 대사 유전자의 조절은 [60]1961년에 최초로 기술된 메커니즘이었다.
RNA유전자
전형적인 단백질 부호화 유전자는 우선 최종 단백질 [32]: 6.1 생성물의 제조 과정에서 중간체로서 RNA에 복사된다.다른 경우, RNA 분자는 리보솜 RNA와 전달 RNA의 합성과 같이 실제 기능적 산물이다. 리보자임으로 알려진 일부 RNA는 효소적 기능을 할 수 있으며, 마이크로RNA는 조절적 역할을 한다.그러한 RNA가 전사되는 DNA 배열을 비코드 RNA [56]유전자라고 한다.
일부 바이러스는 전체 게놈을 RNA 형태로 저장하고 DNA를 [61][62]전혀 포함하지 않는다.그들이 유전자를 저장하기 위해 RNA를 사용하기 때문에, 그들의 세포 숙주는 그들이 감염되자마자 그리고 [63]전사를 기다리는 지연 없이 그들의 단백질을 합성할 수 있다.반면, HIV와 같은 RNA 레트로바이러스는 단백질이 합성되기 전에 RNA에서 DNA로 게놈의 역전사를 요구한다.RNA 매개 후생유전 또한 식물에서 관찰되었고 [64]동물에서 매우 드물게 관찰되었다.
상속
유기체는 부모로부터 유전자를 물려받는다.무성 유기체는 단순히 부모의 게놈의 완전한 사본을 물려받았을 뿐이다.성 유기체는 [32]: 1 부모로부터 하나의 완전한 집합을 물려받기 때문에 각 염색체의 두 개의 복사본을 가지고 있다.
멘델 유전
멘델의 유전에 따르면, 유기체의 표현형의 변화(관찰 가능한 신체적, 행동적 특징)는 부분적으로 유전자형의 변화(특히 유전자 세트)에 기인한다.각 유전자는 다른 표현형을 발생시키는 다른 유전자(알레)의 배열로 특정한 특성을 지정한다.대부분의 진핵 생물들은 각각의 특징에 대해 두 개의 대립 유전자를 가지고 있는데, 하나는 각각의 [32]: 20 부모로부터 물려받습니다.
한 궤적의 대립 유전자는 우세하거나 열성일 수 있다; 지배적 대립 유전자는 동일한 특성에 대해 다른 대립 유전자와 짝을 지을 때 대응하는 표현형을 생성하는 반면, 열성 대립 유전자는 동일한 대립 유전자의 다른 복사본과 짝을 이룰 때만 대응하는 표현형을 생성한다.유기체의 유전자형을 알면 어떤 대립 유전자가 우세하고 어떤 대립 유전자가 열성인지 알 수 있습니다.예를 들어, 완두콩 식물의 키 큰 줄기를 지정하는 대립 유전자가 짧은 줄기를 지정하는 대립 유전자에 우세하다면, 한쪽 부모로부터 키 큰 대립 유전자와 다른 한쪽 부모로부터 키 작은 대립 유전자를 물려받는 완두콩 식물도 키 큰 줄기를 갖게 될 것이다.멘델의 연구는 대립 유전자가 배우자, 즉 생식세포의 생성에서 독립적으로 분류되어 다음 세대의 변화를 보장한다는 것을 증명했다.멘델 유전은 단일 유전자에 의해 결정되는 많은 특징들에 대한 좋은 모델로 남아있지만, 그것은 DNA 복제와 세포 [65][66]분열의 물리적 과정을 포함하지 않는다.
DNA복제와 세포분열
유기체의 성장, 발달, 번식은 세포 분열에 의존합니다; 단일 세포가 보통 동일한 두 개의 딸 세포로 분열하는 과정입니다.이것은 우선 DNA [32]: 5.2 복제라고 불리는 과정에서 게놈에 있는 모든 유전자의 복제를 필요로 한다.복제물은 DNA 중합효소라고 알려진 특수 효소에 의해 만들어지며, 이것은 템플릿 가닥으로 알려진 이중 나선 DNA의 한 가닥을 "읽어내고" 새로운 상보적인 가닥을 합성한다.DNA 이중나선은 염기쌍에 의해 결합되기 때문에, 한 가닥의 배열은 그 보체의 배열을 완전히 규정하기 때문에, 효소에 의해 한 가닥만 읽혀지면 충실한 복사가 된다.DNA 복제 과정은 반보수적이다. 즉, 각 딸 세포에 의해 유전되는 게놈의 복사본에는 원본 [32]: 5.2 DNA와 새로 합성된 DNA 가닥이 하나씩 포함되어 있다.
살아있는 세포의 DNA 복제 속도는 파지에 감염된 대장균의 파지 T4 DNA 신장률로 처음 측정되었고 매우 [67]빠른 것으로 밝혀졌다.37°C에서 DNA가 기하급수적으로 증가하는 동안, 신장 속도는 초당 749 뉴클레오티드였다.
DNA 복제가 완료된 후, 세포는 게놈의 두 복사본을 물리적으로 분리하여 두 개의 분리된 막 결합 [32]: 18.2 세포로 분할해야 한다.원핵생물에서 이것은 보통 각각의 원형 게놈이 세포막에 부착되고 세포질이 두 개의 막으로 묶인 부분으로 분열되도록 자극하면서 딸 세포로 분리되는 바이너리 핵분열이라고 불리는 비교적 간단한 과정을 통해 발생합니다.2분열은 진핵생물의 세포분열 속도에 비해 매우 빠르다.진핵 세포 분열은 세포 순환으로 알려진 더 복잡한 과정이다; DNA 복제는 염색체를 분리하고 세포질을 분열시키는 과정이 M [32]: 18.1 단계 동안 일어난다.
분자 유전
한 세대의 세포에서 다음 세대로 유전 물질의 복제와 전달은 분자 유전의 기초이자 유전자의 고전과 분자 사진 사이의 연결이다.유기체는 부모의 특성을 물려받는다. 왜냐하면 자손의 세포는 부모의 세포에 유전자의 복사를 포함하고 있기 때문이다.무성 생식 유기체에서 자손은 부모 유기체의 유전자 복제 또는 복제품이 될 것이다.성적으로 번식하는 유기체에서, 감수 분열이라고 불리는 특별한 형태의 세포 분열은 생식세포 또는 생식세포라고 불리는 세포들을 생산하는데, 이것은 반수체이거나 [32]: 20.2 각 유전자의 오직 한 개의 복사본만을 포함합니다.암컷에 의해 생성되는 생식체는 난자 또는 난자라고 불리며, 수컷에 의해 생성되는 생식체는 정자라고 불린다.두 개의 생식체가 융합하여 두 세트의 유전자를 가진 단일 세포인 이배체 수정란을 형성하고, 각 유전자의 복사본은 어머니로부터, 다른 하나는 [32]: 20 아버지로부터 얻는다.
감수 세포 분열의 과정 동안, 유전자 재조합 또는 교차라고 불리는 사건은 때때로 일어날 수 있는데, 이것은 하나의 염색체 상의 DNA의 길이가 대응하는 상동성 비 자매 염색체 상의 DNA의 길이와 교환된다.이것은 다른 방법으로 연결된 대립 유전자의 [32]: 5.5 재분배를 초래할 수 있다.멘델의 독립적 배합 원칙은 각각의 특징에 대한 부모의 두 유전자가 독립적으로 배우자로 분류될 것이라고 주장한다; 그것은 유기체가 다른 특징에 대해 유전하는 것과 관련이 없다.이것은 사실 같은 염색체에 존재하지 않거나 같은 염색체에 서로 매우 멀리 있는 유전자에만 해당된다.두 유전자가 같은 염색체에 가까울수록, 그들은 배우자에 더 밀접하게 연관되어 더 자주 함께 나타날 것입니다.[68]매우 가까운 유전자들은 본질적으로 절대 분리되지 않는다.[68] 왜냐하면 그들 사이에 교차점이 발생할 가능성이 극히 낮기 때문이다.
분자 진화
돌연변이
DNA 복제는 대부분 매우 정확하지만 오류([32]: 7.6 변형)가 발생합니다.진핵세포의 오류율은 [69][70]복제당 뉴클레오티드당 10만큼 낮을−8 수 있는 반면, 일부 RNA 바이러스의 경우 [71]10만큼 높을−3 수 있습니다.이는 각 세대, 각 인간 게놈이 1-2개의 새로운 [71]돌연변이를 축적한다는 것을 의미한다.작은 돌연변이는 DNA 복제와 DNA 손상의 여파에 의해 발생할 수 있으며 단일 염기가 변경되는 점 돌연변이와 단일 염기가 삽입 또는 삭제되는 프레임시프트 돌연변이를 포함한다.이 돌연변이들 중 하나는 잘못된 감각으로 유전자를 바꿀 수 있고, 다른 아미노산을 인코딩하기 위해 코돈을 [72]바꾼다.더 큰 돌연변이는 염색체의 큰 부분의 복제, 삭제, 재배열 또는 반전을 포함한 염색체 이상을 일으키는 재조합 오류에 의해 야기될 수 있다.또한 DNA 복구 메커니즘은 분자의 물리적 손상을 복구할 때 돌연변이 오류를 일으킬 수 있습니다.이중 스트랜드 [32]: 5.4 파손을 복구하는 경우 등 정확한 복사본을 복원하는 것보다 돌연변이가 있더라도 복구가 생존에 더 중요합니다.
한 종의 개체군에 유전자에 대한 여러 가지 다른 대립 유전자가 존재할 때, 그것은 다형성이라고 불립니다.대부분의 다른 대립 유전자는 기능적으로 동등하지만, 일부 대립 유전자는 다른 표현형 특성을 발생시킬 수 있습니다.유전자의 가장 흔한 대립 유전자는 야생형이라고 불리며, 희귀한 대립 유전자는 돌연변이라고 불립니다.집단 내 다른 대립 유전자의 상대적인 빈도의 유전적 변화는 자연 선택과 유전적 [73]표류 둘 다에 기인한다.야생형 대립 유전자는 반드시 덜 흔한 대립 유전자의 조상일 뿐만 아니라 더 적합한 것도 아닙니다.
유전자 내의 대부분의 돌연변이는 중립적이며, 유기체의 표현형(침묵 돌연변이)에 영향을 미치지 않는다.몇몇 돌연변이는 여러 코돈이 동일한 아미노산을 암호화하기 때문에 아미노산 염기서열을 바꾸지 않는다.다른 돌연변이는 아미노산 배열 변화를 초래할 경우 중성적일 수 있지만 단백질은 여전히 새로운 아미노산(예: 보존 돌연변이)과 유사하게 기능한다.그러나 많은 돌연변이는 유해하거나 심지어 치명적이며 자연선택에 의해 개체군으로부터 제거된다.유전적 장애는 유해한 돌연변이의 결과이며, 영향을 받은 개인의 자발적 돌연변이에 의한 것일 수도 있고, 유전될 수도 있습니다.마지막으로, 돌연변이의 작은 부분은 유기체의 적합성을 향상시키며, 그들의 방향 선택이 [32]: 7.6 적응 진화를 이끌기 때문에 진화에 매우 중요합니다.
시퀀스 호몰로지
가장 최근의 공통 조상을 가진 유전자와, 따라서 공통의 진화 조상을 가진 유전자는 [74]호몰로지로 알려져 있다.이 유전자들은 평행 유전자로 알려진 유기체의 게놈 내의 유전자 복제로부터 나타나거나, 또는 그것들이 직교 [32]: 7.6 유전자로 알려진 분화 사건 이후 유전자의 분리의 결과이며, 종종 관련된 유기체에서 동일하거나 유사한 기능을 수행한다.종종 직교 유전자의 기능은 차이는 [75][76]미미하지만 평행 유전자의 기능보다 더 유사하다고 가정한다.
유전자 간의 관계는 DNA의 [32]: 7.6 배열 정렬을 비교함으로써 측정될 수 있다.상동 유전자 사이의 배열 유사성의 정도를 보존된 배열이라고 한다.유전자의 배열에 대한 대부분의 변화는 기능에 영향을 미치지 않기 때문에 유전자는 중성 분자 진화에 의해 시간이 지남에 따라 돌연변이를 축적합니다.게다가, 유전자에 대한 어떤 선택도 다른 속도로 유전자 서열을 분산시킬 것이다.안정화 선택 중인 유전자는 구속되기 때문에 더 느리게 변하는 반면 방향 선택 중인 유전자는 더 [77]빠르게 염기서열이 변화한다.유전자 간의 배열 차이는 그 유전자들이 어떻게 진화했는지 그리고 그들이 유래한 유기체들이 어떻게 [78][79]관련되어 있는지를 연구하기 위해 계통학 분석에 사용될 수 있다.
새로운 유전자의 기원
진핵생물 계통에서 새로운 유전자의 가장 흔한 원천은 유전자 복제인데,[80][81] 이것은 게놈에 있는 기존 유전자의 복제 번호의 변화를 일으킨다.그 결과 생성된 유전자(패럴로그)는 순서와 기능에 따라 분리될 수 있다.이렇게 형성된 유전자 세트는 유전자 패밀리를 구성한다.유전자 복제와 가족 내에서의 손실은 흔하고 진화적 생물 [82]다양성의 주요 원천이다.때때로, 유전자 복제는 유전자의 기능하지 않는 복사를 야기할 수도 있고 기능하지 않는 복사는 기능 상실을 초래하는 돌연변이의 영향을 받을 수도 있다. 그러한 기능하지 않는 유전자는 [32]: 7.6 의사 유전자라고 불린다.
기존 유전자와 배열이 유사하지 않은 '오르판' 유전자는 복제 유전자보다 흔하지 않다.인간 게놈에는 1860개[84] 정도의[83] 유전자가 포함되어 있으며, 인간 이외에는 식별 가능한 호몰로지가 없다.고아 유전자는 주로 이전에 코드화되지 않은 배열로부터의 새로운 출현이나 유전자 복제에 따른 급격한 배열 변화로 [85]인해 원래의 관계가 검출되지 않게 된다.데노보 유전자는 전형적으로 대부분의 진핵생물 유전자보다 짧고 구조가 단순하며, 침입자는 거의 [80]없다.오랜 진화 기간 동안, de novo 유전자의 탄생은 분류학적으로 제한된 유전자 [86]패밀리의 중요한 부분에 책임이 있을 수 있습니다.
수평적 유전자 이동은 유전자 물질이 번식 이외의 메커니즘을 통해 전달되는 것을 말한다.이 메커니즘은 원핵생물에서 새로운 유전자의 공통적인 원천이며, 때때로 유전자 [87]복제보다 유전적 변이에 더 많이 기여한다고 생각됩니다.그것은 항생제 내성, 독성, 그리고 적응 대사 [36][88]기능을 확산시키는 일반적인 수단이다.진핵생물에서는 수평적 유전자 이동이 드물지만 박테리아 [89][90]기원의 유전자를 포함하는 원생생물 및 조류 게놈의 예가 확인되었습니다.
게놈
게놈은 유기체의 총 유전 물질이며 유전자와 비암호화 [91]염기서열 모두를 포함한다.진핵생물 유전자는 [92]FINDER를 사용하여 주석을 달 수 있습니다.
유전자수
게놈 크기와 그것이 암호화하는 유전자의 수는 유기체마다 매우 다양하다.가장 작은 게놈은 [101]바이러스와 바이로이드에서 발생한다.[102]반대로, 식물은 매우 큰 [103]게놈을 가질 수 있으며, 쌀은 46,000개 이상의 단백질 코드 [97]유전자를 가지고 있다.총 단백질 코드 유전자의 수는 5백만 [104]개로 추정된다.
인간 게놈의 염기쌍 수는 1960년대부터 알려져 왔지만 유전자의 정의에 따라 추정 유전자 수가 달라지고 검출 방법도 개선됐다.인간 유전자의 수에 대한 초기 이론적인 예측은 200만 [105]개에 달했다.초기 실험 측정 결과 50,000~100,000개의 전사 유전자(표현 배열 태그)[106]가 있는 것으로 나타났다.그 후, 인간 게놈 프로젝트의 염기서열 분석 결과, 이러한 성분의 많은 부분이 같은 유전자의 대체 변종임을 알 수 있었고, 총 단백질 코드 유전자의 수는 미토콘드리아 [98]게놈에 13개의 유전자가 암호화되어 약 20,000개로[100] 수정되었다.GENCODE 주석 프로젝트에서는 그 견적은 계속 [107]19,000까지 떨어졌습니다.인간 게놈 중 1~2%만이 단백질 코드 배열로 [108]구성되고 나머지는 인트론, 역트랜스포존,[108][109] 비코드 RNA와 같은 '비코드' DNA이다.모든 다세포 유기체는 몸의 각 세포에 모든 유전자를 가지고 있지만 모든 유전자가 모든 세포에서 기능을 하는 것은 아니다.
필수 유전자
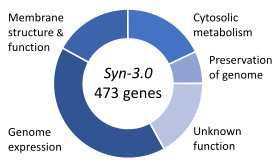
필수 유전자는 유기체의 [111]생존에 중요하다고 생각되는 유전자 세트이다.이 정의는 모든 관련 영양소의 풍부한 가용성과 환경적 스트레스가 없다고 가정한다.유기체 유전자의 극히 일부만이 필수적이다.박테리아에서는 250~400개의 유전자가 대장균과 아틸루스균에 필수적이며,[112][113][114] 이는 유전자의 10% 미만이다.이들 유전자의 절반은 두 유기체의 정형어이고 단백질 [114]합성에 주로 관여한다.발아 효모인 사카로미세스 세레비시아에는 필수 유전자의 수가 1000개([115]유전자의 20%)로 약간 더 많다.진핵생물에서는 그 수를 측정하기가 더 어렵지만, 생쥐와 인간은 약 2000개의 필수 유전자를 가지고 있는 것으로 추정됩니다(유전자의 [116]약 10%).합성 유기체인 Syn 3는 473개의 필수 유전자와 (빠른 성장에 필요한) 준 필수 유전자로 구성된 최소한의 게놈을 가지고 있지만,[110] 149개의 기능은 알려지지 않았다.
필수 유전자는 (기본 세포 기능에 중요한) 하우스키핑 유전자뿐만 아니라 유기체의 발달이나 라이프 [118]사이클에서 다른 시기에 [117]발현되는 유전자를 포함한다.하우스키핑 유전자는 비교적 일정한 수준에서 발현되기 때문에 유전자 발현을 분석할 때 실험적인 대조군으로 사용된다.
유전자 및 게놈 명명법
유전자 명명법은 인간 게놈 기구의 위원회인 HUGO Geneclature Committee(HGNC)에 의해 승인된 유전자 이름과 기호(단축형 약어)의 형태로 각 알려진 인간 유전자에 대해 확립되었으며, HGNC에 의해 유지되는 데이터베이스를 통해 접근할 수 있으며, 각각의 기호는 고유하도록 선택되었다.하나의 기호(단, 승인된 기호가 변경될 수 있음)기호는 유전자 패밀리의 다른 멤버 및 다른 종, 특히 마우스가 공통 모델 [119]유기체로서의 역할을 하기 때문에 상동성과 일치하는 것이 바람직하다.
유전공학
유전공학은 생명공학을 통해 유기체의 게놈을 변형하는 것이다.1970년대 이후,[120] 유기체의 유전자를 특별히 추가, 제거, 편집하기 위한 다양한 기술들이 개발되어 왔다.최근 개발된 게놈 공학적 기술은 염색체에서 표적 DNA 수복을 만들기 위해 조작된 핵산가수분해효소 효소를 사용한다.[121][122][123][124]관련된 용어는 때때로 유기체의 [125]광범위한 유전자 공학을 지칭하기 위해 사용된다.
유전자 공학은 이제 모델 유기체의 일상적인 연구 도구이다.예를 들어, 유전자는 박테리아에 쉽게[126] 추가되며, 특정 유전자의 기능이 파괴된 녹아웃 생쥐의 혈통은 그 유전자의 [127][128]기능을 조사하기 위해 사용된다.많은 유기체들이 농업, 산업 생명공학, 의학 분야에서의 응용을 위해 유전적으로 변형되었다.
다세포 유기체의 경우 전형적으로 배아는 성체 유전자 변형 [129]유기체로 성장하도록 조작된다.그러나 성체 유기체의 세포 게놈은 유전자 치료 기술을 이용해 유전자 질환을 치료해 편집할 수 있다.
「 」를 참조해 주세요.
레퍼런스
인용문
- ^ a b "1909: The Word Gene Coined". genome.gov. Retrieved 8 March 2021. 윌헬름 요한센은 유전의 단위인 멘델을 설명하기 위해 유전자라는 단어를 만들었다.."
- ^ Roth SC (July 2019). "What is genomic medicine?". Journal of the Medical Library Association. University Library System, University of Pittsburgh. 107 (3): 442–448. doi:10.5195/jmla.2019.604. PMC 6579593. PMID 31258451.
- ^ "What is a gene?: MedlinePlus Genetics". MedlinePlus. 17 September 2020. Retrieved 4 January 2021.
- ^ Hirsch ED (2002). The new dictionary of cultural literacy. Boston: Houghton Mifflin. ISBN 0-618-22647-8. OCLC 50166721.
- ^ "Studying Genes". nigms.nih.gov. Retrieved 15 January 2021.
- ^ Elston RC, Satagopan JM, Sun S (2012). "Genetic terminology". Statistical Human Genetics. Methods in Molecular Biology. Vol. 850. Humana Press. pp. 1–9. doi:10.1007/978-1-61779-555-8_1. ISBN 978-1-61779-554-1. PMC 4450815. PMID 22307690.
- ^ a b Gericke N, Hagberg M (5 December 2006). "Definition of historical models of gene function and their relation to students' understanding of genetics". Science & Education. 16 (7–8): 849–881. Bibcode:2007Sc&Ed..16..849G. doi:10.1007/s11191-006-9064-4. S2CID 144613322.
- ^ Pearson H (May 2006). "Genetics: what is a gene?". Nature. 441 (7092): 398–401. Bibcode:2006Natur.441..398P. doi:10.1038/441398a. PMID 16724031. S2CID 4420674.
- ^ a b c Pennisi E (June 2007). "Genomics. DNA study forces rethink of what it means to be a gene". Science. 316 (5831): 1556–7. doi:10.1126/science.316.5831.1556. PMID 17569836. S2CID 36463252.
- ^ a b Johannsen W (1909). Elemente der exakten Erblichkeitslehre [Elements of the exact theory of heredity] (in German). Jena, Germany: Gustav Fischer. p. 124. 페이지 124부터 : "Dieses et was" in den Gameten bezw. der Zygote에서, … – kurz는 wir eben Gene nennen wollen – bedingt sind였다." (이 생식체 또는 접합체에서의 "뭔가"는 보통 상당히 애매한 용어 Anlagen [계획, 대략적인 배치]에 의해 불리고 있다.많은 다른 용어들이 제시되어 왔는데, 불행하게도 대부분 특정한 가설적인 의견들과 더 밀접하게 관련되어 있다.다윈에 의해 소개된 "판게네"라는 단어는 아마도 앤라겐 대신 가장 자주 사용된다.However, the word "pangene" was not well chosen, as it is a compound word containing the roots pan (the neuter form of Πας all, every) and gen (from γί-γ(ε)ν-ομαι, to become).여기서는 후자의 의미만 고려됩니다; 발달하는 유기체의 특성이 결정될 수 있거나 배우자의 "뭔가"에 의해 영향을 받는다는 기본적인 생각만 표현되어야 합니다.이 "뭔가"의 본질에 대한 어떠한 가설도 그것에 의해 가정되거나 뒷받침되어서는 안 된다.그렇기 때문에 다윈의 유명한 단어에서 마지막 음절 gen을 분리해서 사용하는 것이 가장 간단해 보입니다. 이 단어만으로도 우리는 가난하고 애매한 단어 Anlage를 대체할 수 있습니다.따라서 우리는 단순히 "pangene"과 "pangenes"에 대해 "gene"과 "genes"라고 말할 것이다.유전자라는 단어는 어떤 가설로부터도 완전히 자유롭다; 그것은 어떤 경우에도 유기체의 많은 특징들이 특정되고, 분리 가능하며, 따라서 독립적인 "조건", "기반", "계획"에 의해 결정된다는 확립된 사실만을 표현한다 – 간단히 말해서, 우리가 유전자라고 부르고 싶은 것이다.)
- ^ Noble D (September 2008). "Genes and causation". Philosophical Transactions. Series A, Mathematical, Physical, and Engineering Sciences. 366 (1878): 3001–15. Bibcode:2008RSPTA.366.3001N. doi:10.1098/rsta.2008.0086. PMID 18559318.
- ^ "Blending Inheritance - an overview ScienceDirect Topics".
- ^ "genesis". Oxford English Dictionary (Online ed.). Oxford University Press. (가입 또는 참여기관 회원가입 필요)
- ^ Magner LN (2002). A History of the Life Sciences (Third ed.). Marcel Dekker, CRC Press. p. 371. ISBN 978-0-203-91100-6.
- ^ Henig RM (2000). The Monk in the Garden: The Lost and Found Genius of Gregor Mendel, the Father of Genetics. Boston: Houghton Mifflin. pp. 1–9. ISBN 978-0395-97765-1.
- ^ a b de Vries H (1889). Intracellulare Pangenese [Intracellular Pangenesis] (in German). Translated by Gager CS. Jena: Verlag von Gustav Fischer. 1908년 시카고 오픈 코트 출판사에 의해 독일어에서 영어로 번역됨, 1910년
- ^ Bateson W.(1906) "유전자 연구의 진보" 제3회 유전학 국제회의 1906년 W. Wilks, ed.런던, 영국:왕립원예학회, 90~97페이지.91페이지부터: "…과학 그 자체는 아직 이름조차 없다.[즉, 식물의 사육과 교배 연구] 그리고 우리는 우리의 추구를 누적되고 종종 오해의 소지가 있는 주변환경에 의해서만 설명할 수 있다.이 난관에 대처하기 위해 나는 이 의회의 검토를 위해 유전학이라는 용어를 제안하고 싶다.이것은 유전과 변이 현상의 해명에 우리의 노력이 바쳐지고 있음을 충분히 나타낸다.즉, 진화론자와 계통론자의 이론적인 문제에 암묵적으로 관여하고 있다.동물이든 식물이든 사육자의 실제 문제에 적용할 수 있습니다."
- ^ a b c Gerstein MB, Bruce C, Rozowsky JS, Zheng D, Du J, Korbel JO, et al. (June 2007). "What is a gene, post-ENCODE? History and updated definition". Genome Research. 17 (6): 669–81. doi:10.1101/gr.6339607. PMID 17567988.
- ^ 에이버리 OT, 매 클라우드 고객 관리와 맥카트니 M(2월 1944년)."그 화학 자연 약물 유도 변화 Pneumococcal 유형에 관한 연구:변화의 Desoxyribonucleic, 아미노산, 단지 격리된 FromPneumococcus 3에 의해 귀납 법".그 실험 의학 저널. 79(2):137–58. doi:10.1084/jem.79.2.137. 1.2135445.PMID 19871359.Reprint:에이버리 OT, 매 클라우드 고객 관리와 맥카트니 M(2월 1979년)."은 물질의 화학적 성질에 관한 연구 폐렴 구균의 종류의 변환을 유도하는.변화의 회복이desoxyribonucleic 산성 일부pneumococcus형 III"로부터 격리에 의해 Inductions.그 실험 의학 저널.149(2):297–326. doi:10.1084/jem.149.2.297. 1.2184805.PMID 33226.
- ^ Hershey AD, Chase M (May 1952). "Independent functions of viral protein and nucleic acid in growth of bacteriophage". The Journal of General Physiology. 36 (1): 39–56. doi:10.1085/jgp.36.1.39. PMC 2147348. PMID 12981234.
- ^ Judson H (1979). The Eighth Day of Creation: Makers of the Revolution in Biology. Cold Spring Harbor Laboratory Press. pp. 51–169. ISBN 978-0-87969-477-7.
- ^ Watson JD, Crick FH (April 1953). "Molecular structure of nucleic acids; a structure for deoxyribose nucleic acid" (PDF). Nature. 171 (4356): 737–8. Bibcode:1953Natur.171..737W. doi:10.1038/171737a0. PMID 13054692. S2CID 4253007.
- ^ Benzer S (June 1955). "Fine Structure of a Genetic Region in Bacteriophage". Proceedings of the National Academy of Sciences of the United States of America. 41 (6): 344–54. Bibcode:1955PNAS...41..344B. doi:10.1073/pnas.41.6.344. PMC 528093. PMID 16589677.
- ^ Benzer S (November 1959). "On the Topology of the Genetic Fine Structure". Proceedings of the National Academy of Sciences of the United States of America. 45 (11): 1607–20. Bibcode:1959PNAS...45.1607B. doi:10.1073/pnas.45.11.1607. PMC 222769. PMID 16590553.
- ^ Min Jou W, Haegeman G, Ysebaert M, Fiers W (May 1972). "Nucleotide sequence of the gene coding for the bacteriophage MS2 coat protein". Nature. 237 (5350): 82–8. Bibcode:1972Natur.237...82J. doi:10.1038/237082a0. PMID 4555447. S2CID 4153893.
- ^ Sanger F, Nicklen S, Coulson AR (December 1977). "DNA sequencing with chain-terminating inhibitors". Proceedings of the National Academy of Sciences of the United States of America. 74 (12): 5463–7. Bibcode:1977PNAS...74.5463S. doi:10.1073/pnas.74.12.5463. PMC 431765. PMID 271968.
- ^ Adams JU (2008). "DNA Sequencing Technologies". Nature Education Knowledge. SciTable. Nature Publishing Group. 1 (1): 193.
- ^ Huxley J (1942). Evolution: the Modern Synthesis. Cambridge, Massachusetts: MIT Press. ISBN 978-0262513661.
- ^ Williams GC (2001). Adaptation and Natural Selection a Critique of Some Current Evolutionary Thought (Online ed.). Princeton: Princeton University Press. ISBN 9781400820108.
- ^ Dawkins R (1977). The selfish gene (Repr. (with corr.) ed.). London: Oxford University Press. ISBN 978-0-19-857519-1.
- ^ Dawkins R (1989). The extended phenotype (Paperback ed.). Oxford: Oxford University Press. ISBN 978-0-19-286088-0.
- ^ a b c d e f g h i j k l m n o p q r s t u v w x y z aa ab ac ad ae af ag ah ai aj ak Alberts B, Johnson A, Lewis J, Raff M, Roberts K, Walter P (2002). Molecular Biology of the Cell (Fourth ed.). New York: Garland Science. ISBN 978-0-8153-3218-3.
- ^ Stryer L, Berg JM, Tymoczko JL (2002). Biochemistry (5th ed.). San Francisco: W.H. Freeman. ISBN 978-0-7167-4955-4.
- ^ Bolzer A, Kreth G, Solovei I, Koehler D, Saracoglu K, Fauth C, et al. (May 2005). "Three-dimensional maps of all chromosomes in human male fibroblast nuclei and prometaphase rosettes". PLOS Biology. 3 (5): e157. doi:10.1371/journal.pbio.0030157. PMC 1084335. PMID 15839726.

- ^ Braig M, Schmitt CA (March 2006). "Oncogene-induced senescence: putting the brakes on tumor development". Cancer Research. 66 (6): 2881–4. doi:10.1158/0008-5472.CAN-05-4006. PMID 16540631.
- ^ a b Bennett PM (March 2008). "Plasmid encoded antibiotic resistance: acquisition and transfer of antibiotic resistance genes in bacteria". British Journal of Pharmacology. 153 (Suppl 1): S347-57. doi:10.1038/sj.bjp.0707607. PMC 2268074. PMID 18193080.
- ^ International Human Genome Sequencing Consortium (October 2004). "Finishing the euchromatic sequence of the human genome". Nature. 431 (7011): 931–45. Bibcode:2004Natur.431..931H. doi:10.1038/nature03001. PMID 15496913.
- ^ a b Shafee, Thomas; Lowe, Rohan (2017). "Eukaryotic and prokaryotic gene structure". WikiJournal of Medicine. 4 (1). doi:10.15347/wjm/2017.002. ISSN 2002-4436.
- ^ Mortazavi A, Williams BA, McCue K, Schaeffer L, Wold B (July 2008). "Mapping and quantifying mammalian transcriptomes by RNA-Seq". Nature Methods. 5 (7): 621–8. doi:10.1038/nmeth.1226. PMID 18516045. S2CID 205418589.
- ^ Pennacchio LA, Bickmore W, Dean A, Nobrega MA, Bejerano G (April 2013). "Enhancers: five essential questions". Nature Reviews. Genetics. 14 (4): 288–95. doi:10.1038/nrg3458. PMC 4445073. PMID 23503198.
- ^ Maston GA, Evans SK, Green MR (2006). "Transcriptional regulatory elements in the human genome". Annual Review of Genomics and Human Genetics. 7: 29–59. doi:10.1146/annurev.genom.7.080505.115623. PMID 16719718.
- ^ Mignone F, Gissi C, Liuni S, Pesole G (28 February 2002). "Untranslated regions of mRNAs". Genome Biology. 3 (3): REVIEWS0004. doi:10.1186/gb-2002-3-3-reviews0004. PMC 139023. PMID 11897027.
- ^ Bicknell AA, Cenik C, Chua HN, Roth FP, Moore MJ (December 2012). "Introns in UTRs: why we should stop ignoring them". BioEssays. 34 (12): 1025–34. doi:10.1002/bies.201200073. PMID 23108796. S2CID 5808466.
- ^ Shkurin A, Hughes TR (June 2021). "Known sequence features can explain half of all human gene ends". NAR Genomics and Bioinformatics. 3 (2): lqab042. doi:10.1093/nargab/lqab042. PMC 8176999. PMID 34104882.
- ^ Salgado H, Moreno-Hagelsieb G, Smith TF, Collado-Vides J (June 2000). "Operons in Escherichia coli: genomic analyses and predictions". Proceedings of the National Academy of Sciences of the United States of America. 97 (12): 6652–7. Bibcode:2000PNAS...97.6652S. doi:10.1073/pnas.110147297. PMC 18690. PMID 10823905.
- ^ Blumenthal T (November 2004). "Operons in eukaryotes". Briefings in Functional Genomics & Proteomics. 3 (3): 199–211. doi:10.1093/bfgp/3.3.199. PMID 15642184.
- ^ Jacob F, Monod J (June 1961). "Genetic regulatory mechanisms in the synthesis of proteins". Journal of Molecular Biology. 3 (3): 318–56. doi:10.1016/S0022-2836(61)80072-7. PMID 13718526.
- ^ Kellis M, Wold B, Snyder MP, Bernstein BE, Kundaje A, Marinov GK, et al. (April 2014). "Defining functional DNA elements in the human genome". Proceedings of the National Academy of Sciences of the United States of America. 111 (17): 6131–8. Bibcode:2014PNAS..111.6131K. doi:10.1073/pnas.1318948111. PMC 4035993. PMID 24753594.
- ^ Spilianakis CG, Lalioti MD, Town T, Lee GR, Flavell RA (June 2005). "Interchromosomal associations between alternatively expressed loci". Nature. 435 (7042): 637–45. Bibcode:2005Natur.435..637S. doi:10.1038/nature03574. PMID 15880101. S2CID 1755326.
- ^ Williams A, Spilianakis CG, Flavell RA (April 2010). "Interchromosomal association and gene regulation in trans". Trends in Genetics. 26 (4): 188–97. doi:10.1016/j.tig.2010.01.007. PMC 2865229. PMID 20236724.
- ^ Beadle GW, Tatum EL (November 1941). "Genetic Control of Biochemical Reactions in Neurospora". Proceedings of the National Academy of Sciences of the United States of America. 27 (11): 499–506. Bibcode:1941PNAS...27..499B. doi:10.1073/pnas.27.11.499. PMC 1078370. PMID 16588492.
- ^ Horowitz NH, Berg P, Singer M, Lederberg J, Susman M, Doebley J, Crow JF (January 2004). "A centennial: George W. Beadle, 1903-1989". Genetics. 166 (1): 1–10. doi:10.1534/genetics.166.1.1. PMC 1470705. PMID 15020400.
- ^ Marande W, Burger G (October 2007). "Mitochondrial DNA as a genomic jigsaw puzzle". Science. AAAS. 318 (5849): 415. Bibcode:2007Sci...318..415M. doi:10.1126/science.1148033. PMID 17947575. S2CID 30948765.
- ^ Parra G, Reymond A, Dabbouseh N, Dermitzakis ET, Castelo R, Thomson TM, et al. (January 2006). "Tandem chimerism as a means to increase protein complexity in the human genome". Genome Research. 16 (1): 37–44. doi:10.1101/gr.4145906. PMC 1356127. PMID 16344564.
- ^ a b Wright BW, Molloy MP, Jaschke PR (5 October 2021). "Overlapping genes in natural and engineered genomes". Nature Reviews Genetics. 23 (3): 154–168. doi:10.1038/s41576-021-00417-w. ISSN 1471-0064. PMC 8490965. PMID 34611352.
- ^ a b Eddy SR (December 2001). "Non-coding RNA genes and the modern RNA world". Nature Reviews. Genetics. 2 (12): 919–29. doi:10.1038/35103511. PMID 11733745. S2CID 18347629.
- ^ Crick FH, Barnett L, Brenner S, Watts-Tobin RJ (December 1961). "General nature of the genetic code for proteins". Nature. 192 (4809): 1227–32. Bibcode:1961Natur.192.1227C. doi:10.1038/1921227a0. PMID 13882203. S2CID 4276146.
- ^ Crick FH (October 1962). "The genetic code". Scientific American. WH Freeman and Company. 207 (4): 66–74. Bibcode:1962SciAm.207d..66C. doi:10.1038/scientificamerican1062-66. PMID 13882204.
- ^ Woodson SA (May 1998). "Ironing out the kinks: splicing and translation in bacteria". Genes & Development. 12 (9): 1243–7. doi:10.1101/gad.12.9.1243. PMID 9573040.
- ^ Jacob F, Monod J (June 1961). "Genetic regulatory mechanisms in the synthesis of proteins". Journal of Molecular Biology. 3 (3): 318–56. doi:10.1016/S0022-2836(61)80072-7. PMID 13718526.
- ^ Koonin EV, Dolja VV (January 1993). "Evolution and taxonomy of positive-strand RNA viruses: implications of comparative analysis of amino acid sequences". Critical Reviews in Biochemistry and Molecular Biology. 28 (5): 375–430. doi:10.3109/10409239309078440. PMID 8269709.
- ^ Domingo E (2001). "RNA Virus Genomes". eLS. doi:10.1002/9780470015902.a0001488.pub2. ISBN 978-0470016176.
- ^ Domingo E, Escarmís C, Sevilla N, Moya A, Elena SF, Quer J, et al. (June 1996). "Basic concepts in RNA virus evolution". FASEB Journal. 10 (8): 859–64. doi:10.1096/fasebj.10.8.8666162. PMID 8666162. S2CID 20865732.
- ^ Morris KV, Mattick JS (June 2014). "The rise of regulatory RNA". Nature Reviews. Genetics. 15 (6): 423–37. doi:10.1038/nrg3722. PMC 4314111. PMID 24776770.
- ^ Miko I (2008). "Gregor Mendel and the Principles of Inheritance". Nature Education Knowledge. SciTable. Nature Publishing Group. 1 (1): 134.
- ^ Chial H (2008). "Mendelian Genetics: Patterns of Inheritance and Single-Gene Disorders". Nature Education Knowledge. SciTable. Nature Publishing Group. 1 (1): 63.
- ^ McCarthy D, Minner C, Bernstein H, Bernstein C (October 1976). "DNA elongation rates and growing point distributions of wild-type phage T4 and a DNA-delay amber mutant". Journal of Molecular Biology. 106 (4): 963–81. doi:10.1016/0022-2836(76)90346-6. PMID 789903.
- ^ a b Lobo I, Shaw K (2008). "Discovery and Types of Genetic Linkage". Nature Education Knowledge. SciTable. Nature Publishing Group. 1 (1): 139.
- ^ Nachman MW, Crowell SL (September 2000). "Estimate of the mutation rate per nucleotide in humans". Genetics. 156 (1): 297–304. doi:10.1093/genetics/156.1.297. PMC 1461236. PMID 10978293.
- ^ Roach JC, Glusman G, Smit AF, Huff CD, Hubley R, Shannon PT, et al. (April 2010). "Analysis of genetic inheritance in a family quartet by whole-genome sequencing". Science. 328 (5978): 636–9. Bibcode:2010Sci...328..636R. doi:10.1126/science.1186802. PMC 3037280. PMID 20220176.
- ^ a b Drake JW, Charlesworth B, Charlesworth D, Crow JF (April 1998). "Rates of spontaneous mutation". Genetics. 148 (4): 1667–86. doi:10.1093/genetics/148.4.1667. PMC 1460098. PMID 9560386.
- ^ "What kinds of gene mutations are possible?". Genetics Home Reference. United States National Library of Medicine. 11 May 2015. Retrieved 19 May 2015.
- ^ Andrews CA (2010). "Natural Selection, Genetic Drift, and Gene Flow Do Not Act in Isolation in Natural Populations". Nature Education Knowledge. SciTable. Nature Publishing Group. 3 (10): 5.
- ^ Patterson C (November 1988). "Homology in classical and molecular biology". Molecular Biology and Evolution. 5 (6): 603–25. doi:10.1093/oxfordjournals.molbev.a040523. PMID 3065587.
- ^ Studer RA, Robinson-Rechavi M (May 2009). "How confident can we be that orthologs are similar, but paralogs differ?". Trends in Genetics. 25 (5): 210–6. doi:10.1016/j.tig.2009.03.004. PMID 19368988.
- ^ Altenhoff AM, Studer RA, Robinson-Rechavi M, Dessimoz C (2012). "Resolving the ortholog conjecture: orthologs tend to be weakly, but significantly, more similar in function than paralogs". PLOS Computational Biology. 8 (5): e1002514. Bibcode:2012PLSCB...8E2514A. doi:10.1371/journal.pcbi.1002514. PMC 3355068. PMID 22615551.

- ^ Nosil P, Funk DJ, Ortiz-Barrientos D (February 2009). "Divergent selection and heterogeneous genomic divergence". Molecular Ecology. 18 (3): 375–402. doi:10.1111/j.1365-294X.2008.03946.x. PMID 19143936.
- ^ Emery L (5 December 2014). "Introduction to Phylogenetics". EMBL-EBI. Retrieved 19 May 2015.
- ^ Mitchell MW, Gonder MK (2013). "Primate Speciation: A Case Study of African Apes". Nature Education Knowledge. SciTable. Nature Publishing Group. 4 (2): 1.
- ^ a b Guerzoni D, McLysaght A (November 2011). "De novo origins of human genes". PLOS Genetics. 7 (11): e1002381. doi:10.1371/journal.pgen.1002381. PMC 3213182. PMID 22102832.

- ^ Reams AB, Roth JR (February 2015). "Mechanisms of gene duplication and amplification". Cold Spring Harbor Perspectives in Biology. 7 (2): a016592. doi:10.1101/cshperspect.a016592. PMC 4315931. PMID 25646380.
- ^ Demuth JP, De Bie T, Stajich JE, Cristianini N, Hahn MW (December 2006). "The evolution of mammalian gene families". PLOS ONE. 1 (1): e85. Bibcode:2006PLoSO...1...85D. doi:10.1371/journal.pone.0000085. PMC 1762380. PMID 17183716.

- ^ Knowles DG, McLysaght A (October 2009). "Recent de novo origin of human protein-coding genes". Genome Research. 19 (10): 1752–9. doi:10.1101/gr.095026.109. PMC 2765279. PMID 19726446.
- ^ Wu DD, Irwin DM, Zhang YP (November 2011). "De novo origin of human protein-coding genes". PLOS Genetics. 7 (11): e1002379. doi:10.1371/journal.pgen.1002379. PMC 3213175. PMID 22102831.

- ^ McLysaght A, Guerzoni D (September 2015). "New genes from non-coding sequence: the role of de novo protein-coding genes in eukaryotic evolutionary innovation". Philosophical Transactions of the Royal Society of London. Series B, Biological Sciences. 370 (1678): 20140332. doi:10.1098/rstb.2014.0332. PMC 4571571. PMID 26323763.
- ^ Neme R, Tautz D (February 2013). "Phylogenetic patterns of emergence of new genes support a model of frequent de novo evolution". BMC Genomics. 14 (1): 117. doi:10.1186/1471-2164-14-117. PMC 3616865. PMID 23433480.
- ^ Treangen TJ, Rocha EP (January 2011). "Horizontal transfer, not duplication, drives the expansion of protein families in prokaryotes". PLOS Genetics. 7 (1): e1001284. doi:10.1371/journal.pgen.1001284. PMC 3029252. PMID 21298028.

- ^ Ochman H, Lawrence JG, Groisman EA (May 2000). "Lateral gene transfer and the nature of bacterial innovation". Nature. 405 (6784): 299–304. Bibcode:2000Natur.405..299O. doi:10.1038/35012500. PMID 10830951. S2CID 85739173.
- ^ Keeling PJ, Palmer JD (August 2008). "Horizontal gene transfer in eukaryotic evolution". Nature Reviews. Genetics. 9 (8): 605–18. doi:10.1038/nrg2386. PMID 18591983. S2CID 213613.
- ^ Schönknecht G, Chen WH, Ternes CM, Barbier GG, Shrestha RP, Stanke M, et al. (March 2013). "Gene transfer from bacteria and archaea facilitated evolution of an extremophilic eukaryote". Science. 339 (6124): 1207–10. Bibcode:2013Sci...339.1207S. doi:10.1126/science.1231707. PMID 23471408. S2CID 5502148.
- ^ 리들리, M. (2006)게놈.뉴욕, 뉴욕: 하퍼 다년생.ISBN 0-06-019497-9
- ^ Banerjee S, Bhandary P, Woodhouse M, Sen TZ, Wise RP, Andorf CM (April 2021). "FINDER: an automated software package to annotate eukaryotic genes from RNA-Seq data and associated protein sequences". BMC Bioinformatics. 44 (9): e89. doi:10.1186/s12859-021-04120-9. PMC 8056616. PMID 33879057.
- ^ Watson, JD, Baker TA, Bell SP, Gann A, Levine M, Losick R. (2004)"Ch9-10", 유전자의 분자생물학, 제5판, 피슨 벤자민 커밍스, CSHL 프레스.
- ^ "Integr8 – A.thaliana Genome Statistics".
- ^ "Understanding the Basics". The Human Genome Project. Retrieved 26 April 2015.
- ^ "WS227 Release Letter". WormBase. 10 August 2011. Archived from the original on 28 November 2013. Retrieved 19 November 2013.
- ^ a b Yu J, Hu S, Wang J, Wong GK, Li S, Liu B, et al. (April 2002). "A draft sequence of the rice genome (Oryza sativa L. ssp. indica)". Science. 296 (5565): 79–92. Bibcode:2002Sci...296...79Y. doi:10.1126/science.1068037. PMID 11935017. S2CID 208529258.
- ^ a b Anderson S, Bankier AT, Barrell BG, de Bruijn MH, Coulson AR, Drouin J, et al. (April 1981). "Sequence and organization of the human mitochondrial genome". Nature. 290 (5806): 457–65. Bibcode:1981Natur.290..457A. doi:10.1038/290457a0. PMID 7219534. S2CID 4355527.
- ^ Adams MD, Celniker SE, Holt RA, Evans CA, Gocayne JD, Amanatides PG, et al. (March 2000). "The genome sequence of Drosophila melanogaster". Science. 287 (5461): 2185–95. Bibcode:2000Sci...287.2185.. CiteSeerX 10.1.1.549.8639. doi:10.1126/science.287.5461.2185. PMID 10731132.
- ^ a b Pertea M, Salzberg SL (2010). "Between a chicken and a grape: estimating the number of human genes". Genome Biology. 11 (5): 206. doi:10.1186/gb-2010-11-5-206. PMC 2898077. PMID 20441615.
- ^ Belyi VA, Levine AJ, Skalka AM (December 2010). "Sequences from ancestral single-stranded DNA viruses in vertebrate genomes: the parvoviridae and circoviridae are more than 40 to 50 million years old". Journal of Virology. 84 (23): 12458–62. doi:10.1128/JVI.01789-10. PMC 2976387. PMID 20861255.
- ^ Flores R, Di Serio F, Hernández C (February 1997). "Viroids: The Noncoding Genomes". Seminars in Virology. 8 (1): 65–73. doi:10.1006/smvy.1997.0107.
- ^ Zonneveld BJ (2010). "New Record Holders for Maximum Genome Size in Eudicots and Monocots". Journal of Botany. 2010: 1–4. doi:10.1155/2010/527357.
- ^ Perez-Iratxeta C, Palidwor G, Andrade-Navarro MA (December 2007). "Towards completion of the Earth's proteome". EMBO Reports. 8 (12): 1135–41. doi:10.1038/sj.embor.7401117. PMC 2267224. PMID 18059312.
- ^ Kauffman SA (March 1969). "Metabolic stability and epigenesis in randomly constructed genetic nets". Journal of Theoretical Biology. Elsevier. 22 (3): 437–67. Bibcode:1969JThBi..22..437K. doi:10.1016/0022-5193(69)90015-0. PMID 5803332.
- ^ Schuler GD, Boguski MS, Stewart EA, Stein LD, Gyapay G, Rice K, et al. (October 1996). "A gene map of the human genome". Science. 274 (5287): 540–6. Bibcode:1996Sci...274..540S. doi:10.1126/science.274.5287.540. PMID 8849440. S2CID 22619.
- ^ Chi KR (October 2016). "The dark side of the human genome". Nature. 538 (7624): 275–277. Bibcode:2016Natur.538..275C. doi:10.1038/538275a. PMID 27734873.
- ^ a b Claverie JM (September 2005). "Fewer genes, more noncoding RNA". Science. 309 (5740): 1529–30. Bibcode:2005Sci...309.1529C. doi:10.1126/science.1116800. PMID 16141064. S2CID 28359091.
- ^ Carninci P, Hayashizaki Y (April 2007). "Noncoding RNA transcription beyond annotated genes". Current Opinion in Genetics & Development. 17 (2): 139–44. doi:10.1016/j.gde.2007.02.008. PMID 17317145.
- ^ a b Hutchison CA, Chuang RY, Noskov VN, Assad-Garcia N, Deerinck TJ, Ellisman MH, et al. (March 2016). "Design and synthesis of a minimal bacterial genome". Science. 351 (6280): aad6253. Bibcode:2016Sci...351.....H. doi:10.1126/science.aad6253. PMID 27013737.
- ^ Glass JI, Assad-Garcia N, Alperovich N, Yooseph S, Lewis MR, Maruf M, et al. (January 2006). "Essential genes of a minimal bacterium". Proceedings of the National Academy of Sciences of the United States of America. 103 (2): 425–30. Bibcode:2006PNAS..103..425G. doi:10.1073/pnas.0510013103. PMC 1324956. PMID 16407165.
- ^ Gerdes SY, Scholle MD, Campbell JW, Balázsi G, Ravasz E, Daugherty MD, et al. (October 2003). "Experimental determination and system level analysis of essential genes in Escherichia coli MG1655". Journal of Bacteriology. 185 (19): 5673–84. doi:10.1128/jb.185.19.5673-5684.2003. PMC 193955. PMID 13129938.
- ^ Baba T, Ara T, Hasegawa M, Takai Y, Okumura Y, Baba M, et al. (2006). "Construction of Escherichia coli K-12 in-frame, single-gene knockout mutants: the Keio collection". Molecular Systems Biology. 2: 2006.0008. doi:10.1038/msb4100050. PMC 1681482. PMID 16738554.
- ^ a b Juhas M, Reuß DR, Zhu B, Commichau FM (November 2014). "Bacillus subtilis and Escherichia coli essential genes and minimal cell factories after one decade of genome engineering". Microbiology. 160 (Pt 11): 2341–2351. doi:10.1099/mic.0.079376-0. PMID 25092907.
- ^ Tu Z, Wang L, Xu M, Zhou X, Chen T, Sun F (February 2006). "Further understanding human disease genes by comparing with housekeeping genes and other genes". BMC Genomics. 7: 31. doi:10.1186/1471-2164-7-31. PMC 1397819. PMID 16504025.

- ^ Georgi B, Voight BF, Bućan M (May 2013). "From mouse to human: evolutionary genomics analysis of human orthologs of essential genes". PLOS Genetics. 9 (5): e1003484. doi:10.1371/journal.pgen.1003484. PMC 3649967. PMID 23675308.

- ^ Eisenberg E, Levanon EY (October 2013). "Human housekeeping genes, revisited". Trends in Genetics. 29 (10): 569–74. doi:10.1016/j.tig.2013.05.010. PMID 23810203.
- ^ Amsterdam A, Hopkins N (September 2006). "Mutagenesis strategies in zebrafish for identifying genes involved in development and disease". Trends in Genetics. 22 (9): 473–8. doi:10.1016/j.tig.2006.06.011. PMID 16844256.
- ^ "About the HGNC". HGNC Database of Human Gene Names. HUGO Gene Nomenclature Committee. Retrieved 14 May 2015.
- ^ Cohen SN, Chang AC (May 1973). "Recircularization and autonomous replication of a sheared R-factor DNA segment in Escherichia coli transformants". Proceedings of the National Academy of Sciences of the United States of America. 70 (5): 1293–7. Bibcode:1973PNAS...70.1293C. doi:10.1073/pnas.70.5.1293. PMC 433482. PMID 4576014.
- ^ Esvelt KM, Wang HH (2013). "Genome-scale engineering for systems and synthetic biology". Molecular Systems Biology. 9 (1): 641. doi:10.1038/msb.2012.66. PMC 3564264. PMID 23340847.
- ^ Tan WS, Carlson DF, Walton MW, Fahrenkrug SC, Hackett PB (2012). "Precision editing of large animal genomes". Advances in Genetics Volume 80. Advances in Genetics. Vol. 80. pp. 37–97. doi:10.1016/B978-0-12-404742-6.00002-8. ISBN 9780124047426. PMC 3683964. PMID 23084873.
- ^ Puchta H, Fauser F (2013). "Gene targeting in plants: 25 years later". The International Journal of Developmental Biology. 57 (6–8): 629–37. doi:10.1387/ijdb.130194hp. PMID 24166445.
- ^ Ran FA, Hsu PD, Wright J, Agarwala V, Scott DA, Zhang F (November 2013). "Genome engineering using the CRISPR-Cas9 system". Nature Protocols. 8 (11): 2281–2308. doi:10.1038/nprot.2013.143. PMC 3969860. PMID 24157548.
- ^ Kittleson JT, Wu GC, Anderson JC (August 2012). "Successes and failures in modular genetic engineering". Current Opinion in Chemical Biology. 16 (3–4): 329–36. doi:10.1016/j.cbpa.2012.06.009. PMID 22818777.
- ^ Berg P, Mertz JE (January 2010). "Personal reflections on the origins and emergence of recombinant DNA technology". Genetics. 184 (1): 9–17. doi:10.1534/genetics.109.112144. PMC 2815933. PMID 20061565.
- ^ Austin CP, Battey JF, Bradley A, Bucan M, Capecchi M, Collins FS, et al. (September 2004). "The knockout mouse project". Nature Genetics. 36 (9): 921–4. doi:10.1038/ng0904-921. PMC 2716027. PMID 15340423.
- ^ Guan C, Ye C, Yang X, Gao J (February 2010). "A review of current large-scale mouse knockout efforts". Genesis. 48 (2): 73–85. doi:10.1002/dvg.20594. PMID 20095055. S2CID 34470273.
- ^ Deng C (October 2007). "In celebration of Dr. Mario R. Capecchi's Nobel Prize". International Journal of Biological Sciences. 3 (7): 417–9. doi:10.7150/ijbs.3.417. PMC 2043165. PMID 17998949.
원천
- 본교과서
- Alberts B, Johnson A, Lewis J, Raff M, Roberts K, Walter P (2002). Molecular Biology of the Cell (Fourth ed.). New York: Garland Science. ISBN 978-0-8153-3218-3. – NCBI
 Bookbooks에서 무료로 온라인으로 분자생물학 교재 제공.
Bookbooks에서 무료로 온라인으로 분자생물학 교재 제공.
추가 정보
- Watson JD, Baker TA, Bell SP, Gann A, Levine M, Losick R (2013). Molecular Biology of the Gene (7th ed.). Benjamin Cummings. ISBN 978-0-321-90537-6.
- Dawkins R (1990). The Selfish Gene. Oxford University Press. ISBN 978-0-19-286092-7. Google Book Search, 1976년 초판.
- Ridley M (1999). Genome: The Autobiography of a Species in 23 Chapters. Fourth Estate. ISBN 978-0-00-763573-3.
- Brown T (2002). Genomes (2nd ed.). New York: Wiley-Liss. ISBN 978-0-471-25046-3.
외부 링크
- 비교독성유전체학 데이터베이스
- 처음부터 DNA – 유전자와 DNA에 대한 입문서
- Entrez Gene – 검색 가능한 유전자 데이터베이스
- ID 변환기– 공용 데이터베이스 간에 유전자 ID 변환
- iHOP – 단백질과 하이퍼링크된 정보
- Transcriptome Browser – 유전자 발현 프로파일 분석
- 그 단백질 명 유틸리티, 데이터베이스를 해결하는 부족한 유전자 이름을 식별한다
- 유전자는 오픈 액세스 저널 –.
- IMPC(국제 마우스 Phenotyping 컨소시엄)– 백과 사전 포유류의 유전자 기능의.
- 글로벌 Genes 프로젝트 – 주요한 비영리 단체는 사람들이 유전병을 생활을 지원한다.
- ENCODE Explorer특성화 유전자 간 지역과 유전자 정의의 threads.자연.