염색질
Chromatin오래된 정보로 인해 이 문서의 사실 정확도가 저하될 수 있습니다.(2022년 7월) |
염색질은 진핵세포에서 [1]발견되는 DNA와 단백질의 복합체이다.주요 기능은 긴 DNA 분자를 보다 작고 밀도 높은 구조로 포장하는 것입니다.이것은 가닥이 엉키는 것을 방지하고 또한 세포 분열 중에 DNA를 강화시키고, DNA 손상을 방지하고, 유전자 발현과 DNA 복제를 조절하는 중요한 역할을 한다.유사분열과 감수분열 동안, 염색질은 무지상 염색체의 적절한 분리를 촉진합니다; 이 단계에서 보이는 염색체의 특징적인 모양은 DNA가 고도로 응축된 염색질로 감겨진 결과입니다.
크로마틴의 주요 단백질 성분은 히스톤이다.4개의 히스톤 코어(히스톤 H2A, 히스톤 H2B, 히스톤 H3, 및 히스톤 H4)의 2세트로 이루어진 옥타머는 DNA에 결합되어 가닥이 [2]감기는 "앵커"로서 기능한다.일반적으로 염색질 구성에는 세 가지 레벨이 있습니다.
- DNA는 히스톤 단백질을 감싸고, 뉴클레오솜과 소위 구슬을 형성합니다.
- 여러 히스톤은 가장 콤팩트한 형태(헤테로크로마틴)[a]로 뉴클레오솜 배열로 구성된 30나노미터 섬유로 싸여 있습니다.
- 30-nm 섬유의 높은 수준의 DNA 초코일은 (유분열과 감수 분열 동안) 중기 염색체를 생성한다.
그러나 많은 유기체들은 이 조직 체계를 따르지 않는다.예를 들어 정자와 조류 적혈구는 대부분의 진핵세포보다 염색질이 촘촘히 채워져 있고, 트리파노소마티스 원생동물은 그들의 염색질을 눈에 보이는 염색체로 전혀 응축하지 않는다.원핵세포는 DNA를 조직하기 위한 완전히 다른 구조를 가지고 있다.
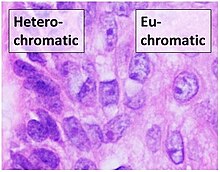
염색질 네트워크의 전체 구조는 세포 주기의 단계에 따라 더욱 달라진다.중간 단계 동안, 염색질은 DNA를 전사하고 복제하는 RNA와 DNA 중합 효소에 접근할 수 있도록 구조적으로 느슨하다.중간 단계 동안 염색질의 국소 구조는 DNA에 존재하는 특정 유전자에 따라 달라집니다.활발하게 전사되는(켜지는) 유전자를 포함하는 DNA 영역은 에우크로마틴으로 알려진 구조에서 RNA 중합효소와의 긴밀성이 떨어지고 밀접하게 연관되어 있는 반면, 비활성 유전자를 포함하는 영역("꺼진")은 일반적으로 더 응축되어 [4]헤테로크로마틴의 구조 단백질과 관련이 있다.메틸화 및 아세틸화를 통한 크로마틴의 구조단백질 후생유전학적 변형은 국소 크로마틴 구조를 변화시켜 유전자 발현을 변화시킨다.염색질 네트워크의 구조는 현재 잘 알려져 있지 않으며 분자 생물학에서 여전히 활발한 연구 분야로 남아 있다.
동적 크로마틴 구조 및 계층
염색질은 세포 주기 동안 다양한 구조적 변화를 겪는다.히스톤 단백질은 크로마틴의 기본 포장제 및 배열제이며, 다양한 번역 후 변형을 통해 크로마틴 패킹을 변경할 수 있다(히스톤 변형).대부분의 수정은 히스톤 꼬리에서 발생합니다.양전하를 띤 히스톤 코어는 DNA 인산염 백본의 음전하를 부분적으로만 상쇄하여 전체 구조의 음전하를 생성한다.고분자 내에서의 전하 불균형은 양전하를 띤 단백질, 분자 및 양이온과의 상호작용을 촉진하는 인접한 염색질 영역 사이에 정전기의 반발을 일으킨다.이러한 변화가 일어나면 크로마틴을 둘러싼 정전기 환경이 변화하고 크로마틴 압축 수준이 [2]변화합니다.염색질 접근성 및 압축성의 결과는 변형된 아미노산과 변형 유형에 따라 달라집니다.예를 들어 히스톤 아세틸화는 복제 및 전사를 위한 염색질의 이완 및 접근성을 증가시킨다.리신 트리메틸화는 전사 활성 증가(히스톤 H3 리신 4의 트리메틸화) 또는 전사 억제 및 염색질 압축(히스톤 H3 리신 9 또는 27의 트리메틸화)을 초래할 수 있다.여러 연구에 따르면 다른 수정이 동시에 발생할 수 있습니다.예를 들어, (히스톤 H3에서 리신 4와 27의 트리메틸화를 모두 갖는) 2가 구조가 포유류의 초기 발달에 관여하는 것이 제안되었다.또 다른 연구에서는 염색질 구조에 대한 H4K16ac의 역할을 테스트한 결과, 균질 아세틸화가 30nm 염색질 형성을 억제하고 아데노신 삼인산 리모델링을 막았다.이 단수 변형은 염색질의 역학을 변화시켰으며, 이는 K16에서 H4의 아세틸화가 염색질 [5]구조의 적절한 기능 내 및 기능 간 기능에 필수적이라는 것을 보여준다.
번스타인 BE, 미켈센 TS, 위험 X, 카말 M, Huebert DJ, 수갑 채워 J, FryB, 마이스너 A, Wernig M, 플래스 K, 재니치 R, Wagschal A, Feil R, 슈라이버 SL, 랜더 ES(2006년 4월)."배아 줄기 세포의 이가 염색질 구조 자국 주요 발달 유전자".세포. 125(2):315–26. doi:10.1016/j.cell.2006.02.041.ISSN 0092-8674.PMID 16630819.S2CID 9993008.<>/ref>.
폴리콤 그룹 단백질은 염색질 구조의 [6]변조를 통해 유전자를 조절하는 역할을 한다.
자세한 내용은 Chromatin 변이체, Chromatin 조절 히스톤 변이체 및 Chromatin 구조에 의한 RNA 중합효소 제어를 참조한다.
DNA구조
DNA는 A, B, Z-DNA의 세 가지 구조를 형성할 수 있으며, A와 B-DNA는 매우 유사하여 오른손 나선을 형성하고 있는 반면 Z-DNA는 지그재그 모양의 인산염 골격을 가진 왼손나선이다.Z-DNA는 B-와 Z-DNA 사이의 접합부의 특성 때문에 염색질 구조와 전사에 특정한 역할을 하는 것으로 생각된다.
B-와 Z-DNA의 접합부에서는 1쌍의 염기가 통상의 결합에서 플립아웃된다.이것들은 많은 단백질에 의해 인식되는 부위와 RNA 중합효소 또는 뉴클레오솜 결합에 의한 비틀림 스트레스의 흡수원으로서 이중 역할을 한다.
뉴클레오솜 및 구슬-온-a-string
염색질의 기본 반복 요소는 링커 DNA의 단면에 의해 상호 연결된 뉴클레오솜으로, 용액 속의 순수한 DNA보다 훨씬 짧은 배열입니다.
코어 히스톤 외에 뉴클레오솜상의 DNA 가닥의 출구/입구와 접촉하는 링커 히스톤 H1이 존재한다.뉴클레오솜 코어 입자는 히스톤 H1과 함께 크로마토솜으로 알려져 있다.약 20에서 60쌍의 염기쌍의 링커 DNA를 가진 뉴클레오솜은 비생리학적인 조건에서 끈 섬유에 약 10 nm의 구슬을 형성할 수 있다.
뉴클레오솜은 일반 DNA 포장의 기능에 따라 DNA를 비특이적으로 결합한다.그러나 뉴클레오좀의 위치를 결정하는 큰 DNA 배열 선호도가 있다.이는 주로 다양한 DNA 배열의 다양한 물리적 특성에 기인합니다.예를 들어 아데닌(A)과 티민(T)을 내부 소구내에 보다 잘 압축한다.이는 뉴클레오솜이 약 10개의 염기쌍(DNA의 나선형 반복)마다 1개의 위치에서 우선적으로 결합할 수 있다는 것을 의미한다. 여기서 DNA는 내부 작은 홈에 있는 A 및 T 염기의 수를 최대화하기 위해 회전한다(핵산 구조 참조).
30나노미터 크로마틴 섬유
H1을 추가하면 코일은 30nm 직경의 나선형 구조인 30nm 파이버 또는 필라멘트로 알려져 있습니다.세포 내 염색질 섬유의 정확한 구조는 [7]자세히 알려져 있지 않다.
이러한 수준의 염색질 구조는 대부분 전사적으로 조용한 유전자를 포함하는 헤테로크로마틴의 형태라고 생각됩니다.전자현미경 연구에 따르면 30nm 섬유는 전사에 관여하는 RNA 중합효소에 의해 변환될 때 10nm 섬유 비즈-on-a-string 구조로 전개될 정도로 매우 역동적인 것으로 나타났다.
기존 모델은 일반적으로 뉴클레오솜이 섬유 축에 수직으로 놓여 있고 링커 히스톤이 내부에 배열되어 있다는 것을 인정한다.안정적인 30 nm 섬유는 DNA를 따라 뉴클레오좀의 규칙적인 위치에 의존한다.링커 DNA는 상대적으로 굽힘과 회전에 강하다.이것은 섬유의 안정성에 링커 DNA의 길이를 중요하게 만들고, DNA에 과도한 스트레스 없이 필요한 방향으로 회전하고 접을 수 있는 길이로 뉴클레오솜을 분리해야 한다.이러한 관점에서 링커 DNA의 다른 길이는 염색질 섬유의 다른 접힘 토폴로지를 생성해야 한다.재구성된 섬유의 전자현미경 이미지를[8] 기반으로 한 최근의 이론적 연구는 이러한 [9]관점을 뒷받침한다.
세포핵 내 염색질 공간구성
핵 내 염색질의 공간 배치는 무작위적이지 않다. 염색질의 특정 영역은 특정 영역에서 찾을 수 있다.영역에는 예를 들어 단백질 [10]복합체에 의해 결합되는 라미나 관련 도메인(LADs)과 위상 관련 도메인(TADs)이 있습니다.현시점에서는 Strings & Binders Switch(SBS[11]) 모델이나 Dynamic Loop(DL) 모델[12] 등의 폴리머 모델을 사용하여 핵 내에서 크로마틴의 접힘을 설명하고 있습니다.핵 내 염색질의 배열은 또한 핵 응력과 기계적 응력에 의한 핵막 변형을 복원하는 역할을 할 수 있다.염색질이 응축되면, 핵은 더 단단해진다.염색질이 축합될 때 핵은 내부 핵막에 가해지는 힘을 줄이면서 더 탄성을 갖게 된다.이 관찰은 게놈 [2]조절을 벗어난 염색질 조직의 다른 가능한 세포 기능을 조명한다.
세포주기 의존 구조조직
- 단계 간:유사분열의 중간 단계 동안 염색질의 구조는 DNA를 핵으로 압축하면서 DNA에 대한 전사 및 DNA 복구 인자의 간단한 접근을 허용하도록 최적화되었다.구조는 DNA에 필요한 접근에 따라 달라집니다.RNA 중합효소에 의한 정기적인 접근을 필요로 하는 유전자는 에크로마틴에 의해 제공되는 더 느슨한 구조를 필요로 한다.
- 메타페이즈:크로마틴의 중기의 구조는 상간 구조와 크게 다르다.그것은 핵형에서 볼 수 있는 전형적인 염색체 구조를 형성하면서, 체력과[citation needed] 관리성을 위해 최적화되었다.응축 크로마틴의 구조는 단백질의 중앙 골격에 30 nm 섬유로 이루어진 루프인 것으로 생각됩니다.그러나 그것은 잘 특징지어지지 않았다.염색체 발판은 염색질을 콤팩트한 염색체로 고정시키는 중요한 역할을 한다.30 nm 구조의 루프는 비계와 함께 더 높은 [13]차수의 구조로 응축됩니다.염색체 비계는 응축소, IIA형 토포이소머라아제 및 키네신 패밀리 멤버 4(KIF4)[14]를 포함한 단백질로 이루어진다.염색질의 물리적 강도는 딸 염색체가 분리될 때 DNA의 전단 손상을 막기 위해 이 분열 단계에서 필수적이다.강도를 최대화하기 위해 염색질의 구성은 주로 대체 히스톤 H1 유사체를 통해 동원체에 접근하면서 변화한다.유사분열 동안, 대부분의 염색질은 단단하게 압축되어 있지만, 그렇게 단단하게 압축되어 있지 않은 작은 영역이 있습니다.이러한 영역은 종종 염색질이 형성되기 전에 해당 세포 유형에서 활성화되었던 유전자의 프로모터 영역에 해당합니다.이러한 영역의 결합의 결여는 북마크라고 불리며, 이것은 유사분열로 [15]들어가기 전에 유전자가 활성화되었던 "기억력"을 딸 세포에 전달하는 데 중요한 것으로 여겨지는 후생유전 메커니즘이다.이 북마크메커니즘은 유사분열 중에 문자 변환이 중지되기 때문에 이 메모리를 전송하기 위해 필요합니다.
염색질 및 전사 버스트
크로마틴과 효소의 상호작용이 연구되어 유전자 발현에 관련성이 있고 중요한 요소라는 결론이 내려지고 있다.록펠러 대학의 빈센트 올프리 교수는 RNA 합성이 히스톤 아세틸화와 관련이 [16]있다고 말했다.히스톤 끝에 부착된 리신 아미노산은 양전하를 띤다.이 꼬리의 아세틸화는 염색질 끝을 중립으로 만들어 DNA 접근을 가능하게 할 것이다.
염색질이 축합되면 DNA는 분자 기계로 들어갈 수 있게 된다.개방 크로마틴과 폐쇄 크로마틴 사이의 변동은 전사 중단 또는 전사 파열의 원인이 될 수 있다.크로마틴과의 전사 인자 복합체의 연관성 및 해리 등과 같은 다른 인자가 관련될 수 있다.특히 RNA 중합효소 및 전사 단백질은 상분리를 통해 물방울에 모이는 것으로 나타났으며, 최근 연구에 따르면 10nm 염색질은 [17]게놈 DNA의 표적성을 높이는 액체와 같은 행동을 보인다.링커 히스톤과 무질서한 꼬리 영역 사이의 상호작용은 대규모 크로마틴을 동적인 액체 같은 영역으로 조직하는 정전 접착제 역할을 합니다.염색질 압축의 감소는 염색질 이동성의 증가와 DNA에 [2]대한 전사적 접근의 용이성을 수반한다.단순한 전사의 확률론적 모델과 달리, 이 현상은 등원성 [18]모집단의 세포 사이에서 일어나는 유전자 발현에서 높은 변동성을 설명할 수 있다.
대체 염색질 조직
메타조아 정자 형성 동안, 정자체의 염색질은 더 간격을 두고, 더 넓고, 거의 결정과 같은 구조로 개조됩니다.이 과정은 전사의 중단과 관련이 있으며 핵단백질 교환을 포함한다.히스톤은 대부분 치환되고 프로타민으로 대체된다.[19]효모에서 히스톤이 없는 영역은 전사 후 매우 취약해진다는 것이 제안되었다. HMG 상자 단백질인 HMO1은 뉴클레오솜이 없는 크로마틴을 [20][21]안정시키는 데 도움을 준다.
염색질 및 DNA 복구
진핵생물 DNA를 염색질로 포장하는 것은 효소의 활동 부위로의 공급을 필요로 하는 모든 DNA 기반 과정에 장벽을 제공합니다.DNA 수복의 중요한 세포 과정을 허용하기 위해, 염색질은 개조되어야 한다.진핵생물에서 ATP 의존성 크로마틴 리모델링 복합체와 히스톤 수식 효소는 이러한 리모델링 [22]과정을 달성하기 위해 사용되는 두 가지 주요 요인이다.
염색질 이완은 DNA [23]손상 부위에서 빠르게 일어난다.이 과정은 1초 이내에 DNA 손상에서 나타나기 시작하는 PARP1 단백질에 의해 시작되며 손상 [24]발생 후 1.6초 이내에 최대 절반이 축적됩니다.다음으로 염색질 리모델링제 Alc1은 PARP1의 제품에 신속하게 부착되어 손상 [23]후 10초 이내에 DNA 손상 도착을 완료한다.최대 염색질 이완의 약 절반은 아마도 Alc1의 작용에 의해 10초 [23]동안 발생한다.이것은 DNA 복구 효소 MRE11을 모집하여 [24]13초 이내에 DNA 복구를 시작할 수 있게 합니다.
또한 H2AX의 인산화 형태인 δH2AX는 DNA 손상 발생 후 염색질 탈축합으로 이어지는 초기 단계에 관여한다.히스톤 변종 H2AX는 인간 염색질에서 [25]H2A 히스톤의 약 10%를 구성한다.δH2AX(세린139에서 인산화되는 H2AX)는 세포 조사 후 20초 이내에 검출할 수 있으며([25]DNA 이중사슬 절단 형성), 1분 이내에 절반의 최대 축적이 발생한다.인산화 δH2AX를 가진 염색질의 범위는 DNA 이중 가닥 절단 [25]부위에서 약 2백만 개의 염기쌍이다.γH2AX 자체는 염색질 탈축합을 일으키지 않지만 조사 후 30초 이내에 γH2AX와 [26]관련된 RNF8 단백질을 검출할 수 있다.RNF8은 뉴클레오솜 리모델링 및 탈아세틸화효소 복합체 NuRD의 성분인 CHD4와의 [27]후속 상호작용을 통해 광범위한 크로마틴 탈축합을 매개한다.
염색질은 DNA 손상 후 이완 후 DNA 수복 후 약 20분 [23]후에 손상 전 수준에 가까운 압축 상태로 회복된다.
염색질 조사방법
- ChIP-seq(Chromatin 면역침습 배열)는 다른 히스톤 변형을 목표로 게놈 전체의 염색질 상태를 식별하기 위해 사용될 수 있다.염색질의 다양한 상태와 다른 변형들이 연관되어 왔다.
- DNase-seq(DNAase I 과민성 부위 시퀀싱)는 DNAase I 효소에 대한 게놈 내 접근 가능한 영역의 민감도를 사용하여 게놈에서 개방되거나 접근 가능한 영역을 매핑합니다.
- FAIR-seq(Form Aldehde-Assisted Isolation of Regulatory Elements Sequencing)는 단백질 결합 DNA의 화학적 성질을 2상 분리법으로 사용하여 [28]게놈에서 뉴클레오솜 고갈 영역을 추출한다.
- ATAC-seq(Assay for Transposable Accessible Chromatin Sequencing)는 Tn5 트랜스포스포지스를 사용하여 게놈의 접근 가능한 영역에 트랜스포존을 통합(합성)하여 결과적으로 게놈 전체의 뉴클레오솜과 전사 인자의 국재화를 강조합니다.
- DNA 풋프린팅은 단백질 결합 DNA를 식별하기 위한 방법이다.그것은 [29]단백질에 의해 결합되어 있는 게놈의 영역을 식별하기 위해 겔 전기영동과 결합된 라벨링과 파편화를 사용한다.
- MNase-seq(Micrococcal Nuclease Sequencing)는 [30][31]게놈 전체에서 뉴클레오솜의 위치를 식별하기 위해 마이크로코칼 뉴클레아제 효소를 사용한다.
- 염색체 배치 포착은 물리적으로 상호작용하는 게놈 위치를 유추함으로써 핵에서 염색질의 공간적 구성을 결정한다.
- MACC 프로파일링(Micrococcal nuclease ACessibility profiling)은 염색질 소화를 마이크로코칼 뉴클레아제와의 적정 시리즈를 사용하여 염색질 접근성을 식별하고 게놈의 [32]개방 및 폐쇄 영역 모두에서 뉴클레오솜과 비히스톤 DNA 결합 단백질을 매핑한다.
염색질과 매듭
어떻게 결로된 상간 염색체가 기본적으로 매듭이 없는 상태로 남아 있는지는 수수께끼였다.자연스런 기대는 서로를 통과하는 이중 가닥 DNA 영역의 통과를 허용하는 타입 II DNA 토포이소머라아제 존재 하에서, 모든 염색체가 위상 평형 상태에 도달해야 한다는 것이다.염색체 영역을 형성하는 매우 혼잡한 상간 염색체에서의 위상 균형은 고도로 뭉친 염색질 섬유의 형성을 초래할 것이다.그러나 염색체 구성 캡처(3C) 방법은 긴 폴리머가 매듭을 형성하지 않고 응축될 때 형성되는 구겨진 구근 상태에서 염색체 간 거리와의 접촉 붕괴가 실질적으로 동일하다는 것을 밝혀냈다.매우 혼잡한 크로마틴에서 매듭을 제거하려면 시스템을 위상 평형 상태에서 이동시키는 에너지를 제공할 뿐만 아니라 매듭을 더 복잡하게 만드는 대신 효율적으로 매듭을 풀 수 있도록 토포이소머라아제 매개 통로를 안내해야 하는 활성 프로세스가 필요하다.염색질-루프 압출 과정은 [33]상간 염색체에서 염색질 섬유를 능동적으로 분리하는 데 이상적으로 적합하다는 것이 입증되었다.
Chromatin: 대체 정의
Walther Flemming에 의해 도입된 이 용어의 의미는 다음과 같다.
- 심플하고 간결한 정의:염색질은 DNA 고분자와 단백질 고분자의 고분자 복합체이다.단백질은 DNA를 포장하고 배열하며 세포핵 내의 기능을 조절한다.
- 생화학자의 작업 정의:크로마틴은 진핵생물 용해된 상간핵에서 추출된 DNA/단백질/RNA 복합체이다.핵에 존재하는 여러 물질 중 어떤 것이 추출된 물질의 일부를 구성하느냐는 부분적으로는 각 연구자가 사용하는 기술에 달려 있다.또한 크로마틴의 조성 및 성질은 세포 유형에 따라, 특정 세포 유형의 발달 중 및 세포 사이클의 다른 단계에서 변화한다.
- DNA + 히스톤 = 염색질 정의:세포핵의 DNA 이중나선은 히스톤이라고 불리는 특별한 단백질에 의해 포장된다.형성된 단백질/DNA 복합체는 크로마틴이라고 불립니다.염색질의 기본 구조 단위는 뉴클레오솜이다.
첫 번째 정의는 분자를 응축시키는 DNA 결합 단백질을 사용하여 박테리아와 고세균과 같은 생명의 다른 영역에서 "크로마틴"을 정의할 수 있도록 합니다.이들 단백질은 보통 핵관련단백질(NAPs)로 언급된다. 예를 들어 HU와 함께 AsnC/LrpC를 포함한다. 또한, 일부 고세균은 진핵 히스톤과 [34]상동하는 단백질로부터 뉴클레오솜을 생성한다.
노벨상
다음 과학자들은 노벨상과 함께 염색질 연구에 기여한 공로를 인정받았다.
| 연도 | 누구 | 상 |
|---|---|---|
| 1910 | 알브레히트 코셀 (하이델베르크 대학교) | 아데닌, 시토신, 구아닌, 티민, 우라실 등 5가지 핵기반을 발견한 공로로 노벨 생리의학상. |
| 1933 | 토마스 헌트 모건(캘리포니아 공과대학) | 초파리 드로소필라의 [35]흰눈 돌연변이에 대한 연구를 바탕으로 유전에서 유전자와 염색체의 역할을 발견한 공로로 노벨 생리의학상. |
| 1962 | 프랜시스 크릭, 제임스 왓슨, 모리스 윌킨스(MRC 분자생물연구소, 하버드대, 런던대) | DNA 이중나선 구조의 발견과 살아있는 물질에서의 정보 전달에 대한 의의로 노벨 생리의학상. |
| 1982 | Aaron Klug (MRC 분자생물연구소) | 노벨 화학상 "결정학 전자 현미경 연구 개발과 생물학적으로 중요한 핵산-단백질 복합체 구조 설명" |
| 1993 | 리처드 J. 로버츠와 필립 A. 샤프 | "분할 유전자에 대한 독립적인 발견으로" 노벨 생리학상은 엑손이라고 불리는 DNA 부분이 단백질을 발현하지 않는 인트론이라고 불리는 DNA 부분에 의해 중단된다. |
| 2006 | Roger Kornberg (Stanford University | DNA가 메신저 RNA로 변환되는 메커니즘을 발견한 공로로 노벨 화학상. |
「 」를 참조해 주세요.
메모들
레퍼런스
- ^ Monday, Tanmoy (July 2010). "Characterization of the RNA content of chromatin". Genome Res. 20 (7): 899–907. doi:10.1101/gr.103473.109. PMC 2892091. PMID 20404130.
- ^ a b c d Maeshima, K., Ide, S. 및 Babokhov, M.(2019).30 nm 파이버를 사용하지 않는 다이내믹크로마틴 구성세포생물학계의 현재 견해는 58, 95~104입니다https://doi.org/10.1016/j.ceb.2019.02.003
- ^ Hansen, Jeffrey (March 2012). "Human mitotic chromosome structure: what happened to the 30-nm fibre?". The EMBO Journal. 31 (7): 1621–1623. doi:10.1038/emboj.2012.66. PMC 3321215. PMID 22415369.
- ^ Dame, R.T. (May 2005). "The role of nucleoid-associated proteins in the organization and compaction of bacterial chromatin". Molecular Microbiology. 56 (4): 858–870. doi:10.1111/j.1365-2958.2005.04598.x. PMID 15853876. S2CID 26965112.
- ^ 쇼그렌 크나크, M., 이시이, H., 선, J. M., 파진, M. J., 데이비, J. R. & 피터슨, C. L. (2006)히스톤 H4-K16 아세틸화는 염색질 구조와 단백질 상호작용을 제어한다.사이언스, 311 (5762), 844 ~847.https://doi.org/10.1126/science.1124000
- ^ Portoso M, Cavalli G (2008). "The Role of RNAi and Noncoding RNAs in Polycomb Mediated Control of Gene Expression and Genomic Programming". RNA and the Regulation of Gene Expression: A Hidden Layer of Complexity. Caister Academic Press. ISBN 978-1-904455-25-7.
- ^ Annunziato, Anthony T. "DNA Packaging: Nucleosomes and Chromatin". Scitable. Nature Education. Retrieved 2015-10-29.
- ^ Robinson DJ; Fairall L; Huynh VA; Rhodes D. (April 2006). "EM measurements define the dimensions of the "30-nm" chromatin fiber: Evidence for a compact, interdigitated structure". Proceedings of the National Academy of Sciences of the United States of America. 103 (17): 6506–11. Bibcode:2006PNAS..103.6506R. doi:10.1073/pnas.0601212103. PMC 1436021. PMID 16617109.
- ^ Wong H, Victor JM, Mozziconacci J (September 2007). Chen P (ed.). "An All-Atom Model of the Chromatin Fiber Containing Linker Histones Reveals a Versatile Structure Tuned by the Nucleosomal Repeat Length". PLoS ONE. 2 (9): e877. Bibcode:2007PLoSO...2..877W. doi:10.1371/journal.pone.0000877. PMC 1963316. PMID 17849006.

- ^ Nicodemi M, Pombo A (June 2014). "Models of chromosome structure" (PDF). Curr. Opin. Cell Biol. 28: 90–5. doi:10.1016/j.ceb.2014.04.004. PMID 24804566.
- ^ Nicodemi M, Panning B, Prisco A (May 2008). "A thermodynamic switch for chromosome colocalization". Genetics. 179 (1): 717–21. arXiv:0809.4788. doi:10.1534/genetics.107.083154. PMC 2390650. PMID 18493085.
- ^ Bohn M, Heermann DW (2010). "Diffusion-driven looping provides a consistent framework for chromatin organization". PLOS ONE. 5 (8): e12218. Bibcode:2010PLoSO...512218B. doi:10.1371/journal.pone.0012218. PMC 2928267. PMID 20811620.
- ^ Lodish, Harvey F. (2016). Molecular Cell Biology (8th ed.). New York: W. H. Freeman and Company. p. 339. ISBN 978-1-4641-8339-3.
- ^ Poonperm, R; Takata, H; Hamano, T; Matsuda, A; Uchiyama, S; Hiraoka, Y; Fukui, K (1 July 2015). "Chromosome Scaffold is a Double-Stranded Assembly of Scaffold Proteins". Scientific Reports. 5: 11916. Bibcode:2015NatSR...511916P. doi:10.1038/srep11916. PMC 4487240. PMID 26132639.
- ^ Xing H, Vanderford NL, Sarge KD (November 2008). "The TBP-PP2A mitotic complex bookmarks genes by preventing condensin action". Nat. Cell Biol. 10 (11): 1318–23. doi:10.1038/ncb1790. PMC 2577711. PMID 18931662.
- ^ Allfrey VG, Faulkner R, Mirsky AE (May 1964). "Acetylation and Methylation of Histones and Their Possible Role in the Regulation of RNA Synthesis". Proc. Natl. Acad. Sci. U.S.A. 51 (5): 786–94. Bibcode:1964PNAS...51..786A. doi:10.1073/pnas.51.5.786. PMC 300163. PMID 14172992.
- ^ 마에시마·K.·이데·S.·히비노·K.·사사이(2016년).염색질의 액체 같은 거동.현재 유전학 및 발달학 의견, 37세, 36세~45세https://doi.org/10.1016/j.gde.2015.11.006
- ^ Kaochar S, Tu BP (November 2012). "Gatekeepers of chromatin: Small metabolites elicit big changes in gene expression". Trends Biochem. Sci. 37 (11): 477–83. doi:10.1016/j.tibs.2012.07.008. PMC 3482309. PMID 22944281.
- ^ De Vries M, Ramos L, Housein Z, De Boer P (May 2012). "Chromatin remodelling initiation during human spermiogenesis". Biol Open. 1 (5): 446–57. doi:10.1242/bio.2012844. PMC 3507207. PMID 23213436.
- ^ Murugesapillai D, McCauley MJ, Huo R, Nelson Holte MH, Stepanyants A, Maher LJ, Israeloff NE, Williams MC (August 2014). "DNA bridging and looping by HMO1 provides a mechanism for stabilizing nucleosome-free chromatin". Nucleic Acids Research. 42 (14): 8996–9004. doi:10.1093/nar/gku635. PMC 4132745. PMID 25063301.
- ^ Murugesapillai D, McCauley MJ, Maher LJ, Williams MC (February 2017). "Single-molecule studies of high-mobility group B architectural DNA bending proteins". Biophysical Reviews. 9 (1): 17–40. doi:10.1007/s12551-016-0236-4. PMC 5331113. PMID 28303166.
- ^ Liu B, Yip RK, Zhou Z (2012). "Chromatin remodeling, DNA damage repair and aging". Curr. Genomics. 13 (7): 533–47. doi:10.2174/138920212803251373. PMC 3468886. PMID 23633913.
- ^ a b c d Sellou H, Lebeaupin T, Chapuis C, Smith R, Hegele A, Singh HR, Kozlowski M, Bultmann S, Ladurner AG, Timinszky G, Huet S (2016). "The poly(ADP-ribose)-dependent chromatin remodeler Alc1 induces local chromatin relaxation upon DNA damage". Mol. Biol. Cell. 27 (24): 3791–3799. doi:10.1091/mbc.E16-05-0269. PMC 5170603. PMID 27733626.
- ^ a b Haince JF, McDonald D, Rodrigue A, Déry U, Masson JY, Hendzel MJ, Poirier GG (2008). "PARP1-dependent kinetics of recruitment of MRE11 and NBS1 proteins to multiple DNA damage sites". J. Biol. Chem. 283 (2): 1197–208. doi:10.1074/jbc.M706734200. PMID 18025084.
- ^ a b c Rogakou EP, Pilch DR, Orr AH, Ivanova VS, Bonner WM (1998). "DNA double-stranded breaks induce histone H2AX phosphorylation on serine 139". J. Biol. Chem. 273 (10): 5858–68. doi:10.1074/jbc.273.10.5858. PMID 9488723.
- ^ Mailand N, Bekker-Jensen S, Faustrup H, Melander F, Bartek J, Lukas C, Lukas J (2007). "RNF8 ubiquitylates histones at DNA double-strand breaks and promotes assembly of repair proteins". Cell. 131 (5): 887–900. doi:10.1016/j.cell.2007.09.040. PMID 18001824. S2CID 14232192.
- ^ Luijsterburg MS, Acs K, Ackermann L, Wiegant WW, Bekker-Jensen S, Larsen DH, Khanna KK, van Attikum H, Mailand N, Dantuma NP (2012). "A new non-catalytic role for ubiquitin ligase RNF8 in unfolding higher-order chromatin structure". EMBO J. 31 (11): 2511–27. doi:10.1038/emboj.2012.104. PMC 3365417. PMID 22531782.
- ^ Giresi, Paul G.; Kim, Jonghwan; McDaniell, Ryan M.; Iyer, Vishwanath R.; Lieb, Jason D. (2007-06-01). "FAIRE (Formaldehyde-Assisted Isolation of Regulatory Elements) isolates active regulatory elements from human chromatin". Genome Research. 17 (6): 877–885. doi:10.1101/gr.5533506. ISSN 1088-9051. PMC 1891346. PMID 17179217.
- ^ Galas, D. J.; Schmitz, A. (1978-09-01). "DNAse footprinting: a simple method for the detection of protein-DNA binding specificity". Nucleic Acids Research. 5 (9): 3157–3170. doi:10.1093/nar/5.9.3157. ISSN 0305-1048. PMC 342238. PMID 212715.
- ^ Cui, Kairong; Zhao, Keji (2012-01-01). Genome-wide approaches to determining nucleosome occupancy in metazoans using MNase-Seq. Methods in Molecular Biology. Vol. 833. pp. 413–419. doi:10.1007/978-1-61779-477-3_24. ISBN 978-1-61779-476-6. ISSN 1940-6029. PMC 3541821. PMID 22183607.
- ^ Buenrostro, Jason D.; Giresi, Paul G.; Zaba, Lisa C.; Chang, Howard Y.; Greenleaf, William J. (2013-12-01). "Transposition of native chromatin for fast and sensitive epigenomic profiling of open chromatin, DNA-binding proteins and nucleosome position". Nature Methods. 10 (12): 1213–1218. doi:10.1038/nmeth.2688. ISSN 1548-7105. PMC 3959825. PMID 24097267.
- ^ Mieczkowski J, Cook A, Bowman SK, Mueller B, Alver BH, Kundu S, Deaton AM, Urban JA, Larschan E, Park PJ, Kingston RE, Tolstorukov MY (2016-05-06). "MNase titration reveals differences between nucleosome occupancy and chromatin accessibility". Nature Communications. 7: 11485. Bibcode:2016NatCo...711485M. doi:10.1038/ncomms11485. PMC 4859066. PMID 27151365.
- ^ Racko D, Benedetti F, Goundaroulis D, Stasiak A (2018). "Chromatin Loop Extrusion and Chromatin Unknotting". Polymers. 10 (10): 1126–1137. doi:10.3390/polym10101126. PMC 6403842. PMID 30961051.
- ^ Luijsterburg, Martijn S.; White, Malcolm F.; van Driel, Roel; Dame, Remus Th. (8 January 2009). "The Major Architects of Chromatin: Architectural Proteins in Bacteria, Archaea and Eukaryotes". Critical Reviews in Biochemistry and Molecular Biology. 43 (6): 393–418. doi:10.1080/10409230802528488. PMID 19037758. S2CID 85874882.
- ^ '토마스 헌트 모건과 그의 유산'Nobelprize.org. 2012년 9월 7일
기타 소스
- 쿠퍼, 제프리 M. 2000더 셀, 제2판, 분자적 접근.4.2 염색체와 염색질.
- Corces, V. G. (1995). "Chromatin insulators. Keeping enhancers under control". Nature. 376 (6540): 462–463. Bibcode:1995Natur.376..462C. doi:10.1038/376462a0. PMID 7637775. S2CID 26494996.
- 크레머, T. 1985Von der Zellenlehre zur Chromotomentheory:Fer Frühen Zell-und Verererbungsforschung, Veröffentlichen aus der Forschungsstelle für Convertische Pathologie der Heidelberger Akademie der Wissenschaften의 Erkentis undientschschschel.스프링거-블로그, 베를린, 하이델베르크
- 엘긴, S.C.R. (에드). 1995.Chromatin Structure and Gene Express, vol. 9. 옥스포드, 뉴욕, 도쿄, IRL Press.
- Gerasimova, T. I.; Corces, V. G. (1996). "Boundary and insulator elements in chromosomes". Curr. Opin. Genet. Dev. 6 (2): 185–192. doi:10.1016/s0959-437x(96)80049-9. PMID 8722175.
- Gerasimova, T. I.; Corces, V. G. (1998). "Polycomb and Trithorax group proteins mediate the function of a chromatin insulator". Cell. 92 (4): 511–521. doi:10.1016/s0092-8674(00)80944-7. PMID 9491892. S2CID 8192263.
- Gerasimova, T. I.; Corces, V. G. (2001). "CHROMATIN INSULATORS AND BOUNDARIES: Effects on Transcription and Nuclear Organization". Annu Rev Genet. 35: 193–208. doi:10.1146/annurev.genet.35.102401.090349. PMID 11700282. S2CID 22738830.
- Gerasimova, T. I.; Byrd, K.; Corces, V. G. (2000). "A chromatin insulator determines the nuclear localization of DNA [In Process Citation]". Mol Cell. 6 (5): 1025–35. doi:10.1016/s1097-2765(00)00101-5. PMID 11106742.
- Ha, S. C.; Lowenhaupt, K.; Rich, A.; Kim, Y. G.; Kim, K. K. (2005). "Crystal structure of a junction between B-DNA and Z-DNA reveals two extruded bases". Nature. 437 (7062): 1183–6. Bibcode:2005Natur.437.1183H. doi:10.1038/nature04088. PMID 16237447. S2CID 2539819.
- 폴라드, T., W. 언쇼.2002년, 세포생물학손더스.
- Saumweber, H. 1987.세포간핵에서의 염색체 배열, 페이지 223-234.W. 헤닉(ed.), 진핵 염색체의 구조와 기능, 제14권.스프링거-벨라그, 베를린, 하이델베르크
- Sinden, R. R. (2005). "Molecular biology: DNA twists and flips". Nature. 437 (7062): 1097–8. Bibcode:2005Natur.437.1097S. doi:10.1038/4371097a. PMID 16237426. S2CID 4409092.
- 반 홀데 KE 1989년염색질뉴욕: Springer-Verlag.ISBN 0-387-96694-3.
- Van Holde, K., J. 즐라타노바, G. 아렌츠, E.무드리아나키스.1995. 염색질 구조의 요소: 히스톤, 뉴클레오솜 및 섬유, 페이지 1-26.S. C. R. Elgin(ed.)에서 염색질 구조와 유전자 발현.옥스포드 대학 출판부의 IRL 프레스입니다.
외부 링크
- 크로마틴, 히스톤 및 카테신; PMAP 단백질 분해 지도 애니메이션
- 네이처 저널: 최근 크로마틴 출판물 및 뉴스
- 체외 염색질 조립체 프로토콜
- ENCODE는 전사인자 바인딩 사이트에서 탐색기 크로마틴 패턴을 스레드합니다.자연 (저널)