TEAD1
TEAD1| TEAD1 | |||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
 | |||||||||||||||||||||||||||||||||||||||||||||||||||
| |||||||||||||||||||||||||||||||||||||||||||||||||||
| 식별자 | |||||||||||||||||||||||||||||||||||||||||||||||||||
| 에일리어스 | TEAD1, AA, NTEF-1, REF1, TCF-13, TCF13, TEAD-1, TEF-1, TEA 도메인 전사 인자 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||
| 외부 ID | OMIM : 189967 MGI : 101876 HomoloGene : 2418 GeneCard : TEAD 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||
| |||||||||||||||||||||||||||||||||||||||||||||||||||
| |||||||||||||||||||||||||||||||||||||||||||||||||||
| |||||||||||||||||||||||||||||||||||||||||||||||||||
| |||||||||||||||||||||||||||||||||||||||||||||||||||
| |||||||||||||||||||||||||||||||||||||||||||||||||||
| 위키데이터 | |||||||||||||||||||||||||||||||||||||||||||||||||||
| |||||||||||||||||||||||||||||||||||||||||||||||||||
TEA 도메인 패밀리 멤버 1(TEAD1) 및 TECF-13(TCF-13)이라고도 불리는 전사 인자인자 TEF-1은 TEAD1 [5][6][7][8]유전자에 의해 인체 내에서 암호화되는 단백질이다.TEAD1은 TEAD 계열의 전사 인자가 확인된 [5][9]첫 번째 멤버였다.
구조.
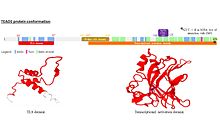
TEAD 패밀리의 모든 구성원은 TEA [10]도메인이라고 불리는 고도로 보존된 DNA 결합 도메인을 공유합니다.이 DNA 결합 도메인은 MCAT [11]요소라고 불리는 컨센서스 DNA 배열 5'-CATTCCA/T-3'을 가지고 있다.TEA 도메인의 3차원 구조가 확인되었다[5].그 배치는 호메오도메인에 가깝고 3개의 α헬릭스(H1, H2, H3)를 포함한다.TEAD 단백질이 [12]DNA와 결합할 수 있도록 하는 것은 H3나선이다.
TEAD1의 또 다른 보존 도메인은 단백질의 C 말단에 위치한다.이것은 보조 인자의 결합을 허용하고 YAP1 결합 도메인으로 불리는데, 그 이유는 TEAD 단백질의 식별을 이끈 잘 알려진 결합 인자의 결합 능력이기 때문이다.실제로 TEAD 단백질은 스스로 유전자 발현을 유도할 수 없다.그들은 행동할 수 있도록[13] 보조 요인들과 연계해야 한다.
조직 분포
TEAD1은 골격근, 췌장, 태반, 폐,[14][15][16][17][18][19][20] 심장을 포함한 다양한 조직에서 발현된다.
맞춤법
TEAD 단백질은 다른 기능을 가정하여 다른 이름으로 많은 유기체에서 발견됩니다.예를 들어, 사카로미세스 세레비시아에 TEC-1은 전이성 요소 TY1을 조절하고 의사 하이팔 성장(영양소가 [21]부족한 조건에서 성장했을 때 효모가 취하는 가늘고 긴 형태)에 관여한다.Aspergillus nidulans에서 TEA 도메인 단백질 ABAA는 원추세포의 [22]분화를 조절한다.드로소필라에서 스칼라형 전사인자는 날개 디스크의 발달, 생존 및 세포 [23]성장에 관여합니다.마지막으로 Xenopus에서는 TEAD1의 맞춤법이 근육 [24]분화를 조절한다는 것이 입증되었다.
기능.
- 심장발달(심근의 [25]분화),
- 골격근 발달(골격근의 알파액틴),[26][27][28]
- 평활근육 발달(평활근의 [26][29]알파액틴),
- 미오신 중쇄유전자,[30] 심근유전자 트로포닌T 및 I의 조절
- 증식 [31][32][33]조절,
- 아포토시스 조절,[34][35]
번역 후 수정
단백질인산화효소A(pKA)는 TEA 도메인 후 세린102에서 TEAD1을 인산화할 수 있다.이 인산화 작용은 [36]α MyHC 유전자의 전사 활성화에 필요하다.단백질인산화효소C(pKC)는 TEA 도메인의 마지막 알파 루프 옆에 있는 세린과 트레오닌에서 TEAD1을 인산화한다.이 인산화 작용은 GTIIC [37]강화제에 대한 TEAD1 결합을 감소시킨다.TEAD1은 단백질의 C단계의 보존된 시스테인 상에서 팔미토일화될 수 있다.이 번역 후 수정은 TEAD 단백질의 적절한 접힘과 [38]안정성에 매우 중요하다.
보조 요인
TEAD 단백질은 표적 [14]유전자의 전사를 유도하기 위해 보조 인자를 필요로 한다.TEAD1은 SRC 계열의 모든 스테로이드 수용체 공활성제와 상호작용한다.HeLa 세포 TEAD1 및 SRC가 유전자 [39]발현을 유도하는 경우 TEAD1은 PARP(Poly-ADP 리보오스 중합효소)와 상호작용하여 평활근α-액틴 발현을 조절한다.PARP는 또한 TEAD 단백질을 ADP-리보실화하여 히스톤 [40]수식을 통해 전사에 유리한 크로마틴 컨텍스트, SRF(Serum response factor) 및 TEAD1을 함께 유전자 [41]발현을 조절한다.
TEAD 단백질과 MEF2(근구증강인자 2)는 물리적으로 상호작용한다.MEF2의 DNA 결합은 MEF2 결합 부위에 인접한 MCAT 배열에서 TEAD1의 신병을 유도하고 강화한다.이 모집은 MLC2v(Myosin Light Chain 2 v) 및 βMHC([42]β-myosin 중쇄) 프로모터의 억제로 이어진다.TEAD1과 인단백질 MAX는 체내 및 체외에서 상호작용한다.일단 이 복합체가 형성되면, 이 두 단백질은 알파 미오신 헤비 체인(α-MHC) 유전자 [43]발현을 조절할 수 있다.
4개의 베스티지얼 유사(VGLL) 단백질은 모든 [44]TEAD와 상호작용할 수 있다.TEAD와 VGLL의 상호작용의 정확한 기능은 아직 잘 알려져 있지 않다.TEAD/VGLL1 복합체는 전립선암 세포주에서 암 진행의 역할을 시사하는 앵커리지 비의존적 세포 증식을 촉진하는 것으로 나타났다. 또한 TEAD1과의 VGLL2 상호작용은 C2C12 분화 시 근육 프로모터를 활성화하고 10T1/[46]2에서 MyoD 매개 근원성을 강화한다.마지막으로 복잡한 TEAD/VGLL4는 기본 문자 변환 억제기로 [47]기능합니다.
YAP(Yes Associated Protein 65), TAZ(Yes Associated Protein 65), YAP에 대한 전사공활성화제 패럴로그(TAZ) 및 모든 TEAD 단백질 간의 상호작용이 시험관내 및 생체내 모두에서 입증되었다.두 경우 모두 단백질의 상호작용은 TEAD 전사 [47][48]활성을 증가시킨다.YAP/TAZ는 세포 증식을 억제하고 포유류와 드로소필라에서 [31][49]아포토시스를 촉진함으로써 장기 성장을 제한하는 히포 종양 억제 경로의 효과자이다.
암에서의 역할
암 전사체 데이터베이스(www.ebi.ac.uk/gxa))의 분석 결과 TEAD1은 여러 유형의 암에서 조절이 잘 되지 않았다.먼저 카포시 육종에서 TEAD1 수치가 300배 증가했다.또한 기저유사 유방암, 나팔관암,[50][51][52] 생식세포 [53]종양에서 TEAD 발현 증가를 검출할 수 있다.그렇지 않으면 다른 유형의 암, 예를 들어 다른 유형의 유방암 및 신장암 또는 방광암에서 TEAD 발현이 감소합니다.이 이중 역할은 TEAD 전사 [35][54]인자에 의한 표적 유전자의 차이 조절과 다른 표적에 의해 설명될 수 있다.마지막으로 최근 연구에서 난소암에서 TEAD1과 YAP가 세포줄기와 화학저항을 [55]유도할 수 있다는 것이 밝혀졌다.TEAD 단백질과 YAP의 유전적 변이가 일부 [56]암에서 풍부합니다.
메모들
이 기사의 2016년 버전은 외부 전문가에 의해 이중 출판 모델로 업데이트되었습니다.대응하는 학술 동료 리뷰 기사는 Gene에 게재되었으며 다음과 같이 인용할 수 있다. André Landin-Malt; Ataaillah Benhaddou; Alain Zider; Domenico Flagiello (14 July 2016). "An evolutionary, structural and functional overview of the mammalian TEAD1 and TEAD2 transcription factors". Gene. Gene Wiki Review Series. 591 (1): 292–303. doi:10.1016/J.GENE.2016.07.028. ISSN 0378-1119. PMC 7034536. PMID 27421669. Wikidata Q30276357. |
레퍼런스
- ^ a b c GRCh38: 앙상블 릴리즈 89: ENSG00000187079 - 앙상블, 2017년 5월
- ^ a b c GRCm38: 앙상블 릴리즈 89: ENSMUSG000055320 - 앙상블, 2017년 5월
- ^ "Human PubMed Reference:". National Center for Biotechnology Information, U.S. National Library of Medicine.
- ^ "Mouse PubMed Reference:". National Center for Biotechnology Information, U.S. National Library of Medicine.
- ^ a b Xiao JH, Davidson I, Matthes H, Garnier JM, Chambon P (May 1991). "Cloning, expression, and transcriptional properties of the human enhancer factor TEF-1". Cell. 65 (4): 551–68. doi:10.1016/0092-8674(91)90088-G. PMID 1851669. S2CID 34258565.
- ^ Jacquemin P, Depetris D, Mattei MG, Martial JA, Davidson I (Jan 1999). "Localization of human transcription factor TEF-4 and TEF-5 (TEAD2, TEAD3) genes to chromosomes 19q13.3 and 6p21.2 using fluorescence in situ hybridization and radiation hybrid analysis". Genomics. 55 (1): 127–9. doi:10.1006/geno.1998.5628. PMID 9889009.
- ^ Fossdal R, Jonasson F, Kristjansdottir GT, Kong A, Stefansson H, Gosh S, Gulcher JR, Stefansson K (May 2004). "A novel TEAD1 mutation is the causative allele in Sveinsson's chorioretinal atrophy (helicoid peripapillary chorioretinal degeneration)". Human Molecular Genetics. 13 (9): 975–81. doi:10.1093/hmg/ddh106. PMID 15016762.
- ^ "Entrez Gene: TEAD1 TEA domain family member 1 (SV40 transcriptional enhancer factor)".
- ^ a b Mar JH, Ordahl CP (September 1988). "A conserved CATTCCT motif is required for skeletal muscle-specific activity of the cardiac troponin T gene promoter". Proceedings of the National Academy of Sciences of the United States of America. 85 (17): 6404–8. Bibcode:1988PNAS...85.6404M. doi:10.1073/pnas.85.17.6404. PMC 281980. PMID 3413104.
- ^ Hwang JJ, Chambon P, Davidson I (June 1993). "Characterization of the transcription activation function and the DNA binding domain of transcriptional enhancer factor-1". The EMBO Journal. 12 (6): 2337–48. doi:10.1002/j.1460-2075.1993.tb05888.x. PMC 413464. PMID 8389695.
- ^ Farrance IK, Mar JH, Ordahl CP (August 1992). "M-CAT binding factor is related to the SV40 enhancer binding factor, TEF-1". The Journal of Biological Chemistry. 267 (24): 17234–40. doi:10.1016/S0021-9258(18)41917-5. PMID 1324927.
- ^ Anbanandam A, Albarado DC, Nguyen CT, Halder G, Gao X, Veeraraghavan S (November 2006). "Insights into transcription enhancer factor 1 (TEF-1) activity from the solution structure of the TEA domain". Proceedings of the National Academy of Sciences of the United States of America. 103 (46): 17225–30. Bibcode:2006PNAS..10317225A. doi:10.1073/pnas.0607171103. PMC 1859914. PMID 17085591.
- ^ Azakie A, Lamont L, Fineman JR, He Y (December 2005). "Divergent transcriptional enhancer factor-1 regulates the cardiac troponin T promoter". American Journal of Physiology. Cell Physiology. 289 (6): C1522–34. doi:10.1152/ajpcell.00126.2005. PMID 16049055.
- ^ a b Xiao JH, Davidson I, Matthes H, Garnier JM, Chambon P (May 1991). "Cloning, expression, and transcriptional properties of the human enhancer factor TEF-1". Cell. 65 (4): 551–68. doi:10.1016/0092-8674(91)90088-g. PMID 1851669. S2CID 34258565.
- ^ Jacquemin P, Hwang JJ, Martial JA, Dollé P, Davidson I (September 1996). "A novel family of developmentally regulated mammalian transcription factors containing the TEA/ATTS DNA binding domain". The Journal of Biological Chemistry. 271 (36): 21775–85. doi:10.1074/jbc.271.36.21775. PMID 8702974.
- ^ Stewart AF, Richard CW, Suzow J, Stephan D, Weremowicz S, Morton CC, Adra CN (October 1996). "Cloning of human RTEF-1, a transcriptional enhancer factor-1-related gene preferentially expressed in skeletal muscle: evidence for an ancient multigene family". Genomics. 37 (1): 68–76. doi:10.1006/geno.1996.0522. PMID 8921372.
- ^ Yasunami M, Suzuki K, Houtani T, Sugimoto T, Ohkubo H (August 1995). "Molecular characterization of cDNA encoding a novel protein related to transcriptional enhancer factor-1 from neural precursor cells". The Journal of Biological Chemistry. 270 (31): 18649–54. doi:10.1074/jbc.270.31.18649. PMID 7629195.
- ^ Yasunami M, Suzuki K, Ohkubo H (November 1996). "A novel family of TEA domain-containing transcription factors with distinct spatiotemporal expression patterns". Biochemical and Biophysical Research Communications. 228 (2): 365–70. doi:10.1006/bbrc.1996.1667. PMID 8920920.
- ^ Yockey CE, Smith G, Izumo S, Shimizu N (February 1996). "cDNA cloning and characterization of murine transcriptional enhancer factor-1-related protein 1, a transcription factor that binds to the M-CAT motif". The Journal of Biological Chemistry. 271 (7): 3727–36. doi:10.1074/jbc.271.7.3727. PMID 8631987.
- ^ Azakie A, Lamont L, Fineman JR, He Y (December 2005). "Divergent transcriptional enhancer factor-1 regulates the cardiac troponin T promoter". American Journal of Physiology. Cell Physiology. 289 (6): C1522-34. doi:10.1152/ajpcell.00126.2005. PMID 16049055.
- ^ Laloux I, Dubois E, Dewerchin M, Jacobs E (July 1990). "TEC1, a gene involved in the activation of Ty1 and Ty1-mediated gene expression in Saccharomyces cerevisiae: cloning and molecular analysis". Molecular and Cellular Biology. 10 (7): 3541–50. doi:10.1128/mcb.10.7.3541. PMC 360789. PMID 2192259.
- ^ Boylan MT, Mirabito PM, Willett CE, Zimmerman CR, Timberlake WE (September 1987). "Isolation and physical characterization of three essential conidiation genes from Aspergillus nidulans". Molecular and Cellular Biology. 7 (9): 3113–8. doi:10.1128/mcb.7.9.3113. PMC 367944. PMID 2823119.
- ^ Goulev Y, Fauny JD, Gonzalez-Marti B, Flagiello D, Silber J, Zider A (March 2008). "SCALLOPED interacts with YORKIE, the nuclear effector of the hippo tumor-suppressor pathway in Drosophila". Current Biology. 18 (6): 435–41. doi:10.1016/j.cub.2008.02.034. PMID 18313299. S2CID 16369642.
- ^ Naye F, Tréguer K, Soulet F, Faucheux C, Fédou S, Thézé N, Thiébaud P (2007). "Differential expression of two TEF-1 (TEAD) genes during Xenopus laevis development and in response to inducing factors". The International Journal of Developmental Biology. 51 (8): 745–52. doi:10.1387/ijdb.072375fn. PMID 17939122.
- ^ Chen Z, Friedrich GA, Soriano P (October 1994). "Transcriptional enhancer factor 1 disruption by a retroviral gene trap leads to heart defects and embryonic lethality in mice". Genes & Development. 8 (19): 2293–301. doi:10.1101/gad.8.19.2293. PMID 7958896.
- ^ a b Jiang SW, Trujillo MA, Sakagashira M, Wilke RA, Eberhardt NL (March 2000). "Novel human TEF-1 isoforms exhibit altered DNA binding and functional properties". Biochemistry. 39 (12): 3505–13. doi:10.1021/bi991048w. PMID 10727247.
- ^ Karns LR, Kariya K, Simpson PC (January 1995). "M-CAT, CArG, and Sp1 elements are required for alpha 1-adrenergic induction of the skeletal alpha-actin promoter during cardiac myocyte hypertrophy. Transcriptional enhancer factor-1 and protein kinase C as conserved transducers of the fetal program in cardiac growth". The Journal of Biological Chemistry. 270 (1): 410–7. doi:10.1074/jbc.270.1.410. PMID 7814403.
- ^ Benhaddou A, Keime C, Ye T, Morlon A, Michel I, Jost B, Mengus G, Davidson I (February 2012). "Transcription factor TEAD4 regulates expression of myogenin and the unfolded protein response genes during C2C12 cell differentiation". Cell Death and Differentiation. 19 (2): 220–31. doi:10.1038/cdd.2011.87. PMC 3263497. PMID 21701496.
- ^ Swartz EA, Johnson AD, Owens GK (August 1998). "Two MCAT elements of the SM alpha-actin promoter function differentially in SM vs. non-SM cells". The American Journal of Physiology. 275 (2 Pt 1): C608-18. doi:10.1152/ajpcell.1998.275.2.C608. PMID 9688616.
- ^ Rindt H, Gulick J, Knotts S, Neumann J, Robbins J (March 1993). "In vivo analysis of the murine beta-myosin heavy chain gene promoter". The Journal of Biological Chemistry. 268 (7): 5332–8. doi:10.1016/S0021-9258(18)53537-7. PMID 8444907.
- ^ a b Yu FX, Zhao B, Guan KL (November 2015). "Hippo Pathway in Organ Size Control, Tissue Homeostasis, and Cancer". Cell. 163 (4): 811–28. doi:10.1016/j.cell.2015.10.044. PMC 4638384. PMID 26544935.
- ^ Landin Malt A, Cagliero J, Legent K, Silber J, Zider A, Flagiello D (2012). "Alteration of TEAD1 expression levels confers apoptotic resistance through the transcriptional up-regulation of Livin". PLOS ONE. 7 (9): e45498. Bibcode:2012PLoSO...745498L. doi:10.1371/journal.pone.0045498. PMC 3454436. PMID 23029054.
- ^ Zhao B, Li L, Lei Q, Guan KL (May 2010). "The Hippo-YAP pathway in organ size control and tumorigenesis: an updated version". Genes & Development. 24 (9): 862–74. doi:10.1101/gad.1909210. PMC 2861185. PMID 20439427.
- ^ Landin Malt A, Cagliero J, Legent K, Silber J, Zider A, Flagiello D (2012). "Alteration of TEAD1 expression levels confers apoptotic resistance through the transcriptional up-regulation of Livin". PLOS ONE. 7 (9): e45498. Bibcode:2012PLoSO...745498L. doi:10.1371/journal.pone.0045498. PMC 3454436. PMID 23029054.
- ^ a b Landin Malt A, Georges A, Silber J, Zider A, Flagiello D (October 2013). "Interaction with the Yes-associated protein (YAP) allows TEAD1 to positively regulate NAIP expression". FEBS Letters. 587 (19): 3216–23. doi:10.1016/j.febslet.2013.08.013. PMID 23994529. S2CID 23797089.
- ^ Gupta MP, Gupta M, Dizon E, Zak R (1996). "Sympathetic control of cardiac myosin heavy chain gene expression". Molecular and Cellular Biochemistry. 157 (1–2): 117–24. doi:10.1007/bf00227889. PMID 8739237. S2CID 10103037.
- ^ Jiang SW, Dong M, Trujillo MA, Miller LJ, Eberhardt NL (June 2001). "DNA binding of TEA/ATTS domain factors is regulated by protein kinase C phosphorylation in human choriocarcinoma cells". The Journal of Biological Chemistry. 276 (26): 23464–70. doi:10.1074/jbc.M010934200. PMID 11313339.
- ^ Noland CL, Gierke S, Schnier PD, Murray J, Sandoval WN, Sagolla M, Dey A, Hannoush RN, Fairbrother WJ, Cunningham CN (January 2016). "Palmitoylation of TEAD Transcription Factors Is Required for Their Stability and Function in Hippo Pathway Signaling". Structure. 24 (1): 179–86. doi:10.1016/j.str.2015.11.005. PMID 26724994.
- ^ Belandia B, Parker MG (October 2000). "Functional interaction between the p160 coactivator proteins and the transcriptional enhancer factor family of transcription factors". The Journal of Biological Chemistry. 275 (40): 30801–5. doi:10.1074/jbc.C000484200. PMID 10934189.
- ^ Butler AJ, Ordahl CP (January 1999). "Poly(ADP-ribose) polymerase binds with transcription enhancer factor 1 to MCAT1 elements to regulate muscle-specific transcription". Molecular and Cellular Biology. 19 (1): 296–306. doi:10.1128/mcb.19.1.296. PMC 83887. PMID 9858553.
- ^ MacLellan WR, Lee TC, Schwartz RJ, Schneider MD (June 1994). "Transforming growth factor-beta response elements of the skeletal alpha-actin gene. Combinatorial action of serum response factor, YY1, and the SV40 enhancer-binding protein, TEF-1". The Journal of Biological Chemistry. 269 (24): 16754–60. doi:10.1016/S0021-9258(19)89455-3. PMID 8206998.
- ^ Maeda T, Chapman DL, Stewart AF (December 2002). "Mammalian vestigial-like 2, a cofactor of TEF-1 and MEF2 transcription factors that promotes skeletal muscle differentiation". The Journal of Biological Chemistry. 277 (50): 48889–98. doi:10.1074/jbc.M206858200. PMID 12376544.
- ^ Gupta MP, Amin CS, Gupta M, Hay N, Zak R (July 1997). "Transcription enhancer factor 1 interacts with a basic helix-loop-helix zipper protein, Max, for positive regulation of cardiac alpha-myosin heavy-chain gene expression". Molecular and Cellular Biology. 17 (7): 3924–36. doi:10.1128/mcb.17.7.3924. PMC 232245. PMID 9199327.
- ^ Chen L, Chan SW, Zhang X, Walsh M, Lim CJ, Hong W, Song H (February 2010). "Structural basis of YAP recognition by TEAD4 in the hippo pathway". Genes & Development. 24 (3): 290–300. doi:10.1101/gad.1865310. PMC 2811830. PMID 20123908.
- ^ Pobbati AV, Chan SW, Lee I, Song H, Hong W (July 2012). "Structural and functional similarity between the Vgll1-TEAD and the YAP-TEAD complexes". Structure. 20 (7): 1135–40. doi:10.1016/j.str.2012.04.004. PMID 22632831.
- ^ Günther S, Mielcarek M, Krüger M, Braun T (2004). "VITO-1 is an essential cofactor of TEF1-dependent muscle-specific gene regulation". Nucleic Acids Research. 32 (2): 791–802. doi:10.1093/nar/gkh248. PMC 373362. PMID 14762206.
- ^ a b Koontz LM, Liu-Chittenden Y, Yin F, Zheng Y, Yu J, Huang B, Chen Q, Wu S, Pan D (May 2013). "The Hippo effector Yorkie controls normal tissue growth by antagonizing scalloped-mediated default repression". Developmental Cell. 25 (4): 388–401. doi:10.1016/j.devcel.2013.04.021. PMC 3705890. PMID 23725764.
- ^ Vassilev A, Kaneko KJ, Shu H, Zhao Y, DePamphilis ML (May 2001). "TEAD/TEF transcription factors utilize the activation domain of YAP65, a Src/Yes-associated protein localized in the cytoplasm". Genes & Development. 15 (10): 1229–41. doi:10.1101/gad.888601. PMC 313800. PMID 11358867.
- ^ Zhao B, Li L, Lei Q, Guan KL (May 2010). "The Hippo-YAP pathway in organ size control and tumorigenesis: an updated version". Genes & Development. 24 (9): 862–74. doi:10.1101/gad.1909210. PMC 2861185. PMID 20439427.
- ^ Han W, Jung EM, Cho J, Lee JW, Hwang KT, Yang SJ, Kang JJ, Bae JY, Jeon YK, Park IA, Nicolau M, Jeffrey SS, Noh DY (June 2008). "DNA copy number alterations and expression of relevant genes in triple-negative breast cancer". Genes, Chromosomes & Cancer. 47 (6): 490–9. doi:10.1002/gcc.20550. PMID 18314908. S2CID 24749682.
- ^ Richardson AL, Wang ZC, De Nicolo A, Lu X, Brown M, Miron A, Liao X, Iglehart JD, Livingston DM, Ganesan S (February 2006). "X chromosomal abnormalities in basal-like human breast cancer". Cancer Cell. 9 (2): 121–32. doi:10.1016/j.ccr.2006.01.013. PMID 16473279.
- ^ Nowee ME, Snijders AM, Rockx DA, de Wit RM, Kosma VM, Hämäläinen K, Schouten JP, Verheijen RH, van Diest PJ, Albertson DG, Dorsman JC (September 2007). "DNA profiling of primary serous ovarian and fallopian tube carcinomas with array comparative genomic hybridization and multiplex ligation-dependent probe amplification". The Journal of Pathology. 213 (1): 46–55. doi:10.1002/path.2217. PMID 17668415. S2CID 27198301.
- ^ Skotheim RI, Autio R, Lind GE, Kraggerud SM, Andrews PW, Monni O, Kallioniemi O, Lothe RA (2006). "Novel genomic aberrations in testicular germ cell tumors by array-CGH, and associated gene expression changes". Cellular Oncology. 28 (5–6): 315–26. doi:10.1155/2006/219786. PMC 4615958. PMID 17167184.
- ^ Landin Malt A, Cagliero J, Legent K, Silber J, Zider A, Flagiello D (2012). "Alteration of TEAD1 expression levels confers apoptotic resistance through the transcriptional up-regulation of Livin". PLOS ONE. 7 (9): e45498. Bibcode:2012PLoSO...745498L. doi:10.1371/journal.pone.0045498. PMC 3454436. PMID 23029054.
- ^ Xia Y, Zhang YL, Yu C, Chang T, Fan HY (2014). "YAP/TEAD co-activator regulated pluripotency and chemoresistance in ovarian cancer initiated cells". PLOS ONE. 9 (11): e109575. Bibcode:2014PLoSO...9j9575X. doi:10.1371/journal.pone.0109575. PMC 4219672. PMID 25369529.
- ^ Yuan H, Liu H, Liu Z, Zhu D, Amos CI, Fang S, Lee JE, Wei Q (August 2015). "Genetic variants in Hippo pathway genes YAP1, TEAD1 and TEAD4 are associated with melanoma-specific survival". International Journal of Cancer. 137 (3): 638–45. doi:10.1002/ijc.29429. PMC 4437894. PMID 25628125.
추가 정보
- Boam DS, Davidson I, Chambon P (August 1995). "A TATA-less promoter containing binding sites for ubiquitous transcription factors mediates cell type-specific regulation of the gene for transcription enhancer factor-1 (TEF-1)". The Journal of Biological Chemistry. 270 (33): 19487–94. doi:10.1074/jbc.270.33.19487. PMID 7642633.
- Fossdal R, Magnússon L, Weber JL, Jensson O (March 1995). "Mapping the locus of atrophia areata, a helicoid peripapillary chorioretinal degeneration with autosomal dominant inheritance, to chromosome 11p15". Human Molecular Genetics. 4 (3): 479–83. doi:10.1093/hmg/4.3.479. PMID 7795606.
- Kariya K, Farrance IK, Simpson PC (December 1993). "Transcriptional enhancer factor-1 in cardiac myocytes interacts with an alpha 1-adrenergic- and beta-protein kinase C-inducible element in the rat beta-myosin heavy chain promoter". The Journal of Biological Chemistry. 268 (35): 26658–62. doi:10.1016/S0021-9258(19)74362-2. PMID 8253797.
- Shimizu N, Smith G, Izumo S (August 1993). "Both a ubiquitous factor mTEF-1 and a distinct muscle-specific factor bind to the M-CAT motif of the myosin heavy chain beta gene". Nucleic Acids Research. 21 (17): 4103–10. doi:10.1093/nar/21.17.4103. PMC 310013. PMID 8396764.
- Stewart AF, Richard CW, Suzow J, Stephan D, Weremowicz S, Morton CC, Adra CN (October 1996). "Cloning of human RTEF-1, a transcriptional enhancer factor-1-related gene preferentially expressed in skeletal muscle: evidence for an ancient multigene family". Genomics. 37 (1): 68–76. doi:10.1006/geno.1996.0522. PMID 8921372.
- Gupta MP, Amin CS, Gupta M, Hay N, Zak R (July 1997). "Transcription enhancer factor 1 interacts with a basic helix-loop-helix zipper protein, Max, for positive regulation of cardiac alpha-myosin heavy-chain gene expression". Molecular and Cellular Biology. 17 (7): 3924–36. doi:10.1128/mcb.17.7.3924. PMC 232245. PMID 9199327.
- Simmonds AJ, Liu X, Soanes KH, Krause HM, Irvine KD, Bell JB (December 1998). "Molecular interactions between Vestigial and Scalloped promote wing formation in Drosophila". Genes & Development. 12 (24): 3815–20. doi:10.1101/gad.12.24.3815. PMC 317270. PMID 9869635.
- Vaudin P, Delanoue R, Davidson I, Silber J, Zider A (November 1999). "TONDU (TDU), a novel human protein related to the product of vestigial (vg) gene of Drosophila melanogaster interacts with vertebrate TEF factors and substitutes for Vg function in wing formation". Development. 126 (21): 4807–16. doi:10.1242/dev.126.21.4807. PMID 10518497.
- Gupta M, Kogut P, Davis FJ, Belaguli NS, Schwartz RJ, Gupta MP (March 2001). "Physical interaction between the MADS box of serum response factor and the TEA/ATTS DNA-binding domain of transcription enhancer factor-1". The Journal of Biological Chemistry. 276 (13): 10413–22. doi:10.1074/jbc.M008625200. PMID 11136726.
- Vassilev A, Kaneko KJ, Shu H, Zhao Y, DePamphilis ML (May 2001). "TEAD/TEF transcription factors utilize the activation domain of YAP65, a Src/Yes-associated protein localized in the cytoplasm". Genes & Development. 15 (10): 1229–41. doi:10.1101/gad.888601. PMC 313800. PMID 11358867.
- Carlini LE, Getz MJ, Strauch AR, Kelm RJ (March 2002). "Cryptic MCAT enhancer regulation in fibroblasts and smooth muscle cells. Suppression of TEF-1 mediated activation by the single-stranded DNA-binding proteins, Pur alpha, Pur beta, and MSY1". The Journal of Biological Chemistry. 277 (10): 8682–92. doi:10.1074/jbc.M109754200. PMID 11751932.
- Maeda T, Gupta MP, Stewart AF (June 2002). "TEF-1 and MEF2 transcription factors interact to regulate muscle-specific promoters". Biochemical and Biophysical Research Communications. 294 (4): 791–7. doi:10.1016/S0006-291X(02)00556-9. PMID 12061776.
- Maeda T, Chapman DL, Stewart AF (December 2002). "Mammalian vestigial-like 2, a cofactor of TEF-1 and MEF2 transcription factors that promotes skeletal muscle differentiation". The Journal of Biological Chemistry. 277 (50): 48889–98. doi:10.1074/jbc.M206858200. PMID 12376544.
- Thompson M, Andrade VA, Andrade SJ, Pusl T, Ortega JM, Goes AM, Leite MF (February 2003). "Inhibition of the TEF/TEAD transcription factor activity by nuclear calcium and distinct kinase pathways". Biochemical and Biophysical Research Communications. 301 (2): 267–74. doi:10.1016/S0006-291X(02)03024-3. PMID 12565854.
- Karasseva N, Tsika G, Ji J, Zhang A, Mao X, Tsika R (August 2003). "Transcription enhancer factor 1 binds multiple muscle MEF2 and A/T-rich elements during fast-to-slow skeletal muscle fiber type transitions". Molecular and Cellular Biology. 23 (15): 5143–64. doi:10.1128/MCB.23.15.5143-5164.2003. PMC 165722. PMID 12861002.
- Günther S, Mielcarek M, Krüger M, Braun T (2004). "VITO-1 is an essential cofactor of TEF1-dependent muscle-specific gene regulation". Nucleic Acids Research. 32 (2): 791–802. doi:10.1093/nar/gkh248. PMC 373362. PMID 14762206.