CTCF
CTCF| CTCF | |||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
 | |||||||||||||||||||||||||||||||
| |||||||||||||||||||||||||||||||
| 식별자 | |||||||||||||||||||||||||||||||
| 에일리어스 | CTCF, MRD21, CCCTC 결합 계수, FAP108, CFAP108 | ||||||||||||||||||||||||||||||
| 외부 ID | OMIM : 604167 MGI : 109447 HomoloGene : 4786 GenCard : CTCF | ||||||||||||||||||||||||||||||
| |||||||||||||||||||||||||||||||
| |||||||||||||||||||||||||||||||
| |||||||||||||||||||||||||||||||
| |||||||||||||||||||||||||||||||
| 위키데이터 | |||||||||||||||||||||||||||||||
| |||||||||||||||||||||||||||||||
11-아연 핑거 단백질 또는 CCCTC 결합 인자로 알려진 전사 억제제 CTCF는 사람에게서 CTCF [5][6]유전자에 의해 암호화되는 전사 인자이다.CTCF는 전사 조절, 절연체 활성, V(D)J 재조합[7] 및 염색질 구조의 [8]조절을 포함한 많은 세포 과정에 관여합니다.
검출
CCCTC-결합인자(CTCF)는 닭 c-myc 유전자의 음성 조절인자로 처음 발견되었다.이 단백질은 코어 배열 CCCTC의 세 번의 규칙적인 간격의 반복에 결합하는 것으로 발견되어 CCCTC 결합 [9]인자로 명명되었다.
기능.
CTCF의 주요 역할은 [8]크로마틴의 3D 구조를 조절하는 것으로 생각됩니다.CTCF는 DNA 가닥을 결합하여 염색질 루프를 형성하고 DNA를 핵 라미나와 [10]같은 세포 구조에 고정시킵니다.그것은 또한 활성 DNA와 이색 DNA 사이의 경계를 정의합니다.
DNA의 3D 구조가 유전자의 조절에 영향을 미치기 때문에 CTCF의 활동은 유전자의 발현에 영향을 미친다.CTCF는 인핸서와 프로모터 간의 상호작용을 차단하는 시퀀스인 절연체 활동의 주요 부분으로 생각됩니다.CTCF 결합은 또한 유전자 발현을 촉진하고 억제하는 것으로 나타났다.CTCF가 단지 그것의 루프 활성을 통해 유전자 발현에 영향을 미치는지, 아니면 알려지지 않은 다른 [8]활성을 가지고 있는지는 알려지지 않았다.최근 연구에서 CTCF는 TAD를 구분하는 것 외에 프로모터-인핸서 루프를 매개하여 종종 프로모터-인핸서 상호작용을 촉진하는 것으로 나타났다.[11]이는 CTCF의 하위 집단이 전사를 활성화하기 위해 RNA 중합효소 II(Pol II) 단백질 복합체와 연관되어 있다는 개념과 일치한다.CTCF는 전사인자 결합 인핸서를 전사 개시 사이트 근위 조절 요소에 브리지하고 Pol II와 대화함으로써 전사를 개시하는 데 도움이 될 수 있습니다.따라서 전사 규제 시퀀스 간의 컨택을 촉진하기 위한 CTCF의 역할을 지원합니다.이 모델은 베타글로빈 궤적에 대한 이전 연구에 의해 입증되었다.
관찰된 활동
CTCF의 바인딩은 다음과 같은 많은 영향을 미치는 것으로 나타났습니다.어느 경우든 CTCF가 직접 결과를 호출하는지, 간접적으로 호출하는지(특히 루프 역할을 통해) 알 수 없습니다.
전사 규제
단백질 CTCF는 차등메틸화영역-1(DMR1) 및 [12][13]MAR3과 함께 H-19 임프린트 제어영역(ICR)에 결합함으로써 인슐린 유사 성장인자 2 유전자를 억제하는 데 큰 역할을 한다.
단열재
CTCF에 의한 타겟팅 시퀀스 요소의 결합은 인핸서와 프로모터 간의 상호작용을 차단할 수 있으며, 따라서 인핸서의 활성을 특정 기능 영역으로 제한할 수 있다.CTCF는 인핸서 블로킹으로 작용할 뿐만 아니라 헤테로크로마틴 구조의 확산을 방지함으로써 염색질[14] 장벽으로도 작용할 수 있다.
염색질 구조 조절
CTCF는 물리적으로 자기 자신과 결합해 호모디머를 [15]형성하고, 이로 인해 결합된 DNA가 [16]루프를 형성합니다.CTCF는 또한 핵 라미나에 [10]결합된 DNA 단면의 경계에서 자주 발생한다.ChIP-seq에 이어 ChIP-seq(Chromatin Immuno-preciptation)를 사용하여 CTCF는 게놈 전체에 걸쳐 응집되어 유전자 조절 메커니즘과 고차 염색질 [17]구조에 영향을 미치는 것으로 밝혀졌다.현재 DNA 루프는 "루프 압출" 메커니즘에 의해 형성되며, 이로 인해 응집된 고리는 CTCF와 만날 때까지 DNA를 따라 활발하게 이동됩니다.CTCF는 응집력을 멈추기 위해 적절한 방향에 있어야 합니다.
RNA 스플라이싱 조절
CTCF 결합은 mRNA [18]스플라이싱에 영향을 미치는 것으로 나타났습니다.
DNA결합
CTCF는 컨센서스 시퀀스 CCGGGNGGCAG에 바인드합니다(IUPAC 표기).[19][20]이 순서는 구조에서 11개의 아연 핑거 모티브로 정의된다.CTCF의 결합은 [21]결합하는 DNA의 CpG 메틸화에 의해 파괴된다.한편, CTCF 결합은 DNA [22]메틸화의 확산을 위한 경계를 설정할 수 있다.최근 연구에서 CTCF 결합 손실은 인간 [23][24][25]게놈에서 CTCF의 또 다른 후생적 리모델링 역할을 반영하는 국소적인 CpG 메틸화를 증가시키는 것으로 보고되었다.
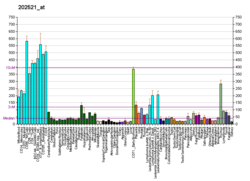
CTCF는 19개의 다양한 세포 유형(정상 12개, 불멸 7개)에서 평균 약 55,000개의 DNA 부위와 19개의 모든 세포 [26]유형에 걸쳐 총 77,811개의 별개의 결합 부위에서 결합합니다.CTCF는 아연 손가락의 다양한 조합을 사용하여 여러 배열에 결합할 수 있는 능력으로 "다가 단백질"[5]의 지위를 얻었다.30,000개 이상의 CTCF 바인딩 사이트가 [27]특징지어졌습니다.인간 게놈은 세포 유형에 따라 15,000개에서 40,000개의 CTCF 결합 부위를 포함하고 있으며, 이는 유전자 [14][19][28]조절에서 CTCF가 광범위한 역할을 한다는 것을 암시한다.또한 CTCF 결합부위는 뉴클레오좀 위치결정 앵커로서 작용하여 다양한 게놈 신호를 배열하기 위해 사용될 때 여러 측면의 뉴클레오솜을 쉽게 [14][29]동정할 수 있다.한편, 고해상도 뉴클레오솜 매핑 연구는 세포 유형 간의 CTCF 결합의 차이가 뉴클레오솜 [30]위치의 차이에 기인할 수 있다는 것을 보여주었다.일부 유전자의 CTCF 결합 부위의 메틸화 손실은 남성 [20]불임을 포함한 인간의 질병과 관련이 있는 것으로 밝혀졌다.
단백질-단백질 상호작용
CTCF는 그 자체에 바인드하여 [15]호모디머를 형성합니다.CTCF는 또한 Y박스 결합 단백질 [31]1과 상호작용하는 것으로 나타났다. CTCF는 또한 적절한 방향으로 [32]CTCF와 만날 때까지 링 모양의 구조를 통해 하나 또는 두 개의 DNA 가닥을 능동적으로 전위시킴으로써 크로마틴 루프를 밀어내는 응집체와 함께 국재화된다.CTCF는 Chd4 및 Snf2h(SMARCA5)[33]와 같은 염색질 재모듈러와도 상호작용하는 것으로 알려져 있다.
레퍼런스
- ^ a b c GRCh38: 앙상블 릴리즈 89: ENSG00000102974 - 앙상블, 2017년 5월
- ^ a b c GRCm38: 앙상블 릴리즈 89: ENSMUSG00000005698 - 앙상블, 2017년 5월
- ^ "Human PubMed Reference:". National Center for Biotechnology Information, U.S. National Library of Medicine.
- ^ "Mouse PubMed Reference:". National Center for Biotechnology Information, U.S. National Library of Medicine.
- ^ a b Filippova GN, Fagerlie S, Klenova EM, Myers C, Dehner Y, Goodwin G, Neiman PE, Collins SJ, Lobanenkov VV (June 1996). "An exceptionally conserved transcriptional repressor, CTCF, employs different combinations of zinc fingers to bind diverged promoter sequences of avian and mammalian c-myc oncogenes". Mol. Cell. Biol. 16 (6): 2802–13. doi:10.1128/mcb.16.6.2802. PMC 231272. PMID 8649389.
- ^ Rubio ED, Reiss DJ, Welcsh PL, Disteche CM, Filippova GN, Baliga NS, Aebersold R, Ranish JA, Krumm A (June 2008). "CTCF physically links cohesin to chromatin". Proc. Natl. Acad. Sci. U.S.A. 105 (24): 8309–14. Bibcode:2008PNAS..105.8309R. doi:10.1073/pnas.0801273105. PMC 2448833. PMID 18550811.
- ^ Chaumeil J, Skok JA (April 2012). "The role of CTCF in regulating V(D)J recombination". Curr. Opin. Immunol. 24 (2): 153–9. doi:10.1016/j.coi.2012.01.003. PMC 3444155. PMID 22424610.
- ^ a b c Phillips JE, Corces VG (June 2009). "CTCF: master weaver of the genome". Cell. 137 (7): 1194–211. doi:10.1016/j.cell.2009.06.001. PMC 3040116. PMID 19563753.
- ^ Lobanenkov VV, Nicolas RH, Adler VV, Paterson H, Klenova EM, Polotskaja AV, Goodwin GH (December 1990). "A novel sequence-specific DNA binding protein which interacts with three regularly spaced direct repeats of the CCCTC-motif in the 5'-flanking sequence of the chicken c-myc gene". Oncogene. 5 (12): 1743–53. PMID 2284094.
- ^ a b Guelen L, Pagie L, Brasset E, Meuleman W, Faza MB, Talhout W, Eussen BH, de Klein A, Wessels L, de Laat W, van Steensel B (June 2008). "Domain organization of human chromosomes revealed by mapping of nuclear lamina interactions". Nature. 453 (7197): 948–51. Bibcode:2008Natur.453..948G. doi:10.1038/nature06947. PMID 18463634. S2CID 4429401.
- ^ Qu J, Yi G, Zhou H (June 2019). "p63 cooperates with CTCF to modulate chromatin architecture in skin keratinocytes". Epigenetics & Chromatin. 12 (1): 31. doi:10.1186/s13072-019-0280-y. PMC 6547520. PMID 31164150.
- ^ Ohlsson R, Renkawitz R, Lobanenkov V (2001). "CTCF is a uniquely versatile transcription regulator linked to epigenetics and disease". Trends Genet. 17 (9): 520–7. doi:10.1016/S0168-9525(01)02366-6. PMID 11525835.
- ^ Dunn KL, Davie JR (2003). "The many roles of the transcriptional regulator CTCF". Biochem. Cell Biol. 81 (3): 161–7. doi:10.1139/o03-052. PMID 12897849.
- ^ a b c Cuddapah S, Jothi R, Schones DE, Roh TY, Cui K, Zhao K (2009). "Global analysis of the insulator binding protein CTCF in chromatin barrier regions reveals demarcation of active and repressive domains". Genome Res. 19 (1): 24–32. doi:10.1101/gr.082800.108. PMC 2612964. PMID 19056695.
- ^ a b Yusufzai TM, Tagami H, Nakatani Y, Felsenfeld G (January 2004). "CTCF tethers an insulator to subnuclear sites, suggesting shared insulator mechanisms across species". Mol. Cell. 13 (2): 291–8. doi:10.1016/S1097-2765(04)00029-2. PMID 14759373.
- ^ Hou C, Zhao H, Tanimoto K, Dean A (December 2008). "CTCF-dependent enhancer-blocking by alternative chromatin loop formation". Proc. Natl. Acad. Sci. U.S.A. 105 (51): 20398–403. Bibcode:2008PNAS..10520398H. doi:10.1073/pnas.0808506106. PMC 2629272. PMID 19074263.
- ^ Lee BK, Iyer VR (September 2012). "Genome-wide studies of CCCTC-binding factor (CTCF) and cohesin provide insight into chromatin structure and regulation". J. Biol. Chem. 287 (37): 30906–13. doi:10.1074/jbc.R111.324962. PMC 3438923. PMID 22952237.
- ^ Shukla S, Kavak E, Gregory M, Imashimizu M, Shutinoski B, Kashlev M, Oberdoerffer P, Sandberg R, Oberdoerffer S (November 2011). "CTCF-promoted RNA polymerase II pausing links DNA methylation to splicing". Nature. 479 (7371): 74–9. Bibcode:2011Natur.479...74S. doi:10.1038/nature10442. PMC 7398428. PMID 21964334.
- ^ a b Kim TH, Abdullaev ZK, Smith AD, Ching KA, Loukinov DI, Green RD, Zhang MQ, Lobanenkov VV, Ren B (March 2007). "Analysis of the vertebrate insulator protein CTCF-binding sites in the human genome". Cell. 128 (6): 1231–45. doi:10.1016/j.cell.2006.12.048. PMC 2572726. PMID 17382889.
- ^ a b Rotondo JC, Selvatici R, Di Domenico M, Marci R, Vesce F, Tognon M, Martini F (September 2013). "Methylation loss at H19 imprinted gene correlates with methylenetetrahydrofolate reductase gene promoter hypermethylation in semen samples from infertile males". Epigenetics. 8 (9): 990–7. doi:10.4161/epi.25798. PMC 3883776. PMID 23975186.
- ^ Bell AC, Felsenfeld G (May 2000). "Methylation of a CTCF-dependent boundary controls imprinted expression of the Igf2 gene". Nature. 405 (6785): 482–5. Bibcode:2000Natur.405..482B. doi:10.1038/35013100. PMID 10839546. S2CID 4387329.
- ^ Wiehle L, Thorn GJ, Raddatz G, Clarkson CT, Rippe K, Lyko F, Breiling A, Teif VB (May 2019). "DNA de-methylation in embryonic stem cells controls CTCF-dependent chromatin boundaries". Genome Research. 29 (5): 750–61. doi:10.1101/gr.239707.118. PMC 6499307. PMID 30948436.
- ^ Tian, Yijun; Soupir, Alex; Liu, Qian; Wu, Lang; Huang, Chiang-Ching; Park, Jong Y; Wang, Liang (29 November 2021). "Novel role of prostate cancer risk variant rs7247241 on PPP1R14A isoform transition through allelic TF binding and CpG methylation". Human Molecular Genetics. 31 (10): 1610–1621. doi:10.1093/hmg/ddab347. PMC 9122641. PMID 34849858.
- ^ Damaschke, Nathan A.; Gawdzik, Joseph; Avilla, Mele; Yang, Bing; Svaren, John; Roopra, Avtar; Luo, Jian-Hua; Yu, Yan P.; Keles, Sunduz; Jarrard, David F. (5 June 2020). "CTCF loss mediates unique DNA hypermethylation landscapes in human cancers". Clinical Epigenetics. 12 (1): 80. doi:10.1186/s13148-020-00869-7. PMC 7275597. PMID 32503656.
- ^ Kemp, CJ; Moore, JM; Moser, R; Bernard, B; Teater, M; Smith, LE; Rabaia, NA; Gurley, KE; Guinney, J; Busch, SE; Shaknovich, R; Lobanenkov, VV; Liggitt, D; Shmulevich, I; Melnick, A; Filippova, GN (22 May 2014). "CTCF haploinsufficiency destabilizes DNA methylation and predisposes to cancer". Cell Reports. 7 (4): 1020–9. doi:10.1016/j.celrep.2014.04.004. PMC 4040130. PMID 24794443.
- ^ Wang H, Maurano MT, Qu H, Varley KE, Gertz J, Pauli F, Lee K, Canfield T, Weaver M, Sandstrom R, Thurman RE, Kaul R, Myers RM, Stamatoyannopoulos JA (September 2012). "Widespread plasticity in CTCF occupancy linked to DNA methylation". Genome Res. 22 (9): 1680–8. doi:10.1101/gr.136101.111. PMC 3431485. PMID 22955980.
- ^ Bao L, Zhou M, Cui Y (January 2008). "CTCFBSDB: a CTCF-binding site database for characterization of vertebrate genomic insulators". Nucleic Acids Res. 36 (Database issue): D83–7. doi:10.1093/nar/gkm875. PMC 2238977. PMID 17981843.
- ^ Xie X, Mikkelsen TS, Gnirke A, Lindblad-Toh K, Kellis M, Lander ES (2007). "Systematic discovery of regulatory motifs in conserved regions of the human genome, including thousands of CTCF insulator sites". Proc. Natl. Acad. Sci. U.S.A. 104 (17): 7145–50. Bibcode:2007PNAS..104.7145X. doi:10.1073/pnas.0701811104. PMC 1852749. PMID 17442748.
- ^ Fu Y, Sinha M, Peterson CL, Weng Z (2008). "The insulator binding protein CTCF positions 20 nucleosomes around its binding sites across the human genome". PLOS Genetics. 4 (7): e1000138. doi:10.1371/journal.pgen.1000138. PMC 2453330. PMID 18654629.
- ^ Teif VB, Vainshtein Y, Caudron-Herger M, Mallm JP, Marth C, Höfer T, Rippe K (2012). "Genome-wide nucleosome positioning during embryonic stem cell development". Nat Struct Mol Biol. 19 (11): 1185–92. doi:10.1038/nsmb.2419. PMID 23085715. S2CID 34509771.
- ^ Chernukhin IV, Shamsuddin S, Robinson AF, Carne AF, Paul A, El-Kady AI, Lobanenkov VV, Klenova EM (September 2000). "Physical and functional interaction between two pluripotent proteins, the Y-box DNA/RNA-binding factor, YB-1, and the multivalent zinc finger factor, CTCF". J. Biol. Chem. 275 (38): 29915–21. doi:10.1074/jbc.M001538200. PMID 10906122.
- ^ Kagey MH, Newman JJ, Bilodeau S, Zhan Y, Orlando DA, van Berkum NL, Ebmeier CC, Goossens J, Rahl PB, Levine SS, Taatjes DJ, Dekker J, Young RA (September 2010). "Mediator and cohesin connect gene expression and chromatin architecture". Nature. 467 (7314): 430–5. Bibcode:2010Natur.467..430K. doi:10.1038/nature09380. PMC 2953795. PMID 20720539.
- ^ Clarkson CT, Deeks EA, Samarista R, Mamayusupova H, Zhurkin VB, Teif VB (September 2019). "CTCF-dependent chromatin boundaries formed by asymmetric nucleosome arrays with decreased linker length". Nucleic Acids Research. 47 (21): 11181–11196. doi:10.1093/nar/gkz908. PMC 6868436. PMID 31665434.
추가 정보
- Ohlsson R, Renkawitz R, Lobanenkov V (2001). "CTCF is a uniquely versatile transcription regulator linked to epigenetics and disease". Trends Genet. 17 (9): 520–7. doi:10.1016/S0168-9525(01)02366-6. PMID 11525835.
- Klenova EM, Morse HC, Ohlsson R, Lobanenkov VV (2003). "The novel BORIS + CTCF gene family is uniquely involved in the epigenetics of normal biology and cancer". Semin. Cancer Biol. 12 (5): 399–414. doi:10.1016/S1044-579X(02)00060-3. PMID 12191639.
- Kuhn EJ, Geyer PK (2004). "Genomic insulators: connecting properties to mechanism". Curr. Opin. Cell Biol. 15 (3): 259–65. doi:10.1016/S0955-0674(03)00039-5. PMID 12787766.
- Recillas-Targa F, De La Rosa-Velázquez IA, Soto-Reyes E, Benítez-Bribiesca L (2007). "Epigenetic boundaries of tumour suppressor gene promoters: the CTCF connection and its role in carcinogenesis". J. Cell. Mol. Med. 10 (3): 554–68. doi:10.1111/j.1582-4934.2006.tb00420.x. PMC 3933142. PMID 16989720.
- Vostrov AA, Quitschke WW (1998). "The zinc finger protein CTCF binds to the APBbeta domain of the amyloid beta-protein precursor promoter. Evidence for a role in transcriptional activation". J. Biol. Chem. 272 (52): 33353–9. doi:10.1074/jbc.272.52.33353. PMID 9407128.
- Filippova GN, Lindblom A, Meincke LJ, Klenova EM, Neiman PE, Collins SJ, Doggett NA, Lobanenkov VV (1998). "A widely expressed transcription factor with multiple DNA sequence specificity, CTCF, is localized at chromosome segment 16q22.1 within one of the smallest regions of overlap for common deletions in breast and prostate cancers". Genes Chromosomes Cancer. 22 (1): 26–36. doi:10.1002/(SICI)1098-2264(199805)22:1<26::AID-GCC4>3.0.CO;2-9. PMID 9591631. S2CID 34221526.
- Bell AC, West AG, Felsenfeld G (1999). "The protein CTCF is required for the enhancer blocking activity of vertebrate insulators". Cell. 98 (3): 387–96. doi:10.1016/S0092-8674(00)81967-4. PMID 10458613. S2CID 18266832.
- Pérez-Juste G, García-Silva S, Aranda A (2000). "An element in the region responsible for premature termination of transcription mediates repression of c-myc gene expression by thyroid hormone in neuroblastoma cells". J. Biol. Chem. 275 (2): 1307–14. doi:10.1074/jbc.275.2.1307. PMID 10625678.
- Lutz M, Burke LJ, Barreto G, Goeman F, Greb H, Arnold R, Schultheiss H, Brehm A, Kouzarides T, Lobanenkov V, Renkawitz R (2000). "Transcriptional repression by the insulator protein CTCF involves histone deacetylases". Nucleic Acids Res. 28 (8): 1707–13. doi:10.1093/nar/28.8.1707. PMC 102824. PMID 10734189.
- Bell AC, Felsenfeld G (2000). "Methylation of a CTCF-dependent boundary controls imprinted expression of the Igf2 gene". Nature. 405 (6785): 482–5. Bibcode:2000Natur.405..482B. doi:10.1038/35013100. PMID 10839546. S2CID 4387329.
- Hark AT, Schoenherr CJ, Katz DJ, Ingram RS, Levorse JM, Tilghman SM (2000). "CTCF mediates methylation-sensitive enhancer-blocking activity at the H19/Igf2 locus". Nature. 405 (6785): 486–9. Bibcode:2000Natur.405..486H. doi:10.1038/35013106. PMID 10839547. S2CID 4421547.
- Chernukhin IV, Shamsuddin S, Robinson AF, Carne AF, Paul A, El-Kady AI, Lobanenkov VV, Klenova EM (2000). "Physical and functional interaction between two pluripotent proteins, the Y-box DNA/RNA-binding factor, YB-1, and the multivalent zinc finger factor, CTCF". J. Biol. Chem. 275 (38): 29915–21. doi:10.1074/jbc.M001538200. PMID 10906122.
- Chao W, Huynh KD, Spencer RJ, Davidow LS, Lee JT (2002). "CTCF, a candidate trans-acting factor for X-inactivation choice". Science. 295 (5553): 345–7. doi:10.1126/science.1065982. PMID 11743158. S2CID 27442721.
- Dintilhac A, Bernués J (2002). "HMGB1 interacts with many apparently unrelated proteins by recognizing short amino acid sequences" (PDF). J. Biol. Chem. 277 (9): 7021–8. doi:10.1074/jbc.M108417200. PMID 11748221. S2CID 39560486.
- Filippova GN, Qi CF, Ulmer JE, Moore JM, Ward MD, Hu YJ, Loukinov DI, Pugacheva EM, Klenova EM, Grundy PE, Feinberg AP, Cleton-Jansen AM, Moerland EW, Cornelisse CJ, Suzuki H, Komiya A, Lindblom A, Dorion-Bonnet F, Neiman PE, Morse HC, Collins SJ, Lobanenkov VV (2002). "Tumor-associated zinc finger mutations in the CTCF transcription factor selectively alter tts DNA-binding specificity". Cancer Res. 62 (1): 48–52. PMID 11782357.
- Kanduri M, Kanduri C, Mariano P, Vostrov AA, Quitschke W, Lobanenkov V, Ohlsson R (2002). "Multiple nucleosome positioning sites regulate the CTCF-mediated insulator function of the H19 imprinting control region". Mol. Cell. Biol. 22 (10): 3339–44. doi:10.1128/MCB.22.10.3339-3344.2002. PMC 133793. PMID 11971967.
- Farrell CM, West AG, Felsenfeld G (2002). "Conserved CTCF insulator elements flank the mouse and human beta-globin loci". Mol. Cell. Biol. 22 (11): 3820–31. doi:10.1128/MCB.22.11.3820-3831.2002. PMC 133827. PMID 11997516.