WRKY단백질영역
WRKY protein domain| 흔들리다 | |||||||||
|---|---|---|---|---|---|---|---|---|---|
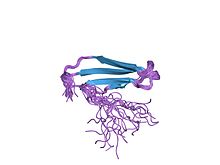 atwrky4의 c 말단 wrky 도메인의 용액 구조 | |||||||||
| 식별자 | |||||||||
| 기호. | 흔들리다 | ||||||||
| 팜 | PF03106 | ||||||||
| 빠맘 클랜 | CL0274 | ||||||||
| 인터프로 | IPR003657 | ||||||||
| |||||||||
WRKY 도메인은 전사 [1]인자의 클래스인 WRKY 전사 인자 패밀리에 있습니다.WRKY 도메인은 일부 디플로마나드, 사회적 아메바 및 다른 아메보조아, 곰팡이 인서테세디스 등에 WRKY 유전자가 존재하지만 거의 식물에서만 발견된다.그것들은 다른 비식물 종에는 없는 것으로 보인다.WRKY 전사 인자는 지난 20년간 [2]식물 연구의 중요한 영역이었다.WRKY DNA 결합 도메인은 W-box(T) TGAC(C/T)(및 이 배열의 변형) 시스 조절 요소를 인식합니다.
구조.
WRKY 전사인자는 하나 또는 두 개의 WRKY 단백질 도메인을 포함합니다.WRKY 단백질 도메인은 60~70개의 아미노산 길이 DNA 결합 도메인입니다.이 도메인은 보존성이 높은 코어 WRKYGQK 모티브와 아연 핑거 영역을 특징으로 합니다.시스테인과 히스티딘 아연 핑거 도메인은 CXCXHXH4-522-23 또는 CXCXHXC723 유형으로 발생하며, 여기서 X는 아미노산일 [3]수 있습니다.아연 손가락은 단백질 [4]기능에 필요한 Zn+2 이온과 결합합니다.WRKYGQK는 대부분의 WRKY 도메인에서 고도로 보존되어 있지만 코어 시퀀스의 변화는 [2][5]문서화되어 있습니다.코어 시퀀스의 자주 발생하는 변종은 WRKYGK이며, 이는 대부분의 식물 [2][3][5][6][7]종에 존재한다.
WRKY 단백질 도메인의 구조는 2005년에 핵자기공명(NMR)을 사용하여 처음 결정학으로 [4][8]결정되었다.WRKY 단백질 도메인은 5개의 반평행 β-스트랜드로 구성된 구상 형태이다.핵심 WRKYGQK 모티브는 두 번째 [8]β-스트랜드에서 찾을 수 있다.코어 모티브, 아연-핑거 결합 시스테인 및 히스티딘, DWK 소금 [8]브릿지를 형성하는 트라이어드를 포함한 18개의 아미노산이 WRKY 단백질 영역에 고보존되어 있다.코어 모티브의 보존된 트립토판(W)과 업스트림의 아스파르트산(D) 4개의 아미노산, 다운스트림의 리신(K) 29개의 아미노산으로 구성되어 도메인 [8]전체를 안정화한다.세 번째 β-스트랜드(PRSYY) 상의 5개의 아미노산도 WRKY [8]도메인에 잘 보존되어 있다.중요한 것은 WRKY 유전자는 PRSYY 아미노산 [3]배열의 PR을 코드하는 위치에서 발생하는 WRKY 도메인에 보존된 인트론을 포함하고 있으며, 따라서 이 모티프의 보존을 설명한다.
WRKY-DNA 상호작용
WRKY 도메인은 DNA [9]가닥의 주요 홈에 수직으로 들어가는 독특한 쐐기 모양의 구조를 형성합니다.WRKY 단백질 도메인은 W-box라고도 불리는 (T/A) TGAC([1][10][11]T/A) cis-element와 상호작용합니다.최근의 증거는 W-box의 GAC 코어가 WRKY 도메인의 주요 대상이며, 측면 배열은 매우 특정한 WRKY [12]단백질과의 DNA 상호작용을 지시하는 데 도움을 준다는 것을 암시한다.코어 모티브의 RKYGQK 잔기와 WRKY 도메인의 추가 아르기닌 및 리신 잔기는 GAC [9][12]코어를 포함한 7개의 연속 DNA 염기쌍의 인산염 골격과의 상호작용을 담당한다.WRKYGQK 모티브의 트립토판, 티로신 또는 리신 중 하나를 알라닌으로 변경하면 DNA [8][13]결합이 완전히 폐지되므로 이들 아미노산이 W박스 원소를 인식하는 데 필수적이라는 것을 알 수 있다.필수는 아니지만 WRKYGQK 모티브 아르기닌, 글리신 또는 글루타민을 알라닌으로 변경하면 W-박스에 [8][13]대한 DNA 결합을 감소시킨다.전반적으로, 이러한 복잡한 WRKY 단백질 도메인-DNA 상호작용은 식물 발달과 방어의 많은 측면에 필요한 유전자 활성화를 초래한다.
외부 링크
- 공장의 WRKY 패밀리TFDB: 플랜트 트랜스크립션 팩터 데이터베이스
- Arabidopsis 정보 자원의 WRKY 전사 인자 패밀리
- 러쉬턴 랩
- 솜시치 랩
- 심랩
- 솜시치의 WRKY 관련 출판물 목록
- 을검연구실
레퍼런스
- ^ a b Rushton PJ, Torres JT, Parniske M, Wernert P, Hahlbrock K, Somssich IE (October 1996). "Interaction of elicitor-induced DNA-binding proteins with elicitor response elements in the promoters of parsley PR1 genes". The EMBO Journal. 15 (20): 5690–700. doi:10.1002/j.1460-2075.1996.tb00953.x. PMC 452313. PMID 8896462.
- ^ a b c Schluttenhofer C, Yuan L (February 2015). "Regulation of specialized metabolism by WRKY transcription factors". Plant Physiology. 167 (2): 295–306. doi:10.1104/pp.114.251769. PMC 4326757. PMID 25501946.
- ^ a b c Eulgem T, Rushton PJ, Robatzek S, Somssich IE (May 2000). "The WRKY superfamily of plant transcription factors". Trends in Plant Science. 5 (5): 199–206. doi:10.1016/s1360-1385(00)01600-9. PMID 10785665.
- ^ a b Yamasaki K, Kigawa T, Inoue M, Tateno M, Yamasaki T, Yabuki T, et al. (March 2005). "Solution structure of an Arabidopsis WRKY DNA binding domain". The Plant Cell. 17 (3): 944–56. doi:10.1105/tpc.104.026435. PMC 1069710. PMID 15705956.
- ^ a b Zhang Y, Wang L (January 2005). "The WRKY transcription factor superfamily: its origin in eukaryotes and expansion in plants". BMC Evolutionary Biology. 5: 1. doi:10.1186/1471-2148-5-1. PMC 544883. PMID 15629062.
- ^ Song H, Wang P, Nan Z, Wang X (2014). "The WRKY Transcription Factor Genes in Lotus japonicus". International Journal of Genomics. 2014: 420128. doi:10.1155/2014/420128. PMC 3976811. PMID 24745006.
- ^ Xiong W, Xu X, Zhang L, Wu P, Chen Y, Li M, Jiang H, Wu G (July 2013). "Genome-wide analysis of the WRKY gene family in physic nut (Jatropha curcas L.)". Gene. 524 (2): 124–32. doi:10.1016/j.gene.2013.04.047. PMID 23644253.
- ^ a b c d e f g Duan MR, Nan J, Liang YH, Mao P, Lu L, Li L, Wei C, Lai L, Li Y, Su XD (2007). "DNA binding mechanism revealed by high resolution crystal structure of Arabidopsis thaliana WRKY1 protein". Nucleic Acids Research. 35 (4): 1145–54. doi:10.1093/nar/gkm001. PMC 1851648. PMID 17264121.
- ^ a b Yamasaki K, Kigawa T, Watanabe S, Inoue M, Yamasaki T, Seki M, Shinozaki K, Yokoyama S (March 2012). "Structural basis for sequence-specific DNA recognition by an Arabidopsis WRKY transcription factor". The Journal of Biological Chemistry. 287 (10): 7683–91. doi:10.1074/jbc.M111.279844. PMC 3293589. PMID 22219184.
- ^ Eulgem T, Rushton PJ, Schmelzer E, Hahlbrock K, Somssich IE (September 1999). "Early nuclear events in plant defence signalling: rapid gene activation by WRKY transcription factors". The EMBO Journal. 18 (17): 4689–99. doi:10.1093/emboj/18.17.4689. PMC 1171542. PMID 10469648.
- ^ de Pater S, Greco V, Pham K, Memelink J, Kijne J (December 1996). "Characterization of a zinc-dependent transcriptional activator from Arabidopsis". Nucleic Acids Research. 24 (23): 4624–31. doi:10.1093/nar/24.23.4624. PMC 146317. PMID 8972846.
- ^ a b Brand LH, Fischer NM, Harter K, Kohlbacher O, Wanke D (November 2013). "Elucidating the evolutionary conserved DNA-binding specificities of WRKY transcription factors by molecular dynamics and in vitro binding assays". Nucleic Acids Research. 41 (21): 9764–78. doi:10.1093/nar/gkt732. PMC 3834811. PMID 23975197.
- ^ a b Maeo K, Hayashi S, Kojima-Suzuki H, Morikami A, Nakamura K (November 2001). "Role of conserved residues of the WRKY domain in the DNA-binding of tobacco WRKY family proteins". Bioscience, Biotechnology, and Biochemistry. 65 (11): 2428–36. doi:10.1271/bbb.65.2428. PMID 11791715. S2CID 22671192.


