단백질 폴딩
Protein folding단백질 접힘은 단백질 사슬이 본래의 3차원 구조로 변환되는 물리적 과정으로, 전형적으로 단백질이 생물학적으로 기능하게 되는 "접힌" 형태이다.폴리펩타이드는 신속하고 재현 가능한 프로세스를 통해 랜덤 [1]코일로부터 특징적인 3차원 구조로 접힙니다.각 단백질은 먼저 일련의 mRNA에서 아미노산의 선형사슬에 번역된 후 전개 폴리펩타이드 또는 랜덤코일로 존재한다.이 단계에서 폴리펩타이드는 안정적인(장시간 지속되는) 3차원 구조(첫 번째 그림의 왼쪽)가 없다.폴리펩타이드 사슬이 리보솜에 의해 합성됨에 따라 선형 사슬이 3차원 구조로 접히기 시작한다.
많은 단백질의 접힘은 심지어 폴리펩타이드 사슬의 번역 동안에도 시작된다.아미노산은 서로 상호작용하여 잘 정의된 3차원 구조인 접힌 단백질(그림의 오른쪽)을 생성하며, 이는 자연 상태로 알려져 있다.결과적인 3차원 구조는 아미노산 배열 또는 1차 구조(Anfinsen의 도그마)[2]에 의해 결정된다.
비록 기능성 단백질의 일부 부분이 [3]펼쳐져 있을 수 있지만, 올바른 3차원 구조는 기능하기 위해 필수적이다. 그래서 단백질 역학은 중요하다.자연 구조로 접히지 않으면 일반적으로 비활성 단백질이 생성되지만, 잘못 접힌 단백질이 변형되거나 독성 기능을 가진 경우도 있다.몇몇 신경 변성 및 다른 질병들은 잘못 접힌 단백질에 의해 형성되는 아밀로이드 섬유소의 축적에서 기인하는 것으로 믿어지는데, 이 아밀로이드 섬유소의 감염 종류는 [4]프리온으로 알려져 있다.면역체계가 특정 단백질 [5]구조에 대한 항체를 생성하지 않기 때문에 많은 알레르기가 일부 단백질의 잘못된 접힘에 의해 발생합니다.
단백질의 변성은 접힌 상태에서 펼쳐진 상태로의 전환 과정이다.그것은 요리, 화상, 단백질 병, 그리고 다른 맥락에서 일어난다.
접히는 과정은 관심 단백질에 따라 크게 달라집니다.세포 밖에서 연구될 때, 가장 느린 접힘 단백질은 주로 프롤린 이성질화로 인해 접히는 데 몇 분 또는 몇 시간이 걸리고, 과정이 [6]완료되기 전에 체크포인트와 같은 여러 중간 상태를 통과해야 합니다.반면에, 최대 100개의 아미노산 길이를 가진 매우 작은 단일 도메인 단백질은 일반적으로 한 [7]번에 접힙니다.밀리초의 시간 척도가 표준이고 가장 빠른 것으로 알려진 단백질 접힘 반응은 몇 마이크로초 [8]안에 완료됩니다.단백질의 접는 시간 척도는 단백질의 크기, 접촉 순서 및 회로 [9]위상에 따라 달라집니다.
단백질 접힘 과정을 이해하고 시뮬레이션하는 것은 1960년대 후반부터 컴퓨터 생물학에서 중요한 과제가 되었다.
단백질 접힘 과정
일차 구조
단백질의 1차 구조인 선형 아미노산 배열이 단백질의 고유 [10]형태를 결정합니다.특정 아미노산 잔류물과 폴리펩타이드 사슬에서 이들의 위치는 단백질의 일부가 서로 밀접하게 접혀져 3차원 구조를 형성하는 결정 요인이다.아미노산 조성은 [11]배열만큼 중요하지 않다.그러나 접힘의 본질적인 사실은 각 단백질의 아미노산 배열이 고유 구조와 그 상태를 달성하기 위한 경로를 지정하는 정보를 포함하고 있다는 것이다.이것은 거의 동일한 아미노산 배열이 항상 비슷하게 [12]접힌다는 것을 말하는 것은 아니다.배치는 환경 요인에 따라 다르다; 유사한 단백질은 그들이 발견되는 위치에 따라 다르게 접힌다.
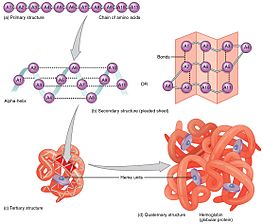
이차 구조
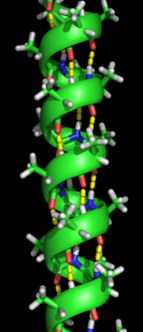

2차 구조의 형성은 단백질이 본래의 구조를 가정하기 위해 취하는 접힘 과정의 첫 번째 단계이다.2차 구조의 특징은 알파 나선 및 베타 시트로 알려진 구조인데, 이는 라이너스 폴링에 의해 처음 특징지어졌던 것처럼 분자 내 수소 결합에 의해 안정화되기 때문이다.분자 내 수소 결합의 형성은 단백질 [13]안정성에 또 다른 중요한 기여를 제공한다.α-염색체는 골격의 수소 결합에 의해 형성되어 나선형으로 형성된다(오른쪽 [11]그림 참조).β 주름 시트는 골격이 스스로 구부러져 수소 결합을 형성하는 구조이다(왼쪽 그림 참조).수소 결합은 펩타이드 결합의 아미드 수소와 카르보닐 산소 사이에 있습니다.평행 [11]시트에 의해 형성된 비스듬한 수소 결합에 비해 수소 결합의 안정성이 이상적인 180도 각도로 결합하기 때문에 반평행 β 시트에서 더 강한 반평행 β 주름 시트와 평행 β 주름 시트가 존재한다.
3차 구조
α-헬리체 및 β-시트는 일반적으로 양친매성이며, 친수성과 소수성 부분을 가지고 있다.이 능력은 친수성 면이 단백질을 둘러싼 수성 환경을 향하고 소수성 면이 [14]단백질의 소수성 코어를 향하도록 접히는 단백질의 3차 구조를 형성하는데 도움을 준다.2차 구조는 계층적으로 3차 구조 형성에 자리를 내준다.일단 단백질의 3차 구조가 소수성 상호작용에 의해 형성되고 안정화되면, 두 개의 시스테인 잔류물 사이에 형성된 디술피드 브리지 형태의 공유 결합이 있을 수도 있다.이러한 비공유 및 공유 결합 접촉은 단백질의 고유 구조에서 특정한 위상 배열을 취한다.단백질의 3차 구조는 단일 폴리펩타이드 사슬을 포함하지만 접힌 폴리펩타이드 사슬의 추가 상호작용은 4차 구조를 [15]형성한다.
4차 구조
3차 구조는 일부 단백질에서 4차 구조의 형성에 자리를 내줄 수 있으며, 이는 보통 이미 접힌 서브유닛의 "조립" 또는 "조립"을 포함한다. 즉, 여러 폴리펩타이드 사슬이 상호작용하여 완전한 기능 4차 [11]단백질을 형성할 수 있다.
단백질 접힘의 추진력
접힘은 주로 소수성 상호작용, 분자 내 수소 결합의 형성, 판 데르 발스 힘에 의해 유도되는 자발적 과정이며, 그것은 구조 [16]엔트로피에 의해 반대된다.접히는 과정은 종종 공번역적으로 시작되어 단백질의 N 말단이 접히기 시작하고 단백질의 C 말단 부분은 리보솜에 의해 아직 합성되고 있다. 그러나 단백질 분자는 생합성 [17]중에 또는 생합성 후에 자연스럽게 접힐 수 있다.이러한 고분자는 "접힘"으로 간주될 수 있지만, 이 과정은 또한 용매(물 또는 지질 이중층),[18] 염분 농도, pH, 온도, 보조인자 및 분자 샤페론의 존재 가능성에 따라 달라집니다.
단백질은 제한된 굽힘 각도 또는 가능한 형태에 의해 접히는 능력에 한계가 있을 것이다.단백질 접힘의 이러한 허용 각도는 Ramachandran 플롯으로 알려진 2차원 플롯으로 설명되며, 허용 [19]회전의 psi 및 phi 각도로 표현된다.
소수성 효과

단백질 접힘이 자발적 반응이 되기 위해서는 세포 내에서 열역학적으로 유리해야 한다.단백질 접힘은 자발적인 반응으로 알려져 있기 때문에, 그것은 반드시 음의 깁스 자유 에너지 값을 가정해야 한다.단백질 접힘에서의 깁스 자유 에너지는 엔탈피와 [11]엔트로피와 직접적으로 관련이 있다.음의 델타 G가 발생하고 단백질 접힘이 열역학적으로 유리해지려면 엔탈피, 엔트로피 또는 두 항 중 하나가 유리해야 한다.
물에 노출되는 소수성 사이드 체인의 수를 최소화하는 것이 접힘 과정의 중요한 [20]원동력입니다.소수성 효과는 단백질의 소수성 사슬이 (친수성 환경에서)[11] 단백질의 핵심으로 붕괴되는 현상이다.수성 환경에서, 물 분자는 단백질의 소수성 영역이나 곁사슬 주위에 모여 질서 있는 물 [21]분자의 물껍질을 만드는 경향이 있습니다.소수성 영역 주변의 물 분자의 순서는 시스템 내 질서를 증가시키고 따라서 엔트로피의 음의 변화(시스템 내 엔트로피 감소)에 기여합니다.물 분자는 소수성 붕괴나 소수성 그룹의 안쪽으로 접히는 물 우리에 고정된다.소수성 붕괴는 질서 있는 물 [11]분자를 해방시키는 물 우리들의 파괴를 통해 엔트로피를 시스템에 다시 유입시킵니다.구상 접힌 단백질의 코어 내에서 상호작용하는 다수의 소수성 그룹은 엄청나게 축적된 반데르발스 힘(특히 런던 분산력)[11] 때문에 접힌 후 단백질 안정성에 상당한 양을 기여한다.소수성 효과는 열역학에서 [22]큰 소수성 영역을 포함하는 양친매 분자와 함께 수성 매체가 존재하는 경우에만 동력으로 존재합니다.수소 결합의 강도는 환경에 따라 달라지며, 따라서 소수성 코어에 둘러싸인 H 결합은 수성 환경에 노출된 H 결합보다 더 많은 자연 [23]상태의 안정성에 기여한다.
구상주름이 있는 단백질에서 소수성 아미노산은 무작위로 분포되거나 함께 [24][25]뭉쳐지는 대신 1차 염기서열을 따라 삽입되는 경향이 있다.그러나 본질적으로 [26][27]무질서한 경향이 있는 데노보에서 최근에 태어난 단백질은 1차 [28]염기서열을 따라 소수성 아미노산 군집의 반대 패턴을 보인다.
샤페로네스

분자 샤페론은 생체 내에서 다른 단백질의 올바른 접힘을 돕는 단백질의 한 종류이다.샤페론은 모든 세포 구획에 존재하며 폴리펩타이드 사슬과 상호작용하여 단백질의 자연스러운 3차원 배열을 형성한다. 그러나 샤페론 자체는 그들이 [29]보조하는 단백질의 최종 구조에 포함되지 않는다.샤페론은 초기 폴리펩타이드가 리보솜에 [30]의해 합성될 때에도 접히는 것을 도울 수 있다.분자 샤페론은 접히는 경로에서 단백질의 불안정한 구조를 안정시키기 위해 결합함으로써 작동하지만, 샤페론은 그들이 돕고 있는 단백질의 정확한 고유 구조를 알기 위해 필요한 정보를 포함하지 않는다; 대신, 잘못된 접히는 [30]구조를 방지함으로써 작동한다.이러한 방법으로, 샤페론은 실제로 자연 구조를 향한 접힘 경로에 포함된 개별 단계의 속도를 증가시키지 않는다; 대신에, 그들은 적절한 중간체를 찾는 것을 늦출 수 있는 폴리펩타이드 사슬의 가능한 원치 않는 집단을 감소시킴으로써 작용하고 그들은 폴을 위한 더 효율적인 경로를 제공한다.올바른 [29]구성을 가정하기 위해 펩타이드 체인.샤페론은 접히는 촉매 단백질과 혼동해서는 안 되며, 접히는 경로의 느린 단계를 담당하는 화학 반응을 촉매합니다.접이식 촉매의 예로는 단백질 이황화물 이성질화효소 및 펩티딜-프롤릴 이성질화효소가 있으며, 이들은 이황화물 결합의 형성 또는 펩티드 그룹의 [30]시스와 트랜스 입체 이성질 간의 상호 변환에 관여할 수 있다.샤페론은 단백질이 생체 내에서 접히는 과정에서 매우 중요한 것으로 보여진다. 왜냐하면 샤페론은 "생물학적 [31]관련성"이 있을 만큼 적절한 배열과 구조를 효율적으로 가정하는데 필요한 도움을 단백질을 제공하기 때문이다.이것은 [31]시험관 내에서 수행된 단백질 접힘 실험에서 증명되었듯이 폴리펩타이드 사슬이 이론적으로 샤페론의 도움 없이 원래의 구조로 접힐 수 있다는 것을 의미한다. 그러나, 이 과정은 생물학적 시스템에 존재하기에는 너무 비효율적이거나 너무 느리다는 것이 입증되었다. 따라서, 샤페론은 생체 내에서 단백질 접힘에 필요하다.자연 구조 형성을 돕는 역할과 함께, 샤페론은 단백질 수송, 분해와 같은 다양한 역할에 관여하는 것으로 보여지며, 심지어 특정 외부 변성 인자에 노출되는 변성 단백질을 그들의 올바른 자연 [32]구조에 다시 결합할 수 있는 기회를 허용한다.
완전히 변성된 단백질은 3차 및 2차 구조가 모두 결여되어 있으며, 이른바 랜덤 코일로 존재한다.특정 조건 하에서 일부 단백질은 재결합할 수 있지만, 많은 경우 변성은 되돌릴 [33]수 없다.세포는 때때로 열 쇼크 단백질로 알려진 효소로 열의 변성 영향으로부터 그들의 단백질을 보호하는데, 이것은 접히는 것과 접힌 채로 있는 다른 단백질을 돕는다.열충격 단백질은 박테리아에서 인간에 이르기까지 검사된 모든 종에서 발견되었으며, 이는 그들이 매우 일찍 진화했고 중요한 기능을 가지고 있다는 것을 암시한다.일부 단백질은 세포 내에서 절대 접히지 않으며, 다른 단백질과의 상호작용에 의해 각각의 단백질을 분리하거나 잘못 접힌 단백질을 펼치도록 돕는 샤페론의 도움을 제외하고는 세포 내에서 [34]접히지 않는다.이 기능은 불용성 비정질 골재로의 침전 위험을 방지하기 위해 중요하다.단백질 변성 또는 자연 상태의 파괴와 관련된 외부 요인에는 온도, 외부장(전기, 자기),[35] 분자 밀집,[36] 그리고 단백질의 [37]접힘에 큰 영향을 미칠 수 있는 공간의 한계(예: 제한)가 포함됩니다.고농도의 용질, 극도의 pH, 기계적 힘 및 화학적 변성제의 존재도 단백질 변성의 원인이 될 수 있습니다.이러한 개별 요인은 스트레스로 분류됩니다.샤페론은 세포 스트레스 시간 동안 농도가 증가하는 것으로 나타나며, 변성되거나 잘못 접힌 [29]단백질뿐만 아니라 새로운 단백질의 적절한 접힘을 돕는다.
어떤 조건에서는 단백질이 생화학적으로 기능하는 형태로 접히지 않을 것이다.세포가 살기 쉬운 범위를 넘거나 밑도는 온도는 열적으로 불안정한 단백질을 펼치거나 변성시킨다.그러나 단백질의 열 안정성은 일정하지 않다. 예를 들어, 122°[38]C까지 높은 온도에서 자라는 온열성 박테리아가 발견되었으며, 이는 물론 필수 단백질과 단백질 어셈블리의 완전한 보체가 해당 온도 이상에서 안정되어야 한다.
대장균은 박테리오파지 T4의 숙주이며, 파지 부호화 gp31 단백질(P17313)은 구조 및 기능적으로 대장균 샤페론 단백질 GroES와 상동성이 있으며,[39] 감염 중 박테리오파지 T4 바이러스 입자의 집합에서 이를 대체할 수 있는 것으로 보인다.gp31은 GroES와 마찬가지로 박테리오파지 T4 주요 캡시드 단백질 gp23의 [39]생체 내 접힘 및 조립에 절대적으로 필요한 GroEL 샤페로닌과 안정된 복합체를 형성한다.
폴드 스위칭
일부 단백질은 여러 개의 고유 구조를 가지고 있으며, 일부 외부 요인에 따라 접힘을 변화시킨다.예를 들어, KaiB 단백질 스위치는 하루 종일 접히면서 시아노박테리아를 위한 시계 역할을 합니다.PDB(Protein Data Bank) 단백질의 약 0.5~4%가 [40]접히는 것으로 추정되었다.
단백질 오접힘 및 신경변성 질환
단백질은 정상적인 자연 상태에 도달할 수 없는 경우 잘못 접힌 것으로 간주됩니다.이는 아미노산 배열의 돌연변이 또는 외부 [41]요인에 의한 정상적인 접힘 과정의 방해 때문일 수 있습니다.잘못 접힌 단백질은 일반적으로 교차 β 구조로 알려진 초분자 배열로 구성된 β-시트를 포함한다.이러한 β-시트가 풍부한 어셈블리는 매우 안정적이고, 매우 불용성이며, 일반적으로 단백질 [42]분해에 내성이 있습니다.이러한 섬유질 어셈블리의 구조적 안정성은 단백질 단량체 간의 광범위한 상호작용에 의해 발생하며, β-스트랜드 사이의 [42]골격 수소 결합에 의해 형성된다.단백질의 잘못된 접힘은 다른 단백질의 응집체 또는 올리고머로의 추가적인 잘못된 접힘과 축적을 촉발할 수 있다.세포에서 응집된 단백질의 증가된 수치는 퇴행성 장애와 세포 [41]사멸을 야기할 수 있는 아밀로이드와 같은 구조의 형성을 이끈다.아밀로이드는 고도로 불용성이고 변환된 단백질 [41]응집체로 만들어진 분자간 수소 결합을 포함하는 섬유질 구조입니다.따라서 프로테아솜 경로는 응집 전에 잘못 접힌 단백질을 분해하기에 충분히 효율적이지 않을 수 있다.잘못 접힌 단백질은 서로 상호작용하고 구조화된 집계를 형성하며 분자간 [41]상호작용을 통해 독성을 얻을 수 있다.
집합 단백질 Creutzfeldt–Jakob 병, 광우병(광우병), 알츠하이머 병 및 가정 아밀로이드 심근 경색증 또는 polyneuropathy,[43]뿐만 아니라 헌팅턴과 파킨슨 병의 경멸하다 같은 세포 내 통합 질환과 같이amyloid-related 질병 같은prion-related 질환과 연관되어 있다.완화이러한 노화 발생 퇴행성 질환은 교차 베타 아밀로이드 섬유를 포함한 불용성 세포외 골재 및/또는 세포내 포접물로 잘못 접힌 단백질의 응집과 관련이 있다.[4][44]골재가 단백질 항상성 상실, 합성, 접힘, 응집 및 단백질 교체의 균형에 대한 원인인지 단순한 반영인지는 완전히 명확하지 않다.최근 유럽의약청은 트란스테레틴 아밀로이드 질환의 치료를 위해 타파미디스 또는 빈다켈(사중합체 트란스테레틴의 운동안정제)의 사용을 승인했다.이것은 아밀로이드 섬유화 과정이 (섬유 자체가 아닌)[45] 인간 아밀로이드 질환에서 유사분열 후 조직의 변성을 일으킨다는 것을 암시한다.접힘과 기능 대신 접힘과 과도한 열화는 기능 상실이 원인인 항트립신 관련 폐기종, 낭포성 섬유증 및 리소좀 저장 질환과 같은 많은 단백질 질환으로 이어진다.단백질 대체 요법은 역사적으로 후자의 장애를 고치기 위해 사용되어 왔지만, 새로운 접근법은 약물 샤페론을 사용하여 돌연변이 단백질을 접어서 기능하게 만드는 것입니다.
단백질 접힘 연구를 위한 실험 기술
단백질 접힘에 대한 추론은 돌연변이 연구를 통해 만들어질 수 있지만, 전형적으로 단백질 접힘을 연구하기 위한 실험 기술은 단백질의 점진적인 전개 또는 접힘에 의존하며 표준 비결정학적 기술을 사용하여 구조 변화를 관찰한다.
X선 결정학

X선 결정학은 접힌 [46]단백질의 3차원 구조를 해독하기 위한 보다 효율적이고 중요한 방법 중 하나입니다.X선 결정학을 수행하기 위해서는 조사 대상 단백질이 결정 격자 안에 위치해야 합니다.결정격자 안에 단백질을 넣으려면 결정화에 적합한 용매가 있어야 하며 용액에서 과포화 수준의 순수 단백질을 얻고 용액에서 [47]결정을 침전시켜야 한다.단백질이 결정화되면 X선 빔이 결정 격자를 통해 집중되어 빔을 회절시키거나 여러 방향으로 바깥쪽으로 쏠 수 있습니다.이러한 외부 빔은 내부에 포함된 단백질의 특정 3차원 구성과 관련이 있습니다.X선은 특히 단백질 결정 격자 내의 개별 원자를 둘러싼 전자 구름과 상호작용하여 식별 가능한 회절 [14]패턴을 생성합니다.전자 밀도 구름과 X선의 진폭을 관련지어야만 이 패턴을 읽을 수 있고 이 [48]방법을 복잡하게 만드는 위상 또는 위상각의 가정으로 이어질 수 있다.푸리에 변환이라고 알려진 수학적 기초를 통해 확립된 관계가 없다면, "위상 문제"는 회절 패턴을 예측하는 것을 매우 [14]어렵게 만들 것입니다.다중 동형 치환과 같은 새로운 방법은 중금속 이온의 존재를 사용하여 X선을 보다 예측 가능한 방식으로 회절하여 관련된 변수의 수를 줄이고 위상 [46]문제를 해결합니다.
형광 분광법
형광 분광법은 단백질의 접힘 상태를 연구하기 위한 매우 민감한 방법이다.페닐알라닌(Phe), 티로신(Tyr), 트립토판(Trp) 등 세 가지 아미노산은 고유 형광 특성을 가지고 있지만, 이들의 양자 수율이 좋은 형광 신호를 줄 수 있을 만큼 높기 때문에 실험적으로 사용되는 것은 Tyr과 Trp뿐이다.Trp와 Tyr는 모두 280nm의 파장으로 들뜬 반면 Trp만 295nm의 파장으로 들뜬다.방향족 특성 때문에 Trp 및 Tyr 잔기는 종종 단백질의 소수성 코어, 두 단백질 도메인 사이의 인터페이스 또는 올리고머 단백질의 서브유닛 사이의 인터페이스에 완전히 또는 부분적으로 묻혀 있는 것으로 발견됩니다.이러한 단극 환경에서, 그것들은 높은 양자 수율을 가지고 있고, 따라서 높은 형광 강도를 가지고 있다.단백질의 3차 또는 4차 구조가 파괴되면, 이러한 곁사슬은 용제의 친수성 환경에 더 많이 노출되고, 그들의 양자 수율이 감소하여 낮은 형광 강도로 이어진다.Trp 잔기의 경우 최대 형광 방출 파장도 환경에 따라 달라진다.
형광분광법은 변성치의 [49][50]함수로서 형광방출 강도 또는 최대방출 파장의 변화를 측정함으로써 단백질의 평형전개를 특징짓는 데 사용할 수 있다.변성제는 화학 분자(요소, 염산구아니디늄), 온도, pH, 압력 등이 될 수 있습니다.서로 다르지만 분리된 단백질 상태 사이의 균형, 즉 고유 상태, 중간 상태, 전개된 상태는 변성 값에 따라 달라진다. 따라서 평형 혼합물의 전역 형광 신호도 이 값에 따라 달라진다.이것에 의해, 글로벌 단백질 신호를 변성치에 관련짓는 프로파일을 얻을 수 있다.평형 전개 프로파일을 통해 [51][52]전개 중간을 검출하고 식별할 수 있습니다.일반 방정식은 호모머 또는 헤테로머 단백질의 전개 평형을 특징짓는 열역학적 매개변수를 그러한 [49]프로파일로부터 삼량체 및 잠재적으로 4량체까지 얻기 위해 Hugues Bedoule에 의해 개발되었습니다.형광분광법은 정지된 흐름과 같은 고속혼합장치와 조합하여 단백질 [53]접힘역동학을 측정하고 쉐브론 플롯을 생성하여 Phi 값 분석을 도출할 수 있다.
순환 이분법
원형 이분법은 단백질 접힘을 연구하는 가장 일반적이고 기본적인 도구 중 하나이다.원형 이색성 분광법은 원형 편광의 흡수를 측정한다.단백질에서 알파 헬리클이나 베타 시트 등의 구조는 키랄이며, 따라서 그러한 빛을 흡수한다.이 빛의 흡수는 단백질 합성의 접힘 정도를 나타내는 지표 역할을 합니다.이 기술은 변성제 농도 또는 온도의 함수로서 이 흡수량의 변화를 측정하여 단백질의 평형 전개를 측정하는 데 사용되어 왔다.변성 용융은 전개 시의 자유 에너지와 단백질의 m 값, 즉 변성 의존성을 측정합니다.온도 용해는 단백질의 [49]변성 온도(Tm)를 측정한다.형광분광법에 대해서는 원형디크로이즘분광법을 정지흐름 등의 고속혼합장치에 조합하여 단백질 접힘역동학을 측정하여 쉐브론 플롯을 생성할 수 있다.
단백질의 진동 원형 이색성
현재 푸리에 변환(FT) 기기와 관련된 단백질에 대한 진동 원형 이분법(VCD) 기술의 보다 최근의 발전은 매우 큰 단백질 분자에 대해서도 용액에서 단백질 구조를 결정하는 강력한 수단을 제공한다.단백질에 대한 이러한 VCD 연구는 단백질 결정에 대한 X선 회절 데이터, 중수(DO)에2 있는 단백질 용액에 대한 FT-IR 데이터 또는 양자 계산과 결합될 수 있습니다.
단백질핵자기공명분광법
단백질핵자기공명(NMR)은 농축 단백질 시료를 통해 자석장을 유도함으로써 단백질 구조 데이터를 수집할 수 있다.NMR에서는 화학적 환경에 따라 특정 핵이 특정 무선 [54][55]주파수를 흡수합니다.단백질 구조 변화는 ns에서 ms까지의 시간 척도로 작동하기 때문에, NMR은 ps에서 [56]s까지의 시간 척도로 중간 구조를 연구하기 위해 특별히 갖춰진다.단백질 구조와 비접힘 단백질 구조 변화를 연구하기 위한 주요 기술로는 COSY, TOCSY, HSQC, 시간 완화(T1 및 T2)가 있으며, NOE는 [54]공간적으로 근접한 수소 간에 자화 전달이 [54]관찰될 수 있기 때문에 특히 유용하다.다양한 NMR 실험은 다양한 단백질 구조 변화에 적합한 다양한 수준의 시간 척도 민감도를 가진다.NOE는 결합 진동이나 측쇄 회전을 감지할 수 있지만, NOE는 너무 민감해서 단백질 접힘을 감지할 수 없습니다. 왜냐하면 더 큰 [56]시간대에 발생하기 때문입니다.
단백질 접힘은 약 50~3000초−1 동안 이루어지기 때문에 CPMG 완화 분산과 화학 교환 포화 전달은 [55]접힘의 NMR 분석의 주요 기술 중 일부가 되었다.또한 두 기술은 단백질 접힘 환경에서 [57]들뜬 중간 상태를 발견하기 위해 사용됩니다.이를 위해 CPMG 완화 분산은 스핀 에코 현상을 활용합니다.이 기술은 대상 핵을 90펄스에 노출시킨 후 하나 이상의 [58]180펄스에 노출시킵니다.원자핵의 재초점으로서 넓은 분포는 대상 원자핵이 중간 들뜸 상태에 있음을 나타낸다.완화 분산은 들뜬 상태와 [58][57]지면 사이의 열역학 및 동력학에 대한 데이터 수집 정보를 표시합니다.포화 전송은 들뜬 상태가 교란됨에 따라 접지 상태에서 신호의 변화를 측정합니다.그것은 약한 무선 주파수 조사를 사용하여 특정 원자핵의 들뜬 상태를 포화시켜 포화를 지면 상태로 [55]전환한다.이 신호는 접지 상태의 [55][57]자화(및 신호)를 감소시킴으로써 증폭됩니다.
NMR의 주요 한계는 분해능이 25kDa보다 크고 X선 [55]결정학만큼 상세하지 않은 단백질에 따라 감소한다는 것이다.또한 단백질 NMR 분석은 상당히 어려우며 동일한 NMR [54]스펙트럼에서 여러 솔루션을 제안할 수 있다.
단백질 SOD1과 관련된 근위축성 측삭경화증의 접힘에 초점을 맞춘 연구에서 들뜬 중간체들은 완화 분산과 포화 [59]전달을 통해 연구되었다.SOD1은 이전에 단백질 응집과 관련된 것으로 추정되는 돌연변이를 일으키는 많은 질병과 연관되어 있었지만, 그 메커니즘은 여전히 알려지지 않았다.완화 분산 및 포화 전달 실험을 사용하여 SOD1 [59]돌연변이에서 많은 들뜬 중간 상태가 잘못 접히는 것을 발견했습니다.
이중 편파 간섭계
이중 편파 간섭계는 분자층의 광학 특성을 측정하기 위한 표면 기반 기술이다.단백질 접힘의 특성화를 위해 사용될 때, 단백질 접힘의 역학적 실시간 측정은 ~10Hz보다 느리게 발생하는 과정으로 제한되지만, 단백질의 단분자층의 전체 크기와 그 밀도를 서브 앵스트롬 [60]분해능에서 실시간으로 결정함으로써 배열을 측정한다.원형 이색성과 유사하게 접힘에 대한 자극은 변성제 또는 온도일 수 있습니다.
높은 시간 분해능으로 접는 방법에 관한 연구
단백질 접힘에 대한 연구는 최근 몇 년 동안 빠르고 시간 분해된 기술의 개발로 크게 발전해 왔다.실험자들은 전개된 단백질 샘플의 접힘을 빠르게 트리거하고 그 결과 발생하는 역학을 관찰합니다.사용되는 빠른 기술에는 중성자 산란,[61] 용액의 초고속 혼합, 광화학 방법 및 레이저 온도 점프 분광법이 포함됩니다.이러한 기술의 개발에 기여한 많은 과학자 중에는 제레미 쿡, 하인리히 로더, 해리 그레이, 마틴 그루벨, 브라이언 다이어, 윌리엄 이튼, 쉬나 래드포드, 크리스 돕슨, 앨런 퍼쉬, 벵트 욜팅, 라르스 코네르만 등이 있다.
단백질 분해
단백질 분해는 다양한 용액 조건(예: 고속 병렬 단백질 분해(FASTPP))[62][63]에서 펼쳐지는 분율을 탐색하는 데 일상적으로 사용됩니다.
단분자력분광법
광학 핀셋과 AFM과 같은 단일 분자 기술은 샤페론을 [64]가진 단백질뿐만 아니라 분리된 단백질의 단백질 접힘 메커니즘을 이해하기 위해 사용되어 왔다.광학 핀셋은 C- 및 N-termini에서 단일 단백질 분자를 늘린 후 전개하여 후속 [65]리폴딩을 연구하기 위해 사용되었다.예를 들어, 광학 핀셋은 혈액 응고에 관련된 단백질의 접힘과 전개를 연구하기 위해 최근 적용되었습니다.von Willebrand 인자(vWF)는 혈전 형성 과정에서 필수적인 역할을 하는 단백질이다.단일 분자 광학 핀셋 측정을 사용하여 칼슘 결합 vWF가 혈액에서 전단력 센서 역할을 한다는 것을 발견했습니다.전단력은 vWF의 A2 영역을 펼치도록 유도하며,[66] 칼슘이 있을 때 리폴딩 속도가 극적으로 향상된다.최근에는 단순한 src SH3 도메인이 [67]여러 전개 경로에 강제로 액세스하는 것으로 나타났다.
비오틴 그림
비오틴 페인팅은 접힌 단백질의 상태별 세포 스냅샷을 가능하게 한다.비오틴 '페인팅'은 예측된 선천적으로 무질서한 [68]단백질에 대한 편견을 보여준다.
단백질 접힘 계산 연구
단백질 접힘의 계산 연구는 단백질 안정성, 속도론 및 구조의 예측과 관련된 세 가지 주요 측면을 포함한다.2013년 검토는 단백질 접힘에 사용할 수 있는 계산 방법을 요약한다.[69]
레빈탈의 역설
1969년, 사이러스 레빈탈은 펼쳐진 폴리펩타이드 사슬의 자유도가 매우 크기 때문에, 분자는 천문학적으로 많은 가능한 형태를 가지고 있다고 언급했다.그의 논문 [70]중 한 편에서 3에서143 10으로300 추정되었다.레빈탈의 역설은 만약 단백질이 가능한 모든 배열을 순차적으로 샘플링하여 접힌다면, 비록 그 배열을 [71]빠른 속도로 샘플링하더라도, 그렇게 하는데 천문학적인 시간이 걸릴 것이라는 관찰에 기초한 사고 실험이다.레빈탈은 단백질이 이것보다 훨씬 더 빨리 접힌다는 관찰에 기초하여 무작위 배좌 탐색은 일어나지 않으며, 따라서 단백질은 일련의 메타 안정적 중간 상태를 통해 접혀야 한다고 제안했다.
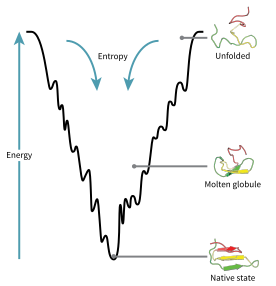
단백질 접힘의 에너지 풍경
접는 동안 단백질의 구성 공간은 에너지 풍경으로 시각화할 수 있습니다.조셉 브린겔슨과 피터 울린스에 따르면, 단백질은 최소한의 좌절의 원리를 따르는데, 이는 자연적으로 진화한 단백질이 접힌 에너지 [72]환경을 최적화했고, 자연이 아미노산 서열을 선택해서 단백질의 접힌 상태가 충분히 안정되었다는 것을 의미한다.또한 접힌 상태의 획득은 충분히 빠른 프로세스가 되어야 했습니다.비록 자연이 단백질의 좌절 수준을 감소시켰지만, 단백질의 에너지 지형에서 국소적 최소치의 존재에서 관찰될 수 있는 것 중 어느 정도는 현재까지 남아 있다.
이러한 진화적으로 선택된 배열의 결과는 일반적으로 단백질이 토종 상태를 향해 주로 향하는 "터널링 에너지 풍경" (José Onuchic에 [73]의해 만들어진 용어)을 가지고 있다고 생각됩니다.이러한 "접는 깔때기" 풍경은 단백질이 단일 메커니즘으로 제한되는 대신 많은 경로와 중간 중 하나를 통해 자연 상태로 접힐 수 있도록 합니다.이 이론은 모델 단백질의 컴퓨터 시뮬레이션과 실험 [72]연구 모두에 의해 뒷받침되며 단백질 구조 예측과 [72]설계를 위한 방법을 개선하는 데 사용되어 왔다.수평 자유 에너지 경관에 의한 단백질 접힘의 설명은 열역학 [74]제2법칙과도 일치합니다.물리적으로는 지리적인 풍경과 마찬가지로 최대치, 안장점, 최소치, 깔때기만으로 시각화할 수 있는 잠재력 또는 총 에너지 표면의 관점에서 풍경을 생각하는 것은 다소 오해의 소지가 있다.관련 설명은 다양체가 다양한 보다 복잡한 위상 [75]형태를 취할 수 있는 고차원 위상 공간입니다.
전개된 폴리펩타이드 사슬은 전개된 변형이 가장 많을 것으로 추측되는 깔때기 꼭대기에서 시작하여 가장 높은 에너지 상태에 있습니다.이러한 에너지 환경은 많은 초기 가능성이 있지만, 오직 하나의 원어민 상태만이 가능하다는 것을 나타낸다. 그러나 가능한 수많은 접힘 경로를 보여주지는 않는다.동일한 정확한 단백질의 다른 분자는 동일한 고유 구조에 [76]도달하는 한 서로 다른 낮은 에너지 중간체를 찾아 약간 다른 접힘 경로를 따를 수 있다.다른 경로들은 각 경로의 열역학적 선호도에 따라 다른 이용 빈도를 가질 수 있다.이것은 한 경로가 다른 경로보다 열역학적으로 더 유리한 것으로 판명되면, [76]자연 구조를 추구하는 데 더 자주 사용될 가능성이 높다는 것을 의미한다.단백질이 접혀지고 다양한 형태를 띠기 시작하면서, 항상 전보다 열역학적으로 유리한 구조를 찾고, 따라서 에너지 깔때기를 통해 계속된다.2차 구조의 형성은 단백질 내에서 안정성이 증가했음을 나타내는 강력한 지표이며, 폴리펩타이드 골격에 의해 가정된 2차 구조의 단 하나의 조합만이 가장 낮은 에너지를 가지므로 단백질의 [76]고유 상태에 존재한다.폴리펩타이드가 접히기 시작하면 형성되는 첫 번째 구조로는 알파나선 및 베타나선이 있으며, 알파나선은 100나노초,[29] 베타나선은 1마이크로초 안에 형성될 수 있습니다.
에너지 깔때기 풍경에는 특정 단백질의 전이 상태를 [29]확인할 수 있는 안장점이 존재합니다.에너지 깔때기 다이어그램의 전이 상태는 단백질이 최종적으로 자연 구조를 가정하기를 원하는 경우 해당 단백질의 모든 분자에 의해 가정되어야 하는 구성입니다.어떤 단백질도 먼저 전이 [29]상태를 통과하지 않고는 자연 구조를 가정할 수 없다.전이 상태는 단순히 다른 중간 [77]단계가 아닌 변형 또는 초기 형태의 네이티브 상태로 참조할 수 있습니다.전이 상태의 접힘은 속도를 결정하는 것으로 나타나며, 네이티브 폴딩보다 높은 에너지 상태로 존재하지만 네이티브 구조와 매우 유사합니다.전이 상태 내에는 단백질이 접힐 수 있는 핵이 존재하며, 핵 [77]위로 구조가 붕괴되기 시작하는 "핵 응축"이라고 불리는 과정에 의해 형성된다.
단백질 접힘 모델링
단백질 접힘의 다양한 측면을 시뮬레이션하기 위해 계산 단백질 구조 예측을 위한 de novo 또는 ab initio 기술을 사용할 수 있다.Molecular Dynamics(MD; 분자역학)는 실리콘에서 [78]단백질 접힘 및 동적 시뮬레이션에 사용되었다.첫 번째 평형 폴딩 시뮬레이션은 암묵적 용제 모델과 포괄적 [79]샘플링을 사용하여 수행되었다.계산 비용 때문에 명시적 물로 ab initio MD 폴딩 시뮬레이션은 펩타이드와 매우 작은 [80][81]단백질로 제한된다.더 큰 단백질의 MD 시뮬레이션은 실험 구조의 역학이나 고온 전개로 제한된다.작은 크기 단백질(약 50개 잔류물) 이상의 접힘과 같은 긴 시간 접힘 과정(약 1밀리초 이상)은 거친 입자 [82][83][84]모델을 사용하여 접근할 수 있다.
Rosetta@home,[85] Folding@home[86] 및 Foldit과 [87]같은 여러 대규모 계산 프로젝트는 단백질 접기를 목표로 합니다.
D. E. Show Research에 의해 커스텀 ASIC와 인터커넥트를 중심으로 설계 및 구축된 대규모 병렬 슈퍼컴퓨터 Anton에 대해 장시간 연속적인 시뮬레이션이 수행되었습니다.안톤을 사용하여 수행된 시뮬레이션의 가장 긴 공개 결과는 355K에서 [88]NTL9의 2.936밀리초 시뮬레이션입니다.
「 」를 참조해 주세요.
레퍼런스
- ^ Alberts B, Johnson A, Lewis J, Raff M, Roberts K, Walters P (2002). "The Shape and Structure of Proteins". Molecular Biology of the Cell; Fourth Edition. New York and London: Garland Science. ISBN 978-0-8153-3218-3.
- ^ Anfinsen CB (July 1972). "The formation and stabilization of protein structure". The Biochemical Journal. 128 (4): 737–49. doi:10.1042/bj1280737. PMC 1173893. PMID 4565129.
- ^ Berg JM, Tymoczko JL, Stryer L (2002). "3. Protein Structure and Function". Biochemistry. San Francisco: W. H. Freeman. ISBN 978-0-7167-4684-3.
- ^ a b Selkoe DJ (December 2003). "Folding proteins in fatal ways". Nature. 426 (6968): 900–4. Bibcode:2003Natur.426..900S. doi:10.1038/nature02264. PMID 14685251. S2CID 6451881.
- ^ Alberts B, Bray D, Hopkin K, Johnson A, Lewis J, Raff M, Roberts K, Walter P (2010). "Protein Structure and Function". Essential cell biology (Third ed.). New York, NY: Garland Science. pp. 120–70. ISBN 978-0-8153-4454-4.
- ^ Kim PS, Baldwin RL (1990). "Intermediates in the folding reactions of small proteins". Annual Review of Biochemistry. 59: 631–60. doi:10.1146/annurev.bi.59.070190.003215. PMID 2197986.
- ^ Jackson SE (1998). "How do small single-domain proteins fold?". Folding & Design. 3 (4): R81-91. doi:10.1016/S1359-0278(98)00033-9. PMID 9710577.
- ^ Kubelka J, Hofrichter J, Eaton WA (February 2004). "The protein folding 'speed limit'". Current Opinion in Structural Biology. 14 (1): 76–88. doi:10.1016/j.sbi.2004.01.013. PMID 15102453.
- ^ Scalvini, Barbara; Sheikhhassani, Vahid; Mashaghi, Alireza (2021). "Topological principles of protein folding". Physical Chemistry Chemical Physics. 23 (37): 21316–21328. Bibcode:2021PCCP...2321316S. doi:10.1039/D1CP03390E. hdl:1887/3277889. PMID 34545868. S2CID 237583577.
- ^ Anfinsen CB (July 1973). "Principles that govern the folding of protein chains". Science. 181 (4096): 223–30. Bibcode:1973Sci...181..223A. doi:10.1126/science.181.4096.223. PMID 4124164.
- ^ a b c d e f g h Voet D, Voet JG, Pratt CW (2016). Principles of Biochemistry (Fifth ed.). Wiley. ISBN 978-1-118-91840-1.
- ^ Alexander PA, He Y, Chen Y, Orban J, Bryan PN (July 2007). "The design and characterization of two proteins with 88% sequence identity but different structure and function". Proceedings of the National Academy of Sciences of the United States of America. 104 (29): 11963–8. Bibcode:2007PNAS..10411963A. doi:10.1073/pnas.0700922104. PMC 1906725. PMID 17609385.
- ^ Rose GD, Fleming PJ, Banavar JR, Maritan A (November 2006). "A backbone-based theory of protein folding". Proceedings of the National Academy of Sciences of the United States of America. 103 (45): 16623–33. Bibcode:2006PNAS..10316623R. CiteSeerX 10.1.1.630.5487. doi:10.1073/pnas.0606843103. PMC 1636505. PMID 17075053.
- ^ a b c Fersht A (1999). Structure and Mechanism in Protein Science: A Guide to Enzyme Catalysis and Protein Folding. Macmillan. ISBN 978-0-7167-3268-6.
- ^ "Protein Structure". Scitable. Nature Education. Retrieved 2016-11-26.
- ^ Pratt C, Cornely K (2004). "Thermodynamics". Essential Biochemistry. Wiley. ISBN 978-0-471-39387-0. Retrieved 2016-11-26.
- ^ Zhang G, Ignatova Z (February 2011). "Folding at the birth of the nascent chain: coordinating translation with co-translational folding". Current Opinion in Structural Biology. 21 (1): 25–31. doi:10.1016/j.sbi.2010.10.008. PMID 21111607.
- ^ van den Berg B, Wain R, Dobson CM, Ellis RJ (August 2000). "Macromolecular crowding perturbs protein refolding kinetics: implications for folding inside the cell". The EMBO Journal. 19 (15): 3870–5. doi:10.1093/emboj/19.15.3870. PMC 306593. PMID 10921869.
- ^ Al-Karadaghi S. "Torsion Angles and the Ramachnadran Plot in Protein Structures". www.proteinstructures.com. Retrieved 2016-11-26.
- ^ Pace CN, Shirley BA, McNutt M, Gajiwala K (January 1996). "Forces contributing to the conformational stability of proteins". FASEB Journal. 10 (1): 75–83. doi:10.1096/fasebj.10.1.8566551. PMID 8566551. S2CID 20021399.
- ^ Cui D, Ou S, Patel S (December 2014). "Protein-spanning water networks and implications for prediction of protein-protein interactions mediated through hydrophobic effects". Proteins. 82 (12): 3312–26. doi:10.1002/prot.24683. PMID 25204743. S2CID 27113763.
- ^ Tanford C (June 1978). "The hydrophobic effect and the organization of living matter". Science. 200 (4345): 1012–8. Bibcode:1978Sci...200.1012T. doi:10.1126/science.653353. PMID 653353.
- ^ Deechongkit S, Nguyen H, Powers ET, Dawson PE, Gruebele M, Kelly JW (July 2004). "Context-dependent contributions of backbone hydrogen bonding to beta-sheet folding energetics". Nature. 430 (6995): 101–5. Bibcode:2004Natur.430..101D. doi:10.1038/nature02611. PMID 15229605. S2CID 4315026.
- ^ Irbäck A, Sandelin E (November 2000). "On hydrophobicity correlations in protein chains". Biophysical Journal. 79 (5): 2252–8. arXiv:cond-mat/0010390. Bibcode:2000BpJ....79.2252I. doi:10.1016/S0006-3495(00)76472-1. PMC 1301114. PMID 11053106.
- ^ Irbäck A, Peterson C, Potthast F (September 1996). "Evidence for nonrandom hydrophobicity structures in protein chains". Proceedings of the National Academy of Sciences of the United States of America. 93 (18): 9533–8. arXiv:chem-ph/9512004. Bibcode:1996PNAS...93.9533I. doi:10.1073/pnas.93.18.9533. PMC 38463. PMID 8790365.
- ^ Wilson BA, Foy SG, Neme R, Masel J (June 2017). "De Novo Gene Birth". Nature Ecology & Evolution. 1 (6): 0146–146. doi:10.1038/s41559-017-0146. PMC 5476217. PMID 28642936.
- ^ Willis S, Masel J (September 2018). "Gene Birth Contributes to Structural Disorder Encoded by Overlapping Genes". Genetics. 210 (1): 303–313. doi:10.1534/genetics.118.301249. PMC 6116962. PMID 30026186.
- ^ Foy SG, Wilson BA, Bertram J, Cordes MH, Masel J (April 2019). "A Shift in Aggregation Avoidance Strategy Marks a Long-Term Direction to Protein Evolution". Genetics. 211 (4): 1345–1355. doi:10.1534/genetics.118.301719. PMC 6456324. PMID 30692195.
- ^ a b c d e f Dobson CM (December 2003). "Protein folding and misfolding". Nature. 426 (6968): 884–90. Bibcode:2003Natur.426..884D. doi:10.1038/nature02261. PMID 14685248. S2CID 1036192.
- ^ a b c Hartl FU (June 1996). "Molecular chaperones in cellular protein folding". Nature. 381 (6583): 571–9. Bibcode:1996Natur.381..571H. doi:10.1038/381571a0. PMID 8637592. S2CID 4347271.
- ^ a b Hartl FU, Bracher A, Hayer-Hartl M (July 2011). "Molecular chaperones in protein folding and proteostasis". Nature. 475 (7356): 324–32. doi:10.1038/nature10317. PMID 21776078. S2CID 4337671.
- ^ Kim YE, Hipp MS, Bracher A, Hayer-Hartl M, Hartl FU (2013). "Molecular chaperone functions in protein folding and proteostasis". Annual Review of Biochemistry. 82: 323–55. doi:10.1146/annurev-biochem-060208-092442. PMID 23746257.
- ^ Shortle D (January 1996). "The denatured state (the other half of the folding equation) and its role in protein stability". FASEB Journal. 10 (1): 27–34. doi:10.1096/fasebj.10.1.8566543. PMID 8566543. S2CID 24066207.
- ^ Lee S, Tsai FT (2005). "Molecular chaperones in protein quality control". Journal of Biochemistry and Molecular Biology. 38 (3): 259–65. doi:10.5483/BMBRep.2005.38.3.259. PMID 15943899.
- ^ Ojeda-May P, Garcia ME (July 2010). "Electric field-driven disruption of a native beta-sheet protein conformation and generation of a helix-structure". Biophysical Journal. 99 (2): 595–9. Bibcode:2010BpJ....99..595O. doi:10.1016/j.bpj.2010.04.040. PMC 2905109. PMID 20643079.
- ^ van den Berg B, Ellis RJ, Dobson CM (December 1999). "Effects of macromolecular crowding on protein folding and aggregation". The EMBO Journal. 18 (24): 6927–33. doi:10.1093/emboj/18.24.6927. PMC 1171756. PMID 10601015.
- ^ Ellis RJ (July 2006). "Molecular chaperones: assisting assembly in addition to folding". Trends in Biochemical Sciences. 31 (7): 395–401. doi:10.1016/j.tibs.2006.05.001. PMID 16716593.
- ^ Takai K, Nakamura K, Toki T, Tsunogai U, Miyazaki M, Miyazaki J, Hirayama H, Nakagawa S, Nunoura T, Horikoshi K (August 2008). "Cell proliferation at 122 degrees C and isotopically heavy CH4 production by a hyperthermophilic methanogen under high-pressure cultivation". Proceedings of the National Academy of Sciences of the United States of America. 105 (31): 10949–54. Bibcode:2008PNAS..10510949T. doi:10.1073/pnas.0712334105. PMC 2490668. PMID 18664583.
- ^ a b 마루시치 EI, 쿠로치키나 LP, 메시안지노프 VV박테리오파지 T4 어셈블리의 샤페론.생화학(모스크)1998;63(4):399-406
- ^ Porter, Lauren L.; Looger, Loren L. (5 June 2018). "Extant fold-switching proteins are widespread". Proceedings of the National Academy of Sciences. 115 (23): 5968–5973. doi:10.1073/pnas.1800168115. PMC 6003340. PMID 29784778.
- ^ a b c d Chaudhuri TK, Paul S (April 2006). "Protein-misfolding diseases and chaperone-based therapeutic approaches". The FEBS Journal. 273 (7): 1331–49. doi:10.1111/j.1742-4658.2006.05181.x. PMID 16689923. S2CID 23370420.
- ^ a b Soto C, Estrada L, Castilla J (March 2006). "Amyloids, prions and the inherent infectious nature of misfolded protein aggregates". Trends in Biochemical Sciences. 31 (3): 150–5. doi:10.1016/j.tibs.2006.01.002. PMID 16473510.
- ^ Hammarström P, Wiseman RL, Powers ET, Kelly JW (January 2003). "Prevention of transthyretin amyloid disease by changing protein misfolding energetics". Science. 299 (5607): 713–6. Bibcode:2003Sci...299..713H. doi:10.1126/science.1079589. PMID 12560553. S2CID 30829998.
- ^ Chiti F, Dobson CM (2006). "Protein misfolding, functional amyloid, and human disease". Annual Review of Biochemistry. 75: 333–66. doi:10.1146/annurev.biochem.75.101304.123901. PMID 16756495.
- ^ Johnson SM, Wiseman RL, Sekijima Y, Green NS, Adamski-Werner SL, Kelly JW (December 2005). "Native state kinetic stabilization as a strategy to ameliorate protein misfolding diseases: a focus on the transthyretin amyloidoses". Accounts of Chemical Research. 38 (12): 911–21. doi:10.1021/ar020073i. PMID 16359163.
- ^ a b Cowtan K (2001). "Phase Problem in X-ray Crystallography, and Its Solution" (PDF). Encyclopedia of Life Sciences. Macmillan Publishers Ltd, Nature Publishing Group. Retrieved November 3, 2016.
- ^ Drenth J (2007-04-05). Principles of Protein X-Ray Crystallography. Springer Science & Business Media. ISBN 978-0-387-33746-3.
- ^ Taylor G (2003). "The phase problem". Acta Crystallographica Section D. 59 (11): 1881–90. doi:10.1107/S0907444903017815. PMID 14573942.
- ^ a b c Bedouelle H (February 2016). "Principles and equations for measuring and interpreting protein stability: From monomer to tetramer". Biochimie. 121: 29–37. doi:10.1016/j.biochi.2015.11.013. PMID 26607240.
- ^ Monsellier E, Bedouelle H (September 2005). "Quantitative measurement of protein stability from unfolding equilibria monitored with the fluorescence maximum wavelength". Protein Engineering, Design & Selection. 18 (9): 445–56. doi:10.1093/protein/gzi046. PMID 16087653.
- ^ Park YC, Bedouelle H (July 1998). "Dimeric tyrosyl-tRNA synthetase from Bacillus stearothermophilus unfolds through a monomeric intermediate. A quantitative analysis under equilibrium conditions". The Journal of Biological Chemistry. 273 (29): 18052–9. doi:10.1074/jbc.273.29.18052. PMID 9660761.
- ^ Ould-Abeih MB, Petit-Topin I, Zidane N, Baron B, Bedouelle H (June 2012). "Multiple folding states and disorder of ribosomal protein SA, a membrane receptor for laminin, anticarcinogens, and pathogens". Biochemistry. 51 (24): 4807–21. doi:10.1021/bi300335r. PMID 22640394.
- ^ Royer CA (May 2006). "Probing protein folding and conformational transitions with fluorescence". Chemical Reviews. 106 (5): 1769–84. doi:10.1021/cr0404390. PMID 16683754.
- ^ a b c d Wüthrich K (December 1990). "Protein structure determination in solution by NMR spectroscopy". The Journal of Biological Chemistry. 265 (36): 22059–62. doi:10.1016/S0021-9258(18)45665-7. PMID 2266107.
- ^ a b c d e Zhuravleva A, Korzhnev DM (May 2017). "Protein folding by NMR". Progress in Nuclear Magnetic Resonance Spectroscopy. 100: 52–77. doi:10.1016/j.pnmrs.2016.10.002. PMID 28552172.
- ^ a b Ortega G, Pons M, Millet O (2013-01-01). Karabencheva-Christova T (ed.). "Protein functional dynamics in multiple timescales as studied by NMR spectroscopy". Advances in Protein Chemistry and Structural Biology. Dynamics of Proteins and Nucleic Acids. Academic Press. 92: 219–51. doi:10.1016/b978-0-12-411636-8.00006-7. ISBN 9780124116368. PMID 23954103.
- ^ a b c Vallurupalli P, Bouvignies G, Kay LE (May 2012). "Studying "invisible" excited protein states in slow exchange with a major state conformation". Journal of the American Chemical Society. 134 (19): 8148–61. doi:10.1021/ja3001419. PMID 22554188.
- ^ a b Neudecker P, Lundström P, Kay LE (March 2009). "Relaxation dispersion NMR spectroscopy as a tool for detailed studies of protein folding". Biophysical Journal. 96 (6): 2045–54. Bibcode:2009BpJ....96.2045N. doi:10.1016/j.bpj.2008.12.3907. PMC 2717354. PMID 19289032.
- ^ a b Sekhar A, Rumfeldt JA, Broom HR, Doyle CM, Sobering RE, Meiering EM, Kay LE (November 2016). "Probing the free energy landscapes of ALS disease mutants of SOD1 by NMR spectroscopy". Proceedings of the National Academy of Sciences of the United States of America. 113 (45): E6939–E6945. doi:10.1073/pnas.1611418113. PMC 5111666. PMID 27791136.
- ^ Cross GH, Freeman NJ, Swann MJ (2008). "Dual Polarization Interferometry: A Real-Time Optical Technique for Measuring (Bio)molecular Orientation, Structure and Function at the Solid/Liquid Interface". Handbook of Biosensors and Biochips. doi:10.1002/9780470061565.hbb055. ISBN 978-0-470-01905-4.
- ^ Bu Z, Cook J, Callaway DJ (September 2001). "Dynamic regimes and correlated structural dynamics in native and denatured alpha-lactalbumin". Journal of Molecular Biology. 312 (4): 865–73. doi:10.1006/jmbi.2001.5006. PMID 11575938.
- ^ Minde DP, Maurice MM, Rüdiger SG (2012). "Determining biophysical protein stability in lysates by a fast proteolysis assay, FASTpp". PLOS ONE. 7 (10): e46147. Bibcode:2012PLoSO...746147M. doi:10.1371/journal.pone.0046147. PMC 3463568. PMID 23056252.
- ^ Park C, Marqusee S (March 2005). "Pulse proteolysis: a simple method for quantitative determination of protein stability and ligand binding". Nature Methods. 2 (3): 207–12. doi:10.1038/nmeth740. PMID 15782190. S2CID 21364478.
- ^ Mashaghi A, Kramer G, Lamb DC, Mayer MP, Tans SJ (January 2014). "Chaperone action at the single-molecule level". Chemical Reviews. 114 (1): 660–76. doi:10.1021/cr400326k. PMID 24001118.
- ^ Jagannathan B, Marqusee S (November 2013). "Protein folding and unfolding under force". Biopolymers. 99 (11): 860–9. doi:10.1002/bip.22321. PMC 4065244. PMID 23784721.
- ^ Jakobi AJ, Mashaghi A, Tans SJ, Huizinga EG (July 2011). "Calcium modulates force sensing by the von Willebrand factor A2 domain". Nature Communications. 2: 385. Bibcode:2011NatCo...2..385J. doi:10.1038/ncomms1385. PMC 3144584. PMID 21750539.
- ^ Jagannathan B, Elms PJ, Bustamante C, Marqusee S (October 2012). "Direct observation of a force-induced switch in the anisotropic mechanical unfolding pathway of a protein". Proceedings of the National Academy of Sciences of the United States of America. 109 (44): 17820–5. Bibcode:2012PNAS..10917820J. doi:10.1073/pnas.1201800109. PMC 3497811. PMID 22949695.
- ^ Minde DP, Ramakrishna M, Lilley KS (2018). "Biotinylation by proximity labelling favours unfolded proteins". bioRxiv. doi:10.1101/274761.
- ^ Compiani M, Capriotti E (December 2013). "Computational and theoretical methods for protein folding". Biochemistry. 52 (48): 8601–24. doi:10.1021/bi4001529. PMID 24187909.
- ^ "Structural Biochemistry/Proteins/Protein Folding - Wikibooks, open books for an open world". en.wikibooks.org. Retrieved 2016-11-05.
- ^ Levinthal C (1968). "Are there pathways for protein folding?" (PDF). Journal de Chimie Physique et de Physico-Chimie Biologique. 65: 44–45. Bibcode:1968JCP....65...44L. doi:10.1051/jcp/1968650044. Archived from the original (PDF) on 2009-09-02.
- ^ a b c Bryngelson JD, Onuchic JN, Socci ND, Wolynes PG (March 1995). "Funnels, pathways, and the energy landscape of protein folding: a synthesis". Proteins. 21 (3): 167–95. arXiv:chem-ph/9411008. doi:10.1002/prot.340210302. PMID 7784423. S2CID 13838095.
- ^ Leopold PE, Montal M, Onuchic JN (September 1992). "Protein folding funnels: a kinetic approach to the sequence-structure relationship". Proceedings of the National Academy of Sciences of the United States of America. 89 (18): 8721–5. Bibcode:1992PNAS...89.8721L. doi:10.1073/pnas.89.18.8721. PMC 49992. PMID 1528885.
- ^ Sharma V, Kaila VR, Annila A (2009). "Protein folding as an evolutionary process". Physica A: Statistical Mechanics and Its Applications. 388 (6): 851–62. Bibcode:2009PhyA..388..851S. doi:10.1016/j.physa.2008.12.004.
- ^ Robson B, Vaithilingam A (2008). "Protein Folding Revisited". Molecular Biology of Protein Folding, Part B. Progress in Molecular Biology and Translational Science. Vol. 84. pp. 161–202. doi:10.1016/S0079-6603(08)00405-4. ISBN 978-0-12-374595-8. PMID 19121702.
- ^ a b c Dill KA, MacCallum JL (November 2012). "The protein-folding problem, 50 years on". Science. 338 (6110): 1042–6. Bibcode:2012Sci...338.1042D. doi:10.1126/science.1219021. PMID 23180855. S2CID 5756068.
- ^ a b Fersht AR (February 2000). "Transition-state structure as a unifying basis in protein-folding mechanisms: contact order, chain topology, stability, and the extended nucleus mechanism". Proceedings of the National Academy of Sciences of the United States of America. 97 (4): 1525–9. Bibcode:2000PNAS...97.1525F. doi:10.1073/pnas.97.4.1525. PMC 26468. PMID 10677494.
- ^ Rizzuti B, Daggett V (March 2013). "Using simulations to provide the framework for experimental protein folding studies". Archives of Biochemistry and Biophysics. 531 (1–2): 128–35. doi:10.1016/j.abb.2012.12.015. PMC 4084838. PMID 23266569.
- ^ Schaefer M, Bartels C, Karplus M (December 1998). "Solution conformations and thermodynamics of structured peptides: molecular dynamics simulation with an implicit solvation model". Journal of Molecular Biology. 284 (3): 835–48. doi:10.1006/jmbi.1998.2172. PMID 9826519.
- ^ Jones D. "Fragment-based Protein Folding Simulations". University College London.
- ^ "Protein folding" (by Molecular Dynamics).
- ^ Kmiecik S, Gront D, Kolinski M, Wieteska L, Dawid AE, Kolinski A (July 2016). "Coarse-Grained Protein Models and Their Applications". Chemical Reviews. 116 (14): 7898–936. doi:10.1021/acs.chemrev.6b00163. PMID 27333362.
- ^ Kmiecik S, Kolinski A (July 2007). "Characterization of protein-folding pathways by reduced-space modeling". Proceedings of the National Academy of Sciences of the United States of America. 104 (30): 12330–5. Bibcode:2007PNAS..10412330K. doi:10.1073/pnas.0702265104. PMC 1941469. PMID 17636132.
- ^ Adhikari AN, Freed KF, Sosnick TR (October 2012). "De novo prediction of protein folding pathways and structure using the principle of sequential stabilization". Proceedings of the National Academy of Sciences of the United States of America. 109 (43): 17442–7. Bibcode:2012PNAS..10917442A. doi:10.1073/pnas.1209000109. PMC 3491489. PMID 23045636.
- ^ 로제타@집입니다
- ^ 접기@집입니다
- ^ FoldIt - 접이식 단백질 게임
- ^ Lindorff-Larsen K, Piana S, Dror RO, Shaw DE (October 2011). "How fast-folding proteins fold". Science. 334 (6055): 517–20. Bibcode:2011Sci...334..517L. doi:10.1126/science.1208351. PMID 22034434. S2CID 27988268.