토폴로지 연관 도메인
Topologically associating domain위상학적으로 연관된 도메인(TAD)은 자가 상호작용 유전체 영역으로, TAD 내의 DNA 시퀀스가 TAD 외부의 시퀀스와 보다 물리적으로 더 자주 상호 작용한다는 것을 의미한다.[1]쥐세포에서 TAD의 중앙 크기는 880kb이며, 비마민종에서도 비슷한 크기를 가지고 있다.[2]이러한 영역의 양쪽에 있는 경계는 서로 다른 포유류 세포 유형과 심지어[2] 종 간에 보존되며 CCCTC 결합 계수(CTCF)와 응집 결합 부위로 고농축된다.[1]게다가, 어떤 종류의 유전자(전송 RNA 유전자와 하우스키핑 유전자와 같은)는 우연히 예상할 수 있는 것보다 더 자주 TAD 경계 근처에 나타난다.[3][4]
TAD의 기능은 충분히 이해되지 않고 여전히 논쟁의 대상이다.대부분의 연구는 TADs가 각 TAD에 대한 엔핸서-프로모터 상호작용을 제한함으로써 유전자 발현을 조절한다는 것을 보여주지만,[5] 최근의 연구는 TAD의 조직과 유전자 발현을 분리한다.[6]TAD 경계의 붕괴는 암과 같은 광범위한 질병,[7][8][9] 시네플리닥틸리, 쿡스증후군, F신드롬과 같은 다양한 사지 기형,[10] 그리고 히포플라스틱 코퍼스 콜로섬, 성인-온셋 탈수 백혈구역질증과 같은 뇌 질환의 수와 관련이 있는 것으로 밝혀졌다.[10]
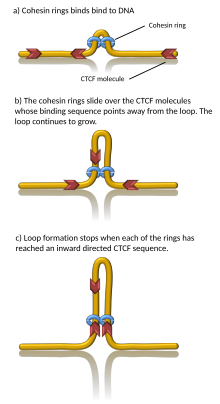
비록 많은 단백질 복합체들과 DNA 요소들이 TAD 경계와 연관되어 있지만, TAD 형성의 기초는 복잡하고 아직 완전히 설명되지 않았다.그러나 수갑 모델과 루프 압출 모델은 CTCF와 응집 단백질에 의한 TAD 형성을 설명한다.[11]나아가, TAD 경계 자체의 경직성이 도메인 절연과 TAD 형성을 야기할 수 있다는 제안이 제기되었다.[11]
발견과 다양성
TAD는 DNA 서열이 서로 우선적으로 접촉하는 지역으로 정의된다.그것들은 Hi-C를 포함한 염색체 순응 포획 기술을 사용하여 2012년에 발견되었다.[3][12][4]이들은 초파리([14]드로소필라), 쥐,[3] 식물, 곰팡이, 인간[4] 게놈 [13]등 다양한 종에 존재하는 것으로 나타났다.박테리아에서는 염색체 상호 작용 도메인(CIDs)이라고 한다.[13]
분석 도구 및 데이터베이스
TAD 위치는 Hi-C 데이터에 알고리즘을 적용하여 정의한다.예를 들어, 흔히 TAD를 소위 "방향성 지수"[4]에 따라 부른다.방향성 지수는 개별 40kb bin에 대해 계산되며, bin에 포함된 판독치를 수집하고, 쌍으로 구성된 판독치가 bin의 업스트림 또는 다운스트림 지도(읽기 쌍은 2Mb 이하까지 확장해야 함)를 관찰한다.양의 값은 업스트림보다 더 많은 읽기 쌍이 다운스트림에 놓여 있음을 나타내며, 음의 값은 그 반대 값을 나타낸다.수학적으로 방향성 지수는 서명된 카이-제곱 통계량이다.
쥬스박스,[16] 하이글래스[17]/하이필러,[18] 3D 게놈브라우저, [19]3DIV,[20] 3D-GNOME,[21] TADKB[22] 등 전문 게놈 브라우저와 시각화[15] 도구를 개발함으로써 서로 다른 세포 유형에 관심 있는 영역의 TAD 조직을 시각화할 수 있었다.
형성 메커니즘
CTCF 단백질과 복합 단백질 응집력을 포함한 많은 단백질들이 TAD 형성과 관련이 있는 것으로 알려져 있다.[1]또한 TAD 경계에서 어떤 성분이 필요한지는 알 수 없지만, 포유류 세포의 경우 이러한 경계 영역은 비교적 높은 수준의 CTCF 결합을 가지고 있는 것으로 나타났다.게다가, 어떤 종류의 유전자(전송 RNA 유전자와 하우스키핑 유전자와 같은)는 우연히 예상할 수 있는 것보다 더 자주 TAD 경계 근처에 나타난다.[3][4]
컴퓨터 시뮬레이션에 따르면 전사가 발생한 슈퍼코일링에 의해 구동되는 염색질 루프 압출은 응집력이 빠르게 다시 계산되고 루프가 합리적인 속도로 좋은 방향으로 성장하도록 보장한다.또한 슈퍼코일링 구동 루프 압출 메커니즘은 보존된 CTCF 바인딩 사이트 옆에 있는 TAD가 다른 CTCF 바인딩 사이트 옆에 있는 TAD보다 더 안정적인 염색질 루프를 형성하는 이유를 제안하는 이전의 설명과 일치한다.이 모델에서 슈퍼코일링은 또한 엔핸서 프로모터 접점을 자극하며, eRNA의 필사본이 주어진 TAD에서 mRNA 전사를 활성화할 수 있는 1단계 슈퍼코일링을 보낼 것을 제안한다.[23][24]컴퓨터 모델도 응집력 고리가 매우 효율적인 분자 빗처럼 작용한다는 것을 보여주었고, 결합력 고리는 Topoisomeras의 작용에 의해 제거되는 TAD의 경계로 캐테나네기와 같은 얽힘과 같은 것을 밀어낸다.일관되게, 루프 압출 중 얽힘 제거는 염색체 간의 분리 정도를 증가시킨다.[25]그러나 DNA 루프 압출에 대한 증거는 지금까지 콘덴신(코신 자매 단백질 복합체)에만 한정되어 있다.[26]
특성.
보존
TAD는 (예를 들어 줄기 세포와 혈액 세포에서) 서로 다른 세포 유형 간에, 그리고 심지어 특정한 경우 종 간에 비교적 일정하다고 보고되어 왔다.[27][28]
프로모터-인핸서 연락처와의 관계
촉진자와 촉진자 사이의 관찰된 상호작용의 대부분은 TAD 경계를 넘지 않는다.TAD 경계를 제거하면(예: CRISPR을 사용하여 게놈의 관련 영역을 삭제함) 새로운 촉진제-증진제 접점이 형성될 수 있다.이것은 근처의 유전자 발현에 영향을 미칠 수 있다 - 그러한 잘못된 규제는 인간과 생쥐에서 사지 기형을 유발하는 것으로 밝혀졌다.[27]
컴퓨터 시뮬레이션에 따르면 전사에 의한 염색질 섬유 초코팅은 어떻게 TAD가 형성되는지 그리고 그것들이 같은 TAD에 위치한 엔핸서들과 그들의 인지 촉진자들 사이의 매우 효율적인 상호작용을 어떻게 보장할 수 있는지를 설명할 수 있다.[24]
게놈의 다른 구조적 특징과의 관계
복제 타이밍 도메인의 경계는 구획의 양쪽에 위치한 TAD의 경계와 함께 지역화되기 때문에 TAD와 관련된 것으로 나타났다.[29]절연된 이웃들, 즉 CTCF/코신 결합 지역에 의해 형성된 DNA 루프는 기능적으로 TAD의 기반이 되는 것으로 제안된다.[30]
질병에서의 역할
TAD 경계의 붕괴는 인근 유전자의 발현에 영향을 줄 수 있으며, 이로 인해 질병이 발생할 수 있다.[31]
예를 들어, TAD 경계를 교란하는 유전학적 구조변형은 인간의 사지 기형과 같은 발달장애를 유발하는 것으로 보고되었다.[32][33][34]또한 여러 연구에서 T-세포 급성 림프성 백혈병([35]T-ALL), 글리오마,[36] 폐암과 같은 특정 암에 성장 이점을 제공할 수 있다는 증거를 제공했다.[37]
라미나 관련 도메인
라미나 관련 도메인(LADs)은 핵의 내부 막에 있는 네트워크와 같은 구조인 라미나(Lamina)와 크게 상호작용하는 염색질의 부분이다.[38]LAD는 대부분 전사적으로 무성 크로마틴으로 구성되며, 히스톤 H3에 트리메틸화 라이스27로 농축되는데, 이는 헤테로크로마틴의 일반적인 변환 후 히스톤 수정이다.[39]LADs는 주변부에 CTCF 바인딩 사이트를 가지고 있다.[38]
참고 항목
참조
- ^ a b c Pombo A, Dillon N (April 2015). "Three-dimensional genome architecture: players and mechanisms". Nature Reviews. Molecular Cell Biology. 16 (4): 245–57. doi:10.1038/nrm3965. PMID 25757416. S2CID 6713103.
- ^ a b Yu M, Ren B (October 2017). "The Three-Dimensional Organization of Mammalian Genomes". Annual Review of Cell and Developmental Biology. 33: 265–289. doi:10.1146/annurev-cellbio-100616-060531. PMC 5837811. PMID 28783961.
- ^ a b c d Nora EP, Lajoie BR, Schulz EG, Giorgetti L, Okamoto I, Servant N, et al. (April 2012). "Spatial partitioning of the regulatory landscape of the X-inactivation centre". Nature. 485 (7398): 381–5. Bibcode:2012Natur.485..381N. doi:10.1038/nature11049. PMC 3555144. PMID 22495304.
- ^ a b c d e Dixon JR, Selvaraj S, Yue F, Kim A, Li Y, Shen Y, et al. (April 2012). "Topological domains in mammalian genomes identified by analysis of chromatin interactions". Nature. 485 (7398): 376–80. Bibcode:2012Natur.485..376D. doi:10.1038/nature11082. PMC 3356448. PMID 22495300.
- ^ Krijger PH, de Laat W (December 2016). "Regulation of disease-associated gene expression in the 3D genome". Nature Reviews. Molecular Cell Biology. 17 (12): 771–782. doi:10.1038/nrm.2016.138. PMID 27826147. S2CID 11484886.
- ^ Ghavi-Helm Y, Jankowski A, Meiers S, Viales RR, Korbel JO, Furlong EE (August 2019). "Highly rearranged chromosomes reveal uncoupling between genome topology and gene expression". Nature Genetics. 51 (8): 1272–1282. doi:10.1038/s41588-019-0462-3. PMC 7116017. PMID 31308546.
- ^ Corces MR, Corces VG (February 2016). "The three-dimensional cancer genome". Current Opinion in Genetics & Development. 36: 1–7. doi:10.1016/j.gde.2016.01.002. PMC 4880523. PMID 26855137.
- ^ Valton AL, Dekker J (February 2016). "TAD disruption as oncogenic driver". Current Opinion in Genetics & Development. 36: 34–40. doi:10.1016/j.gde.2016.03.008. PMC 4880504. PMID 27111891.
- ^ Achinger-Kawecka J, Clark SJ (January 2017). "Disruption of the 3D cancer genome blueprint". Epigenomics. 9 (1): 47–55. doi:10.2217/epi-2016-0111. PMID 27936932.
- ^ a b Spielmann M, Lupiáñez DG, Mundlos S (July 2018). "Structural variation in the 3D genome". Nature Reviews. Genetics. 19 (7): 453–467. doi:10.1038/s41576-018-0007-0. hdl:21.11116/0000-0003-610A-5. PMID 29692413. S2CID 22325904.
- ^ a b Dixon JR, Gorkin DU, Ren B (June 2016). "Chromatin Domains: The Unit of Chromosome Organization". Molecular Cell. 62 (5): 668–80. doi:10.1016/j.molcel.2016.05.018. PMC 5371509. PMID 27259200.
- ^ de Laat W, Duboule D (October 2013). "Topology of mammalian developmental enhancers and their regulatory landscapes". Nature. 502 (7472): 499–506. Bibcode:2013Natur.502..499D. doi:10.1038/nature12753. PMID 24153303. S2CID 4468533.
- ^ a b Szabo Q, Bantignies F, Cavalli G (April 2019). "Principles of genome folding into topologically associating domains". Science Advances. 5 (4): eaaw1668. Bibcode:2019SciA....5.1668S. doi:10.1126/sciadv.aaw1668. PMC 6457944. PMID 30989119.
- ^ Sexton T, Yaffe E, Kenigsberg E, Bantignies F, Leblanc B, Hoichman M, et al. (February 2012). "Three-dimensional folding and functional organization principles of the Drosophila genome". Cell. 148 (3): 458–72. doi:10.1016/j.cell.2012.01.010. PMID 22265598.
- ^ Ing-Simmons E, Vaquerizas JM (July 2019). "Visualising three-dimensional genome organisation in two dimensions". Development. 146 (19): 99–101. doi:10.1242/dev.177162. PMID 31558569.
- ^ Durand NC, Robinson JT, Shamim MS, Machol I, Mesirov JP, Lander ES, et al. (July 2016). "Juicebox provides a visualization system for Hi-C contact maps with unlimited zoom". Cell Systems. 3 (1): 99–101. doi:10.1016/j.cels.2015.07.012. PMC 5596920. PMID 27467250.
- ^ Kerpedjiev P, Abdennur N, Lekschas F, McCallum C, Dinkla K, Strobelt H, et al. (August 2018). "HiGlass: Web-Based Visual Exploration and Analysis of Genome Interaction Maps". Genome Biology. 3 (1): 99–101. doi:10.1186/s13059-018-1486-1. PMC 6109259. PMID 30143029.
- ^ Lekschas F, Bach B, Kerpedjiev P, Gehlenborg N, Pfister H (January 2018). "HiPiler: Visual Exploration of Large Genome Interaction Matrices with Interactive Small Multiples". IEEE Transactions on Visualization and Computer Graphics. 24 (1): 522--521. doi:10.1109/TVCG.2017.2745978. PMC 6038708. PMID 28866592.
- ^ Wang Y, Song F, Zhang B, Zhang L, Xu J, Kuang D, et al. (October 2018). "The 3D Genome Browser: a web-based browser for visualizing 3D genome organization and long-range chromatin interactions". Genome Biology. 19 (1): 151. doi:10.1186/s13059-018-1519-9. PMC 6172833. PMID 30286773.
- ^ Yang D, Jang I, Choi J, Kim MS, Lee AJ, Kim H, et al. (January 2018). "3DIV: A 3D-genome Interaction Viewer and database". Nucleic Acids Research. 46 (D1): D52–D57. doi:10.1093/nar/gkx1017. PMC 5753379. PMID 29106613.
- ^ Szalaj P, Michalski PJ, Wróblewski P, Tang Z, Kadlof M, Mazzocco G, et al. (July 2016). "3D-GNOME: an integrated web service for structural modeling of the 3D genome". Nucleic Acids Research. 44 (W1): W288-93. doi:10.1093/nar/gkw437. PMC 4987952. PMID 27185892.
- ^ 류, T, 포터, J, 자오, C 등.TADKB: 패밀리 분류 및 토폴로지 연계 도메인의 지식 기반.BMC Genomics 20, 217 (2019)https://doi.org/10.1186/s12864-019-5551-2
- ^ Racko D, 베네데티 F, Dorier J스테이지액 A(2018년 2월)."Transcription-induced 염색질 루프 압출의 원동력으로 TADs의 형성되는 동안에 간기 상동 염색체 내 supercoiling".핵산 연구 46(4):1648–1660. doi:10.1093/nar/gkx1123.PMC5829651.PMID 29140466.재료는 창조적 공용 귀인 4.0국제 라이센스 하에 가능하다 이 원본에서 복사되었다.
- ^ a b Racko D, Benedetti F, Dorier J, Stasiak A (January 2019). "Are TADs supercoiled?". Nucleic Acids Research. 47 (2): 521–532. doi:10.1093/nar/gky1091. PMC 6344874. PMID 30395328.
- ^ Racko D, Benedetti F, Goundaroulis D, Stasiak A (October 2018). "Chromatin Loop Extrusion and Chromatin Unknotting". Polymers. 10 (10): 1126–1137. doi:10.3390/polym10101126. PMC 6403842. PMID 30961051.
- ^ Ganji M, Shaltiel IA, Bisht S, Kim E, Kalichava A, Haering CH, Dekker C (April 2018). "Real-time imaging of DNA loop extrusion by condensin". Science. 360 (6384): 102–105. Bibcode:2018Sci...360..102G. doi:10.1126/science.aar7831. PMC 6329450. PMID 29472443.
- ^ a b Jost D, Vaillant C, Meister P (February 2017). "Coupling 1D modifications and 3D nuclear organization: data, models and function". Current Opinion in Cell Biology. 44: 20–27. doi:10.1016/j.ceb.2016.12.001. PMID 28040646.
- ^ Yang Y, Zhang Y, Ren B, Dixon JR, Ma J (June 2019). "Comparing 3D Genome Organization in Multiple Species Using Phylo-HMRF". Cell Systems. 8 (6): 494–505.e14. doi:10.1016/j.cels.2019.05.011. PMC 6706282. PMID 31229558.
- ^ Marchal C, Sima J, Gilbert DM (December 2019). "Control of DNA replication timing in the 3D genome". Nature Reviews. Molecular Cell Biology. 20 (12): 721–737. doi:10.1038/s41580-019-0162-y. PMID 31477886. S2CID 201714312.
- ^ Ji X, Dadon DB, Powell BE, Fan ZP, Borges-Rivera D, Shachar S, et al. (February 2016). "3D Chromosome Regulatory Landscape of Human Pluripotent Cells". Cell Stem Cell. 18 (2): 262–75. doi:10.1016/j.stem.2015.11.007. PMC 4848748. PMID 26686465.
- ^ Lupiáñez DG, Spielmann M, Mundlos S (April 2016). "Breaking TADs: How Alterations of Chromatin Domains Result in Disease". Trends in Genetics. 32 (4): 225–237. doi:10.1016/j.tig.2016.01.003. hdl:11858/00-001M-0000-002E-1D1D-D. PMID 26862051.
- ^ Lupiáñez DG, Kraft K, Heinrich V, Krawitz P, Brancati F, Klopocki E, et al. (May 2015). "Disruptions of topological chromatin domains cause pathogenic rewiring of gene-enhancer interactions". Cell. 161 (5): 1012–1025. doi:10.1016/j.cell.2015.04.004. PMC 4791538. PMID 25959774.
- ^ Angier N (2017-01-09). "A Family's Shared Defect Sheds Light on the Human Genome". The New York Times.
- ^ Franke M, Ibrahim DM, Andrey G, Schwarzer W, Heinrich V, Schöpflin R, et al. (October 2016). "Formation of new chromatin domains determines pathogenicity of genomic duplications". Nature. 538 (7624): 265–269. Bibcode:2016Natur.538..265F. doi:10.1038/nature19800. hdl:11858/00-001M-0000-002C-010A-3. PMID 27706140. S2CID 4463482.
- ^ Hnisz D, Weintraub AS, Day DS, Valton AL, Bak RO, Li CH, et al. (March 2016). "Activation of proto-oncogenes by disruption of chromosome neighborhoods". Science. 351 (6280): 1454–1458. Bibcode:2016Sci...351.1454H. doi:10.1126/science.aad9024. PMC 4884612. PMID 26940867.
- ^ Flavahan WA, Drier Y, Liau BB, Gillespie SM, Venteicher AS, Stemmer-Rachamimov AO, et al. (January 2016). "Insulator dysfunction and oncogene activation in IDH mutant gliomas". Nature. 529 (7584): 110–4. Bibcode:2016Natur.529..110F. doi:10.1038/nature16490. PMC 4831574. PMID 26700815.
- ^ Weischenfeldt J, Dubash T, Drainas AP, Mardin BR, Chen Y, Stütz AM, et al. (January 2017). "Pan-cancer analysis of somatic copy-number alterations implicates IRS4 and IGF2 in enhancer hijacking". Nature Genetics. 49 (1): 65–74. doi:10.1038/ng.3722. PMC 5791882. PMID 27869826.
- ^ a b Gonzalez-Sandoval A, Gasser SM (August 2016). "On TADs and LADs: Spatial Control Over Gene Expression". Trends in Genetics. 32 (8): 485–495. doi:10.1016/j.tig.2016.05.004. PMID 27312344.
- ^ Li M, Liu GH, Izpisua Belmonte JC (July 2012). "Navigating the epigenetic landscape of pluripotent stem cells". Nature Reviews. Molecular Cell Biology. 13 (8): 524–35. doi:10.1038/nrm3393. PMID 22820889. S2CID 22524502.