유전자 발현 조절
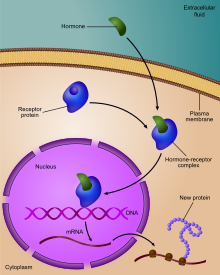
Regulation of gene expression유전자 발현 조절 또는 유전자 [1]조절은 특정 유전자 생성물(단백질 또는 RNA)의 생산을 증가 또는 감소시키기 위해 세포에 의해 사용되는 광범위한 메커니즘을 포함한다.유전자 발현에 대한 정교한 프로그램은 생물학에서 널리 관찰되는데, 예를 들어, 발달 경로를 촉발하거나, 환경 자극에 반응하거나, 새로운 음식 자원에 적응하는 것이다.유전자 발현의 거의 모든 단계는 전사 개시부터 RNA 처리 및 단백질의 번역 후 수식에 이르기까지 변조될 수 있다.유전자 조절 네트워크에서는 하나의 유전자 조절기가 다른 유전자 조절 네트워크를 제어하는 경우가 많습니다.
유전자 조절은 필요할 때 세포가 단백질을 발현하도록 함으로써 생물의 다양성과 적응성을 증가시키기 때문에 바이러스, 원핵생물, 진핵생물에게 필수적이다.1951년 바바라 맥클린톡은 옥수수 씨앗의 색 형성에 있어서 두 유전자 위치, Activator (Ac)와 Disoliator (Ds) 사이의 상호작용을 보였지만, 유전자 조절 시스템의 첫 발견은 1961년 프랑수아 제이콥과 자크 모노드에 의해 발견된 라콘의 식별으로 널리 고려되고 있다.유당 대사에 관여하는 효소는 유당이 존재하고 포도당이 없는 경우에만 대장균에 의해 발현된다.
다세포 유기체에서, 유전자 조절은 배아의 세포 분화와 형태 형성을 촉진하고, 동일한 게놈 배열로부터 다른 유전자 발현 프로파일을 가진 다른 세포 유형을 생성하도록 이끈다.비록 이것이 유전자 조절이 어떻게 시작되었는지를 설명하지는 않지만, 진화 생물학자들은 진화가 분자 수준에서 어떻게 작용하는지에 대한 부분적인 설명으로 그것을 포함시키고, 그것은 진화 발달 생물학의 과학의 중심이다.
유전자 발현 조절 단계
단백질의 시그널링에서 전사, 번역 후 수식까지 유전자 발현의 모든 단계가 변조될 수 있다.다음은 유전자 발현이 조절되는 단계 목록이며, 가장 널리 사용되는 지점이 Transcription Initiation입니다.
DNA의 수정
진핵생물에서, DNA의 큰 영역의 접근성은 DNA 메틸화, ncRNA 또는 DNA 결합 단백질에 의해 지시되는 히스톤 변형의 결과로 바뀔 수 있는 염색질 구조에 의존할 수 있다.따라서 이러한 변형은 유전자의 발현을 상향 또는 하향 조정할 수 있다.유전자 발현을 조절하는 이러한 변형들 중 일부는 유전될 수 있으며 후생적 조절이라고 불린다.
구조
DNA의 전사는 그 구조에 의해 결정된다.일반적으로 패킹의 밀도는 전사 빈도를 나타낸다.히스톤이라고 불리는 8개의 히스톤 단백질 주위에 감겨진 DNA의 일부와 함께 히스톤이라고 불리는 8가지 단백질 복합체는 DNA의 초코일의 양을 책임지고, 이러한 복합체는 인산화와 같은 과정이나 메티와 같은 과정에 의해 일시적으로 수정될 수 있습니다.이러한 수정은 유전자 발현 [2]수준의 다소 영구적인 변화에 책임이 있는 것으로 여겨진다.
화학의
DNA의 메틸화는 유전자 소음의 일반적인 방법이다.DNA는 일반적으로 CpG 디뉴클레오티드 배열(조밀하게 군집될 경우 "CpG 아일랜드"라고도 함)에서 시토신 뉴클레오티드의 메틸전달효소 효소에 의해 메틸화된다.(촉진제일 수 있는) DNA의 특정 영역에서의 메틸화 패턴의 분석은 중황산염 매핑이라고 불리는 방법을 통해 달성될 수 있다.메틸화 시토신 잔기는 처리에 의해 변화하지 않지만, 비메틸화 시토신 잔기는 우라실로 변화한다.차이는 DNA 염기서열 분석 또는 파이로시퀀싱(Biotage) 또는 MassArray(Sequenom)와 같이 SNP를 정량화하기 위해 개발된 방법으로 분석되며 CG 디뉴클레오타이드에서 C/T의 상대적 양을 측정한다.비정상적인 메틸화 패턴은 [3]발암에 관여하는 것으로 생각된다.
히스톤 아세틸화는 또한 전사의 중요한 과정이다.CREB 결합 단백질과 같은 히스톤 아세틸전달효소(HAT)도 히스톤 복합체로부터 DNA를 분리하여 전사가 진행되도록 한다.종종, DNA 메틸화와 히스톤 탈아세틸화는 유전자 사일링에 함께 작용한다.이 둘의 조합은 DNA가 더 조밀하게 채워져 유전자 [citation needed]발현을 낮추기 위한 신호인 것으로 보인다.
전사 규제

따라서 전사의 조절은 언제 전사가 일어나며 RNA가 얼마나 생성되는지를 제어합니다.RNA 중합효소에 의한 유전자 전사는 몇 가지 메커니즘에 의해 조절될 수 있다.특이성 인자는 특정 프로모터 또는 프로모터 세트에 대한 RNA 중합효소의 특이성을 변화시켜, 원핵생물 전사에 사용되는 시그마 인자(sigma factors)와 결합할 가능성을 다소 낮춘다.억제제는 오퍼레이터에 결합하여 프로모터 영역에 가깝거나 겹치는 DNA 가닥의 염기서열을 코드화하고, 가닥을 따라 RNA 중합효소의 진행을 방해하여 유전자의 발현을 방해합니다.오른쪽 이미지는 열상 오퍼론의 억제기에 의한 조절을 보여줍니다.일반적인 전사 인자는 단백질 코드 배열의 시작에서 RNA 중합효소를 위치시킨 다음 중합효소를 방출하여 mRNA를 전사합니다.활성제는 RNA 중합효소와 특정 프로모터 간의 상호작용을 강화하여 유전자의 발현을 촉진한다.활성제는 프로모터에 대한 RNA 중합효소의 흡인력을 증가시키거나 RNA 중합효소의 서브유닛과의 상호작용을 통해 또는 DNA의 구조를 변경함으로써 간접적으로 이를 수행한다.증강제는 특정 프로모터를 시작 복합체로 가져오는 DNA를 루프시키기 위해 활성제에 의해 결합된 DNA 나선 부위이다.강화제는 원핵생물보다 진핵생물에서 훨씬 더 흔하며,[4] 몇 가지 예만 존재합니다.소음기는 특정 전사 인자에 의해 결합되었을 때 유전자의 발현을 침묵시킬 수 있는 DNA 배열의 영역입니다.
RNA에 의한 조절
RNA는 마이크로RNA(miRNA), 안티센스-RNA 또는 긴 비코드 RNA(lncRNA)와 같은 유전자 활성의 중요한 조절제가 될 수 있다.LncRNA는 세포 내 위치 및 기능을 지정한다는 점에서 mRNA와 다릅니다.그것들은 핵과 염색질에 있는 것으로 처음 발견되었고, 현재는 국소화와 기능이 매우 다양합니다.일부는 단백질과 상호작용하는 염색질에 여전히 존재한다.이 lncRNA는 궁극적으로 파킨슨, 헌팅턴, 알츠하이머와 같은 신경 질환의 유전자 발현에 영향을 미치지만, PNCTR(피리미딘이 풍부한 비암호화 전사체)와 같은 다른 것들은 폐암에서 역할을 한다.질병에서의 그들의 역할을 고려할 때, lncRNA는 잠재적인 바이오마커이며 약물이나 유전자 치료의 유용한 표적이 될 수 있다. 비록 아직 lncRNA를 지연시키는 승인된 약은 없다.인간 게놈에는 lncRNA의 수가 제대로 정의되지 않은 채로 남아 있지만, 일부 추정치는 16,000에서 100,000개의 lnc [5]유전자에 이른다.
후생유전자조절
후생유전학은 DNA나 RNA 서열을 바꾸지 않는 유전자의 변형을 말한다.후생유전학적 변형 또한 유전자 발현에 영향을 미치는 핵심 요인이다.그것들은 게놈 DNA와 히스톤에서 발생하며 그들의 화학적 변형은 보다 효율적인 방법으로 유전자 발현을 조절한다.포유류의 세포에는 DNA(보통 메틸화)의 여러 가지 변형과 100개 이상의 RNA의 변형 등이 있습니다."이러한 변형은 DNA에 대한 단백질 결합의 변화와 RNA의 안정성과 번역 [6]효율의 변화를 초래한다.
인간 생물학 및 질병의 특수한 경우
암의 전사 조절
척추동물에서 대부분의 유전자 촉진제는 다수의 CpG [7]부위가 있는 CpG 섬을 포함한다.유전자의 프로모터 CpG 부위의 많은 부분이 메틸화되면 유전자는 [8]침묵하게 된다.대장암은 일반적으로 3~6명의 운전자 돌연변이와 33~66명의 히치하이커 또는 승객 [9]돌연변이가 있다.그러나 전사 소음은 암으로의 진행을 유발하는데 돌연변이보다 더 중요할 수 있다.예를 들어 대장암의 경우 약 600~800개의 유전자가 CpG 섬 메틸화에 의해 전사적으로 침묵된다(암에서의 전사 조절 참조).암에서의 전사 억제는 마이크로RNA의 [10]발현 변화와 같은 다른 후생유전 메커니즘에 의해서도 발생할 수 있다.유방암에서 BRCA1의 전사 억제는 BRCA1 프로모터의 과메틸화보다 과잉 발현된 마이크로RNA-182에 의해 더 자주 발생할 수 있다(유방암과 난소암에서 BRCA1의 저발현 참조).
중독에서의 전사 규제
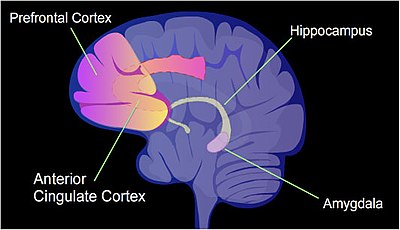
중독의 주요 특징 중 하나는 지속성이다.지속적인 행동 변화는 뇌의 [11]특정 영역 내에서 유전자 발현에 영향을 미치는 후생유전학적 변화에서 비롯되는 장기적인 변화 때문인 것으로 보인다.약물 남용은 뇌에서 세 가지 유형의 후생유전적 변화를 일으킨다.(1) 히스톤 아세틸화 및 히스톤 메틸화, (2) CpG 부위의 DNA 메틸화, (3) 마이크로RNA의 [11][12]후생유전학적 하향조절 또는 상향조절이다(자세한 내용은 코카인 중독 후생유전학 참조).
생쥐의 만성 니코틴 섭취는 히스톤의 아세틸화를 통해 유전자 발현에 대한 뇌세포 후생적 제어를 변화시킨다.이것은 [13]중독에 중요한 단백질 FosB의 뇌에서 발현을 증가시킨다.담배 중독은 또한 절대 흡연하지 않는 사람, 현재 흡연자, 그리고 최장 30년 [14]동안 담배를 끊은 사람들을 포함하여 약 16,000명의 사람들을 대상으로 연구되었다.혈구에서는 18,000개 이상의 CpG 사이트(게놈의 약 450,000개 분석된 CpG 사이트 중)가 현재 흡연자들 사이에서 메틸화를 자주 변경했다.이러한 CpG 부위는 7,000개가 넘는 유전자, 즉 알려진 인간 유전자의 약 1/3에서 발생했다.차등적으로 메틸화된 CpG 사이트의 대부분은 금연 후 5년 이내에 절대 흡연자 수준으로 되돌아갔다.그러나, 942개의 유전자 중 2,568개의 CpGs는 이전 흡연자와 전혀 흡연하지 않은 사람의 차이에서 메틸화 상태를 유지했다.이러한 후생유전학적 변화는 유전자 발현에 영향을 미칠 수 있는 "분자 흉터"[12]로 볼 수 있다.
설치류 모델에서는 코카인,[15] 필로폰,[16][17] 알코올[18],[19] 담배 연기 제품을 포함한 약물 남용은 모두 뇌의 DNA 손상을 일으킨다.DNA 손상 복구 중 일부 개별 복구 이벤트는 손상 부위에서 DNA의 메틸화 및/또는 히스톤의 아세틸화 또는 메틸화를 변경할 수 있으며, 따라서 염색질에 [20]후생유전적 흉터를 남기는 데 기여할 수 있다.
이러한 후생유전적 흉터는 중독에서 발견되는 지속적인 후생유전적 변화에 기여할 수 있다.
학습 및 기억에서의 전사 조절
포유동물에서 DNA에서 시토신의 메틸화는 주요 조절 매개체이다.메틸화 사이토신은 주로 디뉴클레오티드 배열에서 발생하며, 시토신은 CpG 부위인 구아닌에 이어진다.인간 게놈의 총 CpG 사이트 수는 약 2800만 [21]개입니다.그리고 일반적으로 모든 CpG 부위의 약 70%가 메틸화 시토신을 [22]가지고 있다.
쥐에게 있어서, 고통스런 학습 경험, 상황별 두려움 조절은 단 한 번의 훈련 [23]후에 평생의 두려운 기억을 야기할 수 있습니다.시토신 메틸화는 짧은 공포 조절 경험을 [24]거친 쥐의 해마 뉴런 DNA 내 모든 유전자의 약 9.17%의 프로모터 영역에서 변화한다.해마는 새로운 기억이 처음 저장되는 곳이다.
유전자의 프로모터 영역에서의 CpGs의 메틸화는 전사를[25] 억제하고 유전자 체내의 CpGs의 메틸화는 [26]발현을 증가시킨다.TET 효소는 메틸화된 세포신의 탈메틸화에 중심적인 역할을 한다.TET 효소 활성에 의한 유전자 프로모터의 CpGs의 탈메틸화는 유전자의 [27]전사를 증가시킨다.
쥐에게 상황별 공포 조절을 적용하면,[24] 해마의 조절 후 1시간과 24시간 모두 5,000개 이상의 차등 메틸화 영역(DMRs, 각각 500개의 뉴클레오티드)이 쥐의 신경 게놈에서 발생합니다.이것은 약 500개의 유전자가 상향 조절되고(종종 프로모터 영역의 CpG 부위의 탈메틸화로 인해), 약 1,000개의 유전자가 하향 조절되는(종종 프로모터 영역의 CpG 부위에서 새롭게 형성된 5-메틸시토신 때문에) 원인이 된다.뉴런 내의 유도 및 억제된 유전자의 패턴은 쥐 [24]뇌의 해마에서 이 훈련 사건의 첫 번째 일시적인 기억을 형성하기 위한 분자 기반을 제공하는 것으로 보입니다.
전사 후 규정
DNA가 전사되고 mRNA가 형성된 후, mRNA가 단백질로 변환되는 양에 대한 일종의 조절이 있어야 한다.세포는 캡핑, 스플라이싱, 폴리(A) 테일의 추가, 배열별 핵 수출 속도, 그리고 여러 맥락에서 RNA 전사체의 격리를 조절함으로써 이를 수행한다.이러한 과정은 진핵생물에서 일어나지만 원핵생물에서는 일어나지 않는다.이 변조는 단백질 또는 전사체의 결과이며, 이는 차례로 조절되고 특정 배열에 대한 친화력을 가질 수 있습니다.
3개의 주요 미번역 영역과 마이크로RNA
메신저 RNA(mRNA)의 세 가지 주요 미번역 영역(3'-UTR)은 종종 전사 후 유전자 [28]발현에 영향을 미치는 조절 서열을 포함한다.이러한 3'-UTR은 종종 조절 단백질뿐만 아니라 마이크로RNA(miRNA)에 대한 결합 부위를 모두 포함한다.miRNA는 3'-UTR 내의 특정 부위에 결합함으로써 번역을 저해하거나 전사체의 분해를 직접 유발함으로써 다양한 mRNA의 유전자 발현을 감소시킬 수 있다.또한 3'-UTR은 mRNA의 발현을 억제하는 억제 단백질과 결합하는 소음기 영역을 가질 수 있다.
3'-UTR에는 종종 miRNA 응답 요소(MRE)가 포함되어 있습니다.MRE는 miRNA가 바인드되는 시퀀스입니다.이것들은 3'-UTR 내에서 널리 사용되는 모티브이다.3'-UTR(소음기 영역 포함) 내의 모든 규제 모티브 중 MRE는 모티브의 약 절반을 차지한다.
2014년 현재, miRNA 염기서열 및 주석의 아카이브인 miRBase [29]웹사이트에는 233종의 생물종이 28,645개의 항목을 열거했다.이 중 1,881개의 miRNA가 인간의 miRNA 위치에 있었으며, miRNA는 평균 약 400개의 표적 mRNA(수백개의 [30]유전자에 영향을 미치는 발현)를 가질 것으로 예측되었다.Freidman et al.[30]은 인간 mRNA 3'-UTR 내의 45,000개 이상의 miRNA 표적 부위가 배경 수준 이상으로 보존되고 인간 단백질 코드 유전자의 60% 이상이 miRNA와의 쌍을 유지하기 위해 선택적 압력을 받고 있다고 추정했다.
직접 실험에 따르면 단일 miRNA가 수백 [31]개의 고유한 mRNA의 안정성을 감소시킬 수 있습니다.다른 실험들은 단일 miRNA가 수백 개의 단백질 생성을 억제할 수 있지만, 이러한 억제 효과는 종종 비교적 경미하다는 것을 보여준다.[32][33]
miRNA 유전자 발현 조절 장애의 영향은 [34]암에서 중요한 것으로 보인다.예를 들어, 위장암의 경우, 2015년 논문은 9개의 miRNA가 후생적으로 변화하고 DNA 복구 [35]효소를 하향 조절하는 데 효과적인 것으로 식별했다.
miRNA 유전자 발현 조절 장애의 영향은 정신 분열증, 양극성 장애, 주요 우울증 장애, 파킨슨병, 알츠하이머병, 자폐 스펙트럼 [36][37][38]장애와 같은 신경 정신 질환에서도 중요한 것으로 보인다.
번역 규제
mRNA의 변환은 많은 메커니즘에 의해 제어될 수도 있습니다.대부분은 개시 레벨입니다.mRNA 2차 구조, 안티센스 RNA 결합 또는 단백질 결합에 의해 작은 리보솜 서브유닛의 신병을 조정할 수 있다.원핵생물 및 진핵생물에는 다수의 RNA 결합 단백질이 존재하며, 이는 종종 전사의 2차 구조에 의해 목표 배열로 향하며, 이는 온도 또는 리간드(aptamer)의 존재와 같은 특정 조건에 따라 변할 수 있다.몇몇 성전사는 리보자임으로 작용하고 그들의 발현을 스스로 조절한다.
유전자 조절의 예
- 효소 유도는 분자(예: 약물)가 효소의 발현을 유도(즉, 시작 또는 강화)하는 과정이다.
- 초파리 드로소필라 멜라노가스터의 열충격 단백질 유도.
- Lac 오퍼론은 유전자 발현이 어떻게 조절될 수 있는지에 대한 흥미로운 예이다.
- 바이러스는 몇 개의 유전자만 가지고 있음에도 불구하고 항종단자(lambda page) 또는 스플라이싱 변조제(HIV)에 의해 조절되는 공선 시스템을 사용하여 일반적으로 초기 및 후기에 유전자 발현을 조절하는 메커니즘을 가지고 있습니다.
- Gal4는 GAL1, GAL7, GAL10의 발현을 제어하는 전사 활성제이다(이 모든 코드는 효모에서 갈락토스의 신진대사에 대한 코드).GAL4/UAS 시스템은 유전자 [39]발현을 연구하기 위해 다양한 Phyla에 걸쳐 다양한 유기체에서 사용되어 왔다.
발달생물학
많은 연구된 규제 시스템은 발달 생물학에서 유래한다.예를 들어 다음과 같습니다.
- Hox 유전자 클러스터의 중첩된 전후 패턴과 함께 공진성
- 손의 패턴 생성(자릿수 - 자릿수 간): 사지의 편광 활동 영역에서 음파 고슴도치(분비 유도 인자)의 구배를 만들어 사지에서 분비되는 BMP를 억제하는 활성 Gli3의 구배를 만들어 결과적으로 교대로 활동 패턴을 형성한다.이 반응-반응-반응 계통의
- 소미토제네시스(Somitogenesis)는 균일한 조직(소미트 전 중배엽)에서 세그먼트(소미트)를 생성하는 것입니다.그것들은 전방에서 후방으로 순차적으로 형성된다.이는 양막에서 FGF + 노치 및 역위상 [40]Wnt로 구성된 진동 패턴(분할 클럭)과 결합되어 전방(파장 전방)의 레티노산과 후방(Wnt)의 두 가지 상반된 구배를 통해 달성될 수 있다.
- 드로소필라의 소마에서의 성결정은 성염색체 부호화 유전자에 대한 상염색체 유전자의 비율을 감지하는 것을 필요로 하며, 이는 여성에서 성별이 없는 스플라이싱 인자를 생산하여 더블섹스의 [41]여성 아이소폼을 발생시킨다.
회로
업 레귤레이션 및 다운 레귤레이션
상향 조절은 신호(세포 내부 또는 외부에서 발생)에 의해 유발되는 세포 내에서 일어나는 과정으로, 하나 이상의 유전자의 발현을 증가시키고 그 결과 유전자에 의해 암호화된 단백질을 증가시킨다.반대로 다운조절은 유전자 감소와 그에 따른 단백질 발현을 초래하는 과정이다.
- 업 레귤레이션은 예를 들어 세포가 어떤 종류의 수용체에 결핍되어 있을 때 발생합니다.이 경우, 보다 많은 수용체 단백질이 합성되어 세포막으로 운반되어 세포의 감수성이 정상으로 돌아와 항상성을 재정립한다.
- 다운조절은 예를 들어 세포가 신경전달물질, 호르몬 또는 약물에 의해 장기간 과잉자극되어 세포를 보호하기 위해 수용체 단백질의 발현을 감소시킬 때 발생한다(빈맥증 참조).
유도 가능한 시스템과 억제 가능한 시스템
유전자 조절은 각 시스템의 반응으로 요약할 수 있다.
- 유도 시스템 - 유도 시스템은 유전자 발현을 허용하는 어떤 분자가 존재하지 않는 한 꺼집니다.그 분자는 "발현을 유도한다"고 한다.이것이 일어나는 방법은 원핵세포와 진핵세포 사이의 차이뿐만 아니라 제어 메커니즘에 달려있다.
- 억제할 수 있는 시스템 - 억제할 수 있는 시스템은 유전자 발현을 억제하는 분자 (코어프레서라고 불리는)의 존재를 제외하고 작동한다.그 분자는 "발현을 억제한다"고 한다.이것이 일어나는 방법은 원핵세포와 진핵세포 사이의 차이뿐만 아니라 제어 메커니즘에 달려있다.
GAL4/UAS 시스템은 유도 가능한 시스템과 억제 가능한 시스템 모두의 예입니다.Gal4는 업스트림 활성화 시퀀스(UAS)를 결합하여 GAL1/GAL7/GAL10 카세트의 전사를 활성화합니다.한편 포도당의 존재에 대한 MIG1 응답은 GAL4를 억제할 수 있으므로 GAL1/GAL7/GAL10 [42]카세트의 발현을 정지시킬 수 있다.
이론 회로
- 억제제/유도제: 센서의 활성화는 유전자의 발현 변화를 일으킨다.
- 부정적인 피드백: 유전자 생산물은 그 자신의 생산을 직간접적으로 하향 조절하고, 이것은 결과를 가져올 수 있다.
- 인자에 대해 전사 수준을 일정하게/비례적으로 유지
- 양의 피드백 루프와 결합했을 때의 도망 반응 억제
- mRNA와 단백질의 반감기가 짧기 때문에 전사 및 번역의 시간 지연을 이용하여 발진기를 만드는
- 긍정적인 피드백: 유전자 생산물은 그 자신의 생산을 직간접적으로 상향 조정하고, 이것은 결과를 가져올 수 있다.
- 신호 증폭
- 두 유전자가 서로를 억제하고 둘 다 긍정적인 피드백을 가질 때 쌍안정 스위치
- 패턴 생성
연구 방법
일반적으로, 분화 발현을 조사하는 대부분의 실험은 어떤 유전자가 얼마나 변했는지 결정하기 위해 정상 상태 수준이라고 불리는 RNA의 전체 세포 추출물을 사용했습니다.그러나 이러한 정보는 규제가 발생한 위치에 대한 정보를 제공하지 않으며 상충되는 규제 과정을 가릴 수 있다(전사 후 규제 참조). 그러나 여전히 가장 일반적으로 분석된다(정량 PCR 및 DNA 마이크로 어레이).
유전자 발현을 연구할 때 여러 단계를 살펴보는 방법이 있다.진핵생물에서는 다음이 포함됩니다.
- 이 지역의 국소 크로마틴 환경은 RNA 중합효소 II, 히스톤 3 변형, 트리토락스 그룹 단백질, 폴리콤 그룹 단백질 또는 좋은 항체를 사용할 수 있는 다른 DNA 결합 요소를 끌어내림으로써 ChIP-칩 분석에 의해 결정될 수 있다.
- 인식적 상호작용은 합성 유전자 배열 분석에 의해 조사될 수 있다.
- 전사 후 조절로 인해 전사 속도와 총 RNA 수준이 크게 다릅니다.전사율을 측정하기 위해 방사능 [43]대신 티올 라벨링을 사용하여 새로운 고처리량 방법을 개발하고 있다.
- 핵에서 중합된 RNA의 5%만이 빠져나가며,[44] 인트론, 중단 생성물 및 비감각 전사물이 분해된다.따라서 핵과 세포질 수준의 차이는 완만한 [45]용해에 의해 두 분율을 분리함으로써 확인할 수 있다.
- 대체 스플라이싱은 스플라이싱 어레이 또는 타일링 어레이를 사용하여 분석할 수 있습니다(DNA 마이크로 어레이 참조).
- 모든 생체내 RNA는 RNPs로 복합화된다.특정 단백질에 결합된 전사물의 양은 RIP-Chip으로 분석할 수도 있습니다.예를 들어, DCP2는 격리된 단백질의 징후를 제공할 것이다; 리보솜 결합은 전사에서 활성 전사를 제공하고 지시할 것이다(다소 다분화라고 불리는 더 오래된 방법이 일부 실험실에서 여전히 인기 있다).
- 단백질 수준은 질량분석법으로 분석할 수 있으며, 마이크로 어레이 데이터는 절대적이지 않고 상대적이므로 정량 PCR 데이터에만 비교할 수 있다.
- RNA 및 단백질 분해율은 각각 전사억제제(액티노마이신D 또는 α-아마니틴) 또는 번역억제제(시클로헥시미드)에 의해 측정된다.
「 」를 참조해 주세요.
- 인공전사인자(전사인자 단백질을 모방한 작은 분자)
- 셀룰러 모델
- 보존된 비코드 DNA 배열
- 인핸서(유전자)
- 유전자 구조
- 시공간 유전자 발현
주 및 참고 자료
- ^ "Can genes be turned on and off in cells?". Genetics Home Reference.
- ^ Bell JT, Pai AA, Pickrell JK, Gaffney DJ, Pique-Regi R, Degner JF, et al. (2011). "DNA methylation patterns associate with genetic and gene expression variation in HapMap cell lines". Genome Biology. 12 (1): R10. doi:10.1186/gb-2011-12-1-r10. PMC 3091299. PMID 21251332.
- ^ Vertino PM, Spillare EA, Harris CC, Baylin SB (April 1993). "Altered chromosomal methylation patterns accompany oncogene-induced transformation of human bronchial epithelial cells" (PDF). Cancer Research. 53 (7): 1684–9. PMID 8453642.
- ^ Austin S, Dixon R (June 1992). "The prokaryotic enhancer binding protein NTRC has an ATPase activity which is phosphorylation and DNA dependent". The EMBO Journal. 11 (6): 2219–28. doi:10.1002/j.1460-2075.1992.tb05281.x. PMC 556689. PMID 1534752.
- ^ Statello L, Guo CJ, Chen LL, Huarte M (February 2021). "Gene regulation by long non-coding RNAs and its biological functions". Nature Reviews. Molecular Cell Biology. 22 (2): 96–118. doi:10.1038/s41580-020-00315-9. ISSN 1471-0072. PMC 7754182. PMID 33353982.
- ^ Kan RL, Chen J, Sallam T (July 2021). "Crosstalk between epitranscriptomic and epigenetic mechanisms in gene regulation". Trends in Genetics. 38 (2): 182–193. doi:10.1016/j.tig.2021.06.014. PMID 34294427. S2CID 236200223.
- ^ Saxonov S, Berg P, Brutlag DL (January 2006). "A genome-wide analysis of CpG dinucleotides in the human genome distinguishes two distinct classes of promoters". Proceedings of the National Academy of Sciences of the United States of America. 103 (5): 1412–7. Bibcode:2006PNAS..103.1412S. doi:10.1073/pnas.0510310103. PMC 1345710. PMID 16432200.
- ^ Bird A (January 2002). "DNA methylation patterns and epigenetic memory". Genes & Development. 16 (1): 6–21. doi:10.1101/gad.947102. PMID 11782440.
- ^ Vogelstein B, Papadopoulos N, Velculescu VE, Zhou S, Diaz LA, Kinzler KW (March 2013). "Cancer genome landscapes". Science. 339 (6127): 1546–58. Bibcode:2013Sci...339.1546V. doi:10.1126/science.1235122. PMC 3749880. PMID 23539594.
- ^ Tessitore A, Cicciarelli G, Del Vecchio F, Gaggiano A, Verzella D, Fischietti M, et al. (2014). "MicroRNAs in the DNA Damage/Repair Network and Cancer". International Journal of Genomics. 2014: 820248. doi:10.1155/2014/820248. PMC 3926391. PMID 24616890.
- ^ a b Nestler EJ (January 2014). "Epigenetic mechanisms of drug addiction". Neuropharmacology. 76 Pt B: 259–68. doi:10.1016/j.neuropharm.2013.04.004. PMC 3766384. PMID 23643695.
- ^ a b Robison AJ, Nestler EJ (October 2011). "Transcriptional and epigenetic mechanisms of addiction". Nature Reviews. Neuroscience. 12 (11): 623–37. doi:10.1038/nrn3111. PMC 3272277. PMID 21989194.
- ^ Levine A, Huang Y, Drisaldi B, Griffin EA, Pollak DD, Xu S, et al. (November 2011). "Molecular mechanism for a gateway drug: epigenetic changes initiated by nicotine prime gene expression by cocaine". Science Translational Medicine. 3 (107): 107ra109. doi:10.1126/scitranslmed.3003062. PMC 4042673. PMID 22049069.
- ^ Joehanes R, Just AC, Marioni RE, Pilling LC, Reynolds LM, Mandaviya PR, et al. (October 2016). "Epigenetic Signatures of Cigarette Smoking". Circulation: Cardiovascular Genetics. 9 (5): 436–447. doi:10.1161/CIRCGENETICS.116.001506. PMC 5267325. PMID 27651444.
- ^ de Souza MF, Gonçales TA, Steinmetz A, Moura DJ, Saffi J, Gomez R, Barros HM (April 2014). "Cocaine induces DNA damage in distinct brain areas of female rats under different hormonal conditions". Clinical and Experimental Pharmacology & Physiology. 41 (4): 265–9. doi:10.1111/1440-1681.12218. PMID 24552452. S2CID 20849951.
- ^ Johnson Z, Venters J, Guarraci FA, Zewail-Foote M (June 2015). "Methamphetamine induces DNA damage in specific regions of the female rat brain". Clinical and Experimental Pharmacology & Physiology. 42 (6): 570–5. doi:10.1111/1440-1681.12404. PMID 25867833. S2CID 24182756.
- ^ Tokunaga I, Ishigami A, Kubo S, Gotohda T, Kitamura O (August 2008). "The peroxidative DNA damage and apoptosis in methamphetamine-treated rat brain". The Journal of Medical Investigation. 55 (3–4): 241–5. doi:10.2152/jmi.55.241. PMID 18797138.
- ^ Rulten SL, Hodder E, Ripley TL, Stephens DN, Mayne LV (July 2008). "Alcohol induces DNA damage and the Fanconi anemia D2 protein implicating FANCD2 in the DNA damage response pathways in brain". Alcoholism, Clinical and Experimental Research. 32 (7): 1186–96. doi:10.1111/j.1530-0277.2008.00673.x. PMID 18482162.
- ^ Adhami N, Chen Y, Martins-Green M (October 2017). "Biomarkers of disease can be detected in mice as early as 4 weeks after initiation of exposure to third-hand smoke levels equivalent to those found in homes of smokers". Clinical Science. 131 (19): 2409–2426. doi:10.1042/CS20171053. PMID 28912356.
- ^ Dabin J, Fortuny A, Polo SE (June 2016). "Epigenome Maintenance in Response to DNA Damage". Molecular Cell. 62 (5): 712–27. doi:10.1016/j.molcel.2016.04.006. PMC 5476208. PMID 27259203.
- ^ Lövkvist C, Dodd IB, Sneppen K, Haerter JO (June 2016). "DNA methylation in human epigenomes depends on local topology of CpG sites". Nucleic Acids Research. 44 (11): 5123–32. doi:10.1093/nar/gkw124. PMC 4914085. PMID 26932361.
- ^ Jabbari K, Bernardi G (May 2004). "Cytosine methylation and CpG, TpG (CpA) and TpA frequencies". Gene. 333: 143–9. doi:10.1016/j.gene.2004.02.043. PMID 15177689.
- ^ Kim JJ, Jung MW (2006). "Neural circuits and mechanisms involved in Pavlovian fear conditioning: a critical review". Neuroscience and Biobehavioral Reviews. 30 (2): 188–202. doi:10.1016/j.neubiorev.2005.06.005. PMC 4342048. PMID 16120461.
- ^ a b c Duke CG, Kennedy AJ, Gavin CF, Day JJ, Sweatt JD (July 2017). "Experience-dependent epigenomic reorganization in the hippocampus". Learning & Memory. 24 (7): 278–288. doi:10.1101/lm.045112.117. PMC 5473107. PMID 28620075.
- ^ Weber M, Hellmann I, Stadler MB, Ramos L, Pääbo S, Rebhan M, Schübeler D (April 2007). "Distribution, silencing potential and evolutionary impact of promoter DNA methylation in the human genome". Nat. Genet. 39 (4): 457–66. doi:10.1038/ng1990. PMID 17334365. S2CID 22446734.
- ^ Yang X, Han H, De Carvalho DD, Lay FD, Jones PA, Liang G (October 2014). "Gene body methylation can alter gene expression and is a therapeutic target in cancer". Cancer Cell. 26 (4): 577–90. doi:10.1016/j.ccr.2014.07.028. PMC 4224113. PMID 25263941.
- ^ Maeder ML, Angstman JF, Richardson ME, Linder SJ, Cascio VM, Tsai SQ, Ho QH, Sander JD, Reyon D, Bernstein BE, Costello JF, Wilkinson MF, Joung JK (December 2013). "Targeted DNA demethylation and activation of endogenous genes using programmable TALE-TET1 fusion proteins". Nat. Biotechnol. 31 (12): 1137–42. doi:10.1038/nbt.2726. PMC 3858462. PMID 24108092.
- ^ Ogorodnikov A, Kargapolova Y, Danckwardt S (June 2016). "Processing and transcriptome expansion at the mRNA 3' end in health and disease: finding the right end". Pflügers Archiv. 468 (6): 993–1012. doi:10.1007/s00424-016-1828-3. PMC 4893057. PMID 27220521.
- ^ miRBase.org
- ^ a b Friedman RC, Farh KK, Burge CB, Bartel DP (January 2009). "Most mammalian mRNAs are conserved targets of microRNAs". Genome Research. 19 (1): 92–105. doi:10.1101/gr.082701.108. PMC 2612969. PMID 18955434.
- ^ Lim LP, Lau NC, Garrett-Engele P, Grimson A, Schelter JM, Castle J, et al. (February 2005). "Microarray analysis shows that some microRNAs downregulate large numbers of target mRNAs". Nature. 433 (7027): 769–73. Bibcode:2005Natur.433..769L. doi:10.1038/nature03315. PMID 15685193. S2CID 4430576.
- ^ Selbach M, Schwanhäusser B, Thierfelder N, Fang Z, Khanin R, Rajewsky N (September 2008). "Widespread changes in protein synthesis induced by microRNAs". Nature. 455 (7209): 58–63. Bibcode:2008Natur.455...58S. doi:10.1038/nature07228. PMID 18668040. S2CID 4429008.
- ^ Baek D, Villén J, Shin C, Camargo FD, Gygi SP, Bartel DP (September 2008). "The impact of microRNAs on protein output". Nature. 455 (7209): 64–71. Bibcode:2008Natur.455...64B. doi:10.1038/nature07242. PMC 2745094. PMID 18668037.
- ^ Palmero EI, de Campos SG, Campos M, de Souza NC, Guerreiro ID, Carvalho AL, Marques MM (July 2011). "Mechanisms and role of microRNA deregulation in cancer onset and progression". Genetics and Molecular Biology. 34 (3): 363–70. doi:10.1590/S1415-47572011000300001. PMC 3168173. PMID 21931505.
- ^ Bernstein C, Bernstein H (May 2015). "Epigenetic reduction of DNA repair in progression to gastrointestinal cancer". World Journal of Gastrointestinal Oncology. 7 (5): 30–46. doi:10.4251/wjgo.v7.i5.30. PMC 4434036. PMID 25987950.
- ^ Maffioletti E, Tardito D, Gennarelli M, Bocchio-Chiavetto L (2014). "Micro spies from the brain to the periphery: new clues from studies on microRNAs in neuropsychiatric disorders". Frontiers in Cellular Neuroscience. 8: 75. doi:10.3389/fncel.2014.00075. PMC 3949217. PMID 24653674.
- ^ Mellios N, Sur M (2012). "The Emerging Role of microRNAs in Schizophrenia and Autism Spectrum Disorders". Frontiers in Psychiatry. 3: 39. doi:10.3389/fpsyt.2012.00039. PMC 3336189. PMID 22539927.
- ^ Geaghan M, Cairns MJ (August 2015). "MicroRNA and Posttranscriptional Dysregulation in Psychiatry". Biological Psychiatry. 78 (4): 231–9. doi:10.1016/j.biopsych.2014.12.009. PMID 25636176.
- ^ Barnett JA (July 2004). "A history of research on yeasts 7: enzymic adaptation and regulation". Yeast. 21 (9): 703–46. doi:10.1002/yea.1113. PMID 15282797. S2CID 36606279.
- ^ Dequéant ML, Pourquié O (May 2008). "Segmental patterning of the vertebrate embryonic axis". Nature Reviews. Genetics. 9 (5): 370–82. doi:10.1038/nrg2320. PMID 18414404. S2CID 2526914.
- ^ 길버트 SF(2003).발달생물학, 제7판, 선덜랜드, 매스: 시나우어 어소시에이트, 65~6.ISBN 0-87893-258-5.
- ^ Nehlin JO, Carlberg M, Ronne H (November 1991). "Control of yeast GAL genes by MIG1 repressor: a transcriptional cascade in the glucose response". The EMBO Journal. 10 (11): 3373–7. doi:10.1002/j.1460-2075.1991.tb04901.x. PMC 453065. PMID 1915298.
- ^ Cheadle C, Fan J, Cho-Chung YS, Werner T, Ray J, Do L, et al. (May 2005). "Control of gene expression during T cell activation: alternate regulation of mRNA transcription and mRNA stability". BMC Genomics. 6: 75. doi:10.1186/1471-2164-6-75. PMC 1156890. PMID 15907206.
- ^ Jackson DA, Pombo A, Iborra F (February 2000). "The balance sheet for transcription: an analysis of nuclear RNA metabolism in mammalian cells". FASEB Journal. 14 (2): 242–54. doi:10.1096/fasebj.14.2.242. PMID 10657981. S2CID 23518786.
- ^ Schwanekamp JA, Sartor MA, Karyala S, Halbleib D, Medvedovic M, Tomlinson CR (2006). "Genome-wide analyses show that nuclear and cytoplasmic RNA levels are differentially affected by dioxin". Biochimica et Biophysica Acta (BBA) - Gene Structure and Expression. 1759 (8–9): 388–402. doi:10.1016/j.bbaexp.2006.07.005. PMID 16962184.
참고 문헌
- Latchman, David S. (2005). Gene regulation: a eukaryotic perspective. Psychology Press. ISBN 978-0-415-36510-9.
외부 링크
- 플랜트 전사 계수 데이터베이스 및 플랜트 전사 규제 데이터 및 분석 플랫폼
- 미국 국립의학도서관의 유전자 발현 규제(MeSH)
- ChIPBase ChIP-seq 데이터에서 비코딩 RNA 및 단백질 코딩 유전자의 전사 조절 네트워크를 해독하기 위한 개방형 데이터베이스.