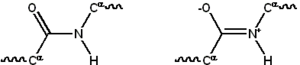단백질
Protein단백질은 하나 이상의 아미노산 잔류물의 긴 사슬을 구성하는 큰 생체 분자와 고분자입니다.단백질은 촉매 대사 반응, DNA 복제, 자극에 대한 반응, 세포와 유기체에 구조를 제공하고 한 위치에서 다른 위치로 분자를 운반하는 것을 포함하여 유기체 내에서 광범위한 기능을 수행합니다.단백질은 주로 유전자의 뉴클레오티드 배열에 의해 결정되는 아미노산 배열에서 서로 다르며, 일반적으로 단백질은 활동을 결정하는 특정 3D 구조로 접히는 결과를 낳는다.
아미노산 잔류물의 선형 사슬을 폴리펩타이드라고 한다.단백질은 적어도 하나의 긴 폴리펩타이드를 포함한다.20~30개 미만의 잔류물을 포함하는 짧은 폴리펩타이드는 단백질로 간주되지 않으며 일반적으로 펩타이드 또는 때로는 올리고펩타이드로 불린다.개별 아미노산 잔기는 펩타이드 결합과 인접한 아미노산 잔기에 의해 결합된다.단백질 내 아미노산 잔류물의 배열은 유전자 코드에 암호화되어 있는 유전자의 배열에 의해 정의된다.일반적으로, 유전 코드는 20개의 표준 아미노산을 지정하지만, 특정 유기체에서 유전 코드는 셀레노시스테인과 특정 고세균에서 필로리신을 포함할 수 있습니다.합성 직후 또는 합성 중에 단백질의 잔류물은 종종 번역 후 변성에 의해 화학적으로 변형되며, 이는 물리적 및 화학적 특성, 접힘, 안정성, 활성, 그리고 궁극적으로 단백질의 기능을 변화시킨다.일부 단백질은 보철물 그룹 또는 보조 인자라고 할 수 있는 비펩타이드 그룹이 붙어 있습니다.단백질은 또한 특정한 기능을 달성하기 위해 함께 일할 수 있고, 그들은 종종 안정적인 단백질 복합체를 형성하기 위해 결합합니다.
일단 단백질이 형성되면, 단백질은 특정 기간 동안만 존재하며, 단백질 교체 과정을 통해 세포의 기계에 의해 분해되고 재활용된다.단백질의 수명은 반감기로 측정되며 광범위한 범위를 차지한다.그들은 포유류의 세포에서 평균 수명은 1-2일이고 몇 분 또는 몇 년 동안 존재할 수 있습니다.비정상적이거나 잘못 접힌 단백질은 파괴 대상이 되거나 불안정하기 때문에 더 빠르게 분해된다.
다당류와 핵산과 같은 다른 생물학적 고분자처럼, 단백질은 유기체의 필수적인 부분이고 세포 내의 거의 모든 과정에 참여합니다.많은 단백질은 생화학 반응을 촉매하는 효소이며 신진대사에 필수적입니다.단백질은 또한 근육의 액틴과 미오신과 세포골격의 단백질과 같은 구조적 또는 기계적 기능을 가지고 있으며, 세포 형태를 유지하는 발판 시스템을 형성합니다.다른 단백질들은 세포 신호 전달, 면역 반응, 세포 접착, 그리고 세포 순환에서 중요하다.동물에서 단백질은 합성할 수 없는 필수 아미노산을 공급하기 위해 식단에 필요하다.소화는 신진대사를 위해 단백질을 분해한다.
단백질은 초원심응고, 침전, 전기영동 및 크로마토그래피와 같은 다양한 기술을 사용하여 다른 세포 성분으로부터 정제될 수 있다. 유전공학의 출현은 정화를 용이하게 하는 많은 방법을 가능하게 했다.단백질 구조와 기능을 연구하기 위해 일반적으로 사용되는 방법에는 면역 조직 화학, 부위 지향 돌연변이 유발, X선 결정학, 핵자기 공명 및 질량 분석 등이 있다.
역사와 어원
단백질은 18세기에 Antoine Fourcroy와 다른 사람들에 의해 독특한 종류의 생물학적 분자로 인식되었고, 열이나 산으로 [1]처리되어 응고되거나 응집되는 분자의 능력으로 구별되었다.그 당시 주목할 만한 예로는 달걀 흰자의 알부민, 혈청 알부민, 피브린, 밀 글루텐 등이 있었다.
단백질은 네덜란드의 화학자 Gerardus Johannes Mulder에 의해 처음 설명되었고 [2][3]1838년 스웨덴의 화학자 Jöns Jacob Berzelius에 의해 명명되었다.멀더는 일반 단백질의 원소 분석을 수행했고 거의 모든 단백질이 동일한 경험식인 CHNOPS를40062010012011 [4]가지고 있다는 것을 발견했다.그는 그것들이 단일 유형의 (매우 큰) 분자로 구성되었을지도 모른다는 잘못된 결론에 도달했다.The term "protein" to describe these molecules was proposed by Mulder's associate Berzelius; protein is derived from the Greek word πρώτειος (proteios), meaning "primary",[5] "in the lead", or "standing in front",[6] + -in.멀더는 이어 아미노산 류신과 같은 단백질 분해 생성물을 확인했는데, 이 류신은 131Da의 [4]분자량을 발견했다. "단백질" 이전에는 "알부민" 또는 "알부민 물질"(독일어로 [7]Eweisskörper)과 같은 다른 이름이 사용되었다.
독일의 칼 폰 보이트와 같은 초기 영양학자들은 단백질이 신체의 구조를 유지하는데 가장 중요한 영양소라고 믿었다. 왜냐하면 일반적으로 "살은 [8]살을 만든다"고 믿었기 때문이다. 칼 하인리히 리트하우젠은 글루탐산의 확인으로 알려진 단백질 형태를 확장시켰다.코네티컷 농업 실험소에서 식물성 단백질에 대한 자세한 검토는 토마스 버 오스본에 의해 작성되었다.Lafayette Mendel과 협력하여 실험용 쥐에게 먹이를 주는 데 있어서 Liebig의 법칙을 적용함으로써 영양학적으로 필수적인 아미노산이 확립되었습니다.그 작업은 윌리엄 커밍 로즈가 계속하여 전달하였다.단백질을 폴리펩타이드로 이해하는 것은 1902년 [9][10]Franz Hofmeister와 Herman Emil Fischer의 연구를 통해 이루어졌다.살아있는 유기체에서 효소로써 단백질의 중심적인 역할은 제임스 B가 1926년까지 완전히 인식되지 않았다. 섬너는 우레아제 효소가 사실상 [11]단백질이라는 것을 보여주었다.
단백질을 대량으로 정제하는 것의 어려움은 초기 단백질 생화학자들이 단백질을 연구하는 것을 매우 어렵게 만들었다.따라서 초기 연구는 대량으로 정제될 수 있는 단백질, 예를 들어 혈액, 난백색, 다양한 독소 및 도축장에서 얻은 소화/대사 효소에 초점을 맞췄다.1950년대에 Armour Hot Dog사는 순수한 소 췌장핵산가수분해효소 A를 1kg 정제하여 과학자들이 자유롭게 사용할 수 있도록 하였다. 이러한 제스처는 리보핵산가수분해효소 A가 다음 수십 [4]년 동안 생화학 연구의 주요 표적이 되는 데 도움을 주었다.
라이너스 폴링은 1933년 [12]윌리엄 애스트베리에 의해 처음 제시된 수소 결합에 기초한 규칙적인 단백질 2차 구조를 성공적으로 예측한 공로를 인정받고 있다.Kaj Linderström-Lang의 [15]이전 연구에 부분적으로 기반을 둔 [13][14]변성에 대한 Walter Kauzmann의 이후 연구는 소수성 상호작용에 의해 매개되는 단백질 접힘과 구조에 대한 이해에 기여했다.
첫 번째 단백질은 1949년 프레드릭 생거가 만든 인슐린이다.생어는 인슐린의 아미노산 배열을 정확하게 결정하여 단백질이 분지 사슬, 콜로이드 [16]또는 사이클롤이 아닌 아미노산의 선형 중합체로 구성되었음을 결론적으로 입증했다.그는 1958년에 [17]이 업적으로 노벨상을 받았다.

X선 결정학의 발달로 단백질 [18]구조의 배열이 가능해졌다.최초로 [19][20]해결된 단백질 구조는 1958년 Max Perutz의 헤모글로빈과 John Cowdery Kendrew 경의 미오글로빈이었다.컴퓨터의 사용과 컴퓨팅 능력의 향상도 복잡한 단백질의 염기서열 분석을 지원했다.1999년 Roger Kornberg는 싱크로트론에서 [18]나오는 고강도 X선을 사용하여 RNA 중합효소의 매우 복잡한 구조를 배열하는 데 성공했다.
이후 대형 고분자 조립체의[21] 극저온 전자 현미경이 개발됐다.Cryo-EM은 결정보다는 냉동된 단백질 샘플, X선보다는 전자 빔을 사용한다.이것은 샘플에 손상을 덜 입히므로 과학자들이 더 많은 정보를 얻고 더 큰 [18]구조물을 분석할 수 있다.작은 단백질[22] 도메인의 계산 단백질 구조 예측은 또한 연구자들이 단백질 구조의 원자 수준 분해능에 접근하는 데 도움을 주었다.2017년 현재[update], 단백질 데이터 뱅크는 126,060개 이상의 단백질 [23]원자 분해능 구조를 가지고 있다.
게놈에 암호화되어 있는 단백질의 수
게놈에 암호화되어 있는 단백질의 수는 대략 유전자의 수와 일치한다(단백질의 RNA를 암호화하는 유전자의 수가 상당할 수 있지만, 예를 들어 리보솜 RNA).바이러스는 전형적으로 수백 개의 단백질, 고세균, 박테리아를 수백에서 수천 개의 단백질로 인코딩하는 반면, 진핵생물은 전형적으로 수천에서 수만 개의 단백질을 인코딩합니다.
생화학
대부분의 단백질은 최대 20개의 서로 다른 L-α- 아미노산으로 구성된 선형 고분자로 구성된다.모든 단백질 발생 아미노산은 아미노기, 카르복실기 및 가변 측쇄가 결합되어 있는 α-탄소를 포함한 공통의 구조적 특징을 가진다.프롤린만이 이 기본 구조와 다르다. 왜냐하면 프롤린은 CO-NH 아미드 부분을 고정된 [24]형태로 강제하는 N-end 아민 그룹에 대한 특이한 고리를 포함하고 있기 때문이다.표준 아미노산 목록에 자세히 나와 있는 표준 아미노산의 곁사슬은 매우 다양한 화학적 구조와 특성을 가지고 있습니다. 이것은 단백질의 모든 아미노산 곁사슬의 결합 효과로 궁극적으로 3차원 구조와 화학적 [25]반응성을 결정합니다.폴리펩타이드 사슬의 아미노산은 펩타이드 결합에 의해 결합된다.일단 단백질 사슬에 결합되면, 개별 아미노산은 잔기라고 불리며, 연결된 탄소, 질소, 산소 원자는 주 사슬 또는 단백질 [26]: 19 골격으로 알려져 있습니다.
펩타이드 결합은 이중 결합 특성을 기여하고 축을 중심으로의 회전을 억제하는 두 가지 공명 형태를 가지며, 따라서 알파 탄소는 대략 동일 평면이다.펩타이드 결합의 다른 2면체 각도는 단백질 [26]: 31 골격에 의해 가정된 국소 형태를 결정한다.활성 아미노기가 있는 끝은 N 말단 또는 아미노 말단이라고 알려진 반면, 활성 카르복실기가 있는 단백질의 끝은 C 말단 또는 카르복시 말단으로 알려져 있다(단백질의 배열은 N 말단에서 C 말단으로 왼쪽에서 오른쪽으로 쓰여진다).
단백질, 폴리펩타이드, 펩타이드라는 단어는 조금 애매하고 의미가 중복될 수 있다.단백질은 일반적으로 안정적인 구성의 완전한 생물학적 분자를 참조하기 위해 사용되며, 펩타이드는 일반적으로 안정적인 3D 구조가 결여된 짧은 아미노산 올리고머를 위해 저장된다.그러나 둘 사이의 경계는 잘 정의되지 않았으며 일반적으로 20~30개의 [27]잔류물 근처에 있다.폴리펩타이드는 보통 길이에 관계없이 아미노산의 단일 선형 사슬을 나타낼 수 있지만, 종종 정의된 배치가 없음을 암시합니다.
상호 작용
단백질은 다른 단백질, 지질, 탄수화물, 그리고 [28][29][26][30]DNA를 포함한 많은 종류의 분자와 상호작용할 수 있습니다.
세포의 풍부함
평균 크기의 박테리아는 세포당 약 2백만 개의 단백질을 포함하고 있는 것으로 추정되었습니다(예: 대장균과 황색포도상구균).미코플라스마나 스피로체트와 같은 작은 박테리아는 약 50,000에서 100만 개의 분자를 포함하고 있다.반면에, 진핵 세포는 더 크고 따라서 훨씬 더 많은 단백질을 포함하고 있다.예를 들어, 효모 세포는 약 5천만 개의 단백질과 약 30억 [31]개의 인간 세포를 포함하는 것으로 추정되어 왔다.개별 단백질 복제의 농도는 세포당 몇 개의 분자에서 2천만 [32]개까지 다양하다.단백질을 코드하는 모든 유전자가 대부분의 세포에서 발현되는 것은 아니며, 그 수는 예를 들어 세포 유형과 외부 자극에 따라 달라집니다.예를 들어 인간 게놈에 의해 암호화되는 약 2만 개의 단백질 중 림프아세포에서 [33]검출되는 단백질은 6,000 개에 불과하다.
합성
생합성
단백질은 유전자에 암호화된 정보를 사용하여 아미노산으로부터 조립된다.각 단백질은 이 단백질을 코드하는 유전자의 뉴클레오티드 배열에 의해 특정되는 고유한 아미노산 배열을 가진다.유전자 코드는 코돈이라고 불리는 3개의 뉴클레오티드 세트의 집합이고 각각의 3개의 뉴클레오티드 조합은 아미노산을 나타냅니다. 예를 들어, AUG는 메티오닌의 코드입니다.DNA는 4개의 뉴클레오티드를 포함하고 있기 때문에, 가능한 코돈의 총 수는 64개이다. 따라서 유전자 코드에는 몇 가지 중복성이 있으며, 일부 아미노산은 하나 이상의 [30]: 1002–42 코돈에 의해 지정된다.DNA로 암호화된 유전자는 우선 RNA 중합효소 같은 단백질에 의해 프리메신저 RNA(mRNA)로 전사된다.그리고 나서 대부분의 유기체는 리보솜에 의한 단백질 합성을 위한 템플릿으로 사용되는 성숙한 mRNA를 형성하기 위해 다양한 형태의 전사 후 변형을 사용하여 사전 mRNA(일차 전사물로도 알려져 있음)를 처리한다.원핵생물에서 mRNA는 생성되자마자 사용되거나 핵에서 떨어진 후 리보솜에 의해 결합될 수 있다.반대로, 진핵생물은 세포핵에서 mRNA를 만든 후 핵막을 가로질러 단백질 합성이 이루어지는 세포질로 이동시킨다.단백질 합성 속도는 진핵생물보다 원핵생물에서 더 높고 [34]초당 최대 20개의 아미노산에 도달할 수 있다.
mRNA 템플릿에서 단백질을 합성하는 과정을 번역이라고 한다.mRNA는 리보솜에 로드되어 각각의 코돈을 그것이 인식하는 코돈에 대응하는 아미노산을 운반하는 전달 RNA 분자에 위치한 염기쌍 안티코돈에 매칭함으로써 한 번에 세 개의 뉴클레오티드를 읽는다.아미노아실 tRNA 합성효소는 tRNA 분자를 올바른 아미노산으로 "충전"시킵니다.성장하는 폴리펩타이드는 종종 초기 사슬이라고 불린다.단백질은 항상 N 말단에서 C [30]: 1002–42 말단으로 생합성된다.
합성 단백질의 크기는 아미노산이 함유된 수와 총 분자량에 의해 측정될 수 있으며, 이는 보통 달톤 단위(원자 질량 단위와 동의어) 또는 유도체 단위 킬로달톤(kDa)으로 보고된다.단백질의 평균 크기는 고등 [35]생물에서 단백질을 구성하는 단백질 도메인의 수가 많아짐에 따라 고세균에서 박테리아로 진핵생물(각각 283, 311, 438잔기, 31, 34, 49kDa)으로 증가한다.예를 들어 효모 단백질의 길이는 평균 466개이고 질량은 [27]53kDa이다.가장 큰 것으로 알려진 단백질은 근육의 성분인 티틴으로, 분자량은 거의 3,000 kDa이고 총 길이는 거의 27,000 아미노산입니다.[36]
화학 합성
짧은 단백질은 또한 [37]펩타이드 합성이라고 알려진 방법군에 의해 화학적으로 합성될 수 있으며, 펩타이드를 높은 수율로 생산하기 위해 화학적 결합과 같은 유기 합성 기술에 의존한다.화학 합성은 아미노산 [38]측쇄에 형광 탐침을 부착하는 것과 같이 폴리펩타이드 사슬에 비천연 아미노산을 도입할 수 있게 한다.이 방법들은 일반적으로 상업적인 용도에는 아니지만 실험실 생화학 및 세포생물학에서는 유용하다.약 300개의 아미노산보다 긴 폴리펩타이드에 대한 화학적 합성은 비효율적이며, 합성된 단백질은 그들의 고유 3차 구조를 쉽게 가정하지 못할 수 있다.대부분의 화학 합성 방법은 생물학적 [39]반응의 반대인 C 말단에서 N 말단으로 진행됩니다.
구조.


대부분의 단백질은 독특한 3D 구조로 접힙니다.단백질이 자연스럽게 접히는 모양을 토종 [26]: 36 형태라고 한다.비록 많은 단백질이 보조 없이 접힐 수 있지만, 단순히 아미노산의 화학적 성질을 통해, 다른 단백질들은 그들의 고유 상태로 접히기 위해 분자 [26]: 37 샤페론의 도움을 필요로 합니다.생화학자는 종종 단백질 구조의 [26]: 30–34 네 가지 뚜렷한 측면을 언급한다.
- 1차 구조: 아미노산 배열.단백질은 폴리아미드이다.
- 2차 구조: 수소 결합에 의해 안정화된 국소 구조를 정기적으로 반복한다.가장 일반적인 예는 α-나선, β-시트 및 턴입니다.2차 구조가 국소적이기 때문에, 다른 2차 구조의 많은 영역이 동일한 단백질 분자에 존재할 수 있다.
- 3차 구조: 단일 단백질 분자의 전체적인 형태; 2차 구조의 공간적 관계 서로.3차 구조는 일반적으로 비국소적 상호작용, 가장 일반적으로 소수성 코어의 형성에 의해 안정화되지만 염교, 수소 결합, 이황화물 결합, 그리고 심지어 번역 후 변형을 통해서도 안정화된다."teriary structure"라는 용어는 종종 접힘이라는 용어와 동의어로 사용된다.3차 구조는 단백질의 기본 기능을 조절하는 것이다.
- 4차 구조: 여러 단백질 분자(폴리펩타이드 사슬)에 의해 형성되는 구조. 보통 이 맥락에서 단백질 서브유닛이라고 불리며, 단일 단백질 복합체로 기능합니다.
- 4진 구조: 복잡한 세포 내부를 구성하는 단백질 표면의 특징.2진 구조는 살아있는 세포 안에서 일어나는 일시적이지만 필수적인 고분자 상호작용에 의존합니다.
단백질은 완전히 단단한 분자는 아니다.이러한 수준의 구조 외에도, 단백질은 기능을 수행하는 동안 관련된 여러 구조 사이에서 이동할 수 있다.이러한 기능 재배열의 맥락에서, 이러한 3차 또는 4차 구조는 보통 "구성"이라고 불리며, 이들 사이의 전이는 구조 변화라고 불린다.이러한 변화는 종종 효소의 활성 부위 또는 화학적 촉매 작용에 참여하는 단백질의 물리적 영역에 대한 기질 분자의 결합에 의해 유발된다.용액에서 단백질은 또한 열진동과 다른 [30]: 368–75 분자와의 충돌을 통해 구조의 변화를 겪는다.
단백질은 비공식적으로 구상 단백질, 섬유 단백질, 막 단백질의 전형적인 3차 구조와 상관관계가 있는 세 가지 주요 클래스로 나눌 수 있습니다.거의 모든 구상 단백질은 용해성이며 많은 것들이 효소이다.섬유질 단백질은 종종 결합 조직의 주요 성분인 콜라겐이나 머리카락과 손톱의 단백질 성분인 케라틴과 같은 구조적인 단백질입니다.막 단백질은 종종 수용체 역할을 하거나 극성 또는 하전된 분자가 [30]: 165–85 세포막을 통과할 수 있는 채널을 제공한다.
단백질 내 분자 내 수소 결합의 특별한 경우, 물의 공격으로부터 잘 보호되지 않고, 따라서 그들 자신의 탈수를 촉진하는 것은 [40]탈수소라고 불립니다.
단백질 도메인
많은 단백질은 여러 단백질 도메인, 즉 뚜렷한 구조 단위로 접히는 단백질의 세그먼트로 구성되어 있다.도메인은 또한 일반적으로 효소 활성(예: 키나제)과 같은 특정 기능을 가지거나 결합 모듈 역할을 한다(예: SH3 도메인은 다른 단백질의 프롤린이 풍부한 배열에 결합한다).
시퀀스 모티브
단백질 이내의 쇼트 아미노산 서열은 종종 다른 단백질을 위한 인지 사이트의 역할도 한다.[41]예를 들어, SH3 도메인 일반적으로 짧은 PxxP의 모티브(즉 2prolines[P], 2지정되지 않은 아미노산[)]에 의해 분리되지만 그 주변의 아미노산은 정확한 바인딩의 특이성을 결정할 수 있)를 에 바인딩 하여.많은 이러한 모티브는 진핵 선형 동기(ELM)데이터베이스에 수집되었다고.
셀룰러 기능
그 세포들은 이내에 단백질은 최고 배우들이 업무가 정보 유전자에 인코딩된에 의해 지정된를 수행할 것이라고 말했다.[27]RNA의 특정 형식에서 예외로 하고, 가장 다른 생물학적인 분자들은 상대적으로 불활성 요소에 단백질 작용한다.반면 DNA와 RNA와 같은 다른 거대 분자만 3%이고 20%각각 만드는 단백질은, 대장 균 세포의 반 정도의 건조 중량을 차지하고 있다.[42]단백질의 세트가 특별한 세포나 세포 타입은 proteome로 알려진 것을 표현했다.
단백질의 기능의 그들의 다양한 정리되게 할 수 있는 주요한 특징은 그들의 능력 구체적으로 단단히 다른 분자하지 않다.단백질은 다른 분자 결합에 책임이 있는 이 지역은 결합 부위로 분자 표면에 종종 우울증이나"주머니" 알려져 있다.이 결합 능력이 단백질의 결합 부위 주머니를 정의하는 삼차 구조와 주변 아미노산의 편 체인점들의 화학적 성질에 의해에 의해 전달된다.단백질은 바인딩 유달리고 구체적인;예를 들어, 리보 핵산 가수 분해 효소 억제제 단백질 자식은 인간 angiogenin에sub-femtomolar 해리 상수(<>10−15 M)로 보지는 양서류 homologonconase(>1M)에 바인딩 하지 않는다. 단일 메틸 그룹의 빈디에 추가와 같이 극도로 경미한 화학 변화를 빠듯할 수 있다.공개 partn어 때때로 거의 바인딩을 제거하기 위해;예를 들어 발린 아미노산 이소류신의 매우 유사한 곁 사슬을 차별화는 아미노산에 아미노 아실 tRNA합성 효소 특정한 충분할 수 있다.[43]
단백질은 작은 분자 기질뿐만 아니라 다른 단백질에도 결합할 수 있다.단백질이 같은 분자의 다른 복제물과 특이하게 결합할 때, 그들은 섬유소를 형성하기 위해 올리고머를 형성할 수 있습니다; 이 과정은 종종 단단한 섬유를 형성하기 위해 자가 결합하는 구상 단량체로 구성된 구조 단백질에서 발생합니다.단백질-단백질 상호작용은 또한 효소 활성을 조절하고, 세포 사이클을 통한 진행을 제어하며, 공통의 생물학적 기능과 밀접하게 관련된 많은 반응을 수행하는 큰 단백질 복합체들의 집합을 가능하게 한다.단백질은 또한 세포막에 결합하거나 세포막에 통합될 수 있다.단백질의 구조 변화를 유도하는 결합 파트너의 능력은 엄청나게 복잡한 [30]: 830–49 시그널링 네트워크의 구축을 가능하게 한다.단백질 사이의 상호작용은 가역적이며, 분리된 기능 세트를 수행할 수 있는 집계를 형성하기 위해 파트너 단백질의 다른 그룹의 가용성에 크게 의존하기 때문에, 특정 단백질 사이의 상호작용 연구는 세포 기능의 중요한 측면, 그리고 궁극적으로 속성을 이해하는 열쇠입니다.특정 세포 [44][45]유형을 구별하는 데 사용됩니다.
효소
세포에서 단백질의 가장 잘 알려진 역할은 화학 반응을 촉매하는 효소이다.효소는 보통 매우 특이하며 오직 한 가지 또는 몇 가지 화학 반응만 가속화한다.효소는 DNA 복제, DNA 복구, 그리고 전사와 같은 과정에서 DNA를 조작하는 것뿐만 아니라 신진대사와 관련된 대부분의 반응을 수행합니다.일부 효소는 번역 후 변형으로 알려진 과정에서 화학 그룹을 추가하거나 제거하기 위해 다른 단백질에 작용합니다.약 4,000개의 반응이 [46]효소에 의해 촉매되는 것으로 알려져 있다.효소 촉매 작용에 의해 부여되는 속도 가속은 종종 엄청나며, 오로틴산 탈탄산화효소의 경우 비촉매 반응보다 속도가 10배나17 증가한다(효소가 없는 7800만 년, [47]효소가 있는 18밀리초).
효소에 의해 결합되고 작용하는 분자를 기질이라고 한다.효소는 수백 개의 아미노산으로 구성될 수 있지만, 일반적으로 기질에 접촉하는 잔류물의 극히 일부에 불과하고 촉매 [48]작용에 직접적으로 관여하는 잔류물은 훨씬 더 적다(평균적으로 3~4개).기질을 결합하고 촉매 잔류물을 포함하는 효소의 영역을 활성 부위라고 합니다.
디리겐트 단백질은 다른 [49]효소에 의해 합성된 화합물의 입체 화학을 지시하는 단백질의 한 부류의 구성원이다.
세포신호 및 배위자 결합
많은 단백질은 세포 신호 전달과 신호 전달 과정에 관여한다.인슐린과 같은 일부 단백질은 세포 외 단백질로, 먼 조직의 다른 세포로 합성된 세포로부터 신호를 전달한다.다른 것들은 신호 분자와 결합하고 세포에서 생화학 반응을 유도하는 주요 기능을 하는 수용체 역할을 하는 막 단백질이다.많은 수용체는 세포 표면에 노출된 결합 부위와 세포 내에 효소 활성을 가질 수 있는 이펙터 도메인을 가지고 있거나 세포 [29]: 251–81 내의 다른 단백질에 의해 검출된 구조 변화를 겪을 수 있다.
항체는 적응형 면역체계의 단백질 성분으로, 주요 기능은 항원이나 체내의 이물질을 결합시켜 파괴 대상으로 삼는 것이다.항체는 세포외 환경으로 분비되거나 플라즈마 세포로 알려진 특수 B 세포의 막에 고정될 수 있습니다.효소는 반응의 필요성에 의해 기질에 대한 결합 친화력이 제한되는 반면, 항체는 그러한 제약을 받지 않는다.항체의 표적에 대한 결합 친화력은 매우 높다.[30]: 275–50
많은 배위자 수송 단백질은 특정한 작은 생체 분자와 결합하고 다세포 유기체의 신체에 있는 다른 위치로 운반합니다.이러한 단백질은 배위자가 고농도로 존재할 때 높은 결합 친화력을 가져야 하지만, 표적 조직에 저농도로 존재할 때 배위자를 방출해야 한다.리간드 결합 단백질의 전형적인 예는 헤모글로빈입니다. 헤모글로빈은 폐에서 모든 척추동물의 다른 기관과 조직으로 산소를 운반하고 모든 생물학적 [30]: 222–29 왕국에서 밀접한 상동성을 가지고 있습니다.렉틴은 당분과 결합하는 단백질로 당분에 매우 특이하다.렉틴은 전형적으로 세포와 [50]단백질을 포함한 생물학적 인식 현상에 역할을 한다.수용체와 호르몬은 매우 특이적인 결합 단백질이다.
막 통과 단백질은 또한 작은 분자와 이온에 대한 세포막의 투과성을 변화시키는 리간드 수송 단백질의 역할을 할 수 있습니다.막은 극성 또는 하전된 분자가 확산할 수 없는 소수성 코어를 가지고 있다.막 단백질은 그러한 분자들이 세포로 들어오고 나가는 것을 허용하는 내부 채널을 포함합니다.많은 이온 채널 단백질은 특정 이온만을 선택하도록 특화되어 있습니다. 예를 들어, 칼륨과 나트륨 채널은 종종 두 [29]: 232–34 이온 중 하나만 구별합니다.
구조단백질
구조 단백질은 그렇지 않으면 유동적인 생물학적 구성요소에 강성과 강성을 부여한다.대부분의 구조 단백질은 섬유 단백질이다; 예를 들어, 콜라겐과 엘라스틴은 연골과 같은 결합 조직의 중요한 구성 요소이고, 케라틴은 털, 손톱, 깃털, 발굽, 그리고 일부 동물 [30]: 178–81 껍질과 같은 단단하거나 필라멘트 구조에서 발견됩니다.일부 구상 단백질은 또한 구조적인 기능을 할 수 있는데, 예를 들어, 액틴과 튜불린은 구상이고 모노머로 용해되지만, 세포 골격을 구성하는 길고 단단한 섬유를 형성하기 위해 중합되어 세포가 모양과 크기를 유지할 수 있게 합니다.
구조적인 기능을 하는 다른 단백질로는 미오신, 키네신, 다이네인과 같은 운동 단백질들이 있는데, 이들은 기계적 힘을 발생시킬 수 있다.이 단백질들은 단세포 유기체의 세포 운동성과 성적으로 번식하는 많은 다세포 유기체의 정자에 매우 중요하다.그들은 또한 근육을 수축시킴으로써[30]: 258–64, 272 가해지는 힘을 발생시키고 세포 내 수송에 필수적인 역할을 한다.
단백질 진화
분자생물학의 핵심 질문은 단백질이 어떻게 진화하느냐이다. 즉, 돌연변이(또는 아미노산 배열의 변화)가 어떻게 새로운 구조와 기능을 이끌 수 있는가?단백질의 대부분의 아미노산은 종에 걸친 수많은 상동 단백질에서 볼 수 있듯이 활동이나 기능을 방해하지 않고 변할 수 있다(예: [51]PFAM과 같은 단백질 패밀리에 대한 전문 데이터베이스에서 수집).돌연변이의 극적인 결과를 막기 위해 유전자는 자유롭게 변이하기 전에 복제될 수 있다.그러나, 이것은 유전자 기능의 완전한 상실로 이어질 수도 있고, 따라서 의사 유전자가 [52]될 수도 있다.보다 일반적으로, 어떤 것들은 단백질 기능을 크게 바꿀 수 있지만, 특히 효소에서 단일 아미노산의 변화는 제한적인 결과를 가져온다.예를 들어, 많은 효소는 1개 또는 몇 [53]개의 돌연변이에 의해 기질특이성을 바꿀 수 있다.기질특이성의 변화는 기질문란성, 즉 여러 기질을 결합 및 처리하는 많은 효소의 능력에 의해 촉진된다.돌연변이가 발생하면 효소의 특이성이 증가(또는 감소)하여 효소의 효소 [53]활성이 증가할 수 있습니다.따라서 박테리아(또는 다른 유기체)는 플라스틱과 [54]같은 부자연스러운 기질을 포함한 다양한 식품원에 적응할 수 있다.
학습 방법
단백질의 활성과 구조는 시험관내, 생체내 및 실리코에서 검사될 수 있다.통제된 환경에서 정제된 단백질에 대한 시험관 내 연구는 단백질이 어떻게 그 기능을 수행하는지를 배우는데 유용하다: 예를 들어, 효소 역학 연구는 효소의 촉매 활성의 화학적 메커니즘과 다양한 가능한 기질 분자에 대한 상대적 친화력을 탐구한다.반대로 생체내 실험은 세포 또는 전체 유기체의 맥락에서 단백질의 생리학적 역할에 대한 정보를 제공할 수 있다.실리코 연구에서 단백질을 연구하기 위해 계산 방법을 사용한다.
단백질 정제
시험관내 분석을 실시하기 위해서는 단백질을 다른 세포 성분으로부터 분리하여 정제해야 한다.이 과정은 보통 세포의 막이 파괴되고 그 내부 내용물이 조용액으로 알려진 용액으로 방출되는 세포 용해로 시작된다.그 결과 생성된 혼합물은 다양한 세포 구성 요소를 수용성 단백질, 막 지질 및 단백질, 세포 기관 및 핵산을 포함하는 분율로 분류하는 초원심 침지를 사용하여 정제될 수 있습니다.염분 처리라고 알려진 방법에 의한 침전은 이 용해액으로부터 단백질을 농축할 수 있다.다음으로 분자량, 순 전하 및 결합 [26]: 21–24 친화성과 같은 특성에 기초하여 관심 있는 단백질 또는 단백질을 분리하기 위해 다양한 유형의 크로마토그래피가 사용된다.원하는 단백질의 분자량과 등전점이 알려진 경우, 단백질이 구별할 수 있는 분광학적 특징을 가지고 있는 경우 분광학, 단백질이 효소 활성을 가지고 있는 경우 효소 분석을 통해 다양한 유형의 겔 전기영동을 통해 정제 수준을 모니터링할 수 있다.또한 전기초점법을 이용하여 [55]전하에 따라 단백질을 분리할 수 있다.
천연 단백질의 경우, 실험실 적용에 충분한 순수 단백질을 얻기 위해 일련의 정제 단계가 필요할 수 있다.이 과정을 단순화하기 위해, 유전자 공학은 종종 단백질의 구조나 활동에 영향을 주지 않고 정화하기 쉽게 하는 화학적 특징을 단백질에 추가하는 데 사용된다.여기서 특정 아미노산 배열, 종종 일련의 히스티딘 잔기(His-tag)로 구성된 "태그"가 단백질의 한 말단에 부착된다.그 결과, 리세이트가 니켈을 포함한 크로마토그래피 컬럼에 통과하면 히스티딘 잔류물이 니켈을 염화시켜 리세이트의 태그 부착되지 않은 성분이 방해받지 않고 통과하면서 컬럼에 부착된다.연구자들이 복잡한 [56]혼합물로부터 특정 단백질을 정화하는 것을 돕기 위해 많은 다른 태그들이 개발되었습니다.
셀룰러 현지화
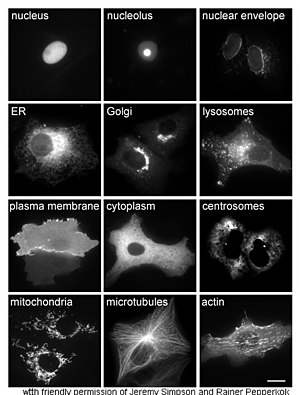
생체 내 단백질 연구는 종종 세포 내 단백질의 합성과 국부화와 관련이 있다.많은 세포내 단백질이 세포질 및 소포체의 막결합 또는 분비 단백질에서 합성되지만, 단백질이 어떻게 특정 기관이나 세포구조를 목표로 하는지에 대한 자세한 내용은 종종 불분명하다.세포 국재성 평가를 위한 유용한 기술은 유전자 공학을 사용하여 녹색 형광단백질(GFP)[57]과 같은 "리포터"에 연결된 천연 단백질로 이루어진 융합단백질 또는 키메라(kimera)를 세포에 발현시킨다.세포 내에서 융합된 단백질의 위치는 반대쪽 그림과 같이 현미경을 [58]사용하여 깨끗하고 효율적으로 시각화할 수 있습니다.
단백질의 세포 위치를 설명하기 위한 다른 방법으로는 ER, 골지, 리소좀 또는 액포, 미토콘드리아, 엽록체, 혈장막 등과 같은 영역에 대해 알려진 구획 마커를 사용해야 한다.형광 태그 부착 버전의 마커 또는 알려진 마커에 대한 항체를 사용하면 관심 있는 단백질의 국소화를 식별하는 것이 훨씬 쉬워집니다.예를 들어 간접면역형광은 형광의 총초점화 및 위치표현을 가능하게 한다.형광 염료는 [59]유사한 목적으로 세포 구획에 라벨을 붙이는 데 사용됩니다.
다른 가능성도 존재한다.예를 들어 면역조직화학은 보통 하나 이상의 관심 단백질에 대한 항체를 이용하는데, 이 항체는 샘플 간에 비교할 수 있는 발광 신호 또는 발색 신호를 생성하는 효소와 결합되어 위치 정보를 가능하게 한다.또 다른 적용 가능한 기법은 이소픽닉 [60]원심분리를 이용한 수크로스(또는 기타 재료) 구배에서의 결합이다.이 기술은 이미 알려진 밀도와 관심 단백질 구획의 혈소 침착을 증명하지는 않지만, 가능성을 높이고 대규모 연구에 더 잘 적응한다.
마지막으로 세포 국재화의 금본위법은 면역전자현미경법이다.이 기술은 또한 기존의 전자 현미경 검사 기술과 함께 관심 단백질에 대한 항체를 사용합니다.샘플은 일반적인 전자 현미경 검사를 위해 준비되고, 그 후 매우 전기 밀도가 높은 물질(일반적으로 금)과 결합되는 관심 단백질에 대한 항체로 처리됩니다.이를 통해 관심 [61]단백질뿐만 아니라 초미세 구조 세부 사항의 위치 파악이 가능합니다.
부위 지향적 돌연변이 유발로 알려진 또 다른 유전자 공학 응용 프로그램을 통해, 연구자들은 단백질 배열과 그 구조, 세포 위치 및 조절에 대한 민감성을 바꿀 수 있다.이 기술은 심지어 변형된 [62]tRNA를 사용하여 단백질에 부자연스러운 아미노산의 통합을 허용하고, 새로운 [63]특성을 가진 새로운 단백질의 합리적인 설계를 허용할 수 있다.
프로테오믹스
세포 또는 세포 유형에서 한 번에 존재하는 단백질의 전체 보체는 프로테옴으로 알려져 있으며, 그러한 대규모 데이터 세트의 연구는 게놈의 관련 분야와 유추하여 명명된 프로테오믹스 분야를 정의한다.단백질 체학의 핵심 실험 방식들은 많은 단백질의 분리, 단백질의 vari의 상대적인 수준의 검출할 수 있는 펩티드(가장 자주 후in-gel 소화), 단백질 마이크로 어레이 빨리 실험해 식별 및 순서 허용 질량 spectrometry,[65]수 있도록 2Delectrophoresis,[64]을 포함한다.용어 프로세포에 존재하는 티인 및 단백질-피질 [66]상호작용의 체계적인 탐구를 가능하게 하는 2-피질 스크리닝.생물학적으로 가능한 그러한 상호작용의 총 보완체를 [67]인터랙텀이라고 한다.모든 가능한 접힘을 나타내는 단백질의 구조를 결정하는 체계적인 시도는 구조 [68]유전체학으로 알려져 있다.
구조 결정
단백질의 3차 구조 또는 복합체의 4차 구조를 발견하는 것은 단백질이 어떻게 그 기능을 수행하고 어떻게 영향을 받을 수 있는지에 대한 중요한 단서를 제공할 수 있다. 즉, 의약품 설계에서 말이다.단백질이 너무 작아서 광현미경으로 볼 수 없기 때문에, 단백질의 구조를 결정하기 위해서는 다른 방법들이 사용되어야 한다.일반적인 실험 방법으로는 X선 결정학과 NMR 분광법이 있으며, 두 방법 모두 원자 분해능에서 구조 정보를 생성할 수 있다.그러나 NMR 실험은 원자 쌍 사이의 거리의 서브셋을 추정할 수 있는 정보를 제공할 수 있으며, 단백질에 대한 최종 가능한 배치는 거리 기하학 문제를 해결함으로써 결정된다.이중 편파 간섭계는 상호작용이나 다른 자극에 의한 전체적인 단백질 배치 및 배치 변화를 측정하기 위한 정량적 분석 방법이다.원형 이색성은 단백질의 내부 β-시트/α-나선조성을 결정하기 위한 또 다른 실험실 기술이다.극저온전자현미경법은 [29]: 340–41 조립된 바이러스를 포함한 매우 큰 단백질 복합체에 대한 저해상도 구조 정보를 생산하기 위해 사용된다; 전자 결정학으로 알려진 변종 또한 몇몇 경우에, 특히 [69]막 단백질의 2차원 결정에 대해 고해상도 정보를 생산할 수 있다.해결된 구조는 보통 단백질의 [70]각 원자에 대한 데카르트 좌표의 형태로 수천 개의 단백질에 대한 구조 데이터를 얻을 수 있는 자유롭게 이용 가능한 자원인 단백질 데이터 뱅크(PDB)에 축적된다.
단백질 구조보다 훨씬 더 많은 유전자 배열이 알려져 있다.또한 분해된 구조 세트는 주요 구조 결정 방법 중 하나인 X선 결정학에서 요구되는 조건에 쉽게 노출될 수 있는 단백질에 편향되어 있다.특히 구상 단백질은 X선 결정학에 대비하여 비교적 결정화가 용이하다.반면 막 단백질과 큰 단백질 복합체는 결정화하기가 어렵고 [71]PDB에서 충분히 표현되지 않는다.구조 유전체학 이니셔티브는 주요 접이식 클래스의 대표 구조를 체계적으로 해결함으로써 이러한 결함을 해결하려고 시도했다.단백질 구조 예측 방법은 구조가 실험적으로 [72]결정되지 않은 단백질에 대해 그럴듯한 구조를 생성하는 수단을 제공하려고 시도한다.
구조 예측

구조 유전체학 분야를 보완하는 단백질 구조 예측은 실험실에서 [73]관찰한 구조를 발견하는 대신 이론상 분자 형성을 계산적으로 예측하기 위해 단백질의 효율적인 수학적 모델을 개발합니다.호몰로지 모델링으로 알려진 구조 예측의 가장 성공적인 유형은 모델링되는 단백질과 배열 유사성을 가진 "템플릿" 구조의 존재에 의존합니다; 구조 유전체학의 목표는 [74]남아있는 대부분의 구조를 모델링하기 위해 해결된 구조에서 충분한 표현을 제공하는 것입니다.멀리 관련된 템플릿 구조만 사용할 수 있는 경우 정확한 모델을 생성하는 것은 여전히 어려운 과제이지만, "완벽한" 시퀀스 정렬을 [75]알면 꽤 정확한 모델을 생성할 수 있기 때문에 이 프로세스에서 시퀀스 정렬이 병목 현상이라고 제안되어 왔다.많은 구조 예측 방법들이 새로운 단백질 접힘이 이미 [76]설계되어 있는 단백질 공학의 새로운 분야를 알리는 역할을 해왔다.또한 단백질(진핵생물에서 33%)은 구조화되지 않았지만 생물학적으로 기능하는 큰 세그먼트를 포함하고 있으며 본질적으로 무질서한 [77]단백질로 분류될 수 있다.따라서 단백질 장애를 예측하고 분석하는 것은 단백질 구조 [78]특성화의 중요한 부분이다.
생물정보학
단백질의 구조, 기능 및 진화를 분석하기 위해 광범위한 계산 방법이 개발되었습니다.이러한 도구의 개발은 인간 게놈을 포함한 다양한 유기체가 이용할 수 있는 대량의 게놈 및 단백질 게놈 데이터에 의해 추진되어 왔다.모든 단백질을 실험적으로 연구하는 것은 단순히 불가능하기 때문에, 오직 소수만이 실험의 대상이 되며, 계산 도구는 유사한 단백질에 대한 추론을 위해 사용된다.이러한 상동성 단백질은 배열 배열에 의해 원거리 관련 유기체에서 효율적으로 식별될 수 있다.게놈과 유전자 배열은 특정 성질을 위해 다양한 도구에 의해 검색될 수 있다.배열 프로파일링 도구는 제한 효소 부위를 찾고, 뉴클레오티드 배열의 판독 프레임을 열고, 2차 구조를 예측할 수 있습니다.현생 생물들의 조상과 그들이 발현하는 유전자에 관한 ClusteralW와 같은 특별한 소프트웨어를 사용하여 계통수를 구성하고 진화 가설을 개발할 수 있다.생물정보학 분야는 이제 유전자와 단백질 분석에 필수적이다.
동적 프로세스의 silico 시뮬레이션
보다 복잡한 계산 문제는 분자 도킹,[79] 단백질 폴딩, 단백질-단백질 상호작용 및 화학 반응성과 같은 분자 간 상호작용의 예측이다.이러한 동적 과정을 시뮬레이션하기 위한 수학적 모델은 분자 역학, 특히 분자 역학을 포함합니다.이와 관련하여, 실리코 시뮬레이션에서 빌린 헤드피스,[80] HIV 보조[81] 단백질 및 표준 분자 역학과 양자 역학을 결합한 하이브리드 방법 등 작은 α-나선 [82]단백질 영역의 접힘을 발견했다.
고전적인 분자역학 이외에도 양자역학 방법은 양자역학적 효과에 대한 정확한 설명과 함께 원자론적 세부사항에서 단백질의 시뮬레이션을 가능하게 한다.예로는 식물 크립토크롬과[83] 박테리아 광수확 [84]복합체에 각각 적용된 다층 다구성 시간 의존 하트리(MCTDH) 방법과 계층 운동 방정식(HEOM) 접근법을 들 수 있다.생물학적 스케일 시스템의 양자 및 고전적 기계적 시뮬레이션은 모두 계산적으로 매우 까다롭기 때문에 분산 컴퓨팅 이니셔티브(예: Folding@home[85] 프로젝트)는 GPU 병렬 처리 및 몬테카를로 기술의 진보를 활용하여 분자 모델링을 촉진합니다.
화학 분석
유기물의 총 질소 함량은 주로 단백질의 아미노기에 의해 형성된다.TKN(Total Kjeldahl Nitroon)은 일반적으로 물, 토양, 식품, 사료 및 유기물 분석에 널리 사용되는 질소의 측정값입니다.이름에서 알 수 있듯이 Kjeldahl 방법이 적용됩니다.보다 민감한 방법을 사용할 [86][87]수 있습니다.
영양
대부분의 미생물과 식물은 20개의 표준 아미노산을 모두 생합성할 수 있는 반면, 동물(사람 포함)은 [42]식단에서 아미노산의 일부를 얻어야 한다.유기체가 스스로 합성할 수 없는 아미노산을 필수 아미노산이라고 한다.특정 아미노산을 합성하는 핵심 효소는 아스파르트키나제와 같은 동물에는 존재하지 않는다. 아스파르트키나제는 아스파르트산으로부터 리신, 메티오닌, 트레오닌을 합성하는 첫 단계를 촉매한다.만약 아미노산이 환경에 존재한다면, 미생물은 주변의 아미노산을 흡수하고 그들의 생합성 경로를 하향 조절함으로써 에너지를 보존할 수 있다.
동물에서 아미노산은 단백질을 함유한 식품을 섭취함으로써 얻어진다.섭취된 단백질은 소화를 통해 아미노산으로 분해되는데, 이것은 전형적으로 단백질 분해효소라고 불리는 효소에 의한 산 노출과 가수 분해를 통해 단백질의 변성을 포함합니다.일부 섭취된 아미노산은 단백질 생합성을 위해 사용되는 반면, 다른 아미노산은 글루코스로 전환되거나 구연산 회로로 공급된다.이러한 단백질의 연료 사용은 특히 [88]근육에서 발견되는 생명 유지에 신체 자신의 단백질을 사용할 수 있게 해주기 때문에 기아 상황에서 특히 중요하다.
개나 고양이와 같은 동물에서 단백질은 모낭의 성장과 각화를 촉진하여 피부 [89]트러블의 발생 가능성을 감소시킴으로써 피부의 건강과 품질을 유지한다.질 나쁜 단백질은 소화되지 않은 상태에서 단백질이 대장에 도달하면 발효되어 황화수소 가스, 인돌, 스카톨을 [90]생성하기 때문에 개의 위장 건강에 대한 역할도 한다.개와 고양이는 식물보다 동물성 단백질을 더 잘 소화시키지만 피부, 깃털, 결합조직 [90]등 질 낮은 동물성 단백질은 제대로 소화되지 않는다.
「 」를 참조해 주세요.
레퍼런스
- ^ Thomas Bur Osborne (1909) :식물성 단백질은 archive.org에서 웨이백 머신에 보관된 2016-03-22 이력 페이지 1~6
- ^ Mulder GJ (1838). "Sur la composition de quelques substances animales". Bulletin des Sciences Physiques et Naturelles en Néerlande: 104.
- ^ Harold H (1951). "Origin of the Word 'Protein.'". Nature. 168 (4267): 244. Bibcode:1951Natur.168..244H. doi:10.1038/168244a0. PMID 14875059. S2CID 4271525.
- ^ a b c Perrett D (August 2007). "From 'protein' to the beginnings of clinical proteomics". Proteomics: Clinical Applications. 1 (8): 720–38. doi:10.1002/prca.200700525. PMID 21136729. S2CID 32843102.
- ^ 새 옥스퍼드 영어사전
- ^ Reynolds JA, Tanford C (2003). Nature's Robots: A History of Proteins (Oxford Paperbacks). New York, New York: Oxford University Press. p. 15. ISBN 978-0-19-860694-9.
- ^ 레이놀즈와 탠포드(2003).
- ^ Bischoff TL, Voit C (1860). Die Gesetze der Ernaehrung des Pflanzenfressers durch neue Untersuchungen festgestellt (in German). Leipzig, Heidelberg.
- ^ "Hofmeister, Franz". encyclopedia.com. Archived from the original on 5 April 2017. Retrieved 4 April 2017.
- ^ "Protein, section: Classification of protein". britannica.com. Archived from the original on 4 April 2017. Retrieved 4 April 2017.
- ^ Sumner JB (1926). "The isolation and crystallization of the enzyme urease. Preliminary paper" (PDF). Journal of Biological Chemistry. 69 (2): 435–41. doi:10.1016/S0021-9258(18)84560-4. Archived from the original on 2011-03-25. Retrieved 2011-01-16.
- ^ Pauling L, Corey RB (May 1951). "Atomic coordinates and structure factors for two helical configurations of polypeptide chains" (PDF). Proceedings of the National Academy of Sciences of the United States of America. 37 (5): 235–40. Bibcode:1951PNAS...37..235P. doi:10.1073/pnas.37.5.235. PMC 1063348. PMID 14834145. Archived (PDF) from the original on 2012-11-28. Retrieved 2009-04-14.
- ^ Kauzmann W (May 1956). "Structural factors in protein denaturation". Journal of Cellular Physiology. 47 (Suppl 1): 113–31. doi:10.1002/jcp.1030470410. PMID 13332017.
- ^ Kauzmann W (1959). "Some factors in the interpretation of protein denaturation". Advances in Protein Chemistry Volume 14. Advances in Protein Chemistry. Vol. 14. pp. 1–63. doi:10.1016/S0065-3233(08)60608-7. ISBN 978-0-12-034214-3. PMID 14404936.
- ^ Kalman SM, Linderstrøm-Lang K, Ottesen M, Richards FM (February 1955). "Degradation of ribonuclease by subtilisin". Biochimica et Biophysica Acta. 16 (2): 297–99. doi:10.1016/0006-3002(55)90224-9. PMID 14363272.
- ^ Sanger F (1949). "The terminal peptides of insulin". The Biochemical Journal. 45 (5): 563–74. doi:10.1042/bj0450563. PMC 1275055. PMID 15396627.
- ^ Sanger F. (1958), Nobel lecture: The chemistry of insulin (PDF), Nobelprize.org, archived (PDF) from the original on 2013-03-19, retrieved 2016-02-09
- ^ a b c Stoddart, Charlotte (1 March 2022). "Structural biology: How proteins got their close-up". Knowable Magazine. doi:10.1146/knowable-022822-1. Retrieved 25 March 2022.
- ^ Muirhead H, Perutz MF (August 1963). "Structure of hemoglobin. A three-dimensional fourier synthesis of reduced human hemoglobin at 5.5 Å resolution". Nature. 199 (4894): 633–38. Bibcode:1963Natur.199..633M. doi:10.1038/199633a0. PMID 14074546. S2CID 4257461.
- ^ Kendrew JC, Bodo G, Dintzis HM, Parrish RG, Wyckoff H, Phillips DC (March 1958). "A three-dimensional model of the myoglobin molecule obtained by x-ray analysis". Nature. 181 (4610): 662–66. Bibcode:1958Natur.181..662K. doi:10.1038/181662a0. PMID 13517261. S2CID 4162786.
- ^ Zhou ZH (April 2008). "Towards atomic resolution structural determination by single-particle cryo-electron microscopy". Current Opinion in Structural Biology. 18 (2): 218–28. doi:10.1016/j.sbi.2008.03.004. PMC 2714865. PMID 18403197.
- ^ Keskin O, Tuncbag N, Gursoy A (April 2008). "Characterization and prediction of protein interfaces to infer protein-protein interaction networks". Current Pharmaceutical Biotechnology. 9 (2): 67–76. doi:10.2174/138920108783955191. hdl:11511/32640. PMID 18393863.
- ^ "RCSB Protein Data Bank". Archived from the original on 2015-04-18. Retrieved 2017-01-19.
- ^ Nelson DL, Cox MM (2005). Lehninger's Principles of Biochemistry (4th ed.). New York, New York: W. H. Freeman and Company.
- ^ Gutteridge A, Thornton JM (November 2005). "Understanding nature's catalytic toolkit". Trends in Biochemical Sciences. 30 (11): 622–29. doi:10.1016/j.tibs.2005.09.006. PMID 16214343.
- ^ a b c d e f g Murray RF, Harper HW, Granner DK, Mayes PA, Rodwell VW (2006). Harper's Illustrated Biochemistry. New York: Lange Medical Books/McGraw-Hill. ISBN 978-0-07-146197-9.
- ^ a b c Lodish H, Berk A, Matsudaira P, Kaiser CA, Krieger M, Scott MP, Zipurksy SL, Darnell J (2004). Molecular Cell Biology (5th ed.). New York, New York: WH Freeman and Company.
- ^ Ardejani MS, Powers ET, Kelly JW (August 2017). "Using Cooperatively Folded Peptides To Measure Interaction Energies and Conformational Propensities". Accounts of Chemical Research. 50 (8): 1875–1882. doi:10.1021/acs.accounts.7b00195. PMC 5584629. PMID 28723063.
- ^ a b c d Branden C, Tooze J (1999). Introduction to Protein Structure. New York: Garland Pub. ISBN 978-0-8153-2305-1.
- ^ a b c d e f g h i j Van Holde KE, Mathews CK (1996). Biochemistry. Menlo Park, California: Benjamin/Cummings Pub. Co., Inc. ISBN 978-0-8053-3931-4.
- ^ Milo R (December 2013). "What is the total number of protein molecules per cell volume? A call to rethink some published values". BioEssays. 35 (12): 1050–55. doi:10.1002/bies.201300066. PMC 3910158. PMID 24114984.
- ^ Beck M, Schmidt A, Malmstroem J, Claassen M, Ori A, Szymborska A, Herzog F, Rinner O, Ellenberg J, Aebersold R (November 2011). "The quantitative proteome of a human cell line". Molecular Systems Biology. 7: 549. doi:10.1038/msb.2011.82. PMC 3261713. PMID 22068332.
- ^ Wu L, Candille SI, Choi Y, Xie D, Jiang L, Li-Pook-Than J, Tang H, Snyder M (July 2013). "Variation and genetic control of protein abundance in humans". Nature. 499 (7456): 79–82. Bibcode:2013Natur.499...79W. doi:10.1038/nature12223. PMC 3789121. PMID 23676674.
- ^ Dobson CM (2000). "The nature and significance of protein folding". In Pain RH (ed.). Mechanisms of Protein Folding. Oxford, Oxfordshire: Oxford University Press. pp. 1–28. ISBN 978-0-19-963789-8.
- ^ Kozlowski LP (January 2017). "Proteome-pI: proteome isoelectric point database". Nucleic Acids Research. 45 (D1): D1112–D1116. doi:10.1093/nar/gkw978. PMC 5210655. PMID 27789699.
- ^ Fulton AB, Isaacs WB (April 1991). "Titin, a huge, elastic sarcomeric protein with a probable role in morphogenesis". BioEssays. 13 (4): 157–61. doi:10.1002/bies.950130403. PMID 1859393. S2CID 20237314.
- ^ Bruckdorfer T, Marder O, Albericio F (February 2004). "From production of peptides in milligram amounts for research to multi-tons quantities for drugs of the future". Current Pharmaceutical Biotechnology. 5 (1): 29–43. doi:10.2174/1389201043489620. PMID 14965208.
- ^ Schwarzer D, Cole PA (December 2005). "Protein semisynthesis and expressed protein ligation: chasing a protein's tail". Current Opinion in Chemical Biology. 9 (6): 561–69. doi:10.1016/j.cbpa.2005.09.018. PMID 16226484.
- ^ Kent SB (February 2009). "Total chemical synthesis of proteins". Chemical Society Reviews. 38 (2): 338–51. doi:10.1039/b700141j. PMID 19169452. S2CID 5432012.
- ^ Fernández A, Scott R (September 2003). "Dehydron: a structurally encoded signal for protein interaction". Biophysical Journal. 85 (3): 1914–28. Bibcode:2003BpJ....85.1914F. doi:10.1016/S0006-3495(03)74619-0. PMC 1303363. PMID 12944304.
- ^ Davey NE, Van Roey K, Weatheritt RJ, Toedt G, Uyar B, Altenberg B, Budd A, Diella F, Dinkel H, Gibson TJ (January 2012). "Attributes of short linear motifs". Molecular BioSystems. 8 (1): 268–81. doi:10.1039/c1mb05231d. PMID 21909575.
- ^ a b Voet D, Voet JG. (2004년)생화학 제1권 제3판호보켄, 뉴저지
- ^ Sankaranarayanan R, Moras D (2001). "The fidelity of the translation of the genetic code". Acta Biochimica Polonica. 48 (2): 323–35. doi:10.18388/abp.2001_3918. PMID 11732604.
- ^ Copland JA, Sheffield-Moore M, Koldzic-Zivanovic N, Gentry S, Lamprou G, Tzortzatou-Stathopoulou F, Zoumpourlis V, Urban RJ, Vlahopoulos SA (June 2009). "Sex steroid receptors in skeletal differentiation and epithelial neoplasia: is tissue-specific intervention possible?". BioEssays. 31 (6): 629–41. doi:10.1002/bies.200800138. PMID 19382224. S2CID 205469320.
- ^ Samarin S, Nusrat A (January 2009). "Regulation of epithelial apical junctional complex by Rho family GTPases". Frontiers in Bioscience. 14 (14): 1129–42. doi:10.2741/3298. PMID 19273120.
- ^ Bairoch A (January 2000). "The ENZYME database in 2000" (PDF). Nucleic Acids Research. 28 (1): 304–05. doi:10.1093/nar/28.1.304. PMC 102465. PMID 10592255. Archived from the original (PDF) on June 1, 2011.
- ^ Radzicka A, Wolfenden R (January 1995). "A proficient enzyme". Science. 267 (5194): 90–3. Bibcode:1995Sci...267...90R. doi:10.1126/science.7809611. PMID 7809611.
- ^ EBI External Services (2010-01-20). "The Catalytic Site Atlas at The European Bioinformatics Institute". Ebi.ac.uk. Archived from the original on 2013-08-03. Retrieved 2011-01-16.
- ^ Pickel B, Schaller A (October 2013). "Dirigent proteins: molecular characteristics and potential biotechnological applications". Applied Microbiology and Biotechnology. 97 (19): 8427–38. doi:10.1007/s00253-013-5167-4. PMID 23989917. S2CID 1896003.
- ^ Rüdiger H, Siebert HC, Solís D, Jiménez-Barbero J, Romero A, von der Lieth CW, Diaz-Mariño T, Gabius HJ (April 2000). "Medicinal chemistry based on the sugar code: fundamentals of lectinology and experimental strategies with lectins as targets". Current Medicinal Chemistry. 7 (4): 389–416. doi:10.2174/0929867003375164. PMID 10702616.
- ^ Mulder NJ (2007-09-28). "Protein Family Databases". eLS. Chichester, UK: John Wiley & Sons, Ltd. pp. a0003058.pub2. doi:10.1002/9780470015902.a0003058.pub2. ISBN 978-0-470-01617-6.
- ^ Sisu C, Pei B, Leng J, Frankish A, Zhang Y, Balasubramanian S, et al. (September 2014). "Comparative analysis of pseudogenes across three phyla". Proceedings of the National Academy of Sciences of the United States of America. 111 (37): 13361–6. Bibcode:2014PNAS..11113361S. doi:10.1073/pnas.1407293111. PMC 4169933. PMID 25157146.
- ^ a b Guzmán GI, Sandberg TE, LaCroix RA, Nyerges Á, Papp H, de Raad M, et al. (April 2019). "Enzyme promiscuity shapes adaptation to novel growth substrates". Molecular Systems Biology. 15 (4): e8462. doi:10.15252/msb.20188462. PMC 6452873. PMID 30962359.
- ^ Roohi, Bano K, Kuddus M, Zaheer MR, Zia Q, Khan MF, Ashraf GM, Gupta A, Aliev G (2017). "Microbial Enzymatic Degradation of Biodegradable Plastics". Current Pharmaceutical Biotechnology. 18 (5): 429–440. doi:10.2174/1389201018666170523165742. PMID 28545359.
- ^ Hey J, Posch A, Cohen A, Liu N, Harbers A (2008). "Fractionation of complex protein mixtures by liquid-phase isoelectric focusing". 2D PAGE: Sample Preparation and Fractionation. Methods in Molecular Biology. Vol. 424. pp. 225–39. doi:10.1007/978-1-60327-064-9_19. ISBN 978-1-58829-722-8. PMID 18369866.
- ^ Terpe K (January 2003). "Overview of tag protein fusions: from molecular and biochemical fundamentals to commercial systems". Applied Microbiology and Biotechnology. 60 (5): 523–33. doi:10.1007/s00253-002-1158-6. PMID 12536251. S2CID 206934268.
- ^ Stepanenko OV, Verkhusha VV, Kuznetsova IM, Uversky VN, Turoverov KK (August 2008). "Fluorescent proteins as biomarkers and biosensors: throwing color lights on molecular and cellular processes". Current Protein & Peptide Science. 9 (4): 338–69. doi:10.2174/138920308785132668. PMC 2904242. PMID 18691124.
- ^ Yuste R (December 2005). "Fluorescence microscopy today". Nature Methods. 2 (12): 902–4. doi:10.1038/nmeth1205-902. PMID 16299474. S2CID 205418407.
- ^ Margolin W (January 2000). "Green fluorescent protein as a reporter for macromolecular localization in bacterial cells". Methods. 20 (1): 62–72. doi:10.1006/meth.1999.0906. PMID 10610805.
- ^ Walker JH, Wilson K (2000). Principles and Techniques of Practical Biochemistry. Cambridge, UK: Cambridge University Press. pp. 287–89. ISBN 978-0-521-65873-7.
- ^ Mayhew TM, Lucocq JM (August 2008). "Developments in cell biology for quantitative immunoelectron microscopy based on thin sections: a review". Histochemistry and Cell Biology. 130 (2): 299–313. doi:10.1007/s00418-008-0451-6. PMC 2491712. PMID 18553098.
- ^ Hohsaka T, Sisido M (December 2002). "Incorporation of non-natural amino acids into proteins". Current Opinion in Chemical Biology. 6 (6): 809–15. doi:10.1016/S1367-5931(02)00376-9. PMID 12470735.
- ^ Cedrone F, Ménez A, Quéméneur E (August 2000). "Tailoring new enzyme functions by rational redesign". Current Opinion in Structural Biology. 10 (4): 405–10. doi:10.1016/S0959-440X(00)00106-8. PMID 10981626.
- ^ Görg A, Weiss W, Dunn MJ (December 2004). "Current two-dimensional electrophoresis technology for proteomics". Proteomics. 4 (12): 3665–85. doi:10.1002/pmic.200401031. PMID 15543535. S2CID 28594824.
- ^ Conrotto P, Souchelnytskyi S (September 2008). "Proteomic approaches in biological and medical sciences: principles and applications". Experimental Oncology. 30 (3): 171–80. PMID 18806738.
- ^ Koegl M, Uetz P (December 2007). "Improving yeast two-hybrid screening systems". Briefings in Functional Genomics & Proteomics. 6 (4): 302–12. doi:10.1093/bfgp/elm035. PMID 18218650. Archived from the original on 2017-09-11. Retrieved 2017-07-23.
- ^ Plewczyński D, Ginalski K (2009). "The interactome: predicting the protein-protein interactions in cells". Cellular & Molecular Biology Letters. 14 (1): 1–22. doi:10.2478/s11658-008-0024-7. PMC 6275871. PMID 18839074.
- ^ Zhang C, Kim SH (February 2003). "Overview of structural genomics: from structure to function". Current Opinion in Chemical Biology. 7 (1): 28–32. doi:10.1016/S1367-5931(02)00015-7. PMID 12547423. Archived from the original on 2018-11-19. Retrieved 2019-06-29.
- ^ Gonen T, Cheng Y, Sliz P, Hiroaki Y, Fujiyoshi Y, Harrison SC, Walz T (December 2005). "Lipid-protein interactions in double-layered two-dimensional AQP0 crystals". Nature. 438 (7068): 633–38. Bibcode:2005Natur.438..633G. doi:10.1038/nature04321. PMC 1350984. PMID 16319884.
- ^ Standley DM, Kinjo AR, Kinoshita K, Nakamura H (July 2008). "Protein structure databases with new web services for structural biology and biomedical research". Briefings in Bioinformatics. 9 (4): 276–85. doi:10.1093/bib/bbn015. PMID 18430752.
- ^ Walian P, Cross TA, Jap BK (2004). "Structural genomics of membrane proteins". Genome Biology. 5 (4): 215. doi:10.1186/gb-2004-5-4-215. PMC 395774. PMID 15059248.
- ^ Sleator RD (2012). "Prediction of protein functions". Functional Genomics. Methods in Molecular Biology. Vol. 815. pp. 15–24. doi:10.1007/978-1-61779-424-7_2. ISBN 978-1-61779-423-0. PMID 22130980.
- ^ Zhang Y (June 2008). "Progress and challenges in protein structure prediction". Current Opinion in Structural Biology. 18 (3): 342–48. doi:10.1016/j.sbi.2008.02.004. PMC 2680823. PMID 18436442.
- ^ Xiang Z (June 2006). "Advances in homology protein structure modeling". Current Protein & Peptide Science. 7 (3): 217–27. doi:10.2174/138920306777452312. PMC 1839925. PMID 16787261.
- ^ Zhang Y, Skolnick J (January 2005). "The protein structure prediction problem could be solved using the current PDB library". Proceedings of the National Academy of Sciences of the United States of America. 102 (4): 1029–34. Bibcode:2005PNAS..102.1029Z. doi:10.1073/pnas.0407152101. PMC 545829. PMID 15653774.
- ^ Kuhlman B, Dantas G, Ireton GC, Varani G, Stoddard BL, Baker D (November 2003). "Design of a novel globular protein fold with atomic-level accuracy". Science. 302 (5649): 1364–68. Bibcode:2003Sci...302.1364K. doi:10.1126/science.1089427. PMID 14631033. S2CID 1939390.
- ^ Ward JJ, Sodhi JS, McGuffin LJ, Buxton BF, Jones DT (March 2004). "Prediction and functional analysis of native disorder in proteins from the three kingdoms of life". Journal of Molecular Biology. 337 (3): 635–45. CiteSeerX 10.1.1.120.5605. doi:10.1016/j.jmb.2004.02.002. PMID 15019783.
- ^ Tompa P, Fersht A (18 November 2009). Structure and Function of Intrinsically Disordered Proteins. CRC Press. ISBN 978-1-4200-7893-0. Archived from the original on 19 April 2017. Retrieved 19 October 2016.
- ^ Ritchie DW (February 2008). "Recent progress and future directions in protein-protein docking". Current Protein & Peptide Science. 9 (1): 1–15. CiteSeerX 10.1.1.211.4946. doi:10.2174/138920308783565741. PMID 18336319.
- ^ Zagrovic B, Snow CD, Shirts MR, Pande VS (November 2002). "Simulation of folding of a small alpha-helical protein in atomistic detail using worldwide-distributed computing". Journal of Molecular Biology. 323 (5): 927–37. CiteSeerX 10.1.1.142.8664. doi:10.1016/S0022-2836(02)00997-X. PMID 12417204.
- ^ Herges T, Wenzel W (January 2005). "In silico folding of a three helix protein and characterization of its free-energy landscape in an all-atom force field". Physical Review Letters. 94 (1): 018101. arXiv:physics/0310146. Bibcode:2005PhRvL..94a8101H. doi:10.1103/PhysRevLett.94.018101. PMID 15698135. S2CID 1477100.
- ^ Hoffmann M, Wanko M, Strodel P, König PH, Frauenheim T, Schulten K, Thiel W, Tajkhorshid E, Elstner M (August 2006). "Color tuning in rhodopsins: the mechanism for the spectral shift between bacteriorhodopsin and sensory rhodopsin II". Journal of the American Chemical Society. 128 (33): 10808–18. doi:10.1021/ja062082i. PMID 16910676.
- ^ Mendive-Tapia D, Mangaud E, Firmino T, de la Lande A, Desouter-Lecomte M, Meyer HD, Gatti F (2018). "Multidimensional Quantum Mechanical Modeling of Electron Transfer and Electronic Coherence in Plant Cryptochromes: The Role of Initial Bath Conditions". J. Phys. Chem. B. 122 (1): 126–136. doi:10.1021/acs.jpcb.7b10412. PMID 29216421.
- ^ Strümpfer J, Schulten K (2012). "Open Quantum Dynamics Calculations with the Hierarchy Equations of Motion on Parallel Computers". J. Chem. Theory Comput. 8 (8): 2808–2816. doi:10.1021/ct3003833. PMC 3480185. PMID 23105920.
- ^ Scheraga HA, Khalili M, Liwo A (2007). "Protein-folding dynamics: overview of molecular simulation techniques". Annual Review of Physical Chemistry. 58: 57–83. Bibcode:2007ARPC...58...57S. doi:10.1146/annurev.physchem.58.032806.104614. PMID 17034338.
- ^ Muñoz-Huerta, Rafael F.; Guevara-Gonzalez, Ramon G.; Contreras-Medina, Luis M.; Torres-Pacheco, Irineo; Prado-Olivarez, Juan; Ocampo-Velazquez, Rosalia V. (Aug 16, 2013). "A Review of Methods for Sensing the Nitrogen Status in Plants: Advantages, Disadvantages and Recent Advances". Sensors (Basel, Switzerland). 13 (8): 10823–10843. Bibcode:2013Senso..1310823M. doi:10.3390/s130810823. PMC 3812630. PMID 23959242.
- ^ Martin, P D; Malley, D F; Manning, G.; Fuller, L. (Nov 1, 2002). "Determination of soil organic carbon and nitrogen at the field level using near-infrared spectroscopy". Canadian Journal of Soil Science. 82 (4): 413–422. doi:10.4141/S01-054 – via DOI.org (Crossref).
- ^ Brosnan JT (June 2003). "Interorgan amino acid transport and its regulation". The Journal of Nutrition. 133 (6 Suppl 1): 2068S–72S. doi:10.1093/jn/133.6.2068S. PMID 12771367.
- ^ Watson TD (1998). "Diet and skin disease in dogs and cats". The Journal of Nutrition. 128 (12 Suppl): 2783S–89S. doi:10.1093/jn/128.12.2783S. PMID 9868266.
- ^ a b Case LP, Daristotle L, Hayek MG, Raasch MF (2010). Canine and Feline Nutrition-E-Book: A Resource for Companion Animal Professionals. Elsevier Health Sciences.
추가 정보
- 교재
- Branden C, Tooze J (1999). Introduction to Protein Structure. New York: Garland Pub. ISBN 978-0-8153-2305-1.
- Murray RF, Harper HW, Granner DK, Mayes PA, Rodwell VW (2006). Harper's Illustrated Biochemistry. New York: Lange Medical Books/McGraw-Hill. ISBN 978-0-07-146197-9.
- Van Holde KE, Mathews CK (1996). Biochemistry. Menlo Park, California: Benjamin/Cummings Pub. Co., Inc. ISBN 978-0-8053-3931-4.
외부 링크
데이터베이스 및 프로젝트
- NCBI Entrez 단백질 데이터베이스
- NCBI 단백질 구조 데이터베이스
- 인간단백질참조데이터베이스
- 인간단백질미디어
- 접기@홈(Stanford University)
- 유럽의 단백질 데이터뱅크(PDBeQuips, 흥미로운 PDB 구조에 대한 짧은 기사 및 튜토리얼 참조)
- 구조 생물 정보학을 위한 Research Collaboratory (PDB의 선택된 단백질에 대한 짧은 설명으로 Wayback Machine에서 보관된 2020-07-24 이달의 분자 참조)
- Proteopedia – Life in 3D: 모든 알려진 단백질 분자 구조에 대한 Wiki 주석이 있는 회전 및 줌 가능한 3D 모델.
- UniProt 범용 단백질 자원
튜토리얼 및 교육용 웹사이트
- HOPERS(Stanford 교육을 위한 Huntington's Disease Outreach Project)의 '단백질 소개'
- 단백질:바이오제네이션에서 열화로 – 생화학 및 세포생물학의 가상 라이브러리