진화
Evolution| 생물학 시리즈의 일부 |
| 진화 |
|---|
 |
| 메커니즘 및 프로세스 |
| 연구와 역사 |
| 진화생물학 분야 |
생물학에서 진화는 생물학적 집단의 유전적 특성이 연속적인 세대에 걸쳐 변화하는 것입니다.[1][2]진화는 자연 선택(성적 선택 포함) 및 유전적 이동과 같은 진화 과정이 유전적 변이에 영향을 미쳐 특정 특성이 연속 세대에 걸쳐 인구 내에서 어느 정도 일반화될 때 발생합니다.[3]진화의 과정은 생물학적 조직의 모든 단계에서 생물학적 다양성을 낳았습니다.[4][5]
자연선택에 의한 진화론은 19세기 중반 찰스 다윈과 알프레드 러셀 월리스에 의해 독립적으로 생물체가 물리적, 생물학적 환경에 적응하는 이유에 대한 설명으로 고안되었습니다.이 이론은 다윈의 '종의 기원'이라는 책에서 처음 자세히 설명되었습니다.[6]자연 선택에 의한 진화는 생물체에 대한 관찰 가능한 사실에 의해 확립됩니다. (1) 더 많은 자손이 종종 생존할 수 있는 것보다 더 많이 생산됩니다. (2) 그들의 형태, 생리학, 그리고 개인에 따라 특성이 다릅니다.그리고 행동; (3) 다른 특성들이 다른 생존율과 재생산율을 부여합니다 (차등적 적합성); (4) 특성들이 세대에서 세대로 전달될 수 있습니다 (적합성의 herit성).따라서 연속적인 세대에서, 모집단의 구성원은 해당 환경에 유리한 특성을 가진 부모의 자손으로 대체될 가능성이 더 높습니다.
20세기 초, 경쟁적인 진화론은 반박되었고 진화론은 멘델의 유전과 인구 유전학과 결합되어 현대 진화론을 낳았습니다.[8]이 합성에서 유전의 기초는 세대에서 세대로 정보를 전달하는 DNA 분자에 있습니다.집단에서 DNA를 변화시키는 과정은 자연선택, 유전적 이동, 돌연변이, 그리고 유전자 흐름을 포함합니다.[3]
인류를 포함한 지구상의 모든 생명체는 약 35억년에서 38억년 전에 살았던 마지막 보편적 공통 조상(LUCA)을 공유하고 있습니다.[9][10][11][12]화석 기록에는 초기 생합성 흑연에서[13] 미생물 매트 화석으로[14][15][16], 화석화된 다세포 생물로의 진행이 포함되어 있습니다.생물다양성의 기존 패턴은 지구 생명체의 진화 역사를 통해 새로운 종의 반복적인 형성(종종), 종 내의 변화(아나제네시스), 종의 손실(멸종)에 의해 형성되었습니다.[17]형태학적, 생화학적 특징은 더 최근의 공통 조상을 공유하는 종들 사이에서 더 유사한 경향이 있는데, 역사적으로 계통수를 재구성하는 데 사용되었지만, 유전자 서열의 직접적인 비교는 오늘날 더 일반적인 방법입니다.[18][19]
진화생물학자들은 현장이나 실험실의 증거와 수학적이고 이론적인 생물학의 방법에 의해 생성된 데이터에 근거하여 이론을 구성할 뿐만 아니라 가설을 형성하고 시험함으로써 진화의 다양한 측면을 계속 연구해 왔습니다.그들의 발견은 생물학의 발전뿐만 아니라 농업, 의학, 컴퓨터 과학을 포함한 다른 분야에도 영향을 미쳤습니다.[20]
세습
유기체의 진화는 유전적 특성인 유기체의 유전적 특성의 변화를 통해 일어납니다.예를 들어, 인간의 경우, 눈 색깔은 유전되는 특성이고 개인은 부모 중 한 명으로부터 "갈색 눈의 특성"을 물려받을 수 있습니다.[21]유전된 형질은 유전자에 의해 통제되고 한 유기체의 게놈 (유전 물질) 안에 있는 완전한 유전자 집합은 그것의 유전자형이라고 불립니다.[22]
유기체의 구조와 행동을 구성하는 관찰 가능한 특성의 완전한 집합을 표현형이라고 합니다.이러한 특성 중 일부는 유전자형이 환경과 상호작용하는 반면 다른 것들은 중립적이기 때문입니다.[23]일부 관측 가능한 특성은 상속되지 않습니다.예를 들어, 햇볕에 그을린 피부는 사람의 유전자형과 햇빛 사이의 상호작용에서 비롯되므로, 선탠은 사람들의 아이들에게 전해지지 않습니다.표현형은 햇빛에 노출되었을 때 피부가 그을리는 능력입니다.그러나 유전자형 변이의 차이로 인해 어떤 사람들은 다른 사람들보다 더 쉽게 태웁니다; 두드러진 예는 전혀 태닝을 하지 않고 햇볕에 매우 민감한 알비니즘의 유전적 특성을 가진 사람들입니다.[24]
유전적 특성은 유전 정보를 암호화하는 분자인 DNA를 통해 한 세대에서 다음 세대로 전달됩니다.[22]DNA는 네 가지 종류의 염기로 구성된 긴 생물 중합체입니다.특정 DNA 분자에 따른 염기서열은 문장의 철자를 쓰는 글자열과 유사한 방식으로 유전 정보를 지정합니다.세포가 분열하기 전에, DNA는 복사되고, 그 결과로 만들어진 두 개의 세포는 각각 DNA 서열을 물려받게 됩니다.하나의 기능적인 단위를 특정하는 DNA 분자의 부분들은 유전자라고 불립니다; 다른 유전자들은 다른 염기서열을 가지고 있습니다.세포 안에서, DNA의 각 긴 가닥은 염색체라고 불립니다.염색체 내의 DNA 염기서열의 특정한 위치는 로커스(locus.만약 위치에 있는 DNA 염기서열이 개인들 사이에서 다양하다면, 이 염기서열의 다른 형태는 대립유전자라고 불립니다.DNA 염기서열은 새로운 대립유전자를 생성하면서 돌연변이를 통해 변화할 수 있습니다.만약 유전자 안에서 돌연변이가 발생한다면, 새로운 대립유전자는 유전자가 통제하는 특성에 영향을 미쳐 유기체의 표현형을 바꿀 수 있습니다.[25]그러나, 대립유전자와 형질 간의 단순한 일치는 어떤 경우에는 효과가 있지만, 대부분의 형질은 양적 또는 서파적인 방식으로 여러 유전자에 의해 영향을 받습니다.[26][27]
변이원
진화는 개체 내에 유전적 변이가 있을 경우 일어날 수 있습니다.변이는 유전체의 돌연변이, 성적 생식을 통한 유전자의 재편성, 집단 간의 이동(유전자 흐름)에서 비롯됩니다.돌연변이와 유전자 흐름을 통한 새로운 변이의 지속적인 도입에도 불구하고, 한 종의 게놈의 대부분은 그 종의 모든 개체들 사이에서 매우 비슷합니다.[28]하지만, 진화 발달 생물학 분야에서의 발견은 유전자형의 비교적 작은 차이라도 종 내와 종 간 표현형의 극적인 차이로 이어질 수 있다는 것을 보여주었습니다.
개별 유기체의 표현형은 그것의 유전자형과 그것이 살아왔던 환경의 영향 모두에서 비롯됩니다.[27]현대의 진화론적 종합은 진화를 이러한 유전적 변이의 시간에 따른 변화로 정의합니다.특정한 대립유전자의 빈도는 그 유전자의 다른 형태에 비해 다소 널리 퍼질 것입니다.변동은 새로운 대립 유전자가 고정 지점에 도달하면 사라집니다. 즉, 모집단에서 사라지거나 조상 대립 유전자를 완전히 대체합니다.[29]
돌연변이

돌연변이는 세포의 게놈의 DNA 서열의 변화이며 모든 유기체에서 유전적 변이의 궁극적인 원천입니다.[30]돌연변이가 일어나면, 유전자의 생성물을 바꾸거나, 유전자의 기능을 방해하거나, 영향을 미치지 않을 수 있습니다.
단백질을 코딩하는 유전자의 코딩 영역에 있는 돌연변이의 약 절반은 유해하고 나머지 절반은 중성입니다.이 지역의 전체 돌연변이 중 적은 비율이 적합성 이점을 제공합니다.[31]게놈의 다른 부분의 돌연변이 중 일부는 유해하지만 대부분은 중성입니다.몇 가지가 도움이 됩니다.
돌연변이는 염색체의 큰 부분이 복제되는 것을 수반할 수 있으며(보통 유전자 재조합에 의해), 유전자의 추가 복사본을 게놈에 도입할 수 있습니다.[32]여분의 유전자 복제물은 새로운 유전자가 진화하는 데 필요한 원료의 주요 원천입니다.[33]이것은 대부분의 새로운 유전자들이 공통된 조상을 공유하는 기존의 유전자들로부터 유전자 가족 내에서 진화하기 때문에 중요합니다.[34]예를 들어, 인간의 눈은 빛을 감지하는 구조를 만들기 위해 3개의 유전자와 1개의 야간 시력을 위해 4개의 유전자를 사용합니다; 4개의 유전자 모두 하나는 모두 하나의 조상 유전자의 후손입니다.[35]
복제된 유전자가 변형되어 새로운 기능을 획득할 때 조상의 유전자로부터 새로운 유전자가 생성될 수 있습니다.한 유전자가 복제되면 시스템의 중복성이 증가하기 때문에 이 과정이 더 쉬워집니다. 한 쌍의 유전자는 다른 유전자가 원래 기능을 계속 수행하는 동안 새로운 기능을 획득할 수 있습니다.[36][37]다른 종류의 돌연변이는 심지어 이전에 코딩되지 않은 DNA로부터 완전히 새로운 유전자를 만들어낼 수 있는데, 이것은 de novo 유전자 탄생이라고 불리는 현상입니다.[38][39]
새로운 유전자의 생성은 또한 여러 유전자들의 작은 부분들이 복제되는 것을 포함할 수 있고, 이 조각들은 새로운 기능을 가진 새로운 조합을 형성하기 위해 재조합됩니다(exon shuffling).[40][41]새로운 유전자가 기존의 부품들을 섞는 것으로부터 조립될 때, 도메인들은 단순한 독립적인 기능을 가진 모듈로서 작용하고, 이것들은 새로운 기능과 복잡한 기능을 가진 새로운 조합을 생산하기 위해 함께 혼합될 수 있습니다.[42]예를 들어, 폴리케타이드 합성효소는 항생제를 만드는 큰 효소입니다. 그들은 조립 라인의 단계와 같이 전체 공정에서 각각 한 단계씩 촉매 작용하는 최대 100개의 독립적인 도메인을 포함합니다.[43]
돌연변이의 한 예는 멧돼지 돼지입니다.그들은 위장색을 띄고 있으며 어둡고 가벼운 세로 줄무늬의 특징적인 패턴을 보여줍니다.그러나 멜라노코르틴 1 수용체(MC1R)의 돌연변이는 패턴을 방해합니다.돼지 품종의 대부분은 야생형 색을 방해하는 MC1R 돌연변이와 검은 색을 지배하는 다른 돌연변이를 가지고 있습니다.[44]
섹스와 재조합
무성 생물에서 유전자는 함께 유전되거나 연결되어 있는데, 이는 번식하는 동안 다른 생물의 유전자와 섞일 수 없기 때문입니다.이와는 대조적으로, 성적 유기체의 자손은 독립적인 분류를 통해 생성된 부모의 염색체의 무작위 혼합물을 포함합니다.상동 재조합이라고 불리는 관련 과정에서, 성적 유기체들은 두 개의 일치하는 염색체 사이에서 DNA를 교환합니다.[45]재조합과 재분류는 대립유전자의 빈도를 변화시키지 않고, 대신 어떤 대립유전자가 서로 연관되어 있는지를 변화시켜 새로운 대립유전자 조합을 가진 자손을 생산합니다.[46]성은 보통 유전적 변이를 증가시키고 진화 속도를 증가시킬 수 있습니다.[47][48]
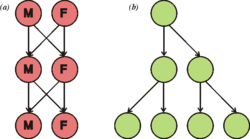
두 배의 성관계 비용은 John Maynard Smith에 의해 처음 묘사되었습니다.[49]첫 번째 비용은 성적 이형성 종에서 두 성별 중 한 명만이 어린 아이를 가질 수 있다는 것입니다.이 비용은 대부분의 식물과 많은 무척추동물처럼 자양동물 종에는 적용되지 않습니다.두 번째 비용은 성적으로 번식하는 모든 개체는 자신의 유전자의 50%만 자손에게 물려줄 수 있으며, 각 새로운 세대가 지나갈수록 더 적게 물려줍니다.[50]그러나 성 생식은 진핵생물과 다세포 생물 사이에서 더 일반적인 생식 수단입니다.레드 퀸 가설은 끊임없이 변화하는 환경에서 다른 종들과의 공진화에 대응하여 지속적인 진화와 적응을 가능하게 하는 수단으로서 성 생식의 중요성을 설명하기 위해 사용되어 왔습니다.[50][51][52][53]또 다른 가설은 성 생식이 주로 생식 DNA의 손상에 대한 정확한 재조합 복구를 촉진하기 위한 적응이며, 다양성의 증가는 때때로 적응적으로 도움이 될 수 있는 이 과정의 부산물이라는 것입니다.[54][55]
유전자 흐름
유전자 흐름은 집단과 종들 간의 유전자 교환입니다.[56]따라서 개체수나 종에 있어서는 새로운 변이의 근원이 될 수 있습니다.유전자 흐름은 내륙 개체군과 해안 개체군 사이의 쥐 이동 또는 중금속에 민감한 풀 개체군과 중금속에 민감한 개체군 사이의 꽃가루 이동에 의해 발생할 수 있습니다.
종간의 유전자 전달은 잡종 생물의 형성과 수평적인 유전자 전달을 포함합니다.수평적 유전자 전달은 한 유기체로부터 그 자손이 아닌 다른 유기체로 유전 물질을 전달하는 것입니다; 이것은 박테리아 중에서 가장 흔합니다.[57]의학에서, 이것은 항생제 내성의 확산에 기여하는데, 한 박테리아가 내성 유전자를 얻을 때 그것들을 다른 종으로 빠르게 옮길 수 있기 때문입니다.[58]효모 Saccharomyces cerevisiae 및 adzuki bean callosobruchus chinensis와 같은 진핵생물로 유전자의 수평적 전이가 발생했습니다.[59][60]더 큰 규모의 이동의 예로는 박테리아, 곰팡이 및 식물로부터 다양한 유전자를 받은 진핵생물 bdelloid 회전체가 있습니다.[61]바이러스는 생물학적 영역을 가로질러서도 유전자의 이동을 가능하게 하면서, 유기체들 사이에 DNA를 운반할 수도 있습니다.[62]
또한 엽록체와 미토콘드리아를 획득하는 동안 진핵세포와 박테리아의 조상 사이에 대규모 유전자 전달이 발생했습니다.진핵생물 자체가 박테리아와 고세균 사이의 수평적인 유전자 이동에서 비롯되었을 가능성이 있습니다.[63]
후생유전학
DNA의 뉴클레오티드 배열에 대한 변화로 일부 유전적인 변화를 설명할 수 없습니다.이러한 현상들은 후생유전학적 유전체계로 분류됩니다.[64]염색체를 표시하는 DNA 메틸화, 자생적 대사 루프, RNA 간섭에 의한 유전자 침묵, 단백질(프리온 등)의 3차원 입체 형태는 생물학적 수준에서 후생유전학적 유전 시스템이 발견된 분야입니다.[65]발달 생물학자들은 유전자 네트워크와 세포들 사이의 의사소통에서 복잡한 상호작용이 발달 가소성과 운하화의 일부 역학의 기초가 될 수 있는 유전적인 변화로 이어질 수 있다고 제안합니다.[66]유전성은 훨씬 더 큰 규모에서도 발생할 수 있습니다.예를 들어, 틈새 건설 과정을 통한 생태적 유산은 생물들이 그들의 환경에서 규칙적이고 반복적인 활동에 의해 정의됩니다.이는 후속 세대의 선택 체제를 수정하고 피드백하는 효과의 유산을 생성합니다.[67]유전자의 직접적인 통제 하에 있지 않은 진화에서의 유전성의 다른 예들은 문화적인 특성의 유전과 공생을 포함합니다.[68][69]
진화력

신다윈적 관점에서 보면, 진화는 교배 유기체 집단 내에서 대립 유전자의 빈도에 변화가 있을 때 발생합니다. 예를 들어 나방 집단의 검은 색에 대한 대립 유전자가 점점 더 일반화되고 있습니다.[70]모든 유전자 빈도의 변화를 초래할 수 있는 메커니즘으로는 자연선택, 유전자 이동, 돌연변이 편향 등이 있습니다.
자연선택
자연 선택에 의한 진화는 생존과 번식을 향상시키는 특성들이 인구의 연속적인 세대에서 더 흔해지는 과정입니다.세 가지 원칙을 구현합니다.[7]
- 형태학, 생리학, 행동학(현상학적 변이)과 관련하여 생물군 내에 변이가 존재합니다.
- 다른 특성들은 생존과 번식의 다른 비율을 제공합니다. (차등 적합).
- 이러한 특성은 대대로 전달될 수 있습니다.
생존할 수 있는 것보다 더 많은 자손이 생산되고, 이러한 조건들은 생존과 번식을 위한 유기체들 사이의 경쟁을 만듭니다.결과적으로, 경쟁자보다 유리한 특성을 가진 유기체는 유리하지 않은 특성을 가진 유기체보다 다음 세대에 자신의 특성을 물려줄 가능성이 더 높습니다.[71]이 텔레노미는 자연 선택 과정이 그들이 수행하는 기능적 역할에 적합해 보이는 특성을 생성하고 보존하는 품질입니다.[72]선택의 결과는 비임의 짝짓기와[73] 유전적 히치하이킹을 포함합니다.
자연 선택의 중심 개념은 유기체의 진화 적합성입니다.[74]체력은 다음 세대에 대한 유전적 기여의 크기를 결정하는 생물체의 생존 및 번식 능력에 의해 측정됩니다.[74]하지만, 건강은 자손의 총 숫자와 같지 않습니다: 대신 건강은 유기체의 유전자를 지니고 있는 다음 세대의 비율로 표시됩니다.[75]예를 들어, 한 유기체가 잘 생존하고 빠르게 번식할 수 있지만, 그 자손들이 모두 생존하기에는 너무 작고 약하다면, 이 유기체는 미래 세대에 유전적인 기여를 거의 하지 못할 것이고 따라서 적합성이 낮을 것입니다.[74]
대립 유전자가 그 유전자의 다른 대립 유전자보다 적합도를 더 높이면, 각 세대에 따라 이 대립 유전자가 집단 내에서 일반화될 확률이 더 높아집니다.이러한 특성들은 "선택된 것"이라고 합니다. 건강을 증진시킬 수 있는 특성들의 예들은 향상된 생존력과 증가된 다산입니다.반대로, 덜 유익하거나 해로운 대립 유전자를 가지고 있는 것으로 인해 발생하는 적합도가 낮을수록 이 대립 유전자는 더 희귀해질 가능성이 있습니다. 즉, 그들은 "반대로 선택됩니다.[76]
중요한 것은 대립유전자의 적합성이 고정된 특성이 아니라는 것입니다. 환경이 바뀌면 이전에 중성적이거나 해로운 특성이 유익해지고 이전에 유익한 특성이 해로울 수 있습니다.[25]그러나 이와 같이 선택의 방향이 반대로 된다고 하더라도 과거에 상실되었던 특성들이 동일한 형태로 재진화되지 않을 수 있습니다.[77][78]그러나 휴면 유전자가 게놈에서 제거되지 않고 수백 세대 동안만 억제되는 한, 휴면 유전자의 재활성화는 돌고래의 뒷다리, 닭의 이빨, 날개 없는 막대기 곤충의 날개, 꼬리 그리고 인간의 추가적인 젖꼭지와 같은 것으로 생각되는 형질의 재발생으로 이어질 수 있습니다.이와 같은 "되돌리기"는 발작이라고 알려져 있습니다.[79]

·그래프 1은 단일 극단 표현형을 선호하는 방향 선택을 보여줍니다.
·그래프 2는 안정화 선택을 설명하는데, 여기서 중간 표현형은 극단적인 특성보다 선호됩니다.
·그래프 3은 극단적 표현형이 중간체보다 선호되는 파괴적 선택을 보여줍니다.
키와 같은 값의 범위에 따라 달라질 수 있는 특성에 대한 모집단 내의 자연 선택은 세 가지 유형으로 분류될 수 있습니다.첫 번째는 방향성 선택으로, 시간이 지남에 따라 특성의 평균값이 변화하는 것입니다. 예를 들어, 유기체가 서서히 키가 커지는 것입니다.[80]둘째, 파괴적 선택은 극단적인 특성 값에 대한 선택이며 종종 두 개의 다른 값이 가장 일반적이 되며 평균 값에 대한 선택이 나타납니다.이것은 키가 작거나 큰 생물체가 이점이 있지만 중간 키의 생물체는 이점이 없을 때입니다.마지막으로, 선택 안정화에서는 양 끝의 극단적인 특성 값에 대한 선택이 있는데, 이는 평균값 주위의 분산을 감소시키고 더 적은 다양성을 야기합니다.[71][81]예를 들어, 이것은 유기체가 결국 비슷한 키를 갖게 할 것입니다.
자연 선택은 일반적으로 자연을 개인과 개인의 특성이 생존 가능성이 높은 척도로 만듭니다.이러한 의미에서 "자연"은 생태계, 즉 생물체가 그들의 지역 환경에서 물리적인 것뿐만 아니라 생물학적인 것까지 모든 다른 요소들과 상호작용하는 시스템을 말합니다.생태학의 창시자인 유진 오둠은 생태계를 다음과 같이 정의했습니다: "에너지의 흐름이 명확하게 정의된 영양 구조, 생물학적 다양성, 물질 순환(즉, 살아있는 부분과 그렇지 않은 부분 사이의 물질 교환)을 이끌도록 물리적 환경과 상호 작용하는 주어진 영역에 있는 모든 유기체를 포함하는 모든 단위"시스템..."[82] 생태계 내의 각 인구는 시스템의 다른 부분들과 서로 다른 관계를 가지고, 서로 다른 틈새 또는 위치를 차지합니다.이러한 관계는 유기체의 생명력, 먹이 사슬에서의 위치, 지리적 범위와 관련이 있습니다.자연에 대한 이러한 광범위한 이해는 과학자들이 함께 자연 선택을 구성하는 특정한 힘을 묘사할 수 있게 해줍니다.
자연 선택은 유전자, 세포, 개별 유기체, 유기체의 그룹, 종과 같은 다양한 수준의 조직에서 작용할 수 있습니다.[83][84][85]여러 수준에서 동시에 선택할 수 있습니다.[86]개별 유기체의 수준 이하에서 발생하는 선택의 예로는 유전자를 복제하고 게놈 전체에 퍼질 수 있는 트랜스포존이라고 불리는 유전자가 있습니다.[87]집단 선택과 같이 개인 이상의 수준에서의 선택은 협력의 진화를 가능하게 할 수 있습니다.[88]
유전적 이동
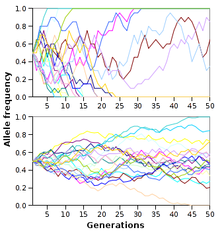
유전적 이동은 한 세대에서 다음 세대로 모집단 내의 모든 유전자 빈도의 무작위 변동입니다.[89]선택적 힘이 없거나 상대적으로 약한 경우, 대립유전자가 표본오차를 받기 때문에 각 연속 세대에서 대립유전자 주파수는 동일하게 위쪽 또는 아래쪽으로[clarification needed] 표류할 가능성이 있습니다.[90]모집단에서 사라지거나 다른 대립 유전자를 완전히 대체하여 대립 유전자가 최종적으로 고정되면 이 드리프트가 중단됩니다.따라서 유전적 이동은 우연 때문에 모집단에서 일부 대립유전자를 제거할 수 있습니다.선택적인 힘이 없을 때에도, 유전적인 이동은 같은 유전 구조로 시작하는 두 개의 분리된 집단이 다른 대립 유전자 집합을 가진 두 개의 다른 집단으로 이동하게 할 수 있습니다.[91]
중성 분자 진화 이론에 따르면 대부분의 진화적 변화는 유전적 이동에 의한 중성 돌연변이의 고정의 결과입니다.[92]이 모델에서, 모집단의 대부분의 유전적 변화는 일정한 돌연변이 압력과 유전적 이동의 결과입니다.[93]이러한 중립 이론의 형태는 자연에서 볼 수 있는 유전적인 변화에 맞지 않는 것으로 보이기 때문에 논의되어 왔습니다.[94][95]이 모델의 더 잘 뒷받침되는 버전은 거의 중립적인 이론으로, 적은 모집단에서 효과적으로 중립적인 돌연변이가 많은 모집단에서 반드시 중립적이지는 않습니다.[71]다른 이론들은 유전적 드리프트가 유전자 드래프트라고도 알려진 유전적 히치하이킹과 같은 진화의 다른 확률적 힘에 의해 왜소해진다고 제안합니다.[90][96][97]또 다른 개념은 건설적 중립 진화(CNE)인데, 이는 용량 과잉, 전제, 래칫의 원리로 인해 중립적 전환을 통해 복잡한 시스템이 출현하여 인구로 확산될 수 있다고 설명하고,[98][99][100]그리고 그것은 스플라이소좀의 기원에서부터 미생물 군집의 복잡한 상호 의존에 이르는 영역에 적용되어 왔습니다.[101][102][103]
유전적 이동에 의해 고정되기 위해 중성 대립자가 필요한 시간은 모집단 크기에 따라 다릅니다. 고정은 더 작은 모집단에서 더 빠릅니다.[104]모집단의 개체 수는 중요한 것이 아니라 유효 모집단 크기로 알려진 측도입니다.[105]유효인구는 일반적으로 교배정도와 인구가 가장 적은 수명주기 단계 등의 요인을 고려하기 때문에 총인구보다 적습니다.[105]유효 모집단 크기가 동일한 모집단의 모든 유전자에 대해 동일하지 않을 수 있습니다.[106]
드리프트를 포함한 선택 및 중립 공정의 상대적 중요도를 측정하는 것은 일반적으로 어렵습니다.[107]진화적 변화를 이끄는 적응력과 비적응력의 비교적 중요성은 현재 연구의 한 분야입니다.[108]
변이편향
돌연변이 바이어스는 일반적으로 전이-변환 바이어스, GC-AT 바이어스, 삭제-삽입 바이어스와 같은 두 가지 다른 종류의 돌연변이에 대한 예상 속도의 차이로 생각됩니다.이것은 발전적 편견에 관한 생각과 관련이 있습니다.Haldane과[109] Fisher는[110] 돌연변이는 선택에 의해 쉽게 극복되는 약한 압력이기 때문에, 돌연변이의 경향은 중립적인 진화의 조건이나 비정상적으로 높은 돌연변이 비율을 제외하고는 효과가 없을 것이라고 주장했습니다.분자 시대가 중립적 진화에 대한 새로운 관심을 불러일으킬 때까지 [111]이 반대 압력 주장은 진화의 내적 경향의 가능성을 무시하기 위해 오랫동안 사용되었습니다.
Noboru Sueoka와[112] Ernst Freese는[113] 돌연변이의 체계적인 편향이 종 간의 게놈 GC 구성의 체계적인 차이에 책임이 있을 수 있다고 제안했습니다.1967년 GC 편향 대장균 변이주의 확인은 중성 이론의 제안과 [114]함께 분자 패턴에 대한 돌연변이 설명의 신뢰성을 확립했으며, 이는 현재 분자 진화 문헌에서 일반적입니다.
예를 들어 돌연변이 편향은 코돈 사용 모델에서 자주 발생합니다.[115]이러한 모델에는 돌연변이 편향과 번역 효과에 따른 차등 선택을 모두 허용하는 돌연변이 선택-이동 모델을 따르는 선택의 효과도 포함됩니다.[116]돌연변이 편견의 가설들은 아이소코어를 포함한 게놈 구성의 진화에 대한 사고의 발전에 중요한 역할을 했습니다.[117]다른 분류군에서 다른 삽입 대 삭제 편향은 다른 게놈 크기의 진화로 이어질 수 있습니다.[118][119]유전체 크기에 관한 린치의 가설은 유전체 크기의 증가 또는 감소에 대한 돌연변이 편향에 의존합니다.
그러나, (1) GC 편향 유전자 전환이 포유류와[120] 같은 이배체 생물에서 조성에 중요한 기여를 하고 (2) 박테리아 유전자는 종종 AT 편향 돌연변이를 갖는다는 것이 밝혀지면서 조성 진화에 대한 돌연변이 가설은 범위의 감소를 겪었습니다.[121]
돌연변이 편향의 역할에 대한 현대적인 사고는 할데인과 피셔의 이론과는 다른 이론을 반영합니다.더 최근의 연구는[111] 원래의 "압력" 이론이 진화가 정상적인 변이에 기초한다고 가정한다는 것을 보여주었습니다: 진화가 새로운 대립유전자를 도입하는 돌연변이의 사건에 의존할 때,변이의 도입에 있어서의 변이 및 발전적 편향(arrival 편향)은 중립적인 진화나 높은 변이율을 요구하지 않고도 진화에 편향을 부과할 수 있습니다.여러 연구에서는 적응에 관련된 돌연변이가 일반적인 돌연변이 편향을[123][124][125] 반영한다고 보고하지만 다른 연구에서는 이러한 해석에 이의를 제기합니다.[126]
유전적 히치하이킹
재조합은 같은 DNA 가닥의 대립유전자를 분리할 수 있게 해줍니다.그러나 재조합 비율은 낮습니다(세대당 염색체당 약 2개의 사건).결과적으로, 염색체 상에 가까이 있는 유전자들이 항상 서로에게서 멀어지지 않을 수도 있고, 가까이 있는 유전자들이 함께 유전되는 경향이 있는데, 이것은 연결이라고 알려진 현상입니다.[127]이러한 경향은 기대에 비해 두 개의 대립유전자가 하나의 염색체에 얼마나 자주 함께 발생하는지를 발견함으로써 측정되는데, 이를 연결 불균형이라고 합니다.한 집단에서 보통 유전되는 대립유전자의 집합을 하플로타입이라고 합니다.이것은 특정한 하플로타입의 한 대립유전자가 매우 유익할 때 중요할 수 있습니다: 자연적인 선택은 하플로타입의 다른 대립유전자가 인구에서 더 흔해지도록 할 선택적인 소탕을 유발할 수 있습니다; 이 효과는 유전적 히치하이킹 또는 유전자 드래프트라고 불립니다.[128]일부 중립적인 유전자가 선택 중인 다른 유전자와 유전적으로 연결되어 있다는 사실로 인해 발생하는 유전자 초안은 적절한 유효 모집단 크기에 의해 부분적으로 포착될 수 있습니다.[96]
성적선택

자연 선택의 특별한 경우는 성적 선택인데, 이것은 유기체의 잠재적인 짝에 대한 매력을 증가시킴으로써 짝짓기 성공을 증가시키는 어떤 특성에 대한 선택입니다.[130]성적 선택을 통해 진화한 특징은 여러 동물 종의 수컷들에게서 특히 두드러집니다.성적으로 선호되지만, 번거로운 뿔, 짝짓기 울음소리, 큰 몸집과 밝은 색과 같은 특징들은 종종 포식을 유도하며, 이는 수컷 개개인의 생존을 위협합니다.[131][132]이 생존 불리함은 가짜로 만들어지기 어렵고 성적으로 선택된 이런 특징들을 보여주는 남성들의 높은 생식 성공에 의해 균형을 이루게 됩니다.[133]
당연한 결과
진화는 유기체의 형태와 행동의 모든 측면에 영향을 미칩니다.가장 눈에 띄는 것은 자연 선택의 결과인 특정 행동 및 신체적 적응입니다.이러한 적응은 음식을 찾고, 포식자를 피하거나 짝을 유혹하는 등의 활동을 도와 건강을 증진시킵니다.유기체들은 또한 서로 협력함으로써 선택에 반응할 수 있는데, 보통 그들의 친척을 돕거나 서로 유익한 공생에 참여함으로써 반응할 수 있습니다.장기적으로, 진화는 유기체의 조상 집단을 교배할 수 없거나 교배하지 않을 새로운 집단으로 나누는 것을 통해 새로운 종을 만들어냅니다.이러한 진화의 결과는 시간 규모에 따라 거시 진화 대 미시 진화로 구분됩니다.거시 진화는 종의 수준 이상에서 일어나는 진화, 특히 종의 종과 멸종을 의미하는 반면, 미시 진화는 종 또는 개체군 내의 작은 진화 변화, 특히 모든 유전자 빈도와 적응의 변화를 의미합니다.[135]거시적 진화는 오랜 기간의 미시적 진화의 결과입니다.[136]따라서 미시적 진화와 거시적 진화의 구별은 근본적인 것이 아니며, 그 차이는 단순히 관련된 시간입니다.[137]하지만, 거시적 진화에서는 종 전체의 특성이 중요할 수 있습니다.예를 들어, 개체 간의 큰 변화는 종이 새로운 서식지에 빠르게 적응할 수 있도록 하여 멸종될 가능성을 줄이는 반면, 넓은 지리적 범위는 개체군의 일부가 고립될 가능성을 높임으로써 종의 가능성을 높입니다.이런 의미에서, 미세 진화와 거시 진화는 유전자와 유기체에 작용하는 미세 진화와 전체 종에 작용하는 종 선택과 같은 거시 진화 과정에 비해 다양한 수준의 선택을 포함할 수 있습니다.[138][139][140]
일반적인 오해는 진화가 목표, 장기 계획, 또는 "진보"에 대한 선천적인 경향을 가지고 있다는 것입니다. 그러나 현실적으로 진화는 장기적인 목표가 없으며 반드시 더 큰 복잡성을 발생시키지도 않습니다.[141][142][143]비록 복잡한 종들이 진화해왔지만, 그것들은 전체적인 유기체의 숫자가 증가하는 것의 부작용으로 발생하고 생물권에서 단순한 형태의 생명체는 여전히 더 흔합니다.[144]예를 들어, 압도적으로 많은 종들이 미세한 원핵생물들인데, 이 원핵생물들은 작은 크기에도 불구하고 전 세계 생물체의 절반 가량을 형성하고 있으며,[145] 지구 생물 다양성의 대부분을 차지하고 있습니다.[146]따라서 단순한 유기체는 역사를 통해 지구상의 지배적인 생명체였으며 현재까지 계속해서 주요한 생명체의 형태를 유지하고 있으며, 복잡한 생명체는 더 눈에 띄기 때문에 더 다양하게 나타날 뿐입니다.[147]실제로 미생물의 진화는 실험적 진화를 연구하고 실시간으로 진화와 적응을 관찰할 수 있기 때문에 진화 연구에 특히 중요합니다.[148][149]
적응.
적응은 생물체가 서식지에 더 적합하도록 만드는 과정입니다.[150][151]또한 적응이라는 용어는 유기체의 생존에 중요한 특성을 나타낼 수도 있습니다.예를 들면, 말의 이빨이 풀을 갈아내는 것에 적응하는 것입니다.진화과정에 적응(adaptation)이라는 용어와 제품(신체의 부위 또는 기능)에 적응(adaptive)이라는 용어를 사용하여 단어의 두 가지 감각을 구분할 수 있습니다.적응은 자연 선택에 의해 이루어집니다.[152]테오도시우스 돕잔스키에 의한 정의는 다음과 같습니다.
- 적응은 유기체가 서식지나 서식지에서 더 잘 살 수 있게 되는 진화 과정입니다.[153]
- 적응성(adaptiveness)은 적응하는 상태를 말합니다: 유기체가 주어진 서식지 집합에서 살고 번식할 수 있는 정도입니다.[154]
- 적응적 특성은 유기체가 생존하고 번식할 확률을 가능하게 하거나 향상시키는 유기체의 발달 패턴의 한 측면입니다.[155]
적응은 새 피쳐의 이득 또는 조상 피쳐의 손실을 초래할 수 있습니다.두 종류의 변화를 보여주는 한 예는 항생제 선택에 대한 박테리아 적응인데, 유전자 변화는 약물의 표적을 수정하거나 세포 밖으로 약물을 퍼내는 운반자의 활동을 증가시킴으로써 항생제 내성을 유발합니다.[156]다른 두드러진 예로는 장기 실험실 실험에서 구연산을 영양소로 사용하는 능력을 진화시키는 박테리아 대장균,[157] 나일론 제조의 부산물에서 이러한 박테리아가 자라도록 하는 새로운 효소를 진화시키는 플라보박테리움,[158][159]그리고 토양 박테리아 스핑고비움은 합성 농약 펜타클로로페놀을 분해하는 완전히 새로운 대사 경로를 진화시킵니다.[160][161]흥미롭지만 여전히 논란의 여지가 있는 아이디어는 일부 적응이 유전적 다양성을 생성하고 자연 선택에 의해 적응하는 유기체의 능력을 증가시킬 수 있다는 것입니다.[162][163][164][165]
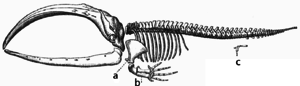
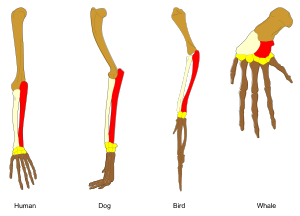
적응은 기존 구조의 점진적인 수정을 통해 이루어집니다.결과적으로, 유사한 내부 조직을 갖는 구조는 관련 유기체에서 다른 기능을 가질 수 있습니다.이것은 하나의 조상 구조가 다른 방식으로 기능하도록 적응된 결과입니다.예를 들어 박쥐 날개 안에 있는 뼈들은 쥐의 발과 영장류의 손에 있는 뼈들과 매우 유사한데, 이는 공통된 포유류 조상으로부터 이 모든 구조들이 내려왔기 때문입니다.[167]그러나 모든 생명체는 어느 정도 관련이 있기 때문에 [168]절지동물, 오징어 및 척추동물의 눈 등 구조적 유사성이 거의 없거나 전혀 없는 것으로 보이는 기관이라 하더라도 절지동물 및 척추동물의 사지 및 날개,그들의 집합과 기능을 조절하는 공통적인 상동 유전자 집합에 의존할 수 있습니다; 이것은 깊은 상동학이라고 불립니다.[169][170]
진화하는 동안 일부 구조는 원래의 기능을 상실하고 흔적 구조가 될 수 있습니다.[171]이러한 구조는 현재의 종에서는 기능이 거의 없거나 아예 없을 수도 있지만, 조상의 종 또는 가까운 다른 종에서는 분명한 기능을 가지고 있습니다.그 예로는 가짜 유전자,[172] 눈먼 동굴에 사는 물고기의 눈의 기능적이지 않은 잔해, 날지 못하는 [173]새의 날개, 고래와 뱀의 엉덩이뼈의 존재,[174][166] 무성생식을 통해 번식하는 생물의 성적 특징 등이 있습니다.[175]인간의 흔적 구조의 예로는 사랑니,[176] 고치,[171] 충수돌기,[171] 그리고 소름과[177][178] 원시 반사와 같은 다른 행동 흔적들이 있습니다.[179][180][181]
그러나 단순한 적응으로 보이는 많은 특성들은 사실 구조가 원래 하나의 기능에 맞게 적응되었지만 그 과정에서 우연히 다른 기능에 어느 정도 유용하게 되었습니다.[182]한 예로, 아프리카 도마뱀 Holaspis guentheri가 있는데, 이 도마뱀은 가까운 동족을 보면 알 수 있듯이 틈에 숨기 위해 극도로 평평한 머리를 발달시켰습니다.하지만, 이 종에서 머리는 매우 납작해져서 이 나무에서 저 나무로 활공하는 것을 돕습니다. 즉, 날렵한 것입니다.[182]세포 내에서, 박테리아[183] 편모와 단백질 분류[184] 기계와 같은 분자 기계는 이전에 다른 기능을 가지고 있던 여러 기존의 단백질의 모집에 의해 진화했습니다.[135]또 다른 예는 유기체의 눈의 렌즈 안에서 크리스탈린이라고 불리는 구조적인 단백질의 역할을 하기 위해 해당과정과 이종생물 대사로부터 효소를 모집하는 것입니다.[185][186]
진화 발달 생물학에서 현재 연구되고 있는 분야는 적응과 적응의 발달적 기반입니다.[187]이 연구는 배아 발달의 기원과 진화, 그리고 발달과 발달 과정의 수정이 어떻게 새로운 특징을 만들어 내는지를 다루고 있습니다.[188]이 연구들은 진화가 포유류에서 중이의 일부를 형성하는 대신 다른 동물의 턱으로 발달하는 배아 뼈 구조와 같은 새로운 구조를 생산하기 위해 발달을 바꿀 수 있다는 것을 보여주었습니다.[189]닭의 돌연변이로 배아가 악어의 치아와 비슷한 치아를 자라게 하는 등 발달 유전자의 변화로 인해 진화 과정에서 잃어버린 구조물이 다시 나타나는 것도 가능합니다.[190]유기체의 형태에 있어서의 대부분의 변화는 보존된 유전자들의 작은 집합의 변화에 기인한다는 것이 이제 분명해지고 있습니다.[191]
공진화

유기체 간의 상호작용은 갈등과 협력을 둘 다 만들어 낼 수 있습니다.병원체와 숙주, 또는 포식자와 그 먹이와 같은 종의 쌍 사이의 상호작용이 있을 때, 이 종들은 일치하는 적응 세트를 발달시킬 수 있습니다.여기서 한 종의 진화는 두 번째 종의 적응을 일으킵니다.두 번째 종의 이러한 변화는 결국 첫 번째 종에 새로운 적응을 일으킵니다.이러한 선택과 반응의 순환을 공진화라고 합니다.[192]예를 들면 거친 피부의 쐐기풀에서 테트로도톡신이 생성되고 포식자인 보통 가터뱀에서 테트로도톡신 저항성이 진화하는 것입니다.이 포식자와 먹이의 쌍에서 진화론적 무기 경쟁은 쐐기풀에서 높은 수준의 독소를 생성하고 뱀에서 상응하게 높은 수준의 독소 저항성을 생성했습니다.[193]
협력.
종들 간의 모든 공진화된 상호작용이 갈등을 수반하는 것은 아닙니다.[194]상호 이익이 되는 상호작용의 많은 사례들이 발전해 왔습니다.예를 들어, 식물과 뿌리에서 자라고 식물이 토양으로부터 영양분을 흡수하는 것을 돕는 균류 사이에는 극단적인 협력이 존재합니다.[195]이것은 식물이 균류에 광합성에서 나온 당을 제공하기 때문에 상호적인 관계입니다.여기서, 곰팡이는 실제로 식물 세포 안에서 자라며, 식물 면역 체계를 억제하는 신호를 보내는 동시에 숙주와 영양분을 교환할 수 있게 해줍니다.[196]
같은 종의 생물들 간의 연합 또한 진화해왔습니다.극단적인 경우는 벌, 흰개미, 개미와 같은 사회적 곤충에서 볼 수 있는 유소셜리티(eusociality)로, 무균 곤충이 번식할 수 있는 군집 내의 소수의 유기체를 먹고 지키는 것입니다.훨씬 더 작은 규모로, 동물의 몸을 구성하는 체세포는 그들의 번식을 제한하여 안정적인 유기체를 유지할 수 있고, 그 후 소수의 동물의 생식 세포가 자손을 생산하도록 지원합니다.여기서 체세포는 성장할 것인지, 그대로 남아있을 것인지, 아니면 죽을 것인지를 지시하는 특정한 신호에 반응합니다.만약 세포들이 이러한 신호들을 무시하고 부적절하게 증식한다면, 그들의 조절되지 않는 성장은 암을 유발합니다.[197]
종내의 이러한 협력은 친척의 자손을 기르는 데 한 유기체가 작용하는 친족 선택의 과정을 통해 진화했을 수도 있습니다.[198]이 활동은 도움을 주는 개인이 도움을 주는 활동을 촉진하는 대립유전자를 포함하고 있다면, 그의 친족 또한 이러한 대립유전자를 포함하고 따라서 그러한 대립유전자를 전달될 가능성이 높기 때문에 선택됩니다.[199]협력을 촉진할 수 있는 다른 과정에는 협력이 유기체 집단에 혜택을 제공하는 집단 선택이 포함됩니다.[200]
스펙

종분화는 한 종이 두 개 이상의 후예 종으로 갈라지는 과정입니다.[201]
"종"의 개념을 정의하는 데는 여러 가지 방법이 있습니다.정의의 선택은 해당 종의 특수성에 따라 달라집니다.[202]예를 들어, 어떤 종의 개념은 성적으로 번식하는 유기체에 더 쉽게 적용되는 반면 다른 종들은 무성 생물에 더 잘 적응합니다.다양한 종 개념의 다양성에도 불구하고, 이 다양한 개념들은 교배, 생태학, 계통발생학의 세 가지 광범위한 철학적 접근 중 하나로 배치될 수 있습니다.[203]생물학적 종 개념(BSC)은 교배 접근법의 전형적인 예입니다.1942년 진화생물학자 에른스트 메이어(Ernst Mayr)에 의해 정의된 BSC는 "종들은 실제로 또는 잠재적으로 교배된 자연 개체군의 그룹이며, 이들은 다른 그러한 그룹으로부터 생식적으로 분리되어 있습니다."[204]그것의 광범위하고 장기간의 사용에도 불구하고, 다른 종 개념들처럼 BSC도 논란의 여지가 없는 것은 아닙니다. 예를 들어, 원핵생물들 사이의 유전자 재조합은 번식의 본질적인 측면이 아니기 때문입니다;[205] 이것은 종 문제라고 불립니다.[202]일부 연구자들은 종에 대한 일원론적 정의를 시도한 반면, 다른 연구자들은 다원론적 접근법을 채택하고 종의 정의를 논리적으로 해석하는 다른 방법이 있을 수 있다고 제안합니다.[202][203]
개체수가 새로운 종이 되기 위해서는 서로 다른 두 성군 사이의 번식에 대한 장벽이 필요합니다.유전자 흐름은 새로운 유전자 변형을 다른 집단들에게도 전파함으로써 이 과정을 늦출 수 있습니다.가장 최근의 공통 조상 이후 두 종이 얼마나 멀리 갈라졌는가에 따라, 말과 당나귀가 노새를 만들기 위해 짝짓기를 하는 것처럼, 그들이 자손을 낳는 것은 여전히 가능할지도 모릅니다.[206]그러한 잡종들은 일반적으로 불임입니다.이런 경우, 밀접하게 관련된 종들은 정기적으로 교배할 수 있지만, 잡종들은 반대로 선택될 것이고 그 종들은 별개로 남을 것입니다.하지만, 생존 가능한 잡종들이 때때로 형성되고 이 새로운 종들은 그들의 부모 종들 사이의 중간적인 특성을 갖거나 완전히 새로운 표현형을 가질 수 있습니다.[207]새로운 종의 동물을 생산하는 데 있어 교배의 중요성은 불분명하지만, 많은 종류의 동물에서 사례가 발견되고 있으며,[208] 특히 회색 청개구리가 잘 연구된 예입니다.[209]
종분화는 통제된 실험실 조건과 자연에서 여러 번 관찰되었습니다.[210]성적으로 생식하는 유기체에서 종분화는 생식 격리에 따른 계보적 분화로 발생합니다.네 가지 주요한 지리학적 사양이 있습니다.동물에서 가장 흔한 것은 동종 종파로, 서식지 파편화나 이주와 같이 처음에 지리적으로 고립된 개체군에서 발생합니다.이러한 조건하에서 선택은 유기체의 겉모습과 행동에 매우 빠른 변화를 일으킬 수 있습니다.[211][212]선택과 표류는 그들의 나머지 종들로부터 고립된 집단들에게 독립적으로 작용하기 때문에, 분리는 결국 교배할 수 없는 유기체를 만들어 낼 수 있습니다.[213]
두 번째 종분화 방식은 주변부 종분화로, 적은 개체수가 새로운 환경에서 고립될 때 발생합니다.이는 고립된 모집단이 부모 모집단보다 수치적으로 훨씬 작다는 점에서 동종업계 사양과는 다릅니다.여기서 창시자 효과는 교배의 증가가 호모지게이트에 대한 선택을 증가시키고 급격한 유전자 변화를 초래한 후 급격한 종분화를 야기합니다.[214]
세 번째 모드는 파라패트릭 스펙입니다.이는 작은 개체수가 새로운 서식지로 들어온다는 점에서 주변부 종과 유사하지만, 이 두 개체수 사이에 물리적인 분리가 없다는 점에서 차이가 있습니다.대신, 종분화는 두 집단 사이의 유전자 흐름을 줄이는 메커니즘의 진화에서 비롯됩니다.[201]일반적으로 이러한 현상은 부모 종의 서식지 내 환경에 급격한 변화가 있을 때 발생합니다.한 가지 예는 풀 안톡산툼 냄새로 광산에서 발생하는 국부적인 금속 오염에 대응하여 파라패트릭 사양화를 거칠 수 있습니다.[215]여기서 식물들은 토양의 높은 수준의 금속에 저항력을 가지는 진화를 합니다.금속에 민감한 부모 집단과의 교배에 반대하는 선택은 금속 저항성 식물의 개화 시기에 점진적인 변화를 가져왔고, 결국 완전한 생식 격리를 만들었습니다.두 집단 사이의 잡종에 대한 선택은 두 종의 외모가 더 뚜렷해지는 특징 이동뿐만 아니라 종 내에서 짝짓기를 촉진하는 특성의 진화인 강화를 일으킬 수 있습니다.[216]
마지막으로, 동종 종들은 지리적 고립이나 서식지의 변화 없이 분화합니다.적은 양의 유전자 흐름 조차도 집단의 일부 사이의 유전적 차이를 제거할 수 있기 때문에 이 형태는 희귀합니다.[217]일반적으로, 동물의 동정적인 종분화는 생식 격리가 진화할 수 있도록 유전적인 차이와 무작위적이지 않은 교미의 진화를 요구합니다.[218]
한 종류의 동정종은 새로운 교배종을 생산하기 위해 관련된 두 종의 교배를 포함합니다.이것은 동물의 잡종들이 보통 무균이기 때문에 동물들에게 흔하지 않습니다.이것은 감수분열 동안 각각의 부모로부터의 상동 염색체가 다른 종의 것이고 성공적으로 짝을 이룰 수 없기 때문입니다.하지만, 식물들은 종종 염색체 수를 두 배로 늘려 다종체를 형성하기 때문에 이것은 식물들에서 더 흔합니다.[219]이것은 각 부모의 염색체가 이미 한 쌍으로 표현되기 때문에 감수분열 동안 각 부모 종의 염색체가 일치하는 쌍을 형성할 수 있게 해줍니다.[220]이러한 종묘행사의 한 예로, 식물종인 Arabidopsis thaliana와 Arabidopsis arenosa가 교배하여 새로운 종인 Arabidopsis suecica를 줄 때를 들 수 있습니다.[221]이런 일이 약 2만 년 전에 일어났고,[222] 이 과정에 관련된 유전적 메커니즘을 연구할 수 있는 종분화 과정이 실험실에서 반복돼 왔습니다.[223]실제로, 한 종 내에서 염색체가 두 배로 증가하는 것은 생식 격리의 흔한 원인일 수 있습니다. 두 배로 증가한 염색체의 절반은 두 배가 되지 않은 유기체와 함께 번식할 때 일치하지 않을 것이기 때문입니다.[224]
종들이 상대적으로 변하지 않는 비교적 긴 기간의 정지 기간 사이에 있는 진화의 짧은 "폭발"이라는 화석 기록의 패턴을 설명하는 간헐적 평형 이론에서 종의 사건은 중요합니다.[225]이 이론에서, 종분화와 빠른 진화는 연결되어 있는데, 자연 선택과 유전적 이동은 새로운 서식지나 작은 인구에서 종분화를 겪고 있는 유기체에 가장 강하게 작용합니다.결과적으로 화석 기록에서 정체기는 부모 개체수에 해당하고 종분화와 급격한 진화를 겪고 있는 생물들은 작은 개체수나 지리적으로 제한된 서식지에서 발견되어 화석으로 보존되는 경우가 거의 없습니다.[139]
소멸

멸종이란 종 전체가 사라지는 것을 말합니다.멸종은 특이한 사건이 아닌데, 종들은 종파를 통해 정기적으로 나타나고 멸종을 통해 사라지기 때문입니다.[226]지구상에 살아왔던 거의 모든 동물과 식물 종들은 이제 멸종되었고,[227] 멸종은 모든 종들의 궁극적인 운명인 것으로 보입니다.[228]이러한 멸종은 생명의 역사를 통틀어 지속적으로 발생해 왔지만, 때때로 대멸종 사건에서 멸종의 속도가 급증하기도 합니다.[229]가장 잘 알려진 것은 비조류 공룡이 멸종한 백악기-팔레오기 멸종 사건이지만, 초기 페름기-트라이아스기 멸종 사건은 더욱 심각하여 전체 해양 종의 약 96%가 멸종 위기에 처했습니다.[229]홀로세 멸종 사건은 지난 수천 년 동안 인류의 전 세계로의 확장과 관련된 진행 중인 대멸종입니다.현재의 멸종률은 배경 비율의 100배에서 1000배에 달하며 21세기 중반에는 현재 종의 30%까지 멸종될 수 있습니다.[230]인간의 활동은 현재 진행중인 멸종 사건의 주요 원인입니다;[231][232] 지구 온난화는 미래에 지구 온난화를 더욱 가속화 시킬지도 모릅니다.[233]지구상에 살았던 모든 생물 종의 99% 이상이 멸종된 것으로 추정되고 있음에도 불구하고,[234][235] 현재 지구상에 약 1조 종이 있는 것으로 추정되고 있으며, 1천 분의 1%만이 설명되어 있습니다.[236]
진화에서 멸종의 역할은 잘 이해되어 있지 않으며 어떤 종류의 멸종을 고려하느냐에 따라 달라질 수 있습니다.[229]멸종의 대부분을 이루는 지속적인 '저준위' 멸종 사건의 원인은 제한된 자원을 놓고 종 간 경쟁(경쟁적 배제 원칙) 때문일 것입니다.[237]만약 한 종이 다른 종과 경쟁할 수 있다면, 이것은 더 적합한 종들이 살아남고 다른 종들은 멸종으로 내몰리는 종 선택을 만들어 낼 수 있습니다.[84]간헐적인 대멸종도 중요하지만 선택적인 힘으로 작용하는 대신 비특이적인 방식으로 다양성을 급격히 감소시키고 생존자들의 빠른 진화와 종분화를 촉진합니다.[238]
적용들
자연 선택과 같은 진화 생물학에서 사용되는 개념과 모델은 많은 응용을 가지고 있습니다.[239]
인위적 선택은 유기체 집단에서 의도적으로 형질을 선택하는 것입니다.이것은 수천 년 동안 식물과 동물을 길들이는 데 사용되어 왔습니다.[240]더 최근에는, 그러한 선택은 DNA를 조작하기 위해 사용되는 항생제 내성 유전자와 같은 선택 가능한 마커들과 함께 유전 공학의 중요한 부분이 되었습니다. 가치 있는 특성을 가진 단백질은 방향 진화라고 불리는 과정에서 반복적인 돌연변이와 선택 (예를 들어 변형 효소와 새로운 항체)에 의해 진화해왔습니다.[241]
유기체가 진화하는 동안 일어난 변화를 이해하는 것은 신체의 일부를 구성하는 데 필요한 유전자, 즉 인간의 유전적 장애에 관련되어 있을지도 모르는 유전자를 밝힐 수 있습니다.[242]예를 들면, 멕시코 테트라는 알비노 동굴 물고기로 진화하는 동안 시력을 잃었습니다.다른 동굴에서 진화한 고립된 개체군에서 다른 돌연변이가 발생했기 때문에 이 맹물고기의 다른 개체군을 함께 교배하여 기능적인 눈을 가진 일부 자손을 낳았습니다.[243]이것은 시력과 색소 침착에 필요한 유전자를 알아보는데 도움이 되었습니다.[244]
진화론은 의학에 많은 응용을 가지고 있습니다.인간의 많은 질병들은 정적인 현상이 아니라 진화가 가능합니다.바이러스, 박테리아, 곰팡이 및 암은 약학적 약물뿐만 아니라 숙주 면역 방어에 내성을 갖도록 진화합니다.[245][246][247]농약과[248] 제초제[249] 내성을 가진 농업에서도 이와 같은 문제가 발생합니다.우리가 대부분의 이용 가능한[250] 항생제의 유효 수명의 끝에 직면해 있고 병원균의 진화와 진화[251] 가능성을 예측하고 그것을 늦추거나 회피하기 위한 전략을 고안하는 것은 분자 수준에서 진화를 이끄는 복잡한 힘에 대한 더 깊은 지식을 요구하는 것일 수 있습니다.[252]
컴퓨터 과학에서 진화 알고리즘과 인공 생명체를 이용한 진화 시뮬레이션은 1960년대에 시작되어 인공 선택 시뮬레이션으로 확장되었습니다.[253]1960년대 잉고 레첸베르크의 연구 결과로 인공 진화는 널리 알려진 최적화 방법이 되었습니다.그는 복잡한 공학적 문제를 해결하기 위해 진화 전략을 사용했습니다.[254]특히 유전자 알고리즘은 존 헨리 홀랜드의 글을 통해 대중화되었습니다.[255]실용적인 응용에는 컴퓨터 프로그램의 자동 진화도 포함됩니다.[256]진화 알고리즘은 이제 인간 설계자가 제작한 소프트웨어보다 다차원 문제를 더 효율적으로 해결하고 시스템 설계를 최적화하는 데 사용됩니다.[257]
생명의 진화사
−4500 — – — – −4000 — – — – −3500 — – — – −3000 — – — – −2500 — – — – −2000 — – — – −1500 — – — – −1000 — – — – −500 — – — – 0 — |
| |||||||||||||||||||||||||||||||||||||||||||||
생명의 기원
지구는 약 45억 4천만년 정도 되었습니다.[258][259][260]지구상 생명체에 대한 최초의 논란의 여지가 없는 증거는 적어도 35억년 전으로 거슬러 올라가는데,[12][261] 이는 초기에 녹은 하데스 이온 이후 지질학적 지각이 굳어지기 시작한 이후 에오아르키안 시대 동안입니다.웨스턴 오스트레일리아의 34억 8천만 년 된 사암에서 미생물 매트 화석이 발견되었습니다.[14][15][16]생물 발생 물질에 대한 또 다른 초기의 물리적 증거는 그린란드[13] 서부에서 발견된 37억 년 된 퇴적암의 흑연과 호주 서부의 41억 년 된 암석에서 발견된 "생물 생물의 잔해"입니다.[262][263]호주의 발견에 대해 논평하면서, 스티븐 블레어 헤지스는 다음과 같이 썼습니다: "만약 생명체가 지구에서 비교적 빨리 발생했다면, 그것은 우주에서 흔한 일이 될 수 있었을 것입니다.[262][264]2016년 7월, 과학자들은 지구에 살고 있는 모든 유기체의 마지막 보편적 공통 조상(LUCA)으로부터 355개의 유전자를 확인했다고 보고했습니다.[265]
지구상에 살았던 모든 종의 99% 이상, 약 50억 종 이상이 멸종된 것으로 추정되고 있습니다.[266][234][235]지구의 현재 종의 추정치는 1,000만에서 1,400만에 이르며,[267][268] 이 중 약 190만 종의 이름이[269] 지어졌을 것으로 추정되며,[270] 현재까지 160만 종이 중앙 데이터베이스에 기록되어 있으며, 적어도 80%는 아직 기술되지 않은 상태입니다.
매우 에너지가 넘치는 화학은 약 40억 년 전에 자기 복제 분자를 만들어 낸 것으로 생각되며, 5억 년 후에는 모든 생명체의 마지막 공통 조상이 존재했습니다.[10]현재 과학적 합의는 생명체를 구성하는 복잡한 생화학이 단순한 화학 반응에서 비롯되었다는 것입니다.[271][272]생명의 시작은 RNA와[273] 단순한 세포의 조립과 같은 자기 복제 분자를 포함했을지도 모릅니다.[274]
공통 혈통
지구상의 모든 생물체는 공통된 조상 또는 조상의 유전자 풀의 후손입니다.[168][275]현재의 종은 진화 과정의 단계로, 그들의 다양성은 긴 일련의 종파와 멸종 사건의 산물입니다.[276]유기체의 공통적인 혈통은 유기체에 관한 네 가지 단순한 사실로부터 처음 도출되었습니다.첫째, 그들은 지역 적응으로 설명할 수 없는 지리적 분포를 가지고 있습니다.둘째, 생명의 다양성은 완전히 독특한 유기체의 집합이 아니라 형태학적 유사성을 공유하는 유기체입니다.셋째, 뚜렷한 목적이 없는 잔존적 특성은 기능적 조상적 특성과 유사합니다.넷째, 이러한 유사성을 이용하여 생물을 가계도와 유사한 중첩집단의 계층으로 분류할 수 있습니다.[277]

수평적인 유전자 전달 때문에, 이 "생명의 나무"는 일부 유전자들이 멀리 관련된 종들 사이에 독립적으로 퍼져있기 때문에 단순한 가지 나무보다 더 복잡할 수 있습니다.[278][279]이 문제 등을 해결하기 위해 일부 저자들은 '생명의 산호'를 은유 혹은 수학적 모델로 사용하여 생명의 진화를 설명하기를 선호합니다.이 견해는 다윈이 잠시 언급했다가 나중에 포기한 아이디어로 거슬러 올라갑니다.[280]
과거의 종들은 또한 진화의 역사에 대한 기록을 남겼습니다.화석은 오늘날의 유기체의 비교 해부학과 함께 형태학적, 즉 해부학적 기록을 구성합니다.[281]고생물학자들은 현대 종과 멸종 종의 해부학적 구조를 비교함으로써 그 종의 혈통을 추론할 수 있습니다.그러나, 이 접근법은 껍질, 뼈 또는 치아와 같이 딱딱한 신체 부위를 가지고 있던 유기체들에게 가장 성공적입니다.게다가, 박테리아와 고세균과 같은 원핵 생물들은 공통적인 형태학의 제한된 집합을 공유하기 때문에, 그들의 화석은 그들의 조상에 대한 정보를 제공하지 않습니다.
더 최근에, 공통적인 혈통에 대한 증거는 유기체들 사이의 생화학적 유사성에 대한 연구로부터 나왔습니다.예를 들어, 모든 살아있는 세포는 뉴클레오티드와 아미노산으로 이루어진 같은 기본 세트를 사용합니다.[282]분자유전학의 발전은 생물체의 게놈에 남아있는 진화의 기록을 밝혀냈습니다: 돌연변이에 의해 만들어진 분자 시계를 통해 종들이 갈라졌을 때의 연대측정입니다.[283]예를 들어, 이러한 DNA 염기서열 비교는 인간과 침팬지가 그들의 게놈의 98%를 공유한다는 것을 밝혀냈고, 그들이 다른 소수의 영역을 분석하는 것은 이 종들의 공통 조상이 언제 존재했는지를 밝히는데 도움이 됩니다.[284]
생명의 진화
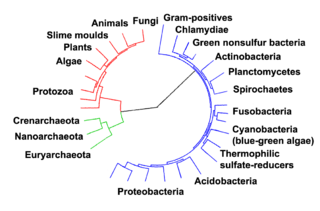
원핵생물은 대략 30억년에서 40억년 전에 지구에 살고 있었습니다.[286][287]다음 몇 십억 년 동안 이 유기체들에서 형태학이나 세포 조직에 뚜렷한 변화는 일어나지 않았습니다.[288]진핵세포는 16억년에서 27억년 전 사이에 출현했습니다.세포 구조의 다음 주요한 변화는 박테리아가 진핵세포에 의해 삼켜졌을 때, 내심비증이라고 불리는 협력적인 연합체에서 왔습니다.[289][290]그리고 나서 삼킨 박테리아와 숙주 세포는 박테리아가 미토콘드리아 또는 수소오솜으로 진화하면서 공진화를 겪었습니다.[291]남조류와 식물에서 엽록체가 형성된 것은 시아노박테리아와 같은 유기체를 집어삼킨 또 다른 사건입니다.[292]
생명의 역사는 에디아카라 시대에 다세포 유기체가 바다에 나타나기 시작한 약 6억 1천만년 전까지 단세포 진핵생물, 원핵생물, 고균의 역사였습니다.[286][293]다세포성의 진화는 해면동물, 갈조류, 남조류, 시아노박테리아, 점액곰팡이 및 균류와 같이 다양한 생물체에서 여러 독립적인 사건에서 발생했습니다.[294]2016년 1월, 과학자들은 약 8억년 전, GK-PID라고 불리는 하나의 분자의 작은 유전적인 변화가 유기체들이 하나의 세포 유기체에서 많은 세포들 중 하나로 가도록 했을지도 모른다고 보고했습니다.[295]
이들 최초의 다세포 생물이 출현한 직후, 캄브리아기 폭발이라고 불리는 사건으로 약 천만 년에 걸쳐 놀라운 양의 생물학적 다양성이 나타났습니다.여기에서, 대부분의 현대 동물의 종류가 화석 기록에 나타났을 뿐만 아니라 이후 멸종된 독특한 계통들도 있습니다.[296]광합성으로 대기 중에 산소가 축적되는 것을 포함하여 캄브리아기 폭발의 다양한 계기들이 제안되었습니다.[297]
약 5억년 전, 식물과 곰팡이가 이 땅을 점령했고 곧 절지동물과 다른 동물들이 뒤따랐습니다.[298]곤충들은 특히 성공적이었고 심지어 오늘날에도 동물 종의 대부분을 차지하고 있습니다.[299]양서류는 약 3억 6천 4백만 년 전에 처음 출현했고, 그 뒤로 초기 양막류와 조류가 약 1억 5천 5백만 년 전에 출현했고, 포유류는 약 1억 2천 9백만 년 전에 출현했고, 현생 인류는 약 25만 년 전에 출현했습니다.[300][301][302]그러나, 이러한 큰 동물들의 진화에도 불구하고, 이 과정에서 초기에 진화한 종류와 유사한 작은 생물들은 계속해서 큰 성공을 거두며 지구를 지배하고 있으며, 바이오매스와 종의 대부분은 원핵생물입니다.[146]
진화사상사




고대
한 종류의 유기체가 다른 종류의 유기체에서 기원할 수 있다는 제안은 아낙시만데르나 엠페도클레스 같은 소크라테스 이전의 그리스 철학자들에게 거슬러 올라갑니다.[304]그러한 제안들은 로마시대까지 살아남았습니다.시인이자 철학자인 루크레티우스는 그의 걸작인 "데레룸 나투라"에서 엠페도클레스를 따랐습니다.[305][306]
중세
이러한 유물론적 견해와는 대조적으로, 아리스토텔레스주의는 모든 자연적인 것들을 형태로 알려진 고정된 자연적 가능성의 실현으로 간주했습니다.[307][308]이것은 모든 사물이 신성한 우주 질서 속에서 의도된 역할을 하는 자연에 대한 중세 목적론적 이해의 일부가 되었습니다.이 생각의 변형은 중세의 표준적인 이해가 되었고 기독교 학습에 통합되었지만 아리스토텔레스는 실제 유형의 유기체가 항상 정확한 형이상학적 형태와 일대일로 대응할 것을 요구하지 않았고 구체적으로 어떻게 새로운 유형의 생물이 될 수 있는지에 대한 예를 제시했습니다.[309]
다수의 아랍 무슬림 학자들은 진화에 대해 저술했는데, 특히 이븐 칼둔은 서기 1377년에 무카디마라는 책을 썼는데, 이 책에서 그는 인간이 "원숭이의 세계"로부터 발전했으며, 이 과정에서 "종들이 더 많아지는" 과정이라고 주장했습니다.[310]
다르위니아 이전의
17세기의 "신과학"은 아리스토텔레스적 접근법을 거부했습니다.그것은 모든 가시적인 것들에 대해 동일하고 어떤 고정된 자연적 범주나 신성한 우주 질서의 존재를 요구하지 않는 물리적 법칙의 관점에서 자연 현상을 설명하고자 했습니다.그러나, 이 새로운 접근법은 생물 과학에 뿌리내리기는 더뎠습니다: 고정된 자연형 개념의 마지막 보루.존 레이(John Ray)는 고정된 자연 유형에 대한 이전의 보다 일반적인 용어 중 하나인 "종"을 식물과 동물 유형에 적용했지만, 그는 생물의 각 유형을 엄격하게 하나의 종으로 식별하고 각 종은 세대를 거쳐 자신을 영속시키는 특징에 의해 정의될 수 있다고 제안했습니다.[311]1735년에 칼 린네가 도입한 생물학적 분류는 종 관계의 계층적 성격을 명시적으로 인정했지만, 여전히 종들을 신의 계획에 따라 고정된 것으로 간주했습니다.[312]
이 시기의 다른 자연주의자들은 자연 법칙에 따라 시간이 지남에 따라 종의 진화적 변화에 대해 추측했습니다.1751년, 피에르 루이 모페르튀아는 번식 중에 일어나는 자연적인 변형과 새로운 종을 만들기 위해 여러 세대에 걸쳐 축적되는 것에 대해 썼습니다.[313]Georges-Louis Leclerc, Comte de Buffon은 종들이 다른 유기체로 변질될 수 있다고 제안했고, Erasmus Darwin은 모든 온혈 동물들이 하나의 미생물 (또는 "필라멘트")[314]의 후손일 수 있다고 제안했습니다.최초의 본격적인 진화 계획은 1809년 장 밥티스트 라마르크의 "변환" 이론으로,[315] 자발적인 생성이 지속적으로 단순한 형태의 생명을 생성하는 것을 상상하고, 고유한 진보적 경향을 가진 평행선에서 더 큰 복잡성을 발전시키고, 지역적 수준에서,이러한 계통들은 부모들에게 그들의 사용 또는 사용하지 않음으로 인해 야기된 변화를 물려받음으로써 환경에 적응하였습니다.[316](후의 과정은 후에 라마르크주의라고 불렸습니다.)[316][317][318]기존의 자연주의자들은 경험적 뒷받침이 부족한 추측이라고 비판했습니다.특히, 조르주 큐비에(Georges Cubier)는 종들이 관련이 없고 고정되어 있으며, 그 유사성은 기능적 필요에 대한 신성한 디자인을 반영한다고 주장했습니다.한편, 레이의 자비로운 디자인 아이디어는 윌리엄 페일리에 의해 자연신학 또는 신의 존재와 속성의 증거(1802)로 발전되었고, 이것은 복잡한 적응을 신의 디자인의 증거로 제안하고 찰스 다윈에 의해 존경을 받았습니다.[319][320]
다윈 혁명
생물학에서 일정한 유형학적 계급 또는 유형의 개념으로부터의 결정적인 단절은 찰스 다윈과 알프레드 월리스에 의해 가변적인 집단의 관점에서 공식화된 자연 선택을 통한 진화 이론과 함께 왔습니다.다윈은 "진화"라는 표현 대신 "수정된 하강"이라는 표현을 사용했습니다.[321]다윈은 토마스 로버트 맬서스(Thomas Robert Malthus)의 인구 원리에 대한 에세이(1798)에 부분적으로 영향을 받아 인구 증가가 "존재를 위한 투쟁"으로 이어질 것이며, 이는 다른 사람들이 죽으면서 유리한 변화가 만연할 것이라고 언급했습니다.각각의 세대에서, 많은 자손들은 제한된 자원 때문에 번식의 나이까지 살아남지 못합니다.이것은 모든 종류의 유기체에 대해 동일한 방식으로 자연법의 작동을 통해 공통된 혈통의 식물과 동물의 다양성을 설명할 수 있습니다.[322][323][324][325]다윈은 1838년부터 그의 "자연 선택"에 대한 이론을 발전시켰고 1858년 알프레드 러셀 월리스가 그에게 사실상 같은 이론의 버전을 보내왔을 때 그 주제에 대한 그의 "큰 책"을 쓰고 있었습니다.그들의 별개의 논문들은 1858년 런던 린네 학회의 회의에서 함께 발표되었습니다.[326]1859년 말, 다윈이 종의 기원에 관하여라는 그의 "추상"을 출판한 것은 자연 선택을 상세하게 그리고 대안 이론의 희생으로 다윈의 진화 개념을 점점 더 광범위하게 받아들이게 하는 방식으로 설명했습니다.토마스 헨리 헉슬리는 인간과 유인원이 같은 조상이라는 강력한 증거를 제공하기 위해 고생물학과 비교 해부학을 사용하여 다윈의 아이디어를 인간에게 적용시켰습니다.어떤 사람들은 이것이 인간이 우주에서 특별한 위치를 가지고 있지 않다는 것을 암시했기 때문에 이것에 대해 불안해 했습니다.[327]
범생과 유전
생식 유전의 메커니즘과 새로운 특성의 기원은 수수께끼로 남아 있었습니다.이를 위해 다윈은 판게네시스에 대한 잠정적인 이론을 발전시켰습니다.[328]1865년 그레고르 멘델(Gregor Mendel)은 형질이 (후에 유전자로 알려진) 독립적인 배열과 분리를 통해 예측 가능한 방식으로 유전된다고 보고했습니다.멘델의 유전법칙은 결국 다윈의 범생론의 대부분을 대체했습니다.[329]August Weismann은 유전이 오직 생식선을 통과한다는 것을 증명하면서, 생식선(정자와 난자 세포와 같은)을 만드는 생식세포와 신체의 체세포 사이의 중요한 구분을 만들었습니다.휴고 드 브리스(Hugo de Vries)는 다윈의 판게네시스 이론을 바이스만(Weismann)의 세균/소마 세포 구별과 연결하고 다윈의 판게네가 세포핵에 집중되어 발현될 때 세포질로 이동하여 세포의 구조를 변화시킬 수 있다고 제안했습니다.드 브리즈는 또한 멘델의 특성이 유전적인 변이가 생식선을 따라 전달되는 것과 일치한다고 믿으면서 멘델의 연구를 널리 알린 연구자들 중 한 명이었습니다.[330]새로운 변종이 어떻게 생겨났는지 설명하기 위해 드브리즈는 다윈의 진화를 받아들인 사람들과 드브리즈와 동맹을 맺은 생체인식자들 사이에 일시적인 균열을 가져온 돌연변이 이론을 개발했습니다.[331][332]1930년대에 로널드 피셔, 시월 라이트, J. B. S. 홀데인과 같은 인구 유전학 분야의 선구자들은 강력한 통계 철학에 진화의 기초를 세웠습니다.이로써 다윈의 이론과 유전적 돌연변이, 멘델의 유산 사이의 잘못된 모순이 조정되었습니다.[333]
'현대적 합성'
1920년대와 1930년대에, 현대의 종합은 멘델의 유전에 기초한 자연 선택과 집단 유전학을 임의의 유전적 이동, 돌연변이, 그리고 유전자 흐름을 포함하는 통일된 이론으로 연결시켰습니다.이 새로운 버전의 진화론은 인구의 모든 유전자 빈도의 변화에 초점을 맞췄습니다.그것은 고생물학의 화석 전이를 통해 집단의 종에 걸쳐 관찰되는 패턴을 설명했습니다.[333]
추가합성
그 이후로, 추가적인 합성은 유전자에서 집단에 이르기까지 생물학적 계층 전체에 걸친 생물학적 현상을 포괄하기 위해 수많은 발견에 비추어 진화의 설명력을 확장시켰습니다.[334]
1953년에 James Watson과 Francis Crick에 의한 DNA의 구조의 발표는 상속을 위한 물리적인 메커니즘을 증명했습니다.[335]분자생물학은 유전자형과 표현형 사이의 관계에 대한 이해를 향상시켰습니다.진화 나무의 출판과 사용을 통해 형질 전환을 비교 가능하고 시험 가능한 틀로 매핑하는 계통발생학에서도 발전이 이루어졌습니다.[336]1973년 진화생물학자 테오도시우스 도브잔스키는 "진화의 관점을 제외하고는 생물학에서 어떤 것도 말이 되지 않습니다." 왜냐하면 그것은 자연사에서 처음으로 서로 단절된 것처럼 보였던 사실들의 관계를 이 행성의 생명체에 대한 많은 관찰 가능한 사실들을 설명하고 예측하는 일관성 있는 설명적인 지식의 단체로 밝혀냈기 때문입니다.[337]
진화 발달 생물학으로 알려져 있고 비공식적으로 "에보데보"라고 불리는 한 가지 확장은 세대 간의 변화(진화)가 개별 유기체(개발) 내의 변화 패턴에 어떻게 작용하는지를 강조합니다.[237][338]21세기 초부터, 일부 생물학자들은 후성유전학, 부모 효과, 생태학적 상속 및 문화적 상속, 진화 가능성과 같은 비유전적 상속 방식의 효과를 설명하는 확장된 진화론적 합성을 주장해 왔습니다.[339][340]
사회·문화적 대응

19세기, 특히 1859년 종의 기원에 관한 출판 이후, 생명체가 진화했다는 생각은 진화의 철학적, 사회적, 종교적 의미를 중심으로 한 활발한 학문적 논쟁의 원천이었습니다.오늘날 현대의 진화론적 종합은 대다수의 과학자들에 의해 받아들여지고 있습니다.[237]그러나 진화론은 일부 유신론자들에게 논쟁의 여지가 있는 개념으로 남아있습니다.[342]
다양한 종교와 종파들이 유신론적 진화론 등의 개념을 통해 진화론과 자신들의 신념을 조화시킨 반면, 자신들의 종교에서 발견되는 창조신화와는 반대된다고 믿고 진화론에 대해 다양한 이의를 제기하는 창조론자들이 있습니다.[135][343][344]1844년 창조의 자연사의 흔적의 출판에 대한 반응에서 입증되었듯이,진화 생물학의 가장 논쟁적인 측면은 인간이 유인원과 공통적인 조상을 공유하고 인간의 정신적 그리고 도덕적 능력이 동물의 다른 유전적인 특징들과 같은 유형의 자연적인 원인들을 가지고 있다는 인간 진화의 함의입니다.[345]일부 국가, 특히 미국에서는 과학과 종교 간의 이러한 긴장이 정치와 공교육에 초점을 맞춘 종교적 갈등인 창조-진화 논쟁을 부채질하고 있습니다.[346]우주론이나[347] 지구과학과[348] 같은 다른 과학 분야들도 많은 종교적인 문헌들에 대한 문자적인 해석과 상충되는 반면, 진화 생물학은 종교적인 문자주의자들로부터 훨씬 더 많은 반대를 경험합니다.
미국 중등학교 생물학 수업에서 진화에 대한 가르침은 20세기 전반 대부분에서 흔하지 않았습니다.1925년의 Scopes Trial 결정은 한 세대 동안 미국의 중등 생물학 교과서에서 그 주제가 매우 희귀하게 만들었지만, 그것은 나중에 점차 다시 소개되었고 1968년 Epperson v. Arkansas 결정으로 법적으로 보호받게 되었습니다.이후 1970~80년대 다양한 결정으로 중등학교 교육과정에서 창조주의의 경쟁적인 종교적 신념이 법적으로 허용되지 않았으나, 의사과학적 형태로 돌아와 지능형 디자인(ID)으로 2005년 Kitzmiller v.[349] Dover Area 학군 사건에서 다시 한 번 제외되었습니다.다윈의 사상에 대한 논쟁은 중국에서 큰 논란을 일으키지 않았습니다.[350]
참고 항목
- 진화(생물학) – 종이 원시적인 형태로 되돌아갈 수 있다는 개념
참고문헌
- ^ Hall & Hallgrimsson 2008, pp.
- ^ "Evolution Resources". Washington, DC: National Academies of Sciences, Engineering, and Medicine. 2016. Archived from the original on 3 June 2016.
- ^ a b Scott-Phillips, Thomas C.; Laland, Kevin N.; Shuker, David M.; et al. (May 2014). "The Niche Construction Perspective: A Critical Appraisal". Evolution. 68 (5): 1231–1243. doi:10.1111/evo.12332. ISSN 0014-3820. PMC 4261998. PMID 24325256.
Evolutionary processes are generally thought of as processes by which these changes occur. Four such processes are widely recognized: natural selection (in the broad sense, to include sexual selection), genetic drift, mutation, and migration (Fisher 1930; Haldane 1932). The latter two generate variation; the first two sort it.
- ^ Hall & Hallgrimsson 2008, pp.
- ^ Voet, Voet & Pratt 2016, pp. 1-22, 1장: 생명화학개론
- ^ 다윈 1859
- ^ a b Lewontin, Richard C. (November 1970). "The Units of Selection" (PDF). Annual Review of Ecology and Systematics. 1: 1–18. doi:10.1146/annurev.es.01.110170.000245. JSTOR 2096764. Archived (PDF) from the original on 6 February 2015.
- ^ Futuyma & Kirkpatrick 2017, pp. 3-26, 1장: 진화생물학
- ^ 캄푸라키스 2014, 127-129쪽
- ^ a b Doolittle, W. Ford (February 2000). "Uprooting the Tree of Life" (PDF). Scientific American. 282 (2): 90–95. Bibcode:2000SciAm.282b..90D. doi:10.1038/scientificamerican0200-90. ISSN 0036-8733. PMID 10710791. Archived from the original (PDF) on 7 September 2006. Retrieved 5 April 2015.
- ^ Glansdorff, Nicolas; Ying Xu; Labedan, Bernard (9 July 2008). "The Last Universal Common Ancestor: emergence, constitution and genetic legacy of an elusive forerunner". Biology Direct. 3: 29. doi:10.1186/1745-6150-3-29. ISSN 1745-6150. PMC 2478661. PMID 18613974.
- ^ a b Schopf, J. William; Kudryavtsev, Anatoliy B.; Czaja, Andrew D.; Tripathi, Abhishek B. (5 October 2007). "Evidence of Archean life: Stromatolites and microfossils". Precambrian Research. 158 (3–4): 141–155. Bibcode:2007PreR..158..141S. doi:10.1016/j.precamres.2007.04.009. ISSN 0301-9268.
- ^ a b Ohtomo, Yoko; Kakegawa, Takeshi; Ishida, Akizumi; et al. (January 2014). "Evidence for biogenic graphite in early Archaean Isua metasedimentary rocks". Nature Geoscience. 7 (1): 25–28. Bibcode:2014NatGe...7...25O. doi:10.1038/ngeo2025. ISSN 1752-0894.
- ^ a b Borenstein, Seth (13 November 2013). "Oldest fossil found: Meet your microbial mom". Excite. Yonkers, New York: Mindspark Interactive Network. Associated Press. Archived from the original on 29 June 2015. Retrieved 31 May 2015.
- ^ a b Pearlman, Jonathan (13 November 2013). "Oldest signs of life on Earth found". The Daily Telegraph. London. Archived from the original on 16 December 2014. Retrieved 15 December 2014.
- ^ a b Noffke, Nora; Christian, Daniel; Wacey, David; Hazen, Robert M. (16 November 2013). "Microbially Induced Sedimentary Structures Recording an Ancient Ecosystem in the ca. 3.48 Billion-Year-Old Dresser Formation, Pilbara, Western Australia". Astrobiology. 13 (12): 1103–1124. Bibcode:2013AsBio..13.1103N. doi:10.1089/ast.2013.1030. ISSN 1531-1074. PMC 3870916. PMID 24205812.
- ^ Futuyma 2004, 페이지 33
- ^ Panno 2005, pp. xv-16
- ^ NAS 2008, p. 17 2015년 6월 30일 Wayback Machine에서 아카이브
- ^ Futuyma, Douglas J., ed. (1999). "Evolution, Science, and Society: Evolutionary Biology and the National Research Agenda" (PDF) (Executive summary). New Brunswick, New Jersey: Office of University Publications, Rutgers, The State University of New Jersey. OCLC 43422991. Archived from the original (PDF) on 31 January 2012. Retrieved 24 November 2014.
- ^ Sturm, Richard A.; Frudakis, Tony N. (August 2004). "Eye colour: portals into pigmentation genes and ancestry". Trends in Genetics. 20 (8): 327–332. doi:10.1016/j.tig.2004.06.010. ISSN 0168-9525. PMID 15262401.
- ^ a b Pearson, Helen (25 May 2006). "Genetics: What is a gene?". Nature. 441 (7092): 398–401. Bibcode:2006Natur.441..398P. doi:10.1038/441398a. ISSN 0028-0836. PMID 16724031. S2CID 4420674.
- ^ Visscher, Peter M.; Hill, William G.; Wray, Naomi R. (April 2008). "Heritability in the genomics era — concepts and misconceptions". Nature Reviews Genetics. 9 (4): 255–266. doi:10.1038/nrg2322. ISSN 1471-0056. PMID 18319743. S2CID 690431.
- ^ Oetting, William S.; Brilliant, Murray H.; King, Richard A. (August 1996). "The clinical spectrum of albinism in humans". Molecular Medicine Today. 2 (8): 330–335. doi:10.1016/1357-4310(96)81798-9. ISSN 1357-4310. PMID 8796918.
- ^ a b 2005년 후투이마[페이지 필요]
- ^ Phillips, Patrick C. (November 2008). "Epistasis—the essential role of gene interactions in the structure and evolution of genetic systems". Nature Reviews Genetics. 9 (11): 855–867. doi:10.1038/nrg2452. ISSN 1471-0056. PMC 2689140. PMID 18852697.
- ^ a b Rongling Wu; Min Lin (March 2006). "Functional mapping — how to map and study the genetic architecture of dynamic complex traits". Nature Reviews Genetics. 7 (3): 229–237. doi:10.1038/nrg1804. ISSN 1471-0056. PMID 16485021. S2CID 24301815.
- ^ Butlin, Roger K.; Tregenza, Tom (28 February 1998). "Levels of genetic polymorphism: marker loci versus quantitative traits". Philosophical Transactions of the Royal Society B. 353 (1366): 187–198. doi:10.1098/rstb.1998.0201. ISSN 0962-8436. PMC 1692210. PMID 9533123.
- Butlin, Roger K.; Tregenza, Tom (29 December 2000). "Correction for Butlin and Tregenza, Levels of genetic polymorphism: marker loci versus quantitative traits". Philosophical Transactions of the Royal Society B. 355 (1404): 1865. doi:10.1098/rstb.2000.2000. ISSN 0962-8436.
Some of the values in table 1 on p. 193 were given incorrectly. The errors do not affect the conclusions drawn in the paper. The corrected table is reproduced below.
- Butlin, Roger K.; Tregenza, Tom (29 December 2000). "Correction for Butlin and Tregenza, Levels of genetic polymorphism: marker loci versus quantitative traits". Philosophical Transactions of the Royal Society B. 355 (1404): 1865. doi:10.1098/rstb.2000.2000. ISSN 0962-8436.
- ^ Amos, William; Harwood, John (28 February 1998). "Factors affecting levels of genetic diversity in natural populations". Philosophical Transactions of the Royal Society B. 353 (1366): 177–186. doi:10.1098/rstb.1998.0200. ISSN 0962-8436. PMC 1692205. PMID 9533122.
- ^ Futuyma & Kirkpatrick 2017, pp. 79–102, 4장: 돌연변이와 변이
- ^ Keightley, PD (2012). "Rates and fitness consequences of new mutations in humans". Genetics. 190 (2): 295–304. doi:10.1534/genetics.111.134668. PMC 3276617. PMID 22345605.
- ^ Hastings, P. J.; Lupski, James R.; Rosenberg, Susan M.; Ira, Grzegorz (August 2009). "Mechanisms of change in gene copy number". Nature Reviews Genetics. 10 (8): 551–564. doi:10.1038/nrg2593. ISSN 1471-0056. PMC 2864001. PMID 19597530.
- ^ 캐럴, 그레니어 & 웨더비 2005[페이지 필요]
- ^ Harrison, Paul M.; Gerstein, Mark (17 May 2002). "Studying Genomes Through the Aeons: Protein Families, Pseudogenes and Proteome Evolution". Journal of Molecular Biology. 318 (5): 1155–1174. doi:10.1016/S0022-2836(02)00109-2. ISSN 0022-2836. PMID 12083509.
- ^ Bowmaker, James K. (May 1998). "Evolution of colour vision in vertebrates". Eye. 12 (3b): 541–547. doi:10.1038/eye.1998.143. ISSN 0950-222X. PMID 9775215. S2CID 12851209.
- ^ Gregory, T. Ryan; Hebert, Paul D. N. (April 1999). "The Modulation of DNA Content: Proximate Causes and Ultimate Consequences". Genome Research. 9 (4): 317–324. doi:10.1101/gr.9.4.317. ISSN 1088-9051. PMID 10207154. S2CID 16791399. Archived from the original on 23 August 2014. Retrieved 11 December 2014.
- ^ Hurles, Matthew (13 July 2004). "Gene Duplication: The Genomic Trade in Spare Parts". PLOS Biology. 2 (7): e206. doi:10.1371/journal.pbio.0020206. ISSN 1545-7885. PMC 449868. PMID 15252449.
- ^ Liu, Na; Okamura, Katsutomo; Tyler, David M.; et al. (October 2008). "The evolution and functional diversification of animal microRNA genes". Cell Research. 18 (10): 985–996. doi:10.1038/cr.2008.278. ISSN 1001-0602. PMC 2712117. PMID 18711447.
- ^ Siepel, Adam (October 2009). "Darwinian alchemy: Human genes from noncoding DNA". Genome Research. 19 (10): 1693–1695. doi:10.1101/gr.098376.109. ISSN 1088-9051. PMC 2765273. PMID 19797681.
- ^ Orengo, Christine A.; Thornton, Janet M. (July 2005). "Protein families and their evolution—a structural perspective". Annual Review of Biochemistry. Annual Reviews. 74: 867–900. doi:10.1146/annurev.biochem.74.082803.133029. ISSN 0066-4154. PMID 15954844. S2CID 7483470.
- ^ Long, Manyuan; Betrán, Esther; Thornton, Kevin; Wang, Wen (November 2003). "The origin of new genes: glimpses from the young and old". Nature Reviews Genetics. 4 (11): 865–875. doi:10.1038/nrg1204. ISSN 1471-0056. PMID 14634634. S2CID 33999892.
- ^ Wang, Minglei; Caetano-Anollés, Gustavo (14 January 2009). "The Evolutionary Mechanics of Domain Organization in Proteomes and the Rise of Modularity in the Protein World". Structure. 17 (1): 66–78. doi:10.1016/j.str.2008.11.008. ISSN 1357-4310. PMID 19141283.
- ^ Weissman, Kira J.; Müller, Rolf (14 April 2008). "Protein–Protein Interactions in Multienzyme Megasynthetases". ChemBioChem. 9 (6): 826–848. doi:10.1002/cbic.200700751. ISSN 1439-4227. PMID 18357594. S2CID 205552778.
- ^ Andersson, Leif (2020). "Mutations in Domestic Animals Disrupting or Creating Pigmentation Patterns". Frontiers in Ecology and Evolution. 8. doi:10.3389/fevo.2020.00116. ISSN 2296-701X.
- ^ Radding, Charles M. (December 1982). "Homologous Pairing and Strand Exchange in Genetic Recombination". Annual Review of Genetics. 16: 405–437. doi:10.1146/annurev.ge.16.120182.002201. ISSN 0066-4197. PMID 6297377.
- ^ Agrawal, Aneil F. (5 September 2006). "Evolution of Sex: Why Do Organisms Shuffle Their Genotypes?". Current Biology. 16 (17): R696–R704. CiteSeerX 10.1.1.475.9645. doi:10.1016/j.cub.2006.07.063. ISSN 0960-9822. PMID 16950096. S2CID 14739487.
- ^ Peters, Andrew D.; Otto, Sarah P. (June 2003). "Liberating genetic variance through sex". BioEssays. 25 (6): 533–537. doi:10.1002/bies.10291. ISSN 0265-9247. PMID 12766942.
- ^ Goddard, Matthew R.; Godfray, H. Charles J.; Burt, Austin (31 March 2005). "Sex increases the efficacy of natural selection in experimental yeast populations". Nature. 434 (7033): 636–640. Bibcode:2005Natur.434..636G. doi:10.1038/nature03405. ISSN 0028-0836. PMID 15800622. S2CID 4397491.
- ^ 메이너드 스미스 1978[페이지 필요]
- ^ a b 리들리 2004, 페이지 314
- ^ Van Valen, Leigh (1973). "A New Evolutionary Law" (PDF). Evolutionary Theory. 1: 1–30. ISSN 0093-4755. Archived from the original (PDF) on 22 December 2014. Retrieved 24 December 2014.
- ^ Hamilton, W. D.; Axelrod, Robert; Tanese, Reiko (1 May 1990). "Sexual reproduction as an adaptation to resist parasites (a review)". PNAS. 87 (9): 3566–3573. Bibcode:1990PNAS...87.3566H. doi:10.1073/pnas.87.9.3566. ISSN 0027-8424. PMC 53943. PMID 2185476.
- ^ Birdsell & Wills 2003, pp. 113–117
- ^ 번스타인 H, 바이얼리 HC, 호프 FA, 미코드 RE.유전적 손상, 돌연변이 그리고 성의 진화.과학. 1985. 9. 20. 229(4719): 1277-81. 도이: 10.1126/과학.3898363.PMID 3898363
- ^ 번스타인 H, 호프 FA, 미코드 RE.섹스의 진화의 분자적 기초.Adv Genet. 1987;24:323-70. Doi:10.1016/s0065-2660(08)60012-7PMID 3324702
- ^ Morjan, Carrie L.; Rieseberg, Loren H. (June 2004). "How species evolve collectively: implications of gene flow and selection for the spread of advantageous alleles". Molecular Ecology. 13 (6): 1341–1356. doi:10.1111/j.1365-294X.2004.02164.x. ISSN 0962-1083. PMC 2600545. PMID 15140081.
- ^ Boucher, Yan; Douady, Christophe J.; Papke, R. Thane; et al. (December 2003). "Lateral gene transfer and the origins of prokaryotic groups". Annual Review of Genetics. 37: 283–328. doi:10.1146/annurev.genet.37.050503.084247. ISSN 0066-4197. PMID 14616063.
- ^ Walsh, Timothy R. (October 2006). "Combinatorial genetic evolution of multiresistance". Current Opinion in Microbiology. 9 (5): 476–482. doi:10.1016/j.mib.2006.08.009. ISSN 1369-5274. PMID 16942901.
- ^ Kondo, Natsuko; Nikoh, Naruo; Ijichi, Nobuyuki; et al. (29 October 2002). "Genome fragment of Wolbachia endosymbiont transferred to X chromosome of host insect". PNAS. 99 (22): 14280–14285. Bibcode:2002PNAS...9914280K. doi:10.1073/pnas.222228199. ISSN 0027-8424. PMC 137875. PMID 12386340.
- ^ Sprague, George F. Jr. (December 1991). "Genetic exchange between kingdoms". Current Opinion in Genetics & Development. 1 (4): 530–533. doi:10.1016/S0959-437X(05)80203-5. ISSN 0959-437X. PMID 1822285.
- ^ Gladyshev, Eugene A.; Meselson, Matthew; Arkhipova, Irina R. (30 May 2008). "Massive Horizontal Gene Transfer in Bdelloid Rotifers". Science. 320 (5880): 1210–1213. Bibcode:2008Sci...320.1210G. doi:10.1126/science.1156407. ISSN 0036-8075. PMID 18511688. S2CID 11862013. Archived from the original on 30 July 2022. Retrieved 30 July 2022.
- ^ Baldo, Angela M.; McClure, Marcella A. (September 1999). "Evolution and Horizontal Transfer of dUTPase-Encoding Genes in Viruses and Their Hosts". Journal of Virology. 73 (9): 7710–7721. doi:10.1128/JVI.73.9.7710-7721.1999. ISSN 0022-538X. PMC 104298. PMID 10438861.
- ^ Rivera, Maria C.; Lake, James A. (9 September 2004). "The ring of life provides evidence for a genome fusion origin of eukaryotes". Nature. 431 (7005): 152–155. Bibcode:2004Natur.431..152R. doi:10.1038/nature02848. ISSN 0028-0836. PMID 15356622. S2CID 4349149.
- ^ Jablonka, Eva; Raz, Gal (June 2009). "Transgenerational Epigenetic Inheritance: Prevalence, Mechanisms, and Implications for the Study of Heredity and Evolution" (PDF). The Quarterly Review of Biology. 84 (2): 131–176. CiteSeerX 10.1.1.617.6333. doi:10.1086/598822. ISSN 0033-5770. PMID 19606595. S2CID 7233550. Archived (PDF) from the original on 15 July 2011. Retrieved 30 July 2022.
- ^ Bossdorf, Oliver; Arcuri, Davide; Richards, Christina L.; Pigliucci, Massimo (May 2010). "Experimental alteration of DNA methylation affects the phenotypic plasticity of ecologically relevant traits in Arabidopsis thaliana" (PDF). Evolutionary Ecology. 24 (3): 541–553. doi:10.1007/s10682-010-9372-7. ISSN 0269-7653. S2CID 15763479. Archived (PDF) from the original on 5 June 2022. Retrieved 30 July 2022.
- ^ Jablonka, Eva; Lamb, Marion J. (December 2002). "The Changing Concept of Epigenetics". Annals of the New York Academy of Sciences. 981 (1): 82–96. Bibcode:2002NYASA.981...82J. doi:10.1111/j.1749-6632.2002.tb04913.x. ISSN 0077-8923. PMID 12547675. S2CID 12561900.
- ^ Laland, Kevin N.; Sterelny, Kim (September 2006). "Perspective: Seven Reasons (Not) to Neglect Niche Construction". Evolution. 60 (9): 1751–1762. doi:10.1111/j.0014-3820.2006.tb00520.x. ISSN 0014-3820. PMID 17089961. S2CID 22997236.
- ^ Chapman, Michael J.; Margulis, Lynn (December 1998). "Morphogenesis by symbiogenesis" (PDF). International Microbiology. 1 (4): 319–326. ISSN 1139-6709. PMID 10943381. Archived from the original (PDF) on 23 August 2014. Retrieved 9 December 2014.
- ^ Wilson, David Sloan; Wilson, Edward O. (December 2007). "Rethinking the Theoretical Foundation of Sociobiology" (PDF). The Quarterly Review of Biology. 82 (4): 327–348. doi:10.1086/522809. ISSN 0033-5770. PMID 18217526. S2CID 37774648. Archived from the original (PDF) on 11 May 2011.
- ^ 에웬스 2004[페이지 필요]
- ^ a b c Hurst, Laurence D. (February 2009). "Fundamental concepts in genetics: genetics and the understanding of selection". Nature Reviews Genetics. 10 (2): 83–93. doi:10.1038/nrg2506. PMID 19119264. S2CID 1670587.
- ^ 다윈 1859, 제14장
- ^ Otto, Sarah P.; Servedio, Maria R.; Nuismer, Scott L. (August 2008). "Frequency-Dependent Selection and the Evolution of Assortative Mating". Genetics. 179 (4): 2091–2112. doi:10.1534/genetics.107.084418. PMC 2516082. PMID 18660541.
- ^ a b c Orr, H. Allen (August 2009). "Fitness and its role in evolutionary genetics". Nature Reviews Genetics. 10 (8): 531–539. doi:10.1038/nrg2603. ISSN 1471-0056. PMC 2753274. PMID 19546856.
- ^ Haldane, J. B. S. (14 March 1959). "The Theory of Natural Selection To-Day". Nature. 183 (4663): 710–713. Bibcode:1959Natur.183..710H. doi:10.1038/183710a0. PMID 13644170. S2CID 4185793.
- ^ Lande, Russell; Arnold, Stevan J. (November 1983). "The Measurement of Selection on Correlated Characters". Evolution. 37 (6): 1210–1226. doi:10.1111/j.1558-5646.1983.tb00236.x. ISSN 0014-3820. JSTOR 2408842. PMID 28556011. S2CID 36544045.
- ^ Goldberg, Emma E.; Igić, Boris (November 2008). "On phylogenetic tests of irreversible evolution". Evolution. 62 (11): 2727–2741. doi:10.1111/j.1558-5646.2008.00505.x. ISSN 0014-3820. PMID 18764918. S2CID 30703407.
- ^ Collin, Rachel; Miglietta, Maria Pia (November 2008). "Reversing opinions on Dollo's Law". Trends in Ecology & Evolution. 23 (11): 602–609. doi:10.1016/j.tree.2008.06.013. PMID 18814933.
- ^ Tomić, Nenad; Meyer-Rochow, Victor Benno (2011). "Atavisms: Medical, Genetic, and Evolutionary Implications". Perspectives in Biology and Medicine. 54 (3): 332–353. doi:10.1353/pbm.2011.0034. PMID 21857125. S2CID 40851098.
- ^ Hoekstra, Hopi E.; Hoekstra, Jonathan M.; Berrigan, David; et al. (31 July 2001). "Strength and tempo of directional selection in the wild". PNAS. 98 (16): 9157–9160. Bibcode:2001PNAS...98.9157H. doi:10.1073/pnas.161281098. PMC 55389. PMID 11470913.
- ^ Felsenstein, Joseph (November 1979). "Excursions along the Interface between Disruptive and Stabilizing Selection". Genetics. 93 (3): 773–795. doi:10.1093/genetics/93.3.773. PMC 1214112. PMID 17248980.
- ^ Odum 1971, p. 8
- ^ 오카샤 2006년
- ^ a b Gould, Stephen Jay (28 February 1998). "Gulliver's further travels: the necessity and difficulty of a hierarchical theory of selection". Philosophical Transactions of the Royal Society B. 353 (1366): 307–314. doi:10.1098/rstb.1998.0211. ISSN 0962-8436. PMC 1692213. PMID 9533127.
- ^ Mayr, Ernst (18 March 1997). "The objects of selection". PNAS. 94 (6): 2091–2094. Bibcode:1997PNAS...94.2091M. doi:10.1073/pnas.94.6.2091. ISSN 0027-8424. PMC 33654. PMID 9122151.
- ^ Maynard Smith 1998, 페이지 203-211, 토론 211-217
- ^ Hickey, Donal A. (1992). "Evolutionary dynamics of transposable elements in prokaryotes and eukaryotes". Genetica. 86 (1–3): 269–274. doi:10.1007/BF00133725. ISSN 0016-6707. PMID 1334911. S2CID 6583945.
- ^ Gould, Stephen Jay; Lloyd, Elisabeth A. (12 October 1999). "Individuality and adaptation across levels of selection: how shall we name and generalise the unit of Darwinism?". PNAS. 96 (21): 11904–11909. Bibcode:1999PNAS...9611904G. doi:10.1073/pnas.96.21.11904. ISSN 0027-8424. PMC 18385. PMID 10518549.
- ^ Futuyma & Kirkpatrick 2017, pp. 55-66, 3장: 자연선택과 적응
- ^ a b Masel, Joanna (25 October 2011). "Genetic drift". Current Biology. 21 (20): R837–R838. doi:10.1016/j.cub.2011.08.007. ISSN 0960-9822. PMID 22032182. S2CID 17619958.
- ^ Lande, Russell (1989). "Fisherian and Wrightian theories of speciation". Genome. 31 (1): 221–227. doi:10.1139/g89-037. ISSN 0831-2796. PMID 2687093.
- ^ Kimura, Motoo (1991). "The neutral theory of molecular evolution: a review of recent evidence". Japanese Journal of Human Genetics. 66 (4): 367–386. doi:10.1266/jjg.66.367. PMID 1954033. Archived from the original on 5 June 2022.
- ^ Kimura, Motoo (1989). "The neutral theory of molecular evolution and the world view of the neutralists". Genome. 31 (1): 24–31. doi:10.1139/g89-009. ISSN 0831-2796. PMID 2687096.
- ^ Kreitman, Martin (August 1996). "The neutral theory is dead. Long live the neutral theory". BioEssays. 18 (8): 678–683, discussion 683. doi:10.1002/bies.950180812. ISSN 0265-9247. PMID 8760341.
- ^ Leigh, E. G. Jr. (November 2007). "Neutral theory: a historical perspective". Journal of Evolutionary Biology. 20 (6): 2075–2091. doi:10.1111/j.1420-9101.2007.01410.x. ISSN 1010-061X. PMID 17956380. S2CID 2081042.
- ^ a b Gillespie, John H. (November 2001). "Is the population size of a species relevant to its evolution?". Evolution. 55 (11): 2161–2169. doi:10.1111/j.0014-3820.2001.tb00732.x. ISSN 0014-3820. PMID 11794777. S2CID 221735887.
- ^ Neher, Richard A.; Shraiman, Boris I. (August 2011). "Genetic Draft and Quasi-Neutrality in Large Facultatively Sexual Populations". Genetics. 188 (4): 975–996. arXiv:1108.1635. Bibcode:2011arXiv1108.1635N. doi:10.1534/genetics.111.128876. PMC 3176096. PMID 21625002.
- ^ Stoltzfus, Arlin (1999). "On the Possibility of Constructive Neutral Evolution". Journal of Molecular Evolution. 49 (2): 169–181. Bibcode:1999JMolE..49..169S. doi:10.1007/PL00006540. PMID 10441669. S2CID 1743092. Archived from the original on 30 July 2022. Retrieved 30 July 2022.
- ^ Stoltzfus, Arlin (13 October 2012). "Constructive neutral evolution: exploring evolutionary theory's curious disconnect". Biology Direct. 7 (1): 35. doi:10.1186/1745-6150-7-35. PMC 3534586. PMID 23062217.
- ^ Muñoz-Gómez, Sergio A.; Bilolikar, Gaurav; Wideman, Jeremy G.; et al. (1 April 2021). "Constructive Neutral Evolution 20 Years Later". Journal of Molecular Evolution. 89 (3): 172–182. Bibcode:2021JMolE..89..172M. doi:10.1007/s00239-021-09996-y. PMC 7982386. PMID 33604782. Archived from the original on 30 July 2022. Retrieved 30 July 2022.
- ^ Lukeš, Julius; Archibald, John M.; Keeling, Patrick J.; et al. (2011). "How a neutral evolutionary ratchet can build cellular complexity". IUBMB Life. 63 (7): 528–537. doi:10.1002/iub.489. PMID 21698757. S2CID 7306575.
- ^ Vosseberg, Julian; Snel, Berend (1 December 2017). "Domestication of self-splicing introns during eukaryogenesis: the rise of the complex spliceosomal machinery". Biology Direct. 12 (1): 30. doi:10.1186/s13062-017-0201-6. PMC 5709842. PMID 29191215.
- ^ Brunet, T. D. P.; Doolittle, W. Ford (19 March 2018). "The generality of Constructive Neutral Evolution". Biology & Philosophy. 33 (1): 2. doi:10.1007/s10539-018-9614-6. S2CID 90290787. Archived from the original on 30 July 2022. Retrieved 30 July 2022.
- ^ Otto, Sarah P.; Whitlock, Michael C. (June 1997). "The Probability of Fixation in Populations of Changing Size" (PDF). Genetics. 146 (2): 723–733. doi:10.1093/genetics/146.2.723. PMC 1208011. PMID 9178020. Archived (PDF) from the original on 19 March 2015. Retrieved 18 December 2014.
- ^ a b Charlesworth, Brian (March 2009). "Fundamental concepts in genetics: effective population size and patterns of molecular evolution and variation". Nature Reviews Genetics. 10 (3): 195–205. doi:10.1038/nrg2526. PMID 19204717. S2CID 205484393.
- ^ Cutter, Asher D.; Choi, Jae Young (August 2010). "Natural selection shapes nucleotide polymorphism across the genome of the nematode Caenorhabditis briggsae". Genome Research. 20 (8): 1103–1111. doi:10.1101/gr.104331.109. PMC 2909573. PMID 20508143.
- ^ Mitchell-Olds, Thomas; Willis, John H.; Goldstein, David B. (November 2007). "Which evolutionary processes influence natural genetic variation for phenotypic traits?". Nature Reviews Genetics. 8 (11): 845–856. doi:10.1038/nrg2207. ISSN 1471-0056. PMID 17943192. S2CID 14914998.
- ^ Nei, Masatoshi (December 2005). "Selectionism and Neutralism in Molecular Evolution". Molecular Biology and Evolution. 22 (12): 2318–2342. doi:10.1093/molbev/msi242. ISSN 0737-4038. PMC 1513187. PMID 16120807.
- Nei, Masatoshi (May 2006). "Selectionism and Neutralism in Molecular Evolution". Molecular Biology and Evolution (Erratum). 23 (5): 2318–42. doi:10.1093/molbev/msk009. ISSN 0737-4038. PMC 1513187. PMID 16120807.
- ^ Haldane, J.B.S. (July 1927). "A Mathematical Theory of Natural and Artificial Selection, Part V: Selection and Mutation". Proceedings of the Cambridge Philosophical Society. 26 (7): 838–844. Bibcode:1927PCPS...23..838H. doi:10.1017/S0305004100015644. S2CID 86716613.
- ^ 피셔 1930
- ^ a b c Yampolsky, Lev Y.; Stoltzfus, Arlin (20 December 2001). "Bias in the introduction of variation as an orienting factor in evolution". Evolution & Development. 3 (2): 73–83. doi:10.1046/j.1525-142x.2001.003002073.x. PMID 11341676. S2CID 26956345.
- ^ Sueoka, Noboru (1 April 1962). "On the Genetic Basis of Variation and Heterogeneity of DNA Base Composition". PNAS. 48 (4): 582–592. Bibcode:1962PNAS...48..582S. doi:10.1073/pnas.48.4.582. PMC 220819. PMID 13918161.
- ^ Freese, Ernst (July 1962). "On the Evolution of the Base Composition of DNA". Journal of Theoretical Biology. 3 (1): 82–101. Bibcode:1962JThBi...3...82F. doi:10.1016/S0022-5193(62)80005-8.
- ^ Cox, Edward C.; Yanofsky, Charles (1 November 1967). "Altered base ratios in the DNA of an Escherichia coli mutator strain". Proc. Natl. Acad. Sci. USA. 58 (5): 1895–1902. Bibcode:1967PNAS...58.1895C. doi:10.1073/pnas.58.5.1895. PMC 223881. PMID 4866980.
- ^ Shah, Premal; Gilchrist, Michael A. (21 June 2011). "Explaining complex codon usage patterns with selection for translational efficiency, mutation bias, and genetic drift". PNAS. 108 (25): 10231–10236. Bibcode:2011PNAS..10810231S. doi:10.1073/pnas.1016719108. PMC 3121864. PMID 21646514.
- ^ Bulmer, Michael G. (November 1991). "The selection-mutation-drift theory of synonymous codon usage". Genetics. 129 (3): 897–907. doi:10.1093/genetics/129.3.897. PMC 1204756. PMID 1752426.
- ^ Fryxell, Karl J.; Zuckerkandl, Emile (September 2000). "Cytosine Deamination Plays a Primary Role in the Evolution of Mammalian Isochores". Molecular Biology and Evolution. 17 (9): 1371–1383. doi:10.1093/oxfordjournals.molbev.a026420. PMID 10958853.
- ^ Petrov, Dmitri A.; Sangster, Todd A.; Johnston, J. Spencer; et al. (11 February 2000). "Evidence for DNA Loss as a Determinant of Genome Size". Science. 287 (5455): 1060–1062. Bibcode:2000Sci...287.1060P. doi:10.1126/science.287.5455.1060. ISSN 0036-8075. PMID 10669421. S2CID 12021662.
- ^ Petrov, Dmitri A. (May 2002). "DNA loss and evolution of genome size in Drosophila". Genetica. 115 (1): 81–91. doi:10.1023/A:1016076215168. ISSN 0016-6707. PMID 12188050. S2CID 5314242.
- ^ Duret, Laurent; Galtier, Nicolas (September 2009). "Biased Gene Conversion and the Evolution of Mammalian Genomic Landscapes". Annual Review of Genomics and Human Genetics. Annual Reviews. 10: 285–311. doi:10.1146/annurev-genom-082908-150001. PMID 19630562. S2CID 9126286.
- ^ Hershberg, Ruth; Petrov, Dmitri A. (9 September 2010). "Evidence That Mutation Is Universally Biased towards AT in Bacteria". PLOS Genetics. 6 (9): e1001115. doi:10.1371/journal.pgen.1001115. PMC 2936535. PMID 20838599.
- ^ A. Stoltzfus (2019). "Understanding bias in the introduction of variation as an evolutionary cause". In Uller, T.; Laland, K.N. (eds.). Evolutionary Causation: Biological and Philosophical Reflections. Cambridge, MA: MIT Press.
- ^ Stoltzfus, Arlin; McCandlish, David M. (September 2017). "Mutational Biases Influence Parallel Adaptation". Molecular Biology and Evolution. 34 (9): 2163–2172. doi:10.1093/molbev/msx180. PMC 5850294. PMID 28645195.
- ^ Payne, Joshua L.; Menardo, Fabrizio; Trauner, Andrej; et al. (13 May 2019). "Transition bias influences the evolution of antibiotic resistance in Mycobacterium tuberculosis". PLOS Biology. 17 (5): e3000265. doi:10.1371/journal.pbio.3000265. PMC 6532934. PMID 31083647.
- ^ Storz, Jay F.; Natarajan, Chandrasekhar; Signore, Anthony V.; et al. (22 July 2019). "The role of mutation bias in adaptive molecular evolution: insights from convergent changes in protein function". Philosophical Transactions of the Royal Society B. 374 (1777): 20180238. doi:10.1098/rstb.2018.0238. PMC 6560279. PMID 31154983.
- ^ Svensson, Erik I.; Berger, David (1 May 2019). "The Role of Mutation Bias in Adaptive Evolution". Trends in Ecology & Evolution. 34 (5): 422–434. doi:10.1016/j.tree.2019.01.015. PMID 31003616. S2CID 125066709.
- ^ Lien, Sigbjørn; Szyda, Joanna; Schechinger, Birgit; et al. (February 2000). "Evidence for Heterogeneity in Recombination in the Human Pseudoautosomal Region: High Resolution Analysis by Sperm Typing and Radiation-Hybrid Mapping". American Journal of Human Genetics. 66 (2): 557–566. doi:10.1086/302754. ISSN 0002-9297. PMC 1288109. PMID 10677316.
- ^ Barton, Nicholas H. (29 November 2000). "Genetic hitchhiking". Philosophical Transactions of the Royal Society B. 355 (1403): 1553–1562. doi:10.1098/rstb.2000.0716. ISSN 0962-8436. PMC 1692896. PMID 11127900.
- ^ Ries, C; Spaethe, J; Sztatecsny, M; Strondl, C; Hödl, W (20 October 2008). "Turning blue and ultraviolet: sex-specific colour change during the mating season in the Balkan moor frog". Journal of Zoology. 276 (3): 229–236. doi:10.1111/j.1469-7998.2008.00456.x – via Google Scholar.
- ^ Andersson, Malte; Simmons, Leigh W. (June 2006). "Sexual selection and mate choice" (PDF). Trends in Ecology & Evolution. 21 (6): 296–302. CiteSeerX 10.1.1.595.4050. doi:10.1016/j.tree.2006.03.015. ISSN 0169-5347. PMID 16769428. Archived (PDF) from the original on 9 March 2013.
- ^ Kokko, Hanna; Brooks, Robert; McNamara, John M.; Houston, Alasdair I. (7 July 2002). "The sexual selection continuum". Proceedings of the Royal Society B. 269 (1498): 1331–1340. doi:10.1098/rspb.2002.2020. ISSN 0962-8452. PMC 1691039. PMID 12079655.
- ^ Quinn, Thomas P.; Hendry, Andrew P.; Buck, Gregory B. (2001). "Balancing natural and sexual selection in sockeye salmon: interactions between body size, reproductive opportunity and vulnerability to predation by bears" (PDF). Evolutionary Ecology Research. 3: 917–937. ISSN 1522-0613. Archived (PDF) from the original on 5 March 2016. Retrieved 15 December 2014.
- ^ Hunt, John; Brooks, Robert; Jennions, Michael D.; et al. (23 December 2004). "High-quality male field crickets invest heavily in sexual display but die young". Nature. 432 (7020): 1024–1027. Bibcode:2004Natur.432.1024H. doi:10.1038/nature03084. ISSN 0028-0836. PMID 15616562. S2CID 4417867.
- ^ Baym, Michael; Lieberman, Tami D.; Kelsic, Eric D.; et al. (9 September 2016). "Spatiotemporal microbial evolution on antibiotic landscapes". Science. 353 (6304): 1147–1151. Bibcode:2016Sci...353.1147B. doi:10.1126/science.aag0822. ISSN 0036-8075. PMC 5534434. PMID 27609891.
- ^ a b c Scott, Eugenie C.; Matzke, Nicholas J. (15 May 2007). "Biological design in science classrooms". PNAS. 104 (Suppl. 1): 8669–8676. Bibcode:2007PNAS..104.8669S. doi:10.1073/pnas.0701505104. PMC 1876445. PMID 17494747.
- ^ Hendry, Andrew Paul; Kinnison, Michael T. (November 2001). "An introduction to microevolution: rate, pattern, process". Genetica. 112–113 (1): 1–8. doi:10.1023/A:1013368628607. ISSN 0016-6707. PMID 11838760. S2CID 24485535.
- ^ Leroi, Armand M. (March–April 2000). "The scale independence of evolution". Evolution & Development. 2 (2): 67–77. CiteSeerX 10.1.1.120.1020. doi:10.1046/j.1525-142x.2000.00044.x. ISSN 1520-541X. PMID 11258392. S2CID 17289010.
- ^ Gould 2002, pp. 657–658.
- ^ a b Gould, Stephen Jay (19 July 1994). "Tempo and mode in the macroevolutionary reconstruction of Darwinism". PNAS. 91 (15): 6764–6771. Bibcode:1994PNAS...91.6764G. doi:10.1073/pnas.91.15.6764. PMC 44281. PMID 8041695.
- ^ Jablonski, David (2000). "Micro- and macroevolution: scale and hierarchy in evolutionary biology and paleobiology". Paleobiology. 26 (sp4): 15–52. doi:10.1666/0094-8373(2000)26[15:MAMSAH]2.0.CO;2. S2CID 53451360.
- ^ Dougherty, Michael J. (20 July 1998). "Is the human race evolving or devolving?". Scientific American. ISSN 0036-8733. Archived from the original on 6 May 2014. Retrieved 11 September 2015.
- ^ Isaak, Mark, ed. (22 July 2003). "Claim CB932: Evolution of degenerate forms". TalkOrigins Archive. Houston, Texas: The TalkOrigins Foundation. Archived from the original on 23 August 2014. Retrieved 19 December 2014.
- ^ 레인 1996, 페이지 61
- ^ Carroll, Sean B. (22 February 2001). "Chance and necessity: the evolution of morphological complexity and diversity". Nature. 409 (6823): 1102–1109. Bibcode:2001Natur.409.1102C. doi:10.1038/35059227. PMID 11234024. S2CID 4319886.
- ^ Whitman, William B.; Coleman, David C.; Wiebe, William J. (9 June 1998). "Prokaryotes: The unseen majority". PNAS. 95 (12): 6578–6583. Bibcode:1998PNAS...95.6578W. doi:10.1073/pnas.95.12.6578. ISSN 0027-8424. PMC 33863. PMID 9618454.
- ^ a b Schloss, Patrick D.; Handelsman, Jo (December 2004). "Status of the Microbial Census". Microbiology and Molecular Biology Reviews. 68 (4): 686–691. doi:10.1128/MMBR.68.4.686-691.2004. PMC 539005. PMID 15590780.
- ^ Nealson, Kenneth H. (January 1999). "Post-Viking microbiology: new approaches, new data, new insights". Origins of Life and Evolution of Biospheres. 29 (1): 73–93. Bibcode:1999OLEB...29...73N. doi:10.1023/A:1006515817767. ISSN 0169-6149. PMID 11536899. S2CID 12289639.
- ^ Buckling, Angus; MacLean, R. Craig; Brockhurst, Michael A.; Colegrave, Nick (12 February 2009). "The Beagle in a bottle". Nature. 457 (7231): 824–829. Bibcode:2009Natur.457..824B. doi:10.1038/nature07892. ISSN 0028-0836. PMID 19212400. S2CID 205216404.
- ^ Elena, Santiago F.; Lenski, Richard E. (June 2003). "Evolution experiments with microorganisms: the dynamics and genetic bases of adaptation". Nature Reviews Genetics. 4 (6): 457–469. doi:10.1038/nrg1088. ISSN 1471-0056. PMID 12776215. S2CID 209727.
- ^ 1982년 5월, 페이지 483: "적응...더 이상 정적인 조건, 창의적인 과거의 산물로 간주될 수 없으며 대신 지속적인 동적 과정이 되었습니다."
- ^ 옥스포드 과학사전 제6판(2010)은 적응을 "인구의 연속적인 세대의 구조나 기능의 변화가 환경에 더 잘 적합하게 만드는 것"으로 정의합니다.
- ^ Orr, H. Allen (February 2005). "The genetic theory of adaptation: a brief history". Nature Reviews Genetics. 6 (2): 119–127. doi:10.1038/nrg1523. ISSN 1471-0056. PMID 15716908. S2CID 17772950.
- ^ Dobzhansky 1968, 페이지 1-34
- ^ Dobzhansky 1970, pp. 4–6, 79–82, 84–87
- ^ Dobzhansky, Theodosius (March 1956). "Genetics of Natural Populations. XXV. Genetic Changes in Populations of Drosophila pseudoobscura and Drosophila persimilis in Some Localities in California". Evolution. 10 (1): 82–92. doi:10.2307/2406099. ISSN 0014-3820. JSTOR 2406099.
- ^ Nakajima, Akira; Sugimoto, Yohko; Yoneyama, Hiroshi; et al. (June 2002). "High-Level Fluoroquinolone Resistance in Pseudomonas aeruginosa Due to Interplay of the MexAB-OprM Efflux Pump and the DNA Gyrase Mutation". Microbiology and Immunology. 46 (6): 391–395. doi:10.1111/j.1348-0421.2002.tb02711.x. ISSN 1348-0421. PMID 12153116. S2CID 22593331.
- ^ Blount, Zachary D.; Borland, Christina Z.; Lenski, Richard E. (10 June 2008). "Inaugural Article: Historical contingency and the evolution of a key innovation in an experimental population of Escherichia coli". PNAS. 105 (23): 7899–7906. Bibcode:2008PNAS..105.7899B. doi:10.1073/pnas.0803151105. ISSN 0027-8424. PMC 2430337. PMID 18524956.
- ^ Okada, Hirosuke; Negoro, Seiji; Kimura, Hiroyuki; et al. (10 November 1983). "Evolutionary adaptation of plasmid-encoded enzymes for degrading nylon oligomers". Nature. 306 (5939): 203–206. Bibcode:1983Natur.306..203O. doi:10.1038/306203a0. ISSN 0028-0836. PMID 6646204. S2CID 4364682.
- ^ Ohno, Susumu (April 1984). "Birth of a unique enzyme from an alternative reading frame of the preexisted, internally repetitious coding sequence". PNAS. 81 (8): 2421–2425. Bibcode:1984PNAS...81.2421O. doi:10.1073/pnas.81.8.2421. ISSN 0027-8424. PMC 345072. PMID 6585807.
- ^ Copley, Shelley D. (June 2000). "Evolution of a metabolic pathway for degradation of a toxic xenobiotic: the patchwork approach". Trends in Biochemical Sciences. 25 (6): 261–265. doi:10.1016/S0968-0004(00)01562-0. ISSN 0968-0004. PMID 10838562.
- ^ Crawford, Ronald L.; Jung, Carina M.; Strap, Janice L. (October 2007). "The recent evolution of pentachlorophenol (PCP)-4-monooxygenase (PcpB) and associated pathways for bacterial degradation of PCP". Biodegradation. 18 (5): 525–539. doi:10.1007/s10532-006-9090-6. ISSN 0923-9820. PMID 17123025. S2CID 8174462.
- ^ 알텐베르크 1995, 페이지 205-259
- ^ Masel, Joanna; Bergman, Aviv (July 2003). "The evolution of the evolvability properties of the yeast prion [PSI+]". Evolution. 57 (7): 1498–1512. doi:10.1111/j.0014-3820.2003.tb00358.x. PMID 12940355. S2CID 30954684.
- ^ Lancaster, Alex K.; Bardill, J. Patrick; True, Heather L.; Masel, Joanna (February 2010). "The Spontaneous Appearance Rate of the Yeast Prion [PSI+] and Its Implications for the Evolution of the Evolvability Properties of the [PSI+] System". Genetics. 184 (2): 393–400. doi:10.1534/genetics.109.110213. ISSN 0016-6731. PMC 2828720. PMID 19917766.
- ^ Draghi, Jeremy; Wagner, Günter P. (February 2008). "Evolution of evolvability in a developmental model". Evolution. 62 (2): 301–315. doi:10.1111/j.1558-5646.2007.00303.x. PMID 18031304. S2CID 11560256.
- ^ a b Bejder, Lars; Hall, Brian K. (November 2002). "Limbs in whales and limblessness in other vertebrates: mechanisms of evolutionary and developmental transformation and loss". Evolution & Development. 4 (6): 445–458. doi:10.1046/j.1525-142X.2002.02033.x. PMID 12492145. S2CID 8448387.
- ^ Young, Nathan M.; HallgrÍmsson, Benedikt (December 2005). "Serial homology and the evolution of mammalian limb covariation structure". Evolution. 59 (12): 2691–2704. doi:10.1554/05-233.1. ISSN 0014-3820. PMID 16526515. S2CID 198156135.
- ^ a b Penny, David; Poole, Anthony (December 1999). "The nature of the last universal common ancestor". Current Opinion in Genetics & Development. 9 (6): 672–677. doi:10.1016/S0959-437X(99)00020-9. PMID 10607605.
- ^ Hall, Brian K. (August 2003). "Descent with modification: the unity underlying homology and homoplasy as seen through an analysis of development and evolution". Biological Reviews. 78 (3): 409–433. doi:10.1017/S1464793102006097. ISSN 1464-7931. PMID 14558591. S2CID 22142786.
- ^ Shubin, Neil; Tabin, Clifford J.; Carroll, Sean B. (12 February 2009). "Deep homology and the origins of evolutionary novelty". Nature. 457 (7231): 818–823. Bibcode:2009Natur.457..818S. doi:10.1038/nature07891. PMID 19212399. S2CID 205216390.
- ^ a b c Fong, Daniel F.; Kane, Thomas C.; Culver, David C. (November 1995). "Vestigialization and Loss of Nonfunctional Characters". Annual Review of Ecology and Systematics. 26: 249–268. doi:10.1146/annurev.es.26.110195.001341.
- ^ ZhaoLei Zhang; Gerstein, Mark (August 2004). "Large-scale analysis of pseudogenes in the human genome". Current Opinion in Genetics & Development. 14 (4): 328–335. doi:10.1016/j.gde.2004.06.003. ISSN 0959-437X. PMID 15261647.
- ^ Jeffery, William R. (May–June 2005). "Adaptive Evolution of Eye Degeneration in the Mexican Blind Cavefish". Journal of Heredity. 96 (3): 185–196. CiteSeerX 10.1.1.572.6605. doi:10.1093/jhered/esi028. PMID 15653557.
- ^ Maxwell, Erin E.; Larsson, Hans C.E. (May 2007). "Osteology and myology of the wing of the Emu (Dromaius novaehollandiae) and its bearing on the evolution of vestigial structures". Journal of Morphology. 268 (5): 423–441. doi:10.1002/jmor.10527. ISSN 0362-2525. PMID 17390336. S2CID 12494187.
- ^ van der Kooi, Casper J.; Schwander, Tanja (November 2014). "On the fate of sexual traits under asexuality" (PDF). Biological Reviews. 89 (4): 805–819. doi:10.1111/brv.12078. ISSN 1464-7931. PMID 24443922. S2CID 33644494. Archived (PDF) from the original on 23 July 2015. Retrieved 5 August 2015.
- ^ Silvestri, Anthony R. Jr.; Singh, Iqbal (April 2003). "The unresolved problem of the third molar: Would people be better off without it?". Journal of the American Dental Association. 134 (4): 450–455. doi:10.14219/jada.archive.2003.0194. PMID 12733778. Archived from the original on 23 August 2014.
- ^ Coyne 2009, 페이지 62
- ^ 다윈 1872, 페이지 101, 103
- ^ 그레이 2007, 페이지 66
- ^ Coyne 2009, 페이지 85-86
- ^ Stevens 1982, 페이지 87
- ^ a b Gould 2002, pp. 1235–1236.
- ^ Pallen, Mark J.; Matzke, Nicholas J. (October 2006). "From The Origin of Species to the origin of bacterial flagella" (PDF). Nature Reviews Microbiology (PDF). 4 (10): 784–790. doi:10.1038/nrmicro1493. ISSN 1740-1526. PMID 16953248. S2CID 24057949. Archived from the original (PDF) on 26 December 2014. Retrieved 25 December 2014.
- ^ Clements, Abigail; Bursac, Dejan; Gatsos, Xenia; et al. (15 September 2009). "The reducible complexity of a mitochondrial molecular machine". PNAS. 106 (37): 15791–15795. Bibcode:2009PNAS..10615791C. doi:10.1073/pnas.0908264106. PMC 2747197. PMID 19717453.
- ^ Piatigorsky et al. 1994, pp. 241-250
- ^ Wistow, Graeme (August 1993). "Lens crystallins: gene recruitment and evolutionary dynamism". Trends in Biochemical Sciences. 18 (8): 301–306. doi:10.1016/0968-0004(93)90041-K. ISSN 0968-0004. PMID 8236445.
- ^ Johnson, Norman A.; Porter, Adam H. (November 2001). "Toward a new synthesis: population genetics and evolutionary developmental biology". Genetica. 112–113 (1): 45–58. doi:10.1023/A:1013371201773. ISSN 0016-6707. PMID 11838782. S2CID 1651351.
- ^ Baguñà, Jaume; Garcia-Fernàndez, Jordi (2003). "Evo-Devo: the long and winding road". The International Journal of Developmental Biology. 47 (7–8): 705–713. ISSN 0214-6282. PMID 14756346. Archived from the original on 28 November 2014.
- Love, Alan C. (March 2003). "Evolutionary Morphology, Innovation and the Synthesis of Evolutionary and Developmental Biology". Biology and Philosophy. 18 (2): 309–345. doi:10.1023/A:1023940220348. S2CID 82307503.
- ^ Allin, Edgar F. (December 1975). "Evolution of the mammalian middle ear". Journal of Morphology. 147 (4): 403–437. doi:10.1002/jmor.1051470404. ISSN 0362-2525. PMID 1202224. S2CID 25886311.
- ^ Harris, Matthew P.; Hasso, Sean M.; Ferguson, Mark W.J.; Fallon, John F. (21 February 2006). "The Development of Archosaurian First-Generation Teeth in a Chicken Mutant". Current Biology. 16 (4): 371–377. doi:10.1016/j.cub.2005.12.047. PMID 16488870. S2CID 15733491.
- ^ Carroll, Sean B. (11 July 2008). "Evo-Devo and an Expanding Evolutionary Synthesis: A Genetic Theory of Morphological Evolution". Cell. 134 (1): 25–36. doi:10.1016/j.cell.2008.06.030. PMID 18614008. S2CID 2513041.
- ^ Wade, Michael J. (March 2007). "The co-evolutionary genetics of ecological communities". Nature Reviews Genetics. 8 (3): 185–195. doi:10.1038/nrg2031. PMID 17279094. S2CID 36705246.
- ^ Geffeney, Shana; Brodie, Edmund D. Jr.; Ruben, Peter C.; Brodie, Edmund D., III (23 August 2002). "Mechanisms of Adaptation in a Predator-Prey Arms Race: TTX-Resistant Sodium Channels". Science. 297 (5585): 1336–1339. Bibcode:2002Sci...297.1336G. doi:10.1126/science.1074310. PMID 12193784. S2CID 8816337.
{{cite journal}}: CS1 유지 : 여러 이름 : 저자 목록 (링크)- Brodie, Edmund D. Jr.; Ridenhour, Benjamin J.; Brodie, Edmund D., III (October 2002). "The evolutionary response of predators to dangerous prey: hotspots and coldspots in the geographic mosaic of coevolution between garter snakes and newts". Evolution. 56 (10): 2067–2082. doi:10.1554/0014-3820(2002)056[2067:teropt]2.0.co;2. PMID 12449493. S2CID 8251443.
{{cite journal}}: CS1 유지 : 여러 이름 : 저자 목록 (링크) - Carroll, Sean B. (21 December 2009). "Whatever Doesn't Kill Some Animals Can Make Them Deadly". The New York Times. New York. Archived from the original on 23 April 2015. Retrieved 26 December 2014.
- Brodie, Edmund D. Jr.; Ridenhour, Benjamin J.; Brodie, Edmund D., III (October 2002). "The evolutionary response of predators to dangerous prey: hotspots and coldspots in the geographic mosaic of coevolution between garter snakes and newts". Evolution. 56 (10): 2067–2082. doi:10.1554/0014-3820(2002)056[2067:teropt]2.0.co;2. PMID 12449493. S2CID 8251443.
- ^ Sachs, Joel L. (September 2006). "Cooperation within and among species". Journal of Evolutionary Biology. 19 (5): 1415–1418, discussion 1426–1436. doi:10.1111/j.1420-9101.2006.01152.x. PMID 16910971. S2CID 4828678.
- Nowak, Martin A. (8 December 2006). "Five Rules for the Evolution of Cooperation". Science. 314 (5805): 1560–1563. Bibcode:2006Sci...314.1560N. doi:10.1126/science.1133755. PMC 3279745. PMID 17158317.
- ^ Paszkowski, Uta (August 2006). "Mutualism and parasitism: the yin and yang of plant symbioses". Current Opinion in Plant Biology. 9 (4): 364–370. doi:10.1016/j.pbi.2006.05.008. ISSN 1369-5266. PMID 16713732.
- ^ Hause, Bettina; Fester, Thomas (May 2005). "Molecular and cell biology of arbuscular mycorrhizal symbiosis". Planta. 221 (2): 184–196. doi:10.1007/s00425-004-1436-x. PMID 15871030. S2CID 20082902.
- ^ Bertram, John S. (December 2000). "The molecular biology of cancer". Molecular Aspects of Medicine. 21 (6): 167–223. doi:10.1016/S0098-2997(00)00007-8. PMID 11173079. S2CID 24155688.
- ^ Reeve, H. Kern; Hölldobler, Bert (5 June 2007). "The emergence of a superorganism through intergroup competition". PNAS. 104 (23): 9736–9740. Bibcode:2007PNAS..104.9736R. doi:10.1073/pnas.0703466104. ISSN 0027-8424. PMC 1887545. PMID 17517608.
- ^ Axelrod, Robert; Hamilton, W. D. (27 March 1981). "The evolution of cooperation". Science. 211 (4489): 1390–1396. Bibcode:1981Sci...211.1390A. doi:10.1126/science.7466396. PMID 7466396.
- ^ Wilson, Edward O.; Hölldobler, Bert (20 September 2005). "Eusociality: Origin and consequences". PNAS. 102 (38): 13367–1371. Bibcode:2005PNAS..10213367W. doi:10.1073/pnas.0505858102. PMC 1224642. PMID 16157878.
- ^ a b Gavrilets, Sergey (October 2003). "Perspective: models of speciation: what have we learned in 40 years?". Evolution. 57 (10): 2197–2215. doi:10.1554/02-727. PMID 14628909. S2CID 198158082.
- ^ a b c de Queiroz, Kevin (3 May 2005). "Ernst Mayr and the modern concept of species". PNAS. 102 (Suppl. 1): 6600–6607. Bibcode:2005PNAS..102.6600D. doi:10.1073/pnas.0502030102. PMC 1131873. PMID 15851674.
- ^ a b Ereshefsky, Marc (December 1992). "Eliminative pluralism". Philosophy of Science. 59 (4): 671–690. doi:10.1086/289701. JSTOR 188136. S2CID 224829314.
- ^ 1942년 5월 120쪽
- ^ Fraser, Christophe; Alm, Eric J.; Polz, Martin F.; et al. (6 February 2009). "The Bacterial Species Challenge: Making Sense of Genetic and Ecological Diversity". Science. 323 (5915): 741–746. Bibcode:2009Sci...323..741F. doi:10.1126/science.1159388. PMID 19197054. S2CID 15763831.
- ^ Short, Roger Valentine (October 1975). "The contribution of the mule to scientific thought". Journal of Reproduction and Fertility. Supplement (23): 359–364. OCLC 1639439. PMID 1107543.
- ^ Gross, Briana L.; Rieseberg, Loren H. (May–June 2005). "The Ecological Genetics of Homoploid Hybrid Speciation". Journal of Heredity. 96 (3): 241–252. doi:10.1093/jhered/esi026. ISSN 0022-1503. PMC 2517139. PMID 15618301.
- ^ Burke, John M.; Arnold, Michael L. (December 2001). "Genetics and the fitness of hybrids". Annual Review of Genetics. 35: 31–52. doi:10.1146/annurev.genet.35.102401.085719. ISSN 0066-4197. PMID 11700276. S2CID 26683922.
- ^ Vrijenhoek, Robert C. (4 April 2006). "Polyploid Hybrids: Multiple Origins of a Treefrog Species". Current Biology. 16 (7): R245–R247. doi:10.1016/j.cub.2006.03.005. ISSN 0960-9822. PMID 16581499. S2CID 11657663.
- ^ Rice, William R.; Hostert, Ellen E. (December 1993). "Laboratory Experiments on Speciation: What Have We Learned in 40 Years?". Evolution. 47 (6): 1637–1653. doi:10.1111/j.1558-5646.1993.tb01257.x. ISSN 0014-3820. JSTOR 2410209. PMID 28568007. S2CID 42100751.
- Jiggins, Chris D.; Bridle, Jon R. (March 2004). "Speciation in the apple maggot fly: a blend of vintages?". Trends in Ecology & Evolution. 19 (3): 111–114. doi:10.1016/j.tree.2003.12.008. ISSN 0169-5347. PMID 16701238.
- Boxhorn, Joseph (1 September 1995). "Observed Instances of Speciation". TalkOrigins Archive. Houston, Texas: The TalkOrigins Foundation, Inc. Archived from the original on 22 January 2009. Retrieved 26 December 2008.
- Weinberg, James R.; Starczak, Victoria R.; Jörg, Daniele (August 1992). "Evidence for Rapid Speciation Following a Founder Event in the Laboratory". Evolution. 46 (4): 1214–1220. doi:10.2307/2409766. ISSN 0014-3820. JSTOR 2409766. PMID 28564398.
- ^ Herrel, Anthony; Huyghe, Katleen; Vanhooydonck, Bieke; et al. (25 March 2008). "Rapid large-scale evolutionary divergence in morphology and performance associated with exploitation of a different dietary resource". PNAS. 105 (12): 4792–4795. Bibcode:2008PNAS..105.4792H. doi:10.1073/pnas.0711998105. ISSN 0027-8424. PMC 2290806. PMID 18344323.
- ^ Losos, Jonathan B.; Warhelt, Kenneth I.; Schoener, Thomas W. (1 May 1997). "Adaptive differentiation following experimental island colonization in Anolis lizards". Nature. 387 (6628): 70–73. Bibcode:1997Natur.387...70L. doi:10.1038/387070a0. ISSN 0028-0836. S2CID 4242248.
- ^ Hoskin, Conrad J.; Higgle, Megan; McDonald, Keith R.; Moritz, Craig (27 October 2005). "Reinforcement drives rapid allopatric speciation". Nature. 437 (7063): 1353–1356. Bibcode:2005Natur.437.1353H. doi:10.1038/nature04004. PMID 16251964. S2CID 4417281.
- ^ Templeton, Alan R. (April 1980). "The Theory of Speciation VIA the Founder Principle". Genetics. 94 (4): 1011–1038. doi:10.1093/genetics/94.4.1011. PMC 1214177. PMID 6777243. Archived from the original on 23 August 2014. Retrieved 29 December 2014.
- ^ Antonovics, Janis (July 2006). "Evolution in closely adjacent plant populations X: long-term persistence of prereproductive isolation at a mine boundary". Heredity. 97 (1): 33–37. doi:10.1038/sj.hdy.6800835. ISSN 0018-067X. PMID 16639420. S2CID 12291411.
- ^ Nosil, Patrik; Crespi, Bernard J.; Gries, Regine; Gries, Gerhard (March 2007). "Natural selection and divergence in mate preference during speciation". Genetica. 129 (3): 309–327. doi:10.1007/s10709-006-0013-6. ISSN 0016-6707. PMID 16900317. S2CID 10808041.
- ^ Savolainen, Vincent; Anstett, Marie-Charlotte; Lexer, Christian; et al. (11 May 2006). "Sympatric speciation in palms on an oceanic island". Nature. 441 (7090): 210–213. Bibcode:2006Natur.441..210S. doi:10.1038/nature04566. ISSN 0028-0836. PMID 16467788. S2CID 867216.
- Barluenga, Marta; Stölting, Kai N.; Salzburger, Walter; et al. (9 February 2006). "Sympatric speciation in Nicaraguan crater lake cichlid fish". Nature. 439 (7077): 719–23. Bibcode:2006Natur.439..719B. doi:10.1038/nature04325. ISSN 0028-0836. PMID 16467837. S2CID 3165729. Archived from the original on 30 July 2022. Retrieved 30 July 2022.
- ^ Gavrilets, Sergey (21 March 2006). "The Maynard Smith model of sympatric speciation". Journal of Theoretical Biology. 239 (2): 172–182. Bibcode:2006JThBi.239..172G. doi:10.1016/j.jtbi.2005.08.041. ISSN 0022-5193. PMID 16242727.
- ^ Wood, Troy E.; Takebayashi, Naoki; Barker, Michael S.; et al. (18 August 2009). "The frequency of polyploid speciation in vascular plants". PNAS. 106 (33): 13875–13879. Bibcode:2009PNAS..10613875W. doi:10.1073/pnas.0811575106. ISSN 0027-8424. PMC 2728988. PMID 19667210.
- ^ Hegarty, Matthew J.; Hiscock, Simon J. (20 May 2008). "Genomic Clues to the Evolutionary Success of Polyploid Plants". Current Biology. 18 (10): R435–R444. doi:10.1016/j.cub.2008.03.043. ISSN 0960-9822. PMID 18492478. S2CID 1584282.
- ^ Jakobsson, Mattias; Hagenblad, Jenny; Tavaré, Simon; et al. (June 2006). "A Unique Recent Origin of the Allotetraploid Species Arabidopsis suecica: Evidence from Nuclear DNA Markers". Molecular Biology and Evolution. 23 (6): 1217–1231. doi:10.1093/molbev/msk006. PMID 16549398. Archived from the original on 15 February 2022. Retrieved 30 July 2022.
- ^ Säll, Torbjörn; Jakobsson, Mattias; Lind-Halldén, Christina; Halldén, Christer (September 2003). "Chloroplast DNA indicates a single origin of the allotetraploid Arabidopsis suecica". Journal of Evolutionary Biology. 16 (5): 1019–1029. doi:10.1046/j.1420-9101.2003.00554.x. PMID 14635917. S2CID 29281998.
- ^ Bomblies, Kirsten; Weigel, Detlef (December 2007). "Arabidopsis—a model genus for speciation". Current Opinion in Genetics & Development. 17 (6): 500–504. doi:10.1016/j.gde.2007.09.006. PMID 18006296.
- ^ Sémon, Marie; Wolfe, Kenneth H. (December 2007). "Consequences of genome duplication". Current Opinion in Genetics & Development. 17 (6): 505–512. doi:10.1016/j.gde.2007.09.007. PMID 18006297.
- ^ Eldredge & Gould 1972, pp. 82–115
- ^ Benton, Michael J. (7 April 1995). "Diversification and extinction in the history of life". Science. 268 (5207): 52–58. Bibcode:1995Sci...268...52B. doi:10.1126/science.7701342. ISSN 0036-8075. PMID 7701342.
- ^ Raup, David M. (28 March 1986). "Biological extinction in Earth history". Science. 231 (4745): 1528–1533. Bibcode:1986Sci...231.1528R. doi:10.1126/science.11542058. PMID 11542058. S2CID 23012011.
- ^ Avise, John C.; Hubbell, Stephen P.; Ayala, Francisco J. (12 August 2008). "In the light of evolution II: Biodiversity and extinction". PNAS. 105 (Suppl. 1): 11453–11457. Bibcode:2008PNAS..10511453A. doi:10.1073/pnas.0802504105. PMC 2556414. PMID 18695213.
- ^ a b c Raup, David M. (19 July 1994). "The role of extinction in evolution". PNAS. 91 (15): 6758–6763. Bibcode:1994PNAS...91.6758R. doi:10.1073/pnas.91.15.6758. PMC 44280. PMID 8041694.
- ^ Novacek, Michael J.; Cleland, Elsa E. (8 May 2001). "The current biodiversity extinction event: scenarios for mitigation and recovery". PNAS. 98 (10): 5466–5470. Bibcode:2001PNAS...98.5466N. doi:10.1073/pnas.091093698. ISSN 0027-8424. PMC 33235. PMID 11344295.
- ^ Pimm, Stuart; Raven, Peter; Peterson, Alan; et al. (18 July 2006). "Human impacts on the rates of recent, present and future bird extinctions". PNAS. 103 (29): 10941–10946. Bibcode:2006PNAS..10310941P. doi:10.1073/pnas.0604181103. ISSN 0027-8424. PMC 1544153. PMID 16829570.
- ^ Barnosky, Anthony D.; Koch, Paul L.; Feranec, Robert S.; et al. (1 October 2004). "Assessing the Causes of Late Pleistocene Extinctions on the Continents". Science. 306 (5693): 70–75. Bibcode:2004Sci...306...70B. CiteSeerX 10.1.1.574.332. doi:10.1126/science.1101476. ISSN 0036-8075. PMID 15459379. S2CID 36156087.
- ^ Lewis, Owen T. (29 January 2006). "Climate change, species–area curves and the extinction crisis". Philosophical Transactions of the Royal Society B. 361 (1465): 163–171. doi:10.1098/rstb.2005.1712. ISSN 0962-8436. PMC 1831839. PMID 16553315.
- ^ a b Stearns & Stearns 1999, p. X
- ^ a b Novacek, Michael J. (8 November 2014). "Prehistory's Brilliant Future". The New York Times. New York. ISSN 0362-4331. Archived from the original on 29 December 2014. Retrieved 25 December 2014.
- ^ "Researchers find that Earth may be home to 1 trillion species". National Science Foundation. Arlington County, Virginia. 2 May 2016. Archived from the original on 4 May 2016. Retrieved 6 May 2016.
- ^ a b c Kutschera, Ulrich; Niklas, Karl J. (June 2004). "The modern theory of biological evolution: an expanded synthesis". Naturwissenschaften. 91 (6): 255–276. Bibcode:2004NW.....91..255K. doi:10.1007/s00114-004-0515-y. ISSN 1432-1904. PMID 15241603. S2CID 10731711.
- ^ Jablonski, David (8 May 2001). "Lessons from the past: Evolutionary impacts of mass extinctions". PNAS. 98 (10): 5393–5398. Bibcode:2001PNAS...98.5393J. doi:10.1073/pnas.101092598. PMC 33224. PMID 11344284.
- ^ Bull, James J.; Wichman, Holly A. (November 2001). "Applied evolution". Annual Review of Ecology and Systematics. 32: 183–217. doi:10.1146/annurev.ecolsys.32.081501.114020. ISSN 1545-2069.
- ^ Doebley, John F.; Gaut, Brandon S.; Smith, Bruce D. (29 December 2006). "The Molecular Genetics of Crop Domestication". Cell. 127 (7): 1309–1321. doi:10.1016/j.cell.2006.12.006. ISSN 0092-8674. PMID 17190597. S2CID 278993.
- ^ Jäckel, Christian; Kast, Peter; Hilvert, Donald (June 2008). "Protein Design by Directed Evolution". Annual Review of Biophysics. 37: 153–173. doi:10.1146/annurev.biophys.37.032807.125832. ISSN 1936-122X. PMID 18573077.
- ^ Maher, Brendan (8 April 2009). "Evolution: Biology's next top model?". Nature. 458 (7239): 695–698. doi:10.1038/458695a. ISSN 0028-0836. PMID 19360058. S2CID 41648315.
- ^ Borowsky, Richard (8 January 2008). "Restoring sight in blind cavefish". Current Biology. 18 (1): R23–R24. doi:10.1016/j.cub.2007.11.023. ISSN 0960-9822. PMID 18177707. S2CID 16967690.
- ^ Gross, Joshua B.; Borowsky, Richard; Tabin, Clifford J. (2 January 2009). Barsh, Gregory S. (ed.). "A novel role for Mc1r in the parallel evolution of depigmentation in independent populations of the cavefish Astyanax mexicanus". PLOS Genetics. 5 (1): e1000326. doi:10.1371/journal.pgen.1000326. ISSN 1553-7390. PMC 2603666. PMID 19119422.
- ^ Merlo, Lauren M.F.; Pepper, John W.; Reid, Brian J.; Maley, Carlo C. (December 2006). "Cancer as an evolutionary and ecological process". Nature Reviews Cancer. 6 (12): 924–935. doi:10.1038/nrc2013. ISSN 1474-175X. PMID 17109012. S2CID 8040576.
- ^ Pan, Dabo; Weiwei Xue; Wenqi Zhang; et al. (October 2012). "Understanding the drug resistance mechanism of hepatitis C virus NS3/4A to ITMN-191 due to R155K, A156V, D168A/E mutations: a computational study". Biochimica et Biophysica Acta (BBA) - General Subjects. 1820 (10): 1526–1534. doi:10.1016/j.bbagen.2012.06.001. ISSN 0304-4165. PMID 22698669.
- ^ Woodford, Neil; Ellington, Matthew J. (January 2007). "The emergence of antibiotic resistance by mutation". Clinical Microbiology and Infection. 13 (1): 5–18. doi:10.1111/j.1469-0691.2006.01492.x. ISSN 1198-743X. PMID 17184282.
- ^ Labbé, Pierrick; Berticat, Claire; Berthomieu, Arnaud; et al. (16 November 2007). "Forty Years of Erratic Insecticide Resistance Evolution in the Mosquito Culex pipiens". PLOS Genetics. 3 (11): e205. doi:10.1371/journal.pgen.0030205. ISSN 1553-7390. PMC 2077897. PMID 18020711.
- ^ Neve, Paul (October 2007). "Challenges for herbicide resistance evolution and management: 50 years after Harper". Weed Research. 47 (5): 365–369. doi:10.1111/j.1365-3180.2007.00581.x. ISSN 0043-1737.
- ^ Rodríguez-Rojas, Alexandro; Rodríguez-Beltrán, Jerónimo; Couce, Alejandro; Blázquez, Jesús (August 2013). "Antibiotics and antibiotic resistance: A bitter fight against evolution". International Journal of Medical Microbiology. 303 (6–7): 293–297. doi:10.1016/j.ijmm.2013.02.004. ISSN 1438-4221. PMID 23517688.
- ^ Schenk, Martijn F.; Szendro, Ivan G.; Krug, Joachim; de Visser, J. Arjan G.M. (28 June 2012). "Quantifying the Adaptive Potential of an Antibiotic Resistance Enzyme". PLOS Genetics. 8 (6): e1002783. doi:10.1371/journal.pgen.1002783. ISSN 1553-7390. PMC 3386231. PMID 22761587.
- ^ Read, Andrew F.; Lynch, Penelope A.; Thomas, Matthew B. (7 April 2009). "How to Make Evolution-Proof Insecticides for Malaria Control". PLOS Biology. 7 (4): e1000058. doi:10.1371/journal.pbio.1000058. PMC 3279047. PMID 19355786.
- ^ Fraser, Alex S. (18 January 1958). "Monte Carlo Analyses of Genetic Models". Nature. 181 (4603): 208–209. Bibcode:1958Natur.181..208F. doi:10.1038/181208a0. ISSN 0028-0836. PMID 13504138. S2CID 4211563.
- ^ 레첸베르크 1973
- ^ 네덜란드 1975년
- ^ 코자 1992
- ^ Jamshidi, Mo (15 August 2003). "Tools for intelligent control: fuzzy controllers, neural networks and genetic algorithms". Philosophical Transactions of the Royal Society A. 361 (1809): 1781–1808. Bibcode:2003RSPTA.361.1781J. doi:10.1098/rsta.2003.1225. PMID 12952685. S2CID 34259612.
- ^ "Age of the Earth". United States Geological Survey. 9 July 2007. Archived from the original on 23 December 2005. Retrieved 31 May 2015.
- ^ Dalrymple 2001, 페이지 205-221
- ^ Manhesa, Gérard; Allègre, Claude J.; Dupréa, Bernard; Hamelin, Bruno (May 1980). "Lead isotope study of basic-ultrabasic layered complexes: Speculations about the age of the earth and primitive mantle characteristics". Earth and Planetary Science Letters. 47 (3): 370–382. Bibcode:1980E&PSL..47..370M. doi:10.1016/0012-821X(80)90024-2. ISSN 0012-821X.
- ^ 레이븐 & 존슨 2002, 페이지 68
- ^ a b Borenstein, Seth (19 October 2015). "Hints of life on what was thought to be desolate early Earth". Excite. Yonkers, NY: Mindspark Interactive Network. Associated Press. Archived from the original on 23 October 2015. Retrieved 8 October 2018.
- ^ Bell, Elizabeth A.; Boehnike, Patrick; Harrison, T. Mark; Mao, Wendy L. (24 November 2015). "Potentially biogenic carbon preserved in a 4.1 billion-year-old zircon" (PDF). PNAS. 112 (47): 14518–14521. Bibcode:2015PNAS..11214518B. doi:10.1073/pnas.1517557112. ISSN 0027-8424. PMC 4664351. PMID 26483481. Archived (PDF) from the original on 6 November 2015. Retrieved 30 December 2015.
- ^ Schouten, Lucy (20 October 2015). "When did life first emerge on Earth? Maybe a lot earlier than we thought". The Christian Science Monitor. Boston, Massachusetts: Christian Science Publishing Society. ISSN 0882-7729. Archived from the original on 22 March 2016. Retrieved 11 July 2018.
- ^ Wade, Nicholas (25 July 2016). "Meet Luca, the Ancestor of All Living Things". The New York Times. New York. ISSN 0362-4331. Archived from the original on 28 July 2016. Retrieved 25 July 2016.
- ^ 맥키니 1997, 페이지 110
- ^ Mora, Camilo; Tittensor, Derek P.; Adl, Sina; et al. (23 August 2011). "How Many Species Are There on Earth and in the Ocean?". PLOS Biology. 9 (8): e1001127. doi:10.1371/journal.pbio.1001127. ISSN 1545-7885. PMC 3160336. PMID 21886479.
- ^ Miller & Spoolman 2012, 페이지 62
- ^ 채프먼 2009
- ^ Roskov, Y.; Abucay, L.; Orrell, T.; Nicolson, D.; et al., eds. (2016). "Species 2000 & ITIS Catalogue of Life, 2016 Annual Checklist". Species 2000. Leiden, Netherlands: Naturalis Biodiversity Center. ISSN 2405-884X. Archived from the original on 12 November 2016. Retrieved 6 November 2016.
- ^ Peretó, Juli (March 2005). "Controversies on the origin of life" (PDF). International Microbiology. 8 (1): 23–31. ISSN 1139-6709. PMID 15906258. Archived from the original (PDF) on 24 August 2015.
- ^ Marshall, Michael (11 November 2020). "Charles Darwin's hunch about early life was probably right – In a few scrawled notes to a friend, biologist Charles Darwin theorised how life began. Not only was it probably correct, his theory was a century ahead of its time". BBC News. Archived from the original on 11 November 2020. Retrieved 11 November 2020.
- ^ Joyce, Gerald F. (11 July 2002). "The antiquity of RNA-based evolution". Nature. 418 (6894): 214–221. Bibcode:2002Natur.418..214J. doi:10.1038/418214a. PMID 12110897. S2CID 4331004.
- ^ Trevors, Jack T.; Psenner, Roland (December 2001). "From self-assembly of life to present-day bacteria: a possible role for nanocells". FEMS Microbiology Reviews. 25 (5): 573–582. doi:10.1111/j.1574-6976.2001.tb00592.x. ISSN 1574-6976. PMID 11742692.
- ^ Theobald, Douglas L. (13 May 2010). "A formal test of the theory of universal common ancestry". Nature. 465 (7295): 219–222. Bibcode:2010Natur.465..219T. doi:10.1038/nature09014. ISSN 0028-0836. PMID 20463738. S2CID 4422345.
- ^ Bapteste, Eric; Walsh, David A. (June 2005). "Does the 'Ring of Life' ring true?". Trends in Microbiology. 13 (6): 256–261. doi:10.1016/j.tim.2005.03.012. ISSN 0966-842X. PMID 15936656.
- ^ 다윈 1859, p. 1
- ^ Doolittle, W. Ford; Bapteste, Eric (13 February 2007). "Pattern pluralism and the Tree of Life hypothesis". PNAS. 104 (7): 2043–2049. Bibcode:2007PNAS..104.2043D. doi:10.1073/pnas.0610699104. ISSN 0027-8424. PMC 1892968. PMID 17261804.
- ^ Kunin, Victor; Goldovsky, Leon; Darzentas, Nikos; Ouzounis, Christos A. (July 2005). "The net of life: Reconstructing the microbial phylogenetic network". Genome Research. 15 (7): 954–959. doi:10.1101/gr.3666505. ISSN 1088-9051. PMC 1172039. PMID 15965028.
- ^ 다윈 1837, 페이지 25
- ^ Jablonski, David (25 June 1999). "The Future of the Fossil Record". Science. 284 (5423): 2114–2116. doi:10.1126/science.284.5423.2114. ISSN 0036-8075. PMID 10381868. S2CID 43388925.
- ^ Mason, Stephen F. (6 September 1984). "Origins of biomolecular handedness". Nature. 311 (5981): 19–23. Bibcode:1984Natur.311...19M. doi:10.1038/311019a0. ISSN 0028-0836. PMID 6472461. S2CID 103653.
- ^ Wolf, Yuri I.; Rogozin, Igor B.; Grishin, Nick V.; Koonin, Eugene V. (1 September 2002). "Genome trees and the tree of life". Trends in Genetics. 18 (9): 472–479. doi:10.1016/S0168-9525(02)02744-0. ISSN 0168-9525. PMID 12175808.
- ^ Varki, Ajit; Altheide, Tasha K. (December 2005). "Comparing the human and chimpanzee genomes: searching for needles in a haystack". Genome Research. 15 (12): 1746–1758. CiteSeerX 10.1.1.673.9212. doi:10.1101/gr.3737405. ISSN 1088-9051. PMID 16339373.
- ^ Ciccarelli, Francesca D.; Doerks, Tobias; von Mering, Christian; et al. (3 March 2006). "Toward Automatic Reconstruction of a Highly Resolved Tree of Life" (PDF). Science. 311 (5765): 1283–1287. Bibcode:2006Sci...311.1283C. CiteSeerX 10.1.1.381.9514. doi:10.1126/science.1123061. ISSN 0036-8075. PMID 16513982. S2CID 1615592. Archived (PDF) from the original on 4 March 2016.
- ^ a b Cavalier-Smith, Thomas (29 June 2006). "Cell evolution and Earth history: stasis and revolution". Philosophical Transactions of the Royal Society B. 361 (1470): 969–1006. doi:10.1098/rstb.2006.1842. ISSN 0962-8436. PMC 1578732. PMID 16754610.
- ^ Schopf, J. William (29 June 2006). "Fossil evidence of Archaean life". Philosophical Transactions of the Royal Society B. 361 (1470): 869–885. doi:10.1098/rstb.2006.1834. PMC 1578735. PMID 16754604.
- Altermann, Wladyslaw; Kazmierczak, Józef (November 2003). "Archean microfossils: a reappraisal of early life on Earth". Research in Microbiology. 154 (9): 611–617. doi:10.1016/j.resmic.2003.08.006. PMID 14596897.
- ^ Schopf, J. William (19 July 1994). "Disparate rates, differing fates: tempo and mode of evolution changed from the Precambrian to the Phanerozoic". PNAS. 91 (15): 6735–6742. Bibcode:1994PNAS...91.6735S. doi:10.1073/pnas.91.15.6735. PMC 44277. PMID 8041691.
- ^ Poole, Anthony M.; Penny, David (January 2007). "Evaluating hypotheses for the origin of eukaryotes". BioEssays. 29 (1): 74–84. doi:10.1002/bies.20516. ISSN 0265-9247. PMID 17187354.
- ^ Dyall, Sabrina D.; Brown, Mark T.; Johnson, Patricia J. (9 April 2004). "Ancient Invasions: From Endosymbionts to Organelles". Science. 304 (5668): 253–257. Bibcode:2004Sci...304..253D. doi:10.1126/science.1094884. PMID 15073369. S2CID 19424594.
- ^ Martin, William (October 2005). "The missing link between hydrogenosomes and mitochondria". Trends in Microbiology. 13 (10): 457–459. doi:10.1016/j.tim.2005.08.005. PMID 16109488.
- ^ Lang, B. Franz; Gray, Michael W.; Burger, Gertraud (December 1999). "Mitochondrial genome evolution and the origin of eukaryotes". Annual Review of Genetics. 33: 351–397. doi:10.1146/annurev.genet.33.1.351. ISSN 0066-4197. PMID 10690412.
- McFadden, Geoffrey Ian (1 December 1999). "Endosymbiosis and evolution of the plant cell". Current Opinion in Plant Biology. 2 (6): 513–519. doi:10.1016/S1369-5266(99)00025-4. PMID 10607659.
- ^ DeLong, Edward F.; Pace, Norman R. (1 August 2001). "Environmental Diversity of Bacteria and Archaea". Systematic Biology. 50 (4): 470–478. CiteSeerX 10.1.1.321.8828. doi:10.1080/106351501750435040. ISSN 1063-5157. PMID 12116647.
- ^ Kaiser, Dale (December 2001). "Building a multicellular organism". Annual Review of Genetics. 35: 103–123. doi:10.1146/annurev.genet.35.102401.090145. ISSN 0066-4197. PMID 11700279. S2CID 18276422.
- ^ Zimmer, Carl (7 January 2016). "Genetic Flip Helped Organisms Go From One Cell to Many". The New York Times. New York. ISSN 0362-4331. Archived from the original on 7 January 2016. Retrieved 7 January 2016.
- ^ Valentine, James W.; Jablonski, David; Erwin, Douglas H. (1 March 1999). "Fossils, molecules and embryos: new perspectives on the Cambrian explosion". Development. 126 (5): 851–859. doi:10.1242/dev.126.5.851. ISSN 0950-1991. PMID 9927587. Archived from the original on 1 March 2015. Retrieved 30 December 2014.
- ^ Ohno, Susumu (January 1997). "The reason for as well as the consequence of the Cambrian explosion in animal evolution". Journal of Molecular Evolution. 44 (Suppl. 1): S23–S27. Bibcode:1997JMolE..44S..23O. doi:10.1007/PL00000055. ISSN 0022-2844. PMID 9071008. S2CID 21879320.
- Valentine, James W.; Jablonski, David (2003). "Morphological and developmental macroevolution: a paleontological perspective". The International Journal of Developmental Biology. 47 (7–8): 517–522. ISSN 0214-6282. PMID 14756327. Archived from the original on 24 October 2014. Retrieved 30 December 2014.
- ^ Waters, Elizabeth R. (December 2003). "Molecular adaptation and the origin of land plants". Molecular Phylogenetics and Evolution. 29 (3): 456–463. doi:10.1016/j.ympev.2003.07.018. ISSN 1055-7903. PMID 14615186.
- ^ Mayhew, Peter J. (August 2007). "Why are there so many insect species? Perspectives from fossils and phylogenies". Biological Reviews. 82 (3): 425–454. doi:10.1111/j.1469-185X.2007.00018.x. ISSN 1464-7931. PMID 17624962. S2CID 9356614.
- ^ Carroll, Robert L. (May 2007). "The Palaeozoic Ancestry of Salamanders, Frogs and Caecilians". Zoological Journal of the Linnean Society. 150 (Supplement s1): 1–140. doi:10.1111/j.1096-3642.2007.00246.x. ISSN 1096-3642.
- ^ Wible, John R.; Rougier, Guillermo W.; Novacek, Michael J.; Asher, Robert J. (21 June 2007). "Cretaceous eutherians and Laurasian origin for placental mammals near the K/T boundary". Nature. 447 (7147): 1003–1006. Bibcode:2007Natur.447.1003W. doi:10.1038/nature05854. ISSN 0028-0836. PMID 17581585. S2CID 4334424.
- ^ Witmer, Lawrence M. (28 July 2011). "Palaeontology: An icon knocked from its perch". Nature. 475 (7357): 458–459. doi:10.1038/475458a. ISSN 0028-0836. PMID 21796198. S2CID 205066360.
- ^ 다윈 1909, 페이지 53
- ^ 커크, 레이븐 & 스코필드 1983, 페이지 100–142, 280–321
- ^ 루크레티우스
- ^ Sedley, David (2003). "Lucretius and the New Empedocles" (PDF). Leeds International Classical Studies. 2 (4). ISSN 1477-3643. Archived from the original (PDF) on 23 August 2014. Retrieved 25 November 2014.
- ^ Torrey, Harry Beal; Felin, Frances (March 1937). "Was Aristotle an Evolutionist?". The Quarterly Review of Biology. 12 (1): 1–18. doi:10.1086/394520. ISSN 0033-5770. JSTOR 2808399. S2CID 170831302.
- ^ Hull, David L. (December 1967). "The Metaphysics of Evolution". The British Journal for the History of Science. Cambridge: Cambridge University Press on behalf of The British Society for the History of Science. 3 (4): 309–337. doi:10.1017/S0007087400002892. JSTOR 4024958. S2CID 170328394.
- ^ Mason 1962, 페이지 43-44
- ^ 키로스, 테오드로스.아프리카 정치사상의 탐구.2001년 55페이지
- ^ 1982년 5월 페이지 256-257
- ^ Waggoner, Ben (7 July 2000). "Carl Linnaeus (1707–1778)". Evolution (Online exhibit). Berkeley, California: University of California Museum of Paleontology. Archived from the original on 30 April 2011. Retrieved 11 February 2012.
- ^ Bowler 2003, 페이지 73–75
- ^ "Erasmus Darwin (1731–1802)". Evolution (Online exhibit). Berkeley, California: University of California Museum of Paleontology. 4 October 1995. Archived from the original on 19 January 2012. Retrieved 11 February 2012.
- ^ 1809년 라마르크
- ^ a b Nardon & Grenier 1991, 페이지 162
- ^ Ghiselin, Michael T. (September–October 1994). "The Imaginary Lamarck: A Look at Bogus 'History' in Schoolbooks". The Textbook Letter. OCLC 23228649. Archived from the original on 12 February 2008. Retrieved 23 January 2008.
- ^ Jablonka, Eva; Lamb, Marion J. (August 2007). "Précis of Evolution in Four Dimensions". Behavioral and Brain Sciences. 30 (4): 353–365. doi:10.1017/S0140525X07002221. ISSN 0140-525X. PMID 18081952. S2CID 15879804.
- ^ 버크하르트 앤 스미스 1991
- "Darwin, C. R. to Lubbock, John". Darwin Correspondence Project. Cambridge: University of Cambridge. Archived from the original on 15 December 2014. Retrieved 1 December 2014. 편지 2532, 1859년 11월 22일.
- ^ Sulloway, Frank J. (June 2009). "Why Darwin rejected intelligent design". Journal of Biosciences. 34 (2): 173–183. doi:10.1007/s12038-009-0020-8. ISSN 0250-5991. PMID 19550032. S2CID 12289290.
- ^ "Search results for "descent with modification" – The Complete Work of Charles Darwin Online". Archived from the original on 5 June 2022. Retrieved 30 July 2022.
- ^ Sober, Elliott (16 June 2009). "Did Darwin write the Origin backwards?". PNAS. 106 (Suppl. 1): 10048–10055. Bibcode:2009PNAS..10610048S. doi:10.1073/pnas.0901109106. ISSN 0027-8424. PMC 2702806. PMID 19528655.
- ^ 2002년 5월, 페이지 165
- ^ Bowler 2003, 페이지 145-146
- ^ Sokal, Robert R.; Crovello, Theodore J. (March–April 1970). "The Biological Species Concept: A Critical Evaluation". The American Naturalist. 104 (936): 127–153. doi:10.1086/282646. ISSN 0003-0147. JSTOR 2459191. S2CID 83528114.
- ^ Darwin, Charles; Wallace, Alfred (20 August 1858). "On the Tendency of Species to form Varieties; and on the Perpetuation of Varieties and Species by Natural Means of Selection". Journal of the Proceedings of the Linnean Society of London. Zoology. 3 (9): 45–62. doi:10.1111/j.1096-3642.1858.tb02500.x. ISSN 1096-3642. Archived from the original on 14 July 2007. Retrieved 13 May 2007.
- ^ Desmond, Adrian J. (17 July 2014). "Thomas Henry Huxley". Encyclopædia Britannica Online. Chicago, Illinois: Encyclopædia Britannica, Inc. Archived from the original on 19 January 2015. Retrieved 2 December 2014.
- ^ Y. -S. Liu; X. M. Zhou; M. X. Zhi; X. J. Li; Q. L. Wang (September 2009). "Darwin's contributions to genetics". Journal of Applied Genetics. 50 (3): 177–184. doi:10.1007/BF03195671. ISSN 1234-1983. PMID 19638672. S2CID 19919317.
- ^ Weiling, Franz (July 1991). "Historical study: Johann Gregor Mendel 1822–1884". American Journal of Medical Genetics. 40 (1): 1–25, discussion 26. doi:10.1002/ajmg.1320400103. PMID 1887835.
- ^ 라이트 1984, 페이지 480
- ^ 1971년 프로빈
- ^ Stamhuis, Ida H.; Meijer, Onno G.; Zevenhuizen, Erik J. A. (June 1999). "Hugo de Vries on Heredity, 1889–1903: Statistics, Mendelian Laws, Pangenes, Mutations". Isis. 90 (2): 238–267. doi:10.1086/384323. JSTOR 237050. PMID 10439561. S2CID 20200394.
- ^ a b Bowler 1989, pp. 307–318.
- ^ 레빈슨 2019년.
- ^ Watson, J. D.; Crick, F. H. C. (25 April 1953). "Molecular Structure of Nucleic Acids: A Structure for Deoxyribose Nucleic Acid" (PDF). Nature. 171 (4356): 737–738. Bibcode:1953Natur.171..737W. doi:10.1038/171737a0. ISSN 0028-0836. PMID 13054692. S2CID 4253007. Archived (PDF) from the original on 23 August 2014. Retrieved 4 December 2014.
It has not escaped our notice that the specific pairing we have postulated immediately suggests a possible copying mechanism for the genetic material.
- ^ 헤니그 1999, 페이지 280
- ^ Dobzhansky, Theodosius (March 1973). "Nothing in Biology Makes Sense Except in the Light of Evolution" (PDF). The American Biology Teacher. 35 (3): 125–129. CiteSeerX 10.1.1.324.2891. doi:10.2307/4444260. JSTOR 4444260. S2CID 207358177. Archived (PDF) from the original on 23 October 2015.
- ^ Avise, John C.; Ayala, Francisco J. (11 May 2010). "In the light of evolution IV: The human condition" (PDF). PNAS. 107 (Suppl. 2): 8897–8901. doi:10.1073/pnas.1003214107. ISSN 0027-8424. PMC 3024015. PMID 20460311. Archived (PDF) from the original on 23 August 2014. Retrieved 29 December 2014.
- ^ Danchin, Étienne; Charmantier, Anne; Champagne, Frances A.; Mesoudi, Alex; Pujol, Benoit; Blanchet, Simon (June 2011). "Beyond DNA: integrating inclusive inheritance into an extended theory of evolution". Nature Reviews Genetics. 12 (7): 475–486. doi:10.1038/nrg3028. ISSN 1471-0056. PMID 21681209. S2CID 8837202.
- ^ 피글루치 & 뮐러 2010
- ^ Browne 2003, 페이지 376-379
- ^ 철학적, 종교적, 우주론적 논쟁에 대한 개요는 다음을 참조하십시오.19세기와 20세기 초의 진화에 대한 과학적, 사회적 반응은 다음을 참조하십시오.
- Johnston, Ian C. (1999). "Section Three: The Origins of Evolutionary Theory". ... And Still We Evolve: A Handbook for the Early History of Modern Science (3rd revised ed.). Nanaimo, BC: Liberal Studies Department, Malaspina University-College. Archived from the original on 16 April 2016. Retrieved 1 January 2015.
- 볼러 2003
- Zuckerkandl, Emile (30 December 2006). "Intelligent design and biological complexity". Gene. 385: 2–18. doi:10.1016/j.gene.2006.03.025. ISSN 0378-1119. PMID 17011142.
- ^ Ross, Marcus R. (May 2005). "Who Believes What? Clearing up Confusion over Intelligent Design and Young-Earth Creationism" (PDF). Journal of Geoscience Education. 53 (3): 319–323. Bibcode:2005JGeEd..53..319R. CiteSeerX 10.1.1.404.1340. doi:10.5408/1089-9995-53.3.319. ISSN 1089-9995. S2CID 14208021. Archived (PDF) from the original on 11 May 2008. Retrieved 28 April 2008.
- ^ Hameed, Salman (12 December 2008). "Bracing for Islamic Creationism" (PDF). Science. 322 (5908): 1637–1638. doi:10.1126/science.1163672. ISSN 0036-8075. PMID 19074331. S2CID 206515329. Archived from the original (PDF) on 10 November 2014.
- ^ 볼러 2003
- ^ Miller, Jon D.; Scott, Eugenie C.; Okamoto, Shinji (11 August 2006). "Public Acceptance of Evolution". Science. 313 (5788): 765–766. doi:10.1126/science.1126746. ISSN 0036-8075. PMID 16902112. S2CID 152990938.
- ^ Spergel, David Nathaniel; Verde, Licia; Peiris, Hiranya V.; et al. (2003). "First-Year Wilkinson Microwave Anisotropy Probe (WMAP) Observations: Determination of Cosmological Parameters". The Astrophysical Journal Supplement Series. 148 (1): 175–194. arXiv:astro-ph/0302209. Bibcode:2003ApJS..148..175S. doi:10.1086/377226. S2CID 10794058.
- ^ Wilde, Simon A.; Valley, John W.; Peck, William H.; Graham, Colin M. (11 January 2001). "Evidence from detrital zircons for the existence of continental crust and oceans on the Earth 4.4 Gyr ago". Nature. 409 (6817): 175–178. Bibcode:2001Natur.409..175W. doi:10.1038/35051550. ISSN 0028-0836. PMID 11196637. S2CID 4319774.
- ^ Branch, Glenn (March 2007). "Understanding Creationism after Kitzmiller". BioScience. 57 (3): 278–284. doi:10.1641/B570313. ISSN 0006-3568. S2CID 86665329.
- ^ Xiaoxing Jin (March 2019). "Translation and transmutation: the Origin of Species in China". The British Journal for the History of Science. Cambridge: Cambridge University Press on behalf of The British Society for the History of Science. 52 (1): 117–141. doi:10.1017/S0007087418000808. PMID 30587253. S2CID 58605626.
서지학
- Altenberg, Lee (1995). "Genome growth and the evolution of the genotype–phenotype map". In Banzhaf, Wolfgang; Eeckman, Frank H. (eds.). Evolution and Biocomputation: Computational Models of Evolution. Lecture Notes in Computer Science. Vol. 899. Berlin; New York: Springer-Verlag Berlin Heidelberg. pp. 205–259. CiteSeerX 10.1.1.493.6534. doi:10.1007/3-540-59046-3_11. ISBN 978-3-540-59046-0. ISSN 0302-9743. LCCN 95005970. OCLC 32049812.
- Birdsell, John A.; Wills, Christopher (2003). "The Evolutionary Origin and Maintenance of Sexual Recombination: A Review of Contemporary Models". In MacIntyre, Ross J.; Clegg, Michael T. (eds.). Evolutionary Biology. Vol. 33. New York: Springer Science+Business Media. ISBN 978-1-4419-3385-0. ISSN 0071-3260. OCLC 751583918.
- Bowler, Peter J. (1989). The Mendelian Revolution: The Emergence of Hereditarian Concepts in Modern Science and Society. Baltimore, Maryland: Johns Hopkins University Press. ISBN 978-0-8018-3888-0. LCCN 89030914. OCLC 19322402.
- Bowler, Peter J. (2003). Evolution: The History of an Idea (3rd completely rev. and expanded ed.). Berkeley, California: University of California Press. ISBN 978-0-520-23693-6. LCCN 2002007569. OCLC 49824702.
- Browne, Janet (2003). Charles Darwin: The Power of Place. Vol. 2. London: Pimlico. ISBN 978-0-7126-6837-8. LCCN 94006598. OCLC 52327000.
- Burkhardt, Frederick; Smith, Sydney, eds. (1991). The Correspondence of Charles Darwin. Vol. 7: 1858–1859. Cambridge: Cambridge University Press. ISBN 978-0-521-38564-0. LCCN 84045347. OCLC 185662993.
- Carroll, Sean B.; Grenier, Jennifer K.; Weatherbee, Scott D. (2005). From DNA to Diversity: Molecular Genetics and the Evolution of Animal Design (2nd ed.). Malden, Massachusetts: Blackwell Publishing. ISBN 978-1-4051-1950-4. LCCN 2003027991. OCLC 53972564.
- Chapman, Arthur D. (2009). Numbers of Living Species in Australia and the World (2nd ed.). Canberra: Department of the Environment, Water, Heritage and the Arts: Australian Biological Resources Study. ISBN 978-0-642-56860-1. OCLC 780539206. Archived from the original on 25 December 2016. Retrieved 6 November 2016.
- Coyne, Jerry A. (2009). Why Evolution is True. New York: Viking. ISBN 978-0-670-02053-9. LCCN 2008033973. OCLC 233549529.
- Dalrymple, G. Brent (2001). "The age of the Earth in the twentieth century: a problem (mostly) solved". In Lewis, C.L.E.; Knell, S.J. (eds.). The Age of the Earth: from 4004 BC to AD 2002. Geological Society Special Publication. Vol. 190. pp. 205–221. Bibcode:2001GSLSP.190..205D. doi:10.1144/gsl.sp.2001.190.01.14. ISBN 978-1-86239-093-5. LCCN 2003464816. OCLC 48570033. S2CID 130092094.
- Darwin, Charles (1837). "B" Notebook. 이 노트북은 Wayback Machine에 있는 The Complete Work of Charles Darwin Online Archived 2022-03-18에서 구입할 수 있습니다.2019-10-09 검색.
- Darwin, Charles (1859). On the Origin of Species by Means of Natural Selection, or the Preservation of Favoured Races in the Struggle for Life (1st ed.). London: John Murray. LCCN 06017473. OCLC 741260650. 이 책은 Wayback Machine에 있는 The Complete Work of Charles Darwin Online Archived 2015-01-27에서 구입할 수 있습니다.2014-11-21 검색.
- Darwin, Charles (1872). The Expression of the Emotions in Man and Animals. London: John Murray. LCCN 04002793. OCLC 1102785.
- Darwin, Francis, ed. (1909). The foundations of The origin of species, a sketch written in 1842 (PDF). Cambridge: Printed at the University Press. LCCN 61057537. OCLC 1184581. Archived (PDF) from the original on 4 March 2016. Retrieved 27 November 2014.
- Dennett, Daniel (1995). Darwin's Dangerous Idea: Evolution and the Meanings of Life. New York: Simon & Schuster. ISBN 978-0-684-80290-9. LCCN 94049158. OCLC 31867409.
- Dobzhansky, Theodosius (1968). "On Some Fundamental Concepts of Darwinian Biology". In Dobzhansky, Theodosius; Hecht, Max K.; Steere, William C. (eds.). Evolutionary Biology (1st ed.). New York: Appleton-Century-Crofts. pp. 1–34. doi:10.1007/978-1-4684-8094-8_1. ISBN 978-1-4684-8096-2. OCLC 24875357.
- Dobzhansky, Theodosius (1970). Genetics of the Evolutionary Process. New York: Columbia University Press. ISBN 978-0-231-02837-0. LCCN 72127363. OCLC 97663.
- Eldredge, Niles; Gould, Stephen Jay (1972). "Punctuated equilibria: an alternative to phyletic gradualism". In Schopf, Thomas J.M. (ed.). Models in Paleobiology. San Francisco, California: Freeman, Cooper. ISBN 978-0-87735-325-6. LCCN 72078387. OCLC 572084.
- Eldredge, Niles (1985). Time Frames: The Rethinking of Darwinian Evolution and the Theory of Punctuated Equilibria. New York: Simon & Schuster. ISBN 978-0-671-49555-8. LCCN 84023632. OCLC 11443805.
- Ewens, Warren J. (2004). Mathematical Population Genetics. Interdisciplinary Applied Mathematics. Vol. I. Theoretical Introduction (2nd ed.). New York: Springer-Verlag New York. ISBN 978-0-387-20191-7. LCCN 2003065728. OCLC 53231891.
- Fisher, Ronald A. (1930). The Genetical Theory of Natural Selection. Oxford: The Clarendon Press. ISBN 978-0-19-850440-5. LCCN 30029177. OCLC 18500548.
- Futuyma, Douglas J. (2004). "The Fruit of the Tree of Life: Insights into Evolution and Ecology". In Cracraft, Joel; Donoghue, Michael J. (eds.). Assembling the Tree of Life. Oxford; New York: Oxford University Press. ISBN 978-0-19-517234-8. LCCN 2003058012. OCLC 61342697. "2002년 뉴욕 미국 자연사 박물관에서 열린 심포지엄의 진행 상황"
- Futuyma, Douglas J. (2005). Evolution. Sunderland, Massachusetts: Sinauer Associates. ISBN 978-0-87893-187-3. LCCN 2004029808. OCLC 57311264.
- Futuyma, Douglas J.; Kirkpatrick, Mark (2017). Evolution (Fourth ed.). Sunderland, Massachusetts: Sinauer Associates. ISBN 978-1-60535-605-1. LCCN 2017000562. OCLC 969439375.
- Gould, Stephen Jay (2002). The Structure of Evolutionary Theory. Cambridge, Massachusetts: Belknap Press of Harvard University Press. ISBN 978-0-674-00613-3. LCCN 2001043556. OCLC 47869352.
- Gray, Peter (2007). Psychology (5th ed.). New York: Worth Publishers. ISBN 978-0-7167-0617-5. LCCN 2006921149. OCLC 76872504.
- Hall, Brian K.; Hallgrímsson, Benedikt (2008). Strickberger's Evolution (4th ed.). Sudbury, Massachusetts: Jones and Bartlett Publishers. ISBN 978-0-7637-0066-9. LCCN 2007008981. OCLC 85814089.
- Hennig, Willi (1999) [Originally published 1966 (reprinted 1979); translated from the author's unpublished revision of Grundzüge einer Theorie der phylogenetischen Systematik, published in 1950]. Phylogenetic Systematics. Translation by D. Dwight Davis and Rainer Zangerl; foreword by Donn E. Rosen, Gareth Nelson, and Colin Patterson (Reissue ed.). Urbana, Illinois: University of Illinois Press. ISBN 978-0-252-06814-0. LCCN 78031969. OCLC 722701473.
- Holland, John H. (1975). Adaptation in Natural and Artificial Systems: An Introductory Analysis with Applications to Biology, Control, and Artificial Intelligence. Ann Arbor, Michigan: University of Michigan Press. ISBN 978-0-472-08460-9. LCCN 74078988. OCLC 1531617.
- Kampourakis, Kostas (2014). Understanding Evolution. Cambridge; New York: Cambridge University Press. ISBN 978-1-107-03491-4. LCCN 2013034917. OCLC 855585457.
- Kirk, Geoffrey; Raven, John; Schofield, Malcolm (1983). The Presocratic Philosophers: A Critical History with a Selection of Texts (2nd ed.). Cambridge; New York: Cambridge University Press. ISBN 978-0-521-27455-5. LCCN 82023505. OCLC 9081712.
- Koza, John R. (1992). Genetic Programming: On the Programming of Computers by Means of Natural Selection. Complex Adaptive Systems. Cambridge, Massachusetts: MIT Press. ISBN 978-0-262-11170-6. LCCN 92025785. OCLC 26263956.
- Lamarck, Jean-Baptiste (1809). Philosophie Zoologique. Paris: Dentu et L'Auteur. OCLC 2210044. 인터넷 아카이브의 철학 동물학 (1809)2014-11-29 검색.
- Lane, David H. (1996). The Phenomenon of Teilhard: Prophet for a New Age (1st ed.). Macon, Georgia: Mercer University Press. ISBN 978-0-86554-498-7. LCCN 96008777. OCLC 34710780.
- Levinson, Gene (2019). Rethinking Evolution: The Revolution That's Hiding in Plain Sight. Hackensack, New Jersey: World Scientific. ISBN 978-1-78634-726-8. LCCN 2019013762. OCLC 1138095098. Archived from the original on 21 May 2022. Retrieved 30 July 2022.
- Lucretius. "Book V, lines 855–877". De Rerum Natura. Edited and translated by William Ellery Leonard (1916). Medford/Somerville, Massachusetts: Tufts University. OCLC 33233743. Archived from the original on 4 September 2014. Retrieved 25 November 2014 – via Perseus Digital Library.
- Mason, Stephen F. (1962). A History of the Sciences. Collier Books. Science Library, CS9 (New rev. ed.). New York: Collier Books. LCCN 62003378. OCLC 568032626.
- Maynard Smith, John (1978). The Evolution of Sex. Cambridge; New York: Cambridge University Press. ISBN 978-0-521-29302-0. LCCN 77085689. OCLC 3413793.
- Maynard Smith, John (1998). "The Units of Selection". In Bock, Gregory R.; Goode, Jamie A. (eds.). The Limits of Reductionism in Biology. Novartis Foundation Symposia. Vol. 213. Chichester; New York: John Wiley & Sons. pp. 203–221. doi:10.1002/9780470515488.ch15. ISBN 978-0-471-97770-4. LCCN 98002779. OCLC 38311600. PMID 9653725. "1997년 5월 13일부터 15일까지 런던 노바티스 재단에서 열린 생물학의 환원주의 한계에 관한 심포지엄의 논문."
- Mayr, Ernst (1942). Systematics and the Origin of Species from the Viewpoint of a Zoologist. Columbia Biological Series. Vol. 13. New York: Columbia University Press. LCCN 43001098. OCLC 766053.
- Mayr, Ernst (1982). The Growth of Biological Thought: Diversity, Evolution, and Inheritance. Translation of John Ray by E. Silk. Cambridge, Massachusetts: Belknap Press. ISBN 978-0-674-36445-5. LCCN 81013204. OCLC 7875904.
- Mayr, Ernst (2002) [Originally published 2001; New York: Basic Books]. What Evolution Is. Science Masters. London: Weidenfeld & Nicolson. ISBN 978-0-297-60741-0. LCCN 2001036562. OCLC 248107061.
- McKinney, Michael L. (1997). "How do rare species avoid extinction? A paleontological view". In Kunin, William E.; Gaston, Kevin J. (eds.). The Biology of Rarity: Causes and consequences of rare—common differences (1st ed.). London; New York: Chapman & Hall. ISBN 978-0-412-63380-5. LCCN 96071014. OCLC 36442106.
- Miller, G. Tyler; Spoolman, Scott E. (2012). Environmental Science (14th ed.). Belmont, California: Brooks/Cole. ISBN 978-1-111-98893-7. LCCN 2011934330. OCLC 741539226. Archived from the original on 2 May 2019. Retrieved 27 December 2014.
- Nardon, Paul; Grenier, Anne-Marie (1991). "Serial Endosymbiosis Theory and Weevil Evolution: The Role of Symbiosis". In Margulis, Lynn; Fester, René (eds.). Symbiosis as a Source of Evolutionary Innovation: Speciation and Morphogenesis. Cambridge, Massachusetts: MIT Press. ISBN 978-0-262-13269-5. LCCN 90020439. OCLC 22597587. "1989년 6월 25일부터 30일까지 이탈리아 벨라지오에서 열린 회의를 기준으로"
- National Academy of Sciences; Institute of Medicine (2008). Science, Evolution, and Creationism. Washington, DC: National Academy Press. ISBN 978-0-309-10586-6. LCCN 2007015904. OCLC 123539346. Retrieved 22 November 2014.
- Odum, Eugene P. (1971). Fundamentals of Ecology (3rd ed.). Philadelphia, Pennsylvania: Saunders. ISBN 978-0-7216-6941-0. LCCN 76081826. OCLC 154846.
- Okasha, Samir (2006). Evolution and the Levels of Selection. Oxford; New York: Oxford University Press. ISBN 978-0-19-926797-2. LCCN 2006039679. OCLC 70985413.
- Panno, Joseph (2005). The Cell: Evolution of the First Organism. Facts on File science library. New York: Facts on File. ISBN 978-0-8160-4946-2. LCCN 2003025841. OCLC 53901436.
- Piatigorsky, Joram; Kantorow, Marc; Gopal-Srivastava, Rashmi; Tomarev, Stanislav I. (1994). "Recruitment of enzymes and stress proteins as lens crystallins". In Jansson, Bengt; Jörnvall, Hans; Rydberg, Ulf; et al. (eds.). Toward a Molecular Basis of Alcohol Use and Abuse. Experientia. Vol. 71. Basel; Boston: Birkhäuser Verlag. pp. 241–50. doi:10.1007/978-3-0348-7330-7_24. ISBN 978-3-7643-2940-2. LCCN 94010167. OCLC 30030941. PMID 8032155.
- Pigliucci, Massimo; Müller, Gerd B., eds. (2010). Evolution, the Extended Synthesis. Cambridge, Massachusetts: MIT Press. ISBN 978-0-262-51367-8. LCCN 2009024587. OCLC 804875316. Archived from the original on 18 September 2015.
- Provine, William B. (1971). The Origins of Theoretical Population Genetics. Chicago History of Science and Medicine (2nd ed.). Chicago, Illinois: University of Chicago Press. ISBN 978-0-226-68464-2. LCCN 2001027561. OCLC 46660910.
- Raven, Peter H.; Johnson, George B. (2002). Biology (6th ed.). Boston, Massachusetts: McGraw-Hill. ISBN 978-0-07-112261-0. LCCN 2001030052. OCLC 45806501.
- Ray, John (1686). Historia Plantarum [History of Plants]. Vol. I. Londini: Typis Mariæ Clark. LCCN agr11000774. OCLC 2126030.
- Rechenberg, Ingo (1973). Evolutionsstrategie; Optimierung technischer Systeme nach Prinzipien der biologischen Evolution (PhD thesis). Problemata (in German). Vol. 15. Afterword by Manfred Eigen. Stuttgart-Bad Cannstatt: Frommann-Holzboog. ISBN 978-3-7728-0373-4. LCCN 74320689. OCLC 9020616.
- Ridley, Mark (2004). Evolution. Oxford: Blackwell. ISBN 978-1-4051-0345-9.
- Stearns, Beverly Peterson; Stearns, Stephen C. (1999). Watching, from the Edge of Extinction. New Haven, Connecticut: Yale University Press. ISBN 978-0-300-08469-6. LCCN 98034087. OCLC 803522914.
- Stevens, Anthony (1982). Archetype: A Natural History of the Self. London: Routledge & Kegan Paul. ISBN 978-0-7100-0980-7. LCCN 84672250. OCLC 10458367.
- Voet, Donald; Voet, Judith G.; Pratt, Charlotte W. (2016). Fundamentals of Biochemistry: Life at the Molecular Level (Fifth ed.). Hoboken, New Jersey: John Wiley & Sons. ISBN 978-1-118-91840-1. LCCN 2016002847. OCLC 939245154.
- Wright, Sewall (1984). Genetic and Biometric Foundations. Evolution and the Genetics of Populations. Vol. 1. Chicago, Illinois: University of Chicago Press. ISBN 978-0-226-91038-3. LCCN 67025533. OCLC 246124737.
추가열람
| 라이브러리 리소스정보 진화 |
- 입문서 읽기
- Barrett, Paul H.; Weinshank, Donald J.; Gottleber, Timothy T., eds. (1981). A Concordance to Darwin's Origin of Species, First Edition. Ithaca, New York: Cornell University Press. ISBN 978-0-8014-1319-3. LCCN 80066893. OCLC 610057960.
- Carroll, Sean B. (2005). Endless Forms Most Beautiful: The New Science of Evo Devo and the Making of the Animal Kingdom. illustrations by Jamie W. Carroll, Josh P. Klaiss, Leanne M. Olds (1st ed.). New York: W.W. Norton & Company. ISBN 978-0-393-06016-4. LCCN 2004029388. OCLC 57316841.
- Charlesworth, Brian; Charlesworth, Deborah (2003). Evolution: A Very Short Introduction. Very Short Introductions. Oxford; New York: Oxford University Press. ISBN 978-0-19-280251-4. LCCN 2003272247. OCLC 51668497.
- Gould, Stephen Jay (1989). Wonderful Life: The Burgess Shale and the Nature of History (1st ed.). New York: W.W. Norton & Company. ISBN 978-0-393-02705-1. LCCN 88037469. OCLC 18983518.
- Jones, Steve (1999). Almost Like a Whale: The Origin of Species Updated. London; New York: Doubleday. ISBN 978-0-385-40985-8. LCCN 2002391059. OCLC 41420544.
- —— (2000). Darwin's Ghost: The Origin of Species Updated (1st ed.). New York: Random House. ISBN 978-0-375-50103-6. LCCN 99053246. OCLC 42690131. 미국판.
- Mader, Sylvia S. (2007). Biology. Significant contributions by Murray P. Pendarvis (9th ed.). Boston, Massachusetts: McGraw-Hill Higher Education. ISBN 978-0-07-246463-4. LCCN 2005027781. OCLC 61748307.
- Maynard Smith, John (1993). The Theory of Evolution (Canto ed.). Cambridge; New York: Cambridge University Press. ISBN 978-0-521-45128-4. LCCN 93020358. OCLC 27676642.
- Pallen, Mark J. (2009). The Rough Guide to Evolution. Rough Guides Reference Guides. London; New York: Rough Guides. ISBN 978-1-85828-946-5. LCCN 2009288090. OCLC 233547316.
- 고급판독서
- Barton, Nicholas H.; Briggs, Derek E.G.; Eisen, Jonathan A.; et al. (2007). Evolution. Cold Spring Harbor, New York: Cold Spring Harbor Laboratory Press. ISBN 978-0-87969-684-9. LCCN 2007010767. OCLC 86090399.
- Coyne, Jerry A.; Orr, H. Allen (2004). Speciation. Sunderland, Massachusetts: Sinauer Associates. ISBN 978-0-87893-089-0. LCCN 2004009505. OCLC 55078441.
- Bergstrom, Carl T.; Dugatkin, Lee Alan (2012). Evolution (1st ed.). New York: W.W. Norton & Company. ISBN 978-0-393-91341-5. LCCN 2011036572. OCLC 729341924.
- Hall, Brian K.; Olson, Wendy, eds. (2003). Keywords and Concepts in Evolutionary Developmental Biology. Cambridge, Massachusetts: Harvard University Press. ISBN 978-0-674-00904-2. LCCN 2002192201. OCLC 50761342.
- Kauffman, Stuart A. (1993). The Origins of Order: Self-organization and Selection in Evolution. New York; Oxford: Oxford University Press. ISBN 978-0-19-507951-7. LCCN 91011148. OCLC 895048122.
- Maynard Smith, John; Szathmáry, Eörs (1995). The Major Transitions in Evolution. Oxford; New York: W.H. Freeman Spektrum. ISBN 978-0-7167-4525-9. LCCN 94026965. OCLC 30894392.
- Mayr, Ernst (2001). What Evolution Is. New York: Basic Books. ISBN 978-0-465-04426-9. LCCN 2001036562. OCLC 47443814.
- Minelli, Alessandro (2009). Forms of Becoming: The Evolutionary Biology of Development. Translation by Mark Epstein. Princeton, New Jersey; Oxford: Princeton University Press. ISBN 978-0-691-13568-7. LCCN 2008028825. OCLC 233030259.
외부 링크
- 일반정보
- BBC에서 우리의 시간에 "진화"
- "Evolution Resources from the National Academies". Washington, DC: National Academy of Sciences. Retrieved 30 May 2011.
- "Understanding Evolution: your one-stop resource for information on evolution". Berkeley, California: University of California, Berkeley. Retrieved 30 May 2011.
- "Evolution of Evolution – 150 Years of Darwin's 'On the Origin of Species'". Arlington County, Virginia: National Science Foundation. Archived from the original on 19 May 2011. Retrieved 30 May 2011.
- "Human Evolution Timeline Interactive". Smithsonian Institution, National Museum of Natural History. 28 January 2010. Retrieved 14 July 2018. Adobe Flash가 필요합니다.
- "미국 진화의 역사".살롱 (검색어 2021-08-24)
- 비디오 (1980; 코스모스 애니메이션; 8:01): "진화" – 칼 세이건 유튜브
- 실험
- Lenski, Richard E. "Experimental Evolution". East Lansing, Michigan: Michigan State University. Retrieved 31 July 2013.
- Chastain, Erick; Livnat, Adi; Papadimitriou, Christos; Vazirani, Umesh (22 July 2014). "Algorithms, games, and evolution". PNAS. 111 (29): 10620–10623. Bibcode:2014PNAS..11110620C. doi:10.1073/pnas.1406556111. ISSN 0027-8424. PMC 4115542. PMID 24979793.
- 온라인강의
- "Evolution Matters Lecture Series". Harvard Online Learning Portal. Cambridge, Massachusetts: Harvard University. Archived from the original on 18 December 2017. Retrieved 15 July 2018.
- Stearns, Stephen C. "EEB 122: Principles of Evolution, Ecology and Behavior". Open Yale Courses. New Haven, Connecticut: Yale University. Archived from the original on 1 December 2017. Retrieved 14 July 2018.




