위 유전자.
Pseudogene
유사유전자는 기능적인 유전자를 닮은 DNA의 비기능적인 부분입니다. 대부분은 유전자 복제에 의해 직접적으로 또는 mRNA 전사체의 역전사에 의해 간접적으로 기능적 유전자의 불필요한 사본으로 발생합니다. 유사유전자는 일반적으로 유전체 서열 분석에서 전사 또는 번역에 필요한 조절 서열이 결여되어 있거나 프레임 시프트 또는 조기 정지 코돈으로 인해 코딩 서열에 명백한 결함이 있는 유전자 유사 서열을 발견할 때 식별됩니다. 유사유전자는 정크 DNA의 한 종류입니다.
대부분의 비박테리아 유전체는 기능 유전자만큼 많은 유사 유전자를 포함하고 있습니다. 이것은 놀라운 일이 아닙니다. 왜냐하면 다양한 생물학적 과정들이 실수로 유사유전자를 만들어낼 것으로 예상되고, 유전체에서 그것들을 제거할 전문적인 메커니즘이 없기 때문입니다. 결국 유사유전자는 DNA 복제나 DNA 복구 오류로 인해 유전체에서 삭제되거나 돌연변이가 너무 많이 발생하여 더 이상 이전 유전자로 인식할 수 없게 될 수 있습니다. 이러한 변성 사건의 분석은 유전체에서 비선택적 과정의 영향을 명확히 하는 데 도움이 됩니다.
유사유전자 서열은 조상 유전자로부터 유전되거나 새로운 돌연변이에 의해 발생하는 프로모터 요소로 인해 낮은 수준에서 RNA로 전사될 수 있습니다. 이러한 전사체의 대부분은 유전체의 다른 부분의 우연한 전사체보다 더 기능적인 중요성은 없을 것이지만, 일부는 유익한 조절 RNA와 새로운 단백질을 탄생시켰습니다.
특성.
이 섹션에는 돌연변이의 더 빠른 축적에 대한 정보가 없습니다. (2023년 12월) |
유사유전자는 일반적으로 알려진 유전자와의 유사성과 일부 기능 상실의 조합으로 특징지어집니다. 즉, 모든 유사 유전자는 일부 기능 유전자와 유사한 DNA 서열을 가지고 있지만, 일반적으로 기능적인 최종 단백질 제품을 생산할 수 없습니다.[1] 유사성과 기능 상실이라는 두 가지 요구 사항은 보통 생물학적으로 입증된 것이 아니라 서열 정렬을 통해 암시되기 때문에 유사 유전자는 유전체에서 식별하고 특성화하기 어려운 경우가 있습니다.
- 상동성은 유사유전자의 DNA 서열과 알려진 유전자 사이의 서열 유사성에 의해 암시됩니다. 두 시퀀스를 정렬한 후 동일한 염기쌍의 백분율을 계산합니다. 서열 동일성이 높다는 것은 이 두 서열이 공통 조상 서열에서 분기되었을 가능성이 높고(상동적임), 이 두 서열이 독립적으로 진화했을 가능성은 매우 낮다는 것을 의미합니다(수렴 진화 참조).
- 비기능성은 여러 가지 방법으로 나타날 수 있습니다. 일반적으로 유전자는 완전히 기능하는 단백질로 여러 단계를 거쳐야 합니다. 전사, 사전 mRNA 처리, 번역 및 단백질 접힘이 모두 이 과정에서 필요한 부분입니다. 이러한 단계 중 하나라도 실패하면 시퀀스가 작동하지 않는 것으로 간주될 수 있습니다. 고처리량 유사유전자 식별에서 가장 일반적으로 확인되는 비활성화는 조기 정지 코돈 및 프레임 시프트이며, 이는 거의 보편적으로 기능성 단백질 생성물의 번역을 방지합니다.
RNA 유전자에 대한 유사유전자는 번역할 필요가 없기 때문에 "판독 프레임"이 없기 때문에 일반적으로 발견하기가 더 어렵습니다. rDNA 배열 말단의 변화에 기초하여 다수의 rRNA 유사유전자가 확인되었습니다.[2]
유사유전자는 분자 유전학 연구를 복잡하게 할 수 있습니다. 예를 들어 PCR에 의한 유전자 증폭은 유사한 서열을 공유하는 유사 유전자를 동시에 증폭시킬 수 있습니다. 이를 PCR 바이어스 또는 증폭 바이어스라고 합니다. 유사하게, 유사유전자는 유전체 서열에서 유전자로서 주석을 달기도 합니다.
가공된 유사유전자는 종종 유전자 예측 프로그램에 문제를 제기하며 종종 실제 유전자 또는 엑손으로 오인됩니다. 가공된 유사유전자의 동정이 유전자 예측 방법의 정확도 향상에 도움을 줄 수 있다고 제안되었습니다.[3]
2014년에 140개의 인간 유사유전자가 번역된 것으로 나타났습니다.[4] 그러나 단백질 제품의 기능은 알려져 있지 않습니다.
종류 및 원산지
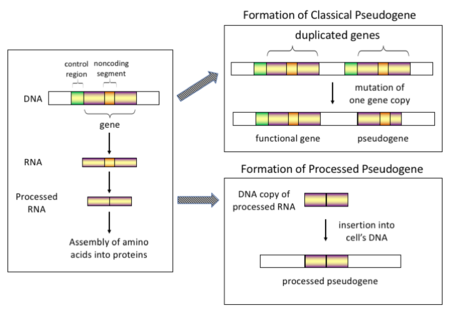
유사유전자에는 크게 네 가지 유형이 있으며, 모두 기원의 메커니즘과 특징적인 특징을 가지고 있습니다. 유사유전자의 분류는 다음과 같습니다.
가공된

고등 진핵생물, 특히 포유류에서 역전사는 유전체 구성에 큰 영향을 미친 상당히 일반적인 사건입니다. 예를 들어 인간 게놈의 30~44%는 SINE 및 LINE과 같은 반복 요소로 구성됩니다(레트로트랜스포존 참조).[7][8] 역전사 과정에서 유전자의 mRNA나 hnRNA 전사체의 일부가 자발적으로 다시 DNA로 역전사되어 염색체 DNA에 삽입됩니다. 레트로트랜스포존은 일반적으로 자신의 사본을 생성하지만, 무작위 유전자의 레트로트랜스포존 사본도 생성할 수 있다는 것이 시험관 내 시스템에서 밝혀졌습니다.[9] 일단 이 유사유전자들이 유전체에 다시 삽입되면, 그것들은 보통 poly-A 꼬리를 포함하고 있고, 대개 그들의 인트론을 접합하였습니다; 이것들은 모두 cDNA의 특징적인 특징들입니다. 그러나 가공된 유사유전자는 RNA 산물에서 유래하기 때문에 정상 유전자의 업스트림 프로모터가 부족하기 때문에 "도착 시 사망"으로 간주되어 역전사 사건이 발생하는 즉시 비기능적 유사유전자가 됩니다.[10] 그러나 이러한 삽입은 때때로 대체적으로 접합된 전사체를 통해 기존 유전자에 엑손을 기여합니다.[11] 가공된 유사유전자의 또 다른 특징은 가공된 유사유전자를 생성하는 상대적으로 비가공적인 역전이 메커니즘의 결과인 부모 서열에 대한 5' 말단의 일반적인 절단입니다.[12] 가공된 유사 유전자는 영장류에서 지속적으로 생성되고 있습니다.[13] 예를 들어, 인간 개체군은 개체에 걸쳐 처리된 유사 유전자 세트가 서로 다릅니다.[14]
가공된 유사유전자는 가공되지 않은 유사유전자보다 돌연변이를 더 빨리 축적하는 것으로 나타났습니다.[15]
미가공(중복)

유전자 복제는 유전체 진화에서 또 다른 일반적이고 중요한 과정입니다. 기능적인 유전자의 사본은 예를 들어, 잘못된 정렬된 염색체에서 반복적인 SINE 서열에서 상동 재조합에 의해 야기된 유전자 복제 사건의 결과로 발생할 수 있고, 그 결과로 사본이 원래 유전자의 기능을 상실하게 하는 돌연변이를 획득합니다. 복제된 유사유전자는 일반적으로 온전한 엑손-인트론 구조 및 조절 서열을 포함하여 유전자와 동일한 특성을 갖습니다. 복제된 유전자의 기능 상실은 온전한 기능적 사본이 여전히 존재하기 때문에 보통 유기체의 적합성에 거의 영향을 미치지 않습니다. 일부 진화 모델에 따르면, 공유된 복제 유사 유전자는 인간과 다른 영장류의 진화적 관련성을 나타냅니다.[16] 유사유전자화가 유전자 복제로 인한 것이라면, 유전자가 선택 압력을 받지 않았다면 유전자 복제 후 처음 몇 백만 년 동안 발생합니다.[17] 유전자 복제는 기능적 중복을 발생시키며 두 개의 동일한 유전자를 보유하는 것은 일반적으로 유리하지 않습니다. 두 유전자 중 하나의 구조나 기능을 방해하는 돌연변이는 유해하지 않으며 선택 과정을 통해 제거되지 않습니다. 그 결과 돌연변이를 일으킨 유전자는 점차 유사유전자가 되어 발현되지 않거나 기능을 상실하게 됩니다. 이런 종류의 진화적 운명은 집단 유전자 모델링과[18][19] 유전체 분석을 통해서도 나타납니다.[17][20] 진화적 맥락에 따르면, 이 유사 유전자들은 삭제되거나 부모 유전자들과 너무 구별되어 더 이상 식별할 수 없게 될 것입니다. 비교적 젊은 유사 유전자는 서열 유사성으로 인해 인식될 수 있습니다.[21]
유니터리 유사유전자

(인델이나 넌센스 돌연변이와 같은) 다양한 돌연변이는 유전자가 정상적으로 전사되거나 번역되는 것을 막을 수 있으므로 유전자가 덜 기능적이거나 비기능적이거나 "비활성화"될 수 있습니다. 이것들은 가공되지 않은 유전자가 유사유전자가 되는 것과 같은 메커니즘이지만, 이 경우의 차이점은 유사유전자화가 되기 전에 유전자가 복제되지 않았다는 것입니다. 일반적으로 이러한 유사 유전자는 개체군에서 고정될 가능성이 적지만 유전적 드리프트, 개체군 병목 현상 또는 경우에 따라 자연 선택과 같은 다양한 개체군 효과로 인해 고정될 수 있습니다. 단일 유사유전자의 전형적인 예는 영장류에서 L-gulono- γ-락톤 산화효소(GULO)를 코딩한 것으로 추정되는 유전자입니다. 영장류 외에 연구된 모든 포유류(기니피그 제외)에서 GULO는 아스코르브산(비타민 C)의 생합성을 돕지만 인간과 다른 영장류에서는 장애 유전자(GULOP)로 존재합니다.[22][23] 장애 유전자의 또 다른 최근 예는 (말도 안 되는 돌연변이를 통해) 카스파제 12 유전자의 비활성화를 인간의 양성 선택과 연결시킵니다.[24]
유사유전자 함수의 예
대다수의 유사유전자가 기능을 상실한 반면, 유사유전자가 원래 또는 유사한 기능을 다시 얻거나 새로운 기능을 진화시킨 사례가 나타났습니다. 인간 게놈에서, 원래는 유사유전자로 분류되었지만 나중에 반드시 단백질 코딩은 아니지만 기능적인 역할을 하는 것으로 밝혀진 많은 예들이 확인되었습니다.[25][26]
예를 들면 다음과 같습니다.
단백질 코딩:"유사 pseud 유전자"

DNA 염기서열 분석 기술의 급속한 확산으로 유전자 예측 기술을 사용하여 많은 명백한 유사 유전자가 확인되었습니다. 유사유전자는 종종 예측된 mRNA 서열에서 조기 정지 코돈의 출현으로 식별되며, 이는 이론적으로 원래 유전자의 정상적인 단백질 생성물의 합성(번역)을 방지합니다. 포유류에서 이러한 조기 정지 코돈의 번역 판독에 대한 일부 보고가 있었습니다. 위의 그림에서 언급된 바와 같이, 이러한 판독의 소량의 단백질 생성물은 여전히 어느 수준에서 인식되고 기능할 수 있습니다. 그렇다면 의사 유전자는 자연 선택의 대상이 될 수 있습니다. 그것은 초파리 종의 진화 과정에서 일어난 것으로 보입니다.
2016년에 여러 초파리 종에서 예측된 4개의 유사유전자가 실제로 생물학적으로 중요한 기능을 가진 단백질을 암호화한다고 보고되었으며,[27] "이러한 '유사유전자'가 광범위한 현상을 나타낼 수 있음을 시사합니다." 예를 들어, Ir75a 유전자의 기능성 단백질(글루타메이트 후각 수용체)은 뉴런에서만 발견됩니다. 인 실리코 분석으로 유사유전자로 분류될 수 있었던 조직 특이적 생물학적 기능 유전자의 이러한 발견은 서열 데이터 분석을 복잡하게 만듭니다.[27] 또 다른 Drosophilia pseudogene은 생체 내에서 기능성 알코올 탈수소효소 효소를 암호화하는 [28][29]jingwei입니다.[30]
2012년 현재 인간 게놈에는 약 12,000~14,000개의 유사유전자가 있는 것으로 나타났습니다.[31] 펩타이드의 질량분석을 이용한 2016년 프로테오제노믹스 분석은 16,271개의 유전자 또는 유전자 군집에서 생성된 최소 19,262개의 인간 단백질을 확인했으며, 이전에 유사유전자로 간주되었던 8개의 새로운 단백질 코딩 유전자를 확인했습니다.[32] 이전의 분석에서는 이전에 유사 유전자로 생각되었던 [33]인간 PGAM4(포스포글리세르산 뮤테이스)가 재미있을 뿐만 아니라 돌연변이를 일으키면 불임을 유발한다는 것을 발견했습니다.[34][35]
많은 유사 유전자가 원핵생물에서도 발견되었으며, 필수 유전자의 일부 정지 코돈 치환은 유지되는 것으로 보이며, 심지어 긍정적으로 선택됩니다.[36][37]
비단백질 코딩
siRNAs. 일부 내인성 siRNA는 유사유전자에서 파생된 것으로 보이며, 따라서 일부 유사유전자는 검토된 바와 같이 단백질 코딩 전사체를 조절하는 역할을 합니다.[38] 많은 예 중 하나가 psiPPM1K입니다. psiPPM1K에서 전사된 RNA를 처리하면 가장 흔한 간암인 간세포암종을 억제하는 작용을 하는 siRNA가 생성됩니다.[39] 이것과 다른 많은 연구들은 치료제를[40] 사용하는/치료제로서 유사유전자를 표적으로 하는 가능성에 대한 상당한 흥분을 불러왔습니다.
piRNAs. 일부 piRNA는 piRNA 군집에 위치한 유사유전자에서 파생됩니다.[41] 이러한 piRNA는 포유류의 고환에서 piRNA 경로를 통해 유전자를 조절하고 유전체에 대한 전위 요소 손상을 제한하는 데 중요합니다.[42]

마이크로 RNA. microRNA의 decoid 역할을 하는 유사유전자 전사체에 대한 보고가 많습니다. 아마도 암에 관련된 그러한 유사유전자의 가장 초기의 결정적인 예는 BRAF의 유사유전자일 것입니다. BRAF 유전자는 돌연변이를 일으키면 많은 암과 관련이 있는 원발암 유전자입니다. 일반적으로 BRAF 단백질의 양은 miRNA의 작용을 통해 세포 내에서 조절됩니다. 정상적인 상황에서는 BRAF와 유사유전자 BRAFP1의 RNA 양이 miRNA를 놓고 경쟁하지만, 두 RNA의 균형은 세포가 정상적으로 성장하는 정도입니다. 그러나 BRAFP1 RNA 발현이 증가하면 (실험적으로 또는 자연적인 돌연변이에 의해) BRAF의 발현을 조절하기 위해 더 적은 miRNA를 이용할 수 있고, 증가된 BRAF 단백질의 양은 암을 유발합니다.[43] 유전체에 내생적인 RNA에 의한 이런 종류의 조절 요소 경쟁은 ceRNA라는 용어를 만들어 냈습니다.
PTEN PTEN 유전자는 종양 억제 유전자로 알려져 있습니다. PTEN 유사유전자인 PTENP1은 야생형 유전자와 유전자 서열이 매우 유사한 가공된 유사유전자입니다. 그러나 PTENP1은 메티오닌을 개시하는 코돈을 제거하여 정상적인 PTEN 단백질의 번역을 막는 미스센스 돌연변이를 가지고 있습니다.[44] 그럼에도 불구하고 PTENP1은 종양 발생에 역할을 하는 것으로 보입니다. PTENP1 mRNA의 3' UTR은 PTEN 유전자와 유사하여 마이크로 RNA를 표적으로 하여 PTEN mRNA의 미끼로 기능하며, 3' UTR의 과발현으로 PTEN 단백질 수준이 증가하였습니다.[45] 즉, PTENP13' UTR의 과발현은 암성 종양의 조절 및 억제를 증가시킵니다. 이 시스템의 생물학은 기본적으로 위에서 설명한 BRAF 시스템의 역입니다.
포토제네스. 유사유전자는 진화적 시간 척도에 따라 유전자 전환 및 새로운 또는 새로운 기능 유전자를 생성할 수 있는 다른 돌연변이 사건에 참여할 수 있습니다. 이것은 유사유전자가 진화적 다양화를 위한 잠재적인 유전자인 포토유전자로 간주될 수 있다는 개념으로 이어졌습니다.[46]
세균유사유전자
유사유전자는 박테리아에서 발견됩니다.[47] 대부분은 자유 생활을 하지 않는 박테리아에서 발견됩니다. 즉, 공생체이거나 절대 세포 내 기생충입니다. 따라서 대사 및 DNA 복구와 관련된 유전자와 같이 자유 생활 박테리아가 필요로 하는 많은 유전자를 필요로 하지 않습니다. 하지만 기능적인 유전자가 먼저 없어지는 순서는 없습니다. 예를 들어, Mycobacterium leprae에서 가장 오래된 유사유전자는 RNA 중합효소와 2차 대사산물의 생합성에 있고, Shigella flexneri와 Shigella typhi에서 가장 오래된 유사유전자는 DNA 복제, 재조합 및 복구에 있습니다.[48]
유사유전자를 운반하는 대부분의 박테리아는 공생체이거나 절대적인 세포 내 기생충이기 때문에 결국 유전체 크기가 줄어듭니다. 절대 기생충이자 한센병의 원인균인 마이코박테리움 레프래의 유전체가 극단적인 예입니다. 전사체의 약 50%를 생성하는 1,133개의 유사 유전자를 가지고 있는 것으로 보고되었습니다.[48] 동종 병원체인 마이코박테리움 마리눔과 비교해보면 유사유전자와 유전체 감소 효과를 더욱 확인할 수 있습니다. 마이코박테리움 마리눔은 숙주 밖에서도 생존할 수 있기 때문에 마이코박테리움 레프래에 비해 유전체가 크기 때문에 유전체에 필요한 유전자가 포함되어 있어야 합니다.[49]
비록 게놈 감소는 유사유전자를 제거함으로써 필요하지 않은 유전자에 초점을 맞추지만, 숙주로부터의 선택적인 압력은 유지되는 것을 흔들 수 있습니다. Verrucomicrobiota 문의 공생체의 경우 mandelalide 경로를 코딩하는 유전자의 7개의 사본이 추가로 있습니다.[50] Lissoclinum의 종인 숙주는 mandelalides를 방어 메커니즘의 일부로 사용합니다.[50]
부흐네라 aphidicola에서 에피스타시스와 유전자 손실 도미노 이론의 관계가 관찰되었습니다. 도미노 이론은 세포 과정의 한 유전자가 비활성화되면 관련된 다른 유전자의 선택이 완화되어 유전자 손실이 발생한다고 제안합니다.[48] 부흐네라 aphidicola와 Escherichia coli를 비교했을 때, 양성 에피스타시스는 유전자 손실을 더 촉진하고 음성 에피스타시스는 이를 방해하는 것으로 나타났습니다.

이 기사에는 고세균에 대한 정보가 누락되어 있습니다. (2023년 12월) |
참고 항목
참고문헌
- ^ Mighell AJ, Smith NR, Robinson PA, Markham AF (February 2000). "Vertebrate pseudogenes". FEBS Letters. 468 (2–3): 109–114. doi:10.1016/S0014-5793(00)01199-6. PMID 10692568. S2CID 42204036.
- ^ Robicheau BM, Susko E, Harrigan AM, Snyder M (February 2017). "Ribosomal RNA Genes Contribute to the Formation of Pseudogenes and Junk DNA in the Human Genome". Genome Biology and Evolution. 9 (2): 380–397. doi:10.1093/gbe/evw307. PMC 5381670. PMID 28204512.
- ^ van Baren MJ, Brent MR (May 2006). "Iterative gene prediction and pseudogene removal improves genome annotation". Genome Research. 16 (5): 678–685. doi:10.1101/gr.4766206. PMC 1457044. PMID 16651666.
- ^ Kim MS, Pinto SM, Getnet D, Nirujogi RS, Manda SS, Chaerkady R, et al. (May 2014). "A draft map of the human proteome". Nature. 509 (7502): 575–581. Bibcode:2014Natur.509..575K. doi:10.1038/nature13302. PMC 4403737. PMID 24870542.
- ^ Max EE (1986). "Plagiarized Errors and Molecular Genetics". Creation Evolution Journal. 6 (3): 34–46.
- ^ Chandrasekaran C, Betrán E (2008). "Origins of new genes and pseudogenes". Nature Education. 1 (1): 181.
- ^ Jurka J (December 2004). "Evolutionary impact of human Alu repetitive elements". Current Opinion in Genetics & Development. 14 (6): 603–608. doi:10.1016/j.gde.2004.08.008. PMID 15531153.
- ^ Dewannieux M, Heidmann T (2005). "LINEs, SINEs and processed pseudogenes: parasitic strategies for genome modeling". Cytogenetic and Genome Research. 110 (1–4): 35–48. doi:10.1159/000084936. PMID 16093656. S2CID 25083962.
- ^ Dewannieux M, Esnault C, Heidmann T (September 2003). "LINE-mediated retrotransposition of marked Alu sequences". Nature Genetics. 35 (1): 41–48. doi:10.1038/ng1223. PMID 12897783. S2CID 32151696.
- ^ Graur D, Shuali Y, Li WH (April 1989). "Deletions in processed pseudogenes accumulate faster in rodents than in humans". Journal of Molecular Evolution. 28 (4): 279–285. Bibcode:1989JMolE..28..279G. doi:10.1007/BF02103423. PMID 2499684. S2CID 22437436.
- ^ Baertsch R, Diekhans M, Kent WJ, Haussler D, Brosius J (October 2008). "Retrocopy contributions to the evolution of the human genome". BMC Genomics. 9: 466. doi:10.1186/1471-2164-9-466. PMC 2584115. PMID 18842134.
- ^ Pavlícek A, Paces J, Zíka R, Hejnar J (October 2002). "Length distribution of long interspersed nucleotide elements (LINEs) and processed pseudogenes of human endogenous retroviruses: implications for retrotransposition and pseudogene detection". Gene. 300 (1–2): 189–194. doi:10.1016/S0378-1119(02)01047-8. PMID 12468100.
- ^ Navarro FC, Galante PA (July 2015). "A Genome-Wide Landscape of Retrocopies in Primate Genomes". Genome Biology and Evolution. 7 (8): 2265–2275. doi:10.1093/gbe/evv142. PMC 4558860. PMID 26224704.
- ^ Schrider DR, Navarro FC, Galante PA, Parmigiani RB, Camargo AA, Hahn MW, de Souza SJ (2013-01-24). "Gene copy-number polymorphism caused by retrotransposition in humans". PLOS Genetics. 9 (1): e1003242. doi:10.1371/journal.pgen.1003242. PMC 3554589. PMID 23359205.
- ^ Zheng D, Frankish A, Baertsch R, Kapranov P, Reymond A, Choo SW, et al. (June 2007). "Pseudogenes in the ENCODE regions: consensus annotation, analysis of transcription, and evolution". Genome Research. 17 (6): 839–851. doi:10.1101/gr.5586307. PMC 1891343. PMID 17568002.
- ^ Max EE (2003-05-05). "Plagiarized Errors and Molecular Genetics". TalkOrigins Archive. Retrieved 2008-07-22.
- ^ a b Lynch M, Conery JS (November 2000). "The evolutionary fate and consequences of duplicate genes". Science. 290 (5494): 1151–1155. Bibcode:2000Sci...290.1151L. doi:10.1126/science.290.5494.1151. PMID 11073452.
- ^ Walsh JB (January 1995). "How often do duplicated genes evolve new functions?". Genetics. 139 (1): 421–428. doi:10.1093/genetics/139.1.421. PMC 1206338. PMID 7705642.
- ^ Lynch M, O'Hely M, Walsh B, Force A (December 2001). "The probability of preservation of a newly arisen gene duplicate". Genetics. 159 (4): 1789–1804. doi:10.1093/genetics/159.4.1789. PMC 1461922. PMID 11779815.
- ^ Harrison PM, Hegyi H, Balasubramanian S, Luscombe NM, Bertone P, Echols N, et al. (February 2002). "Molecular fossils in the human genome: identification and analysis of the pseudogenes in chromosomes 21 and 22". Genome Research. 12 (2): 272–280. doi:10.1101/gr.207102. PMC 155275. PMID 11827946.
- ^ Zhang J (2003). "Evolution by gene duplication: an update". Trends in Ecology and Evolution. 18 (6): 292–298. doi:10.1016/S0169-5347(03)00033-8.
- ^ Nishikimi M, Kawai T, Yagi K (October 1992). "Guinea pigs possess a highly mutated gene for L-gulono-gamma-lactone oxidase, the key enzyme for L-ascorbic acid biosynthesis missing in this species". The Journal of Biological Chemistry. 267 (30): 21967–21972. doi:10.1016/S0021-9258(19)36707-9. PMID 1400507.
- ^ Nishikimi M, Fukuyama R, Minoshima S, Shimizu N, Yagi K (May 1994). "Cloning and chromosomal mapping of the human nonfunctional gene for L-gulono-gamma-lactone oxidase, the enzyme for L-ascorbic acid biosynthesis missing in man". The Journal of Biological Chemistry. 269 (18): 13685–13688. doi:10.1016/S0021-9258(17)36884-9. PMID 8175804.
- ^ Xue Y, Daly A, Yngvadottir B, Liu M, Coop G, Kim Y, et al. (April 2006). "Spread of an inactive form of caspase-12 in humans is due to recent positive selection". American Journal of Human Genetics. 78 (4): 659–670. doi:10.1086/503116. PMC 1424700. PMID 16532395.
- ^ Cheetham SW, Faulkner GJ, Dinger ME (March 2020). "Overcoming challenges and dogmas to understand the functions of pseudogenes". Nature Reviews. Genetics. 21 (3): 191–201. doi:10.1038/s41576-019-0196-1. PMID 31848477. S2CID 209393216.
- ^ Zerbino DR, Frankish A, Flicek P (August 2020). "Progress, Challenges, and Surprises in Annotating the Human Genome". Annual Review of Genomics and Human Genetics. 21 (1): 55–79. doi:10.1146/annurev-genom-121119-083418. PMC 7116059. PMID 32421357.
- ^ a b Prieto-Godino LL, Rytz R, Bargeton B, Abuin L, Arguello JR, Peraro MD, Benton R (November 2016). "Olfactory receptor pseudo-pseudogenes". Nature. 539 (7627): 93–97. Bibcode:2016Natur.539...93P. doi:10.1038/nature19824. PMC 5164928. PMID 27776356.
- ^ Jeffs P, Ashburner M (May 1991). "Processed pseudogenes in Drosophila". Proceedings. Biological Sciences. 244 (1310): 151–159. Bibcode:1991RSPSB.244..151J. doi:10.1098/rspb.1991.0064. PMID 1679549. S2CID 1665885.
- ^ Wang W, Zhang J, Alvarez C, Llopart A, Long M (September 2000). "The origin of the Jingwei gene and the complex modular structure of its parental gene, yellow emperor, in Drosophila melanogaster". Molecular Biology and Evolution. 17 (9): 1294–1301. doi:10.1093/oxfordjournals.molbev.a026413. PMID 10958846.
- ^ Long M, Langley CH (April 1993). "Natural selection and the origin of jingwei, a chimeric processed functional gene in Drosophila". Science. 260 (5104): 91–95. Bibcode:1993Sci...260...91L. doi:10.1126/science.7682012. PMID 7682012.
- ^ Pei B, Sisu C, Frankish A, Howald C, Habegger L, Mu XJ, et al. (September 2012). "The GENCODE pseudogene resource". Genome Biology. 13 (9): R51. doi:10.1186/gb-2012-13-9-r51. PMC 3491395. PMID 22951037.
- ^ Wright JC, Mudge J, Weisser H, Barzine MP, Gonzalez JM, Brazma A, et al. (June 2016). "Improving GENCODE reference gene annotation using a high-stringency proteogenomics workflow". Nature Communications. 7: 11778. Bibcode:2016NatCo...711778W. doi:10.1038/ncomms11778. PMC 4895710. PMID 27250503.
- ^ Dierick HA, Mercer JF, Glover TW (October 1997). "A phosphoglycerate mutase brain isoform (PGAM 1) pseudogene is localized within the human Menkes disease gene (ATP7 A)". Gene. 198 (1–2): 37–41. doi:10.1016/s0378-1119(97)00289-8. PMID 9370262.
- ^ Betrán E, Wang W, Jin L, Long M (May 2002). "Evolution of the phosphoglycerate mutase processed gene in human and chimpanzee revealing the origin of a new primate gene". Molecular Biology and Evolution. 19 (5): 654–663. doi:10.1093/oxfordjournals.molbev.a004124. PMID 11961099.
- ^ Okuda H, Tsujimura A, Irie S, Yamamoto K, Fukuhara S, Matsuoka Y, et al. (2012). "A single nucleotide polymorphism within the novel sex-linked testis-specific retrotransposed PGAM4 gene influences human male fertility". PLOS ONE. 7 (5): e35195. Bibcode:2012PLoSO...735195O. doi:10.1371/journal.pone.0035195. PMC 3348931. PMID 22590500.
- ^ Belinky F, Ganguly I, Poliakov E, Yurchenko V, Rogozin IB (February 2021). "Analysis of Stop Codons within Prokaryotic Protein-Coding Genes Suggests Frequent Readthrough Events". International Journal of Molecular Sciences. 22 (4): 1876. doi:10.3390/ijms22041876. PMID 33672790.
- ^ Feng Y, Wang Z, Chien KY, Chen HL, Liang YH, Hua X, Chiu CH (May 2022). ""Pseudo-pseudogenes" in bacterial genomes: Proteogenomics reveals a wide but low protein expression of pseudogenes in Salmonella enterica". Nucleic Acids Research. 50 (9): 5158–5170. doi:10.1093/nar/gkac302. PMID 35489061.
- ^ Chan WL, Chang JG (2014). "Pseudogene-Derived Endogenous siRNAs and Their Function". Pseudogenes. Methods in Molecular Biology. Vol. 1167. pp. 227–39. doi:10.1007/978-1-4939-0835-6_15. ISBN 978-1-4939-0834-9. PMID 24823781.
- ^ Chan WL, Yuo CY, Yang WK, Hung SY, Chang YS, Chiu CC, et al. (April 2013). "Transcribed pseudogene ψPPM1K generates endogenous siRNA to suppress oncogenic cell growth in hepatocellular carcinoma". Nucleic Acids Research. 41 (6): 3734–3747. doi:10.1093/nar/gkt047. PMC 3616710. PMID 23376929.
- ^ Roberts TC, Morris KV (December 2013). "Not so pseudo anymore: pseudogenes as therapeutic targets". Pharmacogenomics. 14 (16): 2023–2034. doi:10.2217/pgs.13.172. PMC 4068744. PMID 24279857.
- ^ Olovnikov I, Le Thomas A, Aravin AA (2014). "A Framework for piRNA Cluster Manipulation". PIWI-Interacting RNAs. Methods in Molecular Biology. Vol. 1093. pp. 47–58. doi:10.1007/978-1-62703-694-8_5. ISBN 978-1-62703-693-1. PMID 24178556.
- ^ Siomi MC, Sato K, Pezic D, Aravin AA (April 2011). "PIWI-interacting small RNAs: the vanguard of genome defence". Nature Reviews. Molecular Cell Biology. 12 (4): 246–258. doi:10.1038/nrm3089. PMID 21427766. S2CID 5710813.
- ^ Karreth FA, Reschke M, Ruocco A, Ng C, Chapuy B, Léopold V, et al. (April 2015). "The BRAF pseudogene functions as a competitive endogenous RNA and induces lymphoma in vivo". Cell. 161 (2): 319–332. doi:10.1016/j.cell.2015.02.043. PMC 6922011. PMID 25843629.
- ^ Dahia PL, FitzGerald MG, Zhang X, Marsh DJ, Zheng Z, Pietsch T, et al. (May 1998). "A highly conserved processed PTEN pseudogene is located on chromosome band 9p21". Oncogene. 16 (18): 2403–2406. doi:10.1038/sj.onc.1201762. PMID 9620558.
- ^ Poliseno L, Salmena L, Zhang J, Carver B, Haveman WJ, Pandolfi PP (June 2010). "A coding-independent function of gene and pseudogene mRNAs regulates tumour biology". Nature. 465 (7301): 1033–1038. Bibcode:2010Natur.465.1033P. doi:10.1038/nature09144. PMC 3206313. PMID 20577206.
- ^ Balakirev ES, Ayala FJ (2003). "Pseudogenes: are they "junk" or functional DNA?". Annual Review of Genetics. 37: 123–151. doi:10.1146/annurev.genet.37.040103.103949. PMID 14616058.
- ^ Goodhead I, Darby AC (February 2015). "Taking the pseudo out of pseudogenes" (PDF). Current Opinion in Microbiology. 23: 102–109. doi:10.1016/j.mib.2014.11.012. PMID 25461580.
- ^ a b c Dagan T, Blekhman R, Graur D (February 2006). "The "domino theory" of gene death: gradual and mass gene extinction events in three lineages of obligate symbiotic bacterial pathogens". Molecular Biology and Evolution. 23 (2): 310–316. doi:10.1093/molbev/msj036. PMID 16237210.
- ^ Malhotra S, Vedithi SC, Blundell TL (August 2017). "Decoding the similarities and differences among mycobacterial species". PLOS Neglected Tropical Diseases. 11 (8): e0005883. doi:10.1371/journal.pntd.0005883. PMC 5595346. PMID 28854187.
- ^ a b Lopera J, Miller IJ, McPhail KL, Kwan JC (November 21, 2017). "Increased Biosynthetic Gene Dosage in a Genome-Reduced Defensive Bacterial Symbiont". mSystems. 2 (6): 1–18. doi:10.1128/msystems.00096-17. PMC 5698493. PMID 29181447.
- ^ Cole ST, Eiglmeier K, Parkhill J, James KD, Thomson NR, Wheeler PR, et al. (February 2001). "Massive gene decay in the leprosy bacillus". Nature. 409 (6823): 1007–1011. Bibcode:2001Natur.409.1007C. doi:10.1038/35059006. PMID 11234002. S2CID 4307207.
더보기
- Gerstein M, Zheng D (August 2006). "The real life of pseudogenes". Scientific American. 295 (2): 48–55. Bibcode:2006SciAm.295b..48G. doi:10.1038/scientificamerican0806-48. PMID 16866288.
- Torrents D, Suyama M, Zdobnov E, Bork P (December 2003). "A genome-wide survey of human pseudogenes". Genome Research. 13 (12): 2559–2567. doi:10.1101/gr.1455503. PMC 403797. PMID 14656963.
- Bischof JM, Chiang AP, Scheetz TE, Stone EM, Casavant TL, Sheffield VC, Braun TA (June 2006). "Genome-wide identification of pseudogenes capable of disease-causing gene conversion". Human Mutation. 27 (6): 545–552. doi:10.1002/humu.20335. PMID 16671097. S2CID 20219423.
- Syberg-Olsen MJ, Garber AI, Keeling PJ, McCutcheon JP, Husnik F (July 2022). "Pseudofinder: Detection of Pseudogenes in Prokaryotic Genomes". Molecular Biology and Evolution. 39 (7). doi:10.1093/molbev/msac153. PMID 35801562.


