인구유전학
Population genetics| 다음에 대한 시리즈 일부 |
| 진화생물학 |
|---|
 |
| 다음에 대한 시리즈 일부 |
| 유전적 계보 |
|---|
| 개념 |
| 관련 항목 |
인구유전학은 인구 내부와 인구 사이의 유전적 차이를 다루는 유전학의 하위 분야로 진화생물학의 한 부분이다.생물학의 이 학과의 연구는 적응, 특정화, 인구구조와 같은 현상을 조사한다.[1]
인구유전학은 현대 진화합성의 출현에 중요한 요소였다.이것의 주요 설립자는 Sewall Wright, J. B. S. Haldane, Ronald Fisher 등이었는데, 이들도 양적 유전학의 관련 규율의 기초를 닦았다.전통적으로 고도의 수학적 학문인 현대 인구 유전학은 이론, 실험실, 현장 연구를 포함한다.모집단 유전자 모델은 DNA 시퀀스 데이터로부터의 통계적 추론과 개념의 증명/분석에 모두 사용된다.[2]
진화적 게임 이론과 적응적 역학 같은 모델링 진화에 대한 더 새롭고 표현적인 접근법과는 인구 유전학을 구분하는 것은 지배력, 인식론, 유전적 재조합이 불균형 관계를 끊는 정도, 돌연변이와 유전적 표류의 무작위적 현상 등과 같은 유전적 현상에 중점을 두고 있다.이는 모집단 게놈 데이터와의 비교에 적합하게 만든다.
역사
인구유전학은 멘델의 상속과 생물통계학적 모델의 조화로서 시작되었다.자연 선택은 한 모집단에 충분한 유전적 변화가 있을 경우에만 진화를 일으킬 것이다.멘델 유전학의 발견 이전에, 한 가지 일반적인 가설은 유산을 혼합하는 것이었다.그러나 혼합 유전과 함께 유전적 분산이 급격히 감소하여 자연적 또는 성적인 선택에 의한 진화를 신뢰할 수 없게 된다.하디-웨인버그 원칙은 멘델 유산을 가진 인구에서 변화가 어떻게 유지되는지에 대한 해결책을 제공한다.이 원리에 따르면 알레르기의 빈도(유전자 내 변이)는 선택, 돌연변이, 이주 및 유전적 표류가 없는 경우에도 일정하게 유지된다.[3]
다음 핵심 단계는 영국의 생물학자 겸 통계학자 로널드 피셔의 작업이었다.피셔는 1918년부터 시작하여 1930년 저서 자연선택의 유전학 이론에서 생물학자들이 측정한 연속적인 변화는 많은 이산 유전자의 결합 작용에 의해 생성될 수 있으며 자연선택은 모집단의 알레르 주파수를 변화시켜 진화를 초래할 수 있다는 것을 보여주었다.이온. 1924년에 시작된 일련의 논문에서 또 다른 영국의 유전학자 J. B. S. Haldane은 광범위한 조건 하에서 하나의 유전자 위치에서 알레 주파수 변화의 수학을 연구했다.할데인은 또 페퍼링나방 진화, 산업적 흑색증 등 자연선택의 실제 사례에 통계적 분석을 적용했으며, 선택계수가 피셔가 추정하는 것보다 클 수 있다는 것을 보여 공해증가에 따른 위장전략으로서 보다 신속한 적응적 진화를 이끌어냈다.[4][5]
동물 사육 실험의 배경을 가지고 있던 미국의 생물학자 Sewall Wright는 상호작용하는 유전자의 조합과 유전적 표류를 보이는 작고 상대적으로 고립된 개체군에 대한 교배 효과에 초점을 맞췄다.1932년 라이트는 적응형 경관의 개념을 도입했고 유전적 표류와 교배는 적응형 정점에서 작고 고립된 하위 집단을 멀어지게 할 수 있으며, 자연 선택이 그것을 다른 적응형 정점을 향해 몰고 갈 수 있다고 주장했다.[citation needed]
피셔, 할데인, 라이트의 연구는 인구유전학의 규율을 확립했다.이러한 자연 선택은 멘델의 유전학과 통합되었는데, 이것은 진화가 어떻게 작용하는지에 대한 통일된 이론을 개발하는 데 있어서 중요한 첫 단계였다.[4][5]존 메이너드 스미스는 할데인의 제자였고, W. D. 해밀턴은 피셔의 저술에 영향을 받았다.미국인 조지 R. 프라이스는 해밀턴과 메이너드 스미스 양쪽에서 일했다.미국인 리처드 레원틴과 일본인 기무라 모투는 라이트, 할다인의 영향을 받았다.[citation needed]
현대합성
인구유전학의 수학은 원래 현대 합성의 시초로 개발되었다.베티와[6] 같은 저자들은 인구유전학이 현대 합성의 핵심을 규정한다고 주장해왔다.20세기의 처음 몇 십 년 동안, 대부분의 현장 자연학자들은 라마르크주의와 정형외과가 그들이 살아 있는 세계에서 관찰한 복잡성에 대해 최고의 설명을 제공한다고 계속해서 믿었다.[7]현대 합성이 진행되는 동안 이러한 사상은 숙청되었고, 인구유전학의 수학적 틀에서 표현할 수 있는 진화적 원인만 남아 있었다.[8]어떤 진화적 요인이 진화에 영향을 미칠 수 있는지에 대해서는 의견이 일치했지만, 다양한 요인의 상대적 중요성에 대해서는 의견이 일치하지 않았다.[8]
테오도시우스 도브잔스키 T. H. 모건 연구소의 박사 후기 노동자는 세르게이 체트베리코프 등 러시아 유전학자들의 유전적 다양성에 대한 연구로 영향을 받아 왔다.그는 1937년 저서 '유전학'과 '종의 기원'을 통해 인구유전학자들이 개발한 미세진화의 기반과 현장 생물학자들이 관찰한 거시진화의 패턴 사이의 차이를 메우는데 일조했다.도브잔스키는 야생 개체군의 유전적 다양성을 조사하여 개체군 유전학자들의 가정과는 달리 이들 개체군의 유전적 다양성이 크고, 하위 개체군 간의 차이가 현저하다는 것을 보여주었다.이 책은 또한 인구유전학자들의 고도로 수학적인 연구결과를 받아들여 보다 접근하기 쉬운 형태로 만들었다.더 많은 생물학자들이 도브잔스키를 통해 인구유전학의 영향을 받았으며, 이는 원작의 고도로 수학적인 작품을 읽을 수 있었던 것보다 더 많은 생물학자들이 도브잔스키를 통해 인구유전학의 영향을 받았다.[9]
영국에서는 E. B. 생태유전학의 선구자인 포드는 1930년대와 1940년대 내내 인간 혈액형 등 유전적 다형성을 통해 유전적 다양성을 유지하는 능력 등 생태적 요인 때문에 선택력을 실증적으로 입증했다.[10]포드의 작품은 피셔와 협력하여 현대적 종합 과정에서 지배적인 힘으로서 자연선택을 향한 강조의 전환에 기여했다.[4][5][11][12]
중립 이론 및 원점 수정 역학
인구유전학의 독창적이고 현대적인 종합관점은 돌연변이가 풍부한 원료를 제공한다고 가정하고, 단지 모집단 내의 알레르기의 빈도 변화에만 초점을 맞춘다.[13]알레르 주파수에 영향을 미치는 주요 과정은 자연 선택, 유전적 변화, 유전자 흐름, 재발 돌연변이 등이다.피셔와 라이트는 선택과 표류라는 상대적 역할에 대해 근본적인 의견 불일치가 있었다.[14]모든 유전적 차이에 대한 분자 데이터의 가용성은 분자 진화의 중립적 이론으로 이어졌다.이러한 관점에서 많은 돌연변이는 유해하고 따라서 관찰되지 않으며, 나머지 대부분은 중립적이며, 즉 선택 대상이 아니다.각각의 중립 돌연변이의 운명이 우연한(유전적 표류)에 맡겨진 상태에서 진화적 변화의 방향은 돌연변이가 일어나는 것에 의해 추진되기 때문에 (기존) 대립의 빈도 변화 모델만으로는 포착할 수 없다.[13][15]
인구유전학의 원점 수정 관점은 엄밀히 중립적 돌연변이를 넘어 이 접근법을 일반화하고, 특정한 변화가 일어나는 속도를 돌연변이율과 고정 확률의 산물로 본다.[13]
네 가지 공정
선택
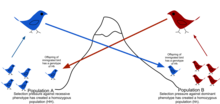
성적 선택을 포함하는 자연선택은 어떤 특성들이 유기체가 생존하고 번식할 가능성을 더 높여준다는 사실이다.인구유전학은 특정 환경에서 체력을 생존과 번식의 성향이나 확률로 정의함으로써 자연선택을 기술한다.일반적으로 적합성은 기호 w=1-s로 주어지며 여기서 s는 선택 계수다.자연선택은 표현형에 작용하기 때문에 모집단 유전모델은 표현형과 그에 따른 알레르기의 적합성을 한 개 또는 적은 수의 로키로 예측하기 위해 비교적 단순한 관계를 가정한다.이와 같이 자연선택은 다른 표현형을 가진 개개인의 건강상의 차이를 연속 세대에 걸쳐 모집단의 알레르빈도 변화로 전환시킨다.[citation needed]
인구유전학이 등장하기 전, 많은 생물학자들은 체력의 작은 차이가 진화에 큰 변화를 일으키기에 충분하다고 의심했다.[9]인구유전학자들은 선택을 유전적 표류와 비교함으로써 부분적으로 이러한 우려를 해결했다.s가 1보다 크면 선택으로 유전자 표류를 극복할 수 있다.이 기준을 충족하면 새로운 유리한 돌연변이가 고정될 확률은 대략 2초와 같다.[16][17]이러한 알레르기가 고정될 때까지 걸리는 시간은 유전적 이동에 거의 의존하지 않으며 대략 로그(sN)/s에 비례한다.[18]
우세
우위는 한 장소에서 하나의 알레르기의 표현형 및/또는 피트니스 효과가 두 번째 카피에 있는 그 알레르기에 따라 결정된다는 것을 의미한다.다음과 같은 피트니스[19] 값을 가진 세 가지 유전자형을 한 장소에서 고려하십시오.
| 유전자형: | A1A1 | A1A2 | A2A2 |
| 상대적 적합성: | 1 | 10분의 1 | 1-s |
| 인구유전 용어집 | |
|---|---|
s는 선택 계수, h는 우세 계수다.h 값은 다음과 같은 정보를 산출한다.
| h=0 | 지배적인1, 열성적인2 |
| h=1 | 지배적인2, 열성적인1 |
| 0[1] | 불완전한 우세 |
| h(0] | 과잉 재정 |
| h>1 | 저자본주의 |
인식론
경구란 한 위치에서 알레의 표현형 및/또는 피트니스 효과가 다른 위치에 어떤 알레르기가 존재하는가에 따라 달라진다는 것을 의미한다.선택은 하나의 중심에서 작용하는 것이 아니라 완전한 유전자형에서 발달을 통해 발생하는 표현형에서 작용한다.[20]그러나, 성종의 많은 인구유전학적 모델은 "단일한 위치" 모델이며, 여기서 개인의 적합성은 각 위치의 기여의 산물로 계산된다(효과적으로 인식하지 않는다고 가정함).
사실, 피트니스 풍경에 대한 유전자형은 더 복잡하다.인구유전학은 이 복잡성을 상세히 모형화하거나, 아니면 어떤 단순한 평균 법칙에 의해 그것을 포착해야 한다.경험적으로, 유익한 돌연변이는 이미 높은 건강상태를 가지고 있는 유전적 배경에 추가될 때 더 적은 건강상의 이점을 갖는 경향이 있는데, 이것은 이익 인식의 감소라고 알려져 있다.[21]유해한 돌연변이가 높은 체력적 배경에도 작은 체력적 영향을 미칠 때 이를 "합리적 인식"이라고 한다.그러나 유해한 돌연변이의 영향은 평균적으로 승수에 매우 가까운 경향이 있거나 심지어 "반동적 인식론"[22]으로 알려진 반대 패턴을 보일 수도 있다.
시너지 인식은 돌연변이의[23] 부하를 제거한다는 일부 이론과 성적 재생산의 진화에 중심적이다.
돌연변이
돌연변이는 새로운 알레르기의 형태로 유전적 변동의 궁극적인 근원이다.또한 돌연변이는 돌연변이 편향이 있을 때, 즉 다른 돌연변이가 발생할 가능성이 다를 때 진화 방향에 영향을 미칠 수 있다.예를 들어 선택과 반대 방향에 있는 경향이 있는 반복적인 돌연변이는 돌연변이-선택 균형으로 이어질 수 있다.분자 수준에서 G에서 A로 돌연변이가 A에서 G로 돌연변이보다 더 자주 일어나면 A를 가진 유전자형이 진화하는 경향이 있다.[24]서로 다른 세사에서 다른 삽입 대 삭제 돌연변이 편향은 다른 게놈 크기의 진화로 이어질 수 있다.[25][26]발달적 또는 돌연변이적 편견은 형태학적 진화에서도 관찰되었다.[27][28]예를 들어 표현형 우선 진화론에 따르면 돌연변이는 결국 환경에 의해 이전에 유도된 형질의 유전적 동화를 유발할 수 있다.[29][30]
돌연변이 바이어스 효과는 다른 과정에 중첩된다.만약 선택이 두 개의 돌연변이 중 한 개에게 유리하지만 둘 다 갖는 것에 추가적인 이점이 없다면, 가장 자주 발생하는 돌연변이는 한 모집단에서 고정될 가능성이 가장 높은 돌연변이다.[31][32]
돌연변이는 아무런 효과도 없고, 유전자의 산물을 바꾸거나, 유전자가 기능하는 것을 막을 수 있다.파리 드로필라 멜라노가스터에 대한 연구는 돌연변이가 유전자에 의해 생성된 단백질을 변화시키면, 이것은 아마도 해로울 것이며, 이러한 돌연변이의 약 70%가 손상 효과를 가지고 있고, 나머지는 중립적이거나 약하게 유익하다고 제안한다.[33]대부분의 기능 돌연변이는 다음에 대해 선택된다.그러나 선택이 약할 때, 기능 상실에 대한 돌연변이 편향은 진화에 영향을 미칠 수 있다.[34]예를 들어, 색소는 동물들이 동굴의 어둠 속에서 살 때 더 이상 유용하지 않고, 길을 잃기 쉽다.[35]이러한 종류의 기능 상실은 돌연변이 치우침으로 인해 발생할 수 있으며, 또는 그 기능에 원가가 있었기 때문에 발생할 수 있으며, 일단 기능의 효익이 사라지면 자연 선택이 손실을 초래한다.실험실 진화 중 박테리아에서 산발 능력 상실은 산발 능력 유지 비용에 대한 자연 선택이 아닌 돌연변이 편견에 의해 발생한 것으로 보인다.[36]기능 상실에 대한 선택이 없을 때 손실이 진화하는 속도는 유효 모집단 규모에 비해 돌연변이 속도에 더 의존하여 유전적 표류보다는 돌연변이 편향에 의해 더 많이 추진된다는 것을 나타낸다.[37]
돌연변이는 대개 유전적 재조합을 통해 DNA의 많은 부분이 복제되는 것을 수반할 수 있다.[38]이것은 모집단 내에서 복사 번호 변동을 초래한다.복제는 새로운 유전자를 진화시키는 주요 원료다.[39]다른 종류의 돌연변이는 때때로 이전에 부호화되지 않은 DNA로부터 새로운 유전자를 만들어낸다.[40][41]
유전적 표류
유전자 표류는 무작위 표본 추출에 의해 야기되는 알레르기의 빈도의 변화다.[42]즉, 자손의 알레르기는 부모의 알레르기의 무작위 표본이다.[43]유전적 표류는 유전자의 변형이 완전히 사라지게 하여 유전적 가변성을 감소시킬 수 있다.생식 성공에 따라 유전자 변형을 더 흔하게 하거나 덜 흔하게 만드는 자연적 선택과는 대조적으로,[44] 유전적 표류에 의한 변화는 환경적 압력이나 적응적 압력에 의해 추진되지 않으며, 마찬가지로 덜 흔하게 알레르기를 더 흔하게 만들 가능성이 있다.
유전적 표류의 영향은 알레르기가 많은 카피에 존재할 때보다 적은 카피에 존재하는 알레르기에 더 크다.유전적 표류의 인구유전학은 가지 과정이나 알레르기의 변화를 설명하는 확산방정식을 사용하여 설명된다.[45]이러한 접근방식은 대개 인구유전학의 라이트 피셔와 모란 모델에 적용된다.유전적 표류가 알레르기에 작용하는 유일한 진화력이라고 가정하면, 많은 복제 모집단에서 t세대를 거쳐 p와 q의 알레르기로 시작하여, 해당 모집단 전체에서 알레르 빈도의 분산이 된다.
로널드 피셔는 유전적 표류가 진화에서 가장 작은 역할을 한다는 견해를 가지고 있었고, 이것은 수십 년 동안 지배적인 견해로 남아 있었다.어떤 인구유전학적 관점에서도 유전적 표류 자체로 중심적 역할을 부여한 적은 없지만, 몇몇은 다른 비선택적 힘과 결합하여 유전적 표류를 중요하게 만들었다.세월 라이트의 시프트 밸런스 이론은 인구 구조와 유전적 표류의 결합이 중요하다고 주장했다.기무라 모투의 중립적인 분자 진화론은 개체군 내부와 개체군 사이의 대부분의 유전적 차이는 중성 돌연변이와 유전적 표류의 결합에 의해 발생한다고 주장한다.[47]
진화의 표본오차에 의한 유전자 표류의 역할은 존 길레스피와[48] 윌 프로빈에 의해 비판 받아왔다.[49] 그는 전통적으로 표본오차에 의해 유전적 표류에 기인하는 작업을 하면서 연계된 사이트에서 선택하는 것이 더 중요한 확률적 힘이라고 주장한다.유전자 초안의 수학적 성질은 유전적 표류와는 다르다.[50]알레르 빈도의 무작위 변화의 방향은 세대에 걸쳐 자동 상관관계가 있다.[42]
유전자 흐름
이동에 대한 물리적 장벽과 함께 개인이 이동하거나 확산하는 제한된 경향(취약성), 나탈 장소로 남거나 돌아오는 경향(철학) 때문에 자연 인구는 이론적 무작위 모델(판믹스)에서 가정할 수 있는 것처럼 모든 교배종을 거의 하지 않는다.[51]일반적으로 일반 인구에서 무작위로 선택된 사람들보다 개인이 서로 더 밀접하게 연관되어 있는 지리적 범위가 있다.이것은 한 집단이 유전적으로 구조되는 정도라고 설명된다.[52]
유전적 구조는 역사적 기후 변화, 종 범위 확장 또는 현재 서식지의 이용가능성에 의한 이동에 의해 발생할 수 있다.유전자의 흐름은 산맥, 바다, 사막이나 심지어 만리장성과 같은 인공 구조물에 의해 방해되어 식물 유전자의 흐름을 방해해 왔다.[53]
유전자 흐름은 개체군이나 종 사이의 유전자를 교환하는 것으로 구조를 무너뜨리는 것이다.한 종 내의 유전자 흐름의 예로는 유기체의 이동과 번식을 들 수 있으며, 꽃가루의 교환을 들 수 있다.종간 유전자 전이에는 잡종생물의 형성과 수평적 유전자 전이가 포함된다.인구유전모델은 어느 집단이 서로 유의한 유전적 고립을 보이는지를 식별하고 그들의 역사를 재구성하는 데 사용될 수 있다.[54]
인구에게 고립을 강요하는 것은 교배 우울증을 초래한다.인구로 이동하는 것은 새로운 유전적 변형을 도입할 수 있으며,[55] 잠재적으로 진화적 구조에 기여할 수 있다.개인이나 게이머의 상당한 비율이 이주하는 경우, 그것은 또한 이동 부하를 발생시키는 등 알레르 주파수를 변화시킬 수 있다.[56]
유전자의 흐름이 존재하는 상황에서 개체군이 새로운 종이 되기 위해서는 두 개체군 사이의 잡종화에 대한 다른 장벽이 필요하다.
수평유전자전달
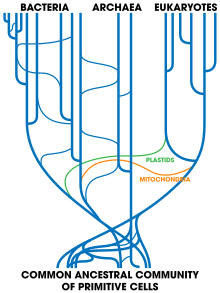
수평 유전자 전달은 유전 물질을 한 유기체에서 그 자손이 아닌 다른 유기체로 옮기는 것이다; 이것은 원핵생물들 사이에서 가장 흔하다.[57]의학에서 이것은 항생제 내성의 확산에 기여하는데, 한 박테리아가 내성 유전자를 획득할 때 다른 종으로 빠르게 옮겨갈 수 있기 때문이다.[58]박테리아에서 효모 사카로마이오스 세레비시아아, adzuki 콩 딱정벌레 Callosobruchs chinensis와 같은 진핵생물로 유전자가 수평적으로 전이되는 현상도 일어났을 수 있다.[59][60]더 큰 규모의 이송의 예로는 진핵생물인 벨로이드 로티페어가 있는데, 이 로티페어는 박테리아, 곰팡이, 식물로부터 다양한 유전자를 받은 것으로 보인다.[61]바이러스는 또한 유기체들 사이에 DNA를 운반할 수 있어 생물학적 영역을 넘나들어도 유전자가 전달될 수 있다.[62]엽록체와 미토콘드리아를 획득하는 동안 진핵세포와 원핵생물 사이의 대규모 유전자 전이도 일어났다.[63]
연결
모든 유전자가 연결 평형 상태에 있는 경우, 한 위치에서의 알레르기의 효과는 다른 위치의 유전자 풀 전체에서 평균화될 수 있다.실제로 한 개의 알레르기는 다른 로키에 있는 유전자와 불안정성을 연결하는데서 자주 발견되는데, 특히 같은 염색체 근처에 위치한 유전자와 관련이 있다.재조합은 유전적 히치하이킹을 피하기 위해 이 연결 고리를 너무 느리게 분해하는데, 이 연결 고리는 가까운 연결 고리에서 선택 중인 연결 고리와 연결되기 때문에 한 위치의 균열이 높은 빈도로 상승한다.연결은 또한 성적인 모집단에서도 적응 속도를 늦춘다.[64][65][66]적응 진화 속도를 늦추는 데 있어 연결 불균형의 영향은 힐-로버트슨 효과(유익한 돌연변이를 함께 가져오는 지연)와 배경 선택(유익한 돌연변이를 해로운 히치하이커와 분리하는 지연)의 조합에서 발생한다.
연계는 한 번에 하나의 유전자 위치를 처리하는 인구 유전자 모델의 문제다.그러나 선택적 스위프를 통해 자연선택의 작용을 탐지하는 방법으로 악용될 수 있다.
무성 인구의 극단적인 경우 연계는 완전하며, 단순한 피트니스 경관을 따라 유전형 주파수의 이동 파동 측면에서 모집단 유전자 방정식을 도출하고 해결할 수 있다.[67]박테리아와 같은 대부분의 미생물은 무성생식이다.그들의 적응에 대한 인구유전학은 두 가지 대조적인 체제를 가지고 있다.유익한 돌연변이율과 인구크기의 산물이 작을 때, 무성 개체군은 원산지 수정 역학의 "성공적 체제"를 따르며, 적응율은 이 제품에 크게 의존한다.제품이 훨씬 클 때, 무성 개체군은 제품에 덜 의존하는 "동류 돌연변이" 체제를 따르며, 마지막 돌연변이가 고정되기 전에 집단 간섭과 새로운 유익한 돌연변이의 출현을 특징으로 한다.
적용들
유전적 변동의 수준 설명
중성 이론은 모집단의 뉴클레오티드 다양성 수준이 모집단 크기와 중성 돌연변이 비율의 산물에 비례할 것이라고 예측한다.유전적 다양성의 수준이 인구 규모보다 훨씬 덜 다르다는 사실은 "변동의 파라독스"[68]로 알려져 있다.높은 수준의 유전적 다양성이 중립 이론에 찬성하는 원래의 주장 중 하나였지만, 변동의 역설은 중립 이론에 반대하는 가장 강력한 주장 중 하나이다.
유전적 다양성의 수준은 유전적 히치하이킹과 배경 선택 둘 다로 인해 지역적 재조합 비율의 함수로서 한 종 내에서 크게 다르다는 것은 분명하다.변동의 역설에 대한 가장 최근의 해결책은 링크된 사이트에서 어느 정도의 선택을 유발한다.[69]예를 들어, 한 분석은 더 큰 모집단이 더 많은 선택적 스위프를 가지고 있으며, 이것은 더 많은 중립적인 유전적 다양성을 제거한다.[70]돌연변이 비율과 모집단 크기 사이의 부정적인 상관관계도 기여할 수 있다.[71]
생명사는 인구사보다 유전적 다양성에 더 큰 영향을 미친다. 예를 들어 r전략가는 유전적 다양성을 더 많이 가지고 있다.[69]
선택 항목 탐지
인구유전학 모델은 어떤 유전자가 선택되고 있는지 유추하는 데 사용된다.한 가지 일반적인 접근법은 염색체를 따라 높은 연관성과 낮은 유전적 분산을 가진 지역을 찾아 최근의 선택적 스위프를 탐지하는 것이다.
두 번째 일반적인 접근법은 맥도날드-크리트만 시험으로, 두 가지 유형의 현장에서 한 종(폴리모르퍼시즘) 내 변동의 양과 종 간(대체)의 차이를 비교하는 것이다. 하나는 중립적이라고 가정한다.일반적으로 동의어 사이트는 중립적인 것으로 가정한다.[72]양성 선정이 진행 중인 유전자는 다형성 부지에 비해 서로 다른 부위가 과다하다.이 테스트는 또한 양성 선택인 α에 의해 고정된 대체물의 비율에 대한 게놈 전체 추정치를 얻는 데 사용될 수 있다.[73][74]분자 진화의 중립 이론에 따르면 이 숫자는 0에 가까워야 한다.따라서 높은 수치는 중립 이론의 게놈 폭의 위변조로 해석되어 왔다.[75]
인구추론
성적으로 재생성되고 디플로이드 종에서 모집단 구조에 대한 가장 간단한 테스트는 유전자형 주파수가 알레 주파수의 함수로 하디 와인버그 비율을 따르는지를 확인하는 것이다.예를 들어, 두 개의 알레르기가 A와 주파수 p와 q로 표시된 단일 위치의 가장 단순한 경우, 무작위 짝짓기는 AA 동족상체의 경우 freq(AA) = p2, aa 동족상자의 경우 freq(Aa) = q2, 그리고 이형상체의 경우 freq(Aa) = 2pq를 예측한다.인구구조가 없는 경우 하디-웨인버그 비율은 무작위 짝짓기 1~2세대 이내에서 도달한다.더 전형적으로, 인구 구조를 나타내는 동형질의 초과가 있다.이 초과분의 정도는 교배계수 F로 정량화할 수 있다.
개인은 K 하위집단으로 군집화될 수 있다.[76][77]그런 다음 인구구조의 정도는 인구구조에 의해 설명될 수 있는 유전적 분산의 비율에 대한 척도인 F를ST 이용하여 계산할 수 있다.그러면 유전적 인구구조가 지리적 구조와 관련될 수 있고, 유전적 혼화물이 검출될 수 있다.
결합 이론은 표본의 유전적 다양성과 그것이 채취된 인구의 인구학적 역사를 연관시킨다.일반적으로 중립성을 가정하기 때문에 게놈의 중성 진화하는 부분의 순서는 그러한 분석을 위해 선택된다.그것은 종 사이의 관계뿐만 아니라 종들 사이의 관계(생체유전학), 인구통계학적 역사(예: 인구 병목 현상, 인구 증가), 생물학적 분산, 원천-싱크 역학[78] 및 종 내부의 내향성을 유추하는 데 사용될 수 있다.
인구통계학적 추론에 대한 또 다른 접근방식은 알레르 주파수 스펙트럼에 의존한다.[79]
유전체계의 진화
유전 시스템 자체를 제어하는 로키가 있다고 가정함으로써, 지배와 다른 형태의 강건성의 진화, 성적 재생산과 재조합 비율의 진화, 돌연변이 비율의 진화, 진화 콘덴서의 진화, 값비싼 시그널린의 진화를 기술하는 인구 유전적 모델이 만들어진다.g 특성, 노화의 진화, 협력의 진화.예를 들어, 대부분의 돌연변이는 유해성이 있기 때문에 종에 대한 최적의 돌연변이 비율은 높은 유해 돌연변이의 피해와 DNA 수리 효소와 같이 돌연변이의 비율을 줄이기 위한 시스템 유지에 따른 대사 비용 사이의 절충일 수 있다.[80]
그러한 모델의 한 가지 중요한 측면은 선택 계수가 유효 모집단 크기의 역수보다 클 경우 유해한 돌연변이를 제거하기에 충분할 뿐이고 따라서 선택 계수가 열화에 대한 돌연변이 편향을 압도한다는 것이다.이것은 표류 장벽으로 알려져 있으며 거의 중립적인 분자 진화 이론과 관련이 있다.표류장벽 이론은 유효인구 크기가 큰 종은 매우 효율적이고 능률적인 유전자 시스템을 가질 것이며, 반면에 개체 크기가 작은 종은 인트론이나 전이 가능한 요소들을 포함하는 비대하고 복잡한 게놈을 가질 것이라고 예측한다.[81]그러나 다소 역설적으로 모집단 크기가 큰 종은 특정 유형의 오류의 결과에 매우 내성적이어서 작은 모집단보다 전사 및 번역 등에서 더 높은 오류율을 진화시킬 수 있다.[82]
참고 항목
참조
- ^ "Population genetics - Latest research and news". www.nature.com. Retrieved 2018-01-29.
- ^ Servedio, Maria R.; Brandvain, Yaniv; Dhole, Sumit; Fitzpatrick, Courtney L.; Goldberg, Emma E.; Stern, Caitlin A.; Van Cleve, Jeremy; Yeh, D. Justin (9 December 2014). "Not Just a Theory—The Utility of Mathematical Models in Evolutionary Biology". PLOS Biology. 12 (12): e1002017. doi:10.1371/journal.pbio.1002017. PMC 4260780. PMID 25489940.
- ^ Ewens, W.J. (2004). Mathematical Population Genetics (2nd ed.). New York: Springer. ISBN 978-0-387-20191-7.
- ^ a b c Bowler, Peter J. (2003). Evolution : the history of an idea (3rd ed.). Berkeley: University of California Press. pp. 325–339. ISBN 978-0-520-23693-6.
- ^ a b c Larson, Edward J. (2004). Evolution : the remarkable history of a scientific theory (Modern Library ed.). New York: Modern Library. pp. 221–243. ISBN 978-0-679-64288-6.
- ^ Beatty, John (1986). "The Synthesis and the Synthetic Theory". Integrating Scientific Disciplines. Science and Philosophy. Vol. 2. Springer Netherlands. pp. 125–135. doi:10.1007/978-94-010-9435-1_7. ISBN 9789024733422.
- ^ Mayr, Ernst; Provine, William B., eds. (1998). The Evolutionary synthesis : perspectives on the unification of biology ([New ed]. ed.). Cambridge, Massachusetts: Harvard University Press. pp. 295–298. ISBN 9780674272262.
- ^ a b Provine, W. B. (1988). "Progress in evolution and meaning in life". Evolutionary progress. University of Chicago Press. pp. 49–79.
- ^ a b Provine, William B. (1978). "The role of mathematical population geneticists in the evolutionary synthesis of the 1930s and 1940s". Studies of the History of Biology. 2: 167–192. PMID 11610409.
- ^ Ford, E. B. (1975) [1964]. Ecological genetics (4th ed.). London: Chapman and Hall. pp. 1ff.
- ^ Mayr, Ernst (1988). Toward a New Philosophy of Biology: Observations of an Evolutionist. Cambridge, Massachusetts: Belknap Press of Harvard University Press. p. 402. ISBN 978-0-674-89665-9.
- ^ Mayr, Ernst; Provine, William B., eds. (1998). The Evolutionary Synthesis : perspectives on the unification of biology ([New ed]. ed.). Cambridge, Massachusetts: Harvard University Press. pp. 338–341. ISBN 9780674272262.
- ^ a b c McCandlish, David M.; Stoltzfus, Arlin (September 2014). "Modeling Evolution Using the Probability of Fixation: History and Implications". The Quarterly Review of Biology. 89 (3): 225–252. doi:10.1086/677571. PMID 25195318. S2CID 19619966.
- ^ Crow, James F. (2010). "Wright and Fisher on Inbreeding and Random Drift". Genetics. 184 (3): 609–611. doi:10.1534/genetics.109.110023. ISSN 0016-6731. PMC 2845331. PMID 20332416.
- ^ Casillas, Sònia; Barbadilla, Antonio (2017). "Molecular Population Genetics". Genetics. 205 (3): 1003–1035. doi:10.1534/genetics.116.196493. PMC 5340319. PMID 28270526.
- ^ Haldane, J. B. S. (1927). "A Mathematical Theory of Natural and Artificial Selection, Part V: Selection and Mutation". Mathematical Proceedings of the Cambridge Philosophical Society. 23 (7): 838–844. Bibcode:1927PCPS...23..838H. doi:10.1017/S0305004100015644.
- ^ Orr, H. A. (2010). "The population genetics of beneficial mutations". Philosophical Transactions of the Royal Society B: Biological Sciences. 365 (1544): 1195–1201. doi:10.1098/rstb.2009.0282. PMC 2871816. PMID 20308094.
- ^ Hermisson, J.; Pennings, P. S. (2005). "Soft sweeps: molecular population genetics of adaptation from standing genetic variation". Genetics. 169 (4): 2335–2352. doi:10.1534/genetics.104.036947. PMC 1449620. PMID 15716498.
- ^ Gillespie, John (2004). Population Genetics: A Concise Guide (2nd ed.). Johns Hopkins University Press. ISBN 978-0-8018-8008-7.
- ^ Miko, I. (2008). "Epistasis: Gene interaction and phenotype effects". Nature Education. 1 (1): 197.
- ^ Berger, D.; Postma, E. (13 October 2014). "Biased Estimates of Diminishing-Returns Epistasis? Empirical Evidence Revisited". Genetics. 198 (4): 1417–1420. doi:10.1534/genetics.114.169870. PMC 4256761. PMID 25313131.
- ^ Kouyos, Roger D.; Silander, Olin K.; Bonhoeffer, Sebastian (June 2007). "Epistasis between deleterious mutations and the evolution of recombination". Trends in Ecology & Evolution. 22 (6): 308–315. doi:10.1016/j.tree.2007.02.014. PMID 17337087.
- ^ Crow, J. F. (5 August 1997). "The high spontaneous mutation rate: is it a health risk?". Proceedings of the National Academy of Sciences of the United States of America. 94 (16): 8380–8386. Bibcode:1997PNAS...94.8380C. doi:10.1073/pnas.94.16.8380. PMC 33757. PMID 9237985.
- ^ Smith, N. G. C.; Webster, M. T.; Ellegren, H. (2002). "Deterministic Mutation Rate Variation in the Human Genome". Genome Research. 12 (9): 1350–1356. doi:10.1101/gr.220502. PMC 186654. PMID 12213772.
- ^ Petrov, D. A. (2000). "Evidence for DNA Loss as a Determinant of Genome Size". Science. 287 (5455): 1060–1062. Bibcode:2000Sci...287.1060P. doi:10.1126/science.287.5455.1060. ISSN 0036-8075. PMID 10669421.
- ^ Petrov, D. A. (2002). "DNA loss and evolution of genome size in Drosophila". Genetica. 115 (1): 81–91. doi:10.1023/A:1016076215168. PMID 12188050. S2CID 5314242.
- ^ Kiontke, K.; Barrière, A.; Kolotuev, I.; Podbilewicz, B.; Sommer, R.; Fitch, D. H. A.; Félix, M. A. (2007). "Trends, stasis, and drift in the evolution of nematode vulva development". Current Biology. 17 (22): 1925–1937. doi:10.1016/j.cub.2007.10.061. PMID 18024125. S2CID 4503181.
- ^ Braendle, C.; Baer, C. F.; Félix, M. A. (2010). Barsh, Gregory S. (ed.). "Bias and Evolution of the Mutationally Accessible Phenotypic Space in a Developmental System". PLOS Genetics. 6 (3): e1000877. doi:10.1371/journal.pgen.1000877. PMC 2837400. PMID 20300655.
- ^ Palmer, RA (2004). "Symmetry breaking and the evolution of development". Science. 306 (5697): 828–833. Bibcode:2004Sci...306..828P. CiteSeerX 10.1.1.631.4256. doi:10.1126/science.1103707. PMID 15514148. S2CID 32054147.
- ^ West-Eberhard, M-J. (2003). Developmental plasticity and evolution. New York: Oxford University Press. ISBN 978-0-19-512235-0.
- ^ Stoltzfus, A.; Yampolsky, L. Y. (2009). "Climbing Mount Probable: Mutation as a Cause of Nonrandomness in Evolution". Journal of Heredity. 100 (5): 637–647. doi:10.1093/jhered/esp048. PMID 19625453.
- ^ Yampolsky, L. Y.; Stoltzfus, A. (2001). "Bias in the introduction of variation as an orienting factor in evolution". Evol Dev. 3 (2): 73–83. doi:10.1046/j.1525-142x.2001.003002073.x. PMID 11341676. S2CID 26956345.
- ^ Sawyer, S. A.; Parsch, J.; Zhang, Z.; Hartl, D. L. (2007). "Prevalence of positive selection among nearly neutral amino acid replacements in Drosophila". Proceedings of the National Academy of Sciences. 104 (16): 6504–6510. doi:10.1073/pnas.0701572104. ISSN 0027-8424. PMC 1871816. PMID 17409186.
- ^ Haldane, J. B. S. (1933). "The Part Played by Recurrent Mutation in Evolution". American Naturalist. 67 (708): 5–19. doi:10.1086/280465. JSTOR 2457127. S2CID 84059440.
- ^ Protas, Meredith; Conrad, M.; Gross, J. B.; Tabin, C.; Borowsky, R (2007). "Regressive evolution in the Mexican cave tetra, Astyanax mexicanus". Current Biology. 17 (5): 452–454. doi:10.1016/j.cub.2007.01.051. PMC 2570642. PMID 17306543.
- ^ Maughan, H.; Masel, J.; Birky, W. C.; Nicholson, W. L. (2007). "The roles of mutation accumulation and selection in loss of sporulation in experimental populations of Bacillus subtilis". Genetics. 177 (2): 937–948. doi:10.1534/genetics.107.075663. PMC 2034656. PMID 17720926.
- ^ Masel, J.; King, O. D.; Maughan, H. (2007). "The loss of adaptive plasticity during long periods of environmental stasis". American Naturalist. 169 (1): 38–46. doi:10.1086/510212. PMC 1766558. PMID 17206583.
- ^ Hastings, P. J.; Lupski, J. R.; Rosenberg, S. M.; Ira, G. (2009). "Mechanisms of change in gene copy number". Nature Reviews Genetics. 10 (8): 551–564. doi:10.1038/nrg2593. PMC 2864001. PMID 19597530.
- ^ M., Long; Betrán, E.; Thornton, K.; Wang, W. (November 2003). "The origin of new genes: glimpses from the young and old". Nat. Rev. Genet. 4 (11): 865–75. doi:10.1038/nrg1204. PMID 14634634. S2CID 33999892.
- ^ Liu, N.; Okamura, K.; Tyler, D. M.; Phillips; Chung; Lai (2008). "The evolution and functional diversification of animal microRNA genes". Cell Research. 18 (10): 985–996. doi:10.1038/cr.2008.278. PMC 2712117. PMID 18711447.
- ^ McLysaght, Aoife; Hurst, Laurence D. (25 July 2016). "Open questions in the study of de novo genes: what, how and why". Nature Reviews Genetics. 17 (9): 567–578. doi:10.1038/nrg.2016.78. PMID 27452112. S2CID 6033249.
- ^ a b Masel, J. (2011). "Genetic drift". Current Biology. 21 (20): R837–R838. doi:10.1016/j.cub.2011.08.007. PMID 22032182.
- ^ Futuyma, Douglas (1998). Evolutionary Biology. Sinauer Associates. p. Glossary. ISBN 978-0-87893-189-7.
- ^ Avers, Charlotte (1989). Process and Pattern in Evolution. Oxford University Press.
- ^ Wahl, L. M. (2011). "Fixation when N and s Vary: Classic Approaches Give Elegant New Results". Genetics. 188 (4): 783–785. doi:10.1534/genetics.111.131748. PMC 3176088. PMID 21828279.
- ^ Barton, Nicholas H.; Briggs, Derek E. G.; Eisen, Jonathan A.; Goldstein, David B.; Patel, Nipam H. (2007). Evolution. Cold Spring Harbor Laboratory Press. p. 417. ISBN 978-0-87969-684-9.
- ^ Futuyma, Douglas (1998). Evolutionary Biology. Sinauer Associates. p. 320. ISBN 978-0-87893-189-7.
- ^ Gillespie, J. H. (2000). "Genetic Drift in an Infinite Population: The Pseudohitchhiking Model". Genetics. 155 (2): 909–919. doi:10.1093/genetics/155.2.909. PMC 1461093. PMID 10835409.
- ^ Provine, William B. The "Random Genetic Drift" Fallacy. CreateSpace.
- ^ Neher, Richard A.; Shraiman, Boris I. (August 2011). "Genetic Draft and Quasi-Neutrality in Large Facultatively Sexual Populations". Genetics. 188 (4): 975–996. arXiv:1108.1635. doi:10.1534/genetics.111.128876. ISSN 0016-6731. PMC 3176096. PMID 21625002.
- ^ Buston, P. M.; Pilkington, J. G.; et al. (2007). "Are clownfish groups composed of close relatives? An analysis of microsatellite DNA vraiation in Amphiprion percula". Molecular Ecology. 12 (3): 733–742. doi:10.1046/j.1365-294X.2003.01762.x. PMID 12675828. S2CID 35546810.
- ^ Repaci, V.; Stow, A. J.; Briscoe, D. A. (2007). "Fine-scale genetic structure, co-founding and multiple mating in the Australian allodapine bee (Ramphocinclus brachyurus)". Journal of Zoology. 270 (4): 687–691. doi:10.1111/j.1469-7998.2006.00191.x.
- ^ a b Su, H.; Qu, L.-J.; He, K.; Zhang, Z.; Wang, J; Chen, Z.; Gu, H. (2003). "The Great Wall of China: a physical barrier to gene flow?". Heredity. 90 (3): 212–219. doi:10.1038/sj.hdy.6800237. ISSN 0018-067X. PMID 12634804. S2CID 13367320.
- ^ Gravel, S. (2012). "Population Genetics Models of Local Ancestry". Genetics. 1202 (2): 607–619. arXiv:1202.4811. Bibcode:2012arXiv1202.4811G. doi:10.1534/genetics.112.139808. PMC 3374321. PMID 22491189.
- ^ Morjan, C.; Rieseberg, L. (2004). "How species evolve collectively: implications of gene flow and selection for the spread of advantageous alleles". Molecular Ecology. 13 (6): 1341–56. doi:10.1111/j.1365-294X.2004.02164.x. PMC 2600545. PMID 15140081.
- ^ Bolnick, Daniel I.; Nosil, Patrik (September 2007). "Natural Selection in Populations Subject to a Migration Load". Evolution. 61 (9): 2229–2243. doi:10.1111/j.1558-5646.2007.00179.x. PMID 17767592. S2CID 25685919.
- ^ Boucher, Yan; Douady, Christophe J.; Papke, R. Thane; Walsh, David A.; Boudreau, Mary Ellen R.; Nesbø, Camilla L.; Case, Rebecca J.; Doolittle, W. Ford (2003). "Lateral Gene Transfer and the Origins of Prokaryotic Groups". Annual Review of Genetics. 37 (1): 283–328. doi:10.1146/annurev.genet.37.050503.084247. ISSN 0066-4197. PMID 14616063.
- ^ Walsh, T. (2006). "Combinatorial genetic evolution of multiresistance". Current Opinion in Microbiology. 9 (5): 476–82. doi:10.1016/j.mib.2006.08.009. PMID 16942901.
- ^ Kondo, N.; Nikoh, N.; Ijichi, N.; Shimada, M.; Fukatsu, T. (2002). "Genome fragment of Wolbachia endosymbiont transferred to X chromosome of host insect". Proceedings of the National Academy of Sciences. 99 (22): 14280–14285. Bibcode:2002PNAS...9914280K. doi:10.1073/pnas.222228199. ISSN 0027-8424. PMC 137875. PMID 12386340.
- ^ Sprague, G. (1991). "Genetic exchange between kingdoms". Current Opinion in Genetics & Development. 1 (4): 530–533. doi:10.1016/S0959-437X(05)80203-5. PMID 1822285.
- ^ Gladyshev, E. A.; Meselson, M.; Arkhipova, I. R. (2008). "Massive Horizontal Gene Transfer in Bdelloid Rotifers". Science. 320 (5880): 1210–1213. Bibcode:2008Sci...320.1210G. doi:10.1126/science.1156407. ISSN 0036-8075. PMID 18511688. S2CID 11862013.
- ^ Baldo, A.; McClure, M. (1 September 1999). "Evolution and horizontal transfer of dUTPase-encoding genes in viruses and their hosts". Journal of Virology. 73 (9): 7710–7721. doi:10.1128/JVI.73.9.7710-7721.1999. PMC 104298. PMID 10438861.
- ^ Poole, A.; Penny, D. (2007). "Evaluating hypotheses for the origin of eukaryotes". BioEssays. 29 (1): 74–84. doi:10.1002/bies.20516. PMID 17187354.
- ^ Weissman, D. B.; Hallatschek, O. (15 January 2014). "The Rate of Adaptation in Large Sexual Populations with Linear Chromosomes". Genetics. 196 (4): 1167–1183. doi:10.1534/genetics.113.160705. PMC 3982688. PMID 24429280.
- ^ Weissman, Daniel B.; Barton, Nicholas H.; McVean, Gil (7 June 2012). "Limits to the Rate of Adaptive Substitution in Sexual Populations". PLOS Genetics. 8 (6): e1002740. doi:10.1371/journal.pgen.1002740. PMC 3369949. PMID 22685419.
- ^ Neher, R. A.; Shraiman, B. I.; Fisher, D. S. (30 November 2009). "Rate of Adaptation in Large Sexual Populations". Genetics. 184 (2): 467–481. arXiv:1108.3464. doi:10.1534/genetics.109.109009. PMC 2828726. PMID 19948891.
- ^ Desai, Michael M.; Fisher, Daniel S. (2007). "Beneficial Mutation Selection Balance and the Effect of Linkage on Positive Selection". Genetics. 176 (3): 1759–1798. doi:10.1534/genetics.106.067678. PMC 1931526. PMID 17483432.
- ^ Lewontin, R. C. (1973). The genetic basis of evolutionary change ([4th printing.] ed.). New York: Columbia University Press. ISBN 978-0231033923.
- ^ a b Ellegren, Hans; Galtier, Nicolas (6 June 2016). "Determinants of genetic diversity". Nature Reviews Genetics. 17 (7): 422–433. doi:10.1038/nrg.2016.58. PMID 27265362. S2CID 23531428.
- ^ Corbett-Detig, Russell B.; Hartl, Daniel L.; Sackton, Timothy B.; Barton, Nick H. (10 April 2015). "Natural Selection Constrains Neutral Diversity across A Wide Range of Species". PLOS Biology. 13 (4): e1002112. doi:10.1371/journal.pbio.1002112. PMC 4393120. PMID 25859758.
- ^ Sung, W.; Ackerman, M. S.; Miller, S. F.; Doak, T. G.; Lynch, M. (17 October 2012). "Drift-barrier hypothesis and mutation-rate evolution" (PDF). Proceedings of the National Academy of Sciences. 109 (45): 18488–18492. Bibcode:2012PNAS..10918488S. doi:10.1073/pnas.1216223109. PMC 3494944. PMID 23077252.
- ^ Charlesworth, J. Eyre-Walker (2008). "The McDonald–Kreitman Test and Slightly Deleterious Mutations". Molecular Biology and Evolution. 25 (6): 1007–1015. doi:10.1093/molbev/msn005. PMID 18195052.
- ^ Eyre-Walker, A. (2006). "The genomic rate of adaptive evolution" (PDF). Trends in Ecology and Evolution. 21 (10): 569–575. doi:10.1016/j.tree.2006.06.015. PMID 16820244.
- ^ Smith, N. G. C.; Eyre-Walker, A. (2002). "Adaptive protein evolution in Drosophila". Nature. 415 (6875): 1022–1024. Bibcode:2002Natur.415.1022S. doi:10.1038/4151022a. PMID 11875568. S2CID 4426258.
- ^ Hahn, M. W. (2008). "Toward a selection theory of molecular evolution". Evolution. 62 (2): 255–265. doi:10.1111/j.1558-5646.2007.00308.x. PMID 18302709. S2CID 5986211.
- ^ Pritchard, J. K.; Stephens, M.; Donnelly, P. (June 2000). "Inference of population structure using multilocus genotype data". Genetics. 155 (2): 945–959. doi:10.1093/genetics/155.2.945. ISSN 0016-6731. PMC 1461096. PMID 10835412.
- ^ Verity, Robert; Nichols, Richard A. (August 2016). "Estimating the Number of Subpopulations (K) in Structured Populations". Genetics. 203 (4): 1827–1839. doi:10.1534/genetics.115.180992. ISSN 0016-6731. PMC 4981280. PMID 27317680.
- ^ Manlik, Oliver; Chabanne, Delphine; Daniel, Claire; Bejder, Lars; Allen, Simon J.; Sherwin, William B. (13 November 2018). "Demography and genetics suggest reversal of dolphin source–sink dynamics, with implications for conservation". Marine Mammal Science. 35 (3): 732–759. doi:10.1111/mms.12555.
- ^ Gutenkunst, Ryan N.; Hernandez, Ryan D.; Williamson, Scott H.; Bustamante, Carlos D.; McVean, Gil (23 October 2009). "Inferring the Joint Demographic History of Multiple Populations from Multidimensional SNP Frequency Data". PLOS Genetics. 5 (10): e1000695. arXiv:0909.0925. doi:10.1371/journal.pgen.1000695. PMC 2760211. PMID 19851460.
- ^ Sniegowski, P. (2000). Gerrish P.; Johnson T;. Shaver A. "The evolution of mutation rates: separating causes from consequences". BioEssays. 22 (12): 1057–1066. doi:10.1002/1521-1878(200012)22:12<1057::AID-BIES3>3.0.CO;2-W. PMID 11084621.
- ^ Lynch, Michael; Conery, John S. (2003). "The origins of genome complexity". Science. 302 (5649): 1401–1404. Bibcode:2003Sci...302.1401L. CiteSeerX 10.1.1.135.974. doi:10.1126/science.1089370. PMID 14631042. S2CID 11246091.
- ^ Rajon, E.; Masel, J. (3 January 2011). "Evolution of molecular error rates and the consequences for evolvability". Proceedings of the National Academy of Sciences. 108 (3): 1082–1087. Bibcode:2011PNAS..108.1082R. doi:10.1073/pnas.1012918108. PMC 3024668. PMID 21199946.
외부 링크
| 위키미디어 커먼즈에는 인구유전학과 관련된 미디어가 있다. |
- 인구유전학 자습서
- 분자인구유전학
- 예일대학교의 ALlele FREQUency Database
- EHSTRAFD.org - Earth Human STR Alle Frequences 데이터베이스
- 인구유전학의 역사
- 선택으로 인구의 유전적 구성이 어떻게 변화하는지, 스티븐 C의 강의 영상. 스턴스 (예일 대학교)
- 내셔널 지오그래픽:인간 여정의 지도 (Happlogroup 기반 인간 이주 지도)