돌연변이
Mutation| 시리즈의 일부 |
| 유전학 |
|---|
 |
| 시리즈의 일부 |
| 진화생물학 |
|---|
 |


생물학에서 돌연변이는 생물, 바이러스 또는 염색체 외 [1]DNA의 게놈의 핵산 배열의 변화이다.바이러스 게놈. 이러한 오류의 DNA나 바이러스 복제, 체세포 분열, 또는 감수 분열이나 DNA( 같은 피리미딘 dimers에 의해 노출되어 자외선 방사선)에 미치는 피해, 그(특히 microhomology-mediated 끝에 가입하는)[2] 다른 시간에 오류를 유발하는 오류 유발 수선을 거칠 수 있는 다른 종류의 결과 DNA또는 RNA을 포함하고 있다. 항의라도rms의 복구 [3][4]또는 복제 중 오류 발생(변환 합성)돌연변이는 또한 이동성 유전 [5][6][7]요소 때문에 DNA 세그먼트의 삽입 또는 삭제로 인해 발생할 수 있다.
돌연변이는 유기체의 관찰 가능한 특성(페노타입)에 감지 가능한 변화를 일으킬 수도 있고 그렇지 않을 수도 있다.돌연변이는 진화, 암, 그리고 접합 다양성을 포함한 면역체계의 발달과 같은 정상적이고 비정상적인 생물학적 과정 모두에서 역할을 한다.돌연변이는 모든 유전자 변이의 궁극적인 원천이며, 자연 선택과 같은 진화적 힘이 작용할 수 있는 원료를 제공한다.
돌연변이는 시퀀스에 다양한 유형의 변화를 일으킬 수 있습니다.유전자의 돌연변이는 아무런 영향도 미치지 못하고, 유전자의 생성물을 바꾸거나, 유전자가 제대로 또는 완전히 기능하는 것을 방해할 수 있다.유전자가 아닌 영역에서도 돌연변이가 발생할 수 있습니다.2007년 드로소필라의 다른 종들 간의 유전자 변이에 대한 연구는 돌연변이가 유전자에 의해 생성된 단백질을 변화시킨다면, 그 결과는 유해할 가능성이 있으며, 해로운 영향을 미치는 아미노산 다형성의 약 70%가 있고, 나머지는 중립적이거나 약간 [8]이롭다고 제안했다.돌연변이가 유전자에 미칠 수 있는 해로운 영향 때문에, 유기체는 돌연변이의 염기서열을 원래 [5]상태로 되돌림으로써 돌연변이를 방지하거나 교정하는 DNA 복구와 같은 메커니즘을 가지고 있다.
개요
돌연변이는 보통 유전자 [9]재조합을 통해 DNA의 큰 부분의 복제를 포함할 수 있습니다.이러한 복제는 진화하는 새로운 유전자의 주요 원료이며,[10] 수백만 년마다 동물 게놈에서 수십에서 수백 개의 유전자가 복제됩니다.대부분의 유전자는 같은 조상의 더 큰 유전자군에 속하며, 배열 호몰로지에 [11]의해 검출된다.새로운 유전자는 보통 조상 유전자의 복제와 돌연변이를 통해, 또는 새로운 [12][13]기능을 가진 새로운 조합을 형성하기 위해 다른 유전자의 일부를 재조합함으로써 여러 가지 방법으로 생산된다.
여기서 단백질 도메인은 모듈로서 작용하며, 각각은 특수하고 독립적인 기능을 가지고 있으며, 함께 혼합되어 새로운 특성을 [14]가진 새로운 단백질을 코드하는 유전자를 생산할 수 있다.예를 들어, 인간의 눈은 빛을 감지하는 구조를 만들기 위해 네 개의 유전자를 사용합니다: 세 개는 원추 세포나 색각을 위한 것이고 한 개는 막대 세포나 야각을 위한 것이고; 네 개는 모두 하나의 조상 [15]유전자에서 유래했습니다.유전자 복제의 또 다른 장점은 이것이 공학적 중복성을 증가시킨다는 것이다; 이것은 쌍의 한 유전자가 새로운 기능을 얻는 반면 다른 유전자는 원래의 기능을 [16][17]수행할 수 있게 한다.다른 종류의 돌연변이는 때때로 이전에 코드화되지 않은 [18][19]DNA로부터 새로운 유전자를 만들어낸다.
염색체 수의 변화는 염색체 내의 DNA 조각이 부서지고 다시 정렬되는 훨씬 더 큰 돌연변이를 포함할 수 있다.예를 들어, Homininae에서는 두 개의 염색체가 융합되어 인간 염색체 2가 생성되었다; 이 융합은 다른 유인원의 혈통에서 일어나지 않았고, 그들은 이러한 분리된 [20]염색체를 유지한다.진화에서, 그러한 염색체 재배열의 가장 중요한 역할은 개체군을 교배할 가능성이 낮아지게 함으로써 개체군의 새로운 종으로의 분리를 가속화하고,[21] 따라서 이들 개체군 사이의 유전적 차이를 보존하는 것일 수 있다.
트랜스포존과 같이 게놈 주위를 이동할 수 있는 DNA의 배열은 식물과 동물의 유전 물질의 주요 부분을 구성하며,[22] 게놈의 진화에 중요했을지도 모른다.예를 들어, 100만 개 이상의 알루 배열이 인간 게놈에 존재하며, 이러한 배열은 유전자 [23]발현 조절과 같은 기능을 수행하기 위해 모집되었다.이 이동 DNA 배열의 또 다른 효과는 게놈 내에서 움직일 때 기존 유전자를 돌연변이를 일으키거나 삭제하여 유전적 [6]다양성을 만들어 낼 수 있다는 것이다.
비살상 돌연변이는 유전자 풀에 축적되어 유전자 [24]변이의 양을 증가시킨다.유전자 풀 내의 일부 유전자 변화의 풍부함은 자연 선택에 의해 감소될 수 있는 반면, 다른 "더 유리한" 돌연변이는 축적되어 적응적인 변화를 초래할 수 있다.
예를 들어, 나비는 새로운 돌연변이를 가진 자손을 낳을 수 있다.이러한 돌연변이의 대부분은 아무런 영향을 미치지 않을 것이다; 하지만 어떤 돌연변이는 나비의 자손들 중 한 명의 색을 바꾸어서 포식자들이 더 보기 어렵게 할 수도 있다.만약 이 색의 변화가 유리하다면, 이 나비가 살아남아 자신의 새끼를 낳을 가능성은 조금 더 높아지며, 시간이 지나면서 이 돌연변이를 가진 나비의 수가 개체수의 더 많은 비율을 형성할 수 있을 것이다.
중성 돌연변이는 효과가 개인의 적합성에 영향을 미치지 않는 돌연변이로 정의된다.이것들은 유전적 표류로 인해 시간이 지남에 따라 빈도가 증가할 수 있다.돌연변이의 압도적 다수가 유기체의 [25][26]적합성에 큰 영향을 미치지 않는다고 믿어진다.또한, DNA 복구 메커니즘은 영구 돌연변이가 되기 전에 대부분의 변화를 복구할 수 있으며, 많은 유기체는 영구히 변이된 체세포를 제거하는 메커니즘을 가지고 있다.
유익한 돌연변이는 생식 [27][28]성공을 향상시킬 수 있다.
원인들
돌연변이의 4가지 클래스는 (1) 자연발생적인 돌연변이(분자붕괴), (2) 자연발생적인 DNA 손상의 복제 바이패스에 의한 돌연변이(일명 오류 발생성 전이 합성), (3) DNA 복구 중에 도입된 오류 및 (4) 돌연변이 유발이다.과학자들은 또한 과학 실험을 위해 DNA 조작을 통해 의도적으로 돌연변이 염기서열을 도입할 수도 있다.
2017년의 한 연구는 암을 유발하는 돌연변이의 66%가 무작위이며, 29%는 환경 때문이며(69개국에 걸쳐 연구된 모집단), [29]5%는 유전된다고 주장했다.
인간은 평균 60개의 새로운 돌연변이를 자녀에게 전달하지만, 아버지는 나이에 따라 더 많은 돌연변이를 전달하고, 매년 두 개의 새로운 돌연변이를 자녀에게 [30]추가한다.
자연 돌연변이
자연 돌연변이는 오염되지 않은 건강한 세포에서도 0이 아닌 확률로 발생합니다.자연적으로 발생하는 산화 DNA 손상은 인간의 경우 세포당 하루에 10,000회, [31]쥐의 경우 세포당 하루에 100,000회 발생하는 것으로 추정됩니다.자발적 돌연변이는 특정 변화에 [32]의해 특징지을 수 있습니다.
- 호변이성 – 염기는 수소 원자의 위치에 따라 변화하며, 그 염기의 수소 결합 패턴을 변화시켜 [33]복제 중에 잘못된 염기 쌍을 일으킨다.이론적 결과에 따르면 양성자 터널링은 GC [34]호변이성체의 자발적 생성에 중요한 요소이다.
- 탈수 – 푸린 염기(A 또는 G)가 손실되어 아푸린성 부위(AP 부위)가 형성됩니다.
- 탈아미네이션 – 가수분해는 정상 염기를 원래 아민기 대신 케토기를 포함하는 비정형 염기로 변화시킨다.예를 들어 C → U 및 A → HX(하이포크산틴), 5Mec(5-메틸시토신) → T는 티민이 정상적인 DNA 염기이기 때문에 돌연변이로 검출될 가능성이 낮다.
- Slip strand mispairing – 복제 중에 템플릿에서 새 스트랜드가 변성된 후 다른 스폿에서 재변성("슬립")됩니다.이로 인해 삽입 또는 삭제가 발생할 수 있습니다.
오류가 발생하기 쉬운 복제 바이패스
자발적으로 발생하는 돌연변이의 대부분이 템플릿 가닥의 과거 DNA 손상으로 인한 오류 발생 가능성이 높은 복제(변환 합성) 때문이라는 증거가 증가하고 있다.생쥐에서 돌연변이의 대부분은 전이 [35]합성에 의해 발생한다.마찬가지로 효모에서 Kunz [36]등은 자발적인 단일 염기쌍 치환 및 결실의 60% 이상이 전이 합성에 의해 발생한다는 것을 발견했다.
DNA 복구 중 발생한 오류
자연발생적인 이중가닥 절단은 DNA에서 비교적 낮은 빈도로 발생하지만, 이들의 복구는 종종 돌연변이를 일으킨다.Non-Homologous End Joining(NHEJ; 비호몰로지 엔드 조인)은 이중 스트랜드 절단을 복구하기 위한 주요 경로입니다.NHEJ는 재결합을 위한 양 끝의 다소 부정확한 정렬을 가능하게 하기 위해 몇 가지 뉴클레오티드를 제거한 후 간격을 메우기 위해 뉴클레오티드를 추가하는 것을 포함한다.그 결과 NHEJ는 [37]돌연변이를 일으키는 경우가 많다.
유도 돌연변이
유도 돌연변이는 돌연변이 및 환경적 원인과 접촉한 후 유전자의 변화이다.
분자 수준의 유도 돌연변이는 다음과 같이 발생할 수 있습니다.
- 화학 물질들
- 히드록실아민
- 염기 아날로그(예: 브로모데옥시우리딘(BrdU))
- 알킬화제(예를 들어 N-에틸-N-니트로소요소(ENU)).이 약물은 복제 DNA와 비복제 DNA를 모두 변형시킬 수 있다.이와는 대조적으로 염기성 아날로그는 DNA 복제에 아날로그가 포함되어 있을 때만 DNA를 돌연변이시킬 수 있다.이러한 화학 돌연변이의 각 등급은 전환, 변환 또는 결실을 초래하는 특정 효과를 가지고 있습니다.
- DNA 부가물을 형성하는 약제(예: 오크라톡신 A)[39]
- DNA간격제(예를 들어 브롬화 에티듐)
- DNA 가교제
- 산화적 손상
- 아질산은 A와 C의 아민기를 디아조기로 전환시켜 수소 결합 패턴을 변화시키고, 이는 복제 시 잘못된 염기쌍을 초래한다.
- ★★★★
과거에는 돌연변이가 우연히 발생하거나 돌연변이에 의해 유발된다고 가정한 반면, 돌연변이의 분자 메커니즘은 박테리아와 생명의 나무에서 발견되었다.S. Rosenberg가 언급했듯이, "이러한 메커니즘은 고도로 조절된 돌연변이 유발의 그림을 보여준다. 스트레스 반응에 의해 일시적으로 상향 조절되고 세포/기구가 환경에 부적응될 때 활성화된다(스트레스가 있을 때 잠재적으로 [41]적응을 가속화한다).유기체의 적응률을 높이는 자기유도형 돌연변이 발생 메커니즘이기 때문에 적응형 돌연변이 발생 메커니즘으로 명명되기도 하며 [42]박테리아에서의 SOS 반응, 이소성 염색체 내 재조합[43] 및 [41]복제와 같은 다른 염색체 이벤트를 포함한다.
종류 구분
구조에 의한 영향
유전자의 배열은 여러 [45]가지 방법으로 바뀔 수 있다.유전자 돌연변이는 어디에서 발생하는지, 그리고 그것이 필수 단백질의 기능을 변화시키는지에 따라 건강에 다양한 영향을 미친다.유전자 구조의 돌연변이는 몇 가지 유형으로 분류할 수 있다.
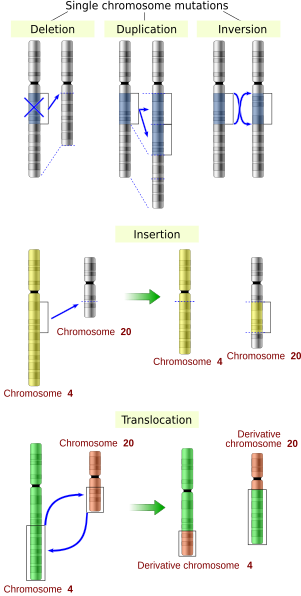
대규모 돌연변이
염색체 구조의 대규모 돌연변이는 다음을 포함한다.
- 염색체 부분의 증폭(또는 유전자 복제) 또는 반복 또는 염색체의 염색체 조각의 여분의 조각의 존재는 상동 또는 비상동 염색체에 부착되어 유전자의 일부가 모든 염색체 영역의 다중 복사를 유도하는 두 개 이상의 용량으로 존재할 수 있다.그 안에 위치한 유전자의 e.
- 다배체, 전체 염색체 집합의 복제로, 잠재적으로 별도의 번식 개체와 종분화를 야기합니다.
- 큰 염색체 영역의 결실로 인해 해당 영역 내의 유전자가 손실됩니다.
- 이전에 분리된 DNA 조각을 병렬 배치하는 효과가 있는 돌연변이, 잠재적으로 분리된 유전자를 결합하여 기능적으로 구별되는 융합 유전자(예: bcr-abl)를 형성한다.
- 염색체는 적합성의 감소라 고립된, 피를 집단에서 종 분화로 이어질 수 있는 염색체 재배열로 불리는 구조물에 큰 규모 변화.이들이 포함됩니다.
- 염색체 translocations:비상동 염색체의 유전적 부분의 교류.
- 염색체 역전:염색체 단편의 방위.
- Non-homologous 염색체 교차.
- Interstitial 삭제:한개의 염색체의 DNA의 세그먼트를 제거한intra-chromosomal 삭제하여 이전에 먼 유전자 apposing.예를 들어, 세포들은 사람의 성상 세포종 뇌 종양의 일종인로부터 격리된 염색체 결실은 Glioblastoma(FIG)유전자와 수용체로는 티로신 인산화 효소(run), 융합 단백질(FIG-ROS)를 생산하는데Fused 사이의 시퀀스를 제거한 것으로 드러났다.그 비정상적인 FIG-ROS 융합 단백질은 종양 형성의 변환(정상 세포와 암 세포에 변화)을 일으키는 성분을 이루어 활동적인 인산화 효소 활동을 가지고 있다.
- 이형 접합성의 상실:한 유전자의 삭제 또는 유전적 재조합 행사 중 하나에 두개의 다른 alleles 생물체 내에서 손실을,.
소규모 돌연변이
Small-scale 돌연변이들을 한번에 한개 유전자나 몇가지 뉴클레오티드에 영향을 미친다.(만약 오직 단일 nucleotide 영향을 받는다, 그들은 점 돌연변이라고 불린다.).Small-scale 돌연변이들이 포함됩니다.
- Insertions는 DNA에 하나 이상의 여분의 뉴클레오티드를 더한다.그들은 보통 전치할 수 있는 요소, 또는 오류에 의해 반복되는 요소의 복제 중에 발생하고 있다.하나의 유전자 부호화가 지역에서 Insertions, 또는 둘 다 크게 유전자 산물을 바꿀 수 있는 독서 프레임(프레임 시프트)에 달라지게 하는 mRNA(이음 사이트 돌연변이)의 이음 개조할 수 있다.Insertions가 전이 인자 꽃대 절제 법에 의해 회복이 가능하다.
- 결실은 DNA에서 하나 이상의 뉴클레오티드를 제거한다.삽입과 마찬가지로, 이러한 돌연변이는 유전자의 판독 범위를 바꿀 수 있습니다.일반적으로는 되돌릴 수 없습니다.정확히 동일한 시퀀스가 삽입에 의해 복원될 수 있지만, 어떤 위치에서든 매우 짧은 결실(예: 1-2 염기)을 되돌릴 수 있는 전이성 요소는 존재할 가능성이 매우 낮거나 아예 존재하지 않는다.
- 종종 화학 물질이나 DNA 복제의 오작동에 의해 야기되는 치환 돌연변이는 하나의 뉴클레오티드를 다른 [46]뉴클레오티드와 교환합니다.이러한 변경은 전환 또는 [47]변환으로 분류됩니다.가장 일반적인 것은 퓨린을 푸린(A ↔ G)으로 교환하거나 피리미딘을 피리미딘(C ↔ T)으로 교환하는 전이이다.전이는 아질산, 염기 오쌍 또는 BrdU와 같은 돌연변이 염기 유사체에 의해 발생할 수 있습니다.덜 일반적인 것은 퓨린을 피리미딘으로 교환하거나 피리미딘을 퓨린으로 교환하는 전이이다(C/T ↔ A/G).전환의 예로는 아데닌(A)을 시토신(C)으로 변환하는 것이 있다.점 돌연변이는 DNA의 단일 염기쌍 또는 유전자 내의 다른 작은 염기쌍의 변형이다.포인트 돌연변이는 뉴클레오티드가 원래 상태로 되돌아가는 다른 포인트 돌연변이(진정한 복귀) 또는 2차 부위 복귀(다른 곳에서 유전자 기능을 회복시키는 상보적 돌연변이)에 의해 반전될 수 있다.아래에서 논의한 바와 같이, 유전자의 단백질 코드 영역 내에서 발생하는 점 돌연변이는 동의어 또는 비익명 치환으로 분류될 수 있으며, 후자는 다시 미스센스 또는 넌센스 돌연변이로 분류될 수 있다.
단백질 배열에 대한 영향
단백질 염기서열에 대한 돌연변이의 영향은 유전체 내 어디에서 변이가 일어나는지에 따라 부분적으로 좌우되며, 특히 변이가 코드화 영역인지 비코드화 영역인지에 따라 다르다.촉진제, 인핸서, 소음기와 같은 유전자의 비코드 조절 배열의 돌연변이는 유전자 발현 수준을 바꿀 수 있지만 단백질 서열을 바꿀 가능성은 낮다.intron 내 및 알려진 생물학적 기능이 없는 영역(예: 의사 유전자, 역트랜스포존)의 돌연변이는 일반적으로 중성이며, 표현형에 영향을 미치지 않는다. 그러나 intron 돌연변이는 mRNA 스플라이싱에 영향을 미칠 경우 단백질 생성물을 변화시킬 수 있다.
게놈의 코딩 영역에서 발생하는 돌연변이는 단백질 생성물을 변화시킬 가능성이 높으며 아미노산 배열에 미치는 영향에 따라 분류할 수 있다.
- 프레임시프트 돌연변이는 DNA 배열에서 3으로 균등하게 분할되지 않는 다수의 뉴클레오티드를 삽입 또는 삭제함으로써 발생한다.코돈에 의한 유전자 발현의 삼중항 특성으로 인해 삽입 또는 결실은 코돈의 읽기 프레임 또는 그룹화를 방해하여 [48]원문과 전혀 다른 번역 결과를 초래할 수 있다.결실 또는 삽입이 조기에 발생할수록 생성되는 단백질은 더욱 변화한다(예를 들어 아미노산 프롤린, 아스파라긴산, 티로신, 류신의 CCU GAC UAC CUA 코드).CCU의 U가 삭제되면 CCG ACU UAx가 되고, 대신 프롤린, 트레오닌, 트레오닌 및 다른 아미노산의 일부 또는 아마도 정지 코돈(여기서 x는 다음 뉴클레오티드를 나타낸다)을 코드화한다.반대로 3으로 균등하게 나눌 수 있는 삽입 또는 삭제는 인프레임 변환이라고 불립니다.
- 포인트 치환 돌연변이는 단일 뉴클레오티드의 변화를 가져오며 동의어 또는 비익명일 수 있다.
- 동의어 치환은 코돈을 동일한 아미노산을 코드하는 다른 코돈으로 치환하여 생성된 아미노산 배열을 변형시키지 않는다.동의어 돌연변이는 유전 코드의 퇴화 특성 때문에 발생한다.만약 이 돌연변이가 어떤 표현형 효과를 초래하지 않는다면, 그것은 침묵이라고 불리고, 그러나 모든 동의어 치환들이 침묵하는 것은 아니다. (정확한 뉴클레오티드 배열이 코딩 영역만큼 중요하지 않기 때문에 인트론 같은 코딩 영역 밖의 뉴클레오티드에는 침묵의 돌연변이가 있을 수 있지만, 이것들은 동일하지 않다.dered 동의어 치환).
- 비익명 치환은 코돈을 다른 아미노산을 코드하는 다른 코돈으로 치환하여 생성된 아미노산 배열을 수정한다.익명이 아닌 대체는 난센스 또는 오센스 돌연변이로 분류할 수 있다.
- 미센스 돌연변이는 다른 아미노산의 치환을 일으키도록 뉴클레오티드를 변화시킨다.이것은 결과적으로 결과적인 단백질을 기능하지 않게 만들 수 있다.이러한 돌연변이는 표피분해성 황소증, 겸상적혈구병, SOD1 매개 [49]ALS와 같은 질병의 원인이 된다.한편 아미노산 코돈에서 미센스 돌연변이가 일어나 다른 화학적으로 유사한 아미노산을 사용하게 되면 단백질에 변화가 거의 또는 전혀 없다.예를 들어 AAA에서 AGA로 변경되면 의도된 리신과 화학적으로 유사한 분자인 아르기닌이 인코딩된다.후자의 경우 돌연변이는 표현형에 거의 또는 전혀 영향을 미치지 않으므로 중립적이다.
- 난센스 돌연변이는 전사된 mRNA에 난센스 코돈 또는 난센스 코돈을 초래하는 일련의 DNA에서 점 돌연변이를 의미하며, 아마도 잘리고 종종 기능하지 않는 단백질 생성물을 초래할 수 있다.이런 종류의 돌연변이는 선천성 부신 과형성과 같은 다른 질병들과 연관되어 있다.(「코돈 정지」를 참조해 주세요).
기능에 의한 영향
- 불활성화 돌연변이라고도 불리는 기능 상실은 유전자 생성물이 기능을 거의 또는 전혀 갖지 못하게 만든다.대립 유전자가 기능을 완전히 상실했을 때(늘 대립 유전체), 뮬러의 형태 스키마에서 종종 비정형 또는 비정형 돌연변이라고 불립니다.이러한 돌연변이와 관련된 표현형은 가장 자주 열성적이다.예외는 유기체가 반수체이거나, 정상적인 유전자 생성물의 감소된 용량이 정상적인 표현형에 충분하지 않은 경우이다.
- 활성 돌연변이라고도 불리는 기능성 돌연변이는 유전자 생성물을 변화시켜 그 효과가 더 강해지거나 심지어 다른 비정상적인 기능으로 대체된다.새로운 대립 유전자가 만들어질 때, 원래의 대립 유전자와 함께 새롭게 생성된 대립 유전자를 포함한 헤테로 접합자가 새로운 대립 유전자를 발현할 것이다; 이것은 돌연변이를 지배적인 표현형으로 정의한다.뮬러의 형태 중 일부는 기능 이득에 해당하며, 여기에는 하이퍼모프(유전자 발현 증가)와 네오모프(노벨 기능)가 포함된다.2017년 12월, 미국 정부는 "조류 인플루엔자, 사스, 중동호흡기증후군 또는 메르스 [50][51]바이러스와 같은" 병원체를 강화하는 새로운 "기능성" 실험에 대한 연방정부의 자금 지원을 금지했던 2014년 시행된 임시 금지를 해제했다.
- 지배적인 음성 돌연변이는 야생형 대립 유전자에 대항하는 변화된 유전자 생성물을 가지고 있다.이러한 돌연변이는 보통 분자 기능을 변화시키고(종종 비활성), 지배적 또는 반지배적 표현형으로 특징지어진다.인간에서 지배적인 음성 돌연변이는 암에 관련되었다(예: 유전자 p53,[52] ATM,[53] CEBPA[54] 및 PPARgamma의[55] 돌연변이).마르판 증후군은 세포외 기질의 [56]당단백질 성분인 피브릴린-1을 코드하는 15번 염색체에 위치한 FBN1 유전자의 돌연변이에 의해 발생한다.마르판 증후군은 또한 지배적인 음성 돌연변이와 [57][58]부실의 한 예이다.
- 뮬러 분류 후 하이포머는 야생형 대립 유전자에 비해 유전자 발현을 감소시키는 유전자 산물이 변화한 것이 특징이다.보통, 저형 돌연변이는 열성이지만, 하플로불능은 일부 대립 유전자가 우세하게 만든다.
- 신모형은 새로운 단백질 생성물 합성의 제어에 의해 특징지어진다.
- 치명적인 돌연변이는 돌연변이를 옮기는 유기체의 죽음을 초래하는 돌연변이이다.
- 역돌연변이 또는 역돌연변이란 원래 염기서열과 [59]원래의 표현형을 복원하는 점돌연변이이다.
적합성에 대한 영향(유해, 유익, 중성 돌연변이)
유전학에서는 돌연변이를 유해 또는 유익(또는 중립)으로 분류하는 것이 때로는 유용합니다.
- 유해하거나 유해한 돌연변이는 유기체의 적합성을 떨어뜨린다.많은, 그러나 필수 유전자의 모든 돌연변이가 해로운 것은 아닙니다. 만약 돌연변이가 필수 단백질의 아미노산 염기서열을 바꾸지 않는다면, 대부분의 경우, 그것은 무해하다.
- 유익하거나 유익한 돌연변이는 유기체의 적합성을 증가시킨다.예를 들어 박테리아에 항생제 내성을 일으키는 돌연변이가 있습니다(세균에는 이롭지만 보통 사람에게는 이롭지 않습니다).
- 중성 돌연변이는 유기체에 유해하거나 유익한 영향을 미치지 않는다.이러한 돌연변이는 일정한 속도로 일어나 분자 시계의 기초를 형성합니다.분자 진화의 중성 이론에서, 중성 돌연변이는 분자 수준에서 대부분의 변화의 기초로서 유전적 표류를 제공한다.동물이나 식물에서, 그들의 게놈의 대부분이 비암호화이거나 뚜렷한 기능을 가지지 않는 반복적인 배열로 구성되어 있다는 것을 고려하면, 대부분의 돌연변이는 중립적이다.[60]
수억 개의 돌연변이가 테스트되는 대규모 정량적 돌연변이 발생 화면에서는 반드시 돌연변이의 더 큰 부분이 유해한 영향을 미치지만 항상 유익한 돌연변이를 많이 반환한다는 것을 알 수 있습니다.예를 들어 대장균의 모든 유전자 결손 검사에서 돌연변이의 80%는 음성이었지만, 20%는 양성이었다. 비록 많은 수가 성장에 매우 작은 영향을 끼쳤지만(상태에 따라 다름).[61]유전자 결손은 전체 유전자를 제거하는 것을 포함하므로 점 돌연변이는 거의 항상 훨씬 더 작은 영향을 미칩니다.폐렴 연쇄상구균의 비슷한 화면에서는, 그러나 이번에는 트랜스포존 삽입으로 삽입 돌연변이의 76%가 중립으로 분류되었고, 16%는 적합성이 현저하게 떨어졌지만, 6%가 [62]유리했다.
이 분류는 분명히 상대적이며 다소 인위적이다. 유해한 돌연변이는 조건이 바뀌면 유익한 돌연변이로 빠르게 변할 수 있다.또한, 많은 돌연변이가 작고 대부분 무시할 수 없는 영향을 미칠 수 있지만 특정 조건에서는 관련이 있기 때문에 유해/유익에서 중립으로 기울어진다.또한, 많은 특징들이 수백 개의 유전자(또는 위치)에 의해 결정되기 때문에 각각의 흔적은 아주 작은 영향만을 가진다.예를 들어, 인간의 키는 수백 가지의 유전자 변형("변성")에 의해 결정되지만, 영양의 영향과는 별개로 각각의 키는 [63]키에 매우 작은 영향을 미친다.키(또는 크기) 자체는 동물이나 식물 그룹의 거대한 크기 범위에서 알 수 있듯이 어느 정도 유익할 수 있다.
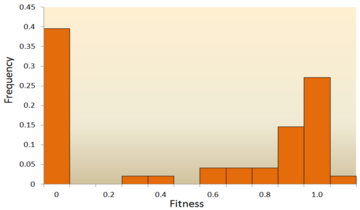
피트니스 효과 분포(DFE)
돌연변이 유발 실험과 분자 배열 데이터에 적용된 이론적 모델을 사용하여 적합성 효과(DFE)의 분포를 추론하려는 시도가 이루어졌다.로 돌연변이(즉, 강하게, 거의하거나 유익한 중립적인 해로운)의 다른 형식의 상대 존재비를 확인하는 데 사용되 DFE,, 유전 variation,[64]의 유지 관리 유전자의 비율 decay,[65]outcrossing 성적 재생산의 유지와 같은 많은 진화적인 질문들 있는 inbreeding[66]에 반대한 관련이 있다.d는 evolu성별과 유전자 [67]재조합DFE는 추정적으로 경미하거나 효과가 [68]없는 돌연변이 분포에 비해 추정적으로 심각한 영향을 미치는 돌연변이 분포의 왜도를 추적하여 추적할 수도 있다.요약하자면, DFE는 진화 [69][70]역학을 예측하는 데 중요한 역할을 한다.이론, 실험 및 분석 방법을 포함한 다양한 접근법이 DFE 연구에 사용되었다.
- 돌연변이 유발 실험:DFE를 조사하는 직접적인 방법은 돌연변이를 유도한 후 돌연변이 피트니스 효과를 측정하는 것인데, 이는 바이러스, 박테리아, 효모 및 드로소필라에서 이미 수행되었다.예를 들어, 바이러스의 DFE에 대한 대부분의 연구는 지점 돌연변이를 생성하고 각 [71][72][73][74]돌연변이의 상대적 적합성을 측정하기 위해 부위 지향 돌연변이 발생을 사용했다.대장균에서 한 연구는 Tn10 [75]유도체의 무작위 삽입 적합성을 직접 측정하기 위해 트랜스포존 돌연변이 유발을 사용했다.효모에서는 고품질의 계통 돌연변이 라이브러리를 생성하고 높은 처리량의 [76]적합성을 측정하기 위해 결합된 돌연변이 유발 및 딥 시퀀싱 접근법이 개발되었습니다.그러나 많은 돌연변이가 검출하기에는[77] 너무 작은 효과를 가지고 있고 돌연변이 유발 실험이 적당히 큰 효과의 돌연변이만 검출할 수 있다는 것을 고려하면, DNA 염기서열 분석은 이러한 돌연변이에 대한 귀중한 정보를 제공할 수 있다.
- 분자 배열 분석:DNA 염기서열 분석 기술의 급속한 발전으로, 엄청난 양의 DNA 염기서열 데이터를 이용할 수 있게 되었고, 앞으로 더 많은 DNA 염기서열 데이터가 제공될 예정입니다.DNA 배열 데이터에서 [78][79][80][81]DFE를 추론하기 위한 다양한 방법이 개발되었습니다.종내 및 종간의 DNA 배열 차이를 조사함으로써, 우리는 중성,[24] 유해 및 유익한 돌연변이에 대한 DFE의 다양한 특성을 추론할 수 있다.구체적으로 말하면, DNA 염기서열 분석 접근방식은 돌연변이 유발 실험을 통해 거의 발견할 수 없는 매우 작은 효과로 돌연변이의 영향을 추정할 수 있게 한다.
피트니스 효과의 분포에 대한 최초의 이론적 연구 중 하나는 영향력 있는 이론 집단 유전학자 Motoo Kimura에 의해 이루어졌다.그의 중립적인 분자 진화 이론은 대부분의 새로운 돌연변이가 매우 유해할 것이며,[25][82] 작은 부분이 중성일 것이라고 제안합니다.이후 아카시 히로시(Hiroshi Akashi)가 제안한 DFE의 바이모달 모델은 매우 유해하고 중성적인 [83]돌연변이를 중심으로 했다.두 이론 모두 새로운 돌연변이의 대부분이 중성 또는 유해하며 유익한 돌연변이는 드물다는 것에 동의하며, 이는 실험 결과에 의해 뒷받침되고 있다.한 가지 예는 소포성 구내염 [71]바이러스의 무작위 돌연변이의 DFE에 대해 수행된 연구이다.전체 돌연변이 중 39.6%는 치사했고 31.2%는 비치사적 유해성, 27.1%는 중성이었다.또 다른 예는 [76]효모를 사용한 높은 throughput 돌연변이 유발 실험입니다.이 실험에서 전체 DFE는 중성 돌연변이 클러스터와 광범위한 유해 돌연변이 분포를 가진 바이모달인 것으로 나타났다.
비교적 적은 수의 돌연변이가 유리하지만,[84] 이러한 돌연변이는 진화적 변화에 중요한 역할을 한다.중성 돌연변이와 마찬가지로, 약하게 선택된 유리한 돌연변이는 무작위 유전자 표류에 의해 손실될 수 있지만, 강하게 선택된 유리한 돌연변이는 고정될 가능성이 더 높다.유리한 돌연변이의 DFE를 아는 것은 진화 역학을 예측하는 능력의 증가로 이어질 수 있다.유리한 돌연변이에 대한 DFE에 대한 이론적 연구는 John H. Gillespie와[85] H에 의해 수행되었다. 알렌 [86]오르그들은 유리한 돌연변이에 대한 분포가 적어도 강하게 선택된 유리한 [87][88][89]돌연변이에 대해 일반적으로 실험 연구에서 뒷받침된 광범위한 조건에서 지수적이어야 한다고 제안했다.
일반적으로 대다수의 돌연변이는 중성 또는 유해하며, 유리한 돌연변이는 드물다. 그러나 돌연변이 유형의 비율은 종마다 다르다.이것은 두 가지 중요한 점을 나타낸다: 첫째, 효과적으로 중성적인 돌연변이의 비율은 종마다 다를 수 있으며, 유효 개체 크기에 의존하여 발생한다. 둘째, 유해한 돌연변이의 평균 효과는 [24]종마다 극적으로 달라진다.또, DFE는 부호화 영역과 비부호화 영역도 다르며, 비부호화 DNA의 DFE는 보다 약하게 선택된 [24]돌연변이를 포함한다.
상속별
헌신적인 생식 세포와 다세포 유기체에서, 돌연변이는 자손에게 그들의 생식 세포를 통해 전달할 수 있는 보통 d.에게 전염되지 않는 헌신적인 생식 집단 이외에는 세포가 있는 생식 세포 돌연변이들, 체세포 돌연변이(또한 획득한 돌연변이라고 불리는)[90]으로 나눌 수 있escendants.
이배체 유기체(예: 인간)는 각 유전자의 두 복사본, 즉 부계 대립 유전자와 모계 대립 유전자를 포함합니다.각 염색체에서의 돌연변이의 발생에 근거하여 우리는 돌연변이를 세 가지 유형으로 분류할 수 있다.야생형 또는 호모 접합성 무변성 생물은 어느 대립 유전자도 돌연변이되지 않은 생물이다.
- 헤테로 접합 돌연변이는 오직 하나의 대립 유전자의 돌연변이이다.
- 호모 접합 돌연변이는 부계 대립 유전자와 모계 대립 유전자의 동일한 돌연변이이다.
- 복합 헤테로 접합 돌연변이 또는 유전자 화합물은 부계 및 모계 대립 유전자의 [91]두 가지 다른 돌연변이로 구성된다.
생식선 돌연변이
개인의 생식세포의 생식세포 돌연변이는 자손의 체질 돌연변이, 즉 모든 세포에 존재하는 돌연변이를 일으킨다.체질 돌연변이는 수정 직후에 발생할 수도 [92]있고 부모의 이전 체질 돌연변이로 이어질 수도 있다.생식계 돌연변이는 다음 세대의 유기체를 통해 유전될 수 있다.
생식세포와 체세포 돌연변이의 구별은 생식세포를 생산하기 위한 전용 생식세포를 가진 동물에게 중요하다.그러나 전용 생식선이 없는 식물에서 돌연변이의 영향을 이해하는 데는 거의 가치가 없다.또한 발아 등의 메커니즘을 통해 무성생식을 하는 동물들에서는 구별이 모호하다. 왜냐하면 딸 유기체를 낳는 세포는 그 유기체의 생식선도 생성하기 때문이다.
부모로부터 유전되지 않은 새로운 생식계 돌연변이를 de novo 돌연변이라고 한다.
체세포 돌연변이
부모로부터 물려받지 않고 자손에게도 물려주지 않는 유전자 구조의 변화를 체세포 [90]돌연변이라고 한다.체세포 돌연변이는 생식선에 영향을 주지 않기 때문에 유기체의 자손에게 유전되지 않는다.그러나 유사분열 중에 같은 유기체 내에 있는 돌연변이 세포의 모든 자손에게 유전된다.그러므로 유기체의 주요 부분은 같은 돌연변이를 가지고 있을 수 있다.이러한 유형의 돌연변이는 보통 자외선이나 특정 유해 화학 물질에 노출되는 것과 같은 환경적 원인에 의해 유발되며 [93]암을 포함한 질병을 유발할 수 있습니다.
식물의 경우, 예를 들어 접붙임과 줄기 절단을 통해 씨앗 생산 없이 일부 체세포 돌연변이가 번식할 수 있다.이러한 종류의 돌연변이는 "맛있는" 사과와 "워싱턴"[94] 배꼽 오렌지와 같은 새로운 종류의 과일로 이어졌다.
인간과 생쥐의 체세포는 두 종의 생식세포 돌연변이율보다 10배 이상 높은 돌연변이율을 가지고 있다. 생쥐는 인간보다 세포 분열당 체세포와 생식세포 돌연변이율이 모두 높다.생식선과 체세포 조직 사이의 돌연변이율 차이는 [95]생식선에서 소마보다 게놈 유지의 중요성이 더 크다는 것을 반영한다.
특수 클래스
- 조건부 돌연변이는 특정한 "허용적인" 환경 조건 하에서 야생형(또는 덜 심각한) 표현형과 특정한 "제한적인" 조건 하에서 돌연변이 표현형을 가진 돌연변이이다.예를 들어, 온도에 민감한 돌연변이는 높은 온도(제한적 조건)에서는 세포 사멸을 일으킬 수 있지만 낮은 온도(허용 [96]조건)에서는 유해한 결과를 초래하지 않을 수 있습니다.이러한 돌연변이는 자율적으로 [97]나타나는 다른 돌연변이와 달리 특정 조건의 존재에 따라 나타나기 때문에 비자율적입니다.허용 조건은 온도,[98] 특정 화학 물질,[99] 빛[99] 또는 [97]게놈의 다른 부분의 돌연변이일 수 있습니다.전사 스위치와 같은 생체 내 메커니즘은 조건부 돌연변이를 만들 수 있다.예를 들어 스테로이드 결합 도메인의 결합은 스테로이드 [100]배위자의 존재에 기초하여 유전자의 발현을 바꿀 수 있는 전사 스위치를 만들 수 있다.조건부 돌연변이는 유전자 발현을 제어할 수 있게 해주기 때문에 연구에 응용된다.이것은 특히 성인의 질병을 연구하는데 유용한데, 일정 기간 성장한 후 발현을 허용함으로써 모델 [99]유기체의 발달 단계에서 나타나는 유전자 발현의 해로운 영향을 제거한다.DNA 특정 조건에서 활성화된 촉진제와 함께 사용되는 Cre-Lox 재조합과 같은 재조합 시스템은 조건부 돌연변이를 생성할 수 있습니다.이중 재조합효소 기술은 여러 유전자의 동시 [99]돌연변이의 결과로 나타나는 질병을 연구하기 위해 여러 조건 돌연변이를 유도하기 위해 사용될 수 있다.특정 내장은 특정 허용 온도에서만 접합되어 부적절한 단백질 합성을 초래하고, 따라서 [101]다른 온도에서 기능 상실을 초래하는 것으로 확인되었다.조건부 돌연변이는 또한 노화와 관련된 유전자 연구에서도 사용될 수 있는데, 그 이유는 생물의 수명 [98]중 특정 기간 후에 발현을 바꿀 수 있기 때문이다.
- 복제 타이밍의 정량적 특성 궤적은 DNA 복제에 영향을 미칩니다.
명명법
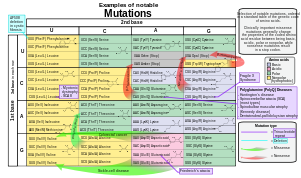
돌연변이를 그렇게 분류하기 위해, "정상" 또는 "건강" 유기체의 DNA로부터 "정상" 염기서열을 얻어야 하며, 그것은 확인되고 보고되어야 한다; 이상적으로는, 뉴클레오티드에 의한 직접적인 비교를 위해 공개되어야 하며, 과학자들에 의해 동의되어야 한다.전문 유전학자 및 생물학자 그룹이 표준 또는 소위 "유전자" 염기서열을 확립할 책임이 있습니다.이 단계에는 엄청난 과학적 노력이 필요하다.일단 합의 서열이 알려지면, 게놈의 돌연변이를 정확히 파악하고, 기술하고, 분류할 수 있다.인간 게놈 변이 학회(HGVS)의 위원회는 연구원과 DNA 진단 센터에서 명확한 돌연변이 기술을 생성하기 위해 사용해야 하는 표준 인간 배열 변이 [102]명명법을 개발했다.원칙적으로, 이 명명법은 다른 유기체의 돌연변이를 묘사하는데도 사용될 수 있다.명명법은 돌연변이의 종류와 염기 또는 아미노산 변화를 규정한다.
- 뉴클레오티드 치환(예: 76A>T) – 숫자는 5' 말단부터의 뉴클레오티드 위치이며, 첫 번째 문자는 야생형 뉴클레오티드를, 두 번째 문자는 야생형을 대체한 뉴클레오티드를 나타낸다.주어진 예에서 76번째 위치의 아데닌은 티민으로 대체되었다.
- 게놈 DNA, 미토콘드리아 DNA, RNA의 돌연변이를 구별할 필요가 있는 경우에는 간단한 규약을 사용한다.예를 들어 뉴클레오티드 배열의 100번째 염기가 G에서 C로 변이된 경우 게놈 DNA에서 변이가 발생한 경우 g.100G>C, 미토콘드리아 DNA에서 변이가 발생한 경우 m.100G>C, RNA에서 변이가 발생한 경우 r.100g>c로 기록된다.
- 아미노산 치환(예: D111E) – 첫 번째 문자는 야생형 아미노산의 한 글자 코드, 숫자는 N-말단 아미노산의 위치, 두 번째 문자는 돌연변이에 존재하는 아미노산의 한 글자 코드입니다.난센스 돌연변이는 두 번째 아미노산(예를 들어 D111X)에 대해 X로 나타난다.
- 아미노산 결실(예: δF508) – 그리스 문자 δ(델타)는 결실을 나타낸다.이 문자는 야생형에 존재하는 아미노산을 가리키며, 숫자는 야생형에 존재하는 아미노산의 N 말단으로부터의 위치이다.
돌연변이율
돌연변이율은 종에 따라 크게 다르며, 일반적으로 돌연변이를 결정하는 진화력이 지속적인 연구의 대상이다.
인간에서 돌연변이율은 세대당 게놈당 약 5090개의 de novo 돌연변이로, 즉 사람마다 부모에게는 없었던 5090개의 새로운 돌연변이가 축적된다.이 숫자는 수천 개의 인간 트리오, 즉 두 명의 부모와 적어도 한 명의 [103]자녀들의 시퀀스를 통해 확립되었습니다.
RNA 바이러스의 게놈은 DNA가 아닌 RNA를 기반으로 한다.RNA 바이러스 게놈은 (DNA에서와 같이) 이중가닥이거나 단일가닥일 수 있습니다.이러한 바이러스 중 일부(예: 단일 가닥 인간 면역 결핍 바이러스)에서는 복제가 빠르게 이루어지며 게놈의 정확성을 확인하는 메커니즘이 없습니다.오류가 발생하기 쉬운 프로세스로 인해 돌연변이가 발생하는 경우가 많습니다.
돌연변이의 무작위성
돌연변이가 (확률 측면에서) 결과와 관련하여 (전적으로) 무작위라는 가정이 널리 퍼져 있다.이러한 DNA 복구 및 돌연변이 바이어스는 다양한 인자와 관련되므로 돌연변이 빈도가 게놈의 영역에 따라 달라질 수 있기 때문에 이는 잘못된 것으로 나타났다.예를 들어, 생물학적으로 중요한 영역은 돌연변이로부터 보호되고 연구된 식물에 유익한 돌연변이는 더 가능성이 높은 것으로 밝혀졌다. 즉, 돌연변이는 "식물에게 이익이 되는 방식으로 무작위적이지 않다."[104][105]
질병원인
DNA의 코드화 영역의 돌연변이에 의해 야기되는 DNA의 변화는 부분적 또는 완전히 기능하지 않는 단백질을 야기할 수 있는 단백질 배열의 오류를 야기할 수 있다.각각의 세포는, 올바르게 기능하기 위해서, 적절한 시기에 적절한 장소에서 기능하기 위해 수천 개의 단백질에 의존합니다.돌연변이가 신체에서 중요한 역할을 하는 단백질을 변화시키면, 의학적 상태가 발생할 수 있다.드로소필라의 다른 종들 간의 유전자 비교에 대한 한 연구는 돌연변이가 단백질을 변화시킨다면, 그 돌연변이는 가장 해로울 것이며, 아미노산 다형성의 [8]약 70%가 해로운 영향을 끼치고 나머지는 중립적이거나 약하게 유익할 것이라고 시사한다.어떤 돌연변이는 유전자의 DNA 염기서열을 바꾸지만 유전자에 의해 만들어진 단백질은 바꾸지 않는다.연구에 따르면 효모의 코드화되지 않은 DNA에서 점 돌연변이의 7%만이 유해하고 DNA 코드화에서 12%만이 유해한 것으로 나타났다.나머지 돌연변이는 중성적이거나 약간 [106]이롭습니다
유전성 질환
만약 돌연변이가 생식세포에 존재한다면, 그것은 모든 세포에서 돌연변이를 옮기는 자손을 낳을 수 있다.이것은 유전병의 경우입니다.특히 생식세포 내에서 DNA 수복 유전자에 돌연변이가 있는 경우, 그러한 생식계 돌연변이를 가진 인간은 암에 걸릴 위험이 높아질 수 있다.이러한 생식계 돌연변이 34개의 리스트는 DNA 복구결핍장애에 기재되어 있다.하나의 예는 OCA1 또는 OCA2 유전자에서 발생하는 돌연변이인 알비니즘이다.이 장애를 가진 사람들은 많은 종류의 암이나 다른 장애에 걸리기 쉽고 시력이 저하됩니다.
DNA 손상은 DNA가 복제될 때 오류를 일으킬 수 있고, 이러한 복제 오류는 유전자 돌연변이를 일으킬 수 있으며, 이는 다시 유전적인 장애를 일으킬 수 있다.DNA 손상은 세포의 DNA 복구 시스템에 의해 복구된다.각각의 세포는 효소가 DNA의 손상을 인식하고 복구하는 많은 경로를 가지고 있다.DNA는 여러 가지 방법으로 손상될 수 있기 때문에, DNA 복구 과정은 신체가 질병으로부터 스스로를 보호하는 중요한 방법이다.일단 DNA 손상이 돌연변이를 일으키면, 그 돌연변이는 복구할 수 없다.
발암에 대한 역할
한편, 돌연변이는 생물의 체세포에서 발생할 수 있다.그러한 돌연변이는 같은 유기체 내의 이 세포의 모든 자손에게 존재할 것이다.체세포의 세대에 걸친 특정 돌연변이의 축적은 정상 세포에서 암 [107]세포로의 악성 변형의 원인 중 일부이다.
헤테로 접합 기능 상실을 가진 세포(유전자의 좋은 복사본 1개와 돌연변이된 복사본 1개)는 좋은 복사본이 자발적으로 신체적으로 돌연변이가 될 때까지 변환되지 않은 복사본과 함께 정상적으로 기능할 수 있다.이런 돌연변이는 생물에서 자주 발생하지만 그 비율을 측정하기는 어렵다.이 비율을 측정하는 것은 사람들이 [108]암에 걸릴 수 있는 비율을 예측하는 데 중요하다.
점 돌연변이는 DNA 복제 중에 발생하는 자발적 돌연변이로 인해 발생할 수 있습니다.돌연변이의 속도는 돌연변이에 의해 증가할 수 있다.돌연변이는 자외선, X선 또는 극도의 열 또는 화학 물질(염기 쌍을 잘못 배치하거나 DNA의 나선 모양을 교란하는 분자)에서 나오는 방사선과 같은 물리적 물질일 수 있다.암과 관련된 돌연변이는 암과 그 예방에 대해 배우기 위해 종종 연구된다.
프리온 돌연변이
프리온은 단백질로 유전 물질을 포함하고 있지 않다.그러나 프리온 복제는 다른 [109]복제 형태와 마찬가지로 돌연변이와 자연선택의 영향을 받는 것으로 나타났다.인간 유전자 PRNP는 주요 프리온 단백질인 PrP를 코드화하며 질병을 유발하는 프리온을 발생시킬 수 있는 돌연변이를 일으킨다.
유익한 돌연변이
단백질 배열의 변화를 일으키는 돌연변이는 유기체에 해로울 수 있지만, 경우에 따라 그 영향은 주어진 환경에서 긍정적일 수 있다.이 경우 돌연변이는 돌연변이 유기체가 야생형 유기체보다 특정한 환경적 스트레스를 더 잘 견디거나 더 빨리 번식할 수 있게 할 수 있다.이러한 경우 돌연변이는 자연 선택을 통해 집단에서 더 흔해지는 경향이 있습니다.예를 들어 다음과 같습니다.
HIV 내성: 인간 CCR5(CCR5-δ32)의 특정 32개의 염기쌍 결실은 호모 접합체에 대한 HIV 내성을 부여하고 헤테로 [110]접합체의 에이즈 발병을 지연시킵니다.유럽 인구에서 CCR5-δ32의 비교적 높은 빈도의 병인에 대한 한 가지 가능한 설명은 그것이 14세기 중반 유럽에서 부보닉 페스트에 내성을 부여했다는 것이다.이 돌연변이를 가진 사람들은 감염에서 살아남을 가능성이 더 높았고, 따라서 인구에서 그 빈도는 증가했다.[111]이 이론은 왜 이 돌연변이가 남부 아프리카에서 발견되지 않는지 설명할 수 있는데, 남부 아프리카는 부보닉 페스트의 영향을 받지 않았다.새로운 이론은 CCR5 Delta 32 돌연변이에 대한 선택적 압력이 선종성 [112]페스트가 아닌 천연두에 의해 발생했다는 것을 시사한다.
말라리아 내성:유해한 돌연변이의 예로는 적혈구 내의 산소 운반 물질인 헤모글로빈의 비정상적인 유형을 만들어 내는 혈액 질환인 겸상적혈구 질환이 있다.사하라 사막 이남 아프리카 원주민의 3분의 1이 이 대립 유전자를 가지고 있는데, 말라리아가 흔한 지역에서는 겸상적혈구 [113]대립 유전자 하나만 가지고 있는 생존 가치가 있기 때문입니다.겸상 적혈구 질환의 두 가지 대립 유전자 중 하나만 있는 사람들은 말라리아에 더 잘 저항합니다. 말라리아 플라스모듐의 감염은 말라리아에 감염된 세포의 병균에 의해 중단되기 때문입니다.
항생제 내성: 사실상 모든 박테리아가 항생제에 노출되면 항생제 내성이 생깁니다.사실, 박테리아 집단은 이미 항생제 [114]선택 하에 선택되는 그러한 돌연변이를 가지고 있습니다.분명히, 그러한 돌연변이는 박테리아에게만 유익할 뿐 감염된 사람들에게는 유익하지 않다.
락타아제 지속성.돌연변이는 인간이 모유에서 자연적으로 젖을 떼고 난 후에 락타아제 효소를 발현할 수 있게 해주었고, 성인들은 최근의 인류 [115]진화에서 가장 유익한 돌연변이들 중 하나인 락토스를 소화시킬 수 있게 해주었다.
역사
돌연변이론은 찰스 다윈의 1859년 책 "종의 기원"의 출판 이전과 이후 모두 존재했던 다윈 진화론의 여러 대안들 중 하나이다.이 이론에서 돌연변이는 새로운 형태와 새로운 종을 만들어 내는 신기함의 원천이었다. 어쩌면 순식간에,[116] 갑작스럽게.[117]이는 돌연변이의 공급에 의해 제한되었던 진화를 주도하는 것으로 예상되었다.
다윈 이전에 생물학자들은 염분론을 믿었는데, 염분론은 즉각적인 분화를 포함한 큰 진화적 도약의 가능성이다.예를 들어, 1822년 에티엔 조프로이 생힐레르는 종이 갑작스런 변형에 의해 형성될 수 있다고 주장했고, 나중에 [118]고색화라고 불리게 되었다.다윈은 지질학에서처럼 진화에서 점진주의를 주장하면서 염화에 반대했다.1864년, 알버트 폰 쾰리커는 조프로이의 [119]이론을 부활시켰다.1901년에 가 휘호 더 프리스 갑자기 저녁 환락의 달맞이 꽃 속 lamarckiana에 그의 실험했고, 20세기, mutationism, 또는 다위니즘에 휘호 더 프리스라는 이름을 가진 mutationstheorie,[116][120]가 된 라이벌이 잠시 동안 유전 학자들 incl에 있는 요소의 첫 십년에서 발생하는 새로운 형태들에 이름을"돌연변이"을 주었다.Wil uding리암 베이트슨,[121] 토마스 헌트 모건, 레지날드 [116][122]패넷입니다
돌연변이론에 대한 이해는 다윈 진화론의 반대자들과 선택이 지속적인 변이에 의해 작동한다고 주장한 생물 통계학 학파의 경쟁자들로서 현대 합성의 지지자들에 의해 20세기 중반의 초기 돌연변이론자들의 묘사에 의해 흐려진다.이 묘사에서 돌연변이론은 1918년경 수학자 로널드 [123][124][125][126]피셔의 작품에서 시작된 것으로 추정되는 유전학과 자연 도태의 통합에 의해 좌절되었다.그러나 멘델 유전학과 자연 도태의 정렬은 1902년 Udny Yule의 [127]논문으로 시작되었고 유럽과 미국에서 이론적이고 실험적인 연구로 구축되었다.논란에도 불구하고, 초기 돌연변이학자들은 1918년까지 이미 자연 도태를 받아들였고 [124][125]키와 같은 같은 같은 특징에 작용하는 여러 유전자의 결과로 지속적인 변화를 설명했습니다.
돌연변이는 라마르크주의와 직교 형성과 같은 다윈주의에 대한 다른 대안들과 함께, 멘델의 유전학과 자연 선택이 쉽게 함께 작동할 수 있다는 것을 알게 되면서 대부분의 생물학자들에 의해 폐기되었다; 돌연변이는 자연 선택에 필수적인 유전자 변이의 원천으로 대체되었다.그러나 돌연변이주의가 완전히 사라진 것은 아니다.1940년, 리처드 골드슈미트는 고색화에 의한 단일 단계 분화를 다시 주장하면서, 이렇게 만들어진 유기체를 "희망적인 괴물"이라고 묘사했고, 널리 [128][129]조롱을 받았다.1987년, 네이 마사토시는 진화가 종종 돌연변이에 [130]제한적이라고 논쟁적으로 주장했다.Douglas J와 같은 현대 생물학자. Futuyma는 본질적으로 큰 돌연변이에 의해 움직이는 진화에 대한 모든 주장들이 다윈의 [131]진화에 의해 설명될 수 있다고 결론지었다.
「 」를 참조해 주세요.
레퍼런스
- ^ "mutation Learn Science at Scitable". Nature. Nature Education. Retrieved 24 September 2018.
- ^ Sfeir, A; Symington, LS (November 2015). "Microhomology-Mediated End Joining: A Back-up Survival Mechanism or Dedicated Pathway?". Trends in Biochemical Sciences. 40 (11): 701–714. doi:10.1016/j.tibs.2015.08.006. PMC 4638128. PMID 26439531.
- ^ Chen J, Miller BF, Furano AV (April 2014). "Repair of naturally occurring mismatches can induce mutations in flanking DNA". eLife. 3: e02001. doi:10.7554/elife.02001. PMC 3999860. PMID 24843013.
- ^ Rodgers K, McVey M (January 2016). "Error-Prone Repair of DNA Double-Strand Breaks". Journal of Cellular Physiology. 231 (1): 15–24. doi:10.1002/jcp.25053. PMC 4586358. PMID 26033759.
- ^ a b Bertram JS (December 2000). "The molecular biology of cancer". Molecular Aspects of Medicine. 21 (6): 167–223. doi:10.1016/S0098-2997(00)00007-8. PMID 11173079. S2CID 24155688.
- ^ a b Aminetzach YT, Macpherson JM, Petrov DA (July 2005). "Pesticide resistance via transposition-mediated adaptive gene truncation in Drosophila". Science. 309 (5735): 764–7. Bibcode:2005Sci...309..764A. doi:10.1126/science.1112699. PMID 16051794. S2CID 11640993.
- ^ Burrus V, Waldor MK (June 2004). "Shaping bacterial genomes with integrative and conjugative elements". Research in Microbiology. 155 (5): 376–86. doi:10.1016/j.resmic.2004.01.012. PMID 15207870.
- ^ a b Sawyer SA, Parsch J, Zhang Z, Hartl DL (April 2007). "Prevalence of positive selection among nearly neutral amino acid replacements in Drosophila". Proceedings of the National Academy of Sciences of the United States of America. 104 (16): 6504–10. Bibcode:2007PNAS..104.6504S. doi:10.1073/pnas.0701572104. PMC 1871816. PMID 17409186.
- ^ Hastings PJ, Lupski JR, Rosenberg SM, Ira G (August 2009). "Mechanisms of change in gene copy number". Nature Reviews. Genetics. 10 (8): 551–64. doi:10.1038/nrg2593. PMC 2864001. PMID 19597530.
- ^ Carroll SB, Grenier JK, Weatherbee SD (2005). From DNA to Diversity: Molecular Genetics and the Evolution of Animal Design (2nd ed.). Malden, MA: Blackwell Publishing. ISBN 978-1-4051-1950-4. LCCN 2003027991. OCLC 53972564.
- ^ Harrison PM, Gerstein M (May 2002). "Studying genomes through the aeons: protein families, pseudogenes and proteome evolution". Journal of Molecular Biology. 318 (5): 1155–74. doi:10.1016/S0022-2836(02)00109-2. PMID 12083509.
- ^ Orengo CA, Thornton JM (July 2005). "Protein families and their evolution-a structural perspective". Annual Review of Biochemistry. 74: 867–900. doi:10.1146/annurev.biochem.74.082803.133029. PMID 15954844.
- ^ Long M, Betrán E, Thornton K, Wang W (November 2003). "The origin of new genes: glimpses from the young and old". Nature Reviews. Genetics. 4 (11): 865–75. doi:10.1038/nrg1204. PMID 14634634. S2CID 33999892.
- ^ Wang M, Caetano-Anollés G (January 2009). "The evolutionary mechanics of domain organization in proteomes and the rise of modularity in the protein world". Structure. 17 (1): 66–78. doi:10.1016/j.str.2008.11.008. PMID 19141283.
- ^ Bowmaker JK (May 1998). "Evolution of colour vision in vertebrates". Eye. 12 (Pt 3b): 541–7. doi:10.1038/eye.1998.143. PMID 9775215. S2CID 12851209.
- ^ Gregory TR, Hebert PD (April 1999). "The modulation of DNA content: proximate causes and ultimate consequences". Genome Research. 9 (4): 317–24. doi:10.1101/gr.9.4.317. PMID 10207154. S2CID 16791399.
- ^ Hurles M (July 2004). "Gene duplication: the genomic trade in spare parts". PLOS Biology. 2 (7): E206. doi:10.1371/journal.pbio.0020206. PMC 449868. PMID 15252449.
- ^ Liu N, Okamura K, Tyler DM, Phillips MD, Chung WJ, Lai EC (October 2008). "The evolution and functional diversification of animal microRNA genes". Cell Research. 18 (10): 985–96. doi:10.1038/cr.2008.278. PMC 2712117. PMID 18711447.
- ^ Siepel A (October 2009). "Darwinian alchemy: Human genes from noncoding DNA". Genome Research. 19 (10): 1693–5. doi:10.1101/gr.098376.109. PMC 2765273. PMID 19797681.
- ^ Zhang J, Wang X, Podlaha O (May 2004). "Testing the chromosomal speciation hypothesis for humans and chimpanzees". Genome Research. 14 (5): 845–51. doi:10.1101/gr.1891104. PMC 479111. PMID 15123584.
- ^ Ayala FJ, Coluzzi M (May 2005). "Chromosome speciation: humans, Drosophila, and mosquitoes". Proceedings of the National Academy of Sciences of the United States of America. 102 Suppl 1 (Suppl 1): 6535–42. Bibcode:2005PNAS..102.6535A. doi:10.1073/pnas.0501847102. PMC 1131864. PMID 15851677.
- ^ Hurst GD, Werren JH (August 2001). "The role of selfish genetic elements in eukaryotic evolution". Nature Reviews Genetics. 2 (8): 597–606. doi:10.1038/35084545. PMID 11483984. S2CID 2715605.
- ^ Häsler J, Strub K (November 2006). "Alu elements as regulators of gene expression". Nucleic Acids Research. 34 (19): 5491–7. doi:10.1093/nar/gkl706. PMC 1636486. PMID 17020921.
- ^ a b c d Eyre-Walker A, Keightley PD (August 2007). "The distribution of fitness effects of new mutations" (PDF). Nature Reviews Genetics. 8 (8): 610–8. doi:10.1038/nrg2146. PMID 17637733. S2CID 10868777. Archived from the original (PDF) on 4 March 2016. Retrieved 6 September 2010.
- ^ a b Kimura M (1983). The Neutral Theory of Molecular Evolution. Cambridge, UK; New York: Cambridge University Press. ISBN 978-0-521-23109-1. LCCN 82022225. OCLC 9081989.
- ^ Bohidar HB (January 2015). Fundamentals of Polymer Physics and Molecular Biophysics. Cambridge University Press. ISBN 978-1-316-09302-3.
- ^ Dover GA, Darwin C (2000). Dear Mr. Darwin: Letters on the Evolution of Life and Human Nature. University of California Press. ISBN 9780520227903.
- ^ Tibayrenc M (12 January 2017). Genetics and Evolution of Infectious Diseases. Elsevier. ISBN 9780128001530.
- ^ "Cancer Is Partly Caused By Bad Luck, Study Finds". NPR.org. Archived from the original on 13 July 2017.
- ^ Jha A (22 August 2012). "Older fathers pass on more genetic mutations, study shows". The Guardian.
- ^ Ames BN, Shigenaga MK, Hagen TM (September 1993). "Oxidants, antioxidants, and the degenerative diseases of aging". Proceedings of the National Academy of Sciences of the United States of America. 90 (17): 7915–22. Bibcode:1993PNAS...90.7915A. doi:10.1073/pnas.90.17.7915. PMC 47258. PMID 8367443.
- ^ Montelone BA (1998). "Mutation, Mutagens, and DNA Repair". www-personal.ksu.edu. Archived from the original on 26 September 2015. Retrieved 2 October 2015.
- ^ Slocombe L, Al-Khalili JS, Sacchi M (February 2021). "Quantum and classical effects in DNA point mutations: Watson-Crick tautomerism in AT and GC base pairs". Physical Chemistry Chemical Physics. 23 (7): 4141–4150. Bibcode:2021PCCP...23.4141S. doi:10.1039/D0CP05781A. ISSN 1463-9076. PMID 33533770. S2CID 231788542.
- ^ Slocombe, Louie; Sacchi, Marco; Al-Khalili, Jim (5 May 2022). "An open quantum systems approach to proton tunnelling in DNA". Communications Physics. 5 (1): 109. arXiv:2110.00113. Bibcode:2022CmPhy...5..109S. doi:10.1038/s42005-022-00881-8. ISSN 2399-3650. S2CID 238253421.
- ^ Stuart GR, Oda Y, de Boer JG, Glickman BW (March 2000). "Mutation frequency and specificity with age in liver, bladder and brain of lacI transgenic mice". Genetics. 154 (3): 1291–300. doi:10.1093/genetics/154.3.1291. PMC 1460990. PMID 10757770.
- ^ Kunz BA, Ramachandran K, Vonarx EJ (April 1998). "DNA sequence analysis of spontaneous mutagenesis in Saccharomyces cerevisiae". Genetics. 148 (4): 1491–505. doi:10.1093/genetics/148.4.1491. PMC 1460101. PMID 9560369.
- ^ Lieber MR (July 2010). "The mechanism of double-strand DNA break repair by the nonhomologous DNA end-joining pathway". Annual Review of Biochemistry. 79: 181–211. doi:10.1146/annurev.biochem.052308.093131. PMC 3079308. PMID 20192759.
- ^ PDB 1JDG 2015년 12월 31일 Wayback Machine에서 작성
- ^ Pfohl-Leszkowicz A, Manderville RA (January 2007). "Ochratoxin A: An overview on toxicity and carcinogenicity in animals and humans". Molecular Nutrition & Food Research. 51 (1): 61–99. doi:10.1002/mnfr.200600137. PMID 17195275.
- ^ Kozmin S, Slezak G, Reynaud-Angelin A, Elie C, de Rycke Y, Boiteux S, Sage E (September 2005). "UVA radiation is highly mutagenic in cells that are unable to repair 7,8-dihydro-8-oxoguanine in Saccharomyces cerevisiae". Proceedings of the National Academy of Sciences of the United States of America. 102 (38): 13538–43. Bibcode:2005PNAS..10213538K. doi:10.1073/pnas.0504497102. PMC 1224634. PMID 16157879.
- ^ a b Fitzgerald DM, Rosenberg SM (April 2019). "What is mutation? A chapter in the series: How microbes "jeopardize" the modern synthesis". PLOS Genetics. 15 (4): e1007995. doi:10.1371/journal.pgen.1007995. PMC 6443146. PMID 30933985.
- ^ Galhardo RS, Hastings PJ, Rosenberg SM (1 January 2007). "Mutation as a stress response and the regulation of evolvability". Critical Reviews in Biochemistry and Molecular Biology. 42 (5): 399–435. doi:10.1080/10409230701648502. PMC 3319127. PMID 17917874.
- ^ Quinto-Alemany D, Canerina-Amaro A, Hernández-Abad LG, Machín F, Romesberg FE, Gil-Lamaignere C (31 July 2012). Sturtevant J (ed.). "Yeasts acquire resistance secondary to antifungal drug treatment by adaptive mutagenesis". PLOS ONE. 7 (7): e42279. Bibcode:2012PLoSO...742279Q. doi:10.1371/journal.pone.0042279. PMC 3409178. PMID 22860105.
- ^ 이미지에 대한 참조는 Wikimedia Commons 페이지의 Commons:파일:Notable morties.svg#References.
- ^ Rahman N. "The clinical impact of DNA sequence changes". Transforming Genetic Medicine Initiative. Archived from the original on 4 August 2017. Retrieved 27 June 2017.
- ^ Freese E (April 1959). "The Difference Between Spontaneous and Base-Analogue Induced Mutations of Phage T4". Proceedings of the National Academy of Sciences of the United States of America. 45 (4): 622–33. Bibcode:1959PNAS...45..622F. doi:10.1073/pnas.45.4.622. PMC 222607. PMID 16590424.
- ^ Freese E (June 1959). "The specific mutagenic effect of base analogues on Phage T4". Journal of Molecular Biology. 1 (2): 87–105. doi:10.1016/S0022-2836(59)80038-3.
- ^ Hogan CM (12 October 2010). "Mutation". In Monosson E (ed.). Encyclopedia of Earth. Washington, D.C.: Environmental Information Coalition, National Council for Science and the Environment. OCLC 72808636. Archived from the original on 14 November 2015. Retrieved 8 October 2015.
- ^ Boillée S, Vande Velde C, Cleveland DW (October 2006). "ALS: a disease of motor neurons and their nonneuronal neighbors". Neuron. 52 (1): 39–59. CiteSeerX 10.1.1.325.7514. doi:10.1016/j.neuron.2006.09.018. PMID 17015226. S2CID 12968143.
- ^ Steenhuysen J (19 December 2017). "U.S. Lifts Funding Ban on Studies That Enhance Dangerous Germs". U.S. News & World Report. Retrieved 15 January 2018.
- ^ Collins FS (19 December 2017). "NIH Lifts Funding Pause on Gain-of-Function Research". National Institutes of Health. Archived from the original on 22 December 2017.
- ^ Goh AM, Coffill CR, Lane DP (January 2011). "The role of mutant p53 in human cancer". The Journal of Pathology. 223 (2): 116–26. doi:10.1002/path.2784. PMID 21125670. S2CID 23998813.
- ^ Chenevix-Trench G, Spurdle AB, Gatei M, Kelly H, Marsh A, Chen X, Donn K, Cummings M, Nyholt D, Jenkins MA, Scott C, Pupo GM, Dörk T, Bendix R, Kirk J, Tucker K, McCredie MR, Hopper JL, Sambrook J, Mann GJ, Khanna KK (February 2002). "Dominant negative ATM mutations in breast cancer families". Journal of the National Cancer Institute. 94 (3): 205–15. CiteSeerX 10.1.1.557.6394. doi:10.1093/jnci/94.3.205. PMID 11830610.
- ^ Paz-Priel I, Friedman A (2011). "C/EBPα dysregulation in AML and ALL". Critical Reviews in Oncogenesis. 16 (1–2): 93–102. doi:10.1615/critrevoncog.v16.i1-2.90. PMC 3243939. PMID 22150310.
- ^ Capaccio D, Ciccodicola A, Sabatino L, Casamassimi A, Pancione M, Fucci A, Febbraro A, Merlino A, Graziano G, Colantuoni V (June 2010). "A novel germline mutation in peroxisome proliferator-activated receptor gamma gene associated with large intestine polyp formation and dyslipidemia". Biochimica et Biophysica Acta (BBA) - Molecular Basis of Disease. 1802 (6): 572–81. doi:10.1016/j.bbadis.2010.01.012. PMID 20123124.
- ^ McKusick VA (July 1991). "The defect in Marfan syndrome". Nature. 352 (6333): 279–81. Bibcode:1991Natur.352..279M. doi:10.1038/352279a0. PMID 1852198. S2CID 4341743.
- ^ Judge DP, Biery NJ, Keene DR, Geubtner J, Myers L, Huso DL, Sakai LY, Dietz HC (July 2004). "Evidence for a critical contribution of haploinsufficiency in the complex pathogenesis of Marfan syndrome". The Journal of Clinical Investigation. 114 (2): 172–81. doi:10.1172/JCI20641. PMC 449744. PMID 15254584.
- ^ Judge DP, Dietz HC (December 2005). "Marfan's syndrome". Lancet. 366 (9501): 1965–76. doi:10.1016/S0140-6736(05)67789-6. PMC 1513064. PMID 16325700.
- ^ Ellis NA, Ciocci S, German J (February 2001). "Back mutation can produce phenotype reversion in Bloom syndrome somatic cells". Human Genetics. 108 (2): 167–73. doi:10.1007/s004390000447. PMID 11281456. S2CID 22290041.
- ^ Doolittle WF, Brunet TD (December 2017). "On causal roles and selected effects: our genome is mostly junk". BMC Biology. 15 (1): 116. doi:10.1186/s12915-017-0460-9. PMC 5718017. PMID 29207982.
- ^ Nichols RJ, Sen S, Choo YJ, Beltrao P, Zietek M, Chaba R, et al. (January 2011). "Phenotypic landscape of a bacterial cell". Cell. 144 (1): 143–56. doi:10.1016/j.cell.2010.11.052. PMC 3060659. PMID 21185072.
- ^ van Opijnen T, Bodi KL, Camilli A (October 2009). "Tn-seq: high-throughput parallel sequencing for fitness and genetic interaction studies in microorganisms". Nature Methods. 6 (10): 767–72. doi:10.1038/nmeth.1377. PMC 2957483. PMID 19767758.
- ^ Allen HL, Estrada K, Lettre G, Berndt SI, Weedon MN, Rivadeneira F, et al. (October 2010). "Hundreds of variants clustered in genomic loci and biological pathways affect human height". Nature. 467 (7317): 832–8. Bibcode:2010Natur.467..832L. doi:10.1038/nature09410. PMC 2955183. PMID 20881960.
- ^ Charlesworth D, Charlesworth B, Morgan MT (December 1995). "The pattern of neutral molecular variation under the background selection model". Genetics. 141 (4): 1619–32. doi:10.1093/genetics/141.4.1619. PMC 1206892. PMID 8601499.
- ^ Loewe L (April 2006). "Quantifying the genomic decay paradox due to Muller's ratchet in human mitochondrial DNA". Genetical Research. 87 (2): 133–59. doi:10.1017/S0016672306008123. PMID 16709275.
- ^ Bernstein H, Hopf FA, Michod RE (1987). "The molecular basis of the evolution of sex". Molecular Genetics of Development. Advances in Genetics. Vol. 24. pp. 323–70. doi:10.1016/s0065-2660(08)60012-7. ISBN 9780120176243. PMID 3324702.
- ^ Peck JR, Barreau G, Heath SC (April 1997). "Imperfect genes, Fisherian mutation and the evolution of sex". Genetics. 145 (4): 1171–99. doi:10.1093/genetics/145.4.1171. PMC 1207886. PMID 9093868.
- ^ Simcikova D, Heneberg P (December 2019). "Refinement of evolutionary medicine predictions based on clinical evidence for the manifestations of Mendelian diseases". Scientific Reports. 9 (1): 18577. Bibcode:2019NatSR...918577S. doi:10.1038/s41598-019-54976-4. PMC 6901466. PMID 31819097.
- ^ Keightley PD, Lynch M (March 2003). "Toward a realistic model of mutations affecting fitness". Evolution; International Journal of Organic Evolution. 57 (3): 683–5, discussion 686–9. doi:10.1554/0014-3820(2003)057[0683:tarmom]2.0.co;2. JSTOR 3094781. PMID 12703958. S2CID 198157678.
- ^ Barton NH, Keightley PD (January 2002). "Understanding quantitative genetic variation". Nature Reviews Genetics. 3 (1): 11–21. doi:10.1038/nrg700. PMID 11823787. S2CID 8934412.
- ^ a b c Sanjuán R, Moya A, Elena SF (June 2004). "The distribution of fitness effects caused by single-nucleotide substitutions in an RNA virus". Proceedings of the National Academy of Sciences of the United States of America. 101 (22): 8396–401. Bibcode:2004PNAS..101.8396S. doi:10.1073/pnas.0400146101. PMC 420405. PMID 15159545.
- ^ Carrasco P, de la Iglesia F, Elena SF (December 2007). "Distribution of fitness and virulence effects caused by single-nucleotide substitutions in Tobacco Etch virus". Journal of Virology. 81 (23): 12979–84. doi:10.1128/JVI.00524-07. PMC 2169111. PMID 17898073.
- ^ Sanjuán R (June 2010). "Mutational fitness effects in RNA and single-stranded DNA viruses: common patterns revealed by site-directed mutagenesis studies". Philosophical Transactions of the Royal Society of London. Series B, Biological Sciences. 365 (1548): 1975–82. doi:10.1098/rstb.2010.0063. PMC 2880115. PMID 20478892.
- ^ Peris JB, Davis P, Cuevas JM, Nebot MR, Sanjuán R (June 2010). "Distribution of fitness effects caused by single-nucleotide substitutions in bacteriophage f1". Genetics. 185 (2): 603–9. doi:10.1534/genetics.110.115162. PMC 2881140. PMID 20382832.
- ^ Elena SF, Ekunwe L, Hajela N, Oden SA, Lenski RE (March 1998). "Distribution of fitness effects caused by random insertion mutations in Escherichia coli". Genetica. 102–103 (1–6): 349–58. doi:10.1023/A:1017031008316. PMID 9720287. S2CID 2267064.
- ^ a b Hietpas RT, Jensen JD, Bolon DN (May 2011). "Experimental illumination of a fitness landscape". Proceedings of the National Academy of Sciences of the United States of America. 108 (19): 7896–901. Bibcode:2011PNAS..108.7896H. doi:10.1073/pnas.1016024108. PMC 3093508. PMID 21464309.
- ^ Davies EK, Peters AD, Keightley PD (September 1999). "High frequency of cryptic deleterious mutations in Caenorhabditis elegans". Science. 285 (5434): 1748–51. doi:10.1126/science.285.5434.1748. PMID 10481013.
- ^ Loewe L, Charlesworth B (September 2006). "Inferring the distribution of mutational effects on fitness in Drosophila". Biology Letters. 2 (3): 426–30. doi:10.1098/rsbl.2006.0481. PMC 1686194. PMID 17148422.
- ^ Eyre-Walker A, Woolfit M, Phelps T (June 2006). "The distribution of fitness effects of new deleterious amino acid mutations in humans". Genetics. 173 (2): 891–900. doi:10.1534/genetics.106.057570. PMC 1526495. PMID 16547091.
- ^ Sawyer SA, Kulathinal RJ, Bustamante CD, Hartl DL (August 2003). "Bayesian analysis suggests that most amino acid replacements in Drosophila are driven by positive selection". Journal of Molecular Evolution. 57 Suppl 1 (1): S154–64. Bibcode:2003JMolE..57S.154S. CiteSeerX 10.1.1.78.65. doi:10.1007/s00239-003-0022-3. PMID 15008412. S2CID 18051307.
- ^ Piganeau G, Eyre-Walker A (September 2003). "Estimating the distribution of fitness effects from DNA sequence data: implications for the molecular clock". Proceedings of the National Academy of Sciences of the United States of America. 100 (18): 10335–40. Bibcode:2003PNAS..10010335P. doi:10.1073/pnas.1833064100. PMC 193562. PMID 12925735.
- ^ Kimura M (February 1968). "Evolutionary rate at the molecular level". Nature. 217 (5129): 624–6. Bibcode:1968Natur.217..624K. doi:10.1038/217624a0. PMID 5637732. S2CID 4161261.
- ^ Akashi H (September 1999). "Within- and between-species DNA sequence variation and the 'footprint' of natural selection". Gene. 238 (1): 39–51. doi:10.1016/S0378-1119(99)00294-2. PMID 10570982.
- ^ Eyre-Walker A (October 2006). "The genomic rate of adaptive evolution". Trends in Ecology & Evolution. 21 (10): 569–75. doi:10.1016/j.tree.2006.06.015. PMID 16820244.
- ^ Gillespie JH (September 1984). "Molecular Evolution Over the Mutational Landscape". Evolution. 38 (5): 1116–1129. doi:10.2307/2408444. JSTOR 2408444. PMID 28555784.
- ^ Orr HA (April 2003). "The distribution of fitness effects among beneficial mutations". Genetics. 163 (4): 1519–26. doi:10.1093/genetics/163.4.1519. PMC 1462510. PMID 12702694.
- ^ Kassen R, Bataillon T (April 2006). "Distribution of fitness effects among beneficial mutations before selection in experimental populations of bacteria". Nature Genetics. 38 (4): 484–8. doi:10.1038/ng1751. PMID 16550173. S2CID 6954765.
- ^ Rokyta DR, Joyce P, Caudle SB, Wichman HA (April 2005). "An empirical test of the mutational landscape model of adaptation using a single-stranded DNA virus". Nature Genetics. 37 (4): 441–4. doi:10.1038/ng1535. PMID 15778707. S2CID 20296781.
- ^ Imhof M, Schlotterer C (January 2001). "Fitness effects of advantageous mutations in evolving Escherichia coli populations". Proceedings of the National Academy of Sciences of the United States of America. 98 (3): 1113–7. Bibcode:2001PNAS...98.1113I. doi:10.1073/pnas.98.3.1113. PMC 14717. PMID 11158603.
- ^ a b "Somatic cell genetic mutation". Genome Dictionary. Athens, Greece: Information Technology Associates. 30 June 2007. Archived from the original on 24 February 2010. Retrieved 6 June 2010.
- ^ "Compound heterozygote". MedTerms. New York: WebMD. 14 June 2012. Archived from the original on 4 March 2016. Retrieved 9 October 2015.
- ^ "RB1 Genetics". Daisy's Eye Cancer Fund. Oxford, UK. Archived from the original on 26 November 2011. Retrieved 9 October 2015.
- ^ "somatic mutation genetics". Encyclopædia Britannica. Archived from the original on 31 March 2017. Retrieved 31 March 2017.
- ^ Hartl L, Jones EW (1998). Genetics Principles and Analysis. Sudbury, Massachusetts: Jones and Bartlett Publishers. pp. 556. ISBN 978-0-7637-0489-6.
- ^ Milholland B, Dong X, Zhang L, Hao X, Suh Y, Vijg J (May 2017). "Differences between germline and somatic mutation rates in humans and mice". Nature Communications. 8: 15183. Bibcode:2017NatCo...815183M. doi:10.1038/ncomms15183. PMC 5436103. PMID 28485371.
- ^ Alberts B (2014). Molecular Biology of the Cell (6 ed.). Garland Science. p. 487. ISBN 9780815344322.
- ^ a b Chadov BF, Fedorova NB, Chadova EV (1 July 2015). "Conditional mutations in Drosophila melanogaster: On the occasion of the 150th anniversary of G. Mendel's report in Brünn". Mutation Research/Reviews in Mutation Research. 765: 40–55. doi:10.1016/j.mrrev.2015.06.001. PMID 26281767.
- ^ a b Landis G, Bhole D, Lu L, Tower J (July 2001). "High-frequency generation of conditional mutations affecting Drosophila melanogaster development and life span". Genetics. 158 (3): 1167–76. doi:10.1093/genetics/158.3.1167. PMC 1461716. PMID 11454765. Archived from the original on 22 March 2017. Retrieved 21 March 2017.
- ^ a b c d Gierut JJ, Jacks TE, Haigis KM (April 2014). "Strategies to achieve conditional gene mutation in mice". Cold Spring Harbor Protocols. 2014 (4): 339–49. doi:10.1101/pdb.top069807. PMC 4142476. PMID 24692485.
- ^ Spencer DM (May 1996). "Creating conditional mutations in mammals". Trends in Genetics. 12 (5): 181–7. doi:10.1016/0168-9525(96)10013-5. PMID 8984733.
- ^ Tan G, Chen M, Foote C, Tan C (September 2009). "Temperature-sensitive mutations made easy: generating conditional mutations by using temperature-sensitive inteins that function within different temperature ranges". Genetics. 183 (1): 13–22. doi:10.1534/genetics.109.104794. PMC 2746138. PMID 19596904.
- ^ den Dunnen JT, Antonarakis SE (January 2000). "Mutation nomenclature extensions and suggestions to describe complex mutations: a discussion". Human Mutation. 15 (1): 7–12. doi:10.1002/(SICI)1098-1004(200001)15:1<7::AID-HUMU4>3.0.CO;2-N. PMID 10612815. S2CID 84706224.
- ^ Jónsson H, Sulem P, Kehr B, Kristmundsdottir S, Zink F, Hjartarson E, et al. (September 2017). "Parental influence on human germline de novo mutations in 1,548 trios from Iceland". Nature. 549 (7673): 519–522. Bibcode:2017Natur.549..519J. doi:10.1038/nature24018. PMID 28959963. S2CID 205260431.
- ^ "Study challenges evolutionary theory that DNA mutations are random". U.C. Davis. Retrieved 12 February 2022.
- ^ Monroe, J. Grey; Srikant, Thanvi; Carbonell-Bejerano, Pablo; Becker, Claude; Lensink, Mariele; Exposito-Alonso, Moises; Klein, Marie; Hildebrandt, Julia; Neumann, Manuela; Kliebenstein, Daniel; Weng, Mao-Lun; Imbert, Eric; Ågren, Jon; Rutter, Matthew T.; Fenster, Charles B.; Weigel, Detlef (February 2022). "Mutation bias reflects natural selection in Arabidopsis thaliana". Nature. 602 (7895): 101–105. Bibcode:2022Natur.602..101M. doi:10.1038/s41586-021-04269-6. ISSN 1476-4687. PMC 8810380. PMID 35022609.
- ^ Doniger SW, Kim HS, Swain D, Corcuera D, Williams M, Yang SP, Fay JC (August 2008). Pritchard JK (ed.). "A catalog of neutral and deleterious polymorphism in yeast". PLOS Genetics. 4 (8): e1000183. doi:10.1371/journal.pgen.1000183. PMC 2515631. PMID 18769710.
- ^ Ionov Y, Peinado MA, Malkhosyan S, Shibata D, Perucho M (June 1993). "Ubiquitous somatic mutations in simple repeated sequences reveal a new mechanism for colonic carcinogenesis". Nature. 363 (6429): 558–61. Bibcode:1993Natur.363..558I. doi:10.1038/363558a0. PMID 8505985. S2CID 4254940.
- ^ Araten DJ, Golde DW, Zhang RH, Thaler HT, Gargiulo L, Notaro R, Luzzatto L (September 2005). "A quantitative measurement of the human somatic mutation rate". Cancer Research. 65 (18): 8111–7. doi:10.1158/0008-5472.CAN-04-1198. PMID 16166284.
- ^ "'Lifeless' prion proteins are 'capable of evolution'". Health. BBC News. London. 1 January 2010. Archived from the original on 25 September 2015. Retrieved 10 October 2015.
- ^ Sullivan AD, Wigginton J, Kirschner D (August 2001). "The coreceptor mutation CCR5Delta32 influences the dynamics of HIV epidemics and is selected for by HIV". Proceedings of the National Academy of Sciences of the United States of America. 98 (18): 10214–9. Bibcode:2001PNAS...9810214S. doi:10.1073/pnas.181325198. PMC 56941. PMID 11517319.
- ^ "Mystery of the Black Death". Secrets of the Dead. Season 3. Episode 2. 30 October 2002. PBS. Archived from the original on 12 October 2015. Retrieved 10 October 2015. 에피소드 배경
- ^ Galvani AP, Slatkin M (December 2003). "Evaluating plague and smallpox as historical selective pressures for the CCR5-Delta 32 HIV-resistance allele". Proceedings of the National Academy of Sciences of the United States of America. 100 (25): 15276–9. Bibcode:2003PNAS..10015276G. doi:10.1073/pnas.2435085100. PMC 299980. PMID 14645720.
- ^ Konotey-Ahulu F. "Frequently Asked Questions [FAQ's]". sicklecell.md. Archived from the original on 30 April 2011. Retrieved 16 April 2010.
- ^ Hughes D, Andersson DI (September 2017). "Evolutionary Trajectories to Antibiotic Resistance". Annual Review of Microbiology. 71: 579–596. doi:10.1146/annurev-micro-090816-093813. PMID 28697667.
- ^ Ségurel L, Bon C (August 2017). "On the Evolution of Lactase Persistence in Humans". Annual Review of Genomics and Human Genetics. 18: 297–319. doi:10.1146/annurev-genom-091416-035340. PMID 28426286.
- ^ a b c Bowler PJ (1992) [1983]. The Eclipse of Darwinism. p. 198.
- ^ Smocovitis VB (1996). "Unifying biology: the evolutionary synthesis and evolutionary biology". Journal of the History of Biology. Princeton University Press. 25 (1): 1–65. doi:10.1007/bf01947504. ISBN 978-0-691-03343-3. LCCN 96005605. OCLC 34411399. PMID 11623198. S2CID 189833728.
- ^ Hallgrímsson B, Hall BK (2011). Variation: A Central Concept in Biology. Academic Press. p. 18.
- ^ Sewall Wright(1984)진화와 모집단의 유전학: 유전학 및 바이오메트릭 재단 제1권시카고 대학교 출판부 10페이지
- ^ De Vries H (1905). Species and Varieties: Their Origin by Mutation.
- ^ Bateson W (1894). Materials for the Study of Variation, Treated with Especial Regard to Discontinuity in the Origin of Species.
- ^ Punnett RC (1915). Mimicry in Butterflies. Cambridge University Press.
- ^ Mayr E (2007). What Makes Biology Unique?: Considerations on the Autonomy of a Scientific Discipline. Cambridge University Press.
- ^ a b Provine WB (2001). The Origins of Theoretical Population Genetics, with a new afterword. University of Chicago Press, Chicago. pp. 56–107.
- ^ a b Stoltzfus A, Cable K (2014). "Mendelian-mutationism: the forgotten evolutionary synthesis". Journal of the History of Biology. 47 (4): 501–46. doi:10.1007/s10739-014-9383-2. PMID 24811736. S2CID 23263558.
- ^ Hull DL (1985). "Darwinism as an historical entity: A historiographic proposal". In Kohn D (ed.). The Darwinian Heritage. Princeton University Press. pp. 773–812. ISBN 9780691024141.
- ^ Yule GU (1902). "Mendel's Laws and their probable relations to inter-racial heredity". New Phytologist. 1 (10): 226–227. doi:10.1111/j.1469-8137.1902.tb07336.x.
- ^ Gould SJ (1982). The uses of heresey; an introduction to Richard Goldschmidt's The Material Basis of Evolution. Yale University Press. pp. xiii–xlii. ISBN 0300028237.
- ^ Ruse M (1996). Monad to man: the Concept of Progress in Evolutionary Biology. Harvard University Press. pp. 412–413. ISBN 978-0-674-03248-4.
- ^ Stoltzfus A (2014). "In search of mutation-driven evolution". Evolution & Development. 16: 57–59. doi:10.1111/ede.12062.
- ^ Futuyma DJ (2015). Serrelli E, Gontier N (eds.). Can Modern Evolutionary Theory Explain Macroevolution? (PDF). Macroevolution. Springer. pp. 29–85. Archived from the original (PDF) on 23 February 2020. Retrieved 31 October 2017.
( )
- Jones S, Woolfson A, Partridge L (6 December 2007). "Genetic Mutation". In Our Time. BBC Radio 4. Retrieved 18 October 2015.
- Liou S (5 February 2011). "All About Mutations". HOPES. Huntington's Disease Outreach Project for Education at Stanford. Retrieved 18 October 2015.
- "Locus Specific Mutation Databases". Leiden, the Netherlands: Leiden University Medical Center. Retrieved 18 October 2015.
- "Welcome to the Mutalyzer website". Leiden, the Netherlands: Leiden University Medical Center. Retrieved 18 October 2015. – Mutalyzer 웹사이트.