호몰로 재조합
Homologous recombination동질 재조합은 유전 정보가 두 개의 이중 가닥 또는 단일 가닥 핵산의 유사하거나 동일한 두 분자 사이에서 교환되는 유전적 재조합의 한 유형이다(대개 세포 유기체에서와 같은 DNA이지만 바이러스에서도 RNA일 수 있다).이것은 DNA의 양쪽 가닥, 즉 DSB(이중 스트랜드 파손)에서 발생하는 유해한 균열을 균일재조합수리(HRR)라고 하는 과정에서 세포가 정확하게 치료하기 위해 널리 사용된다.[1]동질 재조합은 또한 감수분열 동안 DNA 서열의 새로운 조합을 만들어내는데, 이는 진핵생물이 동물의 정자와 난자 세포와 같은 생식세포 세포를 만드는 과정이다.DNA의 이러한 새로운 조합은 자손의 유전적 변이를 나타내며, 이는 진화 과정 동안 개체군이 적응할 수 있게 한다.[2]균질 재조합은 다른 종과 박테리아와 바이러스 사이의 유전 물질을 교환하기 위해 수평 유전자 전달에도 사용된다.
균질 재조합은 유기체와 세포 유형에 따라 매우 다양하지만, 이중 가닥 DNA(dsDNA)의 경우 대부분의 형태는 동일한 기본 단계를 포함한다.이중 끈이 끊어진 후, 절개라는 과정에서 5의 끝부분의 DNA 부분이 잘려 나간다.이어지는 Strand 침공 단계에서, 부서진 DNA 분자의 3분의 1의 끝부분에 돌출한 후, 깨지지 않은 유사하거나 동일한 DNA 분자를 "침입"한다.Strand 침공 후 추가 사건 순서는 아래에서 논의되는 두 가지 주요 경로(모델 참조) 즉 DSBR(이중 스트랜드 파손 수리) 경로 또는 SDSA(합성 의존 스트랜드 어닐링) 경로 중 하나를 따를 수 있다.DNA 수리 중에 발생하는 동질 재조합은 비크로스오버 제품을 유발하는 경향이 있으며, 실제로 손상된 DNA 분자는 이중 가닥이 깨지기 전에 존재했던 것처럼 회복된다.
동질 재조합은 DNA와 RNA 바이러스뿐만 아니라 생명의 세 영역 모두에 걸쳐 보존되어 있어 거의 보편적인 생물학적 메커니즘임을 시사한다.다양한 진핵 미생물의 집단인 양성자에서 동질 재조합을 위한 유전자의 발견은 진핵생물의 진화 초기에 감수분열이 나타났다는 증거로 해석되어 왔다.그들의 기능장애는 여러 종류의 암에 대한 민감성 증가와 강하게 연관되어 왔기 때문에, 동질 재조합을 용이하게 하는 단백질은 활발한 연구의 주제들이다.대상 유기체에 유전적 변화를 도입하는 기술인 유전자 타겟팅에도 동질 재조합이 사용된다.이 기술을 그들의 발전을 위해 마리오 카페키, 마틴 에번스, 올리버 스미디즈와, Capecchi[3]과 Smithies[4]독립적으로, 애플리케이션 배아 줄기 세포 mouse에서 유니폼 homologous을 포함한 하지만 이 고도로 보호되기 메커니즘이 양측 파대 수리 모델을 기초로, 발견한 2007년 노벨 생리 의학 상을 수여하였다.int변형된 DNA의 소거(gene therapy)는 오르-위버, 스조스택, 로스슈타인의 플라스미드 실험에서 처음 나타났다.[5][6][7]1970년대부터 1980년대까지 ir-비방사선을[8] 이용한 플라스미드 유도 DSB를 연구한 결과, 효모보다 비호몰로 재조합이 빈번한 포유류 세포의 유전공학을 위해 염색체를 절단하기 위해 엔도뉴클레아제(예: I-SceI)를 사용한 후기 실험으로 이어졌다.[9]
역사와 발견

1900년대 초 윌리엄 베이슨과 레지날드 푸넷은 1860년대에 그레고르 멘델이 원래 설명한 상속의 원칙 중 하나에 예외를 발견했다.예를 들어 고양이의 털 색깔과 꼬리 길이가 서로 독립적으로 유전된다는 멘델의 생각과는 대조적으로, 베이트슨과 푸넷은 신체적 특징과 관련된 특정 유전자가 함께 유전되거나 유전적으로 연관될 수 있다는 것을 보여주었다.[10][11]1911년, 토마스 헌트 모건은 연결된 형질이 때로는 별도로 유전될 수 있다는 것을 관찰한 후,[12] 연결된 유전자들 중 하나가 물리적으로 다른 염색체로 넘어가는 링크된 유전자들 사이에서 "크로스소버"가 발생할 수 있다고 제안했다.20년 후, 바바라 맥클린톡과 해리엇 크리톤은 정자와 난자가 만들어지는 세포 분열 과정인 [13][14]감수분열 중에 염색체 교차 현상이 일어난다는 것을 증명했다.맥클린톡의 발견과 같은 해 안에 커트 스턴은 백혈구와 유사분열로 분열되는 피부 세포와 같은 체세포에서도 ("재조합"이라고 불리는) 교차 현상이 발생할 수 있다는 것을 보여주었다.[13][15]
1947년, 미생물학자 조슈아 레더버그는 이항분열을 통해 무성으로만 번식한다고 가정되어 온 박테리아가 유전적 재조합이 가능하다는 것을 보여주었는데, 이는 성생식과 더욱 유사하다.이 작품은 대장균을 유전학의 모범생물로 확립했고,[16] 레더버그가 1958년 노벨 생리의학상을 수상하는 데 도움을 주었다.[17]곰팡이 연구를 기반으로 한 1964년 로빈 홀리데이는 감수분열 재조합 모델을 제안했는데, 이 모델은 홀리데이 결합을 통해 염색체들 간의 물질 교환을 포함한 과정이 어떻게 작동할 수 있는지에 대한 핵심 세부사항을 소개했다.[18]1983년에 잭 소스타크와 동료들은 현재 DSBR 경로로 알려진 모델을 제시했는데, 이 모델은 홀리데이 모델에 의해 설명되지 않은 관측치를 설명하였다.[18][7]다음 10년 동안, 드로소필라, 싹트기 효모, 포유류 세포에서의 실험은 항상 홀리데이 결합에 의존하지 않는 SDSA 경로라고 불리는 다른 호몰로 재조합의 모델을 출현시켰다.[18]
그 과정에 관여하는 단백질을 식별하고 그 메커니즘을 결정하는 이후의 많은 작업은 제임스 해버, 패트릭 성, 스테판 코왈츠키 등을 포함한 많은 사람들에 의해 수행되었다.
eukaryotes에서
동질 재조합(HR)은 식물, 동물, 곰팡이, 양성자와 같은 진핵생물의 세포분열에 필수적이다.유사분열을 통해 분열되는 세포에서 균질 재조합은 전리방사선이나 DNA 손상 화학물질에 의해 야기되는 DNA의 이중스트랜드를 복구한다.[19]이런 이중 가닥이 손상되지 않은 채 방치되면 체세포에서 염색체가 대규모로 재배열돼 결국 암으로 이어질 수 있다.[20][21]
DNA를 수리하는 것 외에도, 균질 재조합은 세포가 감수분열에서 분열되어 동물의 sperm 또는 난자 세포, 식물의 꽃가루 또는 난자, 곰팡이의 포자 등 특수한 생식세포가 될 때 유전적 다양성을 생성하는데 도움을 준다.그것은 유사하지만 동일하지 않은 DNA의 영역이 동질 염색체들 사이에서 교환되는 염색체 교차을 촉진함으로써 그렇게 한다.[22][23]이것은 새로운, 어쩌면 유익한 유전자의 조합을 만들어내는데, 이것은 자손들에게 진화적인 이점을 줄 수 있다.[24]염색체 크로스오버는 종종 스포11이라는 단백질이 DNA에서 표적형 이중 가닥을 깨뜨릴 때 시작된다.[25]이러한 부위는 염색체에 무작위로 위치하지 않는다. 대개 유전자간 촉진자 지역 및 GC가 풍부한 도메인[26] 우선이다. 이러한 이중 가닥 파괴 부위는 종종 재결합 핫스팟에서 발생하는데, 길이는 약 1,000–2,000 염기쌍이고 재결합률이 높은 염색체 지역이다.같은 염색체에 있는 두 유전자 사이에 재조합 핫스팟이 없다는 것은 종종 그러한 유전자들이 동일한 비율로 미래 세대에 의해 유전될 것이라는 것을 의미한다.이것은 감수분열 동안 독립적으로 분석되는 유전자로부터 기대되는 것보다 더 큰 두 유전자 사이의 연관성을 나타낸다.[27]
유사 세포 주기 내의 타이밍
이중 스트랜드 파손은 균질 재조합, 중합효소 세타 매개 엔드 결합(TMEJ) 또는 비호몰 엔드 결합(NHEJ)을 통해 수리할 수 있다.[28]NHEJ는 동질 재조합과 달리 수리를 안내하는 데 긴 동질 시퀀스가 필요하지 않은 DNA 수리 메커니즘이다.균질 재조합 또는 NHEJ를 사용하여 이중 스트랜드 파손을 수리하는지는 주로 셀 사이클의 단계에 의해 결정된다.동질 재조합은 세포가 유사분열(M상)에 들어가기 전에 DNA를 복구한다.그것은 DNA 복제 기간과 직후, 세포 주기의 S와 G2 단계에서, 자매 크로마티드를 더 쉽게 이용할 수 있을 때 발생한다.[29]다른 염색체와 유사하지만 알레르기가 다른 경우가 많은 동음이의 염색체에 비해 자매 크로마티드는 주어진 염색체의 동일한 복제품이기 때문에 동음이의 재조합에 이상적인 템플릿이다.균질 템플릿을 사용할 수 없거나 균질 재조합의 결함으로 인해 템플릿에 접근할 수 없는 경우, 파단은 셀 주기의 S 및 G 단계에서2 TMEJ를 통해 수리된다.동질 재조합과 TMEJ와는 대조적으로, NHEJ는 세포가 성장하지만 아직 분열할 준비가 되어 있지 않은 세포 주기의1 G 단계에서 우세하다.G1 단계 이후 덜 자주 발생하지만, 셀 주기 동안 최소한 어느 정도의 활동을 유지한다.세포 주기 전체에 걸쳐 동질 재조합과 NHEJ를 조절하는 메커니즘은 종마다 크게 다르다.[30]
인산염 그룹을 (즉, 인산염)에 첨가하여 다른 단백질의 활동을 수정하는 사이클린 의존성 키나제(CDK)는 진핵생물에서 동질 재조합의 중요한 규제기관이다.[30]DNA 복제가 싹트는 효모에서 시작되면, 사이클린 의존성 키나제 Cdc28은 Sae2 단백질을 인산화하여 동질 재조합을 시작한다.[31]인산염의 첨가로 그렇게 활성화 된 Sae2는 DNA의 이중 가닥이 깨진 근처에서 깨끗한 절단 작업을 하게 한다.이 절단을 담당하는 엔도뉴클레스가 Sae2 자체인지 또 다른 단백질인 Mre11인지는 확실하지 않다.[32]이를 통해 MRX 콤플렉스로 알려진 Mre11을 포함한 단백질 복합체가 DNA에 결합할 수 있게 되고, 두 DNA 분자 사이에서 물질을 교환하는 일련의 단백질 구동 반응을 시작한다.[33]
염색체의 역할
진핵 DNA를 크로마틴으로 포장하는 것은 그들의 행동 현장에 효소를 모아야 하는 모든 DNA 기반 과정에 장벽을 제공한다.균질 재조합(HR) DNA 수리를 허용하려면 염색질을 개조해야 한다.eukaryotes에서 ATP 의존성 크로마틴 리모델링 단지와 히스톤 수정 효소는 이러한 리모델링 과정을 수행하기 위해 채택된 두 가지 주요 요인이다.[34]
크로마틴 이완은 DNA 손상 부위에서 빠르게 일어난다.[35]초기 단계 중 하나인 응력활성화 단백질 키나아제, c-Jun N-terminal kinase(JNK), 인산염은 이중 가닥 파손이나 기타 DNA 손상에 대응하여 세린 10에 SIRT6를 만든다.[36]이러한 변환 후 수정은 DNA 손상 부위에 SIRT6의 동원을 용이하게 하며, DNA 손상 부위에 폴리(ADP-리보스) 중합효소 1(PARP1)을 효율적으로 채용하고 DSB의 효율적인 수리를 위해 필요하다.[36]PARP1 단백질은 1초 이내에 DNA 손상 부위에 나타나기 시작하며, 손상 발생 후 1.6초 이내에 최대 축적량이 절반에 이른다.[37]다음으로 염색질 리모델링기 Alc1은 PARP1 작용 제품에 빠르게 부착되며, 폴리-ADP 리보스 체인이며, Alc1은 손상 발생 후 10초 이내에 DNA 손상 도착을 완료한다.[35]알c1의 작용으로 추측되는 최대 염색체 이완의 약 절반은 10초 정도 발생한다.[35]이를 통해 13초 이내에 DNA 복구 효소 MRE11을 모집할 수 있다.[37]
γH2AX, H2AX의 인산화 형태는 DNA 이중 가닥이 깨진 후 염색질 감응으로 이어지는 초기 단계에도 관여한다.히스톤 변종 H2AX는 인간 염색질에서 H2A 히스톤의 약 10%를 구성한다.[38]γH2AX(세린 139에 H2AX인산화효소)는 세포 조사(DNA 이중스트랜드와 파손형성) 후 20초 이내에 검출할 수 있으며, 1분 이내에 최대 2H2AX의 절반의 축적량이 발생한다.[38]인산염 hH2AX를 함유한 염색질의 범위는 DNA 이중 스트랜드 파단 부위에서 약 200만 염기쌍이다.[38]γH2AX 자체는 염색질 감응을 일으키지 않지만, 조사 후 30초 이내에 hH2AX와 연계하여 RNF8 단백질을 검출할 수 있다.[39]RNF8은 뉴클레오솜 리모델링과 탈세틸라제 복합체 NuRD의 구성요소인 [40]CHD4와의 후속적 상호작용을 통해 광범위한 염색질 감응을 매개한다.
DNA 손상에 이어 DNA 수리에 이어 이완까지 거쳐 약 20분 후 염색질은 손상 전 수준에 가까운 응축 상태로 회복된다.[35]
감수분열 중 호몰로 재조합
척추동물에서 재조합이 발생하는 위치는 아연 핑거 배열에 의해 특정 시퀀스를 인식하는 단백질인 PRDM9의 결합 위치에 의해 결정된다.[41]이러한 현장에서 또 다른 단백질인 SPO11 카탈리시스(SPO11 catalyses)는 DSB(double strand breaks)를 재조합하는데, 그 중 일부는 동질 염색체와의 재조합에 의해 수리된다.PRDM9는 H3K4me3와 H3K36me3 히스톤 메틸화 마크를 결합 사이트에 모두 퇴적시키며, 이러한 메틸전달효소 활동은 DSB 포지셔닝에서 그것의 역할을 위해 필수적이다.Following their formation, DSB sites are processed by resection, resulting in single-stranded DNA (ssDNA) that becomes decorated with DMC1. From mid-zygotene to early pachytene, as part of the recombinational repair process, DMC1 dissociates from the ssDNA and counts decrease until all breaks (except those on the XY chromosomes) are repaired at la티 파키테네이 과정에는 PRDM9의 이중 히스톤 마크에 의해 직접 배치된 최초의 단백질인 ZCWPW1을 포함한 몇 가지 다른 단백질들이 관여하고 있다.ZCWPW1은 위치가 아닌 동음이의 DSB 수리에 중요하다.
모델
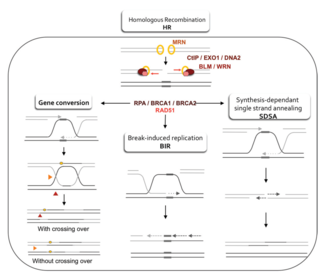
균질 재조합이 DNA에서 이중 가닥을 어떻게 수리하는지에 대한 두 가지 주요 모델은 이중 가닥 파손 수리(DSBR) 경로(double strand break repair, double Hollyday 접합 모델이라고도 함)와 합성 의존적 Strand annealing(SDSA) 경로다.[43]그 두 경로는 처음 몇 단계에서 비슷하다.이중 끈이 끊어진 후, MRX 콤플렉스(인간 내 MRN 콤플렉스)는 끊어진 양쪽의 DNA에 결합한다.다음으로 절제가 이루어지는데, 이 절제가 5의 끝부분의 DNA를 잘라낸다.이것은 두 가지 뚜렷한 단계로 일어난다: 첫째, MRX 콤플렉스는 Sae2 단백질을 모집하고, 이 두 단백질은 균열의 양쪽에 있는 5' 끝부분을 잘라내어 짧은 3'의 단일 가닥과 DNA의 돌출부를 만들어낸다; 둘째 단계에서는 5'→3'의 절제술은 Sgs1 헬리코아제와 Exo1과 dna2 핵에 의해 계속된다.헬리코아제로서 Sgs1은 이중 가닥 DNA를 '풀지'하고, Exo1과 dna2의 누클레스 활성은 Sgs1이 생산한 단일 가닥 DNA를 절단할 수 있게 한다.[31]
단일 가닥 DNA에 친화력이 높은 RPA 단백질은 3의 돌출부를 묶는다.[44]그 과정을 중재하는 몇 개의 다른 단백질들의 도움으로 RDPA로 코팅된 DNA의 단일 가닥에 Rad51 단백질(그리고 Dmc1, 감수분열)이 핵산과 단백질의 필라멘트를 형성한다.그리고 나서 이 뉴클레오프로테인 필라멘트는 3' 오버행의 그것과 유사한 DNA 서열을 찾기 시작한다.그러한 시퀀스를 발견한 후, 단일 가닥의 뉴클레오프로틴 필라멘트는 Strand 침공이라고 불리는 과정에서 유사하거나 동일한 수신자 DNA 듀플렉스로 이동한다.유사분열을 통해 분열되는 세포에서 받는 DNA 듀플렉스(dna duplex)는 일반적으로 자매 크로마티드로 손상된 DNA 분자와 동일하며 수리를 위한 템플릿을 제공한다.그러나 감수분열에서 수령인 DNA는 유사하지만 반드시 동일하지는 않은 동질 염색체에서 오는 경향이 있다.[43]변위 루프(D-루프)는 침입한 3' 오버행 스트랜드와 동질 염색체 사이에 가닥이 침입하는 동안 형성된다.Strand 침공 후 DNA 중합효소는 새로운 DNA를 합성하여 침공 3' Strand의 끝을 확장한다.이것은 D-루프를 홀리데이 분기점이라고 알려진 십자형 구조로 바꾼다.이후 침입한 스트랜드(즉, 원래 3의 돌출부 중 하나)에서 더 많은 DNA 합성이 일어나, 스트랜드 침공 중에 변위된 동질 염색체의 스트랜드가 효과적으로 복원된다.[43]
DSBR 경로
절제, Strand 침입 및 DNA 합성 단계 이후 DSBR 및 SDSA 경로가 구별된다.[43]DSBR 경로 역시 두 번째 3' 오버행(스트랜드 침입에 관여하지 않은 것)이 동질 염색체와의 홀리데이 접점을 형성한다는 점에서 독특하다.그런 다음 이중 홀리데이 결합은 제한 엔도뉴클레아제(ndonuclease)의 일종인 DNA 가닥 하나만 자르는 재조합 제품으로 전환된다.DSBR 경로로 인해 간혹 비크로스오버 제품이 발생할 수 있지만, 일반적으로 교차되지 않는 제품이 발생할 수 있다. 분리된 기증자 로키에서 시퀀스를 수집하는 부서진 DNA 분자의 능력은 염색체 이벤트의 플라스미드 또는 내분자 유도를 사용하는 유사 싹트기 효모에서 나타났다.[45][46]이러한 염색체 교차 경향 때문에 DSBR 경로는 감수분열 동안 교차 호몰로 재조합이 어떻게 발생하는지를 보여주는 유력한 모델이다.[22]
DSBR 경로의 재결합이 염색체 교차 여부를 결정하는 것은 이중 홀리데이 분기점을 절단하는 방법 또는 "해결" 방법에 의해 결정된다.염색체 교차점은 한 홀리데이 분기점이 교차 가닥에서 절단되고 다른 홀리데이 분기점은 비 교차 가닥에서 절단되는 경우(그림 5에서 한 홀리데이 분기점에서 수평 보라색 화살촉을 따라 다른 한 홀리데이 분기점에서 수직 주황색 화살촉을 따라 절단된다).또는 두 개의 Hollyday 접합부를 교차 가닥(그림 5의 양쪽 Hollyday 접합부의 수평 보라색 화살촉을 따라)에 절단하면 교차되지 않는 염색체가 생성된다.[47]
SDSA 경로
SDSA 경로를 통한 호몰로 재조합은 유사분열과 감수분열을 통해 분열되어 비크로스오버 제품이 되는 세포에서 발생한다.이 모델에서 침입한 3' 가닥은 DNA 중합효소에 의해 수신자 DNA 듀플렉스를 따라 연장되며, 기증자와 수령자 DNA 분자 사이의 홀리데이 접합부가 분기 이동이라는 과정에서 미끄러지면서 방출된다.새로 합성된 침공 가닥의 3' 끝부분은 보완적인 염기쌍을 통해 손상된 염색체에서 다른 3'의 돌출부를 제거할 수 있다.가닥이 잡힌 후 DNA의 작은 플랩이 남아있을 수 있다.그러한 플랩은 모두 제거되고 SDSA 경로로 남은 단일 가닥 틈새의 재봉합(레이징이라고도 함)[48]으로 마무리된다.
유사분열 중 DNA 이중 가닥 파손을 수리하기 위한 주요 동질 재결합 경로는 (DSBR 경로가 아닌) SDSA 경로로 나타난다.[49]SDSA 경로에 의해 비크로스오버 재조합물이 생성된다(그림 5).감수분열 동안 비크로스오버 재조합도 자주 발생하며 이러한 재조합은 주로 SDSA 경로에 의해서도 발생하는 것으로 보인다.[49][50]감수분열 중에 발생하는 비크로스오버 재결합 사건은 DNA 이중 가닥 손상 또는 다른 유형의 DNA 손상 수리 사례를 반영할 가능성이 높다.
SSA 경로
균질 재조합의 단일 가닥(SSA) 경로로 두 반복 시퀀스 사이에서 이중 가닥을 수선한다.SSA 경로에는 동질 재조합의 DSBR 또는 SDSA 경로와 같이 유사하거나 동일한 별도의 DNA 분자가 필요하지 않다는 점에서 독특하다.그 대신 SSA 경로에는 단일 DNA 듀플렉스만 필요하며, 반복 시퀀스를 동질 재조합이 수리에 필요한 동일한 시퀀스로 사용한다.경로의 개념은 비교적 간단하다: 동일한 DNA 이중 가닥의 두 가닥이 이중 가닥이 끊어진 후, 그 결과로 생긴 두 가닥의 돌출부가 서로 정렬되고 안느러미가 되어 DNA를 연속 이중으로 복원한다.[48][51]
이중 가닥 주변의 DNA가 잘려나가면서 생산되는 단일 가닥 3의 오버행은 RPA 단백질로 코팅돼 3의 오버행이 스스로 달라붙는 것을 막는다.[52]그런 다음 Rad52라는 단백질은 각 반복 시퀀스를 브레이크의 양쪽에 결합하고, 이를 정렬하여 두 개의 상호 보완적인 반복 시퀀스를 안네알로 사용할 수 있게 한다.[52]어닐링이 완료된 후, 3의 돌출부의 잔여 비호몰성 플랩은 Saw1과 Slx4 단백질에 의해 플랩으로 가져오는 Rad1/Rad10으로 알려진 핵물질 세트에 의해 잘려진다.[52][53]새로운 DNA 합성은 어떤 틈새에서도 채워지며, 두 개의 연속 가닥으로 DNA 복층을 회복시킨다.[54]두 개의 반복 중 하나와 마찬가지로 반복 사이의 DNA 염기서열은 항상 손실된다.SSA 경로로 인해 유전 물질이 삭제되기 때문에 돌연변이 유발 물질로 간주된다.[48]
BIR 경로
DNA 복제 중 DNA 헬리코아제가 템플릿 가닥의 지퍼를 열 때 복제 포크에서 이중 스트랜드 파손이 발생할 수 있다.이러한 결함은 균질 재조합의 BIR 경로에서 수리된다.BIR 경로의 정확한 분자 메커니즘은 여전히 불명확하다.제안된 세 가지 메커니즘은 초기 단계로서 Strand 침투를 가지고 있지만, D-루프의 이동과 재조합의 이후의 단계를 모델링하는 방법에는 차이가 있다.[55]
또한 BIR 경로는 텔로머레이스가 없는 경우(또는 협력하는 경우) 텔로메르(eukaryotic 염색체 끝의 DNA 영역)의 길이를 유지하는 데 도움이 될 수 있다.텔로머라아제 효소의 작업복사가 없으면 일반적으로 말단소립은 각 유사분열 주기에 따라 짧아지고, 이는 결국 세포분열을 막고 노쇠로 이어진다.말단소화효소가 돌연변이를 통해 비활성화된 신생 효모세포에서는 두 가지 유형의 "생존자" 세포가 BIR 경로를 통해 말단소화를 연장함으로써 예상보다 오래 노화를 피하는 것이 관찰되었다.[55]
말단소립 길이를 유지하는 것은 암의 주요 특징인 세포 불멸화에 매우 중요하다.대부분의 암은 텔로머레이스를 상향 조절하여 텔로미어를 유지한다.그러나 인간 암의 여러 가지 유형에서, BIR과 같은 경로는 말단소립 유지의 대안적 메커니즘으로 작용하여 일부 종양을 지탱하는 데 도움이 된다.[56]이러한 사실은 과학자들이 그러한 재조합 기반의 말단소체 유지 메커니즘이 말단소체 억제제와 같은 항암제를 좌절시킬 수 있는지 여부를 조사하도록 이끌었다.[57]
박테리아에
균질 재조합은 박테리아에서 중요한 DNA 회복 과정이다.DNA 손상을 수리하고 진핵 게놈의 다양성을 가져오는 감수성 재조합과는 상당한 차이가 있지만 세균 개체군의 유전적 다양성을 생산하는 데도 중요하다.균질 재조합은 가장 많이 연구되어 왔고 대장균에 대해 가장 잘 이해된다.[59]박테리아에서 이중 가닥과 DNA 균열은 균질 재조합의 RecBCD 경로에 의해 수리된다.단일 가닥 간극으로 알려진 두 개의 DNA 가닥 중 하나에서만 발생하는 파손은 RecF 경로에 의해 수리되는 것으로 생각된다.[60]RecBCD와 RecF 경로 모두 두 개의 교차 DNA 분자 간에 단일 DNA 가닥이 교환되는 분기 이동과 두 개의 교차 DNA 분자가 분리되어 정상적인 이중 변형 상태로 복원되는 분해능으로 알려진 일련의 반응을 포함한다.
RecBCD 경로


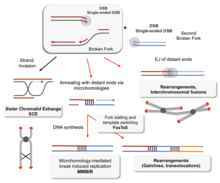
RecBCD 경로(RecBCD pathway)는 DNA의 이중 가닥 파손을 복구하기 위해 많은 박테리아에서 사용되는 주요 재조합 경로로 단백질은 광범위한 박테리아에서 발견된다.[63][64][65]이러한 이중 가닥 균열은 화학 돌연변이뿐만 아니라 자외선과 다른 방사선에 의해 발생할 수 있다.이중 가닥 균열은 단일 가닥의 흠집이나 틈새를 통한 DNA 복제에 의해서도 발생할 수 있다.그러한 상황은 붕괴된 복제 포크라고 알려진 것을 야기하며, RecBCD 경로를 포함한 몇 가지 동음이의 재결합 경로에 의해 고정된다.[66]
이 경로에서 3단위 효소 복합체 RecBCD는 이중 스트랜드 DNA의 파단부위 또는 거의 무딘 단부에 결합하여 재조합을 시작한다. RecBCD가 DNA 끝을 결합한 후, RecB와 RecD 하위 단위는 헬리코아제 활동을 통해 DNA 이중부의 지퍼를 풀기 시작한다.RecB 하위 유닛은 또한 Nuclease 도메인을 가지고 있는데, 이 영역은 지퍼를 해제하는 과정에서 나오는 DNA의 한 가닥을 절단한다.이러한 지퍼링은 RecBCD가 Chi 사이트로 알려진 특정 뉴클레오티드 시퀀스(5'-GCTGGGG-3)를 만날 때까지 계속된다.[65]
Chi 사이트를 접하면 RecBCD 효소의 활동이 급격히 변화한다.[64][61][67]DNA를 풀면 몇 초 동안 멈췄다가 초기 속도의 절반 정도에서 재개된다.이는 RecB 헬리코아제가 느릴수록 DNA가 Chi보다 Chi 다음으로 분해되기 때문이며, 즉 RecD 헬리코아제가 먼저 분해되기 때문일 것이다.[68][69]Chi 사이트의 인식은 또한 RecBCD 효소를 변화시켜 그것이 Chi와 함께 DNA 가닥을 자르고 새로 생성된 3의 끝을 가진 단일 가닥의 DNA에 복수의 RecA 단백질을 싣기 시작한다.그 결과 RecA-코팅 뉴클레오프로틴 필라멘트는 동질 염색체에서 유사한 DNA 서열을 찾아낸다.이 검색 과정은 DNA 이중의 확장을 유도하여 동종학 인식(정합성 교정이라고[70][71][72] 하는 메커니즘)을 강화한다.그러한 시퀀스를 발견하자마자, 단일 가닥의 핵단백질 필라멘트는 Strand 침공이라고 불리는 과정에서 동음이의 수신자 DNA 듀플렉스 안으로 이동한다.[73]침입 3' 오버행은 수신자 DNA 복층의 가닥 중 하나를 이동시켜 D-루프를 형성하게 한다.D루프가 잘리면 또 다른 가닥의 스와핑이 홀리데이 분기점이라는 십자형 구조를 형성한다.[65]RubABC 또는 RecG의 어떤 조합에 의한 Hollyday 접속점의 분해능은 상호작용을 하는 두 개의 DNA 분자가 유전적으로 다른 경우, 상호유전유형을 가진 두 개의 재조합 DNA 분자를 생성할 수 있다.또는 치 근처에 있는 침공 3의 끝부분은 DNA 합성을 프라이밍하고 복제 포크를 형성할 수 있다.이러한 유형의 분해능은 오직 한 가지 유형의 재조합(비호환산)만 생성한다.
RecF 경로
박테리아는 DNA의 단일 가닥 간극을 복구하기 위해 균질 재조합의 RecF 경로를 이용하는 것으로 보인다.SBCCD 및 ExoI 핵들을 비활성화하는 돌연변이와 추가 돌연변이에 의해 RecBCD 경로가 비활성화될 때, RecF 경로도 DNA 이중 가닥을 복구할 수 있다.[74]RecF 경로에서 RecQ 헬리코아제는 DNA를 풀고 RecJ nuclease는 5인치 끝의 가닥을 분해하여 3인치 끝의 가닥을 그대로 유지한다.RecA 단백질은 이 가닥에 결합되어 RecF, RecO, RecR 단백질의 도움을 받거나 그들에 의해 안정화된다.그런 다음 RecA 뉴클레오프로테인 필라멘트는 동질의 DNA를 검색하고 동질의 DNA에서 거의 동일한 가닥 또는 거의 동일한 가닥과 위치를 교환한다.
비록 초기 단계에 관여하는 단백질과 특정한 메커니즘은 다르지만, 두 경로 모두 3'의 끝을 가진 단일 가닥 DNA와 스트랜드 침투를 위한 RecA 단백질을 필요로 한다는 점에서 유사하다.홀리데이 분기점이 한 방향으로 미끄러지는 지점 이동 단계와 홀리데이 접합부가 효소에 의해 갈라지는 분해능도 유사하다.[75][76]대안적인 비회수 유형의 분해능도 어느 경로에 의해서도 발생할 수 있다.
분기 마이그레이션
스트랜드 침공 직후 홀리데이 분기점은 분기 이동 과정에서 연결된 DNA를 따라 이동한다.이 홀리데이 분기점 운동에서 두 동질의 DNA 복층 사이의 염기쌍이 교환된다.가지 이주를 촉진하기 위해 루바 단백질이 먼저 인지하여 홀리데이 접합부에 결합하고 루바브 단백질을 모집하여 루바브 복합체를 형성한다.각각 고리 모양의 ATPase를 형성하는 RuvB 단백질 2세트가 Hollyday 분기점 맞은편에 탑재되어 분기 이주를 위한 힘을 제공하는 트윈 펌프 역할을 한다.RuvB의 두 링 사이에서, 두 세트의 RuvA 단백질은 각 RuvA의 세트 사이에 접합부의 DNA가 끼워지도록 Hollyday 분기점 중앙에 모인다.두 DNA 듀플렉스인 "도너"와 "수취적" 듀플렉스 가닥은 한 듀플렉스로부터 다른 듀플렉스까지 단백질에 의해 인도될 때 루바 표면에서 풀린다.[77][78]
해상도
재조합의 해결 단계에서는 스트랜드 침공 과정에 의해 형성된 홀리데이 결합을 절단하여 두 개의 분리된 DNA 분자를 복원한다.이 갈라짐은 루바브 콤플렉스가 루바브크 콤플렉스를 형성하는 루바브와 상호 작용하여 이루어진다.RuvC는 변질된 시퀀스 5'-(A/T)TT(G/C)-3'을 자르는 내분비선이다.이 염기서열은 DNA에서 자주 발견되는데, 약 64개의 뉴클레오티드에 한 번꼴이다.[78]RuvC는 DNA를 덮는 두 개의 RuvA 테트라머 중 하나를 교체하여 Hollyday 분기점에 접근할 수 있다.[77]재조합은 RuvC가 Hollyday 분기점을 어떻게 분할하느냐에 따라 "분할" 또는 "패치" 제품이 된다.[78]스플라이스 제품은 교차 생산물로서, 재조합 현장 주위에 유전 물질의 재배열이 있다.반면 패치 제품은 이러한 재배열이 없고 재조합 제품에는 하이브리드 DNA의 '패치'만 존재하는 비크로스오버 제품이다.[79]
유전적 전이 촉진
균질 재조합은 수평적 유전자 전달에서 기증자 DNA를 수령자 유기체의 게놈에 통합하는 중요한 방법인데, 이 과정에서 유기체가 다른 유기체의 다른 DNA를 그 유기체의 자손이 되지 않고 통합하는 과정이다.동질 재조합은 유입 DNA가 수신자 게놈과 매우 유사해야 하므로 수평 유전자 전달은 대개 유사한 박테리아로 제한된다.[80]여러 종의 박테리아에 대한 연구는 숙주와 수취인 DNA 사이의 시퀀스 차이가 증가하면서 재결합 빈도가 로그 선형으로 감소한다는 것을 밝혀냈다.[81][82][83]
세포와 세포간의 직접 접촉을 통해 박테리아 사이에 DNA가 전달되는 박테리아 결합에서, 동질 재조합은 RecBCD 경로를 통해 외래 DNA를 숙주 게놈에 통합하는데 도움을 준다.RecBCD 효소는 DNA가 복제 중에 원래 박테리아에 들어간 단일 가닥 DNA에서 이중 가닥 DNA로 변환된 후 재조합을 촉진한다.RecBCD 경로도 바이러스에 의해 DNA가 한 박테리아에서 다른 박테리아로 옮겨지는 수평 유전자 전달의 한 종류인 전도의 최종 단계에 필수적이다.바이러스 복제 중에 DNA가 새로운 박테리오파지로 포장됨에 따라 외래 박테리아 DNA가 박테리오파지 바이러스 입자의 캡시드 머리 부분에 잘못 결합되기도 한다.이 새로운 박테리아들이 다른 박테리아들을 감염시키면, 이전 숙주 박테리아의 DNA가 이중 가닥 DNA로 새로운 박테리아 숙주에 주입된다.RecBCD 효소는 이 이중 스트랜드 DNA를 새로운 박테리아 숙주의 게놈에 통합한다.[65]
세균 변형
자연적인 박테리아 변형은 DNA가 기증자 박테리아에서 수령자 박테리아로 옮겨지는 것을 포함한다. 기증자와 수령자 모두 보통 같은 종이다.변형은 박테리아 결합과 전도와 달리 이 과정을 수행하기 위해 특별히 상호작용하는 수많은 박테리아 유전자 생산물에 의존한다.[84]따라서 변형은 분명히 DNA 전달에 대한 박테리아 적응이다.균이 균질 재조합에 의해 기증자 DNA를 결합, 점유, 결합하기 위해서는 우선 능력이라는 특수한 생리학적 상태에 들어가야 한다.RecA/Rad51/DMC1 유전자 계열은 진핵 감수분열과 유사분열에서와 같이 박테리아 변형 시 동음이의 재결합에서 중심적인 역할을 한다.예를 들어, 레카 단백질은 바실러스 하위조직과 스트렙토코쿠스 폐렴에서 변형에 필수적이며,[85] 이들 유기체에서 변형을 위한 역량의 발달 과정에서 레카 유전자의 발현이 유도된다.
변환 과정의 일부로, RecA 단백질은 단일 가닥 DNA(ssDNA) 입력과 상호작용하여 레지던트 염색체를 검사하여 동질학 영역을 검사하고 입력된 ssDNA를 해당 영역으로 가져오는 RecA/ssDNA 핵분석을 형성하며, 여기서 Strand 교환과 동질 재조합이 발생한다.[86]따라서 박테리아 변형 중 동음이의 재조합 과정은 감수분열 중 동음이의 재조합과 근본적인 유사성을 갖는다.
바이러스에서
동질 재조합은 여러 바이러스 그룹에서 발생한다.헤르페스 바이러스와 같은 DNA 바이러스에서, 재조합은 박테리아와 진핵생물에서와 같은 파괴와 재결합 메커니즘을 통해 발생한다.[87]일부 RNA 바이러스, 특히 레트로바이러스, 피코나비루스, 코로나비루스와 같은 양성 반응 ssRNA 바이러스에서도 재조합을 위한 증거가 있다.인플루엔자 같은 부정적 감각의 ssRNA 바이러스에서 동질 재조합이 발생하는지 논란이 일고 있다.[88]
RNA 바이러스에서 동질 재조합은 정밀하거나 부정확할 수 있다.RNA-RNA 재조합의 정밀한 유형에서는, 두 부모의 RNA 시퀀스와 그로 인한 교차 RNA 영역 사이에 차이가 없다.이 때문에, 재결합 RNA 시퀀스 사이의 교차 이벤트의 위치를 결정하기 어려운 경우가 많다.부정확한 RNA 동질 재결합에서, 교차 영역은 뉴클레오티드의 추가, 삭제 또는 기타 수정으로 인해 발생하는 부모의 RNA 시퀀스와 약간의 차이를 가진다.교차 정밀도 수준은 RNA의 두 재결합 가닥의 시퀀스 컨텍스트에 의해 제어된다: 아데닌과 우라실이 풍부한 시퀀스는 교차 정밀도를 감소시킨다.[89][90]
균질 재조합은 바이러스 진화를 촉진하는 데 중요하다.[89][91]예를 들어, 서로 다른 불리한 돌연변이를 가진 두 바이러스의 게놈들이 재조합을 거치면, 그들은 완전한 기능의 게놈을 재생할 수 있을 것이다.대안적으로, 만약 두 개의 유사한 바이러스가 동일한 호스트 세포를 감염했다면, 동질 재조합은 그 두 바이러스가 유전자를 교환하도록 허용하고, 따라서 그들 자신의 더 강력한 변형을 진화시킬 수 있다.[91]
동질 재조합은 DNA 바이러스의 인간 헤르페스 바이러스-6가 인간의 말단소립자에 통합되는 제안된 메커니즘이다.[92]
각각 치명적인 유전체 손상을 포함하는 두 개 이상의 바이러스가 동일한 숙주 세포를 감염시킬 때, 바이러스 게놈은 종종 서로 짝을 지어 생존 가능한 유전체를 생산하기 위해 동질 재조합 수리를 받을 수 있다.다중성 재활성화라고 알려진 이 과정은 페이징 T4를 포함한 여러 박테리오파지에서 연구되어 왔다.[93]Page T4에서 재조합 수리에 사용되는 효소는 박테리아 및 진핵 재조합 수리에 사용되는 효소와 기능적으로 동질적이다.[94]특히 동질 재조합 수리의 핵심 단계인 스트랜드 교환반응에 필요한 유전자에 관해서는 바이러스로부터 인간에 이르는 기능적 호몰로지(즉, page T4에서는 uvsX, 대장균과 다른 박테리아에서는 recA, 효모 및 기타 진핵생물에는 rad51과 dmc1이 있다)가 있다.[95]다중성 재활성화는 수많은 병원성 바이러스에서도 입증되었다.[96]
코로나비루스는 같은 감염된 세포에 적어도 두 개의 바이러스 게놈들이 존재할 때 유전적으로 재조합할 수 있다.RNA 재결합은 (1) CoV 종 내 유전적 가변성을 결정하는 주요 원동력으로 보이며, (2) CoV 종이 한 호스트에서 다른 호스트로 뛰어드는 능력, (3) 간헐적으로 새로운 CoVs의 출현으로 나타난다.[97]CoVs의 재조합 메커니즘은 게놈 복제 중 템플릿 전환과 관련이 있을 가능성이 높다.[97]RNA 바이러스의 재결합은 게놈 손상에 대처하기 위한 적응으로 보인다.[98]
유행성 사스-CoV-2의 전체 수용체 결합 모티브는 판골린의 코로나비루스에서 재조합을 통해 도입된 것으로 보인다.[99]이러한 재조합 사건은 SARS-CoV-2가 인간을 감염시키는 능력을 진화하는 데 중요한 단계가 되었을지도 모른다.[99]재조합 사건은 새로운 인간 코로나비루스의 출현으로 이어지는 진화 과정의 핵심 단계일 가능성이 높다.[100]
2020년에는 COVID-19 전염병 동안, 호주 SARS‐CoV‐2 소이의 많은 유전자 서열, 삭제 또는 돌연변이(29742G> 또는 29742G>다.U;Coronavirus 3′ stem-loopII-like의 모티프(s2m), 바이러스 게놈의 3'번역되지 않은 지역의 한 RNA의 모티프, RNA재결합 이벤트 SARS-CoV-2의 s2m에서 일어나지 것을 제안하는 것에"G19A"또는"G19U"cm이다.Recco 알고리즘(https://recco.bioinf.mpi-inf.mpg.de/), 29742G("G19"), 29744G("G21") 및 29751G("G28")를 이용한 1319 호주 SARS–CoV–2 시퀀스의 컴퓨터 분석을 바탕으로 재결합 핫스팟으로 예측되었다.[101]
다이아몬드 프린세스 크루즈에서 SARS-CoV-2가 발생한 것은 우한 WIV04와 동일한 바이러스 변종에 감염된 한 사람 또는 11083G > T 돌연변이가 포함된 바이러스에 감염된 또 다른 일차적인 사례와 동시에 발생한 것으로 보인다.연계불균형 분석 결과, 11083G > T 돌연변이와 RNA 재결합도 바이러스 자손 사이에서 돌연변이가 증가하는 데 기여했다는 사실이 확인되었다.이번 연구결과는 사스-CoV-2의 11083G > T 돌연변이가 선상방역 중 확산됐으며, 양성 선택압력 하에서 de novo RNA 재조합을 통해 발생했음을 보여준다.또한 이 크루즈에 탑승한 환자 3명 중 'G28'이 호주 사스-CoV-2 돌연변이의 재결합 핫스팟으로 예측되어 코로나바이러스 3 3 줄기-루프 II 같은 모티브(s2m)에도 돌연변이 29736G > T, 29751G > T("G13" 및 "G28")가 위치하였다.많은 코로나바이러스 종들 사이에서 s2m는 RNA 모티브로 여겨지지만, 이 결과는 SARS-CoV-2의 s2m가 오히려 RNA 재결합/혼합 핫스팟이라는 것을 시사한다.[102]
기능장애의 영향
적절한 동음이의 재조합 없이, 염색체는 감수분열에서 세포분열의 첫 번째 단계에 잘못 정렬되는 경우가 많다.이것은 염색체가 분리되지 않는다는 것을 불연속이라고 불리는 과정에서 적절하게 분리되지 못하게 한다.다시 말해서, 정자와 난자가 너무 적거나 너무 많은 염색체를 갖게 할 수 있다.21번 염색체의 추가 복제에 의해 발생하는 다운증후군은 감수분열에서 이처럼 동음이의 재조합이 실패하여 생기는 많은 이상 현상 중 하나이다.[78][103]
동음이의 재조합의 결핍은 인간의 암 형성과 강하게 연관되어 왔다.예를 들어, 각각의 암 관련 질환인 블룸 증후군, 베르너 증후군, 로스문트-톰슨 증후군은 각각 BLM, WRN, RECQL4와 같은 동질 재조합 규제에 관여하는 RecQ 헬리코아제 유전자의 복사본이 오작동하여 발생한다.[104]BLM 단백질의 작업 복사본이 부족한 블룸증후군 환자들의 세포에서는 동음이의 재조합 비율이 높아진다.[105]BLM이 부족한 생쥐에 대한 실험은 돌연변이가 동질 재조합의 증가로 인한 이질성 상실을 통해 암을 발생시킨다는 것을 시사했다.[106]이형성 상실은 유전자의 두 가지 버전 중 하나 또는 알레르기가 손실되는 것을 말한다.예를 들어 레티노블레스토마 단백질의 유전자처럼 잃어버린 알레르기가 종양을 억제하는데 도움을 준다면, 이질성의 상실은 암을 유발할 수 있다.[107]: 1236
동질 재조합 비율이 감소하면 비효율적인 DNA 수리를 초래하고,[107]: 310 이것은 또한 암을 유발할 수 있다.[108]오작동이 유방암과 난소암의 위험 증가와 관련이 있는 유사한 종양 억제기 두 개 유전자인 BRCA1과 BRCA2의 경우가 이에 해당한다.BRCA1과 BRCA2가 누락된 세포는 동질 재조합률이 감소하고 전리방사선에 대한 민감도가 증가하여 동질 재조합이 감소하면 암에 대한 민감도가 증가한다는 것을 시사한다.[108]BRCA2의 유일한 기능은 동질 재조합을 시작하는 것을 돕는 것이기 때문에, 연구자들은 동질 재조합에 대한 BRCA2의 역할에 대한 보다 상세한 지식이 유방암과 난소암의 원인을 이해하는 열쇠가 될 수 있다고 추측했다.[108]
동질 재조합결핍(BRCA 결함 포함)이 있는 종양을 HRD 양성이라고 한다.[109]
진화보존
그 경로는 기계적으로 다를 수 있지만, 균질 재조합을 수행하는 유기체의 능력은 삶의 모든 영역에 걸쳐 보편적으로 보존된다.[110]그들의 아미노산 염기서열의 유사성에 기초하여, 많은 단백질의 호몰로로그는 그들이 오래 전에 진화했다는 것을 나타내는 생명의 여러 영역에서 발견될 수 있으며, 그 이후로 공통의 조상 단백질로부터 분리되어 왔다.[110]
RecA 재조합효소 가족 구성원은 박테리아에 RecA, eukaryotes에 Rad51과 DMC1, 고고학에 RadA, T4 페이지에 UvsX가 있는 거의 모든 유기체에서 발견된다.[111]
균질 재조합에 중요한 관련 단일 좌초 결합 단백질과 다른 많은 과정들도 삶의 모든 영역에서 발견된다.[112]
Rad54, Mre11, Rad50, 그리고 많은 다른 단백질들이 고고학과 진핵생물 모두에서 발견된다.[110][111][113]
RecA 재조합 제품군
단백질의 레카 재조합효소 계열의 단백질은 공통의 조상 재조합효소의 자손인 것으로 생각된다.[110]RecA 재조합효소 제품군에는 박테리아에서 추출한 RecA 단백질, eukaryotes에서 추출한 Rad51과 Dmc1 단백질, 고고에서 추출한 RadA, 그리고 재조합 파라로그 단백질이 들어 있다.Rad51, Dmc1 및 RadA 단백질 사이의 진화 관계를 모델링한 연구는 그것들이 단극성이거나 공통의 분자 조상을 공유한다는 것을 보여준다.[110]이 단백질 제품군 내에서 Rad51과 Dmc1은 RadA와는 별도의 클래드로 함께 그룹화된다.이 세 가지 단백질을 함께 그룹화하는 이유 중 하나는 그들 모두가 단백질들이 그들의 N-단자 끝을 향해 DNA와 결합하는 것을 돕는 변형 나선-회전-헬릭스 모티브를 가지고 있기 때문이다.[110]진핵 RecA 유전자의 고대 유전자 복제 사건과 이후의 돌연변이가 현대의 RAD51과 DMC1 유전자의 유력한 기원으로 제안되었다.[110]
단백질은 일반적으로 RecA/Rad51 영역으로 알려진 보존된 긴 지역을 공유한다.이 단백질 영역 안에는 워커 A 모티브와 워커 B 모티브의 두 가지 시퀀스 모티브가 있다.Walker A와 B 모티브를 통해 RecA/Rad51 단백질 제품군의 구성원이 ATP 결합 및 ATP 가수분해를 할 수 있다.[110][114]
감수분열 특이 단백질
진핵생물로 분화된 최초의 양성자 중 하나인 Giardia의 여러 종에서 Dmc1이 발견된 것은 진핵생성 진화에서 매우 일찍 나타난 감수성 동질 재조합, 즉 감수분열 그 자체임을 시사한다.[115]Dmc1에 대한 연구 외에도, 스포11 단백질에 대한 연구는 감수성 재조합의 기원에 대한 정보를 제공했다.[116]제2종 topoisomerase인 spo11은 DNA에서 표적형 이중 가닥을 끊음으로써 감수분열에서 동질 재조합을 시작할 수 있다.[25]동물, 곰팡이, 식물, 원생, 고고학에서 SPO11과 유사한 유전자의 순서를 바탕으로 한 계통생식 나무는 과학자들이 현재 진핵생물에 있는 SPO11 버전이 진핵생물과 고대의 마지막 공통 조상에 나타났다고 믿게 만들었다.[116]
기술 응용 프로그램
유전자 타겟팅
DNA 시퀀스를 유기체에 도입하여 재조합 DNA와 유전자 변형 유기체를 만드는 많은 방법들이 동질 재조합 과정을 이용한다.[117]유전자 타겟팅이라고도 불리는 이 방법은 특히 효모와 생쥐 유전학에서 흔하다.녹아웃 생쥐의 유전자 표적법은 쥐 배아줄기세포를 이용해 인공유전물질(대부분의 치료적 관심사)을 전달하는데, 이는 쥐의 표적 유전자를 호몰로 재조합 원리에 의해 억제한다.따라서 마우스는 특정 포유류 유전자의 영향을 이해하는 작업 모델 역할을 한다.배아줄기세포를 통해 쥐에게 유전적 변형을 도입하는 데 어떻게 동질적 재조합이 이용될 수 있는지를 발견한 공로를 인정받아 마리오 카페치, 마틴 에반스, 올리버 스미스가 2007년 노벨 생리학 또는 의학상을 받았다.[118]
세포의 동질 재조합 역학을 가로채는 유전자 표적 기술의 발전은 이제 더 정확하고 이질적인 인간 질병 모델의 새로운 파장을 이끌고 있다.이러한 공학적 인간 세포 모델은 이전 쥐 모델보다 인간 질병의 유전학을 더 정확하게 반영하는 것으로 생각된다.이는 관심의 돌연변이가 실제 환자에서 일어나는 것처럼 내생 유전자에 유입되기 때문이며, 쥐 게놈보다는 인간의 게놈에 기반하기 때문이다.게다가, 특정 기술은 오래된 유전자 표적 기술과 관련된 단순한 녹아웃이 아니라 특정한 돌연변이의 노크인을 가능하게 한다.
단백질공학
동음이의 재조합을 가진 단백질 공학은 두 부모의 단백질 사이에 조각들을 교환함으로써 치메릭 단백질을 개발한다.이러한 기법들은 재조합이 단백질의 3차 구조, 즉 3차원 모양으로 접히는 능력을 보존하면서 높은 수준의 염기서열 다양성을 도입할 수 있다는 사실을 이용한다.[119]이는 아미노산 대체량이 증가함에 따라 단백질 함수를 유지할 확률은 기하급수적으로 감소하는 무작위 포인트 돌연변이 유발과 같은 다른 단백질 공학적 기법과는 대조적이다.[120]재조합 기술에 의해 생성된 키메라들은 그들의 스와핑된 부모 조각들이 구조적으로 그리고 진화적으로 보존되어 있기 때문에 접히는 능력을 유지할 수 있다.이러한 재조합 가능한 "건물 블록"은 단백질 구조에서 다른 아미노산 사이의 물리적 접촉 지점과 같은 구조적으로 중요한 상호작용을 보존한다.SCHEMA 및 통계 결합 분석과 같은 계산 방법을 사용하여 재조합에 적합한 구조 하위 단위를 식별할 수 있다.[121][122][123]
동질 재조합에 의존하는 기술은 새로운 단백질을 개발하기 위해 사용되어 왔다.[121]2007년에 발표된 연구에서, 연구원들은 호르몬, 시각 색소, 특정 페로몬을 포함한 다양한 종류의 화합물인 이소프로노이드의 생합성에 관여하는 두 효소의 키메라를 만들 수 있었다.치맥 단백질은 자연에서 발견되는 가장 다양한 생합성 경로 중 하나인 이소프레노이드 생합성에서 모체 단백질에 없는 필수적인 반응을 촉진하는 능력을 얻었다.[124]재조합을 통한 단백질 공학은 또한 사이토크롬 P450 계열로 알려진 단백질 그룹의 구성원에서 새로운 기능을 가진 치메르 효소를 생산했는데,[125] 이 효소는 인간에게 있어서 약물, 식품 첨가물, 방부제 같은 외국 화합물의 해독에 관여하고 있다.[22]
암치료
동질 재조합 능숙한(HRP) 암세포는 시스플라틴 등 화학요법에 의해 발생하는 DNA 손상을 치료할 수 있다.따라서 HRP암은 치료하기 어렵다.연구에 따르면 동질 재조합은 c-Abl 억제를 통해 대상이 될 수 있다.[126][127]BRCA 돌연변이를 가진 암세포는 균질 재조합에 결함이 있으며, 그러한 결함을 이용하기 위한 약물이 개발되어 임상시험에 성공적으로 사용되고 있다.[128][129]PARP1 억제제인 올라파립은 HR에 필요한 BRCA1이나 BRCA2 유전자의 돌연변이로 인한 유방암, 난소암, 전립선암으로 인한 종양의 성장을 위축시키거나 중단시켰다.BRCA1 또는 BRCA2가 없을 경우, 정지된 복제 포크에 대한 베이스 절연 수리(BER) 또는 이중 스트랜드 파손에 대한 비호몰성 엔드 결합(NHEJ)과 같은 다른 유형의 DNA 수리 메커니즘은 HR의 결함을 보상해야 한다.[128]HR-결핍 세포에서 BER을 억제함으로써, 올라파립은 특히 표적 암세포에 합성 치사성의 개념을 적용한다.PARP1 억제제는 암 치료에 대한 새로운 접근방식을 나타내지만, 연구원들은 그것들이 말기 전이암을 치료하는데 불충분하다는 것을 증명할 수 있다고 경고했다.[128]암세포는 BRCA2에서 돌연변이를 삭제하면 PARP1 억제제에 내성이 생겨 암세포의 DNA 복구 능력을 HR로 회복시켜 약물의 합성적 치사성을 훼손할 수 있다.[130]
참고 항목
참조
- ^ Thompson LH, Schild D (June 2001). "Homologous recombinational repair of DNA ensures mammalian chromosome stability". Mutation Research. 477 (1–2): 131–53. doi:10.1016/S0027-5107(01)00115-4. PMID 11376695.
- ^ Alberts B, Johnson A, Lewis J, Raff M, Roberts K, Walter P, et al. (2002). "Chapter 5: DNA Replication, Repair, and Recombination". Molecular Biology of the Cell (4th ed.). New York: Garland Science. p. 845. ISBN 978-0-8153-3218-3. OCLC 145080076.
- ^ Capecchi MR (June 1989). "Altering the genome by homologous recombination". Science. 244 (4910): 1288–92. Bibcode:1989Sci...244.1288C. doi:10.1126/science.2660260. PMID 2660260.
- ^ Smithies O, Gregg RG, Boggs SS, Koralewski MA, Kucherlapati RS (1985-09-19). "Insertion of DNA sequences into the human chromosomal beta-globin locus by homologous recombination". Nature. 317 (6034): 230–4. Bibcode:1985Natur.317..230S. doi:10.1038/317230a0. PMID 2995814. S2CID 30212766.
- ^ Orr-Weaver TL, Szostak JW, Rothstein RJ (October 1981). "Yeast transformation: a model system for the study of recombination". Proceedings of the National Academy of Sciences of the United States of America. 78 (10): 6354–8. Bibcode:1981PNAS...78.6354O. doi:10.1073/pnas.78.10.6354. PMC 349037. PMID 6273866.
- ^ Orr-Weaver TL, Szostak JW (July 1983). "Yeast recombination: the association between double-strand gap repair and crossing-over". Proceedings of the National Academy of Sciences of the United States of America. 80 (14): 4417–21. Bibcode:1983PNAS...80.4417O. doi:10.1073/pnas.80.14.4417. PMC 384049. PMID 6308623.
- ^ a b Szostak JW, Orr-Weaver TL, Rothstein RJ, Stahl FW (May 1983). "The double-strand-break repair model for recombination". Cell. 33 (1): 25–35. doi:10.1016/0092-8674(83)90331-8. PMID 6380756. S2CID 39590123.
- ^ Resnick MA (June 1976). "The repair of double-strand breaks in DNA; a model involving recombination". Journal of Theoretical Biology. 59 (1): 97–106. Bibcode:1976JThBi..59...97R. doi:10.1016/s0022-5193(76)80025-2. PMID 940351.
- ^ Jasin M, Rothstein R (November 2013). "Repair of strand breaks by homologous recombination". Cold Spring Harbor Perspectives in Biology. 5 (11): a012740. doi:10.1101/cshperspect.a012740. PMC 3809576. PMID 24097900.
- ^ Bateson P (August 2002). "William Bateson: a biologist ahead of his time" (PDF). Journal of Genetics. 81 (2): 49–58. doi:10.1007/BF02715900. PMID 12532036. S2CID 26806110.
- ^ "Reginald Crundall Punnett". NAHSTE, University of Edinburgh. Retrieved 3 July 2010.
- ^ Lobo I, Shaw K (2008). "Thomas Hunt Morgan, genetic recombination, and gene mapping". Nature Education. 1 (1).
- ^ a b Coe E, Kass LB (May 2005). "Proof of physical exchange of genes on the chromosomes". Proceedings of the National Academy of Sciences of the United States of America. 102 (19): 6641–6. Bibcode:2005PNAS..102.6641C. doi:10.1073/pnas.0407340102. PMC 1100733. PMID 15867161.
- ^ Creighton HB, McClintock B (August 1931). "A Correlation of Cytological and Genetical Crossing-Over in Zea Mays". Proceedings of the National Academy of Sciences of the United States of America. 17 (8): 492–7. Bibcode:1931PNAS...17..492C. doi:10.1073/pnas.17.8.492. PMC 1076098. PMID 16587654.
- ^ Stern, C (1931). "Zytologisch-genetische untersuchungen alsbeweise fur die Morgansche theorie des faktoraustauschs". Biologisches Zentralblatt. 51: 547–587.
- ^ "The development of bacterial genetics". US National Library of Medicine. Retrieved 3 July 2010.
- ^ "The Nobel Prize in Physiology or Medicine 1958". Nobelprize.org. Retrieved 3 July 2010.
- ^ a b c Haber JE, Ira G, Malkova A, Sugawara N (January 2004). "Repairing a double-strand chromosome break by homologous recombination: revisiting Robin Holliday's model". Philosophical Transactions of the Royal Society of London. Series B, Biological Sciences. 359 (1441): 79–86. doi:10.1098/rstb.2003.1367. PMC 1693306. PMID 15065659.
- ^ Lodish H, Berk A, Zipursky SL, Matsudaira P, Baltimore D, Darnell J (2000). "12.5: Recombination between Homologous DNA Sites: Double-Strand Breaks in DNA Initiate Recombination". Molecular Cell Biology (4th ed.). W. H. Freeman and Company. ISBN 978-0-7167-3136-8.
- ^ Griffiths A, et al. (1999). "8: Chromosome Mutations: Chromosomal Rearrangements". Modern Genetic Analysis. W. H. Freeman and Company. ISBN 978-0-7167-3118-4.
- ^ Khanna KK, Jackson SP (March 2001). "DNA double-strand breaks: signaling, repair and the cancer connection". Nature Genetics. 27 (3): 247–54. doi:10.1038/85798. PMID 11242102. S2CID 3012823.
- ^ a b c Nelson DL, Cox MM (2005). Principles of Biochemistry (4th ed.). Freeman. pp. 980–981. ISBN 978-0-7167-4339-2.
- ^ Marcon E, Moens PB (August 2005). "The evolution of meiosis: recruitment and modification of somatic DNA-repair proteins". BioEssays. 27 (8): 795–808. doi:10.1002/bies.20264. PMID 16015600. S2CID 27658497.
- ^ Alberts B, Johnson A, Lewis J, Raff M, Roberts K, Walter P (2008). Molecular Biology of the Cell (5th ed.). Garland Science. p. 305. ISBN 978-0-8153-4105-5.
- ^ a b Keeney S, Giroux CN, Kleckner N (February 1997). "Meiosis-specific DNA double-strand breaks are catalyzed by Spo11, a member of a widely conserved protein family". Cell. 88 (3): 375–84. doi:10.1016/S0092-8674(00)81876-0. PMID 9039264. S2CID 8294596.
- ^ Longhese MP, Bonetti D, Guerini I, Manfrini N, Clerici M (September 2009). "DNA double-strand breaks in meiosis: checking their formation, processing and repair". DNA Repair. 8 (9): 1127–38. doi:10.1016/j.dnarep.2009.04.005. PMID 19464965.
- ^ Cahill LP, Mariana JC, Mauléon P (January 1979). "Total follicular populations in ewes of high and low ovulation rates". Journal of Reproduction and Fertility. 55 (1): 27–36. doi:10.1530/jrf.0.0550027. PMID 423159.
- ^ Schimmel J, van Schendel R, den Dunnen JT, Tijsterman M (September 2019). "Templated Insertions: A Smoking Gun for Polymerase Theta-Mediated End Joining". Trends in Genetics. 35 (9): 632–644. doi:10.1016/j.tig.2019.06.001. PMID 31296341. S2CID 195892718.
- ^ Alberts B, Johnson A, Lewis J, Raff M, Roberts K, Walter P (2008). Molecular Biology of the Cell (5th ed.). Garland Science. p. 303. ISBN 978-0-8153-4105-5.
- ^ a b Shrivastav M, De Haro LP, Nickoloff JA (January 2008). "Regulation of DNA double-strand break repair pathway choice". Cell Research. 18 (1): 134–47. doi:10.1038/cr.2007.111. PMID 18157161.
- ^ a b Mimitou EP, Symington LS (May 2009). "Nucleases and helicases take center stage in homologous recombination". Trends in Biochemical Sciences. 34 (5): 264–72. doi:10.1016/j.tibs.2009.01.010. PMID 19375328.
- ^ Andres, Sara N.; Williams, R. Scott (August 2017). "CtIP/Ctp1/Sae2, molecular form fit for function". DNA Repair. 56: 109–117. doi:10.1016/j.dnarep.2017.06.013. PMC 5543718. PMID 28623092.
- ^ Huertas P, Cortés-Ledesma F, Sartori AA, Aguilera A, Jackson SP (October 2008). "CDK targets Sae2 to control DNA-end resection and homologous recombination". Nature. 455 (7213): 689–92. Bibcode:2008Natur.455..689H. doi:10.1038/nature07215. PMC 2635538. PMID 18716619.
- ^ Liu B, Yip RK, Zhou Z (2012). "Chromatin remodeling, DNA damage repair and aging". Curr. Genomics. 13 (7): 533–47. doi:10.2174/138920212803251373. PMC 3468886. PMID 23633913.
- ^ a b c d Sellou H, Lebeaupin T, Chapuis C, Smith R, Hegele A, Singh HR, Kozlowski M, Bultmann S, Ladurner AG, Timinszky G, Huet S (2016). "The poly(ADP-ribose)-dependent chromatin remodeler Alc1 induces local chromatin relaxation upon DNA damage". Mol. Biol. Cell. 27 (24): 3791–3799. doi:10.1091/mbc.E16-05-0269. PMC 5170603. PMID 27733626.
- ^ a b Van Meter M, Simon M, Tombline G, May A, Morello TD, Hubbard BP, Bredbenner K, Park R, Sinclair DA, Bohr VA, Gorbunova V, Seluanov A (2016). "JNK Phosphorylates SIRT6 to Stimulate DNA Double-Strand Break Repair in Response to Oxidative Stress by Recruiting PARP1 to DNA Breaks". Cell Rep. 16 (10): 2641–50. doi:10.1016/j.celrep.2016.08.006. PMC 5089070. PMID 27568560.
- ^ a b Haince JF, McDonald D, Rodrigue A, Déry U, Masson JY, Hendzel MJ, Poirier GG (2008). "PARP1-dependent kinetics of recruitment of MRE11 and NBS1 proteins to multiple DNA damage sites". J. Biol. Chem. 283 (2): 1197–208. doi:10.1074/jbc.M706734200. PMID 18025084.
- ^ a b c Rogakou EP, Pilch DR, Orr AH, Ivanova VS, Bonner WM (1998). "DNA double-stranded breaks induce histone H2AX phosphorylation on serine 139". J. Biol. Chem. 273 (10): 5858–68. doi:10.1074/jbc.273.10.5858. PMID 9488723.
- ^ Mailand N, Bekker-Jensen S, Faustrup H, Melander F, Bartek J, Lukas C, Lukas J (2007). "RNF8 ubiquitylates histones at DNA double-strand breaks and promotes assembly of repair proteins". Cell. 131 (5): 887–900. doi:10.1016/j.cell.2007.09.040. PMID 18001824. S2CID 14232192.
- ^ Luijsterburg MS, Acs K, Ackermann L, Wiegant WW, Bekker-Jensen S, Larsen DH, Khanna KK, van Attikum H, Mailand N, Dantuma NP (2012). "A new non-catalytic role for ubiquitin ligase RNF8 in unfolding higher-order chromatin structure". EMBO J. 31 (11): 2511–27. doi:10.1038/emboj.2012.104. PMC 3365417. PMID 22531782.
- ^ Baudat F, Buard J, Grey C, Fledel-Alon A, Ober C, Przeworski M, et al. (February 2010). "PRDM9 is a major determinant of meiotic recombination hotspots in humans and mice". Science. 327 (5967): 836–40. Bibcode:2010Sci...327..836B. doi:10.1126/science.1183439. PMC 4295902. PMID 20044539.
- ^ Wells D, Bitoun E, Moralli D, Zhang G, Hinch A, Jankowska J, et al. (August 2020). "ZCWPW1 is recruited to recombination hotspots by PRDM9, and is essential for meiotic double strand break repair". eLife. 9: e53392. doi:10.7554/eLife.53392. PMC 7494361. PMID 32744506.
- ^ a b c d Sung P, Klein H (October 2006). "Mechanism of homologous recombination: mediators and helicases take on regulatory functions". Nature Reviews Molecular Cell Biology. 7 (10): 739–50. doi:10.1038/nrm2008. PMID 16926856. S2CID 30324005.
- ^ Wold MS (1997). "Replication protein A: a heterotrimeric, single-stranded DNA-binding protein required for eukaryotic DNA metabolism". Annual Review of Biochemistry. 66: 61–92. doi:10.1146/annurev.biochem.66.1.61. PMID 9242902.
- ^ McMahill MS, Sham CW, Bishop DK (November 2007). "Synthesis-dependent strand annealing in meiosis". PLOS Biology. 5 (11): e299. doi:10.1371/journal.pbio.0050299. PMC 2062477. PMID 17988174.

- ^ Bärtsch S, Kang LE, Symington LS (February 2000). "RAD51 is required for the repair of plasmid double-stranded DNA gaps from either plasmid or chromosomal templates". Molecular and Cellular Biology. 20 (4): 1194–205. doi:10.1128/MCB.20.4.1194-1205.2000. PMC 85244. PMID 10648605.
- ^ Alberts B, Johnson A, Lewis J, Raff M, Roberts K, Walter P (2008). Molecular Biology of the Cell (5th ed.). Garland Science. pp. 312–313. ISBN 978-0-8153-4105-5.
- ^ a b c Helleday T, Lo J, van Gent DC, Engelward BP (July 2007). "DNA double-strand break repair: from mechanistic understanding to cancer treatment". DNA Repair. 6 (7): 923–35. doi:10.1016/j.dnarep.2007.02.006. PMID 17363343.
- ^ a b Andersen SL, Sekelsky J (December 2010). "Meiotic versus mitotic recombination: two different routes for double-strand break repair: the different functions of meiotic versus mitotic DSB repair are reflected in different pathway usage and different outcomes". BioEssays. 32 (12): 1058–66. doi:10.1002/bies.201000087. PMC 3090628. PMID 20967781.
- ^ Allers T, Lichten M (July 2001). "Differential timing and control of noncrossover and crossover recombination during meiosis". Cell. 106 (1): 47–57. doi:10.1016/s0092-8674(01)00416-0. PMID 11461701. S2CID 1878863.
- ^ Haber lab. "Single-strand annealing". Brandeis University. Retrieved 3 July 2010.
- ^ a b c Lyndaker AM, Alani E (March 2009). "A tale of tails: insights into the coordination of 3' end processing during homologous recombination". BioEssays. 31 (3): 315–21. doi:10.1002/bies.200800195. PMC 2958051. PMID 19260026.
- ^ Mimitou EP, Symington LS (September 2009). "DNA end resection: many nucleases make light work". DNA Repair. 8 (9): 983–95. doi:10.1016/j.dnarep.2009.04.017. PMC 2760233. PMID 19473888.
- ^ Pâques F, Haber JE (June 1999). "Multiple pathways of recombination induced by double-strand breaks in Saccharomyces cerevisiae". Microbiology and Molecular Biology Reviews. 63 (2): 349–404. doi:10.1128/MMBR.63.2.349-404.1999. PMC 98970. PMID 10357855.
- ^ a b McEachern MJ, Haber JE (2006). "Break-induced replication and recombinational telomere elongation in yeast". Annual Review of Biochemistry. 75: 111–35. doi:10.1146/annurev.biochem.74.082803.133234. PMID 16756487.
- ^ Morrish TA, Greider CW (January 2009). Haber JE (ed.). "Short telomeres initiate telomere recombination in primary and tumor cells". PLOS Genetics. 5 (1): e1000357. doi:10.1371/journal.pgen.1000357. PMC 2627939. PMID 19180191.

- ^ Muntoni A, Reddel RR (October 2005). "The first molecular details of ALT in human tumor cells". Human Molecular Genetics. 14 Spec No. 2 (Review Issue 2): R191–6. doi:10.1093/hmg/ddi266. PMID 16244317.
- ^ PDB: 3cmt; Chen Z, Yang H, Pavletich NP (May 2008). "Mechanism of homologous recombination from the RecA-ssDNA/dsDNA structures". Nature. 453 (7194): 489–4. Bibcode:2008Natur.453..489C. doi:10.1038/nature06971. PMID 18497818. S2CID 4416531.
- ^ Kowalczykowski SC, Dixon DA, Eggleston AK, Lauder SD, Rehrauer WM (September 1994). "Biochemistry of homologous recombination in Escherichia coli". Microbiological Reviews. 58 (3): 401–65. doi:10.1128/MMBR.58.3.401-465.1994. PMC 372975. PMID 7968921.
- ^ Rocha EP, Cornet E, Michel B (August 2005). "Comparative and evolutionary analysis of the bacterial homologous recombination systems". PLOS Genetics. 1 (2): e15. doi:10.1371/journal.pgen.0010015. PMC 1193525. PMID 16132081.

- ^ a b Amundsen SK, Taylor AF, Reddy M, Smith GR (December 2007). "Intersubunit signaling in RecBCD enzyme, a complex protein machine regulated by Chi hot spots". Genes & Development. 21 (24): 3296–307. doi:10.1101/gad.1605807. PMC 2113030. PMID 18079176.
- ^ Singleton MR, Dillingham MS, Gaudier M, Kowalczykowski SC, Wigley DB (November 2004). "Crystal structure of RecBCD enzyme reveals a machine for processing DNA breaks" (PDF). Nature. 432 (7014): 187–93. Bibcode:2004Natur.432..187S. doi:10.1038/nature02988. PMID 15538360. S2CID 2916995. Archived from the original (PDF) on 2004-05-25.
- ^ Cromie GA (August 2009). "Phylogenetic ubiquity and shuffling of the bacterial RecBCD and AddAB recombination complexes". Journal of Bacteriology. 191 (16): 5076–84. doi:10.1128/JB.00254-09. PMC 2725590. PMID 19542287.
- ^ a b Smith GR (June 2012). "How RecBCD enzyme and Chi promote DNA break repair and recombination: a molecular biologist's view". Microbiology and Molecular Biology Reviews. 76 (2): 217–28. doi:10.1128/MMBR.05026-11. PMC 3372252. PMID 22688812.
- ^ a b c d Dillingham MS, Kowalczykowski SC (December 2008). "RecBCD enzyme and the repair of double-stranded DNA breaks". Microbiology and Molecular Biology Reviews. 72 (4): 642–71, Table of Contents. doi:10.1128/MMBR.00020-08. PMC 2593567. PMID 19052323.
- ^ Michel B, Boubakri H, Baharoglu Z, LeMasson M, Lestini R (July 2007). "Recombination proteins and rescue of arrested replication forks". DNA Repair. 6 (7): 967–80. doi:10.1016/j.dnarep.2007.02.016. PMID 17395553.
- ^ Spies M, Bianco PR, Dillingham MS, Handa N, Baskin RJ, Kowalczykowski SC (September 2003). "A molecular throttle: the recombination hotspot chi controls DNA translocation by the RecBCD helicase". Cell. 114 (5): 647–54. doi:10.1016/S0092-8674(03)00681-0. PMID 13678587. S2CID 16662983.
- ^ Taylor AF, Smith GR (June 2003). "RecBCD enzyme is a DNA helicase with fast and slow motors of opposite polarity". Nature. 423 (6942): 889–93. Bibcode:2003Natur.423..889T. doi:10.1038/nature01674. PMID 12815437. S2CID 4302346.
- ^ Spies M, Amitani I, Baskin RJ, Kowalczykowski SC (November 2007). "RecBCD enzyme switches lead motor subunits in response to chi recognition". Cell. 131 (4): 694–705. doi:10.1016/j.cell.2007.09.023. PMC 2151923. PMID 18022364.
- ^ Savir Y, Tlusty T (November 2010). "RecA-mediated homology search as a nearly optimal signal detection system" (PDF). Molecular Cell. 40 (3): 388–96. arXiv:1011.4382. Bibcode:2010arXiv1011.4382S. doi:10.1016/j.molcel.2010.10.020. PMID 21070965. S2CID 1682936. Archived from the original (PDF) on 2012-10-07. Retrieved 2011-08-31.
- ^ Rambo RP, Williams GJ, Tainer JA (November 2010). "Achieving fidelity in homologous recombination despite extreme complexity: informed decisions by molecular profiling" (PDF). Molecular Cell. 40 (3): 347–8. doi:10.1016/j.molcel.2010.10.032. PMC 3003302. PMID 21070960. Archived from the original (PDF) on 2012-10-07. Retrieved 2011-08-31.
- ^ De Vlaminck I, van Loenhout MT, Zweifel L, den Blanken J, Hooning K, Hage S, et al. (June 2012). "Mechanism of homology recognition in DNA recombination from dual-molecule experiments". Molecular Cell. 46 (5): 616–24. doi:10.1016/j.molcel.2012.03.029. PMID 22560720.
- ^ Alberts B, Johnson A, Lewis J, Raff M, Roberts K, Walter P (2008). Molecular Biology of the Cell (5th ed.). Garland Science. p. 307. ISBN 978-0-8153-4105-5.
- ^ Morimatsu K, Kowalczykowski SC (May 2003). "RecFOR proteins load RecA protein onto gapped DNA to accelerate DNA strand exchange: a universal step of recombinational repair". Molecular Cell. 11 (5): 1337–47. doi:10.1016/S1097-2765(03)00188-6. PMID 12769856.
- ^ Hiom K (July 2009). "DNA repair: common approaches to fixing double-strand breaks". Current Biology. 19 (13): R523–5. doi:10.1016/j.cub.2009.06.009. PMID 19602417. S2CID 2221866.
- ^ Handa N, Morimatsu K, Lovett ST, Kowalczykowski SC (May 2009). "Reconstitution of initial steps of dsDNA break repair by the RecF pathway of E. coli". Genes & Development. 23 (10): 1234–45. doi:10.1101/gad.1780709. PMC 2685532. PMID 19451222.
- ^ a b West SC (June 2003). "Molecular views of recombination proteins and their control". Nature Reviews Molecular Cell Biology. 4 (6): 435–45. doi:10.1038/nrm1127. PMID 12778123. S2CID 28474965.
- ^ a b c d Watson JD, Baker TA, Bell SP, Gann A, Levine M, Losick R (2003). Molecular Biology of the Gene (5th ed.). Pearson/Benjamin Cummings. pp. 259–291. ISBN 978-0-8053-4635-0.
- ^ Gumbiner-Russo LM, Rosenberg SM (28 November 2007). Sandler S (ed.). "Physical analyses of E. coli heteroduplex recombination products in vivo: on the prevalence of 5' and 3' patches". PLOS ONE. 2 (11): e1242. Bibcode:2007PLoSO...2.1242G. doi:10.1371/journal.pone.0001242. PMC 2082072. PMID 18043749.

- ^ Thomas CM, Nielsen KM (September 2005). "Mechanisms of, and barriers to, horizontal gene transfer between bacteria" (PDF). Nature Reviews. Microbiology. 3 (9): 711–21. doi:10.1038/nrmicro1234. PMID 16138099. S2CID 1231127. Archived from the original (PDF) on 2010-06-01.
- ^ Vulić M, Dionisio F, Taddei F, Radman M (September 1997). "Molecular keys to speciation: DNA polymorphism and the control of genetic exchange in enterobacteria". Proceedings of the National Academy of Sciences of the United States of America. 94 (18): 9763–7. Bibcode:1997PNAS...94.9763V. doi:10.1073/pnas.94.18.9763. PMC 23264. PMID 9275198.
- ^ Majewski J, Cohan FM (January 1998). "The effect of mismatch repair and heteroduplex formation on sexual isolation in Bacillus". Genetics. 148 (1): 13–8. doi:10.1093/genetics/148.1.13. PMC 1459767. PMID 9475717.
- ^ Majewski J, Zawadzki P, Pickerill P, Cohan FM, Dowson CG (February 2000). "Barriers to genetic exchange between bacterial species: Streptococcus pneumoniae transformation". Journal of Bacteriology. 182 (4): 1016–23. doi:10.1128/JB.182.4.1016-1023.2000. PMC 94378. PMID 10648528.
- ^ Chen I, Dubnau D (March 2004). "DNA uptake during bacterial transformation". Nature Reviews. Microbiology. 2 (3): 241–9. doi:10.1038/nrmicro844. PMID 15083159. S2CID 205499369.
- ^ Claverys JP, Martin B, Polard P (May 2009). "The genetic transformation machinery: composition, localization, and mechanism". FEMS Microbiology Reviews. 33 (3): 643–56. doi:10.1111/j.1574-6976.2009.00164.x. PMID 19228200.
- ^ Kidane D, Graumann PL (July 2005). "Intracellular protein and DNA dynamics in competent Bacillus subtilis cells". Cell. 122 (1): 73–84. doi:10.1016/j.cell.2005.04.036. PMID 16009134. S2CID 17272331.
- ^ Fleischmann Jr WR (1996). "43". Medical Microbiology (4th ed.). University of Texas Medical Branch at Galveston. ISBN 978-0-9631172-1-2.
- ^ Boni MF, de Jong MD, van Doorn HR, Holmes EC (3 May 2010). Martin DP (ed.). "Guidelines for identifying homologous recombination events in influenza A virus". PLOS ONE. 5 (5): e10434. Bibcode:2010PLoSO...510434B. doi:10.1371/journal.pone.0010434. PMC 2862710. PMID 20454662.

- ^ a b Nagy PD, Bujarski JJ (January 1996). "Homologous RNA recombination in brome mosaic virus: AU-rich sequences decrease the accuracy of crossovers". Journal of Virology. 70 (1): 415–26. doi:10.1128/JVI.70.1.415-426.1996. PMC 189831. PMID 8523555.
- ^ Chetverin AB (October 1999). "The puzzle of RNA recombination". FEBS Letters. 460 (1): 1–5. doi:10.1016/S0014-5793(99)01282-X. PMC 7163957. PMID 10571050.
- ^ a b Roossinck MJ (September 1997). "Mechanisms of plant virus evolution". Annual Review of Phytopathology. 35: 191–209. doi:10.1146/annurev.phyto.35.1.191. PMID 15012521.
- ^ Arbuckle JH, Medveczky PG (August 2011). "The molecular biology of human herpesvirus-6 latency and telomere integration". Microbes and Infection / Institut Pasteur. 13 (8–9): 731–41. doi:10.1016/j.micinf.2011.03.006. PMC 3130849. PMID 21458587.
- ^ Bernstein C (March 1981). "Deoxyribonucleic acid repair in bacteriophage". Microbiological Reviews. 45 (1): 72–98. doi:10.1128/MMBR.45.1.72-98.1981. PMC 281499. PMID 6261109.
- ^ Bernstein C, Bernstein H(2001).박테리오파지에서 DNA를 치료한다.인: Nickoloff JA, Hoekstra MF(Eds).DNA 손상 및 수리, vol.3. 페이지에서 인간으로 발전.Humana Press, Totawa, NJ, 페이지 1-19.ISBN 978-0896038035
- ^ Story RM, Bishop DK, Kleckner N, Steitz TA (March 1993). "Structural relationship of bacterial RecA proteins to recombination proteins from bacteriophage T4 and yeast". Science. 259 (5103): 1892–6. Bibcode:1993Sci...259.1892S. doi:10.1126/science.8456313. PMID 8456313.
- ^ Michod RE, Bernstein H, Nedelcu AM (May 2008). "Adaptive value of sex in microbial pathogens". Infection, Genetics and Evolution. 8 (3): 267–85. doi:10.1016/j.meegid.2008.01.002. PMID 18295550.http://www.hummingbirds.arizona.edu/Faculty/Michod/Downloads/IGE%20review%20sex.pdf
- ^ a b Su S, Wong G, Si W, Liu J, Lai ACK, Zou J, Liu W, Bi Y, Gao GF.코로나비루스의 역학, 유전적 재조합, 병태생성.Trends Microbiol. 2016년 6월 24일:490-502.doi:10.1016/j.tim.2016.03.003.003.Epub 2016년 3월 21일.검토PMID 27012512
- ^ 바 JN, Fearns R.RNA 바이러스가 게놈 무결성을 유지하는 방법.J Gen Virol. 2010년 6월;91(Pt 6):1373-87. doi: 10.1099/vir.020818-0.Epub 2010년 3월 24일.검토PMID 20335491
- ^ a b LiX, Giorgi EE, Marichanngowda MH, Foley B, Xiao C, Kong XP, Chen Y, Gnanakaran S, Korber B, Gao F. 재조합과 강력한 정화를 통한 SARS-CoV-2 출현.Sci Adv. 2020년 7월 1일;6일(27):eabb9153. doi: 10.1126/sciadv.abb9153.PMID 32937441
- ^ 레만 SU, 샤피크 L, 이흐산 A, 류큐.소설 코로나바이러스 SARS-CoV-2 출현을 위한 진화 궤적병원균.2020년 3월 23일 (3):240. doi: 10.3390/병원균 9030240.PMID 32210130; PMCID: PMC7157669
- ^ Yeh TY, Contreras GP (July 2020). "Emerging viral mutants in Australia suggest RNA recombination event in the SARS-CoV-2 genome". The Medical Journal of Australia. 213 (1): 44–44.e1. doi:10.5694/mja2.50657. PMC 7300921. PMID 32506536.
- ^ Yeh TY, Contreras GP (1 July 2021). "Viral transmission and evolution dynamics of SARS-CoV-2 in shipboard quarantine". Bull. World Health Organ. 99 (7): 486–495. doi:10.2471/BLT.20.255752. PMC 8243027. PMID 34248221.
- ^ Lamb NE, Yu K, Shaffer J, Feingold E, Sherman SL (January 2005). "Association between maternal age and meiotic recombination for trisomy 21". American Journal of Human Genetics. 76 (1): 91–9. doi:10.1086/427266. PMC 1196437. PMID 15551222.
- ^ Cold Spring Harbor Laboratory (2007). "Human RecQ Helicases, Homologous Recombination And Genomic Instability". ScienceDaily. Retrieved 3 July 2010.
- ^ Modesti M, Kanaar R (2001). "Homologous recombination: from model organisms to human disease". Genome Biology. 2 (5): REVIEWS1014. doi:10.1186/gb-2001-2-5-reviews1014. PMC 138934. PMID 11387040.
- ^ Luo G, Santoro IM, McDaniel LD, Nishijima I, Mills M, Youssoufian H, Vogel H, Schultz RA, Bradley A (December 2000). "Cancer predisposition caused by elevated mitotic recombination in Bloom mice". Nature Genetics. 26 (4): 424–9. doi:10.1038/82548. PMID 11101838. S2CID 21218975.
- ^ a b Alberts B, Johnson A, Lewis J, Raff M, Roberts K, Walter P (2007). Molecular Biology of the Cell (5th ed.). Garland Science. ISBN 978-0-8153-4110-9.
- ^ a b c Powell SN, Kachnic LA (September 2003). "Roles of BRCA1 and BRCA2 in homologous recombination, DNA replication fidelity and the cellular response to ionizing radiation". Oncogene. 22 (37): 5784–91. doi:10.1038/sj.onc.1206678. PMID 12947386.
- ^ "Use of homologous recombination deficiency (HRD) score to enrich for niraparib sensitive high grade ovarian tumors". Archived from the original on 2017-04-30. Retrieved 2016-12-30.
- ^ a b c d e f g h Lin Z, Kong H, Nei M, Ma H (July 2006). "Origins and evolution of the recA/RAD51 gene family: evidence for ancient gene duplication and endosymbiotic gene transfer". Proceedings of the National Academy of Sciences of the United States of America. 103 (27): 10328–33. Bibcode:2006PNAS..10310328L. doi:10.1073/pnas.0604232103. PMC 1502457. PMID 16798872.
- ^ a b Haseltine CA, Kowalczykowski SC (May 2009). "An archaeal Rad54 protein remodels DNA and stimulates DNA strand exchange by RadA". Nucleic Acids Research. 37 (8): 2757–70. doi:10.1093/nar/gkp068. PMC 2677860. PMID 19282450.
- ^ Rolfsmeier ML, Haseltine CA (March 2010). "The single-stranded DNA binding protein of Sulfolobus solfataricus acts in the presynaptic step of homologous recombination". Journal of Molecular Biology. 397 (1): 31–45. doi:10.1016/j.jmb.2010.01.004. PMID 20080104.
- ^ Huang Q, Liu L, Liu J, Ni J, She Q, Shen Y (2015). "Efficient 5'-3' DNA end resection by HerA and NurA is essential for cell viability in the crenarchaeon Sulfolobus islandicus". BMC Molecular Biology. 16: 2. doi:10.1186/s12867-015-0030-z. PMC 4351679. PMID 25880130.

- ^ Jain SK, Cox MM, Inman RB (August 1994). "On the role of ATP hydrolysis in RecA protein-mediated DNA strand exchange. III. Unidirectional branch migration and extensive hybrid DNA formation". The Journal of Biological Chemistry. 269 (32): 20653–61. doi:10.1016/S0021-9258(17)32043-4. PMID 8051165.
- ^ Ramesh MA, Malik SB, Logsdon JM (January 2005). "A phylogenomic inventory of meiotic genes; evidence for sex in Giardia and an early eukaryotic origin of meiosis". Current Biology. 15 (2): 185–91. doi:10.1016/j.cub.2005.01.003. PMID 15668177. S2CID 17013247.
- ^ a b Malik SB, Ramesh MA, Hulstrand AM, Logsdon JM (December 2007). "Protist homologs of the meiotic Spo11 gene and topoisomerase VI reveal an evolutionary history of gene duplication and lineage-specific loss". Molecular Biology and Evolution. 24 (12): 2827–41. doi:10.1093/molbev/msm217. PMID 17921483.
- ^ Lodish H, Berk A, Zipursky SL, Matsudaira P, Baltimore D, Darnell J (2000). "Chapter 8.5: Gene Replacement and Transgenic Animals: DNA Is Transferred into Eukaryotic Cells in Various Ways". Molecular Cell Biology (4th ed.). W. H. Freeman and Company. ISBN 978-0-7167-3136-8.
- ^ "The Nobel Prize in Physiology or Medicine 2007". The Nobel Foundation. Retrieved December 15, 2008.
- ^ Drummond DA, Silberg JJ, Meyer MM, Wilke CO, Arnold FH (April 2005). "On the conservative nature of intragenic recombination". Proceedings of the National Academy of Sciences of the United States of America. 102 (15): 5380–5. Bibcode:2005PNAS..102.5380D. doi:10.1073/pnas.0500729102. PMC 556249. PMID 15809422.
- ^ Bloom JD, Silberg JJ, Wilke CO, Drummond DA, Adami C, Arnold FH (January 2005). "Thermodynamic prediction of protein neutrality". Proceedings of the National Academy of Sciences of the United States of America. 102 (3): 606–11. arXiv:q-bio/0409013. Bibcode:2005PNAS..102..606B. doi:10.1073/pnas.0406744102. PMC 545518. PMID 15644440.
- ^ a b Carbone MN, Arnold FH (August 2007). "Engineering by homologous recombination: exploring sequence and function within a conserved fold". Current Opinion in Structural Biology. 17 (4): 454–9. doi:10.1016/j.sbi.2007.08.005. PMID 17884462.
- ^ Otey CR, Landwehr M, Endelman JB, Hiraga K, Bloom JD, Arnold FH (May 2006). "Structure-guided recombination creates an artificial family of cytochromes P450". PLOS Biology. 4 (5): e112. doi:10.1371/journal.pbio.0040112. PMC 1431580. PMID 16594730.

- ^ Socolich M, Lockless SW, Russ WP, Lee H, Gardner KH, Ranganathan R (September 2005). "Evolutionary information for specifying a protein fold". Nature. 437 (7058): 512–8. Bibcode:2005Natur.437..512S. doi:10.1038/nature03991. PMID 16177782. S2CID 4363255.
- ^ Thulasiram HV, Erickson HK, Poulter CD (April 2007). "Chimeras of two isoprenoid synthases catalyze all four coupling reactions in isoprenoid biosynthesis". Science. 316 (5821): 73–6. Bibcode:2007Sci...316...73T. doi:10.1126/science.1137786. PMID 17412950. S2CID 43516273.
- ^ Landwehr M, Carbone M, Otey CR, Li Y, Arnold FH (March 2007). "Diversification of catalytic function in a synthetic family of chimeric cytochrome p450s". Chemistry & Biology. 14 (3): 269–78. doi:10.1016/j.chembiol.2007.01.009. PMC 1991292. PMID 17379142.
- ^ Choudhury, Ananya; Zhao, Helen; Jalali, Farid; Rashid, Shahnaz AL; Ran, Jane; Supiot, Stephane; Kiltie, Anne E.; Bristow, Robert G. (1 January 2009). "Targeting homologous recombination using imatinib results in enhanced tumor cell chemosensitivity and radiosensitivity". Molecular Cancer Therapeutics. 8 (1): 203–213. doi:10.1158/1535-7163.MCT-08-0959. ISSN 1535-7163. PMID 19139130. S2CID 6493434.
- ^ Siddiqui, Arafat; Tumiati, Manuela; Joko, Alia; Sandholm, Jouko; Roering, Pia; Aakko, Sofia; Vainionpää, Reetta; Kaipio, Katja; Huhtinen, Kaisa; Kauppi, Liisa; Tuomela, Johanna; Hietanen, Sakari (2021). "Targeting DNA Homologous Repair Proficiency With Concomitant Topoisomerase II and c-Abl Inhibition". Frontiers in Oncology. 11: 3666. doi:10.3389/fonc.2021.733700. ISSN 2234-943X. PMC 8488401. PMID 34616682.
- ^ a b c Iglehart JD, Silver DP (July 2009). "Synthetic lethality--a new direction in cancer-drug development". The New England Journal of Medicine. 361 (2): 189–91. doi:10.1056/NEJMe0903044. PMID 19553640.
- ^ Fong PC, Boss DS, Yap TA, Tutt A, Wu P, Mergui-Roelvink M, Mortimer P, Swaisland H, Lau A, O'Connor MJ, Ashworth A, Carmichael J, Kaye SB, Schellens JH, de Bono JS (July 2009). "Inhibition of poly(ADP-ribose) polymerase in tumors from BRCA mutation carriers". The New England Journal of Medicine. 361 (2): 123–34. doi:10.1056/NEJMoa0900212. PMID 19553641.
- ^ Edwards SL, Brough R, Lord CJ, Natrajan R, Vatcheva R, Levine DA, Boyd J, Reis-Filho JS, Ashworth A (February 2008). "Resistance to therapy caused by intragenic deletion in BRCA2". Nature. 451 (7182): 1111–5. Bibcode:2008Natur.451.1111E. doi:10.1038/nature06548. PMID 18264088. S2CID 205212044.
외부 링크
| 라이브러리 리소스 정보 호몰로 재조합 |
| Wikimedia Commons는 호몰로게이션 재조합과 관련된 미디어를 가지고 있다. |