발암
Carcinogenesis
암 발생 또는 종양 발생이라고도 불리는 발암은 정상적인 세포가 암세포로 변환되는 암의 형성이다.이 과정은 세포, 유전적, 후생유전적 수준의 변화와 비정상적인 세포 분열로 특징지어진다.세포 분열은 거의 모든 조직과 다양한 상황에서 일어나는 생리학적 과정이다.일반적으로 세포자멸의 형태로 증식과 프로그램된 세포사이의 균형은 조직과 장기의 무결성을 보장하기 위해 유지된다.널리 받아들여지고 있는 발암 이론에 따르면, 체세포 돌연변이 이론, DNA의 돌연변이 그리고 암을 유발하는 에피메이션은 이러한 과정을 조절하는 프로그래밍을 방해함으로써 이러한 질서 있는 과정을 방해하고, 증식과 세포 죽음 [1][2][3][4][5]사이의 정상적인 균형을 깨뜨린다.이는 통제되지 않은 세포 분열과 신체 내 자연 선택에 의한 세포 진화를 초래한다.특정 돌연변이만이 암을 유발하지만 대부분의 돌연변이는 그렇지 않다.[citation needed]
유전 유전자의 변종은 개인에게 암을 유발시킬 수 있다.또한 발암물질과 방사선과 같은 환경적 요인은 암 발생에 기여할 수 있는 돌연변이를 일으킨다.마지막으로, 정상적인 DNA 복제에서 무작위적인 실수가 [6]돌연변이를 일으키는 암을 유발할 수 있습니다.보통 정상 세포가 암세포로 [7][8][9][10][11]변하기 전에 특정 종류의 유전자에 대한 일련의 돌연변이가 필요하다.TCGA 코호트에서 운전자 이벤트의 최근 환자 수준 분류 및 정량화에 따르면 종양당 운전자 이벤트는 평균 12개이며, 그 중 0.6개는 종양유전자의 포인트 돌연변이, 1.5개는 종양유전자의 포인트 돌연변이, 1.2개는 종양억제자의 포인트 돌연변이, 2.1개는 종양억제자의 결실, 1.5개는 드라이버이다.염색체 손실, 1은 드라이버 염색체 증가, 2는 드라이버 염색체 팔 손실, 1.5는 드라이버 염색체 팔 [12]증가입니다.세포분열, 세포자멸(세포사망), DNA수복을 조절하는 유전자의 돌연변이는 통제되지 않은 세포증식과 암을 초래할 수 있다.
암은 근본적으로 조직의 성장을 조절하는 질병이다.정상적인 세포가 암세포로 변하기 위해서는 세포의 성장과 분화를 조절하는 유전자가 [13]바뀌어야 한다.유전자 및 후생유전학적 변화는 전체 염색체의 획득 또는 손실, 단일 DNA 뉴클레오티드에 영향을 미치는 돌연변이, 또는 100에서 [14][15]500개의 유전자의 발현을 제어하는 마이크로RNA를 침묵시키거나 활성화하는 등 다양한 수준에서 발생할 수 있다.이러한 변화에 영향을 받는 유전자에는 크게 두 가지 범주가 있다.종양유전자는 부적절하게 높은 수준으로 발현되는 정상적인 유전자이거나 새로운 특성을 가진 변형된 유전자일 수 있다.어느 경우든, 이러한 유전자의 발현은 암세포의 악성 표현형을 촉진한다.종양 억제 유전자는 암세포의 세포 분열, 생존 또는 다른 특성을 억제하는 유전자이다.종양 억제 유전자는 암을 유발하는 유전자 변화로 인해 종종 장애를 일으킨다.마지막으로 Oncovirinae는 종양유전자를 포함하는 바이러스로서 숙주의 종양조직의 성장을 유발하기 때문에 종양유전자로 분류된다.이 과정을 바이러스 변형이라고도 합니다.
원인들
유전적 및 후생적
암세포 생성에 기여할 수 있는 다양한 게놈 변화에 대한 다양한 분류 체계가 있다.이러한 변화들 중 다수는 돌연변이, 즉 게놈 DNA의 뉴클레오티드 배열의 변화이다.유전자 발현 여부를 바꾸는 후생유전학적 변화도 많다.비정상적인 수의 염색체의 존재인 무배수는 돌연변이가 아닌 하나의 게놈 변화이며 유사분열 오류를 통해 하나 이상의 염색체를 얻거나 잃을 수 있다.대규모 돌연변이는 염색체의 일부분을 제거하거나 복제하는 것을 포함한다.게놈 증폭은 세포가 보통 하나 이상의 종양 유전자와 인접한 유전 물질을 포함하는 작은 염색체 영역의 많은 복사본(종종 20개 이상)을 획득할 때 발생합니다.전위는 두 개의 분리된 염색체 영역이 비정상적으로 융합될 때 종종 특징적인 위치에서 발생한다.이것의 잘 알려진 예는 만성 골수성 백혈병에서 발생하고 발암성 티로신 키나아제인 BCR-abl 융합 단백질의 생산을 초래하는 필라델피아 염색체 또는 9번과 22번 염색체의 전위이다.소규모 돌연변이는 유전자의 프로모터에서 일어나 발현에 영향을 미치거나 유전자의 코드 배열에서 일어나 단백질 생성물의 기능이나 안정성을 변화시킬 수 있는 점 돌연변이, 결실 및 삽입을 포함한다.단일 유전자의 파괴는 DNA 바이러스 또는 레트로 바이러스로부터의 게놈 물질의 통합에 의해서도 발생할 수 있으며, 그러한 현상은 또한 영향을 받는 세포와 [citation needed]그 자손의 바이러스성 종양 유전자의 발현을 초래할 수도 있다.
DNA손상
DNA 손상은 [16]암의 주요 원인으로 여겨진다.내인성 세포 작용에 의해 하루에 평균 60,000건 이상의 자연 발생 DNA 손상 사례가 발생한다(자연 발생 기사 DNA 손상 참조).
외인성 물질에 노출되면 추가적인 DNA 손상이 발생할 수 있습니다.외인성 발암물질의 한 예로 담배연기는 DNA 손상을 증가시키고, 이 DNA 손상은 [17]흡연으로 인한 폐암의 증가를 일으킬 수 있다.다른 예로는 태양 복사로 인한 자외선은 [18]흑색종에서 중요한 DNA 손상을 일으키고 헬리코박터균 감염은 DNA를 손상시켜 [19]위암에 기여하는 고농도의 활성산소를 생성하며 아스페르길루스 플라버스 대사물 아플라톡신은 [20]간암을 일으키는 DNA 손상제이다.
DNA 손상은 또한 체내에서 생성된 물질에 의해서도 발생할 수 있다.염증이 있는 대장상피 속의 대식세포와 호중구는 활성산소의 원천으로 대장종양생성을 시작하는 [21]DNA 손상을 일으킨다.그리고 고지방식을 하는 인간의 대장에서도 높은 수준의 담즙산이 DNA 손상을 일으키고 대장암에 [22]기여한다.
이러한 외인성 및 내인성 DNA 손상의 원인은 이 섹션의 그림 상단에 있는 상자에 나와 있습니다.암으로 진행되는 DNA 손상의 중심적인 역할은 그림의 두 번째 수준에 나타나 있다.DNA 손상, 후생유전학적 변화 및 암으로 진행되는 부족한 DNA 수복의 중심 요소는 빨간색으로 표시됩니다.
DNA 수복의 결핍은 더 많은 DNA 손상을 축적시키고 암 발병 위험을 증가시킨다.예를 들어, 34개의 DNA 복구 유전자 중 하나에서 유전적인 장애가 있는 개인(기사 DNA 복구-결핍 장애 참조)은 암에 걸릴 위험이 높아지며, 일부 결함으로 인해 평생 암에 걸릴 확률이 최대 100%가 된다(예: p53 돌연변이).[23]이러한 생식선 돌연변이는 그림 왼쪽에 있는 상자에 표시되며 DNA 복구 결핍에 대한 기여도를 나타냅니다.하지만, 그러한 생식계 돌연변이는 암의 [24]약 1%의 원인일 뿐이다.
대부분의 암은 비유전성 암 또는 "포자성 암"이라고 불립니다.산발성 암의 약 30%는 현재 정의되지 않은 유전 성분을 가지고 있는 반면, 산발성 암의 70%인 대부분은 유전 성분을 [25]가지고 있지 않다.
산발성 암에서, DNA 수복의 결핍은 때때로 DNA 수복 유전자의 돌연변이에 기인한다; 훨씬 더 자주, DNA 수복 유전자의 발현 감소 또는 부재는 유전자 발현을 감소시키거나 침묵시키는 후생 유전자의 변화 때문이다.이것은 위에서 세 번째 레벨의 그림에 나타나 있습니다.예를 들어 순차적으로 검사한 대장암 113개의 경우, DNA 복구 유전자 MGMT에서 미센스 돌연변이를 보인 사람은 4명뿐이었고, 대부분은 MGMT 프로모터 영역의 메틸화로 인해 MGMT 발현을 감소시켰다(후생유전자 [26]변화).
DNA 수복 유전자의 발현이 감소하면 DNA 수복 부족이 발생한다.이것은 위에서 4번째 레벨의 그림에 나와 있습니다.DNA 복구 결핍이 있는 경우, DNA 손상은 일반적인 수준보다 높은 수준(그림 상단에서 5번째 수준)에서 세포에 지속되며, 이러한 초과 손상은 돌연변이 및/또는 발모(그림 상단에서 6번째 수준)의 빈도를 증가시킨다.실험적으로, 돌연변이율은 DNA 불일치[27][28] 복구 또는 호몰로지 재조합 복구(HRR)[29]에서 결함이 있는 세포에서 상당히 증가한다.염색체 재배열과 무배수도 또한 HRR 결손[30] 세포의 증가 DNA 이중 가닥 파괴의 복구 또는 다른 DNA 손상의 복구 동안, 불완전하게 제거된 복구 부위는 후생 유전자의 [31][32]침묵을 야기할 수 있습니다.
DNA 손상과 DNA 수복결핍으로 인한 체세포 돌연변이와 후생유전적 변화는 현장결함에 축적된다.필드 결함은 여러 가지 변화가 있는 정상 출현 조직(아래 절에서 설명)으로, 암에서 조직의 무질서하고 과잉 증식하는 클론의 발달에 대한 일반적인 전조이다.이러한 필드 결함(그림의 아래쪽에서 두 번째 레벨)은 수많은 돌연변이와 후생유전학적 변화를 가질 수 있다.
대부분의 특정 암의 초기 원인을 규명하는 것은 불가능하다.몇 가지 경우, 단 하나의 원인만 존재합니다. 예를 들어 바이러스 HHV-8이 모든 카포시 육종을 유발합니다.그러나, 암 역학 기법과 정보의 도움으로, 더 많은 상황에서 가능한 원인에 대한 추정치를 도출할 수 있다.예를 들어, 폐암은 담배 사용과 라돈 가스를 포함한 여러 가지 원인이 있다.현재 담배를 피우고 있는 남성은 담배를 피우지 않은 남성의 14배 비율로 폐암에 걸린다.현재 흡연자의 폐암이 흡연에 의해 발생할 확률은 약 93%이다. 흡연자의 폐암이 라돈 가스나 다른 비담배 [33]원인에 의해 유발될 확률은 7%이다.이러한 통계적 상관관계는 연구자들이 특정 물질이나 행동이 발암성이라고 추론하는 것을 가능하게 했다.담배 연기는 외인성 DNA 손상을 증가시키고, 이 DNA 손상이 흡연으로 인한 폐암의 원인일 가능성이 높다.담배연기 중 5,000개 이상의 화합물 중 가장 높은 농도로 발생하며 가장 강한 돌연변이 유발 효과를 가진 유전독성 DNA 손상제는 아크로레인, 포름알데히드, 아크릴로니트릴, 1,3-부타디엔, 아세트알데히드, 산화 [17]에틸렌, 이소프렌이다.
분자생물학적 기법을 이용해 종양 내 돌연변이, 에피메이션, 염색체 이상을 특징지을 수 있고, 돌연변이 스펙트럼을 바탕으로 특정 암 환자의 예후를 예측하는 분야에서도 비약적인 진전이 이루어지고 있다.예를 들어, 모든 종양의 절반까지 p53 유전자가 결함이 있다.이 돌연변이는 치료로 손상되었을 때 종양세포가 아포토시스나 프로그램된 세포사망으로 갈 가능성이 낮기 때문에 나쁜 예후와 관련이 있다.텔로머라아제 돌연변이는 추가적인 장벽을 제거하여 세포가 분열할 수 있는 횟수를 늘린다.다른 돌연변이는 종양이 더 많은 영양분을 공급하기 위해 새로운 혈관을 자라게 하거나 신체의 다른 부분으로 전이되도록 한다.그러나 일단 암이 형성되면 암은 계속 진화하고 하위 클론을 생성한다.2012년 9개의 다른 영역에서 표본 추출된 단일 신장암 검체는 9개 영역 모두에서 40개의 "유비쿼터스" 돌연변이를 발견했으며, 일부 영역에서 59개의 돌연변이가 발견되었지만 9개 영역 모두에서 발견된 것은 아니었다. 그리고 29개의 "개인" 돌연변이가 한 [34]영역에서만 존재했다고 보고되었다.
이러한 모든 DNA 변화가 축적되는 세포의 혈통은 추적하기 어렵지만, 최근 두 가지 증거에 따르면 정상 줄기세포가 [35][36]암에서 유래한 세포일 수 있다.첫째, 조직 내 암 발병 위험과 동일한 조직에서 발생하는 정상 줄기세포 분열 수 사이에는 매우 양의 상관관계(Spearman's rho = 0.81, P < 3.5 × 10-8)가 존재한다.상관관계는 31가지 암 유형에 적용되었고 5가지 [37]등급으로 확대되었다.이 상관관계는 조직의 정상적인 줄기세포가 한 번 분열하면 그 조직의 암 위험이 약 1배라는 것을 의미한다.1,000번 나누면 발암 위험이 1,000배입니다.만약 정상 줄기세포가 10만 번 분열한다면그 조직의 암 위험은 약 10만 배입니다이는 암 발생의 주요 요인이 "정상"[36] 줄기세포가 분열한다는 사실이라는 것을 강하게 시사한다. 이는 암이 정상적이고 건강한 줄기세포에서 발생한다는 것을 의미한다.
둘째, 통계는 대부분의 인간 암은 노인에게서 진단된다는 것을 보여준다.가능한 설명은 세포가 시간이 지나면서 손상을 축적하기 때문에 암이 발생한다는 것이다.DNA는 생명체의 전 과정에 걸쳐 손상을 축적할 수 있는 유일한 세포 구성 요소이고, 줄기세포는 접합자에서 늦게 세포로 DNA를 전달할 수 있는 유일한 세포이다.줄기세포에서 파생된 다른 세포들은 암이 발생할 가능성이 있을 때까지 DNA를 생명 초기부터 보관하지 않는다.이는 대부분의 암이 정상적인 줄기세포에서 [35][36]발생한다는 것을 의미한다.
필드 결함의 기여
"장암화"라는 용어는 1953년에 상피의 한 영역 또는 "장"을 설명하기 위해 처음 사용되었으며, 상피가 [38]암 발병으로 발전하기 위해 (당시) 거의 알려지지 않은 과정에 의해 사전 조정되었다.그 이후로, "장암화"와 "장결함"이라는 용어는 새로운 [citation needed]암이 발생할 가능성이 있는 악성 조직 전(前) 조직을 설명하기 위해 사용되어 왔다.
현장결함은 암과 관련하여 확인되었으며 [39][40]암의 진행에 중요하다.그러나[41] Rubin은 "암 연구에서 대부분의 연구는 생체 내 잘 정의된 종양 또는 체외 이산 종양에 대해 수행되었다"고 지적했다.그러나 돌연변이 표현형 인간 대장종양에서 발견되는 체세포 돌연변이의 80% 이상이 말기 클론 [42]확장이 시작되기 전에 발생한다는 증거가 있다…." 종양에서 확인된 체세포 돌연변이의 절반 이상은 명백한 정상세포의 성장 중에 종양이 생기기 전 단계에서 발생했다.또한 종양에 존재하는 후생유전적 변화 중 다수는 종양이 생기기 전의 [43]장결함에서 발생할 수 있다.
대장 내부 표면의 장내 크립트 중 하나의 기부에 있는 줄기세포 중 돌연변이 세포 또는 후생유전적으로 변경된 세포가 자연선택됨으로써 장내 결함이 발생할 수 있다.돌연변이 또는 후생유전적으로 변경된 줄기세포는 자연선택에 의해 근처의 다른 줄기세포를 대체할 수 있다.이로 인해 비정상적인 조직의 패치가 발생할 수 있습니다.이 섹션의 그림에는 대장암과 용종 4개를 보여주는 새로 절제된 세로 방향으로 열린 결장 부분의 사진이 포함되어 있습니다.사진 아래에는 돌연변이 세포 또는 후생유전적으로 변경된 세포의 큰 패치가 어떻게 형성되었는지에 대한 도식도가 있으며, 다이어그램의 노란색으로 표시된 큰 면적이 표시되어 있습니다.이 다이어그램의 첫 번째 큰 패치(세포의 큰 클론) 내에서 두 번째 그러한 돌연변이 또는 후생유전적 변화가 일어날 수 있으며, 따라서 주어진 줄기세포가 그 이웃에 비해 이점을 획득할 수 있으며, 이 변경된 줄기세포는 복제적으로 확장되어 원래의 패치 내에서 2차 패치 또는 서브 클론을 형성할 수 있다.이는 큰 노란색 원래 영역 내에 다른 색상의 4개의 작은 패치로 다이어그램에 나타나 있습니다.이러한 새로운 패치(서브클론) 내에서, 작은 용종 또는 악성 종양(암)을 생성하는 줄기세포가 발생할 때까지 복제적으로 확장되는 4개의 2차 패치(그래픽에서 여전히 다른 색상으로) 내의 더 작은 패치로 나타나는 과정을 여러 번 반복할 수 있다.사진에서 결장의 이 부분에서 명백한 필드 결손이 4개의 용종을 생성했습니다(용종의 크기, 6mm, 5mm, 3mm의 2개, 가장 긴 치수로 약 3cm의 암으로 라벨 표시됨).이러한 종양은 또한 (사진 아래 다이어그램에서) 4개의 작은 황갈색 원(용종)과 더 큰 빨간색 영역(암)으로 표시됩니다.사진 속 암은 대장이 소장과 결합하고(표지) 충수가 발생하는(표지) 결장의 맹장 부위에서 발생했다.사진 속의 지방은 결장의 외벽 바깥쪽에 있다.여기에 표시된 결장 부분에서는 대장을 세로로 절단하여 내부 표면을 드러내고 [citation needed]대장의 내부 상피 라이닝 내에서 발생하는 암과 용종을 표시하였다.
만약 산발적인 대장암이 발생하는 일반적인 과정이 자연선택에 의해 퍼지는 종양이 생기기 전 클론의 형성이고, 그 후 초기 클론 내의 내부 서브클론 및 그 내부의 서브클론의 형성이라면, 대장암은 일반적으로 비정상성 증가의 장과 관련되어야 하며, 그 앞에 와야 한다.예전의 사건의 연속을 반영하는 y.가장 광범위한 이상 영역(도표에서 가장 바깥쪽 노란색 불규칙 영역)은 악성 종양 형성의 가장 이른 사건을 반영한다.
암의 특정 DNA 복구 결핍에 대한 실험 평가에서, 많은 특정 DNA 복구 결핍이 해당 암을 둘러싼 현장 결함에 발생하는 것으로 나타났다.아래 표는 암의 DNA 복구 결핍이 후생유전학적 변화에 의해 유발된 것으로 나타난 예와, 주변 필드 결함에서 동일한 후생유전적으로 DNA 복구 결핍을 야기한 빈도가 다소 낮은 예시를 보여준다.
| 암 | 진 | 암 발생 빈도 | 필드 결함의 빈도 | 언급 |
|---|---|---|---|---|
| 대장균 | 관리 | 46% | 34% | [44] |
| 대장균 | 관리 | 47% | 11% | [45] |
| 대장균 | 관리 | 70% | 60% | [46] |
| 대장균 | MSH2 | 13% | 5% | [45] |
| 대장균 | ERCC1 | 100% | 40% | [47] |
| 대장균 | PMS2 | 88% | 50% | [47] |
| 대장균 | XPF | 55% | 40% | [47] |
| 머리와 목 | 관리 | 54% | 38% | [48] |
| 머리와 목 | MLH1 | 33% | 25% | [49] |
| 머리와 목 | MLH1 | 31% | 20% | [50] |
| 배 | 관리 | 88% | 78% | [51] |
| 배 | MLH1 | 73% | 20% | [52] |
| 식도 | MLH1 | 77%–100% | 23%–79% | [53] |
개방된 대장 세그먼트의 사진에 나타난 필드 결함의 작은 용종 중 일부는 비교적 양성인 종양일 수 있다.1996년 대장내시경 검사에서 발견된 10mm 미만의 용종을 대상으로 3년간 반복 대장내시경 검사를 실시한 결과 25%는 크기가 변하지 않았고 35%는 크기가 퇴보 또는 축소되었으며 40%는 [54]크기가 커졌다.
게놈 불안정성
암은 게놈 불안정 또는 "변이자 표현형"[55]을 보이는 것으로 알려져 있다.핵 내의 단백질 코드 DNA는 총 게놈 [56]DNA의 약 1.5%이다.이 단백질 코드 DNA (엑솜이라고 불림) 안에서, 유방 또는 결장의 평균 암은 약 60에서 70개의 단백질을 변화시키는 돌연변이를 가질 수 있으며, 그 중 약 3 또는 4개는 "운전자" 돌연변이일 수 있고, 나머지 것들은 "승객"[43] 돌연변이일 수 있다.그러나 유방암 조직 샘플 내 전체 게놈(비단백질 코드화 영역 포함)의 평균 DNA 염기서열 돌연변이 수는 [57]약 20,000개이다.평균 흑색종 조직 샘플에서(멜라노마는 더 높은 엑솜 돌연변이 [43]빈도를 가진다) 총 DNA 배열 돌연변이 수는 약 80,[58]000개입니다.암 내 전체 뉴클레오티드 배열에서 이러한 높은 빈도의 돌연변이는 종종 암을 유발하는 필드 결함의 초기 변화(예: 이전 섹션의 다이어그램의 노란색 영역)가 DNA 수복의 부족임을 시사한다.대장암을 둘러싼 대규모 필드 결손(암 양쪽에서 약 10cm까지 연장)은 필드 결손의 전체 영역에서 2개 또는 3개의 DNA 복구 단백질(ERCC1, ERCC4(XPF) 및/또는 PMS2)에서 후생유전학적 결함을 자주 가진다[47].DNA 수복 유전자의 발현이 감소하면 세포에 정상보다 높은 속도로 DNA 손상이 축적되고, 이 과잉 손상은 돌연변이 및/또는 발모 빈도를 증가시킨다.DNA 불일치[27][28] 복구 또는 상동 재조합 복구(HRR)[29]에서 결함이 있는 세포에서 돌연변이율이 크게 증가한다.DNA 복구 자체의 결핍은 DNA 손상이 축적될 수 있으며, 손상 부위의 오류 발생 가능성이 높은 전이 합성이 돌연변이를 일으킬 수 있다.또한, 축적된 DNA 손상에 대한 잘못된 복구는 에피메이션이 발생할 수 있습니다.이러한 새로운 돌연변이 및/또는 에피메이션은 증식상의 이점을 제공하여 필드 결함을 발생시킬 수 있습니다.DNA 수복 유전자의 돌연변이/분열 자체는 선택적인 이점을 주지 않지만, 세포가 증식적 [citation needed]이점을 제공하는 추가적인 돌연변이/분열을 획득할 때 세포 내 이동자로서 운반될 수 있다.
비주류 이론
과학적 근거, 논리 또는 근거의 부족으로 인해 과학적 의견의 주류를 벗어난 발암과 암 치료의 많은 이론들이 있다.이 이론들은 다양한 대체 암 치료법을 정당화하기 위해 사용될 수 있다.그것들은 주류 암 생물학 내에서 논리적인 근거를 가지고 있고, 전통적으로 시험 가능한 가설을 만들 [citation needed]수 있는 발암 이론과 구별되어야 한다.
그러나 발암에 대한 몇 가지 대체 이론은 과학적 증거에 기초하고 있으며 점점 더 인정받고 있다.일부 연구자들은 암이 돌연변이나 외형에 의한 것이 아니라 반수체([59]염색체의 수치적이고 구조적인 이상)에 의해 유발될 수 있다고 믿는다.암은 또한 산소의 세포대사가 에너지를 생성하는 경로(산소인산화)에서 활성산소종을 [60]생성하는 경로로 전환되는 대사 질환으로 간주되어 왔다.이는 산화적 인산화에서 호기성 당분해로 에너지 전환(워버그 가설)을 유발하고 산화적 스트레스("[60]암의 산화적 스트레스 이론")로 이어지는 활성 산소 종의 축적을 유발한다.암 발생의 또 다른 개념은 약한 자기장과 전자기장에 대한 노출과 그들이 산화적 스트레스에 미치는 영향에 기초하고 있는데, 이는 자기암 [61]발생으로 알려져 있다.
많은 저자들은 암이 순차적 무작위 돌연변이에 의해 발생한다는 가정에 대해 지나치게 단순하다고 의문을 제기했고, 대신에 암이 선천적이고 프로그램된 증식 [62]경향을 억제하는 신체의 실패에서 비롯된다는 것을 시사했다.관련된 이론은 암이 격세지감이며, 초기 형태의 다세포 [63]생명체로의 진화적 역행이라는 것을 암시한다.제어되지 않은 세포 성장과 암세포 간의 협력을 담당하는 유전자는 최초의 다세포 생명체가 함께 모여 번성할 수 있게 해준 유전자와 매우 유사합니다.이 유전자들은 인간과 같은 더 복잡한 메타조아들의 게놈 안에 존재하지만, 더 최근에 진화한 유전자들은 그것들을 억제한다.새로운 조절 유전자가 어떤 이유로든 실패하면, 세포는 더 원시적인 프로그래밍으로 돌아가 통제 불능 상태로 번식할 수 있다.이 이론은 암이 몸 안에서 진화를 겪는 불량 세포에서 시작된다는 개념의 대안이다.대신에, 그들은 점진적으로 활성화되는 고정된 수의 원시 유전자를 가지고 있어,[64] 그들에게 유한한 가변성을 준다.또 다른 진화론은 숙주에 의해 감염 바이러스의 게놈이 절단되었지만 면역 보호 차원에서 그들의 조각이 숙주의 게놈에 통합되었을 때 거대한 수평 유전자 전달에 의해 암의 뿌리를 진핵세포의 기원으로 되돌린다.따라서 암은 희귀한 체세포 돌연변이가 이러한 조각들을 세포 [65]증식의 기능적 동인으로 재조합할 때 발생한다.
암세포생물학
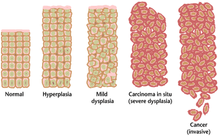
종종 암을 일으키는 여러 가지 유전자 변화가 축적되는 데 오랜 시간이 걸릴 수 있다.이 기간 동안, 악성화 전세포의 생물학적 행동은 정상세포의 특성에서 암과 유사한 특성으로 서서히 변화한다.상처받기 전의 조직은 현미경 아래서도 뚜렷한 외관을 가질 수 있다.전악성 병변의 구별되는 특징으로는 분열세포의 수 증가, 핵의 크기와 형상의 변화, 세포 크기와 형상의 변화, 특수한 세포 특징의 상실 및 정상 조직 조직의 손실이 있다.이형성증은 정상적인 조직 배열과 악성화 전 세포 구조의 상실을 특징으로 하는 과도한 세포 증식의 비정상적인 유형이다.이러한 초기 종양 변화는 호르몬 불균형이나 만성 [citation needed]자극과 같은 외부 자극에 의해 야기되는 세포 분열의 가역적 증가인 과형성과 구별되어야 한다.
가장 심각한 이형성증 환자는 현장 암종이라고 한다.라틴어로 in situit라는 용어는 "in sit"를 의미하며, in situit의 암은 원래 위치에 남아 다른 조직에 침입을 보이지 않는 이형성 세포의 통제되지 않은 성장을 의미합니다.위치암은 침습성 악성 종양으로 발전할 수 있으며 발견되면 대개 수술로 제거된다.
클론 진화
동물의 집단이 진화를 겪듯이, 세포들의 통제되지 않은 집단 또한 "진화"를 겪을 수 있다.이 바람직하지 않은 과정은 체세포 진화라고 불리며,[66] 시간이 지남에 따라 암이 발병하고 더 악성으로 변하는 방법이다.
세포가 무질서하게 자라게 하는 세포대사의 변화는 세포사망으로 이어진다.하지만, 일단 암이 시작되면, 암세포는 자연 선택 과정을 거친다: 암세포의 생존이나 번식을 증진시키는 새로운 유전적 변화를 가진 소수의 세포들은 더 빨리 증식하고, 곧 덜 유리한 유전적 변화를 가진 세포들이 경쟁적으로 [67]성장하면서 종양을 지배하게 된다.이는 MRSA와 같은 병원성 생물종이 항생제에 내성을 갖게 되고 HIV가 약물에 내성을 갖게 되는 메커니즘과 식물성 질병과 곤충이 살충제에 내성을 갖게 되는 메커니즘과 같다.이러한 진화는 암-약물 내성 또는 방사선 치료에 대한 내성을 획득한 세포가 암 재발에 관여하는 이유를 설명한다.
암 세포의 생물학적 속성입니다.
2000년 하나한과 와인버그의 기사에서 악성 종양 세포의 생물학적 특성은 다음과 [68]같이 요약되었다.
- 성장 시그널의 자급률 획득은 억제된 성장으로 이어진다.
- 반성장 신호에 대한 민감성 상실은 또한 억제되지 않은 성장으로 이어진다.
- 아포토시스 능력 상실, 유전자 오류 및 외부 반성장 신호에도 불구하고 성장을 가능하게 한다.
- 노후화에 따른 용량 손실, 무한 리플리케이션 가능성(불멸성)으로 이어짐
- 지속적인 혈관신생을 획득하여 종양이 수동적 영양소 확산의 한계를 넘어 성장할 수 있도록 합니다.
- 침습성 암종의 결정적인 특성인 인접 조직을 침범하는 능력의 획득.
- 일부 악성 종양(암종 또는 기타)의 늦게 나타나는 특성인 먼 곳에서 종자 전이를 획득하는 능력.
이러한 여러 단계를 완료하는 것은 매우 드문 일이며, 드문 일입니다.
- 유전자 오류를 복구하는 능력의 손실은 돌연변이율(유전체 불안정성)을 증가시켜 다른 모든 변화를 가속화한다.
이러한 생물학적 변화는 암종에서 고전적이다; 다른 악성 종양들은 그것들을 모두 달성할 필요가 없을지도 모른다.예를 들어, 조직의 침입과 먼 곳으로의 이동은 백혈구의 정상적인 특성이기 때문에, 백혈병 발병에는 이러한 단계가 필요하지 않다.또한 다른 단계가 반드시 개별 돌연변이를 나타내는 것은 아닙니다.예를 들어, p53 단백질을 코드하는 단일 유전자의 불활성화는 게놈 불안정, 아포토시스 회피 및 혈관신생을 증가시킨다.게다가 모든 암세포가 분열하고 있는 것은 아니다.오히려, 암 줄기세포라고 불리는 종양의 세포 중 일부는 분화된 [69]세포를 생성하면서 스스로를 복제합니다.
세포 상호 작용에 결함이 암.
일반적으로 조직이 손상되거나 감염되면 손상된 세포는 주변 [70][71]세포에서 효소 활성의 특정 패턴과 사이토카인 유전자 발현을 자극하여 염증을 유발한다.분자의 이산 클러스터("사이토카인 클러스터")가 분비되며, 이는 매개자로 작용하여 생화학적 [72]변화의 후속 단계적 활동을 유도한다.각 사이토카인은 다양한 세포 타입의 특정 수용체에 결합하고, 각 세포 타입은 세포가 발현하는 수용체와 [73][74]세포 내부에 존재하는 신호 분자에 따라 세포 내 신호 전달 경로의 활성을 변화시킴으로써 차례로 반응한다.집합적으로, 이 재프로그래밍 과정은 세포 표현형의 단계적 변화를 유도하며, 이는 궁극적으로 조직 기능의 회복과 필수적인 구조적 [75][76]무결성을 되찾는 방향으로 이어질 것이다.이것에 의해, 손상 부위에 존재하는 [77]세포와 면역계와의 생산적인 통신에 의해서, 조직이 치유될 수 있다.치유에 있어 중요한 요소 중 하나는 사이토카인 유전자 발현을 조절하는 것인데, 이것은 상보적인 세포 그룹이 점차 조직 [78][79][80]생리학에 필수적인 변화를 일으키는 방식으로 염증 매개자에 반응할 수 있게 한다.암세포는 게놈에 영구적인 (유전적) 또는 가역적 (유전적) 변화를 가지고 있으며, 이는 부분적으로 주변 세포와 면역 [81][82]체계와의 통신을 방해한다.암세포는 조직의 무결성을 보호하는 방식으로 조직의 미세 환경과 소통하지 않는다; 대신에, 암세포의 움직임과 생존은 그들이 조직의 [83][84]기능을 손상시킬 수 있는 위치에서 가능해진다.암세포는 일반적으로 면역 체계로부터 조직을 보호하는 신호 경로를 "재연결"함으로써 생존한다.면역반응의 이러한 변화는 악성종양의 초기 단계에서도 뚜렷하게 나타난다.[85][86]
암에서 조직 기능이 재배선되는 한 가지 예는 전사 인자 NF-δB의 [87]활성이다.NF-δB는 세포 운명을 바꿀 수 있는 사이토카인, 접착인자 [88]및 다른 분자를 코드하는 염증과 재생 사이의 전환에 관여하는 수많은 유전자의 발현을 활성화한다.세포 표현형의 이러한 재프로그래밍은 일반적으로 완전히 기능하는 온전한 [89]조직의 개발을 가능하게 한다.NF-δB 활성은 여러 단백질에 의해 엄격하게 제어되며, 이는 주어진 세포와 시간에 [90]NF-δB에 의해 유도되는 유전자의 이산 클러스터만 집합적으로 보장한다.세포 간 신호 교환의 이 엄격한 조절은 조직을 과도한 염증으로부터 보호하고, 다른 종류의 세포들이 점차적으로 상호 보완적인 기능과 특정한 위치를 획득하도록 합니다.유전자 재프로그래밍과 세포 상호작용 사이의 상호 조절이 실패하면 암세포가 전이를 일으킬 수 있다.암세포는 사이토카인에 비정상적으로 반응하고 면역체계로부터 [87][91]보호할 수 있는 신호 캐스케이드를 활성화한다.
물고기에서
해산물([92][93]요오드 함량이 풍부한)과 민물고기(요오드 결핍증)에서 요오드의 역할은 완전히 이해되지 않았지만, 민물고기는 해산물보다 전염성, 특히 신형성 및 아테롬성 질환에 더 취약하다고 보고되었다.상어, 가오리와 같은 해양 엘라스모가지 어류는 민물고기보다 암의 영향을 훨씬 덜 받기 때문에 [94]발암을 더 잘 이해하기 위한 의학 연구를 촉진하고 있다.
메커니즘
세포들이 통제할 수 없이 분열하기 시작하기 위해서는, 세포 성장을 조절하는 유전자들은 조절이 [95]잘 되지 않아야 한다.원생유전자는 세포 성장과 유사분열을 촉진하는 유전자인데 반해 종양억제유전자는 세포 성장을 저해하거나 DNA 복구를 위해 세포 분열을 일시적으로 중단시킨다.일반적으로, 이러한 유전자에 [10]대한 일련의 돌연변이는 정상 세포가 암세포로 변하기 전에 필요합니다.이 개념은 때때로 "온코에볼루션"이라고 불립니다.이 유전자에 대한 돌연변이는 종양세포가 걷잡을 수 없이 분열하기 시작하는 신호를 제공한다.그러나 암을 특징짓는 통제되지 않은 세포분열은 또한 분열세포가 두 개의 딸세포를 만들기 위해 모든 세포성분을 복제해야 한다.원시온코겐과 종양억제유전자 [96]돌연변이에 의해 반드시 유발되지 않는 혐기성 당분해(워버그 효과)의 활성화는 분열세포의 세포성분을 복제하는데 필요한 대부분의 구성 요소를 제공하며, 따라서 [60]발암에도 필수적이다.
Oncogenes
종양유전자는 다양한 방법으로 세포 성장을 촉진한다.많은 세포들이 세포들 사이에 유사분열을 촉진하는 "화학적 전달자"인 호르몬을 생산할 수 있는데, 그 효과는 수용 조직이나 세포의 신호 전달에 달려 있다.즉, 수용세포상의 호르몬 수용체가 자극되면 세포표면에서 세포핵으로 신호가 전도되어 핵수준에서의 유전자 전사조절의 변화에 영향을 준다.일부 종양유전자는 신호 전달 시스템 자체 또는 세포와 조직 자체의 신호 수용체의 일부이며, 따라서 그러한 호르몬에 대한 민감도를 조절합니다.종양유전자는 종종 유사분열원을 생산하거나 단백질 합성의 DNA 전사에 관여하며, 이것은 생산물과 생화학 세포가 사용하고 상호작용하는 것을 책임지는 단백질과 효소를 생성한다.
종양유전자의 정상 정지 상태의 대응물인 프로토온코제네스의 돌연변이는 생성물 단백질의 양이나 활성을 증가시키면서 그들의 발현과 기능을 수정할 수 있다.이것이 일어날 때, 원생 원생 원생 원생 원생 원생 원생 원생 원생 원생 원생 원생 원생 원생 원생 원생 원생 원생 원생 원생 원생 원생 원생 원생 원생 원생 원생 원생 원생 원생 원생 원생 원생 원생 원생 원생 세포비록 이것이 가능하더라도, 원생 원생 원생 원생 원생 원생 원생 원생 원생 원생 원생 원생 원생 원생 원생 원생 원생 원생 원생 원생 원생 원생 원생 원생 원생 원생 원생 원생 원생 원생 원생 원생 원생 원생 원생 원생 원생그들이 돌연변이가 되었을 때만이 성장의 신호가 과도해지는 것이다.
암 연구에서 정의된 최초의 종양유전자 중 하나는 라스 종양유전자이다.원생 온코제네의 Ras과 돌연변이(H-Ras, N-Ras, K-Ras로 구성됨)는 인간 [97]종양의 20~30%에서 매우 흔하다.Ras는 원래 Harvey 육종 바이러스 게놈에서 확인되었고, 연구원들은 이 유전자가 인간 게놈에 존재할 뿐만 아니라 자극적인 제어 요소에 결합하면 세포주 [98]배양에 암을 유발할 수 있다는 것에 놀랐다.
Proto-oncogenes
원생 식물들은 다양한 방법으로 세포 성장을 촉진한다.많은 호르몬은 세포 간에 유사분열을 촉진하는 "화학적 전달자"를 생성할 수 있으며, 그 효과는 수용 조직이나 세포의 신호 전달에 따라 달라집니다.일부는 세포와 조직 자체의 신호 전달 시스템과 신호 수용체를 담당하며, 따라서 그러한 호르몬에 대한 민감도를 조절한다.그들은 종종 유사분열원을 생산하거나 단백질 합성의 DNA 전사에 관여하여 생화학 세포가 사용하고 상호작용하는 생산물과 생화학 제품을 생산하는 데 책임이 있는 단백질과 효소를 생성한다.
원생 원생 원생 원생 원생 원생 원생 원생 원생 원생 원생 원생 원생 원생 원생 원생 원생 원생 원생 원생 원생 원생 원생 원생 원생 원생 원생이것이 일어나면, 그것들은 종양유전자가 되고, 따라서 세포는 과도하게 통제 불능으로 분열할 가능성이 높아진다.유전자에서 원생 원생 원생 원생 원생 원생 원생 원생 원생 원생 원생 원생 원생 원생 원생 원생 원생 원생 원생 원생 원생 원생 원생 원생 원생 원생 원생 원생 원생 원생 원생 원생 원생그들이 돌연변이가 되었을 때만이 성장의 신호가 과도해지는 것이다.성장을 촉진하는 역할을 가진 유전자는 성장을 허용하는 모든 필요한 세포 메커니즘이 [99]활성화된다는 조건 하에서 세포의 발암 가능성을 증가시킬 수 있다는 점에 주목하는 것이 중요하다.이 조건에는 특정 종양 억제 유전자의 불활성화도 포함됩니다(아래 참조).조건이 충족되지 않으면 세포가 성장을 멈추고 죽을 수 있습니다.이것은 주어진 종양 유전자의 통제 하에 성장하는 암세포의 단계와 유형을 식별하는 것을 치료 전략 개발에 중요하게 만든다.
종양 억제 유전자

종양 억제 유전자는 증식 방지 신호와 유사분열과 세포 성장을 억제하는 단백질을 암호화한다.일반적으로 종양 억제제는 세포 스트레스나 DNA 손상에 의해 활성화되는 전사 인자입니다.종종 DNA 손상은 자유유동 유전물질의 존재와 다른 징후를 야기하고, 종양억제 유전자의 활성화를 이끄는 효소와 경로를 촉발할 것이다.이러한 유전자의 기능은 DNA 복구를 위해 세포주기의 진행을 억제하여 돌연변이가 딸세포로 전달되는 것을 막는 것이다.연구된 가장 중요한 종양 억제 유전자 중 하나인 p53 단백질은 저산소증과 자외선 방사선 손상을 포함한 많은 세포 스트레스 인자에 의해 활성화된 전사 인자입니다.
p53의 변화를 수반하는 모든 암의 거의 절반에도 불구하고, 그것의 종양 억제제 기능은 잘 알려져 있지 않다. p53은 분명히 두 가지 기능을 가지고 있다: 하나는 전사인자로서의 핵 역할이고 다른 하나는 세포 주기, 세포 분열 및 세포자멸을 조절하는 세포질 역할이다.
워버그 가설은 암 성장을 지속하기 위한 에너지로 해당과정을 우선적으로 사용하는 것이다.p53은 호흡에서 [100]해당과정 경로로의 이동을 조절하는 것으로 나타났다.
그러나 돌연변이는 종양 억제 유전자 자체 또는 이를 활성화하는 신호 경로를 손상시켜 "끄기" 할 수 있습니다.이것의 불변의 결과는 DNA 복구가 방해되거나 억제된다는 것이다: DNA 손상은 복구 없이 축적되어 불가피하게 암을 초래한다.
생식세포에서 발생하는 종양억제 유전자의 돌연변이는 자손에게 전해져 다음 세대에 암 진단 가능성을 높인다.이 가족들의 구성원들은 다발성 종양의 발생률을 증가시키고 지연 시간을 줄였다.종양 유형은 종양 억제 유전자 돌연변이의 각 유형에 대해 전형적이며, 일부 돌연변이는 특정 암을 유발하고 다른 돌연변이는 다른 돌연변이를 일으킨다.돌연변이 종양 억제제의 유전 양태는, 영향을 받은 멤버가 한쪽 부모로부터 결함 카피를, 다른 한쪽 부모로부터 정상적인 카피를 상속하는 것이다.예를 들어, 하나의 돌연변이 p53 대립 유전자를 물려받은 개인은 Li-Fraumeni 증후군으로 알려진 흑색종과 췌장암에 걸릴 수 있습니다.다른 유전적인 종양 억제 유전자 증후군은 망막아종에 연결된 Rb 돌연변이와 대장암에 연결된 APC 유전자 돌연변이를 포함한다.선용종성 대장암은 어릴 때 대장 내 수천 개의 용종과 관련이 있어 비교적 이른 나이에 대장암으로 이어진다.마지막으로, BRCA1과 BRCA2의 유전 돌연변이는 유방암의 조기 발병으로 이어진다.
암의 개발은 1971년에 적어도 두 가지 돌연변이 사건에 의존하기 위해 제안되었다.크누드슨 투히트 가설로 알려지게 된 이 가설에서는 종양 억제 유전자에서 유전되는 생식계 돌연변이는 종양 억제 [101]유전자의 다른 대립 유전자가 활성화되지 않고 나중에 또 다른 돌연변이 사건이 발생할 경우에만 암을 일으킬 것이다.
일반적으로 종양유전자는 기능성 변이를 포함하고 있기 때문에 우세한 반면, 돌연변이 종양억제제는 기능성 변이를 포함하고 있기 때문에 열성적이다.각 세포는 부모로부터 각각 하나씩, 같은 유전자의 두 개의 복사본을 가지고 있으며, 대부분의 경우 특정 원생 유전자의 한 복사본에서 기능 돌연변이를 얻는 것만으로도 그 유전자를 진정한 종양 유전자로 만들기에 충분하다.반면에, 기능 돌연변이의 상실은 그 유전자를 완전히 기능하지 않게 만들기 위해 종양 억제 유전자의 두 복사본 모두에서 발생할 필요가 있다.단, 종양억제유전자의 돌연변이복사 중 하나가 다른 야생형 복사를 기능하지 않게 할 수 있는 경우가 존재한다.이 현상은 지배적인 부정적 효과라고 불리며 많은 p53 돌연변이에 관찰된다.
Knudson의 두 히트 모델은 최근 여러 조사관들에 의해 도전을 받아왔다.일부 종양억제유전자 중 하나의 대립 유전자의 불활성화는 종양을 일으키기에 충분하다.이러한 현상을 하플로우 기능이라고 하며 여러 가지 실험적인 접근법에 의해 입증되었습니다.부실로 인한 종양은 보통 두 번의 적중 [102]과정에 의한 종양에 비해 발병 연령이 늦다.
여러 변이

일반적으로 암이 발생하기 위해서는 두 유전자 모두 돌연변이가 필요하다.예를 들어, 하나의 종양 유전자로 제한된 돌연변이는 Knudson [8]가설에 의해 최초로 가정된 정상적인 유사분열 제어와 종양 억제 유전자에 의해 억제될 것이다.하나의 종양 억제 유전자에 대한 돌연변이 또한 암을 유발하지 않을 것이다. 왜냐하면 그 기능을 복제하는 많은 "백업" 유전자들이 존재하기 때문이다.충분한 수의 원생 유전자가 종양 유전자로 변이되고 충분한 종양 억제 유전자가 비활성화되거나 손상되었을 때에만 세포 성장에 대한 신호가 그것을 조절하는 신호를 압도하고, 세포 성장은 빠르게 [10]통제 불능으로 치솟는다.종종, 이 유전자들이 유전자 자체의 손상을 막는 과정을 조절하기 때문에, DNA 손상은 피드백 루프를 형성하기 때문에, 나이가 들면서 돌연변이의 비율이 증가한다.
종양억제 유전자의 돌연변이가 다음 세대의 세포뿐만 아니라 그 자손에게 전해지는 것은 암이 유전될 가능성을 높일 수 있다.이러한 가족 내 구성원들은 다발성 종양의 발생률을 증가시키고 지연 시간을 줄였다.돌연변이 종양 억제제의 유전 양태는 영향을 받은 구성원이 한쪽 부모로부터 결함 있는 복사본을 상속하고 다른 한쪽 부모로부터 정상적인 복사본을 상속하는 것입니다.종양 억제제의 돌연변이는 열성적인 방식으로 작용하기 때문에(단, 예외가 있습니다), 정상 복사본의 손실은 암 표현형을 생성합니다.예를 들어 p53 돌연변이에 대해 헤테로 접합된 개인은 종종 Li-Fraumeni 증후군의 피해자이며, Rb 돌연변이에 대해 헤테로 접합된 망막아세포종이 발생한다.비슷한 방식으로, 선종성 대장균 유전자의 돌연변이는 어릴 때 대장에 수천 개의 용종이 있는 선종성 대장암과 관련이 있는 반면, BRCA1과 BRCA2의 돌연변이는 유방암의 조기 발병을 이끈다.
2011년에 발표된 새로운 아이디어는 다중 돌연변이의 극단적인 버전이며, 그 지지자들에 의해 색소 침착증이라고 불린다.이 아이디어는 골격암의 25%에 달하지만 암의 2~3%에만 영향을 미치며, 염색체를 수십 또는 수백 조각으로 분할한 후 다시 잘못 패치해야 한다.이 분쇄는 정상적인 세포 분열 중에 염색체가 압축되었을 때 일어날 수 있지만, 분쇄의 계기는 알려지지 않았습니다.이 모델에서 암은 다중 돌연변이의 느린 축적이 아닌 단일 격리된 사건의 결과로 발생한다.[103]
Non-mutagenic 발암 물질
많은 돌연변이 물질도 발암 물질이지만, 일부 발암 물질은 돌연변이 물질이 아닙니다.돌연변이가 아닌 발암물질의 예로는 알코올과 에스트로겐이 있다.이것들은 세포 유사분열 속도에 대한 자극적인 효과를 통해 암을 촉진하는 것으로 생각된다.유사분열 속도가 빨라짐에 따라 DNA 복제 중에 손상된 DNA를 복구할 수 있는 복구 효소의 기회가 점점 줄어들어 유전자 오류의 가능성이 높아집니다.유사분열 과정에서 실수를 하면 딸세포가 잘못된 수의 염색체를 받게 되고, 이로 인해 배수가 잘 안 되고, 암으로 이어질 수 있다.
역할 감염의
박테리아
헬리코박터균은 위암을 일으킬 수 있다.데이터는 나라마다 다르지만, 헬리코박터균에 감염된 사람의 약 1~3%가 평생 위암에 걸리는 반면, H.필로리균에 [104][105]감염된 사람은 0.13%였다.H. pylori 감염은 매우 흔하다.2002년에 평가한 바와 같이, 개발도상국에서는 중년 성인의 74%, 선진국에서는 [106]58%의 위 조직에 존재한다.감염자의 1%~3%가 위암에 [107]걸릴 가능성이 높기 때문에 2018년 [108]기준으로 전 세계 암 사망률 3위다.
H. pylori에 의한 감염은 [109]감염자의 약 80%에서 증상을 일으키지 않는다.H. pylori에 감염된 사람의 약 75%가 위염에 [110]걸린다.따라서 H. pylori 감염의 일반적인 결과는 만성 무증상 [111]위염이다.평소 증상이 없기 때문에 최종적으로 위암이 진단되면 상당히 진행된 경우가 많습니다.위암 환자의 절반 이상이 최초 진단 [112]시 림프절 전이가 나타난다.
H. pylori에 의한 위염은 위상피로의 호중구 및 대식세포의 침윤을 특징으로 하는 염증을 동반하며, 이는 소염성 사이토카인과 활성산소종/반응성 질소종(ROS/RNS)[113]의 축적을 선호한다.ROS/RNS의 상당한 존재는 8-oxo-2'-디옥시구아노신(8-OHDG)[113]을 포함한 DNA 손상을 일으킨다.감염성 H. pylori가 세포독성 cagA 유전자(서양 분리주 약 60%, 아시아 분리주 더 높은 비율)를 가지고 있으면 위세포의 8-OHDG 수치를 8배 높일 수 있고, H. pylori가 cagA 유전자를 가지고 있지 않으면 8-OHDG의 [114]증가량은 약 4배이다.산화성 DNA 손상 8-OHDG 외에 H. pylori 감염은 DNA 이중사슬 [115]파손을 포함한 다른 특징적인 DNA 손상을 일으킨다.
H. pylori는 또한 암 [116][117]발병과 관련된 많은 후생유전학적 변화를 일으킨다.이러한 후생유전학적 변화는 유전자의[116] 촉진제에서 CpG 부위의 H. pylori 유도 메틸화 및 여러 마이크로RNA의 H.[117] pylori 유도 변화 발현에 기인한다.
Santos와 Ribeiro[118] H. pylori 감염은 후생적으로 DNA 복구 기계의 효율 저하와 관련이 있으며, 이는 위암뿐만 아니라 돌연변이의 축적과 게놈 불안정성을 선호한다.특히, Raza [119]등은 H. pylori 감염이 진행되어 소화불량을 유발한 후 ERCC1과 PMS2라는 두 개의 DNA 복구 단백질의 발현이 심각하게 감소했음을 보여주었다.소화불량증은 감염된 사람의 [120]약 20%에서 발생한다.또한 Raza [119]등에 의해 검토된 바와 같이 H. pylori에 의한 인간 위 감염은 DNA 복구 단백질 MLH1, MGMT 및 MRE11의 후생적으로 단백질 발현을 감소시킨다.DNA 손상이 증가했을 때 감소된 DNA 복구는 발암성 돌연변이를 증가시키고 H. pylori 발암의 중요한 원인일 수 있습니다.
바이러스
게다가, 많은 암들은 바이러스 감염에서 비롯된다; 이것은 특히 조류와 같은 동물들에게서 사실이지만, 인간에게는 그렇지 않다.인간 암의 12%는 바이러스 [121]감염에 기인할 수 있다.바이러스성 종양의 양태는 두 가지로 나눌 수 있는데, 급변환 또는 천천히 변형이 된다.급성변형 바이러스에서 바이러스 입자는 viral-oncogen(v-onc)이라 불리는 과활성 종양유전자를 코드하는 유전자를 가지고 있으며 v-onc가 발현되는 즉시 감염된 세포가 변형된다.이와는 대조적으로 천천히 변형을 하는 바이러스에서는 바이러스 게놈이 삽입되는데, 특히 바이러스 게놈 삽입이 레트로바이러스의 필수 부분이며, 숙주 게놈의 원생 유전자 근처에 있다.바이러스 프로모터 또는 다른 전사 조절 요소들은 차례로 그 원형의 과잉 발현을 유발하고, 이는 다시 통제되지 않은 세포 증식을 유발한다.바이러스 게놈 삽입은 원생종에게 특정되지 않고 원생종 근처에 삽입될 가능성이 낮기 때문에, 천천히 변형되는 바이러스는 이미 바이러스-온유전자를 가지고 있는 급성 변형 바이러스에 비해 매우 긴 종양 대기 시간을 가진다.
HPV(경막암), B형 간염(간암), EBV(림프종의 일종) 등 암을 유발하는 것으로 알려진 바이러스는 모두 DNA 바이러스다.바이러스가 세포를 감염시키면 세포 성장 유전자 근처에 자신의 DNA 일부를 삽입해 세포 분열을 일으키는 것으로 알려져 있다.첫 번째 세포가 분열하면서 형성된 변화된 세포 그룹은 세포 성장 유전자 근처에 동일한 바이러스 DNA를 가지고 있다.변화된 세포들의 그룹은 이제 성장에 대한 정상적인 통제 중 하나가 상실되었기 때문에 특별하다.
세포는 위치에 따라 방사선, 담배 연기로 인한 화학물질, 세균 감염이나 다른 바이러스로 인한 염증을 통해 손상될 수 있다.각 세포는 손상 가능성이 있습니다.세포는 종종 손상되면, 중요한 과정이나 면역체계의 기능 상실로 죽지만, 때때로 손상으로 인해 하나의 암 유전자가 녹아웃된다.노인은 수천, 수만 또는 수십만 개의 노크아웃된 세포가 있습니다.누구나 암에 걸릴 확률은 매우 [citation needed]낮다.
바뀐 세포 부위에 손상이 발생하면 뭔가 다른 현상이 발생합니다.각각의 세포는 성장 가능성이 있다.물리적, 화학적 또는 바이러스성 물질에 의해 영역이 손상되면 변경된 세포는 더 빨리 분열됩니다.영역을 손상시키면 변경된 세포가 분열되어 녹아웃될 가능성이 높아진다는 악순환이 형성되었습니다.
이 발암 모델은 암이 왜 자라는지 설명해주기 때문에 인기가 있다.방사선에 의해 손상된 세포들은 죽거나 최소한 더 나쁜 상태에 있을 것으로 예상될 것이다. 왜냐하면 그것들은 작동하는 유전자의 수를 증가시키기 때문이다.
한 가지 생각은 우리의 세포를 바꿀 수 있는 모든 바이러스를 예방하기 위해 수천 개의 백신을 갖게 될 수도 있다는 것이다.바이러스는 신체의 다른 부위에 다른 영향을 미칠 수 있다.하나의 바이러스 약물에 대한 면역 접종을 함으로써 많은 다른 암을 예방할 수 있을 것이다.예를 들어 HPV는 구강 점막암에 관여할 가능성이 높다.
Helminthiasis
어떤 기생충들은 [122]발암성이 있는 것으로 알려져 있다.여기에는 다음이 포함됩니다.
- 클로노르키스 시넨시스(Clonorchis sinensis)와 오피소르키스 비베리니(Opisthorchis viverrini)[123]는 담관암과 관련이 있다.
- 주혈흡충증은 방광암과 관련이 있다.
후성 유전학
후생유전학은 DNA 구조의 화학적, 변이 없는 변화를 통해 유전자 발현을 조절하는 학문이다.암 발병에서의 후생유전학의 이론은 DNA에 대한 변이가 없는 변화가 유전자 발현에 변화를 가져올 수 있다는 것이다.일반적으로 종양유전자는 예를 들어 DNA 메틸화 때문에 침묵한다.메틸화의 상실은 암 발생의 원인이 되는 암 발생의 비정상적인 발현을 유도할 수 있다.후생유전적 변화의 알려진 메커니즘은 특정 위치에서 염색체 DNA에 결합된 히스톤 단백질의 메틸화 또는 아세틸화를 포함한다.HDAC 억제제 및 DNA 메틸전달효소 억제제로 알려진 약물들은 암세포에서 후생유전 신호를 다시 조절할 수 있다.
에피메이션은 유전자의 프로모터 영역의 CpG 섬의 메틸화 또는 탈메틸화를 포함하며, 이는 각각 유전자 [124][125][126]발현을 억제 또는 억제하는 결과를 초래한다.에피메이션은 또한 아세틸화, 메틸화, 인산화 또는 히스톤에 대한 다른 변화에 의해 발생할 수 있으며, 유전자 발현을 억제하거나 활성화하는 히스톤 코드를 생성하며,[127][128] 그러한 히스톤 에피메이션은 암에서 중요한 후생유전 인자가 될 수 있다.또한 발암성 발열은 HMGA2와 [129]같은 단백질에 의해 야기되는 염색체 구조의 변화를 통해 발생할 수 있다. 발열원의 또 다른 원천은 마이크로RNA(miRNA)의 발현 증가 또는 감소에 기인한다.예를 들어 miR-137의 추가 발현으로 491개 유전자의 발현 조절이 저하될 수 있으며 miR-137은 대장암의 [15]32%에서 후생적으로 무음화된다.
암 줄기 세포
발암을 바라보는 새로운 시각은 발달생물학의 개념을 종양학으로 통합하는 것에서 비롯된다.암 줄기세포 가설은 이종 종양의 다른 종류의 세포들이 암 줄기세포라고 불리는 단일 세포에서 발생한다는 것을 제안한다.암 줄기세포는 성체줄기세포의 변형이나 체내 분화세포에서 발생할 수 있다.이 세포들은 종양의 하위 구성요소로 지속되며 주요 줄기세포 특성을 유지합니다.그들은 다양한 세포를 생성하고, 자기 재생과 항상성 조절을 [130]할 수 있다.또한 암의 재발과 전이 발생도 이들 세포에 기인한다.암 줄기세포 가설은 발암의 초기 개념과 모순되지 않는다.암 줄기세포 가설은 종양 이질성에 기여하는 제안된 메커니즘이었다.
클론 진화
종양억제유전자 및 종양유전자의 유전적, 후생적 변화가 세포의 행동을 변화시키는 반면, 결국 이러한 변화는 종양세포의 개체수와 미세환경에 [66]미치는 영향을 통해 암을 유발한다.종양의 돌연변이 세포들은 공간과 자원을 놓고 경쟁한다.따라서, 종양 억제 유전자나 종양 유전자에 돌연변이가 있는 복제는 그 돌연변이가 미세 [131]환경의 다른 복제 세포와 정상 세포에 비해 경쟁 우위를 제공한다면 종양 종양에서만 확장될 것이다.따라서, 발암의 과정은 공식적으로 체세포 또는 복제 [67]진화로 알려진 다윈의 진화 과정이다.게다가, 발암의 다윈론적 메커니즘에 비추어 볼 때, 다양한 형태의 암은 사춘기와 노인기로 분류될 수 있다는 이론이 제기되어 왔다.자연선택이 환경적으로 열등한 표현형을 파괴하는 자연진화 과정으로 현재 암에 대한 인류학 연구가 진행되고 있다.이 이론에 따르면 암은 출생에서 사춘기 말기(약 20세)까지 두 가지 유형으로 구분된다. 즉, 정신적으로 지지적인 그룹 역학으로 기울어지는 것과, 정신적으로 과잉 인구 집단 [citation needed]역학에서 벗어나 중년에서 사망으로 기울어지는 것(약 40세 이상)이다.
「 」를 참조해 주세요.
레퍼런스
- ^ Majérus, Marie-Ange (1 July 2022). "The cause of cancer: The unifying theory". Advances in Cancer Biology - Metastasis. 4: 100034. doi:10.1016/j.adcanc.2022.100034. ISSN 2667-3940.
- ^ Nowell, Peter C. (1 October 1976). "The Clonal Evolution of Tumor Cell Populations: Acquired genetic lability permits stepwise selection of variant sublines and underlies tumor progression". Science. 194 (4260): 23–28. doi:10.1126/science.959840. ISSN 0036-8075.
- ^ Hanahan, Douglas; Weinberg, Robert A (7 January 2000). "The Hallmarks of Cancer". Cell. 100 (1): 57–70. doi:10.1016/s0092-8674(00)81683-9. ISSN 0092-8674.
- ^ Hahn, William C.; Weinberg, Robert A. (14 November 2002). "Rules for Making Human Tumor Cells". New England Journal of Medicine. 347 (20): 1593–1603. doi:10.1056/NEJMra021902. ISSN 0028-4793.
- ^ Calkins, Gary N. (11 December 1914). "Zur Frage der Entstehung maligner Tumoren . By Th. Boveri. Jena, Gustav Fischer. 1914. 64 pages". Science. 40 (1041): 857–859. doi:10.1126/science.40.1041.857. ISSN 0036-8075.
- ^ Tomasetti C, Li L, Vogelstein B (23 March 2017). "Stem cell divisions, somatic mutations, cancer etiology, and cancer prevention". Science. 355 (6331): 1330–1334. Bibcode:2017Sci...355.1330T. doi:10.1126/science.aaf9011. PMC 5852673. PMID 28336671.
- ^ Wood LD, Parsons DW, Jones S, Lin J, Sjöblom T, Leary RJ, et al. (November 2007). "The genomic landscapes of human breast and colorectal cancers". Science. 318 (5853): 1108–13. Bibcode:2007Sci...318.1108W. CiteSeerX 10.1.1.218.5477. doi:10.1126/science.1145720. PMID 17932254. S2CID 7586573.
- ^ a b Knudson AG (November 2001). "Two genetic hits (more or less) to cancer". Nature Reviews. Cancer. 1 (2): 157–62. doi:10.1038/35101031. PMID 11905807. S2CID 20201610.
- ^ Fearon ER, Vogelstein B (June 1990). "A genetic model for colorectal tumorigenesis". Cell. 61 (5): 759–67. doi:10.1016/0092-8674(90)90186-I. PMID 2188735. S2CID 22975880.
- ^ a b c Belikov AV (September 2017). "The number of key carcinogenic events can be predicted from cancer incidence". Scientific Reports. 7 (1): 12170. Bibcode:2017NatSR...712170B. doi:10.1038/s41598-017-12448-7. PMC 5610194. PMID 28939880.
- ^ Belikov AV, Vyatkin A, Leonov SV (6 August 2021). "The Erlang distribution approximates the age distribution of incidence of childhood and young adulthood cancers". PeerJ. 9: e11976. doi:10.7717/peerj.11976. PMC 8351573. PMID 34434669.
- ^ Vyatkin, Alexey D.; Otnyukov, Danila V.; Leonov, Sergey V.; Belikov, Aleksey V. (14 January 2022). "Comprehensive patient-level classification and quantification of driver events in TCGA PanCanAtlas cohorts". PLOS Genetics. 18 (1): e1009996. doi:10.1371/journal.pgen.1009996.
- ^ Croce CM (January 2008). "Oncogenes and cancer". The New England Journal of Medicine. 358 (5): 502–11. doi:10.1056/NEJMra072367. PMID 18234754.
- ^ Lim LP, Lau NC, Garrett-Engele P, Grimson A, Schelter JM, Castle J, Bartel DP, Linsley PS, Johnson JM (February 2005). "Microarray analysis shows that some microRNAs downregulate large numbers of target mRNAs". Nature. 433 (7027): 769–73. Bibcode:2005Natur.433..769L. doi:10.1038/nature03315. PMID 15685193. S2CID 4430576.
- ^ a b Balaguer F, Link A, Lozano JJ, Cuatrecasas M, Nagasaka T, Boland CR, Goel A (August 2010). "Epigenetic silencing of miR-137 is an early event in colorectal carcinogenesis". Cancer Research. 70 (16): 6609–18. doi:10.1158/0008-5472.CAN-10-0622. PMC 2922409. PMID 20682795.
- ^ Kastan MB (April 2008). "DNA damage responses: mechanisms and roles in human disease: 2007 G.H.A. Clowes Memorial Award Lecture". Molecular Cancer Research. 6 (4): 517–24. doi:10.1158/1541-7786.MCR-08-0020. PMID 18403632.
- ^ a b Cunningham FH, Fiebelkorn S, Johnson M, Meredith C (November 2011). "A novel application of the Margin of Exposure approach: segregation of tobacco smoke toxicants". Food and Chemical Toxicology. 49 (11): 2921–33. doi:10.1016/j.fct.2011.07.019. PMID 21802474.
- ^ Kanavy HE, Gerstenblith MR (December 2011). "Ultraviolet radiation and melanoma". Seminars in Cutaneous Medicine and Surgery. 30 (4): 222–8. doi:10.1016/j.sder.2011.08.003. PMID 22123420.
- ^ Handa O, Naito Y, Yoshikawa T (2011). "Redox biology and gastric carcinogenesis: the role of Helicobacter pylori". Redox Report. 16 (1): 1–7. doi:10.1179/174329211X12968219310756. PMC 6837368. PMID 21605492.
- ^ Smela ME, Hamm ML, Henderson PT, Harris CM, Harris TM, Essigmann JM (May 2002). "The aflatoxin B(1) formamidopyrimidine adduct plays a major role in causing the types of mutations observed in human hepatocellular carcinoma". Proceedings of the National Academy of Sciences of the United States of America. 99 (10): 6655–60. Bibcode:2002PNAS...99.6655S. doi:10.1073/pnas.102167699. PMC 124458. PMID 12011430.
- ^ Katsurano M, Niwa T, Yasui Y, Shigematsu Y, Yamashita S, Takeshima H, Lee MS, Kim YJ, Tanaka T, Ushijima T (January 2012). "Early-stage formation of an epigenetic field defect in a mouse colitis model, and non-essential roles of T- and B-cells in DNA methylation induction". Oncogene. 31 (3): 342–51. doi:10.1038/onc.2011.241. PMID 21685942.
- ^ Bernstein C, Holubec H, Bhattacharyya AK, Nguyen H, Payne CM, Zaitlin B, Bernstein H (August 2011). "Carcinogenicity of deoxycholate, a secondary bile acid". Archives of Toxicology. 85 (8): 863–71. doi:10.1007/s00204-011-0648-7. PMC 3149672. PMID 21267546.
- ^ Malkin D (April 2011). "Li-fraumeni syndrome". Genes & Cancer. 2 (4): 475–84. doi:10.1177/1947601911413466. PMC 3135649. PMID 21779515.
- ^ Fearon ER (November 1997). "Human cancer syndromes: clues to the origin and nature of cancer". Science. 278 (5340): 1043–50. Bibcode:1997Sci...278.1043F. doi:10.1126/science.278.5340.1043. PMID 9353177.
- ^ Lichtenstein P, Holm NV, Verkasalo PK, Iliadou A, Kaprio J, Koskenvuo M, Pukkala E, Skytthe A, Hemminki K (July 2000). "Environmental and heritable factors in the causation of cancer--analyses of cohorts of twins from Sweden, Denmark, and Finland". The New England Journal of Medicine. 343 (2): 78–85. doi:10.1056/NEJM200007133430201. PMID 10891514.
- ^ Halford S, Rowan A, Sawyer E, Talbot I, Tomlinson I (June 2005). "O(6)-methylguanine methyltransferase in colorectal cancers: detection of mutations, loss of expression, and weak association with G:C>A:T transitions". Gut. 54 (6): 797–802. doi:10.1136/gut.2004.059535. PMC 1774551. PMID 15888787.
- ^ a b Narayanan L, Fritzell JA, Baker SM, Liskay RM, Glazer PM (April 1997). "Elevated levels of mutation in multiple tissues of mice deficient in the DNA mismatch repair gene Pms2". Proceedings of the National Academy of Sciences of the United States of America. 94 (7): 3122–7. Bibcode:1997PNAS...94.3122N. doi:10.1073/pnas.94.7.3122. PMC 20332. PMID 9096356.
- ^ a b Hegan DC, Narayanan L, Jirik FR, Edelmann W, Liskay RM, Glazer PM (December 2006). "Differing patterns of genetic instability in mice deficient in the mismatch repair genes Pms2, Mlh1, Msh2, Msh3 and Msh6". Carcinogenesis. 27 (12): 2402–8. doi:10.1093/carcin/bgl079. PMC 2612936. PMID 16728433.
- ^ a b Tutt AN, van Oostrom CT, Ross GM, van Steeg H, Ashworth A (March 2002). "Disruption of Brca2 increases the spontaneous mutation rate in vivo: synergism with ionizing radiation". EMBO Reports. 3 (3): 255–60. doi:10.1093/embo-reports/kvf037. PMC 1084010. PMID 11850397.
- ^ German J (March 1969). "Bloom's syndrome. I. Genetical and clinical observations in the first twenty-seven patients". American Journal of Human Genetics. 21 (2): 196–227. PMC 1706430. PMID 5770175.
- ^ O'Hagan HM, Mohammad HP, Baylin SB (August 2008). Lee JT (ed.). "Double strand breaks can initiate gene silencing and SIRT1-dependent onset of DNA methylation in an exogenous promoter CpG island". PLOS Genetics. 4 (8): e1000155. doi:10.1371/journal.pgen.1000155. PMC 2491723. PMID 18704159.
- ^ Cuozzo C, Porcellini A, Angrisano T, Morano A, Lee B, Di Pardo A, Messina S, Iuliano R, Fusco A, Santillo MR, Muller MT, Chiariotti L, Gottesman ME, Avvedimento EV (July 2007). "DNA damage, homology-directed repair, and DNA methylation". PLOS Genetics. 3 (7): e110. doi:10.1371/journal.pgen.0030110. PMC 1913100. PMID 17616978.
- ^ Villeneuve PJ, Mao Y (November 1994). "Lifetime probability of developing lung cancer, by smoking status, Canada". Canadian Journal of Public Health. 85 (6): 385–8. PMID 7895211.
- ^ Gerlinger M, Rowan AJ, Horswell S, Larkin J, Endesfelder D, Gronroos E, et al. (March 2012). "Intratumor heterogeneity and branched evolution revealed by multiregion sequencing". The New England Journal of Medicine. 366 (10): 883–92. doi:10.1056/NEJMoa1113205. PMC 4878653. PMID 22397650.
- ^ a b López-Lázaro M (August 2015). "Stem cell division theory of cancer". Cell Cycle. 14 (16): 2547–8. doi:10.1080/15384101.2015.1062330. PMC 5242319. PMID 26090957.
- ^ a b c López-Lázaro M (May 2015). "The migration ability of stem cells can explain the existence of cancer of unknown primary site. Rethinking metastasis". Oncoscience. 2 (5): 467–75. doi:10.18632/oncoscience.159. PMC 4468332. PMID 26097879.
- ^ Tomasetti C, Vogelstein B (January 2015). "Cancer etiology. Variation in cancer risk among tissues can be explained by the number of stem cell divisions". Science. 347 (6217): 78–81. doi:10.1126/science.1260825. PMC 4446723. PMID 25554788.
- ^ Slaughter DP, Southwick HW, Smejkal W (September 1953). "Field cancerization in oral stratified squamous epithelium; clinical implications of multicentric origin". Cancer. 6 (5): 963–8. doi:10.1002/1097-0142(195309)6:5<963::AID-CNCR2820060515>3.0.CO;2-Q. PMID 13094644.
- ^ Bernstein C, Bernstein H, Payne CM, Dvorak K, Garewal H (February 2008). "Field defects in progression to gastrointestinal tract cancers". review. Cancer Letters. 260 (1–2): 1–10. doi:10.1016/j.canlet.2007.11.027. PMC 2744582. PMID 18164807.
- ^ Nguyen H, Loustaunau C, Facista A, Ramsey L, Hassounah N, Taylor H, Krouse R, Payne CM, Tsikitis VL, Goldschmid S, Banerjee B, Perini RF, Bernstein C (2010). "Deficient Pms2, ERCC1, Ku86, CcOI in field defects during progression to colon cancer". Journal of Visualized Experiments (41): 1931. doi:10.3791/1931. PMC 3149991. PMID 20689513.
- ^ Rubin H (March 2011). "Fields and field cancerization: the preneoplastic origins of cancer: asymptomatic hyperplastic fields are precursors of neoplasia, and their progression to tumors can be tracked by saturation density in culture". BioEssays. 33 (3): 224–31. doi:10.1002/bies.201000067. PMID 21254148. S2CID 44981539.
- ^ Tsao JL, Yatabe Y, Salovaara R, Järvinen HJ, Mecklin JP, Aaltonen LA, Tavaré S, Shibata D (February 2000). "Genetic reconstruction of individual colorectal tumor histories". Proceedings of the National Academy of Sciences of the United States of America. 97 (3): 1236–41. Bibcode:2000PNAS...97.1236T. doi:10.1073/pnas.97.3.1236. PMC 15581. PMID 10655514.
- ^ a b c Vogelstein B, Papadopoulos N, Velculescu VE, Zhou S, Diaz LA, Kinzler KW (March 2013). "Cancer genome landscapes". review. Science. 339 (6127): 1546–58. Bibcode:2013Sci...339.1546V. doi:10.1126/science.1235122. PMC 3749880. PMID 23539594.
- ^ Shen L, Kondo Y, Rosner GL, Xiao L, Hernandez NS, Vilaythong J, Houlihan PS, Krouse RS, Prasad AR, Einspahr JG, Buckmeier J, Alberts DS, Hamilton SR, Issa JP (September 2005). "MGMT promoter methylation and field defect in sporadic colorectal cancer". Journal of the National Cancer Institute. 97 (18): 1330–8. doi:10.1093/jnci/dji275. PMID 16174854.
- ^ a b Lee KH, Lee JS, Nam JH, Choi C, Lee MC, Park CS, Juhng SW, Lee JH (October 2011). "Promoter methylation status of hMLH1, hMSH2, and MGMT genes in colorectal cancer associated with adenoma-carcinoma sequence". Langenbeck's Archives of Surgery. 396 (7): 1017–26. doi:10.1007/s00423-011-0812-9. PMID 21706233. S2CID 8069716.
- ^ Svrcek M, Buhard O, Colas C, Coulet F, Dumont S, Massaoudi I, et al. (November 2010). "Methylation tolerance due to an O6-methylguanine DNA methyltransferase (MGMT) field defect in the colonic mucosa: an initiating step in the development of mismatch repair-deficient colorectal cancers". Gut. 59 (11): 1516–26. doi:10.1136/gut.2009.194787. PMID 20947886. S2CID 206950452.
- ^ a b c d Facista A, Nguyen H, Lewis C, Prasad AR, Ramsey L, Zaitlin B, Nfonsam V, Krouse RS, Bernstein H, Payne CM, Stern S, Oatman N, Banerjee B, Bernstein C (April 2012). "Deficient expression of DNA repair enzymes in early progression to sporadic colon cancer". Genome Integrity. 3 (1): 3. doi:10.1186/2041-9414-3-3. PMC 3351028. PMID 22494821.
- ^ Paluszczak J, Misiak P, Wierzbicka M, Woźniak A, Baer-Dubowska W (February 2011). "Frequent hypermethylation of DAPK, RARbeta, MGMT, RASSF1A and FHIT in laryngeal squamous cell carcinomas and adjacent normal mucosa". Oral Oncology. 47 (2): 104–7. doi:10.1016/j.oraloncology.2010.11.006. PMID 21147548.
- ^ Zuo C, Zhang H, Spencer HJ, Vural E, Suen JY, Schichman SA, Smoller BR, Kokoska MS, Fan CY (October 2009). "Increased microsatellite instability and epigenetic inactivation of the hMLH1 gene in head and neck squamous cell carcinoma". Otolaryngology–Head and Neck Surgery. 141 (4): 484–90. doi:10.1016/j.otohns.2009.07.007. PMID 19786217. S2CID 8357370.
- ^ Tawfik HM, El-Maqsoud NM, Hak BH, El-Sherbiny YM (2011). "Head and neck squamous cell carcinoma: mismatch repair immunohistochemistry and promoter hypermethylation of hMLH1 gene". American Journal of Otolaryngology. 32 (6): 528–36. doi:10.1016/j.amjoto.2010.11.005. PMID 21353335.
- ^ Zou XP, Zhang B, Zhang XQ, Chen M, Cao J, Liu WJ (November 2009). "Promoter hypermethylation of multiple genes in early gastric adenocarcinoma and precancerous lesions". Human Pathology. 40 (11): 1534–42. doi:10.1016/j.humpath.2009.01.029. PMID 19695681.
- ^ Wani M, Afroze D, Makhdoomi M, Hamid I, Wani B, Bhat G, Wani R, Wani K (2012). "Promoter methylation status of DNA repair gene (hMLH1) in gastric carcinoma patients of the Kashmir valley" (PDF). Asian Pacific Journal of Cancer Prevention. 13 (8): 4177–81. doi:10.7314/APJCP.2012.13.8.4177. PMID 23098428.
- ^ Agarwal A, Polineni R, Hussein Z, Vigoda I, Bhagat TD, Bhattacharyya S, Maitra A, Verma A (2012). "Role of epigenetic alterations in the pathogenesis of Barrett's esophagus and esophageal adenocarcinoma". International Journal of Clinical and Experimental Pathology. 5 (5): 382–96. PMC 3396065. PMID 22808291. 복습하다.
- ^ Hofstad B, Vatn MH, Andersen SN, Huitfeldt HS, Rognum T, Larsen S, Osnes M (September 1996). "Growth of colorectal polyps: redetection and evaluation of unresected polyps for a period of three years". Gut. 39 (3): 449–56. doi:10.1136/gut.39.3.449. PMC 1383355. PMID 8949653.
- ^ Schmitt MW, Prindle MJ, Loeb LA (September 2012). "Implications of genetic heterogeneity in cancer". Annals of the New York Academy of Sciences. 1267 (1): 110–6. Bibcode:2012NYASA1267..110S. doi:10.1111/j.1749-6632.2012.06590.x. PMC 3674777. PMID 22954224.
- ^ Lander ES, Linton LM, Birren B, Nusbaum C, Zody MC, Baldwin J, et al. (February 2001). "Initial sequencing and analysis of the human genome". Nature. 409 (6822): 860–921. Bibcode:2001Natur.409..860L. doi:10.1038/35057062. PMID 11237011.
- ^ Yost SE, Smith EN, Schwab RB, Bao L, Jung H, Wang X, Voest E, Pierce JP, Messer K, Parker BA, Harismendy O, Frazer KA (August 2012). "Identification of high-confidence somatic mutations in whole genome sequence of formalin-fixed breast cancer specimens". Nucleic Acids Research. 40 (14): e107. doi:10.1093/nar/gks299. PMC 3413110. PMID 22492626.
- ^ Berger MF, Hodis E, Heffernan TP, Deribe YL, Lawrence MS, Protopopov A, et al. (May 2012). "Melanoma genome sequencing reveals frequent PREX2 mutations". Nature. 485 (7399): 502–6. Bibcode:2012Natur.485..502B. doi:10.1038/nature11071. PMC 3367798. PMID 22622578.
- ^ Rasnick D, Duesberg PH (June 1999). "How aneuploidy affects metabolic control and causes cancer". The Biochemical Journal. 340 (3): 621–30. doi:10.1042/0264-6021:3400621. PMC 1220292. PMID 10359645.
- ^ a b c López-Lázaro M (March 2010). "A new view of carcinogenesis and an alternative approach to cancer therapy". Molecular Medicine. 16 (3–4): 144–53. doi:10.2119/molmed.2009.00162. PMC 2802554. PMID 20062820.
- ^ Juutilainen J, Herrala M, Luukkonen J, Naarala J, Hore PJ (May 2018). "Magnetocarcinogenesis: is there a mechanism for carcinogenic effects of weak magnetic fields?". Proceedings. Biological Sciences. 285 (1879): 20180590. doi:10.1098/rspb.2018.0590. PMC 5998098. PMID 29794049.
- ^ Soto AM, Sonnenschein C (October 2004). "The somatic mutation theory of cancer: growing problems with the paradigm?". BioEssays. 26 (10): 1097–107. doi:10.1002/bies.20087. PMID 15382143.
- ^ Davies PC, Lineweaver CH (February 2011). "Cancer tumors as Metazoa 1.0: tapping genes of ancient ancestors". Physical Biology. 8 (1): 015001. Bibcode:2011PhBio...8a5001D. doi:10.1088/1478-3975/8/1/015001. PMC 3148211. PMID 21301065.
- ^ 딘, 팀. "암은 10억년 전의 생명과 닮았다고 우주생물학자들은 말한다." 호주 생명과학자, 2011년 2월 8일.2011년 2월 15일 취득.
- ^ Sterrer, W (August 2016). "Cancer - Mutational Resurrection of Prokaryote Endofossils" (PDF). Cancer Hypotheses. 1 (1): 1–15.
- ^ a b Nowell PC (October 1976). "The clonal evolution of tumor cell populations". Science. 194 (4260): 23–8. Bibcode:1976Sci...194...23N. doi:10.1126/science.959840. PMID 959840.
- ^ a b Merlo LM, Pepper JW, Reid BJ, Maley CC (December 2006). "Cancer as an evolutionary and ecological process". Nature Reviews. Cancer. 6 (12): 924–35. doi:10.1038/nrc2013. PMID 17109012. S2CID 8040576.
- ^ Hanahan D, Weinberg RA (January 2000). "The hallmarks of cancer". Cell. 100 (1): 57–70. doi:10.1016/S0092-8674(00)81683-9. PMID 10647931.
- ^ Cho RW, Clarke MF (February 2008). "Recent advances in cancer stem cells". Current Opinion in Genetics & Development. 18 (1): 48–53. doi:10.1016/j.gde.2008.01.017. PMID 18356041.
- ^ Taniguchi K, Wu LW, Grivennikov SI, de Jong PR, Lian I, Yu FX, Wang K, Ho SB, Boland BS, Chang JT, Sandborn WJ, Hardiman G, Raz E, Maehara Y, Yoshimura A, Zucman-Rossi J, Guan KL, Karin M (March 2015). "A gp130-Src-YAP module links inflammation to epithelial regeneration". Nature. 519 (7541): 57–62. Bibcode:2015Natur.519...57T. doi:10.1038/nature14228. PMC 4447318. PMID 25731159.
- ^ You H, Lei P, Andreadis ST (December 2013). "JNK is a novel regulator of intercellular adhesion". Tissue Barriers. 1 (5): e26845. doi:10.4161/tisb.26845. PMC 3942331. PMID 24868495.
- ^ Busillo JM, Azzam KM, Cidlowski JA (November 2011). "Glucocorticoids sensitize the innate immune system through regulation of the NLRP3 inflammasome". The Journal of Biological Chemistry. 286 (44): 38703–13. doi:10.1074/jbc.M111.275370. PMC 3207479. PMID 21940629.
- ^ Wang Y, Bugatti M, Ulland TK, Vermi W, Gilfillan S, Colonna M (March 2016). "Nonredundant roles of keratinocyte-derived IL-34 and neutrophil-derived CSF1 in Langerhans cell renewal in the steady state and during inflammation". European Journal of Immunology. 46 (3): 552–9. doi:10.1002/eji.201545917. PMC 5658206. PMID 26634935.
- ^ Siqueira Mietto B, Kroner A, Girolami EI, Santos-Nogueira E, Zhang J, David S (December 2015). "Role of IL-10 in Resolution of Inflammation and Functional Recovery after Peripheral Nerve Injury". The Journal of Neuroscience. 35 (50): 16431–42. doi:10.1523/JNEUROSCI.2119-15.2015. PMC 6605511. PMID 26674868.
- ^ Seifert AW, Maden M (2014). "New insights into vertebrate skin regeneration". International Review of Cell and Molecular Biology. Vol. 310. pp. 129–69. doi:10.1016/B978-0-12-800180-6.00004-9. ISBN 978-0-12-800180-6. PMID 24725426.
- ^ Kwon MJ, Shin HY, Cui Y, Kim H, Thi AH, Choi JY, Kim EY, Hwang DH, Kim BG (December 2015). "CCL2 Mediates Neuron-Macrophage Interactions to Drive Proregenerative Macrophage Activation Following Preconditioning Injury". The Journal of Neuroscience. 35 (48): 15934–47. doi:10.1523/JNEUROSCI.1924-15.2015. PMC 6605453. PMID 26631474.
- ^ Hajishengallis G, Chavakis T (January 2013). "Endogenous modulators of inflammatory cell recruitment". Trends in Immunology. 34 (1): 1–6. doi:10.1016/j.it.2012.08.003. PMC 3703146. PMID 22951309.
- ^ Nelson AM, Katseff AS, Ratliff TS, Garza LA (February 2016). "Interleukin 6 and STAT3 regulate p63 isoform expression in keratinocytes during regeneration". Experimental Dermatology. 25 (2): 155–7. doi:10.1111/exd.12896. PMC 4724264. PMID 26566817.
- ^ Vidal PM, Lemmens E, Dooley D, Hendrix S (February 2013). "The role of "anti-inflammatory" cytokines in axon regeneration". Cytokine & Growth Factor Reviews. 24 (1): 1–12. doi:10.1016/j.cytogfr.2012.08.008. PMID 22985997.
- ^ Hsueh YY, Chang YJ, Huang CW, Handayani F, Chiang YL, Fan SC, Ho CJ, Kuo YM, Yang SH, Chen YL, Lin SC, Huang CC, Wu CC (October 2015). "Synergy of endothelial and neural progenitor cells from adipose-derived stem cells to preserve neurovascular structures in rat hypoxic-ischemic brain injury". Scientific Reports. 5: 14985. Bibcode:2015NatSR...514985H. doi:10.1038/srep14985. PMC 4597209. PMID 26447335.
- ^ Yaniv M (September 2014). "Chromatin remodeling: from transcription to cancer". Cancer Genetics. 207 (9): 352–7. doi:10.1016/j.cancergen.2014.03.006. PMID 24825771.
- ^ Zhang X, He N, Gu D, Wickliffe J, Salazar J, Boldogh I, Xie J (October 2015). "Genetic Evidence for XPC-KRAS Interactions During Lung Cancer Development". Journal of Genetics and Genomics = Yi Chuan Xue Bao. 42 (10): 589–96. doi:10.1016/j.jgg.2015.09.006. PMC 4643398. PMID 26554912.
- ^ Dubois-Pot-Schneider H, Fekir K, Coulouarn C, Glaise D, Aninat C, Jarnouen K, Le Guével R, Kubo T, Ishida S, Morel F, Corlu A (December 2014). "Inflammatory cytokines promote the retrodifferentiation of tumor-derived hepatocyte-like cells to progenitor cells". Hepatology. 60 (6): 2077–90. doi:10.1002/hep.27353. PMID 25098666. S2CID 11182192.
- ^ Finkin S, Yuan D, Stein I, Taniguchi K, Weber A, Unger K, et al. (December 2015). "Ectopic lymphoid structures function as microniches for tumor progenitor cells in hepatocellular carcinoma". Nature Immunology. 16 (12): 1235–44. doi:10.1038/ni.3290. PMC 4653079. PMID 26502405.
- ^ Beane JE, Mazzilli SA, Campbell JD, Duclos G, Krysan K, Moy C, et al. (April 2019). "Molecular subtyping reveals immune alterations associated with progression of bronchial premalignant lesions". Nature Communications. 10 (1): 1856. Bibcode:2019NatCo..10.1856B. doi:10.1038/s41467-019-09834-2. PMC 6478943. PMID 31015447.
- ^ Maoz A, Merenstein C, Koga Y, Potter A, Gower AC, Liu G, et al. (September 2021). "Elevated T cell repertoire diversity is associated with progression of lung squamous cell premalignant lesions". Journal for Immunotherapy of Cancer. 9 (9): e002647. doi:10.1136/jitc-2021-002647. PMC 8477334. PMID 34580161.
- ^ a b Vlahopoulos SA, Cen O, Hengen N, Agan J, Moschovi M, Critselis E, Adamaki M, Bacopoulou F, Copland JA, Boldogh I, Karin M, Chrousos GP (August 2015). "Dynamic aberrant NF-κB spurs tumorigenesis: a new model encompassing the microenvironment". Cytokine & Growth Factor Reviews. 26 (4): 389–403. doi:10.1016/j.cytogfr.2015.06.001. PMC 4526340. PMID 26119834.
- ^ Grivennikov SI, Karin M (February 2010). "Dangerous liaisons: STAT3 and NF-kappaB collaboration and crosstalk in cancer". Cytokine & Growth Factor Reviews. 21 (1): 11–9. doi:10.1016/j.cytogfr.2009.11.005. PMC 2834864. PMID 20018552.
- ^ Rieger S, Zhao H, Martin P, Abe K, Lisse TS (January 2015). "The role of nuclear hormone receptors in cutaneous wound repair". Cell Biochemistry and Function. 33 (1): 1–13. doi:10.1002/cbf.3086. PMC 4357276. PMID 25529612.
- ^ Lu X, Yarbrough WG (February 2015). "Negative regulation of RelA phosphorylation: emerging players and their roles in cancer". Cytokine & Growth Factor Reviews. 26 (1): 7–13. doi:10.1016/j.cytogfr.2014.09.003. PMID 25438737.
- ^ Sionov RV, Fridlender ZG, Granot Z (December 2015). "The Multifaceted Roles Neutrophils Play in the Tumor Microenvironment". Cancer Microenvironment. 8 (3): 125–58. doi:10.1007/s12307-014-0147-5. PMC 4714999. PMID 24895166.
- ^ Venturi, Sebastiano (2011). "Evolutionary Significance of Iodine". Current Chemical Biology. 5 (3): 155–162. doi:10.2174/187231311796765012. ISSN 1872-3136.
- ^ Venturi S (2014). "Iodine, PUFAs and Iodolipids in Health and Disease: An Evolutionary Perspective". Human Evolution. 29 (1–3): 185–205. ISSN 0393-9375.
- ^ Walsh CJ, Luer CA, Bodine AB, Smith CA, Cox HL, Noyes DR, Maura G (December 2006). "Elasmobranch immune cells as a source of novel tumor cell inhibitors: Implications for public health". Integrative and Comparative Biology. 46 (6): 1072–1081. doi:10.1093/icb/icl041. PMC 2664222. PMID 19343108.
- ^ Vogelstein B, Kinzler KW (August 2004). "Cancer genes and the pathways they control". Nature Medicine. 10 (8): 789–99. doi:10.1038/nm1087. PMID 15286780. S2CID 205383514.
- ^ Brand KA, Hermfisse U (April 1997). "Aerobic glycolysis by proliferating cells: a protective strategy against reactive oxygen species". FASEB Journal. 11 (5): 388–95. doi:10.1096/fasebj.11.5.9141507. PMID 9141507. S2CID 16745745.
- ^ Bos JL (September 1989). "ras oncogenes in human cancer: a review". Cancer Research. 49 (17): 4682–9. PMID 2547513.
- ^ Chang EH, Furth ME, Scolnick EM, Lowy DR (June 1982). "Tumorigenic transformation of mammalian cells induced by a normal human gene homologous to the oncogene of Harvey murine sarcoma virus". Nature. 297 (5866): 479–83. Bibcode:1982Natur.297..479C. doi:10.1038/297479a0. PMID 6283358. S2CID 4234621.
- ^ Vlahopoulos SA, Logotheti S, Mikas D, Giarika A, Gorgoulis V, Zoumpourlis V (April 2008). "The role of ATF-2 in oncogenesis". BioEssays. 30 (4): 314–27. doi:10.1002/bies.20734. PMID 18348191. S2CID 678541.
- ^ Matoba S, Kang JG, Patino WD, Wragg A, Boehm M, Gavrilova O, Hurley PJ, Bunz F, Hwang PM (June 2006). "p53 regulates mitochondrial respiration". Science. 312 (5780): 1650–3. Bibcode:2006Sci...312.1650M. doi:10.1126/science.1126863. PMID 16728594. S2CID 36668814.
- ^ Knudson AG (April 1971). "Mutation and cancer: statistical study of retinoblastoma". Proceedings of the National Academy of Sciences of the United States of America. 68 (4): 820–3. Bibcode:1971PNAS...68..820K. doi:10.1073/pnas.68.4.820. PMC 389051. PMID 5279523.
- ^ Fodde R, Smits R (October 2002). "Cancer biology. A matter of dosage". Science. 298 (5594): 761–3. doi:10.1126/science.1077707. PMID 12399571. S2CID 50724.
- ^ Stephens PJ, Greenman CD, Fu B, Yang F, Bignell GR, Mudie LJ, et al. (January 2011). "Massive genomic rearrangement acquired in a single catastrophic event during cancer development". Cell. 144 (1): 27–40. doi:10.1016/j.cell.2010.11.055. PMC 3065307. PMID 21215367.
- 요약 배치 위치:
- ^ Kuipers EJ (March 1999). "Review article: exploring the link between Helicobacter pylori and gastric cancer". Alimentary Pharmacology & Therapeutics. 13 (Suppl 1): 3–11. doi:10.1046/j.1365-2036.1999.00002.x. PMID 10209681.
- ^ Kusters JG, van Vliet AH, Kuipers EJ (July 2006). "Pathogenesis of Helicobacter pylori infection". Clin. Microbiol. Rev. 19 (3): 449–90. doi:10.1128/CMR.00054-05. PMC 1539101. PMID 16847081.
- ^ Parkin DM (June 2006). "The global health burden of infection-associated cancers in the year 2002". Int. J. Cancer. 118 (12): 3030–44. doi:10.1002/ijc.21731. PMID 16404738.
- ^ Wroblewski LE, Peek RM, Wilson KT (October 2010). "Helicobacter pylori and gastric cancer: factors that modulate disease risk". Clin. Microbiol. Rev. 23 (4): 713–39. doi:10.1128/CMR.00011-10. PMC 2952980. PMID 20930071.
- ^ Ferlay J, Colombet M, Soerjomataram I, Mathers C, Parkin DM, Piñeros M, Znaor A, Bray F (April 2019). "Estimating the global cancer incidence and mortality in 2018: GLOBOCAN sources and methods". Int. J. Cancer. 144 (8): 1941–1953. doi:10.1002/ijc.31937. PMID 30350310.
- ^ Meurer LN, Bower DJ (April 2002). "Management of Helicobacter pylori infection". Am Fam Physician. 65 (7): 1327–36. PMID 11996414.
- ^ Prabhu SR, Ranganathan S, Amarapurkar DN (November 1994). "Helicobacter pylori in normal gastric mucosa". J Assoc Physicians India. 42 (11): 863–4. PMID 7868485.
- ^ White JR, Winter JA, Robinson K (2015). "Differential inflammatory response to Helicobacter pylori infection: etiology and clinical outcomes". J Inflamm Res. 8: 137–47. doi:10.2147/JIR.S64888. PMC 4540215. PMID 26316793.
- ^ Deng JY, Liang H (April 2014). "Clinical significance of lymph node metastasis in gastric cancer". World J. Gastroenterol. 20 (14): 3967–75. doi:10.3748/wjg.v20.i14.3967. PMC 3983452. PMID 24744586.
- ^ a b Valenzuela MA, Canales J, Corvalán AH, Quest AF (December 2015). "Helicobacter pylori-induced inflammation and epigenetic changes during gastric carcinogenesis". World J. Gastroenterol. 21 (45): 12742–56. doi:10.3748/wjg.v21.i45.12742. PMC 4671030. PMID 26668499.
- ^ Raza Y, Khan A, Farooqui A, Mubarak M, Facista A, Akhtar SS, Khan S, Kazi JI, Bernstein C, Kazmi SU (2014). "Oxidative DNA damage as a potential early biomarker of Helicobacter pylori associated carcinogenesis". Pathol. Oncol. Res. 20 (4): 839–46. doi:10.1007/s12253-014-9762-1. PMID 24664859. S2CID 18727504.
- ^ Koeppel M, Garcia-Alcalde F, Glowinski F, Schlaermann P, Meyer TF (June 2015). "Helicobacter pylori Infection Causes Characteristic DNA Damage Patterns in Human Cells". Cell Rep. 11 (11): 1703–13. doi:10.1016/j.celrep.2015.05.030. PMID 26074077.
- ^ a b Muhammad JS, Eladl MA, Khoder G (February 2019). "Helicobacter pylori-induced DNA Methylation as an Epigenetic Modulator of Gastric Cancer: Recent Outcomes and Future Direction". Pathogens. 8 (1): 23. doi:10.3390/pathogens8010023. PMC 6471032. PMID 30781778.
- ^ a b Noto JM, Peek RM (2011). "The role of microRNAs in Helicobacter pylori pathogenesis and gastric carcinogenesis". Front Cell Infect Microbiol. 1: 21. doi:10.3389/fcimb.2011.00021. PMC 3417373. PMID 22919587.
- ^ Santos JC, Ribeiro ML (August 2015). "Epigenetic regulation of DNA repair machinery in Helicobacter pylori-induced gastric carcinogenesis". World J. Gastroenterol. 21 (30): 9021–37. doi:10.3748/wjg.v21.i30.9021. PMC 4533035. PMID 26290630.
- ^ a b Raza Y, Ahmed A, Khan A, Chishti AA, Akhter SS, Mubarak M, Bernstein C, Zaitlin B, Kazmi SU (February 2020). "Helicobacter pylori severely reduces expression of DNA repair proteins PMS2 and ERCC1 in gastritis and gastric cancer". DNA Repair (Amst.). 89: 102836. doi:10.1016/j.dnarep.2020.102836. PMID 32143126.
- ^ Dore MP, Pes GM, Bassotti G, Usai-Satta P (2016). "Dyspepsia: When and How to Test for Helicobacter pylori Infection". Gastroenterol Res Pract. 2016: 8463614. doi:10.1155/2016/8463614. PMC 4864555. PMID 27239194.
- ^ Carrillo-Infante C, Abbadessa G, Bagella L, Giordano A (June 2007). "Viral infections as a cause of cancer (review)". International Journal of Oncology. 30 (6): 1521–8. doi:10.3892/ijo.30.6.1521. PMID 17487374.
- ^ Safdar A (2011). Management of Infections in Cancer Patients. Springer. p. 478. ISBN 978-1-60761-643-6.
- ^ Samaras V, Rafailidis PI, Mourtzoukou EG, Peppas G, Falagas ME (June 2010). "Chronic bacterial and parasitic infections and cancer: a review". Journal of Infection in Developing Countries. 4 (5): 267–81. doi:10.3855/jidc.819. PMID 20539059.
- ^ Daniel FI, Cherubini K, Yurgel LS, de Figueiredo MA, Salum FG (February 2011). "The role of epigenetic transcription repression and DNA methyltransferases in cancer". Cancer. 117 (4): 677–87. doi:10.1002/cncr.25482. PMID 20945317. 복습하다.
- ^ Kanwal R, Gupta S (April 2012). "Epigenetic modifications in cancer". Clinical Genetics. 81 (4): 303–11. doi:10.1111/j.1399-0004.2011.01809.x. PMC 3590802. PMID 22082348.
- ^ Pattani KM, Soudry E, Glazer CA, Ochs MF, Wang H, Schussel J, Sun W, Hennessey P, Mydlarz W, Loyo M, Demokan S, Smith IM, Califano JA (2012). Tao Q (ed.). "MAGEB2 is activated by promoter demethylation in head and neck squamous cell carcinoma". PLOS ONE. 7 (9): e45534. Bibcode:2012PLoSO...745534P. doi:10.1371/journal.pone.0045534. PMC 3454438. PMID 23029077.
- ^ Sampath D, Liu C, Vasan K, Sulda M, Puduvalli VK, Wierda WG, Keating MJ (February 2012). "Histone deacetylases mediate the silencing of miR-15a, miR-16, and miR-29b in chronic lymphocytic leukemia". Blood. 119 (5): 1162–72. doi:10.1182/blood-2011-05-351510. PMC 3277352. PMID 22096249.
- ^ Hitchler MJ, Oberley LW, Domann FE (December 2008). "Epigenetic silencing of SOD2 by histone modifications in human breast cancer cells". Free Radical Biology & Medicine. 45 (11): 1573–80. doi:10.1016/j.freeradbiomed.2008.09.005. PMC 2633123. PMID 18845242.
- ^ Baldassarre G, Battista S, Belletti B, Thakur S, Pentimalli F, Trapasso F, Fedele M, Pierantoni G, Croce CM, Fusco A (April 2003). "Negative regulation of BRCA1 gene expression by HMGA1 proteins accounts for the reduced BRCA1 protein levels in sporadic breast carcinoma". Molecular and Cellular Biology. 23 (7): 2225–38. doi:10.1128/MCB.23.7.2225-2238.2003. PMC 150734. PMID 12640109.
- ^ Dalerba P, Cho RW, Clarke MF (2007). "Cancer stem cells: models and concepts". Annual Review of Medicine. 58: 267–84. doi:10.1146/annurev.med.58.062105.204854. PMID 17002552.
- ^ Zhang W, Hanks AN, Boucher K, Florell SR, Allen SM, Alexander A, Brash DE, Grossman D (January 2005). "UVB-induced apoptosis drives clonal expansion during skin tumor development". Carcinogenesis. 26 (1): 249–57. doi:10.1093/carcin/bgh300. PMC 2292404. PMID 15498793.
추가 정보
- Tokar EJ, Benbrahim-Tallaa L, Waalkes MP (2011). "Chepter 14. Metal Ions in Human Cancer Development". In Sigel A, Sigel H, Sigel RK (eds.). Metal ions in toxicology: effects, interactions, interdependencies. Metal Ions in Life Sciences. Vol. 8. RSC Publishing. pp. 375–401. doi:10.1039/9781849732116-00375. ISBN 978-1-84973-091-4.
- Dixon K, Kopras E (December 2004). "Genetic alterations and DNA repair in human carcinogenesis". Seminars in Cancer Biology. 14 (6): 441–8. doi:10.1016/j.semcancer.2004.06.007. PMID 15489137.
- Kleinsmith LJ (2006). Principles of cancer biology. San Francisco: Pearson Benjamin Cummings. ISBN 978-0-8053-4003-7.
- Sarasin A (November 2003). "An overview of the mechanisms of mutagenesis and carcinogenesis". Mutation Research. 544 (2–3): 99–106. doi:10.1016/j.mrrev.2003.06.024. PMID 14644312.
- Schottenfeld D, Beebe-Dimmer JL (2005). "Advances in cancer epidemiology: understanding causal mechanisms and the evidence for implementing interventions". Annual Review of Public Health. 26: 37–60. doi:10.1146/annurev.publhealth.26.021304.144402. PMID 15760280.
- Tannock I, Hill R, Bristow R, Harrington L (2005). The basic science of oncology (4th ed.). New York: McGraw-Hill. ISBN 978-0-07-138774-3.
- Wicha MS, Liu S, Dontu G (February 2006). "Cancer stem cells: an old idea--a paradigm shift". Cancer Research. 66 (4): 1883–90, discussion 1895–6. doi:10.1158/0008-5472.CAN-05-3153. PMID 16488983.