거의 중립적인 분자 진화 이론
Nearly neutral theory of molecular evolution분자 진화의 거의 중립적인 이론은 모든 돌연변이가 무시될 수 있을 정도로 그렇게 해로운 것은 아니라는 사실을 설명하는 분자 진화의 중립 이론을 수정한 것입니다.약간 유해한 돌연변이는 선택 계수를 유효 모집단 크기로 나눈 값보다 클 때만 확실하게 제거된다.더 큰 집단에서, 더 높은 비율의 돌연변이는 유전자 표류가 선택을 압도할 수 없는 이 임계값을 초과하여, 더 적은 고정 이벤트와 더 느린 분자 진화를 초래한다.
거의 중립적인 이론은 1973년 [1]오타 도모코에 의해 제안되었다.돌연변이를 제거하기 위한 개체 수에 의존하는 역치는 마이클 린치에 의해 "떠다니는 장벽"이라고 불리며, 종들 간의 게놈 구조의 차이를 설명하는데 사용되었다.
거의 중립적인 이론의 기원
분자 진화의 중성 이론에 따르면, 종들 사이에 분자 변화가 축적되는 속도는 중성 돌연변이 속도와 같아야 하며, 따라서 종들 간에 비교적 일정해야 합니다.단, 이것은 세대별 비율입니다.더 큰 유기체는 더 긴 생성 시간을 가지고 있기 때문에, 중성 이론은 그들의 분자 진화 속도가 더 느릴 것이라고 예측한다.하지만, 분자 진화론자들은 단백질 진화의 속도가 발생 시간과 상당히 무관하다는 것을 발견했습니다.
오타 토모코(大田oko子)는 모집단 크기가 일반적으로 생성 시간에 반비례한다는 점에 주목하면서 대부분의 아미노산 치환기가 약간 유해한 경우, 이는 작은 모집단에서 효과적으로 중성 돌연변이율을 증가시켜 긴 생성 시간의 영향을 상쇄할 수 있다고 제안했다.그러나 코드화되지 않은 DNA 치환은 더 중립적인 경향이 있기 때문에, 비동명적인 변화 속도와 달리, 그들의 진화 속도는 모집단 크기/[2]세대 시간에 따라 결정될 것으로 정확하게 예측된다.
이 경우, 작은 집단에서 예상되는 단백질에서 중성 진화의 빠른 속도는 (악성 돌연변이를 제거하기 위한 더 관대한 임계값으로 인해) 더 긴 생성 시간만큼 상쇄되지만, 짧은 생성 시간을 가진 큰 집단에서, 비코드 DNA는 선택에 의해 지연되는 동안 더 빨리 진화한다(w1973년,[2] 오타는 다양한 분자 증거가 분자 수준에서 대부분의 돌연변이 사건이 엄격히 중립적이기 보다는 약간 유해하다는 이론을 뒷받침한다는 짧은 편지를[1] 네이처에 발표했다.
그때와 1990년대 초 사이에, 분자 진화에 대한 많은 연구는 돌연변이가 고정될 때 유해한 돌연변이로 인한 집단의 적합성에 대한 부정적인 영향이 원래 값으로 다시 이동하는 "시프트 모델"을 사용했다.1990년대 초, 오타는 유익하고 해로운 돌연변이를 모두 포함하는 "고정 모델"을 개발하여 전체 모집단 적합성의 인위적인 "이동"이 [2]필요하지 않았다.그러나 오타에 따르면, 1980년대 후반에 거의 중립에 가까운 이론은 대부분 지지를 받지 못했는데, 이는 빠른 DNA 염기서열 분석의 등장 이후 널리 퍼진 분자 체계학 연구를 위한 수학적으로 단순한 중립 이론이기 때문이다.1990년대에 보다 상세한 계통학 연구가 강한 선택과 약한 선택 대상 게놈 영역의 진화를 비교하기 시작하면서, 거의 중립적인 이론과 선택과 표류 사이의 상호작용이 다시 한번 연구의 [3]중요한 초점이 되었다.
이론.
대체율은,ρ{\displaystyle \rho} 있다.
- NePg ρ)u¯ fi}{\displaystyle\rho =ugN_{e}{\bar{P}}_{ 고치}x.
어디 너{\displaystyle u}은 변이 속도, g{\displaystyle g}은 세대 시간 및 Ne{\displaystyle N_{e}}은 유효 집단 크기이다.마지막 학기 확률은 새로운 돌연변이 고정이 될 것이다.초기 모델들 그런 식으로{\displaystyle u}종들 사이에, Ne{\displaystyle N_{e}과 g{\displaystyle g}증가}상수입니다. 잡은 고정의 반수체 인구의 확률에 대한 기무라의 방정식:을 준다.
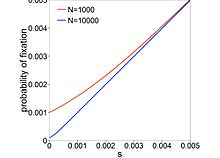
- Pfix=1− es− 1− es− Ne{\displaystyle P_{를 고치}={\frac{1-e^{-s}}{1-e^{-sN_{e}}}}},.
서 ss는 돌연변이의 선택 계수입니다. e \ s \_ { N _ { }} ( 완전 중립), p N \ P_ { } = frac 1 { N _ { { n 및s e \ style - - s - s - s - s - - s - s \ - s - s \ { n { n { n { n { { n { n } { n } { n { N 와 연속적으로 일치하며, ≤ e({와의돌연변이를 거의 중성 돌연변이라고 한다.이러한 돌연변이는 유전적 표류를 통해 의 모집단에서 수정될 수 있다.에서 이러한 돌연변이는 선택으로 삭제됩니다거의 중성 돌연변이가 일반적인 경우 f x 1 e{ }\{} 의 비율은 }}}}에 의존합니다.
거의 중립에 가까운 돌연변이의 영향은의에 따라 달라질 수 있습니다. 초기 작업에서는세대마다 다를 수 " 모델"을 사용했지만 고정 후에는 모집단의 평균 적합성이 0으로 재설정됩니다.이는 기본적으로 ss의 가 일정하다고 가정한다(이러한 의미에서 이전 단락의 주장은 "시프트 모델"에 기초한 것으로 간주할 수 있다).이러한 가정은 단백질 기능의 무기한 개선 또는 저하로 이어질 수 있다.대안으로, 이후의 "고정 모델"[4]은 단백질 기능에 대한 돌연변이 효과의 분포를 고정하지만 모집단의 평균 적합성이 진화하도록 한다.이를 통해 모집단의 평균 적합성에 따라를 변경할 수 있습니다.
"고정 모델"은 단백질의 진화 속도에 대해 약간 다른 설명을 제공한다.에서는 유리한 돌연변이가 선택으로 빠르게 포착되어 모집단의 평균 적합성이 높아진다이에 따라 이들 돌연변이가 선택계수 분포의 꼬리로 제한되기 때문에 거의 중립적인 돌연변이의 돌연변이율이 감소한다.
"고정 모델"은 거의 중립적인 이론을 확장합니다.그“모수 모형”Ne{\displaystyle N_{e}의 제품에}바탕으로 하여 s의 분포{s\displaystyle}:적응 진화에 큰 제품과 일치한 차이 아래 Tachida[5]분류 진화, 중간 제품 거의 중립 진화에 들었고, 작은 생산은 거의 neutr에 해당한다.알진화.이 분류에 따르면, 약간 유리한 돌연변이는 거의 중립적인 진화에 기여할 수 있다.
'떠내려가는 장벽" 이론
마이클 린치는 약간 유해한 돌연변이를 제거하는 능력의 변화(, Ne N_가 게놈의 크기나 돌연변이율 [6]등 종 간의 게놈 구조의 변화를 설명할 수 있다고 제안했다.특히, 모집단이 클수록 돌연변이율이 낮고, 게놈 구조가 합리화되며, 일반적으로 더 미세하게 조정된 적응이 이루어집니다.그러나 전사나 번역 등의 프로세스에서 발생할 수 있는 각 오류의 결과에 대한 견실성이 이러한 오류의 발생 비용을 크게 줄인다면 모집단이 많을수록 글로벌 교정률이 낮아질 수 있으므로 [7]오류율이 높아질 수 있습니다.이것은 왜 대장균이 사카로미세스 세레비시아에 [8][9]비해 전사 오류율이 높은지 설명할 수 있다.이는 대장균의 전사 오류율이 단백질 풍부성(선택의 궤적 특이적 강도를 조절하는 역할을 함)에 따라 달라지지만, S. cerevisae의 [10]높은 오류율 C-U 탈아미네이션 오류에 대해서만 해당된다는 사실에 의해 뒷받침된다.
「 」를 참조해 주세요.
레퍼런스
- ^ a b Ohta T (November 1973). "Slightly deleterious mutant substitutions in evolution". Nature. 246 (5428): 96–8. doi:10.1038/246096a0. PMID 4585855. S2CID 4226804.
- ^ a b c Ohta T, Gillespie JH (April 1996). "Development of Neutral and Nearly Neutral Theories". Theoretical Population Biology. 49 (2): 128–42. CiteSeerX 10.1.1.332.2080. doi:10.1006/tpbi.1996.0007. PMID 8813019.
- ^ Ohta T (August 1996). "The current significance and standing of neutral and neutral theories". BioEssays. 18 (8): 673–7, discussion 683. doi:10.1002/bies.950180811. PMID 8779656.
- ^ Ohta T, Tachida H (September 1990). "Theoretical study of near neutrality. I. Heterozygosity and rate of mutant substitution". Genetics. 126 (1): 219–29. doi:10.1093/genetics/126.1.219. PMC 1204126. PMID 2227381.
- ^ Tachida H (May 1991). "A study on a nearly neutral mutation model in finite populations". Genetics. 128 (1): 183–92. doi:10.1093/genetics/128.1.183. PMC 1204447. PMID 2060776.
- ^ Lynch M (2007). The origins of genome architecture. Sunderland: Sinauer Associates.
- ^ Rajon, E.; Masel, J. (3 January 2011). "Evolution of molecular error rates and the consequences for evolvability". Proceedings of the National Academy of Sciences. 108 (3): 1082–1087. Bibcode:2011PNAS..108.1082R. doi:10.1073/pnas.1012918108. PMC 3024668. PMID 21199946.
- ^ "Correction for Traverse and Ochman, Conserved rates and patterns of transcription errors across bacterial growth states and lifestyles". Proceedings of the National Academy of Sciences. 113 (29): E4257–E4258. 19 July 2016. doi:10.1073/pnas.1609677113. PMC 4961203. PMID 27402746.
- ^ Xiong, Kun; McEntee, Jay P.; Porfirio, David J.; Masel, Joanna (January 2017). "Drift Barriers to Quality Control When Genes Are Expressed at Different Levels". Genetics. 205 (1): 397–407. doi:10.1534/genetics.116.192567. PMC 5223517. PMID 27838629.
- ^ Meer, K M; Nelson, P G; Xiong, K; Masel, J (16 December 2019). "High transcriptional error rates vary as a function of gene expression level". Genome Biology and Evolution. 12 (1): 3754–3761. doi:10.1093/gbe/evz275. PMC 6988749. PMID 31841128.






 비율은
비율은 
