미토콘드리아
Mitochondrion
미토콘드리아는 동물, 식물, 곰팡이와 같은 대부분의 진핵생물의 세포에서 발견되는 소기관입니다.[1] 미토콘드리아는 이중막 구조를 가지고 있으며 호기성 호흡을 이용하여 아데노신 삼인산(ATP)을 생성하고, 이는 세포 전체에서 화학 에너지의 공급원으로 사용됩니다.[2] 그들은 1857년[3] 알버트 폰 쾰리커에 의해 곤충들의 자발적인 근육에서 발견되었습니다. 미토콘드리아라는 용어는 1898년 칼 벤다에 의해 만들어졌습니다. 미토콘드리아는 1957년 필립 시케비츠가 같은 이름의 기사에서 만든 구절인 "세포의 강자"라는 대중적인 별명으로 불립니다.[4]
일부 다세포 생물의 일부 세포에는 미토콘드리아(예: 성숙한 포유류 적혈구)가 부족합니다. 미세포자충, 파라바살리드, 디플로모나드와 같은 많은 단세포 생물이 미토콘드리아를 다른 구조로 줄이거나 변형시켰습니다.[5] 진핵생물인 모노케르코모노이드는 미토콘드리아를 완전히 상실한 것으로 알려져 있으며,[6] 다세포 생물인 헤네구야 살미니콜라는 미토콘드리아 유전체의 완전한 상실과 관련하여 미토콘드리아 관련 소기관을 보유하고 있는 것으로 알려져 있습니다.[6][7][8]
미토콘드리아는 일반적으로 단면이 0.75에서 3μm2 [9]사이이지만 크기와 구조는 상당히 다양합니다. 특별히 얼룩이 지지 않는 한 보이지 않습니다. 미토콘드리아는 세포 에너지를 공급하는 것 외에도 신호전달, 세포 분화, 세포 사멸과 같은 다른 일에 관여하며, 세포 주기와 세포 성장의 제어를 유지합니다.[10] 미토콘드리아 생물 발생은 차례로 이러한 세포 과정과 시간적으로 조정됩니다.[11][12] 미토콘드리아는 미토콘드리아 질환,[13] 심장 기능 장애,[14] 심부전[15] 및 자폐증과 같은 인간의 여러 장애 및 상태와 관련이 있습니다.[16]
세포 내 미토콘드리아의 수는 유기체, 조직 및 세포 유형에 따라 크게 다를 수 있습니다. 성숙한 적혈구는 미토콘드리아가 없는 [17]반면 간세포는 2000개 이상을 가질 수 있습니다.[18][19] 미토콘드리아는 특화된 기능을 수행하는 구획으로 구성되어 있습니다. 이러한 구획 또는 영역에는 외막, 막간 공간, 내막, 크리스타 및 기질이 포함됩니다.
진핵세포의 DNA는 대부분 세포핵에 포함되어 있지만, 미토콘드리아에는 박테리아 유전체와 거의 비슷한 고유의 유전체("mitogenome")가 있습니다.[20] 이 발견은 현대 미토콘드리아의 자유로운 원핵생물 조상이 먼 과거에 진핵세포와 영구적으로 융합되어 현대의 동물, 식물, 곰팡이 및 기타 진핵생물이 호흡하여 세포 에너지를 생성할 수 있도록 진화한다는 내공생 가설의 일반적인 수용으로 이어졌습니다.[21]
구조.
| 세포생물학 | |
|---|---|
| 미토콘드리아 | |
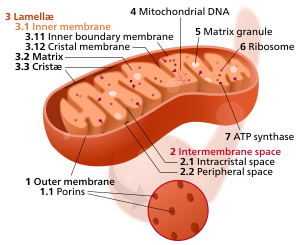 |
미토콘드리아는 여러 가지 다른 모양을 가질 수 있습니다.[22] 미토콘드리아는 인지질 이중층과 단백질로 구성된 외막과 내막을 포함합니다.[18] 두 멤브레인은 서로 다른 특성을 가지고 있습니다. 이 이중막 조직 때문에 미토콘드리아에는 다섯 가지 다른 부분이 있습니다.
미토콘드리아는 표면적을 증가시키기 위해 접힘이 있고, 이것은 다시 ATP(adenosine triphosphate) 생산을 증가시킵니다. 미토콘드리아의 외막이 제거된 것을 미토콘드리아라고 합니다.
외막
세포 소기관 전체를 감싸고 있는 바깥쪽 미토콘드리아 막은 60~75 옹스트롬(Å) 두께입니다. 세포막과 유사한 단백질 대 인지질 비율(중량 기준 약 1:1)을 가지고 있습니다. 포린이라고 불리는 통합 막 단백질이 다량 함유되어 있습니다. 주요 트래피킹 단백질은 기공 형성 전압 의존성 음이온 채널(VDAC)입니다. VDAC는 세포질과 막간 공간 사이의 뉴클레오티드, 이온 및 대사 생성물의 주요 수송체입니다.[23][24] 그람음성세균막과 유사하게 외막에 걸쳐 있는 베타배럴로 형성됩니다.[25] N-말단에 있는 신호전달 서열이 외막의 트랜스로카제라고 불리는 큰 다중소단위 단백질에 결합하면 더 큰 단백질이 미토콘드리아로 들어갈 수 있고, 이 단백질은 막을 가로질러 활발하게 이동합니다.[26] 미토콘드리아 프로 단백질은 전문적인 전위 복합체를 통해 수입됩니다.
외막에는 지방산의 신장, 에피네프린의 산화, 트립토판의 분해와 같은 다양한 활동에 관여하는 효소도 포함되어 있습니다. 이러한 효소에는 모노아민 산화효소, 로테논에 민감하지 않은 NADH-사이토크롬 c-환원효소, 키뉴레닌 하이드록실화효소 및 지방산 Co-A 연결효소가 포함됩니다. 외막이 파괴되면 막간 공간에 있는 단백질이 세포질로 누출되어 세포사멸로 이어집니다.[27] 외부 미토콘드리아 막은 MAM(mitocondria-associated ER-membrane)이라고 불리는 구조에서 소포체(ER) 막과 결합할 수 있습니다. 이것은 ER-미토콘드리아 칼슘 신호전달에 중요하며 ER과 미토콘드리아 사이의 지질 전달에 관여합니다.[28] 외막 바깥에는 파슨의 소단위체라는 이름의 작은(직경: 60Å) 입자들이 있습니다.
막간 공간
미토콘드리아 막간 공간은 외막과 내막 사이의 공간입니다. 주변 연골 공간으로도 알려져 있습니다. 외막은 작은 분자에도 자유롭게 투과되기 때문에 막간 공간의 이온, 당 등 작은 분자의 농도는 세포질과 같습니다.[18] 그러나 큰 단백질은 외막을 가로질러 수송되기 위해서는 특정한 신호전달 서열을 가져야 하므로 이 공간의 단백질 구성은 세포질의 단백질 구성과 다릅니다. 이런 식으로 막간 공간에 국한된 단백질 중 하나가 사이토크롬 c입니다.[27]
내막
내부 미토콘드리아 막은 다음과 같은 세 가지 기능을 가진 단백질을 포함합니다.[18]
151종 이상의 다양한 폴리펩티드를 함유하고 있으며, 매우 높은 단백질 대 인지질 비율(중량 기준 3:1 이상, 인지질 15개에 대해 약 1개의 단백질)을 가지고 있습니다. 내막에는 미토콘드리아에 있는 전체 단백질의 약 1/5이 있습니다.[29] 또한 내막에는 특이한 인지질인 카디오리핀이 풍부합니다. 이 인지질은 원래 1942년 소의 심장에서 발견된 것으로, 보통 미토콘드리아와 세균의 원형질막의 특징입니다.[30] 카디올리핀은 2개가 아닌 4개의 지방산을 함유하고 있으며, 내막을 불투과성으로 만드는 데 도움을 줄 수 있으며,[18] 그 분해는 신경계 질환 및 암을 포함한 여러 임상 질환을 유발할 수 있습니다.[31] 내막은 외막과 달리 포린을 포함하지 않으며, 모든 분자에 대해 불투과성이 높습니다. 거의 모든 이온과 분자는 매트릭스에 들어가거나 나가기 위해 특수한 막 수송체를 필요로 합니다. 단백질은 내막(TIM) 복합체의 트랜스로카제를 통해 또는 OXA1L을 통해 기질로 운반됩니다.[26] 또한 내막을 가로질러 전자 수송 사슬의 효소의 작용에 의해 형성되는 막 전위가 있습니다. 내막 융합은 내막 단백질 OPA1에 의해 매개됩니다.[32]
크리스태

내부 미토콘드리아 막은 크리스타라고 불리는 수많은 주름으로 구획되어 있는데, 크리스타는 내부 미토콘드리아 막의 표면적을 확장시켜 ATP를 생성하는 능력을 향상시킵니다. 전형적인 간 미토콘드리아의 경우 내막의 면적이 외막의 면적에 비해 약 5배 정도 넓습니다. 이 비율은 가변적이며 근육세포와 같이 ATP 수요가 더 많은 세포의 미토콘드리아는 훨씬 더 많은 크리스타를 포함합니다. 같은 세포 내의 미토콘드리아는 크리스타 밀도가 상당히 다를 수 있으며, 더 많은 에너지를 생산하는 데 필요한 미토콘드리아는 훨씬 더 많은 크리스타 막 표면을 가지고 있습니다.[33] 이 주름에는 F1 입자 또는 옥시솜으로 알려진 작고 둥근 몸체가 박혀 있습니다.[34]
매트릭스
매트릭스는 내막으로 둘러싸인 공간입니다. 미토콘드리아에 전체 단백질의 약 2/3를 함유하고 있습니다.[18] 기질은 내막에 포함된 ATP 합성효소의 도움으로 ATP 생성에 중요합니다. 매트릭스에는 수백 개의 효소, 특수 미토콘드리아 리보솜, tRNA 및 미토콘드리아 DNA 유전체의 여러 사본이 고도로 농축된 혼합물이 포함되어 있습니다. 효소 중 주요 기능으로는 피루브산과 지방산의 산화, 시트르산 회로 등이 있습니다.[18] DNA 분자는 단백질에 의해 핵양체로 포장되며, 그 중 하나는 TFAM입니다.[35]
기능.
미토콘드리아의 가장 두드러진 역할은 호흡을 통해 세포의 에너지 화폐인 ATP(즉, ADP의 인산화)를 생성하고 세포 대사를 조절하는 것입니다.[19] ATP 생성에 관련된 중심 반응 세트는 시트르산 회로 또는 크렙스 회로, 산화적 인산화로 통칭됩니다. 그러나 미토콘드리아에는 ATP 생성 외에도 많은 다른 기능이 있습니다.
에너지 변환
미토콘드리아의 지배적인 역할은 ATP의 생산인데, 이는 이 작업을 위해 내막에 있는 많은 수의 단백질에 의해 반영됩니다. 이것은 세포질에서 생성되는 포도당의 주요 생성물인 피루브산과 NADH를 산화시킴으로써 이루어집니다.[19] 호기성 호흡으로 알려진 이러한 유형의 세포 호흡은 산소의 존재에 따라 달라집니다. 산소가 제한되면 해당 생성물은 미토콘드리아와 독립적인 과정인 혐기성 발효에 의해 대사됩니다.[19] 포도당과 산소로부터 ATP의 생성은 발효에 비해 호기성 호흡 동안 약 13배 더 높은 수율을 갖습니다.[36] 식물 미토콘드리아는 또한 광합성 과정에서 생성된 당을 분해하거나 대체 기질인 아질산염을 사용하여 산소 없이 제한된 양의 ATP를 생성할 수 있습니다.[37] ATP는 특정 단백질의 도움으로 내막을 통과하고, 포린을 통해 외막을 통과합니다.[38] 에너지를 방출하는 탈인산화에 의해 ATP가 ADP로 전환된 후, ADP는 같은 경로를 통해 되돌아옵니다.
피루브산과 시트르산 회로
해당과정에 의해 생성된 피루브산 분자는 내부 미토콘드리아 막을 가로질러 기질로 활발하게 수송되고, 이들은 산화되어 조효소 A와2 결합하여 CO, 아세틸-CoA 및 [19]NADH를 형성하거나 (피루브산 카르복실라제에 의해) 카르복실화되어 옥살아세트산을 형성할 수 있습니다. 이 후자의 반응은 시트르산 회로에서 옥살로아세트산염의 양을 "채워"므로, 조직의 에너지 요구량(예: 근육에서)이 갑자기 활성에 의해 증가할 때 아세틸-CoA를 대사하는 회로의 능력을 증가시키는 보충 반응입니다.[39]
시트르산 회로에서 모든 중간체(예: 구연산염, 이소-구연산염, 알파-케토글루타레이트, 숙시네이트, 푸마레이트, 말레이트 및 옥살로아세트산염)는 사이클의 각 턴 동안 재생됩니다. 따라서 미토콘드리아에 이러한 중간체를 더 많이 첨가한다는 것은 추가적인 양이 주기 내에 유지된다는 것을 의미하며, 하나가 다른 하나로 전환됨에 따라 다른 모든 중간체가 증가합니다. 따라서, 이들 중 하나를 사이클에 추가하면 보조 효과가 있고, 그 제거는 보조 효과가 있습니다. 이러한 보충 및 투석 반응은 사이클 과정에서 아세틸-CoA와 결합하여 시트르산을 형성하는 데 사용할 수 있는 옥살아세트산염의 양을 증가시키거나 감소시킬 것입니다. 이것은 차례로 미토콘드리아에 의한 ATP 생성 속도를 증가시키거나 감소시키고, 따라서 세포에 대한 ATP의 가용성을 증가시킵니다.[39]
그러나 시트르산 회로에 들어가는 유일한 연료는 피루브산 산화 또는 지방산의 베타 산화에서 유래하는 아세틸-CoA입니다. 사이클이 바뀔 때마다 한 분자의 아세틸-CoA는 미토콘드리아 기질에 존재하는 옥살로아세트산의 모든 분자에 대해 소비되며, 결코 재생되지 않습니다. CO와 물을 생성하는 아세틸-CoA의 아세테이트2 부분의 산화이며, 방출된 에너지는 ATP의 형태로 포획됩니다.[39]
간에서 세포질 피루브산의 미토콘드리아 내 옥살아세트산으로의 카르복실화는 혈중 글루카곤 및/또는 에피네프린의 고수준의 영향 [19][39]하에 젖산 및 탈아미노화 알라닌을 포도당으로 전환시키는 글루코네오제닉 경로의 초기 단계입니다.[39] 여기서, 미토콘드리아에 옥살로아세트산을 첨가하는 것은 순 아나플러틱 효과를 갖지 않습니다. 다른 시트르산 회로 중간체(말레이트)는 미토콘드리아에서 즉시 제거되어 세포질 옥살로아세트산으로, 그리고 궁극적으로 해당과정의 거의 반대과정에서 포도당으로 전환되기 때문입니다.[39]
복합체 II의 내부 미토콘드리아 막에 결합된 숙시네이트 탈수소효소를 제외하고, 시트르산 회로의 효소는 미토콘드리아 기질에 위치합니다.[40] 시트르산 회로는 아세틸-CoA를 이산화탄소로 산화시키고, 그 과정에서 전자 수송 사슬의 전자 공급원인 환원된 보조인자(NADH 3분자와 FADH2 1분자)와 GTP 1분자(ATP로 쉽게 전환됨)를 생성합니다.[19]
O와2 NADH: 에너지 방출 반응

NADH와 FADH의2 전자는 전자 수송 사슬을 통해 산소(O2)와 수소(양성자)로 여러 단계로 전달됩니다. NADH 및 FADH2 분자는 시트르산 회로를 통해 기질 내에서 생성되고 해당과정에 의해 세포질에서 생성됩니다. 세포질에서 환원 당량은 항포터 단백질의 말산-아스파르트산 셔틀 시스템을 통해 가져오거나 글리세롤 인산 셔틀을 사용하여 전자 수송 사슬로 공급될 수 있습니다.[19]
미토콘드리아를 "세포의 강자"로 만드는 주요 에너지 방출 반응은 미토콘드리아 내부 막의 단백질 복합체 I, III, IV(NADH 탈수소효소(유비퀴논), 시토크롬 c 환원효소, 시토크롬 c 산화효소)에서 발생합니다. 복합체 IV에서 O는2 사이토크롬 c의 환원된 형태의 철과 반응합니다.
유기 연료의 결합을 끊지 않고 반응물로부터 많은 자유 에너지를[42][41] 방출합니다. 사이토크롬 c의 Fe가3+ 반응하여 유비퀴놀(QH2)을 산화시킬 때, Fe에서2+ 전자를 제거하기 위해 투입된 자유 에너지는 복합체 III에서 방출됩니다.
생성된 유비퀴논은 복합체 I에서 NADH와 반응합니다.
반응은 전자 수송 사슬에 의해 제어되지만, 자유 전자는 표시된 세 가지 반응에서 반응물 또는 생성물에 포함되지 않으므로 양성자(H)를+ 막간 공간으로 펌핑하는 데 사용되는 자유 에너지에 영향을 미치지 않습니다. 이 과정은 효율적이지만 적은 비율의 전자가 산소를 조기에 감소시켜 초과산화물과 같은 활성 산소 종을 형성할 수 있습니다.[19] 이것은 미토콘드리아에 산화 스트레스를 유발할 수 있으며 노화와 관련된 미토콘드리아 기능 저하에 기여할 수 있습니다.[43]
막간 공간에서 양성자 농도가 증가함에 따라 내막을 가로질러 강한 전기화학적 구배가 확립됩니다. 양성자는 ATP 합성효소 복합체를 통해 기질로 돌아갈 수 있으며, 이들의 잠재적 에너지는 ADP와 무기 인산염(Pi)으로부터 ATP를 합성하는 데 사용됩니다.[19] 이 과정은 화학삼투증이라고 불리며 1978년 노벨 화학상을 수상한 [44][45]피터 미첼에 의해 처음 기술되었습니다. 이후 1997년 노벨 화학상의 일부가 폴 D에게 수여되었습니다. 보이어와 존 E. ATP 합성효소의 작동 메커니즘을 설명하기 위한 워커.[46]
열생산
특정 조건에서 양성자는 ATP 합성에 기여하지 않고 미토콘드리아 기질에 다시 들어갈 수 있습니다. 이 과정은 양성자 누출 또는 미토콘드리아 분리로 알려져 있으며, 양성자의 매트릭스 내 확산이 촉진되기 때문입니다. 이 과정을 통해 양성자 전기화학적 구배의 연마되지 않은 퍼텐셜 에너지가 열로 방출됩니다.[19] 이 과정은 테르모제닌 또는 UCP1이라는 양성자 채널에 의해 매개됩니다.[47] 테르모제닌은 주로 갈색 지방 조직, 즉 갈색 지방에서 발견되며, 흔들림 없는 열 발생을 담당합니다. 갈색 지방 조직은 포유류에서 발견되며, 초기 생활과 겨울잠을 자는 동물에서 가장 높은 수준에 있습니다. 인간의 경우 갈색 지방 조직은 태어날 때 존재하고 나이가 들수록 감소합니다.[47]
미토콘드리아 지방산 합성
미토콘드리아 지방산 합성(mtFAS)II) 세포 호흡 및 미토콘드리아 생합성에 필수적입니다.[48] 또한 리소인지질이나 스핑고지질과 같은 생체 활성 지질 수준에 영향을 미치기 때문에 세포 내 신호전달의 매개체 역할을 하는 것으로 생각됩니다.[49]
Octanoyl-ACP (C8)는 리포산 생합성의 시작 기질을 형성하기도 하는 mtFAS II의 가장 중요한 최종 생성물로 간주됩니다.[50] 리포산은 피루브산 탈수소효소 복합체(PDC), α-케토글루타레이트 탈수소효소 복합체(OGDC), 분지 사슬 α-케토산 탈수소효소 복합체(BCKDC) 및 글리신 절단 시스템(GCS)과 같은 중요한 미토콘드리아 효소 복합체의 보조인자이기 때문에, mtFASII는 에너지 대사에 영향을 미칩니다.[51]
mtFAS의 기타 제품II는 미토콘드리아 번역, FeS 군집 생합성 및 산화적 인산화 복합체의 조립의 조절에 역할을 합니다.[50]
또한 mtFAS의 도움으로II 및 아실화된 ACP, 아세틸-CoA는 미토콘드리아에서의 소비를 조절합니다.[50]
칼슘 이온의 흡수, 저장 및 방출

세포 내 유리 칼슘의 농도는 일련의 반응을 조절할 수 있으며 세포 내 신호 전달에 중요합니다. 미토콘드리아는 세포의 칼슘 항상성에 기여하는 과정인 칼슘을 일시적으로 저장할 수 있습니다.[52] [53] 나중에 방출될 칼슘을 빠르게 흡수하는 능력은 칼슘을 위한 좋은 "세포질 완충제"로 만듭니다.[54][55][56] 소포체(ER)는 칼슘의 가장 중요한 저장 부위이며,[57] 칼슘과 관련하여 미토콘드리아와 ER 사이에는 중요한 상호 작용이 있습니다.[58] 칼슘은 내부 미토콘드리아 막에 있는 미토콘드리아 칼슘 유니포터에 의해 기질로 흡수됩니다.[59] 그것은 주로 미토콘드리아 막 전위에 의해 구동됩니다.[53] 이 칼슘의 세포 내부로의 방출은 나트륨-칼슘 교환 단백질 또는 "칼슘 유도-칼슘 방출" 경로를 통해 일어날 수 있습니다.[59] 이것은 막 전위의 큰 변화로 칼슘 스파이크 또는 칼슘 파동을 시작할 수 있습니다. 이들은 신경세포의 신경전달물질 방출, 내분비세포의 호르몬 방출 등의 과정을 조율할 수 있는 일련의 제2메신저 시스템 단백질을 활성화할 수 있습니다.[60]
미토콘드리아 기질로의 Ca 유입은 최근 막을 가로지르는 전기화학적 잠재력이 ψ δ이 지배하는 것에서 pH가 지배하는 것으로 일시적으로 "펄스"되어 산화 스트레스의 감소를 촉진함으로써 호흡 생체 에너지를 조절하는 메커니즘으로 연루되었습니다. 뉴런에서 세포질과 미토콘드리아 칼슘의 동반된 증가는 뉴런 활동을 미토콘드리아 에너지 대사와 동기화시키는 작용을 합니다. 미토콘드리아 기질 칼슘 수준은 수십 마이크로몰 수준에 도달할 수 있으며, 이는 크렙스 주기의 핵심 조절 효소 중 하나인 이소시트레이트 탈수소효소의 활성화에 필요합니다.[62]
세포증식조절
세포 증식과 미토콘드리아 사이의 관계가 조사되었습니다. 종양세포는 빠른 증식을 위해 지질, 단백질, 뉴클레오타이드 등 생체 활성 화합물을 합성하기 위해 충분한 ATP가 필요합니다.[63] 종양 세포에서 ATP의 대부분은 산화적 인산화 경로(OxPhos)를 통해 생성됩니다.[64] OxPhos에 대한 간섭은 미토콘드리아가 세포 증식에 역할을 한다는 것을 암시하는 세포 주기 정지를 일으킵니다.[64] 미토콘드리아 ATP 생산은 또한 세포 부피, 용질 농도 및 세포 구조의 조절을 포함한 세포의 기본 기능 외에도 세포 분열 및 감염 분화에 필수적입니다.[66][67][68] ATP 수준은 세포 주기의 여러 단계에서 차이가 있어 ATP의 풍부함과 세포가 새로운 세포 주기에 진입하는 능력 사이에 관계가 있음을 시사합니다.[69] 세포의 기본 기능에서 ATP의 역할은 세포 주기를 미토콘드리아 유래 ATP의 가용성 변화에 민감하게 만듭니다.[69] 세포 주기의 여러 단계에서 ATP 수준의 변화는 미토콘드리아가 세포 주기 조절에 중요한 역할을 한다는 가설을 뒷받침합니다.[69] 미토콘드리아와 세포 주기 조절 사이의 구체적인 메커니즘은 잘 알려져 있지 않지만, 연구에 따르면 에너지가 낮은 세포 주기 체크포인트는 또 다른 세포 분열을 일으키기 전에 에너지 능력을 모니터링합니다.[10]
부가기능
미토콘드리아는 다음과 같은 많은 다른 대사 작용에서 중심적인 역할을 합니다.
- 미토콘드리아 활성산소종을[70] 통한 신호전달
- 막전위[19] 조절
- 세포사멸 프로그램 세포사멸[71]
- 칼슘 신호전달(칼슘 유발 세포자멸사 포함)[72]
- 세포대사의[10] 조절
- 특정 헴 합성 반응[73] (참고: 포르피린)
- 스테로이드 합성[54]
- 호르몬 신호[74] 전달 – 미토콘드리아는 부분적으로 미토콘드리아 에스트로겐 수용체(mtER)의 작용에 의해 호르몬에 민감하고 반응합니다. 이 수용체는 뇌와[75] 심장을[76] 포함한 다양한 조직과 세포 유형에서 발견되었습니다.
- 면역신호전달[77]
- 신경 미토콘드리아는 또한 특화된 체세포 접합을 통해 미세아교세포를 향해 신경 상태를 보고함으로써 세포 품질 관리에 기여합니다.[78]
- 발달 중인 뉴런의 미토콘드리아는 뇌 발달의 적절한 조절에 필수적인 세포간 신호전달에 기여합니다.[79]
일부 미토콘드리아 기능은 특정 유형의 세포에서만 수행됩니다. 예를 들어 간세포의 미토콘드리아에는 단백질 대사의 노폐물인 암모니아를 해독할 수 있는 효소가 들어 있습니다. 이러한 기능을 조절하는 유전자의 돌연변이는 미토콘드리아 질환을 일으킬 수 있습니다.
미토콘드리아 단백질(미토콘드리아 DNA에서 전사된 단백질)은 조직과 종에 따라 다릅니다. 인간의 경우 심장 미토콘드리아에서 615종의 단백질이 확인된 [80]반면 쥐의 경우 940종의 단백질이 확인되었습니다.[81] 미토콘드리아 프로테옴은 동적으로 조절되는 것으로 생각됩니다.[82]
조직 및 분배

미토콘드리아(또는 관련 구조)는 모든 진핵생물(Oxymonad Monocomonoides 제외)에서 발견됩니다.[6] 일반적으로 콩과 같은 구조로 묘사되지만 대부분의 세포에서 매우 역동적인 네트워크를 형성하여 끊임없이 핵분열과 핵융합을 겪습니다. 주어진 세포의 모든 미토콘드리아의 집단이 콘드리옴을 구성합니다.[83] 미토콘드리아는 세포 종류에 따라 그 수와 위치가 다릅니다. 단일 미토콘드리아는 종종 단세포 생물에서 발견되는 반면, 인간의 간세포는 세포당 약 1000-2000개의 미토콘드리아를 가지고 있어 세포 부피의 1/5을 차지합니다.[18] 그렇지 않으면 유사한 세포의 미토콘드리아 함량은 크기와 막 전위가 상당히 다를 수 있으며,[84] 세포 분열에서 불균일한 분할을 포함한 출처에서 차이가 발생하여 ATP 수준 및 다운스트림 세포 과정의 외인적 차이로 이어집니다.[85] 미토콘드리아는 근육의 근섬유 사이에 자리잡거나 정자 편모 주위를 감싼 채 발견될 수 있습니다.[18] 종종, 그들은 세포골격과 함께 세포 내부에 복잡한 3D 분기 네트워크를 형성합니다. 세포골격과의 연관성은 기능에도 영향을 미칠 수 있는 미토콘드리아 모양을 결정합니다.[86] 미토콘드리아 네트워크의 다른 구조는 인구에게 다양한 물리적, 화학적, 신호 전달 이점 또는 단점을 제공할 수 있습니다.[87] 세포 내 미토콘드리아는 항상 미세소관을 따라 분포하며 이러한 소기관의 분포는 소포체와도 상관관계가 있습니다.[88] 최근의 증거는 세포골격의 구성 요소 중 하나인 비멘틴이 세포골격과의 연관성에도 중요하다는 것을 시사합니다.[89]
미토콘드리아 관련 ER 막(MAM)
미토콘드리아 관련 ER 막(MAM)은 세포 생리학 및 항상성에서 중요한 역할을 하는 것으로 점점 더 인정받고 있는 또 다른 구조적 요소입니다. 한때 세포 분획 기술의 기술적인 장애물로 여겨졌던 미토콘드리아 분획에 변함없이 나타난 ER 소포 오염물로 추정되는 물질은 미토콘드리아와 ER 사이의 계면인 MAM에서 유래된 막 구조로 다시 확인되었습니다.[90] 이 두 소기관 사이의 물리적 결합은 이전에 전자 현미경 사진에서 관찰되었으며 최근에는 형광 현미경으로 조사되었습니다.[90] 이러한 연구는 미토콘드리아 외막의 최대 20%를 구성할 수 있는 MAM에서 ER과 미토콘드리아가 단지 10-25 nm만큼 분리되고 단백질 테더링 복합체에 의해 함께 유지된다고 추정합니다.[90][28][91]
세포내 분획으로부터 정제된 MAM은 Ca2+ 신호전달과 관련된 채널 외에 인지질 교환에 관여하는 효소가 풍부합니다.[90][91] 세포 지질 저장 및 신호 전달의 조절에서 MAM의 중요한 역할에 대한 이러한 힌트가 입증되었으며, 아래에서 논의된 바와 같이 미토콘드리아 관련 세포 현상에 대한 중요한 의미가 있습니다. MAM은 내재적 세포자멸사 및 칼슘 신호 전달과 같은 생리학적 과정의 기초가 되는 기계적 기초에 대한 통찰력을 제공했을 뿐만 아니라 미토콘드리아에 대한 보다 정제된 관점을 선호합니다. 종종 고대 내공생 사건을 통해 세포 대사를 위해 납치된 정적이고 고립된 '강국'으로 간주되지만 MAM의 진화는 내막 시스템에 대한 친밀한 물리적 및 기능적 결합과 함께 미토콘드리아가 전반적인 세포 생리학에 통합된 정도를 강조합니다.
인지질전달
MAM은 ER 면의 포스파티딜세린 합성효소 및 미토콘드리아 면의 포스파티딜세린 탈탄산효소와 같은 지질 생합성에 관여하는 효소가 풍부합니다.[92][93] 미토콘드리아는 끊임없이 핵분열과 핵융합을 겪고 있는 동적 소기관이기 때문에 막 무결성을 위해 인지질의 지속적이고 잘 조절된 공급이 필요합니다.[94][95] 그러나 미토콘드리아는 그들이 합성을 완료하는 인지질의 목적지일 뿐만 아니라, 오히려 이 소기관은 인지질 생합성 경로, 세라마이드 및 콜레스테롤 대사, 글리코스핑고지질 동화작용의 중간체 및 생성물의 소기관간 밀매에도 역할을 합니다.[93][95]
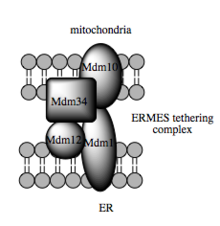
이러한 밀매 능력은 소기관 사이의 지질 중간체 전달을 촉진하는 것으로 나타난 MAM에 달려 있습니다.[92] 지질 전달의 표준 수포 메커니즘과 대조적으로, 증거는 MAM에서 ER 및 미토콘드리아 막의 물리적 근접성이 반대 이중층 사이에서 지질 뒤집기를 허용한다는 것을 나타냅니다.[95] 이 특이하고 겉보기에는 에너지적으로 불리한 메커니즘에도 불구하고 이러한 수송에는 ATP가 필요하지 않습니다.[95] 대신 효모에서 ER-미토콘드리아 조우 구조 또는 ERMES라고 하는 다중 단백질 테더링 구조에 의존하는 것으로 나타났지만, 이 구조가 지질 전달을 직접 매개하는지 또는 지질 뒤집기를 위한 에너지 장벽을 낮추기 위해 막을 충분히 근접하게 유지하는 데 필요한지는 불분명합니다.[95][96]
MAM은 세포내 지질 밀매에서의 역할 외에도 분비 경로의 일부일 수도 있습니다. 특히, MAM은 매우 저밀도 지단백, 즉 VLDL, 조립 및 분비로 이어지는 경로에서 거친 ER과 골지 사이의 중간 목적지인 것으로 보입니다.[93][97] 따라서 MAM은 지질 대사에서 중요한 대사 및 인신매매 허브 역할을 합니다.
칼슘신호전달
부분적으로 미토콘드리아 외부 막에 국한된 Ca2+ 채널의 낮은 친화도가 세포 내 Ca2+ 플럭스의 변화에 대한 이 소기관의 반응성과 모순되는 것으로 보이기 때문에 칼슘 신호 전달에서 ER의 중요한 역할이 널리 받아들여지기 전에 인정되었습니다.[90][57] 그러나 MAM의 존재는 이러한 명백한 모순을 해결합니다: 두 소기관 사이의 긴밀한 물리적 연관성은 ER에서 미토콘드리아로 효율적인 Ca2+ 전달을 촉진하는 접촉점에서2+ Ca 마이크로 도메인을 생성합니다.[90] 전송은 표준 ER 막 Ca2+ 채널인 IP3R의 자발적인 군집화 및 활성화에 의해 생성된 소위 "Ca2+ 퍼프"에 반응하여 발생합니다.[90][28]
특히, 이러한 퍼프의 운명은, 고립된 영역에 국한되어 있는지, 아니면 세포 전체의 전파를 위해 Ca파에2+ 통합되어 있는지에 따라 크게 결정됩니다. ER에 의한 Ca의2+ 재흡수는 (방출과 함께) 퍼프의 강도를 조절하여 높은 Ca2+ 노출로부터 미토콘드리아를 어느 정도 절연시키지만, MAM은 종종 세포질로 방출된 자유 이온이 유입될 수 있는 싱크 역할을 함으로써 Ca2+ 퍼프를 본질적으로 완충하는 방화벽 역할을 합니다.[90][98][99] 이 Ca2+ 터널링은 최근 ER 막의 IP3R 클러스터에 물리적으로 묶여 있고 MAM이 풍부한 것으로 나타난 저친화성 Ca2+ 수용체 VDAC1을 통해 발생합니다.[90][28][100] 산화적 인산화 과정에서 발생하는 전기화학적 구배는 미토콘드리아가 Ca2+ 싱크 역할을 할 수 있게 해주며, 이는 양이온의 터널링을 엑서곤성 과정으로 만듭니다.[100] 정상적이고 가벼운 칼슘이 세포질에서 미토콘드리아 기질로 유입되면 양성자를 펌핑하여 교정되는 일시적인 탈분극이 발생합니다.
그러나2+ Ca의 전송은 단방향이 아니라 양방향 거리입니다.[57] Ca2+ 펌프 SERCA와 ER 막에 존재하는 채널 IP3R의 특성은 MAM 기능에 의해 조정된 피드백 조절을 촉진합니다. 특히, MAM에 의한 Ca의2+ 클리어런스는 Ca가2+ IP3R 활성을 2상 방식으로 변경하기 때문에 Ca2+ 신호의 시공간적 패턴화를 가능하게 합니다.[90] SERCA도 마찬가지로 미토콘드리아 피드백의 영향을 받습니다: MAM에 의한 Ca의2+ 흡수는 ATP 생성을 자극하여 SERCA가 MAM에서 지속적인 Ca2+ 유출을 위해 Ca로2+ ER을 재장전할 수 있는 에너지를 제공합니다.[98][100] 따라서 MAM은 Ca2+ 퍼프를 위한 수동적인 버퍼가 아니라 ER 역학에 영향을 미치는 피드백 루프를 통해 추가적인 Ca2+ 신호를 변조하는 데 도움이 됩니다.
Ca2+ 흡수의 특정 창만이 항상성에서 미토콘드리아, 결과적으로 세포를 유지하기 때문에 MAM에서 Ca의2+ ER 방출을 조절하는 것은 특히 중요합니다. 시트르산 회로를 통한 플럭스에 중요한 탈수소효소를 활성화함으로써 대사를 자극하기 위해서는 충분한 소기관 내 Ca2+ 신호전달이 필요합니다.[101][102] 그러나 일단 미토콘드리아에서2+ Ca 신호전달이 특정 역치를 통과하면 대사에 필요한 미토콘드리아 막 전위를 붕괴시킴으로써 부분적으로 세포자멸사의 고유 경로를 자극합니다.[90] 프로-아포토틱 및 항-아포토틱 인자의 역할을 조사한 연구는 이 모델을 뒷받침합니다. 예를 들어, 항-아포토틱 인자 Bcl-2는 IP3R과 상호작용하여 ER의 Ca2+ 충전을 감소시켜 MAM에서 유출을 감소시키고 미토콘드리아 막 잠재적인 세포자멸 후 자극의 붕괴를 방지하는 것으로 나타났습니다.[90] Ca2+ 신호 전달의 이러한 미세한 조절의 필요성을 고려할 때 조절되지 않은 미토콘드리아 Ca가2+ 여러 신경 퇴행성 질환과 관련이 있는 반면 종양 억제제 목록에는 MAM이 풍부한 몇 가지가 포함되어 있다는 것은 놀라운 일이 아닙니다.[100]
테더링의 분자적 기초
미토콘드리아 막과 ER 막 사이의 테더 식별의 최근 발전은 관련된 분자 요소의 스캐폴딩 기능이 다른 비구조적 기능에 비해 부차적임을 시사합니다. 효모에서 상호작용하는 ER- 및 미토콘드리아 상주 막 단백질의 다중단백질 복합체인 ERMES는 MAM에서 지질 전달에 필요하며 이 원리를 예시합니다. 예를 들어, 그 구성요소 중 하나는 막횡단 베타-배럴 단백질을 지질 이중층에 삽입하는 데 필요한 단백질 복합체의 구성요소이기도 합니다.[95] 그러나 포유류 세포에서는 아직 ERMES 복합체의 상동체가 확인되지 않았습니다. 스캐폴딩에 관련된 다른 단백질들도 마찬가지로 MAM에서 구조적 테더링과 독립적인 기능을 갖습니다. 예를 들어, ER-레지던트 및 미토콘드리아-레지던트 미토푸신은 세포간 접촉 부위의 수를 조절하는 헤테로컴플렉스를 형성합니다. 비록 미토푸신이 개별 미토콘드리아 사이의 핵분열과 핵융합 사건에서의 역할에 대해 처음으로 확인되었습니다.[90] 포도당 관련 단백질 75(grp75)는 또 다른 이중 기능 단백질입니다. grp75의 매트릭스 풀 이외에, 부분은 MAM에서 효율적인 Ca2+ 전송을 위해 미토콘드리아 및 ER Ca2+ 채널 VDAC 및 IP3R을 물리적으로 연결하는 샤페론 역할을 합니다.[90][28] 또 다른 잠재적 테더는 ER-레지던트 IP3R의 안정화가 대사 스트레스 반응 동안 MAM에서의 통신을 보존할 수 있는 비-오피오이드 수용체인 시그마-1R입니다.[103][104]
관점
MAM은 ER과 미토콘드리아 생리학의 통합을 가능하게 하는 세포의 중요한 신호, 대사 및 인신매매 허브입니다. 이러한 소기관 사이의 결합은 단순히 구조적일 뿐만 아니라 기능적일 뿐만 아니라 전반적인 세포 생리학 및 항상성에 중요합니다. 따라서 MAM은 세포에 의해 대사 능력에 적합한 정적이고 고립된 단위로서 이 소기관의 전통적인 관점에서 벗어난 미토콘드리아에 대한 관점을 제공합니다.[105] 대신 이 미토콘드리아-ER 인터페이스는 내공생 이벤트의 산물인 미토콘드리아를 다양한 세포 과정으로 통합하는 것을 강조합니다. 최근에는 뉴런의 미토콘드리아와 MAM-s가 특수한 세포간 통신 부위(소위 체세포 접합부)에 고정되어 있다는 것도 밝혀졌습니다. 미세아교세포 과정은 이러한 부위에서 신경 기능을 감시하고 보호하며, MAM-s는 이러한 유형의 세포 품질 관리에서 중요한 역할을 하는 것으로 추정됩니다.[78]
기원과 진화
미토콘드리아의 기원에 대해서는 내공생과 자가공생이라는 두 가지 가설이 있습니다. 내공생 가설은 미토콘드리아가 원래 원핵세포로서 진핵세포에는 불가능한 산화 메커니즘을 구현할 수 있다는 것을 암시합니다; 그것들은 진핵세포 내부에 살고 있는 내공생체가 되었습니다.[21][106][107][108] 자가 가설에서 미토콘드리아는 원핵생물과 분열할 때 진핵세포의 핵에서 DNA의 일부를 쪼개서 탄생했는데, 이 DNA 부분은 단백질이 교차할 수 없는 막으로 둘러싸여 있었을 것입니다. 미토콘드리아는 세균과 공통적으로 많은 특징을 가지고 있기 때문에 두 가지 설명 중에서 내공생 가설이 더 널리 받아들여지고 있습니다.[108][109]
미토콘드리아는 DNA를 포함하고 있는데, DNA는 보통 원형 염색체 하나의 여러 사본으로 구성되어 있습니다. 이 미토콘드리아 염색체에는 호흡 사슬과 같은 산화 환원 단백질 유전자가 포함되어 있습니다. CoRR 가설은 이 코로케이션이 산화환원 조절에 필요하다는 것을 제안합니다. 미토콘드리아 유전체는 리보솜의 일부 RNA와 mRNA를 단백질로 번역하는 데 필요한 22개의 tRNA를 암호화합니다. 원형 구조는 원핵생물에서도 발견됩니다. 원시 미토콘드리아는 아마도 리케차와 밀접한 관련이 있었을 것입니다.[110][111] 그러나 미토콘드리아 조상과 알파프로테오박테리아의 정확한 관계와 미토콘드리아가 같은 시기에 형성되었는지 또는 핵 이후에 형성되었는지에 대해서는 여전히 논란의 여지가 있습니다.[112] 예를 들어, SAR11 박테리아 분기군은 미토콘드리아와 비교적 최근의 공통 조상을 공유한다고 제안된 반면,[113] 계통발생학적 분석은 미토콘드리아가 알파프로테오박테리아와 밀접한 관련이 있거나 알파프로테오박테리아의 구성체인 Pseudomonadota 계통에서 진화했음을 나타냅니다.[114][115] 일부 논문은 미토콘드리아를 알파프로테오박테리아의 자매로 설명하고, 함께 자매를 해양 프로테오1 그룹을 형성하고, 함께 Magnetocococcidae의 자매를 형성합니다.[116][117][118][119]
| 프로테오박테리아 |
| ||||||||||||||||||||||||
미토콘드리아 DNA에 의해 암호화된 리보솜은 크기와 구조가 박테리아의 리보솜과 비슷합니다.[120] 그들은 핵 DNA에 의해 코딩되는 80S 세포질 리보솜이 아니라 박테리아 70S 리보솜과 매우 유사합니다.
미토콘드리아와 숙주 세포의 내공생 관계는 린 마굴리스에 의해 대중화되었습니다.[121] 내공생 가설은 미토콘드리아가 다른 세포에 의한 세포내이입에서 어떻게든 살아남은 호기성 박테리아의 후손으로 세포질에 통합되었음을 시사합니다. 해당과정과 발효에 의존했던 숙주 세포에서 호흡을 수행하는 이 박테리아의 능력은 상당한 진화적 이점을 제공했을 것입니다. 이 공생 관계는 아마 17억에서 20억 년 전에 발전했을 것입니다.[122][123]

일부 단세포 진핵생물 그룹은 미토콘드리아 또는 파생된 구조만을 가지고 있습니다. 미생물, 메타모나드 및 아르카메바입니다.[124] 이 그룹들은 rRNA 정보를 이용해 구축된 계통수에서 가장 원시적인 진핵생물로 나타나는데, 이는 미토콘드리아의 기원 이전에 나타났음을 시사한 바 있습니다. 그러나, 이것은 현재 긴 가지의 매력을 지닌 유물로 알려져 있습니다. 그들은 파생된 그룹이며 미토콘드리아(예: 미토솜 및 하이드로게노솜)에서 유래된 유전자 또는 소기관을 보유합니다.[5] 일부 loricifera(예: Spinoloricus)[125][126] 및 myxozoa(예: Henneguya zschokkei)에서 발견되는 수소체, 미토솜 및 관련 소기관은 함께 MRO, 미토콘드리아 관련 소기관으로 분류됩니다.[8][127]
모노세르코모노이드는 미토콘드리아를 완전히 잃은 것처럼 보이고 적어도 미토콘드리아 기능 중 일부는 현재 세포질 단백질에 의해 수행되는 것으로 보입니다.[6]
미토콘드리아 유전학

미토콘드리아는 그들 자신의 게놈을 포함합니다. 인간 미토콘드리아 유전체는 약 16킬로베이스의 원형 이중가닥 DNA 분자입니다.[128] 호흡기 복합체 I, III, IV 및 V의 소단위체에 대한 13개, 미토콘드리아 tRNA에 대한 22개(20개 표준 아미노산에 대해 추가 유전자, 류신 및 세린에 대해 추가 유전자), rRNA에 대한 2개(12S 및 16S rRNA)의 37개 유전자를 암호화합니다.[128] 하나의 미토콘드리아는 2~10개의 DNA 사본을 포함할 수 있습니다.[129] 두 개의 미토콘드리아 DNA(mtDNA) 가닥 중 하나는 더 무거운 뉴클레오티드 아데닌과 구아닌의 비율이 불균형적으로 더 높으며, 이것을 헤비 스트랜드(또는 H 스트랜드)라고 하고, 다른 가닥을 라이트 스트랜드(또는 L 스트랜드)라고 합니다. 무게 차이로 인해 두 가닥이 원심분리에 의해 분리될 수 있습니다. mtDNA는 NCR(Non-coding region)로 알려진 하나의 긴 비코딩 스트레치를 가지고 있으며, 여기에는 L 가닥에 국한된 H 가닥(OriH)의 복제 기점인 RNA 전사를 위한 헤비 스트랜드 프로모터(HSP) 및 라이트 스트랜드 프로모터(LSP)가 포함되어 있습니다. 3개의 보존된 시퀀스 박스(CSBs 1-3) 및 종료 관련 시퀀스(TAS). L 가닥(OriL)의 복제 기원은 tRNA를 코딩하는 유전자 군집 내에 위치한 OriH의 11,000 bp 다운스트림 H 가닥에 국한되어 있습니다.[130]
원핵생물에서와 마찬가지로 코딩 DNA의 비율이 매우 높고 반복이 없습니다. 미토콘드리아 유전자는 다중유전자 전사체로 전사되며, 이는 절단되고 폴리아데닐화되어 성숙한 mRNA를 생성합니다. 미토콘드리아 기능에 필요한 대부분의 단백질은 세포핵의 유전자에 의해 암호화되고 해당 단백질은 미토콘드리아로 들여옵니다.[131] 핵과 미토콘드리아 유전체에 의해 암호화된 유전자의 정확한 수는 종에 따라 다릅니다. 대부분의 미토콘드리아 유전체는 원형입니다.[132] 일반적으로, 미토콘드리아 DNA는 인간 미토콘드리아 게놈의 경우와 마찬가지로 인트론이 없지만,[131] 딕티오스텔리움 디스코이데움을 [135]포함한 효모[134] 및 원생생물과 [133]같은 일부 진핵생물 미토콘드리아 DNA에서 인트론이 관찰되었습니다.[136] 단백질 코딩 영역 사이에 tRNA가 존재합니다. 미토콘드리아 tRNA 유전자는 핵 tRNA와 염기서열이 다르지만 염기서열 유사성이 높은 핵염색체에서 미토콘드리아 tRNA의 생김새가 발견됐습니다.[137]
미토콘드리아 유전체는 일반적으로 동물에서 약 16kb 길이의 단일 원형 염색체로 37개의 유전자를 가지고 있습니다. 유전자는 고도로 보존되어 있지만 위치에 따라 다를 수 있습니다. 흥미롭게도, 이 패턴은 사람의 몸에 있는 이(Pediculus humanus)에서는 발견되지 않습니다. 대신 이 미토콘드리아 유전체는 길이가 3~4kb이고 1~3개의 유전자를 가진 18개의 미니 원형 염색체에 배열되어 있습니다.[138] 이 패턴은 다른 빨아들이는 이에서도 발견되지만 씹는 이에서는 발견되지 않습니다. 재조합은 미니 염색체 사이에서 발생하는 것으로 나타났습니다.
인간 집단 유전학 연구
미토콘드리아 DNA에 유전적 재조합이 거의 없기 때문에 집단 유전학과 진화 생물학을 연구하는 데 유용한 정보원이 됩니다.[139] 모든 미토콘드리아 DNA는 단일 단위, 즉 일배체형으로 유전되기 때문에 서로 다른 개체의 미토콘드리아 DNA 사이의 관계를 유전자 트리로 나타낼 수 있습니다. 이러한 유전자 나무의 패턴은 개체군의 진화 역사를 추론하는 데 사용될 수 있습니다. 이것의 고전적인 예는 분자 시계를 사용하여 미토콘드리아 이브의 최근 날짜를 제공할 수 있는 인간 진화 유전학에 있습니다.[140][141] 이것은 종종 아프리카 밖으로 최근의 현대 인간 확장에 대한 강력한 지지로 해석됩니다.[142] 또 다른 인간의 예는 네안데르탈인의 뼈에서 미토콘드리아 DNA의 염기서열을 분석한 것입니다. 네안데르탈인의 미토콘드리아 DNA 염기서열과 살아있는 인간 사이의 진화적 거리가 상대적으로 크다는 것은 네안데르탈인과 현생 인류 사이에 이종교배가 부족하다는 증거로 해석돼 왔습니다.[143]
그러나 미토콘드리아 DNA는 집단 내 여성의 역사만을 반영합니다. 이는 Y 염색체의 비재조합 영역과 같은 부계 유전자 서열을 사용함으로써 부분적으로 극복될 수 있습니다.[142]
미토콘드리아 DNA에[144] 대한 분자 시계의 최근 측정은 인간과 유인원의 가장 최근의 공통 조상까지 거슬러 올라가는 매 7884년마다 1개의 돌연변이 값을 보고했는데, 이는 상염색체 DNA의 돌연변이 비율 추정치(세대당 염기당 10개−8)와 일치합니다.[145]
대체유전자코드
| 유기체 | 코돈 | 표준. | 미토콘드리아 |
|---|---|---|---|
| 포유류 | AGA, AGG | 아르기닌 | 정지 코돈 |
| 무척추동물 | AGA, AGG | 아르기닌 | 세린 |
| 곰팡이 | CUA | 류신 | 트레오닌 |
| 위 사항 모두. | AUA | 이소류신 | 메티오닌 |
| UGA | 정지 코돈 | 트립토판 |
표준 유전자 코드의 약간의 변화는 이전에 예측되었지만,[146] 1979년 인간 미토콘드리아 유전자를 연구하는 연구자들이 대체 코드를 사용했다고 결정하기 전까지는 발견되지 않았습니다.[147] 그럼에도 불구하고 대부분의 식물을 포함한 많은 다른 진핵생물의 미토콘드리아는 표준 코드를 사용합니다.[148] 그 이후로 다양한 대체 미토콘드리아 코드를 [148]포함하여 많은 약간의 변형이 발견되었습니다.[149] 또한 AUA, AUC 및 AUU 코돈은 모두 허용되는 시작 코돈입니다.
이러한 차이 중 일부는 미토콘드리아에서 흔히 볼 수 있는 RNA 편집 현상으로 인한 유전자 코드의 유사 변화로 간주해야 합니다. 고등 식물에서는 CGG가 아르기닌이 아닌 트립토판에 대해 암호화하는 것으로 생각되었지만, 처리된 RNA의 코돈은 UGG 코돈으로 밝혀졌으며, 이는 트립토판에 대한 표준 유전자 코드와 일치합니다.[150] 참고로, 절지동물 미토콘드리아 유전 코드는 문 내에서 병렬 진화를 거쳤으며, 일부 유기체는 AGG를 라이신으로 독특하게 번역했습니다.[151]
복제 및 상속
미토콘드리아는 미토콘드리아 핵분열에 의해 분열되는데, 이는 미토콘드리아 핵분열 과정이 숙주 진핵세포에 의해 엄격하게 조절되고 여러 다른 소기관들과의 통신 및 접촉을 포함하지만 박테리아에[152] 의해 수행되기도 하는 이진 핵분열의 한 형태입니다. 이러한 분열의 조절은 진핵생물마다 다릅니다. 많은 단세포 진핵생물에서 성장과 분열은 세포 주기와 연결되어 있습니다. 예를 들어, 하나의 미토콘드리아가 핵과 동시에 분열할 수 있습니다. 이 분열과 분리 과정은 각 딸 세포가 적어도 하나의 미토콘드리아를 받도록 엄격하게 통제되어야 합니다. 다른 진핵생물(예를 들어 포유류의 경우)에서 미토콘드리아는 DNA를 복제하고 세포 주기의 단계보다는 주로 세포의 에너지 요구에 따라 분열할 수 있습니다. 세포의 에너지 요구량이 높을 때 미토콘드리아는 성장하고 분열합니다. 에너지 사용량이 적으면 미토콘드리아가 파괴되거나 비활성화됩니다. 이러한 예에서 미토콘드리아는 세포질의 분열 동안 딸 세포에 무작위로 분포됩니다. 미토콘드리아 핵융합과 핵분열 사이의 균형인 미토콘드리아 역학은 여러 질병 상태와 관련된 병리학에서 중요한 요소입니다.[153]
미토콘드리아 이진 핵분열의 가설은 형광 현미경과 기존 투과 전자 현미경(TEM)에 의한 시각화에 의존해 왔습니다. 형광현미경(~200nm)의 해상도는 미토콘드리아 분열에 있는 이중 미토콘드리아 막과 같은 구조적 세부 사항을 구별하거나 심지어 여러 개가 가까이 있을 때 개별 미토콘드리아를 구별하기에는 불충분합니다. 기존의 TEM은 미토콘드리아 분열을 검증하는 데에도 몇 가지 기술적 한계가[which?] 있습니다. 최근 냉동 수화된 온전한 세포에서 미토콘드리아 분열을 시각화하기 위해 저온 전자 단층 촬영이 사용되었습니다. 미토콘드리아가 싹을 틔워 분열한다는 사실을 밝혀냈습니다.[154]
개인의 미토콘드리아 유전자는 드문 예외를 제외하고 어머니에게서만 유전됩니다.[155] 인간의 경우 난자 세포가 정자에 의해 수정될 때 미토콘드리아, 따라서 미토콘드리아 DNA는 대개 난자에서만 나옵니다. 정자의 미토콘드리아는 난자에 들어가지만 배아에 유전 정보를 제공하지는 않습니다.[156] 대신, 부계 미토콘드리아는 나중에 배아 안에서 파괴될 수 있도록 선택하기 위해 유비퀴틴으로 표시됩니다.[157] 난자 세포에는 미토콘드리아가 비교적 적게 포함되어 있지만, 이 미토콘드리아는 분열하여 성체의 세포를 채웁니다. 이 방식은 대부분의 동물을 포함한 대부분의 유기체에서 볼 수 있습니다. 그러나 일부 종의 미토콘드리아는 부계 유전되기도 합니다. 이것은 소나무와 주목은 아니지만 특정 침엽수 식물 사이에서 일반적인 것입니다.[158] Mytilids의 경우, 부계 유전은 종의 수컷 내에서만 발생합니다.[159][160][161] 그것은 인간에게 매우 낮은 수준에서 발생한다고 제시되었습니다.[162]
비록 단일 미토콘드리아가 2-10개의 DNA 사본을 포함할 수 있지만, 단일 부모 유전은 미토콘드리아의 다른 계통 사이에서 유전적 재조합의 기회를 거의 가져오지 않습니다.[129] 재조합이 일어나는 것은 다양성을 유지하는 것이 아니라 유전적 무결성을 유지합니다. 하지만 미토콘드리아 DNA에서 재조합의 증거를 보여주는 연구들이 있습니다. 포유류 세포에는 재조합에 필요한 효소가 존재하는 것이 분명합니다.[163] 게다가, 동물의 미토콘드리아가 재조합을 겪을 수 있다는 증거가 있습니다.[164] 재조합에 대한 간접적인 증거가 존재하지만, 이 데이터는 인간에게 더 많은 논란이 되고 있습니다.[165][166]
단부모 유전을 받고 있고 재조합이 거의 또는 전혀 없는 개체는 기능이 상실될 때까지 해로운 돌연변이의 축적인 Muller의 래칫의 대상이 될 것으로 예상할 수 있습니다. 미토콘드리아의 동물 개체군은 mtDNA 병목 현상으로 알려진 발달 과정을 통해 이러한 축적을 방지합니다. 병목현상은 유기체가 발달함에 따라 돌연변이 부하의 세포간 변동성을 증가시키기 위해 세포의 확률적 과정을 이용합니다. 따라서 돌연변이 mtDNA의 일부 비율을 갖는 단일 난자 세포는 서로 다른 돌연변이 부하를 갖는 배아를 생성합니다. 그런 다음 세포 수준 선택은 더 많은 돌연변이 mtDNA를 가진 세포를 제거하는 작용을 하여 세대 간 돌연변이 부하의 안정화 또는 감소로 이어질 수 있습니다. 병목 현상의 기초가 되는 메커니즘은 [167][168][169]최근의 수학적 및 실험적 전이 연구가 세포 분열에서 mtDNA의 무작위 분할과 세포 내 mtDNA 분자의 무작위 전환의 조합에 대한 증거를 제공하면서 논의되고 있습니다.[170]
DNA 복구
미토콘드리아는 세포핵에서 발생하는 것과 유사한 메커니즘으로 산화적 DNA 손상을 복구할 수 있습니다. mtDNA 복구에 사용되는 단백질은 핵 유전자에 의해 암호화되고 미토콘드리아로 전위됩니다. 포유류 미토콘드리아의 DNA 복구 경로에는 염기 절제 복구, 이중 가닥 파손 복구, 직접 역전 및 불일치 복구가 포함됩니다.[171][172] 또는 DNA 손상은 트랜스슬리온 합성에 의해 복구되지 않고 우회될 수 있습니다.
미토콘드리아의 여러 DNA 복구 과정 중에서 염기 절제 복구 경로가 가장 포괄적으로 연구되었습니다.[172] 염기 절제 수리는 손상된 DNA 염기의 인식 및 절제, 생성된 염기 부위의 제거, 말단 처리, 갭 메우기 및 결찰을 포함하는 효소 촉매 단계의 연속으로 수행됩니다. mtDNA에서 염기 절제 복구에 의해 복구되는 일반적인 손상은 구아닌의 산화에 의해 생성되는 8-옥소구아닌입니다.[173]
이중 가닥 파손은 포유류 mtDNA와[174] 식물 mtDNA 모두에서 상동 재조합 복구에 의해 복구될 수 있습니다.[175] mtDNA의 이중 가닥 파손은 또한 미세 상동성 매개 말단 접합에 의해 복구될 수 있습니다.[176] mtDNA에서 직접 역전 및 불일치 복구의 복구 과정에 대한 증거가 있지만 이러한 과정은 잘 특성화되어 있지 않습니다.[172]
미토콘드리아 DNA 부족
일부 유기체는 미토콘드리아 DNA를 완전히 잃었습니다. 이런 경우 미토콘드리아 DNA에 의해 암호화된 유전자가 소실되거나 핵으로 전이됩니다.[128] 와포자충은 DNA가 전혀 없는 미토콘드리아를 가지고 있는데, 아마도 모든 유전자가 손실되거나 전이되었기 때문일 것입니다.[177] Cryptosporidium에서 미토콘드리아는 변형된 ATP 생성 시스템을 가지고 있어 기생충이 시안화물, 아지드 및 아토바콘과 같은 많은 고전적인 미토콘드리아 억제제에 저항하도록 만듭니다.[177] 자체 DNA가 부족한 미토콘드리아는 아메보필라속의 해양 기생 와편모조류에서 발견되었습니다. 이 미생물인 A. cerati는 게놈이 부족한 기능성 미토콘드리아를 가지고 있습니다.[178] 관련 종에서 미토콘드리아 유전체는 여전히 3개의 유전자를 가지고 있지만, A. cerati에서는 단 하나의 미토콘드리아 유전자인 cytochrome coxidase I 유전자(cox1)만 발견되어 핵의 유전체로 이동했습니다.[179]
기능장애와 질병
미토콘드리아 질환
미토콘드리아의 손상과 그에 따른 기능 장애는 세포 대사에 영향을 미치기 때문에 인간의 다양한 질병에서 중요한 요소입니다. 미토콘드리아 장애는 종종 자폐증을 포함한 신경학적 장애로 나타납니다.[16] 또한 근병증, 당뇨병, 다발성 내분비병증 및 기타 다양한 전신 질환으로 나타날 수 있습니다.[180] mtDNA의 변이로 인한 질환으로는 Kearns-Sayre 증후군, MELAS 증후군, Leber 유전성 시신경병증 등이 있습니다.[181] 대부분의 경우, 접합체가 난자에서 미토콘드리아와 mtDNA를 파생하기 때문에 이러한 질병은 여성에 의해 자녀에게 전염됩니다. Kearns-Sayre 증후군, Pearson 증후군, 진행성 외부 안과 통증 등의 질환은 대규모 mtDNA 재배열에 의한 것으로 생각되며, MELAS 증후군, Leber 유전성 시신경병증, MERF 증후군 등의 다른 질환은 mtDNA의 점상 변이에 의한 것으로 생각됩니다.[180]
또한 약물 내성 암세포는 미토콘드리아의 수와 크기가 증가하여 미토콘드리아 생합성의 증가를 시사하는 것으로 보고되었습니다.[182] 네이처 나노테크놀로지의 2022년 연구는 암세포가 물리적 터널링 나노튜브를 통해 면역세포로부터 미토콘드리아를 탈취할 수 있다고 보고했습니다.[183]
다른 질병에서는 핵 유전자의 결함이 미토콘드리아 단백질의 기능 장애로 이어집니다. 프리드리히의 운동실조, 유전성 하반신 마비, 윌슨병 등이 이에 해당합니다.[184] 이러한 질병은 대부분의 다른 유전 질환에 적용되는 것처럼 우성 관계로 유전됩니다. 산화적 인산화 효소의 핵 돌연변이로 인해 코엔자임 Q10 결핍, 바르트 증후군 등 다양한 질환이 발생할 수 있습니다.[180] 환경적 영향은 유전적 소인과 상호 작용하여 미토콘드리아 질환을 유발할 수 있습니다. 예를 들어, 살충제 노출과 파킨슨병의 후기 발병 사이에 연관성이 있을 수 있습니다.[185][186] 미토콘드리아 기능 장애와 관련된 다른 병인으로 조현병, 양극성 장애, 치매, 알츠하이머병, 파킨슨병, 간질, 뇌전증 cardio혈관 질환, 만성 피로 증후군, 망막염, 당뇨병 등이 있습니다.
미토콘드리아 매개 산화 스트레스는 제2형 당뇨병 환자에서 심근병증의 역할을 합니다. 심장으로의 지방산 전달이 증가하면 심근세포에 의한 지방산 흡수가 증가하여 이들 세포에서 지방산 산화가 증가합니다. 이 과정은 미토콘드리아의 전자 수송 사슬이 이용할 수 있는 환원 당량을 증가시켜 궁극적으로 활성 산소종(ROS) 생산을 증가시킵니다. ROS는 분리 단백질(UCP)을 증가시키고, 미토콘드리아를 분리하는 아데닌 뉴클레오티드 트랜스로케이터(ANT)를 통한 양성자 누출을 강화합니다. 그런 다음 분리하면 미토콘드리아에 의한 산소 소비가 증가하여 지방산 산화가 증가합니다. 이로 인해 산소 소비량이 증가하더라도 미토콘드리아가 분리되기 때문에 ATP 합성이 비례적으로 증가하지 않는 등의 악순환이 발생합니다. ATP 가용성이 낮으면 결국 에너지 부족이 발생하여 심장 효율이 저하되고 수축 기능 장애가 발생합니다. 문제를 복잡하게 만드는 것은 손상된 근형질망 칼슘 방출과 감소된 미토콘드리아 재흡수가 근육 수축 동안 중요한 신호 이온의 최고 세포질 수준을 제한합니다. 감소된 미토콘드리아 내 칼슘 농도는 탈수소효소 활성화와 ATP 합성을 증가시킵니다. 그래서 지방산 산화로 ATP 합성이 저하될 뿐만 아니라 칼슘 신호전달이 잘 되지 않아 ATP 합성이 저해되어 당뇨병 환자의 심장에 문제가 생깁니다.[191]
미토콘드리아는 또한 고환 체세포 발달, 정자줄기세포 분화, 내강 산성화, 고환에서의 테스토스테론 생성 등의 과정을 조절합니다. 따라서 정자의 미토콘드리아 기능이상은 불임의 원인이 될 수 있습니다.
미토콘드리아 질환을 퇴치하기 위한 노력으로 미토콘드리아 대체 요법(MRT)이 개발되었습니다. 이런 형태의 체외수정은 기증자 미토콘드리아를 이용하는데, 이는 미토콘드리아 DNA의 돌연변이로 인한 질병의 전염을 피합니다.[193] 그러나 이 치료법은 여전히 연구 중이며 유전자 변형과 안전성 문제를 제기할 수 있습니다. 이러한 질병은 드물지만 극도로 쇠약하고 진행성인 질병일 수 있으므로 공공 정책에 복잡한 윤리적 질문을 제기합니다.[194]
노화와의 관계
활성 산소종을 형성하기 위해 호흡 사슬에 전달된 전자가 약간 누출될 수 있습니다. 이것은 미토콘드리아 DNA의 돌연변이율이 높은 미토콘드리아에서 상당한 산화 스트레스를 초래하는 것으로 생각되었습니다.[195] 노화와 산화 스트레스 사이의 가설적인 연관성은 새로운 것이 아니며 1956년에 제안되었으며,[196] 나중에 노화에 대한 미토콘드리아 자유 라디칼 이론으로 개선되었습니다.[197] 산화 스트레스가 미토콘드리아 DNA 돌연변이로 이어져 효소 이상과 추가 산화 스트레스로 이어질 수 있기 때문에 악순환이 발생하는 것으로 생각되었습니다.
노화 과정에서 미토콘드리아에 여러 변화가 일어날 수 있습니다.[198] 노인의 조직은 호흡 사슬의 단백질의 효소 활성 감소를 보여줍니다.[199] 그러나 돌연변이 mtDNA는 아주 오래된 세포의 약 0.2%에서만 발견할 수 있습니다.[200] 미토콘드리아 유전체의 큰 결손은 파킨슨병에서 높은 수준의 산화 스트레스와 신경세포 사멸로 이어진다는 가설이 세워졌습니다.[201] 미토콘드리아 기능 장애는 근위축성 측색 경화증에서도 발생하는 것으로 나타났습니다.[202][203]
미토콘드리아는 난소 기능의 중추적인 역할을 차지하기 때문에, 배아 소포에서 성숙한 난모세포로의 발달에 필요한 ATP를 제공함으로써 미토콘드리아 기능의 감소는 염증을 유발하여 조기 난소 부전과 가속화된 난소 노화를 초래할 수 있습니다. 결과적인 기능 장애는 난소 노화와 관련이 있을 뿐만 아니라 난소에서 난모세포-응집소 누화를 교란시키는 정량적(mtDNA copy number 및 mtDNA 결손과 같은), 정성적(돌연변이 및 가닥 파손과 같은), 그리고 산화적 손상(ROS로 인한 역기능 미토콘드리아와 같은)에 반영됩니다. 유전적 장애(예: 취약성 X)와 관련이 있으며 배아 선택을 방해할 수 있습니다.[204]
역사
미토콘드리아를 나타낼 가능성이 있는 세포 내 구조에 대한 최초의 관찰은 생리학자 알버트 폰 콜릭커에 의해 1857년에 발표되었습니다.[205][206] Richard Altmann은 1890년에 이들을 세포 소기관으로 확립하고 "바이오블라스트"라고 불렀습니다.[206][207] 1898년 칼 벤다는 그리스 μ ίτος인 미토스(mitos)와 χονδρίον인 콘드리온(chondrion)에서 "미토콘드리아(mitocondria)"라는 용어를 만들었습니다. 레오노르 미카엘리스는 1900년에 야누스 그린이 미토콘드리아의 상염색체로 사용될 수 있다는 것을 발견했습니다.[210] 1904년 프리드리히 메베스(Friedrich Meves)는 흰수박새인 님프해아 알바(Nymphaea alba)의 세포에서 식물에서 미토콘드리아를 처음으로 기록 [206][211]관찰했으며 1908년 클라우디우스 레고(Claudius Regaud)와 함께 단백질과 지질을 함유하고 있다고 제안했습니다. 벤자민 F. 킹즈베리는 1912년에 처음으로 세포 호흡과 관련이 있었지만 거의 독점적으로 형태학적 관찰에 기초했습니다.[212][206] 1913년 오토 하인리히 워버그는 호흡을 기니피그 간 추출물에서 얻은 입자와 연결시켜 "그라나"라고 불렀습니다.[213] 비슷한 입자 메커니즘을 가정했던 워버그와 하인리히 오토 빌랜드는 호흡의 화학적 성질에 대해 의견이 일치하지 않았습니다. 1925년 David Keilin이 사이토크롬을 발견하고서야 호흡 사슬이 기술되었습니다.[206]
1939년에는 다진 근육세포를 이용한 실험을 통해 산소 분자 하나를 이용한 세포 호흡이 4개의 아데노신 삼인산(ATP) 분자를 형성할 수 있음을 입증했고, 1941년에는 세포 대사에서 ATP의 인산 결합이 에너지의 한 형태라는 개념이 프리츠 알버트 립만에 의해 개발되었습니다. 그 다음 몇 년 동안, 미토콘드리아와의 연관성은 알려지지 않았지만 세포 호흡의 메커니즘이 더욱 정교해졌습니다.[206] 알버트 클로드의 조직 분획 도입으로 미토콘드리아를 다른 세포 분획으로부터 분리하고 이에 대한 생화학적 분석을 단독으로 진행할 수 있었습니다. 1946년, 그는 사이토크롬 산화효소와 호흡 사슬을 담당하는 다른 효소들이 미토콘드리아로 분리되었다는 결론을 내렸습니다. 1948년 유진 케네디와 앨버트 레닝거는 미토콘드리아가 진핵생물의 산화적 인산화 부위라는 사실을 발견했습니다. 시간이 지나면서 분획법이 더 발전해 분리된 미토콘드리아의 질이 좋아졌고, 세포 호흡의 다른 요소들이 미토콘드리아에서 일어나는 것으로 밝혀졌습니다.[206]
미토콘드리아를 시각화하는 선호되는 방법으로 야누스 그린 얼룩을 대체하는 최초의 고해상도 전자현미경 사진이 1952년에 나타났습니다.[206] 이것은 미토콘드리아가 막으로 둘러싸여 있다는 것을 확인하는 것을 포함하여 미토콘드리아의 구조를 더 자세히 분석하도록 이끌었습니다. 또한 미토콘드리아 내부에서 융기선으로 접혀 내부 챔버를 나누는 두 번째 막과 미토콘드리아의 크기와 모양이 세포마다 다르다는 것을 보여주었습니다.
"세포의 강자"라는 대중적인 용어는 1957년 필립 시케비츠에 의해 만들어졌습니다.[4][214]
1967년에 미토콘드리아에 리보솜이 포함되어 있다는 것이 밝혀졌습니다.[215] 1968년에는 1976년에 완성된 효모 미토콘드리아 DNA의 유전적, 물리적 지도와 함께 미토콘드리아 유전자를 매핑하는 방법이 개발되었습니다.[206]
참고 항목
참고문헌
- ^ "mitochondrion". Lexico UK English Dictionary. Oxford University Press. Archived from the original on January 2, 2020.
- ^ Campbell NA, Williamson B, Heyden RJ (2006). Biology: Exploring Life. Boston, Massachusetts: Pearson/Prentice Hall. ISBN 978-0132508827.
- ^ "Mighty Mitochondria and Neurodegenerative Diseases". Science in the News. 2012-02-01. Retrieved 2022-04-24.
- ^ a b Siekevitz P (1957). "Powerhouse of the cell". Scientific American. 197 (1): 131–140. Bibcode:1957SciAm.197a.131S. doi:10.1038/scientificamerican0757-131.
- ^ a b Henze K, Martin W (November 2003). "Evolutionary biology: essence of mitochondria". Nature. 426 (6963): 127–128. Bibcode:2003Natur.426..127H. doi:10.1038/426127a. PMID 14614484. S2CID 862398.
- ^ a b c d Karnkowska A, Vacek V, Zubáčová Z, Treitli SC, Petrželková R, Eme L, et al. (May 2016). "A Eukaryote without a Mitochondrial Organelle". Current Biology. 26 (10): 1274–1284. doi:10.1016/j.cub.2016.03.053. PMID 27185558.
- ^ Le Page M. "Animal that doesn't need oxygen to survive discovered New Scientist". New Scientist. Retrieved 2020-02-25.
- ^ a b Yahalomi D, Atkinson SD, Neuhof M, Chang ES, Philippe H, Cartwright P, et al. (March 2020). "A cnidarian parasite of salmon (Myxozoa: Henneguya) lacks a mitochondrial genome". Proceedings of the National Academy of Sciences of the United States of America. 117 (10): 5358–5363. Bibcode:2020PNAS..117.5358Y. doi:10.1073/pnas.1909907117. PMC 7071853. PMID 32094163.
- ^ Wiemerslage L, Lee D (March 2016). "Quantification of mitochondrial morphology in neurites of dopaminergic neurons using multiple parameters". Journal of Neuroscience Methods. 262: 56–65. doi:10.1016/j.jneumeth.2016.01.008. PMC 4775301. PMID 26777473.
- ^ a b c McBride HM, Neuspiel M, Wasiak S (July 2006). "Mitochondria: more than just a powerhouse". Current Biology. 16 (14): R551–R560. doi:10.1016/j.cub.2006.06.054. PMID 16860735. S2CID 16252290.
- ^ Valero T (2014). "Mitochondrial biogenesis: pharmacological approaches". Current Pharmaceutical Design. 20 (35): 5507–5509. doi:10.2174/138161282035140911142118. hdl:10454/13341. PMID 24606795.
Mitochondrial biogenesis is therefore defined as the process via which cells increase their individual mitochondrial mass [3]. ... Mitochondrial biogenesis occurs by growth and division of pre-existing organelles and is temporally coordinated with cell cycle events [1].
- ^ Sanchis-Gomar F, García-Giménez JL, Gómez-Cabrera MC, Pallardó FV (2014). "Mitochondrial biogenesis in health and disease. Molecular and therapeutic approaches". Current Pharmaceutical Design. 20 (35): 5619–5633. doi:10.2174/1381612820666140306095106. PMID 24606801.
Mitochondrial biogenesis (MB) is the essential mechanism by which cells control the number of mitochondria
- ^ Gardner A, Boles RG (2005). "Is a 'Mitochondrial Psychiatry' in the Future? A Review". Curr. Psychiatry Rev. 1 (3): 255–271. doi:10.2174/157340005774575064.
- ^ Lesnefsky EJ, Moghaddas S, Tandler B, Kerner J, Hoppel CL (June 2001). "Mitochondrial dysfunction in cardiac disease: ischemia – reperfusion, aging, and heart failure". Journal of Molecular and Cellular Cardiology. 33 (6): 1065–1089. doi:10.1006/jmcc.2001.1378. PMID 11444914.
- ^ Dorn GW, Vega RB, Kelly DP (October 2015). "Mitochondrial biogenesis and dynamics in the developing and diseased heart". Genes & Development. 29 (19): 1981–1991. doi:10.1101/gad.269894.115. PMC 4604339. PMID 26443844.
- ^ a b Griffiths KK, Levy RJ (2017). "Evidence of Mitochondrial Dysfunction in Autism: Biochemical Links, Genetic-Based Associations, and Non-Energy-Related Mechanisms". Oxidative Medicine and Cellular Longevity. 2017: 4314025. doi:10.1155/2017/4314025. PMC 5467355. PMID 28630658.
- ^ Ney PA (May 2011). "Normal and disordered reticulocyte maturation". Current Opinion in Hematology. 18 (3): 152–157. doi:10.1097/MOH.0b013e328345213e. PMC 3157046. PMID 21423015.
- ^ a b c d e f g h i j Alberts B, Johnson A, Lewis J, Raff M, Roberts K, Walter P (2005). Molecular Biology of the Cell. New York: Garland Publishing Inc. ISBN 978-0815341055.
- ^ a b c d e f g h i j k l Voet D, Voet JC, Pratt CW (2006). Fundamentals of Biochemistry (2nd ed.). John Wiley and Sons, Inc. pp. 547, 556. ISBN 978-0471214953.
- ^ Andersson SG, Karlberg O, Canbäck B, Kurland CG (January 2003). "On the origin of mitochondria: a genomics perspective". Philosophical Transactions of the Royal Society of London. Series B, Biological Sciences. 358 (1429): 165–77, discussion 177–9. doi:10.1098/rstb.2002.1193. PMC 1693097. PMID 12594925.
- ^ a b Gabaldón T (October 2021). "Origin and Early Evolution of the Eukaryotic Cell". Annual Review of Microbiology. 75 (1): 631–647. doi:10.1146/annurev-micro-090817-062213. PMID 34343017. S2CID 236916203.
- ^ "Mitochondrion – much more than an energy converter". British Society for Cell Biology. Retrieved 19 August 2013.
- ^ Blachly-Dyson E, Forte M (September 2001). "VDAC channels". IUBMB Life. 52 (3–5): 113–118. doi:10.1080/15216540152845902. PMID 11798022. S2CID 38314888.
- ^ Hoogenboom BW, Suda K, Engel A, Fotiadis D (July 2007). "The supramolecular assemblies of voltage-dependent anion channels in the native membrane". Journal of Molecular Biology. 370 (2): 246–255. doi:10.1016/j.jmb.2007.04.073. PMID 17524423.
- ^ Zeth K (June 2010). "Structure and evolution of mitochondrial outer membrane proteins of beta-barrel topology". Biochimica et Biophysica Acta (BBA) - Bioenergetics. 1797 (6–7): 1292–1299. doi:10.1016/j.bbabio.2010.04.019. PMID 20450883.
- ^ a b Herrmann JM, Neupert W (April 2000). "Protein transport into mitochondria" (PDF). Current Opinion in Microbiology. 3 (2): 210–214. doi:10.1016/S1369-5274(00)00077-1. PMID 10744987.
- ^ a b Chipuk JE, Bouchier-Hayes L, Green DR (August 2006). "Mitochondrial outer membrane permeabilization during apoptosis: the innocent bystander scenario". Cell Death and Differentiation. 13 (8): 1396–1402. doi:10.1038/sj.cdd.4401963. PMID 16710362.
- ^ a b c d e Hayashi T, Rizzuto R, Hajnoczky G, Su TP (February 2009). "MAM: more than just a housekeeper". Trends in Cell Biology. 19 (2): 81–88. doi:10.1016/j.tcb.2008.12.002. PMC 2750097. PMID 19144519.
- ^ Schenkel LC, Bakovic M (January 2014). "Formation and regulation of mitochondrial membranes". International Journal of Cell Biology. 2014: 709828. doi:10.1155/2014/709828. PMC 3918842. PMID 24578708.
- ^ McMillin JB, Dowhan W (December 2002). "Cardiolipin and apoptosis". Biochimica et Biophysica Acta (BBA) - Molecular and Cell Biology of Lipids. 1585 (2–3): 97–107. doi:10.1016/S1388-1981(02)00329-3. PMID 12531542.
- ^ Bautista JS, Falabella M, Flannery PJ, Hanna MG, Heales SJ, Pope SA, Pitceathly RD (December 2022). "Advances in methods to analyse cardiolipin and their clinical applications". Trends in Analytical Chemistry. 157: 116808. doi:10.1016/j.trac.2022.116808. PMC 7614147. PMID 36751553. S2CID 253211400.
- ^ Youle RJ, van der Bliek AM (August 2012). "Mitochondrial fission, fusion, and stress". Science. 337 (6098): 1062–1065. Bibcode:2012Sci...337.1062Y. doi:10.1126/science.1219855. PMC 4762028. PMID 22936770.
- ^ Cserép C, Pósfai B, Schwarcz AD, Dénes Á (2018). "Mitochondrial Ultrastructure Is Coupled to Synaptic Performance at Axonal Release Sites". eNeuro. 5 (1): ENEURO.0390–17.2018. doi:10.1523/ENEURO.0390-17.2018. PMC 5788698. PMID 29383328.
- ^ Mannella CA (2006). "Structure and dynamics of the mitochondrial inner membrane cristae". Biochimica et Biophysica Acta (BBA) - Molecular Cell Research. 1763 (5–6): 542–548. doi:10.1016/j.bbamcr.2006.04.006. PMID 16730811.
- ^ Bogenhagen DF (September 2012). "Mitochondrial DNA nucleoid structure". Biochimica et Biophysica Acta (BBA) - Gene Regulatory Mechanisms. 1819 (9–10): 914–920. doi:10.1016/j.bbagrm.2011.11.005. PMID 22142616.
- ^ Rich PR (December 2003). "The molecular machinery of Keilin's respiratory chain". Biochemical Society Transactions. 31 (Pt 6): 1095–1105. doi:10.1042/BST0311095. PMID 14641005.
- ^ Stoimenova M, Igamberdiev AU, Gupta KJ, Hill RD (July 2007). "Nitrite-driven anaerobic ATP synthesis in barley and rice root mitochondria". Planta. 226 (2): 465–474. Bibcode:2007Plant.226..465S. doi:10.1007/s00425-007-0496-0. PMID 17333252. S2CID 8963850.
- ^ Neupert W (1997). "Protein import into mitochondria". Annual Review of Biochemistry. 66: 863–917. doi:10.1146/annurev.biochem.66.1.863. PMID 9242927.
- ^ a b c d e f Stryer L (1995). "Citric acid cycle.". In: Biochemistry (Fourth ed.). New York: W.H. Freeman and Company. pp. 509–527, 569–579, 614–616, 638–641, 732–735, 739–748, 770–773. ISBN 0716720094.
- ^ King A, Selak MA, Gottlieb E (August 2006). "Succinate dehydrogenase and fumarate hydratase: linking mitochondrial dysfunction and cancer". Oncogene. 25 (34): 4675–4682. doi:10.1038/sj.onc.1209594. PMID 16892081. S2CID 26263513.
- ^ a b Voet, D; Voet, J. G. (2004) 생화학, 3판, 804쪽, 와일리. ISBN 047119350X
- ^ a b Atkins, P. de Paula, J. (2006) "물리 화학", 8th Ed.; pp. 225-229, Freeman: 뉴욕, 2006.
- ^ Huang H, Manton KG (May 2004). "The role of oxidative damage in mitochondria during aging: a review" (PDF). Frontiers in Bioscience. 9 (1–3): 1100–1117. doi:10.2741/1298. PMID 14977532. S2CID 2278219. Archived from the original (PDF) on 2019-03-03.
- ^ Mitchell P, Moyle J (January 1967). "Chemiosmotic hypothesis of oxidative phosphorylation". Nature. 213 (5072): 137–139. Bibcode:1967Natur.213..137M. doi:10.1038/213137a0. PMID 4291593. S2CID 4149605.
- ^ Mitchell P (June 1967). "Proton current flow in mitochondrial systems". Nature. 214 (5095): 1327–1328. Bibcode:1967Natur.214.1327M. doi:10.1038/2141327a0. PMID 6056845. S2CID 4160146.
- ^ Nobel Foundation. "Chemistry 1997". Retrieved 2007-12-16.
- ^ a b Mozo J, Emre Y, Bouillaud F, Ricquier D, Criscuolo F (November 2005). "Thermoregulation: what role for UCPs in mammals and birds?". Bioscience Reports. 25 (3–4): 227–249. doi:10.1007/s10540-005-2887-4. PMID 16283555. S2CID 164450.
- ^ Kastaniotis AJ, Autio KJ, Kerätär JM, Monteuuis G, Mäkelä AM, Nair RR, et al. (January 2017). "Mitochondrial fatty acid synthesis, fatty acids and mitochondrial physiology". Biochimica et Biophysica Acta (BBA) - Molecular and Cell Biology of Lipids. 1862 (1): 39–48. doi:10.1016/j.bbalip.2016.08.011. PMID 27553474.
- ^ Clay HB, Parl AK, Mitchell SL, Singh L, Bell LN, Murdock DG (March 2016). Peterson J (ed.). "Altering the Mitochondrial Fatty Acid Synthesis (mtFASII) Pathway Modulates Cellular Metabolic States and Bioactive Lipid Profiles as Revealed by Metabolomic Profiling". PLOS ONE. 11 (3): e0151171. Bibcode:2016PLoSO..1151171C. doi:10.1371/journal.pone.0151171. PMC 4786287. PMID 26963735.
- ^ a b c Nowinski SM, Van Vranken JG, Dove KK, Rutter J (October 2018). "Impact of Mitochondrial Fatty Acid Synthesis on Mitochondrial Biogenesis". Current Biology. 28 (20): R1212–R1219. doi:10.1016/j.cub.2018.08.022. PMC 6258005. PMID 30352195.
- ^ Wehbe Z, Behringer S, Alatibi K, Watkins D, Rosenblatt D, Spiekerkoetter U, Tucci S (November 2019). "The emerging role of the mitochondrial fatty-acid synthase (mtFASII) in the regulation of energy metabolism". Biochimica et Biophysica Acta (BBA) - Molecular and Cell Biology of Lipids. 1864 (11): 1629–1643. doi:10.1016/j.bbalip.2019.07.012. PMID 31376476. S2CID 199404906.
- ^ Santulli G, Xie W, Reiken SR, Marks AR (September 2015). "Mitochondrial calcium overload is a key determinant in heart failure". Proceedings of the National Academy of Sciences of the United States of America. 112 (36): 11389–11394. Bibcode:2015PNAS..11211389S. doi:10.1073/pnas.1513047112. PMC 4568687. PMID 26217001.
- ^ a b Siegel GJ, Agranoff BW, Fisher SK, Albers RW, Uhler MD, eds. (1999). Basic Neurochemistry (6 ed.). Lippincott Williams & Wilkins. ISBN 978-0397518203.
- ^ a b Rossier MF (August 2006). "T channels and steroid biosynthesis: in search of a link with mitochondria". Cell Calcium. 40 (2): 155–164. doi:10.1016/j.ceca.2006.04.020. PMID 16759697.
- ^ Brighton CT, Hunt RM (May 1974). "Mitochondrial calcium and its role in calcification. Histochemical localization of calcium in electron micrographs of the epiphyseal growth plate with K-pyroantimonate". Clinical Orthopaedics and Related Research. 100 (5): 406–416. doi:10.1097/00003086-197405000-00057. PMID 4134194.
- ^ Brighton CT, Hunt RM (July 1978). "The role of mitochondria in growth plate calcification as demonstrated in a rachitic model". The Journal of Bone and Joint Surgery. American Volume. 60 (5): 630–639. doi:10.2106/00004623-197860050-00007. PMID 681381.
- ^ a b c Santulli G, Marks AR (2015). "Essential Roles of Intracellular Calcium Release Channels in Muscle, Brain, Metabolism, and Aging". Current Molecular Pharmacology. 8 (2): 206–222. doi:10.2174/1874467208666150507105105. PMID 25966694.
- ^ Pizzo P, Pozzan T (October 2007). "Mitochondria-endoplasmic reticulum choreography: structure and signaling dynamics". Trends in Cell Biology. 17 (10): 511–517. doi:10.1016/j.tcb.2007.07.011. PMID 17851078.
- ^ a b Miller RJ (March 1998). "Mitochondria – the Kraken wakes!". Trends in Neurosciences. 21 (3): 95–97. doi:10.1016/S0166-2236(97)01206-X. PMID 9530913. S2CID 5193821.
- ^ Santulli G, Pagano G, Sardu C, Xie W, Reiken S, D'Ascia SL, et al. (May 2015). "Calcium release channel RyR2 regulates insulin release and glucose homeostasis". The Journal of Clinical Investigation. 125 (5): 1968–1978. doi:10.1172/JCI79273. PMC 4463204. PMID 25844899.
- ^ Schwarzländer M, Logan DC, Johnston IG, Jones NS, Meyer AJ, Fricker MD, Sweetlove LJ (March 2012). "Pulsing of membrane potential in individual mitochondria: a stress-induced mechanism to regulate respiratory bioenergetics in Arabidopsis". The Plant Cell. 24 (3): 1188–1201. doi:10.1105/tpc.112.096438. PMC 3336130. PMID 22395486.
- ^ Ivannikov MV, Macleod GT (June 2013). "Mitochondrial free Ca²⁺ levels and their effects on energy metabolism in Drosophila motor nerve terminals". Biophysical Journal. 104 (11): 2353–2361. Bibcode:2013BpJ...104.2353I. doi:10.1016/j.bpj.2013.03.064. PMC 3672877. PMID 23746507.
- ^ Weinberg F, Chandel NS (October 2009). "Mitochondrial metabolism and cancer". Annals of the New York Academy of Sciences. 1177 (1): 66–73. Bibcode:2009NYASA1177...66W. doi:10.1111/j.1749-6632.2009.05039.x. PMID 19845608. S2CID 29827252.
- ^ a b Moreno-Sánchez R, Rodríguez-Enríquez S, Marín-Hernández A, Saavedra E (March 2007). "Energy metabolism in tumor cells". The FEBS Journal. 274 (6): 1393–1418. doi:10.1111/j.1742-4658.2007.05686.x. PMID 17302740. S2CID 7748115.
- ^ Mistry JJ, Marlein CR, Moore JA, Hellmich C, Wojtowicz EE, Smith JG, et al. (December 2019). "ROS-mediated PI3K activation drives mitochondrial transfer from stromal cells to hematopoietic stem cells in response to infection". Proceedings of the National Academy of Sciences of the United States of America. 116 (49): 24610–24619. Bibcode:2019PNAS..11624610M. doi:10.1073/pnas.1913278116. PMC 6900710. PMID 31727843.
- ^ Pedersen PL (December 1994). "ATP synthase. The machine that makes ATP". Current Biology. 4 (12): 1138–1141. doi:10.1016/S0960-9822(00)00257-8. PMID 7704582. S2CID 10279742.
- ^ Pattappa G, Heywood HK, de Bruijn JD, Lee DA (October 2011). "The metabolism of human mesenchymal stem cells during proliferation and differentiation". Journal of Cellular Physiology. 226 (10): 2562–2570. doi:10.1002/jcp.22605. PMID 21792913. S2CID 22259833.
- ^ Agarwal B (June 2011). "A role for anions in ATP synthesis and its molecular mechanistic interpretation". Journal of Bioenergetics and Biomembranes. 43 (3): 299–310. doi:10.1007/s10863-011-9358-3. PMID 21647635. S2CID 29715383.
- ^ a b c Sweet S, Singh G (July 1999). "Changes in mitochondrial mass, membrane potential, and cellular adenosine triphosphate content during the cell cycle of human leukemic (HL-60) cells". Journal of Cellular Physiology. 180 (1): 91–96. doi:10.1002/(SICI)1097-4652(199907)180:1<91::AID-JCP10>3.0.CO;2-6. PMID 10362021. S2CID 25906279.
- ^ Li X, Fang P, Mai J, Choi ET, Wang H, Yang XF (February 2013). "Targeting mitochondrial reactive oxygen species as novel therapy for inflammatory diseases and cancers". Journal of Hematology & Oncology. 6 (19): 19. doi:10.1186/1756-8722-6-19. PMC 3599349. PMID 23442817.
- ^ Green DR (September 1998). "Apoptotic pathways: the roads to ruin". Cell. 94 (6): 695–698. doi:10.1016/S0092-8674(00)81728-6. PMID 9753316. S2CID 16654082.
- ^ Hajnóczky G, Csordás G, Das S, Garcia-Perez C, Saotome M, Sinha Roy S, Yi M (2006). "Mitochondrial calcium signalling and cell death: approaches for assessing the role of mitochondrial Ca2+ uptake in apoptosis". Cell Calcium. 40 (5–6): 553–560. doi:10.1016/j.ceca.2006.08.016. PMC 2692319. PMID 17074387.
- ^ Oh-hama T (August 1997). "Evolutionary consideration on 5-aminolevulinate synthase in nature". Origins of Life and Evolution of the Biosphere. 27 (4): 405–412. Bibcode:1997OLEB...27..405O. doi:10.1023/A:1006583601341. PMID 9249985. S2CID 13602877.
- ^ Klinge CM (December 2008). "Estrogenic control of mitochondrial function and biogenesis". Journal of Cellular Biochemistry. 105 (6): 1342–1351. doi:10.1002/jcb.21936. PMC 2593138. PMID 18846505.
- ^ Alvarez-Delgado C, Mendoza-Rodríguez CA, Picazo O, Cerbón M (August 2010). "Different expression of alpha and beta mitochondrial estrogen receptors in the aging rat brain: interaction with respiratory complex V". Experimental Gerontology. 45 (7–8): 580–585. doi:10.1016/j.exger.2010.01.015. PMID 20096765. S2CID 30841790.
- ^ Pavón N, Martínez-Abundis E, Hernández L, Gallardo-Pérez JC, Alvarez-Delgado C, Cerbón M, et al. (October 2012). "Sexual hormones: effects on cardiac and mitochondrial activity after ischemia-reperfusion in adult rats. Gender difference". The Journal of Steroid Biochemistry and Molecular Biology. 132 (1–2): 135–146. doi:10.1016/j.jsbmb.2012.05.003. PMID 22609314. S2CID 24794040.
- ^ Breda CN, Davanzo GG, Basso PJ, Saraiva Câmara NO, Moraes-Vieira PM (September 2019). "Mitochondria as central hub of the immune system". Redox Biology. 26: 101255. doi:10.1016/j.redox.2019.101255. PMC 6598836. PMID 31247505.
- ^ a b Cserép C, Pósfai B, Lénárt N, Fekete R, László ZI, Lele Z, et al. (January 2020). "Microglia monitor and protect neuronal function through specialized somatic purinergic junctions". Science. 367 (6477): 528–537. Bibcode:2020Sci...367..528C. doi:10.1126/science.aax6752. PMID 31831638. S2CID 209343260.
- ^ Cserép C, Schwarcz AD, Pósfai B, László ZI, Kellermayer A, Környei Z, et al. (September 2022). "Microglial control of neuronal development via somatic purinergic junctions". Cell Reports. 40 (12): 111369. doi:10.1016/j.celrep.2022.111369. PMC 9513806. PMID 36130488. S2CID 252416407.
- ^ Taylor SW, Fahy E, Zhang B, Glenn GM, Warnock DE, Wiley S, et al. (March 2003). "Characterization of the human heart mitochondrial proteome". Nature Biotechnology. 21 (3): 281–286. doi:10.1038/nbt793. PMID 12592411. S2CID 27329521.
- ^ Zhang J, Li X, Mueller M, Wang Y, Zong C, Deng N, et al. (April 2008). "Systematic characterization of the murine mitochondrial proteome using functionally validated cardiac mitochondria". Proteomics. 8 (8): 1564–1575. doi:10.1002/pmic.200700851. PMC 2799225. PMID 18348319.
- ^ Zhang J, Liem DA, Mueller M, Wang Y, Zong C, Deng N, et al. (June 2008). "Altered proteome biology of cardiac mitochondria under stress conditions". Journal of Proteome Research. 7 (6): 2204–2214. doi:10.1021/pr070371f. PMC 3805274. PMID 18484766.
- ^ Logan DC (June 2010). "Mitochondrial fusion, division and positioning in plants". Biochemical Society Transactions. 38 (3): 789–795. doi:10.1042/bst0380789. PMID 20491666.
- ^ das Neves RP, Jones NS, Andreu L, Gupta R, Enver T, Iborra FJ (December 2010). Weissman JS (ed.). "Connecting variability in global transcription rate to mitochondrial variability". PLOS Biology. 8 (12): e1000560. doi:10.1371/journal.pbio.1000560. PMC 3001896. PMID 21179497.
- ^ Johnston IG, Gaal B, Neves RP, Enver T, Iborra FJ, Jones NS (2012). Haugh JM (ed.). "Mitochondrial variability as a source of extrinsic cellular noise". PLOS Computational Biology. 8 (3): e1002416. arXiv:1107.4499. Bibcode:2012PLSCB...8E2416J. doi:10.1371/journal.pcbi.1002416. PMC 3297557. PMID 22412363.
- ^ Rappaport L, Oliviero P, Samuel JL (July 1998). "Cytoskeleton and mitochondrial morphology and function". Molecular and Cellular Biochemistry. 184 (1–2): 101–105. doi:10.1023/A:1006843113166. PMID 9746315. S2CID 28165195.
- ^ Hoitzing H, Johnston IG, Jones NS (June 2015). "What is the function of mitochondrial networks? A theoretical assessment of hypotheses and proposal for future research". BioEssays. 37 (6): 687–700. doi:10.1002/bies.201400188. PMC 4672710. PMID 25847815.
- ^ Soltys BJ, Gupta RS (1992). "Interrelationships of endoplasmic reticulum, mitochondria, intermediate filaments, and microtubules – a quadruple fluorescence labeling study". Biochemistry and Cell Biology. 70 (10–11): 1174–1186. doi:10.1139/o92-163. PMID 1363623.
- ^ Tang HL, Lung HL, Wu KC, Le AH, Tang HM, Fung MC (February 2008). "Vimentin supports mitochondrial morphology and organization". The Biochemical Journal. 410 (1): 141–146. doi:10.1042/BJ20071072. PMID 17983357.
- ^ a b c d e f g h i j k l m n Rizzuto R, Marchi S, Bonora M, Aguiari P, Bononi A, De Stefani D, et al. (November 2009). "Ca(2+) transfer from the ER to mitochondria: when, how and why". Biochimica et Biophysica Acta (BBA) - Bioenergetics. 1787 (11): 1342–1351. doi:10.1016/j.bbabio.2009.03.015. PMC 2730423. PMID 19341702.
- ^ a b de Brito OM, Scorrano L (August 2010). "An intimate liaison: spatial organization of the endoplasmic reticulum-mitochondria relationship". The EMBO Journal. 29 (16): 2715–2723. doi:10.1038/emboj.2010.177. PMC 2924651. PMID 20717141.
- ^ a b Vance JE, Shiao YJ (1996). "Intracellular trafficking of phospholipids: import of phosphatidylserine into mitochondria". Anticancer Research. 16 (3B): 1333–1339. PMID 8694499.
- ^ a b c Lebiedzinska M, Szabadkai G, Jones AW, Duszynski J, Wieckowski MR (October 2009). "Interactions between the endoplasmic reticulum, mitochondria, plasma membrane and other subcellular organelles". The International Journal of Biochemistry & Cell Biology. 41 (10): 1805–1816. doi:10.1016/j.biocel.2009.02.017. PMID 19703651.
- ^ Twig G, Elorza A, Molina AJ, Mohamed H, Wikstrom JD, Walzer G, et al. (January 2008). "Fission and selective fusion govern mitochondrial segregation and elimination by autophagy". The EMBO Journal. 27 (2): 433–446. doi:10.1038/sj.emboj.7601963. PMC 2234339. PMID 18200046.
- ^ a b c d e f Osman C, Voelker DR, Langer T (January 2011). "Making heads or tails of phospholipids in mitochondria". The Journal of Cell Biology. 192 (1): 7–16. doi:10.1083/jcb.201006159. PMC 3019561. PMID 21220505.
- ^ Kornmann B, Currie E, Collins SR, Schuldiner M, Nunnari J, Weissman JS, Walter P (July 2009). "An ER-mitochondria tethering complex revealed by a synthetic biology screen". Science. 325 (5939): 477–481. Bibcode:2009Sci...325..477K. doi:10.1126/science.1175088. PMC 2933203. PMID 19556461.
- ^ Rusiñol AE, Cui Z, Chen MH, Vance JE (November 1994). "A unique mitochondria-associated membrane fraction from rat liver has a high capacity for lipid synthesis and contains pre-Golgi secretory proteins including nascent lipoproteins". The Journal of Biological Chemistry. 269 (44): 27494–27502. doi:10.1016/S0021-9258(18)47012-3. PMID 7961664.
- ^ a b Kopach O, Kruglikov I, Pivneva T, Voitenko N, Fedirko N (May 2008). "Functional coupling between ryanodine receptors, mitochondria and Ca(2+) ATPases in rat submandibular acinar cells". Cell Calcium. 43 (5): 469–481. doi:10.1016/j.ceca.2007.08.001. PMID 17889347.
- ^ Csordás G, Hajnóczky G (April 2001). "Sorting of calcium signals at the junctions of endoplasmic reticulum and mitochondria". Cell Calcium. 29 (4): 249–262. doi:10.1054/ceca.2000.0191. PMID 11243933.
- ^ a b c d Decuypere JP, Monaco G, Bultynck G, Missiaen L, De Smedt H, Parys JB (May 2011). "The IP(3) receptor-mitochondria connection in apoptosis and autophagy". Biochimica et Biophysica Acta (BBA) - Molecular Cell Research. 1813 (5): 1003–1013. doi:10.1016/j.bbamcr.2010.11.023. PMID 21146562.
- ^ Diercks BP, Fliegert R, Guse AH (June 2017). "Mag-Fluo4 in T cells: Imaging of intra-organelle free Ca2+ concentrations". Biochimica et Biophysica Acta (BBA) - Molecular Cell Research. 1864 (6): 977–986. doi:10.1016/j.bbamcr.2016.11.026. PMID 27913206.
- ^ Hajnóczky G, Csordás G, Yi M (2011). "Old players in a new role: mitochondria-associated membranes, VDAC, and ryanodine receptors as contributors to calcium signal propagation from endoplasmic reticulum to the mitochondria". Cell Calcium. 32 (5–6): 363–377. doi:10.1016/S0143416002001872. PMID 12543096.
- ^ Marriott KS, Prasad M, Thapliyal V, Bose HS (December 2012). "σ-1 receptor at the mitochondrial-associated endoplasmic reticulum membrane is responsible for mitochondrial metabolic regulation". The Journal of Pharmacology and Experimental Therapeutics. 343 (3): 578–586. doi:10.1124/jpet.112.198168. PMC 3500540. PMID 22923735.
- ^ Hayashi T, Su TP (November 2007). "Sigma-1 receptor chaperones at the ER-mitochondrion interface regulate Ca(2+) signaling and cell survival". Cell. 131 (3): 596–610. doi:10.1016/j.cell.2007.08.036. PMID 17981125. S2CID 18885068.
- ^ Cordás et al., Trends Cell Biol. 2018 Jul;28(7):523-540. doi:10.1016/j.tcb. 2018.02.009. 2018년 3월 24일 Epub
- ^ McCutcheon JP (October 2021). "The Genomics and Cell Biology of Host-Beneficial Intracellular Infections". Annual Review of Cell and Developmental Biology. 37 (1): 115–142. doi:10.1146/annurev-cellbio-120219-024122. PMID 34242059. S2CID 235786110.
- ^ Callier V (8 June 2022). "Mitochondria and the origin of eukaryotes". Knowable Magazine. doi:10.1146/knowable-060822-2. S2CID 249526889. Retrieved 18 August 2022.
- ^ a b Margulis L, Sagan D (1986). Origins of Sex: Three billion years of genetic recombination. New Haven, CT: Yale University Press. pp. 69–71, 87. ISBN 978-0300033403.
- ^ Martin WF, Müller M (2007). Origin of mitochondria and hydrogenosomes. Heidelberg, DE: Springer Verlag.
- ^ Emelyanov, V.V. (April 2003). "Mitochondrial connection to the origin of the eukaryotic cell". European Journal of Biochemistry. 270 (8): 1599–1618. doi:10.1046/j.1432-1033.2003.03499.x. PMID 12694174.
- ^ Müller M, Martin W (May 1999). "The genome of Rickettsia prowazekii and some thoughts on the origin of mitochondria and hydrogenosomes". BioEssays. 21 (5): 377–381. doi:10.1002/(sici)1521-1878(199905)21:5<377::aid-bies4>3.0.co;2-w. PMID 10376009.
- ^ Gray MW, Burger G, Lang BF (March 1999). "Mitochondrial evolution". Science. 283 (5407): 1476–1481. Bibcode:1999Sci...283.1476G. doi:10.1126/science.283.5407.1476. PMC 3428767. PMID 10066161.
- ^ Thrash JC, Boyd A, Huggett MJ, Grote J, Carini P, Yoder RJ, et al. (2011-06-14). "Phylogenomic evidence for a common ancestor of mitochondria and the SAR11 clade". Scientific Reports. 1 (1): 13. Bibcode:2011NatSR...1E..13T. doi:10.1038/srep00013. PMC 3216501. PMID 22355532.
- ^ Martijn J, Vosseberg J, Guy L, Offre P, Ettema TJ (May 2018). "Deep mitochondrial origin outside the sampled alphaproteobacteria". Nature. 557 (7703): 101–105. Bibcode:2018Natur.557..101M. doi:10.1038/s41586-018-0059-5. hdl:1874/373336. PMID 29695865. S2CID 13740626.
- ^ Fan L, Wu D, Goremykin V, Xiao J, Xu Y, Garg S, et al. (September 2020). "Phylogenetic analyses with systematic taxon sampling show that mitochondria branch within Alphaproteobacteria". Nature Ecology & Evolution. Springer Science and Business Media LLC. 4 (9): 1213–1219. Bibcode:2020NatEE...4.1213F. bioRxiv 10.1101/715870. doi:10.1038/s41559-020-1239-x. PMID 32661403. S2CID 220507452.
- ^ Wang S, Luo H (June 2021). "Dating Alphaproteobacteria evolution with eukaryotic fossils". Nature Communications. 12 (1): 3324. Bibcode:2021NatCo..12.3324W. doi:10.1038/s41467-021-23645-4. PMC 8175736. PMID 34083540.
- ^ Esposti MD, Geiger O, Sanchez-Flores A (2022-05-16). "On the bacterial ancestry of mitochondria: New insights with triangulated approaches". bioRxiv: 2022.05.15.491939. doi:10.1101/2022.05.15.491939. S2CID 248836055.
- ^ Muñoz-Gómez SA, Susko E, Williamson K, Eme L, Slamovits CH, Moreira D, et al. (March 2022). "Site-and-branch-heterogeneous analyses of an expanded dataset favour mitochondria as sister to known Alphaproteobacteria". Nature Ecology & Evolution. 6 (3): 253–262. Bibcode:2022NatEE...6..253M. doi:10.1038/s41559-021-01638-2. PMID 35027725. S2CID 245958471.
- ^ Schön ME, Martijn J, Vosseberg J, Köstlbacher S, Ettema TJ (August 2022). "The evolutionary origin of host association in the Rickettsiales". Nature Microbiology. 7 (8): 1189–1199. doi:10.1038/s41564-022-01169-x. PMC 9352585. PMID 35798888.
- ^ O'Brien TW (September 2003). "Properties of human mitochondrial ribosomes". IUBMB Life. 55 (9): 505–513. doi:10.1080/15216540310001626610. PMID 14658756. S2CID 43415449.
- ^ Sagan L (March 1967). "On the origin of mitosing cells". Journal of Theoretical Biology. 14 (3): 255–274. Bibcode:1967JThBi..14..225S. doi:10.1016/0022-5193(67)90079-3. PMID 11541392.
- ^ Emelyanov VV (February 2001). "Rickettsiaceae, rickettsia-like endosymbionts, and the origin of mitochondria". Bioscience Reports. 21 (1): 1–17. doi:10.1023/A:1010409415723. PMID 11508688. S2CID 10144020.
- ^ Feng DF, Cho G, Doolittle RF (November 1997). "Determining divergence times with a protein clock: update and reevaluation". Proceedings of the National Academy of Sciences of the United States of America. 94 (24): 13028–13033. Bibcode:1997PNAS...9413028F. doi:10.1073/pnas.94.24.13028. PMC 24257. PMID 9371794.
- ^ Cavalier-Smith T (1991). "Archamoebae: the ancestral eukaryotes?". Bio Systems. 25 (1–2): 25–38. doi:10.1016/0303-2647(91)90010-I. PMID 1854912.
- ^ Danovaro R, Dell'Anno A, Pusceddu A, Gambi C, Heiner I, Kristensen RM (April 2010). "The first metazoa living in permanently anoxic conditions". BMC Biology. 8: 30. doi:10.1186/1741-7007-8-30. PMC 2907586. PMID 20370908.
- ^ Coghaln A (7 April 2010). "The mud creature that lives without oxygen". Zoologger. New Scientist.
- ^ Shiflett AM, Johnson PJ (2010). "Mitochondrion-related organelles in eukaryotic protists". Annual Review of Microbiology. 64: 409–429. doi:10.1146/annurev.micro.62.081307.162826. PMC 3208401. PMID 20528687.
- ^ a b c Chan DC (June 2006). "Mitochondria: dynamic organelles in disease, aging, and development". Cell. 125 (7): 1241–1252. doi:10.1016/j.cell.2006.06.010. PMID 16814712. S2CID 8551160.
- ^ a b Wiesner RJ, Rüegg JC, Morano I (March 1992). "Counting target molecules by exponential polymerase chain reaction: copy number of mitochondrial DNA in rat tissues". Biochemical and Biophysical Research Communications. 183 (2): 553–559. doi:10.1016/0006-291X(92)90517-O. PMID 1550563.
- ^ Falkenberg M (July 2018). "Mitochondrial DNA replication in mammalian cells: overview of the pathway". Essays in Biochemistry. 62 (3): 287–296. doi:10.1042/ebc20170100. PMC 6056714. PMID 29880722.
- ^ a b Anderson S, Bankier AT, Barrell BG, de Bruijn MH, Coulson AR, Drouin J, et al. (April 1981). "Sequence and organization of the human mitochondrial genome". Nature. 290 (5806): 457–465. Bibcode:1981Natur.290..457A. doi:10.1038/290457a0. PMID 7219534. S2CID 4355527.
- ^ Fukuhara H, Sor F, Drissi R, Dinouël N, Miyakawa I, Rousset S, Viola AM (April 1993). "Linear mitochondrial DNAs of yeasts: frequency of occurrence and general features". Molecular and Cellular Biology. 13 (4): 2309–2314. doi:10.1128/mcb.13.4.2309. PMC 359551. PMID 8455612.
- ^ Bernardi G (December 1978). "Intervening sequences in the mitochondrial genome". Nature. 276 (5688): 558–559. Bibcode:1978Natur.276..558B. doi:10.1038/276558a0. PMID 214710. S2CID 4314378.
- ^ Hebbar SK, Belcher SM, Perlman PS (April 1992). "A maturase-encoding group IIA intron of yeast mitochondria self-splices in vitro". Nucleic Acids Research. 20 (7): 1747–1754. doi:10.1093/nar/20.7.1747. PMC 312266. PMID 1579468.
- ^ Gray MW, Lang BF, Cedergren R, Golding GB, Lemieux C, Sankoff D, et al. (February 1998). "Genome structure and gene content in protist mitochondrial DNAs". Nucleic Acids Research. 26 (4): 865–878. doi:10.1093/nar/26.4.865. PMC 147373. PMID 9461442.
- ^ Gray MW, Lang BF, Burger G (2004). "Mitochondria of protists". Annual Review of Genetics. 38: 477–524. doi:10.1146/annurev.genet.37.110801.142526. PMID 15568984.
- ^ Telonis AG, Loher P, Kirino Y, Rigoutsos I (2014). "Nuclear and mitochondrial tRNA-lookalikes in the human genome". Frontiers in Genetics. 5: 344. doi:10.3389/fgene.2014.00344. PMC 4189335. PMID 25339973.
- ^ Shao R, Kirkness EF, Barker SC (May 2009). "The single mitochondrial chromosome typical of animals has evolved into 18 minichromosomes in the human body louse, Pediculus humanus". Genome Research. 19 (5): 904–912. doi:10.1101/gr.083188.108. PMC 2675979. PMID 19336451.
- ^ Castro JA, Picornell A, Ramon M (December 1998). "Mitochondrial DNA: a tool for populational genetics studies". International Microbiology. 1 (4): 327–332. PMID 10943382.
- ^ Cann RL, Stoneking M, Wilson AC (January 1987). "Mitochondrial DNA and human evolution". Nature. 325 (6099): 31–36. Bibcode:1987Natur.325...31C. doi:10.1038/325031a0. PMID 3025745. S2CID 4285418.
- ^ Torroni A, Achilli A, Macaulay V, Richards M, Bandelt HJ (June 2006). "Harvesting the fruit of the human mtDNA tree". Trends in Genetics. 22 (6): 339–345. doi:10.1016/j.tig.2006.04.001. PMID 16678300.
- ^ a b Garrigan D, Hammer MF (September 2006). "Reconstructing human origins in the genomic era". Nature Reviews. Genetics. 7 (9): 669–680. doi:10.1038/nrg1941. PMID 16921345. S2CID 176541.
- ^ Krings M, Stone A, Schmitz RW, Krainitzki H, Stoneking M, Pääbo S (July 1997). "Neandertal DNA sequences and the origin of modern humans". Cell. 90 (1): 19–30. doi:10.1016/S0092-8674(00)80310-4. hdl:11858/00-001M-0000-0025-0960-8. PMID 9230299. S2CID 13581775.
- ^ Soares P, Ermini L, Thomson N, Mormina M, Rito T, Röhl A, et al. (June 2009). "Correcting for purifying selection: an improved human mitochondrial molecular clock". American Journal of Human Genetics. 84 (6): 740–759. doi:10.1016/j.ajhg.2009.05.001. PMC 2694979. PMID 19500773.
- ^ Nachman MW, Crowell SL (September 2000). "Estimate of the mutation rate per nucleotide in humans". Genetics. 156 (1): 297–304. doi:10.1093/genetics/156.1.297. PMC 1461236. PMID 10978293.
- ^ Crick FH, Orgel LE (1973). "Directed panspermia" (PDF). Icarus. 19 (3): 341–346. Bibcode:1973Icar...19..341C. doi:10.1016/0019-1035(73)90110-3.
p. 344: It is a little surprising that organisms with somewhat different codes do not coexist.
좀 더 논의해 보겠습니다. - ^ Barrell BG, Bankier AT, Drouin J (November 1979). "A different genetic code in human mitochondria". Nature. 282 (5735): 189–194. Bibcode:1979Natur.282..189B. doi:10.1038/282189a0. PMID 226894. S2CID 4335828.
- ^ a b Elzanowski A, Ostell J (2019-01-07). "The Genetic Codes". www.ncbi.nlm.nih.gov. Retrieved 2023-02-10.
- ^ Jukes TH, Osawa S (December 1990). "The genetic code in mitochondria and chloroplasts". Experientia. 46 (11–12): 1117–1126. doi:10.1007/BF01936921. PMID 2253709. S2CID 19264964.
- ^ Hiesel R, Wissinger B, Schuster W, Brennicke A (December 1989). "RNA editing in plant mitochondria". Science. 246 (4937): 1632–1634. Bibcode:1989Sci...246.1632H. doi:10.1126/science.2480644. PMID 2480644.
- ^ Abascal F, Posada D, Knight RD, Zardoya R (May 2006). "Parallel evolution of the genetic code in arthropod mitochondrial genomes". PLOS Biology. 4 (5): e127. doi:10.1371/journal.pbio.0040127. PMC 1440934. PMID 16620150.
- ^ Pfeiffer RF (2012). Parkinson's Disease. CRC Press. p. 583. ISBN 978-1439807149.
- ^ Seo AY, Joseph AM, Dutta D, Hwang JC, Aris JP, Leeuwenburgh C (August 2010). "New insights into the role of mitochondria in aging: mitochondrial dynamics and more". Journal of Cell Science. 123 (Pt 15): 2533–2542. doi:10.1242/jcs.070490. PMC 2912461. PMID 20940129.
- ^ Hu GB (August 2014). "Whole cell cryo-electron tomography suggests mitochondria divide by budding". Microscopy and Microanalysis. 20 (4): 1180–1187. Bibcode:2014MiMic..20.1180H. doi:10.1017/S1431927614001317. PMID 24870811. S2CID 7440669.
- ^ McWilliams TG, Suomalainen A (January 2019). "Mitochondrial DNA can be inherited from fathers, not just mothers". Nature. 565 (7739): 296–297. Bibcode:2019Natur.565..296M. doi:10.1038/d41586-019-00093-1. PMID 30643304.
- ^ Kimball, J.W. (2006) "인간에서의 성적 생식: "교배와 수정", 킴볼의 생물학 페이지 (생물학에 기초, 6판, 1996)
- ^ Sutovsky P, Moreno RD, Ramalho-Santos J, Dominko T, Simerly C, Schatten G (November 1999). "Ubiquitin tag for sperm mitochondria". Nature. 402 (6760): 371–372. Bibcode:1999Natur.402..371S. doi:10.1038/46466. PMID 10586873. S2CID 205054671. 과학 뉴스 아카이브 2007-12-19에서 Wayback Machine에서 논의되었습니다.
- ^ Mogensen HL (1996). "The Hows and Whys of Cytoplasmic Inheritance in Seed Plants". American Journal of Botany. 83 (3): 383–404. doi:10.2307/2446172. JSTOR 2446172.
- ^ Zouros E (December 2000). "The exceptional mitochondrial DNA system of the mussel family Mytilidae". Genes & Genetic Systems. 75 (6): 313–318. doi:10.1266/ggs.75.313. PMID 11280005.
- ^ Sutherland B, Stewart D, Kenchington ER, Zouros E (January 1998). "The fate of paternal mitochondrial DNA in developing female mussels, Mytilus edulis: implications for the mechanism of doubly uniparental inheritance of mitochondrial DNA". Genetics. 148 (1): 341–347. doi:10.1093/genetics/148.1.341. PMC 1459795. PMID 9475744.
- ^ 도날드 T에 의해 웨이백 머신에 보관된 블루 홍합(Mytilus edulis) 종군의 암수 미토콘드리아 DNA 계통 2013-05-18. 스튜어트, 카를로스 사베드라, 레베카 R. 스탠우드, 에이미 0.볼, 엘레프테리오스 주로스
- ^ Johns DR (October 2003). "Paternal transmission of mitochondrial DNA is (fortunately) rare". Annals of Neurology. 54 (4): 422–424. doi:10.1002/ana.10771. PMID 14520651. S2CID 90422.
- ^ Thyagarajan B, Padua RA, Campbell C (November 1996). "Mammalian mitochondria possess homologous DNA recombination activity". The Journal of Biological Chemistry. 271 (44): 27536–27543. doi:10.1074/jbc.271.44.27536. PMID 8910339.
- ^ Lunt DH, Hyman BC (May 1997). "Animal mitochondrial DNA recombination". Nature. 387 (6630): 247. Bibcode:1997Natur.387..247L. doi:10.1038/387247a0. PMID 9153388. S2CID 4318161.
- ^ Eyre-Walker A, Smith NH, Smith JM (March 1999). "How clonal are human mitochondria?". Proceedings. Biological Sciences. 266 (1418): 477–483. doi:10.1098/rspb.1999.0662. PMC 1689787. PMID 10189711.
- ^ Awadalla P, Eyre-Walker A, Smith JM (December 1999). "Linkage disequilibrium and recombination in hominid mitochondrial DNA". Science. 286 (5449): 2524–2525. doi:10.1126/science.286.5449.2524. PMID 10617471. S2CID 27685285.
- ^ Cree LM, Samuels DC, de Sousa Lopes SC, Rajasimha HK, Wonnapinij P, Mann JR, et al. (February 2008). "A reduction of mitochondrial DNA molecules during embryogenesis explains the rapid segregation of genotypes". Nature Genetics. 40 (2): 249–254. doi:10.1038/ng.2007.63. PMID 18223651. S2CID 205344980.
- ^ Cao L, Shitara H, Horii T, Nagao Y, Imai H, Abe K, et al. (March 2007). "The mitochondrial bottleneck occurs without reduction of mtDNA content in female mouse germ cells". Nature Genetics. 39 (3): 386–390. doi:10.1038/ng1970. PMID 17293866. S2CID 10686347.
- ^ Wai T, Teoli D, Shoubridge EA (December 2008). "The mitochondrial DNA genetic bottleneck results from replication of a subpopulation of genomes". Nature Genetics. 40 (12): 1484–1488. doi:10.1038/ng.258. PMID 19029901. S2CID 225349.
- ^ Johnston IG, Burgstaller JP, Havlicek V, Kolbe T, Rülicke T, Brem G, et al. (June 2015). "Stochastic modelling, Bayesian inference, and new in vivo measurements elucidate the debated mtDNA bottleneck mechanism". eLife. 4: e07464. arXiv:1512.02988. doi:10.7554/eLife.07464. PMC 4486817. PMID 26035426.
- ^ Gredilla R, Garm C, Stevnsner T (2012). "Nuclear and mitochondrial DNA repair in selected eukaryotic aging model systems". Oxidative Medicine and Cellular Longevity. 2012: 282438. doi:10.1155/2012/282438. PMC 3462412. PMID 23050036.
- ^ a b c Saki M, Prakash A (June 2017). "DNA damage related crosstalk between the nucleus and mitochondria". Free Radical Biology & Medicine. 107: 216–227. doi:10.1016/j.freeradbiomed.2016.11.050. PMC 5449269. PMID 27915046.
- ^ Leon J, Sakumi K, Castillo E, Sheng Z, Oka S, Nakabeppu Y (February 2016). "8-Oxoguanine accumulation in mitochondrial DNA causes mitochondrial dysfunction and impairs neuritogenesis in cultured adult mouse cortical neurons under oxidative conditions". Scientific Reports. 6: 22086. Bibcode:2016NatSR...622086L. doi:10.1038/srep22086. PMC 4766534. PMID 26912170.
- ^ Dahal S, Dubey S, Raghavan SC (May 2018). "Homologous recombination-mediated repair of DNA double-strand breaks operates in mammalian mitochondria". Cellular and Molecular Life Sciences. 75 (9): 1641–1655. doi:10.1007/s00018-017-2702-y. PMID 29116362. S2CID 4779650.
- ^ Odahara M, Inouye T, Fujita T, Hasebe M, Sekine Y (February 2007). "Involvement of mitochondrial-targeted RecA in the repair of mitochondrial DNA in the moss, Physcomitrella patens". Genes & Genetic Systems. 82 (1): 43–51. doi:10.1266/ggs.82.43. PMID 17396019.
- ^ Tadi SK, Sebastian R, Dahal S, Babu RK, Choudhary B, Raghavan SC (January 2016). "Microhomology-mediated end joining is the principal mediator of double-strand break repair during mitochondrial DNA lesions". Molecular Biology of the Cell. 27 (2): 223–235. doi:10.1091/mbc.E15-05-0260. PMC 4713127. PMID 26609070.
- ^ a b Henriquez FL, Richards TA, Roberts F, McLeod R, Roberts CW (February 2005). "The unusual mitochondrial compartment of Cryptosporidium parvum". Trends in Parasitology. 21 (2): 68–74. doi:10.1016/j.pt.2004.11.010. PMID 15664529.
- ^ John U, Lu Y, Wohlrab S, Groth M, Janouškovec J, Kohli GS, et al. (April 2019). "An aerobic eukaryotic parasite with functional mitochondria that likely lacks a mitochondrial genome". Science Advances. 5 (4): eaav1110. Bibcode:2019SciA....5.1110J. doi:10.1126/sciadv.aav1110. PMC 6482013. PMID 31032404.
- ^ "Veritable powerhouse – even without DNA: Parasitic algae from the dinoflagellate lineage have organized their genetic material in an unprecedented way". ScienceDaily.
- ^ a b c Zeviani M, Di Donato S (October 2004). "Mitochondrial disorders". Brain. 127 (Pt 10): 2153–2172. doi:10.1093/brain/awh259. PMID 15358637.
- ^ Taylor RW, Turnbull DM (May 2005). "Mitochondrial DNA mutations in human disease". Nature Reviews. Genetics. 6 (5): 389–402. doi:10.1038/nrg1606. PMC 1762815. PMID 15861210.
- ^ Goldman A, Khiste S, Freinkman E, Dhawan A, Majumder B, Mondal J, et al. (August 2019). "Targeting tumor phenotypic plasticity and metabolic remodeling in adaptive cross-drug tolerance". Science Signaling. 12 (595). doi:10.1126/scisignal.aas8779. PMC 7261372. PMID 31431543.
- ^ Saha T, Dash C, Jayabalan R, Khiste S, Kulkarni A, Kurmi K, et al. (January 2022). "Intercellular nanotubes mediate mitochondrial trafficking between cancer and immune cells". Nature Nanotechnology. 17 (1): 98–106. Bibcode:2022NatNa..17...98S. doi:10.1038/s41565-021-01000-4. PMC 10071558. PMID 34795441. S2CID 244349825.
- ^ Chinnery PF, Schon EA (September 2003). "Mitochondria". Journal of Neurology, Neurosurgery, and Psychiatry. 74 (9): 1188–1199. doi:10.1136/jnnp.74.9.1188. PMC 1738655. PMID 12933917.
- ^ Sherer TB, Betarbet R, Greenamyre JT (June 2002). "Environment, mitochondria, and Parkinson's disease". The Neuroscientist. 8 (3): 192–197. doi:10.1177/1073858402008003004. PMID 12061498. S2CID 24318009.
- ^ Gomez C, Bandez MJ, Navarro A (January 2007). "Pesticides and impairment of mitochondrial function in relation with the parkinsonian syndrome". Frontiers in Bioscience. 12: 1079–1093. doi:10.2741/2128. PMID 17127363.
- ^ Lim YA, Rhein V, Baysang G, Meier F, Poljak A, Raftery MJ, et al. (April 2010). "Abeta and human amylin share a common toxicity pathway via mitochondrial dysfunction". Proteomics. 10 (8): 1621–1633. doi:10.1002/pmic.200900651. PMID 20186753. S2CID 33077667.
- ^ King JV, Liang WG, Scherpelz KP, Schilling AB, Meredith SC, Tang WJ (July 2014). "Molecular basis of substrate recognition and degradation by human presequence protease". Structure. 22 (7): 996–1007. doi:10.1016/j.str.2014.05.003. PMC 4128088. PMID 24931469.
- ^ Schapira AH (July 2006). "Mitochondrial disease". Lancet. 368 (9529): 70–82. doi:10.1016/S0140-6736(06)68970-8. PMID 16815381. S2CID 27039617.
- ^ Pieczenik SR, Neustadt J (August 2007). "Mitochondrial dysfunction and molecular pathways of disease". Experimental and Molecular Pathology. 83 (1): 84–92. doi:10.1016/j.yexmp.2006.09.008. PMID 17239370.
- ^ Bugger H, Abel ED (November 2010). "Mitochondria in the diabetic heart". Cardiovascular Research. 88 (2): 229–240. doi:10.1093/cvr/cvq239. PMC 2952534. PMID 20639213.
- ^ Podolak, Amira; Woclawek-Potocka, Izabela; Lukaszuk, Krzysztof (2022-02-24). "The Role of Mitochondria in Human Fertility and Early Embryo Development: What Can We Learn for Clinical Application of Assessing and Improving Mitochondrial DNA?". Cells. 11 (5): 797. doi:10.3390/cells11050797. ISSN 2073-4409. PMC 8909547. PMID 35269419.
- ^ May-Panloup, Pascale; Boguenet, Magalie; El Hachem, Hady; Bouet, Pierre-Emmanuel; Reynier, Pascal (2021-01-20). "Embryo and Its Mitochondria". Antioxidants. 10 (2): 139. doi:10.3390/antiox10020139. ISSN 2076-3921. PMC 7908991. PMID 33498182.
- ^ Claiborne, Anne; English, Rebecca; Kahn, Jeffrey; Diseases, Committee on the Ethical and Social Policy Considerations of Novel Techniques for Prevention of Maternal Transmission of Mitochondrial DNA; Policy, Board on Health Sciences; Medicine, Institute of; National Academies of Sciences, Engineering (2016-03-17), "Introduction", Mitochondrial Replacement Techniques: Ethical, Social, and Policy Considerations, National Academies Press (US), retrieved 2023-12-05
{{citation}}:first4=일반 이름(도움말)이 있습니다. - ^ Richter C, Park JW, Ames BN (September 1988). "Normal oxidative damage to mitochondrial and nuclear DNA is extensive". Proceedings of the National Academy of Sciences of the United States of America. 85 (17): 6465–6467. Bibcode:1988PNAS...85.6465R. doi:10.1073/pnas.85.17.6465. PMC 281993. PMID 3413108.
- ^ Harman D (July 1956). "Aging: a theory based on free radical and radiation chemistry". Journal of Gerontology. 11 (3): 298–300. CiteSeerX 10.1.1.663.3809. doi:10.1093/geronj/11.3.298. PMID 13332224.
- ^ Harman D (April 1972). "The biologic clock: the mitochondria?". Journal of the American Geriatrics Society. 20 (4): 145–147. doi:10.1111/j.1532-5415.1972.tb00787.x. PMID 5016631. S2CID 396830.
- ^ "Mitochondria and Aging". circuitblue.co.
- ^ Boffoli D, Scacco SC, Vergari R, Solarino G, Santacroce G, Papa S (April 1994). "Decline with age of the respiratory chain activity in human skeletal muscle". Biochimica et Biophysica Acta (BBA) - Molecular Basis of Disease. 1226 (1): 73–82. doi:10.1016/0925-4439(94)90061-2. PMID 8155742.
- ^ de Grey AD (2004). "Mitochondrial mutations in mammalian aging: an over-hasty about-turn?". Rejuvenation Research. 7 (3): 171–174. doi:10.1089/rej.2004.7.171. PMID 15588517.
- ^ Bender A, Krishnan KJ, Morris CM, Taylor GA, Reeve AK, Perry RH, et al. (May 2006). "High levels of mitochondrial DNA deletions in substantia nigra neurons in aging and Parkinson disease". Nature Genetics. 38 (5): 515–517. doi:10.1038/ng1769. PMID 16604074. S2CID 13956928.
- ^ Mehta AR, Walters R, Waldron FM, Pal S, Selvaraj BT, Macleod MR, et al. (August 2019). "Targeting mitochondrial dysfunction in amyotrophic lateral sclerosis: a systematic review and meta-analysis". Brain Communications. 1 (1): fcz009. doi:10.1093/braincomms/fcz009. PMC 7056361. PMID 32133457. S2CID 201168692.
- ^ Mehta AR, Gregory JM, Dando O, Carter RN, Burr K, Nanda J, et al. (February 2021). "Mitochondrial bioenergetic deficits in C9orf72 amyotrophic lateral sclerosis motor neurons cause dysfunctional axonal homeostasis". Acta Neuropathologica. 141 (2): 257–279. doi:10.1007/s00401-020-02252-5. PMC 7847443. PMID 33398403.
- ^ Chiang JL, Shukla P, Pagidas K, Ahmed NS, Karri S, Gunn DD, et al. (November 2020). "Mitochondria in Ovarian Aging and Reproductive Longevity". Ageing Research Reviews. 63: 101168. doi:10.1016/j.arr.2020.101168. PMC 9375691. PMID 32896666. S2CID 221472721.
- ^ Kölliker A (1857). "Einige Bemerkungen über die Endigungen der Hautnerven und den Bau der Muskeln" [Some remarks about the terminations of the cutaneous nerves and the structure of muscles]. Zeitschrift für wissenschaftliche Zoologie (in German). 8: 311–325. 316쪽에서, 쾰리커는 그가 신선한 개구리 근육에서 관찰한 미토콘드리아에 대해 설명했습니다: "…세르 블라스 룬들리체 쾨른첸, 랑겐 리니엔푀르미겐 취겐의 웰체." (…그것들은 긴 선형 열차에서 [근육의] 수축성 물질에 내장된 매우 희미한 둥근 과립입니다." (…그것들은] 매우 희미한 둥근 과립입니다. 이 과립들은 근육 섬유의 전체 두께, 내부와 같은 표면에 위치하고 있으며, 일단 사람들이 이 과립들을 의식하기 시작하면 근육 섬유의 중요하지 않은 요소로 보일 정도로 매우 많습니다.) 쾰리커는 (321쪽) 다른 동물들의 근육에서 미토콘드리아를 발견했다고 말했습니다. 표 XIV의 그림 3에서 쾰리커는 개구리 근육의 미토콘드리아를 묘사했습니다.
- ^ a b c d e f g h i j Ernster L, Schatz G (December 1981). "Mitochondria: a historical review". The Journal of Cell Biology. 91 (3 Pt 2): 227s–255s. doi:10.1083/jcb.91.3.227s. PMC 2112799. PMID 7033239.
- ^ Altmann R (1890). Die Elementarorganismen und ihre Beziehungen zu den Zellen [Elementary Organisms and Their Relations to Cells] (in German). Leipzig, Germany: Veit & Co. p. 125. 125쪽부터: "Dauch sonst Mancherlei Umstände dafür sprechen, dass Microorganismen und Granula einander gleichwerthig und Elementarorganismen vorstellen, Welchesich Uüberall finden, wollebendige Kräfte ausgelöstwerden, wollen sie demeinschaftlichen, Namen der Bioblasten, Daughter sons mancherlei Umstände dafür sprechen, dass Microorganismen und Granula einander gleichwerthig und Elementarorganismen vorstellen, welcheich überall finden, wollebendige K"(그렇지 않은 상황에 따라서는 미생물과 과립이 서로 동등하다는 것을 나타내고, 생명력이 분출되는 곳이라면 어디서나 발견될 수 있는 기본적인 유기체를 암시하기 때문에, 우리는 이들을 "바이오블라스트"라는 총칭으로 지정할 것입니다.)
- ^ "mitochondria". Online Etymology Dictionary.
- ^ Benda C (1898). "Ueber die Spermatogenese der Vertebraten und höherer Evertebraten. II. Theil: Die Histiogenese der Spermien" [On spermatogenesis in vertebrates and higher invertebrates. Part II: The histogenesis of sperm.]. Archiv für Physiologie (in German). 1898: 393–398. 397쪽: 브렌다가 "iich bere ret be vielen Zellarten aler möglichen Thierclassen gefunden habe," (… 저는 이미 모든 가능한 종류의 동물의 많은 세포에서 그들(미토콘드리아)을 발견했습니다."라고 말한 후, 그는 제안합니다: "Ichmöchte vorläufig vorschlagen, Ihnen als Moticotriaine besondere Stellung vorzubehalten," 아르베텐 베그룬덴베르데의 다이이치(die ich in Arbeiten begründen werde)". (저는 그들을 위해 임시로 보존할 것을 제안하고 싶습니다. "미토콘드리아", 이것은 추후 연구에서 정당화할 특별한 지위입니다.)
- Benda C (1899). "Weitere Mitteilungen über die Mitochondria" [Further reports on mitochondria]. Archiv für Physiologie: Verhandlungen der Berliner Physiologischen Gesellschaft (in German). 1899: 376–383.
- ^ Michaelis L (1900). "Die vitale Farbung, eine Darstellungsmethode der Zellgranula" [The vital stain: a method of imaging cell granules]. Archiv für Mikroskopische Anatomie und Entwicklungsgeschichte [Archive for Microscopic Anatomy and Ontogenesis] (in German). 55: 558–575. doi:10.1007/BF02977747. S2CID 85010030.
- ^ Ernster's citation Meves F (May 1908). "Die Chondriosomen als Träger erblicher Anlagen. Cytologische Studien am Hühnerembryo". Archiv für Mikroskopische Anatomie. 72 (1): 816–867. doi:10.1007/BF02982402. S2CID 85047330. is wrong, correct citation is Meves F (1904). "Über das Vorkommen von Mitochondrien bezw. Chondromiten in Pflanzenzellen". Ber. Dtsch. Bot. Ges. 22: 284–286. doi:10.1111/j.1438-8677.1904.tb05237.x. S2CID 257822147., cited in Meves' 1908 paper and in Schmidt EW (1913). "Pflanzliche Mitochondrien". Progressus Rei Botanicae. 4: 164–183. Retrieved 21 September 2012., with confirmation of Nymphaea alba
- ^ Kingsbury BF (1912). "Cytoplasmic fixation". The Anatomical Record. 6 (2): 39–52. doi:10.1002/ar.1090060202. S2CID 221400422. p. 47부터: "… 미토콘드리아는 미토콘드리아의 구조적인 표현입니다 (즉, 세포질에서 화학적 환원 과정의 표현입니다)."
- ^ Warburg O (1913). "Über sauerstoffatmende Körnchen aus Leberzellen und über Sauerstoffatmung in Berkefeld-Filtraten wässriger Leberextrake" [On respiring granules from liver cells and on respiration in Berkefeld filtrates of aqueous liver extracts]. Pflügers Archiv für die gesamte Physiologie des Menschen und der Tiere (Pfluger's Archive for All Physiology of Humans and Animals) (in German). 154: 599–617.
- ^ Milane L, Trivedi M, Singh A, Talekar M, Amiji M (June 2015). "Mitochondrial biology, targets, and drug delivery". Journal of Controlled Release. 207: 40–58. doi:10.1016/j.jconrel.2015.03.036. PMID 25841699.
- ^ Martin WF, Garg S, Zimorski V (September 2015). "Endosymbiotic theories for eukaryote origin". Philosophical Transactions of the Royal Society of London. Series B, Biological Sciences. 370 (1678): 20140330. doi:10.1098/rstb.2014.0330. PMC 4571569. PMID 26323761.
일반
 이 기사는 다음과 같은 퍼블릭 도메인 자료를 포함합니다.
이 기사는 다음과 같은 퍼블릭 도메인 자료를 포함합니다.
외부 링크
- Lane N (2016). The Vital Question: Energy, Evolution, and the Origins of Complex Life. WW Norton & Company. ISBN 978-0393352979.
- 세포 미토콘드리아 전원 공급 – XVIO Scientific Animation
- Mitodb.com – 미토콘드리아 질환 데이터베이스.
- 마인츠 대학교 미토콘드리아 지도
- 미토콘드리아의 미토콘드리아 연구 포털.그물
- 미토콘드리아: 아키텍처는 cytochemistry.net 에서 기능을 결정합니다.
- 앨라배마 대학교 미토콘드리아 연계
- MIP 미토콘드리아 생리학 학회
- 미시간 대학교 미토콘드리아 내막 단백질의 3차원 구조
- 미시건 대학의 미토콘드리아 외막과 관련된 단백질의 3차원 구조
- 위스콘신 대학교 미토콘드리아 단백질 파트너십
- MitoMiner – MRC 미토콘드리아 생물학 유닛의 미토콘드리아 프로테오믹스 데이터베이스[permanent dead link]
- 미토콘드리아 – 세포 중심 데이터베이스
- 샌디에이고 주립대학 전자단층촬영으로 재구성된 미토콘드리아
- 극저온 전자단층촬영에서 나온 쥐-간 미토콘드리아 영상








