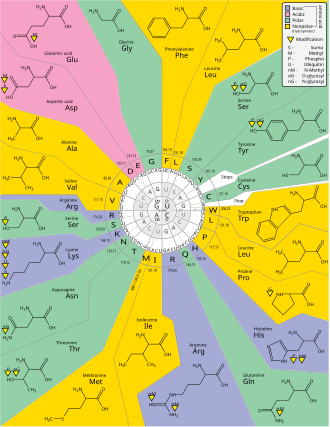
유전자 코드
Genetic code유전자 코드는 살아있는 세포가 유전 물질 내에 암호화된 정보(뉴클레오티드 삼중수소나 코돈의 DNA 또는 mRNA 시퀀스)를 단백질로 변환하기 위해 사용하는 규칙 집합이다.번역은 전령 RNA(mRNA)가 지정한 순서에 따라 단백질 발생 아미노산을 연결하는 리보솜에 의해 이루어지며, 전달 RNA(tRNA) 분자를 사용하여 아미노산을 운반하고 동시에 mRNA 3 뉴클레오티드를 읽는다.유전자 코드는 모든 유기체에서 매우 유사하며 64개의 항목이 포함된 간단한 표로 표현할 수 있다.
코돈은 단백질 합성이 진행되는 동안 다음에 어떤 아미노산이 첨가될 것인지를 명시한다.일부 예외를 제외하고, 핵산 염기서열에서 3-뉴클레오티드 코돈은 단일 아미노산을 지정한다.[1]대부분의 유전자는 단일 체계로 암호화된다(RNA 코돈 표 참조).그 계획은 종종 표준 유전자 코드 또는 단순히 유전자 코드라고 일컬어지지만, 변종 코드(예: 미토콘드리아)는 존재한다.
역사
단백질이 어떻게 암호화되는지 이해하기 위한 노력은 1953년 DNA의 구조가 발견된 이후 시작되었다.George Gamow는 살아있는 세포가 단백질을 형성하기 위해 사용하는 20개의 표준 아미노산을 인코딩하기 위해 3개의 염기 세트가 사용되어야 한다고 가정했는데, 이것은3 최대 4 = 64개의 아미노산을 허용한다.[2]
코돈스
크릭, 브레너, 바넷, 와츠토빈 실험은 코돈들이 세 개의 DNA 염기들로 이루어져 있다는 것을 처음으로 증명했다.1961년 마샬 니렌버그와 하인리히 J. 마태이는 처음으로 코돈의 본질을 드러냈다.[3]
그들은 세포가 없는 시스템을 사용하여 폴리-우라실 RNA 염기서열(즉, UUUUU...)을 번역했고, 합성된 폴리펩타이드 성분이 아미노산 페닐알라닌으로만 구성되었다는 것을 밝혀냈다.[4]따라서 그들은 코돈 UUU가 아미노산 페닐알라닌을 지정했다고 추론했다.
이것은 세베로 오초아의 실험실에서 폴리 아데닌 RNA[5] 염기서열(AAAA...)이 폴리펩타이드 폴리프로라인에 대해 코딩되고 폴리펩타이드 폴리프로라인에 대해 폴리시토신 RNA 염기서열(CCCCCC...)이 코딩되었다는 것을 증명하는 실험이 뒤따랐다.[6]따라서, 코돈 AAA는 아미노산 리신을, 코돈 CCC는 아미노산 프로라인을 명시했다.그 후, 다양한 결합체를 사용하여 나머지 코돈의 대부분이 결정되었다.
하 고빈드 코라나의 후속 연구는 나머지 유전자 코드를 확인했다.얼마 지나지 않아 로버트 W. 홀리는 RNA를 단백질로 변환하는 과정을 용이하게 하는 어댑터 분자인 전이 RNA(transfer RNA, tRNA)의 구조를 결정했다.이 연구는 오초아의 초기 연구에 바탕을 두고 RNA 합성의 효소에 대한 연구로 1959년 노벨 생리의학상을 수여했다.[7]
이 작업을 연장하면서 니렌버그와 필립 레더는 코드의 삼중의 본성을 밝히고 코돈을 해독했다.이러한 실험에서는 RNA를 단백질로 변환하는 세포의 성분인 리보솜이 들어 있는 필터를 통해 mRNA의 다양한 조합이 통과되었다.독특한 세쌍둥이는 리보솜에 특정 tRNA의 결합을 촉진했다.레더와 니렌버그는 실험에서 64개의 코돈 중 54개의 순서를 결정할 수 있었다.[8]코라나, 홀리, 니렌버그는 그들의 업적으로 1968년 노벨상을 받았다.[9]
이 세 정거장 코돈은 발견자인 리차드 엡스타인과 찰스 스타인버그에 의해 이름이 지어졌다."암버"는 그들의 친구 해리스 번스타인의 이름을 따서 지어졌는데, 그의 성은 독일어로 "암버"를 의미한다.[10]나머지 두 정거장 코돈은 "컬러 네임" 테마를 유지하기 위해 "오크레"와 "오팔"로 명명되었다.
확장 유전자 코드(합성생물학)
넓은 학문적 청중에서는 유전코드를 원래의 애매모호한 유전자 코드에서 20 (+2)정규 아미노산의 레퍼토리로 잘 정의된 ("냉동") 코드로 진화하는 개념이 널리 받아들여지고 있다.[11]그러나 다양한 의견과 개념, 접근법, 아이디어가 있어 이를 실험적으로 변화시키는 최선의 방법이다.유전자 코드의 합성 아미노산 침투를 위한 '진입점'을 예측하는 모델까지 제안한다.[12]
2001년부터 40개의 비천연 아미노산이 단백질 구조와 기능을 탐색하는 도구로 이용되거나 새로운 단백질 생성이나 강화된 단백질을 만들기 위해 독특한 코돈(리코딩)과 그에 상응하는 전이-RNA:aminoacyl – tRNA-synthetase 쌍을 만들어 단백질에 첨가되었다.안쪽에[13][14]
H. 무라카미와 M.시시도는 코돈 몇 개를 늘려 4루와 5루로 만들었다.스티븐 A. Benner는 65번째( 생체내) 코돈(코돈)을 만들었다.[15]
2015년 N. Budisa, D. Söll과 동료들은 대장균 박테리아의 유전 코드에서 20,899개의 트립토판 잔류물(UGG 코돈)이 부자연스러운 티에노피롤-알라닌으로 완전히 대체되었다고 보고했다.[16]
2016년에 최초의 안정된 반신성 유기체가 만들어졌다.두 개의 합성 염기(X와 Y라고 한다)를 가진 (단일 세포) 박테리아였다.기지는 세포 분열에서 살아남았다.[17][18]
지난 2017년 국내 연구진이 부자연스러운 아미노산으로 단백질을 생산할 수 있는 확장 유전코드를 가진 생쥐를 조작했다고 보고한 바 있다.[19]
2019년 5월, 연구원들은 대장균 박테리아의 새로운 "Syn61" 변종이 생성되었다고 보고했다.이 변종은 리팩터링(모든 중복이 확대됨), 리코딩(전체 코돈 64개 중 3개를 완전히 제거함)한 완전 합성 게놈을 가지고 있으며, 이제 불필요한 tRNA와 방출 인자를 제거하기 위해 추가로 수정한다.충분히 생존할 수 있고 야생형 상대형 'MDS42'[20][21]보다 1.6배 느리게 성장한다.
특징들
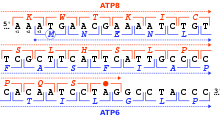
독서 틀
판독 프레임은 번역이 시작되는 뉴클레오티드의 초기 3중으로 정의된다.그것은 "열린 독서 프레임"(ORF)이라고 알려진 연속적이고 겹치지 않는 코돈의 실행을 위한 프레임을 설정한다.예를 들어, 문자열 5'-AAATGACG-3' (그림 참조)는 첫 번째 위치에서 읽었을 경우 코돈 AAA, TGA, ACG를 포함하고, 두 번째 위치에서 읽었을 경우 코돈 AAT 및 GAA를 포함하고, 세 번째 위치에서 읽었을 경우 코돈 ATG와 AC를 포함한다.Every sequence can, thus, be read in its 5' → 3' direction in three reading frames, each producing a possibly distinct amino acid sequence: in the given example, Lys (K)-Trp (W)-Thr (T), Asn (N)-Glu (E), or Met (M)-Asn (N), respectively (when translating with the vertebrate mitochondrial code).DNA가 이중 가닥일 때 6개의 가능한 판독 프레임이 정의되며, 한 가닥의 전방 방향에서 3개, 반대쪽 가닥에서 3개씩이다.[23]: 330 단백질 코딩 프레임은 시작 코돈에 의해 정의되며, 일반적으로 RNA(DNA) 순서에서 첫 번째 AUG(ATG) 코돈에 의해 정의된다.
진핵생물에서 엑손의 ORF는 인트론에 의해 종종 중단된다.
코돈 시작 및 중지
번역은 체인 개시 코돈이나 시작 코돈으로 시작한다.시작 코돈만으로는 과정을 시작하기에 충분하지 않다.대장균의 샤인-달가르노 수열과 같은 인근 수열과 시작 인자도 번역을 시작해야 한다.가장 흔한 시작 코돈은 메티오닌 또는 박테리아에서 포밀메티오닌으로 읽히는 AUG이다.유기체에 따라 다른 출발 코돈은 "GUG" 또는 "UUG"를 포함한다. 이 코돈들은 보통 발린과 류신을 나타낸다. 그러나 출발 코돈으로서 그들은 메티오닌 또는 포밀메티오닌으로 번역된다.[24]
세 정거장 코돈에는 다음과 같은 이름이 있다.UAG는 황색, UGA는 오팔(Umber라고도 함), UAA는 오크레(ochre)이다.스톱 코돈은 "종말" 또는 "말도 안 되는" 코돈이라고도 불린다.그들은 리보솜에서 초기 폴리펩타이드의 방출을 신호한다. 왜냐하면 어떤 인지 tRNA도 이러한 정지 신호를 보완하는 해독제를 가지고 있지 않기 때문에 방출 인자가 대신 리보솜에 결합할 수 있기 때문이다.[25]
돌연변이의 영향
DNA 복제 과정에서 두 번째 가닥의 중합에 오류가 발생하는 경우가 있다.이러한 오류, 돌연변이는 특히 유전자의 단백질 부호화 순서 내에서 발생하는 유기체의 표현형상에 영향을 줄 수 있다.오류율은 일반적으로 1억 염기마다 1개의 오차가 발생하는데, 이는 DNA 중합체의 "검증" 능력 때문이다.[27][28]
미센스 돌연변이와 난센스 돌연변이는 각각 낫세포병, 탈라세미아 등 유전질환을 유발할 수 있는 포인트 돌연변이의 예다.[29][30][31]임상적으로 중요한 오식 돌연변이는 일반적으로 기본, 산성, 극성 또는 비극성 상태 사이에서 코딩된 아미노산 잔류물의 성질을 변화시키는 반면, 터무니없는 돌연변이는 코돈을 멈추게 한다.[23]
3개의 뉴클레오티드 베이스가 아닌 3개의 인델(삽입 또는 삭제)에 의한 판독 프레임 시퀀스를 방해하는 변이를 프레임시프트 돌연변이라고 한다.이러한 돌연변이는 대개 원본과 완전히 다른 번역으로 귀결되며, 단백질을 잘라내는 중지 코돈의 읽기를 유발할 가능성이 있다.[32]이러한 돌연변이는 단백질의 기능을 손상시킬 수 있으며 따라서 생체내 단백질 코딩 시퀀스에서는 드물다.프레임-트래프트 돌연변이의 유전자가 드물게 나타나는 한 가지 이유는, 번역되고 있는 단백질이 유기체가 직면하는 선택적 압력 하에서 성장에 필수적이라면, 기능적 단백질의 부재는 유기체가 생존하기 전에 죽음을 야기할 수 있기 때문이다.[33]프레임시프트 돌연변이는 타이-삭스병 같은 심각한 유전질환을 일으킬 수 있다.[34]
단백질 배열을 변화시키는 대부분의 돌연변이는 해롭거나 중립적이지만, 어떤 돌연변이는 장점을 가지고 있다.[35]이러한 돌연변이는 돌연변이 유기체가 야생형 유기체보다 특정한 환경적 스트레스를 더 잘 견딜 수 있게 하거나 더 빨리 번식하게 할 수 있다.이러한 경우 돌연변이는 자연 선택을 통해 개체군에서 더 흔해지는 경향이 있다.[36]RNA를 유전물질로 사용하는 바이러스는 돌연변이 발생률이 빠른데,[37] 이는 이들 바이러스가 급속하게 진화해 면역체계 방어반응을 회피하기 때문에 장점이 될 수 있다.[38]예를 들어 대장균과 같이 무성으로 생식하는 유기체의 많은 개체군에서 여러 개의 유익한 돌연변이가 함께 발생할 수 있다.이러한 현상을 종횡 간섭이라고 하며 돌연변이들 사이에 경쟁을 유발한다.[39]
퇴보
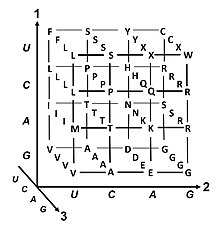
퇴보란 유전 코드의 중복이다.이 용어는 베른필드와 니렌베르크에 의해 주어졌다.유전자 코드는 중복성은 있지만 모호성은 없다(전체 상관관계는 아래 코돈 표를 참조하십시오).예를 들어, 코돈 GAA와 GAG는 둘 다 글루탐산(재분산)을 지정하지만, 다른 아미노산(불확실성)을 지정하지 않는다.하나의 아미노산을 인코딩하는 코돈은 그들의 세 가지 위치 중 어느 하나에서나 다를 수 있다.For example, the amino acid leucine is specified by YUR or CUN (UUA, UUG, CUU, CUC, CUA, or CUG) codons (difference in the first or third position indicated using IUPAC notation), while the amino acid serine is specified by UCN or AGY (UCA, UCG, UCC, UCU, AGU, or AGC) codons (difference in the first, second, or third position).[40]이중화의 실제적인 결과는 3중 코돈의 세 번째 위치에서의 오류는 아미노산의 등가 대체에 의해 친수성 또는 친수성이 유지되기 때문에 단백질에 영향을 주지 않는 돌연변이 또는 오류만을 야기하는 것이다. 예를 들어 NN의 코돈(N = 모든 뉴클레오티드)은 수분에 대해 코딩을 하는 경향이 있다.로포비아 아미노산NCN은 크기가 작고 수질이 보통인 아미노산 잔류물을 생성하며, NAN은 평균 크기의 친수성 잔류물을 암호화한다.유전코드는 수문성을 위해 매우 잘 짜여져 있어 12개 변수(4 뉴클레오티드 x 3개 위치)의 수학적 분석(Singular Value Discolation)은 번역 없이 트리플트 뉴클레오티드 수열에서 직접 인코딩된 아미노산의 수문성을 예측하는 데 현저한 상관관계(C = 0.95)를 산출한다.[41][42]아래 표의 참고문헌에는 8개의 아미노산이 코돈의 세 번째 위치에서 돌연변이에 의해 전혀 영향을 받지 않는 반면, 위의 그림에서는 두 번째 위치에서 돌연변이가 인코딩된 아미노산의 이화학적 특성에 급격한 변화를 일으킬 가능성이 있다.그럼에도 불구하고 코돈의 첫 번째 위치의 변화는 전지구적 규모의 두 번째 위치의 변화보다 더 중요하다.[43]그 이유는 충전 반전이 특정 코돈의 첫 번째 위치에서의 돌연변이에 의해서만 발생할 수 있지만, 두 번째 위치의 변화에는 발생할 수 없기 때문일 수 있다.그러한 전하 반전은 단백질의 구조나 기능에 극적인 결과를 가져올 수 있다.이러한 측면은 이전 연구에서 크게 과소평가되었을 수 있다.[43]
코돈 사용 바이어스
코돈 사용 편향이라고도 알려진 코돈의 빈도는 종마다 다를 수 있으며, 번역의 통제에 대한 기능적 함의가 있을 수 있다.코돈은 유기체에 따라 다르다. 예를 들어 대장균에서 가장 흔한 프로라인 코돈은 CCG인 반면, 인간에서는 이것이 가장 적게 사용되는 프로라인 코돈이다.[44]
인간 게놈 코돈 빈도표 | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
|
대체유전자코드
비표준 아미노산
일부 단백질에서 비표준 아미노산은 메신저 RNA의 관련 신호 순서에 따라 표준 정지 코돈으로 대체된다.예를 들어, UGA는 셀레노시스테인, UAG는 피롤리신을 코딩할 수 있다.셀레노시스테인은 21번째 아미노산으로, 피롤리신은 22번째 아미노산으로 나타났다.[46]셀레노시스테인과 달리, 피롤리신 인코딩 UAG는 전용 아미노실-tRNA 합성효소가 참여하여 번역된다.[47]셀레노시스테인과 피롤리신 모두 같은 유기체에 존재할 수 있다.[46]유전코드는 보통 유기체에서 고정되어 있지만, 아채아 프락카리오테 아세트호알로비움 아라바티쿰은 각기 다른 성장 조건에서 20개에서 21개(피롤리신 포함)의 아미노산으로 유전코드를 확장할 수 있다.[48]
변형
원래 유전 코드는 보편적이어야 한다는 단순하고 널리 받아들여진 주장이 있었다. 즉, 유전 코드의 어떤 변화도 유기체에 치명적일 것이라는 주장이었다(크릭이 바이러스는 예외라고 진술했음에도 불구하고).이는 유전코드의 보편성을 주장하는 '냉동사고' 주장으로 알려져 있다.그러나, 1968년 유전 코드의 기원에 관한 그의 정석 논문에서, 프란시스 크릭은 여전히 모든 유기체에서 유전 코드의 보편성은 증명되지 않은 가정이었고, 어떤 경우에는 아마도 사실이 아니라고 말했다.그는 "코드는 보편적(모든 유기체에서 동일)이거나 거의 그러하다"[50]고 예측했다.첫 번째 변형은 1979년에 인간 미토콘드리아 유전자를 연구하는 연구원들에 의해 발견되었다.[51]이후 다양한 대체 미토콘드리아 코드를 포함하여 [52]많은 작은 변형이 발견되었다.[53]예를 들어 이러한 사소한 변형에는 마이코플라즈마 종의 트립토판으로서 코돈 UGA의 번역과 "CTG 쇄골"의 효모에서 루신이 아닌 세린으로서의 COG의 번역이 포함된다(예:[54][55][56] 칸디다 알비칸).바이러스는 숙주와 동일한 유전코드를 사용해야 하기 때문에 표준 유전코드를 수정하면 바이러스성 단백질 합성이나 기능을 방해할 수 있다.그러나 토티바이러스와 같은 바이러스는 숙주의 유전 암호 개조에 적응했다.[57]박테리아와 고고학에서 GUG와 UUG는 흔한 시작 코돈이다.드문 경우지만, 특정 단백질은 대체 시작 코돈을 사용할 수 있다.[52]놀랍게도, 유전자 코드의 해석의 변화는 인간의 핵 인코딩 유전자에도 존재한다.2016년 말라리아 탈수소효소의 번역을 연구한 연구자들은 이 효소를 인코딩하는 mRNA의 약 4%에서 스톱 코돈이 아미노산 트립토판과 아르기닌을 인코딩하는 데 자연적으로 이용된다는 사실을 발견했다.[58]이러한 유형의 재코딩은 높은 판독 스톱 코돈 컨텍스트에[59] 의해 유도되며 기능적 변환 읽기라고 한다.[60]
이러한 차이에도 불구하고 알려진 자연발생 코드는 모두 매우 유사하다.코딩 메커니즘은 3 베이스 코돈, tRNA, 리보솜, 단방향 판독 및 단일 코돈을 단일 아미노산으로 변환하는 모든 유기체에 대해 동일하다.[61]가장 극단적인 변화는 정지 코돈의 의미가 mRNA 내의 위치에 따라 달라지는 특정 지역에서 발생한다.3의 끝에 가까울 때 내부 위치에 있을 때 종단기 역할을 하며 콘딜로스토마 마그넘에서처럼[62] 아미노산을 코딩하거나 Euplotet에서처럼 리보솜 프레임 쉬핑을 트리거한다.[63]
유전 코드의 진화가능성 이면의 메커니즘을 포함한 유전 코드의 기원과 변화는 널리 연구되어 왔으며,[64][65] 일부 연구에서는 일부 유기체의 유전 코드를 실험적으로 진화하는 연구가 행해졌다.[66][67][68][69]
유추
유기체가 사용하는 변형 유전자 코드는 그 게놈에 암호화되어 있는 보존도가 높은 유전자를 식별하고, 코돈의 사용법을 다른 유기체의 동질 단백질에 있는 아미노산과 비교함으로써 추론할 수 있다.예를 들어, FEXER 프로그램은 어떤 아미노산이 모든 코돈에 가장 많이 정렬되어 있는지를 검색함으로써 유전코드를 주입한다.각 코돈에 대한 결과 아미노산(또는 정지 코돈) 확률은 유전자 코드 로고로 표시된다.[49]
2022년 1월 현재 유전자 코드에 대한 가장 완전한 조사는 슐기나와 에디가 코데타 도구를 이용해 25만 개의 친핵 게놈을 선별했다.이 도구는 더 큰 Pfam 데이터베이스를 가진 PEXCE와 유사한 앱프로치(approach)을 사용한다.이미 NCBI가 33개의 번역표를 제공했음에도 불구하고, 저자들은 새로운 5개의 유전자 코드 변형(tRNA 돌연변이에 의해 변형된 것)을 찾아내고 몇 가지 잘못된 주의사항을 고칠 수 있었다.[70]
기원
유전자 코드는 우리가 알고 있는 것처럼 자기복제 RNA 분자가 생명보다 앞서 있다는 한 버전에 따르면 생명의 역사의 핵심 부분이다.이것은 RNA 세계 가설이다.이 가설에서 유전 코드의 출현에 대한 어떤 모델도 세포의 주요 효소로서 리보임(RNA 효소)에서 단백질로의 전이 모델과 밀접하게 관련되어 있다.RNA 세계 가설과 일치하여, 전달 RNA 분자는 현대의 아미노산-tRNA 합성 전에 진화한 것으로 나타나므로 후자는 그 패턴에 대한 설명의 일부가 될 수 없다.[71]
무작위로 진화한 가상의 유전자 코드는 그 기원에 대한 생화학적 또는 진화적 모델에 더욱 동기를 부여한다.만약 아미노산이 트리플트 코돈에 무작위로 할당되었다면, 1.5 × 10의84 가능한 유전자 코드가 있을 것이다.[72]: 163 이 숫자는 각 품목을 한 번 이상 사용하는 64개의 빈에 21개 품목(아미노산 20개+1개 스톱)을 넣을 수 있는 방법을 계산해 찾아낸다.[73]그러나 유전자 코드에 있는 코돈 배정의 분포는 무작위적이지 않다.[74]특히 유전자 코드는 특정 아미노산 배정을 클러스터링한다.
동일한 생합성 경로를 공유하는 아미노산은 코돈에 동일한 1차 염기를 갖는 경향이 있다.이것은 더 적은 아미노산을 가진 더 단순한 초기 유전자 코드의 진화적인 유물이 될 수 있으며, 후에 더 큰 아미노산 세트를 코딩하기 위해 진화되었다.[75]그것은 또한 진화하는 동안 코돈에 또 다른 영향을 미친 강직하고 화학적인 성질을 반영할 수 있다.물리적 성질이 비슷한 아미노산도 코돈이 비슷한 경향이 있어 포인트 돌연변이와 오역으로 인한 문제를 줄여준다.[76][77][74]
비랜덤 유전 3중 코딩 방식을 고려할 때, 유전자 코드의 기원에 대한 가설이 D-아미노산에 대한 코돈의 부재, 일부 아미노산에 대한 2차 코돈 패턴, 동의어 위치의 제3위치에 대한 구속, (대신) 20개 아미노산의 작은 세트와 같은 코돈 테이블의 여러 측면을 다룰 수 있다.64에 근접하는 숫자)[78] 및 아미노산 코딩 패턴에 대한 중지 코돈 패턴의 관계
세 가지 주요 가설은 유전 코드의 기원을 다루고 있다.많은 모델은 다음 중 하나 또는 하이브리드 모델에 속한다.[79]
- 무작위 동결: 유전자 코드가 무작위로 생성되었다.예를 들어, 초기의 tRNA와 같은 리보임은 아미노산에 대해 서로 다른 친화력을 가졌을 수 있으며, 코돈은 무작위 가변성을 보이는 리보임의 다른 부분에서 생겨났다.일단 충분한 펩타이드들이 코딩되면, 유전 코드의 어떠한 중대한 무작위적인 변화도 치명적일 것이고, 따라서 그것은 "동결"[80]이 되었다.
- 스테레오케미컬 친화력: 유전 코드는 각 아미노산과 그 코돈 또는 안티코돈 사이의 높은 친화력의 결과물이다. 후자의 옵션은 이 친화력에 의해 사전 tRNA 분자가 해당 아미노산과 일치한다는 것을 암시한다.이후 진화 과정에서 이 매칭은 점차 아미노아실-tRNA 합성물에 의해 매칭으로 대체되었다.[78][81][82]
- Optimality(최적성): 유전자 코드는 초기 생성 후 계속 진화하여 현재 코드는 일부 피트니스 기능, 대개 일종의 오류 최소화 기능을 극대화한다.[78][79]
가설은 다음과 같은 다양한 시나리오를 다루었다.[83]
- 화학 원리는 아미노산과의 특정 RNA 상호작용을 관장한다.압타머를 이용한 실험에서는 일부 아미노산이 코돈에 대해 선택적 화학적 친화력을 가지고 있다는 것을 보여주었다.[84]실험 결과 8개의 아미노산 중 6개는 RNA 삼중-아미노산 연관성을 보였다.[72][82]
- 생합성 팽창.유전자 코드는 보다 단순한 초기 코드에서 "생물학적 결합체 확장" 과정을 거쳐 성장했다.원시 생명체는 새로운 아미노산(예를 들어 신진대사의 부산물로써)을 "발견"했고, 후에 이들 중 일부를 유전적 코딩의 기계에 통합시켰다.[85]과거 아미노산 사용량이 적었다는 정황증거가 많이 발견됐지만,[86] 어떤 아미노산이 어떤 순서로 코드에 들어갔는지에 대한 정확하고 상세한 가설이 논란이 되고 있다.[87][88]그러나 여러 연구에서 글리, 알라, 아스프, 발, 세르, 프로, 글루, 르우, 스르는 조기 첨가 아미노산 그룹에 속할 수 있으며, 체스, 메트, 티르, 트르, 히스, 페는 후기 첨가 아미노산 그룹에 속할 수 있다고 제시되었다.[89][90][91][92]
- 자연선택은 돌연변이의 영향을 최소화하는 유전자 코드의 코돈 배정을 이끌어냈다.[93]최근의 가설은[94] 트리플트 코돈이 트리플트 코돈(예: 쿼드러플 코돈)보다 더 긴 코드에서 유래되었다는 것을 암시한다.트리플트 디코딩보다 더 길면 코돈 중복성이 증가하며 오류 저항성이 더 높을 것이다.이 기능은 세포가 리보솜을 만들기 시작하기 전과 같이 리보솜과 같은 복잡한 번역기계가 없는 정확한 디코딩을 가능하게 할 수 있다.
- 정보 채널:정보이론적 접근방식은 유전코드를 해당 아미노산으로 번역하는 과정을 오류가 발생하기 쉬운 정보채널로 모델링한다.[95]채널에 내재된 소음(즉, 오류)은 어떻게 정보를 정확하고 효율적으로 번역하면서 소음에[96] 견딜 수 있도록 유전자 코드를 구성할 수 있는가라는 근본적인 질문을 유기체에 던진다.이러한 "금리 배분" 모델은[97] 유전 코드가 다양한 아미노산의 필요성,[98] 오류 허용[93] 및 최소 자원 비용에 대한 세 가지 상충되는 진화력의 상호 작용의 결과로 발생했음을 시사한다.코돈과 아미노산의 매핑이 무작위적이 되지 않을 때 코드가 나타난다.코드의 출현은 발생 가능한 오류에 의해 정의된 위상에 의해 관리되며 지도 색칠 문제와 관련이 있다.[99]
- 게임 이론: 신호 게임을 기반으로 하는 모델들은 게임 이론, 자연 선택, 정보 채널의 요소들을 결합한다.그러한 모델들은 첫 번째 폴리펩타이드들이 짧을 가능성이 높고 비정전 기능을 가지고 있었다는 것을 암시하기 위해 사용되어 왔다.게임 이론 모델은 RNA 끈을 세포로 조직하는 것이 유전자 코드의 "기만적인" 사용을 막기 위해서, 즉 고대의 바이러스와 동등한 바이러스가 RNA 세계를 압도하는 것을 막기 위해 필요했을 수도 있다고 제안했다.[100]
- 스톱 코돈: 번역 스톱을 위한 코돈은 유전 코드의 기원에 대한 문제에 대한 흥미로운 측면이기도 하다.정지 코돈 진화를 다루기 위한 예로서 정지 코돈은 프레임 이동 오류의 경우 가장 일찍 번역을 종료할 가능성이 높은 코돈이라고 제안되었다.[101]대조적으로, 일부 입체 화학 분자 모델은 정지 코돈의 기원을 "할당 불가능"으로 설명한다.[78]
참고 항목
참조
- ^ Turanov AA, Lobanov AV, Fomenko DE, Morrison HG, Sogin ML, Klobutcher LA, Hatfield DL, Gladyshev VN (January 2009). "Genetic code supports targeted insertion of two amino acids by one codon". Science. 323 (5911): 259–61. doi:10.1126/science.1164748. PMC 3088105. PMID 19131629.
- ^ Crick, Francis (10 July 1990). "Chapter 8: The genetic code". What Mad Pursuit: A Personal View of Scientific Discovery. Basic Books. pp. 89–101. ISBN 978-0-465-09138-6.
- ^ Yanofsky, Charles (9 March 2007). "Establishing the Triplet Nature of the Genetic Code". Cell. 128 (5): 815–818. doi:10.1016/j.cell.2007.02.029. PMID 17350564. S2CID 14249277.
- ^ Nirenberg MW, Matthaei JH (October 1961). "The dependence of cell-free protein synthesis in E. coli upon naturally occurring or synthetic polyribonucleotides". Proceedings of the National Academy of Sciences of the United States of America. 47 (10): 1588–602. Bibcode:1961PNAS...47.1588N. doi:10.1073/pnas.47.10.1588. PMC 223178. PMID 14479932.
- ^ Gardner RS, Wahba AJ, Basilio C, Miller RS, Lengyel P, Speyer JF (December 1962). "Synthetic polynucleotides and the amino acid code. VII". Proceedings of the National Academy of Sciences of the United States of America. 48 (12): 2087–94. Bibcode:1962PNAS...48.2087G. doi:10.1073/pnas.48.12.2087. PMC 221128. PMID 13946552.
- ^ Wahba AJ, Gardner RS, Basilio C, Miller RS, Speyer JF, Lengyel P (January 1963). "Synthetic polynucleotides and the amino acid code. VIII". Proceedings of the National Academy of Sciences of the United States of America. 49 (1): 116–22. Bibcode:1963PNAS...49..116W. doi:10.1073/pnas.49.1.116. PMC 300638. PMID 13998282.
- ^ "The Nobel Prize in Physiology or Medicine 1959" (Press release). The Royal Swedish Academy of Science. 1959. Retrieved 27 February 2010.
The Nobel Prize in Physiology or Medicine 1959 was awarded jointly to Severo Ochoa and Arthur Kornberg 'for their discovery of the mechanisms in the biological synthesis of ribonucleic acid and deoxyribonucleic acid'.
- ^ Nirenberg M, Leder P, Bernfield M, Brimacombe R, Trupin J, Rottman F, O'Neal C (May 1965). "RNA codewords and protein synthesis, VII. On the general nature of the RNA code". Proceedings of the National Academy of Sciences of the United States of America. 53 (5): 1161–8. Bibcode:1965PNAS...53.1161N. doi:10.1073/pnas.53.5.1161. PMC 301388. PMID 5330357.
- ^ "The Nobel Prize in Physiology or Medicine 1968" (Press release). The Royal Swedish Academy of Science. 1968. Retrieved 27 February 2010.
The Nobel Prize in Physiology or Medicine 1968 was awarded jointly to Robert W. Holley, Har Gobind Khorana and Marshall W. Nirenberg 'for their interpretation of the genetic code and its function in protein synthesis'.
- ^ Edgar B (October 2004). "The genome of bacteriophage T4: an archeological dig". Genetics. 168 (2): 575–82. doi:10.1093/genetics/168.2.575. PMC 1448817. PMID 15514035.
- ^ Budisa, Nediljko (23 December 2005). The book at the Wiley Online Library. doi:10.1002/3527607188. ISBN 9783527312436.
- ^ Kubyshkin, V.; Budisa, N. (2017). "Synthetic alienation of microbial organisms by using genetic code engineering: Why and how?". Biotechnology Journal. 12 (8): 1600097. doi:10.1002/biot.201600097. PMID 28671771.
- ^ Xie J, Schultz PG (December 2005). "Adding amino acids to the genetic repertoire". Current Opinion in Chemical Biology. 9 (6): 548–54. doi:10.1016/j.cbpa.2005.10.011. PMID 16260173.
- ^ Wang Q, Parrish AR, Wang L (March 2009). "Expanding the genetic code for biological studies". Chemistry & Biology. 16 (3): 323–36. doi:10.1016/j.chembiol.2009.03.001. PMC 2696486. PMID 19318213.
- ^ Simon M (7 January 2005). Emergent Computation: Emphasizing Bioinformatics. Springer Science & Business Media. pp. 105–106. ISBN 978-0-387-22046-8.
- ^ Hoesl, M. G.; Oehm, S.; Durkin, P.; Darmon, E.; Peil, L.; Aerni, H.-R.; Rappsilber, J.; Rinehart, J.; Leach, D.; Söll, D.; Budisa, N. (2015). "Chemical evolution of a bacterial proteome". Angewandte Chemie International Edition. 54 (34): 10030–10034. doi:10.1002/anie.201502868. PMC 4782924. PMID 26136259. NIHMSID: NIHMS711205
- ^ "First stable semisynthetic organism created KurzweilAI". www.kurzweilai.net. 3 February 2017. Retrieved 9 February 2017.
- ^ Zhang Y, Lamb BM, Feldman AW, Zhou AX, Lavergne T, Li L, Romesberg FE (February 2017). "A semisynthetic organism engineered for the stable expansion of the genetic alphabet". Proceedings of the National Academy of Sciences of the United States of America. 114 (6): 1317–1322. doi:10.1073/pnas.1616443114. PMC 5307467. PMID 28115716.
- ^ Han S, Yang A, Lee S, Lee HW, Park CB, Park HS (February 2017). "Expanding the genetic code of Mus musculus". Nature Communications. 8: 14568. Bibcode:2017NatCo...814568H. doi:10.1038/ncomms14568. PMC 5321798. PMID 28220771.
- ^ Zimmer, Carl (15 May 2019). "Scientists Created Bacteria With a Synthetic Genome. Is This Artificial Life? - In a milestone for synthetic biology, colonies of E. coli thrive with DNA constructed from scratch by humans, not nature". The New York Times. Archived from the original on 2 January 2022. Retrieved 16 May 2019.
- ^ Fredens, Julius; et al. (15 May 2019). "Total synthesis of Escherichia coli with a recoded genome". Nature. 569 (7757): 514–518. Bibcode:2019Natur.569..514F. doi:10.1038/s41586-019-1192-5. PMC 7039709. PMID 31092918. S2CID 205571025.
- ^ 호모 사피엔스 미토콘드리온, 완전한 게놈."개정된 케임브리지 참조 시퀀스(RCRS: 등록 NC_012920)", 국립 생명공학 정보 센터.2017년 12월 27일 회수
- ^ a b King RC, Mulligan P, Stansfield W (10 January 2013). A Dictionary of Genetics. OUP USA. p. 608. ISBN 978-0-19-976644-4.
- ^ Touriol C, Bornes S, Bonnal S, Audigier S, Prats H, Prats AC, Vagner S (2003). "Generation of protein isoform diversity by alternative initiation of translation at non-AUG codons". Biology of the Cell. 95 (3–4): 169–78. doi:10.1016/S0248-4900(03)00033-9. PMID 12867081.
- ^ Maloy S (29 November 2003). "How nonsense mutations got their names". Microbial Genetics Course. San Diego State University. Retrieved 10 March 2010.
- ^ 이미지에 대한 참조는 Wikimedia Commons 페이지(: Commons:파일:알 수 없는 돌연변이.svg#Reference.
- ^ Griffiths AJ, Miller JH, Suzuki DT, Lewontin RC, et al., eds. (2000). "Spontaneous mutations". An Introduction to Genetic Analysis (7th ed.). New York: W. H. Freeman. ISBN 978-0-7167-3520-5.
- ^ Freisinger E, Grollman AP, Miller H, Kisker C (April 2004). "Lesion (in)tolerance reveals insights into DNA replication fidelity". The EMBO Journal. 23 (7): 1494–505. doi:10.1038/sj.emboj.7600158. PMC 391067. PMID 15057282.
- ^ (Boillé 2006, 페이지 39) : no (
- ^ Chang JC, Kan YW (June 1979). "beta 0 thalassemia, a nonsense mutation in man". Proceedings of the National Academy of Sciences of the United States of America. 76 (6): 2886–9. Bibcode:1979PNAS...76.2886C. doi:10.1073/pnas.76.6.2886. PMC 383714. PMID 88735.
- ^ Boillée S, Vande Velde C, Cleveland DW (October 2006). "ALS: a disease of motor neurons and their nonneuronal neighbors". Neuron. 52 (1): 39–59. doi:10.1016/j.neuron.2006.09.018. PMID 17015226.
- ^ Isbrandt D, Hopwood JJ, von Figura K, Peters C (1996). "Two novel frameshift mutations causing premature stop codons in a patient with the severe form of Maroteaux-Lamy syndrome". Human Mutation. 7 (4): 361–3. doi:10.1002/(SICI)1098-1004(1996)7:4<361::AID-HUMU12>3.0.CO;2-0. PMID 8723688.
- ^ Crow JF (1993). "How much do we know about spontaneous human mutation rates?". Environmental and Molecular Mutagenesis. 21 (2): 122–9. doi:10.1002/em.2850210205. PMID 8444142. S2CID 32918971.
- ^ Lewis R (2005). Human Genetics: Concepts and Applications (6th ed.). Boston, Mass: McGraw Hill. pp. 227–228. ISBN 978-0-07-111156-0.
- ^ Sawyer SA, Parsch J, Zhang Z, Hartl DL (April 2007). "Prevalence of positive selection among nearly neutral amino acid replacements in Drosophila". Proceedings of the National Academy of Sciences of the United States of America. 104 (16): 6504–10. Bibcode:2007PNAS..104.6504S. doi:10.1073/pnas.0701572104. PMC 1871816. PMID 17409186.
- ^ Bridges KR (2002). "Malaria and the Red Cell". Harvard. Archived from the original on 27 November 2011.
- ^ Drake JW, Holland JJ (November 1999). "Mutation rates among RNA viruses". Proceedings of the National Academy of Sciences of the United States of America. 96 (24): 13910–3. Bibcode:1999PNAS...9613910D. doi:10.1073/pnas.96.24.13910. PMC 24164. PMID 10570172.
- ^ Holland J, Spindler K, Horodyski F, Grabau E, Nichol S, VandePol S (March 1982). "Rapid evolution of RNA genomes". Science. 215 (4540): 1577–85. Bibcode:1982Sci...215.1577H. doi:10.1126/science.7041255. PMID 7041255.
- ^ de Visser JA, Rozen DE (April 2006). "Clonal interference and the periodic selection of new beneficial mutations in Escherichia coli". Genetics. 172 (4): 2093–100. doi:10.1534/genetics.105.052373. PMC 1456385. PMID 16489229.
- ^ Watson, James D. (2008). Molecular Biology of the Gene. Pearson/Benjamin Cummings. ISBN 978-0-8053-9592-1. : 102–117 : 521–522
- ^ Michel-Beyerle, Maria Elisabeth (1990). Reaction centers of photosynthetic bacteria: Feldafing-II-Meeting. Springer-Verlag. ISBN 978-3-540-53420-4.
- ^ 뮐렌 G, 유반 DC (1994년)."유전자 알고리즘과 단백질 공학에서의 재귀적 앙상블 돌연변이 유발"복잡성 국제 1.
- ^ a b Fricke, Markus (2019). "Global importance of RNA secondary structures in protein coding sequences". Bioinformatics. 35 (4): 579–583. doi:10.1093/bioinformatics/bty678. PMC 7109657. PMID 30101307. S2CID 51968530.
- ^ "Codon Usage Frequency Table(chart)-Genscript". www.genscript.com. Retrieved 4 February 2022.
- ^ "Codon usage table".
- ^ a b Zhang Y, Baranov PV, Atkins JF, Gladyshev VN (May 2005). "Pyrrolysine and selenocysteine use dissimilar decoding strategies". The Journal of Biological Chemistry. 280 (21): 20740–51. doi:10.1074/jbc.M501458200. PMID 15788401.
- ^ Krzycki JA (December 2005). "The direct genetic encoding of pyrrolysine". Current Opinion in Microbiology. 8 (6): 706–12. doi:10.1016/j.mib.2005.10.009. PMID 16256420.
- ^ Prat L, Heinemann IU, Aerni HR, Rinehart J, O'Donoghue P, Söll D (December 2012). "Carbon source-dependent expansion of the genetic code in bacteria". Proceedings of the National Academy of Sciences of the United States of America. 109 (51): 21070–5. Bibcode:2012PNAS..10921070P. doi:10.1073/pnas.1218613110. PMC 3529041. PMID 23185002.
- ^ a b Dutilh BE, Jurgelenaite R, Szklarczyk R, van Hijum SA, Harhangi HR, Schmid M, de Wild B, Françoijs KJ, Stunnenberg HG, Strous M, Jetten MS, Op den Camp HJ, Huynen MA (July 2011). "FACIL: Fast and Accurate Genetic Code Inference and Logo". Bioinformatics. 27 (14): 1929–33. doi:10.1093/bioinformatics/btr316. PMC 3129529. PMID 21653513.
- ^ 프랜시스 크릭, 1968년"유전 코드의 기원"J. Mol. Biol.
- ^ Barrell BG, Bankier AT, Drouin J (1979). "A different genetic code in human mitochondria". Nature. 282 (5735): 189–194. Bibcode:1979Natur.282..189B. doi:10.1038/282189a0. PMID 226894. S2CID 4335828. ([1])
- ^ a b Elzanowski A, Ostell J (7 April 2008). "The Genetic Codes". National Center for Biotechnology Information (NCBI). Retrieved 10 March 2010.
- ^ Jukes TH, Osawa S (December 1990). "The genetic code in mitochondria and chloroplasts". Experientia. 46 (11–12): 1117–26. doi:10.1007/BF01936921. PMID 2253709. S2CID 19264964.
- ^ Fitzpatrick DA, Logue ME, Stajich JE, Butler G (1 January 2006). "A fungal phylogeny based on 42 complete genomes derived from supertree and combined gene analysis". BMC Evolutionary Biology. 6: 99. doi:10.1186/1471-2148-6-99. PMC 1679813. PMID 17121679.
- ^ Santos MA, Tuite MF (May 1995). "The CUG codon is decoded in vivo as serine and not leucine in Candida albicans". Nucleic Acids Research. 23 (9): 1481–6. doi:10.1093/nar/23.9.1481. PMC 306886. PMID 7784200.
- ^ Butler G, Rasmussen MD, Lin MF, et al. (June 2009). "Evolution of pathogenicity and sexual reproduction in eight Candida genomes". Nature. 459 (7247): 657–62. Bibcode:2009Natur.459..657B. doi:10.1038/nature08064. PMC 2834264. PMID 19465905.
- ^ Taylor DJ, Ballinger MJ, Bowman SM, Bruenn JA (2013). "Virus-host co-evolution under a modified nuclear genetic code". PeerJ. 1: e50. doi:10.7717/peerj.50. PMC 3628385. PMID 23638388.
- ^ Hofhuis J, Schueren F, Nötzel C, Lingner T, Gärtner J, Jahn O, Thoms S (2016). "The functional readthrough extension of malate dehydrogenase reveals a modification of the genetic code". Open Biol. 6 (11): 160246. doi:10.1098/rsob.160246. PMC 5133446. PMID 27881739.
- ^ Schueren F, Lingner T, George R, Hofhuis J, Gartner J, Thoms S (2014). "Peroxisomal lactate dehydrogenase is generated by translational readthrough in mammals". eLife. 3: e03640. doi:10.7554/eLife.03640. PMC 4359377. PMID 25247702.
- ^ F. Schueren und S. Thoms (2016). "Functional Translational Readthrough: A Systems Biology Perspective". PLOS Genetics. 12 (8): e1006196. doi:10.1371/journal.pgen.1006196. PMC 4973966. PMID 27490485.
- ^ Kubyshkin V, Acevedo-Rocha CG, Budisa N (February 2018). "On universal coding events in protein biogenesis". Bio Systems. 164: 16–25. doi:10.1016/j.biosystems.2017.10.004. PMID 29030023.
- ^ Heaphy SM, Mariotti M, Gladyshev VN, Atkins JF, Baranov PV (November 2016). "Novel Ciliate Genetic Code Variants Including the Reassignment of All Three Stop Codons to Sense Codons in Condylostoma magnum". Molecular Biology and Evolution. 33 (11): 2885–2889. doi:10.1093/molbev/msw166. PMC 5062323. PMID 27501944.
- ^ Lobanov AV, Heaphy SM, Turanov AA, Gerashchenko MV, Pucciarelli S, Devaraj RR, et al. (January 2017). "Position-dependent termination and widespread obligatory frameshifting in Euplotes translation". Nature Structural & Molecular Biology. 24 (1): 61–68. doi:10.1038/nsmb.3330. PMC 5295771. PMID 27870834.
- ^ Koonin EV, Novozhilov AS (February 2009). "Origin and Evolution of the Genetic Code: The Universal Enigma". IUBMB Life. 61 (2): 91–111. doi:10.1002/iub.146. PMC 3293468. PMID 19117371.
- ^ Sengupta S, Higgs PG (June 2015). "Pathways of Genetic Code Evolution in Ancient and Modern Organisms". Journal of Molecular Evolution. 80 (5–6): 229–243. Bibcode:2015JMolE..80..229S. doi:10.1007/s00239-015-9686-8. PMID 26054480. S2CID 15542587.
- ^ Xie J, Schultz PG (August 2006). "A chemical toolkit for proteins--an expanded genetic code". Nature Reviews Molecular Cell Biology. 7 (10): 775–782. doi:10.1038/nrm2005. PMID 16926858. S2CID 19385756.
- ^ Neumann H, Wang K, Davis L, Garcia-Alai M, Chin JW (March 2010). "Encoding multiple unnatural amino acids via evolution of a quadruplet-decoding ribosome". Nature. 18 (464): 441–444. doi:10.1038/nrm2005. PMID 16926858. S2CID 19385756.
- ^ Liu CC, Schultz PG (2010). "Adding new chemistries to the genetic code". Annual Review of Biochemistry. 79: 413–444. doi:10.1146/annurev.biochem.052308.105824. PMID 20307192.
- ^ Chin JW (February 2014). "Expanding and reprogramming the genetic code of cells and animals". Annual Review of Biochemistry. 83: 379–408. doi:10.1146/annurev-biochem-060713-035737. PMID 24555827.
- ^ Shulgina, Y; Eddy, SR (9 November 2021). "A computational screen for alternative genetic codes in over 250,000 genomes". eLife. 10. doi:10.7554/eLife.71402. PMC 8629427. PMID 34751130.
- ^ Ribas de Pouplana L, Turner RJ, Steer BA, Schimmel P (September 1998). "Genetic code origins: tRNAs older than their synthetases?". Proceedings of the National Academy of Sciences of the United States of America. 95 (19): 11295–300. Bibcode:1998PNAS...9511295D. doi:10.1073/pnas.95.19.11295. PMC 21636. PMID 9736730.
- ^ a b Yarus, Michael (2010). Life from an RNA World: The Ancestor Within. Harvard University Press. ISBN 978-0-674-05075-4.
- ^ "Mathematica function for # possible arrangements of items in bins? – Online Technical Discussion Groups—Wolfram Community". community.wolfram.com. Retrieved 3 February 2017.
- ^ a b Freeland SJ, Hurst LD (September 1998). "The genetic code is one in a million". Journal of Molecular Evolution. 47 (3): 238–48. Bibcode:1998JMolE..47..238F. doi:10.1007/PL00006381. PMID 9732450. S2CID 20130470.
- ^ Taylor FJ, Coates D (1989). "The code within the codons". Bio Systems. 22 (3): 177–87. doi:10.1016/0303-2647(89)90059-2. PMID 2650752.
- ^ Di Giulio M (October 1989). "The extension reached by the minimization of the polarity distances during the evolution of the genetic code". Journal of Molecular Evolution. 29 (4): 288–93. Bibcode:1989JMolE..29..288D. doi:10.1007/BF02103616. PMID 2514270. S2CID 20803686.
- ^ Wong JT (February 1980). "Role of minimization of chemical distances between amino acids in the evolution of the genetic code". Proceedings of the National Academy of Sciences of the United States of America. 77 (2): 1083–6. Bibcode:1980PNAS...77.1083W. doi:10.1073/pnas.77.2.1083. PMC 348428. PMID 6928661.
- ^ a b c d Erives A (August 2011). "A model of proto-anti-codon RNA enzymes requiring L-amino acid homochirality". Journal of Molecular Evolution. 73 (1–2): 10–22. Bibcode:2011JMolE..73...10E. doi:10.1007/s00239-011-9453-4. PMC 3223571. PMID 21779963.
- ^ a b Freeland SJ, Knight RD, Landweber LF, Hurst LD (April 2000). "Early fixation of an optimal genetic code". Molecular Biology and Evolution. 17 (4): 511–18. doi:10.1093/oxfordjournals.molbev.a026331. PMID 10742043.
- ^ Crick FH (December 1968). "The origin of the genetic code". Journal of Molecular Evolution. 38 (3): 367–79. doi:10.1016/0022-2836(68)90392-6. PMID 4887876.
- ^ Hopfield JJ (1978). "Origin of the genetic code: a testable hypothesis based on tRNA structure, sequence, and kinetic proofreading". PNAS. 75 (9): 4334–4338. Bibcode:1978PNAS...75.4334H. doi:10.1073/pnas.75.9.4334. PMC 336109. PMID 279919.
- ^ a b Yarus M, Widmann JJ, Knight R (November 2009). "RNA-amino acid binding: a stereochemical era for the genetic code". Journal of Molecular Evolution. 69 (5): 406–29. Bibcode:2009JMolE..69..406Y. doi:10.1007/s00239-009-9270-1. PMID 19795157.
- ^ Knight RD, Freeland SJ, Landweber LF (June 1999). "Selection, history and chemistry: the three faces of the genetic code" (PDF). Trends in Biochemical Sciences. 24 (6): 241–7. doi:10.1016/S0968-0004(99)01392-4. PMID 10366854.[영구적 데드링크]
- ^ Knight RD, Landweber LF (September 1998). "Rhyme or reason: RNA-arginine interactions and the genetic code". Chemistry & Biology. 5 (9): R215–20. doi:10.1016/S1074-5521(98)90001-1. PMID 9751648.
- ^ Sengupta S, Higgs PG (2015). "Pathways of genetic code evolution in ancient and modern organisms". Journal of Molecular Evolution. 80 (5–6): 229–243. Bibcode:2015JMolE..80..229S. doi:10.1007/s00239-015-9686-8. PMID 26054480. S2CID 15542587.
- ^ Brooks DJ, Fresco JR, Lesk AM, Singh M (October 2002). "Evolution of amino acid frequencies in proteins over deep time: inferred order of introduction of amino acids into the genetic code". Molecular Biology and Evolution. 19 (10): 1645–55. doi:10.1093/oxfordjournals.molbev.a003988. PMID 12270892.
- ^ Amirnovin R (May 1997). "An analysis of the metabolic theory of the origin of the genetic code". Journal of Molecular Evolution. 44 (5): 473–6. Bibcode:1997JMolE..44..473A. doi:10.1007/PL00006170. PMID 9115171. S2CID 23334860.
- ^ Ronneberg TA, Landweber LF, Freeland SJ (December 2000). "Testing a biosynthetic theory of the genetic code: fact or artifact?". Proceedings of the National Academy of Sciences of the United States of America. 97 (25): 13690–5. Bibcode:2000PNAS...9713690R. doi:10.1073/pnas.250403097. PMC 17637. PMID 11087835.
- ^ Trifonov, Edward N. (September 2009). "The origin of the genetic code and of the earliest oligopeptides". Research in Microbiology. 160 (7): 481–486. doi:10.1016/j.resmic.2009.05.004. PMID 19524038.
- ^ Higgs, Paul G.; Pudritz, Ralph E. (June 2009). "A Thermodynamic Basis for Prebiotic Amino Acid Synthesis and the Nature of the First Genetic Code". Astrobiology. 9 (5): 483–490. arXiv:0904.0402. Bibcode:2009AsBio...9..483H. doi:10.1089/ast.2008.0280. ISSN 1531-1074. PMID 19566427. S2CID 9039622.
- ^ Chaliotis, Anargyros; Vlastaridis, Panayotis; Mossialos, Dimitris; Ibba, Michael; Becker, Hubert D.; Stathopoulos, Constantinos; Amoutzias, Grigorios D. (17 February 2017). "The complex evolutionary history of aminoacyl-tRNA synthetases". Nucleic Acids Research. 45 (3): 1059–1068. doi:10.1093/nar/gkw1182. ISSN 0305-1048. PMC 5388404. PMID 28180287.
- ^ Ntountoumi, Chrysa; Vlastaridis, Panayotis; Mossialos, Dimitris; Stathopoulos, Constantinos; Iliopoulos, Ioannis; Promponas, Vasilios; Oliver, Stephen G; Amoutzias, Grigoris D (4 November 2019). "Low complexity regions in the proteins of prokaryotes perform important functional roles and are highly conserved". Nucleic Acids Research. 47 (19): 9998–10009. doi:10.1093/nar/gkz730. ISSN 0305-1048. PMC 6821194. PMID 31504783.
- ^ a b Freeland SJ, Wu T, Keulmann N (October 2003). "The case for an error minimizing standard genetic code". Origins of Life and Evolution of the Biosphere. 33 (4–5): 457–77. Bibcode:2003OLEB...33..457F. doi:10.1023/A:1025771327614. PMID 14604186. S2CID 18823745.
- ^ Baranov PV, Venin M, Provan G (2009). Gemmell NJ (ed.). "Codon size reduction as the origin of the triplet genetic code". PLOS ONE. 4 (5): e5708. Bibcode:2009PLoSO...4.5708B. doi:10.1371/journal.pone.0005708. PMC 2682656. PMID 19479032.
- ^ Tlusty T (November 2007). "A model for the emergence of the genetic code as a transition in a noisy information channel". Journal of Theoretical Biology. 249 (2): 331–42. arXiv:1007.4122. Bibcode:2007JThBi.249..331T. doi:10.1016/j.jtbi.2007.07.029. PMID 17826800. S2CID 12206140.
- ^ Sonneborn TM (1965). Bryson V, Vogel H (eds.). Evolving genes and proteins. New York: Academic Press. pp. 377–397.
- ^ Tlusty T (February 2008). "Rate-distortion scenario for the emergence and evolution of noisy molecular codes". Physical Review Letters. 100 (4): 048101. arXiv:1007.4149. Bibcode:2008PhRvL.100d8101T. doi:10.1103/PhysRevLett.100.048101. PMID 18352335. S2CID 12246664.
- ^ Sella G, Ardell DH (September 2006). "The coevolution of genes and genetic codes: Crick's frozen accident revisited". Journal of Molecular Evolution. 63 (3): 297–313. Bibcode:2006JMolE..63..297S. doi:10.1007/s00239-004-0176-7. PMID 16838217. S2CID 1260806.
- ^ Tlusty T (September 2010). "A colorful origin for the genetic code: information theory, statistical mechanics and the emergence of molecular codes". Physics of Life Reviews. 7 (3): 362–76. arXiv:1007.3906. Bibcode:2010PhLRv...7..362T. doi:10.1016/j.plrev.2010.06.002. PMID 20558115. S2CID 1845965.
- ^ Jee J, Sundstrom A, Massey SE, Mishra B (November 2013). "What can information-asymmetric games tell us about the context of Crick's 'frozen accident'?". Journal of the Royal Society, Interface. 10 (88): 20130614. doi:10.1098/rsif.2013.0614. PMC 3785830. PMID 23985735.
- ^ Itzkovitz S, Alon U (2007). "The genetic code is nearly optimal for allowing additional information within protein-coding sequences". Genome Research. 17 (4): 405–412. doi:10.1101/gr.5987307. PMC 1832087. PMID 17293451.
추가 읽기
- Griffiths AJ, Miller JH, Suzuki DT, Lewontin RC, Gilbert WM (1999). An Introduction to genetic analysis (7th ed.). San Francisco: W.H. Freeman. ISBN 978-0-7167-3771-1.
- Alberts B, Johnson A, Lewis J, Raff M, Roberts K, Walter P (2002). Molecular biology of the cell (4th ed.). New York: Garland Science. ISBN 978-0-8153-3218-3.
- Lodish HF, Berk A, Zipursky SL, Matsudaira P, Baltimore D, Darnell JE (2000). Molecular cell biology (4th ed.). San Francisco: W.H. Freeman. ISBN 978-0-7167-3706-3.
- Caskey CT, Leder P (April 2014). "The RNA code: nature's Rosetta Stone". Proceedings of the National Academy of Sciences of the United States of America. 111 (16): 5758–9. Bibcode:2014PNAS..111.5758C. doi:10.1073/pnas.1404819111. PMC 4000803. PMID 24756939.
외부 링크
| 위키미디어 커먼즈에는 유전자 코드와 관련된 미디어가 있다. |
- 유전자 코드: 유전자 코드 표
- Codon Usage Database - 많은 유기체에 대한 Codon 주파수 표
- 유전암호 해독의 역사