핵산 유사체
Nucleic acid analogue핵산 유사체는 자연적으로 발생하는 RNA 및 DNA와 유사한 (구조적으로 유사한) 화합물이며, 의학 및 분자 생물학 연구에 사용됩니다.핵산은 세 부분으로 구성된 뉴클레오티드의 사슬이며, 인산염 골격, 펜토오스 당, 리보오스 또는 디옥시리보스, 그리고 네 개의 핵염기 중 하나로 구성됩니다.아날로그는 이들 중 하나를 [1]변경할 수 있습니다.일반적으로 아날로그 핵염기는 다른 염기쌍과 염기적층 특성을 부여합니다.예를 들어, 4개의 표준 염기와 모두 쌍을 이룰 수 있는 범용 염기와 사슬의 특성에 영향을 미치는 PNA와 같은 인산-당 골격 유사체(PNA는 3중 [2]나선을 형성할 수도 있다)가 있다.핵산 유사체들은 또한 제노 핵산이라고도 불리며 대체 생화학에 기초한 새로운 형태의 생명체 설계인 제노 생물학의 주요 기둥 중 하나를 나타냅니다.
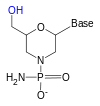
인공핵산에는 펩타이드핵산(PNA), 모르포리노 및 록드핵산(LNA), 글리콜핵산(GNA), 트레오스핵산(TNA) 및 헥시톨핵산(HNA)이 포함된다.이들 각각은 분자의 골격에 대한 변화에 의해 자연적으로 발생하는 DNA 또는 RNA와 구별된다.
2014년 5월, 연구진은 박테리아 DNA에 2개의 새로운 인공 뉴클레오티드를 성공적으로 도입했으며, 배양 배지에 개별 인공 뉴클레오티드를 포함시킴으로써 박테리아를 24번 통과할 수 있었다고 발표했다.; 그들은 mRNA나 인공 뉴클레오티드를 사용할 수 있는 단백질을 생성하지 않았다.인공 뉴클레오티드는 2개의 융합 방향족 고리를 특징으로 한다.
약
몇 가지 뉴클레오시드 유사체가 항바이러스제 또는 항암제로 사용된다.바이러스성 중합효소는 이러한 화합물을 비표준 염기와 결합시킨다.이 화합물들은 세포에서 뉴클레오티드로 전환됨으로써 활성화되고, 하전된 뉴클레오티드가 세포막을 쉽게 통과할 수 없기 때문에 뉴클레오시드로 투여된다.
분자생물학
핵산 유사체는 분자생물학에서 다음과 같은 목적으로 사용된다.생명의 기원을 시나리오에 대한 조사: 다른 전후로 실험하면서원들은 DNA와 RNA의 삶의 사용 시간의 경과에 따라 장점 때문에, 또는 그들은 임의의 기회에 의해서 선택되었다. 선정되었다 그 질문에 대답하기;[3] 도구 특별한 시퀀스를 감지할 수 있는:XNA를 광범위한 태그를 붙이는 것이 사용될 수 없듯이 노력한다. 의효소 DNA나 RNA, XNA는 기질-XNA에 작용하는 높은 특이성과 정확도가 DNA와 RNA구성 요소;[4]은 능력과ligate DNA나 RNA, 다른 XNA분자 RNAribozymes의와 비슷한 행동이 하려고 하는데 보여 주고 있다[3]RNA가수 분해 저항성이 있는 공구, 메커니즘이 효소에 의해 사용된 연구, 인만큼 필요하다.vesti핵산의 구조적 특징의 결합.
백본 유사점
내가수분해성 RNA-아날로그
인산염 연결 3' 히드록시기(RNA가 불안정하여 안정적으로 사용 또는 합성할 수 없음)와 반응하는 리보스의 2' 히드록시기(hydroxy group)를 극복하기 위해 리보스 유사체를 사용한다.가장 일반적인 RNA 유사체는 2'-O-메틸 치환 RNA, 잠긴 핵산(LNA) 또는 브리지드 핵산(BNA), 모르포리노 [5][6]및 펩타이드 핵산(PNA)이다.이들 올리고뉴클레오티드는 다른 백본당 또는 PNA의 경우 리보오스 인산 대신 아미노산 잔류물을 가지고 있지만 왓슨과 크릭의 쌍에 따라 RNA 또는 DNA에 결합하지만 뉴클레아제 활성에 면역이 된다.이들은 효소적으로 합성될 수 없으며, 포스포라마이다이트 전략 또는 PNA의 경우 펩타이드 합성 방법을 통해서만 합성될 수 있다.
도구로서 사용되는 기타 주목할 만한 유사점
디데옥시뉴클레오티드는 염기서열분석에 사용된다.이러한 뉴클레오시드 삼인산염은 일반적으로 DNA에 존재하는 3' 하이드록실기가 없으므로 다음 염기와 결합할 수 없는 비 카논 당인 디데옥시리보스를 가지고 있다.3' 히드록실기의 결핍은 DNA 중합효소가 디옥시리보뉴클레오티드를 일반 디옥시리보뉴클레오티드로 오인하여 연쇄반응을 종료시킨다.3' 수산화기가 없고 아데노신을 모방하는 또 다른 사슬 종단 유사체는 코디세핀이라고 불린다.코디세핀은 RNA 복제를 목표로 하는 항암제이다.염기서열 분석의 또 다른 유사체는 핵염기 유사체인 7-deaza-GTP이며, CG 리치 영역의 염기서열에 사용됩니다. 대신 7-deaza-ATP는 항생제인 투베르시딘이라고 합니다.
RNA 세계의 전구체
RNA는 첫 번째 핵산이 되기에는 너무 복잡할 수 있기 때문에, RNA의 세계 이전에는 TNA, GN, PNA와 같이 골격이 다른 몇 가지 단순한 핵산이 첫 번째 핵산의 후보로 제시되었다.
기본 아날로그
핵염기 구조와 명명법
자연적으로 발생하는 염기는 그 구조에 따라 두 가지 등급으로 나눌 수 있다.
인공 뉴클레오티드(d5SICS UBP 및 dNaM UBP라는 이름의 비자연 염기쌍(UBP))가 세균 DNA에 삽입되었지만 이러한 유전자들은 mRNA를 템플릿화하거나 단백질 합성을 유도하지 않았다.인공 뉴클레오티드는 천연(dG–dC) 염기쌍을 [7][8][9]모방한 (d5SICS–dNaM) 복합체를 형성하는 두 개의 융합 방향족 고리를 특징으로 했다.
뮤타겐스
가장 일반적인 염기 유사체 중 하나는 돌연변이원성 뉴클레오티드 아날로그 BrdU에서 발견되는 이상 염기인 5-브로모우라실(5BU)이다.5-브로모우라실을 포함하는 뉴클레오티드가 DNA에 통합될 때, 그것은 아데닌과 짝을 이룰 가능성이 가장 높지만, 다른 핵염기인 구아닌과 짝을 이루는 다른 이성질체로 자발적으로 이동할 수 있다.만약 이것이 DNA 복제 중에 일어난다면, 구아닌은 반대쪽 염기 유사체로 삽입될 것이고, 다음 DNA 복제에서 구아닌은 시토신과 짝을 이룰 것이다.이것은 하나의 염기쌍의 DNA, 특히 전이 돌연변이를 일으킨다.
또한 HNO2, 즉 아질산은 DNA 복제 및 비복제에 작용하는 강력한 돌연변이 물질이다.그것은 아데닌, 구아닌, 시토신의 아미노기 탈아미노화를 일으킬 수 있다.아데닌은 티민 대신 시토신에 염기쌍을 이루는 하이포산틴과 탈아미노화된다.시토신은 구아닌 대신 아데닌과 염기쌍을 이루는 우라실로 탈아미노화된다.구아닌의 탈아미노화는 돌연변이를 유발하지 않는다.아질산 유도 돌연변이도 아질산을 사용하여 야생형으로 변이하도록 유도된다.
형광체
일반적으로 불소포자(로다민 또는 플루오레세인)는 유연한 팔을 통해 설탕(파라)에 연결된 고리에 연결되며, 아마도 나선의 주요 홈에서 돌출되어 있을 것이다.Taq 중합효소에 의한 플로로포어와 같은 부피가 큰 부가물에 연결된 뉴클레오티드의 낮은 처리성으로 인해, 배열은 일반적으로 팔이 있는 뉴클레오티드를 사용하여 복사되고 나중에 반응성 불소포(간접 라벨링)와 결합됩니다.
- 아민반응성 : 아미노알릴뉴클레오티드는 시아닌 또는 알렉사플루오르염료와 같은 아미노반응성 염료와 반응하는 링커에 1차 아민기를 포함하고 있으며, 이들은 숙시니미딜에스테르(NHS)와 같은 반응성 이탈기를 포함하고 있다. (염기쌍성 아미노기는 영향을 받지 않는다.)
- 티올 반응성: 뉴클레오티드를 포함하는 티올은 말레이미드와 같은 반응성 이탈 그룹에 연결된 불소 포자와 반응합니다.
- 비오틴 연결 뉴클레오티드는 동일한 간접 라벨링 원리(+형광 스트렙타비딘)에 의존하며 Affymetrix DNA칩에 사용된다.
불소 세포는 의학과 생화학에서 다양한 용도를 발견합니다.
형광염기유사체
가장 일반적으로 사용되고 상업적으로 이용 가능한 형광염기 아날로그인 2-아미노푸린(2-AP)은 [10]핵산에 통합될 때 상당히 감소(약 100배지만 염기서열에 크게 의존함)되는 고형광 양자수율(0.68)을 가지고 있다.주변 환경에 대한 2-AP의 방출 민감도는 3-MI, 6-MI, 6-MAP,[11] pyrrolo-dC([13]상업적으로도 이용 가능),[12] pyrrolo-dC의 변형 및 개선된 유도체, fran-modified[14] base 및 기타 많은 형광염기 유사체(최근 [15][16][17][18][19]리뷰 참조)와 같다.미세 환경에 대한 이러한 민감도는 DNA와 RNA 내 구조 및 역학, DNA-단백질 상호작용의 역학 및 동력학, DNA 내 전자 전달 등의 연구에 사용되어 왔다.
주변 환경에 거의 둔감한 형광 양자 수율을 가진 형광 염기 유사체의 새로 개발되고 매우 흥미로운 그룹은 삼환식 시토신족이다. 1,3-디아자-2-옥소페노티아진, tC는 단일 및 이중 스트랜드 모두에서 약 0.2의 형광 양자 수율을 가진다.둥그스[20][21]또한 tC라고 불리는O tC의 옥소호몰로그(1,3-디아자-2-옥소페녹사진)는 이중가닥 시스템에서 0.[22]2의 양자수율을 가진다.그러나 단일 가닥의 주변 염기(양자 수율 0.14–0.41)에 다소 민감하다.이러한 염기성 유사체의 높고 안정적인 양자 수율은 매우 밝으며, 우수한 염기성 유사 특성(DNA 구조 및 방해되지 않은 옆의 안정성을 남김)과 결합하여, 다른 형광 염기성 유사체의 정확도가 낮은 영역인 형광 이방성 및 FLET 측정에 특히 유용합니다.또, 동일 계열의 시토신 유사체에서는, FLET-Acceptor 염기 유사체 tC가nitro [23]개발되고 있다.이것은 FLET 기증자로서O tC와 함께 지금까지 개발된 최초의 핵산염기 아날로그 FLET 쌍을 구성한다.예를 들어, tC군은 중합효소 DNA 결합 및 DNA 중합 메커니즘과 관련된 연구에 사용되어 왔다.
자연 비규격 기저
세포에는 여러 개의 비표준 염기가 존재한다: DNA의 CpG 섬(종종 메틸화), 모든 진핵생물 mRNA(메틸-7-구아노신으로 캡) 및 여러 개의 rRNA 염기(메틸화).종종 tRNA는 특히 안티코돈 내/근처에서 tRNA 또는 염기쌍을 개선하기 위해 전사 후 고도로 변형된다. 이노신은 C, U, 심지어 A와 염기쌍을 형성할 수 있는 반면 티오우리딘(A와 함께)은 유라실(푸린과 [24]함께)보다 특이적이다.다른 일반적인 tRNA 염기 변형으로는 의사우리딘(TΩC 루프에 이름을 붙임), 디히드로우리딘(방향족이지 않아 쌓이지 않음), 큐우오신, 와이오신 등이 있다.그럼에도 불구하고 이것들은 모두 정상 염기에 대한 변형이며 중합효소에 의해 배치되지 않는다.[24]
베이스 페어링
표준 염기는 글리코시드 결합에서 가장 멀리 떨어진 질소 원자를 둘러싼 탄소 위에 카르보닐기 또는 아민기를 가질 수 있으며, 이를 통해 수소 결합(케톤과 푸린, 피리미딘과 푸린)을 통해 염기쌍(왓슨-크릭 염기쌍)을 형성할 수 있다.아데닌과 2-아미노아데닌은 1/2개의 아민기를 가지며, 티민은 2개의 카르보닐기를 가지며, 시토신과 구아닌은 혼합 아민과 카르보닐(서로 반전)이다.
| 내추럴 베이스 페어 | |
|---|---|
 |  |
| GC 염기쌍: 푸린카르보닐/아민은 피리미딘아민/카르보닐과 3분자간 수소결합을 형성한다. | AT염기쌍:푸린아민/-은 피리미딘카르보닐/카르보닐과 2개의 분자간 수소결합을 형성한다. |
왜 4개의 뉴클레오티드가 존재하는지에 대한 정확한 이유는 논의되고 있지만, 사용되지 않는 몇 가지 가능성이 있다.또한 아데닌은 염기쌍을 위한 가장 안정적인 선택이 아닙니다. 시아노파지 S-2L 디아미노퓨린(DAP)은 아데닌 대신 사용됩니다(숙주 회피).[25]디아미노푸린 염기쌍은 아데닌과 동일하지만 위치 2에 아민기가 3개의 분자 내 수소 결합을 형성하여 두 가지 염기쌍 사이의 주요 차이를 제거한다(약점:A-T 및 Strong:C-G).이러한 개선된 안정성은 이러한 차이에 의존하는 단백질 결합 상호작용에 영향을 미친다.기타 조합은 다음과 같습니다.
- 표준 구아닌 및 시토신에 비해 아민과 케톤이 반전된 이소구아닌 및 이소시토신(아마도 호변이체로 사용되지 않지만, isoC 및 isoG는 4개의 표준 [26]염기의 존재에서도 PCR로 올바르게 증폭될 수 있음)
- 디아미노피리미딘과 크산틴으로 2-글루아데닌과 티민처럼 결합하지만 역구조(크산틴은 탈아미네이션 생성물로서 사용되지 않음)
| 미사용 베이스 페어 배치 | ||
|---|---|---|
 | 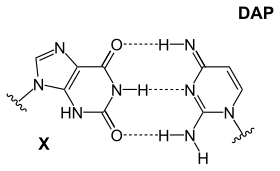 |  |
| DAP-T염기:푸린아민/아민은 피리미딘케톤/케톤과 3분자간 수소결합을 형성한다. | X-DAP 염기: 퓨린케톤/케톤은 피리미딘아민/아민과 3분자간 수소결합을 형성한다. | iG-iC염기:푸린아민/케톤은 피리미딘케톤/아민과 3분자간 수소결합을 형성한다. |
그러나 수소 결합을 통해 염기가 쌍을 이루지 않는 경우에도 올바른 DNA 구조가 형성될 수 있다. 즉, 연구 결과 티민 유사체 2,4-디플루오로톨루엔(F) 또는 아데닌 유사체 4-메틸벤츠이미다졸(Z)[27]과 같이 DNA 이소스테레스(같은 수의 원자를 가진 아날로그)를 사용한 것으로 나타났다.대체 소수성 쌍은 이소퀴놀린과 피롤로[2,3-b]피리딘일[28] 수 있다.
기타 주목할 만한 베이스 페어:
- 2-아미노-6-(2-티에닐) 푸린 및 피롤-2-카르발알데히드 염기쌍과 [29]같은 여러 형광염기도 만들어졌다.
- 중앙 구리 이온에 대한 사각 평면 배위를 통해 피리딘-2,6-디카르복실레이트(삼등배위자)와 [30]피리딘(단일배위자)의 쌍을 이루는 금속배위염기.
- 범용 염기는 다른 염기와 무차별적으로 결합할 수 있지만 일반적으로 배열의 용해 온도를 상당히 낮춥니다. 예를 들어 2'-디옥시이노신(하이포잔틴 디옥시뉴클레오티드) 유도체, 니트로아졸 유사체 및 소수성 방향족 비수소 결합 염기(강적층 효과)가 있습니다.이들은 개념 증명으로 사용되며 일반적으로 퇴화 프라이머(프라이머의 혼합물)에서는 사용되지 않습니다.
- xDNA를 고려할 때 가능한 염기쌍의 수는 두 배로 증가합니다.xDNA는 벤젠 고리가 첨가된 확장된 염기를 포함하고 있으며, 이는 표준 염기와 쌍을 이루며, 4개의 가능한 염기쌍(8개의 염기:xA-T,xT-A,xC-G,xG-C, 사용하지 않는 배열이 사용되는 경우 16개의 염기)을 생성할 수 있습니다.벤젠 첨가 염기의 또 다른 형태는 yDNA로,[31] 벤젠에 의해 염기가 넓어집니다.
| 특수 특성을 가진 새로운 베이스 페어 | ||
|---|---|---|
 |  |  |
| A F-Z염기: 메틸벤즈이미다졸은 톨루엔 F/F와 분자간 수소결합을 형성하지 않는다. | S-Pa 염기: 푸린티에닐/아민은 피롤-/카르발알데히드와 3개의 분자간 수소 결합을 형성한다. | xA-T 베이스: A-T와 동일한 본딩 |
금속 베이스 페어
금속 염기쌍화에서 왓슨-크릭 수소결합은 배위자로 작용하는 뉴클레오시드와 금속이온의 상호작용으로 치환된다.중심 금속 원자 주위에 두 개의 이등분 뉴클레오사이드를 가진 이중화 형성을 허용하는 금속의 가능한 기하학적 구조는 사면체, 십이면체 및 정사각형 평면이다.금속이온에 의한 관여를 수반하는 천연 핵염기로부터의 비표준 염기쌍의 형성 및 왓슨-크릭 염기쌍의 일부인 수소원자를 금속이온에 [32]의한 교환에 의해 DNA와의 금속복합화가 일어날 수 있다.금속 이온을 DNA 이중체에 도입하면 잠재적인 [33]자성과 전도성이 있을 [34]뿐만 아니라 [35]안정성이 향상되는 것으로 나타났습니다.
금속 착화 현상은 천연 핵염기 사이에서 일어나는 것으로 나타났다.잘 문서화된 예는 T-Hg-T의 형성이며, 이는 Hg에 의해2+ 결합되어 연결된 금속-염기 쌍을 형성하는 [36]두 개의 탈양성자 티민 핵염기를 포함한다.이 모티브는 [37]듀플렉스형성보다 스트랜드 내 헤어핀형성처리를 선호하기 때문에 듀플렉스에 쌓인 Hg를2+ 수용하지 않습니다.듀플렉스 내에서 서로 마주보고 있는2개의 티마인은 듀플렉스 내에서 Watson-Crick 베이스쌍을 형성하지 않습니다.이는 Watson-Crick 베이스쌍의 불일치가 금속 베이스쌍의 형성에 의해 안정화되는 예입니다.천연 핵염기로 착화되는 금속의 또 다른 예는 높은 pH에서 A-Zn-T와 G-Zn-C의 형성이며+2, Co와+2 Ni도 이러한 착체를 형성한다.이것들은 왓슨-크릭 염기쌍으로 2가의 양이온이 핵염기에 맞춰져 있다.정확한 구속력은 [38]논의되고 있다.
금속 염기쌍으로 사용하기 위해 다양한 인공 핵산염기들이 개발되어 왔다.이러한 변형된 핵염기는 특정 금속에 최적화될 수 있는 조정 가능한 전자 특성, 크기 및 결합 친화성을 나타냅니다.예를 들어 피리딘-2,6-디카르본산염으로 수식된 뉴클레오시드는 Cu에 단단히2+ 결합하는 반면 다른 2가 이온은 느슨하게 결합되어 있다.이 선택에는 삼등분 문자가 기여합니다.구리 위의 네 번째 배위 부위는 반대로 배열된 피리딘 핵염기에 [39]의해 포화된다.비대칭 금속 베이스 페어링 시스템은 Watson-Crick 베이스 페어와 직교합니다.인공핵산염기의 또 다른 예는 DNA 이중체 내에서 Cu와 결합할2+ 수 있는 히드록시피리돈 핵산염기이다.5개의 연속된 구리-히드록시피리돈 염기쌍이 이중 가닥에 통합되었고, 양 끝에는 오직 하나의 천연 핵염기만이 양옆에 있었다.EPR 데이터에 따르면 구리 중심 간 거리는 3.7 ± 0.1Ω으로 추정되며, 자연 B형 DNA 이중성은 약간 더 크다(3.4Ω).[40]DNA 듀플렉스 안에 금속 이온을 쌓는 것의 매력은 나노경 자체 조립 금속 와이어를 얻는다는 것입니다. 하지만 이것은 아직 실현되지 않았습니다.
부자연스러운 베이스 페어(UBP)
부자연 염기쌍(UBP)은 실험실에서 생성되고 자연에서는 발생하지 않는 DNA의 설계 서브유닛(또는 핵염기)이다.2012년, 캘리포니아 샌디에고에 있는 스크립스 연구소의 화학 생물학자인 플로이드 롬스버그가 이끄는 미국 과학자 그룹은 그의 팀이 부자연스러운 [41]염기쌍을 설계했다고 발표했다.두 개의 새로운 인공 뉴클레오티드 또는 부자연스러운 염기쌍(UBP)은 d5SICS와 dNaM으로 명명되었다.좀 더 기술적으로, 소수성 핵산염기를 가진 이러한 인공 뉴클레오티드는 DNA에서 [9][42](d5SICS–dNaM) 복합체 또는 염기쌍을 형성하는 두 개의 융합 방향족 고리를 특징으로 한다.2014년 스크립스 연구소의 같은 팀은 자연 T-A와 C-G 염기쌍을 포함하는 플라스미드로 알려진 원형 DNA를 UBP 롬스버그의 연구소가 설계한 가장 뛰어난 성능의 UBP Romesberg의 실험실과 합성하여 부자연스러운 복제를 성공적으로 수행한 일반적인 박테리아 대장균의 세포에 삽입했다고 보고했습니다.기본 쌍을 여러 [43]세대에 걸쳐 만듭니다.이것은 살아있는 유기체가 확장된 유전자 코드를 따라 다음 [9][44]세대에 전달되는 최초의 알려진 사례이다.이는 부분적으로 d5SICSTP와 dNaMTP의 트리포스페이트를 대장균에 [9]효율적으로 수입하는 뉴클레오티드 삼인산 트랜스포터를 발현하는 지지 조류 유전자를 추가함으로써 달성되었다.그런 다음 자연 세균 복제 경로는 d5SICS–dNaM을 포함하는 플라스미드를 정확하게 복제하기 위해 이들을 사용한다.
세 번째 염기쌍의 성공적인 통합은 DNA에 의해 암호화될 수 있는 아미노산의 수를 기존의 20개의 아미노산에서 이론적으로 가능한 172개로 크게 확장함으로써 생물들이 새로운 [43]단백질을 생산할 수 있는 가능성을 확장하는 목표를 향한 중요한 돌파구이다.이전에, DNA의 인공 끈은 어떤 것도 암호화하지 않았지만, 과학자들은 그것들이 산업적 또는 [45]의학적 용도를 가질 수 있는 새로운 단백질을 생산하도록 설계될 수 있다고 추측했다.부자연스러운 염기쌍을 포함하는 DNA의 전사 및 대응하는 mRNA의 번역이 실제로 최근에 이루어졌다.2017년 11월, 박테리아 DNA에 두 개의 추가 핵산염기를 처음 도입한 스크립스 연구소의 같은 팀은 그러한 DNA를 사용하여 단백질을 만들 수 있는 반합성 대장균을 구축했다고 보고했다.그것의 DNA는 6개의 서로 다른 핵염기, 즉 4개의 표준염기와 2개의 인공첨가물, dNaM과 dTPT3를 포함하고 있었다.또한, 이 박테리아는 두 개의 새로운 코돈에 포함된 두 개의 대응하는 추가적인 RNA 염기를 가지고 있었고, 이러한 새로운 코돈을 인식하는 추가적인 tRNA와 추가적인 아미노산을 가지고 있었으며, 박테리아는 "자연스럽지 않은"[46][47] 단백질을 합성할 수 있었다.
또 다른 UBP 시연은 히라오 이치로(ira ichiro一郞)의 일본 리켄(RIKEN) 연구소에 의해 이루어졌다.2002년 그들은 비표준 아미노산을 단백질에 [48]부위 특이적으로 결합하기 위해 2-아미노-8-(2-티에닐) 푸린(s)과 피리딘-2-온(y) 사이에 시험관 내 기능을 하는 부자연스러운 염기쌍을 개발했다.2006년에는 복제 및 [49]전사를 위한 세 번째 염기쌍으로 7-(2-티에닐)이미다조[4,5-b]피리딘(Ds)과 피롤-2-카르발알데히드(Pa)를 생성했다.그 후 PCR [50][51]증폭에서 Ds와 4-[3-(6-아미노헥사나미도)-1-프로피닐]-2-니트로피롤(Px)을 고충실성 쌍으로 발견했다.2013년, 그들은 Ds-Px 쌍을 시험관내 선택(SELEX)에 의한 DNA 압타머 생성에 적용하고 유전자 알파벳 확장이 표적 [52]단백질에 대한 DNA 압타머 친화력을 유의하게 증가시킨다는 것을 입증했다.
직교계
이론적으로나 실험적으로나 세포 유전 물질에 의존하지 않는 세포 내부의 직교 시스템을 구현하여 부호화 [54]전위의 증가를 가능하게 하는 완전히 안전한 [53]시스템을 만들 수 있는 가능성이 제안되고 연구되었다.여러 그룹이 서로 다른 측면에 초점을 맞추고 있습니다.
- 위에서 설명한 바와 같이 새로운 백본과 베이스 페어
- 일반적으로 T7 RNA 중합효소로부터[55] 시작되는 XNA(Xeno Nuclear Acid) 인공 복제/전사 중합효소
- 리보솜(변화된 반샤인-달가르노 시퀀스를 가진 16S 시퀀스, 일치하는 변화된 샤인-달가르노 [56]시퀀스를 가진 직교 mRNA만 번역 가능)
- 비천연 아미노산을 코드하는 새로운 tRNA.'확장 유전자 코드
「 」를 참조해 주세요.
레퍼런스
- ^ Singer E (July 19, 2015). "Chemists Invent New Letters for Nature's Genetic Alphabet". Wired. Retrieved July 20, 2015.
- ^ Petersson B, Nielsen BB, Rasmussen H, Larsen IK, Gajhede M, Nielsen PE, Kastrup JS (February 2005). "Crystal structure of a partly self-complementary peptide nucleic acid (PNA) oligomer showing a duplex-triplex network". Journal of the American Chemical Society. 127 (5): 1424–30. doi:10.1021/ja0458726. PMID 15686374.
- ^ a b Taylor AI, Pinheiro VB, Smola MJ, Morgunov AS, Peak-Chew S, Cozens C, Weeks KM, Herdewijn P, Holliger P (February 2015). "Catalysts from synthetic genetic polymers". Nature. 518 (7539): 427–30. Bibcode:2015Natur.518..427T. doi:10.1038/nature13982. PMC 4336857. PMID 25470036.
- ^ Wang Q, Chen L, Long Y, Tian H, Wu J (2013). "Molecular beacons of xeno-nucleic acid for detecting nucleic acid". Theranostics. 3 (6): 395–408. doi:10.7150/thno.5935. PMC 3677410. PMID 23781286.
- ^ Summerton J, Weller D (June 1997). "Morpholino antisense oligomers: design, preparation, and properties". Antisense & Nucleic Acid Drug Development. 7 (3): 187–95. doi:10.1089/oli.1.1997.7.187. PMID 9212909. S2CID 19372403.
- ^ Summerton J (December 1999). "Morpholino antisense oligomers: the case for an RNase H-independent structural type". Biochimica et Biophysica Acta (BBA) - Gene Structure and Expression. 1489 (1): 141–58. doi:10.1016/s0167-4781(99)00150-5. PMID 10807004.
- ^ Pollack A (May 7, 2014). "Researchers Report Breakthrough in Creating Artificial Genetic Code". New York Times. Retrieved May 7, 2014.
- ^ Callaway E (May 7, 2014). "First life with 'alien' DNA". Nature. doi:10.1038/nature.2014.15179. S2CID 86967999.
- ^ a b c d Malyshev DA, Dhami K, Lavergne T, Chen T, Dai N, Foster JM, Corrêa IR, Romesberg FE (May 2014). "A semi-synthetic organism with an expanded genetic alphabet". Nature. 509 (7500): 385–88. Bibcode:2014Natur.509..385M. doi:10.1038/nature13314. PMC 4058825. PMID 24805238.
- ^ Ward DC, Reich E, Stryer L (March 1969). "Fluorescence studies of nucleotides and polynucleotides. I. Formycin, 2-aminopurine riboside, 2,6-diaminopurine riboside, and their derivatives". The Journal of Biological Chemistry. 244 (5): 1228–37. doi:10.1016/S0021-9258(18)91833-8. PMID 5767305.
- ^ Hawkins ME (2001). "Fluorescent pteridine nucleoside analogs: a window on DNA interactions". Cell Biochemistry and Biophysics. 34 (2): 257–81. doi:10.1385/cbb:34:2:257. PMID 11898867. S2CID 12134698.
- ^ Berry DA, Jung KY, Wise DS, Sercel AD, Pearson WH, Mackie H, Randolph JB, Somers RL (2004). "Pyrrolo-dC and pyrrolo-C: fluorescent analogs of cytidine and 2 '-deoxycytidine for the study of oligonucleotides". Tetrahedron Lett. 45 (11): 2457–61. doi:10.1016/j.tetlet.2004.01.108.
- ^ Wojciechowski F, Hudson RH (September 2008). "Fluorescence and hybridization properties of peptide nucleic acid containing a substituted phenylpyrrolocytosine designed to engage Guanine with an additional H-bond". Journal of the American Chemical Society. 130 (38): 12574–75. doi:10.1021/ja804233g. PMID 18761442.
- ^ Greco NJ, Tor Y (August 2005). "Simple fluorescent pyrimidine analogues detect the presence of DNA abasic sites". Journal of the American Chemical Society. 127 (31): 10784–85. doi:10.1021/ja052000a. PMID 16076156.
- ^ Rist MJ, Marino JP (2002). "Fluorescent nucleotide base analogs as probes of nucleic acid structure, dynamics and interactions". Curr. Org. Chem. 6 (9): 775–93. doi:10.2174/1385272023373914.
- ^ Wilson JN, Kool ET (December 2006). "Fluorescent DNA base replacements: Reporters and sensors for biological systems". Organic & Biomolecular Chemistry. 4 (23): 4265–74. doi:10.1039/b612284c. PMID 17102869.
- ^ Wilhelmsson and Tor (2016). Fluorescent Analogs of Biomolecular Building Blocks: Design and Applications. New Jersey: Wiley. ISBN 978-1-118-17586-6.
- ^ Wilhelmsson LM (May 2010). "Fluorescent nucleic acid base analogues". Quarterly Reviews of Biophysics. 43 (2): 159–83. doi:10.1017/s0033583510000090. PMID 20478079. S2CID 10783202.
- ^ Sinkeldam RW, Greco NJ, Tor Y (May 2010). "Fluorescent analogs of biomolecular building blocks: design, properties, and applications". Chemical Reviews. 110 (5): 2579–619. doi:10.1021/cr900301e. PMC 2868948. PMID 20205430.
- ^ Wilhelmsson LM, Holmén A, Lincoln P, Nielsen PE, Nordén B (2001). "A highly fluorescent DNA base analogue that forms Watson-Crick base pairs with guanine". J. Am. Chem. Soc. 123 (10): 2434–35. doi:10.1021/ja0025797. PMID 11456897.
- ^ Sandin P, Wilhelmsson LM, Lincoln P, Powers VE, Brown T, Albinsson B (2005). "Fluorescent properties of DNA base analogue tC upon incorporation into DNA – negligible influence of neighbouring bases on fluorescence quantum yield". Nucleic Acids Research. 33 (16): 5019–25. doi:10.1093/nar/gki790. PMC 1201328. PMID 16147985.
- ^ Sandin P, Börjesson K, Li H, Mårtensson J, Brown T, Wilhelmsson LM, Albinsson B (January 2008). "Characterization and use of an unprecedentedly bright and structurally non-perturbing fluorescent DNA base analogue". Nucleic Acids Research. 36 (1): 157–67. doi:10.1093/nar/gkm1006. PMC 2248743. PMID 18003656.
- ^ Börjesson K, Preus S, El-Sagheer AH, Brown T, Albinsson B, Wilhelmsson LM (April 2009). "Nucleic acid base analog FRET-pair facilitating detailed structural measurements in nucleic acid containing systems". Journal of the American Chemical Society. 131 (12): 4288–93. doi:10.1021/ja806944w. PMID 19317504.
- ^ a b Rodriguez-Hernandez A, Spears JL, Gaston KW, Limbach PA, Gamper H, Hou YM, Kaiser R, Agris PF, Perona JJ (October 2013). "Structural and mechanistic basis for enhanced translational efficiency by 2-thiouridine at the tRNA anticodon wobble position". Journal of Molecular Biology. 425 (20): 3888–906. doi:10.1016/j.jmb.2013.05.018. PMC 4521407. PMID 23727144.
- ^ Kirnos MD, Khudyakov IY, Alexandrushkina NI, Vanyushin BF (November 1977). "2-aminoadenine is an adenine substituting for a base in S-2L cyanophage DNA". Nature. 270 (5635): 369–70. Bibcode:1977Natur.270..369K. doi:10.1038/270369a0. PMID 413053. S2CID 4177449.
- ^ Johnson SC, Sherrill CB, Marshall DJ, Moser MJ, Prudent JR (2004). "A third base pair for the polymerase chain reaction: inserting isoC and isoG". Nucleic Acids Research. 32 (6): 1937–41. doi:10.1093/nar/gkh522. PMC 390373. PMID 15051811.
- ^ Taniguchi Y, Kool ET (July 2007). "Nonpolar isosteres of damaged DNA bases: effective mimicry of mutagenic properties of 8-oxopurines". Journal of the American Chemical Society. 129 (28): 8836–44. doi:10.1021/ja071970q. PMID 17592846.
- ^ Hwang GT, Romesberg FE (November 2008). "Unnatural substrate repertoire of A, B, and X family DNA polymerases". Journal of the American Chemical Society. 130 (44): 14872–82. doi:10.1021/ja803833h. PMC 2675700. PMID 18847263.
- ^ Kimoto M, Mitsui T, Harada Y, Sato A, Yokoyama S, Hirao I (2007). "Fluorescent probing for RNA molecules by an unnatural base-pair system". Nucleic Acids Research. 35 (16): 5360–69. doi:10.1093/nar/gkm508. PMC 2018647. PMID 17693436.
- ^ Atwell, Shane; Meggers, Eric; Spraggon, Glen; Schultz, Peter G. (Dec 2001). "Structure of a Copper-Mediated Base Pair in DNA". Journal of the American Chemical Society. 123 (49): 12364–12367. doi:10.1021/ja011822e. ISSN 0002-7863. PMID 11734038.
- ^ Liu H, Gao J, Lynch SR, Saito YD, Maynard L, Kool ET (October 2003). "A four-base paired genetic helix with expanded size". Science. 302 (5646): 868–71. Bibcode:2003Sci...302..868L. doi:10.1126/science.1088334. PMID 14593180. S2CID 37244007.
- ^ Wettig SD, Lee JS (2003). "Thermodynamic investigation of M-DNA: a novel metal ion–DNA complex". Journal of Inorganic Biochemistry. 94 (1–2): 94–99. doi:10.1016/S0162-0134(02)00624-4. PMID 12620678.
- ^ Zhang HY, Calzolari A, Di Felice R (August 2005). "On the magnetic alignment of metal ions in a DNA-mimic double helix". The Journal of Physical Chemistry B. 109 (32): 15345–48. doi:10.1021/jp052202t. PMID 16852946.
- ^ Aich P, Skinner RJ, Wettig SD, Steer RP, Lee JS (August 2002). "Long range molecular wire behaviour in a metal complex of DNA". Journal of Biomolecular Structure & Dynamics. 20 (1): 93–98. doi:10.1080/07391102.2002.10506826. PMID 12144356. S2CID 41568646.
- ^ Clever GH, Polborn K, Carell T (2005). "Ein hochgradig DNA-Duplex-stabilisierendes Metall-Salen-Basenpaar". Angew. Chem. Int. Ed. 117 (44): 7370–74. Bibcode:2005AngCh.117.7370C. doi:10.1002/ange.200501589.
- ^ Buncel E, Boone C, Joly H, Kumar R, Norris AR (1985). "Metal ion-biomolecule interactions. XII. 1H and 13C NMR evidence for the preferred reaction of thymidine over guanosine in exchange and competition reactions with mercury (II) and methylmercury (II)". Inorg. Biochem. 25: 61–73. doi:10.1016/0162-0134(85)83009-9.
- ^ Ono A, Togashi H (August 2004). "Highly selective oligonucleotide-based sensor for mercury(II) in aqueous solutions". Angewandte Chemie. 43 (33): 4300–02. doi:10.1002/anie.200454172. PMID 15368377.
- ^ Meggers E, Holland PL, Tolman WB, Romesberg FE, Schultz PG (2000). "A Novel Copper-Mediated DNA Base Pair". J. Am. Chem. Soc. 122 (43): 10714–15. doi:10.1021/ja0025806.
- ^ Lee JS, Latimer LJ, Reid RS (1993). "A cooperative conformational change in duplex DNA induced by Zn2+ and other divalent metal ions". Biochemistry and Cell Biology. 71 (3–4): 162–68. doi:10.1139/o93-026. PMID 8398074.
- ^ Tanaka K, Tengeiji A, Kato T, Toyama N, Shionoya M (February 2003). "A discrete self-assembled metal array in artificial DNA". Science. 299 (5610): 1212–13. Bibcode:2003Sci...299.1212T. doi:10.1126/science.1080587. PMID 12595687. S2CID 22413126.
- ^ Malyshev DA, Dhami K, Quach HT, Lavergne T, Ordoukhanian P, Torkamani A, Romesberg FE (July 2012). "Efficient and sequence-independent replication of DNA containing a third base pair establishes a functional six-letter genetic alphabet". Proceedings of the National Academy of Sciences of the United States of America. 109 (30): 12005–10. Bibcode:2012PNAS..10912005M. doi:10.1073/pnas.1205176109. PMC 3409741. PMID 22773812.
- ^ Callaway E (May 7, 2014). "Scientists Create First Living Organism With 'Artificial' DNA". Nature News. Huffington Post. Retrieved 8 May 2014.
- ^ a b Fikes BJ (May 8, 2014). "Life engineered with expanded genetic code". San Diego Union Tribune. Retrieved 8 May 2014.
- ^ Sample I (May 7, 2014). "First life forms to pass on artificial DNA engineered by US scientists". The Guardian. Retrieved 8 May 2014.
- ^ Pollack A (May 7, 2014). "Scientists Add Letters to DNA's Alphabet, Raising Hope and Fear". New York Times. Retrieved 8 May 2014.
- ^ Zhang Y, Ptacin JL, Fischer EC, Aerni HR, Caffaro CE, San Jose K, Feldman AW, Turner CR, Romesberg FE (2017). "A semi-synthetic organism that stores and retrieves increased genetic information". Nature. 551 (7682): 644–47. Bibcode:2017Natur.551..644Z. doi:10.1038/nature24659. PMC 5796663. PMID 29189780.
- ^ '자연스럽지 않은' 미생물은 단백질을 만들 수 있다.BBC 뉴스 2017년 11월 29일
- ^ Hirao I, Ohtsuki T, Fujiwara T, Mitsui T, Yokogawa T, Okuni T, Nakayama H, Takio K, Yabuki T, Kigawa T, Kodama K, Yokogawa T, Nishikawa K, Yokoyama S (February 2002). "An unnatural base pair for incorporating amino acid analogs into proteins". Nature Biotechnology. 20 (2): 177–82. doi:10.1038/nbt0202-177. PMID 11821864. S2CID 22055476.
- ^ Hirao I, Kimoto M, Mitsui T, Fujiwara T, Kawai R, Sato A, Harada Y, Yokoyama S (September 2006). "An unnatural hydrophobic base pair system: site-specific incorporation of nucleotide analogs into DNA and RNA". Nature Methods. 3 (9): 729–35. doi:10.1038/nmeth915. PMID 16929319. S2CID 6494156.
- ^ Kimoto M, Kawai R, Mitsui T, Yokoyama S, Hirao I (February 2009). "An unnatural base pair system for efficient PCR amplification and functionalization of DNA molecules". Nucleic Acids Research. 37 (2): e14. doi:10.1093/nar/gkn956. PMC 2632903. PMID 19073696.
- ^ Yamashige R, Kimoto M, Takezawa Y, Sato A, Mitsui T, Yokoyama S, Hirao I (March 2012). "Highly specific unnatural base pair systems as a third base pair for PCR amplification". Nucleic Acids Research. 40 (6): 2793–806. doi:10.1093/nar/gkr1068. PMC 3315302. PMID 22121213.
- ^ Kimoto M, Yamashige R, Matsunaga K, Yokoyama S, Hirao I (May 2013). "Generation of high-affinity DNA aptamers using an expanded genetic alphabet". Nature Biotechnology. 31 (5): 453–57. doi:10.1038/nbt.2556. PMID 23563318. S2CID 23329867.
- ^ 슈미트 M. "제노바이올로지: 궁극의 바이오 안전성 도구로서의 새로운 형태의 생명체" Biosesses Vol 32(4) : 322 – 31
- ^ Herdewijn P, Marlière P (June 2009). "Toward safe genetically modified organisms through the chemical diversification of nucleic acids". Chemistry & Biodiversity. 6 (6): 791–808. doi:10.1002/cbdv.200900083. PMID 19554563. S2CID 8572188.
- ^ Shinkai A, Patel PH, Loeb LA (June 2001). "The conserved active site motif A of Escherichia coli DNA polymerase I is highly mutable". The Journal of Biological Chemistry. 276 (22): 18836–42. doi:10.1074/jbc.M011472200. PMID 11278911.
- ^ Rackham O, Chin JW (August 2005). "A network of orthogonal ribosome x mRNA pairs". Nature Chemical Biology. 1 (3): 159–66. doi:10.1038/nchembio719. PMID 16408021. S2CID 37181098.