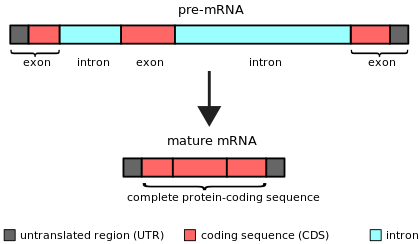
인트론
Intron인트론은 최종 RNA 생성물의 생산 과정에서 RNA 처리에 의해 제거되는 유전자 내의 모든 뉴클레오티드 배열이다.인트론이라는 단어는 유전자 내 영역, 즉 유전자 [1]내 영역에서 유래했습니다.인트론(intron)이라는 용어는 유전자 [2]내 DNA 배열과 RNA 전사의 해당 RNA 배열 모두를 가리킨다.이 RNA 처리에 의해 결합되어 성숙한 RNA를 형성하는 비인트론 배열은 [3]엑손이라고 불립니다.
인트론은 대부분의 유기체와 많은 바이러스의 유전자에서 발견되며 단백질 코드 유전자와 RNA로 기능하는 유전자 둘 다에 위치할 수 있다.인트론에는 tRNA 인트론, 그룹 I 인트론, 그룹 II 인트론 및 스플라이세오솜 인트론의 네 가지 주요 유형이 있습니다(아래 참조).인트론은 박테리아와 고세균에서 드물지만, 대부분의 진핵 생물 유전자는 여러 개의 스플라이솜 인트론을 포함합니다.
발견과 어원
인트론은 아데노바이러스의 [4][5]단백질 부호화 유전자에서 처음 발견되었고, 이후 전달 RNA 및 리보솜 RNA 유전자를 코드하는 유전자에서 확인되었다.인트론은 현재 모든 생물학적 왕국 내의 유기체, 박테리아,[6] 바이러스 전반에 걸쳐 다양한 유전자 내에서 발생하는 것으로 알려져 있다.
유전자가 인트론들에 의해 분열되거나 간섭된다는 사실은 필립 앨런 샤프와 리처드 J. 로버츠에 의해 1977년에 독립적으로 발견되었고,[7] 그들은 1993년에 노벨 생리의학상을 공동 수상했다.인트론이라는 용어는 미국 생화학자인 월터 [1]길버트가 처음 도입했습니다.
"시스트론[즉 유전자]의 개념은 성숙한 메신저에서 손실되는 영역을 포함하는 전사 단위의 개념으로 대체되어야 합니다. 저는 이것을 (인트로네겐 내 영역에 대해) 인트론이라고 부르도록 제안합니다." (길버트 1978)
인트론이라는 용어는 또한 인트라크론,[8] 즉 시스트론 내에서 발생하는 추가적인 DNA 조각을 가리킨다.
비록 인트론은 때때로 중간 배열이라고 [9]불리지만, "간섭 배열"이라는 용어는 인트론뿐만 아니라, 최종 유전자 생성물에 존재하지 않는 내부 핵산 배열의 여러 패밀리를 언급할 수 있습니다.
분배
서로 다른 게놈 내의 인트론의 빈도는 생물학적 유기체의 스펙트럼에 따라 크게 달라지는 것으로 관찰된다.예를 들어, 인트론은 턱뼈가 있는 척추동물(예: 인간과 쥐)의 핵 게놈 내에서 매우 흔하며, 단백질 코드 유전자는 거의 항상 여러 개의 인트론을 포함하고 있는 반면, 인트론은 일부 진핵 미생물([10]예: 제빵사/제빵사/제빵사 효모)의 핵 유전자 내에서 희귀하다.이와는 대조적으로 척추동물의 미토콘드리아 게놈은 완전히 인트론이 없는 반면, 진핵 미생물의 게놈은 많은 [11]인트론을 포함할 수 있다.
특히 극단적인 경우는 3.6메가베이스(Mb) 이상의 인트론을 포함한 드로소필라 dhc7 유전자로 [12][13]전사에는 약 3일이 걸린다.한편 2015년 연구에 따르면 가장 짧은 메타조아 인트론 길이는 인간 MST1L [14]유전자에 속하는 30개의 염기쌍(bp)이다.가장 짧은 것으로 알려진 인트론은 Stentor corpuleus와 같은 헤테로트리치 섬모충에 속하며, 대부분의 인트론은 길이가 [15]15 또는 16 bp이다.
분류
위에서 설명한 바와 같이 모든 인트론 함유 RNA 분자의 스플라이싱은 표면적으로 유사하다.그러나 RNA 스플라이싱 반응의 유전자 및 생화학 분석과 함께 DNA 배열 분석에 의한 인트론 구조 검사를 통해 다른 형태의 인트론이 확인되었다.적어도 네 가지 등급의 인트론이 식별되었다.
- 스플라이세오솜에 의해 제거되는 핵단백질 코드 유전자의 인트론(스플라이세오솜 인트론)
- 단백질에 의해 제거되는 핵 및 고고학적 전달 RNA 유전자의 인트론(tRNA 인트론)
- RNA 촉매 작용에 의해 제거되는 자가 스플라이싱 그룹 I 인트론
- RNA 촉매 작용에 의해 제거되는 자가 스플라이싱 그룹 II 인트론
그룹 III 인트론은 다섯 번째 패밀리로 제안되지만, 이들의 스플라이싱을 매개하는 생화학 기구에 대해서는 거의 알려져 있지 않다.그들은 그룹 II 인트론과 관련이 있는 것으로 보이며, 아마도 스플라이세오솜 [16]인트론과 관련이 있을 수 있다.
스플라이소좀 인트론
핵 사전 mRNA 인트론(스플리세오솜 인트론)은 인트론과 엑손 [17]사이의 경계에 위치한 특정 인트론 서열에 의해 특징지어진다.이러한 배열은 스플라이싱 반응이 [18]시작될 때 스플라이싱 RNA 분자에 의해 인식된다.또한 분기점, 즉 스플라이싱 공정 중에 인트론의 5' 말단과 공유적으로 결합되어 분기된(대형) 인트론을 생성하는 인트론의 3' 말단 부근의 특정 뉴클레오티드 배열을 포함한다.이 세 가지 짧은 보존 요소를 제외하고, 핵 사전 mRNA 인트론 시퀀스는 매우 가변적이다.핵 전 mRNA 인트론은 종종 주변 외부 물질보다 훨씬 더 길다.
tRNA 인트론
제거를 위해 단백질에 의존하는 전달 RNA 인트론은 미첨가 tRNA 전구체의 안티코돈 루프 내의 특정 위치에서 발생하며, tRNA 스플라이싱 엔도핵산가수분해효소에 의해 제거된다.엑손은 두 번째 단백질인 tRNA 스플라이싱 연결 효소에 [19]의해 서로 결합됩니다.자가 분할 인트론은 tRNA [20]유전자 내에서 발견되기도 합니다.
그룹 I 및 그룹 II 인트론
그룹 I 및 그룹 II 인트론은 매우 광범위한 [21][22]생물에서 단백질(메신저 RNA), 전달 RNA 및 리보솜 RNA를 코드하는 유전자에서 발견됩니다.RNA로 전사된 후, 그룹 I과 그룹 II 인트론은 또한 특정하고 복잡한 3차원 구조로 접을 수 있도록 하는 광범위한 내부 상호작용을 합니다.이러한 복잡한 구조를 통해 일부 그룹 I 및 그룹 II 인트론이 자기 분할될 수 있습니다. 즉, 인트론을 포함한 RNA 분자는 인트론을 정밀하게 제거하고 엑손들을 올바른 순서로 연결하기 위해 자신의 공유 구조를 재정렬할 수 있습니다.어떤 경우에, 특정 인트론 결합 단백질은 자가 스플라이싱 활동에 필요한 3차원 구조로 접히는 것을 돕는 방식으로 작용하면서 스플라이싱에 관여한다.그룹 I 및 그룹 II 인트론은 내부 보존 배열과 접힌 구조에 의해 구별되며, 그룹 II 인트론을 포함하는 RNA 분자의 스플라이싱이 분기 인트론을 생성하는 반면 그룹 I 인트론은 스플라이싱되지 않은 구아노신 뉴클레오티드(일반적으로 GTP)를 사용하여 스플라이싱을 개시한다.아이싱, 절제된 인트론의 5' 끝 부분에 추가합니다.
스플라이싱의 정확성에 대하여
스플라이소좀은 100개의 단백질과 5개의 다른 RNA를 포함하는 매우 복잡한 구조이다.반응의 기질은 긴 RNA 분자이고 스플라이세오솜에 의해 촉매되는 에스테르 전이 반응은 수천 개의 뉴클레오티드가 [23][24]떨어져 있을 수 있는 부위의 모음을 필요로 합니다.모든 생화학 반응은 알려진 오류율과 관련이 있으며 반응이 복잡할수록 오류율이 높아집니다.따라서 스플라이스 [25]부위의 우발적인 분열을 억제하는 스플라이스 보조인자가 있어도 스플라이싱 반응이 큰 오차율을 보이는 것은 놀랄 일이 아니다.
이상적인 상황에서는 스플라이싱 반응이 99.999% 정확할 가능성이 높고(오류율 10−5), 올바른 엑손이 결합되어 올바른 인트론이 삭제됩니다.[26]그러나 이러한 이상적인 조건은 최적의 스플라이스 부위 배열과 매우 가까운 매칭을 필요로 하며, 인트론 내에 경쟁하는 암호 스플라이스 부위 배열이 없으며, 이러한 조건은 40킬로베이스 쌍을 포함할 수 있는 큰 진핵생물 유전자에서는 거의 충족되지 않습니다.최근 연구에 따르면 실제 오류율은 10보다 상당히−5 높을 수 있으며 [27][28][29]유전자당 오류율은 2% 또는 3%(오류율−2 2~3x10)에 이를 수 있다.추가 연구에 따르면 오류율은 [30][31]인트론당 0.1% 이상입니다.이 비교적 높은 수준의 스플라이싱 오류는 대부분의 스플라이스 변형이 넌센스 매개 [32][33]붕괴에 의해 급격히 저하되는 이유를 설명합니다.
유전자 내의 엉성한 결합 부위의 존재는 스플라이싱 오류를 야기하고 이러한 부위가 자연 도태에 의해 제거되지 않은 것이 이상하게 보일 수 있다.그들의 집요함에 대한 주장은 정크 [30][34]DNA에 대한 주장과 유사하다.
결합부위를 만들거나 교란시키는 돌연변이는 약간 유해할 수 있지만, 그러한 돌연변이의 많은 수는 집단 내에서 고정되는 것을 불가피하게 만든다.이는 특히 인간과 같이 장기 유효 개체수가 상대적으로 작은 종과 관련이 있다.따라서 인간 게놈이 비정상적인 전사 동소체를 생성하는 차선의 상당한 부하를 운반하는 것은 타당하다.이 연구에서 우리는 이것이 [30]사실이라는 직접적인 증거를 제시한다.
촉매 반응은 대부분의 경우 효과적인 처리를 위해 충분히 정확할 수 있지만 전사 오류는 암호 스플라이스 부위를 만드는 돌연변이를 일으키기 때문에 전체 오류율은 전사의 충실도에 의해 부분적으로 제한될 수 있다.또한 10 - 10의−6 전사−5 오류율은 25,000개의 전사된 엑손 중 1개가 스플라이스 부위 중 하나에서 삽입 오류가 발생하여 스플라이스 인트론 또는 스킵된 엑손이 발생할 정도로 높다.거의 모든 다중 엑손 유전자는 잘못 접합된 전사를 생성하지만, 이 배경 소음의 빈도는 유전자의 크기, 인트론 수, 그리고 접합 부위 [28][31]시퀀스의 품질에 따라 달라집니다.
어떤 경우에는 유전자(DNA)의 돌연변이에 의해 스플라이스 변형이 생성된다.이들은 암호 스플라이스 사이트를 만들거나 기능 사이트를 변환하는 SNP 다형성일 수 있습니다.또한 특정 조직이나 세포주의 [35][36][37]스플라이싱에 영향을 미치는 체세포 돌연변이일 수도 있습니다.돌연변이 대립 유전자가 헤테로 접합 상태일 때, 이것은 두 가지 풍부한 스플라이스 변형을 생산하게 됩니다. 하나는 기능성이고 다른 하나는 비기능성입니다.호모 접합 상태에서 돌연변이 대립 유전자는 빅토리아 여왕의 후손에서 발견되는 혈우병 등의 유전병을 일으킬 수 있으며, 혈액응고인자 유전자 중 하나의 돌연변이가 크립틱 3' 스플라이스 부위를 만들어 이상 스플라이싱을 [38]일으킬 수 있다.질병으로 인한 인간 사망의 상당 부분은 정상적인 스플라이싱을 방해하는 돌연변이에 의해 야기될 수 있다; 대부분 암호 스플라이스 [39][36]부위의 생성에 의해.
잘못 스플라이스된 트랜스크립트는 쉽게 검출되어 그 시퀀스가 온라인 데이터베이스에 입력됩니다.이들은 보통 "대체 스플라이스" 전사물로 설명되는데, 이 용어는 실제, 생물학적으로 관련된 대체 스플라이싱 및 스플라이싱 오류로 인한 처리 노이즈를 구분하지 않기 때문에 혼란스러울 수 있다.대체 스플라이싱 분야에서 가장 중요한 문제 중 하나는 이 두 가지 가능성 사이의 차이를 해결하는 것입니다.많은 과학자들은 귀무 가설은 생물학적으로 관련된 대체 결합을 주장하는 사람들에게 입증의 부담을 지우는 소음 결합이어야 한다고 주장해왔다.그 과학자들에 따르면, 기능의 주장은 여러 기능성 제품들이 [40][41]같은 유전자로부터 생산된다는 설득력 있는 증거를 동반해야 한다.
생물학적 기능과 진화
인트론은 단백질 생성물을 코드하지 않지만 유전자 발현 조절에 필수적이다.일부 인트론들은 기능성 RNA를 스플라이싱 후 추가적인 처리를 통해 암호화하여 비부호화 RNA [42]분자를 생성한다.대체 스플라이싱은 단일 유전자에서 여러 단백질을 생성하기 위해 널리 사용된다.또한 일부 인트론은 난센스 매개[43] 붕괴 및 mRNA [44]수출과 같은 광범위한 유전자 발현 조절 기능에서 필수적인 역할을 한다.
침입자의 생물학적 기원은 불분명하다.진핵핵의 단백질 코드 유전자의 인트론의 최초 발견 후, 현대 유기체의 인트론이 공통의 고대 조상으로부터 물려받은 것인지(인트론-초기 가설로 해석됨), 또는 오히려 진화 과정에서 유전자에 나타난 것인지(인트론-l로 해석됨)에 대한 중요한 논쟁이 있었다.가설을 받아들였습니다).또 다른 이론은 유전자의 스플리세오솜과 인트론-엑손 구조가 RNA 세계의 유물이라는 것이다(인트론 우선 가설).[45]이 가설들 중 어느 것이 가장 옳은지에 대해서는 여전히 상당한 논쟁이 있다.현재 일반적인 공감대는 인트론이 진핵생물 계통 내에서 이기적인 [46]요소로서 발생했다는 것이다.
광범위한 유기체의 게놈 DNA 배열에 대한 초기 연구는 다른 유기체의 상동 유전자의 인트론-엑손 구조가 매우 [47]다양할 수 있다는 것을 보여준다.전체 진핵생물의 게놈에 대한 보다 최근의 연구는 이제 인트론의 길이와 밀도(인트론/유전자)가 관련된 종마다 상당히 다르다는 것을 보여주었다.예를 들어 인간 게놈은 평균 8.4개의 인트론/제네(게놈 139,418개)를 포함하고 있는 반면, 단세포 균류인 뇌조운 큐니쿨리는 0.0075개의 인트론/제네(게놈 [48]15개 인트론)만을 포함하고 있다.진핵생물이 공통의 조상(공통 혈통)에서 [49][50]생겨났기 때문에, 진화 기간 동안 인트론의 광범위한 득실이 있었을 것이다.이 과정은 선택 대상이 될 것으로 생각되며, 개체 수가 작기 때문에 더 큰 종에서는 인트론이 증가하는 경향이 있고, 더 작은 종(특히 단세포)[51] 종에서는 그 반대의 경향이 있다.생물학적 요인은 또한 게놈에서 어떤 유전자가 인트론을 [52][53][54]잃거나 축적하는지에 영향을 미친다.
인트론 절제 후 유전자 내 엑손의 대체 스플라이싱은 단일 유전자 및 단일 전구체 mRNA 전사체로부터 복수의 관련 단백질이 생성될 수 있도록 단일 유전자에서 번역된 단백질 배열의 더 큰 가변성을 도입하는 작용을 한다.대체 RNA 스플라이싱의 제어는 광범위한 세포 내 및 세포 외 신호에 반응하는 복잡한 신호 분자 네트워크에 의해 수행됩니다.
인트론은 스플라이싱에 의한 적절한 스플라이싱에 필요한 분기점 부위뿐만 아니라 인트론의 양 끝에 있는 수용체 및 기증자 부위 등 효율적인 스플라이싱에 중요한 몇 가지 짧은 시퀀스를 포함한다.일부 인트론은 인트론 매개 강화(IME)로 알려진 과정에 의해 그들이 포함된 유전자의 발현을 강화시키는 것으로 알려져 있다.
활발하게 전사된 DNA 영역은 종종 DNA 손상에 취약한 R-루프를 형성합니다.고발현 효모 유전자에서 인트론은 R-루프 형성과 DNA [55]손상의 발생을 억제한다.효모와 인간의 게놈 전체 분석 결과 인트론을 함유한 유전자는 유사한 [55]발현을 가진 인트론리스 유전자에 비해 R-루프 수치를 낮추고 DNA 손상을 줄인 것으로 밝혀졌다.R루프성 유전자에 인트론을 삽입하는 것도 R루프 형성 및 재조합을 억제할 수 있다.보닛 외 연구진(2017)[55]은 유전자 안정성을 유지하는 인트론의 기능이 특정 위치, 특히 고도로 발현된 유전자에서 인트론의 진화적 유지를 설명할 수 있다고 추측했다.
기아 적응
인트론의 물리적 존재는 영양소 감지 [56]경로의 리보솜 단백질 유전자의 인트론 강화 억제를 통해 기아에 대한 세포 저항을 촉진한다.
이동성 유전 요소로서
직교 유전자에 대한 많은 비교 연구에서 알 수 있듯이, 인트론은 진화 과정에서 사라지거나 획득될 수 있다.후속 분석은 인트론 손실과 이득 이벤트의 수천 가지 예를 확인했으며, 진핵생물의 출현 또는 진핵생물 진화의 초기 단계는 인트론 [57]침투를 포함한다고 제안되었다.인트론 손실의 두 가지 결정적 메커니즘, 즉 역전사효소 매개 인트론 손실(RTMIL)과 게놈 결실이 확인되었으며 [58]발생하는 것으로 알려져 있다.그러나 인트론 이득의 최종 메커니즘은 여전히 이해하기 어렵고 논란의 여지가 있다.인트론 게인의 메커니즘은 인트론 전위, 트랜스포존 삽입, 탠덤 게놈 복제, 인트론 전이, 더블 스트랜드 브레이크 리페어(DSBR) 중 인트론 게인, II족 인트론 삽입 및 인트론화 등 적어도 7가지로 보고되었다.이론적으로는 숙주에 의한 돌연변이가 없기 때문에 최근에 얻은 인트론의 기원을 추론하는 것이 가장 쉬워야 하지만, 최근에 얻은 인트론조차도 앞서 언급한 메커니즘 중 어느 것에서도 발생하지 않았다.따라서 이러한 발견들은 제안된 인트론 게인의 메커니즘이 정확한 인트론 게인의 메커니즘이 아니기 때문에 많은 새로운 인트론들의 기계적 기원을 설명하지 못하는지 또는 새로운 인트론을 [59]생성하는 다른, 아직 발견되지 않은 프로세스가 있는지 여부에 대한 의문을 제기한다.
가장 일반적으로 알려진 인트론 이득 메커니즘인 인트론 전위에서는 스플라이스된 인트론은 이전에 인트론이 없었던 위치에서 자신의 mRNA 또는 다른 mRNA로 역스플라이스 되는 것으로 생각된다.이 인트론 함유 mRNA는 역전사되며, 그 결과 인트론 함유 cDNA는 원래의 게놈 궤적과의 완전 또는 부분 재조합을 통해 인트론 이득을 일으킬 수 있다.트랜스포존 삽입으로 인트론이 생성될 수도 있습니다.이러한 삽입은 트랜스포존이 AGGT 배열에 삽입될 때 코드 시퀀스를 중단하지 않고 트랜스포존을 인트로닉화할 수 있으며, 결과적으로 트랜스포존의 양쪽에 이 시퀀스가 복제된다.우연이든 트랜스포존에 의한 일부 우선 작용에 의해 이들 요소가 결합되는 이유는 아직 밝혀지지 않았다.탠덤 게놈 복제에서는 컨센서스 공여체와 수용체 스플라이스 부위의 유사성으로 인해 AGGT 배열을 가진 엑소닉 세그먼트의 탠덤 게놈 복제는 2개의 잠재적 스플라이스 부위를 생성한다.스플라이소솜에 의해 인식될 때, 원래의 AGGT와 중복된 AGGT 사이의 배열을 스플라이스하여 유전자의 코드 배열을 변경하지 않고 인트론을 생성한다.연구진이 다프니아에서 [59]획득된 인트론의 43% 측면에 있는 짧은 직접 반복을 확인했을 때 비호몰로지 엔드 접합을 통한 이중 가닥 절단 수리가 최근 인트론 이득의 원천으로 확인되었다.그러나 통계적 관련성을 위해 이 수치는 다른 유기체의 반복실험을 수반하는 보존된 인트론 수와 비교해야 한다.그룹 II 인트론 삽입의 경우, 그룹 II 인트론의 핵 유전자로의 역호밍이 최근 스플라이소좀 인트론 증가를 유발하도록 제안되었다.
인트론 전달은 패럴로그 또는 의사 유전자가 인트론을 획득하고 재조합을 통해 자매 패럴로그의 인트론 부재 위치로 이 인트론을 전달했을 때 인트론 이득을 초래하는 것으로 가정되어 왔다.인트론라이제이션은 돌연변이가 이전의 외부 배열에서 새로운 인트론을 생성하는 과정이다.따라서, 인트론 게인의 다른 제안된 메커니즘과는 달리, 이 메커니즘은 새로운 [59]인트론을 만들기 위해 DNA의 삽입 또는 생성을 필요로 하지 않는다.
직접적인 증거가 결여된 최근의 인트론 증가의 유일한 가설 메커니즘은 II 그룹 인트론 삽입이며, 이는 생체 내에서 입증될 때 유전자 [60]발현을 폐지한다.따라서 그룹 II 인트론은 부위 특이적 레트로 엘리먼트 역할을 하는 스플라이세오솜 인트론의 추정 조상일 가능성이 높으며, 더 이상 인트론 [61][62]이득에 책임이 없다.탠덤 게놈 복제는 생체 내 실험 증거를 뒷받침하는 유일한 제안된 메커니즘이다. 짧은 인트론 탠덤 복제는 단백질 코드화 유전자에 새로운 인트론을 삽입할 수 있으며, 이에 상응하는 펩타이드 배열은 [63]변경되지 않는다.이 메커니즘은 또한 탠덤 게놈 복제가 인트론 이득을 위한 일반적인 메커니즘이라는 생각을 뒷받침하는 광범위한 간접 증거를 가지고 있다.생체 내에서 제안된 다른 메커니즘, 특히 DSBR, 인트론 전달 및 인트론화 동안 인트론 이득의 실제 메커니즘으로 굳히기 위해 이러한 메커니즘을 생체 내에서 입증해야 하지만, 생체 내에서 테스트가 가능하다.추가적인 게놈 분석은, 특히 모집단 수준에서 실행될 때, 각 메커니즘의 상대적 기여도를 정량화할 수 있으며, 아마도 [59]다른 종들 사이의 다양한 인트론 이득 속도를 밝힐 수 있는 종별 편견을 식별할 수 있다.
「 」를 참조해 주세요.
구조:
스플라이싱:
기능.
기타:
레퍼런스
- ^ a b "시스트론[즉 유전자]의 개념은 성숙한 메신저에서 손실되는 영역을 포함하는 전사 단위의 개념으로 대체되어야 합니다. 저는 이것을 (인트로네겐 내 영역에 대해) 인트론이라고 부르도록 제안합니다." (길버트 1978)Gilbert W (February 1978). "Why genes in pieces?". Nature. 271 (5645): 501. Bibcode:1978Natur.271..501G. doi:10.1038/271501a0. PMID 622185. S2CID 4216649.
- ^ Kinniburgh AJ, Mertz JE, Ross J (July 1978). "The precursor of mouse beta-globin messenger RNA contains two intervening RNA sequences". Cell. 14 (3): 681–693. doi:10.1016/0092-8674(78)90251-9. PMID 688388. S2CID 21897383.
- ^ Lewin B (1987). Genes (3rd ed.). New York: Wiley. pp. 159–179, 386. ISBN 0-471-83278-2. OCLC 14069165.
- ^ Chow LT, Gelinas RE, Broker TR, Roberts RJ (September 1977). "An amazing sequence arrangement at the 5' ends of adenovirus 2 messenger RNA". Cell. 12 (1): 1–8. doi:10.1016/0092-8674(77)90180-5. PMID 902310. S2CID 2099968.
- ^ Berget SM, Moore C, Sharp PA (August 1977). "Spliced segments at the 5' terminus of adenovirus 2 late mRNA". Proceedings of the National Academy of Sciences of the United States of America. 74 (8): 3171–3175. doi:10.1073/pnas.74.8.3171. PMC 431482. PMID 269380.
- ^ Belfort M, Pedersen-Lane J, West D, Ehrenman K, Maley G, Chu F, Maley F (June 1985). "Processing of the intron-containing thymidylate synthase (td) gene of phage T4 is at the RNA level". Cell. 41 (2): 375–382. doi:10.1016/s0092-8674(85)80010-6. PMID 3986907. S2CID 27127017.
- ^ "The Nobel Prize in Physiology or Medicine 1993".
- ^ Tonegawa S, Maxam AM, Tizard R, Bernard O, Gilbert W (March 1978). "Sequence of a mouse germ-line gene for a variable region of an immunoglobulin light chain". Proceedings of the National Academy of Sciences of the United States of America. 75 (3): 1485–1489. Bibcode:1978PNAS...75.1485T. doi:10.1073/pnas.75.3.1485. PMC 411497. PMID 418414.
- ^ Tilghman SM, Tiemeier DC, Seidman JG, Peterlin BM, Sullivan M, Maizel JV, Leder P (February 1978). "Intervening sequence of DNA identified in the structural portion of a mouse beta-globin gene". Proceedings of the National Academy of Sciences of the United States of America. 75 (2): 725–729. Bibcode:1978PNAS...75..725T. doi:10.1073/pnas.75.2.725. PMC 411329. PMID 273235.
- ^ Stajich JE, Dietrich FS, Roy SW (2007). "Comparative genomic analysis of fungal genomes reveals intron-rich ancestors". Genome Biology. 8 (10): R223. doi:10.1186/gb-2007-8-10-r223. PMC 2246297. PMID 17949488.
- ^ Taanman JW (February 1999). "The mitochondrial genome: structure, transcription, translation and replication". Biochimica et Biophysica Acta (BBA) - Bioenergetics. 1410 (2): 103–123. doi:10.1016/s0005-2728(98)00161-3. PMID 10076021.
- ^ Tollervey D, Caceres JF (November 2000). "RNA processing marches on". Cell. 103 (5): 703–709. doi:10.1016/S0092-8674(00)00174-4. PMID 11114327.
- ^ Reugels AM, Kurek R, Lammermann U, Bünemann H (February 2000). "Mega-introns in the dynein gene DhDhc7(Y) on the heterochromatic Y chromosome give rise to the giant threads loops in primary spermatocytes of Drosophila hydei". Genetics. 154 (2): 759–769. doi:10.1093/genetics/154.2.759. PMC 1460963. PMID 10655227.
- ^ Piovesan A, Caracausi M, Ricci M, Strippoli P, Vitale L, Pelleri MC (December 2015). "Identification of minimal eukaryotic introns through GeneBase, a user-friendly tool for parsing the NCBI Gene databank". DNA Research. 22 (6): 495–503. doi:10.1093/dnares/dsv028. PMC 4675715. PMID 26581719.
- ^ Slabodnick MM, Ruby JG, Reiff SB, Swart EC, Gosai S, Prabakaran S, et al. (February 2017). "The Macronuclear Genome of Stentor coeruleus Reveals Tiny Introns in a Giant Cell". Current Biology. 27 (4): 569–575. doi:10.1016/j.cub.2016.12.057. PMC 5659724. PMID 28190732.
- ^ Copertino DW, Hallick RB (December 1993). "Group II and group III introns of twintrons: potential relationships with nuclear pre-mRNA introns". Trends in Biochemical Sciences. 18 (12): 467–471. doi:10.1016/0968-0004(93)90008-b. PMID 8108859.
- ^ Padgett RA, Grabowski PJ, Konarska MM, Seiler S, Sharp PA (1986). "Splicing of messenger RNA precursors". Annual Review of Biochemistry. 55: 1119–1150. doi:10.1146/annurev.bi.55.070186.005351. PMID 2943217.
- ^ Guthrie C, Patterson B (1988). "Spliceosomal snRNAs". Annual Review of Genetics. 22: 387–419. doi:10.1146/annurev.ge.22.120188.002131. PMID 2977088.
- ^ Greer CL, Peebles CL, Gegenheimer P, Abelson J (February 1983). "Mechanism of action of a yeast RNA ligase in tRNA splicing". Cell. 32 (2): 537–546. doi:10.1016/0092-8674(83)90473-7. PMID 6297798. S2CID 44978152.
- ^ Reinhold-Hurek B, Shub DA (May 1992). "Self-splicing introns in tRNA genes of widely divergent bacteria". Nature. 357 (6374): 173–176. Bibcode:1992Natur.357..173R. doi:10.1038/357173a0. PMID 1579169. S2CID 4370160.
- ^ Cech TR (1990). "Self-splicing of group I introns". Annual Review of Biochemistry. 59: 543–568. doi:10.1146/annurev.bi.59.070190.002551. PMID 2197983.
- ^ Michel F, Ferat JL (1995). "Structure and activities of group II introns". Annual Review of Biochemistry. 64: 435–461. doi:10.1146/annurev.bi.64.070195.002251. PMID 7574489.
- ^ Wan R, Bai R, Zhan X, Shi Y (2020). "How is precursor messenger RNA spliced by the spliceosome?". Annual Review of Biochemistry. 89: 333–358. doi:10.1146/annurev-biochem-013118-111024. PMID 31815536. S2CID 209167227.
- ^ Wilkinson ME, Charenton C, Nagai K (2020). "RNA splicing by the spliceosome". Annual Review of Biochemistry. 89: 359–388. doi:10.1146/annurev-biochem-091719-064225. PMID 31794245. S2CID 208626110.
- ^ Sales-Lee J, Perry DS, Bowser BA, Diedrich JK, Rao B, Beusch I, Yates III JR, Roy SW, Madhani HD (2021). "Coupling of spliceosome complexity to intron diversity". Current Biology. 31 (22): 4898–4910 e4894. doi:10.1016/j.cub.2021.09.004. PMC 8967684. PMID 34555349. S2CID 237603074.
- ^ Hsu SN, Hertel KJ (2009). "Spliceosomes walk the line: splicing errors and their impact on cellular function". RNA Biology. 6 (5): 526–530. doi:10.4161/rna.6.5.9860. PMC 3912188. PMID 19829058. S2CID 22592978.
- ^ Melamud E, Moult J (2009). "Stochastic noise in splicing machinery". Nucleic Acids Research. gkp471 (14): 4873–4886. doi:10.1093/nar/gkp471. PMC 2724286. PMID 19546110.
- ^ a b Fox-Walsh KL, Hertel KJ (2009). "Splice-site pairing is an intrinsically high fidelity process". Proceedings of the National Academy of Sciences. 106 (6): 1766–1771. Bibcode:2009PNAS..106.1766F. doi:10.1073/pnas.0813128106. PMC 2644112. PMID 19179398.
- ^ Stepankiw N, Raghavan M, Fogarty EA, Grimson A, Pleiss JA (2015). "Widespread alternative and aberrant splicing revealed by lariat sequencing". Nucleic Acids Research. 43 (17): 8488–8501. doi:10.1093/nar/gkv763. PMC 4787815. PMID 26261211.
- ^ a b c Pickrell JK, Pai AA, Gilad Y, Pritchard JK (2010). "Noisy splicing drives mRNA isoform diversity in human cells". PLOS Genet. 6 (12): e1001236. doi:10.1371/journal.pgen.1001236. PMC 3000347. PMID 21151575.
- ^ a b Skandalis A (2016). "Estimation of the minimum mRNA splicing error rate in vertebrates". Mutation Research/Fundamental and Molecular Mechanisms of Mutagenesis. 784 (1713): 34–38. doi:10.1098/rstb.2015.0474. PMC 5182408. PMID 27994117.
- ^ Zhang Z, Xin D, Wang P, Zhou L, Hu L, Kong X, Hurst LD (2009). "Noisy splicing, more than expression regulation, explains why some exons are subject to nonsense-mediated mRNA decay". BMC Biology. 7: 23. doi:10.1186/1741-7007-7-23. PMC 2697156. PMID 19442261.
- ^ Bitton DA, Atkinson SR, Rallis C, Smith GC, Ellis DA, Chen YY, Malecki M, Codlin S, Lemay JF, Cotobal C (2015). "Widespread exon skipping triggers degradation by nuclear RNA surveillance in fission yeast". Genome Research. 25 (6): 884–896. doi:10.1101/gr.185371.114. PMC 4448684. PMID 25883323.
- ^ Saudemont B, Popa A, Parmley JL, Rocher V, Blugeon C, Necsulea A, Meyer E, Duret L (2017). "The fitness cost of mis-splicing is the main determinant of alternative splicing patterns". Genome Biology. 18 (1): 208. doi:10.1186/s13059-017-1344-6. PMC 5663052. PMID 29084568.
- ^ Scotti MM, Swanson MS (2016). "RNA mis-splicing in disease". Nature Reviews Genetics. 17 (1): 19–32. doi:10.1038/nrg.2015.3. PMC 5993438. PMID 26593421.
- ^ a b Shirley B, Mucaki E, Rogan P (2019). "Pan-cancer repository of validated natural and cryptic mRNA splicing mutations". F1000Research. 7: 1908. doi:10.12688/f1000research.17204.3. PMC 6544075. PMID 31275557. S2CID 202702147.
- ^ Mucaki EJ, Shirley BC, Rogan PK (2020). "Expression changes confirm genomic variants predicted to result in allele-specific, alternative mRNA splicing". Frontiers in Genetics. 11: 109. doi:10.3389/fgene.2020.00109. PMC 7066660. PMID 32211018.
- ^ Rogaev EI, Grigorenko AP, Faskhutdinova G, Kittler EL, Moliaka YK (2009). "Genotype analysis identifies the cause of the "royal disease"". Science. 326 (5954): 817. Bibcode:2009Sci...326..817R. doi:10.1126/science.1180660. PMID 19815722. S2CID 206522975.
- ^ Lynch M (2010). "Rate, molecular spectrum, and consequences of human mutation". Proceedings of the National Academy of Sciences. 107 (3): 961–968. Bibcode:2010PNAS..107..961L. doi:10.1073/pnas.0912629107. PMC 2824313. PMID 20080596.
- ^ Mudge JM, Harrow J (2016). "The state of play in higher eukaryote gene annotation". Nature Reviews Genetics. 17 (12): 758–772. doi:10.1038/nrg.2016.119. PMC 5876476. PMID 27773922.
- ^ Bhuiyan SA, Ly S, Phan M, Huntington B, Hogan E, Liu CC, Liu J, Pavlidis P (2018). "Systematic evaluation of isoform function in literature reports of alternative splicing". BMC Genomics. 19 (1): 637. doi:10.1186/s12864-018-5013-2. PMC 6114036. PMID 30153812.
- ^ Rearick D, Prakash A, McSweeny A, Shepard SS, Fedorova L, Fedorov A (March 2011). "Critical association of ncRNA with introns". Nucleic Acids Research. 39 (6): 2357–2366. doi:10.1093/nar/gkq1080. PMC 3064772. PMID 21071396.
- ^ Bicknell AA, Cenik C, Chua HN, Roth FP, Moore MJ (December 2012). "Introns in UTRs: why we should stop ignoring them". BioEssays. 34 (12): 1025–1034. doi:10.1002/bies.201200073. PMID 23108796. S2CID 5808466.
- ^ Cenik C, Chua HN, Zhang H, Tarnawsky SP, Akef A, Derti A, et al. (April 2011). Snyder M (ed.). "Genome analysis reveals interplay between 5'UTR introns and nuclear mRNA export for secretory and mitochondrial genes". PLOS Genetics. 7 (4): e1001366. doi:10.1371/journal.pgen.1001366. PMC 3077370. PMID 21533221.
- ^ Penny D, Hoeppner MP, Poole AM, Jeffares DC (November 2009). "An overview of the introns-first theory". Journal of Molecular Evolution. 69 (5): 527–540. Bibcode:2009JMolE..69..527P. doi:10.1007/s00239-009-9279-5. PMID 19777149. S2CID 22386774.
- ^ Cavalier-Smith T (1985). "Selfish DNA and the origin of introns". Nature. 315 (6017): 283–284. Bibcode:1985Natur.315..283C. doi:10.1038/315283b0. PMID 2987701. S2CID 4367253.
- ^ Rodríguez-Trelles F, Tarrío R, Ayala FJ (2006). "Origins and evolution of spliceosomal introns". Annual Review of Genetics. 40: 47–76. doi:10.1146/annurev.genet.40.110405.090625. PMID 17094737.
- ^ Mourier T, Jeffares DC (May 2003). "Eukaryotic intron loss". Science. 300 (5624): 1393. doi:10.1126/science.1080559. PMID 12775832. S2CID 7235937.
- ^ Roy SW, Gilbert W (March 2006). "The evolution of spliceosomal introns: patterns, puzzles and progress". Nature Reviews. Genetics. 7 (3): 211–221. doi:10.1038/nrg1807. PMID 16485020. S2CID 33672491.
- ^ de Souza SJ (July 2003). "The emergence of a synthetic theory of intron evolution". Genetica. 118 (2–3): 117–121. doi:10.1023/A:1024193323397. PMID 12868602. S2CID 7539892.
- ^ Lynch M (April 2002). "Intron evolution as a population-genetic process". Proceedings of the National Academy of Sciences of the United States of America. 99 (9): 6118–6123. Bibcode:2002PNAS...99.6118L. doi:10.1073/pnas.092595699. PMC 122912. PMID 11983904.
- ^ Jeffares DC, Mourier T, Penny D (January 2006). "The biology of intron gain and loss". Trends in Genetics. 22 (1): 16–22. doi:10.1016/j.tig.2005.10.006. PMID 16290250.
- ^ Jeffares DC, Penkett CJ, Bähler J (August 2008). "Rapidly regulated genes are intron poor". Trends in Genetics. 24 (8): 375–378. doi:10.1016/j.tig.2008.05.006. PMID 18586348.
- ^ Castillo-Davis CI, Mekhedov SL, Hartl DL, Koonin EV, Kondrashov FA (August 2002). "Selection for short introns in highly expressed genes". Nature Genetics. 31 (4): 415–418. doi:10.1038/ng940. PMID 12134150. S2CID 9057609.
- ^ a b c Bonnet A, Grosso AR, Elkaoutari A, Coleno E, Presle A, Sridhara SC, et al. (August 2017). "Introns Protect Eukaryotic Genomes from Transcription-Associated Genetic Instability". Molecular Cell. 67 (4): 608–621.e6. doi:10.1016/j.molcel.2017.07.002. PMID 28757210.
- ^ Parenteau J, Maignon L, Berthoumieux M, Catala M, Gagnon V, Abou Elela S (January 2019). "Introns are mediators of cell response to starvation". Nature. 565 (7741): 612–617. Bibcode:2019Natur.565..612P. doi:10.1038/s41586-018-0859-7. PMID 30651641. S2CID 58014466.
- ^ Rogozin IB, Carmel L, Csuros M, Koonin EV (April 2012). "Origin and evolution of spliceosomal introns". Biology Direct. 7: 11. doi:10.1186/1745-6150-7-11. PMC 3488318. PMID 22507701.
- ^ Derr LK, Strathern JN (January 1993). "A role for reverse transcripts in gene conversion". Nature. 361 (6408): 170–173. Bibcode:1993Natur.361..170D. doi:10.1038/361170a0. PMID 8380627. S2CID 4364102.
- ^ a b c d Yenerall P, Zhou L (September 2012). "Identifying the mechanisms of intron gain: progress and trends". Biology Direct. 7: 29. doi:10.1186/1745-6150-7-29. PMC 3443670. PMID 22963364.
- ^ Chalamcharla VR, Curcio MJ, Belfort M (April 2010). "Nuclear expression of a group II intron is consistent with spliceosomal intron ancestry". Genes & Development. 24 (8): 827–836. doi:10.1101/gad.1905010. PMC 2854396. PMID 20351053.
- ^ Cech TR (January 1986). "The generality of self-splicing RNA: relationship to nuclear mRNA splicing". Cell. 44 (2): 207–210. doi:10.1016/0092-8674(86)90751-8. PMID 2417724. S2CID 11652546.
- ^ Dickson L, Huang HR, Liu L, Matsuura M, Lambowitz AM, Perlman PS (November 2001). "Retrotransposition of a yeast group II intron occurs by reverse splicing directly into ectopic DNA sites". Proceedings of the National Academy of Sciences of the United States of America. 98 (23): 13207–13212. Bibcode:2001PNAS...9813207D. doi:10.1073/pnas.231494498. PMC 60849. PMID 11687644.
- ^ Hellsten U, Aspden JL, Rio DC, Rokhsar DS (August 2011). "A segmental genomic duplication generates a functional intron". Nature Communications. 2: 454. Bibcode:2011NatCo...2..454H. doi:10.1038/ncomms1461. PMC 3265369. PMID 21878908.
외부 링크
- NCBI에서 정의한 exon/intron 시퀀스 검색 엔진
- Bruce Alberts, Alexander Johnson, Julian Lewis, Martin Raff, Keith Roberts 및 Peter Walter Molecular Biology of the Cell, 2007, ISBN 978-0-8153-4105-5.제4판은 NCBI Bookbooks에서 온라인으로 입수할 수 있습니다.링크
- Jeremy M Berg, John L Tymoczko, Lubert Stryer, 생화학 제5판, 2002년 W H Freeman.NCBI Bookbooks에서 온라인으로 이용 가능: 링크
- 식물 게놈 배열을 위한 인트론 검색 도구
- 엑손 인트론 그래픽 메이커