게놈학
Genomics| 시리즈의 일부(on) |
| 유전학 |
|---|
 |
유전체학은 유전체의 구조, 기능, 진화, 지도 제작, 편집에 초점을 맞춘 학제간 생물학 분야입니다.게놈(genome)은 모든 유전자뿐만 아니라 계층적인 3차원 구조적 구조를 포함하는 유기체의 완전한 DNA 집합입니다.[1][2][3][4] 유전체학은 개별 유전자와 유전에서의 역할에 대한 연구를 의미하는 유전학과는 대조적으로, 유기체의 모든 유전자의 집단적 특성화와 정량화, 그것들의 상호관계와 유기체에 대한 영향력을 목표로 합니다.[5]유전자는 효소와 메신저 분자의 도움으로 단백질의 생성을 지시할 수 있습니다.결국, 단백질은 장기와 조직과 같은 신체 구조를 구성하고 화학 반응을 조절하고 세포 사이에 신호를 전달합니다.유전체학은 또한 전체 유전체의 기능과 구조를 조합하고 분석하기 위해 높은 처리량의 DNA 염기서열결정과 생물정보학을 사용하여 유전체의 염기서열결정과 분석을 포함합니다.[6][7]유전체학의 발전은 뇌와 같은 가장 복잡한 생물학적 시스템을 이해하는 것을 촉진하기 위해 발견 기반 연구 및 시스템 생물학의 혁명을 촉발했습니다.[8]
이 분야는 또한 인지증(한 유전자가 다른 유전자에 미치는 영향), 흉선성(한 유전자가 하나 이상의 형질에 영향을 미치는), 이질성(혼종성), 그리고 게놈 내의 위치와 대립유전자 사이의 다른 상호작용과 같은 게놈 내 (게놈 내) 현상에 대한 연구도 포함합니다.[9]
역사
어원
그리스 γ ε ν 유전자에서 유래한 "유전자" (감마, 엡실론, 누, 엡실론)는 "되다, 창조하다, 창조하다, 탄생하다"를 의미하며, 그 이후의 변형들: 계보, 유전자, 유전자, 게놈, 유전자형, 속 등.유전체(Genome)라는 단어는 일찍이 1926년에 영어로 사용되었지만,[11] 유전체학이라는 용어는 Jim Womack과 맥주를 마시며 Jackson Laboratory (메인주 Bar Harbor)의 유전학자인 Tom Roderick에 의해 만들어졌습니다.1986년 메릴랜드에서 열린 인간 게놈 지도에 관한 회의에서 톰 쇼스와 스티븐 오브라이언.[12]처음에는 새로운 학술지의 이름으로, 그리고 그 다음에는 완전히 새로운 과학 학문으로.[13]
초기 시퀀싱 작업
로잘린드 프랭클린이 DNA의 나선형 구조를 확인한 후 제임스 D. 1953년 Watson과 Francis Crick의 DNA 구조에 대한 발표와 1955년 Fred Sanger의 인슐린 아미노산 서열에 대한 발표, 핵산 서열 분석은 초기 분자 생물학자들의 주요 표적이 되었습니다.[14]1964년 로버트 W. Holley와 동료들은 알라닌 전달 RNA의 리보뉴클레오티드 서열인 최초의 핵산 서열을 발표했습니다.[15][16] 이 연구를 확장하면서 Marshall Nirenberg와 Philip Leader는 유전자 코드의 삼중항 성질을 밝혔고 그들의 실험에서 64개 코돈 중 54개의 코돈의 서열을 결정할 수 있었습니다.[17]1972년, 겐트 대학교(벨기에 겐트) 분자생물학 연구소의 월터 파이어스와 그의 팀은 최초로 박테리오파지 MS2 코트 단백질의 유전자의 염기서열을 밝혀냈습니다.[18]피어스 그룹은 1976년과 1978년 각각 박테리오파지 MS2-RNA(유전체는 3569개 염기쌍 [bp]에 4개의 유전자만 암호화함)와 시미안 바이러스 40의 완전한 뉴클레오타이드 서열을 결정하면서 MS2 코트 단백질 연구를 확장했습니다.[19][20]
DNA 염기서열결정 기술이 개발되었습니다.
인슐린의 아미노산 서열에 대한 그의 획기적인 연구 외에도, Frederick Sanger와 그의 동료들은 포괄적인 게놈 서열 분석 프로젝트의 설립을 가능하게 한 DNA 서열 분석 기술의 개발에 중요한 역할을 했습니다.[9]1975년, 그와 앨런 콜슨은 그가 플러스 앤 마이너스 기법이라고 부르는 방사선 표지 뉴클레오티드를 가진 DNA 중합효소를 이용한 염기서열 분석 방법을 발표했습니다.[21][22]이것은 정의된 3' 말단을 갖는 짧은 올리고뉴클레오티드를 생성하는 두 가지 밀접한 관련이 있는 방법을 포함했습니다.이들은 폴리아크릴아미드 겔 전기영동(폴리아크릴아미드 겔 전기영동이라고 함)에 전기영동에 의해 분류되고 자동 방사선 촬영을 사용하여 시각화될 수 있습니다.이 과정은 한번에 80개의 뉴클레오티드를 배열할 수 있었고 큰 개선이었지만 여전히 매우 힘들었습니다.그럼에도 불구하고, 1977년에 그의 그룹은 단일 가닥 박테리오파지 φX174의 5,386 뉴클레오타이드의 대부분을 배열할 수 있었고, 최초로 완전하게 배열된 DNA 기반 게놈을 완성했습니다.플러스와 마이너스 방법의 개선은 다음 25세기의 연구에서 가장 널리 사용된 DNA 염기서열결정, 유전체 매핑, 데이터 저장 및 생체정보 분석의 기술의 기초를 형성한 연쇄 종결, 즉 생어 방법(아래 참조)으로 이어졌습니다.[24][25]같은 해 하버드 대학의 월터 길버트와 앨런 맥삼은 DNA 염기서열 분석의 맥삼-길버트 방법(화학적 방법으로도 알려져 있음)을 독자적으로 개발했는데, 이 방법은 덜 효율적인 방법입니다.[26][27]핵산 서열 분석의 획기적인 업적으로, 길버트와 생어는 1980년 노벨 화학상의 절반을 폴 버그(재조합 DNA)와 공유했습니다.
완전유전체
이러한 기술의 출현은 게놈 시퀀싱 프로젝트의 수행 범위와 수행 속도의 급속한 강화를 초래했습니다.1981년 인간 미토콘드리아(16,568bp, 약 16.6kb [kilobase])라는 진핵 세포 소기관의 최초 완전한 게놈 서열이 보고되었고,[28] 1986년에 최초의 엽록체 게놈이 보고되었습니다.[29][30]1992년 브루어 효모 Saccharomyces cerevisiae (315 kb)의 첫 번째 진핵 염색체 III 염색체가 서열화되었습니다.[31]최초로 염기서열이 밝혀진 생물은 1995년 Haemophilus influenzae (1.8 Mb [megabase])의 생물이었습니다.[32]다음 해 북미, 유럽, 일본 전역의 실험실에서 온 연구자들로 구성된 컨소시엄은 진핵생물의 첫 번째 완전한 게놈 서열인 S. cerevisiae (12.1 Mb)의 완성을 발표했고, 그 이후 게놈은 기하급수적으로 증가하는 속도로 계속 배열되어 왔습니다.[33]2011년[update] 10월 현재, 완전한 서열은 2,719개의 바이러스, 1,115개의 고균과 박테리아, 그리고 36개의 진핵생물에 이용 가능하며, 그 중 약 절반이 균류입니다.[34][35]

게놈이 완전히 배열된 대부분의 미생물은 Haemophilus influenzae와 같은 문제가 많은 병원체로 미생물 다양성의 폭에 비해 계통발생 분포에 현저한 편향을 초래했습니다.[36][37]배열된 다른 종들 중, 대부분은 잘 연구된 모델 생물이거나 좋은 모델이 되기로 약속했기 때문에 선택되었습니다.효모(Saccharomyces cerevisiae)는 오랫동안 진핵세포의 중요한 모델 유기체였으며 초파리 드로소필라 멜라노가스터는 매우 중요한 도구였습니다(특히 초기 분자 전 유전학에서).벌레 Caenorhabditis elegans는 종종 다세포 생물에 사용되는 간단한 모델입니다.제브라피쉬 브라키다니오 레리오(Brachydanio rerio)는 분자 수준에 대한 많은 발달 연구에 사용되며, 아라비놉시스 탈리아나(Arabidopsis thaliana) 식물은 꽃이 피는 식물의 모델 유기체입니다.일본 복어 (타키푸구 루브립스)와 점박이 초록 복어 (테트라오돈 니그로비리디스)는 대부분의 종에 비해 코딩이 되지 않은 DNA를 거의 포함하지 않은 작고 작은 게놈 때문에 흥미롭습니다.[38][39]포유류 개 (Canis familyis),[40] 갈색 쥐 (Rattus norvegicus), 쥐 (Mus musculus), 그리고 침팬지 (Pantroglodytes)는 모두 의학 연구에서 중요한 모델 동물입니다.[27]
인간 게놈의 초안은 2001년 초 인간 게놈 프로젝트에 의해 완성되어 많은 팡파르를 만들었습니다.[41]2003년에 완성된 이 프로젝트는 특정한 한 사람의 게놈 전체를 배열했고, 2007년까지 이 배열은 "완료"로 선언되었습니다 (20,000개의 염기와 모든 염색체가 조립된 상태에서 하나의 오류 미만).[41]그 이후 몇 년 동안, 다른 많은 개체들의 게놈이 부분적으로 2012년 10월에 1,092개의 게놈의 시퀀싱을 발표한 1000 게놈 프로젝트의 후원 하에 배열되었습니다.[42]이 프로젝트의 완성은 훨씬 더 효율적인 시퀀싱 기술의 개발로 가능해졌으며, 대규모 국제 협력에서 중요한 생물정보학 자원의 투입이 필요했습니다.[43]인간 게놈 데이터에 대한 지속적인 분석은 인간 사회에 깊은 정치적, 사회적 파장을 가져옵니다.[44]
오믹스 혁명
영어 신조어는 비공식적으로 유전체학, 프로테오믹스 또는 대사체학과 같은 생물학으로 끝나는 학문 분야를 말합니다.관련 접미사 -ome은 게놈, 프로테옴 또는 대사체와 같은 분야의 연구 대상을 다루는 데 사용됩니다.분자생물학에서 사용되는 접미사 -ome은 어떤 종류의 총체를 나타냅니다. 유사하게 오믹스는 일반적으로 크고 포괄적인 생물학적 데이터 세트에 대한 연구를 나타내게 되었습니다.이 용어의 사용이 증가함에 따라 일부 과학자들(조나단 아이젠[45] 등)은 이 용어가 지나치게 많이 팔렸다고 주장하고 있지만,[46] 이는 시스템의 모든 구성 요소에 대해 완전하거나 거의 완전한 조합을 정량적으로 분석하는 방향의 변화를 반영합니다.[47]예를 들어, 공생에 대한 연구에서, 한 때 단일 유전자 산물에 대한 연구에만 국한되었던 연구자들은 이제 동시에 여러 종류의 생물학적 분자의 총보수를 비교할 수 있습니다.[48][49]
유전체 분석
유기체가 선택된 후, 게놈 프로젝트는 세 가지 요소를 포함합니다: DNA 염기서열,[9] 원래 염색체의 표현을 만들기 위한 그 염기서열 집합, 그리고 그 표현의 주석과 분석.
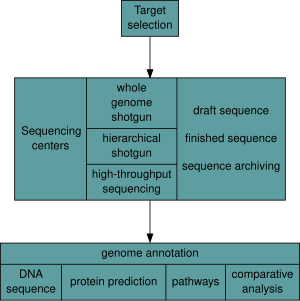
시퀀싱
역사적으로 시퀀싱은 비용이 많이 드는 계측 및 기술 지원과 함께 연구소가 있는 시퀀싱 센터, 중앙 집중식 시설(연간 수십 테라베이스를 시퀀싱하는 조인트 게놈 연구소와 같은 대규모 독립 기관부터 지역 분자 생물학 핵심 시설에 이르기까지)에서 수행되었습니다.그러나 시퀀싱 기술이 계속해서 향상됨에 따라, 효과적인 빠른 턴어라운드 벤치탑 시퀀서의 새로운 세대가 평균적인 학술 연구소에 도달했습니다.[50][51]일반적으로, 게놈 시퀀싱 접근법은 샷건과 고 처리량(또는 차세대) 시퀀싱의 두 가지 광범위한 범주로 나뉩니다.[9]
산탄총 염기서열 분석

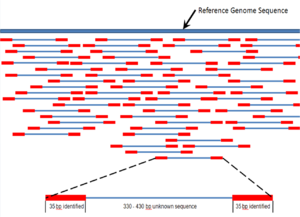
산탄총 염기서열 분석은 1000개 이상의 염기쌍을 분석하고 전체 염색체를 포함하기 위해 고안된 염기서열 분석 방법입니다.[52]그것은 산탄총의 급속하게 팽창하는 준임의 사격 패턴에 비유하여 이름 지어졌습니다.겔 전기영동 시퀀싱은 상당히 짧은 시퀀스(100~1000개 염기쌍)에만 사용할 수 있기 때문에, 긴 DNA 시퀀스를 무작위의 작은 세그먼트로 분할한 다음 판독치를 얻기 위해 시퀀싱해야 합니다.이 단편화 및 시퀀싱의 여러 라운드를 수행하여 표적 DNA에 대한 중복 판독치를 여러 번 획득합니다.그런 다음 컴퓨터 프로그램은 다른 읽기의 겹쳐진 끝을 사용하여 연속적인 시퀀스로 조립합니다.[52][53]산탄총 염기서열 분석은 임의로 추출하는 과정으로, 주어진 뉴클레오티드가 재구성된 염기서열에 나타나는지 확인하기 위해 오버샘플링이 필요합니다. 게놈이 오버샘플링되는 평균 읽기 수를 커버리지(coverage)[54]라고 합니다.
그 역사의 대부분 동안 산탄총 염기서열 분석의 기반이 되는 기술은 고전적인 사슬 종결 방법 또는 시험관 내 DNA 복제 동안 DNA 중합효소에 의한 사슬 종결 디옥시뉴클레오티드의 선택적 통합에 기반을 둔 '생어 방법'이었습니다.[23][55]최근, 샷건 시퀀싱은 특히 대규모의 자동화된 게놈 분석을 위해 높은 처리량의 시퀀싱 방법에 의해 대체되고 있습니다.그러나 생어 방법은 주로 소규모 프로젝트와 특히 긴 연속 DNA 염기서열 판독(>500 뉴클레오티드)을 얻기 위해 널리 사용되고 있습니다.[56]사슬 말단화 방법은 단일 가닥 DNA 템플릿, DNA 프라이머, DNA 중합효소, 정상 디옥시뉴클레오시디트리포스페이트(dNTP) 및 DNA 가닥 신장을 종결시키는 변형된 뉴클레오타이드(dideoxyNTP)를 필요로 합니다.이러한 사슬 말단 뉴클레오티드는 두 뉴클레오티드 사이의 포스포다이에스터 결합 형성에 필요한 3'-OH 기를 결여하고 있어, ddNTP가 포함될 때 DNA 중합효소가 DNA의 확장을 중단하게 합니다.ddNTP는 DNA 염기서열에서 검출을 위해 방사성 또는 형광 표지를 부착할 수 있습니다.[9]일반적으로, 이 기계들은 하루에 최대 48번의 실행으로 한 번의 배치(실행)로 최대 96개의 DNA 샘플을 배열할 수 있습니다.[57]
높은 처리량의 시퀀싱
저비용 시퀀싱에 대한 높은 수요로 인해 시퀀싱 프로세스를 병렬화하는 고 처리량 시퀀싱 기술이 개발되어 수천 또는 수백만 개의 시퀀스를 한 번에 생성할 수 있게 되었습니다.[58][59]높은 처리량의 시퀀싱은 표준 염료 종결자 방법으로 가능한 것 이상으로 DNA 시퀀싱 비용을 낮추기 위한 것입니다.초고 처리량 시퀀싱에서는 합성별 시퀀싱 작업을 최대 500,000회까지 병렬로 실행할 수 있습니다.[60][61]

일루미나 염료 시퀀싱 방법은 가역적 염료 종결자를 기반으로 하며 1996년 제네바 생물의학 연구소에서 파스칼 메이어와 로랑 파리넬리에 의해 개발되었습니다.[62]이 방법에서 DNA 분자와 프라이머는 먼저 슬라이드 위에 부착되고 중합효소로 증폭되어 처음에 "DNA colony"라고 명명된 국소 클론 콜로니가 형성됩니다.서열을 결정하기 위해 네 가지 유형의 가역적 터미네이터 염기(RT-base)가 추가되고 비통합 뉴클레오티드가 세척됩니다.파이로 시퀀싱과는 달리 DNA 사슬은 한 번에 하나의 뉴클레오타이드로 확장되고 이미지 획득이 지연된 순간에 수행될 수 있으므로 매우 큰 배열의 DNA 집단이 단일 카메라에서 촬영된 순차 이미지에 의해 캡처될 수 있습니다.효소 반응과 이미지 캡처를 분리하면 최적의 처리량과 이론적으로 무제한의 시퀀싱 용량을 얻을 수 있습니다. 최적의 구성으로 기기의 궁극적인 처리량은 카메라의 A/D 변환 속도에만 의존합니다.카메라는 형광 표지가 있는 뉴클레오티드의 이미지를 찍은 다음, 염색약과 말단 3' 차단제를 DNA로부터 화학적으로 제거하여 다음 주기를 허용합니다.[63]
대안적인 접근법인 이온 반도체 서열 분석은 표준 DNA 복제 화학을 기반으로 합니다.이 기술은 염기가 들어갈 때마다 수소 이온의 방출을 측정합니다.주형 DNA를 포함하는 마이크로웰은 단일 뉴클레오티드로 넘쳐나며, 만약 뉴클레오티드가 주형 가닥과 상보적이면 그것은 통합되고 수소 이온이 방출됩니다.이 릴리스는 ISFET 이온 센서를 트리거합니다.주형 시퀀스에 단일 중합체가 존재하는 경우 여러 뉴클레오티드가 단일 홍수 주기로 통합되고 감지된 전기 신호는 비례적으로 더 높아집니다.[64]
어셈블
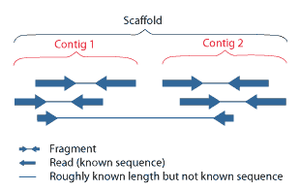
시퀀스 어셈블리는 원래 시퀀스를 재구성하기 위해 훨씬 더 긴 DNA 시퀀스의 조각을 정렬하고 병합하는 것을 말합니다.[9]이것은 현재의 DNA 염기서열결정 기술이 연속적인 서열로서 전체 게놈을 읽을 수 없고, 사용되는 기술에 따라 20개에서 1000개 사이의 염기의 작은 조각을 읽을 수 있기 때문에 필요합니다.PacBio 또는 Oxford Nanopore와 같은 3세대 시퀀싱 기술은 일반적으로 길이가 10kb를 초과하는 시퀀싱 판독치를 생성하지만, 이들은 약 15%의 높은 오류율을 가지고 있습니다.[65][66]일반적으로 읽기라고 불리는 짧은 조각들은 산탄총 염기서열 분석 유전자 DNA 또는 유전자 전사(EST)에서 비롯됩니다.[9]
조립 접근법
조립은 크게 두 가지 접근법으로 분류될 수 있습니다: 과거의 어떤 염기서열과도 유사하지 않은 유전체에 대한 드노보 조립(de novo assembly)[54]과 밀접한 관련이 있는 유기체의 기존 염기서열을 조립 중 기준으로 사용하는 비교 조립(comparative assembly).비교 어셈블리에 비해, novo 어셈블리는 계산적으로 어렵기 때문에(NP-hard), 단판독 NGS 기술에 덜 적합합니다.새로운 조립 패러다임 내에는 조립을 위한 두 가지 주요 전략, 오일러 경로 전략 및 OLC(overlap-layout-consensus) 전략이 있습니다.OLC 전략은 궁극적으로 NP-hard 문제인 중첩 그래프를 통해 해밀턴 경로를 생성하려고 합니다.오일러 경로 전략은 드브루인 그래프를 통해 오일러 경로를 찾으려 하기 때문에 계산적으로 더 다루기 쉽습니다.[54]
마무리
완성된 게놈은 각 복제자를 나타내는 모호성이 없는 연속된 단일 시퀀스를 갖는 것으로 정의됩니다.[67]
주석
DNA 염기서열 집합체는 추가 분석 없이는 거의 가치가 없습니다.[9]게놈 주석은 생물학적 정보를 염기서열에 붙이는 과정으로 크게 세 단계로 구성됩니다.[68]
자동 주석 도구는 인간의 전문 지식과 잠재적인 실험 검증을 수반하는 수동 주석(일명 큐레이션)과는 달리 이러한 단계를 실리코로 수행하려고 합니다.[69]이상적으로, 이 접근법들은 동일한 주석 파이프라인에서 서로 공존하고 보완합니다(아래 참조).
전통적으로 기본적인 주석 수준은 BLAST를 사용하여 유사점을 찾은 다음 상동체를 기반으로 게놈을 주석하는 것입니다.[9]최근에는 주석 플랫폼에 추가 정보가 추가됩니다.추가 정보를 통해 수동 주석자는 동일한 주석이 부여된 유전자 간의 불일치를 해소할 수 있습니다.일부 데이터베이스는 하위 시스템 접근 방식을 통해 게놈 주석을 제공하기 위해 게놈 컨텍스트 정보, 유사도 점수, 실험 데이터 및 다른 리소스의 통합을 사용합니다.다른 데이터베이스(예: Ensemble)는 자동화된 게놈 주석 파이프라인의 다양한 소프트웨어 도구뿐만 아니라 큐레이팅된 데이터 소스 모두에 의존합니다.[70]구조 주석은 주로 ORF와 그 지역화 또는 유전자 구조인 게놈 요소의 식별로 구성됩니다.기능 주석은 생물학적 정보를 게놈 요소에 부착하는 것으로 구성됩니다.
파이프라인 및 데이터베이스 시퀀싱
유전체 프로젝트와 관련된 많은 양의 데이터의 재현성과 효율적인 관리의 필요성은 계산 파이프라인이 유전체학에 중요한 응용 분야를 가지고 있음을 의미합니다.[71]
연구영역
기능유전체학
기능성 유전체학(Functional genomics)은 유전자(및 단백질) 기능과 상호 작용을 설명하기 위해 유전체 프로젝트(게놈 시퀀싱 프로젝트 등)에 의해 생성된 방대한 데이터를 활용하려는 분자생물학의 한 분야입니다.기능성 유전체학은 DNA 염기서열이나 구조와 같은 유전체 정보의 정적인 측면과는 반대로 유전자 전사, 번역, 단백질-단백질 상호작용과 같은 동적인 측면에 초점을 맞추고 있습니다.기능성 유전체학은 유전자, RNA 전사체 및 단백질 제품 수준에서 DNA의 기능에 대한 질문에 답하려고 시도합니다.기능성 게놈 연구의 핵심 특징은 이러한 질문에 대한 게놈 전체 접근 방식이며, 일반적으로 보다 전통적인 "유전자별" 접근 방식이 아닌 높은 처리량의 방법을 포함합니다.
유전체학의 주요 분야는 여전히 다양한 유기체의 유전체 배열 순서와 관련이 있지만, 완전한 유전체에 대한 지식은 주로 다양한 조건 동안의 유전자 발현 패턴과 관련된 기능성 유전체학 분야의 가능성을 만들어냈습니다.여기서 가장 중요한 도구는 마이크로어레이와 생물정보학입니다.
구조유전체학
구조유전체학은 주어진 유전체에 의해 암호화된 모든 단백질의 3차원 구조를 설명하고자 합니다.[72][73]이 게놈 기반 접근법은 실험적 접근법과 모델링적 접근법의 조합에 의한 구조 결정의 높은 처리량 방법을 가능하게 합니다.구조 유전체학과 전통적인 구조 예측의 주요한 차이점은 구조 유전체학이 하나의 특정한 단백질에 집중하는 것이 아니라, 유전체에 의해 암호화된 모든 단백질의 구조를 결정하려고 시도한다는 것입니다.풀-게놈 시퀀스를 사용할 수 있으므로 실험 및 모델링 접근 방식의 조합을 통해 구조 예측을 보다 신속하게 수행할 수 있습니다.특히 많은 수의 서열화된 유전체와 이전에 해결된 단백질 구조의 이용가능성이 과학자들이 이전에 해결된 상동체의 구조에 단백질 구조를 모델링할 수 있게 해주기 때문입니다.구조 유전체학은 알려진 구조의 단백질에 대한 서열 또는 구조 상동성에 기초한 유전체 서열 또는 모델링 기반 접근법을 사용하는 실험 방법을 포함하여 구조 결정에 대한 많은 접근법을 취하는 것을 포함합니다.전통적인 구조 생물학과는 대조적으로, 구조 유전체학적 노력을 통한 단백질 구조의 결정은 종종 (항상은 아니지만) 단백질 기능에 대해 알려진 것보다 우선합니다.이것은 구조 생물 정보학, 즉 3D 구조로부터 단백질 기능을 결정하는 데 새로운 도전을 제기합니다.[74]
후생유전학
후생유전학은 후생유전체로 알려진 세포의 유전물질에 대한 완전한 후생유전학적 변형에 대한 연구입니다.[75]후성유전학적 변형은 DNA 서열을 변경하지 않고 유전자 발현에 영향을 미치는 세포의 DNA 또는 히스톤에 대한 가역적인 변형입니다(Russell 2010 p. 475).후성유전학적 변형 중 가장 특징적인 두 가지는 DNA 메틸화와 히스톤 변형입니다.[76]후생유전학적 변형은 유전자 발현 및 조절에 중요한 역할을 하며, 분화/발달 및[77] 종양 발생과 같은 수많은 세포 과정에 관여합니다.[75]전 세계적인 수준에서 후생유전학에 대한 연구는 게놈 고처리량 분석법의 적용을 통해 최근에야 가능해졌습니다.[78]
메타게노믹스
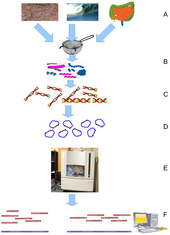
메타게노믹스는 환경 샘플에서 직접 회수한 유전 물질인 메타게노믹스를 연구하는 것입니다.광범위한 분야는 환경 유전체학, 생태 유전체학 또는 공동체 유전체학이라고도 불릴 수 있습니다.전통적인 미생물학과 미생물 게놈 시퀀싱은 배양된 클론 배양에 의존하는 반면, 초기 환경 유전자 시퀀싱은 자연 샘플에서 다양성의 프로파일을 생성하기 위해 복제된 특정 유전자(종종 16S rRNA 유전자)를 복제했습니다.이러한 연구는 미생물 생물 다양성의 대부분이 재배 기반 방법에 의해 누락되었음을 밝혀냈습니다.[79]최근의 연구들은 "샷건" 생어 염기서열 분석 또는 광범위한 병렬 파이로 염기서열 분석을 사용하여 표본이 추출된 공동체의 모든 구성원들로부터 대부분 편견이 없는 모든 유전자의 샘플을 얻습니다.[80]미생물의 다양성을 드러내는 힘 때문에, 메타게노믹스는 미생물 세계를 관찰하는 강력한 렌즈를 제공하며, 이는 생물 세계 전체에 대한 이해를 혁신할 가능성을 가지고 있습니다.[81][82]
모델 시스템
바이러스와 박테리오파지
박테리오파지는 박테리아 유전학과 분자생물학에서 중요한 역할을 해왔고 계속해서 중요한 역할을 하고 있습니다.역사적으로, 그들은 유전자 구조와 유전자 조절을 정의하는데 사용되었습니다.또한 최초로 배열된 게놈은 박테리오파지였습니다.그러나 박테리오파지 연구는 세균유전체학이 분명히 지배하는 유전체학 혁명을 이끌지 못했습니다.아주 최근에서야 박테리오파지 유전체에 대한 연구가 두드러져서, 연구자들이 파지 진화의 기초가 되는 메커니즘을 이해할 수 있게 되었습니다.박테리오파지 유전체 서열은 분리된 박테리오파지의 직접적인 염기서열 분석을 통해 얻을 수 있지만, 미생물 유전체의 일부로서도 유도될 수 있습니다.박테리아 유전체의 분석은 상당한 양의 미생물 DNA가 프로파지 서열과 프로파지 유사 요소로 구성되어 있음을 보여주었습니다.[83]이러한 시퀀스에 대한 상세한 데이터베이스 마이닝은 박테리아 게놈을 형성하는 데 있어 프로파지의 역할에 대한 통찰력을 제공합니다.전반적으로, 이 방법은 알려진 많은 박테리오파지 그룹을 검증했고, 이것을 박테리아 유전체로부터 프로파지의 관계를 예측하는 데 유용한 도구로 만들었습니다.[84][85]
시아노박테리아
현재 총 게놈 서열을 이용할 수 있는 24종의 시아노박테리아가 있습니다.이들 시아노박테리아 중 15종은 해양환경에서 유래합니다.이것들은 6개의 Prochlorococcus 균주, 7개의 해양 Synechoccus 균주, Trichodesmium erythraum IMS101 및 Crocosphaera watsonii WH8501입니다.몇몇 연구들은 어떻게 이 서열들이 해양 시아노박테리아의 중요한 생태학적, 생리학적 특징들을 추론하는데 매우 성공적으로 사용될 수 있는지 보여주었습니다.그러나 현재 진행 중인 유전체 프로젝트는 더 많은 것들이 있는데, 그 중에는 더 많은 Prochlorococcus 및 해양 Synechococcus isolates, Acaryochloris 및 Prochloron, N-fixing2 필라멘트 시아노박테리아 Nodularia spumigena, Lyngbya aestuarii 및 Lyngbya majuscula 및 해양 시아노박테리아 감염 박테리오파지가 있습니다.따라서, 유전체 정보의 증가하는 본체는 비교적 접근법을 적용함으로써 세계적인 문제를 해결하기 위한 보다 일반적인 방법으로 활용될 수 있습니다.이 분야에서 새롭고 흥미로운 진보의 예로는 조절 RNA에 대한 유전자 확인, 광합성의 진화적 기원에 대한 통찰, 또는 분석된 유전체에 대한 수평적 유전자 전달의 기여 추정 등이 있습니다.[86]
적용들

유전체학은 의학, 생명공학, 인류학, 그리고 다른 사회과학을 포함한 많은 분야에서 응용을 제공해 왔습니다.[44]
게놈의학
차세대 유전체 기술을 통해 임상의와 생체의학 연구자들은 대규모 연구 집단에서 수집되는 유전체 데이터의 양을 획기적으로 늘릴 수 있습니다.[87]질병 연구에서 유전체 데이터와 많은 종류의 데이터를 통합하는 새로운 정보학적 접근법과 결합하면 연구자들이 약물 반응과 질병의 유전적 기반을 더 잘 이해할 수 있습니다.[88][89]게놈을 의학에 적용하려는 초기의 노력에는 인간 게놈의 의학적 해석을 위한 최초의 도구를 개발한 유안 애슐리가 이끄는 스탠포드 팀의 노력이 포함되어 있습니다.[90][91][92]지놈즈2유전체학을 건강으로 바꾸는 실증연구를 위해 2012년 브리검 여성병원, 브로드 인스티튜트, 하버드 의과대학의 사람 연구 프로그램이 설립되었습니다.브리검 여성병원은 2019년 8월 예방 유전체학 클리닉을 열었고, 한 달 뒤 매사추세츠 종합병원이 문을 열었습니다.[93][94]All of Us 연구 프로그램은 100만 명의 참가자로부터 유전체 염기서열 데이터를 수집하여 정밀의학 연구 플랫폼의 중요한 구성요소가 되는 것을 목표로 하고 있습니다.[95]
합성생물학 및 생명공학
게놈 지식의 성장은 합성 생물학의 점점 더 정교한 응용을 가능하게 했습니다.[96]2010년 J. Craig Venter Institute의 연구원들은 마이코플라즈마 생식기의 게놈에서 유래한 부분적으로 합성된 박테리아 종인 마이코플라즈마 연구소를 만들었다고 발표했습니다.[97]
인구보전유전체학
인구 유전체학은 인구 유전학에서 전통적으로 사용되는 근거리 PCR 생성물 또는 마이크로위성과 같은 유전적 표지자의 한계를 넘어, 유전자 염기 서열 분석 방법이 인구 간 DNA 서열의 대규모 비교를 수행하는 데 사용되는 인기 있는 연구 분야로 발전했습니다.인구 유전체학은 유전체 전반에 걸친 영향을 연구하여 미세 진화에 대한 이해를 향상시켜 인구의 계통발생학적 역사와 인구통계학을 배울 수 있습니다.[98]집단 게놈 방법은 진화 생물학, 생태학, 생물 지리학, 보존 생물학, 그리고 어업 관리를 포함한 많은 다양한 분야에 사용됩니다.마찬가지로, 풍경 유전체학은 풍경 유전학에서 환경적 변화 패턴과 유전적 변화 간의 관계를 확인하기 위해 유전학적 방법을 사용하는 것으로 발전했습니다.
환경보호론자들은 개체수의 유전적 다양성이나 한 개인이 열성 유전성 질환에 대해 이형접합성이 있는지 여부와 같은 종 보존에 핵심적인 유전적 요인들을 더 잘 평가하기 위해 유전자 염기서열 분석을 통해 수집된 정보를 사용할 수 있습니다.[99]게놈 데이터를 사용하여 진화 과정의 효과를 평가하고 주어진 집단 전체에서 변화의 양상을 탐지함으로써, 보존가들은 표준 유전적 접근법에 의해 다루어지지 않은 변수들만큼 알려지지 않은 채로 주어진 종을 돕기 위한 계획을 세울 수 있습니다.[100]
참고 항목
참고문헌
- ^ Franklin RE, Gosling RG (April 1953). "Molecular configuration in sodium thymonucleate". Nature. 171 (4356): 740–1. Bibcode:1953Natur.171..740F. doi:10.1038/171740a0. PMID 13054694. S2CID 4268222.
- ^ Satzinger H (March 2008). "Theodor and Marcella Boveri: chromosomes and cytoplasm in heredity and development". Nature Reviews. Genetics. 9 (3): 231–238. doi:10.1038/nrg2311. PMID 18268510. S2CID 15829893.
- ^ Cremer T, Cremer C (2006). "Rise, fall and resurrection of chromosome territories: a historical perspective. Part I. The rise of chromosome territories". European Journal of Histochemistry. 50 (3): 161–176. PMID 16920639.
- ^ Rossi, M.J; Kuntala, P.K; Lai, W.K.M; Yamada, N; Badjatia, N; Mittal, C; Kuzu, G; Bocklund, K; Farrell, N.P; Blanda, T.R; Mairose, J.D; Basting, A.V; Mistretta, K.S; Rocco, D.J; Perkinson, E.S; Kellogg, G.D; Mahony, S; Pugh, B.F (March 2021). "A high-resolution protein architecture of the budding yeast genome". Nature. 592 (7853): 309–314. Bibcode:2021Natur.592..309R. doi:10.1038/s41586-021-03314-8. PMC 8035251. PMID 33692541.
- ^ "WHO definitions of genetics and genomics". World Health Organization. Archived from the original on June 30, 2004.
- ^ Concepts of genetics (10th ed.). San Francisco: Pearson Education. 2012. ISBN 978-0-321-72412-0.
- ^ Culver KW, Labow MA (8 November 2002). "Genomics". In Robinson R (ed.). Genetics. Macmillan Science Library. Macmillan Reference USA. ISBN 978-0-02-865606-9.
- ^ Kadakkuzha BM, Puthanveettil SV (July 2013). "Genomics and proteomics in solving brain complexity". Molecular BioSystems. 9 (7): 1807–1821. doi:10.1039/C3MB25391K. PMC 6425491. PMID 23615871.
- ^ a b c d e f g h i Pevsner J (2009). Bioinformatics and functional genomics (2nd ed.). Hoboken, NJ: Wiley-Blackwell. ISBN 978-0-470-08585-1.
- ^ Liddell HG, Scott R (2013). Intermediate Greek-English Lexicon. Martino Fine Books. ISBN 978-1-61427-397-4.
- ^ "Genome, n". Oxford English Dictionary (Third ed.). Oxford University Press. 2008. Retrieved 2012-12-01.(구독 필요)
- ^ Yadav SP (December 2007). "The wholeness in suffix -omics, -omes, and the word om". Journal of Biomolecular Techniques. 18 (5): 277. PMC 2392988. PMID 18166670.
- ^ O'Brien SJ (June 2022). "A decade of GigaScience: A perspective on conservation genetics". GigaScience. 11. doi:10.1093/gigascience/giac055. PMC 9197679. PMID 35701371.
- ^ Ankeny RA (June 2003). "Sequencing the genome from nematode to human: changing methods, changing science". Endeavour. 27 (2): 87–92. doi:10.1016/S0160-9327(03)00061-9. PMID 12798815.
- ^ Holley RW, Everett GA, Madison JT, Zamir A (May 1965). "Nucleotide sequences in the yeast alanine transfer ribonucleic acid". The Journal of Biological Chemistry. 240 (5): 2122–2128. doi:10.1016/S0021-9258(18)97435-1. PMID 14299636.
- ^ Holley RW, Apgar J, Everett GA, Madison JT, Marquisee M, Merrill SH, et al. (March 1965). "Structure of a Ribonucleic Acid". Science. 147 (3664): 1462–1465. Bibcode:1965Sci...147.1462H. doi:10.1126/science.147.3664.1462. PMID 14263761. S2CID 40989800.
- ^ Nirenberg M, Leder P, Bernfield M, Brimacombe R, Trupin J, Rottman F, O'Neal C (May 1965). "RNA codewords and protein synthesis, VII. On the general nature of the RNA code". Proceedings of the National Academy of Sciences of the United States of America. 53 (5): 1161–1168. Bibcode:1965PNAS...53.1161N. doi:10.1073/pnas.53.5.1161. PMC 301388. PMID 5330357.
- ^ Min Jou W, Haegeman G, Ysebaert M, Fiers W (May 1972). "Nucleotide sequence of the gene coding for the bacteriophage MS2 coat protein". Nature. 237 (5350): 82–88. Bibcode:1972Natur.237...82J. doi:10.1038/237082a0. PMID 4555447. S2CID 4153893.
- ^ Fiers W, Contreras R, Duerinck F, Haegeman G, Iserentant D, Merregaert J, et al. (April 1976). "Complete nucleotide sequence of bacteriophage MS2 RNA: primary and secondary structure of the replicase gene". Nature. 260 (5551): 500–507. Bibcode:1976Natur.260..500F. doi:10.1038/260500a0. PMID 1264203. S2CID 4289674.
- ^ Fiers W, Contreras R, Haegemann G, Rogiers R, Van de Voorde A, Van Heuverswyn H, et al. (May 1978). "Complete nucleotide sequence of SV40 DNA". Nature. 273 (5658): 113–120. Bibcode:1978Natur.273..113F. doi:10.1038/273113a0. PMID 205802. S2CID 1634424.
- ^ Tamarin RH (2004). Principles of genetics (7 ed.). London: McGraw Hill. ISBN 978-0-07-124320-9.
- ^ Sanger F (1980). "Nobel lecture: Determination of nucleotide sequences in DNA" (PDF). Nobelprize.org. Retrieved 2010-10-18.
- ^ a b Sanger F, Air GM, Barrell BG, Brown NL, Coulson AR, Fiddes CA, et al. (February 1977). "Nucleotide sequence of bacteriophage phi X174 DNA". Nature. 265 (5596): 687–695. Bibcode:1977Natur.265..687S. doi:10.1038/265687a0. PMID 870828. S2CID 4206886.
- ^ Kaiser O, Bartels D, Bekel T, Goesmann A, Kespohl S, Pühler A, Meyer F (December 2003). "Whole genome shotgun sequencing guided by bioinformatics pipelines--an optimized approach for an established technique". Journal of Biotechnology. 106 (2–3): 121–133. doi:10.1016/j.jbiotec.2003.08.008. PMID 14651855.
- ^ Sanger F, Nicklen S, Coulson AR (December 1977). "DNA sequencing with chain-terminating inhibitors". Proceedings of the National Academy of Sciences of the United States of America. 74 (12): 5463–5467. Bibcode:1977PNAS...74.5463S. doi:10.1073/pnas.74.12.5463. PMC 431765. PMID 271968.
- ^ Maxam AM, Gilbert W (February 1977). "A new method for sequencing DNA". Proceedings of the National Academy of Sciences of the United States of America. 74 (2): 560–564. Bibcode:1977PNAS...74..560M. doi:10.1073/pnas.74.2.560. PMC 392330. PMID 265521.
- ^ a b Darden L, Tabery J (2010). "Molecular Biology". In Zalta EN (ed.). The Stanford Encyclopedia of Philosophy (Fall 2010 ed.).
- ^ Anderson S, Bankier AT, Barrell BG, de Bruijn MH, Coulson AR, Drouin J, et al. (April 1981). "Sequence and organization of the human mitochondrial genome". Nature. 290 (5806): 457–465. Bibcode:1981Natur.290..457A. doi:10.1038/290457a0. PMID 7219534. S2CID 4355527.(구독 필요)
- ^ Shinozaki K, Ohme M, Tanaka M, Wakasugi T, Hayashida N, Matsubayashi T, et al. (September 1986). "The complete nucleotide sequence of the tobacco chloroplast genome: its gene organization and expression". The EMBO Journal. 5 (9): 2043–2049. doi:10.1002/j.1460-2075.1986.tb04464.x. PMC 1167080. PMID 16453699.
- ^ Ohyama K, Fukuzawa H, Kohchi T, Shirai H, Sano T, Sano S, et al. (1986). "Chloroplast gene organization deduced from complete sequence of liverwort Marchantia polymorpha chloroplast DNA". Nature. 322 (6079): 572–574. Bibcode:1986Natur.322..572O. doi:10.1038/322572a0. S2CID 4311952.
- ^ Oliver SG, van der Aart QJ, Agostoni-Carbone ML, Aigle M, Alberghina L, Alexandraki D, et al. (May 1992). "The complete DNA sequence of yeast chromosome III". Nature. 357 (6373): 38–46. Bibcode:1992Natur.357...38O. doi:10.1038/357038a0. PMID 1574125. S2CID 4271784.
- ^ Fleischmann RD, Adams MD, White O, Clayton RA, Kirkness EF, Kerlavage AR, et al. (July 1995). "Whole-genome random sequencing and assembly of Haemophilus influenzae Rd". Science. 269 (5223): 496–512. Bibcode:1995Sci...269..496F. doi:10.1126/science.7542800. PMID 7542800. S2CID 10423613.
- ^ Goffeau A, Barrell BG, Bussey H, Davis RW, Dujon B, Feldmann H, et al. (October 1996). "Life with 6000 genes". Science. 274 (5287): 546, 563–546, 567. Bibcode:1996Sci...274..546G. doi:10.1126/science.274.5287.546. PMID 8849441. S2CID 211123134.(구독 필요)
- ^ "Complete genomes: Viruses". NCBI. 17 November 2011. Retrieved 2011-11-18.
- ^ "Genome Project Statistics". Entrez Genome Project. 7 October 2011. Retrieved 2011-11-18.
- ^ Zimmer C (29 December 2009). "Scientists Start a Genomic Catalog of Earth's Abundant Microbes". The New York Times. ISSN 0362-4331. Retrieved 2012-12-21.
- ^ Wu D, Hugenholtz P, Mavromatis K, Pukall R, Dalin E, Ivanova NN, et al. (December 2009). "A phylogeny-driven genomic encyclopaedia of Bacteria and Archaea". Nature. 462 (7276): 1056–1060. Bibcode:2009Natur.462.1056W. doi:10.1038/nature08656. PMC 3073058. PMID 20033048.
- ^ "Human gene number slashed". BBC. 20 October 2004. Retrieved 2012-12-21.
- ^ Yue GH, Lo LC, Zhu ZY, Lin G, Feng F (April 2006). "The complete nucleotide sequence of the mitochondrial genome of Tetraodon nigroviridis". DNA Sequence. 17 (2): 115–121. doi:10.1080/10425170600700378. PMID 17076253. S2CID 21797344.
- ^ National Human Genome Research Institute (14 July 2004). "Dog Genome Assembled: Canine Genome Now Available to Research Community Worldwide". Genome.gov. Retrieved 2012-01-20.
- ^ a b McElheny V (2010). Drawing the map of life: inside the Human Genome Project. New York NY: Basic Books. ISBN 978-0-465-04333-0.
- ^ Abecasis GR, Auton A, Brooks LD, DePristo MA, Durbin RM, Handsaker RE, et al. (November 2012). "An integrated map of genetic variation from 1,092 human genomes". Nature. 491 (7422): 56–65. Bibcode:2012Natur.491...56T. doi:10.1038/nature11632. PMC 3498066. PMID 23128226.
- ^ Nielsen R (October 2010). "Genomics: In search of rare human variants". Nature. 467 (7319): 1050–1051. Bibcode:2010Natur.467.1050N. doi:10.1038/4671050a. PMID 20981085.
- ^ a b Barnes B, Dupré J (2008). Genomes and what to make of them. Chicago: University of Chicago Press. ISBN 978-0-226-17295-8.
- ^ Eisen JA (July 2012). "Badomics words and the power and peril of the ome-meme". GigaScience. 1 (1): 6. doi:10.1186/2047-217X-1-6. PMC 3617454. PMID 23587201.
- ^ Hotz RL (13 August 2012). "Here"s an Omical Tale: Scientists Discover Spreading Suffix". Wall Street Journal. ISSN 0099-9660. Retrieved 2013-01-04.
- ^ Scudellari M (1 October 2011). "Data Deluge". The Scientist. Retrieved 2013-01-04.
- ^ Chaston J, Douglas AE (August 2012). "Making the most of "omics" for symbiosis research". The Biological Bulletin. 223 (1): 21–29. doi:10.1086/BBLv223n1p21. PMC 3491573. PMID 22983030.
- ^ McCutcheon JP, von Dohlen CD (August 2011). "An interdependent metabolic patchwork in the nested symbiosis of mealybugs". Current Biology. 21 (16): 1366–1372. doi:10.1016/j.cub.2011.06.051. PMC 3169327. PMID 21835622.
- ^ a b Baker M (14 September 2012). "Benchtop sequencers ship off" (Blog). Nature News Blog. Retrieved 2012-12-22.
- ^ Quail MA, Smith M, Coupland P, Otto TD, Harris SR, Connor TR, et al. (July 2012). "A tale of three next generation sequencing platforms: comparison of Ion Torrent, Pacific Biosciences and Illumina MiSeq sequencers". BMC Genomics. 13: 341. doi:10.1186/1471-2164-13-341. PMC 3431227. PMID 22827831.
- ^ a b Staden R (June 1979). "A strategy of DNA sequencing employing computer programs". Nucleic Acids Research. 6 (7): 2601–2610. doi:10.1093/nar/6.7.2601. PMC 327874. PMID 461197.
- ^ Anderson S (July 1981). "Shotgun DNA sequencing using cloned DNase I-generated fragments". Nucleic Acids Research. 9 (13): 3015–3027. doi:10.1093/nar/9.13.3015. PMC 327328. PMID 6269069.
- ^ a b c Pop M (July 2009). "Genome assembly reborn: recent computational challenges". Briefings in Bioinformatics. 10 (4): 354–366. doi:10.1093/bib/bbp026. PMC 2691937. PMID 19482960.
- ^ Sanger F, Coulson AR (May 1975). "A rapid method for determining sequences in DNA by primed synthesis with DNA polymerase". Journal of Molecular Biology. 94 (3): 441–448. doi:10.1016/0022-2836(75)90213-2. PMID 1100841.
- ^ Mavromatis K, Land ML, Brettin TS, Quest DJ, Copeland A, Clum A, et al. (2012). Liu Z (ed.). "The fast changing landscape of sequencing technologies and their impact on microbial genome assemblies and annotation". PLOS ONE. 7 (12): e48837. Bibcode:2012PLoSO...748837M. doi:10.1371/journal.pone.0048837. PMC 3520994. PMID 23251337.
- ^ Illumina, Inc. (28 February 2012). An Introduction to Next-Generation Sequencing Technology (PDF). San Diego, California, USA: Illumina, Inc. p. 12. Retrieved 2012-12-28.
- ^ Hall N (May 2007). "Advanced sequencing technologies and their wider impact in microbiology". The Journal of Experimental Biology. 210 (Pt 9): 1518–1525. doi:10.1242/jeb.001370. PMID 17449817.
- ^ Church GM (January 2006). "Genomes for all". Scientific American. 294 (1): 46–54. Bibcode:2006SciAm.294a..46C. doi:10.1038/scientificamerican0106-46. PMID 16468433. S2CID 28769137.
- ^ ten Bosch JR, Grody WW (November 2008). "Keeping up with the next generation: massively parallel sequencing in clinical diagnostics". The Journal of Molecular Diagnostics. 10 (6): 484–492. doi:10.2353/jmoldx.2008.080027. PMC 2570630. PMID 18832462.
- ^ Tucker T, Marra M, Friedman JM (August 2009). "Massively parallel sequencing: the next big thing in genetic medicine". American Journal of Human Genetics. 85 (2): 142–154. doi:10.1016/j.ajhg.2009.06.022. PMC 2725244. PMID 19679224.
- ^ US 20050100900, 카와시마 EH, 파리넬리 L, 메이어 P, "핵산 증폭 방법", 2005년 5월 12일 출판, 2011년 7월 26일 발행, Sollexa Ltd Great Britain.
- ^ Mardis ER (2008). "Next-generation DNA sequencing methods" (PDF). Annual Review of Genomics and Human Genetics. 9: 387–402. doi:10.1146/annurev.genom.9.081307.164359. PMID 18576944. Archived from the original (PDF) on 2013-05-18. Retrieved 2013-01-04.
- ^ Davies K (2011). "Powering Preventative Medicine". Bio-IT World (September–October).
- ^ "Home". PacBio.
- ^ "home". Oxford Nanopore Technologies.
- ^ Chain PS, Grafham DV, Fulton RS, Fitzgerald MG, Hostetler J, Muzny D, et al. (October 2009). "Genomics. Genome project standards in a new era of sequencing". Science. 326 (5950): 236–237. Bibcode:2009Sci...326..236C. doi:10.1126/science.1180614. PMC 3854948. PMID 19815760.
- ^ Stein L (July 2001). "Genome annotation: from sequence to biology". Nature Reviews. Genetics. 2 (7): 493–503. doi:10.1038/35080529. PMID 11433356. S2CID 12044602.
- ^ Brent MR (January 2008). "Steady progress and recent breakthroughs in the accuracy of automated genome annotation" (PDF). Nature Reviews. Genetics. 9 (1): 62–73. doi:10.1038/nrg2220. PMID 18087260. S2CID 20412451. Archived from the original (PDF) on 2013-05-29. Retrieved 2013-01-04.
- ^ Flicek P, Ahmed I, Amode MR, Barrell D, Beal K, Brent S, et al. (January 2013). "Ensembl 2013". Nucleic Acids Research. 41 (Database issue): D48–D55. doi:10.1093/nar/gks1236. PMC 3531136. PMID 23203987.
- ^ Keith JM (2008). Keith JM (ed.). Bioinformatics. Methods in Molecular Biology. Vol. 453. pp. v–vi. doi:10.1007/978-1-60327-429-6. ISBN 978-1-60327-428-9. PMID 18720577.
- ^ Marsden RL, Lewis TA, Orengo CA (March 2007). "Towards a comprehensive structural coverage of completed genomes: a structural genomics viewpoint". BMC Bioinformatics. 8: 86. doi:10.1186/1471-2105-8-86. PMC 1829165. PMID 17349043.
- ^ Brenner SE, Levitt M (January 2000). "Expectations from structural genomics". Protein Science. 9 (1): 197–200. doi:10.1110/ps.9.1.197. PMC 2144435. PMID 10739263.
- ^ Brenner SE (October 2001). "A tour of structural genomics". Nature Reviews. Genetics. 2 (10): 801–809. doi:10.1038/35093574. PMID 11584296. S2CID 5656447.
- ^ a b Francis RC (2011). Epigenetics: the ultimate mystery of inheritance. New York: WW Norton. ISBN 978-0-393-07005-7.
- ^ Gallego, L.D; Schneider, M; Mittal, C; Romanuska, Anete; Gudino Carrillo, R.M; Schubert, T; Pugh, B.F; Kohler, A (March 2020). "Phase separation directs ubiquitination of gene-body nucleosomes". Nature. 579 (7800): 592–597. Bibcode:2020Natur.579..592G. doi:10.1038/s41586-020-2097-z. PMC 7481934. PMID 32214243.
- ^ Sams, K.L; Mukai, C; Marks, B.A; Mittal, C; Demeter, E.A; Nelissen, S; Grenier, J.K; Tate, A.E; Ahmed, F; Coonrod, S.A (October 2022). "Delayed puberty, gonadotropin abnormalities and subfertility in male Padi2/Padi4 double knockout mice". Reprod Biol Endocrinol. 20 (1): 150. doi:10.1186/s12958-022-01018-w. PMC 9555066. PMID 36224627.
- ^ Laird PW (March 2010). "Principles and challenges of genomewide DNA methylation analysis". Nature Reviews. Genetics. 11 (3): 191–203. doi:10.1038/nrg2732. PMID 20125086. S2CID 6780101.
- ^ Hugenholtz P, Goebel BM, Pace NR (September 1998). "Impact of culture-independent studies on the emerging phylogenetic view of bacterial diversity". Journal of Bacteriology. 180 (18): 4765–4774. doi:10.1128/JB.180.18.4765-4774.1998. PMC 107498. PMID 9733676.
- ^ Eisen JA (March 2007). "Environmental shotgun sequencing: its potential and challenges for studying the hidden world of microbes". PLOS Biology. 5 (3): e82. doi:10.1371/journal.pbio.0050082. PMC 1821061. PMID 17355177.
- ^ Marco D, ed. (2010). Metagenomics: Theory, Methods and Applications. Caister Academic Press. ISBN 978-1-904455-54-7.
- ^ Marco D, ed. (2011). Metagenomics: Current Innovations and Future Trends. Caister Academic Press. ISBN 978-1-904455-87-5.
- ^ Canchaya C, Proux C, Fournous G, Bruttin A, Brüssow H (June 2003). "Prophage genomics". Microbiology and Molecular Biology Reviews. 67 (2): 238–76, table of contents. doi:10.1128/MMBR.67.2.238-276.2003. PMC 156470. PMID 12794192.
- ^ McGrath S, van Sinderen D, eds. (2007). Bacteriophage: Genetics and Molecular Biology (1st ed.). Caister Academic Press. ISBN 978-1-904455-14-1.
- ^ Fouts DE (November 2006). "Phage_Finder: automated identification and classification of prophage regions in complete bacterial genome sequences". Nucleic Acids Research. 34 (20): 5839–5851. doi:10.1093/nar/gkl732. PMC 1635311. PMID 17062630.
- ^ Herrero A, Flores E, eds. (2008). The Cyanobacteria: Molecular Biology, Genomics and Evolution (1st ed.). Caister Academic Press. ISBN 978-1-904455-15-8.
- ^ Hudson KL (September 2011). "Genomics, health care, and society". The New England Journal of Medicine. 365 (11): 1033–1041. doi:10.1056/NEJMra1010517. PMID 21916641.
- ^ O'Donnell CJ, Nabel EG (December 2011). "Genomics of cardiovascular disease". The New England Journal of Medicine. 365 (22): 2098–2109. doi:10.1056/NEJMra1105239. PMID 22129254.
- ^ Lu YF, Goldstein DB, Angrist M, Cavalleri G (July 2014). "Personalized medicine and human genetic diversity". Cold Spring Harbor Perspectives in Medicine. 4 (9): a008581. doi:10.1101/cshperspect.a008581. PMC 4143101. PMID 25059740.
- ^ Ashley EA, Butte AJ, Wheeler MT, Chen R, Klein TE, Dewey FE, et al. (May 2010). "Clinical assessment incorporating a personal genome". Lancet. 375 (9725): 1525–1535. doi:10.1016/S0140-6736(10)60452-7. PMC 2937184. PMID 20435227.
- ^ Dewey FE, Chen R, Cordero SP, Ormond KE, Caleshu C, Karczewski KJ, et al. (September 2011). "Phased whole-genome genetic risk in a family quartet using a major allele reference sequence". PLOS Genetics. 7 (9): e1002280. doi:10.1371/journal.pgen.1002280. PMC 3174201. PMID 21935354.
- ^ Dewey FE, Grove ME, Pan C, Goldstein BA, Bernstein JA, Chaib H, et al. (March 2014). "Clinical interpretation and implications of whole-genome sequencing". JAMA. 311 (10): 1035–1045. doi:10.1001/jama.2014.1717. PMC 4119063. PMID 24618965.
- ^ Robbins R (16 August 2019). "Top U.S. medical centers roll out DNA sequencing clinics for healthy (and often wealthy) clients". STAT News.
- ^ "Two Boston Health Systems Enter the Growing Direct-to-Consumer Gene Sequencing Market by Opening Preventative Genomics Clinics, but Can Patients Afford the Service?". Dark Daily. The Dark Intelligence Group. 3 January 2020.
- ^ "NIH-funded genome centers to accelerate precision medicine discoveries". National Institutes of Health: All of Us Research Program. National Institutes of Health. 25 September 2018.
- ^ Church GM, Regis E (2012). Regenesis: how synthetic biology will reinvent nature and ourselves. New York: Basic Books. ISBN 978-0-465-02175-8.
- ^ Baker M (May 2011). "Synthetic genomes: The next step for the synthetic genome". Nature. 473 (7347): 403, 405–403, 408. Bibcode:2011Natur.473..403B. doi:10.1038/473403a. PMID 21593873. S2CID 205064528.
- ^ Luikart G, England PR, Tallmon D, Jordan S, Taberlet P (December 2003). "The power and promise of population genomics: from genotyping to genome typing". Nature Reviews. Genetics. 4 (12): 981–94. doi:10.1038/nrg1226. PMID 14631358. S2CID 8516357.
- ^ Frankham R (1 September 2010). "Challenges and opportunities of genetic approaches to biological conservation". Biological Conservation. 143 (9): 1922–1923. doi:10.1016/j.biocon.2010.05.011.
- ^ Allendorf FW, Hohenlohe PA, Luikart G (October 2010). "Genomics and the future of conservation genetics". Nature Reviews. Genetics. 11 (10): 697–709. doi:10.1038/nrg2844. PMID 20847747. S2CID 10811958.
추가열람
- Lesk AM (2017). Introduction to Genomics (3rd ed.). New York: Oxford University Press. p. 544. ISBN 978-0-19-107085-3. ASIN 0198754833.
- Stunnenberg HG, Hubner NC (June 2014). "Genomics meets proteomics: identifying the culprits in disease". Human Genetics. 133 (6): 689–700. doi:10.1007/s00439-013-1376-2. PMC 4021166. PMID 24135908.
- Shibata T (October 2012). "Cancer genomics and pathology: all together now". Pathology International. 62 (10): 647–659. doi:10.1111/j.1440-1827.2012.02855.x. PMID 23005591. S2CID 27886018.
- Roychowdhury S, Chinnaiyan AM (2016). "Translating cancer genomes and transcriptomes for precision oncology". CA. 66 (1): 75–88. doi:10.3322/caac.21329. PMC 4713245. PMID 26528881.
- Gladyshev VN, Zhang Y (2013). "Chapter 16 Comparative Genomics Analysis of the Metallomes". In Banci L (ed.). Metallomics and the Cell. Metal Ions in Life Sciences. Vol. 12. Springer. doi:10.1007/978-94-007-5561-10_16 (inactive 1 August 2023). ISBN 978-94-007-5560-4.Gladyshev VN, Zhang Y (2013). "Chapter 16 Comparative Genomics Analysis of the Metallomes". In Banci L (ed.). Metallomics and the Cell. Metal Ions in Life Sciences. Vol. 12. Springer. doi:10.1007/978-94-007-5561-10_16 (inactive 1 August 2023). ISBN 978-94-007-5560-4.
{{cite book}}CS1 메인 : 2023년 8월 현재 DOI 비활성화 (링크) 전자도서 ISBN 978-94-007-5561-1 ISSN 1559-0836 전자-ISSN 1868-0402
외부 링크
- Wayback Machine에서 2009-01-18년 보관된 게놈 및 인간 유전학 연례 리뷰
- BMC 유전체학: 유전체학에 관한 BMC 저널
- 유전체학 저널
- Genomics.org : 열린 무료 유전체학 포털.
- NHGRI: 미국 정부의 게놈 연구소
- JCVI 종합 미생물 자원
- KoreaGenome.org :최초의 한국인 게놈이 출판되었고 염기서열은 자유롭게 이용할 수 있습니다.
- 유전체 네트워크 : 유전체학의 과학기술의 발전과 활용을 살펴봅니다.
- 유전체 과학 연구소:게놈 연구.
- MIT 오픈코스웨어 HST.512 게놈의학 무료, 자습 과정 유전의학자료에는 오디오 강의와 엄선된 강의 노트가 포함됩니다.
- ENCODE 스레드 탐색기 유전체학에 대한 기계 학습 접근법.네이처(저널)
- 유전체학 실험실의 글로벌 지도
- 게놈학:자연교육에 의한 스케이터링