바이로메
Virome| 시리즈의 일부 |
| 마이크로바이옴 |
|---|
 |
바이롬은 특정 생태계, 유기체 또는 홀로비온트와 관련된 바이러스 핵산의 메타제노믹 시퀀싱에 의해 종종 조사되고 기술되는 바이러스의[1][2] 집합체를 말한다.이 단어는 환경 바이러스성 산탄총 메타제놈을 묘사하기 위해 자주 사용된다.박테리오파지를 포함한 바이러스는 모든 환경에서 발견되며, 바이롬에 대한 연구는 용원 전환을 [7]통해 영양 순환,[4][5] 면역 [6]발달, 그리고 유전자의 주요 공급원에 대한 통찰력을 제공해 왔다.
역사
바이러스군에 대한 첫 번째 포괄적인 연구는 엽총 군집 [8]시퀀싱에 의한 것이었는데, 이것은 종종 메타게노믹스로 언급된다.2000년대에 Rower 연구소는 바닷물,[8][9] 해양 퇴적물,[10] 성인 변,[11] 유아 변,[12] 토양,[13] [14]혈액에서 바이롬의 염기서열을 분석했습니다.이 그룹은 또한 싱가포르 [15]유전체 연구소의 공동 연구자들과 함께 첫 번째 RNA 바이롬을 수행했다.이러한 초기 연구로부터, 게놈 다양성의 대부분은 지구 바이러스군에 포함되어 있으며, 이 다양성의 대부분은 [16]특징지어지지 않은 채 남아 있다는 결론이 내려졌다.이러한 견해는 개별 게놈 배열 분석 프로젝트, 특히 마이코박테륨 파지에 [17]의해 뒷받침되었다.
2010년대 후반까지 염기서열 분석 기술의 진보로 바이러스군을 [18]심층적으로 조사할 수 있게 되었다.특히 인간 내장의 바이롬은 이러한 [19][20]발전의 결과로 더욱 주목을 받고 있다.
학습 방법
바이롬을 연구하기 위해, 바이러스 같은 입자들은 세포 성분으로부터 분리되며, 보통 여과, 밀도 원심 분리, 그리고 유리 [21]핵산을 제거하기 위한 효소 처리의 조합을 사용합니다.그런 다음 메타게노믹 방법을 사용하여 핵산을 배열하고 분석합니다.또는 바이러스를 검출하기 위해 직접 메타제노믹 [22]조립 시퀀스를 사용하는 최근 계산 방법이 있습니다.
GOV(Global Ocean Viromes)는 국제 [23]팀이 두 번의 조사 기간 동안 전 세계 해양에서 수집한 150개 이상의 샘플에서 심층 시퀀싱으로 구성된 데이터 세트입니다.
바이러스 호스트
바이러스는 지구상에서 가장 풍부한 생물학적 실체이지만, 알려지지 않은 바이러스를 검출, 분리, 분류하는 데 어려움이 있어 전지구 바이러스군에 [24]대한 철저한 조사를 방해하고 있습니다.바이러스의 글로벌 분포, 계통 다양성 및 숙주 특이성을 평가하기 위해 지리적으로 다양한 3,042개의 샘플에서 [24]5 Tb 이상의 메타게노믹 배열 데이터가 사용되었습니다.
2016년 8월, 지금까지 확인된 가장 큰 파지를 포함한 125,000개 이상의 부분 DNA 바이러스 게놈이 알려진 바이러스 유전자의 수를 16배 [24]증가시켰다.추정 호스트 바이러스 [24]연결을 식별하기 위해 일련의 계산 방법이 사용되었습니다.격리된 바이러스 숙주 정보가 그룹에 투영되어 바이러스 [24]그룹의 2.4%에 숙주가 할당되었습니다.
그리고 이전에 [24]숙주를 감염시킨 페이저(프로토 스페이서)의 게놈 조각의 "라이브러리"를 보유하는 CRISPR-Cas 원핵 면역 체계.메타제노믹 바이러스 콘티그(mVCs)와 일치하는 미생물 게놈에서 분리된 스페이서는 바이러스 그룹의 4.4%, 싱글톤의 [24]1.7%에 대해 확인되었다.바이러스 전달 RNA(tRNA) 유전자가 [24]숙주로부터 유래한다는 가설을 탐구했다.
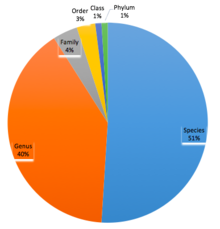
mVC의 7.6%에서 확인된 바이러스 tRNA는 단일 종 또는 [24]속으로부터 게놈을 분리하기 위해 매칭되었다.tRNA 기반 숙주 바이러스 할당의 특이성은 속 수준에서 94% 일치함을 나타내는 CRISPR-Cas 스페이서 매칭에 의해 확인되었다.이러한 접근방식을 통해 mVC의 [24]7.7%에 호스트를 할당할 수 있는 추정 호스트-바이러스 관련성을 9,992개 확인했습니다.이러한 접속의 대부분은 이전에는 알려지지 않았으며, 이전에는 바이러스가 [24]확인되지 않은 16개의 원핵생물 Phyla의 호스트를 포함합니다.
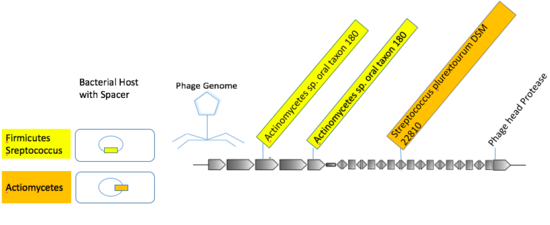
많은 바이러스는 관련 [24]호스트를 감염시키는 것을 전문으로 합니다.여러 분류 체계에 걸쳐 숙주를 감염시키는 바이러스 일반론자가 [24]존재할 수 있습니다.대부분의 CRISPR 스페이서 매칭은 바이러스 염기서열에서 한 종 또는 한 [24]속 내의 숙주까지였습니다.일부 mVC는 더 높은 분류에서 여러 호스트에 링크되었습니다.인간의 구강 샘플에서 추출한 맥으로 구성된 바이러스 그룹에는 Actionbacteria와 Bacilota의 [24]스페이서와 거의 정확히 일치하는 세 개의 다른 광 스페이서가 포함되어 있습니다.
2017년 1월, 가장 큰 인터랙티브 퍼블릭 바이러스 데이터베이스인 IMG/VR 시스템은 265,000개의 메타게노믹 바이러스 염기서열과 격리 바이러스를 포함하였습니다.이 숫자는 2018년 11월에 최대 760,000까지 확장되었습니다(IMG/VR v.2.0).[26]IMG/VR 시스템은 메타제노믹 샘플에서 파생된 바이러스 조각의 시퀀스 분석을 위한 시작 지점 역할을 합니다.
「 」를 참조해 주세요.
레퍼런스
- ^ Anderson NG, Gerin JL, Anderson NL (July 2003). "Global screening for human viral pathogens". Emerging Infectious Diseases. 9 (7): 768–774. doi:10.3201/eid0907.030004. PMC 3023425. PMID 12890315.
- ^ Zárate S, Taboada B, Yocupicio-Monroy M, Arias CF (November 2017). "Human Virome". Archives of Medical Research. 48 (8): 701–716. doi:10.1016/j.arcmed.2018.01.005. PMID 29398104.
- ^ McDaniel L, Breitbart M, Mobberley J, Long A, Haynes M, Rohwer F, Paul JH (September 2008). "Metagenomic analysis of lysogeny in Tampa Bay: implications for prophage gene expression". PLOS ONE. 3 (9): e3263. Bibcode:2008PLoSO...3.3263M. doi:10.1371/journal.pone.0003263. PMC 2533394. PMID 18810270.
- ^ Wilhelm, Steven W.; Suttle, Curtis A. (1999). "Viruses and Nutrient Cycles in the Sea". BioScience. 49 (10): 781–788. doi:10.2307/1313569. ISSN 1525-3244. JSTOR 1313569.
- ^ Wegley L, Edwards R, Rodriguez-Brito B, Liu H, Rohwer F (November 2007). "Metagenomic analysis of the microbial community associated with the coral Porites astreoides". Environmental Microbiology. 9 (11): 2707–2719. doi:10.1111/j.1462-2920.2007.01383.x. PMID 17922755.
- ^ Barr JJ, Auro R, Furlan M, Whiteson KL, Erb ML, Pogliano J, et al. (June 2013). "Bacteriophage adhering to mucus provide a non-host-derived immunity". Proceedings of the National Academy of Sciences of the United States of America. 110 (26): 10771–10776. Bibcode:2013PNAS..11010771B. doi:10.1073/pnas.1305923110. PMC 3696810. PMID 23690590.
- ^ Sharon I, Battchikova N, Aro EM, Giglione C, Meinnel T, Glaser F, et al. (July 2011). "Comparative metagenomics of microbial traits within oceanic viral communities". The ISME Journal. 5 (7): 1178–1190. doi:10.1038/ismej.2011.2. PMC 3146289. PMID 21307954.
- ^ a b Breitbart M, Salamon P, Andresen B, Mahaffy JM, Segall AM, Mead D, et al. (October 2002). "Genomic analysis of uncultured marine viral communities". Proceedings of the National Academy of Sciences of the United States of America. 99 (22): 14250–14255. Bibcode:2002PNAS...9914250B. doi:10.1073/pnas.202488399. PMC 137870. PMID 12384570.
- ^ Angly FE, Felts B, Breitbart M, Salamon P, Edwards RA, Carlson C, et al. (November 2006). "The marine viromes of four oceanic regions". PLOS Biology. 4 (11): e368. doi:10.1371/journal.pbio.0040368. PMC 1634881. PMID 17090214.
- ^ Breitbart M, Felts B, Kelley S, Mahaffy JM, Nulton J, Salamon P, Rohwer F (March 2004). "Diversity and population structure of a near-shore marine-sediment viral community". Proceedings. Biological Sciences. 271 (1539): 565–574. doi:10.1098/rspb.2003.2628. PMC 1691639. PMID 15156913.
- ^ Breitbart M, Hewson I, Felts B, Mahaffy JM, Nulton J, Salamon P, Rohwer F (October 2003). "Metagenomic analyses of an uncultured viral community from human feces". Journal of Bacteriology. 185 (20): 6220–6223. doi:10.1128/jb.185.20.6220-6223.2003. PMC 225035. PMID 14526037.
- ^ Breitbart M, Haynes M, Kelley S, Angly F, Edwards RA, Felts B, et al. (June 2008). "Viral diversity and dynamics in an infant gut". Research in Microbiology. 159 (5): 367–373. doi:10.1016/j.resmic.2008.04.006. PMID 18541415.
- ^ Fierer N, Breitbart M, Nulton J, Salamon P, Lozupone C, Jones R, et al. (November 2007). "Metagenomic and small-subunit rRNA analyses reveal the genetic diversity of bacteria, archaea, fungi, and viruses in soil". Applied and Environmental Microbiology. 73 (21): 7059–7066. Bibcode:2007ApEnM..73.7059F. doi:10.1128/aem.00358-07. PMC 2074941. PMID 17827313.
- ^ Breitbart M, Rohwer F (November 2005). "Method for discovering novel DNA viruses in blood using viral particle selection and shotgun sequencing". BioTechniques. 39 (5): 729–736. doi:10.2144/000112019. PMID 16312220.
- ^ Zhang T, Breitbart M, Lee WH, Run JQ, Wei CL, Soh SW, et al. (January 2006). "RNA viral community in human feces: prevalence of plant pathogenic viruses". PLOS Biology. 4 (1): e3. doi:10.1371/journal.pbio.0040003. PMC 1310650. PMID 16336043.
- ^ Edwards RA, Rohwer F (June 2005). "Viral metagenomics". Nature Reviews. Microbiology. 3 (6): 504–510. doi:10.1038/nrmicro1163. PMID 15886693. S2CID 8059643.
- ^ Rohwer F (April 2003). "Global phage diversity". Cell. 113 (2): 141. doi:10.1016/s0092-8674(03)00276-9. PMID 12705861.
- ^ Garmaeva S, Sinha T, Kurilshikov A, Fu J, Wijmenga C, Zhernakova A (October 2019). "Studying the gut virome in the metagenomic era: challenges and perspectives". BMC Biology. 17 (1): 84. doi:10.1186/s12915-019-0704-y. PMC 6819614. PMID 31660953.
- ^ Shkoporov AN, Clooney AG, Sutton TD, Ryan FJ, Daly KM, Nolan JA, et al. (October 2019). "The Human Gut Virome Is Highly Diverse, Stable, and Individual Specific". Cell Host & Microbe. 26 (4): 527–541.e5. doi:10.1016/j.chom.2019.09.009. PMID 31600503. S2CID 204242937.
- ^ Clooney AG, Sutton TD, Shkoporov AN, Holohan RK, Daly KM, O'Regan O, et al. (December 2019). "Whole-Virome Analysis Sheds Light on Viral Dark Matter in Inflammatory Bowel Disease". Cell Host & Microbe. 26 (6): 764–778.e5. doi:10.1016/j.chom.2019.10.009. PMID 31757768. S2CID 208234961.
- ^ Thurber RV, Haynes M, Breitbart M, Wegley L, Rohwer F (2009). "Laboratory procedures to generate viral metagenomes". Nature Protocols. 4 (4): 470–483. doi:10.1038/nprot.2009.10. PMID 19300441. S2CID 205464352.
- ^ Paez-Espino D, Pavlopoulos GA, Ivanova NN, Kyrpides NC (August 2017). "Nontargeted virus sequence discovery pipeline and virus clustering for metagenomic data". Nature Protocols. 12 (8): 1673–1682. doi:10.1038/nprot.2017.063. PMID 28749930. S2CID 2127494.
- ^ Tang L (July 2019). "Pole-to-pole ocean viromes". Research highlights. Nature Methods (Paper). 16 (7): 575. doi:10.1038/s41592-019-0480-1. PMID 31249411. – Springer Nature 경유 (서브스크립션 필요)
- ^ a b c d e f g h i j k l m n o Paez-Espino D, Eloe-Fadrosh EA, Pavlopoulos GA, Thomas AD, Huntemann M, Mikhailova N, et al. (August 2016). "Uncovering Earth's virome". Nature. 536 (7617): 425–430. Bibcode:2016Natur.536..425P. doi:10.1038/nature19094. PMID 27533034. S2CID 4466854.
- ^ Paez-Espino D, Chen IA, Palaniappan K, Ratner A, Chu K, Szeto E, et al. (January 2017). "IMG/VR: a database of cultured and uncultured DNA Viruses and retroviruses". Nucleic Acids Research. 45 (D1): D457–D465. doi:10.1093/nar/gkw1030. PMC 5210529. PMID 27799466.
- ^ Paez-Espino D, Roux S, Chen IA, Palaniappan K, Ratner A, Chu K, et al. (January 2019). "IMG/VR v.2.0: an integrated data management and analysis system for cultivated and environmental viral genomes". Nucleic Acids Research. 47 (D1): D678–D686. doi:10.1093/nar/gky1127. PMC 6323928. PMID 30407573.