셀 사이클
Cell cycle셀 사이클 또는 셀 분할 사이클은 셀이 두 개의 딸 셀로 분할되도록 하는 일련의 이벤트입니다.이러한 사건들은 DNA와 그 세포들의 복제, 그리고 세포 분열이라고 불리는 과정에서 세포질과 다른 성분들이 두 개의 딸 세포로 분할되는 것을 포함한다.
핵을 가진 세포(즉, 동물, 식물, 곰팡이 및 원생 세포)에서 세포 주기는 상간 및 유사분열(M) 상(분열 및 사이토키네시스 포함)의 2개의 주요 단계로 나뉜다.중간 단계 동안 세포는 성장하며 유사분열에 필요한 영양분을 축적하고 세포 DNA와 세포소기관 일부를 복제합니다.유사분열 단계에서, 복제된 염색체, 세포질, 그리고 세포질은 두 개의 새로운 딸 세포로 분리된다.세포 구성요소와 분열의 적절한 복제를 보장하기 위해, 세포가 다음 단계로 진행할 수 있는지 여부를 결정하는 주기의 각 주요 단계 뒤에 세포 주기 검사점으로 알려진 제어 메커니즘이 있습니다.
핵이 없는 세포(즉, 박테리아와 고세균)에서 세포 주기는 B, C, D 시기로 나뉜다.B 기간은 세포 분열의 끝에서 DNA 복제의 시작까지 연장됩니다.DNA 복제는 C 기간에 일어납니다.D기는 DNA 복제가 끝나고 세균세포가 두 개의 [1]딸세포로 분열되는 단계를 말한다.
세포 분열 주기는 단세포 수정란이 성숙한 유기체로 발달하는 중요한 과정일 뿐만 아니라 머리카락, 피부, 혈구, 그리고 일부 내부 장기가 재생되고 치유되는 과정이다.세포 분열 후, 각각의 딸 세포는 새로운 세포 주기의 중간 단계를 시작한다.상간의 다양한 단계는 보통 형태학적으로 구별할 수 없지만, 세포 주기의 각 상은 세포 분열의 시작을 위해 세포를 준비하는 독특한 일련의 생화학적 과정을 가지고 있다.
단계
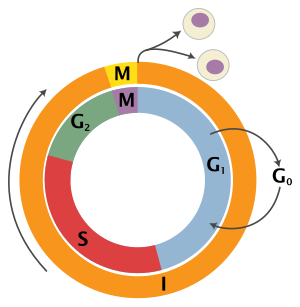
진핵 세포 주기는 4개의 상으로 구성되어1 있다: G상, S상, G상2 그리고 M상.M상은 세포핵이 분열하는 유사분열과 세포의 세포질이 분열하여 두 개의 딸세포를 형성하는 사이토키네시스라는 두 개의 밀접하게 결합된 과정으로 구성되어 있습니다.각 단계의 활성화는 이전 단계의 적절한 진행 및 완료에 따라 달라집니다.일시적으로 또는 가역적으로 분열을 멈춘 세포는 G상이라고 불리는0 정지 상태에 들어갔다고 한다.
| 주 | 단계 | 줄임말 | 묘사 |
|---|---|---|---|
| 휴식하고 있는 | 갭 0 | G0 | 셀이 사이클을 벗어나 분열을 멈춘 단계입니다. |
| 중간상 | 갭 1 | G1 | 세포 성장.G1 체크포인트는 모든 것이 DNA 합성을 위한 준비가 되었음을 보증합니다. |
| 합성 | S | DNA 복제. | |
| 갭 2 | G2 | 유사분열을 위한 성장과 준비.G2 체크포인트는 모든 것이 M(유도증) 단계로 들어가 분할할 준비가 되어 있음을 보증합니다. | |
| 세포분열 | 유사분열 | M | 세포 분열이 일어나다.Metaphase Checkpoint는 셀이 세포분열을 완료할 준비가 되었는지 확인합니다. |
세포 분열 후, 각각의 딸 세포는 새로운 주기의 중간 단계를 시작합니다.상간의 다양한 단계는 보통 형태학적으로 구별할 수 없지만, 세포 주기의 각 상은 세포 분열의 시작을 위해 세포를 준비하는 특별한 생화학적 과정을 가지고 있다.
G상0(질서)
G는0 세포가 사이클을 떠나 분열을 멈춘 정지 단계입니다.셀 사이클은 이 단계부터 시작됩니다.다세포 진핵 생물의 비증식(비분열) 세포는 일반적으로 G에서 대기01 G 상태로 들어가 장기간, 아마도 무기한으로(종종 뉴런의 경우처럼) 정지 상태를 유지할 수 있다.이것은 완전히 분화된 세포에서 매우 일반적입니다.일부 세포는 반영구적으로 G 단계에 들어가0 일부 간, 신장 및 위 세포와 같이 유사 후 세포로 간주됩니다.많은 세포들이 G로 들어가지0 않고 상피 세포와 같이 유기체의 일생 동안 계속 분열한다.
"포스트 미토틱"이라는 단어는 때때로 정지 전지와 노화 전지를 모두 가리키는 데 사용됩니다.세포 노화는 DNA 손상과 외부 스트레스에 반응하여 일어나며, 보통 G에서1 정지를 구성합니다. 세포 노화는 세포의 자손을 생존할 수 없게 만들 수 있습니다. 세포 노화는 종종 아포토시스에 의해 손상된 세포가 스스로 파괴되는 것에 대한 생화학적 대안입니다.
중간상
Interphase는 두 개의 연속된 M 단계 사이의 위상을 나타냅니다.간상(Interphase)은 새로 형성된 세포와 세포핵이 다시 분열하기 전에 일어나는 일련의 변화이다.준비단계 또는 중간증이라고도 합니다.일반적으로 세포 주기에 필요한 총 시간의 91% 이상 간상이 지속됩니다.
간기는 G, S, G의12 3단계로 진행되며, 이어서 유사분열과 사이토키네시스 주기가 이어진다.세포의 핵 DNA 함량은 S 단계 동안 복제된다.
G1 단계(첫 번째 성장 단계 또는 유사분열 후 갭 단계)
이전 M 단계의 끝에서 DNA 합성이 시작될 때까지의 중간 단계 내의 첫 번째 단계를 G(G는 갭을 나타낸다)라고1 한다.그것은 성장기라고도 불린다.이 단계에서는 M상에서는 상당히 느려진 세포의 생합성활동이 고속으로 재개된다.G의 지속1 시간은 심지어 같은 [3]종의 다른 세포들 사이에서도 매우 가변적입니다.이 단계에서, 세포는 단백질의 공급을 증가시키고, 세포 세포(미토콘드리아, 리보솜과 같은)의 수를 증가시키며, 크기가 커진다.G단계에서는1 셀에 3가지 옵션이 있습니다.
결정점을 체크 포인트(제한 포인트)라고 부릅니다.이 체크포인트는 제한점 또는 START라고 불리며 G/S 사이클린에1 의해 조절되며, G에서1 S로 전환됩니다.G1 체크 포인트를 통과하면 셀이 분할됩니다.
S단계(DNA 리플리케이션)
이어지는 S 단계는 DNA 합성이 시작되면 시작됩니다. 완료되면 모든 염색체가 복제됩니다. 즉, 각 염색체는 두 개의 자매 염색체로 구성됩니다.따라서, 이 단계에서, 세포 내 DNA의 양은 두 배로 증가했지만, 배수성과 염색체 수는 변하지 않았다.이 단계에서 RNA 전사 및 단백질 합성의 속도는 매우 낮습니다.예외적으로 히스톤 생산은 대부분 [4][5][6]S 단계에서 발생합니다.
G상2(성장)
G상은2 DNA 복제 후 발생하며 세포분열을 준비하기 위한 단백질 합성과 빠른 세포 성장 기간이다.이 단계 동안 마이크로튜브가 스핀들을 형성하기 위해 재구성되기 시작합니다(프리프로파즈).유사분열 단계로 진행하기 전에, 세포는 염색체 내의 DNA 손상 여부를 G 체크2 포인트에서 확인해야 합니다.G2 체크포인트는 주로 종양 단백질 p53에 의해 조절된다.만약 DNA가 손상된다면, p53은 DNA를 복구하거나 세포의 세포자멸을 촉발시킬 것이다.만약 p53이 기능하지 않거나 돌연변이를 일으키면, 손상된 DNA를 가진 세포는 세포주기를 통해 지속되어 암의 발생을 초래할 수 있다.
유사분열상(염색체분리)
비교적 짧은 M상은 핵분열(핵분열)로 구성된다.이것은 세포주기의 비교적 짧은 기간이다.M 단계는 복잡하고 고도로 규제됩니다.이벤트의 순서는 하나의 활동의 완료와 다음 활동의 시작에 해당하는 단계로 나뉜다.이러한 단계는 순차적으로 다음과 같이 알려져 있습니다.
유사분열은 진핵세포가 세포핵의 염색체를 두 개의 [7]핵에서 동일한 두 세트로 분리하는 과정이다.유사분열 과정 동안 염색체 쌍은 응축되어 자매 염색체를 세포의 [8]반대편으로 끌어당기는 미세관에 부착된다.
유사분열은 오직 진핵세포에서만 발생하지만, 다른 종에서 다른 방식으로 발생한다.예를 들어, 동물 세포는 염색체가 분리되기 전에 핵 외피가 분해되는 "개방" 유사분열을 겪는 반면, 아스페르길루스 니둘란스와 사카로미세스 세레비시아이 같은 균류는 염색체가 온전한 세포핵 [9]내에서 분열되는 "폐쇄" 유사분열을 겪습니다.
사이토카인시스상(모든 세포성분 분리)
유사분열은 즉시 세포질, 세포질, 세포소기관, 세포막을 이러한 세포성분들의 거의 동일한 부분을 포함하는 두 개의 세포로 나누는 세포로 이어지는 세포분열로 이어집니다.유사분열과 사이토키네시스는 함께 모세포를 유전적으로 동일한 두 개의 딸세포로 분할하는 것을 정의합니다.이는 세포주기의 약 10%를 차지한다.
사이토키네시스는 보통 유사분열과 함께 발생하기 때문에, "유도증"은 종종 "M상"과 상호 교환적으로 사용된다.그러나 체세포분열과 사이토키네시스가 따로 일어나 여러 개의 핵을 가진 단세포가 내생식이라고 불리는 과정에서 형성되는 세포들이 많다.이것은 곰팡이와 슬라임 곰팡이 중에서 가장 두드러지게 발생하지만, 다양한 그룹에서 발견된다.동물에서도 사이토키네시스 및 유사분열은 독립적으로 발생할 수 있으며, 예를 들어 초파리 배아 [10]발달의 특정 단계에서 발생할 수 있다.유사분열 오류는 아포토시스를 통해 세포사멸을 초래하거나 암을 유발할 수 있는 돌연변이를 일으킬 수 있다.
진핵세포주기조절

세포 주기의 조절은 제어되지 않은 세포 분열의 방지뿐만 아니라 유전적 손상의 검출과 복구를 포함하여 세포의 생존에 중요한 과정을 포함한다.세포 주기를 제어하는 분자적 사건들은 순서와 방향이다; 즉, 각 과정은 순차적인 방식으로 발생하며 주기를 "역행"하는 것은 불가능하다.
사이클린과 CDK의 역할
 노벨상 수상자 폴 간호사 |  노벨상 수상자 팀 헌트 |
조절 분자의 두 가지 주요 등급인 사이클린과 사이클린 의존성 키나아제(CDK)가 세포 주기 [11]동안 세포의 진행을 결정합니다.릴랜드 H. 하트웰, R. 티모시 헌트, 폴 M. 간호사는 이러한 중심 [12]분자를 발견한 공로로 2001년 노벨 생리의학상을 수상했다.사이클린과 CDK를 코드하는 많은 유전자들은 모든 진핵생물들 사이에서 보존되지만, 일반적으로, 더 복잡한 유기체들은 더 많은 개별적인 요소들을 포함하는 더 정교한 세포 주기 제어 시스템을 가지고 있다.많은 관련 유전자는 효모, 특히 사카로미세스 세레비시아에 [13]대한 연구를 통해 처음 확인되었으며, 효모 더브의 유전자 명명법 cdc ("세포 분열 주기"를 위한) 뒤에 식별 번호 cdc25 또는 cdc20이 이어졌다.
사이클린은 조절 서브유닛을 형성하고 CDK는 활성화된 헤테로다이머의 촉매 서브유닛을 형성합니다. 사이클린은 촉매활성이 없으며 CDK는 파트너 사이클린이 없을 때 비활성화됩니다.결합된 사이클린에 의해 활성화될 때, CDKs는 표적 단백질을 활성화하거나 비활성화하는 인산화라고 불리는 공통 생화학적 반응을 수행하여 세포 주기의 다음 단계로의 조정된 진입을 조정합니다.다른 사이클린-CDK 조합에 따라 타깃 다운스트림 단백질이 결정됩니다.CDK는 세포에서 구성적으로 발현되는 반면, 사이클린은 다양한 분자 [14]신호에 반응하여 세포 주기의 특정 단계에서 합성된다.
사이클린-CDK 상호작용의 일반적인 메커니즘
친유도체 세포외 신호를 수신하면 G 사이클린-CDK 복합체가1 활성화되어 S상을 위한 세포를 준비함으로써 S 사이클린의 발현과 DNA 복제에 필요한 효소의 발현을 촉진한다.G1 사이클린-CDK 복합체는 또한 S상 억제제로서 기능하는 분자를 유비퀴티네이션 대상으로 하여 분자의 분해를 촉진한다.일단 단백질이 유비쿼터스화되면, 그것은 프로테아솜에 의한 단백질 분해의 표적이 된다.그러나 단세포 수준에서 E2F 전사 역학에 대한 최근 연구 결과는 G1 사이클린-CDK 활동, 특히 사이클린 D-CDK4/6의 역할은 세포 주기 [15]진입의 확약보다는 타이밍을 조정하는 것이라고 주장한다.
활성 S 사이클린-CDK 복합체는 DNA 복제 기원에서 G 단계 동안1 조립된 사전 복제 복합체를 구성하는 단백질을 인산화한다.인산화는 이미 조립된 각 사전 복제 복합체를 활성화하는 것과 새로운 복합체가 형성되는 것을 방지하는 두 가지 목적을 제공한다.이것은 세포 게놈의 모든 부분이 한 번만 복제되도록 한다.중요한 유전자의 전부 또는 일부가 빠진 딸세포는 죽게 되기 때문에 복제의 격차를 막는 이유는 매우 명확하다.하지만, 유전자 복제 수 효과와 관련된 이유로, 특정 유전자의 여분의 복제물의 소유는 또한 딸 세포에 해롭다.
S상 및2 G상 동안 합성되지만 비활성화되는 유사분열 사이클린-CDK 복합체는 염색체 응축 및 유사분열 방추 조립에 관여하는 하류 단백질을 자극함으로써 유사분열 개시를 촉진한다.이 과정에서 활성화되는 중요한 복합체는 염색체 키네토코어와 관련된 구조단백질의 분해를 촉진하는 아나파기촉진복합체(APC)로 알려진 유비퀴틴 연결효소이다.APC는 또한 유사분열 사이클린을 분해 대상으로 하여 텔로파기와 사이토키네시스가 [16]진행될 수 있도록 보장합니다.
사이클린-CDK 복합체의 특이적 작용
사이클린 D는 세포외 신호(예: 성장인자)에 반응하여 세포주기에 들어가는 세포에서 생성되는 첫 번째 사이클린이다.사이클린 D 수치는 증식하지 않는 휴면 세포에서 낮게 유지된다.또한 CDK4/6은 INK4 패밀리 멤버(예를 들어 p16)에 의해 결합되어 키나아제 활성을 제한하기 때문에 CDK4/6 및 CDK2도 비활성화된다.한편 CDK2 복합체는 p21, p27 [17]등의 CIP/KIP 단백질에 의해 억제되며, 세포주기에 들어갈 때가 되면 승모원 자극에 의해 유발되는 사이클린 D의 수치가 증가한다.이 트리거에 반응하여 사이클린 D는 기존 CDK4/6에 결합하여 활성 사이클린 D-CDK4/6 복합체를 형성합니다.Cyclin D-CDK4/6 복합체는 망막아종 감수성 단백질(Rb)을 pRb로 모노인산화한다.비인산화 Rb종양억제제는 세포주기 출구를 유도하고 G0 정지(노화)[18]를 유지하는 기능을 한다.
지난 수십 년 동안 pRB 단백질이 사이클린 D-Cdk4/6 매개 인산화로 불활성화되는 모델이 널리 받아들여져 왔다.Rb는 14개 이상의 잠재적 인산화 부위가 있다.Cyclin D-Cdk 4/6은 Rb를 점진적으로 과인산화시켜 pRB-E2F 복합체의 해리를 유발하고, 그 결과 G1/[19]S 세포주기 유전자 발현과 S상으로의 진행을 유도한다.
그러나 최근 연구의 과학적 관찰에 따르면 Rb는 세 가지 유형의 등산화물로 존재한다. (1) G0 상태에서 비인산화 Rb, (2) 단인산화 Rb, "하이포-인산화" 또는 "부분적" 인산화 Rb라고도 함, (3) 초기 G1 상태에서 비인산화 Rb, 그리고 (3) 초인산화 Rb1에서 비인산화 Rb이다.초기 G1 세포에서 모노인산화 Rb는 14개의 서로 다른 동질체로 존재하며, 각각은 뚜렷한 E2F 결합 [22]친화성을 가진다.Rb는 수백 개의 다른[23] 단백질과 관련이 있는 것으로 밝혀졌으며, 서로 다른 단인산화 Rb 동질체가 서로 다른 단백질 파트너를 가지고 있다는 생각은 매우 [24]매력적이었다.최근 보고서는 단일인산이 Rb와 다른 단백질과의 관련성을 제어하고 기능적으로 별개의 형태의 [25]Rb를 발생시킨다는 것을 확인했다.서로 다른 모든 단인산화 Rb 아이소폼은 E2F 전사 프로그램을 억제하고 G1상 세포를 억제할 수 있다.중요한 것은 서로 다른 단일 인산화 형태의 RB가 E2F [25]조절 이상으로 확장되는 별개의 전사 출력을 가지고 있다는 점이다.
일반적으로 pRb와 E2F의 결합은 E형 사이클린을 포함한 특정 G1/S 및 S 전이 유전자의 E2F 표적 유전자 발현을 억제한다.RB의 부분적 인산화로 E2F 표적 유전자 발현에 대한 Rb 매개 억제가 해제되어 사이클린 E의 발현을 시작한다.세포를 사이클린 E 활성화로 전환하는 분자 메커니즘은 현재 알려져 있지 않지만, 사이클린 E 수치가 상승함에 따라 활성 사이클린 E-CDK2 복합체가 형성되어 Rb가 초인산화로 [22]비활성화된다.과인산화 Rb는 E2F에서 완전히 분리되므로 세포를 S상으로 진행시키기 위해서는 광범위한 E2F 표적 유전자의 발현이 필요하다[1].최근에는 사이클린 D-Cdk4/6이 사이클린 E, A 및 [26]B가 아닌 사이클린 D에만 구별 가능한 C 말단 알파-나선 영역 Rb에 결합하는 것이 확인되었다.Rb 인산화 구조 분석에 기초한 이러한 관찰은 Rb가 여러 사이클린-Cdk 복합체를 통해 다른 수준에서 인산화된다는 것을 뒷받침한다.이것은 또한 한 가지 유형의 Rb 초인산화 메커니즘을 통해 모든 단인산화 Rb 아이소폼의 동시 스위치 형태의 불활성화 모델을 실현 가능하게 한다.또한 사이클린 D-Cdk 4/6 특이 Rb C 말단 나선의 돌연변이 분석에 따르면 Rb에 결합하는 사이클린 D-Cdk 4/6의 교란은 Rb 인산화 방지,[26] G1의 세포 정지 및 종양 억제제에서의 Rb의 기능을 강화한다.이 사이클린-Cdk 구동 세포 주기 전이 메커니즘은 세포 증식을 허용하는 세포 주기에 전념하는 세포를 지배합니다.암세포의 성장은 종종 사이클린 D-Cdk 4/6 활성의 규제 완화를 동반한다.
과인산화 Rb는 E2F/DP1/Rb 복합체(E2F 반응 유전자에 결합되어 효과적으로 전사를 차단)에서 분리되어 E2F를 활성화한다.E2F의 활성화는 사이클린 E, 사이클린 A, DNA 중합효소, 티미딘 키나아제 등과 같은 다양한 유전자의 전사를 일으킨다.이렇게 생성된 사이클린 E는 CDK2에 결합하여 사이클린 E-CDK2 복합체를 형성하고, 이는 세포를 G에서1 S 상(G1/S, G/M [27]전이를2 개시)으로 밀어낸다.사이클린 B-cdk1 복합활성화는 핵외피의 파괴와 전기의 개시를 야기하고, 그 불활성화는 세포의 유사분열을 [14]종료시킨다.공학적 형광 리포터 셀을 사용하여 단세포 수준에서 E2F 전사 역학에 대한 정량적 연구는 표준 교과서 모델에 도전하는 세포 주기 진입의 제어 논리를 이해하기 위한 정량적 프레임워크를 제공했다.Myc와 같이 E2F 축적의 진폭을 조절하는 유전자는 세포주기와 S상 진입에서의 관여를 결정한다.G1 사이클린-CDK 활동은 세포 주기 진입의 동인이 아니다.대신, 주로 E2F 증가 타이밍을 조정하여 세포 주기 [15]진행 속도를 조절합니다.
억제제
내인성

cip/kip(CDK 상호작용 단백질/Kinase 억제 단백질) 패밀리와 INK4a/ARF(인산화효소 4/대체 판독 프레임의 억제제) 패밀리의 2가지 유전자 패밀리는 세포주기의 진행을 방해한다.이 유전자들은 종양 형성을 막는 데 중요한 역할을 하기 때문에 종양 억제제로 알려져 있다.
cip/kip 패밀리는 유전자 p21, p27 및 p57을 포함한다.이들은 사이클린-CDK 복합체에 결합 및 비활성화를 통해 G상1 세포주기를 정지시킨다.p21은 p53에 의해 활성화된다(즉, 방사선으로 인한 DNA 손상에 의해 트리거된다).p27은 성장억제제인 TGFβ(Transforming Growth Factor β)에 의해 활성화된다.
INK4a/ARF 패밀리는 CDK4에 결합하여 G상에서의 세포1 사이클을 정지시키는INK4a p16과 p53 열화를 방지하는ARF p14를 포함한다.
합성
또한 Cdc25의 합성억제제는 세포주기의 정지에도 유용할 수 있으며, 따라서 항종양제 및 [28]항암제로도 유용할 수 있다.
많은 인간 암은 초활성 Cdk 4/6 [29]활성을 가지고 있다.사이클린 D-Cdk 4/6 기능이 관찰된 경우, Cdk 4/6을 억제하면 악성 종양이 증식하는 것을 방지할 수 있다.따라서 과학자들은 Cdk4/6이 항종양 효과의 치료 대상으로 특징지어짐에 따라 합성 Cdk4/6 억제제를 발명하려고 시도했다.Palbociclib, ribociclib, abemaciclib 등 3가지 Cdk4/6 억제제는 현재 진행단계 또는 전이성 호르몬 수용체 양성(HR-양성, HR+), HER2 음성(HER2-) 유방암 [30][31]치료를 위한 임상 사용을 위해 FDA 승인을 받았다.예를 들어 palbociclib은 경구 활성 CDK4/6 억제제로서 ER 양성/HER2 음성 진행 유방암에 대한 개선된 결과를 보여준다.주요 부작용은 호중구감소증이며,[32] 이는 선량 감소로 관리할 수 있다.
Cdk4/6 표적 치료는 Rb가 발현된 암 유형만 치료할 것이다.Rb 손실이 있는 암세포는 Cdk4/6 억제제에 대한 1차 내성을 가진다.
문자 변환 규제 네트워크
현재의 증거에 따르면 반자율 전사 네트워크는 CDK 사이클린 기계와 연계하여 세포 사이클을 조절합니다.Saccharomyces cerevisae의 여러 유전자 발현 연구는 세포 [13][33][34]주기의 과정에 걸쳐 발현을 변화시키는 800–1200개의 유전자를 확인했다.그것들은 세포 주기의 특정 지점에서 높은 수준에서 전사되고, 나머지 주기의 내내 낮은 수준에서 유지됩니다.확인된 유전자 세트는 그것들을 식별하기 위해 사용되는 계산 방법과 기준 때문에 연구마다 다르지만, 각각의 연구는 효모 유전자의 많은 부분이 일시적으로 [35]조절된다는 것을 보여준다.
많은 주기적으로 발현되는 유전자들은 주기적으로 발현되는 전사 인자에 의해 구동된다.단일 유전자 녹아웃의 한 화면에서는 세포 주기 진행 [36]결함을 나타내는 48개의 전사 인자(모든 비필수 전사 인자의 약 20%)가 확인되었습니다.높은 throughput 기술을 사용한 게놈 전체의 연구는 효모 유전자의 프로모터에 결합하는 전사 인자를 확인하였고, 이러한 발견과 시간적 발현 패턴의 상관관계를 통해 위상 특이적 유전자 [33][37]발현을 촉진하는 전사 인자를 식별할 수 있었다.이러한 전사 인자의 발현 프로파일은 이전 단계에서 최고조에 달한 전사 인자에 의해 구동되며, 계산 모델은 이러한 전사 인자의 CDK 자율 네트워크가 유전자 [34][38]발현에서 안정된 상태 진동을 일으키기에 충분하다는 것을 보여주었다.)
실험 증거는 또한 유전자 발현이 CDK 기계와는 독립적으로 야생형 세포를 분열시키는 데 나타나는 기간에 따라 진동을 일으킬 수 있다는 것을 암시한다.올란도 등마이크로어레이를 사용하여 야생형 세포와 모든 S상 및 유사분열성 사이클린(clb1,2,3,4,5,6)이 결여된 세포 모두에서 주기적인 것으로 확인된 1,271개 유전자 세트의 발현을 측정했다.검사된 1,271개의 유전자 중 882개는 G상과 S상의 경계에서1 사이클린 결핍 세포가 정지하고 있음에도 불구하고 야생형 세포와 동시에 사이클린 결핍 세포에서 계속 발현되었다.그러나 유전자 중 833개는 야생형과 돌연변이 세포 사이의 변화된 행동을 측정했으며, 이는 이러한 유전자들이 CDK-사이클린 기계에 의해 직간접적으로 조절될 가능성이 높다는 것을 보여준다.돌연변이 세포에서 제시간에 계속 발현된 일부 유전자는 돌연변이 세포와 야생형 세포에서도 다른 수준으로 발현되었다.이러한 연구 결과는 전사 네트워크가 CDK 사이클린 발진기와는 독립적으로 발진할 수 있지만 셀 사이클 [34]이벤트의 적절한 타이밍을 확보하기 위해 두 네트워크가 모두 필요한 방식으로 결합됨을 시사합니다.다른 연구는 Cdk1에 의한 세포 주기 전사 인자의 번역 후 변형인 인산화(phosphalation)가 표적 [36][39][40]유전자의 타이밍을 엄격하게 제어하기 위해 전사 인자의 국재화 또는 활성을 변경할 수 있음을 나타낸다.
반면 동요하는 표기는 효모 세포 주기의 추이에 핵심 역할을 한다, CDK-cyclin 기계 독립적으로 초기 배아 세포 주기에서 운영되고 있다.그 midblastula 전환 전에, 접합자 전사와 B-type cyclins와 같은 모든 필요한 단백질, 어머니와 같이 적재 mRNA.[41]에서 변환 된다 일어나지 않는다.
DNA복제및DNA복제원활동
Saccharomyces cerevisiae에 대하는 문화의 세포 주기 진행을 지연시키지 않고 DNA복제 개시를 막아 주는 조건에서 분석이 원산지 허가하는 그들의 3끝 근처에 기원이 하류로 기원은 상류의 유전자 발현을 조절할 수 있는 유전자 발현을 감소시켜 보여 주었다.[42]이 DNA복제 개시점 활동과 mRNAexpression,[43][44][45]사이에 세계적인 인과 조정의 수학적 모델링에서 DNA마이크로 어레이의 데이터를 수학적 모델링 까지 정확하게 규제의 이전의 알려지지 않은 생물학적 모드 예측할 수 있는지를 보여 줍니다. 이전의 가설을 입증하는 것.
체크 포인트
세포 주기 체크 포인트는 세포에 의하고 조절하는지 세포 주기의 진행 상황을 지켜보기 위해 사용된다.[46]경비 초소가 특정 지점에 필요한 단계 과정과 DNA손상의 수리 검증 허용하는 세포 주기 진행을 예방한다.때까지 검문소 요구 조건을 충족하는 세포는 다음 단계로의 진행될 수 없다.체크 포인트가 대체로 모니터 규제 단백질 네트워크의 세포 세포 주기의 단계를 통해 그 진행을 지시하는 것들로 이루어져 있다.
그것은 평범한 인간의 세포에서 한가닥 사슬 DNA의 손상을 약 1%세포 주기당 세포에 약 50내인성 DNAdouble-strand 휴식 시간을 변환하는 것으로 추정됩니다.[47]비록 이러한 double-strand이 보통 고충실도로 고친다 하더라도 이들의 수리의 오류가 크게 암이 인간의 속도에 기여하는 것으로 간주된다.[47]
몇가지 체크 포인트가 좋거나 불완전한 손상된 DNA 딸세포에 전달되지 않습니다를 보장하기 위함이다.3주요 체크 포인트:G1/S 검문소, G2/M 검문소와 중기(유사 분열)검문소 존재한다.이에 있는 세포들은 성숙 검사를 받는다고 또 다른 검문소는 이동 검문소.만약 세포는 아직 준비되지 않음으로써 이 검문소 통과하지 못하면, 그들은 가르는 폐기된다.
G1/S 전환은 셀 사이클의 속도 제한 단계이며 [14]제한 지점이라고도 합니다.이것은 세포가 DNA를 완전히 복제하기에 충분한 원료를 가지고 있는지 확인하는 곳입니다.건강하지 않거나 영양이 부족한 세포는 이 검문소에 갇힐 것이다.
G2/M 체크포인트는 세포가 두 개의 딸세포를 위한 충분한 세포질과 인지질을 가지고 있는지 확인하는 곳입니다.그러나 더 중요한 것은 지금이 복제하기에 적절한 시기인지 확인하는 것입니다.많은 세포들이 동시에 복제되어야 하는 상황들이 있다. (예를 들어, 성장하는 배아는 그것이 중간 소포 전환에 도달할 때까지 대칭적인 세포 분포를 가져야 한다.)이는 G/M 체크포인트를2 제어함으로써 이루어집니다.
중기의 체크포인트는 세포가 중기의 상태가 되면 유사분열을 겪는다는 점에서 상당히 작은 체크포인트이다.하지만 그렇다고 그것이 중요하지 않다는 것은 아니다.이 체크포인트에서, 세포는 방추체가 형성되었는지,[48] 그리고 무지개가 시작되기 전에 모든 염색체가 방추 적도에 정렬되어 있는지 확인합니다.
이들은 3개의 "주요" 체크포인트이지만 복제하기 위해 모든 셀이 이러한 체크포인트를 통과할 필요는 없습니다.많은 종류의 암은 세포가 다양한 체크포인트를 빠르게 통과하거나 심지어 그것들을 모두 건너뛰게 하는 돌연변이에 의해 발생한다.S에서 M으로 거의 연속적으로 진행됩니다.이 세포들이 체크포인트를 잃었기 때문에, 발생했을지도 모르는 모든 DNA 돌연변이는 무시되고 딸 세포로 전달됩니다.이것이 암세포가 돌연변이를 기하급수적으로 축적하는 경향을 보이는 이유 중 하나이다.암세포를 제외하고, 많은 완전히 분화된 세포들은 더 이상 복제되지 않기 때문에 세포주기를 떠나 죽을 때까지 G에0 머무른다.따라서 셀룰러 체크포인트의 필요성이 없어집니다.DNA 손상에 대한 세포 주기 반응의 대안 모델인 복제 후 체크포인트도 제안되었다.
검문소 조절은 유기체의 발달에 중요한 역할을 한다.성 생식에서는 난자가 수정되면 정자가 난자에 결합하면 수정되었음을 알리는 신호 인자를 방출한다.무엇보다도, 이것은 현재 수정된 난모세포가 이전의 휴면상태인0 G에서 세포주기로 돌아가 유사분열 복제로 돌아가도록 유도한다.
p53은 G/S 체크포인트와2 G/M 체크포인트 모두에서1 제어 메커니즘을 트리거하는데 중요한 역할을 합니다.p53 외에도 검문소 규제기관은 암의 성장과 증식에 대한 그들의 역할을 위해 많은 연구가 이루어지고 있다.
세포주기의 형광 이미징

미야와키 아쓰시와 동료의 선구적인 연구로, 형광 유비퀴티네이션 베이스의 세포 사이클 인디케이터(FUCCI)를 개발해, 세포 사이클의 형광 이미징을 가능하게 했다.당초 녹색 형광단백질 mAG는 hGem(1/110)에, 주황색 형광단백질2(mKO)은 hCdt1(30/120)에 각각 융합됐다.주의: 이러한 융합은 핵 국재 신호와 열화를 위한 유비쿼티네이션 부위가 포함된 단편이지만 기능성 단백질은 아니다.녹색 형광 단백질은 S, G2, M 단계에서 생성되고 G 또는1 G 단계에서0 분해되며, 주황색 형광 단백질은 G 또는01 G 단계에서 생성되어 S,[49] G2 또는 M 단계에서 파괴된다.시아노박테리아유래형광단백질(smURFP)과 박테리오피토크롬유래형광단백질(이 [50]링크에서 발견된 영화)을 사용하여 원적외선 근적외선 FUCCI를 개발했다.
종양 형성에 대한 역할
세포 주기 성분의 규제 해제는 종양의 [51]형성을 초래할 수 있다.이상과 같이 세포주기억제제, RB, p53 등의 일부 유전자가 돌연변이를 일으키면 세포가 제어불능으로 증식하여 종양을 형성할 수 있다.종양세포의 세포주기 지속시간은 정상세포주기보다 같거나 길지만 종양에서 활성세포분열(G상0 정지세포에 비해) 중인 세포의 비율은 [52]정상조직보다 훨씬 높다.따라서 아포토시스나 노화에 의해 죽는 세포의 수는 그대로 유지되므로 세포 수는 순증한다.
세포주기가 활발하게 진행되는 세포는 세포분열 시 DNA가 상대적으로 노출되어 약물이나 방사선에 의한 손상을 받기 쉽기 때문에 암치료의 표적이 된다.이 사실은 암 치료에 이용된다; 디블링이라고 알려진 과정에 의해, 상당한 양의 종양 덩어리가 제거되고, 이것은 (영양소, 산소, 성장 인자의 가용성 증가 등으로 인해) 남은 종양 세포의 상당수를 G 단계에서0 G 단계로1 밀어낸다.디블킹 절차에 따른 방사선 또는 화학요법은 세포주기에 [14]새로 진입한 세포를 죽인다.
배양에서 가장 빠르게 순환하는 포유류의 세포인 장상피 속의 세포는 9시간에서 10시간 정도의 짧은 주기를 가지고 있다.쉬고 있는 쥐의 피부에 있는 줄기세포는 200시간 이상의 주기를 가질 수 있다.이 차이의 대부분은 사이클의 가장 가변적인 위상인 G의 길이가1 다양하기 때문입니다.M과 S는 크게 다르지 않다.
일반적으로 셀은 후기 M상2 및 G상에서는 방사선에 가장 민감하고 후기 S상에서는 가장 내성이 높습니다.
세포주기가 길고 G상이 상당히 긴1 세포는 G 늦게1 두 번째 저항 피크가 있다.
저항과 감도의 패턴은 세포 내 술프하이드릴 화합물의 수준과 관련이 있습니다.술프하이드릴은 방사선 손상으로부터 세포를 보호하는 천연 물질로 S가 가장 높고 근위 분열이 가장 낮은 경향이 있다.
상동재조합(HR)은 DNA 이중사슬 절단을 수리하는 정확한 과정이다.HR은 G1상에서는 거의 존재하지 않고 S상에서는 가장 활성화되며 G/[53]M상에서는2 감소합니다.이중 스트랜드 절단을 수리하기 위한 보다 정확도가 낮고 변이원성이 높은 프로세스인 비호몰로지 엔드 결합은 셀 사이클 전체에서 활성화됩니다.
「 」를 참조해 주세요.
레퍼런스
- ^ Wang JD, Levin PA (November 2009). "Metabolism, cell growth and the bacterial cell cycle". Nature Reviews. Microbiology. 7 (11): 822–7. doi:10.1038/nrmicro2202. PMC 2887316. PMID 19806155.
- ^ Cooper GM (2000). "Chapter 14: The Eukaryotic Cell Cycle". The cell: a molecular approach (2nd ed.). Washington, D.C: ASM Press. ISBN 978-0-87893-106-4.
- ^ Smith JA, Martin L (April 1973). "Do cells cycle?". Proceedings of the National Academy of Sciences of the United States of America. 70 (4): 1263–7. Bibcode:1973PNAS...70.1263S. doi:10.1073/pnas.70.4.1263. PMC 433472. PMID 4515625.
- ^ Wu RS, Bonner WM (December 1981). "Separation of basal histone synthesis from S-phase histone synthesis in dividing cells". Cell. 27 (2 Pt 1): 321–30. doi:10.1016/0092-8674(81)90415-3. PMID 7199388. S2CID 12215040.
- ^ Nelson DM, Ye X, Hall C, Santos H, Ma T, Kao GD, et al. (November 2002). "Coupling of DNA synthesis and histone synthesis in S phase independent of cyclin/cdk2 activity". Molecular and Cellular Biology. 22 (21): 7459–72. doi:10.1128/MCB.22.21.7459-7472.2002. PMC 135676. PMID 12370293.
- ^ Cameron IL, Greulich RC (July 1963). "Evidence for an essentially constant duration of DNA synthesis in renewing epithelia of the adult mouse". The Journal of Cell Biology. 18: 31–40. doi:10.1083/jcb.18.1.31. PMC 2106275. PMID 14018040.
- ^ Rubenstein I, Wick SM (2008). "Cell". World Book Online Reference Center. Archived from the original on 30 May 2011. Retrieved 10 July 2009.
- ^ Maton A, Lahart D, Hopkins J, Warner MQ, Johnson S, Wright JD (1997). Cells: Building Blocks of Life. New Jersey: Prentice Hall. pp. 70–4. ISBN 978-0-13-423476-2.
- ^ De Souza CP, Osmani SA (September 2007). "Mitosis, not just open or closed". Eukaryotic Cell. 6 (9): 1521–7. doi:10.1128/EC.00178-07. PMC 2043359. PMID 17660363.
- ^ Lilly MA, Duronio RJ (April 2005). "New insights into cell cycle control from the Drosophila endocycle". Oncogene. 24 (17): 2765–75. doi:10.1038/sj.onc.1208610. PMID 15838513.
- ^ Nigg EA (June 1995). "Cyclin-dependent protein kinases: key regulators of the eukaryotic cell cycle". BioEssays. 17 (6): 471–80. doi:10.1002/bies.950170603. PMID 7575488. S2CID 44307473.
- ^ "The Nobel Prize in Physiology or Medicine 2001 - Press release". Nobelprize.org.
- ^ a b Spellman PT, Sherlock G, Zhang MQ, Iyer VR, Anders K, Eisen MB, et al. (December 1998). "Comprehensive identification of cell cycle-regulated genes of the yeast Saccharomyces cerevisiae by microarray hybridization". Molecular Biology of the Cell. 9 (12): 3273–97. doi:10.1091/mbc.9.12.3273. PMC 25624. PMID 9843569.
- ^ a b c d Robbins SL, Cotran RS (2004). Kumar V, Abbas AK, Fausto N (eds.). Pathological Basis of Disease. Elsevier. ISBN 978-81-8147-528-2.
- ^ a b Dong P, Maddali MV, Srimani JK, Thélot F, Nevins JR, Mathey-Prevot B, You L (September 2014). "Division of labour between Myc and G1 cyclins in cell cycle commitment and pace control". Nature Communications. 5: 4750. Bibcode:2014NatCo...5.4750D. doi:10.1038/ncomms5750. PMC 4164785. PMID 25175461.
- ^ Mahmoudi M, Azadmanesh K, Shokrgozar MA, Journeay WS, Laurent S (May 2011). "Effect of nanoparticles on the cell life cycle". Chemical Reviews. 111 (5): 3407–32. doi:10.1021/cr1003166. PMID 21401073.
- ^ Goel S, DeCristo MJ, McAllister SS, Zhao JJ (November 2018). "CDK4/6 Inhibition in Cancer: Beyond Cell Cycle Arrest". Trends in Cell Biology. 28 (11): 911–925. doi:10.1016/j.tcb.2018.07.002. PMC 6689321. PMID 30061045.
- ^ Burkhart DL, Sage J (September 2008). "Cellular mechanisms of tumour suppression by the retinoblastoma gene". Nature Reviews. Cancer. 8 (9): 671–82. doi:10.1038/nrc2399. PMC 6996492. PMID 18650841.
- ^ Morgan DO (2007). The cell cycle : principles of control. London: New Science Press. ISBN 978-0-19-920610-0. OCLC 70173205.
- ^ Paternot S, Bockstaele L, Bisteau X, Kooken H, Coulonval K, Roger PP (February 2010). "Rb inactivation in cell cycle and cancer: the puzzle of highly regulated activating phosphorylation of CDK4 versus constitutively active CDK-activating kinase". Cell Cycle. 9 (4): 689–99. doi:10.4161/cc.9.4.10611. PMID 20107323.
- ^ Henley SA, Dick FA (March 2012). "The retinoblastoma family of proteins and their regulatory functions in the mammalian cell division cycle". Cell Division. 7 (1): 10. doi:10.1186/1747-1028-7-10. PMC 3325851. PMID 22417103.
- ^ a b c Narasimha AM, Kaulich M, Shapiro GS, Choi YJ, Sicinski P, Dowdy SF (June 2014). "Cyclin D activates the Rb tumor suppressor by mono-phosphorylation". eLife. 3: e02872. doi:10.7554/eLife.02872. PMC 4076869. PMID 24876129.
- ^ Morris EJ, Dyson NJ (1 January 2001). Retinoblastoma protein partners. Advances in Cancer Research. Vol. 82. Academic Press. pp. 1–54. doi:10.1016/s0065-230x(01)82001-7. ISBN 9780120066827. PMID 11447760.
- ^ Dyson NJ (July 2016). "RB1: a prototype tumor suppressor and an enigma". Genes & Development. 30 (13): 1492–502. doi:10.1101/gad.282145.116. PMC 4949322. PMID 27401552.
- ^ a b Sanidas I, Morris R, Fella KA, Rumde PH, Boukhali M, Tai EC, et al. (March 2019). "A Code of Mono-phosphorylation Modulates the Function of RB". Molecular Cell. 73 (5): 985–1000.e6. doi:10.1016/j.molcel.2019.01.004. PMC 6424368. PMID 30711375.
- ^ a b Topacio BR, Zatulovskiy E, Cristea S, Xie S, Tambo CS, Rubin SM, et al. (May 2019). "Cyclin D-Cdk4,6 Drives Cell-Cycle Progression via the Retinoblastoma Protein's C-Terminal Helix". Molecular Cell. 74 (4): 758–770.e4. doi:10.1016/j.molcel.2019.03.020. PMC 6800134. PMID 30982746.
- ^ Norbury C (1995). "Cdk2 protein kinase (vertebrates)". In Hardie DG, Hanks S (eds.). Protein kinase factsBook. Boston: Academic Press. pp. 184. ISBN 978-0-12-324719-3.
- ^ "Presentation on CDC25 PHOSPHATASES: A Potential Target for Novel Anticancer Agents". Archived from the original on 3 March 2016. Retrieved 11 March 2010.
- ^ Sherr CJ, Beach D, Shapiro GI (April 2016). "Targeting CDK4 and CDK6: From Discovery to Therapy". Cancer Discovery. 6 (4): 353–67. doi:10.1158/2159-8290.cd-15-0894. PMC 4821753. PMID 26658964.
- ^ O'Leary B, Finn RS, Turner NC (July 2016). "Treating cancer with selective CDK4/6 inhibitors". Nature Reviews. Clinical Oncology. 13 (7): 417–30. doi:10.1038/nrclinonc.2016.26. PMID 27030077. S2CID 23646632.
- ^ Bilgin B, Sendur MA, Şener Dede D, Akıncı MB, Yalçın B (September 2017). "A current and comprehensive review of cyclin-dependent kinase inhibitors for the treatment of metastatic breast cancer". Current Medical Research and Opinion. 33 (9): 1559–1569. doi:10.1080/03007995.2017.1348344. PMID 28657360. S2CID 205542255.
- ^ Schmidt M, Sebastian M (August 2018). "Palbociclib-The First of a New Class of Cell Cycle Inhibitors". Recent Results in Cancer Research. Fortschritte der Krebsforschung. Progres dans les Recherches Sur le Cancer. Recent Results in Cancer Research. 211: 153–175. doi:10.1007/978-3-319-91442-8_11. ISBN 978-3-319-91441-1. PMID 30069766.
- ^ a b Pramila T, Wu W, Miles S, Noble WS, Breeden LL (August 2006). "The Forkhead transcription factor Hcm1 regulates chromosome segregation genes and fills the S-phase gap in the transcriptional circuitry of the cell cycle". Genes & Development. 20 (16): 2266–78. doi:10.1101/gad.1450606. PMC 1553209. PMID 16912276.
- ^ a b c Orlando DA, Lin CY, Bernard A, Wang JY, Socolar JE, Iversen ES, et al. (June 2008). "Global control of cell-cycle transcription by coupled CDK and network oscillators". Nature. 453 (7197): 944–7. Bibcode:2008Natur.453..944O. doi:10.1038/nature06955. PMC 2736871. PMID 18463633.
- ^ de Lichtenberg U, Jensen LJ, Fausbøll A, Jensen TS, Bork P, Brunak S (April 2005). "Comparison of computational methods for the identification of cell cycle-regulated genes". Bioinformatics. 21 (7): 1164–71. doi:10.1093/bioinformatics/bti093. PMID 15513999.
- ^ a b White MA, Riles L, Cohen BA (February 2009). "A systematic screen for transcriptional regulators of the yeast cell cycle". Genetics. 181 (2): 435–46. doi:10.1534/genetics.108.098145. PMC 2644938. PMID 19033152.
- ^ Lee TI, Rinaldi NJ, Robert F, Odom DT, Bar-Joseph Z, Gerber GK, et al. (October 2002). "Transcriptional regulatory networks in Saccharomyces cerevisiae". Science. 298 (5594): 799–804. Bibcode:2002Sci...298..799L. doi:10.1126/science.1075090. PMID 12399584. S2CID 4841222.
- ^ Simon I, Barnett J, Hannett N, Harbison CT, Rinaldi NJ, Volkert TL, et al. (September 2001). "Serial regulation of transcriptional regulators in the yeast cell cycle". Cell. 106 (6): 697–708. doi:10.1016/S0092-8674(01)00494-9. PMID 11572776. S2CID 9308235.
- ^ Sidorova JM, Mikesell GE, Breeden LL (December 1995). "Cell cycle-regulated phosphorylation of Swi6 controls its nuclear localization". Molecular Biology of the Cell. 6 (12): 1641–58. doi:10.1091/mbc.6.12.1641. PMC 301322. PMID 8590795.
- ^ Ubersax JA, Woodbury EL, Quang PN, Paraz M, Blethrow JD, Shah K, et al. (October 2003). "Targets of the cyclin-dependent kinase Cdk1". Nature. 425 (6960): 859–64. Bibcode:2003Natur.425..859U. doi:10.1038/nature02062. PMID 14574415. S2CID 4391711.
- ^ Morgan DO (2007). "2–3". The Cell Cycle: Principles of Control. London: New Science Press. p. 18. ISBN 978-0-9539181-2-6.
- ^ Omberg L, Meyerson JR, Kobayashi K, Drury LS, Diffley JF, Alter O (October 2009). "Global effects of DNA replication and DNA replication origin activity on eukaryotic gene expression". Molecular Systems Biology. 5: 312. doi:10.1038/msb.2009.70. PMC 2779084. PMID 19888207.
- ^ Alter O, Golub GH, Brown PO, Botstein D (February 2004). Deutscher MP, Black S, Boehmer PE, D'Urso G, Fletcher TM, Huijing F, et al. (eds.). Novel Genome-Scale Correlation between DNA Replication and RNA Transcription During the Cell Cycle in Yeast is Predicted by Data-Driven Models (PDF). Miami Nature Biotechnology Winter Symposium. Cell Cycle, Chromosomes and Cancer. Vol. 15. Miami Beach, FL: University of Miami School of Medicine.
- ^ Alter O, Golub GH (November 2004). "Integrative analysis of genome-scale data by using pseudoinverse projection predicts novel correlation between DNA replication and RNA transcription". Proceedings of the National Academy of Sciences of the United States of America. 101 (47): 16577–82. Bibcode:2004PNAS..10116577A. doi:10.1073/pnas.0406767101. PMC 534520. PMID 15545604.
- ^ Omberg L, Golub GH, Alter O (November 2007). "A tensor higher-order singular value decomposition for integrative analysis of DNA microarray data from different studies". Proceedings of the National Academy of Sciences of the United States of America. 104 (47): 18371–6. Bibcode:2007PNAS..10418371O. doi:10.1073/pnas.0709146104. PMC 2147680. PMID 18003902.
- ^ Elledge SJ (December 1996). "Cell cycle checkpoints: preventing an identity crisis". Science. 274 (5293): 1664–72. Bibcode:1996Sci...274.1664E. doi:10.1126/science.274.5293.1664. PMID 8939848. S2CID 39235426.
- ^ a b Vilenchik MM, Knudson AG (October 2003). "Endogenous DNA double-strand breaks: production, fidelity of repair, and induction of cancer". Proceedings of the National Academy of Sciences of the United States of America. 100 (22): 12871–6. Bibcode:2003PNAS..10012871V. doi:10.1073/pnas.2135498100. PMC 240711. PMID 14566050.
- ^ LeMaire-Adkins R, Radke K, Hunt PA (December 1997). "Lack of checkpoint control at the metaphase/anaphase transition: a mechanism of meiotic nondisjunction in mammalian females". The Journal of Cell Biology. 139 (7): 1611–9. doi:10.1083/jcb.139.7.1611. PMC 2132649. PMID 9412457.
- ^ Sakaue-Sawano A, Kurokawa H, Morimura T, Hanyu A, Hama H, Osawa H, et al. (February 2008). "Visualizing spatiotemporal dynamics of multicellular cell-cycle progression". Cell. 132 (3): 487–98. doi:10.1016/j.cell.2007.12.033. PMID 18267078. S2CID 15704902.
- ^ Rodriguez EA, Tran GN, Gross LA, Crisp JL, Shu X, Lin JY, Tsien RY (September 2016). "A far-red fluorescent protein evolved from a cyanobacterial phycobiliprotein". Nature Methods. 13 (9): 763–9. doi:10.1038/nmeth.3935. PMC 5007177. PMID 27479328.
- ^ Champeris Tsaniras S, Kanellakis N, Symeonidou IE, Nikolopoulou P, Lygerou Z, Taraviras S (June 2014). "Licensing of DNA replication, cancer, pluripotency and differentiation: an interlinked world?". Seminars in Cell & Developmental Biology. 30: 174–80. doi:10.1016/j.semcdb.2014.03.013. PMID 24641889.
- ^ Baserga R (June 1965). "The Relationship of the Cell Cycle to Tumor Growth and Control of Cell Division". Cancer Research. 25 (5): 581–95. PMID 14347544.
- ^ Mao Z, Bozzella M, Seluanov A, Gorbunova V (September 2008). "DNA repair by nonhomologous end joining and homologous recombination during cell cycle in human cells". Cell Cycle. 7 (18): 2902–6. doi:10.4161/cc.7.18.6679. PMC 2754209. PMID 18769152.
추가 정보
- Morgan DO (2007). The Cell Cycle: Principles of Control. London: Published by New Science Press in association with Oxford University Press. ISBN 978-0-87893-508-6.
- Alberts B, Johnson A, Lewis J, Raff M, Roberts K, Walter P (2008). "Chapter 17". Molecular Biology of the Cell (5th ed.). New York: Garland Science. ISBN 978-0-8153-4111-6.
- Krieger M, Scott MP, Matsudaira PT, Lodish HF, Darnell JE, Zipursky L, Kaiser C, Berk A (2004). Molecular cell biology. New York: W.H. Freeman and CO. ISBN 978-0-7167-4366-8.
- Watson JD, Baker TA, Bell SP, Gann A, Levine M, Losick R (2004). "Chapter 7". Molecular biology of the gene (5th ed.). San Francisco: Pearson/Benjamin Cummings. ISBN 978-0-8053-4642-8.
외부 링크
 이 문서에는 NCBI 문서의 퍼블릭 도메인 자료가 포함되어 있습니다.
이 문서에는 NCBI 문서의 퍼블릭 도메인 자료가 포함되어 있습니다.- David Morgan의 세미나:셀 사이클 제어
- 세포주기와 세포사망
- 셀 사이클의 전사 프로그램: 고해상도 타이밍
- 효모의 세포주기 및 대사주기 조절 전사
- 셀 사이클 애니메이션 1Lec.com
- 셀 사이클
- Fuci: GFP를 사용한 셀 사이클 시각화
- Science Creative Quarterly 세포주기 개요
- KEGG – 인간 세포 주기