DNA트랜스포존
DNA transposonDNA 트랜스포존은 게놈 [1]내의 다른 위치로 이동하고 통합될 수 있는 DNA 배열로, 때때로 "점핑 유전자"라고 불립니다.그들은 RNA [2]중간체를 통과하는 I급 TE와 달리 DNA 중간체를 통과하는 II급 Transposable Elements(TE; 역트랜스포존은 RNA 중간체를 통과하는 역트랜스포존입니다.DNA 트랜스포존은 단일 또는 이중 가닥 DNA [3]중간체를 통해 유기체의 DNA 내에서 이동할 수 있다.DNA 트랜스포존은 원핵 생물과 진핵 생물 모두에서 발견되었다.그들은 유기체의 게놈에서 특히 진핵생물에서 상당한 부분을 차지할 수 있다.원핵생물에서 TE는 항생제 내성이나 독성과 관련된 다른 유전자의 수평 이동을 촉진할 수 있다.숙주 내에서 복제 및 증식한 후, 모든 트랜스포존 복사는 활성화되지 않고, 수평 [4]전달을 수반하는 새로운 라이프 사이클을 개시함으로써 트랜스포존이 게놈에 전달되지 않는 한 손실된다.DNA 트랜스포존이 무작위로 게놈에 삽입되는 것이 아니라 특정 부위에 대한 선호도를 보인다는 것을 주목하는 것이 중요하다.
움직임과 관련하여, DNA 트랜스포존은 자율성과 [5]비자율성으로 분류될 수 있다.자율적인 것은 스스로 움직일 수 있는 반면, 비자율적인 것은 움직이기 위해 또 다른 전이성 요소의 유전자인 트랜스포제(transposase)의 존재를 필요로 한다.DNA 트랜스포존의 움직임에는 크게 세 가지 분류가 있습니다: "컷 [6]앤 페이스트," "롤링 서클,"[7] 그리고 "셀프 싱싱"[8]입니다.이러한 뚜렷한 운동 메커니즘은 그들이 유기체의 게놈 주위를 이동할 수 있게 해준다.DNA 트랜스포존은 DNA를 합성할 수 없기 때문에 숙주 복제 기계를 사용하여 복제한다.이 세 가지 주요 분류는 구조, 순서 및 [9]작용 메커니즘에 의해 특징지어지는 23개의 다른 슈퍼 패밀리로 더 세분화된다.
DNA 트랜스포존은 유전자 발현 변화의 원인이다.활성 코드 배열에 새로 삽입된 DNA는 정상적인 단백질 기능을 방해하고 돌연변이를 일으킬 수 있습니다.Class II TE는 인간 게놈의 약 3%를 차지한다.오늘날, 인간 게놈에는 활성 DNA 트랜스포존이 없다.그러므로 인간 게놈에서 발견되는 요소들은 화석이라고 불린다.
동작 메커니즘
컷 앤 페이스트
전통적으로, DNA 트랜스포존은 잘라내 붙여넣기 방식으로 게놈 안을 돌아다닌다.이 시스템은 게놈의 현재 위치에서 DNA의 움직임을 촉매하고 새로운 위치에 DNA를 삽입하는 트랜스포제 효소를 필요로 한다.전이를 위해서는 트랜스포존에 3개의 DNA 부위가 필요합니다. 즉, 말단 역반복이라고 불리는 트랜스포존의 양 끝에 2개가 있고 목표 부위에 1개가 있습니다.트랜스포자아제는 트랜스포존의 말단 역반복에 결합하고 트랜스포존 말단의 시냅시스를 매개한다.트랜스포자아제 효소는 그 원소를 원래 공여 부위의 옆쪽 DNA로부터 분리하고 트랜스포자를 새로운 삽입 부위로 연결하는 결합 반응을 매개한다.새 DNA를 대상 부위에 추가하면 삽입된 [10]세그먼트의 양쪽에 짧은 간격이 발생합니다.호스트 시스템은 이러한 갭을 복구하여 전이의 특징인 Target Sequence Duplication(TSD; 타깃 시퀀스 복제)을 생성합니다.많은 반응에서, 트랜스포존은 "컷 앤 페이스트"[11] 전이로 불리는 기증자 부위에서 완전히 제거되고 단순한 삽입을 형성하기 위해 표적 DNA에 삽입된다.간혹 원래 트랜스포저블 요소에 포함되지 않은 유전물질이 복사되고 이동되기도 한다.
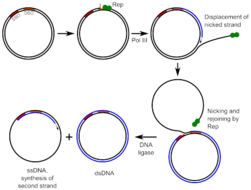
헬리트론
헬리트론은 또한 진핵생물 클래스 II TE의 그룹이다.헬리트론은 기존의 "컷 앤 페이스트" 메커니즘을 따르지 않습니다.대신에, 그들은 유전체 주위를 회전하는 원과 같은 메커니즘으로 움직인다는 가설이 있다.이 과정은 DNA를 두 개의 단일 가닥으로 분리하는 효소에 의해 원형 가닥에 흠집을 내는 것을 포함한다.그런 다음 시작 단백질은 절단된 가닥의 5' 인산염에 부착되어 상보적인 가닥의 3' 수산기를 노출시킨다.이것은 중합효소 효소가 무손상 가닥에서 복제를 시작할 수 있게 해줍니다.최종적으로 전체 가닥은 새로 합성된 DNA가 분리되고 원래의 템플릿 [12]가닥과 병렬로 복제되는 지점에서 복제된다.헬리트론은 HUH 핵산가수분해효소 기능뿐만 아니라 5'~3' 헬리케이스 활성을 갖는 것으로 생각되는 알려지지 않은 단백질을 부호화한다.이 효소는 DNA에 단일 가닥 절단을 만들어내서 Helitrons에서 발견된 표적 부위 중복이 없다는 것을 설명해준다.헬리트론은 또한 계산적으로 발견된 최초의 전이성 원소였고 전체 게놈 연구 [13]방식에 있어 패러다임의 변화를 나타냈다.
폴리온스
폴린톤은 또한 진핵생물 클래스 II TE의 그룹이다.진핵생물에서 가장 복잡한 것으로 알려진 DNA 트랜스포존 중 하나로서, 그들은 각각 엔타메바, 콩 녹, 닭과 같은 원생동물, 곰팡이, 그리고 동물들의 게놈을 구성합니다.그것들은 바이러스 단백질과 상동성을 가진 유전자를 포함하고 중합효소나 레트로바이러스 인테그레이스 같은 진핵생물 게놈에서 종종 발견됩니다.그러나 기능적으로 바이러스 캡시드나 외피 단백질과 유사한 알려진 단백질은 없다.그들은 단백질 프라이밍된 DNA 중합효소를 사용하여 복제되는 선형 플라스미드, 박테리오파지 및 아데노바이러스와 많은 구조적 특성을 공유합니다.폴린톤은 중합효소에 의해 유사한 자기합성을 거치는 것이 제안되었다.15~20kb 길이의 폴리톤은 최대 10개의 개별 단백질을 인코딩한다.복제를 위해 단백질 프라이밍 DNA 중합효소 B, 레트로바이러스 인테그레이스, 시스테인 단백질분해효소 및 ATP화효소를 이용한다.우선, 숙주 게놈 복제중에, 인테그라아제를 이용해, 단가닥 엑스트라 염색체 폴린톤 원소를 숙주 DNA로부터 절제해, 라켓 형태의 구조를 형성한다.둘째, 폴린톤은 DNA 중합효소 B를 사용하여 복제를 수행하며, 시작은 일부 선형 플라스미드에 코드될 수 있는 말단 단백질에 의해 시작됩니다.일단 이중 가닥 폴린톤이 생성되면, 인테그레이스는 그것을 숙주의 게놈에 삽입하는 역할을 한다.폴리톤은 다른 종 간에 높은 변동성을 보이며 엄격하게 규제될 수 있으며, 많은 [14]게놈에서 낮은 빈도로 나타난다.
분류
2015년 최신 업데이트를 기준으로, 유전자 [15]정보 연구소가 유지하는 반복 DNA 요소의 데이터베이스인 Repbase에 23개의 DNA 트랜스포존 슈퍼 패밀리가 인식되고 주석을 달았다.
트랜스포존의 효과
DNA 트랜스포존은 모든 트랜스포존과 마찬가지로 유전자 발현에 매우 영향을 미칩니다.일련의 DNA는 이전에 기능했던 유전자에 삽입되어 돌연변이를 일으킬 수 있다.이것은 세 가지 뚜렷한 방법으로 일어날 수 있다: 1. 기능의 변화, 2. 염색체 재배열, 그리고 3. 새로운 유전 [16]물질의 원천.DNA 트랜스포존이 유전체 배열의 일부를 가지고 갈 수 있기 때문에, 엑손 교환이 일어날 수 있다.엑손 셔플링은 이전에 관련이 없었던 두 엑손이 [17]전이를 통해 새롭게 배치되어 새로운 유전자 생성물이 만들어지는 것이다.DNA 발현을 바꾸는 능력 때문에 트랜스포존은 유전공학에서 중요한 연구 대상이 되었다.
예
옥수수
Barbara McClintock은 1940년대 Zea [18]mays에서 DNA 트랜스포존을 처음 발견하고 묘사했다; 이것은 그녀에게 1983년 노벨상을 안겨준 업적이다.그녀는 AC 단위(활성화제)가 자율적인 Ac/D 시스템을 설명했지만, D 게놈 단위는 이동하기 위해 활성제가 있어야 했다.이 TE는 옥수수 색깔을 노란색에서 갈색으로/개개의 알맹이에 얼룩이 생기게 할 수 있었기 때문에 시각적으로 가장 명백한 것 중 하나입니다.
초파리
마리너/Tc1 트랜스포존은 많은 동물에서 발견되지만 드로소필라에서 연구된 것으로 제이콥슨과 하틀에 [19]의해 처음 기술되었다.마리너는 새로운 [20]유기체에 수평으로 삽입하고 절제할 수 있는 것으로 잘 알려져 있다.수천 개의 TE 사본이 다른 동물들뿐만 아니라 인간 게놈에서 발견되었다.
Drosophila의 Hobo 트랜스포존은 성선발생을 [21]일으키는 능력 때문에 광범위하게 연구되어 왔다.호보 유사 배열의 삽입과 그 이후의 표현은 발달하는 파리의 생식선에서 생식세포의 손실을 초래한다.
박테리아
박테리아 트랜스포존은 미생물 간의 수평적 유전자 이동을 촉진하는 데 특히 뛰어나다.전이는 항생제 내성 유전자의 전달과 축적을 촉진한다.박테리아에서, 트랜스포저블 요소는 염색체 게놈과 플라스미드 사이를 쉽게 이동할 수 있다.1982년 Devaud 등의 연구에서 다제내성 Acinetobacter 균주가 분리 및 조사되었다.증거는 내성 유전자가 염색체 [22]게놈으로 옮겨진 박테리아에 플라스미드가 전달되는 것을 가리켰다.
유전적 다양성
트랜스포존은 많은 유기체의 유전적 다양성을 촉진하는 데 영향을 미칠 수 있다.DNA 트랜스포존은 DNA 염기서열 부분의 재배치를 촉진함으로써 게놈의 진화를 촉진할 수 있다.결과적으로, 이것은 유전자 조절 영역과 표현형을 [23]바꿀 수 있다.트랜스포존의 발견은 바바라 맥클린톡에 의해 이루어졌는데, 그는 이 요소들이 실제로 그녀가 연구하던 옥수수 식물의 색깔을 바꿀 수 있다는 것을 알아채고 트랜스포존 [24]운동으로 인한 한 가지 결과에 대한 빠른 증거를 제공했다.또 다른 예는 색소 침착 [25]패턴의 다양성의 결과로 알려진 메다카 물고기의 톨2 DNA 트랜스포존이다.이러한 예들은 트랜스포존이 게놈의 변화를 빠르게 유도함으로써 진화의 과정에 큰 영향을 미칠 수 있다는 것을 보여준다.
비활성화
모든 DNA 트랜스포존은 인간 [26]게놈에서 활성화되지 않는다.불활성화되거나 무음화된 트랜스포존은 표현형 결과를 초래하지 않으며 게놈 내에서 움직이지 않습니다.어떤 것들은 염색체 사이를 이동하는 능력에 영향을 미치는 돌연변이를 가지고 있기 때문에 비활동적인 반면, 다른 것들은 움직일 수 있지만 DNA 메틸화와 염색질 리모델링과 같은 후생적 방어 때문에 비활동적인 상태로 남아 있다.예를 들어, DNA의 화학적 수정은 전사 효소가 게놈에 도달할 수 없도록 게놈의 특정 영역을 수축시킬 수 있습니다.RNAi, 특히 siRNA와 miRNA 소음은 진핵생물 유전자 발현을 조절하는 것 외에 DNA 트랜스포존의 전사를 막는 자연적으로 발생하는 메커니즘이다.불활성화의 또 다른 방법은 과잉 생산 억제이다.트랜스포자아제가 역치 농도를 초과하면 트랜스포자 활성은 [27]감소한다.트랜스포사아제는 전체적으로 전위 활성을 감소시키는 비활성 또는 덜 활성 단량체를 형성할 수 있기 때문에, 트랜스포사아제 생산의 감소는 또한 숙주 게놈에서 덜 활성인 요소의 큰 복사본이 증가할 때 발생할 것이다.
수평 전송
수평 이동은 다른 유기체의 세포들 사이에서 DNA 정보가 이동하는 것을 말한다.수평 이동은 한 유기체에서 다른 유기체의 게놈으로 TE가 이동하는 것을 포함할 수 있다.삽입 자체는 TE가 새로운 숙주의 활성화된 유전자가 되도록 한다.수평 전달은 트랜스포존의 불활성화와 완전한 손실을 방지하기 위해 DNA 트랜스포존에 의해 사용된다.이 불활성화를 수직 불활성화라고 하는데, 이는 DNA 트랜스포존이 비활성 상태이고 화석으로 남아있다는 것을 의미합니다.이러한 유형의 전달은 가장 흔하지는 않지만, 서로 다른 균류 병원 Parastagonospora nodorum, Pyrenophora tritici-repentis [28]및 Bolifolis sorokiniana 사이에서 전달된 밀 독성 단백질 ToxA의 경우에 나타났다.다른 예로는 해양 갑각류, 다른 목의 곤충들, 그리고 인간과 [29]선충과 같은 서로 다른 식물의 유기체들 사이의 이동을 포함한다.
진화
진핵생물 게놈은 TE 함량이 다르다.최근 TE의 다른 슈퍼패밀리에 대한 연구는 그룹 사이에 현저한 유사성이 있다는 것을 보여준다.그들 중 많은 수가 두 개 이상의 진핵생물의 슈퍼그룹으로 표현된다는 가설이 있다.이것은 트랜스포존 슈퍼패밀리의 차이가 진핵 슈퍼그룹의 [30]차이보다 앞서 있을 수 있다는 것을 의미한다.
V(D)J 재결합
V(D)J 재조합은 DNA TE는 아니지만 트랜스포존과 현저하게 유사하다.V(D)J 재조합은 항체 결합 부위에 큰 변화가 생기는 과정이다.이 메커니즘에서 DNA는 유전자 [31]다양성을 만들기 위해 재조합된다.이 때문에 이들 단백질, 특히 Rag1과 Rag2는[32] 전이성 [33]요소에서 유래한다는 가설이 제기되어 왔다.
인간 게놈의 멸종
포유류 방사선과 초기 영장류 혈통 동안 적어도 40개의 인간 DNA 트랜스포존 가족이 활동했다는 증거가 있다.그 후, 영장류 방사선의 후반부에서 전위 활동이 중단되었고, 인체형 영장류 조상의 트랜스포존 이동이 완전히 중단되었다.약 3700만년 [34]전의 전이성 원소에 대한 증거는 없다.
레퍼런스
- ^ "Transposon genetics". Encyclopedia Britannica. Retrieved 2019-10-28.
- ^ Wicker, Thomas; Sabot, François; Hua-Van, Aurélie; Bennetzen, Jeffrey L.; Capy, Pierre; Chalhoub, Boulos; Flavell, Andrew; Leroy, Philippe; Morgante, Michele (2007). "A unified classification system for eukaryotic transposable elements". Nature Reviews Genetics. 8 (12): 973–982. doi:10.1038/nrg2165. PMID 17984973. S2CID 32132898.
- ^ Feschotte, Cédric; Pritham, Ellen J. (December 2007). "DNA Transposons and the Evolution of Eukaryotic Genomes". Annual Review of Genetics. 41 (1): 331–368. doi:10.1146/annurev.genet.40.110405.090448. PMC 2167627. PMID 18076328.
- ^ Muñoz-López, Martín; García-Pérez, José L. (April 2010). "DNA Transposons: Nature and Applications in Genomics". Current Genomics. 11 (2): 115–128. doi:10.2174/138920210790886871. ISSN 1389-2029. PMC 2874221. PMID 20885819.
- ^ "Transposons Learn Science at Scitable". www.nature.com. Retrieved 2019-10-28.
- ^ Craig, Nancy L. (1995-10-13). "Unity in Transposition Reactions". Science. 270 (5234): 253–4. Bibcode:1995Sci...270..253C. doi:10.1126/science.270.5234.253. ISSN 0036-8075. PMID 7569973. S2CID 29930180.
- ^ Kapitonov, Vladimir V.; Jurka, Jerzy (2001-07-17). "Rolling-circle transposons in eukaryotes". Proceedings of the National Academy of Sciences of the United States of America. 98 (15): 8714–8719. Bibcode:2001PNAS...98.8714K. doi:10.1073/pnas.151269298. ISSN 0027-8424. PMC 37501. PMID 11447285.
- ^ Kapitonov, Vladimir V.; Jurka, Jerzy (2006-03-21). "Self-synthesizing DNA transposons in eukaryotes". Proceedings of the National Academy of Sciences of the United States of America. 103 (12): 4540–4545. Bibcode:2006PNAS..103.4540K. doi:10.1073/pnas.0600833103. ISSN 0027-8424. PMC 1450207. PMID 16537396.
- ^ Kapitonov, Vladimir V.; Jurka, Jerzy (2006-03-21). "Self-synthesizing DNA transposons in eukaryotes". Proceedings of the National Academy of Sciences of the United States of America. 103 (12): 4540–4545. Bibcode:2006PNAS..103.4540K. doi:10.1073/pnas.0600833103. ISSN 0027-8424. PMC 1450207. PMID 16537396.
- ^ Berg and Howe, Douglas E. and Martha M. (1989). Mobile DNA II. ASM Press. p. 98. ISBN 9781555812096.
- ^ Madigan M, Martinko J, eds. (2006). Brock Biolog of Microorganisms (11th ed.). Prentice Hall. ISBN 0-13-144329-1.
- ^ "Rolling circle replication - The School of Biomedical Sciences Wiki". teaching.ncl.ac.uk. Retrieved 2019-10-06.
- ^ Thomas J, Pritham EJ (August 2015). "Helitrons, the Eukaryotic Rolling-circle Transposable Elements". Microbiology Spectrum. 3 (4): 893–926. doi:10.1128/microbiolspec.MDNA3-0049-2014. ISBN 9781555819200. PMID 26350323.
- ^ Kapitonov VV, Jurka J (March 2006). "Self-synthesizing DNA transposons in eukaryotes". Proceedings of the National Academy of Sciences of the United States of America. 103 (12): 4540–5. Bibcode:2006PNAS..103.4540K. doi:10.1073/pnas.0600833103. PMC 1450207. PMID 16537396.
- ^ Bao W, Kojima KK, Kohany O (2 June 2015). "Repbase Update, a database of repetitive elements in eukaryotic genomes". Mobile DNA. 6 (1): 11. doi:10.1186/s13100-015-0041-9. PMC 4455052. PMID 26045719.
- ^ Feschotte C, Pritham EJ (2007). "DNA transposons and the evolution of eukaryotic genomes". Annual Review of Genetics. 41: 331–68. doi:10.1146/annurev.genet.40.110405.090448. PMC 2167627. PMID 18076328.
- ^ Morgante M, Brunner S, Pea G, Fengler K, Zuccolo A, Rafalski A (September 2005). "Gene duplication and exon shuffling by helitron-like transposons generate intraspecies diversity in maize". Nature Genetics. 37 (9): 997–1002. doi:10.1038/ng1615. PMID 16056225. S2CID 10401931.
- ^ McClintock, Barbara (1950). "The origin and behavior of mutable loci in maize". Proc Natl Acad Sci U S A. 36 (6): 344–55. Bibcode:1950PNAS...36..344M. doi:10.1073/pnas.36.6.344. PMC 1063197. PMID 15430309.
- ^ Jacobson, JW; Medhora, MM; Hartl, DL (Nov 1986). "Molecular structure of a somatically unstable transposable element in Drosophila". Proc Natl Acad Sci U S A. 83 (22): 8684–8. Bibcode:1986PNAS...83.8684J. doi:10.1073/pnas.83.22.8684. PMC 386995. PMID 3022302.
- ^ Lohe, AR; Moriyama, EN; Lidholm, DA; Hartl, DL (1995). "Horizontal transmission, vertical inactivation, and stochastic loss of mariner-like transposable elements". Mol. Biol. Evol. 12 (1): 62–72. doi:10.1093/oxfordjournals.molbev.a040191. PMID 7877497.
- ^ Deprá, M; Valente, VL; Margis, R; Loreto, EL (2009). "The hobo transposon and hobo-related elements are expressed as developmental genes in Drosophila". Gene. 448 (1): 57–63. doi:10.1016/j.gene.2009.08.012. PMID 19720121.
- ^ Devaud, M; Kayser, FH; Bächi, B (Aug 1982). "Transposon-mediated multiple antibiotic resistance in Acinetobacter strains". Antimicrob Agents Chemother. 22 (2): 323–9. doi:10.1128/aac.22.2.323. PMC 183733. PMID 6100428.
- ^ "Transposons Learn Science at Scitable". www.nature.com. Retrieved 2019-11-01.
- ^ "Barbara McClintock and the Discovery of Jumping Genes (Transposons) Learn Science at Scitable". www.nature.com. Retrieved 2019-10-07.
- ^ Koga, Akihiko; Iida, Atsuo; Hori, Hiroshi; Shimada, Atsuko; Shima, Akihiro (July 2006). "Vertebrate DNA transposon as a natural mutator: the medaka fish Tol2 element contributes to genetic variation without recognizable traces". Molecular Biology and Evolution. 23 (7): 1414–1419. doi:10.1093/molbev/msl003. ISSN 0737-4038. PMID 16672286.
- ^ "Transposons Learn Science at Scitable". www.nature.com. Retrieved 2019-11-01.
- ^ Muñoz-López, Martín; García-Pérez, José L. (April 2010). "DNA Transposons: Nature and Applications in Genomics". Current Genomics. 11 (2): 115–128. doi:10.2174/138920210790886871. ISSN 1389-2029. PMC 2874221. PMID 20885819.
- ^ McDonald, Megan C.; Taranto, Adam P.; Hill, Erin; Schwessinger, Benjamin; Liu, Zhaohui; Simpfendorfer, Steven; Milgate, Andrew; Solomon, Peter S. (2019-10-29). "Transposon-Mediated Horizontal Transfer of the Host-Specific Virulence Protein ToxA between Three Fungal Wheat Pathogens". mBio. 10 (5). doi:10.1128/mBio.01515-19. ISSN 2150-7511. PMC 6737239. PMID 31506307.
- ^ Muñoz-López, Martín; García-Pérez, José L. (April 2010). "DNA Transposons: Nature and Applications in Genomics". Current Genomics. 11 (2): 115–128. doi:10.2174/138920210790886871. ISSN 1389-2029. PMC 2874221. PMID 20885819.
- ^ Feschotte C, Pritham EJ (2007). "DNA transposons and the evolution of eukaryotic genomes". Annual Review of Genetics. 41: 331–68. doi:10.1146/annurev.genet.40.110405.090448. PMC 2167627. PMID 18076328.
- ^ Jung D, Alt FW (January 2004). "Unraveling V(D)J recombination; insights into gene regulation". Cell. 116 (2): 299–311. doi:10.1016/S0092-8674(04)00039-X. PMID 14744439. S2CID 16890458.
- ^ Bourque G, Burns KH, Gehring M, Gorbunova V, Seluanov A, Hammell M, et al. (November 2018). "Ten things you should know about transposable elements". Genome Biology. 19 (1): 199. doi:10.1186/s13059-018-1577-z. PMC 6240941. PMID 30454069.
- ^ Muñoz-López M, García-Pérez JL (April 2010). "DNA transposons: nature and applications in genomics". Current Genomics. 11 (2): 115–28. doi:10.2174/138920210790886871. PMC 2874221. PMID 20885819.
- ^ Pace JK, Feschotte C (April 2007). "The evolutionary history of human DNA transposons: evidence for intense activity in the primate lineage". Genome Research. 17 (4): 422–32. doi:10.1101/gr.5826307. PMC 1832089. PMID 17339369.