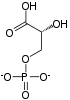
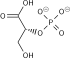
화농산탈수소효소 복합체
Pyruvate dehydrogenase complex피루베이트 탈수소효소 복합체(PDC)는 피루베이트 디카르복시화라는 공정에 의해 피루베이트를 아세틸-CoA로 변환하는 3가지 효소의 복합체다.[1] 아세틸-CoA는 구연산 사이클에서 세포호흡을 수행하기 위해 사용될 수 있으며, 이 복합체는 글리코 분해 대사 경로를 구연산 사이클과 연결한다. 피루베이트 디카르복시화(Pyruvate detarboxylation)는 피루베이트의 산화 작용도 수반되기 때문에 "피루베이트 탈수소효소 반응"으로도 알려져 있다.[2]
이 멀티엔자임 콤플렉스는 옥소글루타레이트 탈수소효소 및 브랜치 체인 옥소산탈수소효소 멀티엔자임 콤플렉스와 구조적으로 기능적으로 관련이 있다.
반응
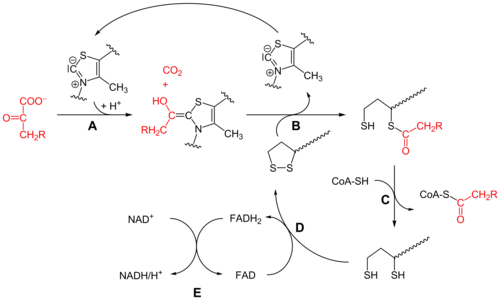
화농산 탈수소효소 복합체에 의해 촉매되는 반응은 다음과 같다.
| 화농성의 | 화농산탈수소효소 복합체 | 아세틸 코아 | |
 |  | ||
| CoA-SH + NAD+ | CO2 + NADH + H+ | ||
 | |||
구조
화농산탈수소효소(E1)
화농산 탈수소효소 소단위라 불리는 E1 소단위에는 두 개의 체인("ɑ"와 "ꞵ" 체인)으로 구성된 구조가 있다. 마그네슘 이온은 3개의 극성 아미노산 잔류물(Asp, Asn, Tyr)과 피루베이트의 디카르복시화에 직접 관여하는 티아민 디파인산(TPP) 공동 인자(coactor)를 가진 4-코멘트 콤플렉스를 형성한다.[3][4]
디하이드롤리포일 트랜스아세틸라아제(E2)
원핵생물 및 진핵생물 모두에 대한 E2 소단위 또는 디히드롤리포일 아세틸전달효소는 일반적으로 세 개의 영역으로 구성된다. N-단자 영역(the lipoyl domain)은 각각 약 80개의 아미노산으로 구성된 1-3개의 지질군으로 구성되어 있다. 주변 서브유닛 바인딩 도메인(PBSD)은 E1 및 E3 서브유닛의 다른 도메인의 선택적 바인딩 사이트 역할을 한다. 마지막으로 C-단자(catalytic) 영역은 아세틸 그룹의 전송과 아세틸-CoA 합성을 촉진한다.[5]
디하이드롤리포일탈수소효소(E3)
디하이드롤리포일 탈수소효소라고 불리는 E3 소분위는 이황화 결합에 관여하는 두 개의 시스테인 잔류물에서 산화촉매로서 주요 목적을 촉진하는 호모디머 단백질로 특징지어진다. Phyomonas putida에서 발견된 E3 구조의 한 예는 각각의 호모디머 서브유닛이 FAD 바인딩과 NAD 바인딩을 담당하는 두 개의 바인딩 도메인과 중앙 도메인과 인터페이스 도메인을 포함하도록 형성된다.[6][7]
디하이드롤리포일탈수소효소결합단백질(E3BP)
대부분의 진핵생물에 고유한 보조 단백질은 E3 결합 단백질(E3BP)으로 E3 서브 유닛을 PDC 콤플렉스에 바인딩하는 역할을 한다. 인간 E3BP의 경우, BP의 소수성 프로라인과 류신 잔여물은 동일한 E3 단량체의 결합에 의해 형성된 표면 인식 부위와 상호작용한다.[8]
메커니즘
| 효소 | 줄여서. | 공동 인자 | # 원핵생물들 | # eukaryotes 하위유닛 |
|---|---|---|---|---|
| 화농산탈수소효소 (EC1.2.4.1) | E1 | TPP(Thiamine Pyrophosphate) | 24 | 30 |
| 디하이드롤리포일 트랜스 아세틸라아제 (EC2.3.1.12) | E2 | 지방질의 코엔자임 A | 24 | 60 |
| 디하이드롤리포일탈수소효소 (EC1.8.1.4) | E3 | FAD NAD+ | 12 | 12 |
화농산탈수소효소(E1)
초기에는 피루베이트와 티아민 피로인산염(TPP 또는 비타민 B1)은 피루베이트 탈수소효소 서브유닛에 의해 결합된다.[1] TPP의 티아졸륨 링은 zwitterionic 형태로 되어 있으며 음이온 C2 탄소는 피루베이트의 C2(케톤) 카보닐에 핵포필러 공격을 실시한다. 그 결과 헤미티오아세탈은 아킬 음이온 등가물을 생성하기 위해 데카르복시술을 거친다(벤조인 응축은 물론 시아노리딘 또는 알데히드디티아네 umpolung 화학 참조). 이 음이온은 리신 잔여물에 부착된 산화 지포종의 S1을 공격한다. 링 개구부 S2와N 같은 메커니즘에서 S2는 황화 또는 황하이드릴 모이티로 대체된다. 이후 사면체혈모세포가 붕괴되면 티아졸이 배출되고 TPP 공동요소가 방출되며 지포산염 S1에 티오아세테이트가 생성된다. E1-catalyze 공정은 전체 화농산 탈수소효소 복합체의 속도제한 단계다.
디하이드롤리포일 트랜스아세틸라아제(E2)
이때 지혈-티오에스터 기능성은 디히드로리포일 트랜스 아세틸라제(E2) 활성 부위로 번역되며,[1] 여기서 트랜스액션 반응은 지혈의 "스윙 암"에서 코엔자임 A의 티올로 아세틸을 전달한다. 이것은 아세틸-CoA를 생성하는데, 효소 복합체에서 방출되어 그 후 구연산 사이클로 들어간다. E2는 또한 리포아미드 환원효소-투명세틸라아제라고도 알려져 있다.
디하이드롤리포일탈수소효소(E3)
이 화합물의 리신 잔여물과 여전히 결합되어 있는 디히드로이포이산은 이황화 이소머라아제와 화학적으로 동일한 플라빈 매개 산화를 [1]거치는 디히드로이포일(E3) 활성 부위로 이동한다. 우선 FAD는 디하이드롤리포이트를 다시 지포 휴면 상태로 산화시켜 FADH를2 생성한다. 그 후, NAD의+ 공동 작용자는 FADH를2 다시 FAD 휴면 상태로 산화시켜 NADH와 H를 생산한다.
종간의 구조적 차이
PDC는 종에 따라 3~4개의 서브유닛을 여러 장 복사해 놓은 대규모 복합단지다.
그람 음성 박테리아
그람 음성 박테리아(예: 대장균)에서 PDC는 디하이드롤리포일 트랜스아세틸라아제(E2)의 24개 분자로 구성된 중앙 입방 코어로 구성된다. 최대 24개의 피루브산탈수소효소(E1)와 12개의 디히드로이포일탈수소효소(E3) 분자가 E2코어의 외부에 결합한다.[9]
그램 양성균 및 진핵생물
이와는 대조적으로 그람 양성 박테리아(예: 바실러스 스테아로테모필루스)와 진핵생물에서 중심 PDC 코어는 60개의 E2 분자를 아이코사헤드론으로 배열하여 포함하고 있다. 이 E2 서브유닛 "핵심"은 E1의 서브유닛 30개와 E3의 복사본 12개로 조정된다.
eukaryotes는 또한 E3 하위 유니트를 E2 코어에 바인딩하는 추가 코어 단백질인 E3 결합 단백질(E3BP)을 12개 포함하고 있다.[10] E3BP의 정확한 위치가 완전히 명확하지는 않다. 극저온 현미경 검사에서 E3BP가 효모에 있는 각 이두면체 면에 결합한다는 것이 밝혀졌다.[11] 그러나, 그것은 소 PDC 코어에 있는 동등한 수의 E2 분자를 대체하는 것이 제안되었다.
최대 60개의 E1 또는 E3 분자가 그람 양성 박테리아로부터 E2 코어와 연관될 수 있다 - 결합은 상호 배타적이다. eukaryotes에서 E1은 특히 E2에 의해 구속되는 반면, E3는 E3BP와 연관된다. 정확한 분자의 수는 생체내 다양할 수 있고 종종 해당 조직의 대사 요구 사항을 반영하지만, 최대 30 E1과 6 E3 효소가 존재한다고 생각된다.
규정
ATP/ADP, NADH/NAD, 아세틸-CoA/CoA 등 3가지 비율 중 하나 이상이 증가하면 화농산 탈수소효소가 억제된다.
eukaryotes에서 PDC는 자체 고유 화농산탈수소효소 키나아제(PDK)와 화농산탈수소효소인산효소(PDP)에 의해 엄격하게 규제되어 각각 비활성화 및 활성화된다.[12]
- PDK 인산염은 E1의 세린 잔류물 3개를 서로 다른 친화력으로 처리한다. 그것들 중 하나(ATP 사용)의 인산화 작용은 E1 (그리고 결과적으로 전체 콤플렉스를 비활성으로 만든다.[12]
- PDP에 의한 E1의 탈인산화는 복잡한 활동을 재설치한다.[12]
반응의 생산물은 PDK를 활성화하기 때문에 PDC의 알로스테릭 억제제로 작용한다. 기판은 PDK를 억제하고 PDC를 재활성화한다.
굶주리는 동안 PDK는 유전자 전사의 증가를 통해 골격근육을 포함한 대부분의 조직에서 양이 증가한다. 같은 조건에서 PDP의 양은 감소한다. PDC의 결과적 억제는 근육과 다른 조직들이 포도당과 글루코네제네시스 전구체들을 격분시키는 것을 막는다. 신진대사는 지방활용 쪽으로 이동하고, 글루코네제네시스 전구체를 공급하기 위한 근육단백질 분해는 최소화하며, 이용 가능한 포도당은 뇌가 사용할 수 있도록 절약된다.
칼슘 이온은 근육 조직에서 PDC의 조절에 역할을 하는데, 이는 PDP를 활성화시켜 근육 수축 중에 세포솔로 방출되는 글리콜리분해를 자극하기 때문이다. 이러한 성형의 일부 제품들은 H2를 근육으로 방출한다. 이것은 칼슘 이온을 시간이 지남에 따라 썩게 할 수 있다.
피루베이트 디카복시화의 국산화
진핵 세포에서 피루베이트 디카복시술은 세포솔로부터 기질인 피루베이트를 운반한 후 미토콘드리아 매트릭스 안에서 발생한다. 피루베이트를 미토콘드리아로 운반하는 것은 피루베이트 반투명효소를 운반하는 단백질이다. 피루베이트 반투명효소는 양자와 함께 심볼포트 방식으로 피루베이트를 운반하여 활동적으로 에너지를 소비한다.[citation needed] 대안적 출처에 따르면 "외부 미토콘드리아 막에 걸친 피루브레이트 수송은 수동적 확산을 가능하게 하는 전압 의존적 음이온 채널과 같은 대형 비선택적 채널을 통해 쉽게 달성되는 것으로 보인다"고 하며 내측 미토콘드리아 막에 걸친 수송은 미토콘드리아 피루브레이트 운반체 1(MPC1)와 미토콘드리아에 의해 매개된다.격납 캐리어 2([13]MPC2)
미토콘드리아에 들어갈 때, 피루브산은 아세틸-CoA를 생성하면서 디카르복실화된다. 이 되돌릴 수 없는 반응은 아세틸-CoA를 미토콘드리아 내(아세틸-CoA는 일반적으로 희박한 TCA 중간물인 구연산 셔틀을 통해 높은 옥살로아세테이트 조건에서만 미토콘드리아 매트릭스 밖으로 운반할 수 있다). 이 반응에 의해 생성되는 이산화탄소는 비극성이고 작으며, 미토콘드리아와 세포 밖으로 확산될 수 있다.
미토콘드리아가 없는 원핵생물에서는 이러한 반응이 시토솔에서 수행되거나 전혀 수행되지 않는다.
진화사
진핵세포의 미토콘드리아에서 발견된 피루바이트 탈수소효소 효소가 그램 양성균의 일종인 지오바실러스 스테아로테모필루스(Geaacillus starothermophilus)의 효소와 매우 유사한 것으로 나타났다. 그램 양성 박테리아와 화농산 탈수소효소 복합체가 유사함에도 불구하고 그램 음성 박테리아와 유사한 점은 거의 없다. 그램 양성 박테리아 내 피루베이트 탈수소효소와 효소의 4차 구조물의 유사성은 그램 음성 박테리아에서 발견되는 해당 효소의 진화 이력과 구별되는 공통된 진화 이력을 가리킨다. 내분비생물학적 사건을 통해 진핵 미토콘드리아에서 발견되는 화농산탈수소효소는 그램 양성 박테리아로 거슬러 올라가는 조상 연계를 가리킨다.[14] 피루베이트 탈수소효소 복합체는 특히 알파케토산의 기질특성에서 분기체인 2-옥소산화 탈수소효소(BCOADH)와 많은 유사점을 공유한다. 구체적으로, BCOADH는 아미노산의 저하를 촉진하며 이러한 효소는 풍부한 아미노산 환경이 지배하는 선사시대 지구상에 널리 퍼졌을 것이다. 화농산염 탈수소효소에서 나온 E2 소분위는 BCOADH에서 발견된 E2 유전자에서 진화했지만 E3 유전자가 하나만 존재하기 때문에 두 효소 모두 동일한 E3 소분위를 함유하고 있다. E1 서브유닛은 특정 기질에 대해 독특한 특수성을 가지고 있기 때문에, E1 서브유닛은 화농산 탈수소효소와 BCOADH는 다양하지만 유전적 유사성을 공유한다. 나중에 진핵 세포에서 발견된 미토콘드리아와 엽록체를 발생시킬 그램 양성 박테리아와 시아노박테리아들은 BCOADH 효소에서 발견된 것과 유전적으로 관련된 E1 하위 단위를 유지했다.[15][16]
임상 관련성
화농산탈수소효소 결핍증(PCDC)은 효소나 공동 인자 내의 돌연변이로 인해 발생할 수 있다. 이것의 주된 임상적 발견은 젖산증이다.[17] 그러한 PCDC 돌연변이는 NAD와 FAD 생산의 후속적인 결함으로 이어져 유산소 호흡의 핵심인 산화 인산화 과정을 방해한다. 따라서 아세틸-CoA는 혐기성 메커니즘을 통해 젖산염과 같은 다른 분자로 감소되어 체내 젖산염과 관련된 신경학적 병리학의 과잉으로 이어진다.[18]
화농산탈수소효소 결핍은 드물지만 돌연변이 시 다양한 유전자가 존재하거나 비기능성 유전자가 있어 이 결핍을 유도할 수 있다. 첫째, 화농산 탈수소효소의 E1 소단위에는 E1-알파로 지정된 알파 서브유닛 2개와 E1-베타로 지정된 베타 서브유닛 2개가 들어 있다. E1-알파 하위유닛에서 발견된 PDHA1 유전자는 돌연변이가 E1-알파 단백질을 파괴하기 때문에 피루베이트 탈수소효소 결핍증 사례의 80%를 유발한다. 기능적 E1 알파 감소는 피루베이트 탈수소효소가 피루베이트에 충분히 결합되지 않도록 방지하여 전체 복합체의 활성을 감소시킨다.[19] 복합체의 E1 베타 서브유닛에서 발견된 PDHB 유전자가 돌연변이를 일으키면, 이는 또한 화농산탈수소효소 결핍으로 이어진다.[20] 마찬가지로, E2 서브유닛에서 발견된 DLAT 유전자, E3 서브유닛에서 발견된 PDHX 유전자, 그리고 PDP1로 알려진 화농산탈수소효소인산효소 유전자의 돌연변이와 같이 복합체의 다른 서브유닛에서 발견된 돌연변이는 모두 화농산탈수소효소 결핍에 의해 추적된 반면, 질병에 대한 특정 기여도는 우박산염이다.미지의[21][22][23]
참고 항목
참조
- ^ a b c d DeBrosse, Suzanne D.; Kerr, Douglas S. (2016-01-01), Saneto, Russell P.; Parikh, Sumit; Cohen, Bruce H. (eds.), "Chapter 12 - Pyruvate Dehydrogenase Complex Deficiency", Mitochondrial Case Studies, Boston: Academic Press, pp. 93–101, doi:10.1016/b978-0-12-800877-5.00012-7, ISBN 978-0-12-800877-5, retrieved 2020-11-16
- ^ J. M. Berg; J. L. Tymoczko, L. Stryer (2007). Biochemistry (6 ed.). Freeman. ISBN 978-0-7167-8724-2.
{{cite book}}: CS1 maint : 복수이름 : 작성자 목록(링크) - ^ Sgrignani, J.; Chen, J.; Alimonti, A. (2018). "How phosphorylation influences E1 subunit pyruvate dehydrogenase: A computational study". Scientific Reports. 8 (14683): 14683. Bibcode:2018NatSR...814683S. doi:10.1038/s41598-018-33048-z. PMC 6168537. PMID 30279533. S2CID 52910721.
- ^ [대통령 예산 확정 ID:2QTC]케일, S.;Arjunan, P.;퓨리, W., 요르단, F.(2007년)."E2성분으로 대장 균 피루브산 탈수소 효소 COMPLEX E1요소 Modulates SUBSTRATE 활용 및 CHEMICAL 통신의 적극적인 센터의 한 동적 루프".필기장 생물학 화학의. 282(38):28106–28116. doi:10.1074/jbc.m704326200.PMID 17635929.S2CID 25199383.
- ^ Patel, M. S.; Nemeria, N. S.; Furey, W.; Jordan, F. (2014). "The pyruvate dehydrogenase complexes: structure-based function and regulation". The Journal of Biological Chemistry. 289 (24): 16615–16623. doi:10.1074/jbc.R114.563148. PMC 4059105. PMID 24798336.
- ^ Billgren, E. S.; Cicchillo, R. M.; Nesbitt, N. M.; Booker, S. J. (2010). "Lipoic Acid Biosynthesis and Enzymology". Comprehensive Natural Products. 2 (7): 181–212. doi:10.1016/B978-008045382-8.00137-4.
- ^ [PDB ID: 1LVL] Mattevia, A.; Obmolova, G.; Sokatch, J. R.; Betzel, C.; Hol, W. G. (1992). "The refined crystal STRUCTURE of pseudomonas Putida LIPOAMIDE DEHYDROGENASE complexed with NAD+ at 2.45 Å resolution". Proteins: Structure, Function, and Genetics. 13 (4): 336–351. doi:10.1002/prot.340130406. PMID 1325638. S2CID 23288363.
- ^ Ciszak, E. M.; Makal, A.; Hong, Y. S.; Vettaikkorumakankauv, A. K.; Korotchkina, L. G.; Patel, M. S. (2006). "How dihydrolipoamide dehydrogenase-binding protein binds dihydrolipoamide dehydrogenase in the human pyruvate dehydrogenase complex". Journal of Biological Chemistry. 281 (1): 648–655. doi:10.1074/jbc.m507850200. PMID 16263718. S2CID 26797600.
- ^ Izard T, Aevarsson A, Allen MD, Westphal AH, Perham RN, de Kok A, Hol WG (1999). "Principles of quasi-equivalence and Euclidean geometry govern the assembly of cubic and dodecahedral cores of pyruvate dehydrogenase complexes". Proc. Natl. Acad. Sci. USA. 96 (4): 1240–1245. Bibcode:1999PNAS...96.1240I. doi:10.1073/pnas.96.4.1240. PMC 15447. PMID 9990008. [1]
- ^ Brautigam, C. A.; Wynn, R. M.; Chuang, J. L.; Machius, M.; Tomchick, D. R.; Chuang, D. T. (2006). "Structural insight into interactions between Dihydrolipoamide Dehydrogenase (E3) and E3 binding protein of Human pyruvate dehydrogenase complex". Structure. 14 (3): 611–621. doi:10.1016/j.str.2006.01.001. PMC 2879633. PMID 16442803.
- ^ Stoops, J.K., Cheng, R.H., Yazdi, M.A., Maeng, C.Y., Schroeter, J.P., Klueppelberg, U., Kolodziej, S.J., Baker, T.S., Reed, L.J. (1997) On the unique structural organization of the Saccharomyces cerevisiae pyruvate dehydrogenase complex. J. 비올. 화학 272, 5757-5764
- ^ a b c Pelley, John W. (2012-01-01), Pelley, John W. (ed.), "6 - Glycolysis and Pyruvate Oxidation", Elsevier's Integrated Review Biochemistry (Second Edition), Philadelphia: W.B. Saunders, pp. 49–55, doi:10.1016/b978-0-323-07446-9.00006-4, ISBN 978-0-323-07446-9, retrieved 2020-11-16
- ^ Rutter, Jared (23 January 2013). "The long and winding road to the mitochondrial pyruvate carrier". Cancer & Metabolism. 1 (1): 6. doi:10.1186/2049-3002-1-6. PMC 3834494. PMID 24280073.
- ^ Henderson, Christopher E.; Perham, Richard N.; Finch, John T. (May 1979). "Structure and symmetry of B. stearothermophilus pyruvate dehydrogenase multienzyme complex and implications for eucaryote evolution". Cell. 17 (1): 85–93. doi:10.1016/0092-8674(79)90297-6. ISSN 0092-8674. PMID 455461. S2CID 35282258.
- ^ Schreiner, Mark E.; Fiur, Diana; Holátko, Jiří; Pátek, Miroslav; Eikmanns, Bernhard J. (2005-09-01). "E1 Enzyme of the Pyruvate Dehydrogenase Complex in Corynebacterium glutamicum: Molecular Analysis of the Gene and Phylogenetic Aspects". Journal of Bacteriology. 187 (17): 6005–6018. doi:10.1128/jb.187.17.6005-6018.2005. ISSN 0021-9193. PMC 1196148. PMID 16109942.
- ^ Schnarrenberger, Claus; Martin, William (2002-02-01). "Evolution of the enzymes of the citric acid cycle and the glyoxylate cycle of higher plants". European Journal of Biochemistry. 269 (3): 868–883. doi:10.1046/j.0014-2956.2001.02722.x. ISSN 0014-2956. PMID 11846788.
- ^ "Pyruvate dehydrogenase deficiency". Genetics Home Reference. Retrieved March 17, 2013.
- ^ Gupta, N.; Rutledge, C. (2019). "Pyruvate Dehydrogenase Complex Deficiency: An Unusual Cause of Recurrent Lactic Acidosis in a Paediatric Critical Care Unit". The Journal of Critical Care Medicine. 5 (2): 71–75. doi:10.2478/jccm-2019-0012. PMC 6534940. PMID 31161145.
- ^ Lissens, Willy; De Meirleir, Linda; Seneca, Sara; Liebaers, Inge; Brown, Garry K.; Brown, Ruth M.; Ito, Michinori; Naito, Etsuo; Kuroda, Yasuhiro; Kerr, Douglas S.; Wexler, Isaiah D. (March 2000). <209::aid-humu1>3.0.co;2-k "Mutations in the X-linked pyruvate dehydrogenase (E1) ? subunit gene (PDHA1) in patients with a pyruvate dehydrogenase complex deficiency". Human Mutation. 15 (3): 209–219. doi:10.1002/(sici)1098-1004(200003)15:3<209::aid-humu1>3.0.co;2-k. ISSN 1059-7794.
- ^ Okajima, K.; Korotchkina, L.G.; Prasad, C.; Rupar, T.; Phillips III, J.A.; Ficicioglu, C.; Hertecant, J.; Patel, M.S.; Kerr, D.S. (April 2008). "Mutations of the E1β subunit gene (PDHB) in four families with pyruvate dehydrogenase deficiency". Molecular Genetics and Metabolism. 93 (4): 371–380. doi:10.1016/j.ymgme.2007.10.135. ISSN 1096-7192. PMID 18164639.
- ^ Head, Rosemary A.; Brown, Ruth M.; Zolkipli, Zarazuela; Shahdadpuri, Raveen; King, Mary D.; Clayton, Peter T.; Brown, Garry K. (2005-07-27). "Clinical and genetic spectrum of pyruvate dehydrogenase deficiency: Dihydrolipoamide acetyltransferase (E2) deficiency". Annals of Neurology. 58 (2): 234–241. doi:10.1002/ana.20550. ISSN 0364-5134. PMID 16049940. S2CID 38264402.
- ^ Pavlu-Pereira, Hana; Silva, Maria João; Florindo, Cristina; Sequeira, Sílvia; Ferreira, Ana Cristina; Duarte, Sofia; Rodrigues, Ana Luísa; Janeiro, Patrícia; Oliveira, Anabela (2020-05-29). "Pyruvate dehydrogenase complex deficiency: updating the clinical, metabolic and mutational landscapes". doi:10.21203/rs.3.rs-31232/v1.
{{cite journal}}: Cite 저널은 필요로 한다.journal=(도움말) - ^ Heo, Hye Jin; Kim, Hyoung Kyu; Youm, Jae Boum; Cho, Sung Woo; Song, In-Sung; Lee, Sun Young; Ko, Tae Hee; Kim, Nari; Ko, Kyung Soo; Rhee, Byoung Doo; Han, Jin (August 2016). "Mitochondrial pyruvate dehydrogenase phosphatase 1 regulates the early differentiation of cardiomyocytes from mouse embryonic stem cells". Experimental & Molecular Medicine. 48 (8): e254. doi:10.1038/emm.2016.70. ISSN 2092-6413. PMC 5007642. PMID 27538372.
외부 링크
| 위키미디어 커먼스는 피루베이트 탈수소효소 복합체와 관련된 미디어를 보유하고 있다. |
- https://web.archive.org/web/20070405211049/http://www.dentistry.leeds.ac.uk/biochem/MBWeb/mb1/part2/krebs.htm#animat1 - 리즈 대학교 PDC(오른쪽 위 링크)의 일반 메커니즘 애니메이션
- 피루바테+미국 국립 의학 도서관의 탈수소효소+복합체(MesH)
3D 구조
- 조우, H., 매카시, B, 오코너, M., 리드, J.;Stoops K(12월 2001년)."는 진핵 피루브산 탈수소 효소 단지의 놀라운 및 기능적 구조적 조직".미국 국립 과학 아카데미의 아메리카 미국의 회보. 98(26):14802–14807.Bibcode:2001PNAS...9814802Z.doi:10.1073/pnas.011597698.ISSN 0027-8424.PMC64939.PMID 11752427., 소의 신장 피루브산 탈수소 효소 단지.
- Yu는 X, Hiromasa, Y, Tsen, H.;Stoops, K, 로시, E;조우 H.(1월 2008년)."휴먼 Pyruvate Dehydrogenase 단지 Cores 구조물의:고도로 Conserved Catalytic 센터 가변형 N-Terminal Domains과".구조체입니다. 16(1):104–114. doi:10.1016/j.str.2007.10.024.ISSN 0969-2126.PMC 4807695.PMID 18184588., 인간 full-length와 주 도메인 콘트롤러의들의 길이를 줄였다 E2(tE2)중심부인 대장 균으로 표현된입니다.