공생
Symbiogenesis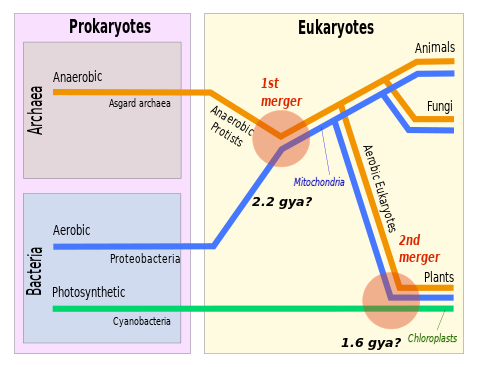
공생(公生)은[2] 원핵 생물로부터 진핵 세포의 기원에 대한 선도적인 진화 이론입니다.[3]이 이론은 미토콘드리아, 엽록체와 같은 플라스티드, 그리고 아마도 진핵 세포의 다른 세포소기관들은 이전에 자유롭게 살아있는 원핵생물들(고균보다 박테리아와 더 밀접한 관련이 있는)의 후손이라고 주장합니다.미토콘드리아는 계통발생학적으로 Rickettsiales 박테리아와 관련이 있는 것으로 보이는 반면 엽록체는 청록균과 관련이 있는 것으로 생각됩니다.
엽록체가 원래 독립적인 유기체였고 다른 단세포 유기체와 공생 관계로 융합되었다는 생각은 안드레아스 심퍼와 같은 연구자들에 의해 지지를 받았던 19세기로 거슬러 올라갑니다.내생균 이론은 1905년과 1910년에 러시아 식물학자 콘스탄틴 메레슈코프스키에 의해 기술되었고 1967년 린 마굴리스에 의해 미생물학적 증거로 발전되고 입증되었습니다.
공생을 뒷받침하는 많은 증거들 중에는 새로운 미토콘드리아와 플라스티즈가 두개로 쪼개져서만 형성된다는 것과 그렇지 않으면 세포들이 새로운 세포를 만들어낼 수 없다는 것이 있습니다; 포린이라고 불리는 운반 단백질이 미토콘드리아의 바깥쪽 막에서 발견된다는 것, 엽록체,그리고 박테리아 세포막; 카디올리핀이 단지 내부 미토콘드리아 막과 박테리아 세포막에서만 발견되는 것; 그리고 일부 미토콘드리아와 플라스티드가 박테리아의 원형 염색체와 유사한 단일 원형 DNA 분자를 포함하는 것.
역사
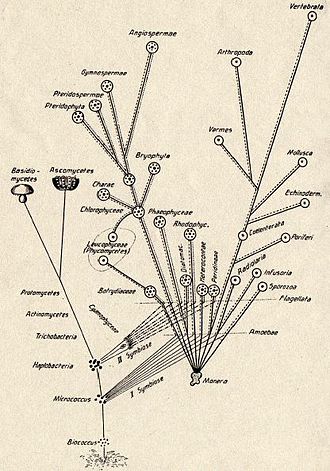
러시아의 식물학자 콘스탄틴 메레슈코프스키는 1905년 저서 "식물 왕국에서 발색단의 본질과 기원"에서 공생성 이론(그리스어: σύν 합성어 "함께", β ίος 바이오스 "생명체", γένεσις 발생 "기원, 탄생"에서)의 개요를 처음 설명한 다음 1910년 저서 "공생성의 기초로서 두 개의 플라스마 이론"에서 자세히 설명했습니다. 생물의 기원에 [5][6][7]관한 새로운 연구메레슈코프스키는 식물학자 안드레아스 심퍼의 업적을 알고 있었습니다.1883년, 심퍼는 녹색 식물에서 엽록체의 구분이 자유롭게 사는 시아노박테리아의 그것과 매우 비슷하다는 것을 관찰했습니다.그는 녹색 식물이 두 유기체의 공생 결합에서 비롯되었다는 것을 잠정적으로 제안했습니다.[8]1918년에 프랑스 과학자 Paul Jules Portier는 Les Symbiotes를 출판했는데, 그는 미토콘드리아가 공생 과정에서 비롯되었다고 주장했습니다.[9][10]이반 월린은 1920년대에 미토콘드리아의 내공생 기원에 대한 아이디어를 옹호했습니다.[11][12]러시아의 식물학자인 보리스 고조-폴리안스키는 다윈의 진화론의 관점에서 이 이론을 설명한 최초의 사람이 되었습니다.[13]그의 1924년 저서 생물학의 새로운 원리에서. 그는 [14]공생이론에 대한 에세이에서 "공생이론은 공생현상에 의존한 선택이론입니다."[15]
이 이론들은 1961년과 1962년의 한스 리스와 같이 시아노박테리아와 엽록체 사이의 보다 상세한 전자현미경 비교가 이루어질 때까지 설득력을 얻지 못했습니다.[16][17]이것들은 플라스티딘과 미토콘드리아가 그들 자신의 DNA를 포함하고 있다는 발견과 함께 1960년대 공생에 대한 생각의 부활로 이끌었습니다.[18]린 마굴리스(Lynn Margulis)는 유사분열 세포의 기원에 관한 1967년 논문에서 미생물학적 증거로 이론을 발전시키고 입증했습니다.[19]그녀의 1981년 연구인 세포 진화에서의 공생(Symbiosis in Cell Evolution)에서 그녀는 진핵 세포가 진핵 편모와 섬모로 발전한 내공생 스피로케아를 포함한 상호작용하는 개체들의 공동체로 시작되었다고 주장했습니다.편모는 DNA가 부족하고 박테리아나 고균과 초구조적 유사성을 보이지 않기 때문에 이 마지막 아이디어는 많은 수용을 받지 못했습니다.편모와 원핵세포골격의 진화).Margulis와 Dorion Sagan에 따르면,[20] "삶은 전투에 의해 지구를 점령한 것이 아니라, 네트워킹에 의해" (즉, 협력에 의해).Christian de Duve는 퍼옥시좀이 지구 대기에서 증가하는 양의 자유 분자 산소를 세포가 견딜 수 있도록 하는 최초의 내생성체였을지도 모른다고 제안했습니다.하지만, 현재 퍼옥시좀은 공생 기원을 가지고 있다는 생각과 모순되어, 드 노보(de novo)로 형성될 수도 있는 것으로 보입니다.[21]미토콘드리아와 엽록체의 기원으로서 공생에 대한 근본적인 이론은 현재 널리 받아들여지고 있습니다.[3]
내상징에서 소기관까지
생물학자들은 대개 게놈 크기를 줄여 소기관을 다른 유기체 안에 사는 전체 유기체인 내생체와 구별합니다.[23]내생동체가 세포 소기관으로 진화하면서, 그 유전자의 대부분은 숙주 세포 유전체로 옮겨집니다.[24]따라서 숙주 세포와 세포소기관은 세포소기관에 의해 필요하지만 지금은 세포에 의해 제조된 단백질 생산물의 반송을 가능하게 하는 운반 메커니즘을 개발할 필요가 있습니다.[25]
자유로운 조상
알파프로테오박테리아는 이전에는 미토콘드리아와 가장 밀접한 관련이 있는 자유 생명체로 여겨졌습니다.[25]이후의 연구는 미토콘드리아가 특히 SAR11 분류군에 속하는 펠라지박테랄레스 박테리아와 가장 밀접한 관련이 있다는 것을 나타냅니다.[26][27]
질소 고정 필라멘트 시아노박테리아는 플라스틱과 가장 밀접한 관련이 있는 자유 생명체입니다.[25][28][29]
시아노박테리아와 알파프로테오박테리아 모두 수천 개의 단백질을 암호화하는 큰 (>6 Mb) 게놈을 유지합니다.[25]플라스틱과 미토콘드리아는 세균의 동족과 비교했을 때 게놈 크기가 극적으로 감소했습니다.[25]광합성 생물의 엽록체 유전체는 보통 20-200 kb[25] 단백질을 암호화하는 120-200[30] kb이고 인간의 미토콘드리아 유전체는 약 16 kb이고 37개의 유전자를 암호화하는데 그 중 13개가 단백질입니다.[31]그러나 민물 아메보이드의 예를 사용하여, 시아노박테리아로부터 진화된 것으로 발견된 크로마토포어를 포함하는 파울리넬라 크로마토포라는 이것이 유일하게 가능한 기준이 아니라고 주장합니다; 다른 하나는 숙주 세포가 이전의 내생성 분열의 조절을 통제하는 것을 가정했다는 것입니다.따라서 세포 자체의 분열과 동기화 할 수 있습니다.[23]Nowack과 그녀의 동료 유전자는 발색단(1.02 Mb)의 염기서열을 분석하여 867개의 단백질만이 광합성 세포에 의해 암호화된다는 것을 발견했습니다.그들의 가장 가까운 자유로운 살아있는 시아노박테리아인 Synechococcus (유전체 크기 3 Mb, 유전자 3300개를 가진)와 비교한 결과, 크로마토포어가 급격한 유전체 수축을 겪었다는 것이 밝혀졌습니다.발색단은 광합성에 책임이 있지만 다른 생합성 기능을 수행할 수 있는 유전자가 부족한 유전자를 포함하고 있습니다. 이러한 관찰은 이러한 내생성 세포가 생존과 성장 메커니즘을 위해 숙주에 크게 의존하고 있음을 시사합니다.따라서 이러한 발색단은 미토콘드리아 및 플라스티즈와 비교했을 때 소기관 특이적인 목적으로 기능하지 않는 것으로 확인되었습니다.이 구분은 광합성 소기관의 초기 진화를 촉진시킬 수 있었을 것입니다.[32]
유전자 자율성의 상실, 즉 내생성으로부터 많은 유전자의 상실은 진화적 시기에 매우 일찍 일어났습니다.[33]원래의 내생성 게놈 전체를 고려하면, 진화의 시간에 걸쳐 유전자에 대해 가능한 세 가지 주요 운명이 있습니다.첫 번째는 기능적으로 중복된 유전자의 손실로,[33] 핵에 이미 존재하는 유전자가 결국 손실됩니다.두 번째는 핵으로 유전자가 옮겨지는 것이고, 세 번째는 한때 유기체였던 세포 소기관에 유전자가 남아있다는 것입니다.[25][33][34][35][36]자율성의 상실과 숙주와의 결합의 통합은 주로 핵 유전자 전이에 기인할 수 있습니다.[36]세포소기관 유전체가 진화의 시간에 걸쳐 크게 감소함에 따라, 핵 유전자는 확장되고 더 복잡해졌습니다.[25]결과적으로, 많은 플라스티드 및 미토콘드리아 과정은 핵 암호화된 유전자 생성물에 의해 구동됩니다.[25]게다가, 내생동체에서 유래한 많은 핵 유전자들은 그들의 소기관과 관계없는 새로운 기능들을 습득해왔습니다.[25][36]
유전자 전달 메커니즘
유전자 전달의 메커니즘은 완전히 알려져 있지 않지만, 이 현상을 설명하기 위한 여러 가설들이 존재합니다.가능한 메커니즘에는 보완 DNA(cDNA) 가설과 대량 흐름 가설이 포함됩니다.[25][34]
cDNA 가설은 유전자를 세포소기관에서 핵으로 운반하기 위해 메신저 RNA (mRNA)를 사용하는 것을 포함하며, 여기서 유전자는 cDNA로 전환되어 게놈에 통합됩니다.[25][34]cDNA 가설은 꽃이 피는 식물의 게놈에 대한 연구를 기반으로 합니다.미토콘드리아의 단백질 코딩 RNA는 소기관 특이적인 스플라이스 및 편집 부위를 사용하여 접합되고 편집됩니다.그러나 일부 미토콘드리아 유전자의 핵 사본은 세포 소기관 특이적인 스플라이스 부위를 포함하지 않으며, 이는 가공된 mRNA 중간체를 시사합니다.편집된 미토콘드리아 cDNA가 핵 유전체와 재조합할 가능성이 낮고 그들의 고유 미토콘드리아 유전체와 재조합할 가능성이 더 높기 때문에 cDNA 가설은 이후 수정되었습니다.만약 편집된 미토콘드리아 서열이 미토콘드리아 유전체와 재결합한다면, 미토콘드리아 스플라이스 부위는 미토콘드리아 유전체에 더 이상 존재하지 않을 것입니다.따라서 후속적인 핵 유전자 전달은 미토콘드리아 스플라이스 부위가 부족할 것입니다.[25]
대량 흐름 가설은 mRNA가 아닌 탈출 DNA가 유전자 전달의 메커니즘이라는 cDNA 가설의 대안입니다.[25][34]이 가설에 따르면, 자가포식(정상적인 세포 파괴), 유전자 형성(유전자의 형성), 세포 스트레스를 포함한 소기관의 교란은 핵으로 수입되어 비동위 말단 접합(이중 좌초된 분열의 수리)을 사용하여 핵 DNA에 통합된 DNA를 방출합니다.[34]예를 들어, 주요 유전자 전달의 부족으로 인해, 내생체의 초기 단계에서, 숙주 세포는 내생체에 대한 통제력이 거의 없었습니다.내생성체는 숙주 세포와 독립적으로 세포 분열을 거쳤고, 이로 인해 숙주 세포 내에서 내생성체의 많은 "복사"가 이루어졌습니다.일부 내상징은 용해되었고(터짐), 높은 수준의 DNA가 핵에 포함되었습니다.비슷한 메커니즘이 높은 유전자 전달률을 보여주고 세포가 다수의 엽록체를 포함하는 담배 식물에서 발생한다고 생각됩니다.[33]또한, 다발성 유동 가설은 소기관 유전자의 비임의 클러스터의 존재에 의해서도 지지되며, 이는 여러 유전자의 동시적인 움직임을 시사합니다.[34]
포드 둘리틀은 (어떤 메커니즘이든) 유전자 전달이 래칫처럼 작용하여 세포 소기관에서 핵 게놈으로 유전자가 단방향으로 전달된다고 제안했습니다.[37]세포소기관의 유전적 물질이 핵 게놈에 포함될 때, 유전자의 세포소기관 또는 핵 사본은 집단으로부터 손실될 수 있습니다.만약 세포소기관 복제가 소실되고 이것이 고쳐지거나 유전자 이동을 통해 소실된다면, 유전자는 성공적으로 핵으로 옮겨집니다.만약 핵 사본을 잃어버리면, 수평적인 유전자 전달이 다시 일어날 수 있고, 세포는 성공적으로 유전자를 핵으로 전달하기 위해 '다시 시도'할 수 있습니다.[37]이러한 래칫과 같은 방식으로, 세포 소기관의 유전자는 진화의 시간이 지남에 따라 핵 게놈에 축적될 것으로 예상됩니다.[37]
원발성 연골어류의 내공생증
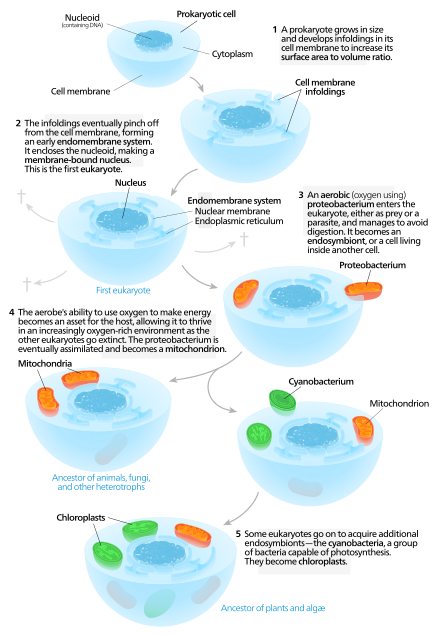
미토콘드리아의 기원에 대한 내생생물학 이론은 원핵생물이 원핵생물을 삼켰고, 이 내생생물은 진핵생물의 생성인 진핵생물 생성의 주요 단계인 세포소기관이 되었음을 시사합니다.[38]
미토콘드리아

미토콘드리아는 탄소 기반의 거대 분자를 대사시킴으로써 세포에 에너지를 전달하는 분자인 ATP를 합성하는 소기관입니다.[39]미토콘드리아에 있는 DNA와 mtDNA로부터 유래된 단백질의 존재는 이 소기관이 원핵생물로 통합되기 전에 원핵생물이었을지도 모른다는 것을 암시합니다.[40]미토콘드리아와 숙주세포가 유전체의 일부를 공유하고 동시에 분열을 겪으며 서로 에너지를 생산할 수 있는 수단을 제공하기 때문에 미토콘드리아는 내공생이 아닌 소기관으로 여겨집니다.[40]내막계와 핵막은 원핵세포에서 유래한 것으로 가정되었습니다.[41][42][43]
핵막
핵의 존재는 진핵생물과 원핵생물 사이의 주요한 차이점 중 하나입니다.[44]진핵생물과 원핵생물 사이에 보존된 일부 핵 단백질들은 이 두 종류가 공통적인 조상을 가지고 있음을 암시합니다.[45]핵생성 뒤에 있는 또 다른 이론은 초기 핵막 단백질이 세포막을 접게 하고 핵 외피와 같은 기공을 가진 구체를 형성하게 했다는 것입니다.[46]핵막을 형성하는 방법으로, 내생체는 세포가 목적을 위해 세포막을 접는 대사 과정을 개발하는 것보다 에너지를 덜 사용할 것으로 예상될 수 있습니다.[42]에너지를 생산하는 미토콘드리아 없이 삼킨 세포를 소화하는 것은 숙주 세포에게 어려운 일이었을 것입니다.[41]이 관점에서, 원형질 콘드리아를 떠나는 막 결합 기포 또는 소포가 핵 외피를 형성했을 수 있습니다.[41]
초기 진핵 세포가 미토콘드리아 원생체를 통합하는 공생 과정에는 활성산소 종의 방출로부터 고고 숙주 유전체를 보호하는 것이 포함되었을 가능성이 있습니다.이들은 원형 미토콘드리아에 의한 산화적 인산화와 ATP 생성 과정에서 형성되었을 것입니다.핵막은 활성 산소종에 의해 야기되는 핵 유전체 DNA 손상으로부터 보호하기 위한 적응적 혁신으로 진화했을 수 있습니다.[47]조상의 미토콘드리아 유전체에서 핵 유전체로의 유전자의 상당한 전이는 초기 진핵생물 진화 동안에 발생했을 가능성이 있습니다.[48]핵막에 의해 제공되는 활성 산소종에 대한 핵 게놈의 더 큰 보호는 이러한 유전자 전달의 적응적 이점을 설명할 수 있습니다.
내막계
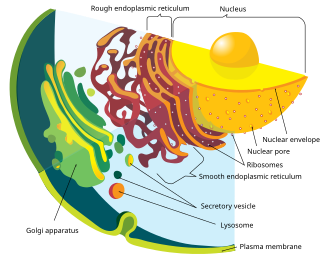
현대의 진핵세포는 세포 내부, 내부 및 외부로 생성물과 폐기물을 운반하기 위해 내막 시스템을 사용합니다.핵포락막과 내막 소포는 유사한 막 단백질로 구성되어 있습니다.[49]이 소포들은 또한 그들이 발생했거나 이동하고 있는 세포 소기관과 비슷한 막 단백질을 공유합니다.[50]이것은 핵막을 형성한 것이 또한 내막 체계를 형성했음을 암시합니다.원핵생물은 진핵생물처럼 복잡한 내부 막 네트워크를 가지고 있지 않지만, 그들은 그들의 외부 막으로부터 세포 외 소포를 생산할 수 있습니다.[41]초기 원핵생물이 원핵생물에 의해 섭취된 후, 원핵생물은 세포 내에 축적된 소포를 계속해서 생산했을 것입니다.[41]소포의 내부 구성 요소들의 상호작용은 소포체와 골지 장치로 이어졌을 수 있으며, 둘 다 내막 시스템의 일부입니다.[41]
세포질
2000년경 로페스 가르시아와 모레이라에 의해 제안된 합성영양학 가설은 진핵생물이 고세균, 발효델타프로테오박테리움, 미토콘드리아가 된 메타노트로피 알파프로테오박테리움의 대사 능력을 결합함으로써 생겨났다고 제시했습니다.2020년에, 같은 팀은 황을 산화시키는 델타프로테오박테리움으로 수소를 생산하는 아스가르드 고생물을 포함하기 위해 합성법 제안서를 업데이트했습니다.세 번째 유기체인 알파 프로테오박테리움은 에어로빅과 혐기성 호흡을 할 수 있고, 미토콘드리아로 발달된 황을 산화시킬 수 있습니다. 또한 광합성도 할 수 있었을 것입니다.[51]
오르간성게놈
플라스톰과 미토게놈

일부 내결합 유전자는 소기관에 남아있습니다.플라스티드와 미토콘드리아는 rRNA, tRNA, 산화환원 반응에 관여하는 단백질, 전사, 번역, 복제에 필요한 단백질을 암호화하는 유전자를 보유하고 있습니다.소기관이 왜 그들의 게놈의 작은 부분을 유지하는지를 설명하기 위한 많은 가설들이 있습니다; 그러나 어떤 가설도 모든 유기체에 적용되지 않을 것이고, 그 주제는 여전히 꽤 논란이 많습니다.소수성 가설은 고도의 소수성(물을 싫어하는) 단백질(예를 들어 산화환원 반응에 관여하는 막 결합 단백질)이 세포질을 통해 쉽게 운반되지 않으며 따라서 이러한 단백질은 각각의 소기관에서 암호화되어야 한다는 것입니다.코드 차이 가설은 전달의 한계가 소기관과 핵 사이의 다른 유전 코드와 RNA 편집 때문이라고 말합니다.레독스 조절 가설은 레독스 반응 단백질을 암호화하는 유전자들이 이러한 단백질의 복구와 합성의 필요성을 효과적으로 결합하기 위해 유지된다는 것을 말합니다.예를 들어, 플라스틱에서 광계 중 하나가 손실될 경우 중간 전자 캐리어가 너무 많은 전자를 잃거나 얻을 수 있으므로 광계 수리의 필요성을 알 수 있습니다.세포질 단백질을 세포 소기관으로 운반하고 핵에 신호를 보내는 데 관련된 시간 지연은 손상된 활성 산소 종을 생성합니다.마지막 가설은 막 단백질의 조립, 특히 산화환원 반응에 관련된 것들은 조절된 합성과 소단위의 조립을 필요로 한다는 것입니다. 그러나 번역과 단백질 전달 조절은 세포질에서 조절하기 더 어렵습니다.[25][30][33][52]
비광합성 플라스티디지놈
미토콘드리아와 플라스티드의 유전자 대부분은 광합성(플라스티드) 또는 세포 호흡(미토콘드리아)에 관여하는 단백질을 암호화하는 유전자의 발현(전사, 번역, 복제)과 관련이 있습니다.광합성이나 세포 호흡의 손실이 각각 플라스틱 게놈이나 미토콘드리아 게놈의 완전한 손실을 가져올 것이라고 예측할 수도 있습니다.[25][30][33]미토콘드리아 후손들(미토솜과 수소오솜)의 많은 예들이 그들의 전체 오르간성 유전체를 잃었지만,[50] 비광합성 플라스티드는 작은 유전체를 보유하는 경향이 있습니다.이러한 현상을 설명하는 가설은 크게 두 가지입니다.[33][53]
본질적인 tRNA 가설은 RNA 제품(tRNA 및 rRNA)을 암호화하는 유전자의 기능적 플라스티-핵 유전자 전달이 문서화되지 않았다는 것에 주목합니다.결과적으로, 플라스티드는 그들 자신의 기능적인 RNA를 만들거나 핵 대응물을 수입해야 합니다.그러나 tRNA-Glu와 tRNA-fmet를 암호화하는 유전자는 필수불가결한 것으로 보입니다.플라스티드는 전구체 분자로서 플라스티드 인코딩된 tRNA-Glu(유전자 trnE로부터)를 필요로 하는 혈액 생합성을 담당합니다.RNA를 암호화하는 다른 유전자들처럼, trnE는 핵으로 전이될 수 없습니다.또한, trnE가 고도로 보존됨에 따라 세포질 tRNA-Glu로 대체될 가능성은 희박합니다. trnE의 단일 염기 변화는 혈액 합성의 손실을 초래했습니다.tRNA-포르밀메티오닌(tRNA-fmet)에 대한 유전자는 또한 플라스티드 유전체에 암호화되어 있으며 플라스티드와 미토콘드리아 모두에서 번역 개시를 위해 필요합니다.미토콘드리아가 단백질을 번역하는 한 tRNA-fmet 유전자를 계속해서 발현시키기 위해서 플라스티드는 필요합니다.[33]
제한된 창 가설은 비광합성 플라스티즈에서 유전자의 보유에 대한 보다 일반적인 설명을 제공합니다.[53]이 가설에 따르면, 유전자는 소기관의 교란에 따라 핵으로 옮겨집니다.[34]교란은 내공생의 초기 단계에서 흔했지만, 숙주 세포가 세포 소기관 분열을 통제하게 되면, 진핵생물은 세포 당 하나의 플라스미드만을 갖도록 진화할 수 있습니다.단 하나의 플라스티드만 있으면 세포사멸을 초래할 가능성이 있기 때문에 유전자 전달이[33] 심각하게 제한됩니다.[33][53]이 가설과 일치하여, 여러 개의 플라스티를 가진 유기체는 단일 플라스티를 가진 유기체에 비해 플라스티-핵 유전자 전달이 80배 증가했습니다.[53]
증거
미토콘드리아와 엽록체를 포함한 플라스틱이 박테리아로부터 발생했다는 많은 증거들이 있습니다.[54][55][56][57][58]
- 새로운 미토콘드리아와 플라스티드는 박테리아와 고균이 사용하는 세포 분열의 형태인 이항 핵분열을 통해서만 형성됩니다.[59]
- 만약 세포의 미토콘드리아나 엽록체가 제거된다면, 그 세포는 새로운 세포를 만들 수 있는 수단을 가지고 있지 않습니다.[60]유글레나(Euglena)와 같은 일부 조류에서는 세포에 다른 영향을 주지 않고 특정 화학물질이나 빛이 오래 없는 경우 플라스틱이 파괴될 수 있습니다. 플라스틱은 재생되지 않습니다.
- 포린이라고 불리는 수송 단백질은 미토콘드리아와 엽록체의 외막에서 발견되며 세균 세포막에서도 발견됩니다.[61][62][63]
- 막 지질 카르디올리핀은 내부 미토콘드리아 막과 세균 세포 막에서만 발견됩니다.[64]
- 일부 미토콘드리아와 일부 플라스티딘은 크기와 구조 모두에서 박테리아의 DNA와 비슷한 단일 원형 DNA 분자를 포함합니다.[65]
- 유전체 비교는 미토콘드리아와 알파프로테오박테리아 사이의 밀접한 관계를 시사합니다.[66]
- 유전체 비교는 플라스틱과 시아노박테리아 사이에 밀접한 관계가 있음을 시사합니다.[67]
- 미토콘드리아와 엽록체의 게놈에 있는 많은 유전자들이 숙주 세포의 핵으로 사라지거나 옮겨졌습니다.결과적으로, 많은 진핵생물의 염색체는 미토콘드리아와 플라스티딘의 게놈에서 유래한 유전자를 포함하고 있습니다.[65]
- 미토콘드리아와 플라스티드는 그들 자신의 리보솜을 포함합니다; 이것들은 진핵생물의 것보다 박테리아의 것(70S)과 더 유사합니다.[68]
- 미토콘드리아와 엽록체에 의해 만들어진 단백질은 N-포르밀메티오닌을 개시 아미노산으로 사용하고, 박테리아에 의해 만들어진 단백질은 진핵세포 핵 유전자나 고세균에 의해 만들어진 단백질은 사용하지 않습니다.[69][70]
이차성내공생증
일차적인 내생성은 또 다른 자유 생명체에 의해 세포를 집어삼키는 것을 포함합니다.이차성 내공생증은 일차성 내공생증의 산물 자체가 또 다른 살아있는 진핵생물에 의해 삼켜지고 유지될 때 발생합니다.이차적인 내생성은 여러 번 발생했고 매우 다양한 그룹의 조류와 다른 진핵생물을 발생시켰습니다.일부 유기체들은 비슷한 과정을 이용할 수 있는데, 조류를 집어삼키고 광합성의 산물을 사용하지만, 일단 먹이 아이템이 죽거나 없어지면 숙주는 자유로운 살아있는 상태로 돌아갑니다.의무적인 2차 내생동체는 그들의 소기관에 의존하게 되고, 그들이 없을 때는 살아남을 수 없습니다.조상의 적조류와 이종영양 진핵생물을 포함한 2차적인 내생합성 사건은 Cryptophyta, Haptophyta, Stramenopiles (또는 Heterokontophyta), 그리고 폐포라타를 포함한 몇몇 다른 광합성 계통의 진화와 다양화를 초래했습니다.[71]
이형영양원생동물인 하테나(Hatena)에서 가능한 2차 내생체가 관찰되었습니다.이 유기체는 편모와 세포골격을 잃지만 계속 공생하는 녹조를 섭취할 때까지 포식자처럼 행동합니다.한편, 이제 숙주가 된 하테나는 광합성 영양으로 전환하고, 빛을 향해 나아가는 능력을 얻고, 먹이를 주는 장치를 잃어버립니다.[72]
플라스티드를 포함하는 유기체의 다양성에도 불구하고, 플라스티드 RNA와 단백질의 형태학, 생화학,[73][74] 게놈 조직, 분자 계통발생은 모든 현존하는 플라스티드의 단일 기원을 시사합니다.
페디큘러스 휴머너스(Pediculus humanus, 이)를 포함한 몇몇 종들은 미토콘드리아에 여러 개의 염색체를 가지고 있습니다.이것과 미토콘드리아 내에 암호화되어 있는 유전자의 계통발생학은 미토콘드리아가 여러 개의 조상을 가지고 있고, 이것들이 단지 한 번이 아니라 여러 번의 내생체에 의해 획득되었다는 것과, 원래의 미토콘드리아 염색체에 유전자들의 광범위한 합병과 재배열이 있었다는 것을 암시합니다.[75]
날짜.
언제 원핵생물에서 진핵생물 형태로의 전이가 일어났고 언제 지구상에 첫 번째 왕관 그룹 진핵생물이 나타났는지에 대한 문제는 아직 해결되지 않았습니다.진핵생물에 긍정적으로 할당될 수 있는 가장 오래된 알려진 신체 화석은 인도의 1.631 Gya Deonar 층에서 발견된 초형화된 무정형의 무정형화석입니다.[76]이 화석들은 미토콘드리아에 의해 유지되는 정교한 형태학 생성 세포골격을 가진 핵 이후 진핵생물로 여전히 확인될 수 있습니다.[77]이 화석 증거는 알파단백질 박테리아의 내생균 획득이 1.6 Gya 이전에 일어났음을 나타냅니다.분자 시계는 또한 마지막 진핵 공통 조상을 추정하는 데 사용되었지만, 이 방법들은 큰 고유의 불확실성을 가지고 있고 광범위한 날짜를 제공합니다.합리적인 결과는 c. 1.8 Gya의 추정치를 포함합니다.[78] 2.3 Gya 추정치[79] 또한 합리적인 것으로 보이며, 지구 역사상 가장 뚜렷한 생물 지구화학적 섭동 중 하나인 초기 고생대대대 산소화 사건과 일치한다는 추가적인 매력을 가지고 있습니다.그 당시 대기 중 산소 농도의 현저한 증가는 산소를 해독하는 미토콘드리아의 진화를 유도하는 진핵 생성의 기여 원인으로 제시되어 왔습니다.[80]또는, Great Oxidation Event는 진핵생물의 결과일 수 있고, 유기 탄소의 수출과 매장에 미치는 영향일 수 있습니다.[81]
참고 항목
- 의무적인 박테리아 공생을 가지고 있는 원생동물인 Angomonas deanei.
- 하테나 아레니콜라, 내생성을 획득하는 과정에 있는 것으로 보이는 종
- 수소 가설, 미토콘드리아가 수소 의존성 고균에 의해 획득되었다는 이론, 이들의 내결합은 기능성 혐기성 박테리아입니다.
- 도벽성형술, 섭취한 해조류에서 플라스티디를 분리하는 것
- Mixotricha paradoxa, 그 자체가 공생체이며, 수많은 내생체 박테리아를 포함하고 있습니다.
- 기생충 이브, 내생충에 관한 소설
- 의무적인 박테리아 공생을 가지고 있는 또 다른 원생동물인 Strigomonas culicis
- 바이러스 진핵생물, 세포핵이 내생체에서 유래했다는 가설
참고문헌
- ^ Latorre, A.; Durban, A.; Moya, A.; Pereto, J. (2011). "The role of symbiosis in eukaryotic evolution". In Gargaud, M.; López-Garcìa, P.; Martin, H. (eds.). Origins and Evolution of Life: An astrobiological perspective. Cambridge: Cambridge University Press. pp. 326–339. ISBN 978-0-521-76131-4. Archived from the original on 24 March 2019. Retrieved 27 August 2017.
- ^ "Serial endosymbiotic theory (SET)". flax.nzdl.org. Archived from the original on 18 August 2021. Retrieved 8 March 2021.
- ^ a b Cornish-Bowden, Athel (7 December 2017). "Lynn Margulis and the origin of the eukaryotes". Journal of Theoretical Biology. The origin of mitosing cells: 50th anniversary of a classic paper by Lynn Sagan (Margulis). 434: 1. Bibcode:2017JThBi.434....1C. doi:10.1016/j.jtbi.2017.09.027. PMID 28992902.
- ^ "Mereschkowsky's Tree of Life". Scientific American. Retrieved 1 May 2017.
- ^ Mereschkowski, Konstantin (15 September 1905). "Über Natur und Ursprung der Chromatophoren im Pflanzenreiche" [On the nature and origin of chromatophores in the plant kingdom]. Biologisches Centralblatt (in German). 25 (18): 593–604.
- ^ 참조:
- Mereschkowski, Konstantin (15 April 1910). "Theorie der zwei Plasmaarten als Grundlage der Symbiogenesis, einer neuen Lehre von der Entstehung der Organismen" [Theory of two types of plasms as the basis of symbiogenesis, a new study of the origin of organisms [part 1 of 4]]. Biologisches Centralblatt (in German). 30 (8): 278–288.
- Mereschkowsky, Konstantin (1 May 1910). "Theorie der zwei Plasmaarten als Grundlage der Symbogenesis, einer neuen Lehre von der Entstehung der Organismen" [Theory of two types of plasms as the basis of symbiogenesis, a new study of the origin of organisms [part 2 of 4]]. Biologisches Centralblatt (in German). 30 (9): 289–303.
- Mereschkowski, Konstantin (15 May 1910). "Theorie der zwei Plasmaarten als Grundlage der Symbiogenesis, einer neuen Lehre von der Entstehung der Organismen" [Theory of two types of plasms as the basis of symbiogenesis, a new study of the origin of organisms [part 3 of 4]]. Biologisches Centralblatt (in German). 30 (10): 321–347.
- Mereschkowsky, Konstantin (1 June 1910). "Theorie der zwei Plasmaarten als Grundlage der Symbiogenesis, einer neuen Lehre von der Entstehung der Organismen" [Theory of two types of plasms as the basis of symbiogenesis, a new study of the origin of organisms [part 4 of 4]]. Biologisches Centralblatt (in German). 30 (11): 353–367.
- ^ Martin, William F.; Roettger, Mayo; Kloesges, Thorsten; et al. "Modern endosymbiotic theory: Getting lateral gene transfer into the equation" (PDF). Journal of Endocytobiosis and Cell Research. 23: 1–5.(저널 URL : [1])
- ^ 참조:
- Schimper, A. F. W. (16 February 1883). "Ueber die Entwicklung der Chlorophyllkörner und Farbkörper" [On the development of chlorophyll granules and colored bodies [part 1 of 4]]. Botanische Zeitung (in German). 41 (7): 105–114. 105쪽: "인즈위센 더 일테미르 헤르 교수 슈미트, dass…die höheren Pflanzen sichebenso overhalten würden" (한편, 교수님.Schmitz는 조류 중에서 세포 플라스마에서 엽록소 과립이 생성되는 것이 아니라 분열에 의해 서로에게서만 생성된다고 보고했습니다.포자들은 어미 식물의 엽록소 과립으로부터 받는데, 이 과립들은 분할에 의해 그로부터 발생하는 식물의 엽록소 과립들을 모두 만듭니다.해조류에서 이 발견은 교수님에게 그것을 가능성 있게 만들었습니다.Schmitz는 고등 식물도 마찬가지로 행동할 것입니다.페이지 106부터: "Meine Untersuchungen haben ergeben, … aus dem Scheitelmeristem sich entwickelnden Gewebeerzeugen" (제 조사에 따르면 식물 생장점은 항상 분화된 엽록소체 또는 그 무색의 기초를 포함하고 있으며, 그것들은 세포 플라스마로부터 생성된 것이 아니라 fr.om은 분열에 의해 서로에게, 그리고 그들은 아피칼 메리스템으로부터 발달하는 조직의 모든 엽록소체와 전분을 형성하는 [bodies]을 생성합니다.)112쪽부터 각주 2: "Solte sich definitive bestätigen, …aine Symbiose erinnern." (난자 세포의 플라스티즈가 새로 형성되지 않는다는 것이 확실히 확인되어야 한다면, 이들을 포함하는 유기체와의 관계는 어느 정도 공생을 시사할 것입니다.)
- Schimper, A. F. W. (23 February 1883). "Ueber die Entwicklung der Chlorophyllkörner und Farbkörper" [On the development of chlorophyll granules and colored bodies [part 2 of 4]]. Botanische Zeitung (in German). 41 (8): 121–131.
- Schimper, A. F. W. (2 March 1883). "Ueber die Entwicklung der Chlorophyllkörner und Farbkörper" [On the development of chlorophyll granules and colored bodies [part 3 of 4]]. Botanische Zeitung (in German). 41 (9): 137–146.
- Schimper, A. F. W. (9 March 1883). "Ueber die Entwicklung der Chlorophyllkörner und Farbkörper" [On the development of chlorophyll granules and colored bodies [part 4 of 4]]. Botanische Zeitung (in German). 41 (10): 153–162.
- ^ Portier, Paul (1918). Les Symbiotes (in French). Paris, France: Masson et Cie. p. 293. p. 293: "Cette modification dans les raports desaparils nucleéair et miconrial peuttre résultat deux mecanism.… Cette la parthénogénése." (핵과 미토콘드리아 시스템의 관계의 이러한 수정은 두 가지 메커니즘의 결과일 수 있습니다: (a) 정자와 환원 분열에 의한 새로운 공생의 기여라는 두 가지 요인의 조합이 있습니다.그것이 바로 수정입니다. (b) 단 하나의 요인이 존재합니다: 환원 분열: 이 경우, 난자는 충분히 활성화된 공생체를 포함합니다.그것이 바로 발육 부전입니다.
- ^ Lane, Nick (2005). Power, Sex, Suicide. Mitochondria and the Meaning of Life. New York: Oxford University Press. p. 14. ISBN 9780199205646.
- ^ Wallin, Ivan E. (1923). "The Mitochondria Problem". The American Naturalist. 57 (650): 255–61. doi:10.1086/279919. S2CID 85144224.
- ^ Wallin, Ivan E. (1927). Symbionticism and the origin of species. Baltimore: Williams & Wilkins. p. 117.
- ^ Margulis, Lynn (2011). "Symbiogenesis. A new principle of evolution rediscovery of Boris Mikhaylovich Kozo-Polyansky (1890–1957)". Paleontological Journal. 44 (12): 1525–1539. doi:10.1134/S0031030110120087. S2CID 86279772.
- ^ Kozo-Polyansky, Boris Mikhaylovich (1924). Новый принцип биологии. Очерк теории симбиогенеза [A New Principle of Biology. Essay on the Theory of Symbiogenesis] (in Russian). Moscow and Leningrad (St. Petersburg), Russia: Пучина (Puchina).
- 영어 번역:
- 검토 대상:
- ^ Corning, Peter A. (2010). Holistic Darwinism: Synergy, Cybernetics, and the Bioeconomics of Evolution. Chicago: University of Chicago Press. p. 81. ISBN 978-0-22611-633-4.
- ^ Ris, Hans; Plaut, Walter (June 1962). "Ultrastructure of DNA-containing areas in the chloroplast of Chlamydomonas". The Journal of Cell Biology. 13 (3): 383–91. doi:10.1083/jcb.13.3.383. PMC 2106071. PMID 14492436.
- ^ Ris, Hans; Singh, R. N. (January 1961). "Electron microscope studies on blue-green algae". The Journal of Biophysical and Biochemical Cytology. 9 (1): 63–80. doi:10.1083/jcb.9.1.63. PMC 2224983. PMID 13741827.
- ^ Stocking, C.; Gifford, E. (1959). "Incorporation of thymidine into chloroplasts of Spirogyra". Biochem. Biophys. Res. Commun. 1 (3): 159–64. doi:10.1016/0006-291X(59)90010-5.
- ^ Sagan, Lynn (March 1967). "On the origin of mitosing cells". Journal of Theoretical Biology. 14 (3): 255–74. Bibcode:1967JThBi..14..225S. doi:10.1016/0022-5193(67)90079-3. PMID 11541392.
- ^ Margulis, Lynn; Sagan, Dorion (1997). Microcosmos: Four Billion Years of Microbial Evolution. Berkeley, Los Angeles, London: University of California Press. p. 29. ISBN 0-520-21064-6.
- ^ Gabaldón, Toni; Snel, Berend; Zimmeren, Frank van; et al. (23 March 2006). "Origin and evolution of the peroxisomal proteome". Biology Direct. 1 (1): 8. doi:10.1186/1745-6150-1-8. PMC 1472686. PMID 16556314. (페록시좀의 내생체 기원과 모순되는 증거를 제공하고, 대신 그것들이 소포체에서 진화적으로 기원했음을 암시합니다.)
- ^ Pisani D, Cotton JA, McInerney JO (August 2007). "Supertrees disentangle the chimerical origin of eukaryotic genomes". Molecular Biology and Evolution. 24 (8): 1752–1760. doi:10.1093/molbev/msm095. PMID 17504772.
- ^ a b Keeling, P. J.; Archibald, J. M. (April 2008). "Organelle evolution: what's in a name?". Current Biology. 18 (8): R345-7. doi:10.1016/j.cub.2008.02.065. PMID 18430636. S2CID 11520942.
- ^ Syvanen, Michael; Kado, Clarence I. (30 January 2002). Horizontal Gene Transfer. Academic Press. p. 405. ISBN 978-0126801262.
- ^ a b c d e f g h i j k l m n o p Timmis, Jeremy N.; Ayliffe, Michael A.; Huang, Chun Y.; Martin, William (2004). "Endosymbiotic gene transfer: organelle genomes forge eukaryotic chromosomes". Nature Reviews Genetics. 5 (2): 123–135. doi:10.1038/nrg1271. PMID 14735123. S2CID 2385111.
- ^ "Mitochondria Share an Ancestor With SAR11, a Globally Significant Marine Microbe". ScienceDaily. July 25, 2011. Retrieved 26 July 2011.
- ^ Thrash, J. Cameron; Boyd, Alex; Huggett, Megan J.; et al. (14 June 2011). "Phylogenomic evidence for a common ancestor of mitochondria and the SAR11 clade". Scientific Reports. 1 (1): 13. Bibcode:2011NatSR...1E..13T. doi:10.1038/srep00013. PMC 3216501. PMID 22355532.
- ^ Deusch, O.; Landan, G.; Roettger, M.; et al. (14 February 2008). "Genes of Cyanobacterial Origin in Plant Nuclear Genomes Point to a Heterocyst-Forming Plastid Ancestor". Molecular Biology and Evolution. 25 (4): 748–761. doi:10.1093/molbev/msn022. PMID 18222943.
- ^ Ochoa de Alda, Jesús A. G.; Esteban, Rocío; Diago, María Luz; et al. (15 September 2014). "The plastid ancestor originated among one of the major cyanobacterial lineages". Nature Communications. 5 (1): 4937. Bibcode:2014NatCo...5.4937O. doi:10.1038/ncomms5937. PMID 25222494.
- ^ a b c Lila Koumandou, V.; Nisbet, R. Ellen R.; Barbrook, Adrian C.; et al. (May 2004). "Dinoflagellate chloroplasts—where have all the genes gone?". Trends in Genetics. 20 (5): 261–267. doi:10.1016/j.tig.2004.03.008. PMID 15109781.
- ^ Taanman, J. W. (February 1999). "The mitochondrial genome: structure, transcription, translation and replication". Biochimica et Biophysica Acta (BBA) - Bioenergetics. 1410 (2): 103–23. doi:10.1016/S0005-2728(98)00161-3. PMID 10076021.
- ^ Nowack, E. C.; Melkonian, M.; Glockner, G. (March 2008). "Chromatophore genome sequence of Paulinella sheds light on acquisition of photosynthesis by eukaryotes". Current Biology. 18 (6): 410–8. doi:10.1016/j.cub.2008.02.051. PMID 18356055. S2CID 15929741.
- ^ a b c d e f g h i j Barbrook, Adrian C.; Howe, Christopher J.; Purton, Saul (February 2006). "Why are plastid genomes retained in non-photosynthetic organisms?". Trends in Plant Science. 11 (2): 101–8. doi:10.1016/j.tplants.2005.12.004. PMID 16406301.
- ^ a b c d e f g Leister, D. (December 2005). "Origin, evolution and genetic effects of nuclear insertions of organelle DNA". Trends in Genetics. 21 (12): 655–63. doi:10.1016/j.tig.2005.09.004. hdl:11858/00-001M-0000-0012-3B56-7. PMID 16216380.
- ^ Keeling, P. J. (October 2004). "Diversity and evolutionary history of plastids and their hosts". American Journal of Botany. 91 (10): 1481–93. doi:10.3732/ajb.91.10.1481. PMID 21652304.
- ^ a b c Archibald, J. M. (January 2009). "The puzzle of plastid evolution". Current Biology. 19 (2): R81–R88. doi:10.1016/j.cub.2008.11.067. PMID 19174147. S2CID 51989.
- ^ a b c Ford Doolittle, W (1998-12-01). "You are what you eat: a gene transfer ratchet could account for bacterial genes in eukaryotic nuclear genomes". Trends in Genetics. 14 (8): 307–311. doi:10.1016/S0168-9525(98)01494-2. ISSN 0168-9525. PMID 9724962.
- ^ Zimorski, Verena; Ku, Chuan; Martin, William F; Gould, Sven B (2014). "Endosymbiotic theory for organelle origins". Current Opinion in Microbiology. 22: 38–48. doi:10.1016/j.mib.2014.09.008. PMID 25306530.
- ^ "Mitochondria, Cell Energy, ATP Synthase: Learn Science at Scitable". www.nature.com. Retrieved 24 March 2019.
- ^ a b Gruber, A. (January 2019). "What's in a name? How organelles of endosymbiotic origin can be distinguished from endosymbionts". Microbial Cell. 6 (2): 123–133. doi:10.15698/mic2019.02.668. PMC 6364258. PMID 30740457.
- ^ a b c d e f Gould, Sven B.; Garg, Sriram G.; Martin, William F. (July 2016). "Bacterial Vesicle Secretion and the Evolutionary Origin of the Eukaryotic Endomembrane System". Trends in Microbiology. 24 (7): 525–534. doi:10.1016/j.tim.2016.03.005. PMID 27040918.
- ^ a b Martin, William F.; Garg, Sriram; Zimorski, Verena (September 2015). "Endosymbiotic theories for eukaryote origin". Philosophical Transactions of the Royal Society of London. Series B, Biological Sciences. 370 (1678): 20140330. doi:10.1098/rstb.2014.0330. PMC 4571569. PMID 26323761.
- ^ Garavís, Miguel; González, Carlos; Villasante, Alfredo (June 2013). "On the origin of the eukaryotic chromosome: the role of noncanonical DNA structures in telomere evolution". Genome Biology and Evolution. 5 (6): 1142–50. doi:10.1093/gbe/evt079. PMC 3698924. PMID 23699225.
- ^ "Typical prokaryotic (left) and eukaryotic (right) cells: Learn Science at Scitable". www.nature.com. Retrieved 2019-03-24.
- ^ Devos, Damien P.; Gräf, Ralph; Field, Mark C. (June 2014). "Evolution of the nucleus". Current Opinion in Cell Biology. 28 (100): 8–15. doi:10.1016/j.ceb.2014.01.004. PMC 4071446. PMID 24508984.
- ^ Wilson, Katherine L.; Dawson, Scott C. (October 2011). "Evolution: functional evolution of nuclear structure". Journal of Cell Biology. 195 (2): 171–81. doi:10.1083/jcb.201103171. PMC 3198171. PMID 22006947.
- ^ Bernstein, H.; Bernstein, C. (2017). "Sexual communication in archaea, the precursor to meiosis". In Witzany, G. (ed.). Biocommunication of Archaea. Springer International Publishing. pp. 103–117. doi:10.1007/978-3-319-65536-9. ISBN 978-3-319-65535-2. S2CID 26593032.
- ^ Gabaldón, T.; Huynen, M. A. (August 2003). "Reconstruction of the proto-mitochondrial metabolism". Science. 301 (5633): 609. doi:10.1126/science.1085463. PMID 12893934. S2CID 28868747.
- ^ Liashkovich, Ivan; Shahin, Victor (August 2017). "Functional implication of the common evolutionary origin of nuclear pore complex and endomembrane management systems". Seminars in Cell and Developmental Biology. 68: 10–17. doi:10.1016/j.semcdb.2017.04.006. PMID 28473267.
- ^ a b Howe, Christopher J. (May 2008). "Cellular evolution: what's in a mitochondrion?". Current Biology. 18 (10): R429–R431. doi:10.1016/j.cub.2008.04.007. PMID 18492476. S2CID 15730462.
- ^ López-García, Purificación; Moreira, David (2020-04-27). "The Syntrophy hypothesis for the origin of eukaryotes revisited" (PDF). Nature Microbiology. 5 (5): 655–667. doi:10.1038/s41564-020-0710-4. ISSN 2058-5276. PMID 32341569. S2CID 81678433.
- ^ Giannakis, Konstantinos; Arrowsmith, Samuel J.; Richards, Luke; et al. (16 September 2022). "Evolutionary inference across eukaryotes identifies universal features shaping organelle gene retention". Cell Systems. 13 (11): 874–884.e5. doi:10.1016/j.cels.2022.08.007. PMID 36115336. S2CID 252337501.
- ^ a b c d Lane, Nick (2011). "Plastids, genomes, and the probability of gene transfer". Genome Biology and Evolution. 3: 372–374. doi:10.1093/gbe/evr003. PMC 3101016. PMID 21292628.
- ^ [2]Wayback Machine Kimball, J. 2010에서 2017-06-22 보관.킴볼의 생물학 페이지.2010년 10월 13일 접속.일반 생물학 텍스트의 저자이자 하버드대 교수의 온라인 오픈소스 생물학 텍스트, John W. Kimball.
- ^ 리스, J. 리사 A.우리, 마이클 L. 케인, 스티븐 A.와서먼, 피터 5세.미니어처 스카이, 로버트 B.잭슨, 2010년캠벨 생물학.제9판 벤자민 커밍스; 제9판 (2010년 10월 7일)
- ^ Raven, P.; Johnson, George; Mason, Kenneth; et al. (January 14, 2010). Biology (9th ed.). McGraw-Hill.
- ^ Gray, M. W. (1992). The endosymbiont hypothesis revisited. pp. 233–357. doi:10.1016/S0074-7696(08)62068-9. ISBN 9780123645449. PMID 1452433.
{{cite book}}:journal=무시됨(도움말) - ^ Zimorski, V.; Ku, C.; Martin, W. F.; Gould, S. B. (December 2014). "Endosymbiotic theory for organelle origins". Current Opinion in Microbiology. 22: 38–48. doi:10.1016/j.mib.2014.09.008. PMID 25306530.
- ^ Margolin, William (November 2005). "FtsZ and the division of prokaryotic cells and organelles". Nature Reviews. Molecular Cell Biology. 6 (11): 862–71. doi:10.1038/nrm1745. PMC 4757588. PMID 16227976.
- ^ Wise, Robert R.; Hoober, J. Kenneth (2007). Structure and function of plastids. Berlin: Springer. p. 104. ISBN 9781402065705.
- ^ Fischer, K.; Weber, A.; Brink, S.; et al. (October 1994). "Porins from plants. Molecular cloning and functional characterization of two new members of the porin family". The Journal of Biological Chemistry. 269 (41): 25754–60. doi:10.1016/S0021-9258(18)47312-7. PMID 7523392.
- ^ Zeth, K.; Thein, M. (October 2010). "Porins in prokaryotes and eukaryotes: common themes and variations". The Biochemical Journal. 431 (1): 13–22. doi:10.1042/BJ20100371. PMID 20836765. S2CID 22073622.
- ^ Fairman, J. W.; Noinaj, N.; Buchanan, S. K. (August 2011). "The structural biology of β-barrel membrane proteins: a summary of recent reports". Current Opinion in Structural Biology. 21 (4): 523–331. doi:10.1016/j.sbi.2011.05.005. PMC 3164749. PMID 21719274.
- ^ Mileykovskaya, E.; Dowhan, W. (October 2009). "Cardiolipin membrane domains in prokaryotes and eukaryotes". Biochimica et Biophysica Acta (BBA) - Biomembranes. 1788 (10): 2084–91. doi:10.1016/j.bbamem.2009.04.003. PMC 2757463. PMID 19371718.
- ^ a b Timmis, Jeremy; Ayliffe, Michael; Huang, Chun; Martin, William (February 2004). "Endosymbiotic gene transfer: organelle genomes forge eukaryotic chromosomes". Nature Reviews. Genetics. 5 (2): 123–35. doi:10.1038/nrg1271. PMID 14735123. S2CID 2385111.
- ^ Munoz-Gomez, Sergio; Susko, Edward; Williamson, Kelsey; et al. (January 2022). "Site-and-branch-heterogeneous analyses of an expanded dataset favour mitochondria as sister to known Alphaproteobacteria". Nature Ecology and Evolution. 6 (3): 253–62. doi:10.1038/s41559-021-01638-2. PMID 35027725. S2CID 245958471.
- ^ Dagan, Tal; Roettger, Mayo; Stucken, Karina; et al. (2013). "Genomes of Stigonematalean cyanobacteria (subsection V) and the evolution of oxygenic photosynthesis from prokaryotes to plastids". Genome Biology and Evolution. 5 (1): 31–44. doi:10.1093/gbe/evs117. PMC 3595030. PMID 23221676.
- ^ Manuell, Andrea L.; Quispe, Joel; Mayfield, Stephen P. (August 2007). "Structure of the chloroplast ribosome: novel domains for translation regulation". PLOS Biology. 5 (8): e209. doi:10.1371/journal.pbio.0050209. PMC 1939882. PMID 17683199.
- ^ Schwartz, James H.; Meyer, Ralph; Eisenstadt, Jerome M.; Brawerman, George (May 1967). "Involvement of N-formylmethionine in initiation of protein synthesis in cell-free extracts of Euglena gracilis". Journal of Molecular Biology. 25 (3): 571–4. doi:10.1016/0022-2836(67)90210-0. PMID 5340700.
- ^ Smith, A.E.; Marcker, K.A. (December 1968). "N-formylmethionyl transfer RNA in mitochondria from yeast and rat liver". Journal of Molecular Biology. 38 (2): 241–3. doi:10.1016/0022-2836(68)90409-9. PMID 5760639.
- ^ McFadden, G. I. (2001). "Primary and secondary endosymbiosis and the origin of plastids". Journal of Phycology. 37 (6): 951–959. doi:10.1046/j.1529-8817.2001.01126.x. S2CID 51945442.
- ^ Okamoto, N.; Inouye, I. (October 2005). "A secondary symbiosis in progress?". Science. 310 (5746): 287. doi:10.1126/science.1116125. PMID 16224014. S2CID 22081618.
- ^ McFadden, G. I.; van Dooren, G. G. (July 2004). "Evolution: red algal genome affirms a common origin of all plastids". Current Biology. 14 (13): R514-6. doi:10.1016/j.cub.2004.06.041. PMID 15242632. S2CID 18131616.
- ^ Gould, Sven B.; Waller, Ross F.; McFadden, Geoffrey I. (2008). "Plastid evolution". Annual Review of Plant Biology. 59 (1): 491–517. doi:10.1146/annurev.arplant.59.032607.092915. PMID 18315522. S2CID 30458113.
- ^ Georgiades, K.; Raoult, D. (October 2011). "The rhizome of Reclinomonas americana, Homo sapiens, Pediculus humanus and Saccharomyces cerevisiae mitochondria". Biology Direct. 6: 55. doi:10.1186/1745-6150-6-55. PMC 3214132. PMID 22014084.
- ^ Prasad, Pijai (August 2005). "Organic-walled microfossils from the Proterozoic Vindhyan Supergroup of Son Valley, Madhya Pradesh, India" (PDF). Paleobotanist. 54.
- ^ Butterfield, Nicholas J. (2014-11-26). "Early evolution of the Eukaryota". Palaeontology. 58 (1): 5–17. doi:10.1111/pala.12139.
- ^ Parfrey, Laura Wegener; Lahr, Daniel J. G.; Knoll, Andrew H.; Katz, Laura A. (August 2011). "Estimating the timing of early eukaryotic diversification with multigene molecular clocks". Proceedings of the National Academy of Sciences of the United States of America. 108 (33): 13624–9. Bibcode:2011PNAS..10813624P. doi:10.1073/pnas.1110633108. PMC 3158185. PMID 21810989.
- ^ Hedges, S. Blair; Blair, Jaime E.; Venturi, Maria L.; Shoe, Jason L. (January 2004). "A molecular timescale of eukaryote evolution and the rise of complex multicellular life". BMC Evolutionary Biology. 4: 2. doi:10.1186/1471-2148-4-2. PMC 341452. PMID 15005799.
- ^ Gross, Jeferson; Bhattacharya, Debashish (August 2010). "Uniting sex and eukaryote origins in an emerging oxygenic world". Biology Direct. 5: 53. doi:10.1186/1745-6150-5-53. PMC 2933680. PMID 20731852.
- ^ Butterfield, Nicholas J. (1997). "Plankton ecology and the Proterozoic-Phanerozoic transition". Paleobiology. 23 (2): 247–262. Bibcode:1997Pbio...23..247B. doi:10.1017/S009483730001681X. S2CID 140642074.
추가열람
- Alberts, Bruce (2002). Molecular Biology of the Cell. New York: Garland Science. ISBN 978-0-8153-3218-3. (일반교재)
- Brinkman, Fiona S. L.; Blanchard, Jeffrey L.; Cherkasov, Artem; et al. (August 2002). "Evidence that plant-like genes in Chlamydia species reflect an ancestral relationship between Chlamydiaceae, cyanobacteria, and the chloroplast". Genome Research. 12 (8): 1159–67. doi:10.1101/gr.341802. PMC 186644. PMID 12176923.
- Cohen, W. E.; Gardner, R. S. (1959). "Viral Theory and Endosymbiosis" (PDF). Archived from the original (PDF) on 2011-07-15. Retrieved 2009-08-26. ('파지 세균 및 추정 바이러스성 미토콘드리아/클로로플라스틱 상호작용'에 중점을 두고 혐기성 세포에 미토콘드리아와 엽록체를 통합하여 진핵세포 기원론을 논의합니다.)
- Jarvis, P. (April 2001). "Intracellular signalling: the chloroplast talks!". Current Biology. 11 (8): R307-10. doi:10.1016/S0960-9822(01)00171-3. PMID 11369220. S2CID 11753648. (미토콘드리아나 엽록체에 영향을 미치는 핵 암호화 단백질의 경우가 더 잘 기록된 것과 달리 엽록체로 암호화된 단백질이 핵 유전자의 전사에 영향을 미친다는 증거를 다시 설명합니다.)
- Blanchard, J. L.; Lynch, M. (July 2000). "Organellar genes: why do they end up in the nucleus?". Trends in Genetics. 16 (7): 315–20. doi:10.1016/S0168-9525(00)02053-9. PMID 10858662. (미토콘드리아와 엽록체 유전자가 어떻게 핵 안으로 전달되는지, 그리고 이 과정을 완성하기 위해 유전자가 어떤 과정을 거쳐야 하는지에 대한 이론을 논의합니다.)
- Okamoto, N.; Inouye, I. (October 2005). "A secondary symbiosis in progress?". Science. 310 (5746): 287. doi:10.1126/science.1116125. PMID 16224014. S2CID 22081618.
- Understanding Science Team. "Cells within cells: An extraordinary claim with extraordinary evidence" (PDF). University of California, Berkeley. Retrieved 16 February 2014.



