I족 촉매 인트론
Group I catalytic intron| I족 촉매 인트론 | |
|---|---|
 | |
| 식별자 | |
| 기호. | Intron_gpi |
| Rfam | RF00028 |
| 기타 데이터 | |
| RNA형 | 인트론 |
| 도메인 | 진핵생물, 박테리아, 바이러스 |
| 가세요 | GO:0000372 |
| 그렇게 | SO:0000587 |
| PDB 구조 | PDBe |
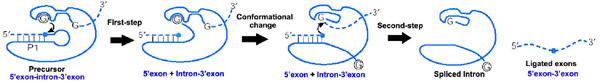
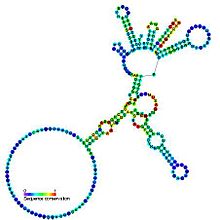
그룹 I 인트론은 큰 자기 분열 리보자임이다.이들은 광범위한 [1][2][3]유기체의 mRNA, tRNA 및 rRNA 전구체로부터 자체 절제를 촉매한다.코어 2차 구조는 9개의 쌍으로 구성된 영역(P1-P9)[4]으로 구성됩니다.이것들은 기본적으로 2개의 도메인, 즉 P4-P6 도메인(P5, P4, P6 및 P6a 헬리크 스택에서 형성)과 P3-P9 도메인(P8,[2] P3, P7, P9 헬리크에서 형성)으로 접힌다.이 패밀리의 세컨더리 구조 마크업은 이 보존된 코어만을 나타냅니다.그룹 I 인트론은 루프 영역에 긴 열린 읽기 프레임을 삽입하는 경우가 많습니다.
촉매 작용
I족 인트론의 스플라이싱은 2개의 순차적 에스테르 전이 [3]반응에 의해 처리된다.외인성 구아노신 또는 구아노신 뉴클레오티드(exoG)는 먼저 P7에 위치한 활성 G결합 부위에 도킹하고, 그 3'-OH는 P1에 위치한 5' 스플라이스 부위의 포스포디에스테르 결합을 공격하도록 배열되어 업스트림 엑손의 3'-OH기를 생성하고, EXOG는 인트론의 5' 말단에 결합한다.다음으로 인트론의 말단 G(오메가 G)가 exoG를 스왑하여 G결합부위를 점유하여 제2에스테르전달반응을 형성한다.P1의 업스트림 엑손의 3'-OH군이 P10의 3' 스플라이스부위를 공격하도록 정렬되어 인접한 업스트림 엑손과 다운스트림 엑손의 결합 및 촉매 인트론의 방출로 이어진다.
단백질 중합효소 및 포스파타아제에서 볼 수 있는 2-금속 이온 메커니즘은 그룹 I 및 그룹 II의 인트론에서 포스포릴 [5]전달 반응을 처리하기 위해 사용되도록 제안되었으며,[6] 이는 2006년 그룹 I의 인트론의 고해상도 구조에 의해 명확하게 입증되었다.


인트론 폴딩
1990년대 초부터 과학자들은 I그룹 인트론이 체외에서 어떻게 본래의 구조를 달성하는지 연구하기 시작했고,[10] 지금까지 RNA 접힘의 일부 메커니즘이 인정되어 왔다.2차 구조를 형성한 후 3차 구조를 접는 것으로 합의한다.접히는 동안 RNA 분자는 다른 접히는 중간체로 빠르게 채워지고, 네이티브 상호작용을 포함하는 중간체는 빠른 접히는 경로를 통해 네이티브 구조로 더욱 접히는 반면, 비네이티브 상호작용을 포함하는 중간체는 준안정적이거나 안정적인 비네이티브 배합으로 갇히고, 대류의 과정원어민 구조로의 회수는 매우 천천히 일어난다.그룹 I은 일련의 주변 요소에서 서로 다른 침입자가 고속 접힘 경로에 진입할 때 서로 다른 가능성을 보이는 것이 분명하다.한편, 제3구조의 공동조립은 토종구조의 접힘에 있어서 중요하다.그럼에도 불구하고, 체외에서 그룹 I의 접힘은 열역학적 및 운동학적 도전에 직면한다.소수의 RNA 결합 단백질과 샤페론은 각각 토종 중간체를 안정화시키고 비토종 구조를 불안정하게 함으로써 체외 및 박테리아 내 I군 침입자의 접힘을 촉진하는 것으로 나타났다.
유통, 계통발생 및 이동성
I그룹 인트론은 박테리아, 낮은 진핵생물, 높은 식물에 분포한다.그러나 박테리아에서 발생하는 것은 낮은 진핵생물보다 더 산발적인 것으로 보이며, 그것들은 높은 식물에서 널리 퍼지고 있다.그룹 I이 간섭하는 유전자는 크게 다릅니다.그들은 박테리아 게놈뿐만 아니라 낮은 진핵생물의 미토콘드리아와 엽록체 게놈의 rRNA, mRNA, tRNA 유전자를 방해하지만, 낮은 진핵생물의 핵 게놈의 rRNA 유전자만 침범한다.고등 식물에서, 이러한 인트론은 엽록체와 미토콘드리아의 몇 개의 tRNA와 mRNA 유전자로 제한되는 것으로 보인다.
그룹 I 인트론은 또한 그램 양성 [11]박테리오파지의 다양한 박테리오파지의 유전자에 삽입된 것으로 발견되었다.단, 그람음성균의 파지 내에서의 분포는 주로 T4, T-짝수 및 T7형 박테리오파지에 [11][12][13][14]한정된다.
인트론-얼리 이론과 인트론-후기 이론 모두 I그룹 인트론의 기원을 설명하는 증거를 발견했다.일부 그룹 I 인트론은 인트론 모빌리티를 촉매하는 호밍핵산가수분해효소(HEG)를 인코딩합니다.HEG는 인트론을 한 곳에서 다른 곳으로, 한 유기체에서 다른 유기체로 이동시켜 이기적인 집단 I의 광범위한 확산을 설명해야 한다고 제안되었다.지금까지 그룹 I의 침입자에 대한 생물학적 역할은 그들이 살고 있는 숙주의 죽음을 막기 위해 전구체로부터 그들 자신을 스플라이싱하는 것 외에는 확인되지 않았다.또한 소수의 I군 인트론은 인트론 스플라이싱을 촉진하는 마투라아제라고 불리는 단백질의 클래스를 코드하는 것으로 밝혀졌다.
「 」를 참조해 주세요.
- 인트론
- Group I Intron 시퀀스 및 구조 데이터베이스
- 스플라이스 사이트
- 핵 인트론
- 그룹 II 인트론
- 그룹 III 인트론
- 트윈트론
- LTRA
- I족 인트론과 함께 작용하여 유전자의 발현을 조절하는 리보스위치의 예가 있는 고리형 di-GMP-II 리보스위치
레퍼런스
- ^ Nielsen H, Johansen SD (2009). "Group I introns: Moving in new directions". RNA Biol. 6 (4): 375–83. doi:10.4161/rna.6.4.9334. PMID 19667762. Retrieved 2010-07-15.
- ^ a b Cate JH, Gooding AR, Podell E, et al. (September 1996). "Crystal structure of a group I ribozyme domain: principles of RNA packing". Science. 273 (5282): 1678–85. Bibcode:1996Sci...273.1678C. doi:10.1126/science.273.5282.1678. PMID 8781224. S2CID 38185676.
- ^ a b Cech TR (1990). "Self-splicing of group I introns". Annu. Rev. Biochem. 59: 543–68. doi:10.1146/annurev.bi.59.070190.002551. PMID 2197983.
- ^ Woodson SA (June 2005). "Structure and assembly of group I introns". Curr. Opin. Struct. Biol. 15 (3): 324–30. doi:10.1016/j.sbi.2005.05.007. PMID 15922592.
- ^ Steitz, TA; Steitz JA (1993). "A general two-metal-ion mechanism for catalytic RNA". Proc Natl Acad Sci USA. 90 (14): 6498–6502. Bibcode:1993PNAS...90.6498S. doi:10.1073/pnas.90.14.6498. PMC 46959. PMID 8341661.
- ^ Stahley, MR; Strobel SA (2006). "RNA splicing: group I intron crystal structures reveal the basis of splice site selection and metal ion catalysis". Curr Opin Struct Biol. 16 (3): 319–326. doi:10.1016/j.sbi.2006.04.005. PMID 16697179.
- ^ Golden BL, Gooding AR, Podell ER, Cech TR (1998). "A preorganized active site in the crystal structure of the Tetrahymena ribozyme". Science. 282 (5387): 259–64. Bibcode:1998Sci...282..259G. doi:10.1126/science.282.5387.259. PMID 9841391.
- ^ Golden BL, Kim H, Chase E (2005). "Crystal structure of a phage Twort group I ribozyme-product complex". Nat Struct Mol Biol. 12 (1): 82–9. doi:10.1038/nsmb868. PMID 15580277. S2CID 33369317.
- ^ Guo F, Gooding AR, Cech TR (2004). "Structure of the Tetrahymena ribozyme: base triple sandwich and metal ion at the active site". Mol Cell. 16 (3): 351–62. doi:10.1016/j.molcel.2004.10.003. PMID 15525509.
- ^ Brion P, Westhof E (1997). "Hierarchy and dynamics of RNA folding". Annu Rev Biophys Biomol Struct. 26: 113–37. doi:10.1146/annurev.biophys.26.1.113. PMID 9241415.
- ^ a b Edgell DR, Belfort M, Shub DA (October 2000). "Barriers to intron promiscuity in bacteria". J. Bacteriol. 182 (19): 5281–9. doi:10.1128/jb.182.19.5281-5289.2000. PMC 110968. PMID 10986228.
- ^ Sandegren L, Sjöberg BM (May 2004). "Distribution, sequence homology, and homing of group I introns among T-even-like bacteriophages: evidence for recent transfer of old introns". J. Biol. Chem. 279 (21): 22218–27. doi:10.1074/jbc.M400929200. PMID 15026408.
- ^ Bonocora RP, Shub DA (December 2004). "A self-splicing group I intron in DNA polymerase genes of T7-like bacteriophages". J. Bacteriol. 186 (23): 8153–5. doi:10.1128/JB.186.23.8153-8155.2004. PMC 529087. PMID 15547290.
- ^ Lee CN, Lin JW, Weng SF, Tseng YH (December 2009). "Genomic characterization of the intron-containing T7-like phage phiL7 of Xanthomonas campestris". Appl. Environ. Microbiol. 75 (24): 7828–37. Bibcode:2009ApEnM..75.7828L. doi:10.1128/AEM.01214-09. PMC 2794104. PMID 19854925.
추가 정보
- Chauhan, S; Caliskan G; Briber RM; Perez-Salas U; Rangan P; Thirumalai D; Woodson SA (2005). "RNA tertiary interactions mediate native collapse of a bacterial group I ribozyme". J Mol Biol. 353 (5): 1199–1209. doi:10.1016/j.jmb.2005.09.015. PMID 16214167.
- Haugen, P; Simon DM; Bhattacharya D (2005). "The natural history of group I introns". Trends in Genetics. 21 (2): 111–119. doi:10.1016/j.tig.2004.12.007. PMID 15661357.
- Rangan, P; Masquida, B; Westhof E; Woodson SA (2003). "Assembly of core helices and rapid tertiary folding of a small bacterial group I ribozyme". Proc Natl Acad Sci USA. 100 (4): 1574–1579. Bibcode:2003PNAS..100.1574R. doi:10.1073/pnas.0337743100. PMC 149874. PMID 12574513.
- Schroeder, R; Barta A; Semrad K (2004). "Strategies for RNA folding and assembly". Nat Rev Mol Cell Biol. 5 (11): 908–919. doi:10.1038/nrm1497. PMID 15520810. S2CID 22030359.
- Thirumalai, D; Lee N; Woodson SA; Klimov D (2001). "Early events in RNA folding". Annu Rev Phys Chem. 52: 751–762. Bibcode:2001ARPC...52..751T. doi:10.1146/annurev.physchem.52.1.751. PMID 11326079.
- Treiber, DK; Williamson JR (1999). "Exposing the kinetic traps in RNA folding". Curr Opin Struct Biol. 9 (3): 339–345. doi:10.1016/S0959-440X(99)80045-1. PMID 10361090.
- Xiao, M; Leibowitz MJ; Zhang Y (2003). "Concerted folding of a Candida ribozyme into the catalytically active structure posterior to a rapid RNA compaction". Nucleic Acids Res. 31 (14): 3901–3908. doi:10.1093/nar/gkg455. PMC 165970. PMID 12853605.