원형 염색체
Circular chromosome원형 염색체는 대부분의 진핵 생물의 선형 염색체와 달리 원형 DNA 분자의 형태로 박테리아, 고세균, 미토콘드리아, 그리고 엽록체의 염색체이다.
대부분의 원핵생물 염색체는 원형 DNA 분자를 포함하고 있다 – DNA에는 자유단이 없다.그렇지 않으면 자유단은 DNA 복제와 안정성에 관해 세포에 중대한 도전을 야기할 것이다.DNA 말단을 가진 염색체를 포함하는 세포 또는 텔로미어(대부분의 진핵생물)는 이러한 문제들을 극복하기 위한 정교한 메커니즘을 획득했습니다.그러나 원형 염색체는 세포에 다른 문제를 제공할 수 있다.복제 후, 두 개의 자손 원형 염색체는 때때로 상호 연결되거나 엉킨 채로 남아있을 수 있으며, 각 세포가 세포 분열 동안 하나의 완전한 염색체 복사본을 상속하도록 분해되어야 한다.
레플리케이션
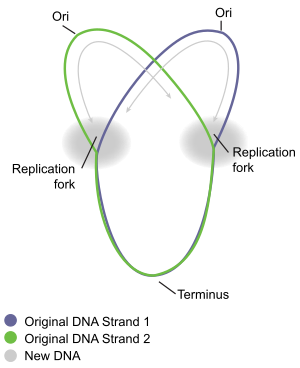
원형세균 염색체 복제는 잘 연구된 대장균과 서브틸리스균에서 가장 잘 이해된다.염색체 복제는 시작, 신장, 종료의 세 가지 주요 단계로 진행됩니다.시작 단계는 oriC라고 불리는 염색체의 원점 영역에서 "개시자" 단백질의 순서 있는 집합으로 시작합니다.이러한 조립 단계는 염색체 복제가 각 세포 주기에 한 번만 발생하도록 조절된다.복제의 신장 단계 동안, 시작 동안 오리C에서 조립된 효소는 염색체의 각 팔("replichore")을 따라 오리C에서 반대 방향으로 진행되며, DNA를 복제하여 두 개의 동일한 복사본을 생성한다.이 프로세스를 쌍방향 복제라고 합니다.각각의 팔에 있는 DNA 복제에 관련된 분자들의 전체 집합은 "복제"라고 불립니다.리플리솜의 선두에는 두 가닥의 DNA를 풀어주는 DNA 헬리케이스가 있어 움직이는 복제 포크(replication fork)를 만든다.풀린 두 개의 단일 DNA 가닥은 각 가닥의 상보적인 복사본을 합성하기 위해 헬리케이스(다른 단백질과 함께)와 함께 이동하는 DNA 중합효소의 템플릿 역할을 합니다.이 방법으로 원본 DNA의 두 개의 동일한 복사본이 만들어집니다.결국 원형 염색체 주위를 이동하는 두 복제 포크는 말단 영역이라고 불리는 염색체의 거의 반대쪽인 특정 구역에서 만난다.신장 효소는 분해되고 두 개의 "딸" 염색체는 세포 분열이 완료되기 전에 분해된다.
개시
oriC라고 불리는 대장균 박테리아 복제 기원은 다른 박테리아 종들 사이에서 잘 보존된 DnaA 단백질에 의해 인식되는 DNA 배열로 구성됩니다.기원에 결합하는 DNAA는 결국 양방향 [1]복제를 위한 두 개의 완전한 리플리솜을 확립하는 것으로 이어지는 다른 효소 및 단백질의 조절된 모집을 시작합니다.
기능에 중요한 오리C 내 DNA 배열 요소는 DnaA 박스를 포함하며, 이는 DnaA 단백질에 의해 인식되는 고도로 보존된 합의 배열 5' – TTATCACA – 3'[2]을 가진 9m 반복이다.DnaA 단백질은 염색체 DNA [3]복제의 시작에 중요한 역할을 한다.ATP에 결합되어 박테리아 히스톤 유사 단백질의 도움으로 DnaA는 3개의 13-mer [4]모티브를 가진 oriC의 왼쪽 경계 근처의 AT가 풍부한 영역을 풀고 다른 복제 [5]단백질의 입구를 위해 이중 가닥 DNA를 개방한다.
이 영역은 또한 DNA 아데닌 메틸라아제(Dam)에 의해 인식되는 4개의 "GATC" 염기서열을 포함하고 있는데, 이 염기서열이 비메틸화 또는 반메틸화되었을 때 아데닌 염기를 수정하는 효소이다.아데닌의 메틸화는 가닥 분리를 [6]촉진하기 위해 DNA의 구조를 변화시키기 때문에 중요하며,[7] 오리C의 이 영역은 자연스럽게 풀리는 경향이 있는 것으로 보인다.
그런 다음 DnaA는 프리 프라이밍 복합체를 형성하기 위해 DnaB-DnaC 복합체에서 [8]미완성 영역으로 복제 헬리케이스인 DnaB를 모집한다.DnaB가 각 복제 포크의 꼭대기로 이동한 후,[9] 헬리케이스는 부모 DNA를 풀고 프리마아제와 순간적으로 상호작용합니다.
DNA 복제가 계속되기 위해서는 단일 가닥의 DNA가 2차 구조를 형성하지 않도록 하고 재결합을 방지하기 위해 단일 가닥 결합 단백질이 필요하다.또한 DNA 자이라제는 DnaB 헬리케이스의 작용에 의해 발생하는 위상 스트레스를 완화하기 위해 필요하다.
신장
레플리케이션 포크가 원을 따라 움직이면 그리스 문자 세타 δ와 같은 구조가 형성된다.John Cairns는 1963년 DNA 복제를 시각화하는 혁신적인 방법을 사용하여 대장균 염색체 복제의 세타 구조를 시연했다.그의 실험에서, 그는 3H-티미딘이 함유된 배지에서 배양하여 염색체에 방사능 라벨을 붙였습니다.뉴클레오시드 염기는 균등하게 세균 염색체에 통합되었다.그런 다음 세포를 부드럽게 용해시켜 염색체를 분리한 후 전자 현미경(EM) 그리드에 두 달 동안 X선 필름에 노출시켰다.이 실험은 원형 세균 [10]염색체의 세타 복제 모델을 명확히 보여준다.
- E.coli의 온전한 복제 염색체의 자동 방사선 촬영 참조 [1]
위와 같이 박테리아 염색체 복제는 양방향으로 일어난다.이것은 최초로 복제 세균 염색체에 방사성 동위원소를 라벨링함으로써 입증되었다.실험 중에 복제되는 DNA의 영역은 자동 방사선 촬영과 현미경으로 현상된 필름을 조사함으로써 시각화되었다.이를 통해 연구원들은 어디에서 복제가 일어나는지 볼 수 있었다.양방향 복제의 첫 번째 결정적인 관찰은 B. 서브틸리스 [11]연구로부터였다.얼마 지나지 않아 대장균 염색체도 양방향으로 [12]복제되는 것으로 나타났다.
- D의 그림 4를 참조한다.M. Prescott 및 P. L. Kuempel(1972) :대장균 염색체가 [3H]티민으로 19분 동안 라벨링된 세포에서 생성된 곡물 궤적, [3H]티민 및 ['H]티미딘으로 라벨링된 2.5분 동안 라벨링된 곡물 궤적.[2]
대장균 DNA 중합효소 III 홀로엔자임은 900kD 복합체로 기본적으로 이합체 구조를 가지고 있다.각 단량체 유닛은 촉매 코어, 이합체 서브유닛 및 [13]처리성분을 가진다.DNA Pol III는 그 코어 서브유닛의 1세트를 사용하여 리드스트랜드를 연속적으로 합성하고, 다른 코어 서브유닛은 루프상 후행스트랜드 상에서 1세트에서 다음 세트로 순환한다.선두 가닥 합성은 프리마아제 효소(DnaG 단백질)에 의한 복제 원점에서의 짧은 RNA 프라이머 합성으로 시작된다.
디옥시뉴클레오티드는 단일 DNA 중합효소 III 이합체에 의해 DnaB 헬리케이스와 통합된 복합체로 이 프라이머에 첨가된다.리플리케이션 포크에서 DNA가 동시에 풀리는 동안 선도 가닥 합성이 연속적으로 진행된다.반면 짧은 오카자키 단편에서는 후행 스트랜드 합성이 이루어진다.우선 프리마아제에 의해 RNA 프라이머를 합성하고, 선두 스트랜드 합성과 마찬가지로 DNA Pol III가 RNA 프라이머에 결합해 디옥시리보뉴클레오티드를 첨가한다.
Okazaki 단편의 합성이 완료되면, 복제가 멈추고 DNA Pol III의 핵심 서브유닛이 β 슬라이딩 클램프에서 분리된다[B 슬라이딩 클랩은 DNA Pol [14]III의 처리성 서브유닛이다].RNA 프라이머는 DNA 중합효소 I에 의해 제거되고 DNA로 대체되며(또한 교정성 엑소핵산가수분해효소 활성을 보유함), 나머지 닉은 DNA 연결효소에 의해 봉인되며, DNA 연결효소는 이러한 단편들을 결합시켜 지연 가닥을 형성한다.
ORIC에서 발생하는 복제 [15]포크의 상당 부분(10~15%)은 (외인성 DNA 손상처리가 없는) 정상적인 실험실 조건 하에서 세포가 성장했을 때 DNA 손상 또는 가닥 파손에 직면한다.조우한 DNA 손상은 일반적으로 재조합 복구 효소에 의해 처리되어 지속적인 복제 포크 [15]진행을 가능하게 합니다.
종료
터미네이션은 복제 포크의 융합과 복제체의 분해를 통해 두 개의 분리된 완전한 DNA 분자를 생성하는 과정입니다.이는 염색체의 오리C와 거의 반대인 말단 영역에서 발생한다(그림 5).터미널 영역에는 여러 DNA 복제 터미네이터 사이트(Ter)가 있습니다.특별한 "복제 종료자" 단백질이 복제를 일시 중지하기 위해 Ter 사이트에 결합되어야 합니다.각 Ter 사이트는 동작의 극성을 가지고 있습니다.즉, 한 방향에서 Ter 사이트로 접근하는 복제 포크를 정지시키지만 다른 방향에서 Ter 사이트를 통해 방해받지 않는 포크 이동을 허용합니다.Ter 사이트의 배열은 두 개의 대립 그룹을 형성하며, 두 개의 포크가 서로 교차하는 영역 내에서 만나게 합니다.이 배치를 "레플리케이션 포크 트랩"[16]이라고 합니다.
- 대장균 복제 말미의 위치와 순서를 참조하십시오.(가) ORI와 10 Ter 사이트를 나타낸 지도 (나) Ter의 컨센서스 시퀀스[3]
Ter 사이트는 특히 [17]대장균의 Tus라고 불리는 복제 종료 단백질과 상호작용합니다.Tus-Ter 복합체는 방향 의존적인 방식으로 [18]DnaB의 DNA 풀림 활동을 방해한다.
반대되는 복제를 분리하는 DNA의 복제는 완성된 염색체를 '카테난' 또는 위상적으로 상호 연결된 원으로 결합시킵니다.원은 공유로 연결되어 있지 않지만 상호 연결되어 있고 각각 공유로 닫혀 있기 때문에 분리할 수 없습니다.결합원은 원을 분리하기 위해 국소 이성질화효소의 작용을 필요로 한다.E.coli에서, DNA 토포이소머라아제 IV는 한 염색체의 양쪽 DNA 가닥을 일시적으로 끊고 다른 염색체가 그 분리를 통과하도록 하는 주요 역할을 한다.
DNA 자이라아제가 탈카테닌에서 하는 역할에 대해 약간의 혼란이 있었다.명명법을 정의하기 위해 두 가지 유형의 토포이소머라아제가 있습니다. 즉, I형은 DNA에서 일시적인 단일 가닥 절단을 생성하고 II형은 일시적인 이중 가닥 절단을 생성합니다.그 결과, 제1형 효소는 DNA에서 한 번에 한 개씩 슈퍼코일을 제거하는 반면, 제2형 효소는 한 번에 두 개씩 슈퍼코일을 제거합니다.원핵생물과 진핵생물의 토포I는 I형 토포이소머라아제이다.진핵생물 토포 II, 세균 자이라아제, 세균 토포 IV는 타입 II에 속합니다.
우리는 종종 DNA 자이라아제가 실제로 토포이소머라아제 타입 II 활성을 가지고 있다는 것을 잊어버린다. 따라서, 토포이소머라아제 IV의 상동체이기 때문에, 우리는 두 단백질의 기능에서 유사성을 기대한다.DNA 자이라아제 예비 역할은 DNA에 음의 슈퍼코일을 도입하여 DNA 복제 중에 발생하는 양성 슈퍼코일을 완화시키는 것입니다.토포이소머라아제 IV는 또한 양성 슈퍼코일을 완화하므로, DNA 자이라아제와 토포이소머라아제 IV는 전이성 DNA 중합효소보다 먼저 양성 슈퍼코일을 제거하는 데 거의 동일한 역할을 하므로, 위상학적 [19]변형에 의해 DNA 복제가 방해받지 않는다.
일부 과학 문헌에서 DNA 자이라아제가 탈카테인을 담당하는 유일한 효소라고 언급할 때 혼란이 발생한다.1997년 치히드리히, 호두르스키, 코자렐리에 의해 수행된 실험에서 [20]토포이소머라아제 IV가 박테리아에서 DNA 복제 중간체의 중요한 분쇄효소라는 것이 발견되었다.이 특별한 실험에서 DNA 자이라아제만 억제되었을 때, 대부분의 카테난은 연결되지 않았다.그러나 토포이소머라아제 IV가 단독으로 억제되었을 때 탈카테닌은 거의 완전히 차단되었다.얻어진 결과는 토포이소머라아제 IV가 생체 내 1차 탈카테나아제이며, DNA 자이라아제는 탈카테나아제 역할을 하지만, 그 기능은 상호 연결된 염색체의 탈카테나아제 IV만큼 필수적이지 않다.
확인 응답
이는 이말다 데바파라남과 데이비드 트리브가 2007년 멜버른 대학 미생물학 및 면역학부의 대학 과정 활동에서 SA 라이선스 조건에 따라 CC에서 제공한 기사에 기초하고 있다.
「 」를 참조해 주세요.
레퍼런스
이 기사에는 Creative Commons Attribution-ShareAlike 3.0 Unported License에 따라 라이선스가 부여되지만 GFDL에는 포함되지 않은 시티즌디움 기사 "원형 세균 염색체 복제"의 자료가 포함되어 있다.
- ^ Jon M. Kaguni DnaA: 세균 DNA 복제 시작 제어 등안누, 마이크로바이올 목사님2006. 60:351–71
- ^ C Weigel, A Schmidt, B Rückert, R Lurz, W Messer.Dna 대장균 복제 기원인 oriC에서 개별 DnaA 상자에 결합하는 단백질.EMBO J. 1997년 11월 3일; 16(21) : 6574~6583.
- ^ Hirota Y, Mordoh J, Jacob F(1970) 대장균 III의 세포 분열 과정에 대하여.대장균의 감열성 돌연변이는 DNA 개시 과정에서 변화했다.J Mol Biol, 53, 369–387
- ^ 브램힐 D, 콘버그 A. 1988년대장균 염색체의 원점에서 복제를 시작할 때 새로운 배열에서 dnaA 단백질에 의한 이중 개방.셀 52:743-55
- ^ 세키미즈 K, 브램힐 D 및 콘버그 A(1987) ATP는 E.coli 염색체의 기원을 가진 플라스미드의 복제를 개시할 때 dnaA 단백질을 활성화한다.셀, 50, 259–265
- ^ 고토오, 타가시라 Y.자연 DNA에서 자주 열리는 영역의 위치와 기능적 위치와의 관계.생체고분자 20:1043-58
- ^ 코왈스키 D, 에디 MJ 1989년DNA 풀림 요소: 대장균 복제 기원의 개방을 촉진하는 새로운 시스 작용 성분.EMBO J. 8:4335~44
- ^ 카 KM, 카구니 JM, 2001년대장균 염색체 기원의 시작에서 DnaA 및 DnaB 단백질의 스토이코메트리.J. Biol.약 276:44919~25
- ^ Tougu K, Marians KJ. 1996년헬리케이스와 프라이마아제 간의 상호작용은 복제 포크 클럭을 설정합니다.J. Biol.약 271:21398–405
- ^ 케언스, J.P:Cold Spring Harbor 심포지엄, 정량생물학 28:44, 1963.
- ^ 일어나, 1972년 R.G.서브틸리스 바실러스 내 재초기화된 염색체 시각화.제이몰 비올.7월 28일;68(3):501-9.
- ^ 1972년 쿰펠 경찰 프레스콧 D.M.대장균 염색체의 양방향 복제.Proc Natl Acad Sci US A. 10월;69(10) : 2842-5.
- ^ O'Donnell M., Jeruzalmi D., Kuriyan J. 클램프 로더 구조는 DNA 중합효소 III 홀로엔자임과 RFC의 아키텍처를 예측합니다.Curr. Biol. 11 R935-R946 2001
- ^ 인디애나 C, 오도넬 M베타 슬라이딩 클램프를 여는 델타 렌치의 메커니즘.J Biol Chem. 2003년 10월 10일;278(41):40272-81.2003년 7월 8일
- ^ a b Cox MM (1998). "A broadening view of recombinational DNA repair in bacteria". Genes Cells. 3 (2): 65–78. doi:10.1046/j.1365-2443.1998.00175.x. PMID 9605402.
- ^ Dugin IG, Wake RG, Bell SD, Hill TM. 2008.복제 포크 트랩 및 염색체 복제 종료.몰 마이크로바이올12월 70일 (6) : 1323 ~ 33일
- ^ 카마다 K, 호리우치 T, 오스미 K, 시마모토 N, 모리카와 K. 1996.DNA와 복합된 복제-종단 단백질 구조.자연, 17;383(6601) : 598~603.
- ^ 카플란 DL, 바스티아 D. 2009레플리케이션 포크의 폴라 어레지 메커니즘.몰 마이크로바이올 72:279-85
- ^ 크리스 울스퍼거와 니콜라스 코자렐리.대장균의 토포이소머라아제 IV와 DNA Gyrase의 효소 활성 비교.제271권, 제49호, 1996년 12월 6일호, 31549-31555페이지
- ^ E L 치히드리치, A B 호두르스키, N R 코자렐리자이라아제가 아닌 토포이소머라아제 IV는 대장균의 부위 특이적 재조합 산물을 분해한다.유전자 개발 1997년 10월 1일; 11일(19): 2580-92 9334322