산화인산화
Oxidative phosphorylation산화 인산화(영국 / /kˈsɪd.ə.tɪv/, US /ˈɑːk.s.ɪˌdeɪ.tɪv/ ) 또는 전자전달연계인산화 또는 말단 산화는 세포가 효소를 사용하여 영양소를 산화시켜 아데노신 3인산염(ATP)을 생성하기 위해 화학적 에너지를 방출하는 대사 경로다.진핵생물에서 이것은 미토콘드리아 안에서 일어난다.거의 모든 유산소 유기체는 산화 인산화 작용을 수행한다.이 길은 혐기성 글리콜리시스 같은 대체 발효 과정보다 더 많은 에너지를 방출하기 때문에(산소에 의해 제공됨)[2] 널리 퍼져 있다.
포도당의 화학적 결합에 저장된 에너지는 구연산 사이클에서 세포에 의해 방출되어 이산화탄소가 생성되며, 에너지 공여 전자공여자인 NADH와 FADH. 산화인산화효소화는 이러한 분자와 O를2 이용하여 에너지가 필요할 때마다 세포 전체에 사용되는 ATP를 생성한다.산화 인산화 동안 전자는 산소로 끝나는 일련의 리독스 반응에서 전자 기증자에서 일련의 전자 수용기로 전달되며, 이 반응으로 인해 전체 에너지의 절반이 방출된다.[3]
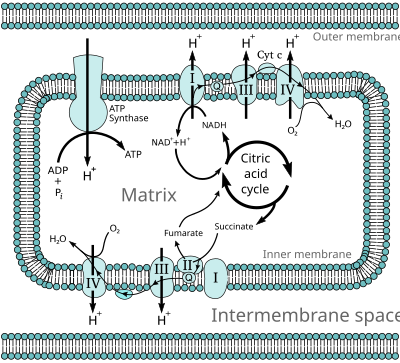
eukaryotes에서 이러한 redox 반응은 세포의 미토콘드리아 내막 내에 있는 일련의 단백질 복합체에 의해 촉매되는 반면, 원핵생물에서는 이러한 단백질들이 세포의 외막에 위치한다.이러한 연결된 단백질 세트를 전자 전송 체인이라고 부른다.eukaryotes에서는 5가지 주요 단백질 복합체가 관여하는 반면, prokaryotes에서는 다양한 전자 기증자와 수용자를 사용하여 많은 다양한 효소가 존재한다.
이 전자전달체인을 통해 흐르는 전자에 의해 전달되는 에너지는 전자전달이라 불리는 과정에서 내측 미토콘드리아 막을 가로질러 양자를 운반하는 데 사용된다.이것은 이 막에 걸쳐 pH 구배와 전기 전위의 형태로 전위 에너지를 생성한다.이 에너지 저장소는 양자가 체미오스메시스라고 불리는 과정에서 ATP synthase라는 큰 효소를 통해 막을 가로질러 잠재적 에너지 구배를 다시 흐를 때 두드려진다.ATP 싱타아제는 에너지를 사용하여 아데노신 디포스포산염(ADP)을 아데노신 트리포산염으로, 인산화 반응으로 변환한다.반응은 효소의 한 부분을 회전시키는 양성자 흐름에 의해 추진된다.ATP 싱타아제는 회전식 기계식 모터다.
산화인산화효소는 신진대사의 필수적인 부분이지만, 과산화수소나 과산화수소와 같은 반응성 산소종을 생성하는데, 이는 활성산소의 전파로 이어져 세포를 손상시키고 질병에 기여하며, 아마도 노화와 노화에 기여할 것이다.이러한 대사 경로를 수행하는 효소는 또한 활동을 억제하는 많은 약물과 독의 대상이기도 하다.
체미오스모스
산화 인산화 작용은 에너지를 방출하는 화학 반응을 이용하여 에너지를 필요로 하는 반응을 유도하는 작용을 한다.두 세트의 반응이 결합되어 있다고 한다.이것은 한 사람이 다른 사람 없이 일어날 수 없다는 것을 의미한다.NADH와 같은 전자 기증자에서 산소와 수소(프로톤)와 같은 전자 수용체에 이르는 전자 전송 체인을 통해 전자의 흐름을 주도하는 리독스 반응 체인은 외기 과정이다.[2] 에너지를 방출하는 반면, ATP의 합성은 내기 과정으로 에너지의 입력이 필요하다.전자전달체인과 ATP신분효소는 모두 막에 내장되어 있으며, 에너지는 이 막에 걸쳐 양자의 움직임에 의해 전자전달체인에서 ATP신분효소로 전달되어 체미오스미오시스라고 불리는 과정을 거친다.[4]양성자의 전류는 전자전달 체인의 양성자 점프 효소를 통해 막의 음의 N측에서 양의 P측으로 구동된다.양자의 움직임은 막 전체에 전기화학적 경사를 일으키는데, 이를 흔히 양성자 기력이라고 한다.양성자 농도 차이(H+ 그라데이션, ΔpH)와 전위차 등 두 가지 성분이 있으며, N측에는 음전하가 있다.[5]
ATP synthase는 회로를 완성하고 양자가 다시 막의 N측으로 전기화학적 경사로를 흐르게 함으로써 저장된 에너지를 방출한다.[6]전기화학적 구배는 효소 구조의 일부의 회전을 유도하고 이 운동을 ATP의 합성에 결합시킨다.
양성자의 두 구성 요소는 열역학적으로 동일하다.미토콘드리아에서, 에너지의 가장 큰 부분은 전위에 의해 제공된다; 알칼리필레 박테리아에서 전기 에너지는 역 pH 차이를 보상해야 한다.반대로 엽록체는 주로 ΔpH에 작용한다.그러나 ATP 합성의 운동학을 위한 작은 막 전위도 필요하다.후소박테리움 프로피오니게늄 모데르툼의 경우 ATP 싱타아제 F 모터의O 서브유닛 a와 c의 역회전을 유도한다.[5]
산화 인산화에 의해 방출되는 에너지의 양은 O의2 높은 에너지로 인해 혐기성 발효에 의해 생성되는 양에 비해 많다.[2]글리콜리시스(Glycolyisis)는 ATP 분자를 2개만 생산하지만, 어느 곳에서는 포도당 1개 분자를 이산화탄소와 물로 변환하여 만든 10 NADH와 2의 산화 인산화 분자에 의해 30~36 ATP가 생성되는 반면,[7] 지방산의 베타 산화 주기는 각각 약 14 ATP를 산출한다.이러한 ATP 수율은 이론적으로 최대값이며, 실제로 일부 양성자가 막을 가로질러 누출되어 ATP 수율이 낮아진다.[8]
전자 및 양성자 전달 분자
전자 전송 체인은 양자와 전자를 모두 운반하며, 기증자에서 수용자로 전자를 전달하고, 양자를 막으로 운반한다.이러한 과정은 수용성 및 단백질 결합 전달 분자를 모두 사용한다.미토콘드리아에서 전자는 수용성 전자전달 단백질 시토크롬 c에 의해 중간 공간 내에서 전달된다.[9]이것은 전자만을 운반하며, 이것들은 단백질이 그 구조에서 헤메 그룹 내에 가지고 있는 철 원자의 감소와 산화에 의해 전달된다.시토크롬 c는 또한 일부 박테리아에서 발견되는데, 그 박테리아는 경막 내에 위치해 있다.[10]
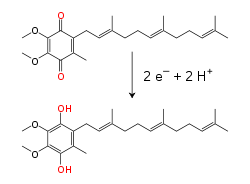
내측 미토콘드리아 멤브레인 내에서 지질 수용성 전자 캐리어 코엔자임 Q10(Q)은 전자와 양자를 모두 리독스 사이클로 운반한다.[11]이 작은 벤조퀴논 분자는 소수성이 매우 강해서 막 안에서 자유롭게 확산된다.Q가 전자 두 개와 양성자 두 개를 받아들이면 유비퀴놀 형태(QH2)로 축소되고, QH가2 전자 두 개와 양성자 두 개를 방출하면 유비퀴논 형태(Q)로 다시 산화된다.그 결과 2개의 효소를 배열하여 Q가 막의 한 면에 감소되고 QH가2 다른 면에 산화되도록 하면 유비퀴논은 이러한 반응과 멤브레인 전체에 걸쳐 셔틀 양자를 결합하게 된다.[12]어떤 박테리아 전자 운송 체인은 유비퀴논 외에도 메나퀴논과 같은 다른 퀴논을 사용한다.[13]
단백질 내에서 전자는 플라빈 코팩터,[6][14] 철-황반, 사이토크롬 사이에서 전달된다.철-황 군집에는 몇 가지 종류가 있다.전자전달망에서 발견되는 가장 단순한 종류는 무기유황의 두 원자와 결합된 두 개의 철 원자로 구성되어 있다. 이들을 [2Fe–2S] 군집이라고 한다.[4Fe–4S]라고 불리는 두 번째 종류는 4개의 철 원자와 4개의 유황 원자로 이루어진 입방체를 포함하고 있다.이들 군집의 각 철 원자는 추가 아미노산에 의해 조정되며, 보통 시스테인의 유황 원자에 의해 조정된다.금속 이온 공효자는 양성자를 결합하거나 방출하지 않고 리독스 반응을 겪기 때문에 전자전달망에서는 오로지 단백질을 통해 전자를 전달하는 역할을 한다.전자는 이러한 공분자의 사슬을 따라 깡충깡충 뛰면서 단백질을 통해 꽤 먼 거리를 이동한다.[15]이것은 1.4×10m−9 미만의 거리에서 빠른 양자 튜닝에 의해 발생한다.[16]
진핵 전자 전송 체인
글리콜리시스, 구연산 주기, 베타 산화 등과 같은 많은 카타볼릭 생화학적 공정은 코엔자임 NADH를 감소시킨다.이 코엔자임은 높은 전달 전위를 가진 전자를 포함하고 있다; 다시 말해서, 그들은 산화 시 많은 양의 에너지를 방출할 것이다.그러나 이것은 걷잡을 수 없는 반응이 될 것이기 때문에 세포는 이 에너지를 한꺼번에 방출하지 않는다.대신, 전자는 NADH에서 제거되고 각각의 효소가 소량의 에너지를 방출하는 일련의 효소를 통해 산소로 전달된다.복합체 I부터 IV까지로 구성된 이 효소 세트는 전자전달체인으로 불리며 미토콘드리온의 내막에서 발견된다.숙신 또한 전자 운송 체인에 의해 산화되지만 다른 지점에서 경로로 공급된다.
진핵생물에서 이 전자전달계통의 효소는 NADH에 의해 O에서2 방출된 에너지를 사용하여 미토콘드리온의 내막을 가로질러 양자를 펌프질한다.이것은 양자가 중간 공간에 쌓이게 하고, 막 전체에 전기화학적 구배를 발생시킨다.이 전위에 저장된 에너지는 ATP synthase에 의해 ATP를 생산하는데 사용된다.진핵 미토콘드리온의 산화 인산화는 이 과정에서 가장 잘 이해되는 예다.미토콘드리온은 거의 모든 진핵생물에 존재하는데, 대신 수소좀이라고 불리는 잔여 미토콘드리온에서 양성자를 수소로 줄이는 트리코모나스 질과 같은 혐기성 원생물을 제외한다.[17]
| 호흡효소 | 리독스 쌍 | 중간점 전위 (볼트) |
|---|---|---|
| NADH 탈수소효소 | NAD+ / NADH | −0.32[18] |
| 숙성탈수소효소 | FMN 또는 FAD/FMNH2 또는 FADH2 | −0.20[18] |
| 시토크롬 bc1 콤플렉스 | 코엔자임 Q10ox / 코엔자임 Q10red | +0.06[18] |
| 시토크롬 bc1 콤플렉스 | 사이토크롬ox bred / 사이토크롬 b | +0.12[18] |
| 콤플렉스 IV | 시토크롬ox cred / 시토크롬 c | +0.22[18] |
| 콤플렉스 IV | 사이토크롬ox ared / 사이토크롬 a | +0.29[18] |
| 콤플렉스 IV | O2 / HO− | +0.82[18] |
| 조건: pH = 7[18] | ||
NADH-코엔자임 Q 산화효소(복합 I)
NADH 탈수소효소 또는 복합 I로도 알려진 NADH-코엔자임 Q 산화효소는 전자전달체인의 첫 번째 단백질이다.[19]콤플렉스 I는 포유류 콤플렉스 I가 46개의 서브유닛과 약 1,000킬로달톤(kDa)의 분자량을 갖는 거대 효소다.[20]이 구조는 박테리아에서만 자세히 알려져 있다;[21][22] 대부분의 유기체에서 이 복합체는 막에서 미토콘드리온으로 튀어나온 커다란 "공"이 있는 부츠와 닮았다.[23][24]개별 단백질을 암호화하는 유전자는 세포핵과 미토콘드리아 게놈에 모두 들어 있는데, 미토콘드리아에 존재하는 많은 효소의 경우처럼 말이다.
이 효소에 의해 촉매되는 반응은 미토콘드리온 막에서 발견되는 지질 용해성 퀴논인 코엔자임 Q10 또는 유비퀴논(아래 방정식에서 Q로 표현)에 의한 NADH의 두 전자 산화다.
-
(1)
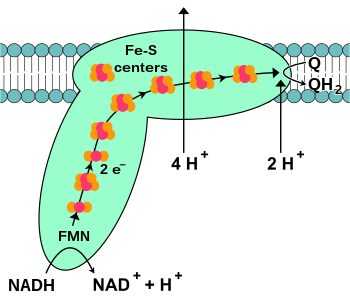
반응의 시작, 그리고 실제로 전체 전자 사슬의 시작은 복잡한 I에 대한 NADH 분자의 결합과 두 전자의 기증이다.전자는 복합 플라빈 모뉴클레오티드(FMN)에 부착된 보형물 그룹을 통해 복합 I로 들어간다.FMN에 전자를 추가하면 축소된 형태인 FMNH로2 변환된다. 그런 다음 전자는 일련의 철-황산 군집을 통해 전달된다. 즉, 복합체에 존재하는 두 번째 종류의 보철 그룹이다.[21]복합 I에는 [2Fe–2S] 및 [4Fe–4S] 철-황색 클러스터가 모두 있다.
전자가 이 콤플렉스를 통과할 때, 네 개의 양성자가 매트릭스에서 중간 공간으로 펌프된다.정확히 어떻게 이런 일이 일어나는지는 불분명하지만, 단백질이 막의 N측에서 양자를 묶어서 막의 P측에서 방출하게 하는 복합 I의 순응적인 변화를 수반하는 것 같다.[25]마지막으로, 전자는 철-황 군집의 체인에서 막 속의 유비퀴논 분자로 전달된다.[19]유비쿼시논의 감소는 또한 양성자 구배 생성에 기여하는데, 그것이 유비쿼시놀(QH2)로 줄어들면서 두 개의 양성자가 매트릭스에서 차지하기 때문이다.
숙신-Q 산화효소(복합 II)

복합 II 또는 숙성 탈수소효소라고도 알려진 숙신-Q 산화효소는 전자 운송 체인의 두 번째 진입점이다.[26]구연산 사이클과 전자 운반 체인의 일부인 유일한 효소이기 때문에 특이하다.콤플렉스 II는 4개의 단백질 서브유닛으로 구성되며 바운드 플라빈 아데닌 디뉴클레오티드(FAD) 공동 인자, 철-술푸르 클러스터, 그리고 코엔자임 Q로의 전자전달에는 참여하지 않지만 활성산소종의 생산을 줄이는 데 중요한 것으로 여겨진다.[27][28]그것은 후마린트에 굴복하여 유비쿼시논을 감소시킨다.이 반응은 NADH의 산화보다 적은 에너지를 방출하기 때문에 복합 II는 막을 가로질러 양자를 운반하지 않으며 양성자 구배에도 기여하지 않는다.
-
(2)
일부 진핵생물에서는 복합 II와 유사한 효소인 기생충 아스카리스수움, 후마레이트 환원효소(메나퀴놀:후마산산화효소, 또는 QFR)와 같이 역작용을 하여 유비쿼시놀을 산화시키고 후마레이트를 감소시킨다.이를 통해 전자수용기로서 후마산염과 함께 혐기성 산화 인산화를 수행하면서 대장의 혐기성 환경에서 벌레가 생존할 수 있게 된다.[29]복합 II의 또 다른 파격적인 기능은 말라리아 기생충 플라스모듐 팔시파룸에서 볼 수 있다.여기서 산화제로 복합 II의 역작용은 기생충이 특이한 형태의 피리미딘 생합성체로 사용하는 유비퀴놀의 재생에 중요하다.[30]
전자전달플라보프로테인Q산화효소
전자전달플라보프로테아인 탈수소효소라고도 알려진 전자전달플라보프로테아인-유비키논 산화효소(ETF-Q 산화효소)는 전자전달체인의 세 번째 진입점이다.미토콘드리아 매트릭스에서 전자전달 플라보프로테인으로부터 전자를 수용하는 효소로, 이러한 전자를 사용하여 유비쿼시논을 감소시킨다.[31]이 효소는 플라빈과 [4Fe–4S] 군집을 함유하고 있으나, 다른 호흡 복합체와 달리 막 표면에 부착되어 지질 빌레이어를 넘지 않는다.[32]
-
(3)
포유류에서 이 대사경로는 여러 아세틸-CoA 탈수소효소에서 나오는 전자를 수용하기 때문에 지방산의 베타 산화와 아미노산과 콜린의 카타볼리즘에 중요하다.[33][34]식물의 경우 ETF-Q 산화효소는 장기간 어둠 속에서 생존할 수 있는 대사 반응에서도 중요하다.[35]
Q-사이토크롬 c 산화효소(복합 III)
Q-사이토크롬 c 산화효소는 시토크롬 creducationctase, 시토크롬 bc1 complex 또는 단순히 complex III로도 알려져 있다.[36][37]포유류에서 이 효소는 조광기로서, 각 소단위 복합체가 11개의 단백질 서브유닛, [2Fe-2S] 철-황산군, 3개의 시토크롬(사이토크롬 c와1 2 b 사이토크롬)을 함유하고 있다.[38]시토크롬은 전자전달 단백질의 일종으로 적어도 1개의 헤미 그룹을 포함하고 있다.콤플렉스 III의 heme 그룹 내부의 철 원자는 전자가 단백질을 통해 전달될 때 감소된 철(+2)과 산화된 철(+3) 상태를 번갈아 본다.
콤플렉스 III에 의해 촉매되는 반응은 유비쿼시놀의 한 분자의 산화와 미토콘드리온과 느슨하게 연관된 헤메 단백질인 시토크롬 c의 두 분자의 감소다.두 개의 전자를 운반하는 코엔자임 Q와 달리 시토크롬 c는 한 개의 전자만 운반한다.
-
(4)
한 번에 1개의 전자만 QH2 기증자에서 사이토크롬 c 수용기로 전달될 수 있기 때문에 콤플렉스 III의 반응 메커니즘은 다른 호흡 복합체의 반응 메커니즘보다 정교하며 Q 사이클이라는 두 단계로 발생한다.[39]첫 번째 단계에서 효소는 세 개의 기판을 묶는데, 첫째, QH는2 산화된 다음, 하나의 전자가 두 번째 기질인 시토크롬 c로 전달된다.QH에서2 방출된 두 양자는 중간 공간으로 통과한다.세 번째 기질은 Q인데 QH로부터2 두 번째 전자를 받아 Q로.− 축소되는데, 이 기질은 유비시퀴논 프리 래디컬이다.처음 2개의 기판은 풀려나지만, 이 유비세미키논 중간 기종은 여전히 묶여 있다.두 번째 단계에서는 QH의2 두 번째 분자가 결합되어 다시 첫 번째 전자를 사이토크롬 c수용체에 전달한다.두 번째 전자는 바운드된 유비세미키논에 전달되어 미토콘드리아 매트릭스에서 두 개의 양성자를 얻으면서 QH로2 감소한다.이 QH는2 효소에서 방출된다.[40]
코엔자임 Q는 막 안쪽의 유비퀴놀로 감소하고 다른 한 쪽에서는 유비퀴논으로 산화되면서 막 전체에 걸쳐 양성자의 순전달이 일어나 양성자 구배를 더한다.[6]이것이 양성자 전달의 효율성을 증가시키므로, 이것이 발생하는 다소 복잡한 2단계 메커니즘이 중요하다.Q 사이클 대신 QH의2 1개 분자를 사용해 시토크롬 c의 2개 분자를 직접 감소시킨다면, 효율은 절반으로 떨어져 시토크롬 c당 1개의 양성자만 감소하게 된다.[6]
시토크롬 c산화효소(복합 IV)
콤플렉스 IV로도 알려진 사이토크롬 c산화효소는 전자전달체인의 최종 단백질 복합체다.[41]포유류 효소는 매우 복잡한 구조를 가지고 있으며 13개의 서브유닛, 2개의 헤미 그룹, 그리고 여러 개의 금속 이온 공효소를 포함하고 있다. 모두 구리 원자 3개, 마그네슘 원자 1개, 아연 원자를 포함하고 있다.[42]
이 효소는 전자전달체인의 최종반응을 매개하여 전자를 산소와 수소(프로톤)로 전달하면서 양자를 세포막으로 펌핑한다.[2][43]전자전달체인에 방출되는 에너지의 대부분을 공급하고 단자 전자수용체라고도 불리는 최종 전자수용체 산소는 이 단계에서 물로 환원되어 모든 에너지의 절반을 유산소호흡에서 방출하게 된다.[2]양자의 직접 펌핑과 산소 감소의 매트릭스 양성자의 소비 모두 양성자 구배에 기여한다.촉매반응은 시토크롬 c의 산화 및 산소 감소다.
-
(5)
대체 환원제 및 산화제
많은 진핵생물들은 위에서 설명한 많은 연구를 거친 포유류 효소와 다른 전자전달 체인을 가지고 있다.예를 들어, 식물은 대체 NADH 산화물을 가지고 있는데, 이것은 미토콘드리아 매트릭스가 아닌 시토솔에서 NADH를 산화시키고, 이러한 전자를 유비쿼터스니온 풀에 전달한다.[44]이러한 효소들은 양성자를 운반하지 않기 때문에 내막을 가로지르는 전기화학적 경사를 바꾸지 않고 유비쿼시논을 감소시킨다.[45]
다른 전자 운송 체인의 또 다른 예는 식물뿐만 아니라 일부 곰팡이, 양성자, 그리고 일부 동물에서도 발견되는 대체 산화제다.[46][47]이 효소는 전자를 유비쿼시놀에서 산소로 직접 전달한다.[48]
이러한 대체 NADH와 유비쿼시논 산화에 의해 생성된 전자전달 경로는 전체 경로보다 ATP 수율이 낮다.단축된 경로로 인한 이점이 완전히 명확하지는 않다.그러나 대체 산화효소는 냉간, 반응성 산소종, 병원균에 의한 감염과 같은 스트레스와 더불어 전자의 전달 체인을 억제하는 다른 요인에 반응하여 생성된다.[49][50]따라서 대체 경로는 산화 스트레스를 감소시킴으로써 유기체의 상해 저항력을 향상시킬 수 있다.[51]
복합체 구성
호흡 체인 콤플렉스가 어떻게 조직되는지에 대한 원래 모델은 미토콘드리아 막에서 자유롭고 독립적으로 확산되는 것이었다.[52]그러나 최근의 자료에 따르면 이 복합체들이 슈퍼콤플렉스 또는 "레스피라솜"[53]이라고 불리는 고차 구조를 형성할 수 있다고 한다.이 모델에서, 다양한 콤플렉스는 상호작용 효소의 조직화된 집합으로 존재한다.[54]이러한 연관성은 다양한 효소 복합체들 사이에 기질들의 채널링을 허용하여 전자 전달의 속도와 효율성을 증가시킬 수 있다.[55]이러한 포유류 복합체 내에서는 일부 구성 요소가 다른 구성 요소보다 더 많은 양으로 존재할 수 있으며, 일부 데이터는 복합체 I/II/[56]IV와 ATP 동기화효소 간의 비율을 나타내는 것으로 약 1:1:3:7:4그러나 일부 데이터가 이 모형에 맞지 않는 것처럼 보이기 때문에, 이 초복소 가설에 대한 논쟁이 완전히 해결된 것은 아니다.[20][57]
원핵전자전달망
진핵생물에서 전자전달 체인의 구조와 기능의 일반적인 유사성과 대조적으로, 박테리아와 고대는 많은 종류의 전자전달 효소를 가지고 있다.이것들은 기판과 마찬가지로 광범위한 화학물질을 사용한다.[58]진핵생물들과 공통적으로 원핵전자전달은 기질의 산화로부터 방출된 에너지를 사용하여 이온을 막에 걸쳐 펌프질하여 전기화학적 경사를 발생시킨다.이 박테리아에서 대장균의 산화 인산염은 가장 자세히 이해되는 반면, 고고학 시스템은 현재 잘 이해되지 않는다.[59]
진핵과 원핵 산화 인산화의 주요한 차이점은 박테리아와 고고학이 전자를 기증하거나 받아들이기 위해 많은 다른 물질을 사용한다는 것이다.이것은 원핵생물이 다양한 환경 조건 하에서 자랄 수 있게 해준다.[60]예를 들어 대장균에서 산화 인산화 작용은 아래에 열거된 다수의 환원제와 산화제 쌍에 의해 추진될 수 있다.화학 물질의 중간점 전위는 산화되거나 감소했을 때 방출되는 에너지의 양을 측정하는데, 음의 전위를 가지는 환원제와 양의 전위를 갖는 산화제가 있다.
| 호흡효소 | 리독스 쌍 | 중간점 전위 (볼트) |
|---|---|---|
| 포메이트탈수소효소 | 중탄산염 / 포메이트 | −0.43 |
| 수소효소 | 양성자 / 수소 | −0.42 |
| NADH 탈수소효소 | NAD+ / NADH | −0.32 |
| 글리세롤-3-인산탈수소효소 | DHAP / 글리-3-P | −0.19 |
| 피루베이트산화효소 | 아세테이트 + 이산화탄소 / 피루베이트 | ? |
| 젖산탈수소효소 | 피루베이트 / 젖산염 | −0.19 |
| 디아미노산탈수소효소 | 2-옥소산 + 암모니아 / D-아미노산 | ? |
| 포도당탈수소효소 | 글루콘산염 / 포도당 | −0.14 |
| 숙성탈수소효소 | 후마레이트 / 굴복 | +0.03 |
| 유비퀴놀산화효소 | 산소 / 물 | +0.82 |
| 질산 환원효소 | 질산염 / 질산염 | +0.42 |
| 아질산 환원효소 | 아질산염 / 암모니아 | +0.36 |
| 황산화디메틸 환원효소 | DMSO / DMS | +0.16 |
| 트리메틸아민 N산화 환원효소 | TMAO / TMA | +0.13 |
| 후마레이트 환원효소 | 후마레이트 / 굴복 | +0.03 |
위와 같이 대장균은 전자공여자로서 포메이트, 수소, 젖산 등의 감소제, 수용체로서의 질산염, DMSO 또는 산소와 함께 성장할 수 있다.[60]산화제와 환원제 사이의 중간점 전위차가 클수록 반응할 때 더 많은 에너지가 방출된다.이러한 화합물 중에서 중간점 전위가 0에 가깝기 때문에 항복/후마산 쌍은 특이하다.따라서 숙주는 산소 등 강한 산화제가 있으면 후마산염으로 산화할 수 있고, 후마산염은 포마이트와 같은 강한 환원제를 사용해 숙사할 수 있다.이러한 대체 반응은 각각 숙성된 탈수소효소와 후마레이트 환원효소에 의해 촉매로 작용한다.[62]
일부 원핵생물들은 중간점 전위차이가 작은 redox 쌍을 사용한다.예를 들어, 니트로박터 같은 질화 박테리아는 질산염을 산화시켜 산소에 전자를 기증한다.이 반응에서 방출되는 소량의 에너지는 양성자를 펌프질하고 ATP를 발생시키기에 충분하지만, 아나볼리즘에 사용하기 위해 NADH나 NADPH를 직접 생산하기에는 충분하지 않다.[63]이 문제는 질산염 산화효소를 사용하여 전자 운송 체인의 일부를 역주행할 수 있는 충분한 양성자를 생산함으로써 해결되어 복합 I가 NADH를 발생시킨다.[64][65]
원핵생물들은 환경 조건에 따라 어떤 효소가 생산되는지를 변화시킴으로써 이러한 전자 기증자와 수용자의 사용을 통제한다.[66]다른 산화물과 환원제가 동일한 유비쿼터스니온 풀을 사용하기 때문에 이러한 유연성이 가능하다.이것은 많은 효소의 조합이 공통의 유비퀴놀 중간재에 의해 연계되어 함께 기능할 수 있게 해준다.[61]그러므로 이러한 호흡 체인은 쉽게 교환할 수 있는 효소 계통 세트를 가진 모듈식 설계를 가지고 있다.
이러한 신진대사 다양성 외에도 원핵생물은 다양한 이소자임(동일한 반응을 촉진하는 다른 효소)을 가지고 있다.예를 들어 대장균에는 산소를 전자수용체로 사용하는 유비쿼터스놀 산화효소가 두 가지 있다.높은 유산소 조건에서, 세포는 전자당 두 개의 양성자를 운반할 수 있는 산소 친화력이 낮은 산화제를 사용한다.그러나 산소 농도가 떨어지면 전자당 1개의 양성자만 전달하는 산화제로 전환되지만 산소에 대한 친화력은 높다.[67]
ATP synthase(복합
복합 V라고도 불리는 ATP 싱타아제는 산화인산화 경로의 최종 효소다.이 효소는 모든 형태의 생명체에서 발견되며 원핵생물과 진핵생물의 동일한 방식으로 기능한다.[68]효소는 ADP와 인산염(Pi)의 ATP 합성을 추진하기 위해 막을 가로질러 양성자 구배 속에 저장된 에너지를 사용한다.1개의 ATP를 합성하는 데 필요한 양성자 수의 추정치는 3개에서 4개까지 다양하며,[69][70] 일부 셀은 다른 조건에 맞게 이 비율을 변경할 수 있다고 제안한다.[71]
-
(6)
이 인산화 반응은 평형인데, 양성자-기동력을 변화시킴으로써 움직일 수 있다.양성자가 없는 경우 ATP 싱타아제 반응은 오른쪽에서 왼쪽으로 진행되어 ATP를 가수분해하고 양성자를 멤브레인 전체에 걸쳐 매트릭스에서 펌핑한다.그러나 양성자가 높을 때는 반대 방향으로 반응할 수밖에 없다. 양자가 농도 구배를 따라 흐르게 하고 ADP를 ATP로 변화시킨다.[68]실제로 밀접하게 연관된 vacuolar 타입 H+-ATPases에서 가수분해 반응은 양성자를 펌프질하고 ATP를 가수분해하여 세포 구획을 산성화하는 데 사용된다.[72]
ATP 신타아제는 버섯 모양의 거대한 단백질 복합체다.포유류 효소 복합체는 16개의 서브유닛을 포함하고 있으며 질량은 약 600킬로달톤이다.[73]멤브레인 안에 내장된 부분을 F라고O 하며 c 서브유닛과 양성자 채널의 링을 포함한다.줄기와 공 모양의 헤드피스는 F라고1 불리며 ATP 합성의 현장이다.F1 부분의 끝에 있는 공 모양의 콤플렉스는 두 종류의 서로 다른 단백질(α 서브유닛 3개와 β 서브유닛 3개) 6개를 포함하고 있는 반면, '스토크'는 하나의 단백질인 γ 서브유닛으로 구성되며, 줄기의 끝이 α와 β 서브유닛의 볼로 확장된다.[74]α와 β 서브유닛은 모두 뉴클레오티드를 결합하지만, β 서브유닛만이 ATP 합성 반응을 촉매한다.F1 부분의 측면을 따라 다시 막 속으로 도달하는 것은 α와 β 서브유닛을 효소의 기저부에 고정시키는 긴 막대형 서브유닛이다.
양자가 ATP 싱타아제 베이스의 채널을 통해 막을 가로지르면서 FO 양성자 구동 모터가 회전한다.[75]회전은 c 서브유닛의 링에서 아미노산의 이온화가 변화하여 c 서브유닛의 링이 양성자 채널을 지나도록 밀어내는 정전기적 상호작용을 야기할 수 있다.[76]이 회전 링은 차례로 α 및 β 서브유닛 내에서 중심 차축(γ 서브유닛 레버)의 회전을 구동한다.α와 β 서브유닛은 스테이터 역할을 하는 사이드 암에 의해 스스로 회전하는 것을 방지한다.α 및 β 서브유닛의 볼 안에서 γ 서브유닛의 팁의 이러한 움직임은 β 서브유닛의 활성 부위가 ATP를 생성했다가 방출하는 이동의 주기를 거치는 에너지를 제공한다.[77]

이 ATP 합성 반응은 결합 변화 메커니즘이라고 불리며 세 상태 사이에서 β 서브유닛 사이클의 활성 현장이 포함된다.[78]"개방" 상태에서 ADP와 인산염은 활성 사이트(도면에 갈색으로 표시)로 들어간다.그런 다음 단백질은 분자 주위를 감싸고 느슨하게 묶는다 – "느슨한" 상태(빨간색으로 표시됨).그러면 효소는 다시 모양을 바꾸어 이 분자들을 함께 강제하게 되는데, 그 결과 "긴장" 상태에서의 활성 부위는 매우 높은 친화력으로 새로 생성된 ATP 분자를 결합시킨다.마지막으로 활성 사이트는 다시 개방 상태로 순환하며, ATP를 해제하고 더 많은 ADP와 인산염을 바인딩하여 다음 사이클을 준비한다.
일부 박테리아와 고고학에서 ATP 합성은 양자의 움직임보다는 세포막을 통한 나트륨 이온의 움직임에 의해 추진된다.[79][80]Methanococcus와 같은 고세아에는 다른 박테리아와 진핵 ATP 싱타아제 서브유닛과 순서에 따라 유사성이 거의 없는 추가 단백질을 포함하는 효소의 한 형태인 AA1o 싱타아제도 들어 있다.일부 종에서는 효소의 AA1o 형태가 나트륨에 의한 특화된 ATP 싱타아제일 수도 있지만,[81] 이것은 모든 경우에 사실이 아닐 수도 있다.[80]
산화인산화 - 정력제
산화 인산화에서 방출되는 에너지는 대부분 상대적으로 약한 이중 결합을 가진2 O에 기인할 수 있다.[2]redox 쌍 NAD+/NADH에서 최종 redox 쌍 1/22 O/ HO까지의2 전자의 전달은 다음과 같이 요약할 수 있다.
1/2 O2 + NADH + H → HO+2 + NAD+
이 두 레독스 쌍 사이의 전위차는 1.14볼트로 O의2 6몰당 -52kcal/mol 또는 -2600kJ에 해당한다.
1 NADH가 전자전달 체인을 통해 산화되면 3개의 ATP가 생성되는데, 이는 7.3kcal/mol x 3 = 21.9kcal/mol에 해당한다.
에너지의 보존은 다음의 공식으로 계산할 수 있다.
효율성 = (21.9 x 100%) / 52 = 42%
따라서 NADH가 산화되면 에너지의 약 42%가 3개의 ATP 형태로 보존되고 나머지(58%) 에너지는 열로 손실된다고 결론을 내릴 수 있다(생리학적 조건에서 ATP의 화학적 에너지를 과소평가하지 않은 경우).
활성산소종
분자 산소는 강한 산화제이기 때문에 이상적인 단자 전자 수용체다.산소의 감소는 잠재적으로 해로운 매개체를 포함한다.[82]전자 4개와 양성자 4개를 전달하면 산소가 물로 감소해 무해하지만 1~2개의 전자가 전달되면 과산화물이나 과산화물 음이온이 생성돼 위험하게 반응한다.
-
(7)
이러한 활성 산소종과 히드록실 라디칼 과 같은 반응성 산소는 단백질을 산화시키고 DNA에 돌연변이를 일으키기 때문에 세포에 매우 해롭다.이러한 세포 손상은 질병의 원인이 될 수 있으며 노화의 한 원인으로 제시된다.[83][84]
시토크롬 c산화효소 복합체는 물로 산소를 줄이는 데 매우 효율적이며 부분적으로 감소된 중간체를 거의 방출하지 않는다. 그러나 소량의 과산화수소 음이온과 과산화수소는 전자 운송 체인에 의해 생성된다.[85]특히 중요한 것은 콤플렉스 III에서 코엔자임 Q의 감소인데, Q 사이클의 중간으로서 고반응성 유비시퀴논 프리 래디컬이 형성되기 때문이다.이 불안정한 종은 전자가 산소로 직접 전달되어 과산화수소를 형성할 때 전자 "유출"로 이어질 수 있다.[86]이러한 양성자 점프 콤플렉스에 의한 활성산소 종의 생산이 높은 막 전위대에서 가장 크기 때문에, 미토콘드리아는 산화제 생성과 균형을 이루는 ATP 생산의 좁은 범위 내에서 막 전위를 유지하기 위해 활동을 조절할 것을 제안했다.[87]예를 들어, 산화제는 막 전위를 감소시키는 분리되지 않은 단백질을 활성화시킬 수 있다.[88]
이러한 활성산소 종에 대항하기 위해 세포에는 비타민C, 비타민E 등의 항산화 비타민과 반응성 종을 해독하는 [82]과산화디푸타아제, 카탈라아제, 과산화효소 등의 항산화 효소가 많이 들어 있어 세포의 손상을 제한한다.
저산소 조건에서의 산화 인산화
산소는 산화 인산염에 기본이기 때문에 O레벨의2 부족은 ATP 생산률을 변화시킬 가능성이 있다.그러나 양성자 동력과 ATP 생산은 세포내 산성화에 의해 유지될 수 있다.[89]ATP 가수분해와 젖산증 등으로 축적된 세포질 양성자는 미토콘드리아 외측엽에 자유롭게 확산되어 기억간 공간을 산성화할 수 있어 양성자 동력과 ATP 생산에 직접적인 기여를 한다.
억제제
산화 인산화를 억제하는 몇 가지 잘 알려진 약물과 독소가 있다.비록 이러한 독소들 중 어떤 것이든 전자 운송 체인의 한 효소만을 억제하지만, 이 과정에서 어떤 단계도 억제하면 나머지 과정이 중단될 것이다.예를 들어 올리고마이신이 ATP 싱타제를 억제하면 양성자는 미토콘드리온으로 되돌아갈 수 없다.[90]그 결과 양자 펌프는 경사가 너무 강해 극복하지 못하기 때문에 작동할 수 없게 된다.NADH는 더 이상 산화되지 않고 구연산 주기가 작동을 멈춘다. NAD의+ 농도가 이들 효소가 사용할 수 있는 농도 아래로 떨어지기 때문이다.
전자 전송 체인의 많은 현장 고유 억제제들은 미토콘드리아 호흡에 대한 현재의 지식에 기여했다.ATP의 합성도 전자 전송 체인에 의존하므로 모든 현장 고유 억제제 역시 ATP 형성을 억제한다.생선독 로테논, 바비투레이트 약 아밀턴, 항생제 피에리딘 A는 NADH와 코엔자임 Q를 억제한다.[91]
일산화탄소, 청산가리, 황화수소, 아지드화물은 시토크롬 산화효소를 효과적으로 억제한다.일산화탄소는 시토크롬의 감소된 형태와 반응하는 반면 청산가리와 아지드화물은 산화된 형태와 반응한다.항생제, 안티마이신A, 화학무기에 사용되는 해독제인 영국 항레시테는 사이토크롬 B와 C1 사이에 있는 부지의 두 가지 중요한 억제제다.[91]
| 화합물 | 사용하다 | 활동지 | 산화 인산화 효과 |
|---|---|---|---|
| 시안화 일산화탄소 아지드 황화수소 | 독 | 콤플렉스 IV | 시토크롬 c산화효소의 Fe-Cu 중심부에 산소보다 더 강하게 결합하여 산소의 감소를 방지함으로써 전자 운송 체인을 억제한다.[92] |
| 올리고마이신 | 항생제 | 콤플렉스 V | Fo 서브유닛을 통한 양자의 흐름을 차단하여 ATP 싱타아제를 억제한다.[90] |
| CCCP 2,4-디니트로페놀 | 독, 체중[N 1] 감소 | 내막 | 막을 가로질러 양자를 운반하여 양성자 구배를 방해하는 이오노포어.이 이오노포레는 내부 미토콘드리아 막을 가로질러 양자를 운반하기 때문에 ATP 합성으로부터 양자를 분리한다.[93] |
| 로테논 | 살충제 | 콤플렉스 I | 유비쿼시논 결합 부지를 차단하여 복합 I에서 유비쿼시논으로 전자가 전달되는 것을 방지한다.[94] |
| 말론산염과 옥살로아세테이트 | 독 | 콤플렉스 II | 숙성 탈수소효소에 대한 경쟁적 억제제(복제 II).[95] |
| 안티마이신 A | 피스케살 | 콤플렉스 III | 시토크롬 환원효소의 Qi 부지에 결합하여 유비쿼시놀의 산화를 억제한다. |
산화인산화 억제제가 모두 독소인 것은 아니다.갈색 지방 조직에서, 분리 단백질이라고 불리는 조절된 양성자 채널은 ATP 합성으로부터 호흡을 분리할 수 있다.[96]비록 이러한 단백질들이 스트레스에 대한 세포들의 반응에 더 일반적인 기능을 가지고 있을지 모르지만, 이 빠른 호흡은 열을 발생시키고, 특히 동면하는 동물들을 위해 체온을 유지하는 방법으로 중요하다.[97]
역사
산화 인산염 분야는 1906년 세포 발효에 인산염의 중요한 역할을 한 아서 하든의 보고로 시작됐지만, 처음에는 설탕 인산염만 관여하는 것으로 알려졌다.[98]그러나 1940년대 초에는 허먼 칼카르에 의해 당분 산화와 ATP 발생의 연계가 확고히 정착되어 1941년 프리츠 알버트 립만이 제안했던 에너지 전달에서 ATP의 중심적 역할을 확인하게 되었다.[99][100]이후 1949년에 모리스 프리드킨과 앨버트 L. 레닝거는 코엔자임 NADH가 구연산 사이클과 ATP 합성 같은 대사 경로를 연결시켰다는 것을 증명했다.[101]산화 인산화라는 용어는 1939년 Volodymyr Belitser에 의해 만들어졌다.[102][103]
또 다른 20년 동안 ATP가 생성되는 메커니즘은 신비한 상태로 남아 있었으며, 과학자들은 산화 반응과 인산화 반응을 연결할 수 있는 이해하기 어려운 "고에너지 중간"을 찾고 있었다.[104]이 퍼즐은 피터 D에 의해 풀렸다. 미첼은 1961년에 체미오스테틱 이론을 출판했다.[105]처음에는 이 제안이 크게 논란이 되었지만 서서히 받아들여져 1978년 미첼이 노벨상을 받았다.[106][107]이후 연구는 데이비드 E가 주요 기여를 하는 등 관련 효소를 정화하고 특성화하는 데 초점을 맞췄다. 전자-트랜스포트 체인의 복합체에 녹색, ATP 싱타아제의 에프레임 라커.[108]ATP synthase의 메커니즘을 해결하기 위한 중요한 단계는 Paul D에 의해 제공되었다. 보이어는 1973년에 "구속적 변화" 메커니즘을 개발하면서 1982년에 그의 급진적인 회전 촉매제 제안이 이어졌다.[78][109]보다 최근의 연구는 존 E에 의한 산화 인산화에 관련된 효소에 대한 구조적 연구를 포함하고 있다. 워커, 워커와 보이어는 1997년에 노벨상을 받았다.[110]
참고 항목
메모들
- ^ DNP는 1930년대에 비만 방지 약으로 널리 사용되었으나, 위험한 부작용 때문에 결국 중단되었다.하지만, 이런 목적을 위한 약의 불법 사용은 오늘날에도 계속되고 있다.자세한 내용은 2,4-Dinitrophenol#Dieting 도움말을 참조하십시오.
참조
- ^ "oxidative Meaning in the Cambridge English Dictionary". dictionary.cambridge.org. Archived from the original on 24 January 2018. Retrieved 28 April 2018.
- ^ a b c d e f Schmidt-Rohr K (2020). "Oxygen Is the High-Energy Molecule Powering Complex Multicellular Life: Fundamental Corrections to Traditional Bioenergetics". ACS Omega. 5 (5): 2221–2233. doi:10.1021/acsomega.9b03352. PMC 7016920. PMID 32064383.
- ^ Voet, D.; Voet, J. G. (2004)"생물화학" 3부 804쪽 와일리ISBN 0-471-19350-X.
- ^ Mitchell P, Moyle J (1967). "Chemiosmotic hypothesis of oxidative phosphorylation". Nature. 213 (5072): 137–9. Bibcode:1967Natur.213..137M. doi:10.1038/213137a0. PMID 4291593. S2CID 4149605.
- ^ a b Dimroth P, Kaim G, Matthey U (1 January 2000). "Crucial role of the membrane potential for ATP synthesis by F(1)F(o) ATP synthases". J. Exp. Biol. 203 (Pt 1): 51–9. doi:10.1242/jeb.203.1.51. PMID 10600673. Archived from the original on 30 September 2007.
- ^ a b c d Schultz BE, Chan SI (2001). "Structures and proton-pumping strategies of mitochondrial respiratory enzymes" (PDF). Annu Rev Biophys Biomol Struct. 30: 23–65. doi:10.1146/annurev.biophys.30.1.23. PMID 11340051.
- ^ Rich PR (2003). "The molecular machinery of Keilin's respiratory chain". Biochem. Soc. Trans. 31 (Pt 6): 1095–105. doi:10.1042/bst0311095. PMID 14641005.
- ^ Porter RK, Brand MD (1995). "Mitochondrial proton conductance and H+/O ratio are independent of electron transport rate in isolated hepatocytes". Biochem. J. 310 (Pt 2): 379–82. doi:10.1042/bj3100379. PMC 1135905. PMID 7654171.
- ^ Mathews FS (1985). "The structure, function and evolution of cytochromes". Prog. Biophys. Mol. Biol. 45 (1): 1–56. doi:10.1016/0079-6107(85)90004-5. PMID 3881803.
- ^ Wood PM (1983). "Why do c-type cytochromes exist?". FEBS Lett. 164 (2): 223–6. doi:10.1016/0014-5793(83)80289-0. PMID 6317447. S2CID 7685958.
- ^ Crane FL (1 December 2001). "Biochemical functions of coenzyme Q10". J Am Coll Nutr. 20 (6): 591–8. doi:10.1080/07315724.2001.10719063. PMID 11771674. S2CID 28013583.
- ^ Mitchell P (1979). "Keilin's respiratory chain concept and its chemiosmotic consequences". Science. 206 (4423): 1148–59. Bibcode:1979Sci...206.1148M. doi:10.1126/science.388618. PMID 388618.
- ^ Søballe B, Poole RK (1999). "Microbial ubiquinones: multiple roles in respiration, gene regulation and oxidative stress management" (PDF). Microbiology. 145 (8): 1817–30. doi:10.1099/13500872-145-8-1817. PMID 10463148. Archived (PDF) from the original on 2008-05-29.
- ^ Johnson DC, Dean DR, Smith AD, Johnson MK (2005). "Structure, function, and formation of biological iron-sulfur clusters". Annu. Rev. Biochem. 74: 247–81. doi:10.1146/annurev.biochem.74.082803.133518. PMID 15952888.
- ^ Page CC, Moser CC, Chen X, Dutton PL (1999). "Natural engineering principles of electron tunnelling in biological oxidation-reduction". Nature. 402 (6757): 47–52. Bibcode:1999Natur.402...47P. doi:10.1038/46972. PMID 10573417. S2CID 4431405.
- ^ Leys D, Scrutton NS (2004). "Electrical circuitry in biology: emerging principles from protein structure". Curr. Opin. Struct. Biol. 14 (6): 642–7. doi:10.1016/j.sbi.2004.10.002. PMID 15582386.
- ^ Boxma B, de Graaf RM, van der Staay GW, van Alen TA, Ricard G, Gabaldón T, van Hoek AH, Moon-van der Staay SY, Koopman WJ, van Hellemond JJ, Tielens AG, Friedrich T, Veenhuis M, Huynen MA, Hackstein JH (2005). "An anaerobic mitochondrion that produces hydrogen" (PDF). Nature. 434 (7029): 74–9. Bibcode:2005Natur.434...74B. doi:10.1038/nature03343. PMID 15744302. S2CID 4401178.
- ^ a b c d e f g h 메디컬 케미컬 컴펜디엄.앤더스 오버가어드 페더슨과 헤닝 닐슨.오르후스 대학교.2008
- ^ a b Hirst J (2005). "Energy transduction by respiratory complex I--an evaluation of current knowledge". Biochem. Soc. Trans. 33 (Pt 3): 525–9. doi:10.1042/BST0330525. PMID 15916556.
- ^ a b Lenaz G, Fato R, Genova ML, Bergamini C, Bianchi C, Biondi A (2006). "Mitochondrial Complex I: structural and functional aspects". Biochim. Biophys. Acta. 1757 (9–10): 1406–20. doi:10.1016/j.bbabio.2006.05.007. PMID 16828051.
- ^ a b Sazanov LA, Hinchliffe P (2006). "Structure of the hydrophilic domain of respiratory complex I from Thermus thermophilus". Science. 311 (5766): 1430–6. Bibcode:2006Sci...311.1430S. doi:10.1126/science.1123809. PMID 16469879. S2CID 1892332.
- ^ Efremov RG, Baradaran R, Sazanov LA (May 2010). "The architecture of respiratory complex I". Nature. 465 (7297): 441–5. Bibcode:2010Natur.465..441E. doi:10.1038/nature09066. PMID 20505720. S2CID 4372778.
- ^ Baranova EA, Holt PJ, Sazanov LA (2007). "Projection structure of the membrane domain of Escherichia coli respiratory complex I at 8 A resolution". J. Mol. Biol. 366 (1): 140–54. doi:10.1016/j.jmb.2006.11.026. PMID 17157874.
- ^ Friedrich T, Böttcher B (2004). "The gross structure of the respiratory complex I: a Lego System". Biochim. Biophys. Acta. 1608 (1): 1–9. doi:10.1016/j.bbabio.2003.10.002. PMID 14741580.
- ^ Hirst J (January 2010). "Towards the molecular mechanism of respiratory complex I". Biochem. J. 425 (2): 327–39. doi:10.1042/BJ20091382. PMID 20025615.
- ^ Cecchini G (2003). "Function and structure of complex II of the respiratory chain". Annu. Rev. Biochem. 72: 77–109. doi:10.1146/annurev.biochem.72.121801.161700. PMID 14527321.
- ^ Yankovskaya V, Horsefield R, Törnroth S, Luna-Chavez C, Miyoshi H, Léger C, Byrne B, Cecchini G, Iwata S, et al. (2003). "Architecture of succinate dehydrogenase and reactive oxygen species generation". Science. 299 (5607): 700–4. Bibcode:2003Sci...299..700Y. doi:10.1126/science.1079605. PMID 12560550. S2CID 29222766.
- ^ Horsefield R, Iwata S, Byrne B (2004). "Complex II from a structural perspective". Curr. Protein Pept. Sci. 5 (2): 107–18. doi:10.2174/1389203043486847. PMID 15078221.
- ^ Kita K, Hirawake H, Miyadera H, Amino H, Takeo S (2002). "Role of complex II in anaerobic respiration of the parasite mitochondria from Ascaris suum and Plasmodium falciparum". Biochim. Biophys. Acta. 1553 (1–2): 123–39. doi:10.1016/S0005-2728(01)00237-7. PMID 11803022.
- ^ Painter HJ, Morrisey JM, Mather MW, Vaidya AB (2007). "Specific role of mitochondrial electron transport in blood-stage Plasmodium falciparum". Nature. 446 (7131): 88–91. Bibcode:2007Natur.446...88P. doi:10.1038/nature05572. PMID 17330044. S2CID 4421676.
- ^ Ramsay RR, Steenkamp DJ, Husain M (1987). "Reactions of electron-transfer flavoprotein and electron-transfer flavoprotein: ubiquinone oxidoreductase". Biochem. J. 241 (3): 883–92. doi:10.1042/bj2410883. PMC 1147643. PMID 3593226.
- ^ Zhang J, Frerman FE, Kim JJ (2006). "Structure of electron transfer flavoprotein-ubiquinone oxidoreductase and electron transfer to the mitochondrial ubiquinone pool". Proc. Natl. Acad. Sci. U.S.A. 103 (44): 16212–7. Bibcode:2006PNAS..10316212Z. doi:10.1073/pnas.0604567103. PMC 1637562. PMID 17050691.
- ^ Ikeda Y, Dabrowski C, Tanaka K (25 January 1983). "Separation and properties of five distinct acyl-CoA dehydrogenases from rat liver mitochondria. Identification of a new 2-methyl branched chain acyl-CoA dehydrogenase". J. Biol. Chem. 258 (2): 1066–76. doi:10.1016/S0021-9258(18)33160-0. PMID 6401712. Archived from the original on 29 September 2007.
- ^ Ruzicka FJ, Beinert H (1977). "A new iron-sulfur flavoprotein of the respiratory chain. A component of the fatty acid beta oxidation pathway" (PDF). J. Biol. Chem. 252 (23): 8440–5. doi:10.1016/S0021-9258(19)75238-7. PMID 925004. Archived (PDF) from the original on 2007-09-27.
- ^ Ishizaki K, Larson TR, Schauer N, Fernie AR, Graham IA, Leaver CJ (2005). "The critical role of Arabidopsis electron-transfer flavoprotein:ubiquinone oxidoreductase during dark-induced starvation". Plant Cell. 17 (9): 2587–600. doi:10.1105/tpc.105.035162. PMC 1197437. PMID 16055629.
- ^ Berry EA, Guergova-Kuras M, Huang LS, Crofts AR (2000). "Structure and function of cytochrome bc complexes" (PDF). Annu. Rev. Biochem. 69: 1005–75. CiteSeerX 10.1.1.319.5709. doi:10.1146/annurev.biochem.69.1.1005. PMID 10966481. Archived (PDF) from the original on 2015-12-28.
- ^ Crofts AR (2004). "The cytochrome bc1 complex: function in the context of structure". Annu. Rev. Physiol. 66: 689–733. doi:10.1146/annurev.physiol.66.032102.150251. PMID 14977419.
- ^ Iwata S, Lee JW, Okada K, Lee JK, Iwata M, Rasmussen B, Link TA, Ramaswamy S, Jap BK (1998). "Complete structure of the 11-subunit bovine mitochondrial cytochrome bc1 complex". Science. 281 (5373): 64–71. Bibcode:1998Sci...281...64I. doi:10.1126/science.281.5373.64. PMID 9651245.
- ^ Trumpower BL (1990). "The protonmotive Q cycle. Energy transduction by coupling of proton translocation to electron transfer by the cytochrome bc1 complex" (PDF). J. Biol. Chem. 265 (20): 11409–12. doi:10.1016/S0021-9258(19)38410-8. PMID 2164001. Archived (PDF) from the original on 2007-09-27.
- ^ Hunte C, Palsdottir H, Trumpower BL (2003). "Protonmotive pathways and mechanisms in the cytochrome bc1 complex". FEBS Lett. 545 (1): 39–46. doi:10.1016/S0014-5793(03)00391-0. PMID 12788490. S2CID 13942619.
- ^ Calhoun MW, Thomas JW, Gennis RB (1994). "The cytochrome oxidase superfamily of redox-driven proton pumps". Trends Biochem. Sci. 19 (8): 325–30. doi:10.1016/0968-0004(94)90071-X. PMID 7940677.
- ^ Tsukihara T, Aoyama H, Yamashita E, Tomizaki T, Yamaguchi H, Shinzawa-Itoh K, Nakashima R, Yaono R, Yoshikawa S (1996). "The whole structure of the 13-subunit oxidized cytochrome c oxidase at 2.8 A". Science. 272 (5265): 1136–44. Bibcode:1996Sci...272.1136T. doi:10.1126/science.272.5265.1136. PMID 8638158. S2CID 20860573.
- ^ Yoshikawa S, Muramoto K, Shinzawa-Itoh K, Aoyama H, Tsukihara T, Shimokata K, Katayama Y, Shimada H (2006). "Proton pumping mechanism of bovine heart cytochrome c oxidase". Biochim. Biophys. Acta. 1757 (9–10): 1110–6. doi:10.1016/j.bbabio.2006.06.004. PMID 16904626.
- ^ Rasmusson AG, Soole KL, Elthon TE (2004). "Alternative NAD(P)H dehydrogenases of plant mitochondria". Annu Rev Plant Biol. 55: 23–39. doi:10.1146/annurev.arplant.55.031903.141720. PMID 15725055.
- ^ Menz RI, Day DA (1996). "Purification and characterization of a 43-kDa rotenone-insensitive NADH dehydrogenase from plant mitochondria". J. Biol. Chem. 271 (38): 23117–20. doi:10.1074/jbc.271.38.23117. PMID 8798503. S2CID 893754.
- ^ McDonald A, Vanlerberghe G (2004). "Branched mitochondrial electron transport in the Animalia: presence of alternative oxidase in several animal phyla". IUBMB Life. 56 (6): 333–41. doi:10.1080/1521-6540400000876. PMID 15370881.
- ^ Sluse FE, Jarmuszkiewicz W (1998). "Alternative oxidase in the branched mitochondrial respiratory network: an overview on structure, function, regulation, and role". Braz. J. Med. Biol. Res. 31 (6): 733–47. doi:10.1590/S0100-879X1998000600003. PMID 9698817.
- ^ Moore AL, Siedow JN (1991). "The regulation and nature of the cyanide-resistant alternative oxidase of plant mitochondria". Biochim. Biophys. Acta. 1059 (2): 121–40. doi:10.1016/S0005-2728(05)80197-5. PMID 1883834.
- ^ Vanlerberghe GC, McIntosh L (1997). "ALTERNATIVE OXIDASE: From Gene to Function". Annu. Rev. Plant Physiol. Plant Mol. Biol. 48: 703–734. doi:10.1146/annurev.arplant.48.1.703. PMID 15012279.
- ^ Ito Y, Saisho D, Nakazono M, Tsutsumi N, Hirai A (1997). "Transcript levels of tandem-arranged alternative oxidase genes in rice are increased by low temperature". Gene. 203 (2): 121–9. doi:10.1016/S0378-1119(97)00502-7. PMID 9426242.
- ^ Maxwell DP, Wang Y, McIntosh L (1999). "The alternative oxidase lowers mitochondrial reactive oxygen production in plant cells". Proc. Natl. Acad. Sci. U.S.A. 96 (14): 8271–6. Bibcode:1999PNAS...96.8271M. doi:10.1073/pnas.96.14.8271. PMC 22224. PMID 10393984.
- ^ Lenaz G (2001). "A critical appraisal of the mitochondrial coenzyme Q pool". FEBS Lett. 509 (2): 151–5. doi:10.1016/S0014-5793(01)03172-6. PMID 11741580. S2CID 46138989.
- ^ Heinemeyer J, Braun HP, Boekema EJ, Kouril R (2007). "A structural model of the cytochrome C reductase/oxidase supercomplex from yeast mitochondria". J. Biol. Chem. 282 (16): 12240–8. doi:10.1074/jbc.M610545200. PMID 17322303. S2CID 18123642.
- ^ Schägger H, Pfeiffer K (2000). "Supercomplexes in the respiratory chains of yeast and mammalian mitochondria". EMBO J. 19 (8): 1777–83. doi:10.1093/emboj/19.8.1777. PMC 302020. PMID 10775262.
- ^ Schägger H (2002). "Respiratory chain supercomplexes of mitochondria and bacteria". Biochim. Biophys. Acta. 1555 (1–3): 154–9. doi:10.1016/S0005-2728(02)00271-2. PMID 12206908.
- ^ Schägger H, Pfeiffer K (2001). "The ratio of oxidative phosphorylation complexes I-V in bovine heart mitochondria and the composition of respiratory chain supercomplexes". J. Biol. Chem. 276 (41): 37861–7. doi:10.1074/jbc.M106474200. PMID 11483615. Archived from the original on 2007-09-29.
- ^ Gupte S, Wu ES, Hoechli L, Hoechli M, Jacobson K, Sowers AE, Hackenbrock CR (1984). "Relationship between lateral diffusion, collision frequency, and electron transfer of mitochondrial inner membrane oxidation-reduction components". Proc. Natl. Acad. Sci. U.S.A. 81 (9): 2606–10. Bibcode:1984PNAS...81.2606G. doi:10.1073/pnas.81.9.2606. PMC 345118. PMID 6326133.
- ^ Nealson KH (1999). "Post-Viking microbiology: new approaches, new data, new insights". Orig Life Evol Biosph. 29 (1): 73–93. Bibcode:1999OLEB...29...73N. doi:10.1023/A:1006515817767. PMID 11536899. S2CID 12289639.
- ^ Schäfer G, Engelhard M, Müller V (1999). "Bioenergetics of the Archaea". Microbiol. Mol. Biol. Rev. 63 (3): 570–620. doi:10.1128/MMBR.63.3.570-620.1999. PMC 103747. PMID 10477309.
- ^ a b Ingledew WJ, Poole RK (1984). "The respiratory chains of Escherichia coli". Microbiol. Rev. 48 (3): 222–71. doi:10.1128/mmbr.48.3.222-271.1984. PMC 373010. PMID 6387427.
- ^ a b Unden G, Bongaerts J (1997). "Alternative respiratory pathways of Escherichia coli: energetics and transcriptional regulation in response to electron acceptors". Biochim. Biophys. Acta. 1320 (3): 217–34. doi:10.1016/S0005-2728(97)00034-0. PMID 9230919.
- ^ Cecchini G, Schröder I, Gunsalus RP, Maklashina E (2002). "Succinate dehydrogenase and fumarate reductase from Escherichia coli". Biochim. Biophys. Acta. 1553 (1–2): 140–57. doi:10.1016/S0005-2728(01)00238-9. PMID 11803023.
- ^ Freitag A, Bock E (1990). "Energy conservation in Nitrobacter". FEMS Microbiology Letters. 66 (1–3): 157–62. doi:10.1111/j.1574-6968.1990.tb03989.x.
- ^ Starkenburg SR, Chain PS, Sayavedra-Soto LA, Hauser L, Land ML, Larimer FW, et al. (March 2006). "Genome sequence of the chemolithoautotrophic nitrite-oxidizing bacterium Nitrobacter winogradskyi Nb-255". Applied and Environmental Microbiology. 72 (3): 2050–63. doi:10.1128/AEM.72.3.2050-2063.2006. PMC 1393235. PMID 16517654.
- ^ Yamanaka T, Fukumori Y (December 1988). "The nitrite oxidizing system of Nitrobacter winogradskyi". FEMS Microbiology Reviews. 4 (4): 259–70. doi:10.1111/j.1574-6968.1988.tb02746.x. PMID 2856189.
- ^ Iuchi S, Lin EC (1993). "Adaptation of Escherichia coli to redox environments by gene expression". Mol. Microbiol. 9 (1): 9–15. doi:10.1111/j.1365-2958.1993.tb01664.x. PMID 8412675. S2CID 39165641.
- ^ Calhoun MW, Oden KL, Gennis RB, de Mattos MJ, Neijssel OM (1993). "Energetic efficiency of Escherichia coli: effects of mutations in components of the aerobic respiratory chain" (PDF). J. Bacteriol. 175 (10): 3020–5. doi:10.1128/jb.175.10.3020-3025.1993. PMC 204621. PMID 8491720. Archived (PDF) from the original on 2007-09-27.
- ^ a b Boyer PD (1997). "The ATP synthase--a splendid molecular machine". Annu. Rev. Biochem. 66: 717–49. doi:10.1146/annurev.biochem.66.1.717. PMID 9242922.
- ^ Van Walraven HS, Strotmann H, Schwarz O, Rumberg B (1996). "The H+/ATP coupling ratio of the ATP synthase from thiol-modulated chloroplasts and two cyanobacterial strains is four". FEBS Lett. 379 (3): 309–13. doi:10.1016/0014-5793(95)01536-1. PMID 8603713. S2CID 35989618.
- ^ Yoshida M, Muneyuki E, Hisabori T (2001). "ATP synthase--a marvellous rotary engine of the cell". Nat. Rev. Mol. Cell Biol. 2 (9): 669–77. doi:10.1038/35089509. PMID 11533724. S2CID 3926411.
- ^ Schemidt RA, Qu J, Williams JR, Brusilow WS (1998). "Effects of carbon source on expression of F0 genes and on the stoichiometry of the c subunit in the F1F0 ATPase of Escherichia coli". J. Bacteriol. 180 (12): 3205–8. doi:10.1128/jb.180.12.3205-3208.1998. PMC 107823. PMID 9620972.
- ^ Nelson N, Perzov N, Cohen A, Hagai K, Padler V, Nelson H (1 January 2000). "The cellular biology of proton-motive force generation by V-ATPases". J. Exp. Biol. 203 (Pt 1): 89–95. doi:10.1242/jeb.203.1.89. PMID 10600677. Archived from the original on 30 September 2007.
- ^ Rubinstein JL, Walker JE, Henderson R (2003). "Structure of the mitochondrial ATP synthase by electron cryomicroscopy". EMBO J. 22 (23): 6182–92. doi:10.1093/emboj/cdg608. PMC 291849. PMID 14633978.
- ^ Leslie AG, Walker JE (2000). "Structural model of F1-ATPase and the implications for rotary catalysis". Philos. Trans. R. Soc. Lond. B Biol. Sci. 355 (1396): 465–71. doi:10.1098/rstb.2000.0588. PMC 1692760. PMID 10836500.
- ^ Noji H, Yoshida M (2001). "The rotary machine in the cell, ATP synthase". J. Biol. Chem. 276 (3): 1665–8. doi:10.1074/jbc.R000021200. PMID 11080505. S2CID 30953216.
- ^ Capaldi RA, Aggeler R (2002). "Mechanism of the F(1)F(0)-type ATP synthase, a biological rotary motor". Trends Biochem. Sci. 27 (3): 154–60. doi:10.1016/S0968-0004(01)02051-5. PMID 11893513.
- ^ Dimroth P, von Ballmoos C, Meier T (2006). "Catalytic and mechanical cycles in F-ATP synthases. Fourth in the Cycles Review Series". EMBO Rep. 7 (3): 276–82. doi:10.1038/sj.embor.7400646. PMC 1456893. PMID 16607397.
- ^ a b Gresser MJ, Myers JA, Boyer PD (25 October 1982). "Catalytic site cooperativity of beef heart mitochondrial F1 adenosine triphosphatase. Correlations of initial velocity, bound intermediate, and oxygen exchange measurements with an alternating three-site model". J. Biol. Chem. 257 (20): 12030–8. doi:10.1016/S0021-9258(18)33672-X. PMID 6214554. Archived from the original on 29 September 2007.
- ^ Dimroth P (1994). "Bacterial sodium ion-coupled energetics". Antonie van Leeuwenhoek. 65 (4): 381–95. doi:10.1007/BF00872221. PMID 7832594. S2CID 23763996.
- ^ a b Becher B, Müller V (1994). "Delta mu Na+ drives the synthesis of ATP via an delta mu Na(+)-translocating F1F0-ATP synthase in membrane vesicles of the archaeon Methanosarcina mazei Gö1". J. Bacteriol. 176 (9): 2543–50. doi:10.1128/jb.176.9.2543-2550.1994. PMC 205391. PMID 8169202.
- ^ Müller V (2004). "An exceptional variability in the motor of archael A1A0 ATPases: from multimeric to monomeric rotors comprising 6-13 ion binding sites". J. Bioenerg. Biomembr. 36 (1): 115–25. doi:10.1023/B:JOBB.0000019603.68282.04. PMID 15168615. S2CID 24887884.
- ^ a b Davies KJ (1995). "Oxidative stress: the paradox of aerobic life". Biochem. Soc. Symp. 61: 1–31. doi:10.1042/bss0610001. PMID 8660387.
- ^ Rattan SI (2006). "Theories of biological aging: genes, proteins, and free radicals" (PDF). Free Radic. Res. 40 (12): 1230–8. CiteSeerX 10.1.1.476.9259. doi:10.1080/10715760600911303. PMID 17090411. S2CID 11125090. Archived from the original (PDF) on 2014-06-14. Retrieved 2017-10-27.
- ^ Valko M, Leibfritz D, Moncol J, Cronin MT, Mazur M, Telser J (2007). "Free radicals and antioxidants in normal physiological functions and human disease". Int. J. Biochem. Cell Biol. 39 (1): 44–84. doi:10.1016/j.biocel.2006.07.001. PMID 16978905.
- ^ Raha S, Robinson BH (2000). "Mitochondria, oxygen free radicals, disease and ageing". Trends Biochem. Sci. 25 (10): 502–8. doi:10.1016/S0968-0004(00)01674-1. PMID 11050436.
- ^ Finkel T, Holbrook NJ (2000). "Oxidants, oxidative stress and the biology of ageing". Nature. 408 (6809): 239–47. Bibcode:2000Natur.408..239F. doi:10.1038/35041687. PMID 11089981. S2CID 2502238.
- ^ Kadenbach B, Ramzan R, Wen L, Vogt S (March 2010). "New extension of the Mitchell Theory for oxidative phosphorylation in mitochondria of living organisms". Biochim. Biophys. Acta. 1800 (3): 205–12. doi:10.1016/j.bbagen.2009.04.019. PMID 19409964.
- ^ Echtay KS, Roussel D, St-Pierre J, Jekabsons MB, Cadenas S, Stuart JA, Harper JA, Roebuck SJ, Morrison A, Pickering S, Clapham JC, Brand MD (January 2002). "Superoxide activates mitochondrial uncoupling proteins". Nature. 415 (6867): 96–9. Bibcode:2002Natur.415...96E. doi:10.1038/415096a. PMID 11780125. S2CID 4349744.
- ^ Devaux JB, Hedges CP, Birch N, Herbert N, Renshaw GM, Hickey AJ (January 2019). "Acidosis Maintains the Function of Brain Mitochondria in Hypoxia-Tolerant Triplefin Fish: A Strategy to Survive Acute Hypoxic Exposure?". Frontiers in Physiology. 9: 1941. doi:10.3389/fphys.2018.01941. PMC 6346031. PMID 30713504.
- ^ a b Joshi S, Huang YG (1991). "ATP synthase complex from bovine heart mitochondria: the oligomycin sensitivity conferring protein is essential for dicyclohexyl carbodiimide-sensitive ATPase". Biochim. Biophys. Acta. 1067 (2): 255–8. doi:10.1016/0005-2736(91)90051-9. PMID 1831660.
- ^ a b Satyanarayana U (2002). Biochemistry (2nd ed.). Kolkata, India: Books and Allied. ISBN 8187134801. OCLC 71209231.
- ^ Tsubaki M (1993). "Fourier-transform infrared study of cyanide binding to the Fea3-CuB binuclear site of bovine heart cytochrome c oxidase: implication of the redox-linked conformational change at the binuclear site". Biochemistry. 32 (1): 164–73. doi:10.1021/bi00052a022. PMID 8380331.
- ^ Heytler PG (1979). "Uncouplers of oxidative phosphorylation". Meth. Enzymol. Methods in Enzymology. 55: 462–42. doi:10.1016/0076-6879(79)55060-5. ISBN 978-0-12-181955-2. PMID 156853.
- ^ Lambert AJ, Brand MD (2004). "Inhibitors of the quinone-binding site allow rapid superoxide production from mitochondrial NADH:ubiquinone oxidoreductase (complex I)". J. Biol. Chem. 279 (38): 39414–20. doi:10.1074/jbc.M406576200. PMID 15262965. S2CID 26620903.
- ^ Dervartanian DV, Veeger C (November 1964). "Studies on succinate dehydrogenase: I. Spectral properties of the purified enzyme and formation of enzyme-competitive inhibitor complexes". Biochim. Biophys. Acta. 92 (2): 233–47. doi:10.1016/0926-6569(64)90182-8. PMID 14249115.
- ^ Ricquier D, Bouillaud F (2000). "The uncoupling protein homologues: UCP1, UCP2, UCP3, StUCP and AtUCP". Biochem. J. 345 (2): 161–79. doi:10.1042/0264-6021:3450161. PMC 1220743. PMID 10620491.
- ^ Borecký J, Vercesi AE (2005). "Plant uncoupling mitochondrial protein and alternative oxidase: energy metabolism and stress". Biosci. Rep. 25 (3–4): 271–86. doi:10.1007/s10540-005-2889-2. PMID 16283557. S2CID 18598358.
- ^ Harden A, Young WJ (1906). "The alcoholic ferment of yeast-juice". Proceedings of the Royal Society. B (77): 405–20. doi:10.1098/rspb.1906.0029.
- ^ Kalckar HM (1974). "Origins of the concept oxidative phosphorylation". Mol. Cell. Biochem. 5 (1–2): 55–63. doi:10.1007/BF01874172. PMID 4279328. S2CID 26999163.
- ^ Lipmann F (1941). "Metabolic generation and utilization of phosphate bond energy". Adv Enzymol. 1: 99–162. doi:10.4159/harvard.9780674366701.c141. ISBN 9780674366701.
- ^ Friedkin M, Lehninger AL (1 April 1949). "Esterification of inorganic phosphate coupled to electron transport between dihydrodiphosphopyridine nucleotide and oxygen". J. Biol. Chem. 178 (2): 611–44. doi:10.1016/S0021-9258(18)56879-4. PMID 18116985. Archived from the original on 16 December 2008.
- ^ Kalckar HM (1991). "50 years of biological research--from oxidative phosphorylation to energy requiring transport regulation". Annual Review of Biochemistry. 60: 1–37. doi:10.1146/annurev.bi.60.070191.000245. PMID 1883194.
- ^ Belitser VA, Tsibakova ET (1939). "About phosphorilation mechanism coupled with respiration". Biokhimiya. 4: 516–534.
- ^ Slater EC (1953). "Mechanism of phosphorylation in the respiratory chain". Nature. 172 (4387): 975–8. Bibcode:1953Natur.172..975S. doi:10.1038/172975a0. PMID 13111237. S2CID 4153659.
- ^ Mitchell P (1961). "Coupling of phosphorylation to electron and hydrogen transfer by a chemi-osmotic type of mechanism". Nature. 191 (4784): 144–8. Bibcode:1961Natur.191..144M. doi:10.1038/191144a0. PMID 13771349. S2CID 1784050.
- ^ Saier Jr MH. Peter Mitchell and the Vital Force. OCLC 55202414.
- ^ Mitchell P (1978). "David Keilin's Respiratory Chain Concept and Its Chemiosmotic Consequences" (PDF). Nobel lecture. Nobel Foundation. Archived (PDF) from the original on 2007-09-27. Retrieved 2007-07-21.
- ^ Pullman ME, Penefsky HS, Datta A, Racker E (1 November 1960). "Partial resolution of the enzymes catalyzing oxidative phosphorylation. I. Purification and properties of soluble dinitrophenol-stimulated adenosine triphosphatase". J. Biol. Chem. 235 (11): 3322–9. doi:10.1016/S0021-9258(20)81361-1. PMID 13738472. Archived from the original on 29 September 2007.
- ^ Boyer PD, Cross RL, Momsen W (1973). "A new concept for energy coupling in oxidative phosphorylation based on a molecular explanation of the oxygen exchange reactions". Proc. Natl. Acad. Sci. U.S.A. 70 (10): 2837–9. Bibcode:1973PNAS...70.2837B. doi:10.1073/pnas.70.10.2837. PMC 427120. PMID 4517936.
- ^ "The Nobel Prize in Chemistry 1997". Nobel Foundation. Archived from the original on 2017-03-25. Retrieved 2007-07-21.
추가 읽기
소개
- Nelson DL, Cox MM (2004). Lehninger Principles of Biochemistry (4th ed.). W. H. Freeman. ISBN 0-7167-4339-6.
- Schneider ED, Sagan D (2006). Into the Cool: Energy Flow, Thermodynamics and Life (1st ed.). University of Chicago Press. ISBN 0-226-73937-6.
- Lane N (2006). Power, Sex, Suicide: Mitochondria and the Meaning of Life (1st ed.). Oxford University Press, USA. ISBN 0-19-920564-7.
고급
- Nicholls DG, Ferguson SJ (2002). Bioenergetics 3 (1st ed.). Academic Press. ISBN 0-12-518121-3.
- Haynie D (2001). Biological Thermodynamics (1st ed.). Cambridge University Press. ISBN 0-521-79549-4.
- Rajan SS (2003). Introduction to Bioenergetics (1st ed.). Anmol. ISBN 81-261-1364-2.
- Wikstrom M, ed. (2005). Biophysical and Structural Aspects of Bioenergetics (1st ed.). Royal Society of Chemistry. ISBN 0-85404-346-2.
일반 자원
- 산화 인산화를 보여주는 애니메이션 도표 와일리앤코 생화학 개념
- 온라인 생물물리학 강의는 일리노이 대학교의 안토니 크로프트스(Antony Crofts, University of Illinois)에서 Urbana-Champaign
- ATP 싱타세 그레이엄 존슨
구조 자원
- 해당 달의 PDB 분자:
- ATP Synthase 2020-07-24 Wayback Machine에 보관
- 시토크롬 c
- 시토크롬산화효소
- 페르난도 페소아의 대화형 분자 모델:








![{\displaystyle {\ce {O2->[{\ce {e^{-}}}]{\underset {Superoxide}{O2^{\underline {\bullet }}}}->[{\ce {e^{-}}}]{\underset {Peroxide}{O2^{2-}}}}}}](https://wikimedia.org/api/rest_v1/media/math/render/svg/a3d9bf9d3a61736aa6207fa53b8ce0165b9eebb6)