시토크롬c산화효소 서브유닛I
Cytochrome c oxidase subunit I| COX1 | |||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 식별자 | |||||||||||||||||||||||||||||||||||||||||||||||||||
| 에일리어스 | COX1, 미토콘드리아 부호화 시토크롬c산화효소 I, COI, MTCO1, 시토크롬c산화효소 주요 서브유닛, COI, 시토크롬c산화효소 서브유닛 I | ||||||||||||||||||||||||||||||||||||||||||||||||||
| 외부 ID | OMIM: 516030 MGI: 102504 HomoloGene: 5016 GeneCard: COX1 | ||||||||||||||||||||||||||||||||||||||||||||||||||
| |||||||||||||||||||||||||||||||||||||||||||||||||||
| |||||||||||||||||||||||||||||||||||||||||||||||||||
| |||||||||||||||||||||||||||||||||||||||||||||||||||
| |||||||||||||||||||||||||||||||||||||||||||||||||||
| |||||||||||||||||||||||||||||||||||||||||||||||||||
| 위키데이터 | |||||||||||||||||||||||||||||||||||||||||||||||||||
| |||||||||||||||||||||||||||||||||||||||||||||||||||
| 시토크롬c산화효소 서브유닛I | |||||||||
|---|---|---|---|---|---|---|---|---|---|
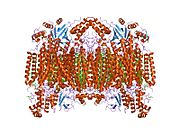
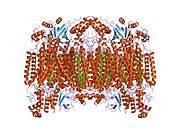
 13-서브유닛 산화 시토크롬c산화효소 [5]구조. | |||||||||
| 식별자 | |||||||||
| 기호. | COX1 또는 COI | ||||||||
| 팜 | PF00115 | ||||||||
| 인터프로 | IPR000883 | ||||||||
| 프로 사이트 | PDOC00074 | ||||||||
| SCOP2 | 1oc/SCOPe/SUPFAM | ||||||||
| TCDB | 3. D.4 | ||||||||
| OPM 슈퍼 패밀리 | 4 | ||||||||
| OPM단백질 | 1v55 | ||||||||
| CDD | cd01663 | ||||||||
| |||||||||
시토크롬c산화효소I(COx1)는 미토콘드리아로 부호화된 시토크롬c산화효소I(MT-CO1)로 알려져 있으며, 사람에게서 MT-CO1 [6]유전자에 의해 부호화되는 단백질이다.다른 진핵생물에서, 그 유전자는 COX1, CO1, 또는 [7]COI라고 불린다.시토크롬c산화효소I는 시토크롬c산화효소복합체의 주요 서브단위이다.MT-CO1의 돌연변이는 레버의 유전성 시신경증(LHON), 후천성 측두엽성 빈혈, 복합체 IV 결핍, 대장암, 감각성 난청, 재발성 미오글로빈뇨와 [8][9][10]관련이 있다.
구조.
37개의 미토콘드리아 유전자 중 하나인 MT-CO1 유전자는 mtDNA의 구아닌이 풍부한 중(H) 부분에 뉴클레오티드 쌍 5904에서 7444까지 있다.이 유전자 제품은 513개의 [11][12]아미노산으로 구성된 57kDa 단백질이다.
기능.
시토크롬c산화효소 서브유닛 I(CO1 또는 MT-CO1)는 호흡복합체 IV의 3개의 미토콘드리아 DNA(mtDNA) 부호화 서브유닛(MT-CO1, MT-CO2, MT-CO3) 중 하나이다.복합체 IV는 미토콘드리아 산화 인산화 [6]전자전달계의 세 번째이자 마지막 효소이다.
시토크롬c산화효소(EC 1.9.3.1)는 호기성 대사의 핵심 효소이다.양성자 펌핑 헴-구리 산화효소는 원핵생물 및 진핵생물에서 호흡 사슬의 말단 에너지 전달 효소를 나타냅니다.시토크롬 c와 유비퀴놀 산화효소(EC 1.10.3.10)의 가장 큰 서브유닛 I와 연관된 CuB-헴 a3(또는 헴o) 2핵 중심은 다이옥시겐 감소와 양성자 [13][14]펌핑 사이의 결합에 직접 관여한다.일부 말단 산화효소는 혈장막(원핵생물) 또는 미토콘드리아 내막(유카리오테스)을 가로질러 막 투과 양성자 구배를 생성한다.
효소 복합체는 최대 13개의 폴리펩타이드(원핵생물)까지 3~4개의 서브유닛(원핵생물)으로 구성되며, 이 중 촉매 서브유닛(포유동물 서브유닛 I(COI)과 동일)만이 모든 헴-구리 호흡 산화효소에서 발견된다.COI 내에서 4개의 막 통과 스팬의 바깥쪽 부근에 보존된 히스티딘 잔기 6개에 연결된 바이메탈 중심(고회전 헴과 구리 B에 의해 형성됨)과 저회전 헴의 존재는 모든 가족 [15][16][17]구성원에게 공통적이다.진핵생물과는 대조적으로 원핵생물의 호흡 사슬은 여러 말단 산화효소로 분기된다.효소 복합체는 헴 및 구리 조성, 기질 유형 및 기질 친화력에서 다양합니다.서로 다른 호흡 산화효소는 세포가 다양한 환경 성장 [13]조건에 따라 호흡 시스템을 맞춤화할 수 있도록 합니다.
그람양성균의 시토크롬c산화효소로부터 유박테리아 퀴놀산화효소가 유도되었으며, 고세균 퀴놀산화효소는 독립적인 기원을 가지고 있는 것으로 밝혀졌다.상당한 양의 증거는 Pseudomonadota(프로테오박테리아 또는 보라색 박테리아로도 알려져 있음)가 그램 양성 [13]박테리아로부터의 측면 유전자 이동을 통해 퀴놀 산화효소를 획득했다는 것을 암시한다.
관련된 산화질소환원효소(EC 1.7.99.7)는 고세균 및 유박테리아 탈질종에 존재하며 시토크롬 b 및 c의 헤테로다이머이다.페나진 메토황산염은 수용체 역할을 할 수 있다.시토크롬 C 산화효소 촉매 서브유닛은 질소와 [18][19]산소를 모두 감소시킬 수 있는 고대 산화질소 환원효소로부터 진화했다고 제안되어 왔다.
임상적 의의
이 유전자의 돌연변이는 레버의 유전성 시신경증(LHON), 후천성 측두엽성 빈혈, 복합체 IV 결핍증, 대장암, 감각성 난청, 재발성 미오글로빈뇨와 [8][9][10]관련이 있다.
레버 유전성 시신경증
MT-CO1의 돌연변이와 상관된 LHON은 아급성 또는 급성 중추 시력 상실을 일으키는 시신경 기능 장애로 특징지어진다.일부 환자는 신경학적 또는 심장 전도 장애를 보일 수 있습니다.이 질병은 미토콘드리아 DNA 돌연변이가 호흡사슬 복합체에 영향을 미치는 결과이기 때문에 [20][9][10]모계 유전이다.
후천성 특발성 시데로아구성 빈혈
MT-CO1은 후천성 특발성 편모세포성 빈혈의 발생에 관여할 수 있다.미토콘드리아 DNA의 돌연변이는 헴의 미토콘드리아 생합성 마지막 단계에 필요한 철을 철 철로 환원하는 것을 방지하면서 호흡 사슬 장애를 일으킬 수 있다.그 결과 미토콘드리아에 철이 축적되고 헴 [21][22][9][10]생산이 불충분합니다.
미토콘드리아 복합체 IV 결핍증(MT-C4D)
이 유전자의 돌연변이는 미토콘드리아 복합체 IV 결핍을 야기할 수 있는데, 미토콘드리아 호흡 사슬의 질병은 고립된 미오패시로부터 여러 장기와 조직에 영향을 미치는 심각한 다계통 질환에 이르기까지 다양한 임상적 징후를 보인다.증상으로는 간기능장애, 간기능저하증, 저혈압, 근육약화, 운동불내증, 운동장애, 운동발달지연, 정신지체, 발달지연, 비후성심근증이 포함될 수 있다.일부 환자의 경우 비후성 심근증은 신생아 단계에서 치명적이다.다른 영향을 받는 사람들은 리 [23][24][9][10]병을 나타낼 수 있다.
대장암(CRC)
MT-CO1 돌연변이는 대장암과 직장 내벽에 악성 병변을 보이는 매우 복잡한 질병이다.그러한 수많은 유전적 변화는 종종 선종, 즉 전분열성 병변의 침습성 선암으로의 진행과 관련이 있다.장기 궤양성 대장염, 대장 용종, 가족력이 대장암의 [25][26][9][10]위험인자입니다.
재발성 미오글로빈뇨 미토콘드리아(RM-MT)
RM-MT는 근육통 및 쇠약과 관련된 횡문근융해증(골격근의 괴사 또는 붕괴), 운동불내성, 산화인산화근능 저하, 소변 내 미오글로빈 배설 등이 반복되는 것이 특징인 질환이다.그것은 미토콘드리아 근병증과 연관되어 왔다.G5920A 돌연변이와 헤테로플라스믹 G6708A 난센스 돌연변이는 COX 결핍 및 [27][28][9][10]RM-MT와 관련되어 있다.
난청, 감음, 미토콘드리아(DFNM)
DFNM은 모성 유전으로 인한 비신드롬성 난청의 한 형태이다.영향을 받는 사람들은 높은 주파수와 관련된 진행성, 발작 후, 감각적인 청력 손실을 보인다.돌연변이 A1555G는 이 [29][9][10]질병과 관련이 있다.
아과
- 시토크롬c산화효소 cbb3-type, 서브유닛I InterPro: IPR004677
- 시토크롬오유비퀴놀산화효소, 서브유닛I InterPro: IPR014207
- 시토크롬aa3 퀴놀산화효소, 서브유닛I InterPro: IPR014233
- 시토크롬c산화효소, 서브유닛I세균형 InterPro : IPR014241
DNA 바코딩에 사용
MT-CO1은 동물의 종을 식별하기 위한 DNA 바코드로 자주 사용되는 유전자이다.MT-CO1 유전자 배열은 돌연변이율이 종종 밀접하게 연관된 종들을 구별할 수 있을 만큼 빠르기 때문에 그리고 또한 그 배열이 동식물들 사이에서 보존되기 때문에 이 역할에 적합하다.MT-CO1 염기서열 차이가 너무 작아서 가까운 종 간에 검출할 수 없다는 회의론자들의 1차 반대와 달리, 일반적으로 2% 이상의 염기서열 차이가 밀접하게 연관된 동물 [30]종 간에 검출되며, 이는 바코드가 많은 동물에게 효과적이라는 것을 시사한다.그러나 모든 종자 식물에서 MT-CO1의 진화 속도는 매우 느리다.
대장암호 내 MT-COI(= CCOI)

CCOI라고도 알려진 MT-COI 단백질은 보통 인간 대장의 결장암 세포질에서 높은 수준으로 발현됩니다.그러나 MT-COI는 사람의 나이가 들면서 대장암에서 자주 손실되며 대장암과 대장암의 [31]일부에서 발생하는 현장결함에도 종종 누락된다.
결장의 상피 내부 표면은 결장암인 침입에 의해 뚫려 있다.대장암괴는 현미경처럼 두꺼운 시험관 모양으로 되어 있으며, 시험관 길이(암괴 내강) 아래에 중앙 구멍이 있습니다.이 섹션의 이미지에는 4개의 조직 섹션이 표시되어 있으며, 2개는 크립트의 긴 축을 가로지르고 2개는 긴 축에 평행하게 절단되어 있습니다.
사진 속 인간 대장암은 대부분 갈색-오렌지색 MT-COI의 높은 발현을 보인다.그러나 일부 결장암괴에서는 모든 세포가 MT-COI가 없고 대부분 흰색으로 보이며, 주요 색깔은 암괴의 외벽에 있는 핵의 청회색 얼룩이다.Greaves 등은 대장암호에서 [32]MT-COI의 결핍이 MT-COI 유전자의 돌연변이에 의한 것임을 보여주었다.패널 B와 같이 3개의 크립트의 줄기세포 일부가 MT-COI 돌연변이를 가지고 있는 것처럼 보이므로 이들 줄기세포에서 발생하는 세포의 40~50%가 횡단영역에서 흰색 세그먼트를 형성한다.
사람의 경우, MT-COI에 부족한 대장암은 40세 이전에는 1% 미만이지만,[31] 그 이후에는 나이와 함께 선형적으로 증가한다.평균적으로 MT-COI가 부족한 대장암은 80-84세까지 [31]여성이 18%, 남성이 23%에 이른다.대장 종양은 종종 MT-COI 결핍 크립트의 대규모 클러스터(410개까지)를 포함하는 크립트 분야에서 발생한다.대장암의 경우 종양세포의 80%가 MT-COI가 [31]부족할 수 있다.
패널 C와 D에서 볼 수 있듯이 크립트의 길이는 약 75에서 약 110셀입니다.평균 암호 둘레는 23 [33]셀입니다.이러한 측정치를 바탕으로 암호에는 1725~2530개의 셀이 있습니다.또 다른 보고서에서는 [34]대장암호당 1500~4900개의 세포 범위를 제시했다.
1700~5000개의 세포에서 MT-COI가 거의 완전히 손실되는 빈번한 암호화가 발생하는 것은 자연선택의 과정을 암시한다.그러나 초기 미토콘드리아 DNA 돌연변이에 의한 특정 크립트 전체의 결핍이 확률적 [35][36]과정을 통해 때때로 발생할 수 있다는 것도 밝혀졌다.그럼에도 불구하고 대장 상피 내의 많은 크립트에서 MT-COI 결핍이 자주 발생하는 것은 MT-COI가 없는 것이 선택적인 이점을 제공할 수 있음을 나타낸다.
MT-COI는 미토콘드리아 염색체에 의해 암호화된다.대부분의 미토콘드리아에는 보통 미토콘드리아당 [37][38][39]2개에서 6개 사이의 염색체가 여러 개 있다.미토콘드리아의 한 염색체에서 MT-COI의 돌연변이가 발생하면, 새로운 미토콘드리아를 생성하기 위해 미토콘드리아 핵분열 중에 염색체의 무작위 분리가 있을 수 있다.이것은 주로 또는 MT-COI 변이 염색체를 가진 미토콘드리아를 발생시킬 수 있다.
MT-COI 변이 염색체를 많이 가진 미토콘드리아는 세포(MT-COI 결핍 호모플라스미 세포)에서 미토콘드리아의 주요 유형이 되기 위해 양의 선택 편향을 가져야 한다.세포 [38][39]유형에 따라 세포당 약 100에서 700개의 미토콘드리아가 있다.게다가 미토콘드리아의 회전이 꽤 빠르기 때문에, MT-COI 변이 염색체와 양성 선택 편향을 가지는 미토콘드리아는, 곧 세포내의 주요한 미토콘드리아의 타입이 될 수 있다.쥐의 미토콘드리아의 평균 반감기는 세포 유형에 따라 9일에서 24일 [40]사이이고 쥐는 약 [41]2일입니다.인간의 경우 미토콘드리아의 반감기가 며칠에서 몇 주 정도 걸릴 수 있다.
주로 MT-COI 결핍이었던 결장암막의 기부에 있는 줄기세포는 줄기세포 틈새를 차지하기 위해 다른 4개 또는 5개의 줄기세포와 경쟁할 수 있다.이 경우 이미지의 패널A, B 및 D에 있는 일부 암호에 나타나 있듯이 1700 ~5000개의 모든 셀에서 대장암호에는 MT-COI가 부족합니다.
결장의 크립트는 2개의 크립트를 형성하기 위해 크립트가 파열하는 패널 C와 적어도 1개의 크립트가 파열하고 있다고 생각되는 패널 B에서 볼 수 있듯이 핵분열에 의해 재생될 수 있다.MT-COI가 부족한 대부분의 암호는 서로 인접한 MT-COI가 부족한 암호와 함께 암호(암호 복제) 클러스터 내에 있습니다(패널 [31]D 참조).이것은 부족한 크립트의 클론이 종종 발생하고, 따라서 인간 대장 상피에서 확산되도록 하는 긍정적인 선택적 편견이 있을 수 있다는 것을 보여준다.
MT-COI의 결핍이 왜 양의 선택적 편향을 가져야 하는지는 명확하지 않다.한 가지 제안은[31] 미토콘드리아에서 MT-COI의 결핍은 활성산소 생산을 감소시키고(그리고 산화적 손상 감소) 이것은 MT-COI 결핍을 위한 동질체를 생성하기 위해 동일한 세포 내의 다른 미토콘드리아와 경쟁하는 데 선택적인 이점을 제공한다는 것이다.또 다른 제안은 시토크롬c산화효소에 결핍된 세포는 아포토시스 내성이 있어 생존 가능성이 더 높다는 것이었다.활성 시토크롬 c 산화효소가 시토크롬 c를 산화시키고, 시토크롬 c는 프로 카스파아제 9를 활성화하여 아포토시스(apoptosis)[42]로 이어지기 때문에 MT-COI와 아포토시스(apotosis)이러한 두 가지 요인은 MT-COI 결핍 대장암세포가 나이가 들면서 또는 인간 대장 발암 중에 자주 발생하는 원인이 될 수 있다.
상호 작용
MITRAC(시토크롬c산화효소 중간연골번역조절조립체) 복합체 내에서 코드화된 단백질은 COA3, SMIM20/MITRAC7과 상호작용한다.SMIM20과의 상호작용은 새롭게 합성된 MT-CO1을 안정시키고 조기 [43]교체를 방지한다.또한 COX20 의존적인 방법으로 [44][9][10]TMEM177과 상호작용합니다.
레퍼런스
- ^ a b c GRCh38: 앙상블 릴리즈 89: ENSG00000198804 - 앙상블, 2017년 5월
- ^ a b c GRCm38: 앙상블 릴리즈 89: ENSMUSG000064351 - 앙상블, 2017년 5월
- ^ "Human PubMed Reference:". National Center for Biotechnology Information, U.S. National Library of Medicine.
- ^ "Mouse PubMed Reference:". National Center for Biotechnology Information, U.S. National Library of Medicine.
- ^ Tsukihara T, Aoyama H, Yamashita E, Tomizaki T, Yamaguchi H, Shinzawa-Itoh K, Nakashima R, Yaono R, Yoshikawa S (May 1996). "The whole structure of the 13-subunit oxidized cytochrome c oxidase at 2.8 A". Science. 272 (5265): 1136–44. Bibcode:1996Sci...272.1136T. doi:10.1126/science.272.5265.1136. PMID 8638158. S2CID 20860573.
- ^ a b "Entrez Gene: Cytochrome c oxidase subunit I".
- ^ Kosakyan A, Heger TJ, Leander BS, Todorov M, Mitchell EA, Lara E (May 2012). "COI barcoding of Nebelid testate amoebae (Amoebozoa: Arcellinida): extensive cryptic diversity and redefinition of the Hyalospheniidae Schultze" (PDF). Protist. 163 (3): 415–34. doi:10.1016/j.protis.2011.10.003. PMID 22130576.
- ^ a b 온라인 Mendelian In Man (OMIM) : 516030
- ^ a b c d e f g h i "MT-CO1 - Cytochrome c oxidase subunit 1 - Homo sapiens (Human) - MT-CO1 gene & protein". www.uniprot.org. Retrieved 2018-08-13.
 이 문서에는 CC BY 4.0 라이센스로 제공되는 텍스트가 포함되어 있습니다.
이 문서에는 CC BY 4.0 라이센스로 제공되는 텍스트가 포함되어 있습니다. - ^ a b c d e f g h i "UniProt: the universal protein knowledgebase". Nucleic Acids Research. 45 (D1): D158–D169. January 2017. doi:10.1093/nar/gkw1099. PMC 5210571. PMID 27899622.
- ^ Zong NC, Li H, Li H, Lam MP, Jimenez RC, Kim CS, Deng N, Kim AK, Choi JH, Zelaya I, Liem D, Meyer D, Odeberg J, Fang C, Lu HJ, Xu T, Weiss J, Duan H, Uhlen M, Yates JR, Apweiler R, Ge J, Hermjakob H, Ping P (October 2013). "Integration of cardiac proteome biology and medicine by a specialized knowledgebase". Circulation Research. 113 (9): 1043–53. doi:10.1161/CIRCRESAHA.113.301151. PMC 4076475. PMID 23965338.
- ^ "MT-CO1 - Cytochrome c oxidase subunit 1". Cardiac Organellar Protein Atlas Knowledgebase (COPaKB).[영구 데드링크]
- ^ a b c García-Horsman JA, Barquera B, Rumbley J, Ma J, Gennis RB (September 1994). "The superfamily of heme-copper respiratory oxidases". Journal of Bacteriology. 176 (18): 5587–600. doi:10.1128/jb.176.18.5587-5600.1994. PMC 196760. PMID 8083153.
- ^ Papa S, Capitanio N, Glaser P, Villani G (May 1994). "The proton pump of heme-copper oxidases". Cell Biology International. 18 (5): 345–55. doi:10.1006/cbir.1994.1084. PMID 8049679. S2CID 36428993.
- ^ Castresana J, Lübben M, Saraste M, Higgins DG (June 1994). "Evolution of cytochrome oxidase, an enzyme older than atmospheric oxygen". The EMBO Journal. 13 (11): 2516–25. doi:10.1002/j.1460-2075.1994.tb06541.x. PMC 395125. PMID 8013452.
- ^ Capaldi RA, Malatesta F, Darley-Usmar VM (July 1983). "Structure of cytochrome c oxidase". Biochimica et Biophysica Acta (BBA) - Reviews on Bioenergetics. 726 (2): 135–48. doi:10.1016/0304-4173(83)90003-4. PMID 6307356.
- ^ Holm L, Saraste M, Wikström M (September 1987). "Structural models of the redox centres in cytochrome oxidase". The EMBO Journal. 6 (9): 2819–23. doi:10.1002/j.1460-2075.1987.tb02578.x. PMC 553708. PMID 2824194.
- ^ Saraste M, Castresana J (March 1994). "Cytochrome oxidase evolved by tinkering with denitrification enzymes". FEBS Letters. 341 (1): 1–4. doi:10.1016/0014-5793(94)80228-9. PMID 8137905. S2CID 1248917.
- ^ Chen J, Strous M (February 2013). "Denitrification and aerobic respiration, hybrid electron transport chains and co-evolution". Biochimica et Biophysica Acta (BBA) - Bioenergetics. 1827 (2): 136–44. doi:10.1016/j.bbabio.2012.10.002. PMID 23044391.
- ^ Brown MD, Yang CC, Trounce I, Torroni A, Lott MT, Wallace DC (August 1992). "A mitochondrial DNA variant, identified in Leber hereditary optic neuropathy patients, which extends the amino acid sequence of cytochrome c oxidase subunit I". American Journal of Human Genetics. 51 (2): 378–85. PMC 1682694. PMID 1322638.
- ^ Gattermann N, Retzlaff S, Wang YL, Hofhaus G, Heinisch J, Aul C, Schneider W (December 1997). "Heteroplasmic point mutations of mitochondrial DNA affecting subunit I of cytochrome c oxidase in two patients with acquired idiopathic sideroblastic anemia". Blood. 90 (12): 4961–72. doi:10.1182/blood.V90.12.4961. PMID 9389715.
- ^ Bröker S, Meunier B, Rich P, Gattermann N, Hofhaus G (November 1998). "MtDNA mutations associated with sideroblastic anaemia cause a defect of mitochondrial cytochrome c oxidase". European Journal of Biochemistry. 258 (1): 132–8. doi:10.1046/j.1432-1327.1998.2580132.x. PMID 9851701.
- ^ Varlamov DA, Kudin AP, Vielhaber S, Schröder R, Sassen R, Becker A, Kunz D, Haug K, Rebstock J, Heils A, Elger CE, Kunz WS (August 2002). "Metabolic consequences of a novel missense mutation of the mtDNA CO I gene". Human Molecular Genetics. 11 (16): 1797–805. doi:10.1093/hmg/11.16.1797. PMID 12140182.
- ^ Lucioli S, Hoffmeier K, Carrozzo R, Tessa A, Ludwig B, Santorelli FM (March 2006). "Introducing a novel human mtDNA mutation into the Paracoccus denitrificans COX I gene explains functional deficits in a patient". Neurogenetics. 7 (1): 51–7. doi:10.1007/s10048-005-0015-z. PMID 16284789. S2CID 21304246.
- ^ Greaves LC, Preston SL, Tadrous PJ, Taylor RW, Barron MJ, Oukrif D, Leedham SJ, Deheragoda M, Sasieni P, Novelli MR, Jankowski JA, Turnbull DM, Wright NA, McDonald SA (January 2006). "Mitochondrial DNA mutations are established in human colonic stem cells, and mutated clones expand by crypt fission". Proceedings of the National Academy of Sciences of the United States of America. 103 (3): 714–9. Bibcode:2006PNAS..103..714G. doi:10.1073/pnas.0505903103. PMC 1325106. PMID 16407113.
- ^ Namslauer I, Brzezinski P (March 2009). "A mitochondrial DNA mutation linked to colon cancer results in proton leaks in cytochrome c oxidase". Proceedings of the National Academy of Sciences of the United States of America. 106 (9): 3402–7. Bibcode:2009PNAS..106.3402N. doi:10.1073/pnas.0811450106. PMC 2651238. PMID 19218458.
- ^ Karadimas CL, Greenstein P, Sue CM, Joseph JT, Tanji K, Haller RG, Taivassalo T, Davidson MM, Shanske S, Bonilla E, DiMauro S (September 2000). "Recurrent myoglobinuria due to a nonsense mutation in the COX I gene of mitochondrial DNA". Neurology. 55 (5): 644–9. doi:10.1212/wnl.55.5.644. PMID 10980727. S2CID 26776388.
- ^ Kollberg G, Moslemi AR, Lindberg C, Holme E, Oldfors A (February 2005). "Mitochondrial myopathy and rhabdomyolysis associated with a novel nonsense mutation in the gene encoding cytochrome c oxidase subunit I". Journal of Neuropathology and Experimental Neurology. 64 (2): 123–8. doi:10.1093/jnen/64.2.123. PMID 15751226.
- ^ Pandya A, Xia XJ, Erdenetungalag R, Amendola M, Landa B, Radnaabazar J, Dangaasuren B, Van Tuyle G, Nance WE (December 1999). "Heterogenous point mutations in the mitochondrial tRNA Ser(UCN) precursor coexisting with the A1555G mutation in deaf students from Mongolia". American Journal of Human Genetics. 65 (6): 1803–6. doi:10.1086/302658. PMC 1288397. PMID 10577941.
- ^ Hebert PD, Ratnasingham S, deWaard JR (August 2003). "Barcoding animal life: cytochrome c oxidase subunit 1 divergences among closely related species". Proceedings. Biological Sciences. 270 Suppl 1: S96–9. doi:10.1098/rsbl.2003.0025. PMC 1698023. PMID 12952648.
- ^ a b c d e f g Bernstein C, Facista A, Nguyen H, Zaitlin B, Hassounah N, Loustaunau C, Payne CM, Banerjee B, Goldschmid S, Tsikitis VL, Krouse R, Bernstein H (December 2010). "Cancer and age related colonic crypt deficiencies in cytochrome c oxidase I". World Journal of Gastrointestinal Oncology. 2 (12): 429–42. doi:10.4251/wjgo.v2.i12.429. PMC 3011097. PMID 21191537.
- ^ Greaves LC, Preston SL, Tadrous PJ, Taylor RW, Barron MJ, Oukrif D, Leedham SJ, Deheragoda M, Sasieni P, Novelli MR, Jankowski JA, Turnbull DM, Wright NA, McDonald SA (January 2006). "Mitochondrial DNA mutations are established in human colonic stem cells, and mutated clones expand by crypt fission". Proceedings of the National Academy of Sciences of the United States of America. 103 (3): 714–9. Bibcode:2006PNAS..103..714G. doi:10.1073/pnas.0505903103. PMC 1325106. PMID 16407113.
- ^ Baker AM, Cereser B, Melton S, Fletcher AG, Rodriguez-Justo M, Tadrous PJ, Humphries A, Elia G, McDonald SA, Wright NA, Simons BD, Jansen M, Graham TA (August 2014). "Quantification of crypt and stem cell evolution in the normal and neoplastic human colon". Cell Reports. 8 (4): 940–7. doi:10.1016/j.celrep.2014.07.019. PMC 4471679. PMID 25127143.
- ^ Nooteboom M, Johnson R, Taylor RW, Wright NA, Lightowlers RN, Kirkwood TB, Mathers JC, Turnbull DM, Greaves LC (February 2010). "Age-associated mitochondrial DNA mutations lead to small but significant changes in cell proliferation and apoptosis in human colonic crypts". Aging Cell. 9 (1): 96–9. doi:10.1111/j.1474-9726.2009.00531.x. PMC 2816353. PMID 19878146.
- ^ Coller HA, Bodyak ND, Khrapko K (April 2002). "Frequent intracellular clonal expansions of somatic mtDNA mutations: significance and mechanisms". Annals of the New York Academy of Sciences. 959 (1): 434–47. Bibcode:2002NYASA.959..434C. doi:10.1111/j.1749-6632.2002.tb02113.x. PMID 11976216. S2CID 40639679.
- ^ Nekhaeva E, Bodyak ND, Kraytsberg Y, McGrath SB, Van Orsouw NJ, Pluzhnikov A, Wei JY, Vijg J, Khrapko K (April 2002). "Clonally expanded mtDNA point mutations are abundant in individual cells of human tissues". Proceedings of the National Academy of Sciences of the United States of America. 99 (8): 5521–6. Bibcode:2002PNAS...99.5521N. doi:10.1073/pnas.072670199. PMC 122802. PMID 11943860.
- ^ Legros F, Malka F, Frachon P, Lombès A, Rojo M (June 2004). "Organization and dynamics of human mitochondrial DNA". Journal of Cell Science. 117 (Pt 13): 2653–62. doi:10.1242/jcs.01134. PMID 15138283.
- ^ a b Robin ED, Wong R (September 1988). "Mitochondrial DNA molecules and virtual number of mitochondria per cell in mammalian cells". Journal of Cellular Physiology. 136 (3): 507–13. doi:10.1002/jcp.1041360316. PMID 3170646. S2CID 2841036.
- ^ a b Satoh M, Kuroiwa T (September 1991). "Organization of multiple nucleoids and DNA molecules in mitochondria of a human cell". Experimental Cell Research. 196 (1): 137–40. doi:10.1016/0014-4827(91)90467-9. PMID 1715276.
- ^ Menzies RA, Gold PH (April 1971). "The turnover of mitochondria in a variety of tissues of young adult and aged rats". The Journal of Biological Chemistry. 246 (8): 2425–9. doi:10.1016/S0021-9258(18)62305-1. PMID 5553400.
- ^ Miwa S, Lawless C, von Zglinicki T (December 2008). "Mitochondrial turnover in liver is fast in vivo and is accelerated by dietary restriction: application of a simple dynamic model". Aging Cell. 7 (6): 920–3. doi:10.1111/j.1474-9726.2008.00426.x. PMC 2659384. PMID 18691181.
- ^ Brown GC, Borutaite V (2008). "Regulation of apoptosis by the redox state of cytochrome c". Biochimica et Biophysica Acta (BBA) - Bioenergetics. 1777 (7–8): 877–81. doi:10.1016/j.bbabio.2008.03.024. PMID 18439415.
- ^ Dennerlein S, Oeljeklaus S, Jans D, Hellwig C, Bareth B, Jakobs S, Deckers M, Warscheid B, Rehling P (September 2015). "MITRAC7 Acts as a COX1-Specific Chaperone and Reveals a Checkpoint during Cytochrome c Oxidase Assembly". Cell Reports. 12 (10): 1644–55. doi:10.1016/j.celrep.2015.08.009. PMID 26321642.
- ^ Lorenzi I, Oeljeklaus S, Aich A, Ronsör C, Callegari S, Dudek J, Warscheid B, Dennerlein S, Rehling P (February 2018). "The mitochondrial TMEM177 associates with COX20 during COX2 biogenesis". Biochimica et Biophysica Acta (BBA) - Molecular Cell Research. 1865 (2): 323–333. doi:10.1016/j.bbamcr.2017.11.010. PMC 5764226. PMID 29154948.
추가 정보
- Torroni A, Achilli A, Macaulay V, Richards M, Bandelt HJ (June 2006). "Harvesting the fruit of the human mtDNA tree". Trends in Genetics. 22 (6): 339–45. doi:10.1016/j.tig.2006.04.001. PMID 16678300.
- Bodenteich A, Mitchell LG, Polymeropoulos MH, Merril CR (May 1992). "Dinucleotide repeat in the human mitochondrial D-loop". Human Molecular Genetics. 1 (2): 140. doi:10.1093/hmg/1.2.140-a. PMID 1301157.
- Brown MD, Yang CC, Trounce I, Torroni A, Lott MT, Wallace DC (August 1992). "A mitochondrial DNA variant, identified in Leber hereditary optic neuropathy patients, which extends the amino acid sequence of cytochrome c oxidase subunit I". American Journal of Human Genetics. 51 (2): 378–85. PMC 1682694. PMID 1322638.
- Lu X, Walker T, MacManus JP, Seligy VL (July 1992). "Differentiation of HT-29 human colonic adenocarcinoma cells correlates with increased expression of mitochondrial RNA: effects of trehalose on cell growth and maturation". Cancer Research. 52 (13): 3718–25. PMID 1377597.
- Marzuki S, Noer AS, Lertrit P, Thyagarajan D, Kapsa R, Utthanaphol P, Byrne E (December 1991). "Normal variants of human mitochondrial DNA and translation products: the building of a reference data base". Human Genetics. 88 (2): 139–45. doi:10.1007/bf00206061. PMID 1757091. S2CID 28048453.
- Moraes CT, Andreetta F, Bonilla E, Shanske S, DiMauro S, Schon EA (March 1991). "Replication-competent human mitochondrial DNA lacking the heavy-strand promoter region". Molecular and Cellular Biology. 11 (3): 1631–7. doi:10.1128/MCB.11.3.1631. PMC 369459. PMID 1996112.
- Attardi G, Chomyn A, Doolittle RF, Mariottini P, Ragan CI (1987). "Seven unidentified reading frames of human mitochondrial DNA encode subunits of the respiratory chain NADH dehydrogenase". Cold Spring Harbor Symposia on Quantitative Biology. 51 (1): 103–14. doi:10.1101/sqb.1986.051.01.013. PMID 3472707.
- Chomyn A, Cleeter MW, Ragan CI, Riley M, Doolittle RF, Attardi G (October 1986). "URF6, last unidentified reading frame of human mtDNA, codes for an NADH dehydrogenase subunit". Science. 234 (4776): 614–8. Bibcode:1986Sci...234..614C. doi:10.1126/science.3764430. PMID 3764430.
- Chomyn A, Mariottini P, Cleeter MW, Ragan CI, Matsuno-Yagi A, Hatefi Y, Doolittle RF, Attardi G (1985). "Six unidentified reading frames of human mitochondrial DNA encode components of the respiratory-chain NADH dehydrogenase". Nature. 314 (6012): 592–7. Bibcode:1985Natur.314..592C. doi:10.1038/314592a0. PMID 3921850. S2CID 32964006.
- Sanger F, Coulson AR, Barrell BG, Smith AJ, Roe BA (October 1980). "Cloning in single-stranded bacteriophage as an aid to rapid DNA sequencing". Journal of Molecular Biology. 143 (2): 161–78. doi:10.1016/0022-2836(80)90196-5. PMID 6260957.
- Montoya J, Ojala D, Attardi G (April 1981). "Distinctive features of the 5'-terminal sequences of the human mitochondrial mRNAs". Nature. 290 (5806): 465–70. Bibcode:1981Natur.290..465M. doi:10.1038/290465a0. PMID 7219535. S2CID 4358928.
- Horai S, Hayasaka K, Kondo R, Tsugane K, Takahata N (January 1995). "Recent African origin of modern humans revealed by complete sequences of hominoid mitochondrial DNAs". Proceedings of the National Academy of Sciences of the United States of America. 92 (2): 532–6. Bibcode:1995PNAS...92..532H. doi:10.1073/pnas.92.2.532. PMC 42775. PMID 7530363.
- Andrews RM, Kubacka I, Chinnery PF, Lightowlers RN, Turnbull DM, Howell N (October 1999). "Reanalysis and revision of the Cambridge reference sequence for human mitochondrial DNA". Nature Genetics. 23 (2): 147. doi:10.1038/13779. PMID 10508508. S2CID 32212178.
- Ingman M, Kaessmann H, Pääbo S, Gyllensten U (December 2000). "Mitochondrial genome variation and the origin of modern humans". Nature. 408 (6813): 708–13. Bibcode:2000Natur.408..708I. doi:10.1038/35047064. PMID 11130070. S2CID 52850476.
- Finnilä S, Lehtonen MS, Majamaa K (June 2001). "Phylogenetic network for European mtDNA". American Journal of Human Genetics. 68 (6): 1475–84. doi:10.1086/320591. PMC 1226134. PMID 11349229.
- Maca-Meyer N, González AM, Larruga JM, Flores C, Cabrera VM (2003). "Major genomic mitochondrial lineages delineate early human expansions". BMC Genetics. 2: 13. doi:10.1186/1471-2156-2-13. PMC 55343. PMID 11553319.