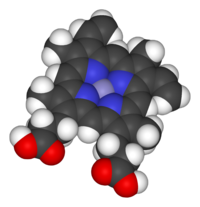
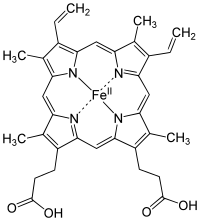
헤메
Heme헤메, 즉 헴은 혈류에서 산소를 결합하는 데 필요한 헤모글로빈의 전구체다.헤메는 골수와 간에서 모두 생체합성형이다.[1]
생화학적 용어로 heme는 "사면체 리간드 역할을 하는 포르피린에 조화된 철이온을, 축이온 1~2개에 결합하는 것"[2]이다.그 정의는 느슨하고, 많은 묘사들은 축 리간드를 생략한다.[3]야금단백질이 보형물 집단으로 배치한 야금포피린 중에서 헤메는 가장 널리 사용되는[4] 것 중 하나이며, 헤모프로테인으로 알려진 단백질군을 정의하고 있다.헤메스는 혈액 속의 붉은 색소인 헤모글로빈의 성분으로 가장 흔하게 인식되지만, 미오글로빈, 사이토크롬, 카탈레이트, 헤메 페록시다아제, 내피질산화물 시네타제와 같은 다른 생물학적으로 중요한 헤모글로빈에서도 발견된다.[5][6]
헴이라는 단어는 "혈액"을 뜻하는 그리스어 αἷμα 하이마에서 유래되었다.
함수
헤모프로테인은 이원자 기체의 운송, 화학적 촉매, 이원자 기체 감지, 전자 전달 등 다양한 생물학적 기능을 가지고 있다.헴 철은 전자전달이나 리독스 화학에서 전자의 원천이나 싱크 역할을 한다.과산화수소효소 반응에서 포르피린 분자는 또한 전자원으로서, 결합 링에서 급진전자를 분해할 수 있다.이산화질소를 운반하거나 검출할 때, 가스는 헤메 철에 결합한다.이원자 기체를 검출하는 동안, 기체 리간드를 헴 철에 결합하면 주변 단백질의 순응적 변화를 유도한다.[7]일반적으로, 이원자 가스는 철분 Fe(II)로서 감소된 hem에만 결합되는 반면, 대부분의 과산화효소는 Fe(III)와 Fe(IV) 사이의 주기와 미토콘드리아 redox, 산화 감소, Fe(II)와 Fe(III) 사이의 주기에 관여한다.
분자 산소가 출현하기 전에 조상 시아노박테리아와 같은 유기체에서 원시 유황에 기초한 광합성 경로에서의 전자전달은 혈단백질의 원래 진화적 기능이었다고 추측되어 왔다.[8]
헤모프로테인은 단백질 매트릭스 내에서 heme 매크로사이클의 환경을 수정하여 주목할 만한 기능적 다양성을 달성한다.[9]예를 들어 헤모글로빈이 효과적으로 산소를 조직에 전달할 수 있는 능력은 헤메 분자 근처에 위치한 특정 아미노산 잔류물 때문이다.[10]헤모글로빈은 pH가 높고 이산화탄소 농도가 낮을 때 폐의 산소에 역행적으로 결합한다.상황이 역전되면(낮은 pH와 높은 이산화탄소 농도) 헤모글로빈이 산소를 조직으로 방출한다.헤모글로빈의 산소 결합 친화력이 이산화탄소의 산도와 농도에 모두 반비례한다는 이 현상을 보어 효과라고 한다.[11]이러한 효과의 뒤에 있는 분자 메커니즘은 글로빈 체인의 견고한 조직이다; 헤메 그룹에 인접한 히스티딘 잔여물은 산성의 조건(작업 근육 등에서 CO가2 용해되어 발생하는 것 등)에서 양전하되어 헤메 그룹에서 산소를 방출한다.[12]
종류들
주요 골칫거리
헤메에는 생물학적으로 중요한 몇 가지 종류가 있다.
| 헤메 A | 헤메 B | 헤메 C | 헤메 오 | ||
|---|---|---|---|---|---|
| 퍼브켐 수 | 7888115 | 444098 | 444125 | 6323367 | |
| 화학식 | 촌페495664 | 촌페343244 | 욘스페3436442 | 촌페495854 | |
| C의3 기능 그룹 |  | –CH(OH)CHFAR2 | –CH=CH2 | –CH(사이슈타인-S-yl)CH3 | –CH(OH)CHFAR2 |
| C의8 기능 그룹 | –CH=CH2 | –CH=CH2 | –CH(사이슈타인-S-yl)CH3 | –CH=CH2 | |
| C의18 기능 그룹 | –CH=O | –CH3 | –CH3 | –CH3 | |
가장 흔한 타입은 헤메 B이다; 다른 중요한 타입은 헤메 A와 헤메 C를 포함한다.고립된 헴은 일반적으로 대문자로 지정되며 단백질에 묶인 헴은 소문자로 지정된다.시토크롬 a는 시토크롬 c 산화효소의 일부를 형성하는 막 단백질과 특정한 조합으로 헤메 A를 말한다.[15]
다른 헤미즈
- 다음의 포르피린 탄소번호 부여 시스템은 생화학자들이 사용하는 오래된 번호 부여 시스템이지 위 표에 나와 있는 IUPAC가 권장하는 1-24 번호 부여 시스템이 아니다.
- 헤메 l는 락토페록시디아제, 어시노필페록시디아제, 갑상선페록시디아제의 단백질에 공칭적으로 붙어 있는 헤메 B의 파생물이다.글루타밀-375와 aspartyl-225를 첨가한 락토페록시디아제는 각각 이러한 아미노산 잔류물과 헤메 1-메틸 그룹과 5-메틸 그룹 사이에 에스테르 결합을 형성한다.[16]이 두 메틸 그룹과 유사한 에스테르 결합은 어시노필과 갑상선 과산화지질에서 형성되는 것으로 생각된다.헤메 l는 동물 과산화효소의 중요한 특성 중 하나이다. 식물 과산화효소는 헤메 B를 포함한다.락토페록시디아제와 어시노필페록시디아제는 침입균과 바이러스의 파괴를 책임지는 보호 효소다.갑상선 과산화효소는 중요한 갑상선 호르몬의 생합성을 촉진하는 효소다.락토페록시디아제는 폐와 배설물에 침입한 유기체를 파괴하기 때문에 중요한 보호 효소로 여겨진다.[17]
- 헤메 m은 골수화효소의 활성 부위에서 공밸런스하게 결합된 헤메 B의 파생물이다.헤메 m은 락토페록시디아제, 어시노필페록시디아제 등 다른 포유류 과산화효소의 헤메 l에도 있는 헤메 1과 5메틸 그룹의 에스테르 결합 2개를 함유하고 있다.또한 메티오닐 아미노산잔류 유황과 헤메 2비닐군 사이에 독특한 설포나미드 이온 연계가 형성되어 염화물과 브롬화 이온을 차아염소산염과 하이포브로마이트로 쉽게 산화시킬 수 있는 독특한 능력을 이 효소에 부여한다.미엘로페록시디아제는 포유류 중성미자에 존재하며 침입 박테리아와 바이러스성 물질을 파괴하는 역할을 한다.그것은 아마도 "실수"에 의해 하이포브로마이트를 합성할 것이다.차아염소산염과 하이포브로마이트 모두 돌연변이 화합물인 할로겐화 뉴클레오시드의 생성을 담당하는 매우 반응성이 높은 종이다.[18][19]
- 헤메 D는 헤메 B의 또 다른 파생물이지만, 위치 6의 탄소에 있는 프로피온산 사이드 체인이 히드록시화 되어 있어 γ-스피롤락톤을 형성한다.또한 링 III는 새로운 락톤 그룹에 대한 순응 트랜스에서 위치 5에서 히드록실화된다.[20]헤메 D는 산소 장력이 낮을 때 많은 종류의 박테리아의 물로 산소를 감소시키는 곳이다.[21]
- 헤메 S는 2비닐 그룹 대신 위치 2에 정식 그룹을 두어 헤메 B와 관련이 있다.헤메 S는 몇 종의 해양 벌레의 헤모글로빈에서 발견된다.heme B와 heme S의 정확한 구조는 독일의 화학자 Hans Fischer에 의해 처음 설명되었다.[22]
사이토크롬의 이름은 일반적으로(그러나 항상은 아님) 그들이 포함하는 헤메의 종류를 반영한다: 시토크롬 a는 헤메 A를 포함하고, 시토크롬 c는 헤메 C를 포함하고 있다.이 협약은 heme A의 구조의 출판과 함께 처음 도입되었을지도 모른다.
헤메의 유형을 지정하는 데 대문자 사용
대문자로 hemes를 지정하는 관행은 Puustinen과 Wikstrom에[23] 의해 각주에 공식화되었는데, 어떤 조건에서 대문자를 사용해야 하는지를 설명하고 있다: "우리는 heme 구조를 고립된 것으로 묘사하기 위해 대문자를 사용하는 것을 선호한다.소문자는 개별 단백질 결합 헤메 그룹(예: 시토크롬 b, bc51 콤플렉스의 헤메 c1, aa3 콤플렉스의 헤메 a 등3)을 설명할 뿐만 아니라 시토크롬과 효소에도 자유롭게 사용될 수 있다."즉, 화학적 화합물은 대문자로 지정되지만 소문자가 있는 구조물에 특정 사례가 지정될 수 있다.따라서 구조물에 두 개의 A 헤메(heme a와 heme3 a)가 있는 시토크롬 산화효소는 몰 단백질당 두 개의 heme A 몰을 함유하고 있다.hemes bH, bL, c와1 함께 시토크롬 bc는1 heme B와 heme C를 2:1 비율로 포함하고 있다.이러한 관행은 Caughey와 York의 논문에서 시토크롬 aa3의 heme에 대한 새로운 격리 절차의 생산물을 이전의 준비와 구별하기 위해 heme A로 지정했던 것으로 보인다: "우리 제품은 hemi의 감소에 의해 다른 노동자들이 용해에서 얻은 heme와 모든 면에서 동일하지 않다.n a는 이전의 (2)와 같이 격리된 경우(2).이 때문에 뚜렷한 차이가 합리화될 수 있을 때까지 제품 heme A를 지정하겠다"[24]고 말했다.이후 논문에서,[25] Caughey의 그룹은 A뿐만 아니라 고립된 Heme B와 C에 대문자를 사용한다.
합성
헤메를 생산하는 효소성 과정은 모든 매개체가 화학적으로 포피린으로 분류되는 테트라피롤이기 때문에 적절하게 포피린 합성이라고 불린다.그 과정은 생물학 전반에 걸쳐 매우 보존되어 있다.인간에게 있어서 이 길은 거의 독점적으로 헤미를 형성하는 역할을 한다.박테리아에서는 또한, 공동 인자 F430과 코발라민(비타민 B12)과 같은 더 복잡한 물질을 생산한다.[26]
경로는 아미노산 글리신에서 Δ-아미노레불린산(dALA 또는 ΔALA)이 합성되고 구연산 사이클(Krebs cycle)에서 수시닐-CoA가 합성되어 시작된다.이 반응을 일으키는 속도제한 효소인 ALA 싱타아제는 포도당과 헤메 농도에 의해 부정적으로 조절된다.헤미 또는 헤민에 의한 ALA의 억제 메커니즘은 mRNA 합성의 안정성을 감소시키고 미토콘드리아에서 mRNA의 섭취를 감소시킴으로써 이루어진다.이 메커니즘은 치료상 중요하다: 헤메 아르진 또는 헤마틴과 포도당을 주입하면 ALA 싱타아제의 전사 감소를 통해 이 과정의 신진대사의 선천적인 오류가 있는 환자의 급성 간헐성 포르피리아 공격을 중단할 수 있다.[27]
헤메 합성에 주로 관여하는 기관들은 비록 모든 세포가 헤메를 적절하게 기능하도록 요구하지만 간(시스템 헤메 풀에 따라 합성 속도가 매우 가변적인 것)과 골수(헤메의 합성 속도가 상대적으로 일정하고 글로빈 체인의 생산에 의존하는 것)이다.그러나 유독성 때문에 철분(Hx)과 같은 단백질이 철분 생리적 저장소를 유지하도록 도와야 합성에 쓰일 수 있다.[28]헤메는 빌리루빈 대사 과정에서 헤모글로빈의 카타볼리즘에서 중간 분자로 보인다.헤메 합성에 있어 다양한 효소의 결함은 포르피리아라고 불리는 장애의 그룹으로 이어질 수 있는데, 이것들은 급성 간헐성 포르피리아, 선천성 적혈구 포르피리아, 포르피리아 커타네아 타르다, 유전성 코프로포피리아, 바리게이트 포르피리아, 적혈구 프로포폴포피리아를 포함한다.[29][citation needed]
식품 합성
식물성 고기 대체물의 생산자인 임파서블 푸드는 콩 뿌리 레그헤모글로빈과 효모를 포함하는 가속 헤메 합성 과정을 사용하며, 그 결과로 나온 헤메를 무육(비건) 임파서블 버거 패티와 같은 품목에 첨가한다.레그헤모글로빈 생성을 위한 DNA는 콩뿌리 결절에서 추출해 효모세포로 표현해 고기 없는 버거에 사용하기 위한 헤메를 과잉 생산했다.[30]이 과정은 결과물에서 육질의 맛을 낸다고 주장한다.[31][32]
분해
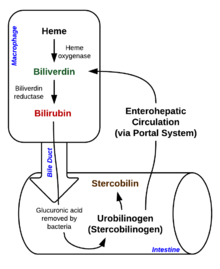
비장의 대식세포 내부에서 열화가 시작되어 혈액순환에서 오래되고 손상된 적혈구가 제거된다.첫 번째 단계에서 헤메는 효소 헤메 산소효소(HO)에 의해 빌리버딘으로 전환된다.[33] NADPH는 환원제로 사용되고, 분자 산소는 반응으로 들어가며, 일산화탄소(CO)는 생성되고, 철분자로부터 철분자 이온(Fe2+)로 방출된다.[34] CO는 세포 전달자 역할을 하며 혈관확장 작용에서 기능한다.[35]
또한, 헤임 열화는 산화 스트레스에 대한 진화적으로 보존된 반응으로 보인다.간단히 말해서, 세포가 활성산소에 노출되었을 때, 스트레스 반응 헤메 산소효소-1 (HMOX1) 이소엔자임 발현이 빠르게 유도되어 heme를 카타볼라화한다(아래 참조).[36]세포가 산화 스트레스에 반응하여 헤메를 분해하는 능력을 기하급수적으로 증가시켜야 하는 이유는 여전히 불분명하지만 이는 자유 헤메의 해로운 영향을 피하는 세포 보호 반응의 일부인 것으로 보인다.다량의 무료 헤미가 축적되면 헤메 해독/분해 시스템이 압도되어 헤메는 그 해로운 효과를 발휘할 수 있게 된다.[28]
| 헤메 | 헤메 산소효소-1 | 빌리버딘2+ + 페 | |
 |  | ||
| H+ + NADPH + O2 | NADP+ + CO | ||
 | |||
두 번째 반응에서 빌리버딘은 빌리버딘 환원효소(BVR)에 의해 빌리루빈으로 전환된다.[37]
| 빌리버딘 | 빌리버딘 환원효소 | 빌리루빈 | |
 |  | ||
| H+ + NADPH | NADP+ | ||
 | |||
빌리루빈은 단백질(세럼 알부민)에 묶인 원활한 확산에 의해 간으로 운반되며, 글루쿠론산과 결합하여 수용성이 높아진다.반응은 UDP-글루쿠로노실전달효소에 의해 촉매된다.[38]
| 빌리루빈 | UDP-글루쿠로노실전달효소 | 빌리루빈 디글루쿠로니드 | |
 |  | ||
| UDP-글루큐로니드 2개 | UMP 2개i + P | ||
 | |||
이러한 형태의 빌리루빈은 간에서 담즙으로 배설된다.빌리루빈을 간에서 담도운하술리까지 배설하는 것은 적극적이고 에너지에 의존하며 요율을 제한하는 과정이다.장내세균 디콘쥬게이트 빌리루빈 디글루쿠로니드(diglucuronide)와 빌리루빈을 우로비린균으로 전환한다.일부 우로비린겐은 장내 세포에 흡수되어 신장으로 운반되어 소변과 함께 배설된다(우로비린겐의 산화제로서 소변의 황색에 책임이 있다).나머지는 소화관을 타고 내려가 스테르코빌리노겐으로 전환된다.이것은 스테르코빌린으로 산화되는데, 스테르코빌린은 배설되어 대변의 갈색을 담당한다.[39]
건강과 질병에 있어서
동종요법 하에서 헤미의 반응성은 헤모프로테인의 "heme pockets"에 삽입되어 제어된다.[citation needed]그러나 산화적 스트레스 하에서 헤모글로빈과 같은 일부 헤모프로테우스는 그들의 헤미 보형물 그룹을 방출할 수 있다.[40][41]이런 식으로 생산되는 비단백질 결합(자유) 헤미는 높은 세포독성이 되는데, 아마도 그것의 원형포도피린 IX 링 안에 들어 있는 철 원자로 인해 유리기의 생성을 제한 없이 촉진시키는 펜톤의 시약 역할을 할 수 있을 것이다.[42]단백질의 산화와 집적, 지질 과산화물에 의한 세포독성 과산화지질의 형성을 촉진하고 산화 스트레스를 통해 DNA를 손상시킨다.지질학적 특성 때문에 미토콘드리아, 핵 등 유기농의 지질 빌레이어를 손상시킨다.[43]이러한 자유 헤메의 성질은 말라리아나[44] 패혈증과 같은 특정 염증성 질환의 병원생성에 중요한 역할을 하는 해로운 효과인 친염증 작용제에 반응하여 프로그램된 세포사멸을 겪기 위해 다양한 세포사멸을 감수할 수 있다.[45]
암
고기로부터 공급된 헴 철분을 많이 섭취하는 것과 대장암의 위험 증가 사이에는 연관성이 있다.[46]붉은 고기의 heme 함량은 닭고기 등 흰 고기의 10배에 이른다.[47]2019년 한 리뷰는 heme 철분 섭취가 유방암 위험 증가와 관련이 있다는 것을 발견했다.[48]
유전자
다음 유전자는 헤메를 만드는 화학적 경로의 일부분이다.
- ALAD: 아미노레불린산, Δ-, 탈수효소(결핍은 알라-탈수효소 결핍증을 유발한다)[49]
- ARAS1: 아미노레불산염, Δ-, 신타제 1
- ARAS2: 아미노레불산염, Δ-, 신타제 2 (결핍은 시데로블라스틱/고혈색성 빈혈의 원인)
- CAPANS: 코프로포피리노겐산산화효소(결핍이 유전성 코프로포피리뇨증을 유발함)[50]
- FECH: 페로셀라타아제(결핍은 적혈구 양성포폴피리아를 유발한다)
- HMBS: 히드록시메틸빌레인 신타아제(결핍이 급성 간헐성 포르피리아를 유발함)[51]
- PUPSO: 프로토폴피피리노겐 산화효소(결핍이 바리게이트 포르피리아를 유발함)[52]
- UROD: Uroporphyinogen decarboxylase (결핍으로 인해 포르피리아 cutanea tarda)[53]
- UROS: uroporphyinogen III synthase (결핍은 선천성 적혈구공피증을 유발한다)
참고 및 참조
- ^ Bloomer, Joseph R. (1998). "Liver metabolism of porphyrins and haem". Journal of Gastroenterology and Hepatology. 13 (3): 324–329. doi:10.1111/j.1440-1746.1998.01548.x. PMID 9570250. S2CID 25224821.
- ^ Chemistry, International Union of Pure and Applied (2009). "Hemes (heme derivatives)". IUPAC Compendium of Chemical Terminology. iupac.org. IUPAC. doi:10.1351/goldbook.H02773. ISBN 978-0-9678550-9-7. Archived from the original on 22 August 2017. Retrieved 28 April 2018.
- ^ 표준 생화학 본문은 헤메를 "헤메 단백질의 철-포피린 보형물 집단"으로 정의한다(넬슨, D. L.; 콕스, M. M. "레닝거, 생화학 원리" 3차 개정).게시할 가치가 있는 항목:2000년 뉴욕ISBN 1-57259-153-6)
- ^ Poulos, Thomas L. (2014-04-09). "Heme Enzyme Structure and Function". Chemical Reviews. 114 (7): 3919–3962. doi:10.1021/cr400415k. ISSN 0009-2665. PMC 3981943. PMID 24400737.
- ^ Paoli, M. (2002). "Structure-function relationships in heme-proteins" (PDF). DNA Cell Biol. 21 (4): 271–280. doi:10.1089/104454902753759690. hdl:20.500.11820/67200894-eb9f-47a2-9542-02877d41fdd7. PMID 12042067.
- ^ Alderton, W.K. (2001). "Nitric oxide synthases: structure, function and inhibition". Biochem. J. 357 (3): 593–615. doi:10.1042/bj3570593. PMC 1221991. PMID 11463332.
- ^ Milani, M. (2005). "Structural bases for heme binding and diatomic ligand recognition in truncated hemoglobins". J. Inorg. Biochem. 99 (1): 97–109. doi:10.1016/j.jinorgbio.2004.10.035. PMID 15598494.
- ^ Hardison, R. (1999). "The Evolution of Hemoglobin: Studies of a very ancient protein suggest that changes in gene regulation are an important part of the evolutionary story". American Scientist. 87 (2): 126. doi:10.1511/1999.20.809.
- ^ Poulos, T. (2014). "Heme Enzyme Structure and Function". Chem. Rev. 114 (7): 3919–3962. doi:10.1021/cr400415k. PMC 3981943. PMID 24400737.
- ^ Thom, C. S. (2013). "Hemoglobin Variants: Biochemical Properties and Clinical Correlates". Cold Spring Harbor Perspectives in Medicine. 3 (3): a011858. doi:10.1101/cshperspect.a011858. PMC 3579210. PMID 23388674.
- ^ Bohr; Hasselbalch, Krogh. "Concerning a Biologically Important Relationship - The Influence of the Carbon Dioxide Content of Blood on its Oxygen Binding". Archived from the original on 2017-04-18.
{{cite journal}}:Cite 저널은 필요로 한다.journal=(도움말) - ^ Ackers, G. K.; Holt, J. M. (2006). "Asymmetric cooperativity in a symmetric tetramer: human hemoglobin". J. Biol. Chem. 281 (17): 11441–3. doi:10.1074/jbc.r500019200. PMID 16423822. S2CID 6696041.
- ^ Caughey, W. S.; Smythe, G. E.; O'Keeffe, D. H.; Maskasky, J. E.; Smith, M. L. (1975). "Heme A of Cytochrome c Oxidase: Structure and properties: comparisons with hemes B, C, and S and derivatives". J. Biol. Chem. 250 (19): 7602–7622. doi:10.1016/S0021-9258(19)40860-0. PMID 170266.
- ^ Hegg, Eric L. (2004). "Heme A Synthase Does Not Incorporate Molecular Oxygen into the Formyl Group of Heme A". Biochemistry. 43 (27): 8616–8624. doi:10.1021/bi049056m. PMID 15236569.
- ^ Yoshikawa, S. (2012). "Structural studies on bovine heart cytochrome c oxidase". Biochim. Biophys. Acta. 1817 (4): 579–589. doi:10.1016/j.bbabio.2011.12.012. PMID 22236806.
- ^ Rae, T.; Goff, H. (1998). "The heme prosthetic group of lactoperoxidase. Structural characteristics of heme l and heme l-peptides". The Journal of Biological Chemistry. 273 (43): 27968–27977. doi:10.1074/jbc.273.43.27968. PMID 9774411. S2CID 25780396.
- ^ Purdy, M.A. (1983). "Effect of growth phase and cell envelope structure on susceptibility of Salmonella triumphant to the lactoperoxidase-thiocyanate-hydrogen peroxide system". Infect. Immun. 39 (3): 1187–95. doi:10.1128/IAI.39.3.1187-1195.1983. PMC 348082. PMID 6341231.
- ^ Ohshima, H. (2003). "Chemical basis of inflammation-induced carcinogenesis". Arch. Biochem. Biophys. 417 (1): 3–11. doi:10.1016/s0003-9861(03)00283-2. PMID 12921773.
- ^ Henderson, J.P. (2003). "Phagocytes produce 5-chlorouracil and 5-bromouracil, two mutagenic products of myeloperoxidase, in human inflammatory tissue". J. Biol. Chem. 278 (26): 23522–8. doi:10.1074/jbc.m303928200. PMID 12707270. S2CID 19631565.
- ^ Murshudov, G.; Grebenko, A.; Barynin, V.; Dauter, Z.; Wilson, K.; Vainshtein, B.; Melik-Adamyan, W.; Bravo, J.; Ferrán, J.; Ferrer, J. C.; Switala, J.; Loewen, P. C.; Fita, I. (1996). "Structure of the heme d of Penicillium vitale and Escherichia coli catalases" (PDF). The Journal of Biological Chemistry. 271 (15): 8863–8868. doi:10.1074/jbc.271.15.8863. PMID 8621527.
- ^ Belevich, I. (2005). "Oxygenated complex of cytochrome bd from Escherichia coli: stability and photolability". FEBS Letters. 579 (21): 4567–70. doi:10.1016/j.febslet.2005.07.011. PMID 16087180. S2CID 36465802.
- ^ Fischer, H.; Orth, H. (1934). Die Chemie des Pyrrols. Liepzig: Ischemia Verlagsgesellschaft.
- ^ Puustinen A, Wikström M. (1991). "The heme groups of cytochrome o from Escherichia coli". Proc. Natl. Acad. Sci. U.S.A. 88 (14): 6122–6. Bibcode:1991PNAS...88.6122P. doi:10.1073/pnas.88.14.6122. PMC 52034. PMID 2068092.
- ^ Caughey WS, York JL (1962). "Isolation and some properties of the green heme of cytochrome oxidase from beef heart muscle". J. Biol. Chem. 237 (7): 2414–6. doi:10.1016/S0021-9258(19)63456-3. PMID 13877421.
- ^ Caughey WS, Smythe GA, O'Keeffe DH, Maskasky JE, Smith ML (1975). "Heme A of cytochrome c oxidase. Structure and properties: comparisons with hemes B, C, and S and derivatives". J. Biol. Chem. 250 (19): 7602–22. doi:10.1016/S0021-9258(19)40860-0. PMID 170266.
- ^ Battersby, Alan R. (2000). "Tetrapyrroles: The pigments of life". Natural Product Reports. 17 (6): 507–526. doi:10.1039/B002635M. PMID 11152419.
- ^ Sridevi, Kolluri (28 April 2018). Upregulation of Heme Pathway Enzyme ALA Synthase-1 by Glutethimide and 4,6-Dioxoheptanoic Acid and Downregulation by Glucose and Heme: A Dissertation. EScholarship@UMMS (Thesis). University of Massachusetts Medical School. doi:10.13028/yyrz-qa79. Archived from the original on 8 August 2016. Retrieved 28 April 2018.
- ^ a b Kumar, Sanjay; Bandyopadhyay, Uday (July 2005). "Free heme toxicity and its detoxification systems in human". Toxicology Letters. 157 (3): 175–188. doi:10.1016/j.toxlet.2005.03.004. PMID 15917143.
- ^ Puy, Hervé; Gouya, Laurent; Deybach, Jean-Charles (March 2010). "Porphyrias". The Lancet. 375 (9718): 924–937. doi:10.1016/S0140-6736(09)61925-5. PMID 20226990. S2CID 208791867.
- ^ Fraser, Rachel Z.; Shitut, Mithila; Agrawal, Puja; Mendes, Odete; Klapholz, Sue (2018-04-11). "Safety Evaluation of Soy Leghemoglobin Protein Preparation Derived FromPichia pastoris, Intended for Use as a Flavor Catalyst in Plant-Based Meat". International Journal of Toxicology. 37 (3): 241–262. doi:10.1177/1091581818766318. ISSN 1091-5818. PMC 5956568. PMID 29642729.
- ^ "Inside the Strange Science of the Fake Meat That 'Bleeds'". Wired. 2017-09-20. Archived from the original on 24 March 2018. Retrieved 28 April 2018.
- ^ "Silicon Valley gets a taste for food". The Economist. 2015-03-05. ISSN 0013-0613. Retrieved 2019-04-08.
- ^ Maines, Mahin D. (July 1988). "Heme oxygenase: function, multiplicity, regulatory mechanisms, and clinical applications". The FASEB Journal. 2 (10): 2557–2568. doi:10.1096/fasebj.2.10.3290025. ISSN 0892-6638. PMID 3290025.
- ^ Lehninger's Principles of Biochemistry (5th ed.). New York: W. H. Freeman and Company. 2008. pp. 876. ISBN 978-0-7167-7108-1.
- ^ Hanafy, K.A. (2013). "Carbon Monoxide and the brain: time to rethink the dogma". Curr. Pharm. Des. 19 (15): 2771–5. doi:10.2174/1381612811319150013. PMC 3672861. PMID 23092321.
- ^ Abraham, N.G.; Kappas, A. (2008). "Pharmacological and clinical aspects of heme oxygenase". Pharmacol. Rev. 60 (1): 79–127. doi:10.1124/pr.107.07104. PMID 18323402. S2CID 12792155.
- ^ Florczyk, U.M.; Jozkowicz, A.; Dulak, J. (January–February 2008). "Biliverdin reductase: new features of an old enzyme and its potential therapeutic significance". Pharmacological Reports. 60 (1): 38–48. PMC 5536200. PMID 18276984.
- ^ King, C.; Rios, G.; Green, M.; Tephly, T. (2000). "UDP-Glucuronosyltransferases". Current Drug Metabolism. 1 (2): 143–161. doi:10.2174/1389200003339171. PMID 11465080.
- ^ Helmenstine, Anne Marie. "The Chemicals Responsible for the Color of Urine and Feces". ThoughtCo. Retrieved 2020-01-24.
- ^ Bunn, H. F.; Jandl, J. H. (Sep 1966). "Exchange of heme among hemoglobin molecules". Proc. Natl. Acad. Sci. USA. 56 (3): 974–978. Bibcode:1966PNAS...56..974B. doi:10.1073/pnas.56.3.974. PMC 219955. PMID 5230192.
- ^ Smith, M. L.; Paul, J.; Ohlsson, P. I.; Hjortsberg, K.; Paul, K. G. (Feb 1991). "Heme-protein fission under nondenaturing conditions". Proc. Natl. Acad. Sci. USA. 88 (3): 882–886. Bibcode:1991PNAS...88..882S. doi:10.1073/pnas.88.3.882. PMC 50918. PMID 1846966.
- ^ Everse, J.; Hsia, N. (1197). "The toxicities of native and modified hemoglobins". Free Radical Biology and Medicine. 22 (6): 1075–1099. doi:10.1016/S0891-5849(96)00499-6. PMID 9034247.
- ^ Kumar, Sanjay; Bandyopadhyay, Uday (July 2005). "Free heme toxicity and its detoxification systems in humans". Toxicology Letters. 157 (3): 175–188. doi:10.1016/j.toxlet.2005.03.004. PMID 15917143.
- ^ Pamplona, A.; Ferreira, A.; Balla, J.; Jeney, V.; Balla, G.; Epiphanio, S.; Chora, A.; Rodrigues, C. D.; Gregoire, I. P.; Cunha-Rodrigues, M.; Portugal, S.; Soares, M. P.; Mota, M. M. (Jun 2007). "Heme oxygenase-1 and carbon monoxide suppress the pathogenesis of experimental cerebral malaria". Nature Medicine. 13 (6): 703–710. doi:10.1038/nm1586. PMID 17496899. S2CID 20675040.
- ^ Larsen, R.; Gozzelino, R.; Jeney, V.; Tokaji, L.; Bozza, F. A.; Japiassú, A. M.; Bonaparte, D.; Cavalcante, M. M.; Chora, A.; Ferreira, A.; Marguti, I.; Cardoso, S.; Sepúlveda, N.; Smith, A.; Soares, M. P. (2010). "A central role for free heme in the pathogenesis of severe sepsis". Science Translational Medicine. 2 (51): 51ra71. doi:10.1126/scitranslmed.3001118. PMID 20881280. S2CID 423446.
- ^ Bastide, N. M.; Pierre, F. H.; Corpet, D. E. (2011). "Heme iron from meat and risk of colorectal cancer: a meta-analysis and a review of the mechanisms involved" (PDF). Cancer Prev. Res. 4 (2): 177–184. doi:10.1158/1940-6207.CAPR-10-0113. PMID 21209396. S2CID 4951579. Archived (PDF) from the original on 2015-09-25.
- ^ Bastide, Nadia M.; Pierre, Fabrice H. F.; Corpet, Denis E. (1 February 2011). "Heme Iron from Meat and Risk of Colorectal Cancer: A Meta-analysis and a Review of the Mechanisms Involved". Cancer Prevention Research. 4 (2): 177–184. doi:10.1158/1940-6207.CAPR-10-0113. PMID 21209396. S2CID 4951579. Archived from the original on 11 October 2017. Retrieved 28 April 2018 – via cancerpreventionresearch.aacrjournals.org.
- ^ Chang, Vicky C; Cotterchio, Michelle; Khoo, Edwin (2019). "Iron intake, body iron status, and risk of breast cancer: a systematic review and meta-analysis". BMC Cancer. 19 (1): 543. doi:10.1186/s12885-019-5642-0. PMC 6555759. PMID 31170936.
{{cite journal}}: CS1 maint : 복수이름 : 작성자 목록(링크) - ^ Plewinska, Magdalena; Thunell, Stig; Holmberg, Lars; Wetmur, James; Desnick, Robert (1991). "delta-Aminolevulinate dehydratase deficient porphyria: identification of the molecular lesions in a severely affected homozygote". American Journal of Human Genetics. 49 (1): 167–174. PMC 1683193. PMID 2063868.
- ^ Aurizi, C.; Lupia Palmieri, G.; Barbieri, L.; Macri, A.; Sorge, F.; Usai, G.; Biolcati, G. (February 2009). "Four novel mutations of the coproporphyrinogen III oxidase gene". Cellular and Molecular Biology. 55 (1): 8–15. PMID 19267996.
- ^ Bustad, H. J.; Vorland, M.; Ronneseth, E.; Sandberg, S.; Martinez, A.; Toska, K. (August 8, 2013). "Conformational stability and activity analysis of two hydroxymethylbilane synthase mutants, K132N and V215E, with different phenotypic association with acute intermittent porphyria". Bioscience Reports. 33 (4): 617–626. doi:10.1042/BSR20130045. PMC 3738108. PMID 23815679.
- ^ Martinez di Montemuros, F.; Di Pierro, E.; Patti, E.; Tavazzi, D.; Danielli, M. G.; Biolcati, G.; Rocchi, E.; Cappllini, M. D. (December 2002). "Molecular characterization of porphyrias in Italy: a diagnostic flow-chart". 48 (8): 867–876.
{{cite journal}}:Cite 저널은 필요로 한다.journal=(도움말) - ^ Badenas, C.; To Figueras, J.; Phillips, J. D.; Warby, C. A.; Muñoz, C.; Herrero, C. (April 2009). "Identification and characterization of novel uroporphyrinogen decarboxylase gene mutations in a large series of porphyria cutanea tarda patients and relatives". Clinical Genetics. 75 (4): 346–353. doi:10.1111/j.1399-0004.2009.01153.x. PMC 3804340. PMID 19419417.