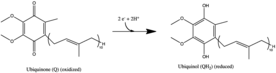호흡 복합체 I
Respiratory complex I호흡 복합체 I, EC 7.1.1.2(NADH:ubiquinone 산화환원효소, Type I NADH 탈수소효소 및 미토콘드리아 복합체 I로도 알려져 있음)는 박테리아에서 인간에 이르는 많은 유기체의 호흡 사슬 중 첫 번째 큰 단백질 복합체이다.NADH에서 코엔자임 Q10(CoQ10)으로 전자의 전달을 촉매하고 진핵생물 또는 박테리아의 혈장막에서 양자를 이동시킨다.
| 호흡 복합체 I | |
|---|---|
| 식별자 | |
| 기호. | 호흡 복합체 I |
| OPM 슈퍼 패밀리 | 246 |
| OPM단백질 | 6g72 |
| 막질 | 255 |
| NADH:유비퀴논 환원효소(H-translationing+). | |||||||||
|---|---|---|---|---|---|---|---|---|---|
| 식별자 | |||||||||
| EC 번호 | 1.6.5.3 | ||||||||
| 데이터베이스 | |||||||||
| 인텐츠 | IntEnz 뷰 | ||||||||
| 브렌다 | 브렌다 엔트리 | ||||||||
| ExPASy | NiceZyme 뷰 | ||||||||
| 케그 | KEGG 엔트리 | ||||||||
| 메타사이크 | 대사 경로 | ||||||||
| 프라이머리 | 프로필 | ||||||||
| PDB 구조 | RCSB PDB PDBe PDBum | ||||||||
| 진 온톨로지 | AmiGO / QuickGO | ||||||||
| |||||||||
이 효소는 세포의 정상적인 기능을 위해 필수적이며, 그 하위 단위의 돌연변이는 광범위한 유전 신경근 및 대사 장애를 야기합니다.이 효소의 결함은 허혈/재유입 손상(뇌졸중 및 심근경색), 파킨슨병 등과 같은 여러 병리학적 과정의 발달에 책임이 있다.
기능.
복합체 I은 미토콘드리아 전자전달계의 첫 번째 효소이다.전자전달계에는 NADH, 유비퀴논 산화환원효소(복합체 I), 코엔자임 Q – 시토크롬c 환원효소(복합체 III), 시토크롬c 산화효소(복합체 [1]IV)의 세 가지 에너지 전달 효소가 있다.복합체 I은 전자전달계에서 [2]가장 크고 복잡한 효소이다.
복합체 I에 의해 촉매되는 반응은 다음과 같습니다.
- NADH + H+ + CoQ + 4H+in → NAD+ + CoQH2 + 4H+out
이 과정에서 복합체는 산화 NADH [3][4][5]분자당 내부 막을 가로질러 4개의 양성자를 이동시켜 ATP 생성에 사용되는 전기화학적 전위차를 만드는 데 도움을 준다.대장의 복잡한 나는(NADH탈수소 효소) 수가의 양성자 전좌에 같은 방향으로 설립되 Δψ, 새끼가에서 실시한 상태, 연결 장치 이온은 수소 이온.반대 방향으로[6]Na+ 이송 및 비록 Na+ 또는 양성자 촉매 운송 활동에 필요한 것이 아니다, 그것의 배치를 늘려 졌다.후자.Paracoccus+ denitrificans complex I에 의해 H가 전이되었으나, 이 경우 H+ 수송은 Na의 영향을+ 받지 않았으며+, Na 수송은 관찰되지 않았다.대장균 복합체 I은 Rhodothermus marinus 복합체 I에서 관찰된 바와 같이 두 개의 에너지 결합 부위(한+ 개는 Na 독립적, 다른 하나는 Nad의존적+)를 가지며, P. 데니트리판스 효소의 결합 메커니즘은 완전히+ Na 독립적이다.또 다른 운반체가 Na의 흡수를 촉매할+ 수도 있다. 양성자 펌핑에 의한 복합 I 에너지 전달은 R. 마리누스 효소에 독점적이지 않을 수 있다.Na/H+ 안티포트 활성은+ 복합체 [6]I의 일반적인 특성이 아닌 것으로 보인다.그러나 복합체 I의 Na-translation+ 활성의 존재는 여전히 의문이다.
이 반응은 높은 막 전위가 존재하는 경우 유비퀴놀에 의한 호기성 숙신산염 지지+ NAD 환원이라고 불리며 역전될 수 있지만, 정확한 촉매 메커니즘은 알려지지 않았다.이 반응의 원동력은 ATP 가수분해 또는 석신산염 [7]산화 중 복합체 III 및 IV에 의해 유지될 수 있는 막을 가로지르는 전위이다.
콤플렉스 나는 아포토시스를 [8]유발하는 역할을 할 수 있다.실제로 체세포 발달 [9]과정에서 미토콘드리아 활동과 프로그램 세포사망(PCD) 사이에 상관관계가 있는 것으로 나타났다.
복합체 I은 Mrp 슈퍼패밀리를 운반하는 Na의+ 구성원인 NADH 탈수소효소(NDH) 패밀리(TC# 3.D.1)와+ 상동하지 않는다.
2개의 NADH 분자가 NAD+로 산화된 결과, 3개의 ATP 분자는 호흡 사슬의 복합체 IV에 의해 생성될 수 있다.
메커니즘
전체적인 메커니즘
모든 산화환원 반응은 복합체 I의 친수성 영역에서 일어난다. NADH는 처음에 복합체 I에 결합하고 효소의 플라빈 모노뉴클레오티드(FMN) 보철물 그룹으로 2개의 전자를 전달하여 FMNH를2 생성한다.그런 다음 전자는 일련의 철-황([10]Fe-S) 클러스터를 통해 FMN을 통해 전달되고, 마지막으로 코엔자임 Q10(유비퀴논)으로 전달됩니다.이 전자 흐름은 단백질의 산화환원 상태를 변화시키고, 이온화 가능한 측쇄의 pK 값을 바꾸는 단백질의 구조 변화를 유발하며, 4개의 수소 이온을 미토콘드리아 매트릭스 [11]밖으로 내보낸다.유비퀴논(CoQ)은 두 개의 전자를 받아들여2 유비퀴놀(CoQH)[1]로 환원한다.
전자 전달 메커니즘
유비퀴논 환원에 앞서 제안된 전자 수송 경로는 다음과 같다. NADH – FMN – N3 – N1b – N4 – N5 – N6a – N6b – N2 – Q. 여기서 Nx는 철 황 [10]클러스터에 대한 라벨 표시 규칙이다.N2 클러스터의 높은 환원 잠재력과 체인 내 다른 클러스터의 상대적 근접성은 단백질에서 장거리(NADH에서 N2 철-황 클러스터로의 전달 속도 약 100μs)[12][13]에 걸쳐 효율적인 전자 전달을 가능하게 한다.
복합체 I의 평형 역학은 주로 퀴논 산화환원 사이클에 의해 구동된다.높은 양성자 원동력 조건(따라서 유비퀴놀 농축 풀)에서 효소는 역방향으로 실행됩니다.유비퀴놀은 유비퀴논으로 산화되고, 그 결과 방출된 양성자는 양성자의 원동력을 [14]감소시킨다.
양성자 전위 메커니즘
복합체 I에서 양성자 전위와 전자 전달의 결합은 현재 직접적(복합체 III [10]및 IV의 헴 그룹에서와 같이 수소 펌프의 레독스 중간체)이 아닌 간접적(장거리 구조 변화)으로 제안되고 있다.복합체 I의 소수성 영역의 구조는 기계적으로 상호 연결된 다중 양성자 전달체를 보여준다.이 장거리 구조 변화에 기여하는 것으로 생각되는 세 가지 중심 구성 요소는 pH 결합 N2 철-황 클러스터, 퀴논 환원 및 막 팔의 막 통과 나선 서브유닛입니다.유비퀴논의 환원 동안 '연결봉'으로 연결된 막간 전달체를 구동하기 위한 구조 변화의 변환은 산화 NADH당 펌핑된 4개의 양성자 중 2개 또는 3개를 설명할 수 있다.나머지 양성자는 유비퀴논 결합 부위에서 직접 결합을 통해 펌핑되어야 한다.직접 및 간접 결합 메커니즘이 4개의 [15]양성자의 펌핑을 설명하는 것이 제안된다.
N2 클러스터의 인근 시스테인 잔기에 대한 근접성은 인근 나선형의 감소에 따른 구조 변화를 초래하고, 전체 단백질 구조에 [16]작지만 중요한 변화를 초래한다.전자 전달에 대한 추가적인 전자 상사성 공명 연구는 후속 CoQ 감소 동안 방출되는 에너지의 대부분이 세미퀴논으로부터 최종 유비퀴놀 형성 단계에 있다는 것을 보여주며, "단일 스트로크+" H 전위 메커니즘에 대한 증거를 제공한다(즉, 4개의 양성자 모두가 막에서 이동함).e동시에)[14][17]대안 이론은 각 환원 단계(세미퀴논과 유비퀴놀)가 막간 공간에 [18][19]두 개의 양성자의 뇌졸중을 일으키는 "투 스트로크 메커니즘"을 제안한다.
막 도메인에 국소화된 유비퀴놀은 막 팔의 음전하 잔류물과 상호작용하여 구조 [10]변화를 안정화시킨다.멤브레인 [20]암에서 보존된 Asp 잔류물의 증거를 사용하여 안티포터 메커니즘(Na/H+ 스왑)이+ 제안되었다.Lys, Glu 및 그의 잔류물의 존재는 [10]잔류물의 pK에a 의해 구동되는 양성자 게이트(막 전체에 걸친 양성자 생성 후 탈양성자 이벤트)를 가능하게 한다.
구성 및 구조
NADH:유비퀴논산화환원효소는 호흡복합체 중 가장 크다.포유동물에서 효소는 44개의 수용성 말초막 단백질을 포함하고 있으며, 이들은 일체형 막 구성 요소에 고정되어 있다.특히 기능적으로 중요한 것은 플라빈 보철기(FMN)와 8개의 철-황 클러스터(FeS)입니다.44개의 서브유닛 중 7개는 미토콘드리아 [21][22][23]게놈에 의해 암호화된다.
구조는 긴 막 도메인(약 60개의 막 나선형)과 친수(또는 말초) 도메인을 가진 "L" 모양이며, 여기에는 알려진 모든 산화환원 중심과 NADH 결합 [24]부위가 포함된다.NADH 탈수소효소 I을 구성하는 대장균 단백질 13개 모두 nuo 오퍼론 내에 암호화되어 있으며, 미토콘드리아 복합체 I 서브유닛과 상동한다.역포터형 서브유닛 NuoL/M/N은 각각 14개의 보존막간(TM)나선을 포함한다.이들 중 2개는 불연속이지만 서브유닛 NuoL은 도메인의 전장에 걸쳐 110Ω 길이의 양성α-나선을 포함한다.서브유닛인 NuoL은 TC# 2.A.63.1.1(PhaA 및 PhaD)의 Na/H+ 안티포터와 관련이+ 있다.
NADH 탈수소효소의 보존된 막결합 서브유닛 중 3개는 서로 관련이 있으며, Mrp 나트륨-프로톤 항균제와 관련이 있다.두 개의 원핵 복합체의 구조 분석 I은 세 개의 서브유닛 각각이 구조 정렬로 중첩되는 14개의 막 통과 나선을 포함하고 있다는 것을 밝혔다. 세 개의 양성자의 전위는 그것들을 연결하는 [25]가로 나선에 의해 조정될 수 있다.
복합 I은 49-kDa 및 PSST 서브유닛의 계면에 유비퀴논 결합 포켓을 포함합니다.유비퀴논에 대해 제안된 직접 전자공여체인 철-황 클러스터 N2에 근접하여 고도로 보존된 티로신이 퀴논 환원 부위의 중요한 요소를 구성한다.가능한 퀴논 교환 경로는 클러스터 N2에서 49-kDa 서브유닛의 [26]N말단 베타시트로 이어진다.소 NDHI의 45개 서브유닛은 모두 [27][28]배열이 확인되었습니다.각 복합체는 비공유 결합 FMN, 조효소 Q 및 몇 가지 철황 중심을 포함한다.박테리아 NDH는 8-9개의 철-황 중심을 가지고 있습니다.
최근의 연구는 친수 영역에 위치한 철-황 착체를 통한 전자 전달 경로를 결정하기 위해 전자-자기 공명(EPR) 스펙트럼과 이중 전자-전자 공명(DEER)을 사용했다.이들 클러스터 중 7개는 플라빈에서 퀴논 결합 부위까지의 사슬을 형성하고, 8번째 클러스터는 플라빈의 다른 쪽에 위치하며, 그 기능은 알려지지 않았다.EPR 및 DER 결과는 활성 부위 간 및 철-황 클러스터를 따라 전자 전달을 위한 대체 또는 "롤러-코스터" 위치 에너지 프로파일을 제시하며, 이는 복합체 [29]I에서 전자 이동 속도를 최적화하고 효율적인 에너지 변환을 가능하게 한다.
| # | 인간/소 서브유닛 | 인간단백질 | 단백질 설명(UniProt) | 인간단백질을 가진 Pfam 패밀리 | |
|---|---|---|---|---|---|
| 코어 서브a 유닛 | |||||
| 1 | NDUFS7/PST/NUKM | NDUS7_인간 | NADH탈수소효소 [ubiquinone] 철황단백질 7, 미토콘드리아 EC 1.6.5.3 EC 1.6.99.3 | 팜 PF01058 | |
| 2 | NDUFS8 / TYKY / NUIM | NDUS8_인간 | NADH탈수소효소 [ubiquinone] 철황단백질 8, 미토콘드리아 EC 1.6.5.3 EC 1.6.99.3 | 팜 PF12838 | |
| 3 | NDUFV2 / 24kD / NUHMc | NDUV2_인간 | NADH탈수소효소[유비퀴논] 플라보단백질2, 미토콘드리아 EC 1.6.5.3 EC 1.6.99.3 | 팜 PF01257 | |
| 4 | NDUFS3/30kD/NUGM | NDUS3_인간 | NADH탈수소효소[유비퀴논]철황단백질3, 미토콘드리아EC 1.6.5.3EC 1.6.99.3 | 팜 PF00329 | |
| 5 | NDUFS2 / 49kD / NUCM | NDUS2_인간 | NADH탈수소효소[유비퀴논]철황단백질2, 미토콘드리아EC 1.6.5.3EC 1.6.99.3 | 팜 PF00346 | |
| 6 | NDUFV1 / 51kD / NUBM | NDUV1_인간 | NADH탈수소효소[유비퀴논] 플라보단백질1, 미토콘드리아 EC 1.6.5.3 EC 1.6.99.3 | 팜 PF01512 | |
| 7 | NDUFS1 / 75kD / NUAM | NDUS1_인간 | NADH-유비퀴논산화환원효소 75kDa 서브유닛, 미토콘드리아 EC 1.6.5.3 EC 1.6.99.3 | 팜 PF00384 | |
| 8 | ND1 / NU1M | NU1M_HUMAN | NADH-유비퀴논산화환원효소사슬1EC 1.6.5.3 | 팜 PF00146 | |
| 9 | ND2 / NU2M | NU2M_HUMAN | NADH-유비퀴논산화환원효소사슬2EC 1.6.5.3 | Pfam PF00361, Pf06444 | |
| 10 | ND3 / NU3M | NU3M_HUMAN | NADH-유비퀴논산화환원효소사슬3EC 1.6.5.3 | 팜 PF00507 | |
| 11 | ND4 / NU4M | NU4M_HUMAN | NADH-유비퀴논산화환원효소사슬4EC 1.6.5.3 | Pfam PF01059, Pfam PF00361 | |
| 12 | ND4L/NULM | NU4LM_HUMAN | NADH-유비퀴논산화환원효소사슬 4L EC 1.6.5.3 | 팜 PF00420 | |
| 13 | ND5 / NU5M | NU5M_HUMAN | NADH-유비퀴논산화환원효소사슬5EC 1.6.5.3 | Pfam PF00361, Pfam PF06455, Pfam PF00662 | |
| 14 | ND6 / NU6M | NU6M_HUMAN | NADH-유비퀴논산화환원효소사슬6EC 1.6.5.3 | 팜 PF00499 | |
| 코어 악세사리b 서브 유닛 | |||||
| 15 | NDUFS6 / 13A | NDUS6_인간 | NADH탈수소효소[유비퀴논]철황단백질6, 미토콘드리아 | 팜 PF10276 | |
| 16 | NDUFA12 / B17.2 | NDUAC_인간 | NADH탈수소효소[유비퀴논]1α 서브콤플렉스 서브유닛12 | 팜 PF05071 | |
| 17 | NDUFS4 / AQDQ | NDUS4_인간 | NADH탈수소효소[유비퀴논]철황단백질4, 미토콘드리아 | 팜 PF04800 | |
| 18 | NDUFA9 / 39kDa | NDUA9_인간 | NADH탈수소효소[유비퀴논]1α 서브콤플렉스 서브유닛 9, 미토콘드리아 | 팜 PF01370 | |
| 19 | NDUFAB1 / ACPM | ACPM_HUMAN | 아실담체단백질, 미토콘드리아 | 팜 PF00550 | |
| 20 | NDUFA2 / B8 | NDUA2_인간 | NADH탈수소효소[유비퀴논]1α 서브콤플렉스 서브유닛2 | 팜 PF05047 | |
| 21 | NDUFA1 / MFWE | NDUA1_인간 | NADH탈수소효소[유비퀴논]1α 서브콤플렉스 서브유닛1 | 팜 PF15879 | |
| 22 | NDUFB3 / B12 | NDUB3_인간 | NADH탈수소효소[유비퀴논]1β 서브콤플렉스 서브유닛3 | 팜 PF08122 | |
| 23 | NDUFA5 / AB13 | NDUA5_인간 | NADH탈수소효소[유비퀴논]1α 서브콤플렉스 서브유닛5 | 팜 PF04716 | |
| 24 | NDUFA6 / B14 | NDUA6_인간 | NADH탈수소효소[유비퀴논]1α 서브콤플렉스 서브유닛6 | 팜 PF05347 | |
| 25 | NDUFA11 / B14.7 | NDUAB_인간 | NADH탈수소효소[유비퀴논]1α 서브콤플렉스 서브유닛11 | 팜 PF02466 | |
| 26 | NDUFB11 / ESS | NDUB_인간 | NADH탈수소효소[유비퀴논] 1β 서브콤플렉스 서브유닛 11, 미토콘드리아 | 팜 PF10183 | |
| 27 | NDUFS5 / PFFD | NDUS5_인간 | NADH탈수소효소[유비퀴논]철황단백질5 | 팜 PF10200 | |
| 28 | NDUFB4 / B15 | NDUB4_인간 | NADH탈수소효소[유비퀴논]1β 서브콤플렉스 서브유닛4 | 팜 PF07225 | |
| 29 | NDUFA13 / A13 | NDUAD_인간 | NADH탈수소효소[유비퀴논]1α 서브콤플렉스 서브유닛 13 | 팜 PF06212 | |
| 30 | NDUFB7 / B18 | NDUB7_인간 | NADH탈수소효소[유비퀴논]1β 서브콤플렉스 서브유닛7 | 팜 PF05676 | |
| 31 | NDUFA8 / PGIV | NDUA8_인간 | NADH탈수소효소[유비퀴논]1α 서브콤플렉스 서브유닛8 | 팜 PF06747 | |
| 32 | NDUFB9 / B22 | NDUB9_인간 | NADH탈수소효소[유비퀴논]1β 서브콤플렉스 서브유닛9 | 팜 PF05347 | |
| 33 | NDUFB10 / PDSW | NDUBA_인간 | NADH탈수소효소[유비퀴논]1β 서브콤플렉스 서브유닛10 | 팜 PF10249 | |
| 34 | NDUFB8/ASHI | NDUB8_인간 | NADH탈수소효소[유비퀴논]1β 서브콤플렉스 서브유닛 8, 미토콘드리아 | 팜 PF05821 | |
| 35 | NDUFC2 / B14.5b | NDUC2_인간 | NADH탈수소효소[유비퀴논]1 서브유닛C2 | 팜 PF06374 | |
| 36 | NDUFB2 / AGGG | NDUB2_인간 | NADH탈수소효소[유비퀴논]1β 서브콤플렉스 서브유닛2, 미토콘드리아 | 팜 PF14813 | |
| 37 | NDUFA7 / B14.5a | NDUA7_인간 | NADH탈수소효소[유비퀴논]1α 서브콤플렉스 서브유닛7 | 팜 PF07347 | |
| 38 | NDUFA3 / B9 | NDUA3_인간 | NADH탈수소효소[유비퀴논]1α 서브콤플렉스 서브유닛3 | 팜 PF14987 | |
| 39 | NDUFA4 / MLRQc,d | NDUA4_인간 | NADH탈수소효소[유비퀴논]1α 서브콤플렉스 서브유닛4 | 팜 PF06522 | |
| 40 | NDUFB5 / SGDH | NDUB5_인간 | NADH탈수소효소[유비퀴논]1β 서브콤플렉스 서브유닛5, 미토콘드리아 | 팜 PF09781 | |
| 41 | NDUFB1 / MNLL | NDUB1_인간 | NADH탈수소효소[유비퀴논]1β 서브콤플렉스 서브유닛1 | 팜 PF08040 | |
| 42 | NDUFC1 / KFYI | NDUC1_인간 | NADH탈수소효소[유비퀴논]1 서브유닛 C1, 미토콘드리아 | 팜 PF15088 | |
| 43 | NDUFA10 / 42kD | NDUAA_인간 | NADH탈수소효소[유비퀴논]1α 서브콤플렉스 서브유닛 10, 미토콘드리아 | 팜 PF01712 | |
| 44 | NDUFA4L2 | NUA4L_인간 | NADH탈수소효소[유비퀴논]1α 서브콤플렉스 서브유닛4 라이크 2 | 팜 PF15880 | |
| 45 | NDUFV3 | NDUV3_인간 | NADH탈수소효소[유비퀴논]플라보단백질3, 10kDa | - | |
| 46 | NDUFB6 | NDUB6_인간 | NADH탈수소효소[유비퀴논]1β 서브콤플렉스 서브유닛6 | 팜 PF09782 | |
| 조립인자단백질[31] | |||||
| 47 | NDUFAF1c | CIA30_인간 | NADH탈수소효소[유비퀴논]1α 서브콤플렉스, 조립인자 1 | 팜 PF08547 | |
| 48 | NDUFAF2 | MIMIT_HUMIT | NADH탈수소효소[유비퀴논]1α 서브콤플렉스, 조립인자2 | 팜 PF05071 | |
| 49 | NDUFAF3 | NDUF3_인간 | NADH탈수소효소[유비퀴논]1α아복소조립인자3 | 팜 PF05071 | |
| 50 | NDUFAF4 | NDUF4_인간 | NADH탈수소효소[유비퀴논]1α 서브콤플렉스, 조립인자 4 | 팜 PF06784 | |
주의:
- a 곰팡이를 제외한 모든 종에서 발견됨
- b 어떤 종에도 존재하거나 존재하지 않을 수 있다
- c 시오당류 폼베와 같은 진균종에서 발견됨
- d 최근 연구에 따르면 NDUFA4는 복합[34] I의 서브유닛이 아니라 복합 IV의 서브유닛이라고 합니다.
억제제
불라타신(아시미나 트릴로바 열매에서 발견되는 아세트게닌)은 NADH 탈수소효소(아시미나 트릴로바 열매)의 가장 강력한 억제제이다(IC50=1.[35]2nM, 로테논보다 강하다).복합체 I의 가장 잘 알려진 억제제는 로테논(일반적으로 유기 농약으로 사용됨)입니다.로테논과 로테노이드는 안토니아(로가니아과), 데리스, 론코카르푸스(파보이데아과, 파바과)와 같은 열대 식물에서 발견되는 이소플라보노이드이다.프랑스령 기아나의 원주민들이 어류독성의 영향으로 로테논이 함유된 식물을 낚시에 사용했다는 보고가 17세기 [36]초에 있었다.로테논은 복합체 I의 유비퀴논 결합부위 및 유비퀴논과 밀접한 구조적 상동성을 가진 또 다른 강력한 억제제인 피에리시딘 A와 결합한다.
Annonaceae의 아세트게닌은 복합체 I의 더욱 강력한 억제제이다.ND2 서브유닛에 가교하여 ND2가 퀴논 [37]결합에 필수적이라는 것을 나타냅니다.아세트게닌인 롤리니아스타틴-2는 로테논과 [38]동일한 결합 부위를 공유하지 않는 최초의 복합 I 억제제이다.
복합체 I에 대한 50년 이상의 연구에도 불구하고, 효소 내부의 전자 흐름을 차단하는 억제제는 발견되지 않았다.로테논 또는 피에리시딘과 같은 소수성 억제제는 말단 FeS 클러스터 N2와 유비퀴논 사이의 전자 전달을 방해할 가능성이 높다.로테논에 의한 복합 I의 장기 전신 억제는 도파민 작동성 [39]뉴런의 선택적 변성을 유도할 수 있는 것으로 나타났다.
복합체 I은 또한 뉴클레오티드 결합 [40]부위에서 효소에 결합함으로써 NADH 산화의 가역적 경쟁 억제제인 아데노신 이인산 리보스에 의해 차단된다.친수성 NADH 및 소수성 유비퀴논 유사체는 각각 내부 전자-수송 경로의 시작과 끝에 작용한다.
항당뇨병 약물인 메트포르민은 미토콘드리아 호흡쇄 복합체 I의 경미하고 일시적인 억제를 유도하는 것으로 나타났으며, 이 억제는 작용 [41]메커니즘에 중요한 역할을 하는 것으로 보인다.
복합체 I의 억제는 플루타미드 및 네파조돈과 [42]같은 다양한 약물과 관련된 간독성과 관련이 있다.
액티브/디액티브 이행
진핵생물 복합체 I의 촉매 특성은 간단하지 않다.효소의 어떤 주어진 조제에도 촉매 및 구조적으로 구별되는 두 가지 형태가 존재합니다. 하나는 완전히 유능하고, 소위 "활성적인" A 형태이고, 다른 하나는 촉매적으로 조용하고, 휴면적이며, "비활성적인" D 형태입니다.기질이 없는 상태에서 유휴 효소가 상승하지만 생리적인 온도(>30 °C)에 노출되면 효소는 D 형태로 변환됩니다.이 형태는 촉매적으로 무능하지만 NADH 산화의 느린 반응(k~4분−1)과 그에 따른 유비퀴논 환원에 의해 활성화될 수 있다.하나 또는 여러 번 턴오버한 후 효소는 활성 상태가 되며 훨씬 높은 속도(k~10분4−1)로 생리학적 NADH:ubiquinone 반응을 촉매할 수 있다.2가 양이온(Mg, Ca2+)이2+ 존재하거나 알칼리 pH에서 활성화되는 데 시간이 훨씬 더 오래 걸립니다.
불활성화 과정의 높은 활성화 에너지(270 kJ/mol)는 복합체 I의 조직에 주요한 구조 변화가 발생했음을 나타낸다.그러나, 지금까지 이 두 형태 사이에서 관찰된 유일한 구조 차이는 효소의 표면에 노출되는 시스테인 잔기의 수이다.술프하이드릴 시약 N-에틸말레이미드 또는 DTNB에 의한 복합체 I의 D형태를 불가역적으로 처리함으로써 임계 시스테인 잔기를 차단하고 활성화에 대한 효소의 반응 능력을 폐지하여 불가역적으로 불활성화시킨다.복합체 I의 A형은 술프하이드릴 [43][44]시약에 민감하지 않습니다.
이러한 구조 변화는 생리적으로 매우 중요한 의미를 가질 수 있는 것으로 밝혀졌다.활성은 아니지만 비활성 형태인 복합체 I은 니트로소티올과 과산화니트라이트에 [45]의해 억제되기 쉬웠다.활성에서 비활성 형태의 복합체 I로의 이행은 저산소증, 허혈과 같은 생리적 온도에서 효소의 교체가 제한되거나 조직의 일산화질소:산소 비율이 증가(즉, 대사 저산소증)[48]하는 병리 조건 중에 발생할 수 있다.
슈퍼옥시드의 제조
최근 조사에 따르면 I 복합체는 활성산소종의 [49]잠재적 공급원이라는 것을 알 수 있다.복합 I은 적어도 두 가지 다른 경로를 통해 과산화수소를 생성할 수 있습니다.순방향 전자 전달 중에 매우 적은 양의 슈퍼옥사이드만 생성됩니다(전체 전자 [49][50][51]흐름의 0.1% 미만일 수 있음).
역전자 이동 중에 복합체 I은 미토콘드리아 내에서 슈퍼옥시드 생성의 가장 중요한 부위가 될 수 있으며, 전자의 3~4%가 슈퍼옥시드 [52]형성으로 전환된다.환원된 유비퀴놀 풀(숙신산탈수소효소, 글리세린-3-인산탈수소효소, 포유류 미토콘드리아의 전자전달 플라보단백질 또는 디히드로오로틴산탈수소효소에 의해 공급됨)의 전자가 복합체 I을 통과하여 NAD를 NADH로 환원하는+ 과정인 역전자전달잠재 전위어떤 병리학적 조건에서 역전자 전달이 생체 내에서 일어날지는 정확히 알려져 있지 않지만, 시험관내 실험에 따르면 석신산염 농도가 높고 옥살로아세테이트 또는 사과산염 농도가 [53]낮을 때 이 과정이 매우 강력한 슈퍼옥시드의 원천이 될 수 있다.이것은 조직 허혈증 중에 산소 공급이 [54]차단될 때 발생할 수 있습니다.
슈퍼옥시드는 세포 산화 스트레스에 기여하고 신경근육 질환과 [55]노화와 관련이 있는 활성 산소종이다.NADH 탈수소효소는 FMNH(또는 반감소 플라빈)에서2 산소(O2)로 하나의 전자를 전달함으로써 과산화물을 생성한다.래디칼 플라빈 잔류물은 불안정하며, 나머지 전자를 철-황 중심부로 이동시킨다.슈퍼옥시드 [56][57]생성 속도를 결정하는 것은 NADH+ 대 NAD의 비율이다.
병리학
복합체 I의 소단위 돌연변이는 리 증후군을 포함한 미토콘드리아 질병을 일으킬 수 있다.미토콘드리아 DNA(mtDNA)에서 파생된 다양한 복합 I 서브유닛의 점 돌연변이도 Leber 유전성 시신경증을 초래할 수 있다.복합 I 결함이 활성 산소 종 때문에 파킨슨병의 병인에 영향을 미칠 수 있다는 몇 가지 증거가 있습니다(복합 I는 복합 III와 같이 산소로 전자를 누출하여 매우 독성이 강한 슈퍼옥시드를 형성할 수 있습니다).
파킨슨병의 정확한 병인은 불분명하지만, 프로테아좀 억제와 환경 독소와 함께 미토콘드리아 기능 장애가 큰 역할을 할 수 있다.실제로 복합체 I의 억제는 과산화물의 생성과 프로테아솜 활성의 감소를 유발하며, 이는 파킨슨병을 [58]유발할 수 있다.또한 Esteves et al. (2010)는 파킨슨병을 가진 세포주가 복합체 I에서 양성자 누출이 증가하여 최대 호흡 능력 감소를 [59]유발한다는 것을 발견했다.
뇌허혈/출혈 손상은 복합 I [60]장애를 통해 치료된다.최근 산소 결핍은 미토콘드리아 복합체 I이 자연적인 보조 인자인 플라빈 모노뉴클레오티드(FMN)를 잃고 [61][62]비활성 상태가 되는 상태로 이어진다는 것이 발견되었다.산소가 존재할 때 효소는 유비퀴논에 의한 NADH 산화의 생리적인 반응을 촉매하여 호흡 사슬의 하류에 전자를 공급합니다(복합체 III 및 IV).허혈은 숙신산염 수치를 극적으로 증가시킨다.숙신산 미토콘드리아의 존재 하에서 역전자 전달을 촉매하여 숙신산염에서 나오는 전자의 일부가 복합체 I의 FMN으로 상류로 향하도록 한다.역전자 전달은 복합 I FMN의 [52]감소, ROS의 증가, 감소 보조인자(FMNH2)의 손실 및 미토콘드리아 에너지 생산의 장애를 초래한다.복합 I 및 I/R 손상에 의한 FMN 손실은 FMN 전구체인 리보플라빈을 [62]투여함으로써 완화될 수 있다.
최근의 연구들은 뇌에서 복합 I 활동의 다른 역할을 조사했다.Andreaza et al. (2010)는 복합 I 활동 수준이 양극성 장애 환자에서는 유의하게 감소했지만 우울증이나 정신분열증 환자에서는 그렇지 않다는 것을 발견했다.그들은 양극성 장애를 가진 환자들이 전전두피질에서 단백질 산화 및 질화 증가를 보인다는 것을 발견했다.이러한 결과는 향후 연구들이 조울증에 [63]대한 잠재적 치료 연구를 위해 복합체 I을 목표로 삼아야 한다는 것을 암시한다.마찬가지로, Moran et al. (2010)는 심각한 복합체 I 결핍증 환자가 산소 소비율이 감소하고 성장 속도가 느려지는 것을 발견했다.그러나, 그들은 복합 I의 다른 유전자의 돌연변이가 다른 표현형으로 이어져 복합 I [64]결핍의 병태 생리학적 징후의 변화를 설명한다는 것을 발견했다.
살충제에 노출되는 것은 또한 복합체 I을 억제하고 질병 증상을 일으킬 수 있다.예를 들어, 농약으로 사용되는 유기인산 디클로로보스의 낮은 수치에 대한 만성적인 노출은 간 기능 장애를 야기하는 것으로 나타났다.이것은 디클로로보스가 복합 I과 II의 활성 수준을 변화시켜 미토콘드리아 전자 전달 활동을 감소시키고 ATP [65]합성을 감소시키기 때문에 발생한다.
엽록체 내
복합체 I에 상동하는 양성자 펌핑, 유비퀴논 사용 NADH 탈수소효소 복합체는 ndh라는 이름으로 대부분의 육상 식물의 엽록체 게놈에서 발견된다.이 복합체는 시아노박테리아로부터 원래의 공생으로부터 물려받았지만, 대부분의 진핵 조류, 몇몇 나체 식물, 그리고 몇몇 아주 어린 혈관 배유류에서 없어졌습니다.이 복합체의 목적은 원래 엽록체가 호흡에 관여하지 않기 때문에 알 수 없지만, 지금은 ndh가 스트레스를 받는 상황에서 광합성을 유지하는 역할을 하는 것으로 알려져 있다.따라서 적어도 부분적으로 양호한 조건에서 사용할 수 있습니다.ndh가 없는 안지오스퍼의 혈통이 어린 나이부터 오래 지속되지 않는다는 것은 분명하지만, 어떻게 ndh가 없는 땅에서 그렇게 오랫동안 생존하는지는 [66]알려져 있지 않다.
유전자
다음은 복합 I의 성분을 인코딩하는 인간 유전자 목록입니다.
- NADH탈수소효소(유비퀴논)1α 서브콤플렉스
- NDUFA1 – NADH 탈수소효소(유비퀴논) 1α 서브콤플렉스, 1, 7.5kDa
- NDUFA2 – NADH 탈수소효소(유비퀴논) 1α 서브콤플렉스, 2, 8kDa
- NDUFA3 – NADH 탈수소효소(유비퀴논) 1α 서브콤플렉스, 3, 9kDa
- NDUFA4 – NADH 탈수소효소(유비퀴논) 1α 서브콤플렉스, 4, 9kDa - 최근 복합체[67] IV의 일부로 설명됨
- NDUFA4L – NADH탈수소효소(유비퀴논) 1α 서브콤플렉스, 4-like
- NDUFA4L2 – NADH 탈수소효소(유비퀴논) 1α 서브콤플렉스, 4-라이크 2
- NDUFA5 – NADH 탈수소효소(유비퀴논) 1α 서브콤플렉스, 5, 13kDa
- NDUFA6 – NADH 탈수소효소(유비퀴논) 1α 서브콤플렉스, 6, 14kDa
- NDUFA7 – NADH 탈수소효소(유비퀴논) 1α 서브콤플렉스, 7, 14.5kDa
- NDUFA8 – NADH 탈수소효소(유비퀴논) 1α 서브콤플렉스, 8, 19kDa
- NDUFA9 – NADH 탈수소효소(유비퀴논) 1α 서브콤플렉스, 9, 39kDa
- NDUFA10 – NADH 탈수소효소(유비퀴논) 1α 서브콤플렉스, 10, 42kDa
- NDUFA11 – NADH 탈수소효소(유비퀴논) 1α 서브콤플렉스, 11, 14.7kDa
- NDUFA12 – NADH 탈수소효소(유비퀴논) 1α 서브콤플렉스, 12
- NDUFA13 – NADH 탈수소효소(유비퀴논) 1α 서브콤플렉스, 13
- NDUFAB1 – NADH 탈수소효소(유비퀴논) 1, 알파/베타 서브콤플렉스, 1, 8kDa
- NDUFAF1 – NADH 탈수소효소(유비퀴논) 1α 서브콤플렉스, 조립인자 1
- NDUFAF2 – NADH 탈수소효소(유비퀴논) 1α 서브콤플렉스, 조립인자 2
- NDUFAF3 – NADH 탈수소효소(유비퀴논) 1α 서브콤플렉스, 조립인자 3
- NDUFAF4 – NADH 탈수소효소(유비퀴논) 1α 서브콤플렉스, 조립인자 4
- NADH탈수소효소(유비퀴논)1 베타 서브콤플렉스
- NDUFB1 – NADH 탈수소효소(유비퀴논) 1 베타 서브콤플렉스, 1, 7kDa
- NDUFB2 – NADH 탈수소효소(유비퀴논) 1 베타 서브콤플렉스, 2, 8kDa
- NDUFB3 – NADH 탈수소효소(유비퀴논) 1 베타 서브콤플렉스, 3, 12kDa
- NDUFB4 – NADH 탈수소효소(유비퀴논) 1 베타 서브콤플렉스, 4, 15kDa
- NDUFB5 – NADH 탈수소효소(유비퀴논) 1 베타 서브콤플렉스, 5, 16kDa
- NDUFB6 – NADH 탈수소효소(유비퀴논) 1 베타 서브콤플렉스, 6, 17kDa
- NDUFB7 – NADH 탈수소효소(유비퀴논) 1 베타 서브콤플렉스, 7, 18kDa
- NDUFB8 – NADH 탈수소효소(유비퀴논) 1 베타 서브콤플렉스, 8, 19kDa
- NDUFB9 – NADH 탈수소효소(유비퀴논) 1 베타 서브콤플렉스, 9, 22kDa
- NDUFB10 – NADH 탈수소효소(유비퀴논) 1 베타 서브콤플렉스, 10, 22kDa
- NDUFB11 – NADH 탈수소효소(유비퀴논) 1 베타 서브콤플렉스, 11, 17.3kDa
- NADH탈수소효소(유비퀴논)1, 아복합체 불명
- NADH탈수소효소(유비퀴논)Fe-S단백질
- NDUFS1 – NADH탈수소효소(유비퀴논) Fe-S단백질1,75kDa(NADH-코엔자임Q환원효소)
- NDUFS2 – NADH탈수소효소(유비퀴논) Fe-S단백질2,49kDa(NADH-코엔자임Q환원효소)
- NDUFS3 – NADH 탈수소효소(유비퀴논) Fe-S 단백질 3, 30kDa(NADH-coenzyme Q 환원효소)
- NDUFS4 – NADH 탈수소효소(유비퀴논) Fe-S 단백질 4, 18kDa(NADH-coenzyme Q 환원효소)
- NDUFS5 – NADH 탈수소효소(유비퀴논) Fe-S 단백질 5, 15kDa(NADH-coenzyme Q 환원효소)
- NDUFS6 – NADH탈수소효소(유비퀴논) Fe-S단백질 6, 13kDa(NADH-코엔자임Q환원효소)
- NDUFS7 – NADH 탈수소효소(유비퀴논) Fe-S 단백질 7, 20kDa(NADH-coenzyme Q 환원효소)
- NDUFS8 – NADH탈수소효소(유비퀴논) Fe-S단백질 8,23kDa(NADH-coenzym Q환원효소)
- NADH탈수소효소(유비퀴논)플라보단백질1
- 미토콘드리아 부호화 NADH탈수소효소 서브유닛
레퍼런스
- ^ a b Berg J, Tymoczko J, Stryer L (2006). Biochemistry (6th ed.). New York: WH Freeman & Company. pp. 509–513.
- ^ Brandt U (2006). "Energy converting NADH:quinone oxidoreductase (complex I)". Annual Review of Biochemistry. 75: 69–92. doi:10.1146/annurev.biochem.75.103004.142539. PMID 16756485.
- ^ Wikström M (April 1984). "Two protons are pumped from the mitochondrial matrix per electron transferred between NADH and ubiquinone". FEBS Letters. 169 (2): 300–4. doi:10.1016/0014-5793(84)80338-5. PMID 6325245.
- ^ Galkin A, Dröse S, Brandt U (December 2006). "The proton pumping stoichiometry of purified mitochondrial complex I reconstituted into proteoliposomes". Biochimica et Biophysica Acta (BBA) - Bioenergetics. 1757 (12): 1575–81. doi:10.1016/j.bbabio.2006.10.001. PMID 17094937.
- ^ Galkin AS, Grivennikova VG, Vinogradov AD (May 1999). "-->H+/2e- stoichiometry in NADH-quinone reductase reactions catalyzed by bovine heart submitochondrial particles". FEBS Letters. 451 (2): 157–61. doi:10.1016/s0014-5793(99)00575-x. PMID 10371157. S2CID 2337382.
- ^ a b Batista AP, Pereira MM (March 2011). "Sodium influence on energy transduction by complexes I from Escherichia coli and Paracoccus denitrificans". Biochimica et Biophysica Acta (BBA) - Bioenergetics. 1807 (3): 286–92. doi:10.1016/j.bbabio.2010.12.008. PMID 21172303.
- ^ Grivennikova VG, Kotlyar AB, Karliner JS, Cecchini G, Vinogradov AD (September 2007). "Redox-dependent change of nucleotide affinity to the active site of the mammalian complex I". Biochemistry. 46 (38): 10971–8. doi:10.1021/bi7009822. PMC 2258335. PMID 17760425.
- ^ Chomova M, Racay P (March 2010). "Mitochondrial complex I in the network of known and unknown facts". General Physiology and Biophysics. 29 (1): 3–11. doi:10.4149/gpb_2010_01_3. PMID 20371875.
- ^ Petrussa E, Bertolini A, Casolo V, Krajnáková J, Macrì F, Vianello A (December 2009). "Mitochondrial bioenergetics linked to the manifestation of programmed cell death during somatic embryogenesis of Abies alba". Planta. 231 (1): 93–107. doi:10.1007/s00425-009-1028-x. PMID 19834734. S2CID 25828432.
- ^ a b c d e Sazanov LA (June 2015). "A giant molecular proton pump: structure and mechanism of respiratory complex I". Nature Reviews. Molecular Cell Biology. 16 (6): 375–88. doi:10.1038/nrm3997. PMID 25991374. S2CID 31633494.
- ^ Voet DJ, Voet GJ, Pratt CW (2008). "Chapter 18, Mitochondrial ATP synthesis". Principles of Biochemistry, 3rd Edition. Wiley. p. 608. ISBN 978-0-470-23396-2.
- ^ Ohnishi T (May 1998). "Iron-sulfur clusters/semiquinones in complex I". Biochimica et Biophysica Acta (BBA) - Bioenergetics. 1364 (2): 186–206. doi:10.1016/s0005-2728(98)00027-9. PMID 9593887.
- ^ Bridges HR, Bill E, Hirst J (January 2012). "Mössbauer spectroscopy on respiratory complex I: the iron-sulfur cluster ensemble in the NADH-reduced enzyme is partially oxidized". Biochemistry. 51 (1): 149–58. doi:10.1021/bi201644x. PMC 3254188. PMID 22122402.
- ^ a b Efremov RG, Sazanov LA (October 2012). "The coupling mechanism of respiratory complex I - a structural and evolutionary perspective". Biochimica et Biophysica Acta (BBA) - Bioenergetics. 1817 (10): 1785–95. doi:10.1016/j.bbabio.2012.02.015. PMID 22386882.
- ^ Treberg JR, Quinlan CL, Brand MD (August 2011). "Evidence for two sites of superoxide production by mitochondrial NADH-ubiquinone oxidoreductase (complex I)". The Journal of Biological Chemistry. 286 (31): 27103–10. doi:10.1074/jbc.M111.252502. PMC 3149303. PMID 21659507.
- ^ Berrisford JM, Sazanov LA (October 2009). "Structural basis for the mechanism of respiratory complex I". The Journal of Biological Chemistry. 284 (43): 29773–83. doi:10.1074/jbc.m109.032144. PMC 2785608. PMID 19635800.
- ^ Baranova EA, Morgan DJ, Sazanov LA (August 2007). "Single particle analysis confirms distal location of subunits NuoL and NuoM in Escherichia coli complex I". Journal of Structural Biology. 159 (2): 238–42. doi:10.1016/j.jsb.2007.01.009. PMID 17360196.
- ^ Brandt U (October 2011). "A two-state stabilization-change mechanism for proton-pumping complex I". Biochimica et Biophysica Acta (BBA) - Bioenergetics. 1807 (10): 1364–9. doi:10.1016/j.bbabio.2011.04.006. PMID 21565159.
- ^ Zickermann V, Wirth C, Nasiri H, Siegmund K, Schwalbe H, Hunte C, Brandt U (January 2015). "Structural biology. Mechanistic insight from the crystal structure of mitochondrial complex I" (PDF). Science. 347 (6217): 44–9. doi:10.1126/science.1259859. PMID 25554780. S2CID 23582849.
- ^ Hunte C, Screpanti E, Venturi M, Rimon A, Padan E, Michel H (June 2005). "Structure of a Na+/H+ antiporter and insights into mechanism of action and regulation by pH". Nature. 435 (7046): 1197–202. Bibcode:2005Natur.435.1197H. doi:10.1038/nature03692. PMID 15988517. S2CID 4372674.
- ^ Voet JG, Voet D (2004). Biochemistry (3rd ed.). New York: J. Wiley & Sons. pp. 813–826. ISBN 0-471-19350-X.
- ^ Carroll J, Fearnley IM, Skehel JM, Shannon RJ, Hirst J, Walker JE (October 2006). "Bovine complex I is a complex of 45 different subunits". The Journal of Biological Chemistry. 281 (43): 32724–7. doi:10.1074/jbc.M607135200. PMID 16950771.
- ^ Balsa E, Marco R, Perales-Clemente E, Szklarczyk R, Calvo E, Landázuri MO, Enríquez JA (September 2012). "NDUFA4 is a subunit of complex IV of the mammalian electron transport chain". Cell Metabolism. 16 (3): 378–86. doi:10.1016/j.cmet.2012.07.015. PMID 22902835.
- ^ Sazanov LA, Hinchliffe P (March 2006). "Structure of the hydrophilic domain of respiratory complex I from Thermus thermophilus". Science. 311 (5766): 1430–6. Bibcode:2006Sci...311.1430S. doi:10.1126/science.1123809. PMID 16469879. S2CID 1892332.
- ^ Efremov RG, Baradaran R, Sazanov LA (May 2010). "The architecture of respiratory complex I". Nature. 465 (7297): 441–5. Bibcode:2010Natur.465..441E. doi:10.1038/nature09066. PMID 20505720. S2CID 4372778.
- ^ Tocilescu MA, Zickermann V, Zwicker K, Brandt U (December 2010). "Quinone binding and reduction by respiratory complex I". Biochimica et Biophysica Acta (BBA) - Bioenergetics. 1797 (12): 1883–90. doi:10.1016/j.bbabio.2010.05.009. PMID 20493164.
- ^ Cardol P, Vanrobaeys F, Devreese B, Van Beeumen J, Matagne RF, Remacle C (October 2004). "Higher plant-like subunit composition of mitochondrial complex I from Chlamydomonas reinhardtii: 31 conserved components among eukaryotes". Biochimica et Biophysica Acta (BBA) - Bioenergetics. 1658 (3): 212–24. doi:10.1016/j.bbabio.2004.06.001. PMID 15450959.
- ^ Gabaldón T, Rainey D, Huynen MA (May 2005). "Tracing the evolution of a large protein complex in the eukaryotes, NADH:ubiquinone oxidoreductase (Complex I)". Journal of Molecular Biology. 348 (4): 857–70. doi:10.1016/j.jmb.2005.02.067. PMID 15843018.
- ^ Roessler MM, King MS, Robinson AJ, Armstrong FA, Harmer J, Hirst J (February 2010). "Direct assignment of EPR spectra to structurally defined iron-sulfur clusters in complex I by double electron-electron resonance". Proceedings of the National Academy of Sciences of the United States of America. 107 (5): 1930–5. Bibcode:2010PNAS..107.1930R. doi:10.1073/pnas.0908050107. PMC 2808219. PMID 20133838.
- ^ Cardol P (November 2011). "Mitochondrial NADH:ubiquinone oxidoreductase (complex I) in eukaryotes: a highly conserved subunit composition highlighted by mining of protein databases". Biochimica et Biophysica Acta (BBA) - Bioenergetics. 1807 (11): 1390–7. doi:10.1016/j.bbabio.2011.06.015. PMID 21749854.
- ^ Ogilvie I, Kennaway NG, Shoubridge EA (October 2005). "A molecular chaperone for mitochondrial complex I assembly is mutated in a progressive encephalopathy". The Journal of Clinical Investigation. 115 (10): 2784–92. doi:10.1172/JCI26020. PMC 1236688. PMID 16200211.
- ^ Dunning CJ, McKenzie M, Sugiana C, Lazarou M, Silke J, Connelly A, Fletcher JM, Kirby DM, Thorburn DR, Ryan MT (July 2007). "Human CIA30 is involved in the early assembly of mitochondrial complex I and mutations in its gene cause disease". The EMBO Journal. 26 (13): 3227–37. doi:10.1038/sj.emboj.7601748. PMC 1914096. PMID 17557076.
- ^ Saada A, Vogel RO, Hoefs SJ, van den Brand MA, Wessels HJ, Willems PH, Venselaar H, Shaag A, Barghuti F, Reish O, Shohat M, Huynen MA, Smeitink JA, van den Heuvel LP, Nijtmans LG (June 2009). "Mutations in NDUFAF3 (C3ORF60), encoding an NDUFAF4 (C6ORF66)-interacting complex I assembly protein, cause fatal neonatal mitochondrial disease". American Journal of Human Genetics. 84 (6): 718–27. doi:10.1016/j.ajhg.2009.04.020. PMC 2694978. PMID 19463981.
- ^ Balsa, Eduardo; Marco, Ricardo; Perales-Clemente, Ester; Szklarczyk, Radek; Calvo, Enrique; Landázuri, Manuel O.; Enríquez, José Antonio (2012-09-05). "NDUFA4 is a subunit of complex IV of the mammalian electron transport chain". Cell Metabolism. 16 (3): 378–386. doi:10.1016/j.cmet.2012.07.015. ISSN 1932-7420. PMID 22902835.
- ^ Miyoshi H, Ohshima M, Shimada H, Akagi T, Iwamura H, McLaughlin JL (July 1998). "Essential structural factors of annonaceous acetogenins as potent inhibitors of mitochondrial complex I". Biochimica et Biophysica Acta (BBA) - Bioenergetics. 1365 (3): 443–52. doi:10.1016/s0005-2728(98)00097-8. PMID 9711297.
- ^ Moretti C, Grenand P (September 1982). "[The "nivrées", or ichthyotoxic plants of French Guyana]". Journal of Ethnopharmacology (in French). 6 (2): 139–60. doi:10.1016/0378-8741(82)90002-2. PMID 7132401.
- ^ Nakamaru-Ogiso E, Han H, Matsuno-Yagi A, Keinan E, Sinha SC, Yagi T, Ohnishi T (March 2010). "The ND2 subunit is labeled by a photoaffinity analogue of asimicin, a potent complex I inhibitor". FEBS Letters. 584 (5): 883–8. doi:10.1016/j.febslet.2010.01.004. PMC 2836797. PMID 20074573.
- ^ Degli Esposti M, Ghelli A, Ratta M, Cortes D, Estornell E (July 1994). "Natural substances (acetogenins) from the family Annonaceae are powerful inhibitors of mitochondrial NADH dehydrogenase (Complex I)". The Biochemical Journal. 301 ( Pt 1): 161–7. doi:10.1042/bj3010161. PMC 1137156. PMID 8037664.
- ^ Watabe M, Nakaki T (October 2008). "Mitochondrial complex I inhibitor rotenone inhibits and redistributes vesicular monoamine transporter 2 via nitration in human dopaminergic SH-SY5Y cells". Molecular Pharmacology. 74 (4): 933–40. doi:10.1124/mol.108.048546. PMID 18599602. S2CID 1844073.
- ^ Zharova TV, Vinogradov AD (July 1997). "A competitive inhibition of the mitochondrial NADH-ubiquinone oxidoreductase (complex I) by ADP-ribose". Biochimica et Biophysica Acta (BBA) - Bioenergetics. 1320 (3): 256–64. doi:10.1016/S0005-2728(97)00029-7. PMID 9230920.
- ^ Viollet B, Guigas B, Sanz Garcia N, Leclerc J, Foretz M, Andreelli F (March 2012). "Cellular and molecular mechanisms of metformin: an overview". Clinical Science. 122 (6): 253–70. doi:10.1042/CS20110386. PMC 3398862. PMID 22117616.
- ^ Nadanaciva S, Will Y (2011). "New insights in drug-induced mitochondrial toxicity". Current Pharmaceutical Design. 17 (20): 2100–12. doi:10.2174/138161211796904795. PMID 21718246.
- ^ Babot M, Birch A, Labarbuta P, Galkin A (July 2014). "Characterisation of the active/de-active transition of mitochondrial complex I". Biochimica et Biophysica Acta (BBA) - Bioenergetics. 1837 (7): 1083–92. doi:10.1016/j.bbabio.2014.02.018. PMC 4331042. PMID 24569053.
- ^ Dröse S, Stepanova A, Galkin A (July 2016). "Ischemic A/D transition of mitochondrial complex I and its role in ROS generation". Biochimica et Biophysica Acta (BBA) - Bioenergetics. 1857 (7): 946–57. doi:10.1016/j.bbabio.2015.12.013. PMC 4893024. PMID 26777588.
- ^ Galkin A, Moncada S (December 2007). "S-nitrosation of mitochondrial complex I depends on its structural conformation". The Journal of Biological Chemistry. 282 (52): 37448–53. doi:10.1074/jbc.M707543200. PMID 17956863.
- ^ Kim M, Stepanova A, Niatsetskaya Z, Sosunov S, Arndt S, Murphy MP, et al. (August 2018). "Attenuation of oxidative damage by targeting mitochondrial complex I in neonatal hypoxic-ischemic brain injury". Free Radical Biology & Medicine. 124: 517–524. doi:10.1016/j.freeradbiomed.2018.06.040. PMC 6389362. PMID 30037775.
- ^ Stepanova A, Konrad C, Guerrero-Castillo S, Manfredi G, Vannucci S, Arnold S, Galkin A (September 2019). "Deactivation of mitochondrial complex I after hypoxia-ischemia in the immature brain". Journal of Cerebral Blood Flow and Metabolism. 39 (9): 1790–1802. doi:10.1177/0271678X18770331. PMC 6727140. PMID 29629602.
- ^ Moncada S, Erusalimsky JD (March 2002). "Does nitric oxide modulate mitochondrial energy generation and apoptosis?". Nature Reviews. Molecular Cell Biology. 3 (3): 214–20. doi:10.1038/nrm762. PMID 11994742. S2CID 29513174.
- ^ a b Murphy MP (January 2009). "How mitochondria produce reactive oxygen species". The Biochemical Journal. 417 (1): 1–13. doi:10.1042/BJ20081386. PMC 2605959. PMID 19061483.
- ^ Hansford RG, Hogue BA, Mildaziene V (February 1997). "Dependence of H2O2 formation by rat heart mitochondria on substrate availability and donor age". Journal of Bioenergetics and Biomembranes. 29 (1): 89–95. doi:10.1023/A:1022420007908. PMID 9067806. S2CID 7501110.
- ^ Stepanova A, Konrad C, Manfredi G, Springett R, Ten V, Galkin A (March 2019). "The dependence of brain mitochondria reactive oxygen species production on oxygen level is linear, except when inhibited by antimycin A". Journal of Neurochemistry. 148 (6): 731–745. doi:10.1111/jnc.14654. PMC 7086484. PMID 30582748.
- ^ a b Stepanova A, Kahl A, Konrad C, Ten V, Starkov AS, Galkin A (December 2017). "Reverse electron transfer results in a loss of flavin from mitochondrial complex I: Potential mechanism for brain ischemia reperfusion injury". Journal of Cerebral Blood Flow and Metabolism. 37 (12): 3649–3658. doi:10.1177/0271678X17730242. PMC 5718331. PMID 28914132.
- ^ Muller FL, Liu Y, Abdul-Ghani MA, Lustgarten MS, Bhattacharya A, Jang YC, Van Remmen H (January 2008). "High rates of superoxide production in skeletal-muscle mitochondria respiring on both complex I- and complex II-linked substrates". The Biochemical Journal. 409 (2): 491–9. doi:10.1042/BJ20071162. PMID 17916065.
- ^ Sahni PV, Zhang J, Sosunov S, Galkin A, Niatsetskaya Z, Starkov A, et al. (February 2018). "Krebs cycle metabolites and preferential succinate oxidation following neonatal hypoxic-ischemic brain injury in mice". Pediatric Research. 83 (2): 491–497. doi:10.1038/pr.2017.277. PMC 5866163. PMID 29211056.
- ^ Esterházy D, King MS, Yakovlev G, Hirst J (March 2008). "Production of reactive oxygen species by complex I (NADH:ubiquinone oxidoreductase) from Escherichia coli and comparison to the enzyme from mitochondria". Biochemistry. 47 (12): 3964–71. doi:10.1021/bi702243b. PMID 18307315.
- ^ Kussmaul L, Hirst J (May 2006). "The mechanism of superoxide production by NADH:ubiquinone oxidoreductase (complex I) from bovine heart mitochondria". Proceedings of the National Academy of Sciences of the United States of America. 103 (20): 7607–12. Bibcode:2006PNAS..103.7607K. doi:10.1073/pnas.0510977103. PMC 1472492. PMID 16682634.
- ^ Galkin A, Brandt U (August 2005). "Superoxide radical formation by pure complex I (NADH:ubiquinone oxidoreductase) from Yarrowia lipolytica". The Journal of Biological Chemistry. 280 (34): 30129–35. doi:10.1074/jbc.M504709200. PMID 15985426.
- ^ Chou AP, Li S, Fitzmaurice AG, Bronstein JM (August 2010). "Mechanisms of rotenone-induced proteasome inhibition". Neurotoxicology. 31 (4): 367–72. doi:10.1016/j.neuro.2010.04.006. PMC 2885979. PMID 20417232.
- ^ Esteves AR, Lu J, Rodova M, Onyango I, Lezi E, Dubinsky R, Lyons KE, Pahwa R, Burns JM, Cardoso SM, Swerdlow RH (May 2010). "Mitochondrial respiration and respiration-associated proteins in cell lines created through Parkinson's subject mitochondrial transfer". Journal of Neurochemistry. 113 (3): 674–82. doi:10.1111/j.1471-4159.2010.06631.x. PMID 20132468.
- ^ Galkin A (November 2019). "Brain Ischemia/Reperfusion Injury and Mitochondrial Complex I Damage". Biochemistry. Biokhimiia. 84 (11): 1411–1423. doi:10.1134/S0006297919110154. PMID 31760927. S2CID 207990089.
- ^ Kahl A, Stepanova A, Konrad C, Anderson C, Manfredi G, Zhou P, et al. (May 2018). "Critical Role of Flavin and Glutathione in Complex I-Mediated Bioenergetic Failure in Brain Ischemia/Reperfusion Injury". Stroke. 49 (5): 1223–1231. doi:10.1161/STROKEAHA.117.019687. PMC 5916474. PMID 29643256.
- ^ a b Stepanova A, Sosunov S, Niatsetskaya Z, Konrad C, Starkov AA, Manfredi G, et al. (September 2019). "Redox-Dependent Loss of Flavin by Mitochondrial Complex I in Brain Ischemia/Reperfusion Injury". Antioxidants & Redox Signaling. 31 (9): 608–622. doi:10.1089/ars.2018.7693. PMC 6657304. PMID 31037949.
- ^ Andreazza AC, Shao L, Wang JF, Young LT (April 2010). "Mitochondrial complex I activity and oxidative damage to mitochondrial proteins in the prefrontal cortex of patients with bipolar disorder". Archives of General Psychiatry. 67 (4): 360–8. doi:10.1001/archgenpsychiatry.2010.22. PMID 20368511.
- ^ Morán M, Rivera H, Sánchez-Aragó M, Blázquez A, Merinero B, Ugalde C, Arenas J, Cuezva JM, Martín MA (May 2010). "Mitochondrial bioenergetics and dynamics interplay in complex I-deficient fibroblasts". Biochimica et Biophysica Acta (BBA) - Molecular Basis of Disease. 1802 (5): 443–53. doi:10.1016/j.bbadis.2010.02.001. PMID 20153825.
- ^ Binukumar BK, Bal A, Kandimalla R, Sunkaria A, Gill KD (April 2010). "Mitochondrial energy metabolism impairment and liver dysfunction following chronic exposure to dichlorvos". Toxicology. 270 (2–3): 77–84. doi:10.1016/j.tox.2010.01.017. PMID 20132858.
- ^ Sabater, B (19 November 2021). "On the Edge of Dispensability, the Chloroplast ndh Genes". International Journal of Molecular Sciences. 22 (22): 12505. doi:10.3390/ijms222212505. PMC 8621559. PMID 34830386.
- ^ Balsa, Eduardo; Marco, Ricardo; Perales-Clemente, Ester; Szklarczyk, Radek; Calvo, Enrique; Landázuri, Manuel O.; Enríquez, José Antonio (2012-09-05). "NDUFA4 is a subunit of complex IV of the mammalian electron transport chain". Cell Metabolism. 16 (3): 378–386. doi:10.1016/j.cmet.2012.07.015. ISSN 1932-7420. PMID 22902835.
외부 링크
- 오스트리아 과학기술연구소(ISTA) : 사자노프 그룹 MRC MBU 사자노프 그룹
- NADH 탈수소효소의 인터랙티브 분자 모델(MDL 차임벨 필요)
- 콤플렉스 I 홈페이지
- Complex I 뉴스 페이스북 페이지
- 미국 국립 의학 도서관(MeSH)의 전자+수송+복합+I
이 편집 시점에서는, 이 기사는, 다음의 컨텐츠를 사용하고 있습니다."3.D.1 H+ 또는 Na+ 변환 NADH 탈수소효소(NDH)Creative Commons Attribution-Share Alike 3.0 Unported License(GFDL)에서는 재사용할 수 있지만 GFDL에서는 재사용할 수 없습니다.모든 관련 조건을 따라야 합니다.