프로테옴
Proteome프로테옴은 특정 시기에 게놈, 세포, 조직, 유기체에 의해 발현되거나 발현될 수 있는 단백질의 전체 세트이다.그것은 정의된 조건 하에서 주어진 시간에 주어진 세포나 유기체의 형태에서 발현된 단백질의 집합이다.프로테오믹스는 프로테옴에 대한 연구이다.
프로테옴의 종류
프로테옴은 일반적으로 유기체의 프로테옴을 지칭하는 반면, 다세포 유기체는 다른 세포에서 매우 다른 프로테옴을 가질 수 있기 때문에 세포와 유기체에서 프로테옴을 구별하는 것이 중요하다.
세포단백질은 호르몬 자극에 대한 노출과 같은 특정 환경 조건 하에서 특정 세포 유형에서 발견되는 단백질의 집합이다.
유기체의 완전한 프로테옴을 고려하는 것도 유용할 수 있는데, 이것은 다양한 세포 프로테옴의 완전한 단백질 집합으로 개념화될 수 있다.이것은 대략 게놈과 같은 단백질입니다.
프로테옴이라는 용어는 또한 세포 내 조직과 같은 특정 하위 세포 시스템에 있는 단백질의 수집을 가리키는 데 사용되어 왔다.예를 들어, 미토콘드리아 프로테옴은 3000개 이상의 단백질로 [1][2]구성될 수 있다.
바이러스 속의 단백질은 바이러스 프로테옴이라고 불릴 수 있다.보통 바이러스 프로테옴은 바이러스[3] 게놈에서 예측되지만, 바이러스 [4]프로테옴에서 발현되는 모든 단백질, 즉 바이러스 프로테옴을 결정하기 위한 몇 가지 시도가 있었다.그러나 바이러스 프로테오믹스는 바이러스 감염 시 숙주 단백질의 변화를 분석하기 때문에 사실상 두 프로테옴([5]바이러스와 숙주)이 연구된다.
암의 중요성
프로테옴은 다른 암세포주를 비교 분석하기 위해 사용될 수 있다.방광암 세포주 KK47과 YTS1의 전이 가능성을 확인하기 위해 단백질학 연구가 사용되었으며 36개의 비조절 [6]단백질과 74개의 저조절 단백질을 가지고 있는 것으로 밝혀졌다.단백질 발현의 차이는 새로운 암 신호 메커니즘을 식별하는데 도움을 줄 수 있다.
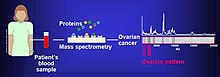
암의 바이오마커는 질량분석에 기초한 단백질 분석에서 발견되었다.프로테오믹스 또는 프로테옴의 연구는 환자의 특정 프로테오믹 및 게놈 [7]프로파일에 맞게 약물 칵테일을 맞춤화하는 개인화된 의학의 한 걸음 진전입니다.난소암 세포주 분석 결과, 난소암의 추정 바이오마커는 "α-에놀라아제(ENOA)", 신장인자 Tu, 미토콘드리아(EFTU), 글리세린알데히드-3-인산탈수소효소(G3P), 스트레스-70 단백질, 미토콘드리아(GRP75), 아폴리포단백신(APOA)를 포함한다.
11개의 세포주를 비교한 단백질 분석 결과, 각 세포주의 대사 과정 사이의 유사성이 입증되었다. 이 연구에서 11,731개의 단백질이 완전히 확인되었다.하우스키핑 단백질은 세포주 [9]간에 더 큰 변화를 보이는 경향이 있다.
특정 암 약물에 대한 내성은 아직 잘 알려져 있지 않다.단백질 분석법은 항암제 특성을 가질 수 있는 단백질, 특히 대장암 치료제 [10]이리노테칸을 식별하기 위해 사용되어 왔다.선암 세포주 LoVo에 대한 연구는 8개의 단백질이 조절되지 않고 7개의 단백질이 하향 조절된다는 것을 보여주었다.차이를 보이는 단백질은 전사, 아포토시스, 세포증식/분화 등의 과정에 관여하였다.
박테리아 시스템의 프로테옴은
단백질 분석법은 다양한 조건에 대한 그들의 대사 반응을 평가하기 위해 다양한 종류의 박테리아에서 수행되었다.예를 들어 클로스트리듐과 바실러스 같은 박테리아에서는 단백질 분석법을 사용하여 다양한 단백질이 장기간의 휴면 [11]후 각각의 박테리아 포자가 어떻게 발아하는지를 조사했다.포자를 적절하게 제거하는 방법을 더 잘 이해하기 위해서는 단백질 분석을 수행해야 합니다.
역사
마크 윌킨스는 1994년 이탈리아 시에나에서 열린 '2D 전기영동: 단백질 지도에서 게놈으로' 심포지엄에서 프로테옴이라는 용어를 만들었다.그것은 [13]1995년에 그의 박사 논문의 일부와 함께 인쇄되었다.윌킨스는 게놈, 세포, 조직 또는 유기체에 의해 발현되는 단백질의 전체 보체를 설명하기 위해 이 용어를 사용했다.
크기 및 내용물
바이러스와 원핵생물의 게놈은 각각의 단백질이 열린 판독 프레임에 근거해 높은 신뢰도로 예측될 수 있기 때문에 비교적 잘 정의된 단백질군을 인코딩한다(바이러스는 약 3개에서 1000개, 박테리아는 약 500개에서 [14]약 10,000개에 이른다.하지만, 대부분의 단백질 예측 알고리즘은 50 또는 100개의 아미노산과 같은 특정 컷오프(cut-off)를 사용하기 때문에, 작은 단백질은 종종 그러한 [15]예측에 의해 놓친다.진핵생물에서 이것은 대안적인 결합으로 인해 대부분의 유전자로부터 하나 이상의 단백질이 생성될 수 있기 때문에 훨씬 더 복잡해진다. (예를 들어 인간 프로테옴은 약 20,000개의 단백질을 암호화하지만, 일부 추정치는 92,179개의[citation needed] 단백질 중 71,173개가 결합[citation needed] [16]변종이라고 예측했다.)
프로테오폼.단백질에 가변성을 더할 수 있는 다른 요소들이 있다.SAPs(단일 아미노산 다형성)와 비동명 단일 뉴클레오티드 다형성(nsSNPs)은 서로 다른 "단백질 형태"[17] 또는 "단백질 형태"를 초래할 수 있다.최근 추정치에 따르면 현재 SwissProt에 포함된 익명의 검증된 CSNP는 약 135,000개입니다.dbSNP에는 470만 개의 후보 cSNP가 있지만, [17]단백질에서 아미노산의 정체를 변화시키는 익명의 cSNP로 설정된 1,000-genomes에서 약 67만 개의 cSNP만이 검증되었다.
다크 프로테옴.페르디강과 동료들에 의해 만들어진 다크 프로테옴이라는 용어는 알려진 3차원 구조의 다른 단백질에 대한 검출 가능한 배열 상동성을 가지지 않는 단백질의 영역을 정의하며, 따라서 호몰로지로 모델링할 수 없다.546,000 스위스-프로트 단백질의 경우, 진핵 생물과 바이러스의 프로테옴 중 44–54%가 "암흑"인 것으로 밝혀졌으며, 이에 비해 고세균과 [18]박테리아는 14%에 불과했다.
인간 프로테옴.현재, 인간 프로테옴 지도, 프로테오믹스를 포함한 여러 프로젝트들이 인간 프로테옴을 지도화하는 것을 목표로 하고 있다.DB 및 HPP(Human Proteome Project)입니다.인간 게놈 프로젝트와 마찬가지로, 이 프로젝트들은 인간 게놈에 있는 모든 예측된 단백질 코드 유전자에 대한 증거를 찾고 수집하려고 한다.Human Proteome Map은 현재(2020년 10월) 17,294개의 단백질과 프로테오믹스를 주장하고 있다.DB 15,479(다른 기준 사용)2020년 10월 16일, HPP는 예측 단백질 코드 유전자의 90% 이상을 포함하는 높은 문자열 청사진을 발표했다.단백질은 조혈세포를 포함한 광범위한 태아 및 성인 조직과 세포 유형에서 확인된다.
프로테옴을 연구하는 방법
단백질 분석은 핵산 염기서열을 분석하는 것보다 더 어렵다는 것이 입증되었다.DNA를 구성하는 뉴클레오티드가 4개밖에 없는 반면, 단백질을 구성할 수 있는 아미노산은 적어도 20개 이상 있습니다.또한 현재 단일 단백질의 복사본을 만드는 높은 처리량 기술은 알려져 있지 않습니다.단백질, 단백질 세트 또는 전체 프로테옴을 연구하기 위해 많은 방법들이 이용 가능하다.사실, 단백질은 종종 간접적으로 연구된다. 예를 들어, 계산 방법과 게놈 분석을 사용한다.아래에 몇 가지 예만 제시하겠습니다.
분리기술 및 전기영동
프로테옴의 연구인 프로테오믹스는 주로 2차원 겔 전기영동에 의한 단백질 분리를 통해 실행되어 왔다.첫 번째 차원에서는 단백질이 아이소 일렉트릭 포커싱에 의해 분리되며, 이는 전하를 기반으로 단백질을 분해한다.두 번째 차원에서는 SDS-PAGE를 이용하여 단백질을 분자량으로 분리한다.겔은 단백질을 시각화하기 위해 쿠마시에 밝은 파란색 또는 은색으로 염색됩니다.겔에 있는 점들은 특정 위치로 이동한 단백질이다.
질량 분석

질량분석법은 프로테옴을 [20]연구하는 중요한 방법 중 하나이다.일부 중요한 질량 분석 방법에는 궤도 질량 분석법, MALDI(매트릭스 보조 레이저 탈착/이온화), ESI(일렉트로스프레이 이온화) 등이 있다.펩타이드 질량 지문은 단백질을 짧은 펩타이드로 쪼개 식별한 후 관찰된 펩타이드 질량을 배열 데이터베이스에 일치시킴으로써 단백질의 동일성을 추론한다.반면 탠덤 질량분석법은 개별 펩타이드를 분리해 비반응성 기체와 충돌시킨 뒤 생성된 [21]단편 이온을 분류함으로써 배열 정보를 얻을 수 있다.
2014년 5월,[22] 인간 프로테옴의 초안 지도가 네이처에 게재되었다.이 지도는 고해상도 푸리에 변환 질량 분석법을 사용하여 생성되었습니다.이 연구는 17,294개의 유전자에 의해 코드화된 단백질을 식별한 30개의 조직학적으로 정상적인 인간 샘플을 프로파일링했다.이것은 주석이 달린 총 단백질 코드 유전자의 약 84%를 차지한다.
크로마토그래피
액체 크로마토그래피는 단백질 연구에 중요한 도구이다.그것은 매트릭스에 대한 그들의 친화력을 바탕으로 다른 종류의 단백질을 매우 민감하게 분리할 수 있게 해준다.단백질의 분리 및 식별을 위한 새로운 방법으로는 모노리식 캐피럴리 컬럼, 고온 크로마토그래피 및 [23]캐피럴리 일렉트로크로마토그래피의 사용이 있다.
블롯팅
웨스턴 블롯은 특정 단백질의 풍부함을 정량화하기 위해 사용될 수 있다.관심 단백질에 특이적인 항체를 사용함으로써 단백질의 혼합물에서 특이적인 단백질의 존재를 탐사할 수 있다.
단백질보완측정 및 상호작용화면
단백질-조각상보법(protein-fragment comparation assay)은 단백질-단백질 상호작용을 검출하기 위해 종종 사용된다.효모 2-하이브리드 어세이(hybrid assay)가 가장 인기 있지만 체외 및 체내 둘 다 사용되는 수많은 변형이 있다.풀다운 어세이(pull-down assay)는 단백질이 [24]어떤 단백질과 상호작용하는지를 결정하는 방법이다.
단백질 데이터베이스
Human Protein Atlas는 세포, 조직, 장기의 인간 단백질에 대한 정보를 담고 있다.지식 자원에 있는 모든 데이터는 공개되어 있어 학계와 산업계의 과학자들이 인간 프로테옴의 탐사를 위해 데이터에 자유롭게 접근할 수 있습니다.조직 ELIX는IR는 광범위한 생명과학 커뮤니티에 근본적인 중요성을 지닌 단백질 지도책을 핵심 자원으로 선정했다.
혈장 프로테옴 데이터베이스에는 혈장 단백질 10,500개에 대한 정보가 포함되어 있습니다.혈장 내 단백질 함량 범위가 매우 넓기 때문에 풍부한 단백질에 비해 부족한 단백질을 검출하기 어렵다.초저농도의 [25]단백질 검출에 장벽이 될 수 있는 분석 한계가 있다.
neXtprot 및 UniProt와 같은 데이터베이스는 인간 단백질 데이터를 위한 중앙 리소스입니다.
「 」를 참조해 주세요.
레퍼런스
- ^ Morgenstern, Marcel; Stiller, Sebastian B.; Lübbert, Philipp; Peikert, Christian D.; Dannenmaier, Stefan; Drepper, Friedel; Weill, Uri; Höß, Philipp; Feuerstein, Reinhild; Gebert, Michael; Bohnert, Maria (June 2017). "Definition of a High-Confidence Mitochondrial Proteome at Quantitative Scale". Cell Reports. 19 (13): 2836–2852. doi:10.1016/j.celrep.2017.06.014. ISSN 2211-1247. PMC 5494306. PMID 28658629.
- ^ Gómez-Serrano, M (November 2018). "Mitoproteomics: Tackling Mitochondrial Dysfunction in Human Disease". Oxid Med Cell Longev. 2018: 1435934. doi:10.1155/2018/1435934. PMC 6250043. PMID 30533169.
- ^ Uetz, P. (2004-10-15). "From ORFeomes to Protein Interaction Maps in Viruses". Genome Research. 14 (10b): 2029–2033. doi:10.1101/gr.2583304. ISSN 1088-9051. PMID 15489322.
- ^ Maxwell, Karen L.; Frappier, Lori (June 2007). "Viral proteomics". Microbiology and Molecular Biology Reviews. 71 (2): 398–411. doi:10.1128/MMBR.00042-06. ISSN 1092-2172. PMC 1899879. PMID 17554050.
- ^ Viswanathan, Kasinath; Früh, Klaus (December 2007). "Viral proteomics: global evaluation of viruses and their interaction with the host". Expert Review of Proteomics. 4 (6): 815–829. doi:10.1586/14789450.4.6.815. ISSN 1744-8387. PMID 18067418. S2CID 25742649.
- ^ Yang, Ganglong; Xu, Zhipeng; Lu, Wei; Li, Xiang; Sun, Chengwen; Guo, Jia; Xue, Peng; Guan, Feng (2015-07-31). "Quantitative Analysis of Differential Proteome Expression in Bladder Cancer vs. Normal Bladder Cells Using SILAC Method". PLOS ONE. 10 (7): e0134727. Bibcode:2015PLoSO..1034727Y. doi:10.1371/journal.pone.0134727. ISSN 1932-6203. PMC 4521931. PMID 26230496.
- ^ An, Yao; Zhou, Li; Huang, Zhao; Nice, Edouard C.; Zhang, Haiyuan; Huang, Canhua (2019-05-04). "Molecular insights into cancer drug resistance from a proteomics perspective". Expert Review of Proteomics. 16 (5): 413–429. doi:10.1080/14789450.2019.1601561. ISSN 1478-9450. PMID 30925852. S2CID 88474614.
- ^ Cruz, Isa N.; Coley, Helen M.; Kramer, Holger B.; Madhuri, Thumuluru Kavitah; Safuwan, Nur a. M.; Angelino, Ana Rita; Yang, Min (2017-01-01). "Proteomics Analysis of Ovarian Cancer Cell Lines and Tissues Reveals Drug Resistance-associated Proteins". Cancer Genomics & Proteomics. 14 (1): 35–51. doi:10.21873/cgp.20017. ISSN 1109-6535. PMC 5267499. PMID 28031236.
- ^ Geiger, Tamar; Wehner, Anja; Schaab, Christoph; Cox, Juergen; Mann, Matthias (March 2012). "Comparative Proteomic Analysis of Eleven Common Cell Lines Reveals Ubiquitous but Varying Expression of Most Proteins". Molecular & Cellular Proteomics. 11 (3): M111.014050. doi:10.1074/mcp.M111.014050. ISSN 1535-9476. PMC 3316730. PMID 22278370.
- ^ Peng, Xing-Chen; Gong, Feng-Ming; Wei, Meng; Chen, Xi; Chen, Ye; Cheng, Ke; Gao, Feng; Xu, Feng; Bi, Feng; Liu, Ji-Yan (December 2010). "Proteomic analysis of cell lines to identify the irinotecan resistance proteins". Journal of Biosciences. 35 (4): 557–564. doi:10.1007/s12038-010-0064-9. ISSN 0250-5991. PMID 21289438. S2CID 6082637.
- ^ Chen, Yan; Barat, Bidisha; Ray, W. Keith; Helm, Richard F.; Melville, Stephen B.; Popham, David L. (2019-03-15). "Membrane Proteomes and Ion Transporters in Bacillus anthracis and Bacillus subtilis Dormant and Germinating Spores". Journal of Bacteriology. 201 (6). doi:10.1128/JB.00662-18. ISSN 0021-9193. PMC 6398275. PMID 30602489.
- ^ Wilkins, Marc (Dec 2009). "Proteomics data mining". Expert Review of Proteomics. England. 6 (6): 599–603. doi:10.1586/epr.09.81. PMID 19929606. S2CID 207211912.
- ^ Wasinger VC, Cordwell SJ, Cerpa-Poljak A, Yan JX, Gooley AA, Wilkins MR, Duncan MW, Harris R, Williams KL, Humphery-Smith I (1995). "Progress with gene-product mapping of the Mollicutes: Mycoplasma genitalium". Electrophoresis. 16 (1): 1090–94. doi:10.1002/elps.11501601185. PMID 7498152. S2CID 9269742.
- ^ Kozlowski, LP (26 October 2016). "Proteome-pI: proteome isoelectric point database". Nucleic Acids Research. 45 (D1): D1112–D1116. doi:10.1093/nar/gkw978. PMC 5210655. PMID 27789699.
- ^ Leslie, Mitch (2019-10-18). "Outsize impact". Science. 366 (6463): 296–299. Bibcode:2019Sci...366..296L. doi:10.1126/science.366.6463.296. ISSN 0036-8075. PMID 31624194. S2CID 204774732.
- ^ "UniProt: a hub for protein information". Nucleic Acids Research. 43 (D1): D204–D212. 2014. doi:10.1093/nar/gku989. ISSN 0305-1048. PMC 4384041. PMID 25348405.
- ^ a b Aebersold, Ruedi; Agar, Jeffrey N; Amster, I Jonathan; Baker, Mark S; Bertozzi, Carolyn R; Boja, Emily S; Costello, Catherine E; Cravatt, Benjamin F; Fenselau, Catherine; Garcia, Benjamin A; Ge, Ying (March 2018). "How many human proteoforms are there?". Nature Chemical Biology. 14 (3): 206–214. doi:10.1038/nchembio.2576. hdl:1721.1/120977. ISSN 1552-4450. PMC 5837046. PMID 29443976.
- ^ Perdigão, Nelson; et al. (2015). "Unexpected features of the dark proteome". PNAS. 112 (52): 15898–15903. Bibcode:2015PNAS..11215898P. doi:10.1073/pnas.1508380112. PMC 4702990. PMID 26578815.
- ^ Adhikari, S (October 2020). "A high-stringency blueprint of the human proteome". Nature Communications. 11 (1): 5301. Bibcode:2020NatCo..11.5301A. doi:10.1038/s41467-020-19045-9. PMC 7568584. PMID 33067450.
- ^ Altelaar, AF; Munoz, J; Heck, AJ (January 2013). "Next-generation proteomics: towards an integrative view of proteome dynamics". Nature Reviews Genetics. 14 (1): 35–48. doi:10.1038/nrg3356. PMID 23207911. S2CID 10248311.
- ^ Wilhelm, Mathias; Schlegl, Judith; Hahne, Hannes; Gholami, Amin Moghaddas; Lieberenz, Marcus; Savitski, Mikhail M.; Ziegler, Emanuel; Butzmann, Lars; Gessulat, Siegfried; Marx, Harald; Mathieson, Toby; Lemeer, Simone; Schnatbaum, Karsten; Reimer, Ulf; Wenschuh, Holger; Mollenhauer, Martin; Slotta-Huspenina, Julia; Boese, Joos-Hendrik; Bantscheff, Marcus; Gerstmair, Anja; Faerber, Franz; Kuster, Bernhard (2014). "Mass-Spectrometry-Based Draft of the Human Proteome". Nature. 509 (7502): 582–7. Bibcode:2014Natur.509..582W. doi:10.1038/nature13319. PMID 24870543. S2CID 4467721.
- ^ Kim, Min-Sik; et al. (May 2014). "A draft map of the human proteome". Nature. 509 (7502): 575–81. Bibcode:2014Natur.509..575K. doi:10.1038/nature13302. PMC 4403737. PMID 24870542.
- ^ Shi, Yang; Xiang, Rong; Horváth, Csaba; Wilkins, James A. (2004-10-22). "The role of liquid chromatography in proteomics". Journal of Chromatography A. Bioanalytical Chemistry: Perspectives and Recent Advances with Recognition of Barry L. Karger. 1053 (1): 27–36. doi:10.1016/j.chroma.2004.07.044. ISSN 0021-9673. PMID 15543969.
- ^ "Pull-Down Assays - US". www.thermofisher.com. Retrieved 2019-12-05.
- ^ Ponomarenko, Elena A.; Poverennaya, Ekaterina V.; Ilgisonis, Ekaterina V.; Pyatnitskiy, Mikhail A.; Kopylov, Arthur T.; Zgoda, Victor G.; Lisitsa, Andrey V.; Archakov, Alexander I. (2016). "The Size of the Human Proteome: The Width and Depth". International Journal of Analytical Chemistry. 2016: 7436849. doi:10.1155/2016/7436849. ISSN 1687-8760. PMC 4889822. PMID 27298622.
외부 링크
- PIR 데이터베이스
- UniProt 데이터베이스
- 의회 도서관 웹 아카이브의 Pfam 데이터베이스(2011-05-06)