메신저 RNA
Messenger RNA분자생물학에서 메신저 리보핵산(mRNA)은 유전자의 유전자 배열에 대응하는 RNA의 단가닥 분자로 단백질을 합성하는 과정에서 리보솜에 의해 읽힌다.
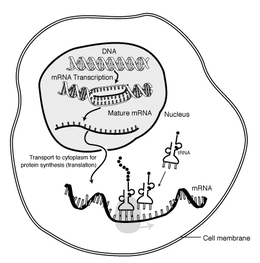
mRNA는 효소(RNA 중합효소)가 유전자를 1차 전사 mRNA(pre-mRNA라고도 함)로 변환하는 전사 과정에서 생성된다.이 사전 mRNA는 보통 최종 아미노산 서열을 위해 코드화되지 않는 영역인 인트론을 포함하고 있습니다.이것들은 RNA 스플라이싱 과정에서 제거되고, 단백질을 코드할 영역인 엑손만 남습니다.이 엑손 배열은 성숙한 mRNA를 구성한다.그 후 성숙한 mRNA는 리보솜에 의해 읽혀지고, tRNA에 의해 운반되는 아미노산을 이용하여 리보솜은 단백질을 생성한다.이 프로세스를 번역이라고 합니다.이 모든 과정은 분자생물학의 중심 교의의 일부를 형성하는데, 이것은 생물학적 시스템에서 유전 정보의 흐름을 묘사한다.
DNA와 마찬가지로 mRNA의 유전자 정보는 3개의 리보뉴클레오티드로 이루어진 코돈으로 배열된 뉴클레오티드의 배열에 포함되어 있다.각 코돈은 단백질 합성을 종료하는 정지 코돈을 제외하고 특정 아미노산을 코드화합니다.코돈을 아미노산으로 변환하려면 코돈을 인식하고 그에 상응하는 아미노산을 제공하는 전달 RNA와 리보솜의 단백질 제조 기계의 중심 성분인 리보솜 RNA(RNA)의 두 가지 다른 RNA가 필요합니다.
mRNA의 개념은 1960년 시드니 브레너와 프란시스 크릭에 의해 개발되었다.실험 검증을 수행하는 동안, 프랑수아 야콥과 자크 모노드는 "메신저 RNA"라는 이름을 만들었다.1961년 제임스 왓슨의 연구팀과 제이콥, 모노드, 매튜 메셀슨 팀에 의해 mRNA가 분리되고 독립적으로 기술되었다.
합성, 처리 및 기능
mRNA 분자의 짧은 존재는 전사에서 시작하여 궁극적으로 분해로 끝납니다.mRNA 분자는 수명 동안 번역 전에 처리, 편집 및 운반될 수 있다.진핵생물 mRNA 분자는 종종 광범위한 처리와 운송을 필요로 하는 반면, 원핵생물 mRNA 분자는 그렇지 않다.진핵생물 mRNA 분자와 그것을 둘러싼 단백질은 함께 메신저 RNP라고 불린다.
문자 변환
전사는 RNA가 DNA에서 복제되는 것이다.전사 중에 RNA 중합효소는 필요에 따라 DNA에서 mRNA로 유전자를 복제합니다.이 과정은 진핵생물과 원핵생물은 약간 다르다.한 가지 주목할 만한 차이점은 원핵 RNA 중합효소가 전사 중에 DNA 처리 효소와 연관되어 전사 중에 처리가 진행될 수 있다는 것이다.따라서, 이것은 tRNA 가닥으로 알려진 상보적인 가닥을 생성함으로써 새로운 mRNA 가닥이 이중 가닥이 되도록 유발하며, 결합되면 염기쌍으로부터 구조를 형성할 수 없다.또한 mRNA용 템플릿은 tRNA의 상보적 가닥으로 DNA가 결합하는 안티코돈 배열과 순차적으로 동일하다.단수명, 미가공 또는 부분적으로 가공된 제품을 전구체 mRNA 또는 사전 mRNA라고 하며, 완전히 가공된 후에는 성숙한 mRNA라고 한다.
진핵생물 사전 mRNA 처리
mRNA의 처리는 진핵생물, 박테리아, 고세균에 따라 크게 다르다.비핵성 mRNA는 본질적으로 전사 후 성숙하며, [1]드문 경우를 제외하고는 가공을 필요로 하지 않는다.그러나 진핵생물 사전 mRNA는 세포질로 전달되고 리보솜에 의해 변환되기 전에 몇 가지 처리 단계를 필요로 한다.
스플라이싱
성숙한 mRNA로 이어지는 진핵생물 사전 mRNA의 광범위한 처리는 RNA 스플라이싱으로, 인트론 또는 아웃론(비부호화 영역)이 제거되고 엑손(부호화 영역)이 결합되는 메커니즘이다.
5인치 캡 추가
5' 캡(RNA 캡, RNA 7-메틸구아노신 캡 또는 RNA mG7 캡이라고도 함)은 전사가 시작된 직후 진핵 전달 RNA의 "전면" 또는 5' 말단에 첨가된 변형 구아닌 뉴클레오티드이다.5' 캡은 5'-5'-3인산 결합을 통해 첫 번째 전사된 뉴클레오티드에 연결된 말단 7-메틸구아노신 잔기로 구성됩니다.리보솜에 의한 인식과 RNases로부터의 보호에 있어서 이 존재는 매우 중요합니다.
캡 첨가는 전사와 결합되어 각각이 서로 영향을 미치도록 동시 전사적으로 발생합니다.전사 개시 직후 합성되는 mRNA의 5' 말단은 RNA 중합효소 관련 캡합성 복합체에 의해 결합된다.이 효소 복합체는 mRNA 캡핑에 필요한 화학 반응을 촉매합니다.합성은 다단계 생화학 반응으로 진행된다.
편집
어떤 경우에는 mRNA가 편집되어 해당 mRNA의 뉴클레오티드 조성이 변경됩니다.인간의 예로는 아폴리포단백질 B mRNA가 있는데, 이것은 일부 조직에서는 편집되지만 다른 조직에서는 편집되지 않습니다.편집에 의해 얼리 스톱 코돈이 생성되고 번역 시 짧은 단백질이 생성됩니다.
폴리아데닐화
폴리아데닐화는 전달 RNA 분자에 대한 폴리아데닐 분자의 공유 결합이다.진핵생물에서 대부분의 메신저 RNA(mRNA) 분자는 3' 말기에 폴리아데닐화되지만, 최근의 연구는 또한 짧은 길이의 우리딘(올리고리딜화)이 [2]흔하다는 것을 보여준다.폴리(A) 꼬리와 그에 결합된 단백질은 mRNA를 엑소뉴클라아제에 의한 분해로부터 보호하는 데 도움이 됩니다.폴리아데닐화는 또한 전사종료, 핵으로부터의 mRNA의 수출 및 번역에도 중요하다.mRNA는 또한 원핵생물에서 폴리아데닐화될 수 있으며, 여기서 폴리(A) 꼬리는 외부핵분해를 방해하는 것이 아니라 촉진하는 역할을 한다.
폴리아데닐화는 DNA를 RNA로 전사하는 동안 및/또는 직후에 발생한다.전사가 종료된 후, mRNA 사슬은 RNA 중합효소 관련 엔도핵산가수분해효소 복합체의 작용을 통해 절단된다.mRNA를 분해한 후 약 250개의 아데노신 잔기를 분할 부위의 유리 3' 말단에 첨가한다.이 반응은 폴리아데닐산 중합효소에 의해 촉매된다.대체 스플라이싱과 마찬가지로 mRNA의 폴리아데닐화 변종이 둘 이상 있을 수 있습니다.
폴리아데닐화 부위 돌연변이도 발생한다.유전자의 1차 RNA 전사체는 poly-A 첨가 부위에서 절단되고, RNA의 3' 말단에 100~200A가 첨가되며, 이 부위가 변경되면 비정상적으로 길고 불안정한 mRNA 구조가 형성된다.
운송
진핵생물과 원핵생물의 또 다른 차이점은 mRNA 수송이다.진핵생물 전사와 번역은 구획적으로 분리되기 때문에, 진핵생물 mRNA는 핵에서 세포질로 내보내져야 한다. 이것은 다른 신호 경로에 [3]의해 조절될 수 있는 과정이다.성숙한 mRNA는 가공된 변형에 의해 인식되며, 그 후 캡결합단백질 CBP20 및 CBP80 [4]및 전사/수출복합체(TREX)[5][6]에 결합함으로써 핵공을 통해 수출된다.진핵생물에서 [7]여러 mRNA 수출 경로가 확인되었다.
공간적으로 복잡한 세포에서, 일부 mRNA는 특정 아세포의 목적지로 운반된다.성숙한 뉴런에서 특정 mRNA는 소마에서 수상돌기로 운반된다.mRNA 번역의 한 부위는 시냅스 [8]아래에 선택적으로 국소화된 폴리리보솜이다.Arc/Arg3.1에 대한 mRNA는 시냅스 활성에 의해 유도되며, NMDA [9]수용체에 의해 생성된 신호에 기초하여 활성 시냅스 근처에서 선택적으로 국부화된다.β-actin mRNA와 [10]같은 외부 자극에 반응하여 다른 mRNA도 덴드라이트 안으로 이동한다. 핵에서 내보내면 액틴 mRNA는 ZBP1 및 40S 서브유닛과 관련된다.복합체는 운동 단백질에 의해 결합되어 세포골격을 따라 표적 위치(뉴라이트 확장)로 운반된다.최종적으로 ZBP1은 Src에 의해 인산화되어 번역이 [11]개시된다.발달하는 뉴런에서, mRNA는 또한 성장하는 축삭, 특히 성장 원추로 운반된다.많은 mRNA에는 특정 [12]장소로 전송되는 것을 목표로 하는 소위 "zip 코드"가 표시되어 있습니다.
번역.
원핵생물 mRNA는 처리되거나 운반될 필요가 없기 때문에, 리보솜에 의한 번역은 전사가 끝난 직후에 시작될 수 있다.따라서 원핵 번역은 전사와 결합되어 동시전사로 일어난다고 할 수 있다.
처리되어 세포질로 운반된 진핵생물 mRNA(성숙한 mRNA)는 리보솜에 의해 번역될 수 있다.번역은 세포질 내에서 자유 부유하는 리보솜에서 일어나거나 신호 인식 입자에 의해 소포체로 유도될 수 있다.그러므로 원핵생물과는 달리 진핵생물 번역은 전사와 직접적으로 연관되어 있지 않다.유방암에서 [13][non-primary source needed]EEF1A1의 mRNA/단백질 수치에서 관찰된 바와 같이, mRNA 수치 감소는 단백질 수치 증가를 동반하는 일부 맥락에서도 가능하다.
구조.
지역 코드화
코딩 영역은 리보솜에 의해 해독되고 단백질로 변환되는 코돈으로 구성되어 있습니다; 진핵생물에서는 보통 하나로, 원핵생물에서는 보통 여러 개로 구성됩니다.코딩 영역은 시작 코돈에서 시작하여 중지 코돈으로 끝납니다.일반적으로 시작 코돈은 AUG 트리플렛이고 중지 코돈은 UAG("암페어"), UAA("ochre") 또는 UGA("opal")입니다.코딩 영역은 내부 염기 쌍에 의해 안정화되는 경향이 있으며,[14][15] 이로 인해 열화가 방지됩니다.코딩 영역의 일부는 단백질 코딩일 뿐만 아니라 엑소닉 스플라이싱 증강제 또는 엑소닉 스플라이싱 소음제로서 프리mRNA의 조절 배열로서 기능할 수 있다.
미번역 영역
미번역영역(UTR)은 시작 코돈 전 및 정지 코돈 후 mRNA의 부분으로 각각 5개의 주요 미번역영역(5' UTR)과 3개의 주요 미번역영역(3' UTR)이라고 불린다.이들 영역은 코드화 영역과 함께 전사되어 성숙한 mRNA에 존재하기 때문에 외부적이다. 유전자 발현에서 mRNA 안정성, mRNA 국재화, 번역 효율 등 미번역 영역에 기인하는 여러 가지 역할이 있다.이러한 기능을 수행하는 UTR의 능력은 UTR의 시퀀스에 따라 다르며 mRNA마다 다를 수 있습니다.또한 3' UTR의 유전자 변이는 RNA 구조와 [16]단백질 번역의 변화로 인해 질병 감수성과 관련이 있다.
mRNA의 안정성은 리보핵산 분해 효소 및 RNA 분해를 촉진하거나 억제할 수 있는 보조 단백질에 대한 다양한 친화력으로 인해 5' UTR 및/또는 3' UTR에 의해 제어될 수 있다.('C 리치 안정성 요소'도 참조).
번역의 완전한 억제를 포함한 번역 효율은 UTR에 의해 제어될 수 있습니다.3' 또는 5' UTR에 결합하는 단백질은 mRNA에 결합하는 리보솜의 능력에 영향을 줌으로써 번역에 영향을 미칠 수 있다. 3' UTR에 결합하는 마이크로RNA도 번역 효율성 또는 mRNA 안정성에 영향을 미칠 수 있다.
mRNA의 세포질 국소화는 3' UTR의 기능인 것으로 생각된다. 세포의 특정 영역에 필요한 단백질도 그곳에서 번역될 수 있다. 이 경우, 3' UTR은 변환을 위해 이 영역에 전사체를 국소화할 수 있는 배열을 포함할 수 있다.
미번역 영역에 포함된 원소 중 일부는 RNA로 변환될 때 특징적인 2차 구조를 형성한다.이러한 구조적 mRNA 요소는 mRNA를 조절하는 데 관여합니다. SECIS 요소와 같은 일부는 단백질이 결합하는 대상입니다.mRNA 요소의 한 종류인 리보스위치는 작은 분자와 직접 결합하며, 전사 또는 번역 수준을 수정하기 위해 접힌 부분을 변경합니다.이 경우 mRNA는 스스로 조절합니다.
폴리(A) 테일
3' 폴리(A) 꼬리는 mRNA의 3' 말단에 첨가된 아데닌 뉴클레오티드의 긴 배열이다.이 꼬리는 핵으로부터의 내보내기와 변환이 촉진되어 mRNA의 열화를 방지합니다.
모노시스트론 대 폴리시스트론 mRNA
mRNA 분자는 단일 단백질 사슬(폴리펩타이드)만 번역하는 유전 정보를 포함할 때 단일istronic이라고 한다.이것은 대부분의 진핵생물 [17][18]mRNA의 경우이다.한편, 폴리시스트론 mRNA는 몇 가지 ORF(Open Reading Frame)를 운반하며, 각각은 폴리펩타이드로 변환됩니다.이러한 폴리펩타이드는 일반적으로 관련된 기능(종종 최종 복합 단백질을 구성하는 서브유닛)을 가지며, 이들의 코드 배열은 프로모터와 연산자를 포함하는 조절 영역에서 함께 그룹화되고 조절된다.박테리아와 고세균에서 발견되는 mRNA의 대부분은 인간의 미토콘드리아 [19]게놈과 마찬가지로 다낭성 [17]물질이다.다이시스트론 또는 바이시스트론 mRNA는 두 가지 단백질만 암호화합니다.
mRNA 순환화
진핵생물에서 mRNA 분자는 eIF4E와 폴리(A) 결합단백질 간의 상호작용에 의해 원형구조를 형성하고, 둘 다 eIF4G에 결합하여 mRNA-단백질-mRNA [20]브릿지를 형성한다.순환화는 시간 효율적인 번역으로 이어지는 mRNA의 리보솜 순환을 촉진하는 것으로 생각되며, 온전한 mRNA만 번역되도록 기능할 수도 있다(부분적으로 분해된 mRNA에는 m7G 캡이 없거나 폴리 A [21]꼬리가 없다).
다른 원형화 메커니즘은 바이러스 mRNA에 특히 존재한다.폴리오바이러스 mRNA는 클로버 잎 단면을 5' 말단으로 하여 폴리(A) 결합 단백질을 결합하는 PCBP2를 결합시켜 친숙한 mRNA-단백질-mRNA 원을 형성한다.보리 황색 왜소 바이러스는 5' 끝과 3' 끝의 mRNA 세그먼트 사이에 결합을 가지며, 단백질 없이 mRNA를 순환시킨다.
RNA 바이러스 게놈(+ 가닥은 mRNA로 번역됨)도 일반적으로 [citation needed]순환됩니다.게놈 복제 동안 원형화는 게놈 복제 속도를 높이기 위해 작용하며, 리보솜과 거의 같은 바이러스 RNA 의존성 RNA 중합효소 순환을 가정합니다.
열화
같은 셀 내의 다른 mRNA는 다른 수명(안정성)을 가진다.박테리아 세포에서, 개별 mRNA는 몇 초에서 한 시간 이상 생존할 수 있다.그러나, 평균 수명은 1분에서 3분 사이이며, 박테리아 mRNA는 진핵생물 [22]mRNA보다 훨씬 덜 안정적이다.포유류의 세포에서 mRNA의 수명은 몇 분에서 [23]며칠에 이른다.mRNA의 안정성이 높을수록 해당 mRNA에서 더 많은 단백질이 생성될 수 있다.mRNA의 제한된 수명은 세포가 변화하는 요구에 반응하여 단백질 합성을 빠르게 변화시킬 수 있게 한다.mRNA의 파괴에는 많은 메커니즘이 있으며, 그 중 일부는 아래에 설명되어 있습니다.
원핵생물 mRNA 분해
일반적으로, 원핵생물에서 mRNA의 수명은 진핵생물보다 훨씬 짧다.원핵생물은 엔도핵산분해효소, 3' 엑소핵산가수분해효소, 5' 엑소핵산가수분해효소를 포함한 리보핵산가수분해효소의 조합을 사용하여 메시지를 분해한다.경우에 따라서는 수십~수백 개의 뉴클레오티드의 작은 RNA 분자(sRNA)가 상보 배열과 염기쌍을 이루고 RNase III에 의한 리보핵산가수분해효소 분해를 촉진함으로써 특정 mRNA의 분해를 촉진할 수 있다.최근 박테리아는 또한 5' 끝에 [24]삼인산으로 구성된 일종의 5' 캡을 가지고 있는 것으로 나타났다.인산염 중 2개를 제거하면 5'1인산염이 남으며, 엑소뉴클레아제 RNase J에 의해 메시지가 파괴되어 5'에서 3'로 분해된다.
진핵생물 mRNA 전환
진핵세포 내부에서는 번역 과정과 mRNA 붕괴 사이에 균형이 있다.능동적으로 번역되는 메시지는 리보솜, 진핵생물 개시인자 eIF-4E 및 eIF-4G 및 폴리(A) 결합단백질에 의해 결합되며, eIF-4E 및 eIF-4G는 디캡핑효소(DCP2)를 차단하고 폴리(A) 결합단백질은 메시지의 말단을 보호한다.번역과 부패 사이의 균형은 P-보디로 [25]알려진 세포질 구조의 크기와 풍부함에 반영됩니다.mRNA의 폴리(A) 꼬리는 RNA의 시스 조절 배열과 전달 작용 RNA 결합 단백질의 조합에 의해 특정 메신저 RNA를 대상으로 하는 특수화된 엑소뉴클라아제들에 의해 짧아진다.폴리(A) 테일 제거는 메시지의 원형 구조를 교란시키고 캡 바인딩 복합체를 불안정하게 하는 것으로 생각됩니다.그 후 메시지는 엑소좀 복합체 또는 디캡핑 복합체에 의해 열화됩니다.이렇게 하면 액티브메시지는 그대로 유지하면서 번역이 비활성화된 메시지는 신속하게 파기할 수 있습니다.변환이 중지되고 메시지가 붕괴 복합체에 전달되는 메커니즘은 자세히 이해되지 않습니다.
AU가 풍부한 원소 붕괴
일부 포유류의 mRNA에서 AU가 풍부한 원소의 존재는 이러한 염기서열을 결합하고 폴리(A) 꼬리 제거를 자극하는 세포 단백질의 작용을 통해 이러한 전달물을 불안정하게 만드는 경향이 있다.폴리(A) 꼬리의 상실은 엑소좀[26] 복합체와 디캡핑 [27]복합체의 공격을 촉진함으로써 mRNA 분해를 촉진하는 것으로 생각된다.AU가 풍부한 원소를 통한 신속한 mRNA 분해는 종양괴사인자(TNF) 및 과립구-대식세포 콜로니 자극인자(GM-CSF)[28]와 같은 강력한 사이토카인의 과잉 생산을 방지하기 위한 중요한 메커니즘이다.AU가 풍부한 원소들은 또한 c-Jun과 c-Fos와 [29]같은 원생전사인자의 생합성을 조절한다.
난센스 매개 붕괴
진핵생물 메시지는 메시지에 조기 정지 코돈(말도 안 되는 코돈)의 존재를 확인하는 넌센스 매개 붕괴(NMD)에 의해 감시된다.이것들은 불완전한 스플라이싱, 적응 면역 체계에서의 V(D)J 재조합, DNA의 돌연변이, 전사 오류, 프레임 이동을 일으키는 리보솜에 의한 누출 스캔, 그리고 다른 원인들에 의해 발생할 수 있습니다.조기정지 코돈의 검출은 5' 디캡핑, 3' 폴리(A) 테일 제거 또는 핵내분해에 [30]의한 mRNA 분해를 유발한다.
소형 간섭 RNA(siRNA)
메타조안에서는 Dicer에 의해 처리되는 작은 간섭 RNA(siRNA)가 RNA 유도 사일런싱 복합체(RISC)로 알려진 복합체에 포함되어 있다.이 복합체는 siRNA가 결합하는 완벽한 보완적 메시지를 분해하는 핵산가수분해효소를 포함한다.생성된 mRNA 조각은 엑소핵산 분해 효소에 의해 파괴된다. siRNA는 세포 배양에서 유전자의 기능을 차단하기 위해 실험실에서 일반적으로 사용된다.그것은 이중가닥 [31]RNA 바이러스에 대한 방어책으로 선천적인 면역체계의 일부로 여겨진다.
MicroRNA(miRNA)
마이크로RNA(miRNA)는 일반적으로 메타조아 메신저 RNA의 [32][33]염기서열을 부분적으로 보완하는 작은 RNA입니다.메시지에 대한 miRNA의 바인딩은 해당 메시지의 번역을 억제하고 폴리(A) 테일 제거를 가속화하여 mRNA 열화를 촉진할 수 있다.miRNA의 작용 메커니즘은 활발한 [34][35]연구의 대상이다.
기타 붕괴 메커니즘
메시지가 저하될 수 있는 다른 방법에는 PiWi-Interacting RNA(piRNA)에 의한 논스톱 붕괴와 사일런싱 등이 있습니다.
적용들
뉴클레오시드 변형 메신저 RNA 배열의 투여는 세포가 단백질을 만들게 할 수 있고, 이는 다시 질병을 직접적으로 치료하거나 백신으로 기능할 수 있다; 보다 간접적으로 단백질은 내생 줄기세포가 원하는 방식으로 [36][37]분화하도록 유도할 수 있다.
RNA 치료의 주요 과제는 적절한 [38]세포에 RNA를 전달하는 데 집중됩니다.도전은 준비 후 벌거벗은 RNA 배열이 자연적으로 분해된다는 사실을 포함합니다; 그것들은 인체의 면역 체계를 자극하여 침입자로 공격할 수 있습니다; 그리고 그들은 세포막에 [37]침투하지 않습니다.일단 세포 안에 들어가면, 그들은 필요한 리보솜을 [36]수용하는 세포질 내에서 행동을 취하기 위해 세포의 운반 메커니즘을 떠나야 합니다.
이러한 과제를 극복하고 치료제로서의 mRNA는 1989년에 "광범위하게 적용 가능한 시험관내 감염 기술의 [39]개발 후" 처음 제시되었다.1990년대에는 비뉴클레오시드 변형 mRNA에 의존하여 개인화 암용 mRNA 백신이 개발되었으며, mRNA 기반 치료법은 자가면역, 대사, 호흡기 염증 질환뿐만 아니라 암의 치료 또는 치료 방법으로도 계속 연구되고 있다.CRISPR과 같은 유전자 편집 요법은 또한 세포가 원하는 Cas [40]단백질을 만들도록 유도하기 위해 mRNA를 사용하는 것으로부터 이익을 얻을 수 있다.
2010년대부터, RNA 백신과 다른 RNA 치료제는 "새로운 종류의 [41]약"으로 여겨져 왔다.첫 번째 mRNA 기반 백신은 제한된 허가를 받았으며 화이자-바이오에 의해 COVID-19 대유행 기간 동안 전 세계에 배포되었다.예를 [42]들어 NTech COVID-19 백신과 Moderna.
역사
1950년대 초부터 분자 연구는 단백질 합성 중에 RNA와 관련된 분자가 있다는 것을 보여 주었다.예를 들어, 초기 보고서 중 하나에서, Jacques Monod와 그의 팀은 RNA 합성이 단백질 합성에, 특히 박테리아 대장균에서 [43]β-갈락토시다아제 생산 중에 필요하다는 것을 보여주었다.Arthur Pardee 또한 [44]1954년에 비슷한 RNA 축적을 발견했다.1953년 알프레드 허시, 준 딕슨, 마사 체이스는 대장균에서 [45]합성된 후 빠르게 소멸된 특정 시토신 함유 DNA(RNA임을 나타냄)를 설명했다.이것은 mRNA의 존재에 대한 첫 번째 기록이었지만,[46] 그렇게 확인되지 않았다.
mRNA의 아이디어는 1960년 4월 15일 시드니 브레너와 프랜시스 크릭이 캠브리지 킹스 칼리지에서 처음 고안했고, 프랑수아 제이콥은 아서 파디, 그 자신, 그리고 모노드가 수행한 최근 실험에 대해 그들에게 말하고 있었다.크릭의 격려로 브레너와 제이콥은 즉시 이 새로운 가설을 시험하기 시작했고 캘리포니아 공과대학의 매튜 메셀슨에게 연락했다.1960년 여름, 브레너, 제이콥, 메셀슨은 칼텍에 있는 메셀슨의 연구실에서 mRNA의 존재를 확립하는 실험을 했다.그 해 가을, 제이콥과 모노드는 "메신저 RNA"라는 이름을 만들고 [46]그 기능을 설명하는 최초의 이론 체계를 개발했습니다.
1961년 2월, 제임스 왓슨은 그의 연구팀이 거의 같은 방향의 유사한 실험을 하면서 그들의 바로 뒤에 있다고 밝혔다; 브레너와 다른 사람들은 그들의 연구 결과의 발표를 연기해 달라는 왓슨의 요청에 동의했다.그 결과, 브레너와 왓슨 기사는 1961년 5월 네이처지의 같은 호에서 동시에 발표되었고, 같은 달 제이콥과 모노드는 분자생물학 저널에 [46]mRNA에 대한 이론적 프레임워크를 발표했다.
「 」를 참조해 주세요.
- mRNA 프로파일링 테크놀로지인 GeneCalling
- 미센스 mRNA
- mRNA 디스플레이
- mRNA 감시
- 전사체, 세포 내 모든 RNA의 총합계
레퍼런스
- ^ Watson JD (February 22, 2013). Molecular Biology of the Gene, 7th edition. Pearson Higher Ed USA. ISBN 9780321851499.
- ^ Choi YS, Patena W, Leavitt AD, McManus MT (March 2012). "Widespread RNA 3'-end oligouridylation in mammals". RNA (New York, N.Y.). 18 (3): 394–401. doi:10.1261/rna.029306.111. PMC 3285928. PMID 22291204.
- ^ Quaresma AJ, Sievert R, Nickerson JA (April 2013). "Regulation of mRNA export by the PI3 kinase/AKT signal transduction pathway". Molecular Biology of the Cell. 24 (8): 1208–1221. doi:10.1091/mbc.E12-06-0450. PMC 3623641. PMID 23427269.
- ^ Kierzkowski D, Kmieciak M, Piontek P, Wojtaszek P, Szweykowska-Kulinska Z, Jarmolowski A (September 2009). "The Arabidopsis CBP20 targets the cap-binding complex to the nucleus, and is stabilized by CBP80". The Plant Journal. 59 (5): 814–825. doi:10.1111/j.1365-313X.2009.03915.x. PMID 19453442.
- ^ Strässer K, Masuda S, Mason P, Pfannstiel J, Oppizzi M, Rodriguez-Navarro S, Rondón AG, Aguilera A, Struhl K, Reed R, Hurt E (May 2002). "TREX is a conserved complex coupling transcription with messenger RNA export". Nature. 417 (6886): 304–308. Bibcode:2002Natur.417..304S. doi:10.1038/nature746. PMID 11979277. S2CID 1112194.
- ^ Katahira J, Yoneda Y (27 October 2014). "Roles of the TREX complex in nuclear export of mRNA". RNA Biology. 6 (2): 149–152. doi:10.4161/rna.6.2.8046. PMID 19229134.
- ^ Cenik C, Chua HN, Zhang H, Tarnawsky SP, Akef A, Derti A, Tasan M, Moore MJ, Palazzo AF, Roth FP (April 2011). "Genome analysis reveals interplay between 5'UTR introns and nuclear mRNA export for secretory and mitochondrial genes". PLOS Genetics. 7 (4): e1001366. doi:10.1371/journal.pgen.1001366. PMC 3077370. PMID 21533221.
- ^ Steward O, Levy WB (March 1982). "Preferential localization of polyribosomes under the base of dendritic spines in granule cells of the dentate gyrus". The Journal of Neuroscience. 2 (3): 284–291. doi:10.1523/JNEUROSCI.02-03-00284.1982. PMC 6564334. PMID 7062109.
- ^ Steward O, Worley PF (April 2001). "Selective targeting of newly synthesized Arc mRNA to active synapses requires NMDA receptor activation". Neuron. 30 (1): 227–240. doi:10.1016/s0896-6273(01)00275-6. PMID 11343657. S2CID 13395819.
- ^ Job C, Eberwine J (December 2001). "Localization and translation of mRNA in dendrites and axons". Nature Reviews. Neuroscience. 2 (12): 889–898. doi:10.1038/35104069. PMID 11733796. S2CID 5275219.
- ^ Hüttelmaier S, Zenklusen D, Lederer M, Dictenberg J, Lorenz M, Meng X, et al. (November 2005). "Spatial regulation of beta-actin translation by Src-dependent phosphorylation of ZBP1". Nature. 438 (7067): 512–5. Bibcode:2005Natur.438..512H. doi:10.1038/nature04115. PMID 16306994. S2CID 2453397.
- ^ Ainger K, Avossa D, Diana AS, Barry C, Barbarese E, Carson JH (September 1997). "Transport and localization elements in myelin basic protein mRNA". The Journal of Cell Biology. 138 (5): 1077–1087. doi:10.1083/jcb.138.5.1077. PMC 2136761. PMID 9281585.
- ^ Lin CY, Beattie A, Baradaran B, Dray E, Duijf PH (September 2018). "Contradictory mRNA and protein misexpression of EEF1A1 in ductal breast carcinoma due to cell cycle regulation and cellular stress". Scientific Reports. 8 (1): 13904. Bibcode:2018NatSR...813904L. doi:10.1038/s41598-018-32272-x. PMC 6141510. PMID 30224719.
- ^ Shabalina SA, Ogurtsov AY, Spiridonov NA (2006). "A periodic pattern of mRNA secondary structure created by the genetic code". Nucleic Acids Research. 34 (8): 2428–2437. doi:10.1093/nar/gkl287. PMC 1458515. PMID 16682450.
- ^ Katz L, Burge CB (September 2003). "Widespread selection for local RNA secondary structure in coding regions of bacterial genes". Genome Research. 13 (9): 2042–2051. doi:10.1101/gr.1257503. PMC 403678. PMID 12952875.
- ^ Lu YF, Mauger DM, Goldstein DB, Urban TJ, Weeks KM, Bradrick SS (November 2015). "IFNL3 mRNA structure is remodeled by a functional non-coding polymorphism associated with hepatitis C virus clearance". Scientific Reports. 5: 16037. Bibcode:2015NatSR...516037L. doi:10.1038/srep16037. PMC 4631997. PMID 26531896.
- ^ a b Kozak M (March 1983). "Comparison of initiation of protein synthesis in procaryotes, eucaryotes, and organelles". Microbiological Reviews. 47 (1): 1–45. doi:10.1128/MMBR.47.1.1-45.1983. PMC 281560. PMID 6343825.
- ^ Niehrs C, Pollet N (December 1999). "Synexpression groups in eukaryotes". Nature. 402 (6761): 483–487. Bibcode:1999Natur.402..483N. doi:10.1038/990025. PMID 10591207. S2CID 4349134.
- ^ Mercer TR, Neph S, Dinger ME, Crawford J, Smith MA, Shearwood AM, Haugen E, Bracken CP, Rackham O, Stamatoyannopoulos JA, Filipovska A, Mattick JS (August 2011). "The human mitochondrial transcriptome". Cell. 146 (4): 645–658. doi:10.1016/j.cell.2011.06.051. PMC 3160626. PMID 21854988.
- ^ Wells SE, Hillner PE, Vale RD, Sachs AB (July 1998). "Circularization of mRNA by eukaryotic translation initiation factors". Molecular Cell. 2 (1): 135–140. CiteSeerX 10.1.1.320.5704. doi:10.1016/S1097-2765(00)80122-7. PMID 9702200.
- ^ López-Lastra M, Rivas A, Barría MI (2005). "Protein synthesis in eukaryotes: the growing biological relevance of cap-independent translation initiation". Biological Research. 38 (2–3): 121–146. doi:10.4067/S0716-97602005000200003. PMID 16238092.
- ^ Lewin B, Krebs JE, Kilpatrick ST, Goldstein ES, eds. (2011). Lewin's genes X (10th ed.). Sudbury, Mass.: Jones and Bartlett. ISBN 9780763766320. OCLC 456641931.
- ^ Yu J, Russell JE (September 2001). "Structural and functional analysis of an mRNP complex that mediates the high stability of human beta-globin mRNA". Molecular and Cellular Biology. 21 (17): 5879–5888. doi:10.1128/mcb.21.17.5879-5888.2001. PMC 87307. PMID 11486027.
- ^ Deana A, Celesnik H, Belasco JG (January 2008). "The bacterial enzyme RppH triggers messenger RNA degradation by 5' pyrophosphate removal". Nature. 451 (7176): 355–358. Bibcode:2008Natur.451..355D. doi:10.1038/nature06475. PMID 18202662. S2CID 4321451.
- ^ Parker R, Sheth U (March 2007). "P bodies and the control of mRNA translation and degradation". Molecular Cell. 25 (5): 635–646. doi:10.1016/j.molcel.2007.02.011. PMID 17349952.
- ^ Chen CY, Gherzi R, Ong SE, Chan EL, Raijmakers R, Pruijn GJ, Stoecklin G, Moroni C, Mann M, Karin M (November 2001). "AU binding proteins recruit the exosome to degrade ARE-containing mRNAs". Cell. 107 (4): 451–464. doi:10.1016/S0092-8674(01)00578-5. PMID 11719186. S2CID 14817671.
- ^ Fenger-Grøn M, Fillman C, Norrild B, Lykke-Andersen J (December 2005). "Multiple processing body factors and the ARE binding protein TTP activate mRNA decapping". Molecular Cell. 20 (6): 905–915. doi:10.1016/j.molcel.2005.10.031. PMID 16364915.
- ^ Shaw G, Kamen R (August 1986). "A conserved AU sequence from the 3' untranslated region of GM-CSF mRNA mediates selective mRNA degradation". Cell. 46 (5): 659–667. doi:10.1016/0092-8674(86)90341-7. PMID 3488815. S2CID 40332253.
- ^ Chen CY, Shyu AB (November 1995). "AU-rich elements: characterization and importance in mRNA degradation". Trends in Biochemical Sciences. 20 (11): 465–470. doi:10.1016/S0968-0004(00)89102-1. PMID 8578590.
- ^ Isken O, Maquat LE (August 2007). "Quality control of eukaryotic mRNA: safeguarding cells from abnormal mRNA function". Genes & Development. 21 (15): 1833–1856. doi:10.1101/gad.1566807. PMID 17671086.
- ^ Obbard DJ, Gordon KH, Buck AH, Jiggins FM (January 2009). "The evolution of RNAi as a defence against viruses and transposable elements". Philosophical Transactions of the Royal Society of London. Series B, Biological Sciences. 364 (1513): 99–115. doi:10.1098/rstb.2008.0168. PMC 2592633. PMID 18926973.
- ^ 로버트 E.Farrell, Jr. RNA 방법론, 제5판학술 출판사, 2017
- ^ Brennecke J, Stark A, Russell RB, Cohen SM (March 2005). "Principles of microRNA-target recognition". PLOS Biology. 3 (3): e85. doi:10.1371/journal.pbio.0030085. PMC 1043860. PMID 15723116.
- ^ 타스쿠 혼조, 마이클 리스, 안드레아스 라드브루치, 프레데릭 알트B세포의 분자생물학 제2판Academic Press, 2014년 ('마이크로RNA에 관한 최신 연구' 포함)
- ^ Eulalio A, Huntzinger E, Nishihara T, Rehwinkel J, Fauser M, Izaurralde E (January 2009). "Deadenylation is a widespread effect of miRNA regulation". RNA. 15 (1): 21–32. doi:10.1261/rna.1399509. PMC 2612776. PMID 19029310.
- ^ a b Hajj KA, Whitehead KA (12 September 2017). "Tools for translation: non-viral materials for therapeutic mRNA delivery". Nature Reviews Materials. 2 (10): 17056. Bibcode:2017NatRM...217056H. doi:10.1038/natrevmats.2017.56.
- ^ a b Gousseinov E, Kozlov M, Scanlan C (September 15, 2015). "RNA-Based Therapeutics and Vaccines". Genetic Engineering News.
- ^ Kaczmarek JC, Kowalski PS, Anderson DG (June 2017). "Advances in the delivery of RNA therapeutics: from concept to clinical reality". Genome Medicine. 9 (1): 60. doi:10.1186/s13073-017-0450-0. PMC 5485616. PMID 28655327.
- ^ Schlake T, Thess A, Fotin-Mleczek M, Kallen KJ (November 2012). "Developing mRNA-vaccine technologies". RNA Biology. 9 (11): 1319–30. doi:10.4161/rna.22269. PMC 3597572. PMID 23064118.
- ^ Haridi R (2021-04-23). "The mRNA revolution: How COVID-19 hit fast-forward on an experimental technology". New Atlas. Retrieved 2021-04-26.
{{cite web}}: CS1 maint :url-status (링크) - ^ Kowalska J, Wypijewska del Nogal A, Darzynkiewicz ZM, Buck J, Nicola C, Kuhn AN, Lukaszewicz M, Zuberek J, Strenkowska M, Ziemniak M, Maciejczyk M, Bojarska E, Rhoads RE, Darzynkiewicz E, Sahin U, Jemielity J (2014), "mRNA-based therapeutics–developing a new class of drugs.", Nature Reviews Drug Discovery, vol. 13, no. 10, pp. 759–780, doi:10.1093/nar/gku757, PMC 4176373, PMID 25150148
- ^ Barbier AJ, Jiang AY, Zhang P, Wooster R, Anderson DG (June 2022). "The clinical progress of mRNA vaccines and immunotherapies". Nature Biotechnology. 40 (6): 840–854. doi:10.1038/s41587-022-01294-2. PMID 35534554.
- ^ Monod J, Pappenheimer AM, Cohen-Bazire G (1952). "La cinétique de la biosynthèse de la β-galactosidase chez E. coli considérée comme fonction de la croissance". Biochimica et Biophysica Acta (in French). 9: 648–660. doi:10.1016/0006-3002(52)90227-8.
- ^ Pardee AB (May 1954). "NUCLEIC ACID PRECURSORS AND PROTEIN SYNTHESIS". Proceedings of the National Academy of Sciences of the United States of America. 40 (5): 263–270. doi:10.1073/pnas.40.5.263. PMC 534118. PMID 16589470.
- ^ Hershey AD, Dixon J, Chase M (July 1953). "Nucleic acid economy in bacteria infected with bacteriophage T2. I. Purine and pyrimidine composition". The Journal of General Physiology. 36 (6): 777–789. doi:10.1085/jgp.36.6.777. PMC 2147416. PMID 13069681.
- ^ a b c Cobb M (29 June 2015). "Who discovered messenger RNA?". Current Biology. 25 (13): R526–R532. doi:10.1016/j.cub.2015.05.032. PMID 26126273.
추가 정보
- Alsaweed M, Lai CT, Hartmann PE, Geddes DT, Kakulas F (February 2016). "Human milk miRNAs primarily originate from the mammary gland resulting in unique miRNA profiles of fractionated milk". Scientific Reports. 6 (1): 20680. Bibcode:2016NatSR...620680A. doi:10.1038/srep20680. PMC 4745068. PMID 26854194.
- Lillycrop KA, Burdge GC (October 2012). "Epigenetic mechanisms linking early nutrition to long term health". Best Practice & Research. Clinical Endocrinology & Metabolism. 26 (5): 667–676. doi:10.1016/j.beem.2012.03.009. PMID 22980048.
- Melnik BC, Kakulas F, Geddes DT, Hartmann PE, John SM, Carrera-Bastos P, Cordain L, Schmitz G (21 June 2016). "Milk miRNAs: simple nutrients or systemic functional regulators?". Nutrition & Metabolism. 13 (1): 42. doi:10.1186/s12986-016-0101-2. PMC 4915038. PMID 27330539.
- Vickers MH (June 2014). "Early life nutrition, epigenetics and programming of later life disease". Nutrients. 6 (6): 2165–2178. doi:10.3390/nu6062165. PMC 4073141. PMID 24892374.
- Zhou Q, Li M, Wang X, Li Q, Wang T, Zhu Q, Zhou X, Wang X, Gao X, Li X (2012). "Immune-related microRNAs are abundant in breast milk exosomes". International Journal of Biological Sciences. 8 (1): 118–123. doi:10.7150/ijbs.8.118. PMC 3248653. PMID 22211110.
외부 링크
- RNAi Atlas: RNAi 라이브러리와 대상 분석 결과의 데이터베이스
- miRSearch 2012-12-04 Wayback Machine에서 아카이브:mRNA를 대상으로 하는 마이크로RNA를 찾기 위한 도구
- mRNA의 코드화 방법:유튜브 동영상
- mRNA란? theconversation.com