유비퀴틴
Ubiquitin| 유비퀴틴족 | |||||||||
|---|---|---|---|---|---|---|---|---|---|
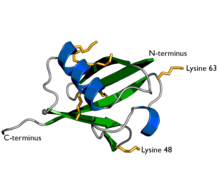 유비퀴틴의 도표.7개의 리신 측쇄는 노란색/주황색으로 표시됩니다. | |||||||||
| 식별자 | |||||||||
| 기호. | 유비퀴틴 | ||||||||
| 팜 | PF00240 | ||||||||
| 인터프로 | IPR000626 | ||||||||
| 프로 사이트 | PDOC00271 | ||||||||
| SCOP2 | 1aar/SCOPe/SUPFAM | ||||||||
| |||||||||
유비퀴틴은 진핵생물 대부분의 조직에서 발견되는 작은 조절 단백질이다. 즉, 어디에서나 발견된다.이것은 1975년[1] Gideon Goldstein에 의해 발견되었고 1970년대 후반과 1980년대에 [2]걸쳐 더욱 특징지어졌습니다.유비퀴틴의 인간 게놈 코드에는 UBB, UBC, UBA52 및 RPS27A의 [3]4가지 유전자가 있습니다.
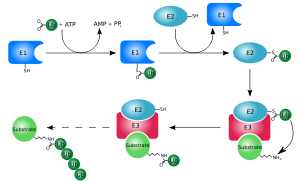
기질 단백질에 유비퀴틴의 첨가는 유비퀴틸화(또는 유비퀴티네이션 또는 유비퀴티닐화)라고 불린다.유비퀴틸화는 단백질에 많은 방식으로 영향을 미칩니다: 단백질은 단백질 분해에 대한 표시를 하고, 세포 위치를 바꾸고, 그들의 활동에 영향을 미치고,[4][5][6] 단백질 상호작용을 촉진하거나 예방할 수 있습니다.유비퀴틸화는 유비퀴틴 활성화 효소(E1s), 유비퀴틴 결합 효소(E2s), 유비퀴틴 결합 효소(E3s)에 의해 수행되는 활성화, 결합, 결합의 세 가지 주요 단계를 포함한다.이 순차적 캐스케이드의 결과는 이소펩타이드 결합을 통해 단백질 기질의 리신 잔류물, 티오에스테르 결합을 통한 시스테인 잔류물, 에스테르 결합을 통해 세린 및 트레오닌 잔류물 또는 펩타이드 [7][8][9]결합을 통해 단백질 N-말단의 아미노기에 결합한다.
단백질 변형은 단일 유비퀴틴 단백질(모노비퀴틸화) 또는 유비퀴틴 사슬(폴리유비퀴틸화)일 수 있다.2차 유비퀴틴 분자는 항상 이전의 유비퀴틴 분자의 7개의 리신 잔기 또는 N말단 메티오닌 중 하나에 연결되어 있다.이러한 '연결' 잔류물은 "K" 또는 "M"(각각 리신과 메티오닌의 1글자 아미노산 표기법)과 K48, K29 또는 M1에서와 같이 유비퀴틴 분자에서의 위치를 참조하는 숫자로 표현된다. 첫 번째 유비퀴틴 분자는 특정 리신 C 말단을 통해 공유 결합된다.표적 단백질의 ne, threonine 또는 N-terminus.폴리유비퀴틸화는 다른 유비퀴틴의 C 말단이 이전에 첨가된 유비퀴틴 분자 상의 7개의 리신 잔기 또는 첫 번째 메티오닌 중 하나에 연결되어 사슬을 형성할 때 발생한다.이 과정이 여러 번 반복되어 여러 유비퀴틴이 추가된다.정의된 리신(주로 K48 및 K29)에 대한 폴리유비퀴틸화만이 프로테아솜에 의한 분해('죽음의 분자 키스'라고 함)와 관련이 있는 반면, 다른 폴리유비퀴틸화(K63, K11, K6 및 M1에 대한 폴리유비퀴틸화는 염증, 종말세포와 같은 과정을 조절할 수 있다.
유비퀴틴 사슬이 단백질을 분해하고 재활용하는 프로테아솜을 표적 단백질로 만든다는 사실은 2004년 [8][11][12]노벨 화학상을 수상했다.
신분증
유비퀴틴(원래 유비쿼터스 면역 폴리펩타이드)은 1975년 모든[1] 진핵 세포에서 발현되는 8.6 kDa 단백질로 처음 확인되었다.유비퀴틴의 기본 기능과 유비퀴틸화 경로의 구성요소는 1980년대 초 Aaron Ciechanover, Avram Hershko, Irwin Rose가 2004년 [11]노벨 화학상을 수상한 Technion에서 설명되었습니다.
유비퀴틸화 시스템은 초기에 세포 추출물에 존재하는 ATP 의존성 단백질 분해 시스템으로 특징지어졌다.이러한 추출물에 존재하는 열안정성 폴리펩타이드인 ATP 의존성 단백질 분해 인자 1(APF-1)은 ATP 의존성 [13]및 Mg2+ 의존성 프로세스에서 모델 단백질 기질인 리조자임에 공유 결합하는 것으로 밝혀졌다.복수의 APF-1 분자는 이소펩타이드 결합에 의해 단일 기질 분자에 연결되었고, 결합체는 유리 APF-1의 방출과 함께 빠르게 분해되는 것으로 확인되었다. APF-1-단백질 결합이 특성화된 직후 APF-1은 유비퀴틴으로 확인되었다.유비퀴틴(Gly76)의 C 말단 글리신 잔기의 카르복실기는 기질 리신 잔기와 결합된 부분으로 확인되었다.
단백질
| 잔류물수 | 76 |
|---|---|
| 분자량 | 8564.8448 Da |
| 등전점(pI) | 6.79 |
| 유전자명 | RPS27A(UBA80, UBCEP1, UBA52(UBCEP2), UBB, UBC |
| 시퀀스(단일 문자) | MQIFVLTG TITLE VEPSTien VKAKIQDKEGIPPD QQ리페아그클리드GRTDYNIQESTLVLRGG |
유비퀴틴은 모든 진핵 세포에 존재하는 작은 단백질이다.그것은 광범위한 표적 단백질에 대한 결합을 통해 무수한 기능을 수행한다.다양한 수정이 발생할 수 있습니다.유비퀴틴 단백질 자체는 76개의 아미노산으로 구성되며 분자량은 약 8.6kDa이다.주요 특징으로는 C 말단 꼬리와 7 리신 잔류물이 있습니다.그것은 진핵생물 진화 내내 잘 보존되어 인간과 효모 유비퀴틴은 96%의 염기서열 [citation needed]정체성을 공유한다.
유전자
유비퀴틴은 포유류의 4가지 유전자에 의해 암호화된다.UBA52 및 RPS27A 유전자는 각각 리보솜 단백질 L40 및 S27a에 융합된 단일 유비퀴틴을 코드한다.UBB 및 UBC 유전자는 폴리유비퀴틴 전구 [3]단백질을 코드화한다.
유비퀴틸화
유비퀴틸화(유비퀴티네이션 또는 유비퀴티닐화라고도 함)는 유비퀴틴 단백질이 기질 단백질에 결합되는 효소적 번역 후 변형이다.이 과정은 가장 일반적으로 유비퀴틴(글리신 76)의 마지막 아미노산을 기질상의 리신 잔기에 결합시킨다.유비퀴틴 글리신의 카르복실기(COO)−와 기판의 리신의 [14]엡실론아미노기(δ-NH+
3) 사이에 이소펩티드 결합을 형성한다.유비퀴틴 결합 기질의 트립신 분할은 유비퀴틸화 [15][16]부위를 식별하기 위해 사용되는 디글리신 "잔량"을 남긴다.유비퀴틴은 또한 "비 카논론적 유비쿼티실화"[9]라고 불리는 전자가 풍부한 친핵성 단백질의 다른 부위에 결합될 수 있다.이것은 단백질 MyoD에서[17] 리신 잔기가 아닌 단백질 N 말단의 아민 그룹이 유비쿼티화에 사용되는 것으로 처음 관찰되었고, 유비퀴틴 [37][38]자체를 포함한 여러 [18][19][20][21][22][23][24][25][26][27][28][29][30][31][32][33][34][35][36]종의 다른 22개 단백질에서 그 이후로 관찰되었다.또한 시스테인의 [33][34][39][40][41][42][43][44][45][46]술프하이드릴기, 트레오닌과 [33][34][39][45][46][47][48][49][50]세린의 수산기와 같은 비아민기를 사용하는 유비퀴틸화 표적으로 비라이신 잔기에 대한 증거가 증가하고 있다.이 과정의 최종 결과는 하나의 유비퀴틴 분자(모노비퀴틸화) 또는 유비퀴틴 분자 사슬(폴리유비퀴티네이션)을 기질 [51]단백질에 추가하는 것이다.
유비퀴티네이션은 유비퀴틴 활성화 효소, 유비퀴틴 결합 효소, 그리고 각각 E1s, E2s, E3s로 알려진 유비퀴틴 결합 효소의 세 가지 유형을 필요로 한다.이 프로세스는 다음 3가지 주요 단계로 구성됩니다.
- 활성화 : 유비퀴틴은 ATP에 의존하는 E1 유비퀴틴 활성화 효소에 의해 2단계 반응으로 활성화된다.첫 단계는 유비퀴틴-아데닐산 중간체의 생산을 포함한다.E1은 ATP와 유비퀴틴을 결합하고 유비퀴틴 분자의 C 말단의 아실 아데닐화를 촉매한다.두 번째 단계는 AMP의 방출과 함께 유비퀴틴을 활성 부위 시스테인 잔기로 이동시킨다. 이 단계는 유비퀴틴의 C 말단 카르복실기와 E1 시스테인 술프하이드릴기 [14][52]사이에 티오에스테르 결합을 일으킨다.인간 게놈은 유비퀴틴을 활성화시킬 수 있는 효소를 생성하는 두 개의 유전자를 포함하고 있다: UBA1과 UBA6.[53]
- 접합: E2 유비퀴틴 결합효소는 트랜스(티오)에스테르화 반응을 통해 E1에서 E2의 활성부위 시스테인으로의 유비퀴틴 전달을 촉매한다.이 반응을 수행하기 위해 E2는 활성화된 유비퀴틴과 E1 효소에 결합한다.인간은 35가지의 다른 E2 효소를 가지고 있는 반면, 다른 진핵 생물들은 16개에서 35개 사이의 효소를 가지고 있다.유비퀴틴 결합 촉매([54]UBC) 접힘으로 알려진 매우 보존된 구조로 특징지어집니다.
- 결합: E3 유비퀴틴 결합효소는 유비퀴티네이션 캐스케이드의 마지막 단계를 촉매합니다.가장 일반적으로, 그들은 표적 단백질의 리신과 유비퀴틴의 C 말단 글리신 사이에 이소펩티드 결합을 형성한다.일반적으로 이 단계에는 수백 개의 E3 중 하나의 액티비티가 필요합니다.E3 효소는 시스템의 기판 인식 모듈로 기능하며 E2 및 기판과 상호 작용할 수 있습니다.일부 E3 효소는 또한 E2 효소를 활성화시킨다.E3 효소는 E6-AP 카르복실 말단(HECT) 도메인과 정말로 흥미로운 새로운 유전자(RING) 도메인(또는 밀접하게 관련된 U-box 도메인)의 두 도메인 중 하나를 가지고 있다.HEC 도메인 E3s는 이 과정에서 유비퀴틴과 일시적으로 결합하는 반면, RING 도메인 E3s는 E2 효소에서 [55]기질로의 직접 전달을 촉매한다.아나베이스 촉진 복합체(APC)와 SCF 복합체(Skp1-Cullin-F-box 단백질 복합체의 경우)는 [56]프로테아솜에 의한 분해에 대한 특정 표적 단백질의 인식 및 유비쿼티화에 관여하는 다중 서브유닛 E3의 두 가지 예이다.
유비쿼티네이션 캐스케이드에서는, E1은 계층적인 방법으로 수백 개의 E3와 바인드 할 수 있는 많은 E2와 바인드 할 수 있습니다.캐스케이드 내에 레벨이 있으면 유비쿼티네이션 [7]기계를 엄격하게 규제할 수 있습니다.다른 유비퀴틴 유사단백질(UBL)도 E1–E2–E3 캐스케이드를 통해 수정되지만, 이러한 시스템의 변화는 존재한다.[57]
E4 효소 또는 유비퀴틴 사슬 신장 인자는 미리 형성된 폴리유비퀴틴 사슬을 기질 [58]단백질에 첨가할 수 있다.예를 들어 Mdm2에 의한 종양억제제[59] p53의 복수 모노비퀴틸화에 이어 p300 및 CBP를 [60][61]이용한 폴리유비퀴틴 사슬을 추가할 수 있다.
종류들
유비퀴티네이션은 단백질의 분해 조절, 단백질의 세포 위치 조정, 단백질 활성화 및 비활성화, 단백질-단백질 [4][5][6]상호작용을 조절함으로써 세포 과정에 영향을 미친다.이러한 효과는 예를 들어 단일 유비퀴틴 분자(모노비퀴티네이션) 또는 다른 유형의 유비퀴티네이션 사슬(폴리유비퀴티네이션)[62]의 추가와 같은 다양한 유형의 기질 유비퀴티네이션에 의해 매개된다.
모노비퀴티네이션
모노비퀴티네이션은 하나의 기질 단백질 잔기에 하나의 유비퀴틴 분자를 추가하는 것이다.다중모노비퀴티네이션은 하나의 유비퀴틴 분자가 여러 기질 잔기에 첨가되는 것이다.단백질의 모노비퀴티네이션은 동일한 단백질의 폴리유비퀴티네이션에 다른 영향을 미칠 수 있다.단일 유비퀴틴 분자의 첨가는 폴리유비퀴틴 [62]사슬을 형성하기 전에 필요한 것으로 생각된다.모노비퀴티네이션은 막 밀매, 세포내구증, 바이러스 [10][63]싹과 같은 세포 과정에 영향을 미친다.
폴리유비퀴틴사슬
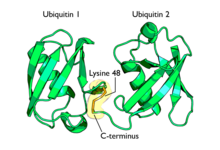
폴리유비퀴티네이션은 기질 단백질 상의 단일 리신 잔기에 유비퀴틴 사슬이 형성되는 것이다.단백질 기질에 단일 유비퀴틴 부분을 첨가한 후 추가로 유비퀴틴 분자를 첨가하여 폴리유비퀴틴 [62]사슬을 생성할 수 있다.이러한 사슬은 유비퀴틴 분자의 글리신 잔기를 기질에 결합된 유비퀴틴의 리신에 연결함으로써 만들어진다.유비퀴틴은 7개의 리신 잔기와 유비퀴티네이션 지점 역할을 하는 N-말단을 가지고 있으며,[8] 이들은 각각 K6, K11, K27, K29, K33, K48, K63 및 M1이다.리신 48 연결 사슬은 최초로 확인되었으며 유비퀴틴 사슬의 가장 잘 특징지어지는 유형이다.K63 사슬은 또한 잘 특징지어지는 반면, 다른 리신 사슬, 혼합 사슬, 분기 사슬, M1 연결 선형 사슬 및 이종 사슬(유비퀴틴과 다른 유비퀴틴 유사 단백질의 혼합물)의 기능은 더 불분명하다.[16][38][62][63][64]
리신 48 연결 폴리유비퀴틴 사슬은 단백질 분해로 알려진 과정에 의해 파괴를 위한 단백질을 목표로 한다.적어도 4개의 유비퀴틴 분자가 긴 멀티유비퀴틴 사슬이 26S 프로테아솜에 [65]인식되기 위해서는 피복단백질 상의 리신 잔기에 부착되어야 한다.이것은 4개의 링 구조로 이루어진 중앙단백질분해코어로 이루어진 배럴형 구조이며, 측면에는 유비쿼티화 단백질의 선택적으로 유입이 가능한 2개의 실린더가 있다.일단 내부로 들어가면 단백질은 작은 펩타이드로 빠르게 분해된다(보통 길이 아미노산 잔류물 3-25).유비퀴틴 분자는 파괴되기 직전에 단백질에서 분리되어 [66]더 사용하기 위해 재활용됩니다.단백질 기질의 대부분은 유비쿼티드화 되어 있지만 프로테아솜을 대상으로 [67]하는 비유비쿼티드 단백질의 예가 있다.폴리유비퀴틴 사슬은 프로테아좀의 서브유닛인 S5a/Rpn10에 의해 인식된다.이는 S5a/Rpn10 장치의 [4]C 말단 영역의 소수성 패치에 있는 유비퀴틴 상호작용 모티브(UIM)에 의해 달성된다.
리신63결합사슬은 기질단백질의 프로테아솜분해와 관련이 없다.대신에, 그들은 내세포 밀매, 염증, 번역, 그리고 DNA [10]복구와 같은 다른 과정들의 조정을 가능하게 한다.세포에서 리신63결합사슬은 ESCRT-0복합체에 의해 결합되어 프로테아솜에 결합하는 것을 방지한다.이 복합체는 UIM을 포함하는 Hrs와 STAM1이라는 두 개의 단백질을 포함하고 있으며, UIM은 리신 63 연결 [68][69]사슬에 결합할 수 있게 해줍니다.
비정형(비리신 48-결합) 유비퀴틴 체인에 대해서는 덜 알려져 있지만 연구는 이러한 [63]체인에 대한 역할을 제안하기 시작했다.리신 6, 11, 27, 29 및 메티오닌 1에 의해 결합된 비정형 사슬이 프로테아솜 [67][70]분해를 유도할 수 있다는 증거가 있다.
복수의 연결형을 포함한 분기형 유비퀴틴 사슬을 [71]형성할 수 있다.이들 체인의 기능은 [8]불분명합니다.
구조.
서로 다르게 연결된 사슬은 단백질 사슬의 구조 차이로 인해 단백질에 특정한 영향을 미친다.K29-, K33-,[72] K63- 및 M1 연결 사슬은 상당히 선형적인 구조를 가지며, 이러한 사슬을 개방형 구성 사슬이라고 한다.K6-, K11- 및 K48 연결 체인은 폐쇄 구성을 형성합니다.오픈 컨피규레이션 사슬의 유비퀴틴 분자는 그것들을 서로 연결하는 공유가 이소펩타이드 결합을 제외하고 서로 상호작용하지 않는다.이와는 대조적으로, 닫힌 배향 사슬은 상호 작용 잔류물과의 계면을 가진다.사슬 구조를 변경하는 것은 유비퀴틴 단백질의 다른 부분을 노출하고 숨기고, 다른 연결은 연결에 고유한 고유한 토폴로지에 특정한 단백질에 의해 인식된다.단백질은 유비퀴틴 결합 도메인(UBD)을 통해 유비퀴틴에 특이적으로 결합할 수 있다.사슬에서 개별 유비퀴틴 단위 사이의 거리는 리신 63-연결 사슬과 48-연결 사슬 사이에서 다르다.UBD는 리신 48 연결 사슬(콤팩트 유비퀴틴 사슬)을 결합하는 유비퀴틴 상호작용 모티브와 리신 63 연결 사슬의 더 큰 스페이서 사이에 작은 스페이서를 가지면서 이를 이용한다.폴리유비퀴틴 사슬의 인식에 관여하는 기계는 또한 K63 연결 사슬과 M1 연결 사슬을 구별할 수 있으며, K63 연결 사슬과 M1 연결 사슬은 [8][10][70]기질의 프로테아솜 분해를 유도할 수 있다는 사실에 의해 증명된다.
기능.
유비쿼티네이션 시스템은 다음을 [73]포함한 다양한 셀룰러 프로세스에서 기능합니다.
- 항원 처리
- 아포토시스
- 세포소기관생성
- 세포주기와 분열
- DNA전사수복
- 차별화 및 개발
- 면역반응과 염증
- 신경 및 근육의 퇴화
- 다능성[74] 유지
- 신경망의 형태 형성
- 세포표면수용체, 이온채널 및 분비경로
- 스트레스 및 세포외 조절제에 대한 반응
- 리보솜생성
- 바이러스 감염
막단백질
다단일화(Multi-monoubiquitation)는 막에서 제거(내화)하기 위해 막 통과 단백질(예: 수용체)을 표시하고 세포 내에서 여러 가지 신호 전달 역할을 수행할 수 있다.세포 표면의 막 통과 분자가 유비퀴틴으로 태그될 때, 단백질의 세포 내 국소화는 변화하고, 종종 리소좀에서 파괴되는 단백질을 목표로 삼는다.이것은 종종 리간드에 의한 수용체의 자극이 유비퀴티네이션과 내부화의 속도를 증가시키기 때문에 부정적인 피드백 메커니즘으로 작용한다.모노비퀴티네이션과 마찬가지로 리신63-결합 폴리유비퀴틴 사슬도 일부 막단백질을 [10][62][65][75]운반하는 역할을 한다.
게놈 유지 보수
증식세포핵항원(PCNA)은 DNA 합성에 관여하는 단백질이다.정상적인 생리 조건 하에서 PCNA는 수모일화된다(유비퀴티네이션과 유사한 번역 후 변형).DNA가 자외선이나 화학물질에 의해 손상되면 리신 잔기에 부착된 SMO 분자가 유비퀴틴으로 대체된다.모노비퀴티드 PCNA는 손상된 DNA로 DNA 합성을 수행할 수 있는 중합효소를 모집하지만, 이것은 매우 오류가 발생하기 쉬우며, 아마도 돌연변이 DNA 합성을 야기할 수 있다.PCNA의 리신63 연결 폴리유비퀴티네이션은 템플릿 전환 경로에 [6][76][77]의해 알려진 오류 발생 가능성이 낮은 돌연변이 바이패스를 실행할 수 있게 한다.
히스톤 H2AX의 유비퀴티화는 DNA 이중 가닥 파괴의 DNA 손상 인식에 관여한다.E2/E3 리가아제 쌍 Ubc13-Mms2/RNF168에 [78][79]의해 H2AX 히스톤 상에 리신63결합 폴리유비퀴틴 사슬을 형성한다.이 K63 체인은 UIM을 포함한 RAP80을 채용하고 RAP80은 BRCA1의 현지화를 지원합니다.이 경로는 결국 상동 재조합 [80]수복을 위해 필요한 단백질을 모집할 것이다.
전사 규제
히스톤은 어디에나 있을 수 있고 이것은 보통 모노비퀴티네이션의 형태이다(다중유비퀴티네이션 형태는 발생하지만).히스톤 유비퀴티네이션은 염색질 구조를 변화시키고 전사에 관여하는 효소의 접근을 가능하게 한다.히스톤의 유비퀴틴은 또한 전사를 활성화하거나 억제하는 단백질의 결합 부위로 작용하고 또한 단백질의 번역 후 추가적인 수정을 유도할 수 있다.이러한 효과는 모두 유전자의 [81][82]전사를 조절할 수 있다.
탈유비퀴티네이션
탈유비퀴타이제 효소(DUB)는 기질 단백질에서 유비퀴틴을 제거함으로써 유비퀴타이제이션의 역할에 반대한다.그것들은 두 단백질 사이의 아미드 결합을 분해하는 시스테인 단백질 분해효소이다.이들은 매우 특이하며, E3 결합 효소와 마찬가지로 효소당 몇 개의 기질만 가지고 유비퀴틴을 결합합니다.이들은 이소펩타이드(유비퀴틴과 리신 사이)와 펩타이드 결합(유비퀴틴과 N-말단 사이)을 모두 분해할 수 있다.기질 단백질에서 유비퀴틴을 제거하는 것 외에도, DUB는 세포 내에서 다른 많은 역할을 한다.유비퀴틴은 사슬(폴리유비퀴틴)에 결합된 여러 복사본으로 발현되거나 리보솜 서브유닛에 부착된다.DUB는 이러한 단백질을 분해하여 활성 유비퀴틴을 생성한다.그들은 또한 유비퀴티네이션 과정 동안 작은 친핵성 분자에 결합되었던 유비퀴틴을 재활용한다.모노비퀴틴은 [83][84]단백질에서 이전에 제거된 유리 폴리유비퀴틴 사슬에서 유비퀴틴을 분리하는 DUB에 의해 형성된다.
유비퀴틴 결합 도메인
| 도메인 | 단백질 수 프로테옴에서 | 길이 (아연산) | 유비퀴틴 결합 어피니 |
|---|---|---|---|
| 큐 | 세레비시아에: 7 H. 사피엔스: 21 | 42–43 | 최대 2 ~ 160 μM |
| 가타리 | 세레비시아에: 2 H. 사피엔스: 14 | 135 | 최대 180μM |
| 딱풀 | 세레비시아에: 사피엔스: | ~135 | 최대 460μM |
| NZF | 세레비시아에: 1 H. 사피엔스: 25 | ~35 | 최대 100 ~ 400 μM |
| PAZ | 세레비시아에: 5 H. 사피엔스: 16 | ~58 | 알 수 |
| UBA | 세레비시아에: 10 H. 사피엔스: 98 | 45–55 | 0.03 ~ 500 μM |
| UEV | 세레비시아에: 2 사피엔스: | ~145 | 최대 100 ~ 500 μM |
| UIM | 세레비시아에: 8 H. 사피엔스: 71 | ~20 | 최대 100 ~ 400 μM |
| VHS | 세레비시아에: 4 H. 사피엔스: 28 | 150 | 알 수 |
유비퀴틴 결합 도메인(UBD)은 유비퀴틴과 비공유적으로 결합하는 모듈러 단백질 도메인이며, 이러한 모티브는 다양한 세포 이벤트를 제어합니다.상세한 분자 구조는 다수의 UBD로 알려져 있으며 결합 특이성은 작용과 조절의 메커니즘과 세포 단백질과 [85][86]과정을 어떻게 조절하는지를 결정합니다.
질병 관련성
병인 발생
유비퀴틴 경로는 다음을 [87]포함한 광범위한 질병 및 장애의 병인과 관련이 있다.
신경 퇴화
유비퀴틴은 알츠하이머병, 운동뉴론병,[88] 헌팅턴병, 파킨슨병 [87]등 단백질 고정장애와 관련된 신경변성 질환에 관여한다.유비퀴린-1의 다른 동질 형태를 코드하는 전사 변이체는 알츠하이머병 [89]및 파킨슨병과 관련된 병변에서 발견된다.뇌에서 높은 수준의 유비퀴린은 알츠하이머병을 [90]유발하는 데 중요한 역할을 하는 아밀로이드 전구 단백질의 기형을 감소시키는 것으로 나타났다.반대로, 뇌에서 유비퀴린-1의 낮은 [90]수치는 APP의 기형의 증가와 관련이 있다.유비퀴틴 B의 프레임시프트 돌연변이는 C 말단 글리신이 결핍된 잘린 펩타이드를 초래할 수 있다.UBB+1로 알려진 이 비정상적인 펩타이드는 알츠하이머병과 다른 타우병에서 선택적으로 축적되는 것으로 나타났다.
감염 및 면역
유비퀴틴과 유비퀴틴 유사 분자는 정상 상태 억제, 감염 중 활성화, 클리어런스 시 감쇠를 포함한 거의 모든 단계에서 면역 신호 전달 경로를 광범위하게 조절합니다.이 조절이 없다면, 병원균에 대한 면역 활성화가 결함이 되어 만성 질환이나 사망을 초래할 수 있습니다.또는 면역계가 과활성화되어 장기 및 조직이 자가면역 손상을 입을 수 있다.
반면 바이러스는 효과적으로 복제하기 위해 면역력을 포함한 숙주 세포 과정을 차단하거나 수정해야 하지만, 질병과 관련된 많은 바이러스들은 정보적으로 제한된 게놈을 가지고 있다.세포 내에서의 역할이 매우 많기 때문에 유비퀴틴 시스템을 조작하는 것은 그러한 바이러스가 자신의 [91]복제를 지원하기 위해 중요한 숙주 세포 프로세스를 차단, 전복 또는 리다이렉트하는 효율적인 방법입니다.
레티노인산유도유전자I(RIG-I) 단백질은 인간 [92]세포의 바이러스 및 기타 침습적 RNA에 대한 1차 면역체계 센서이다.RIG-I-like receptor(LR) 면역 [93]신호 경로는 면역 조절에서 유비퀴틴의 역할 측면에서 가장 광범위하게 연구되는 것 중 하나이다.
유전적 장애
- 엔젤만 증후군은 E6-AP라고 불리는 유비퀴틴 리가아제(E3) 효소를 코드하는 UBE3A의 파괴에 의해 발생한다.
- Von Hippel-Lindau 증후군은 VHL 종양 억제제 또는 VHL 유전자로 불리는 유비퀴틴 E3 연결 효소의 분해를 수반합니다.
- 판코니 빈혈:이 질병을 일으킬 수 있는 13개의 확인된 유전자 중 8개는 큰 유비퀴틴 연결효소(E3) 복합체를 형성하는 단백질을 암호화한다.
- 3-M 증후군은 Cullin7 E3 유비퀴틴 연결효소의 [94]돌연변이와 관련된 상염색체 역성장 지연 장애이다.
진단 용도
유비퀴틴에 대한 항체를 이용한 면역 조직 화학은 이 단백질의 비정상적인 축적을 확인할 수 있으며, 이는 질병 과정을 나타낸다.이러한 단백질 축적은 포접체(세포에서 현미경으로 볼 수 있는 비정상적인 물질의 집합의 총칭)라고 불립니다.예를 들어 다음과 같습니다.
암과의 관련성
단백질의 번역 후 수정은 진핵 세포 [95]신호 전달에서 일반적으로 사용되는 메커니즘이다.유비퀴티네이션 또는 단백질에 대한 유비퀴틴 결합은 세포 주기 진행과 세포 증식과 발달을 위한 중요한 과정이다.유비쿼티네이션은 보통 26S 프로테아솜을 통해 단백질 분해의 신호로 작용하지만, 또한 엔도시스,[96] 효소 활성화[97] 및 DNA [98]복구와 같은 다른 기본적인 세포 과정에도 [95]작용할 수 있다.또한, 유비쿼티네이션은 사이클린의 세포 수준을 엄격하게 조절하는 기능을 하기 때문에, 그 잘못된 조절은 심각한 영향을 미칠 것으로 예상된다.발암 과정에서 유비퀴틴/단백질 경로의 중요성에 대한 첫 번째 증거는 프로테아솜 억제제의 [99][100][101]높은 항종양 활성으로 인해 관찰되었다.다양한 연구에 따르면 유비쿼티네이션 과정의 결함이나 변화는 일반적으로 인간 [102][103][104][105][106][107][108][109]암종과 관련이 있거나 인간 암종에서 나타난다.악성종양은 종양억제유전자에서 직접 기능 돌연변이, 유비퀴티네이션의 활성 증가 및/또는 [110]관련 단백질의 돌연변이에 의한 유비퀴티네이션의 간접 감쇠를 통해 발병할 수 있다.
E3 유비퀴틴 리가아제의 직접적인 기능상실 돌연변이
신장세포암
VHL(Von Hippel-Lindau) 유전자는 E3 유비퀴틴 리가제 성분을 암호화한다.VHL 복합체는 정상독성 조건에서 산소 의존성 파괴 영역과 상호작용함으로써 저산소 유도 전사 인자 패밀리(HIF)의 구성원을 분해 대상으로 한다.HIF는 혈관내피성장인자(VEGF)와 같은 다운스트림 대상을 활성화하여 혈관신생을 촉진합니다.VHL의 돌연변이는 HIF의 저하를 방지하여 고혈관 병변과 신장 [102][110]종양의 형성을 초래한다.
유방암
BRCA1 유전자는 DNA 손상에 반응하는 BRCA1 단백질을 암호화하는 인간의 또 다른 종양 억제 유전자이다.단백질은 E3 유비퀴틴 리가아제 활성의 RING 모티브를 포함하고 있습니다.BRCA1은 유비쿼티네이션 활성을 위해 BARD1 및 BAP1과 같은 다른 분자와 이합체를 형성할 수 있다.연결효소 기능에 영향을 미치는 돌연변이는 종종 발견되고 다양한 [106][110]암과 관련이 있다.
사이클린 E
세포주기 진행 과정은 세포 성장과 분화를 위한 가장 기본적인 과정이며 인간 암종에서 가장 흔하게 변화하기 때문에 세포주기 조절 단백질은 엄격한 규제를 받을 것으로 예상된다.이름에서 알 수 있듯이 사이클린의 수치는 세포 주기 동안 특정 시점에서만 높습니다.이는 유비쿼티네이션 및 열화를 통해 사이클린 또는 CDKs 수준을 지속적으로 제어함으로써 달성됩니다.사이클린 E가 CDK2와 제휴하여 인산화되면 SCF 관련 F박스 단백질 Fbw7이 복합체를 인식하여 분해 대상으로 한다.Fbw7의 돌연변이는 인간 종양의 30% 이상에서 발견되어 종양 억제 [109]단백질로 특징지어진다.
유비쿼티네이션 활동 증가
자궁경부암
인간유두종바이러스(HPV)의 발암성 유형은 바이러스 감염과 복제를 위해 세포 유비퀴틴-단백질 경로를 납치하는 것으로 알려져 있다.HPV의 E6 단백질은 세포 E6-AP E3 유비퀴틴 연결효소의 N 말단에 결합하며, 복합체는 많은 종류의 [104]암에서 불활성화가 발견되는 잘 알려진 종양 억제 유전자인 p53과 결합하도록 수정된다.따라서 p53은 유비쿼티네이션과 프로테아솜 매개 분해를 거친다.한편 초기 발현된 HPV 유전자 중 하나인 E7은 종양 억제 유전자인 Rb와 결합해 분해를 [110]매개한다.세포에서 p53과 Rb의 상실은 세포 증식을 무한히 가능하게 한다.
p53 규제
유전자 증폭은 p53 활성의 하향 조절을 담당하는 RING E3 유비퀴틴 연결효소를 코드하는 MDM2를 포함한 다양한 종양 사례에서 종종 발생한다.MDM2는 p53을 유비쿼티네이션 및 프로테아좀 분해를 목표로 하여 정상적인 세포 상태에 적절한 수준을 유지한다.MDM2가 과도하게 발현되면 p53 액티비티가 손실되므로 셀이 무한한 복제 [105][110]가능성을 가질 수 있습니다.
p27
유전자 증폭의 표적이 되는 또 다른 유전자는 SKP2이다. SKP2는 유비쿼티네이션과 분해의 기질 인식에 역할을 하는 F박스 단백질이다.SKP2는 cyclin-dependent kinase(CDK; 사이클린 의존성 키나아제)의 억제제인 p27을Kip-1 표적으로 한다.CDKs2/4는 각각 사이클린 E/D 계열의 세포 주기 조절기와 결합하여 G1 단계를 통해 세포 주기 진행을 제어한다.p27Kip-1 단백질의 낮은 수치는 다양한 암에서 종종 발견되며, SKP2의 [107][110]과발현을 통한 유비퀴틴 매개 단백질 분해의 과활성화에 기인한다.
Efp
Efp, 즉 에스트로겐을 섭취할 수 없는 링핑거 단백질은 E3 유비퀴틴 연결효소이며 과발현이 에스트로겐 비의존성 유방암의 [101][111]주요 원인인 것으로 나타났다.Efp의 기질은 세포주기를 음으로 조절하는 14-3-3 단백질이다.
유비쿼티네이션 회피
대장암
대장암과 관련된 유전자는 전형적인 종양 억제 유전자인 선종성 용종성 대장균이다.APC 유전자 생성물은 N 말단에서 유비퀴티네이션에 의해 분해되는 베타카테닌을 목표로 하여 세포 수준을 조절한다.대부분의 대장암 환자는 APC 유전자의 돌연변이와 함께 발견됩니다.그러나 APC 유전자가 변이되지 않은 경우 베타카테닌의 N말단에서 돌연변이가 발견되어 유비쿼티네이션이 없어지고 [103][110]활성이 증가한다.
교아세포종
뇌에서 발생한 가장 공격적인 암으로서 교아종 환자에게서 발견되는 돌연변이는 표피성장인자수용체(EGFR)의 세포외 도메인 일부의 결실과 관련이 있다.이러한 결실은 CBL E3 연결효소가 유비퀴틴-리소좀 경로를 통해 수용체를 재활용 및 분해하기 위해 결합할 수 없게 한다.따라서 EGFR은 세포막에서 구성적으로 활성화되며 세포 증식과 [108]이동에 관여하는 하류 효과인자를 활성화한다.
인산화 의존성 유비퀴티네이션
유비퀴티네이션과 인산화 사이의 상호작용은 인산화가 종종 유비퀴티네이션이 [95]분해로 이어지는 지표 역할을 하기 때문에 지속적인 연구 관심사였다.또한 유비퀴티네이션은 단백질의 [112]키나아제 활성을 온/오프하는 작용도 할 수 있다.인산화의 중요한 역할은 Cbl [113]단백질의 자가억제 활성화 및 제거에서 크게 강조된다.Cbl은 RING 핑거 도메인이 있는 E3 유비퀴틴 연결효소이며, 티로신 키나제 결합(TKB) 도메인과 상호작용하여 E2 유비퀴틴 결합 효소와 RING 도메인의 상호작용을 방지한다.이 분자내 상호작용은 다양한 성장인자와 티로신인산화효소 시그널링 및 T세포 [113]활성화의 음의 조절기로서의 역할을 막는 자가억제 조절이다.Y363의 인산화는 자가억제를 완화시키고 E2에 [113]대한 결합을 강화한다.리게아제/종양억제제 기능의 상실과 양성 시그널링/온코제닉 기능의 유지로 인해 Cbl 단백질을 기능장애로 만드는 돌연변이는 [114][115]암 발생을 유발하는 것으로 나타났다.
약물 표적으로서
유비퀴틴리가아제 기질 선별
E3-기질 상호작용의 규제 완화가 많은 경우 주요 원인으로 작용하기 때문에 E3 리가아제 기질의 식별은 인간 질병에서의 그 의미를 이해하는 데 매우 중요하다.E3 유비퀴틴 리가아제의 기질 식별에 사용되는 메커니즘의 한계를 극복하기 위해 2008년 '[116]글로벌 단백질 안정성(GPS) 프로파일링'이라는 시스템이 개발되었습니다.이 높은 처리량 시스템은 수천 개의 잠재적 기질과 독립적으로 융합된 리포터 단백질을 사용했다.리게아제 활성을 억제함으로써(Cul1 우성 음성을 생성함으로써 유비퀴티네이션이 발생하지 않도록 함) 리포터 활성이 증가하면 식별된 기질이 축적되고 있음을 알 수 있다.이 접근방식은 E3 리가아제 기질 목록에 많은 새로운 기질을 추가하였다.
가능한 치료 응용 프로그램
E3 연결효소(예: 보르테조미브)[111]에 의한 특정 기질 인식 차단.
도전
특정 E3 리가아제 및/또는 질병과 관련된 단백질-단백질 상호작용의 활성을 선택적으로 억제하는 특정 분자를 찾는 것은 중요하고 확대되는 연구 영역 중 하나로 남아 있다.게다가 유비쿼티네이션은 다양한 참여자와 중간 형태를 가진 다단계 과정이기 때문에, 소분자 [101]억제제를 설계할 때 성분들 간의 매우 복잡한 상호작용에 대한 고려가 크게 필요하다.
유사단백질
유비퀴틴은 가장 잘 알려진 번역 후 수식제이지만, 유비퀴틴과 평행하지만 구별되는 경로에서 세포 표적을 수정하는 유비퀴틴 유사 단백질(UBLs) 패밀리가 증가하고 있다.알려진 UBL은 다음과 같다: 작은 유비퀴틴 유사 수식제(SUMO), 유비퀴틴 교차반응 단백질(UCRP, 인터페론 자극 유전자-15 ISG15라고도 알려져 있음), 유비퀴틴 관련 수식제 1(URM1), 신경-프리처 세포 발현 저하 단백질 8NED라고도 불린다., autophagy-8(ATG8) 및 -12(ATG12), 소수의 유비퀴틴 유사단백질(FUB1), MUB(멤브레인 앵커링 [117]UBL), 유비퀴틴 접이식자-1(UFM1) 및 유비퀴틴 유사단백질-5(UBL5), 유비퀴티틴 유사단백질(UBL5), 유비퀴틴은 유비퀴틴으로 알려져 있지만 유비퀴틴은 유비퀴틴으로 알려져 있다.이러한 단백질은 유비퀴틴과 약간의 1차 배열 동일성만 공유하지만, 3차원적으로 밀접하게 관련되어 있다.예를 들어, SMO는 18%의 시퀀스 식별성만을 공유하지만 동일한 구조 접힘을 포함한다.이 주름을 "유비퀴틴 접힘"이라고 합니다.FAT10과 UCRP에는 2개가 있습니다.이 콤팩트한 구상 베타 그라프 접힘은 유비퀴틴, UBL 및 유비퀴틴 유사 도메인을 구성하는 단백질, 예를 들어 S. cerevisae 스핀들 극체 복제 단백질, Dsk2, NER 단백질 Rad23에서 발견되며 모두 N-말단 유비퀴틴 도메인을 포함한다.
이러한 관련 분자들은 새로운 기능을 가지고 있고 다양한 생물학적 과정에 영향을 미친다.일부 단백질은 둘 이상의 UBL에 의해 변형될 수 있고, 때로는 동일한 리신 잔기로도 변형될 수 있기 때문에 다양한 결합 경로 사이에 교차 조절이 있다.예를 들어 SMO 수식은 유비퀴티네이션과 대립하여 단백질 기질을 안정시키는 역할을 하는 경우가 많다.UBL에 결합된 단백질은 일반적으로 프로테아솜에 의해 분해되는 것이 아니라 다양한 조절 활동에서 기능한다.UBL의 부착은 기질 배열을 변화시키고, 리간드 또는 다른 상호작용 분자에 대한 친화력에 영향을 미치며, 기질 국재화를 변화시키고, 단백질 안정성에 영향을 미칠 수 있다.
UBL은 구조적으로 유비퀴틴과 유사하며, 유비퀴틴의 상응하는 메커니즘과 유사한 효소적 단계에 의해 처리, 활성화, 결합 및 결합에서 방출된다.UBL은 불변 C단말기 LRG를 공개하기 위해 처리되는 C단말기 확장으로도 변환됩니다.이러한 수식어들은 세포 내 표적에 UBL을 결합시키는 그들만의 특정한 E1(활성화), E2(결합) 및 E3(결합) 효소를 가지고 있다.이러한 결합체는 탈유화효소와 [73]유사한 메커니즘을 가진 UBL 특이적 이소펩티다아제에 의해 역전될 수 있다.
일부 종에서는 유비퀴틴과 관련된 메커니즘을 통해 정자 미토콘드리아를 인식하고 파괴하는 [120]것이 수정 후 정자 미토콘드리아를 폐기하는 역할을 한다.
원핵생물 기원
유비퀴틴은 ThiS(O32583)[121] 또는 MoaD(P30748)[122]와 유사한 박테리아 단백질에서 유래한 것으로 여겨진다.이러한 원핵 단백질은 배열 동일성이 거의 없음에도 불구하고 동일한 단백질 접힘을 공유합니다.이 단백질들은 또한 유비퀴틴과 유황 화학을 공유한다.몰리브도프테린 생합성에 관여하는 MoaD는 MoaD를 위한 E1 유비퀴틴 활성화 효소처럼 작용하는 MoeB와 상호작용하여 이러한 원핵 단백질과 유비퀴틴 시스템 사이의 연결을 강화합니다.E1 유사 효소 TiF를 가진 TiS에도 유사한 시스템이 존재한다.또 유비퀴틴 관련 수식어인 사카로미세스 세레비시아이 단백질 Urm1은 원핵유비퀴틴 [123]유사 분자 및 유비퀴틴과의 진화적 관계를 연결하는 분자화석인 것으로 추정된다.
고세균은 기능적으로 유비퀴틴 수식계의 보다 가까운 상동성을 가지며, 여기서 SAMPs(소형 고세균 수식 단백질)에 의한 "삼필화"가 수행됩니다.삼필화 시스템은 단백질을 프로테오솜으로 [124]유도하기 위해 E1만을 사용한다.진핵생물의 조상과 관련된 프로테오아르카이오타는 모든 E1, E2, E3 효소와 조절된 Rpn11 시스템을 가지고 있다.TiS 또는 MoaD와 더 유사한 SAMP와 달리, 프로테오아르카이오타 유비퀴틴은 진핵생물 호몰로그와 [125]가장 유사하다.
원핵유비퀴틴유사단백질(Pup)과 유비퀴틴세균(UBACT)
원핵유비퀴틴 유사단백질(Pup)은 그램 양성 세균문 방선균에서 발견된 유비퀴틴의 기능적 유사체이다.유비퀴티네이션과 번데기의 효소학은 다르지만, 두 과는 호몰로지를 공유하지 않지만, 동일한 기능(분해를 위한 단백질 표적화)을 한다.유비퀴티네이션의 3단계 반응과는 대조적으로 번데기는 2단계 과정을 필요로 하므로 번데기에는 2가지 효소만 관여한다.
archaeon[126]은 팝 homologs의 시퀀스 매우 팝의 그람 양성의 박테리아에서 씬에서, 유비퀴틴 세균(UBact)이라 칭해 진 차이를 보이고 있지만 차별 아직 되phylo다고 증명되지 않고 2017년에는 팝의 homologs 그람 음성의 박테리아의 5phyla, 7후보 세균 phyla에 있고에 보고되지 않았다.사향 고양이의 일종별도의 진화적 기원에 의해 뒷받침되고 실험적인 [126]증거가 없다.
그Pup/UBact-proteasome 시스템의 그램 음성과 그램 양성 박테리아의 발견은Pup/UBact-proteasome 시스템은 박테리아의 전에 분열 그램도 긍정적이고 부정적인 clades에 3000만년 전 or,[127]은 이러한 시스템은 다른 세균 종에 의해 수평 그는 받았습니다 진화했음을 암시한다.trne아직 알려지지 않은 제3의 유기체로부터 응답한다.두 번째 가능성을 뒷받침하기 위해, 두 개의 UBact 궤적이 배양되지 않은 혐기성 메타노 영양성 시조체(ANME-1;locus CBH38808.1 및 궤적 CBH39258.1)의 게놈에서 발견되었다.
유비퀴틴 도메인을 포함하는 인간 단백질
이것들은 유비퀴틴 유사 단백질을 포함한다.
ANUBL1; BAG1; BAT3/BAG6; C1orf131; DDI1; DDI2; FAU; HERPUD1; HERPUD2; 홉스; IKBKB; ISG15; LOC391257; MIDN; NED8AS;UBFD1, UBL4A, UBL4B, UBL7, UBLCP1, UBQLN1, UBQLN2, UBQLN3, UBQLN4, UBQLNL, UBTD1; UH
관련 단백질
유비쿼티네이션 예측
현재 사용 가능한 예측 프로그램은 다음과 같습니다.
- UbiPred는 유비퀴티네이션 [128]사이트를 예측하기 위해 31가지 물리 화학적 특성을 사용하는 SVM 기반 예측 서버입니다.
- UbPred는 단백질의 잠재적 유비쿼티네이션 부위의 무작위 숲 기반 예측자입니다.그것은 우리의 실험과 두 개의 대규모 단백질학 [129]연구에서 이용할 수 있는 266개의 비중복 실험적으로 검증된 유비쿼티네이션 사이트의 결합 세트에서 훈련되었다.
- CKSAAP_UbSite는 입력으로 쿼리 사이트(즉 쿼리 시퀀스의 모든 리신)를 둘러싼 k 간격 아미노산 쌍의 구성을 사용하는 SVM 기반 예측이며 UbPred와 [130]동일한 데이터 세트를 사용한다.
팟캐스트
- 유비퀴틴 프로테아솜 시스템을 조사하는 것이 치매 연구자 팟캐스트의 [131]초점이었다.이 팟캐스트는 2021년 8월 16일 런던 유니버시티 칼리지의 셀리나 레이 교수가 주최하여 발행되었다.
「 」를 참조해 주세요.
레퍼런스
- ^ a b Goldstein G, Scheid M, Hammerling U, Schlesinger DH, Niall HD, Boyse EA (January 1975). "Isolation of a polypeptide that has lymphocyte-differentiating properties and is probably represented universally in living cells". Proceedings of the National Academy of Sciences of the United States of America. 72 (1): 11–5. Bibcode:1975PNAS...72...11G. doi:10.1073/pnas.72.1.11. PMC 432229. PMID 1078892.
- ^ Wilkinson KD (October 2005). "The discovery of ubiquitin-dependent proteolysis". Proceedings of the National Academy of Sciences of the United States of America. 102 (43): 15280–2. Bibcode:2005PNAS..10215280W. doi:10.1073/pnas.0504842102. PMC 1266097. PMID 16230621.
- ^ a b Kimura Y, Tanaka K (June 2010). "Regulatory mechanisms involved in the control of ubiquitin homeostasis". Journal of Biochemistry. 147 (6): 793–8. doi:10.1093/jb/mvq044. PMID 20418328.
- ^ a b c Glickman MH, Ciechanover A (April 2002). "The ubiquitin-proteasome proteolytic pathway: destruction for the sake of construction". Physiological Reviews. 82 (2): 373–428. doi:10.1152/physrev.00027.2001. PMID 11917093.
- ^ a b Mukhopadhyay D, Riezman H (January 2007). "Proteasome-independent functions of ubiquitin in endocytosis and signaling". Science. 315 (5809): 201–5. Bibcode:2007Sci...315..201M. doi:10.1126/science.1127085. PMID 17218518. S2CID 35434448.
- ^ a b c Schnell JD, Hicke L (September 2003). "Non-traditional functions of ubiquitin and ubiquitin-binding proteins". The Journal of Biological Chemistry. 278 (38): 35857–60. doi:10.1074/jbc.R300018200. PMID 12860974.
- ^ a b Pickart CM, Eddins MJ (November 2004). "Ubiquitin: structures, functions, mechanisms". Biochimica et Biophysica Acta (BBA) - Molecular Cell Research. 1695 (1–3): 55–72. doi:10.1016/j.bbamcr.2004.09.019. PMID 15571809.
- ^ a b c d e Komander D, Rape M (2012). "The ubiquitin code". Annual Review of Biochemistry. 81: 203–29. doi:10.1146/annurev-biochem-060310-170328. PMID 22524316.
- ^ a b McDowell GS, Philpott A (August 2013). "Non-canonical ubiquitylation: mechanisms and consequences". The International Journal of Biochemistry & Cell Biology. 45 (8): 1833–42. doi:10.1016/j.biocel.2013.05.026. PMID 23732108.
- ^ a b c d e Miranda M, Sorkin A (June 2007). "Regulation of receptors and transporters by ubiquitination: new insights into surprisingly similar mechanisms". Molecular Interventions. 7 (3): 157–67. doi:10.1124/mi.7.3.7. PMID 17609522.
- ^ a b "The Nobel Prize in Chemistry 2004". Nobelprize.org. Retrieved 2010-10-16.
- ^ "The Nobel Prize in Chemistry 2004: Popular Information". Nobelprize.org. Retrieved 2013-12-14.
- ^ Ciechanover A, Hod Y, Hershko A (August 2012). "A heat-stable polypeptide component of an ATP-dependent proteolytic system from reticulocytes. 1978". Biochemical and Biophysical Research Communications. 425 (3): 565–70. doi:10.1016/j.bbrc.2012.08.025. PMID 22925675.
- ^ a b Pickart CM (2001). "Mechanisms underlying ubiquitylation". Annual Review of Biochemistry. 70: 503–33. doi:10.1146/annurev.biochem.70.1.503. PMID 11395416.
- ^ Marotti LA, Newitt R, Wang Y, Aebersold R, Dohlman HG (April 2002). "Direct identification of a G protein ubiquitylation site by mass spectrometry". Biochemistry. 41 (16): 5067–74. doi:10.1021/bi015940q. PMID 11955054.
- ^ a b Peng J, Schwartz D, Elias JE, Thoreen CC, Cheng D, Marsischky G, Roelofs J, Finley D, Gygi SP (August 2003). "A proteomics approach to understanding protein ubiquitylation". Nature Biotechnology. 21 (8): 921–6. doi:10.1038/nbt849. PMID 12872131. S2CID 11992443.
- ^ Breitschopf K, Bengal E, Ziv T, Admon A, Ciechanover A (October 1998). "A novel site for ubiquitylation: the N-terminal residue, and not internal lysines of MyoD, is essential for conjugation and degradation of the protein". The EMBO Journal. 17 (20): 5964–73. doi:10.1093/emboj/17.20.5964. PMC 1170923. PMID 9774340.
- ^ Bloom J, Amador V, Bartolini F, DeMartino G, Pagano M (October 2003). "Proteasome-mediated degradation of p21 via N-terminal ubiquitinylation". Cell. 115 (1): 71–82. doi:10.1016/S0092-8674(03)00755-4. PMID 14532004. S2CID 15114828.
- ^ Scaglione KM, Basrur V, Ashraf NS, Konen JR, Elenitoba-Johnson KS, Todi SV, Paulson HL (June 2013). "The ubiquitin-conjugating enzyme (E2) Ube2w ubiquitinates the N terminus of substrates". The Journal of Biological Chemistry. 288 (26): 18784–8. doi:10.1074/jbc.C113.477596. PMC 3696654. PMID 23696636.
- ^ Sadeh R, Breitschopf K, Bercovich B, Zoabi M, Kravtsova-Ivantsiv Y, Kornitzer D, Schwartz A, Ciechanover A (October 2008). "The N-terminal domain of MyoD is necessary and sufficient for its nuclear localization-dependent degradation by the ubiquitin system". Proceedings of the National Academy of Sciences of the United States of America. 105 (41): 15690–5. Bibcode:2008PNAS..10515690S. doi:10.1073/pnas.0808373105. PMC 2560994. PMID 18836078.
- ^ Coulombe P, Rodier G, Bonneil E, Thibault P, Meloche S (July 2004). "N-Terminal ubiquitylation of extracellular signal-regulated kinase 3 and p21 directs their degradation by the proteasome". Molecular and Cellular Biology. 24 (14): 6140–50. doi:10.1128/MCB.24.14.6140-6150.2004. PMC 434260. PMID 15226418.
- ^ Kuo ML, den Besten W, Bertwistle D, Roussel MF, Sherr CJ (August 2004). "N-terminal polyubiquitylation and degradation of the Arf tumor suppressor". Genes & Development. 18 (15): 1862–74. doi:10.1101/gad.1213904. PMC 517406. PMID 15289458.
- ^ Ben-Saadon R, Fajerman I, Ziv T, Hellman U, Schwartz AL, Ciechanover A (October 2004). "The tumor suppressor protein p16(INK4a) and the human papillomavirus oncoprotein-58 E7 are naturally occurring lysine-less proteins that are degraded by the ubiquitin system. Direct evidence for ubiquitylation at the N-terminal residue". The Journal of Biological Chemistry. 279 (40): 41414–21. doi:10.1074/jbc.M407201200. PMID 15254040.
- ^ Li H, Okamoto K, Peart MJ, Prives C (February 2009). "Lysine-independent turnover of cyclin G1 can be stabilized by B'alpha subunits of protein phosphatase 2A". Molecular and Cellular Biology. 29 (3): 919–28. doi:10.1128/MCB.00907-08. PMC 2630686. PMID 18981217.
- ^ Reinstein E, Scheffner M, Oren M, Ciechanover A, Schwartz A (November 2000). "Degradation of the E7 human papillomavirus oncoprotein by the ubiquitin-proteasome system: targeting via ubiquitylation of the N-terminal residue". Oncogene. 19 (51): 5944–50. doi:10.1038/sj.onc.1203989. PMID 11127826.
- ^ Aviel S, Winberg G, Massucci M, Ciechanover A (August 2000). "Degradation of the epstein-barr virus latent membrane protein 1 (LMP1) by the ubiquitin-proteasome pathway. Targeting via ubiquitylation of the N-terminal residue". The Journal of Biological Chemistry. 275 (31): 23491–9. doi:10.1074/jbc.M002052200. PMID 10807912.
- ^ Ikeda M, Ikeda A, Longnecker R (August 2002). "Lysine-independent ubiquitylation of Epstein-Barr virus LMP2A". Virology. 300 (1): 153–9. doi:10.1006/viro.2002.1562. PMID 12202215.
- ^ Yang J, Hong Y, Wang W, Wu W, Chi Y, Zong H, Kong X, Wei Y, Yun X, Cheng C, Chen K, Gu J (May 2009). "HSP70 protects BCL2L12 and BCL2L12A from N-terminal ubiquitylation-mediated proteasomal degradation". FEBS Letters. 583 (9): 1409–14. doi:10.1016/j.febslet.2009.04.011. PMID 19376117. S2CID 32330510.
- ^ Wang Y, Shao Q, Yu X, Kong W, Hildreth JE, Liu B (May 2011). "N-terminal hemagglutinin tag renders lysine-deficient APOBEC3G resistant to HIV-1 Vif-induced degradation by reduced polyubiquitylation". Journal of Virology. 85 (9): 4510–9. doi:10.1128/JVI.01925-10. PMC 3126286. PMID 21345952.
- ^ Trausch-Azar JS, Lingbeck J, Ciechanover A, Schwartz AL (July 2004). "Ubiquitin-Proteasome-mediated degradation of Id1 is modulated by MyoD". The Journal of Biological Chemistry. 279 (31): 32614–9. doi:10.1074/jbc.M403794200. PMID 15163661.
- ^ Trausch-Azar J, Leone TC, Kelly DP, Schwartz AL (December 2010). "Ubiquitin proteasome-dependent degradation of the transcriptional coactivator PGC-1{alpha} via the N-terminal pathway". The Journal of Biological Chemistry. 285 (51): 40192–200. doi:10.1074/jbc.M110.131615. PMC 3001001. PMID 20713359.
- ^ Fajerman I, Schwartz AL, Ciechanover A (February 2004). "Degradation of the Id2 developmental regulator: targeting via N-terminal ubiquitylation". Biochemical and Biophysical Research Communications. 314 (2): 505–12. doi:10.1016/j.bbrc.2003.12.116. PMID 14733935.
- ^ a b c Vosper JM, McDowell GS, Hindley CJ, Fiore-Heriche CS, Kucerova R, Horan I, Philpott A (June 2009). "Ubiquitylation on canonical and non-canonical sites targets the transcription factor neurogenin for ubiquitin-mediated proteolysis". The Journal of Biological Chemistry. 284 (23): 15458–68. doi:10.1074/jbc.M809366200. PMC 2708843. PMID 19336407.
- ^ a b c McDowell GS, Kucerova R, Philpott A (October 2010). "Non-canonical ubiquitylation of the proneural protein Ngn2 occurs in both Xenopus embryos and mammalian cells". Biochemical and Biophysical Research Communications. 400 (4): 655–60. doi:10.1016/j.bbrc.2010.08.122. PMID 20807509.
- ^ Tatham MH, Plechanovová A, Jaffray EG, Salmen H, Hay RT (July 2013). "Ube2W conjugates ubiquitin to α-amino groups of protein N-termini". The Biochemical Journal. 453 (1): 137–45. doi:10.1042/BJ20130244. PMC 3778709. PMID 23560854.
- ^ Vittal V, Shi L, Wenzel DM, Scaglione KM, Duncan ED, Basrur V, Elenitoba-Johnson KS, Baker D, Paulson HL, Brzovic PS, Klevit RE (January 2015). "Intrinsic disorder drives N-terminal ubiquitylation by Ube2w". Nature Chemical Biology. 11 (1): 83–9. doi:10.1038/nchembio.1700. PMC 4270946. PMID 25436519.
- ^ Johnson ES, Ma PC, Ota IM, Varshavsky A (July 1995). "A proteolytic pathway that recognizes ubiquitin as a degradation signal". The Journal of Biological Chemistry. 270 (29): 17442–56. doi:10.1074/jbc.270.29.17442. PMID 7615550.
- ^ a b Kirisako T, Kamei K, Murata S, Kato M, Fukumoto H, Kanie M, Sano S, Tokunaga F, Tanaka K, Iwai K (October 2006). "A ubiquitin ligase complex assembles linear polyubiquitin chains". The EMBO Journal. 25 (20): 4877–87. doi:10.1038/sj.emboj.7601360. PMC 1618115. PMID 17006537.
- ^ a b Wang X, Herr RA, Chua WJ, Lybarger L, Wiertz EJ, Hansen TH (May 2007). "Ubiquitination of serine, threonine, or lysine residues on the cytoplasmic tail can induce ERAD of MHC-I by viral E3 ligase mK3". The Journal of Cell Biology. 177 (4): 613–24. doi:10.1083/jcb.200611063. PMC 2064207. PMID 17502423.
- ^ Cadwell K, Coscoy L (July 2005). "Ubiquitination on nonlysine residues by a viral E3 ubiquitin ligase". Science. 309 (5731): 127–30. Bibcode:2005Sci...309..127C. doi:10.1126/science.1110340. PMID 15994556.
- ^ Cadwell K, Coscoy L (April 2008). "The specificities of Kaposi's sarcoma-associated herpesvirus-encoded E3 ubiquitin ligases are determined by the positions of lysine or cysteine residues within the intracytoplasmic domains of their targets". Journal of Virology. 82 (8): 4184–9. doi:10.1128/JVI.02264-07. PMC 2293015. PMID 18272573.
- ^ Williams C, van den Berg M, Sprenger RR, Distel B (August 2007). "A conserved cysteine is essential for Pex4p-dependent ubiquitination of the peroxisomal import receptor Pex5p". The Journal of Biological Chemistry. 282 (31): 22534–43. doi:10.1074/jbc.M702038200. PMID 17550898.
- ^ Carvalho AF, Pinto MP, Grou CP, Alencastre IS, Fransen M, Sá-Miranda C, Azevedo JE (October 2007). "Ubiquitination of mammalian Pex5p, the peroxisomal import receptor". The Journal of Biological Chemistry. 282 (43): 31267–72. doi:10.1074/jbc.M706325200. PMID 17726030.
- ^ Léon S, Subramani S (March 2007). "A conserved cysteine residue of Pichia pastoris Pex20p is essential for its recycling from the peroxisome to the cytosol". The Journal of Biological Chemistry. 282 (10): 7424–30. doi:10.1074/jbc.M611627200. PMC 3682499. PMID 17209040.
- ^ a b Tait SW, de Vries E, Maas C, Keller AM, D'Santos CS, Borst J (December 2007). "Apoptosis induction by Bid requires unconventional ubiquitination and degradation of its N-terminal fragment". The Journal of Cell Biology. 179 (7): 1453–66. doi:10.1083/jcb.200707063. PMC 2373500. PMID 18166654.
- ^ a b Roark R, Itzhaki L, Philpott A (December 2012). "Complex regulation controls Neurogenin3 proteolysis". Biology Open. 1 (12): 1264–72. doi:10.1242/bio.20121750. PMC 3522888. PMID 23259061.
- ^ Magadán JG, Pérez-Victoria FJ, Sougrat R, Ye Y, Strebel K, Bonifacino JS (April 2010). "Multilayered mechanism of CD4 downregulation by HIV-1 Vpu involving distinct ER retention and ERAD targeting steps". PLOS Pathogens. 6 (4): e1000869. doi:10.1371/journal.ppat.1000869. PMC 2861688. PMID 20442859.
- ^ Tokarev AA, Munguia J, Guatelli JC (January 2011). "Serine-threonine ubiquitination mediates downregulation of BST-2/tetherin and relief of restricted virion release by HIV-1 Vpu". Journal of Virology. 85 (1): 51–63. doi:10.1128/JVI.01795-10. PMC 3014196. PMID 20980512.
- ^ Ishikura S, Weissman AM, Bonifacino JS (July 2010). "Serine residues in the cytosolic tail of the T-cell antigen receptor alpha-chain mediate ubiquitination and endoplasmic reticulum-associated degradation of the unassembled protein". The Journal of Biological Chemistry. 285 (31): 23916–24. doi:10.1074/jbc.M110.127936. PMC 2911338. PMID 20519503.
- ^ Shimizu Y, Okuda-Shimizu Y, Hendershot LM (December 2010). "Ubiquitylation of an ERAD substrate occurs on multiple types of amino acids". Molecular Cell. 40 (6): 917–26. doi:10.1016/j.molcel.2010.11.033. PMC 3031134. PMID 21172657.
- ^ Dikic I, Robertson M (March 2012). "Ubiquitin ligases and beyond". BMC Biology. 10: 22. doi:10.1186/1741-7007-10-22. PMC 3305657. PMID 22420755.
- ^ Schulman BA, Harper JW (May 2009). "Ubiquitin-like protein activation by E1 enzymes: the apex for downstream signalling pathways". Nature Reviews Molecular Cell Biology. 10 (5): 319–31. doi:10.1038/nrm2673. PMC 2712597. PMID 19352404.
- ^ Groettrup M, Pelzer C, Schmidtke G, Hofmann K (May 2008). "Activating the ubiquitin family: UBA6 challenges the field". Trends in Biochemical Sciences. 33 (5): 230–7. doi:10.1016/j.tibs.2008.01.005. PMID 18353650.
- ^ van Wijk SJ, Timmers HT (April 2010). "The family of ubiquitin-conjugating enzymes (E2s): deciding between life and death of proteins". FASEB Journal. 24 (4): 981–93. doi:10.1096/fj.09-136259. PMID 19940261. S2CID 21280193.
- ^ Metzger MB, Hristova VA, Weissman AM (February 2012). "HECT and RING finger families of E3 ubiquitin ligases at a glance". Journal of Cell Science. 125 (Pt 3): 531–7. doi:10.1242/jcs.091777. PMC 3381717. PMID 22389392.
- ^ Skaar JR, Pagano M (December 2009). "Control of cell growth by the SCF and APC/C ubiquitin ligases". Current Opinion in Cell Biology. 21 (6): 816–24. doi:10.1016/j.ceb.2009.08.004. PMC 2805079. PMID 19775879.
- ^ Kerscher O, Felberbaum R, Hochstrasser M (2006). "Modification of proteins by ubiquitin and ubiquitin-like proteins". Annual Review of Cell and Developmental Biology. 22: 159–80. doi:10.1146/annurev.cellbio.22.010605.093503. PMID 16753028.
- ^ Koegl M, Hoppe T, Schlenker S, Ulrich HD, Mayer TU, Jentsch S (March 1999). "A novel ubiquitination factor, E4, is involved in multiubiquitin chain assembly". Cell. 96 (5): 635–44. doi:10.1016/S0092-8674(00)80574-7. PMID 10089879.
- ^ Lai Z, Ferry KV, Diamond MA, Wee KE, Kim YB, Ma J, Yang T, Benfield PA, Copeland RA, Auger KR (August 2001). "Human mdm2 mediates multiple mono-ubiquitination of p53 by a mechanism requiring enzyme isomerization". The Journal of Biological Chemistry. 276 (33): 31357–67. doi:10.1074/jbc.M011517200. PMID 11397792.
- ^ Grossman SR, Deato ME, Brignone C, Chan HM, Kung AL, Tagami H, Nakatani Y, Livingston DM (April 2003). "Polyubiquitination of p53 by a ubiquitin ligase activity of p300". Science. 300 (5617): 342–4. Bibcode:2003Sci...300..342G. doi:10.1126/science.1080386. PMID 12690203. S2CID 11526100.
- ^ Shi D, Pop MS, Kulikov R, Love IM, Kung AL, Kung A, Grossman SR (September 2009). "CBP and p300 are cytoplasmic E4 polyubiquitin ligases for p53". Proceedings of the National Academy of Sciences of the United States of America. 106 (38): 16275–80. Bibcode:2009PNAS..10616275S. doi:10.1073/pnas.0904305106. PMC 2752525. PMID 19805293.
- ^ a b c d e Komander D (October 2009). "The emerging complexity of protein ubiquitination". Biochemical Society Transactions. 37 (Pt 5): 937–53. doi:10.1042/BST0370937. PMID 19754430.
- ^ a b c Ikeda F, Dikic I (June 2008). "Atypical ubiquitin chains: new molecular signals. 'Protein Modifications: Beyond the Usual Suspects' review series". EMBO Reports. 9 (6): 536–42. doi:10.1038/embor.2008.93. PMC 2427391. PMID 18516089.
- ^ Xu P, Peng J (May 2008). "Characterization of polyubiquitin chain structure by middle-down mass spectrometry". Analytical Chemistry. 80 (9): 3438–44. doi:10.1021/ac800016w. PMC 2663523. PMID 18351785.
- ^ a b Hicke L (March 2001). "Protein regulation by monoubiquitin". Nature Reviews Molecular Cell Biology. 2 (3): 195–201. doi:10.1038/35056583. PMID 11265249. S2CID 205013847.
- ^ Lecker SH, Goldberg AL, Mitch WE (July 2006). "Protein degradation by the ubiquitin-proteasome pathway in normal and disease states". Journal of the American Society of Nephrology. 17 (7): 1807–19. doi:10.1681/ASN.2006010083. PMID 16738015.
- ^ a b Kravtsova-Ivantsiv Y, Ciechanover A (February 2012). "Non-canonical ubiquitin-based signals for proteasomal degradation". Journal of Cell Science. 125 (Pt 3): 539–48. doi:10.1242/jcs.093567. PMID 22389393.
- ^ Nathan JA, Kim HT, Ting L, Gygi SP, Goldberg AL (February 2013). "Why do cellular proteins linked to K63-polyubiquitin chains not associate with proteasomes?". The EMBO Journal. 32 (4): 552–65. doi:10.1038/emboj.2012.354. PMC 3579138. PMID 23314748.
- ^ Bache KG, Raiborg C, Mehlum A, Stenmark H (April 2003). "STAM and Hrs are subunits of a multivalent ubiquitin-binding complex on early endosomes". The Journal of Biological Chemistry. 278 (14): 12513–21. doi:10.1074/jbc.M210843200. PMID 12551915.
- ^ a b Zhao S, Ulrich HD (April 2010). "Distinct consequences of posttranslational modification by linear versus K63-linked polyubiquitin chains". Proceedings of the National Academy of Sciences of the United States of America. 107 (17): 7704–9. Bibcode:2010PNAS..107.7704Z. doi:10.1073/pnas.0908764107. PMC 2867854. PMID 20385835.
- ^ Kim HT, Kim KP, Lledias F, Kisselev AF, Scaglione KM, Skowyra D, Gygi SP, Goldberg AL (June 2007). "Certain pairs of ubiquitin-conjugating enzymes (E2s) and ubiquitin-protein ligases (E3s) synthesize nondegradable forked ubiquitin chains containing all possible isopeptide linkages". The Journal of Biological Chemistry. 282 (24): 17375–86. doi:10.1074/jbc.M609659200. PMID 17426036.
- ^ Michel MA, Elliott PR, Swatek KN, Simicek M, Pruneda JN, Wagstaff JL, Freund SM, Komander D (April 2015). "Assembly and specific recognition of k29- and k33-linked polyubiquitin". Molecular Cell. 58 (1): 95–109. doi:10.1016/j.molcel.2015.01.042. PMC 4386031. PMID 25752577.
- ^ a b "Ubiquitin Proteasome Pathway Overview". Archived from the original on 2008-03-30. Retrieved 2008-04-30.
- ^ Bax, M (June 2019). "The Ubiquitin Proteasome System Is a Key Regulator of Pluripotent Stem Cell Survival and Motor Neuron Differentiation". Cells. 8 (6): 581. doi:10.3390/cells8060581. PMC 6627164. PMID 31200561.
- ^ Soni D, Wang DM, Regmi SC, Mittal M, Vogel SM, Schlüter D, Tiruppathi C (May 2018). "Deubiquitinase function of A20 maintains and repairs endothelial barrier after lung vascular injury". Cell Death Discovery. 4 (60): 60. doi:10.1038/s41420-018-0056-3. PMC 5955943. PMID 29796309.
- ^ Shaheen M, Shanmugam I, Hromas R (August 2010). "The Role of PCNA Posttranslational Modifications in Translesion Synthesis". Journal of Nucleic Acids. 2010: 1–8. doi:10.4061/2010/761217. PMC 2935186. PMID 20847899.
- ^ Jackson SP, Durocher D (March 2013). "Regulation of DNA damage responses by ubiquitin and SUMO". Molecular Cell. 49 (5): 795–807. doi:10.1016/j.molcel.2013.01.017. PMID 23416108.
- ^ Campbell SJ, Edwards RA, Leung CC, Neculai D, Hodge CD, Dhe-Paganon S, Glover JN (July 2012). "Molecular insights into the function of RING finger (RNF)-containing proteins hRNF8 and hRNF168 in Ubc13/Mms2-dependent ubiquitylation". The Journal of Biological Chemistry. 287 (28): 23900–10. doi:10.1074/jbc.M112.359653. PMC 3390666. PMID 22589545.
- ^ Ikura T, Tashiro S, Kakino A, Shima H, Jacob N, Amunugama R, Yoder K, Izumi S, Kuraoka I, Tanaka K, Kimura H, Ikura M, Nishikubo S, Ito T, Muto A, Miyagawa K, Takeda S, Fishel R, Igarashi K, Kamiya K (October 2007). "DNA damage-dependent acetylation and ubiquitination of H2AX enhances chromatin dynamics". Molecular and Cellular Biology. 27 (20): 7028–40. doi:10.1128/MCB.00579-07. PMC 2168918. PMID 17709392.
- ^ Kim H, Chen J, Yu X (May 2007). "Ubiquitin-binding protein RAP80 mediates BRCA1-dependent DNA damage response". Science. 316 (5828): 1202–5. Bibcode:2007Sci...316.1202K. doi:10.1126/science.1139621. PMID 17525342. S2CID 31636419.
- ^ Hofmann K (April 2009). "Ubiquitin-binding domains and their role in the DNA damage response". DNA Repair. 8 (4): 544–56. doi:10.1016/j.dnarep.2009.01.003. PMID 19213613.
- ^ Hammond-Martel I, Yu H, Affar el B (February 2012). "Roles of ubiquitin signaling in transcription regulation". Cellular Signalling. 24 (2): 410–21. doi:10.1016/j.cellsig.2011.10.009. PMID 22033037.
- ^ Reyes-Turcu FE, Ventii KH, Wilkinson KD (2009). "Regulation and cellular roles of ubiquitin-specific deubiquitinating enzymes". Annual Review of Biochemistry. 78: 363–97. doi:10.1146/annurev.biochem.78.082307.091526. PMC 2734102. PMID 19489724.
- ^ Nijman SM, Luna-Vargas MP, Velds A, Brummelkamp TR, Dirac AM, Sixma TK, Bernards R (December 2005). "A genomic and functional inventory of deubiquitinating enzymes". Cell. 123 (5): 773–86. doi:10.1016/j.cell.2005.11.007. hdl:1874/20959. PMID 16325574. S2CID 15575576.
- ^ a b Hicke L, Schubert HL, Hill CP (August 2005). "Ubiquitin-binding domains". Nature Reviews Molecular Cell Biology. 6 (8): 610–21. doi:10.1038/nrm1701. PMID 16064137. S2CID 3056635.
- ^ Husnjak K, Dikic I (2012-01-01). "Ubiquitin-binding proteins: decoders of ubiquitin-mediated cellular functions". Annual Review of Biochemistry. 81: 291–322. doi:10.1146/annurev-biochem-051810-094654. PMID 22482907.
- ^ a b Popovic, D (November 2014). "Ubiquitination in disease pathogenesis and treatment". Nature Medicine. 20 (11): 1242–1253. doi:10.1038/nm.3739. PMID 25375928. S2CID 205394130.
- ^ Yerbury, Justin (May 2020). "Proteome Homeostasis Dysfunction: A Unifying Principle in ALS Pathogenesis". Trends in Neurosciences. 43 (5): 274‐284. doi:10.1016/j.tins.2020.03.002. PMID 32353332. S2CID 216095994.
- ^ "UBQLN1 ubiquilin 1 [ Homo sapiens ]". Gene. National Center for Biotechnology Information. Retrieved 9 May 2012.
- ^ a b Stieren ES, El Ayadi A, Xiao Y, Siller E, Landsverk ML, Oberhauser AF, Barral JM, Boehning D (October 2011). "Ubiquilin-1 is a molecular chaperone for the amyloid precursor protein". The Journal of Biological Chemistry. 286 (41): 35689–98. doi:10.1074/jbc.M111.243147. PMC 3195644. PMID 21852239.
- "Alzheimer's brains found to have lower levels of key protein". ScienceDaily (Press release). September 1, 2011.
- ^ Heaton SM, Borg NA, Dixit VM (January 2016). "Ubiquitin in the activation and attenuation of innate antiviral immunity". The Journal of Experimental Medicine. 213 (1): 1–13. doi:10.1084/jem.20151531. PMC 4710203. PMID 26712804.
- ^ Takeuchi O, Akira S (March 2010). "Pattern recognition receptors and inflammation". Cell. 140 (6): 805–20. doi:10.1016/j.cell.2010.01.022. PMID 20303872. S2CID 223338.
- ^ Okamoto M, Kouwaki T, Fukushima Y, Oshiumi H (2018). "Regulation of RIG-I Activation by K63-Linked Polyubiquitination". Frontiers in Immunology. 8: 1942. doi:10.3389/fimmu.2017.01942. PMC 5760545. PMID 29354136.
- ^ Huber C, Dias-Santagata D, Glaser A, O'Sullivan J, Brauner R, Wu K, Xu X, Pearce K, Wang R, Uzielli ML, Dagoneau N, Chemaitilly W, Superti-Furga A, Dos Santos H, Mégarbané A, Morin G, Gillessen-Kaesbach G, Hennekam R, Van der Burgt I, Black GC, Clayton PE, Read A, Le Merrer M, Scambler PJ, Munnich A, Pan ZQ, Winter R, Cormier-Daire V (October 2005). "Identification of mutations in CUL7 in 3-M syndrome". Nature Genetics. 37 (10): 1119–24. doi:10.1038/ng1628. PMID 16142236. S2CID 44003147.
- ^ a b c Nguyen LK, Kolch W, Kholodenko BN (July 2013). "When ubiquitination meets phosphorylation: a systems biology perspective of EGFR/MAPK signalling". Cell Communication and Signaling. 11: 52. doi:10.1186/1478-811X-11-52. PMC 3734146. PMID 23902637.
- ^ Sorkin A, Goh LK (October 2008). "Endocytosis and intracellular trafficking of ErbBs". Experimental Cell Research. 314 (17): 3093–106. doi:10.1016/j.yexcr.2008.07.029. PMC 2605728. PMID 18793634.
- ^ Nguyen LK, Muñoz-García J, Maccario H, Ciechanover A, Kolch W, Kholodenko BN (December 2011). "Switches, excitable responses and oscillations in the Ring1B/Bmi1 ubiquitination system". PLOS Computational Biology. 7 (12): e1002317. Bibcode:2011PLSCB...7E2317N. doi:10.1371/journal.pcbi.1002317. PMC 3240587. PMID 22194680.
- ^ Zhou W, Wang X, Rosenfeld MG (January 2009). "Histone H2A ubiquitination in transcriptional regulation and DNA damage repair". The International Journal of Biochemistry & Cell Biology. 41 (1): 12–5. doi:10.1016/j.biocel.2008.09.016. PMID 18929679.
- ^ Dou QP, Li B (August 1999). "Proteasome inhibitors as potential novel anticancer agents". Drug Resistance Updates. 2 (4): 215–223. doi:10.1054/drup.1999.0095. PMID 11504494.
- ^ Vries EG, Verweij J (2000). "Clinical Cancer Research 2000: New Agents and Therapies". Drug Resistance Updates. 3 (4): 197–201. doi:10.1054/drup.2000.0153. PMID 11498385.
- ^ a b c Pray TR, Parlati F, Huang J, Wong BR, Payan DG, Bennett MK, Issakani SD, Molineaux S, Demo SD (December 2002). "Cell cycle regulatory E3 ubiquitin ligases as anticancer targets". Drug Resistance Updates. 5 (6): 249–58. doi:10.1016/s1368-7646(02)00121-8. PMID 12531181.
- ^ a b Clifford SC, Cockman ME, Smallwood AC, Mole DR, Woodward ER, Maxwell PH, Ratcliffe PJ, Maher ER (2001). "Contrasting effects on HIF-1alpha regulation by disease-causing pVHL mutations correlate with patterns of tumourigenesis in von Hippel-Lindau disease". Human Molecular Genetics. 10 (10): 1029–38. doi:10.1093/hmg/10.10.1029. PMID 11331613.
- ^ a b Sparks AB, Morin PJ, Vogelstein B, Kinzler KW (March 1998). "Mutational analysis of the APC/beta-catenin/Tcf pathway in colorectal cancer". Cancer Research. 58 (6): 1130–4. PMID 9515795.
- ^ a b Scheffner M, Huibregtse JM, Vierstra RD, Howley PM (November 1993). "The HPV-16 E6 and E6-AP complex functions as a ubiquitin-protein ligase in the ubiquitination of p53". Cell. 75 (3): 495–505. doi:10.1016/0092-8674(93)90384-3. PMID 8221889. S2CID 27437768.
- ^ a b Momand J, Jung D, Wilczynski S, Niland J (August 1998). "The MDM2 gene amplification database". Nucleic Acids Research. 26 (15): 3453–9. doi:10.1093/nar/26.15.3453. PMC 147746. PMID 9671804.
- ^ a b Hashizume R, Fukuda M, Maeda I, Nishikawa H, Oyake D, Yabuki Y, Ogata H, Ohta T (May 2001). "The RING heterodimer BRCA1-BARD1 is a ubiquitin ligase inactivated by a breast cancer-derived mutation". The Journal of Biological Chemistry. 276 (18): 14537–40. doi:10.1074/jbc.C000881200. PMID 11278247.
- ^ a b Zhu CQ, Blackhall FH, Pintilie M, Iyengar P, Liu N, Ho J, Chomiak T, Lau D, Winton T, Shepherd FA, Tsao MS (2004). "Skp2 gene copy number aberrations are common in non-small cell lung carcinoma, and its overexpression in tumors with ras mutation is a poor prognostic marker". Clinical Cancer Research. 10 (6): 1984–91. doi:10.1158/1078-0432.ccr-03-0470. PMID 15041716.
- ^ a b Schmidt MH, Furnari FB, Cavenee WK, Bögler O (May 2003). "Epidermal growth factor receptor signaling intensity determines intracellular protein interactions, ubiquitination, and internalization". Proceedings of the National Academy of Sciences of the United States of America. 100 (11): 6505–10. Bibcode:2003PNAS..100.6505S. doi:10.1073/pnas.1031790100. PMC 164476. PMID 12734385.
- ^ a b Knuutila S, Aalto Y, Autio K, Björkqvist AM, El-Rifai W, Hemmer S, Huhta T, Kettunen E, Kiuru-Kuhlefelt S, Larramendy ML, Lushnikova T, Monni O, Pere H, Tapper J, Tarkkanen M, Varis A, Wasenius VM, Wolf M, Zhu Y (September 1999). "DNA copy number losses in human neoplasms". The American Journal of Pathology. 155 (3): 683–94. doi:10.1016/S0002-9440(10)65166-8. PMC 1866903. PMID 10487825.
- ^ a b c d e f g Mani A, Gelmann EP (July 2005). "The ubiquitin-proteasome pathway and its role in cancer". Journal of Clinical Oncology. 23 (21): 4776–89. doi:10.1200/JCO.2005.05.081. PMID 16034054.
- ^ a b Nalepa G, Wade Harper J (May 2003). "Therapeutic anti-cancer targets upstream of the proteasome". Cancer Treatment Reviews. 29 Suppl 1: 49–57. doi:10.1016/s0305-7372(03)00083-5. PMID 12738243.
- ^ Witowsky JA, Johnson GL (January 2003). "Ubiquitylation of MEKK1 inhibits its phosphorylation of MKK1 and MKK4 and activation of the ERK1/2 and JNK pathways". The Journal of Biological Chemistry. 278 (3): 1403–6. doi:10.1074/jbc.C200616200. PMID 12456688.
- ^ a b c Kobashigawa Y, Tomitaka A, Kumeta H, Noda NN, Yamaguchi M, Inagaki F (December 2011). "Autoinhibition and phosphorylation-induced activation mechanisms of human cancer and autoimmune disease-related E3 protein Cbl-b". Proceedings of the National Academy of Sciences of the United States of America. 108 (51): 20579–84. Bibcode:2011PNAS..10820579K. doi:10.1073/pnas.1110712108. PMC 3251137. PMID 22158902.
- ^ Niemeyer CM, Kang MW, Shin DH, Furlan I, Erlacher M, Bunin NJ, Bunda S, Finklestein JZ, Sakamoto KM, Gorr TA, Mehta P, Schmid I, Kropshofer G, Corbacioglu S, Lang PJ, Klein C, Schlegel PG, Heinzmann A, Schneider M, Starý J, van den Heuvel-Eibrink MM, Hasle H, Locatelli F, Sakai D, Archambeault S, Chen L, Russell RC, Sybingco SS, Ohh M, Braun BS, Flotho C, Loh ML (September 2010). "Germline CBL mutations cause developmental abnormalities and predispose to juvenile myelomonocytic leukemia". Nature Genetics. 42 (9): 794–800. doi:10.1038/ng.641. PMC 4297285. PMID 20694012.
- ^ Kales SC, Ryan PE, Nau MM, Lipkowitz S (June 2010). "Cbl and human myeloid neoplasms: the Cbl oncogene comes of age". Cancer Research. 70 (12): 4789–94. doi:10.1158/0008-5472.CAN-10-0610. PMC 2888780. PMID 20501843.
- ^ Yen HC, Elledge SJ (2008). "Identification of SCF ubiquitin ligase substrates by global protein stability profiling". Science. 322 (5903): 923–9. Bibcode:2008Sci...322..923Y. doi:10.1126/science.1160462. PMID 18988848. S2CID 23586705.
- ^ Downes BP, Saracco SA, Lee SS, Crowell DN, Vierstra RD (September 2006). "MUBs, a family of ubiquitin-fold proteins that are plasma membrane-anchored by prenylation". The Journal of Biological Chemistry. 281 (37): 27145–57. doi:10.1074/jbc.M602283200. PMID 16831869.
- ^ Welchman RL, Gordon C, Mayer RJ (August 2005). "Ubiquitin and ubiquitin-like proteins as multifunctional signals". Nature Reviews Molecular Cell Biology. 6 (8): 599–609. doi:10.1038/nrm1700. PMID 16064136. S2CID 7373421.
- ^ Grabbe C, Dikic I (April 2009). "Functional roles of ubiquitin-like domain (ULD) and ubiquitin-binding domain (UBD) containing proteins". Chemical Reviews. 109 (4): 1481–94. doi:10.1021/cr800413p. PMID 19253967.
- ^ Sutovsky P, Moreno RD, Ramalho-Santos J, Dominko T, Simerly C, Schatten G (August 2000). "Ubiquitinated sperm mitochondria, selective proteolysis, and the regulation of mitochondrial inheritance in mammalian embryos". Biology of Reproduction. 63 (2): 582–90. doi:10.1095/biolreprod63.2.582. PMID 10906068.
- ^ Wang C, Xi J, Begley TP, Nicholson LK (January 2001). "Solution structure of ThiS and implications for the evolutionary roots of ubiquitin". Nature Structural Biology. 8 (1): 47–51. doi:10.1038/83041. PMID 11135670. S2CID 29632248.
- ^ Lake MW, Wuebbens MM, Rajagopalan KV, Schindelin H (November 2001). "Mechanism of ubiquitin activation revealed by the structure of a bacterial MoeB-MoaD complex". Nature. 414 (6861): 325–9. Bibcode:2001Natur.414..325L. doi:10.1038/35104586. PMID 11713534. S2CID 3224437.
- ^ Hochstrasser M (March 2009). "Origin and function of ubiquitin-like proteins". Nature. 458 (7237): 422–9. Bibcode:2009Natur.458..422H. doi:10.1038/nature07958. PMC 2819001. PMID 19325621.
- ^ Maupin-Furlow JA (2013). "Archaeal proteasomes and sampylation". Sub-Cellular Biochemistry. 66: 297–327. doi:10.1007/978-94-007-5940-4_11. ISBN 978-94-007-5939-8. PMC 3936409. PMID 23479445.
- ^ Fuchs AC, Maldoner L, Wojtynek M, Hartmann MD, Martin J (July 2018). "Rpn11-mediated ubiquitin processing in an ancestral archaeal ubiquitination system". Nature Communications. 9 (1): 2696. Bibcode:2018NatCo...9.2696F. doi:10.1038/s41467-018-05198-1. PMC 6043591. PMID 30002364.
- ^ a b Lehmann G, Udasin RG, Livneh I, Ciechanover A (February 2017). "Identification of UBact, a ubiquitin-like protein, along with other homologous components of a conjugation system and the proteasome in different gram-negative bacteria". Biochemical and Biophysical Research Communications. 483 (3): 946–950. doi:10.1016/j.bbrc.2017.01.037. PMID 28087277.
- ^ Marin J, Battistuzzi FU, Brown AC, Hedges SB (February 2017). "The Timetree of Prokaryotes: New Insights into Their Evolution and Speciation". Molecular Biology and Evolution. 34 (2): 437–446. doi:10.1093/molbev/msw245. PMID 27965376.
- ^ Tung CW, Ho SY (July 2008). "Computational identification of ubiquitylation sites from protein sequences". BMC Bioinformatics. 9: 310. doi:10.1186/1471-2105-9-310. PMC 2488362. PMID 18625080.
- ^ Radivojac P, Vacic V, Haynes C, Cocklin RR, Mohan A, Heyen JW, Goebl MG, Iakoucheva LM (February 2010). "Identification, analysis, and prediction of protein ubiquitination sites". Proteins. 78 (2): 365–80. doi:10.1002/prot.22555. PMC 3006176. PMID 19722269.
- ^ Chen Z, Chen YZ, Wang XF, Wang C, Yan RX, Zhang Z (2011). Fraternali F (ed.). "Prediction of ubiquitination sites by using the composition of k-spaced amino acid pairs". PLOS ONE. 6 (7): e22930. Bibcode:2011PLoSO...622930C. doi:10.1371/journal.pone.0022930. PMC 3146527. PMID 21829559.
- ^ "Stream episode Investigating the ubiquitin proteasome system by Dementia Researcher podcast Listen online for free on SoundCloud".
외부 링크
- 엔젤만 증후군에 대한 GeneReviews/NCBI/NIH/UW 엔트리
- 엔젤맨 증후군에 대한 OMIM 엔트리
- 유비퀴틴용 UniProt 엔트리
- "7.340 Ubiquitination: The Proteasome and Human Disease". MIT OpenCourseWare. 2004. MIT 코스의 노트.
- 미국 국립 의학 도서관 의학 주제 제목(MeSH)의 유비퀴틴