미토겐활성화단백질인산화효소
Mitogen-activated protein kinase| 미토겐활성화단백질인산화효소 | |||||||||
|---|---|---|---|---|---|---|---|---|---|
| 식별자 | |||||||||
| EC 번호 | 2.7.11.24 | ||||||||
| CAS 번호 | 142243-02-5 | ||||||||
| 데이터베이스 | |||||||||
| 인텐츠 | IntEnz 뷰 | ||||||||
| 브렌다 | 브렌다 엔트리 | ||||||||
| ExPASy | NiceZyme 뷰 | ||||||||
| 케그 | KEGG 엔트리 | ||||||||
| 메타사이크 | 대사 경로 | ||||||||
| 프라이머리 | 프로필 | ||||||||
| PDB 구조 | RCSB PDB PDBe PDBum | ||||||||
| 진 온톨로지 | AmiGO / QuickGO | ||||||||
| |||||||||
마이트젠 활성화 단백질 키나제(MAPK 또는 MAP 키나제)는 아미노산 세린 및 트레오닌(즉, 세린/트레오닌 특이 단백질 키나제)에 특이적인 단백질 키나제의 일종이다.MAPK는 유사체, 삼투압, 열충격 및 염증성 사이토카인과 같은 다양한 자극에 대한 세포 반응을 유도하는 데 관여합니다.그들은 증식, 유전자 발현, 분화, 유사분열, 세포 생존, 아포토시스를 [1]포함한 세포 기능을 조절한다.
MAP 키나아제들은 진핵생물에서만 발견되지만, 그것들은 상당히 다양하고 모든 동물, 곰팡이, 식물, 그리고 심지어 단세포 진핵생물들에서도 [citation needed]발견됩니다.
MAPK는 CMGC(CDK/MAPK/GSK3/CLK) 키나제 그룹에 속한다.MAPKs의 가장 가까운 친척은 사이클린 의존성 키나아제(CDKs)[2]이다.
검출
최초로 발견된 마이트젠 활성 단백질 키나제는 포유동물에서 ERK1(MAPK3)이었다.ERK1과 그 가까운 친척 ERK2(MAPK1)는 모두 성장인자 시그널링에 관여하기 때문에, 이 패밀리는 「미토겐 활성」이라고 불립니다.대부분의 MAPK가 잠재적으로 유해하고 비생물적인 스트레스 자극(고삼투압, 산화적 스트레스, DNA 손상, 낮은 삼투압성, 감염 등)에 대한 반응에 실제로 관여하기 때문에 다른 구성원의 발견으로 인해 이 이름이 잘못된 명칭이라는 것이 점점 분명해졌다.식물은 스트레스로 인해 "날 수" 없기 때문에, 육생 식물은 지금까지 발견된[citation needed] 생물 당 가장 많은 수의 MAPK 유전자를 가지고 있다.따라서 세포 증식의 조절제로서의 포유동물 ERK 1/2 키나아제 역할은 일반적이지 않고 고도로 전문화된 기능이다.
종류들
대부분의 MAPK는 두 개의 인산화 사건, 3층 경로 구조 및 유사한 기질 인식 부위에 의존하는 활성화와 같은 여러 가지 공유 특성을 가지고 있다.이들은 "클래식" MAP 키나아제입니다.그러나 위에서 설명한 바와 같이 그룹의 일부 고대 특이치들은 이중 인산화 부위가 없고, 2계층 경로만 형성하며, 기질 결합에 필요한 다른 MAPK가 부족한 특징을 가지고 있다.이들은 보통 '비정형'[3] MAPK라고 불립니다.비정형 MAPK가 기존의 [clarification needed]MAPK와 달리 단일 그룹을 형성하는지 여부는 아직 불분명하다.
포유류의 MAPK 키나아제 패밀리는 세 가지 아과로 구성된다.
- 세포외신호조절인산화효소(ERK)
- c-Jun N-말단 키나아제(JNK)
- p38 마이트겐활성화단백질인산화효소(p38s)[4][5]
일반적으로 ERK는 성장인자와 승모균에 의해 활성화되며, 세포스트레스와 염증성 사이토카인은 JNK와 p38을 [4]활성화한다.
액티베이션
이 섹션은 확장해야 합니다.추가해서 도와주시면 됩니다. (2019년 2월) |
마이트젠 활성 단백질 키나아제들은 염기 형태로 촉매적으로 불활성화된다.활성 상태가 되기 위해서는 활성화 루프에서 (잠재적으로 여러 가지) 인산화 이벤트가 필요합니다.이것은 STE 단백질 키나제 그룹의 특화된 효소에 의해 수행됩니다.이와 같이 단백질 역학이 장거리 알로스테리를 통해 단백질 구조의 구조 변화를 유도할 수 있다.
고전적인 MAP 키나아제인 경우 활성화 루프는 인산화효소 모두에 필요한 특징적인 TxY(트레오닌-x-티로신) 모티브(포유동물 ERK1 및 ERK2의 TEY, ERK5의 TDY, JNKS의 TPY, p38의 TGY)를 포함한다.체내와 체외에서는 티로신의 인산화보다 종종 트레오닌의 인산화보다 앞서지만,[citation needed] 두 잔기의 인산화 중 하나는 다른 것이 없을 때 발생할 수 있다.
이 탠덤 활성화 루프 인산화(MAP2 키나아제라고도 알려진 Ste7 단백질 키나아제 계열의 구성원에 의해 수행된다.MAP2 키나아제 또한 인산화, 다수의 상류의 세린-트레오닌 키나제(MAP3 키나제)에 의해 활성화된다.MAP2 키나아제들은 그들의 동족 MAPK 이외의 기질에서 거의 활성을 보이지 않기 때문에, 고전적인 MAPK 경로는 다층적이지만 상대적으로 선형 경로를 형성한다.이러한 경로는 세포막(많은 MAP3K가 활성화된 곳)에서 핵(MAPK만 들어갈 수 있는 곳) 또는 다른 많은 세포 하위 [citation needed]표적으로 효과적으로 자극을 전달할 수 있다.
기존의 3계층 MAPK 경로와 비교하여 일부 비정형 MAP 키나아제들은 보다 오래된 2계층 시스템을 가지고 있는 것으로 보인다.ERK3(MAPK6)와 ERK4(MAPK4)는 최근 PAK 키나아제([6]다른 MAP3 키나아제 관련)에 의해 직접 인산화되어 활성화되는 것으로 나타났다.기존의 MAP 키나아제와는 달리, 이러한 비정형 MAPK는 인산화되기 위해 활성화 루프의 단일 잔류물만 필요로 한다.NLK 및 ERK7(MAPK15)의 액티베이션에 대한 자세한 내용은 아직 알려지지 않았습니다.
MAPK의 불활성화는 다수의 포스파타아제에 의해 이루어진다.매우 보존된 전용 포스파타아제 계열은 이중 특이성 포스파타아제(DSP)의 하위 그룹인 소위 MAP 키나제 포스파타아제(MKPs)[7]이다.이름에서 알 수 있듯이, 이러한 효소는 포스포티로신과 포스포트레오닌 잔류물 모두에서 인산염을 가수분해할 수 있다.인산기 중 하나를 제거하면 MAPK 활성이 크게 감소하여 시그널링이 근본적으로 폐지되므로, 일부 티로신 포스파타아제(예: 포유류의 포스파타아제 HePTP, STEP 및 PTPR)는 MAP 키나아제 불활성화에도 관여한다.
시그널링 캐스케이드
위에서 언급한 바와 같이, MAPKs는 일반적으로 다층 경로를 형성하며 실제 MAP 키나아제보다 몇 단계 높은 입력을 받는다.MAPKs 및 MAP2Ks의 비교적 단순한 인산화 의존적 활성화 메커니즘과 대조적으로, MAP3Ks는 놀라울 정도로 복잡한 조절을 가지고 있다.c-Rap, MEKK4 또는 MLK3 등 더 잘 알려진 MAP3K의 대부분은 활성화에 여러 단계를 필요로 합니다.이들은 전형적으로 알로스테릭하게 제어되는 효소들로, 여러 메커니즘에 의해 비활성 상태로 단단히 고정된다.활성화되는 첫 번째 단계는 보다 작은 배위자(예를 들어 Ras for c-Rap, GADD45 for[10] MEKK4 또는 Cdc42 for[11] MLK3)에 의한 자동억제를 완화하는 것입니다.이것은 일반적으로(항상 그렇지는 않지만) 대부분의 활성제가 결합되어 있는 세포막에서 발생한다(작은 G-단백질은 프리닐화로 인해 구성적으로 막과 관련되어 있음).그 단계는 현재 접근 가능한 키나아제 도메인의 측면 호모 및 헤테로다이머화가 뒤따른다.최근에 결정된 복잡한 구조에서 이합체가 양쪽의 기질 결합 영역을 [12]자유롭게 하는 방향으로 형성된다는 것을 알 수 있습니다.중요한 것은, 이합체화 이벤트는 또한 MAP3 키나아제 도메인이 부분적으로 활성화된 배열을 채택하도록 강요한다.완전한 활동은 이합체가 활성화 루프에서 서로 트랜스포스포릴레이트한 후에만 달성됩니다.또한 후자의 단계는 보조 단백질 키나아제(MAP4 키나아제, Ste20 계열의 구성원)에 의해 달성되거나 보조될 수 있습니다.일단 MAP3 키나제가 완전히 활성화되면, MAP2 키나아제 기질을 인산화할 수 있으며, 이는 다시 그들의 MAP 키나아제 [citation needed]기질을 인산화할 것이다.
동물에서
포유류의 ERK 1/2 경로는 아마도 가장 잘 특징지어지는 MAPK 시스템일 것이다.이 경로의 가장 중요한 상류 활성제는 성장인자(EGF, FGF, PDGF 등)에 대한 반응의 핵심 매개체인 Raf 단백질(A-Rap, B-Rap 또는 c-Rap)이지만, c-Mos 및 Tpl2/Cot과 같은 다른 MAP3K도 같은 역할을 할 수 있다.이러한 모든 효소는 ERK1 및 ERK2에 대한 매우 특이적인 활성제인 MKK1 및/또는 MKK2 키나제를 인산화하여 활성화한다.후자는 세포 증식, 세포 주기 진행, 세포 분열 및 분화에 중요한 많은 기질을 인산화한다(RSK 키나아제, Elk-1 전사 인자 등).
비교적 잘 절연된 ERK1/2 경로와 대조적으로 포유류의 p38 및 JNK 키나아제들은 대부분의 활성제를 MAP3K 수준에서 공유한다(MEK1, MEKK4, ASK1, TAK1, MLK3, TAOK1 등).또한 일부 MAP2K 효소는 p38과 JNK(MKK4)를 활성화시킬 수 있으며, 다른 효소는 JNK(MKK7) 또는 p38(MK3 및 MKK6)에 대해 더 특이적이다.이러한 연동으로 인해 p38을 동시에 활성화하거나 [13]반대로 활성화하지 않고 JNK 활성화를 유도할 수 있는 자극은 거의 없다.JNK 및 p38 신호 경로는 모두 사이토카인, 자외선 조사, 열충격 및 삼투압 충격과 같은 스트레스 자극에 반응하며 스트레스, 아포토시스 또는 세포 분화에 대한 적응에 관여한다.JNK는 자신만이 인산화시킬 수 있는 많은 전용 기질(c-Jun, NFAT4 등)을 가지고 있으며, p38은 또한 몇 가지 고유한 표적(예: MAPKAP 키나아제 MK2 및 MK3)을 가지고 있어 스트레스 자극에 반응하기 위해 둘 다 필요하다.
ERK5는 포유류에서 상당히 잘 분리된 경로의 일부이다.MAP3 키나아제 MEKK2 및 MEKK3에 응답하여 유일한 고유 업스트림액티베이터 MKK5가 켜집니다이러한 상호작용의 특이성은 MKK5 및 MEKK2/3의 고유한 아키텍처에 의해 제공되며, 둘 다 N 터미널 PB1 도메인을 포함하고 있어 서로 [14]직접 헤테로다이머라이즈를 가능하게 합니다.MKK5의 PB1 도메인은 ERK5와 MKK5의 상호작용에도 관여합니다.MKK5에서 발견된 D-motif와 더불어 MKK5가 기판 ERK5를 특정 인식할 [15]수 있는 특별한 인터페이스를 제공합니다.분자 수준의 세부 사항은 잘 알려져 있지 않지만, MEKK2와 MEKK3는 내피 형성과 심장 형태 형성을 지시하는 특정 발달 단서에 반응한다.뇌 발달에도 관여하지만 심장 이상으로 인한 ERK5 불활성화 배아 치사율은 포유류의 혈관 [16]형성에 있어 ERK5의 중심적인 역할을 강조한다.성인 동물에서 ERK5의 조건부 녹아웃은 내피 [17]장벽의 광범위한 파괴로 인해 치명적이다.ERK5 경로(CCM 복합체)의 상류 구성요소의 돌연변이는 인간의 뇌 동굴성 기형의 기초가 되는 것으로 생각된다.
곰팡이속
곰팡이의 MAPK 경로도 잘 연구되고 있다.효모에서 Fus3 MAPK는 페로몬 자극에 반응하여 세포 주기 정지 및 교미를 담당한다.페로몬 알파 인자는 7개의 막 통과 수용체에 의해 감지된다.Fus3 경로 성분의 모집 및 활성화는 헤테로 이성질 G단백질 활성화에 전적으로 의존한다.짝짓기 MAPK 경로는 세 가지 계층(Ste11-Ste7-Fus3)으로 구성되지만, MAP2 및 MAP3 키나아제들은 다른 경로인 Kss1 또는 필라멘트 성장 경로와 공유된다.Fus3와 Kss1은 밀접하게 관련된 ERK형 키나아제이지만 효모세포는 접합경로의 G단백질에 의해 선택적으로 포섭되는 발판단백질 Ste5의 도움을 받아 여전히 이들을 개별적으로 활성화시킬 수 있다.문제는 Ste5가 Ste7용 Fus3를 3차 복합체의 기판으로서 관련지어 「잠금 해제」할 수 있는 반면, Kss1에 대해서는 같은 처리를 하지 않고, Ste5 채용이 [18]없는 경우에만 필라멘트 성장 경로가 활성화된다는 것입니다.
곰팡이는 또한 포유류의 JNK/p38 신호를 연상시키는 경로를 가지고 있다.이것은 Hog1 경로이다: 높은 삼투압성(사카로미세스 세레비시아) 또는 다른 많은 비생물적 스트레스(분열당류 폼브)에 의해 활성화된다.이 경로의 MAP2 키나아제는 Pbs2(포유동물 MKK3/4/6/7과 관련), 활성화에 관여하는 전용 MAP3 키나아제들은 Ssk2와 SSk22이다.S. cerevisiae의 시스템은 Sho1과 Sln1 단백질로 구성된 정교한 삼투압 모듈에 의해 활성화되지만, 다른 자극이 Hog1의 활성화를 어떻게 유도할 수 있는지는 아직 불분명하다.효모는 또한 세포벽 무결성 경로(Mpk1/Slt2) 또는 포자 형성 경로(Smk1)[19]와 같이 동물에서 근접 호몰로그가 없는 많은 다른 MAPK 경로를 나타낸다.
식물 내
MAPK 유전자의 수가 많음에도 불구하고 고등식물의 MAPK 경로는 동물이나 균류보다 덜 연구되었다.이들의 시그널링은 매우 복잡해 보이지만 아라비도시스 탈리아나의 MPK3, MPK4, MPK6 키나아제들은 삼투압 쇼크, 산화 스트레스, 추위에 대한 반응 및 항병원체 반응에 [20][21][22]관여하는 주요 매개체이다.Asai 등 2002년 MAPK 매개 면역 모델은 FLS2 me MEKK1 mk MKK4 4 MKK5 mp MPK3 3 MPK6 wr WRKY22 wr WRKY29로부터의 [22]이펙터 인식 신호를 통과시킨다.그러나 Mészarros et al. 2006과 Suarez-Rodriges et al. 2007의 연구는 이 경로에 다른 명령을 제공하며,[22] 이 두 경로가 동시에 작동하는 병렬 경로일 수 있다.또한 MPK4 돌연변이는 심각한 [23]왜소증을 보이기 때문에 형태 형성에도 관여합니다.
진화적 관계
MAPK 패밀리의 구성원들은 지금까지 검사된 모든 진핵 생물에서 발견될 수 있다.특히, 고전적 및 비정형 MAP 키나아제 모두 주요 진핵생물군의 방사선의 근원으로 거슬러 올라갈 수 있다.육생 발전소는 무수한 비생물적 [25]스트레스에 반응하는 4개의 고전적 MAPK 그룹(MAPK-A, MAPK-B, MAPK-C 및 MAPK-D)을 포함한다.그러나 이러한 그룹은 오피스토콘트(fungi와 동물)에서 발견되는 고전 MAPK의 군집과 직접 동일시할 수 없다.후자의 경우, 고전적인 MAPK의 주요 서브그룹은 ERK/Fus3 유사 분기(메타존에서 ERK1/2 및 ERK5 서브그룹으로 더욱 세분화)와 p38/Hog1 유사 키나아제(다세포 [26]동물에서 p38 및 JNK 서브그룹으로 분할됨)를 형성한다.또한 균류와 동물에는 여러 개의 MAPK가 있으며, 그 기원은 높은 발산(예: NLK) 또는 전체 MAPK 제품군(ERK3, ERK4, ERK7)에 대한 초기 분기일 가능성이 있기 때문에 명확하지 않다.척추동물에서는 두족보행/버테동물이 [27]분열된 후 쌍둥이 전체 게놈 복제로 인해 모든 그룹에는 여러 개의 패럴로그가 존재한다.따라서 ERK1과 ERK2는 모두 드로소필라 키나제에 해당하며, JNK1, JNK2, JNK3는 모두 드로소필라의 유전자 바구니에 직교한다.p38 그룹 중 p38 알파와 β는 명백히 평행한 쌍이며 척추동물의 p38 감마 및 델타도 마찬가지이지만, 많은 메타조안이 이미 다중 p38 호몰로그를 가지고 있다는 점을 고려할 때 염기 분할 시기는 덜 명확하다(드로소필라, Mpk2(p38a), p38c에는 3가지 p38형 키나아제가 있다).단일 ERK5 단백질은 존재하는 모든 곳에서 매우 특수한 역할을 채우는 것으로 보입니다(척추동물에서 혈관 발달에 필수적입니다).이 혈통은 다세포 동물의 [28]기원에 가까운 특정 단세포 생물(예: choanoflagellate Monosiga brevicollis)에 분명히 존재하지만, 상류 경로 성분(MEK2/3, MKK5)과 함께 프로토스톰에서 삭제되었다.
고전적 MAP 키나아제 및 일부 비정형 MAP 키나아제 간의 분열은 상당히 일찍 일어났다.이는 현존하는 유전자 간의 높은 차이뿐만 아니라 원시, 기초 진핵생물에서 비정형 MAPKs의 최근 발견에 의해서도 제시된다.Giardia lamblia의 게놈 배열 분석 결과 이미 잘 알려진 포유류 MAPKs(ERKs, p38s 등)와 유사한 두 개의 MAPK 유전자가 존재했고, 다른 하나는 포유류 ERK7 [29]단백질과 유사한 것으로 나타났다.ddERK1 단백질이 고전적인 MAPK인 반면 ddERK2는 ERK7 [30]및 ERK3/4 단백질과 더 가까운 다세포 아메바 딕티오스텔륨 디스코이디움에서도 상황은 유사합니다.비정형 MAPK는 잘 알려져 있지 않지만 고등 식물에서도 발견될 수 있다.포유류의 상황과 마찬가지로, 비정형 MAPK의 대부분은 이 분야에 대한 연구가 집중되지 않기 때문에 특징지어지지 않는다.
기판 및 파트너 인식
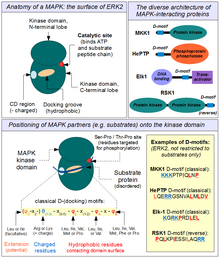
CMGC 키나아제 그룹의 전형적으로 MAP 키나아제 촉매 부위는 기질에 대한 컨센서스 배열이 매우 느슨하다.모든 친척과 마찬가지로, 그들은 표적 세린/트레오닌 아미노산 뒤에 작은 아미노산, 바람직하게는 프롤린(프롤린 유도 키나아제)만 있으면 된다.그러나 SP/TP 부위가 모든 단백질에서 매우 흔하기 때문에, 신호 전달 [31]충실도를 보장하기 위해 추가적인 기질 인식 메커니즘이 진화했다.가장 가까운 친척인 사이클린 의존성 키나제(CDK)와는 달리, 기질이 사이클린 서브유닛에 의해 인식되는 MAPK는 키나아제 도메인 상의 보조 결합 영역을 통해 기질과 결합한다.가장 중요한 영역은 소수성 도킹 홈과 음전하를 띤 CD 영역으로 구성됩니다.그들은 함께 소위 MAPK 도킹 또는 D-모티프(인산화효소 상호작용 모티브 / KIM이라고도 함)를 인식한다. D-모티프는 본질적으로 하나 또는 두 개의 양으로 대전된 아미노산으로 구성되며, 그 다음에 일반적으로 10~[32]50개의 아미노산에 의해 인산화 부위의 상류인 소수성 잔기(대부분의 류신)가 번갈아 이루어진다.기존의 MAPK 기판의 대부분은 특정 MAPK에 바인드할 수 있을 뿐만 아니라 특정 MAPK에 의한 특정 인식도 가능하게 하는 그러한 D모티프를 포함하고 있습니다.D-모티프는 기질에 한정되지 않습니다.MAP2 키나아제에는 MAP2K-MAPK 상호작용 및 MAPK [33]활성화에 절대적으로 필요한 N-termini에도 이러한 모티브가 포함되어 있습니다.마찬가지로, 이중 특이성 MAP 키나제 인산화효소 및 MAP 특이성 티로신 인산화효소 모두 동일한 도킹 부위를 [34][35]통해 MAP 키나제에 결합한다.D-모티프는 특정 MAPK 경로 조절제 및 스캐폴드(포유동물 JIP 단백질 등)에서도 발견될 수 있다.
그 밖에 잘 특징지어지지 않은 기질결합부위도 존재한다.이러한 부위 중 하나(DEF 부위)는 활성화 루프(활성 배합일 때)와 그 아래의 MAP 키나제 특이 삽입물에 의해 형성된다.이 부위는 일반적으로 인산화 [36]부위의 하류인 FxFP 컨센서스 시퀀스를 가진 펩타이드를 수용할 수 있다.후자의 부위는 활성 MAP 키나제를 선택적으로 인식할 필요가 있는 단백질에서만 발견될 수 있으므로, 기질에서 거의 독점적으로 발견된다.다른 모티브는 D-모티프와 FxFP 모티브를 모두 가진 Elk 계열의 전사인자와 같이 서로 협력할 수 있다.KSR1 스칼렛 단백질에 FxFP 모티브가 존재하여 ERK1/2 기질로 만들어 ERK1/2 활성화의 정확한 강도를 설정하기 위한 부귀환 메커니즘을 제공한다.
비계단백질
효모에서 Ste5가 발견된 이후 과학자들은 포유동물에서 유사한 비효소 발판 경로 요소를 발견하기 위해 연구를 진행했습니다.경로의 여러 요소에 결합할 수 있는 ERK 시그널링에는 실제로 많은 단백질이 포함되어 있습니다. MP1은 MKK1/2와 ERK1/2를 결합하고, KSR1과 KSR2는 B-Rap 또는 c-Rap, MKK1/2와 ERK1/2를 결합할 수 있습니다.JNK 경로에도 유사한 단백질이 발견되었다: JIP1/JIP2 및 JIP3/JIP4 단백질군은 모두 MLK, MKK7 및 JNK 키나제에 결합하는 것으로 나타났다.불행히도 효모 Ste5와는 달리 MAPK 활성화를 조절하는 메커니즘은 상당히 덜 알려져 있다.Ste5가 Ste7 및 Fus3의 인산화 촉진을 위해 실제로 3원 복합체를 형성하는 반면, 알려진 포유류의 골격 단백질은 매우 다른 메커니즘에 의해 작용하는 것으로 보입니다.예를 들어 KSR1과 KSR2는 실제로 MAP3 키나아제이며 Raf [37]단백질과 관련이 있습니다.KSR만으로는 MAP3 키나아제 활성이 미미하지만, KSR 단백질은 여전히 Raf 키나제 활성화에 참여할 수 있으며, 이는 각 [38]효소를 활성화하는 알로스테릭 쌍을 제공한다.반면 JIP는 분극세포의 [39]특정 구획에서 MAPK 시그널링 성분의 농도를 담당하는 수송단백질이다.이러한 맥락에서 JIP1(그리고 아마도 JIP2)의 JNK 의존적 인산화(인산화)는 JIP가 JIP 결합 및 비활성 업스트림 경로 성분을 방출하는 신호를 제공하여 강한 국소 양성 피드백 [40]루프를 유도한다.이 정교한 메커니즘은 포유동물뿐만 아니라 초파리 드로소필라 [41]멜라노가스터에서 키네신 의존성 수송을 국소 JNK 활성화에 결합시킵니다.
치료 대상으로서
ERK 시그널링 경로는 생리학적 및 병리학적 세포 증식에 모두 관여하므로 ERK1/2 억제제가 바람직한 항종양제를 나타내는 것은 당연하다.실제로, 많은 원생 "드라이버" 돌연변이는 구성 활성(변형) 수용체 티로신 키나아제, Ras 또는 Raf 단백질과 같은 ERK1/2 시그널링에 결합되어 있다.임상 사용을 위해 개발된 MKK 1/2 또는 ERK 1/2 억제제는 없지만, Raf 키나아제(예: Sorafenib)를 억제하는 키나아제 억제제는 다양한 유형의 [42][43]암에 대한 성공적인 항종양제이다.MEK 억제제인 코비메티니브는 PI3K 경로의 억제와 함께 임상 전 폐암 모델에서 조사되었으며, 두 약물은 상승 [44][45]반응을 이끌어냈다.
JNK 키나아제들은 허혈증 상태 이후의 신경전달물질 흥분독성뿐만 아니라 비만인[46] 사람의 인슐린 저항성의 발달에 관여한다.JNK1의 억제는 특정 동물 모델에서 인슐린 저항을 개선한다.기능성 JNK3 유전자(뇌의 주요 아이소폼)가 부족하도록 유전공학적으로 조작된 쥐들은 강화된 허혈 내성과 뇌졸중 [47]회복을 보인다.소분자 JNK 억제제가 개발 중이지만 인체실험에서 효과가 입증된 것은 아직 없다.펩타이드 기반의 JNK 억제제(AM-111, JIP1의 역방향 D-모티프 펩타이드, 이전에 XG-102로 알려짐)도 감각성 [48]난청을 위해 임상 개발 중이다.
p38은 한때 항염증제의 완벽한 표적으로 여겨졌다.그러나 임상 단계에서 12개 이상의 화학적으로 다른 화합물의 실패는 p38 키나아제들이 자가면역질환에서 좋지 않은 치료 대상이 될 수 있음을 시사한다.이들 화합물 중 다수는 다양한 정도로 간독성이 있는 것으로 밝혀졌으며 항염증 효과에 대한 내성이 몇 [49]주 내에 나타났다.대체 접근법은 ASK1 [50]등의 업스트림 MAPK를 대상으로 하는 가능성을 평가하는 것입니다.염증성 관절염의 동물 모델에 대한 연구는 유망한 결과를 얻었으며, ASK1은 최근 TNF-α와 [50]같은 염증성 사이토카인에 의해 유도된다는 점에서 MAPKs에서 특이하다는 것이 발견되었다.
「 」를 참조해 주세요.
- 신호 변환
- MAP인산화효소
- MAP인산화효소인산화효소
- MAP인산화효소인산화효소인산화효소
- MAPK1(ERK2)
- MAPK3(ERK1)
- MAPK7(ERK5)
- MAPK8(JNK1)
- MAPK9(JNK2)
- MAPK10(JNK3)
- MAPK11(p38-beta)
- MAPK12(p38-gamma)
- MAPK13(p38-delta)
- MAPK14(p38-alpha)
- MAPK4(ERK4: 비정형 MAPK)
- MAPK6(ERK3: 비정형 MAPK)
- MAPK15(ERK7/ERK8: 비정형 MAPK)
- NLK(니모양인산화효소: 비정형 MAPK)
- ERK 1/2 키나아제
- ERK1/2 패스
- JNK 키나아제
- p38 MAP 키나아제
- MEKK2(MAP3K2)
- ASK1(MAP3K5)
레퍼런스
- ^ Pearson G, Robinson F, Beers Gibson T, Xu BE, Karandikar M, Berman K, Cobb MH (Apr 2001). "Mitogen-activated protein (MAP) kinase pathways: regulation and physiological functions". Endocrine Reviews. 22 (2): 153–83. doi:10.1210/edrv.22.2.0428. PMID 11294822.
- ^ Manning G, Whyte DB, Martinez R, Hunter T, Sudarsanam S (Dec 2002). "The protein kinase complement of the human genome". Science. 298 (5600): 1912–34. Bibcode:2002Sci...298.1912M. doi:10.1126/science.1075762. PMID 12471243. S2CID 26554314.
- ^ Coulombe P, Meloche S (Aug 2007). "Atypical mitogen-activated protein kinases: structure, regulation and functions". Biochimica et Biophysica Acta (BBA) - Molecular Cell Research. 1773 (8): 1376–87. doi:10.1016/j.bbamcr.2006.11.001. PMID 17161475.
- ^ a b Yu J, Sun X, Goie J, Zhang Y (2020). "Regulation of Host Immune Responses against Influenza A Virus Infection by Mitogen-Activated Protein Kinases (MAPKs)". Microorganisms. 8 (7): 1067. doi:10.3390/microorganisms8071067. PMC 7409222. PMID 32709018.
- ^ Shi J, Sun S (2018). "Tumor Necrosis Factor Receptor-Associated Factor Regulation of Nuclear Factor κB and Mitogen-Activated Protein Kinase Pathways". Frontiers in Immunology. 9: 1849. doi:10.3389/fimmu.2018.01849. PMC 6094638. PMID 30140268.
- ^ Déléris P, Trost M, Topisirovic I, Tanguay PL, Borden KL, Thibault P, Meloche S (Feb 2011). "Activation loop phosphorylation of ERK3/ERK4 by group I p21-activated kinases (PAKs) defines a novel PAK-ERK3/4-MAPK-activated protein kinase 5 signaling pathway". The Journal of Biological Chemistry. 286 (8): 6470–8. doi:10.1074/jbc.M110.181529. PMC 3057823. PMID 21177870.
- ^ Theodosiou A, Ashworth A (Jun 2002). "MAP kinase phosphatases". Genome Biology. 3 (7): reviews3009.1–reviews3009.10. doi:10.1186/gb-2002-3-7-reviews3009. PMC 139386. PMID 12184814.
- ^ Matallanas D, Birtwistle M, Romano D, et al. (March 2011). "Raf family kinases: old dogs have learned new tricks". Genes Cancer. 2 (3): 232–60. doi:10.1177/1947601911407323. PMC 3128629. PMID 21779496.
- ^ Alexa A, Varga J, Reményi A (November 2010). "Scaffolds are 'active' regulators of signaling modules". FEBS J. 277 (21): 4376–82. doi:10.1111/j.1742-4658.2010.07867.x. PMID 20883493. S2CID 43848866.
- ^ Miyake Z, Takekawa M, Ge Q, Saito H (Apr 2007). "Activation of MTK1/MEKK4 by GADD45 through induced N-C dissociation and dimerization-mediated trans autophosphorylation of the MTK1 kinase domain". Molecular and Cellular Biology. 27 (7): 2765–76. doi:10.1128/MCB.01435-06. PMC 1899887. PMID 17242196.
- ^ Du Y, Böck BC, Schachter KA, Chao M, Gallo KA (Dec 2005). "Cdc42 induces activation loop phosphorylation and membrane targeting of mixed lineage kinase 3". The Journal of Biological Chemistry. 280 (52): 42984–93. doi:10.1074/jbc.M502671200. PMID 16253996.
- ^ Rajakulendran T, Sahmi M, Lefrançois M, Sicheri F, Therrien M (Sep 2009). "A dimerization-dependent mechanism drives RAF catalytic activation". Nature. 461 (7263): 542–5. Bibcode:2009Natur.461..542R. doi:10.1038/nature08314. PMID 19727074. S2CID 427603.
- ^ Cargnello M, Roux PP (Mar 2011). "Activation and function of the MAPKs and their substrates, the MAPK-activated protein kinases". Microbiology and Molecular Biology Reviews. 75 (1): 50–83. doi:10.1128/MMBR.00031-10. PMC 3063353. PMID 21372320.
- ^ Nakamura K, Johnson GL (Sep 2003). "PB1 domains of MEKK2 and MEKK3 interact with the MEK5 PB1 domain for activation of the ERK5 pathway". The Journal of Biological Chemistry. 278 (39): 36989–92. doi:10.1074/jbc.C300313200. PMID 12912994.
- ^ a b Glatz G, Gógl G, Alexa A, Reményi A (Mar 2013). "Structural mechanism for the specific assembly and activation of the extracellular signal regulated kinase 5 (ERK5) module". The Journal of Biological Chemistry. 288 (12): 8596–609. doi:10.1074/jbc.M113.452235. PMC 3605678. PMID 23382384.
- ^ Regan CP, Li W, Boucher DM, Spatz S, Su MS, Kuida K (Jul 2002). "Erk5 null mice display multiple extraembryonic vascular and embryonic cardiovascular defects". Proceedings of the National Academy of Sciences of the United States of America. 99 (14): 9248–53. Bibcode:2002PNAS...99.9248R. doi:10.1073/pnas.142293999. PMC 123126. PMID 12093914.
- ^ Hayashi M, Lee JD (Dec 2004). "Role of the BMK1/ERK5 signaling pathway: lessons from knockout mice". Journal of Molecular Medicine. 82 (12): 800–8. doi:10.1007/s00109-004-0602-8. PMID 15517128. S2CID 8499230.
- ^ Good M, Tang G, Singleton J, Reményi A, Lim WA (Mar 2009). "The Ste5 scaffold directs mating signaling by catalytically unlocking the Fus3 MAP kinase for activation". Cell. 136 (6): 1085–97. doi:10.1016/j.cell.2009.01.049. PMC 2777755. PMID 19303851.
- ^ Gustin MC, Albertyn J, Alexander M, Davenport K (Dec 1998). "MAP kinase pathways in the yeast Saccharomyces cerevisiae". Microbiology and Molecular Biology Reviews. 62 (4): 1264–300. doi:10.1128/MMBR.62.4.1264-1300.1998. PMC 98946. PMID 9841672.
- ^ Sinha AK, Jaggi M, Raghuram B, Tuteja N (Feb 2011). "Mitogen-activated protein kinase signaling in plants under abiotic stress". Plant Signaling & Behavior. 6 (2): 196–203. doi:10.4161/psb.6.2.14701. PMC 3121978. PMID 21512321.
- ^ Rodriguez MC, Petersen M, Mundy J (2010). "Mitogen-activated protein kinase signaling in plants". Annual Review of Plant Biology. 61: 621–49. doi:10.1146/annurev-arplant-042809-112252. PMID 20441529.
- ^ a b c Göhre V, Robatzek S (2008-09-01). "Breaking the Barriers: Microbial Effector Molecules Subvert Plant Immunity". Annual Review of Phytopathology. Annual Reviews. 46 (1): 189–215. doi:10.1146/annurev.phyto.46.120407.110050. ISSN 0066-4286. PMID 18422429.
- ^ Kosetsu K, Matsunaga S, Nakagami H, Colcombet J, Sasabe M, Soyano T, Takahashi Y, Hirt H, Machida Y (Nov 2010). "The MAP kinase MPK4 is required for cytokinesis in Arabidopsis thaliana". The Plant Cell. 22 (11): 3778–90. doi:10.1105/tpc.110.077164. PMC 3015120. PMID 21098735.
- ^ Li M, Liu J, Zhang C (2011). "Evolutionary history of the vertebrate mitogen activated protein kinases family". PLOS ONE. 6 (10): e26999. Bibcode:2011PLoSO...626999L. doi:10.1371/journal.pone.0026999. PMC 3202601. PMID 22046431.
- ^ MAPK Group (Jul 2002). "Mitogen-activated protein kinase cascades in plants: a new nomenclature". Trends in Plant Science. 7 (7): 301–8. doi:10.1016/S1360-1385(02)02302-6. PMID 12119167.
- ^ Caffrey DR, O'Neill LA, Shields DC (Nov 1999). "The evolution of the MAP kinase pathways: coduplication of interacting proteins leads to new signaling cascades". Journal of Molecular Evolution. 49 (5): 567–82. Bibcode:1999JMolE..49..567C. doi:10.1007/PL00006578. PMID 10552038. S2CID 2992439.
- ^ Putnam NH, Butts T, Ferrier DE, Furlong RF, Hellsten U, Kawashima T, et al. (Jun 2008). "The amphioxus genome and the evolution of the chordate karyotype". Nature. 453 (7198): 1064–71. Bibcode:2008Natur.453.1064P. doi:10.1038/nature06967. PMID 18563158.
- ^ King N, Westbrook MJ, Young SL, Kuo A, Abedin M, Chapman J, et al. (Feb 2008). "The genome of the choanoflagellate Monosiga brevicollis and the origin of metazoans". Nature. 451 (7180): 783–8. Bibcode:2008Natur.451..783K. doi:10.1038/nature06617. PMC 2562698. PMID 18273011.
- ^ Ellis JG, Davila M, Chakrabarti R (Jan 2003). "Potential involvement of extracellular signal-regulated kinase 1 and 2 in encystation of a primitive eukaryote, Giardia lamblia. Stage-specific activation and intracellular localization". The Journal of Biological Chemistry. 278 (3): 1936–45. doi:10.1074/jbc.M209274200. PMID 12397063.
- ^ Hadwiger JA, Nguyen HN (Apr 2011). "MAPKs in development: insights from Dictyostelium signaling pathways". Biomolecular Concepts. 2 (1–2): 39–46. doi:10.1515/BMC.2011.004. PMC 3110071. PMID 21666837.
- ^ a b Garai Á, Zeke A, Gógl G, Törő I, Fördős F, Blankenburg H, Bárkai T, Varga J, Alexa A, Emig D, Albrecht M, Reményi A (Oct 2012). "Specificity of linear motifs that bind to a common mitogen-activated protein kinase docking groove". Science Signaling. 5 (245): ra74. doi:10.1126/scisignal.2003004. PMC 3500698. PMID 23047924.
- ^ Reményi A, Good MC, Bhattacharyya RP, Lim WA (Dec 2005). "The role of docking interactions in mediating signaling input, output, and discrimination in the yeast MAPK network". Molecular Cell. 20 (6): 951–62. doi:10.1016/j.molcel.2005.10.030. PMID 16364919.
- ^ Bardwell AJ, Frankson E, Bardwell L (May 2009). "Selectivity of docking sites in MAPK kinases". The Journal of Biological Chemistry. 284 (19): 13165–73. doi:10.1074/jbc.M900080200. PMC 2676048. PMID 19196711.
- ^ Goldsmith EJ (Dec 2011). "Three-dimensional docking in the MAPK p38α". Science Signaling. 4 (204): pe47. doi:10.1126/scisignal.2002697. PMID 22375047. S2CID 206671310.
- ^ Huang Z, Zhou B, Zhang ZY (Dec 2004). "Molecular determinants of substrate recognition in hematopoietic protein-tyrosine phosphatase". The Journal of Biological Chemistry. 279 (50): 52150–9. doi:10.1074/jbc.M407820200. PMID 15466470.
- ^ Sheridan DL, Kong Y, Parker SA, Dalby KN, Turk BE (Jul 2008). "Substrate discrimination among mitogen-activated protein kinases through distinct docking sequence motifs". The Journal of Biological Chemistry. 283 (28): 19511–20. doi:10.1074/jbc.M801074200. PMC 2443660. PMID 18482985.
- ^ McKay MM, Freeman AK, Morrison DK (Sep 2011). "Complexity in KSR function revealed by Raf inhibitor and KSR structure studies". Small GTPases. 2 (5): 276–281. doi:10.4161/sgtp.2.5.17740. PMC 3265819. PMID 22292131.
- ^ Brennan DF, Dar AC, Hertz NT, Chao WC, Burlingame AL, Shokat KM, Barford D (Apr 2011). "A Raf-induced allosteric transition of KSR stimulates phosphorylation of MEK". Nature. 472 (7343): 366–9. Bibcode:2011Natur.472..366B. doi:10.1038/nature09860. PMID 21441910. S2CID 18818.
- ^ Koushika SP (Jan 2008). ""JIP"ing along the axon: the complex roles of JIPs in axonal transport". BioEssays. 30 (1): 10–4. doi:10.1002/bies.20695. PMID 18081006.
- ^ Nihalani D, Wong HN, Holzman LB (Aug 2003). "Recruitment of JNK to JIP1 and JNK-dependent JIP1 phosphorylation regulates JNK module dynamics and activation". The Journal of Biological Chemistry. 278 (31): 28694–702. doi:10.1074/jbc.M304212200. PMID 12756254.
- ^ Horiuchi D, Collins CA, Bhat P, Barkus RV, Diantonio A, Saxton WM (Aug 2007). "Control of a kinesin-cargo linkage mechanism by JNK pathway kinases". Current Biology. 17 (15): 1313–7. doi:10.1016/j.cub.2007.06.062. PMC 2041807. PMID 17658258.
- ^ Kim DH, Sim T (Mar 2012). "Novel small molecule Raf kinase inhibitors for targeted cancer therapeutics". Archives of Pharmacal Research. 35 (4): 605–15. doi:10.1007/s12272-012-0403-5. PMID 22553052. S2CID 26714490.
- ^ Matsuda Y, Fukumoto M (Dec 2011). "Sorafenib: complexities of Raf-dependent and Raf-independent signaling are now unveiled". Medical Molecular Morphology. 44 (4): 183–9. doi:10.1007/s00795-011-0558-z. PMID 22179180. S2CID 23677733.
- ^ Heavey S, Cuffe S, Finn S, Young V, Ryan R, Nicholson S, et al. (November 2016). "In pursuit of synergy: An investigation of the PI3K/mTOR/MEK co-targeted inhibition strategy in NSCLC". Oncotarget. 7 (48): 79526–79543. doi:10.18632/oncotarget.12755. PMC 5346733. PMID 27765909.
- ^ Heavey S, O'Byrne KJ, Gately K (April 2014). "Strategies for co-targeting the PI3K/AKT/mTOR pathway in NSCLC". Cancer Treatment Reviews. 40 (3): 445–456. doi:10.1016/j.ctrv.2013.08.006. PMID 24055012.
- ^ Hirosumi J, Tuncman G, Chang L, Görgün CZ, Uysal KT, Maeda K, Karin M, Hotamisligil GS (Nov 2002). "A central role for JNK in obesity and insulin resistance". Nature. 420 (6913): 333–6. Bibcode:2002Natur.420..333H. doi:10.1038/nature01137. PMID 12447443. S2CID 1659156.
- ^ Bogoyevitch MA, Boehm I, Oakley A, Ketterman AJ, Barr RK (Mar 2004). "Targeting the JNK MAPK cascade for inhibition: basic science and therapeutic potential". Biochimica et Biophysica Acta (BBA) - Proteins and Proteomics. 1697 (1–2): 89–101. doi:10.1016/j.bbapap.2003.11.016. PMID 15023353.
- ^ Wang J, Van De Water TR, Bonny C, de Ribaupierre F, Puel JL, Zine A (Sep 2003). "A peptide inhibitor of c-Jun N-terminal kinase protects against both aminoglycoside and acoustic trauma-induced auditory hair cell death and hearing loss". The Journal of Neuroscience. 23 (24): 8596–607. doi:10.1523/JNEUROSCI.23-24-08596.2003. PMC 6740364. PMID 13679429.
- ^ Genovese MC (Feb 2009). "Inhibition of p38: has the fat lady sung?". Arthritis and Rheumatism. 60 (2): 317–20. doi:10.1002/art.24264. PMID 19180514.
- ^ a b Nygaard G, Di Paolo JA, Hammaker D, Boyle DL, Budas G, Notte GT, et al. (May 2018). "Regulation and function of apoptosis signal-regulating kinase 1 in rheumatoid arthritis". Biochemical Pharmacology. 151: 282–290. doi:10.1016/j.bcp.2018.01.041. PMID 29408488. S2CID 4863537.
외부 링크
- MAP 키나제 리소스.
- 마이트젠 활성 키나아제 이름표.
- MAPK 캐스케이드 그림
- 미 국립의학도서관의 Mitogen-Activated+Protein+Kinase(MeSH) 의학 주제 표제
- 바이오모델 데이터베이스의 MAPK 초음파 모델
- Drosophila 롤 – 인터랙티브 플라이





