하플로그룹
Haplogroup| 시리즈의 일부 |
| 유전계보 |
|---|
| 개념 |
| 관련 토픽 |
A haplotype is a group of alleles in an organism that are inherited together from a single parent,[1][2] and a haplogroup (haploid from the Greek: ἁπλοῦς, haploûs, "onefold, simple" and English: group) is a group of similar haplotypes that share a common ancestor with a single-nucleotide polymorphism mutation.[3][4]구체적으로 말하면, 하플로그룹은 서로 밀접하게 연결되어 있고 함께 유전되는 경향이 있는 다른 염색체 영역의 대립 유전자들의 조합이다.하플로그룹은 유사한 하플로타입으로 구성되어 있기 때문에 보통 하플로타입에서 하플로그룹을 예측할 수 있다.하플로그룹은 단일 혈통에 속합니다.이와 같이, 하플로그룹의 멤버십은, 어느 개인에 의해서도, 그 개인이 가지는 유전 물질의 비교적 작은 비율에 의존하고 있다.
각 하플로그룹은 이전의 단일 하플로그룹(또는 파라그룹)에서 시작되어 그 일부가 됩니다.이와 같이, 하플로그룹의 관련 그룹은 내포된 계층으로 정밀하게 모델링될 수 있으며, 여기서 각 집합(하플로그룹)은 또한 단일 더 넓은 집합의 하위 집합이다(즉, 인간 가계도와 같은 두 부모 모델과는 반대).
하플로그룹은 일반적으로 알파벳의 첫 글자로 식별되며, 미세화는 (예를 들어) A → A1 → A1a와 같은 추가 숫자와 문자 조합으로 구성된다.
인간 유전학에서 가장 일반적으로 연구되는 하플로그룹은 Y-염색체(Y-DNA) 하플로그룹과 미토콘드리아 DNA(mtDNA) 하플로그룹으로, 각각 유전자 집단을 정의하는데 사용될 수 있다.Y-DNA는 아버지에서 아들로, mtDNA는 어머니에서 남녀의 자손으로 모계 계통을 따라 전달된다.둘 다 재결합하지 않기 때문에 Y-DNA와 mtDNA는 부모의 유전자 물질 사이에 혼화물이 없는 각 세대의 우연한 돌연변이에 의해서만 변화한다.
하플로그룹 형성

미토콘드리아는 인간의 세포질처럼 진핵세포의 세포질에 있는 작은 세포소기관이다.그들의 주된 기능은 세포에 에너지를 공급하는 것이다.미토콘드리아는 한때 자유로웠던 공생 박테리아의 후손으로 여겨진다.미토콘드리아가 한때 자유로운 삶이었다는 한 가지 증거는 각각이 진핵생물보다 박테리아와 더 유사한 구조를 가진 미토콘드리아 DNA라고 불리는 원형 DNA를 포함하고 있다는 것이다.인간 DNA의 압도적 대다수는 세포핵의 염색체에 포함되어 있지만 mtDNA는 예외이다.개인은 모란(난자 세포)에서만 세포질과 그 세포질에 포함된 소기관만을 물려받는다.정자는 염색체 DNA에만 전달되며 모든 부계 미토콘드리아는 난모세포에서 소화된다.따라서 mtDNA 분자에서 돌연변이가 발생하면, 돌연변이는 직접적인 여성 혈통으로 전달된다.돌연변이는 DNA 염기서열에서 실수를 복제하는 거야단일 실수는 단일 뉴클레오티드 다형이라고 불린다.[dubious ]
인간의 Y염색체는 남성 특유의 성염색체이다; Y염색체를 가진 거의 모든 인간은 형태학적으로 남성일 것이다.Y염색체는 세포핵에 위치하고 X염색체와 짝을 이루지만 Y염색체 끝의 X염색체와만 재결합하고 나머지 95%의 Y염색체는 재결합하지 않는다.따라서 Y염색체와 Y염색체에서 발생하는 돌연변이는 직계 남성 혈통을 통해 아버지로부터 아들에게로 전달된다.이것은 Y 염색체와 mtDNA가 특정한 특성을 공유한다는 것을 의미한다.
여성의 다른 염색체, 자가 염색체와 X 염색체는 감수 분열 동안 그들의 유전 물질을 공유합니다.사실상 이것은 이 염색체들의 유전 물질이 모든 세대에서 섞여서 새로운 돌연변이가 부모로부터 자손에게 무작위로 전해진다는 것을 의미합니다.
Y염색체와 mtDNA가 보여주는 특별한 특징은 돌연변이가 양쪽 분자의 특정 부분을 따라 축적될 수 있고 이러한 돌연변이는 DNA에 고정된 상태로 유지된다는 것이다.게다가 이러한 돌연변이의 이력 순서도 추측할 수 있다.예를 들어, 10개의 Y염색체 집합이 돌연변이 A를 포함하지만, 이 염색체 중 5개만 두 번째 돌연변이 B를 포함한다면, 돌연변이 B가 돌연변이 A 후에 발생한 경우일 것이다.
게다가, 돌연변이 A를 가진 염색체를 운반하는 10명의 남성들은 모두 이 돌연변이를 최초로 운반한 동일한 남성의 직계 후손들이다.돌연변이 B를 가진 최초의 남성은 이 남성의 직계 후손이기도 하지만, 돌연변이 B를 가진 모든 남성의 직계 남성 조상이기도 하다.이와 같은 일련의 돌연변이는 분자 계통을 형성한다.게다가, 각각의 돌연변이는 하플로그룹이라고 불리는 특정한 Y염색체 세트를 정의한다.
돌연변이 A를 가진 모든 남성은 단일 하플로그룹을 형성하고, 돌연변이 B를 가진 모든 남성은 이 하플로그룹의 일부이지만, 돌연변이 B는 또한 돌연변이 A만을 가진 남성은 소속되지 않는 자신의 보다 최근의 하플로그룹(하위 그룹 또는 하위 그룹)을 정의한다.mtDNA와 Y 염색체는 모두 계통군과 하플로그룹으로 분류되며, 이들은 종종 나무와 같은 그림으로 나타난다.
하플로그룹 집단 유전학
일반적으로 그 선물 day,[표창 필요한]에 극 돌연변이율에서(는 표시 하나에서 다른 다양하게 변한다)집단 유전학 haplotypes의 인구의 비율이 영향을 미치는 주요 요인은 유전 drift—r를 넘기고 살아남은 특정한 일배 체형 돌연변이에 대해 작은 자연 도태는 것으로 가정한다.andom 변동다음 세대의 적절한 성별에게 DNA를 물려주는 표본 추출의 무작위성에 의해 야기된다.
이로 인해 모집단에서 특정 마커의 유병률은 100%에 도달하거나 모집단에서 완전히 떨어질 때까지 계속 변동합니다.일반 대립 유전자의 효율적인 혼합을 가진 큰 개체군에서는 유전적 표류 속도가 매우 낮지만, 매우 작은 이종 교배 개체군에서는 비율이 훨씬 더 빠르게 변할 수 있습니다.따라서 특정 하플로타입과 하플로타입 그룹의 현저한 지리적 변화와 농도는 반복적인 인구 병목현상 또는 인구 분리와 증가에 따른 창시자의 뚜렷한 영향을 목격한다.
현재로부터 거슬러 올라갈 수 있는 혈통은 노년층의 완전한 유전적 변이를 반영하지 않을 것이다: 유전자 표류는 일부 변종들이 사라질 것이라는 것을 의미한다.전체 Y-DNA 및 mtDNA 시퀀스 테스트 비용은 데이터 가용성을 제한했지만, 지난 10년 동안 이러한 비용은 급격히 감소했습니다.하플로타입 결합 시간과 현재 지리적 유병률 모두 상당한 오차 불확실성을 가지고 있다.대부분의 인구유전학자들이 여전히 지보토프스키 방법을 계속 사용하고 있기 때문에, 이것은 특히 합병 시기에 문제가 된다.지보토프스키 방법은 DNA-유전학자들로부터 [citation needed]거짓으로 심하게 비판받고 있다.공생 말벌인 앤지오폴리비아는 위치에 따라 8개의 하플로그룹을 가지고 있다.이것은 유전적 표류 개념을 보여준다.
인간 Y염색체 DNA 하플로그룹
인간 Y염색체 DNA(Y-DNA) 하플로그룹은 A에서 T로 명명되며 숫자와 소문자를 사용하여 더욱 세분화된다.Y염색체 하플로그룹은 Y염색체 컨소시엄에 [5]의해 지정된다.
Y염색체 아담은 모든 살아있는 인간의 가장 최근 흔한 부계 조상인 남성에게 연구자들에 의해 붙여진 이름이다.
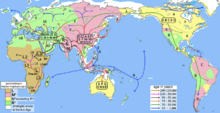
주요 Y염색체 하플로그룹과 발생 지역(최근 유럽 식민지화 이전)은 다음과 같다.
변환 M168이 없는 그룹
변환이 M168인 그룹
(변환 M168은 50,000 bp까지 발생)
- 하플로그룹 C(M130) (오세아니아, 북/중앙/동아시아, 북미 및 남미, 동남아시아, 남아시아, 서아시아, 유럽 등)
- YAP+ 하플로그룹
- 하플로그룹 DE (M1, M145, M203)
- 하플로그룹 D(CTS3946) (티베트, 일본 안다만 제도, 중앙아시아, 나이지리아, 시리아, 사우디아라비아 산발적 존재)
- 하플로그룹 E (M96)
- 하플로그룹 E1b1a(V38) 서아프리카 및 그 주변 지역(이전의 이름은 E3a)
- 아프리카아시아어 확산과 관련된 하플로그룹 E1b1b(M215) 현재는 북아프리카와 아프리카의 뿔, 중동, 지중해, 발칸 반도 일부에 집중되어 있다.이전에는 E3b로 알려져 있었다.
- 하플로그룹 DE (M1, M145, M203)
돌연변이가 M89인 그룹
(변환 M89는 45,000 bp까지 발생)
- 하플로그룹 F(M89) 오세아니아, 유럽, 아시아, 북미 및 남미
- 하플로그룹 G(M201) (보통 유라시아의 많은 민족 집단 사이에 존재하며, 보통 빈도가 낮다; 코카서스, 이란 고원 및 아나톨리아에서 가장 흔하다; 유럽에서는 주로 그리스, 이탈리아, 이베리아, 티롤, 보헤미아, 북유럽에서는 드물다.)
- 하플로그룹 H(L901/M2939)
- 하플로그룹 IJK(L15, L16)
돌연변이 L15 및 L16 그룹
- 하플로그룹 IJK(L15, L16)
돌연변이가 M9인 그룹
(변환 M9은 40,000bp까지 발생)
변환 M526을 가진 그룹
- 하플로그룹 M(P256) (인도네시아 동부 뉴기니, 멜라네시아)
- 하플로그룹 NO (M214)
- 하플로그룹 P-M45(M45)(M45는 35,000bp까지 발생)
- 하플로그룹 Q-M242(M242) (약 15,000~20,000 bp 발생).아시아 및 아메리카에서 발견
- 하플로그룹 Q-M3(M3) (북미, 중미, 남미)
- 하플로그룹 R (M207)
- 하플로그룹 Q-M242(M242) (약 15,000~20,000 bp 발생).아시아 및 아메리카에서 발견
- 하플로그룹 S(M230, P202, P204) (인도네시아 동부 뉴기니)
인간 미토콘드리아 DNA 하플로그룹
인간 mtDNA 하플로그룹에는 A, B, C, CZ, D, E, F, G, H, HV, I, JT, JT, K, L0, L1, L2, L3, L4, L6, L6 등의 문자가 있습니다.mtDNA 트리의 최신 버전은 PhyloTree 웹사이트에 [9]있는 Mannis van Oven에 의해 관리되고 있습니다.
| 인간 미토콘드리아 DNA(mtDNA) 하플로그룹 계통수 | |||||||||||||||||||||||||||||||||||||||
| 미토콘드리아 이브(L) | |||||||||||||||||||||||||||||||||||||||
| L0 | L1 ~ 6 | ||||||||||||||||||||||||||||||||||||||
| L1 | L2 | L3 | L4 | L5 | L6 | ||||||||||||||||||||||||||||||||||
| M | N | ||||||||||||||||||||||||||||||||||||||
| CZ | D | E | G | Q | O | A | S | R | I | W | X | Y | |||||||||||||||||||||||||||
| C | Z | B | F | R0 | JT전 | P | U | ||||||||||||||||||||||||||||||||
| HV | JT | K | |||||||||||||||||||||||||||||||||||||
| H | V | J | T | ||||||||||||||||||||||||||||||||||||
미토콘드리아 이브는 모든 살아있는 인간의 가장 최근의 흔한 모계 조상인 여성에게 연구자들에 의해 붙여진 이름이다.
모집단의 정의

하플로그룹은 유전자 집단을 정의하는 데 사용될 수 있으며 종종 지리적으로 지향적이다.예를 들어 mtDNA 하플로그룹의 일반적인 분할은 다음과 같습니다.
- 아프리카: L0, L1, L2, L3, L4, L5, L6
- 서유라시아: H, T, U, V, X, K, I, J, W(목록된 모든 서유라시아 하플로그룹은 매크로 하플로그룹 [10]N에서 파생됨)
- 동유라시아: A, B, C, D, E, F, G, Y, Z(주: C, D, E, G, Z는 매크로 하플로그룹 M에 속함)
- 아메리칸 네이티브: A, B, C, D, X
- 오스트랄로-멜라네시아어: P, Q, S
미토콘드리아 하플로그룹은 세 개의 주요 그룹으로 나뉘는데, 이는 순차 문자 L, M, N으로 구분된다. 인류는 L0과 L1-6 사이에서 L 그룹 내에서 처음 분할되었다. L1-6은 다른 L 그룹, 그 중 하나인 L3을 M과 N 그룹으로 분할하였다.
M 그룹은 남부 해안 지역을 따라 동쪽으로 가는 경로를 따라 아프리카 밖에서 진화한 것으로 생각되는 인류 이동의 첫 물결을 이루고 있다.하플로그룹 M의 후손 혈통은 현재 아프리카 뿔과 북아프리카의 일부뿐만 아니라 아시아, 아메리카, 멜라네시아 전역에서 발견되고 있다; 유럽에서는 거의 발견되지 않았다.N 하플로그룹은 동쪽이 아닌 북쪽으로 향하는 아프리카 밖에서 진화한 또 다른 거시 혈통을 나타낼 수 있다.이행 직후 큰 R그룹은 N에서 분리되었습니다.
하플로그룹 R은 지리적 분포에 따라 정의된 두 개의 서브그룹으로 구성되어 있다.하나는 동남아시아와 오세아니아에서 발견되고 다른 하나는 거의 모든 현대 유럽 인구를 포함한다.하플로그룹 N(xR), 즉 N 그룹에 속하지만 R 하위 그룹에 속하지 않는 mtDNA는 호주 원주민 집단의 전형이며, 유라시아와 아메리카의 많은 인구 중 낮은 빈도로 존재한다.
L형은 거의 모든 아프리카인으로 구성되어 있다.
M 타입은 다음과 같이 구성됩니다.
M1 – 에티오피아, 소말리아 및 인도 인구.홍해와 아덴만 사이의 좁은 해협에 의해 분리된 아프리카의 뿔과 아라비아 반도(사우디아라비아, 예멘, 오만) 사이의 많은 유전자 흐름 때문일 것이다.
CZ – 많은 시베리아인; 지점 C – 일부 Amerindian; 지점 Z – 많은 Saami, 일부 한국인, 일부 북중국인, 일부 중앙아시아 인구.
D – 일부 미국계, 많은 시베리아인 및 동아시아 북부인
E – 말레이어, 보르네오어, 필리핀, 대만 원주민, 파푸아 뉴기니
G – 많은 북동부 시베리아인, 북동아시아인, 중앙아시아인
Q – 멜라네시아, 폴리네시아, 뉴기니 인구
N타입은 다음과 같이 구성됩니다.
A – 많은 미국인과 일부 동아시아인 및 시베리아인에서 발견됨
I – 북유럽, 동유럽에서 10 % 주파수
S – 일부 호주 원주민
W – 일부 동유럽인, 남아시아인 및 남아시아인
X – 일부 미국계, 남부 시베리아계, 남서 아시아계, 남부 유럽인
Y – 대부분의 니브흐족과 니아족. 많은 아이누스족, 퉁구스족, 오스트로네시안족.시베리아, 동아시아, 중앙아시아의 다른 집단에서도 빈도가 낮다.
R: N타입 내에서 발견된 대규모 그룹.여기에 포함된 개체군은 지리적으로 서유라시아와 동유라시아로 나눌 수 있다.오늘날 거의 모든 유럽 인구와 많은 수의 중동 인구가 이 분파에 포함되어 있다.다른 N타입 그룹에 포함되는 비율은 작습니다(위 참조).다음은 R의 서브레이드입니다.
B – 일부 중국인, 티베트인, 몽골인, 중앙아시아인, 한국인, 아메리카인, 남시베리아인, 일본인, 호주인
F – 주로 동남아시아, 특히 베트남에서 발견되며 크로아티아 [11]흐바르섬에서 8.3% 발견됩니다.
R0 – 아라비아, 에티오피아 및 소말리아에서 발견; HV 지점(지점 H; 지점 V) – 유럽, 서아시아, 북아프리카에서 발견;
JT 이전 – 레반트(현대 레바논 지역), 베두인 개체군의 25% 빈도로 발견; 지점 JT(지점 J; 지점 T) – 북유럽, 동유럽, 인더스, 지중해
U – 서유라시아, 인도 아대륙 및 알제리에서 발견되며, 인도에서 지중해 및 기타 유럽에서 발견됩니다. 특히 U5는 스칸디나비아와 발트해 국가에서 높은 빈도를 보이며, Sami족에서 가장 높은 빈도를 보입니다.
Y염색체 및 MtDNA 지리적 하플로그룹 할당
다음은 2013년 Bekada 등이 제안한 Y-염색체 및 MtDNA 지리적 하플로그룹 할당 목록이다.[12]
Y염색체
SNPS 하플로그룹에 따르면 첫 번째 멸종 사건의 나이는 45-50 킬로그룹 정도인 것으로 나타났다.말타에 따르면 두 번째 멸종 사건의 하플로그룹은 32-35 카야로 갈라지는 것으로 보였다.그라운드 제로 소멸 이벤트는 하플로그룹 CDEF*가 C, DE, F로 분산된 것처럼 보이는 Toba로 보입니다.C와 F는 공통점이 거의 없는 반면 D와 E는 공통점이 많다.현재의 추정에 따르면 멸종 사건 #1은 토바 이후에 발생했지만, 오래된 고대 DNA는 그라운드 제로 멸종 사건을 토바 훨씬 이전으로 밀어넣을 수 있었고, 첫 번째 멸종 사건을 이곳 토바에 다시 밀어넣을 수 있었다.멸종 이벤트 노트를 가진 하플로그룹은 기원이 불분명한데, 이는 멸종 이벤트가 심각한 병목현상을 일으키기 때문에 이들 그룹의 모든 노트는 추측에 불과하기 때문입니다.고대 DNA의 SNP 수치는 매우 가변적일 수 있습니다. 즉, 이 모든 집단이 비슷한 시기에 갈라졌음에도 불구하고 아무도 언제 [13][14]갈라질지 모른다는 것을 의미합니다.
| 기원. | 하플로그룹 | 마커 |
| 유럽(제2의 멸종 사건) | I | M170, M253, P259, M227, M507 |
| 유럽 | I1b | P215, M438, P37.2, M359, P41.2 |
| 유럽 | I1b2 | M26 |
| 유럽 | I1c | M223, M284, P78, P95 |
| 유럽 | J2a1 | M47 |
| 유럽 | J2a2 | M67, M166 |
| 유럽 | J2a2a | M92 |
| 유럽 | J2b | M12, M102, M280, M241 |
| 유럽 | R1b1b1a | M412, P310 |
| 유럽 | R1b1b1a1 | L11 |
| 유럽 | R1b1b1a1a | U106 |
| 유럽 | R1b1b1a1b | U198, P312, S116 |
| 유럽 | R1b1b1a1b1 | U152 |
| 유럽 | R1b1b1a1b2 | M529 |
| 유럽 | R1b1b1a1b3,4 | M65, M153 |
| 유럽 | R1b1b1a1b5 | SRY2627 |
| 남아시아 또는 멜라네시아 | C1(구칭 CxC3) | Z1426 |
| 북아시아 | C2(구칭 C3) | M217+ |
| 인도네시아 또는 남아시아(제1차 멸종 사건)? | F | M89, M282 |
| 유럽(카카스) (제2의 멸종 사건) | G | M201, M285, P15, P16, M406 |
| 남아시아 | H | M69, M52, M82, M197, M370 |
| 유럽 또는 중동(제2의 멸종 사건) | J1 | M304, M267, P58, M365, M368, M369 |
| 유럽 또는 중동(제2의 멸종 사건) | J2 | M172, M410, M158, M319, DYS445=6, M339, M340 |
| 유라시아의 버마 서부(제1차 멸종 사건)?[15] | ||
| 인도네시아(제1차 멸종 사건)[15] | K2(NOPS) | M526 |
| 남아시아 | L | M11, M20, M27, M76, M317, M274, M349, M357 |
| 동아시아, 동남아시아 | N | M231, M214, LLY22g, Tat, M178 |
| 동아시아, 동남아시아, 남아시아(제2의 멸종 사건) | O | M175, M119 |
| 인도네시아, 필리핀 (제1차 멸종 사건) | P(xQR) | 92R7, M207, M173, M45 |
| 남아시아, 시베리아(제2의 멸종 사건)? | R과 Q(QR) 분할 | MEH2, M242, P36.2, M25, M346 |
| 중동, 유럽, 시베리아, 남아시아 | R1a1 | M420, M17, M198, M204, M458 |
| 아나톨리아, 남동유럽? | R1b | M173, M343, P25, M73 |
| 유럽 | R1b1b | M269 |
| 유럽 | R1b1b1 | L23 |
| 파키스탄, 인도 (제2의 멸종 사건) | R2 | M479, M124 |
| 중동 | T | M70 |
| 북아프리카 | E1b1b1 | M35 |
| 북아프리카 | E1b1b1a | M78 |
| 서아시아 | E1b1b1a2 | V13 |
| 북아프리카 | E1b1b1a1 | V12 |
| 북아프리카 | E1b1b1a1b | V32 |
| 북아프리카 | E1b1b1a3 | V22 |
| 북아프리카 | E1b1b1a4 | V65 |
| 북아프리카 | E1b1b1b | M81 |
| 북아프리카 | E1b1b1c | M123, M34 |
| 서아프리카, 북아프리카 | A | M91, M13 |
| 동아프리카 | B | M60, M181, SRY10831.1, M150, M109, M112 |
| 아시아, 아프리카 | DE | M1, YAP, M174, M40, M96, M75, M98 |
| 동아시아, | D | M174 |
| 서아프리카(제1차 멸종 사건) | E1a | M33 |
| 동아프리카(첫 번째 멸종 이벤트는 E1b1과 E1a의 분할, 두 번째 멸종 이벤트는 E1b1b와 E1b1a의 분할) | E1b1 | P2, M2, U175, M191 |
| 중동 | R1b1a | V88, M18 |
mtDNA
| 기원. | 하플로그룹 |
| 유럽 | H1 |
| 유럽 | H11a |
| 유럽 | H1a |
| 유럽 | H1b |
| 유럽 | H2a |
| 유럽 | H3 |
| 유럽 | H5a |
| 유럽 | H6a |
| 유럽 | H7 |
| 유럽 | HV0/HV0a/V |
| 유럽 | I4 |
| 유럽 | J1c7 |
| 유럽 | J2b1 |
| 유럽 | T2b* |
| 유럽 | T2b4 |
| 유럽 | T2e |
| 유럽 | U4c1 |
| 유럽 | U5* |
| 유럽 | U5a |
| 유럽 | U5a1b1 |
| 유럽 | U5b* |
| 유럽 | U5b1b* |
| 유럽 | U5b1c |
| 유럽 | U5b3 |
| 유럽 | X2c'e |
| 중동 | I |
| 중동 | A |
| 중동 | B |
| 중동 | C/Z |
| 중동 | D/G/M9/E |
| 인도 | F |
| 중동 | H* |
| 중동 | H13a1 |
| 중동 | H14a |
| 중동 | H20 |
| 중동 | H2a1 |
| 중동 | H4 |
| 중동 | H6b |
| 중동 | H8 |
| 중동 | HV1 |
| 중동 | I1 |
| 중동 | J/J1c/J2 |
| 중동 | J1a'b'e |
| 중동 | J1b1a1 |
| 중동 | J1b2a |
| 중동 | J1d/J2b |
| 중동 | J1d1 |
| 중동 | J2a |
| 중동 | J2a2a1 |
| 중동 | K* |
| 중동 | K1a* |
| 중동 | K1b1* |
| 중동 | N1a* |
| 중동 | N1b |
| 중동 | N1c |
| 중동 | N2 |
| 중동 | N9 |
| 중동 | R* |
| 중동 | R0a |
| 중동 | T |
| 중동 | T1* |
| T1a | |
| 중동 | T2 |
| 중동 | T2c |
| 중동 | T2i |
| 중동 | U1* |
| 중동 | U2* |
| 중동 | U2e |
| 유라시아 | U3* |
| 중동 | U4 |
| 중동 | U4a* |
| 중동 | U7 |
| 중동 | U8* |
| 중동 | U9a |
| 중동 | X |
| 중동 | X1a |
| 중동 | X2b1 |
| 북아프리카 | L3e5 |
| 북아프리카 | M1 |
| 북아프리카 | M1a1 |
| 북아프리카 | U6a |
| 북아프리카 | U6a1'2'3 |
| 북아프리카 | U6 b'c'd |
| 동아프리카 | L0* |
| 동아프리카 | L0a1 |
| 동아프리카 | L0a1b |
| 동아프리카 | L0a2* |
| 동아프리카 | L3c/L4/M |
| 동아프리카 | L3d1a1 |
| 동아프리카 | L3d1d |
| 동아프리카 | L3e1* |
| 동아프리카 | L3f* |
| 동아프리카 | L3h1b* |
| 동아프리카 | L3i* |
| 동아프리카 | L3x* |
| 동아프리카 | L4a'b* |
| 동아프리카 | L5* |
| 동아프리카 | L6 |
| 동아프리카 | N*/M*/L3* |
| 서아프리카 | L1b* |
| 서아프리카 | L1b3 |
| 서아프리카 | L1c* |
| 서아프리카 | L1c2 |
| 서아프리카 | L2* |
| 서아프리카 | L2a |
| 서아프리카 | L2a1* |
| 서아프리카 | L2a1a2'3'4 |
| 서아프리카 | L2a1b |
| 서아프리카 | L2a1b'f |
| 서아프리카 | L2a1c1'2 |
| 서아프리카 | L2a1(16189) |
| 서아프리카 | L2a2 |
| 서아프리카 | L2b* |
| 서아프리카 | L2c1'2 |
| 서아프리카 | L2d |
| 서아프리카 | L2e |
| 서아프리카 | L3b |
| 서아프리카 | L3b1a3 |
| 서아프리카 | L3b(16124!) |
| 서아프리카 | L3b2a |
| 서아프리카 | L3d* |
| 서아프리카 | L3e2'3'4 |
| 서아프리카 | L3f1b* |
「 」를 참조해 주세요.
- 국제 햅맵 프로젝트
- 분자 진화
- 분자 계통학
- 인류의 진화 유전학
- 경주(생물학)/인종(인간의 범주화).
- 유전계보
- 족보 DNA 검사
- 유전적 족보 주제 목록.
- 저명한 사람들의 haplogroups의 목록.
레퍼런스
- ^ C. 배리 콕스, 피터 D까지무어, 리처드는 국자.Wiley-Blackwell, 2016년.아이 에스비엔 978-1-118-96858-1 페이지의 주 106이다. 생물 지리학:생태학과 진화 접근법이다.
- ^ 편집 위원회, V&, S출판사, 2012년 국제 표준 도서 번호 9381588643 우편 137.간명한 사전 과학의
- ^ Arora, Devender; Singh, Ajeet; Sharma, Vikrant; Bhaduria, Harvendra Singh; Patel, Ram Bahadur (2015). "Hgs Db: Haplogroups Database to understand migration and molecular risk assessment". Bioinformation. 11 (6): 272–75. doi:10.6026/97320630011272. PMC 4512000. PMID 26229286.
- ^ 국제 사회 유전자 계보의 2015년 유전학 용어.
- ^ "Y Chromosome Consortium". Archived from the original on 2017-01-16. Retrieved 2005-07-27.
- ^ Poznik, G. David; Xue, Yali; Mendez, Fernando L.; et al. (2016). "Punctuated bursts in human male demography inferred from 1,244 worldwide Y-chromosome sequences". Nature Genetics. 48 (6): 593–599. doi:10.1038/ng.3559. PMC 4884158. PMID 27111036.
- ^ a b Karmin, Monika; Saag, Lauri; Vicente, Mário; et al. (2015). "A recent bottleneck of Y chromosome diversity coincides with a global change in culture". Genome Research. 25 (4): 459–466. doi:10.1101/gr.186684.114. PMC 4381518. PMID 25770088.
- ^ Rootsi S, Magri C, Kivisild T, Benuzzi G, Help H, Bermisheva M, Kutuev I, Barać L, Pericić M, Balanovsky O, Pshenichnov A, Dion D, Grobei M, Zhivotovsky LA, Battaglia V, Achilli A, Al-Zahery N, Parik J, King R, Cinnioğlu C, Khusnutdinova E, Rudan P, Balanovska E, Scheffrahn W, Simonescu M, Brehm A, Goncalves R, Rosa A, Moisan JP, Chaventre A, Ferak V, Füredi S, Oefner PJ, Shen P, Beckman L, Mikerezi I, Terzić R, Primorac D, Cambon-Thomsen A, Krumina A, Torroni A, Underhill PA, Santachiara-Benerecetti AS, Villems R, Semino O (Jul 2004). "Phylogeography of Y-chromosome haplogroup I reveals distinct domains of prehistoric gene flow in europe" (PDF). American Journal of Human Genetics. 75 (1): 128–37. doi:10.1086/422196. PMC 1181996. PMID 15162323. Archived from the original (PDF) on 2009-06-24. Retrieved 2007-03-08.
- ^ "PhyloTree.org".
- ^ Loogväli EL, Roostalu U, Malyarchuk BA, Derenko MV, Kivisild T, Metspalu E, et al. (2004). "Disuniting uniformity: a pied cladistic canvas of mtDNA haplogroup H in Eurasia". Mol. Biol. Evol. 21 (11): 2012–21. doi:10.1093/molbev/msh209. PMID 15254257.
- ^ Tolk HV, Barac L, Pericic M, Klaric IM, Janicijevic B, Campbell H, Rudan I, Kivisild T, Villems R, Rudan P (Sep 2001). "The evidence of mtDNA haplogroup F in a European population and its ethnohistoric implications". European Journal of Human Genetics. 9 (9): 717–23. doi:10.1038/sj.ejhg.5200709. PMID 11571562.
- ^ Bekada A, Fregel R, Cabrera VM, Larruga JM, Pestano J, Benhamamouch S, González AM (2013). "Introducing the Algerian mitochondrial DNA and Y-chromosome profiles into the North African landscape". PLOS ONE. 8 (2): e56775. doi:10.1371/journal.pone.0056775. PMC 3576335. PMID 23431392.
- ^ "Common genetic ancestors lived during roughly same time period". 1 Aug 2013. Retrieved 23 Jan 2015.
- ^ Raghavan M, Skoglund P, Graf KE, Metspalu M, Albrechtsen A, Moltke I, Rasmussen S, Stafford TW, Orlando L, Metspalu E, Karmin M, Tambets K, Rootsi S, Mägi R, Campos PF, Balanovska E, Balanovsky O, Khusnutdinova E, Litvinov S, Osipova LP, Fedorova SA, Voevoda MI, DeGiorgio M, Sicheritz-Ponten T, Brunak S, Demeshchenko S, Kivisild T, Villems R, Nielsen R, Jakobsson M, Willerslev E (Jan 2014). "Upper Palaeolithic Siberian genome reveals dual ancestry of Native Americans". Nature. 505 (7481): 87–91. doi:10.1038/nature12736. PMC 4105016. PMID 24256729.
- ^ a b c Karafet TM, Mendez FL, Sudoyo H, Lansing JS, Hammer MF (Mar 2015). "Improved phylogenetic resolution and rapid diversification of Y-chromosome haplogroup K-M526 in Southeast Asia". European Journal of Human Genetics. 23 (3): 369–73. doi:10.1038/ejhg.2014.106. PMC 4326703. PMID 24896152.
외부 링크
장군
모든 DNA 하플로그룹
Y염색체
Y염색체DNA하플로그룹
- Y염색체 컨소시엄
- ISOGG Y-DNA 하플로그룹 트리
- PhyloTree의 Y-tree 인간 Y-염색체를 위한 최소한의 기준 계통 발생
- 하플로그룹 프레딕터
- Y염색체 컨소시엄(2002), 인간 Y염색체 이원 하플로그룹 나무의 명명 시스템, 게놈 연구, Vol. 12(2), 339-48, 2002년 2월. (상세 계층도는 이전 명명 체계에서 변환되었다.)
- 세미노 등(2000),현존 유럽인 사이언스 제290권 ('유' 하플로그룹을 소개한 논문)의 구석기 호모 사피엔스 사피엔스의 유전적 유산.
- Y-DNA Etnographic 및 Genographic Atlas 및 오픈 소스 데이터 컴파일
미토콘드리아 DNA 하플로그룹
- PhyloTree – 인간 미토콘드리아 DNA[1] 변이의 계통수
- PhyloD3 – PhyloTree 기반의 D3.js 기반 계통수
- MitoTool – 인간 미토콘드리아 DNA 배열 변이 분석 및 검색용 웹 서버
- HapploGrep – 웨이백 머신에서 아카이브된 PhyloTree에 기반한 미토콘드리아 DNA 하플로그룹 자동분류 2016-06-12
- HappoFind – 인간 미토콘드리아 DNA에 대한 고속 자동 하플로그룹 할당 파이프라인
- 그래피컬 mtDNA 하플로그룹 골격
- 아프리카 mtDNA 풍경 만들기
- mtDNA 하플로그룹 L2의 4개 클래드는 다른 속도로 진화합니까?