사람의 유전적 변이
Human genetic variation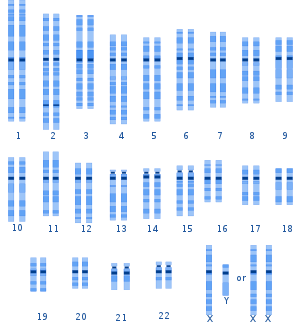

인간의 유전적 변이는 인구와 인구 사이의 유전적 차이입니다.인간 집단(알레알레알)에는 주어진 유전자의 다양한 변형이 있을 수 있는데, 이는 다형성이라고 불리는 상황입니다.
어떤 두 사람도 유전적으로 똑같지 않습니다.심지어 단일접합성 쌍둥이(하나의 접합체에서 발달하는)도 발달 중에 발생하는 돌연변이와 유전자 복사 수 변화로 인해 드물게 유전적 차이가 있습니다.[1]개인 간의 차이, 심지어 밀접한 관련이 있는 개인 간의 차이는 유전자 지문과 같은 기술의 핵심입니다.
인간 게놈은 세포성 미토콘드리아에 있는 17,000bp 미만의 DNA 뿐만 아니라 46개의 DNA 염색체에 걸쳐 약 32억개의 염기쌍을 가지고 있습니다.2015년, 개인의 게놈과 기준 게놈의 일반적인 차이는 2천만 개의 염기쌍(전체의 0.6%)으로 추정되었습니다.[2]2017년 기준으로, 서열화된 인간 유전체로부터 총 3억 2,400만 개의 알려진 변이체가 있습니다.[3]
비교적으로 말하면, 인간은 유전적으로 동질적인 종입니다.비록 소수의 유전자 변형이 특정한 지리적 지역에서 더 자주 발견되거나 그 지역 출신의 조상을 가진 사람들에게서 더 많이 발견되지만, 이 변형은 인간 게놈 가변성의 작은 부분(~15%)을 차지합니다.대부분의 변동은 각 인구 집단의 구성원 내에 존재합니다.비교를 위해, 붉은 마카크 원숭이는 인간에 비해 2.5배 더 큰 DNA 서열 다양성을 보여줍니다.[4]
인간 집단 간 유전적 거리의 불연속성, 인간 종에 분리된 가지의 부재, 그리고 전 세계적으로 인간의 현저한 동질성은 인간의 인종이나 아종을 추론할 수 있는 과학적 근거가 없다는 것을 의미하며, 대부분의 특성에 대해 그들 사이보다 집단 내에서 훨씬 더 많은 차이가 있다는 것을 의미합니다.[5][6][7][8][9][10][11][12]그럼에도 불구하고, 현대의 유전학 연구는 피부색, 신체 치수, 락토오스와 전분 소화, 높은 고도의 적응, 그리고 특정한 질병을 발병시키는 성향과 같은 특성에서 인간 인구에 걸쳐 상당한 평균 유전적 차이를 발견했습니다.[11]가장 큰 다양성은 아프리카의 인구 내부와 인구 사이에서 발견되며,[13] 인류 기원에 대한 아웃 오브 아프리카 이론과 일치하는 아프리카 대륙과의 거리가 증가함에 따라 점차 감소합니다.[14]
인간의 유전적 변이에 대한 연구는 진화론적 의미와 의학적 응용을 가지고 있습니다.과학자들이 과거 인류 이동의 패턴을 재구성하고 이해하는 데 도움을 줄 수 있습니다.의학에서는 질병을 유발하는 대립유전자가 특정 집단에서 더 자주 발생하기 때문에 인간의 유전자 변이에 대한 연구가 중요할 수 있습니다.예를 들어, 낫 세포 빈혈에 대한 돌연변이는 말라리아를 옮기는 모기의 진화적 압력으로 인해 특정 사하라 사막 이남의 아프리카, 남유럽, 아라비아, 그리고 인도 인구의 조상을 가진 사람들에게서 더 자주 발견됩니다.
새로운 연구 결과는 각 인간이 그들의 부모에 비해 평균 60개의 새로운 돌연변이를 가지고 있다는 것을 보여줍니다.[15][16]
변동원인
개체 간 차이의 원인에는 독립적인 분류, 생식 중 유전자의 교환(메이오시스를 통해) 및 다양한 돌연변이 사건이 포함됩니다.
모집단 사이에 유전적 변이가 존재하는 이유는 최소한 세 가지입니다.대립 유전자가 경쟁 우위를 제공한다면 자연 선택은 특정 환경에 있는 개인에게 적응 우위를 부여할 수 있습니다.선택 중인 대립 유전자는 이점을 제공하는 지리적 영역에서만 발생할 가능성이 높습니다.두 번째 중요한 과정은 대부분의 돌연변이가 중립적인 조건에서 유전자 풀의 무작위적인 변화의 영향인 유전적 드리프트입니다(즉, 그들은 유기체에 어떠한 긍정적이거나 부정적인 선택적인 영향을 미치지 않는 것으로 보입니다).마지막으로, 소규모 이주 인구는 그들이 기원한 전체 인구와 통계적인 차이가 있습니다. 이 이주자들이 새로운 지역에 정착할 때, 그들의 후손 인구는 전형적으로 그들의 출신 인구와 다릅니다. 다른 유전자가 우세하고 유전적으로 덜 다양합니다.
인간의 경우, 주요 원인은 유전적 이동입니다.[17]연속적인 창시자 효과와 과거의 작은 모집단 크기(유전적 이동 가능성 증가)는 모집단 간의 중립적인 차이에 중요한 영향을 미쳤을 수 있습니다.[citation needed]유전자 변이의 두 번째 주요 원인은 대부분의 돌연변이의 높은 중립성 때문입니다.작지만 상당한 수의 유전자들이 최근 자연 선택을 거친 것으로 보이며, 이러한 선택적 압력은 때때로 한 지역에 특정적입니다.[18][19]
변동의 측도
인간 핵형의 총체적인 변화에서부터 단일 뉴클레오타이드 변화에 이르기까지 인간 사이의 유전적 변이는 많은 규모에서 발생합니다.[20]160명의 살아있는 사람의 출생 중 1명에게서 염색체 이상이 검출됩니다.성염색체 장애를 제외하고, 대부분의 유엽은 발달하는 태아의 사망을 초래합니다; 살아있는 출생자들 중에서 가장 흔한 상염색체외 염색체는 21, 18 그리고 13입니다.[21]
뉴클레오티드 다양성은 두 개체 사이에 다른 뉴클레오티드의 평균 비율입니다.2004년 기준, 인간 뉴클레오타이드 다양성은 염기쌍의 0.1%[22] 내지 0.4%로 추정되었습니다.[23]2015년, 26개의 인간 모집단으로부터 1,000개의 개체를 염기서열 분석한 1000 게놈 프로젝트는 "일반적인 [개별] 게놈은 410만에서 5,000만 부위의 기준 인간 게놈과 다릅니다… 2,000만 염기서열에 영향을 미칩니다"; 후자의 수치는 전체 염기쌍 수의 0.6%에 해당합니다.[2]이러한 사이트의 거의 대부분(99.9%)은 유전자 서열에서 단일 뉴클레오티드 다형성 또는 간단한 삽입 또는 삭제(인델)와 같은 작은 차이이지만 구조적 변화는 SNP 및 인델보다 더 많은 수의 기본 쌍을 차지합니다.[2][24]
SNP 및 기타 변종을 나열하는 단일 뉴클레오티드 다형성 데이터베이스(dbSNP)는 2017년[update] 기준으로 서열화된 인간 게놈에서 발견된 3억 2,400만 개의 변종을 나열했습니다.[3]
단일 뉴클레오티드 다형성

단일 뉴클레오티드 다형성(single nucleotide polymorphism, SNP)은 인구의 최소 1%에서 발생하는 한 종의 구성원 간의 단일 뉴클레오티드의 차이입니다.이 중 1000 유전체 프로젝트로 특징지어지는 2504명의 SNP는 8470만개였습니다.[2]SNP는 가장 일반적인 유형의 염기서열 변이로, 1998년에 모든 염기서열 변이의 90%를 차지할 것으로 추정되었습니다.[25]다른 시퀀스 변형으로는 단일 베이스 교환, 삭제 및 삽입이 있습니다.[26]SNP는 평균적으로 약 100에서 300개의 베이스[27] 당 발생하며, 이질성의 주요 원인이기도 합니다.
기능적이거나 동의어가 아닌 SNP는 유전자 접합 또는 메신저 RNA와 같은 일부 요소에 영향을 미치는 것이므로 종 구성원 간의 표현형 차이를 유발합니다.인간 SNP의 약 3~5%가 기능적입니다(International HapMap Project(국제 합맵 프로젝트)중성 또는 동의어인 SNP는 순수한 수와 세대에 걸쳐 안정적으로 물려받기 때문에 여전히 유전체 전반의 연관 연구에서 유전적 지표로 유용합니다.[25]
코딩 SNP는 유전자 안에서 일어나는 것입니다.103개 유전자에서 조기 정지 코돈을 초래하는 인간 참조 SNP는 105개입니다.이는 코딩 SNP의 0.5%에 해당합니다.유전체의 분절적 복제로 인해 발생합니다.이러한 SNP는 단백질의 손실을 초래하지만, 이러한 모든 SNP 대립유전자는 일반적이며 음성 선택에서는 정제되지 않습니다.[28]
구조변화
구조적 변이는 유기체의 염색체 구조의 변이입니다.복사 수 변동 및 삭제, 반전, 삽입 및 복제와 같은 구조적 변동은 단일 뉴클레오티드 다양성보다 훨씬 더 많은 인간의 유전적 변동을 설명합니다.이것은 2007년 Craig Venter와 James D, 두 사람의 게놈의 이배체 전체 서열의 분석으로부터 결론을 내렸습니다. 왓슨.이것은 Human Genome Project와 Celera Genomics에 의해 각각 발표된 많은 개인들의 서열의 합병인 두 개의 반배체 서열에 추가되었습니다.[29]
1000 게놈 프로젝트에 따르면, 일반적인 인간은 약 1,000개의 대규모 삭제, 160개의 복사 수 변형, 915개의 Alu 삽입, 128개의 L1 삽입, 51개의 SVA 삽입, 4개의 NUMT 및 10개의 반전을 포함하는 2,100개에서 2,500개의 구조적 변형을 가지고 있습니다.[2]
복사번호변동
카피-넘버 변이(copy-number variation, CNV)는 일부 염색체의 DNA 영역을 삭제하거나 복제하여 게놈의 차이입니다.관련이 없는 인간의 유전체 중 0.4%가 복사 수에 따라 다른 것으로 추정됩니다.복사 수 변이를 포함할 경우 사람 간 유전자 변이는 최소 0.5%(99.5% 유사도)로 추정됩니다.[30][31][32][33]복사 번호 변동은 상속되지만 개발 중에 발생할 수도 있습니다.[34][35][36][37]
프라타스 등은 50k의[38] 네안데르탈인에 비해 현대-인간 기준 어셈블리의 게놈 변이가 큰 지역을 포함하는 시각 지도를 구축했습니다.[39]
후생유전학
후성유전학적 변이는 DNA에 붙어 유전자가 어떻게 읽히는지에 영향을 미치는 화학 태그의 변이입니다."후성유전자 표시라고 불리는 이 꼬리표는 유전자가 어떻게 읽힐 수 있는지를 조절하는 스위치 역할을 합니다."[40]어떤 대립유전자에서 DNA의 후생유전학적 상태와 관련 표현형은 개인의 세대에 걸쳐 유전될 수 있습니다.[41]
유전적 변이성
유전적 변이성(genetic variability)은 모집단에서 개별 유전자형들이 서로 다르게 변화하는 경향을 나타내는 척도입니다.변이성은 특정 모집단에서 볼 수 있는 변이량인 유전적 다양성과는 다릅니다.특성의 가변성은 환경 및 유전적 영향에 따라 특성이 얼마나 다양한 경향을 갖는지에 대한 것입니다.
클라인스
생물학에서 클라인(cline)은 생물학에서 생물의 종, 개체군, 품종 또는 형태의 연속체로, 지리적 영역에 걸쳐 점진적인 표현형 및/또는 유전적 차이를 보이며, 일반적으로 환경적 이질성의 결과입니다.[42][43][44]인간의 유전적 변이에 대한 과학적 연구에서, 유전자 계통은 엄격하게 정의되고 정량적 지표에 따를 수 있습니다.
하플로그룹
분자 진화 연구에서 하플로그룹은 단일 뉴클레오티드 다형성(SNP) 돌연변이와 공통 조상을 공유하는 유사한 하플로타입의 그룹입니다.하플로그룹에 대한 연구는 수천 년 전으로 거슬러 올라가는 조상의 기원에 대한 정보를 제공합니다.[45]
가장 일반적으로 연구되는 인간 하플로그룹은 Y-염색체(Y-DNA) 하플로그룹과 미토콘드리아 DNA(mtDNA) 하플로그룹으로, 둘 다 유전적 집단을 정의하는 데 사용될 수 있습니다.Y-DNA는 아버지에서 아들로, Y-DNA는 어머니에서 딸 또는 아들 모두로, MtDNA는 어머니에서 어머니로, 또는 아들 모두로, 모선을 따라 전달됩니다.Y-DNA와 mtDNA는 각 세대에서 우연히 돌연변이에 의해 변화할 수 있습니다.
가변수 탠덤 반복측정
가변 수 탠덤 반복(VNTR)은 탠덤 반복의 길이 변화입니다.연쇄반복이란 짧은 뉴클레오타이드 서열의 인접한 반복입니다.탠덤 반복은 많은 염색체에 존재하며, 그들의 길이는 개체마다 다릅니다.각 변종은 유전자의 역할을 하므로 개인 또는 부모 식별에 사용됩니다.그들의 분석은 유전학과 생물학 연구, 포렌식 그리고 DNA 지문 채취에 유용합니다.
짧은 탠덤 반복(약 5개의 염기쌍)은 마이크로위성이라고 불리고, 긴 것은 미니위성이라고 불립니다.
역사와 지리적 분포
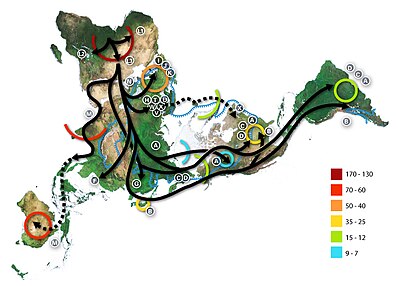

현생 인류의 최근 아프리카 기원
현대 인류 패러다임의 최근 아프리카 기원은 해부학적으로 70,000년 전 이후에 아프리카인이 아닌 현대 인류의 인구가 분산된 것을 가정합니다.아프리카 내의 분산은 적어도 13만년 전에 상당히 일찍 발생했습니다."아웃 오브 아프리카" 이론은 찰스 다윈의 인간의 하강에서 잠정적으로 제안된 것으로 19세기에 시작되었지만,[46] 1980년대에 미토콘드리아 DNA의 연구에 의해 뒷받침되고 고대 표본의 물리적 인류학의 증거가 결합될 때까지 추측으로 남아 있었습니다.
Y-염색체 염기서열 변이에 대한 2000년 연구에 따르면,[47] 인간 Y-염색체는 아프리카의 조상을 추적하고, 파생된 혈통의 후손들은 아프리카를 떠나 유라시아의 고대 인간 Y-염색체로 대체되었습니다.이 연구는 또한 동아프리카와 코이산의 소수의 동시대 인구가 35,000년에서 89,000년 전에 아프리카를 떠난 해부학적으로 현대 인류의 가장 조상적인 혈통의 후손이라는 것을 보여줍니다.[47]이 이론을 뒷받침하는 다른 증거는 두개골 측정의 변화가 유전적 다양성의 감소와 같은 비율로 아프리카로부터 멀어질수록 감소한다는 것입니다.인류의 유전적 다양성은 아프리카로부터 이주 거리가 멀어질수록 토착 인구에서 감소하는데, 이는 일시적으로 인구 규모를 감소시키는 사건인 인간 이주 과정에서 병목 현상이 발생하기 때문으로 판단됩니다.[48][49]
2009년 유전자 군집 연구에서 다양한 아프리카 개체군에서 1327개의 다형성 표지를 유전자형으로 유형화하여 6개의 조상 군집을 확인했습니다.군집은 민족, 문화, 언어와 밀접하게 일치했습니다.[50]2018년 전 세계 인구를 대상으로 한 유전체 염기서열 분석 연구에서는 아프리카 인구들 사이에서 비슷한 군집을 관찰했습니다.K=9에서는 북아프리카와 북동아프리카에 거주하는 아프리카어를 사용하는 인구, 북동아프리카와 동아프리카의 닐로-사하라어를 사용하는 인구, 북동아프리카의 아리 인구, 서-중앙아프리카의 니제르-콩고어를 사용하는 인구,동아프리카와 남아프리카, 중앙아프리카의 피그미 개체군, 남아프리카의 코이족 개체군.[51]
2023년 5월, 과학자들은 유전자 연구를 바탕으로 이전에 이해되었던 것보다 더 복잡한 인간 진화의 경로를 보고했습니다.연구에 의하면, 인간은 단일 장소와 기간이 아니라 아프리카의 다른 장소와 시간에서 진화했다고 합니다.[52][53]
집단유전학
모든 인간의 공통적인 조상 때문에, 소수의 변종만이 모집단 간의 빈도에 큰 차이가 있습니다.그러나, 세계 인구에서 일부 희귀한 변종들은 적어도 한 인구에서 훨씬 더 자주 발생합니다. (5% 이상).[54]

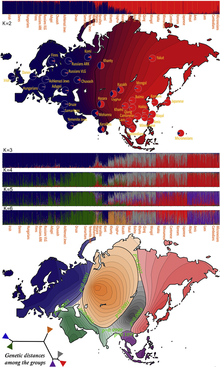
초기 인류가 아프리카를 떠났기 때문에 약 10만 년 전(대략 3,000 세대) 아프리카-유라시아 분열 이전에 인구 병목 현상을 겪었을 것으로 일반적으로 추정됩니다.이전에 적은 인구의 급속한 팽창은 유전자 변이의 분포에 두 가지 중요한 영향을 미칩니다.첫째, 창업자 집단이 조상 집단으로부터 유전적 변이의 부분만 가져올 때 소위 창업자 효과가 발생합니다.둘째, 창업자들이 지리적으로 더 분리됨에 따라, 서로 다른 창업자 집단의 두 사람이 짝짓기를 할 확률은 더 작아집니다.이 다양한 짝짓기의 효과는 지리적 집단 간의 유전자 흐름을 줄이고 집단 간의 유전적 거리를 늘리는 것입니다.[citation needed]
아프리카에서 온 인간의 확대는 두 가지 다른 방법으로 유전적 변이의 분포에 영향을 미쳤습니다.첫째, 작은 (설립자) 집단은 중성 다형성의 증가된 변동으로 인해 더 큰 유전적 이동을 경험합니다.둘째, 한 그룹에서 발생한 새로운 다형성은 유전자 흐름이 제한됨에 따라 다른 그룹으로 전달될 가능성이 적었습니다.[citation needed]
아프리카의 인구는 아프리카 밖의 인구보다 연관 불균형의 양이 적은 경향이 있는데, 이는 부분적으로 인류 역사의 과정에서 아프리카에 있는 인간 인구의 규모가 더 크기 때문이기도 하고, 부분적으로 세계의 나머지 지역을 식민지로 만들기 위해 아프리카를 떠난 현대 인류의 수가 상대적으로 적었기 때문이기도 합니다.[56]대조적으로 과거에 급격한 크기 감소 또는 급속한 팽창을 겪은 집단과 이전에 분리된 조상 집단의 혼합으로 형성된 집단은 비정상적으로 높은 수준의 연결 불균형을[56] 가질 수 있습니다.
변동분포

"집단"의 정의의 어려움, 변이의 임상적 성격, 게놈 전체의 이질성 때문에 인간 집단 내 및 집단 간의 유전적 변이 분포는 간결하게 설명할 수 없습니다(Long and Kittles 2003).그러나 일반적으로 유전자 변이의 평균 85%는 지역 인구 내에 존재하며 ~7%는 같은 대륙 내 지역 인구 간에 존재하며 ~8%의 변이는 다른 대륙에 사는 대규모 집단 간에 발생합니다.[57][58]인간에 대한 최근의 아프리카 기원 이론은 아프리카에는 다른 곳보다 훨씬 더 많은 다양성이 존재하며 아프리카에서 표본이 추출될수록 다양성이 감소할 것이라고 예측할 것입니다.
표현형 변이
사하라 사막 이남의 아프리카는 가장 많은 인간의 유전적 다양성을 가지고 있으며 두개골 형태의 표현형 변화에도 동일한 것으로 나타났습니다.[48][59]표현형은 유전자 발현을 통해 유전자형과 연결됩니다.유전적 다양성은 많은 과학자들이 현생 인류의 기원이라고 믿는 그 지역으로부터의 이동 거리에 따라 순조롭게 감소하고, 그 감소는 표현형 변이의 감소에 의해 반영됩니다.두개골 측정은 아프리카에서 멀어질수록 모집단 내 변동이 감소하는 물리적 속성의 한 예입니다.
많은 신체적 특성의 분포는 인간 집단 내와 집단 간의 유전적 변이의 분포와 유사합니다(미국 물리인류학협회 1996; Keita and Kittles 1997).예를 들어, 인간의 머리 모양 변화의 ~90%는 대륙 그룹 내에서 발생하며, ~10%는 그룹을 분리하며, 최근 아프리카 조상을 가진 개인들 사이에서 머리 모양의 더 큰 변동성을 갖습니다(Lelthford 2002).
집단 내 및 집단 간 신체적 특성의 공통적인 분포에 대한 두드러진 예외는 피부색입니다.피부색의 변화의 약 10%는 그룹 내에서 발생하며, ~90%는 그룹 간에서 발생합니다(Lelthford 2002).이러한 피부색의 분포와 그것의 지리적 패턴 – 주로 적도 근처에 살았던 조상들이 주로 고위도에 살았던 조상들보다 어두운 피부를 가졌다는 – 은 이 속성이 강한 선택적 압력을 받아왔다는 것을 나타냅니다.피부가 어두운 것은 적도 지역에서 햇볕에 타는 것, 피부암, 엽산의 광분해, 땀샘의 손상을 예방하기 위해 강하게 선택되는 것으로 보입니다.[60]
인구의 유전적 다양성이 다양한 유전자 발현 수준에 어떤 영향을 미치는지를 이해하는 것은 활발한 연구 분야입니다.이전의 연구들이 DNA 변이와 RNA 발현 사이의 관계에 초점을 맞춘 반면, 최근의 노력들은 염색질 상태,[61] 번역,[62] 그리고 단백질 수준을 포함한 유전자 발현의 다양한 측면들의 유전자 조절을 특징짓고 있습니다.[63]2007년에 발표된 한 연구는 유전자의 25%가 유럽과 아시아 혈통의 인구 사이에 다른 수준의 유전자 발현을 나타냈다는 것을 발견했습니다.[64][65][66][67][68]이러한 유전자 발현 차이의 주요 원인은 DNA의 유전자 조절 영역에서의 SNPs라고 생각되었습니다.2007년에 발표된 또 다른 연구에서는 유전자의 약 83%가 개인 간에, 그리고 유럽과 아프리카 혈통의 인구 간에 약 17%가 다른 수준으로 발현된다는 것을 발견했습니다.[69][70]
Wright의 변동성 측정치 고정지수
모집단 유전학자인 Sewall Wright는 모집단 간의 유전적 차이를 측정하는 방법으로 고정 지수(종종 F로ST 약칭됨)를 개발했습니다.이 통계량은 개별 유전자 또는 많은 유전자에 대한 모집단 간 및 모집단 간의 유전적 차이를 동시에 측정함으로써 주어진 두 모집단 간의 차이를 비교하기 위해 분류학에서 자주 사용됩니다.[71]인간의 고정 지수는 약 0.15라고 종종 언급됩니다.이는 전체 인구에서 측정된 변이의 약 85%가 동일한 모집단의 개체 내에서 발견되며, 변이의 약 15%가 모집단 간에 발생한다는 것을 의미합니다.이러한 추정치는 서로 다른 모집단의 두 개체가 자신의 그룹에 속한 개체보다 서로 더 유사할 수 있음을 의미합니다.[72][73]"살아있는 인간의 공통된 진화 역사는 모든 살아있는 사람들 사이에서 높은 관련성을 가져왔는데, 예를 들어 살아있는 인간 집단 사이의 매우 낮은 고정 지수(FST)에서 알 수 있습니다."이러한 비율을 확인한 Richard Lewontin은 따라서 "인종"이나 "아종" 모두 인간 개체수를 묘사하는 적절하거나 유용한 방법이 아니라고 결론 내렸습니다.[57]
Wright 자신은 0.25 이상의 값은 매우 큰 유전적 변이를 나타내고 0.15–0.25의 F는ST 큰 변이를 나타낸다고 믿었습니다.그러나, 인간 변이의 약 5%가 대륙 내 인구 사이에서 발생하므로, 일부 연구에서 인간(또는 인종)의 대륙 그룹 간 FST 값이 0.1(또는 가능하면 더 낮은)로 발견되었으며, 이는 더 중간 수준의 유전자 변이를 시사합니다.[71]Graves(1996)는 비록 Wright(1978)도 참조하지만,[71] 이 통계는 모집단 간의 분화 정도를 측정하는 데 사용되기 때문에 F가ST 아종 상태의 표시로 사용되어서는 안 된다고 반박했습니다.[74]
제프리 롱(Jeffrey Long)과 릭 키틀즈(Rick Kittles)는 2003년 논문 "인간의 유전적 다양성과 생물학적 인종의 비존재"에서 인간 집단에 대한 F의ST 적용에 대해 긴 비판을 하고 있습니다.그들은 85%라는 수치는 모든 인간의 인구가 모든 유전적 다양성의 평균 85%를 포함하고 있다는 것을 암시하기 때문에 오해의 소지가 있다는 것을 발견했습니다.그들은 기본 통계 모형이 각 대규모 인구 집단에 대해 동일하고 독립적인 변동 이력을 잘못 가정하고 있다고 주장합니다.더 현실적인 접근법은 일부 인간 그룹이 다른 그룹의 부모이고 이러한 그룹이 그들의 혈통 그룹에 대한 부갑상선 그룹을 나타낸다는 것을 이해하는 것입니다.예를 들어, 최근의 아프리카 기원 이론에 따르면, 아프리카의 인류는 모든 다른 인류 집단에 대해 가류적입니다. 왜냐하면 그것은 모든 비아프리카 인구가 유래한 조상 집단을 대표하지만, 그 이상의 비아프리카 집단은 이 아프리카 인구의 작은 비대표 표본에서만 유래하기 때문입니다.이것은 모든 비아프리카 집단이 다른 집단보다 서로, 그리고 일부 아프리카 집단(아마도 동아프리카인)과 더 밀접한 관련이 있다는 것을 의미하며, 더 나아가 아프리카의 이주는 유전적 병목 현상을 나타냈으며, 아프리카에 존재했던 다양성의 많은 부분이 이주 집단에 의해 아프리카 밖으로 이행되지 않았다는 것을 의미합니다.이 시나리오 하에서, 인간의 인구는 동일한 양의 지역적 변동성을 가지고 있지 않고, 오히려 아프리카에서 더 멀리 떨어진 어떤 인구가 살고 있을 때보다 감소된 양의 다양성을 가지고 있습니다.Long and Kittles는 인간 유전적 다양성의 85%가 모든 인간 인구에 존재하는 것이 아니라, 인간 유전적 다양성의 약 100%가 단일 아프리카 인구에 존재하는 반면, 뉴기니 출신 인구에는 인간 유전적 다양성의 약 70%만이 존재한다는 것을 발견했습니다.롱과 키틀즈는 이것이 여전히 다른 포유류 개체수와 비교하여 유전적으로 동질적인 전세계적인 인간 개체수를 만들어 낸다고 주장했습니다.[75]
고대혼합물
해부학적으로 현대의 인류는 중기 구석기 시대에 네안데르탈인과 교배했습니다.2010년 5월, 네안데르탈인 게놈 프로젝트는 네안데르탈인 혼혈이 일어났고, 현생 유라시아인과 오세아니아인의 DNA에 2-4% 정도의 작지만 중요한 네안데르탈인 혼혈이 존재한다는 유전적 증거를 제시했습니다.[76][77]
멜라네시아인의 게놈(파푸아뉴기니인과 부건빌 섬 주민으로 대표됨)의 4%에서 6% 사이는 데니소반스에서 유래한 것으로 보이며, 이전에는 사피엔스보다 네안데르탈인과 더 가까운 관계에 있습니다.멜라네시아인의 조상들이 동남아시아로 이주한 초기에 도입되었을 가능성이 있습니다.이 상호작용의 역사는 데니소반인들이 한때 동아시아 전역에 광범위하게 분포했음을 암시합니다.[78]
그러므로 멜라네시아인들은 데니소반/네안데르탈인 관련 혼합물이 ~8%[78]로 가장 오래된 혼합 집단 중 하나로 부상합니다.
2013년에 발표된 연구에서 캘리포니아 대학의 제프리 월은 전체 염기서열 게놈 데이터를 연구했고 유럽인에 비해 아시아인의 내성률이 더 높다는 것을 발견했습니다.[79]Hammer et al. 은 현대의 아프리카 게놈이 고대 인류 조상들과 유전자 흐름의 특징을 가지고 있다는 가설을 시험했고 일부 아프리카 그룹의 게놈에서 고대 혼합물의 증거를 발견했으며 해부학적으로 현대 인류의 진화 동안 적당한 양의 유전자 흐름이 시공간을 통해 널리 퍼졌음을 시사했습니다.[80]
2020년에 발표된 한 연구에 따르면, 서아프리카의 요루바와 멘데 개체군은 그들의 게놈의 2%에서 19% 사이에서 현대 인류와 네안데르탈인과 데니소반인의 조상이 갈라지기 전에 갈라졌을 가능성이 있는 아직 밝혀지지 않은 고대 호미닌 개체군에서 유래한 것으로 밝혀졌습니다.[81]잠재적으로 이 집단들을 지금까지 확인된 가장 오래된 adm 혼합 인간 집단으로 만듭니다.
세계인구분류

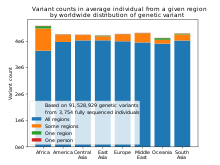
인간의 유전적 변이에 대한 새로운 데이터는 인간을 인종으로 분류하는 가능한 생물학적 근거에 대한 논쟁을 재점화시켰습니다.대부분의 논란은 유전자 데이터를 어떻게 해석할 것인가, 그에 근거한 결론이 타당한가의 문제를 둘러싼 것입니다.일부 연구원들은 자가 확인된 인종이 특정한 건강 위험과 약물에 대한 지리적 조상을 나타내는 지표로 사용될 수 있다고 주장합니다.
인간 그룹 간의 유전적 차이는 비교적 작지만, 혈통 정보 마커(AIM)라고 불리는 더피, ABCC11, SLC24A5와 같은 특정 유전자의 이러한 차이는 광범위하고 지리적으로 기반을 둔 그룹 내에서 많은 개인을 안정적으로 배치하는 데 사용될 수 있습니다.예를 들어, 전 세계적으로 분포된 집단에서 샘플링된 수백 개의 다형성 위치의 컴퓨터 분석은 역사적으로 큰 대륙 및 아대륙 지역을 차지한 집단과 대략적으로 관련된 유전적 군집의 존재를 밝혀냈습니다(Rosenberg et al. 2002; Bamshad et al. 2003).
일부 평론가들은 이러한 변이 패턴이 전통적인 인종 범주의 사용에 대한 생물학적 정당성을 제공한다고 주장했습니다.그들은 대륙 군집이 인간을 사하라 이남의 아프리카인, 유럽인, 서아시아인, 중앙아시아인, 남아시아인, 그리고 북아프리카인, 동아시아인, 동남아시아인, 폴리네시아인, 그리고 아메리카 원주민들, 그리고 오세아니아의 다른 거주자들(멜라네시아인, 미크로네시아인, 그리고 오스트레일리아인)로 나누는 것과 대략 일치한다고 주장합니다.borigines)(Risch et al. 2002).다른 관찰자들은 같은 자료가 인종 집단에 대한 전통적인 개념을 약화시킨다고 말하면서 동의하지 않습니다. (킹 앤 모툴스키 2002; 칼라펠 2003; 티슈코프와 키드 2004[23])예를 들어 인종 또는 인종 내 하위 그룹으로 간주되는 주요 모집단이 반드시 자신의 군집을 형성하지는 않는다는 점을 지적합니다.
한 지역에 국한된 유전적 변이는 그 지역 내에서 드물고, 한 지역 내에서 흔히 볼 수 있는 변이는 전 세계적으로 공유되는 경향이 있으며, 같은 지역이든 다른 지역이든 개인 간의 대부분의 차이는 전 세계적인 변이에 기인한다는 연구 결과로 인해 인종 범주 또한 훼손됩니다.[84]대륙이나 주요 지역 내에 고정되어 있고 다른 곳에서는 발견되지 않은 유전적 변이체는 발견되지 않았습니다.[85]
또한, 인간의 유전적 변이는 임상적이기 때문에 많은 사람들이 2개 이상의 대륙군에 소속되어 있습니다.따라서, 어떤 특정한 사람에게 할당된 유전적 기반의 "생물지리학적 조상"은 일반적으로 광범위하게 분포될 것이고 상당한 불확실성을 수반할 것입니다(Paff et al.2004).
세계의 많은 지역에서, 집단들은 많은 사람들이 광범위하게 분리된 지역에서 비교적 최근의 조상들을 가지고 있는 방식으로 섞였습니다.비록 많은 수의 로키족의 유전학적 분석들이 다양한 대륙 집단들로부터 온 한 사람의 조상들의 비율에 대한 추정치를 산출할 수 있지만(Shriver et al. 2003; Bamshad et al.2004), 이러한 추정은 역사를 통해 인간 그룹이 지역 스케일에서 대륙 스케일로 짝을 교환했기 때문에 부모 집단의 잘못된 구별을 가정할 수 있습니다(Cavalli-Sporza et al. 1994; Hoerder 2002).많은 수의 마커를 사용하더라도 개체 또는 그룹의 혼합물 비율을 추정하기 위한 정보는 제한적이며 추정치는 일반적으로 넓은 신뢰 구간을 갖습니다(Paff et al.2004).
유전자 군집
유전자 데이터는 인구 구조를 추론하고 개인을 종종 그들의 지리적 조상과 일치하는 집단에 할당하는 데 사용될 수 있습니다.Jorde and Wooding (2004)은 "많은 지역을 분석한 결과 인구가 아닌 개인 간 유전적 유사성에 대한 합리적으로 정확한 추정치가 도출되었습니다.개체의 군집은 지리적 기원이나 혈통과 관련이 있습니다."[22]그러나 과거로 거슬러 올라가는 사람들 사이에 공유된 역사적 조상을 고려할 때, 지리적 기원에 의한 식별은 빠르게 분해될 수 있습니다.[86]
2009년 국제 합맵 프로젝트(Phase II) 및 CEPH 인간 게놈 다양성 패널 샘플의 상염색체 SNP 데이터 분석이 발표되었습니다.HapMap과 CEPH 데이터(관련이 없는 개인 1138명)에서 얻은 53명의 모집단에 대한 연구는 자연 선택이 이전에 생각했던 것보다 훨씬 더 느리게 인간 게놈을 형성할 수 있으며, 대륙 내 및 대륙 간 이동과 같은 요인들이 유전적 변이의 분포에 더 큰 영향을 미칠 수 있음을 시사했습니다.[87]2010년에 출판된 비슷한 연구는 특히 극지방 생태지역, 먹이, 그리고 근과 관이 풍부한 식단과 관련하여 생태지역, 식단, 그리고 생존의 변화로 인한 게놈 전체의 강력한 증거를 발견했습니다.[88]2016년 연구에서, 유전체 전반에 걸친 데이터의 주요 성분 분석은 (집단에 대한 사전 정의 없이) 이전에 알려진 양성 선택 대상과 새로운 후보 유전자를 복구할 수 있었습니다.[89]
법의학 인류학
법의학 인류학자들은 가능하면 유전자와 화학적 표식을 사용할 뿐만 아니라 골격 형태학을 분석함으로써 골격 유해의 조상을 평가할 수 있습니다.[90]이러한 평가는 결코 확실하지 않지만, 실제 조상을 결정하는 데 있어 골격 형태학 분석의 정확도는 90%[91]로 추정되었습니다.

유전자 흐름 및 혼합물
두 모집단 사이의 유전자 흐름은 모집단 사이의 평균 유전적 거리를 감소시키며, 완전히 고립된 인간 개체군만이 유전자 흐름을 경험하지 않으며 대부분의 개체군은 대부분의 유전적 변이에 대해 관찰된 임상 분포를 생성하는 다른 이웃 개체군과 지속적인 유전자 흐름을 가지고 있습니다.유전자 흐름이 잘 분화된 유전자 집단 사이에서 일어날 때 그 결과는 "유전적 혼합물"이라고 불립니다.
혼합물 매핑은 유전자 변이가 모집단 간의 질병 비율에 차이를 유발하는 방법을 연구하는 데 사용되는 기술입니다.[92]여러 대륙에서 그들의 조상을 추적하는 최근의 혼합 집단은 부모 집단 간의 유병률에 차이가 있는 특성과 질병에 대한 유전자를 확인하는 데 매우 적합합니다.아프리카계 미국인 인구는 백혈구 수, 체질량 지수, 전립선 암 및 신장 질환과 같은 복잡한 유전적 특성에 대한 연구를 포함한 다양한 인구 유전 및 혼합물 매핑 연구의 초점이 되어 왔습니다.[93]
유럽인과 아프리카인의 접촉 역사가 잘 기록된 케이프 베르데 인구를 대상으로 피부색과 사회 경제적 상태를 포함한 표현형 및 유전적 변이에 대한 분석이 수행되었습니다.연구들은 이 인구 집단의 혼합물의 패턴이 성 편향적이고 (대부분 유럽 남성과 아프리카 여성 사이의 이성교제를 포함한다), 사회경제적 지위와 혈통에 관계없이 피부색 사이에 상당한 상호작용이 있다는 것을 보여주었습니다.[94]또 다른 연구는 인간 백혈구 항원(HLA)과 비 HLA 단백질의 유전적 변이로 인해 이식 후 이식편대 숙주 질환 합병증의 위험이 증가하는 것을 보여줍니다.[95]
헬스
모든 유전자 빈도의 차이는 일부 단원성 질환의 발병률의 집단 차이에 기여하며, 일부 일반적인 질환의 발병률의 차이에 기여할 수 있습니다.[96]단원성 질환의 경우 원인 대립 유전자의 빈도는 보통 가족(예: 펜실베니아 아미쉬 중 엘리스-반 크레벨트 증후군), 인종(아슈케나지 유대인 인구 중 테이-삭스병) 또는 지리(말라리아 지역에 살았던 조상이 있는 사람들 중 혈색소 병)과 가장 잘 관련되어 있습니다.단일원성 질환의 발병률은 인종이나 민족에 따라 분류된 집단에 따라 다를 수 있으며, 의료 전문가들은 일반적으로 이러한 패턴을 고려하여 진단을 수행합니다.[97]
다양한 유전적 변이와 환경적 요인을 포함하는 일반적인 질병에도 불구하고, 조사자들은 작은 효과에서 중간 효과를 가진 차별적으로 분포된 대립 유전자의 개입을 시사하는 증거를 지적합니다.대표적으로 고혈압(Douglas et al. 1996), 당뇨병(Gower et al. 2003), 비만(Fernandez et al. 2003), 전립선암(Platz et al.2000).그러나 이러한 경우 중 어떤 경우에도 민감성 유전자의 대립 유전자 변이도 그룹 간 질병 유병률 차이의 상당 부분을 설명하는 것으로 나타났으며, 이러한 차이를 발생시키는 데 있어서 유전적 요인의 역할은 여전히 불확실합니다(Mountain and Risch 2004).
반면에 어떤 다른 변화들은 특정 질병을 예방하고 환경에 적응할 수 있는 기회를 증가시키기 때문에 인간에게 이롭습니다.예를 들어 에이즈로부터 보호하는 CCR5 유전자의 돌연변이는 세포 표면에 돌연변이로 인하여 CCR5 유전자가 존재하지 않습니다.표면에 CCR5 유전자가 없으면 HIV 바이러스가 잡고 결합할 수 있는 것은 아무것도 없습니다.따라서 CCR5 유전자의 돌연변이는 개인이 에이즈에 걸릴 가능성을 줄여줍니다.CCR5의 돌연변이는 특정 지역에서도 꽤 흔한데, 인구의 14% 이상이 유럽에서, 약 6~10%가 아시아와 북아프리카에서 돌연변이를 옮깁니다.[98]

돌연변이를 제외하고, 고대에 인간을 도왔을지도 모르는 많은 유전자들이 오늘날 인간을 괴롭힙니다.예를 들어, 오늘날 사람들을 비만과 당뇨병에 걸리기 쉽게 만드는 유전자가 인간으로 하여금 음식을 더 효율적으로 처리하게 하는 유전자라고 의심받고 있습니다.[99]
스탠포드 대학의 닐 리쉬(Neil Risch)는 미국에서 공중 보건과 정책적 고려를 위해 인종/민족을 스스로 확인하는 것이 유효한 분류 수단이 될 수 있다고 제안했습니다.[100][96]Noah Rosenberg 그룹의 2002년 논문은 비슷한 주장을 합니다: "인간 개체수의 구조는 다양한 역학적 맥락에서 관련이 있습니다.유전적 및 비유전적 위험 인자의 빈도 변화의 결과로 질병의 비율과 약물 부작용과 같은 표현형은 모집단에 따라 다릅니다.또한 환자의 출신지에 대한 정보는 질병의 직접적인 원인이 알려지지 않은 경우 의료 종사자에게 위험에 대한 정보를 제공할 수 있습니다."[101]그러나 2018년에 노아 로젠버그는 환경적 변이가 더 가능성이 높은[102] 원인이라고 말하면서 인구 간 건강 차이에 대한 유전적 본질주의적인 생각에 반대하는 연구를 발표했습니다.
게놈 프로젝트
인간 게놈 프로젝트는 인간 게놈의 구조를 결정하거나 연구하는 과학적 노력입니다.인간 게놈 프로젝트는 획기적인 게놈 프로젝트였습니다.
참고 항목
지방의
프로젝트
참고문헌
- ^ Bruder CE, Piotrowski A, Gijsbers AA, Andersson R, Erickson S, Diaz de Ståhl T, et al. (March 2008). "Phenotypically concordant and discordant monozygotic twins display different DNA copy-number-variation profiles". American Journal of Human Genetics. 82 (3): 763–71. doi:10.1016/j.ajhg.2007.12.011. PMC 2427204. PMID 18304490.
- ^ a b c d e Auton A, Brooks LD, Durbin RM, Garrison EP, Kang HM, Korbel JO, et al. (October 2015). "A global reference for human genetic variation". Nature. 526 (7571): 68–74. Bibcode:2015Natur.526...68T. doi:10.1038/nature15393. PMC 4750478. PMID 26432245.
- ^ a b NCBI (8 May 2017). "dbSNP's human build 150 has doubled the amount of RefSNP records!". NCBI Insights. Retrieved 16 May 2017.
- ^ Xue, Cheng; Raveendran, Muthuswamy; Harris, R. Alan; Fawcett, Gloria L.; Liu, Xiaoming; White, Simon; Dahdouli, Mahmoud; Deiros, David Rio; Below, Jennifer E.; Salerno, William; Cox, Laura (1 December 2016). "The population genomics of rhesus macaques (Macaca mulatta) based on whole-genome sequences". Genome Research. 26 (12): 1651–1662. doi:10.1101/gr.204255.116. ISSN 1088-9051. PMC 5131817. PMID 27934697.
- ^ Reich, David (23 March 2018). "Opinion How Genetics Is Changing Our Understanding of 'Race'". The New York Times. ISSN 0362-4331. Retrieved 15 August 2022.
- ^ Williams, David R. (1 July 1997). "Race and health: Basic questions, emerging directions". Annals of Epidemiology. Special Issue: Interface Between Molecular and Behavioral Epidemiology. 7 (5): 322–333. doi:10.1016/S1047-2797(97)00051-3. ISSN 1047-2797. PMID 9250627.
- ^ "1". Race and racism in theory and practice. Berel Lang. Lanham, Md.: Rowman & Littlefield. 2000. ISBN 0-8476-9692-8. OCLC 42389561.
{{cite book}}: CS1 메인 : 기타 (링크) - ^ Lee, Jun-Ki; Aini, Rahmi Qurota; Sya’bandari, Yustika; Rusmana, Ai Nurlaelasari; Ha, Minsu; Shin, Sein (1 April 2021). "Biological Conceptualization of Race". Science & Education. 30 (2): 293–316. Bibcode:2021Sc&Ed..30..293L. doi:10.1007/s11191-020-00178-8. ISSN 1573-1901. S2CID 231598896.
- ^ Kolbert, Elizabeth (4 April 2018). "There's No Scientific Basis for Race—It's a Made-Up Label". National Geographic. Retrieved 15 August 2022.
- ^ Templeton, Alan Robert (2018). Human Population Genetics and Genomics. London. pp. 445–446. ISBN 978-0-12-386026-2. OCLC 1062418886.
{{cite book}}: CS1 유지 관리: 위치 누락 게시자(링크) - ^ a b Reich, David (2018). Who we are and how we got here: ancient DNA and the new science of the human past (First ed.). Oxford, United Kingdom. p. 255. ISBN 978-0-19-882125-0. OCLC 1006478846.
{{cite book}}: CS1 유지 관리: 위치 누락 게시자(링크) - ^ Witherspoon, D. J.; Wooding, S.; Rogers, A. R.; Marchani, E. E.; Watkins, W. S.; Batzer, M. A.; Jorde, L. B. (2007). "Genetic Similarities Within and Between Human Populations". Genetics. 176 (1): 351–359. doi:10.1534/genetics.106.067355. ISSN 0016-6731. PMC 1893020. PMID 17339205.
- ^ Campbell, Michael C.; Tishkoff, Sarah A. (2008). "AFRICAN GENETIC DIVERSITY: Implications for Human Demographic History, Modern Human Origins, and Complex Disease Mapping". Annual Review of Genomics and Human Genetics. 9: 403–433. doi:10.1146/annurev.genom.9.081307.164258. ISSN 1527-8204. PMC 2953791. PMID 18593304.
- ^ Campbell, Michael C.; Tishkoff, Sarah A. (2008). "AFRICAN GENETIC DIVERSITY: Implications for Human Demographic History, Modern Human Origins, and Complex Disease Mapping". Annual Review of Genomics and Human Genetics. 9: 403–433. doi:10.1146/annurev.genom.9.081307.164258. ISSN 1527-8204. PMC 2953791. PMID 18593304.
- ^ "We are all mutants: First direct whole-genome measure of human mutation predicts 60 new mutations in each of us". Science Daily. 13 June 2011. Retrieved 5 September 2011.
- ^ Conrad DF, Keebler JE, DePristo MA, Lindsay SJ, Zhang Y, Casals F, et al. (June 2011). "Variation in genome-wide mutation rates within and between human families". Nature Genetics. 43 (7): 712–4. doi:10.1038/ng.862. PMC 3322360. PMID 21666693.
- ^ Ackermann, R. R.; Cheverud, J. M. (16 December 2004). "Detecting genetic drift versus selection in human evolution". Proceedings of the National Academy of Sciences. 101 (52): 17946–17951. Bibcode:2004PNAS..10117946A. doi:10.1073/pnas.0405919102. ISSN 0027-8424. PMC 539739. PMID 15604148.
- ^ Guo J, Wu Y, Zhu Z, Zheng Z, Trzaskowski M, Zeng J, Robinson MR, Visscher PM, Yang J (May 2018). "Global genetic differentiation of complex traits shaped by natural selection in humans". Nature Communications. 9 (1): 1865. Bibcode:2018NatCo...9.1865G. doi:10.1038/s41467-018-04191-y. PMC 5951811. PMID 29760457.
- ^ Wang ET, Kodama G, Baldi P, Moyzis RK (January 2006). "Global landscape of recent inferred Darwinian selection for Homo sapiens". Proceedings of the National Academy of Sciences of the United States of America. 103 (1): 135–40. Bibcode:2006PNAS..103..135W. doi:10.1073/pnas.0509691102. PMC 1317879. PMID 16371466.
By these criteria, 1.6% of Perlegen SNPs were found to exhibit the genetic architecture of selection.
- ^ Kidd JM, Cooper GM, Donahue WF, Hayden HS, Sampas N, Graves T, et al. (May 2008). "Mapping and sequencing of structural variation from eight human genomes". Nature. 453 (7191): 56–64. Bibcode:2008Natur.453...56K. doi:10.1038/nature06862. PMC 2424287. PMID 18451855.
- ^ Driscoll DA, Gross S (June 2009). "Clinical practice. Prenatal screening for aneuploidy". The New England Journal of Medicine. 360 (24): 2556–62. doi:10.1056/NEJMcp0900134. PMID 19516035.
- ^ a b Jorde LB, Wooding SP (November 2004). "Genetic variation, classification and 'race'". Nature Genetics. 36 (11 Suppl): S28–33. doi:10.1038/ng1435. PMID 15508000.
- ^ a b Tishkoff SA, Kidd KK (November 2004). "Implications of biogeography of human populations for 'race' and medicine". Nature Genetics. 36 (11 Suppl): S21–7. doi:10.1038/ng1438. PMID 15507999.
- ^ Mullaney JM, Mills RE, Pittard WS, Devine SE (October 2010). "Small insertions and deletions (INDELs) in human genomes". Human Molecular Genetics. 19 (R2): R131–6. doi:10.1093/hmg/ddq400. PMC 2953750. PMID 20858594.
- ^ a b Collins FS, Brooks LD, Chakravarti A (December 1998). "A DNA polymorphism discovery resource for research on human genetic variation". Genome Research. 8 (12): 1229–31. doi:10.1101/gr.8.12.1229. PMID 9872978.
- ^ Thomas PE, Klinger R, Furlong LI, Hofmann-Apitius M, Friedrich CM (2011). "Challenges in the association of human single nucleotide polymorphism mentions with unique database identifiers". BMC Bioinformatics. 12 (Suppl 4): S4. doi:10.1186/1471-2105-12-S4-S4. PMC 3194196. PMID 21992066.
- ^ Ke X, Taylor MS, Cardon LR (April 2008). "Singleton SNPs in the human genome and implications for genome-wide association studies". European Journal of Human Genetics. 16 (4): 506–15. doi:10.1038/sj.ejhg.5201987. PMID 18197193.
- ^ Ng PC, Levy S, Huang J, Stockwell TB, Walenz BP, Li K, et al. (August 2008). Schork NJ (ed.). "Genetic variation in an individual human exome". PLOS Genetics. 4 (8): e1000160. doi:10.1371/journal.pgen.1000160. PMC 2493042. PMID 18704161.
- ^ Gross L (October 2007). "A new human genome sequence paves the way for individualized genomics". PLOS Biology. 5 (10): e266. doi:10.1371/journal.pbio.0050266. PMC 1964778. PMID 20076646.
- ^ "First Individual Diploid Human Genome Published By Researchers at J. Craig Venter Institute". J. Craig Venter Institute. 3 September 2007. Archived from the original on 16 July 2011. Retrieved 5 September 2011.
- ^ Levy S, Sutton G, Ng PC, Feuk L, Halpern AL, Walenz BP, et al. (September 2007). "The diploid genome sequence of an individual human". PLOS Biology. 5 (10): e254. doi:10.1371/journal.pbio.0050254. PMC 1964779. PMID 17803354.
- ^ "Understanding Genetics: Human Health and the Genome". The Tech Museum of Innovation. 24 January 2008. Archived from the original on 29 April 2012. Retrieved 5 September 2011.
- ^ "First Diploid Human Genome Sequence Shows We're Surprisingly Different". Science Daily. 4 September 2007. Retrieved 5 September 2011.
- ^ "Copy number variation may stem from replication misstep". EurekAlert!. 27 December 2007. Retrieved 5 September 2011.
- ^ Lee JA, Carvalho CM, Lupski JR (December 2007). "A DNA replication mechanism for generating nonrecurrent rearrangements associated with genomic disorders". Cell. 131 (7): 1235–47. doi:10.1016/j.cell.2007.11.037. PMID 18160035. S2CID 9263608.
- ^ Redon R, Ishikawa S, Fitch KR, Feuk L, Perry GH, Andrews TD, et al. (November 2006). "Global variation in copy number in the human genome". Nature. 444 (7118): 444–54. Bibcode:2006Natur.444..444R. doi:10.1038/nature05329. PMC 2669898. PMID 17122850.
- ^ Dumas L, Kim YH, Karimpour-Fard A, Cox M, Hopkins J, Pollack JR, et al. (September 2007). "Gene copy number variation spanning 60 million years of human and primate evolution". Genome Research. 17 (9): 1266–77. doi:10.1101/gr.6557307. PMC 1950895. PMID 17666543.
- ^ Prüfer K, Racimo F, Patterson N, Jay F, Sankararaman S, Sawyer S, et al. (January 2014). "The complete genome sequence of a Neanderthal from the Altai Mountains". Nature. 505 (7481): 43–9. Bibcode:2014Natur.505...43P. doi:10.1038/nature12886. PMC 4031459. PMID 24352235.
- ^ Pratas D, Hosseini M, Silva R, Pinho A, Ferreira P (20–23 June 2017). "Visualization of Distinct DNA Regions of the Modern Human Relatively to a Neanderthal Genome". Pattern Recognition and Image Analysis. Lecture Notes in Computer Science. Vol. 10255. pp. 235–242. doi:10.1007/978-3-319-58838-4_26. ISBN 978-3-319-58837-7.
- ^ "Human Genetic Variation Fact Sheet". National Institute of General Medical Sciences. 19 August 2011. Retrieved 5 September 2011.
- ^ Rakyan V, Whitelaw E (January 2003). "Transgenerational epigenetic inheritance". Current Biology. 13 (1): R6. doi:10.1016/S0960-9822(02)01377-5. PMID 12526754.
- ^ "Cline". Microsoft Encarta Premium. 2009.
- ^ King RC, Stansfield WD, Mulligan PK (2006). "Cline". A dictionary of genetics (7th ed.). Oxford University Press. ISBN 978-0195307610.
- ^ Begon M, Townsend CR, Harper JL (2006). Ecology: From individuals to ecosystems (4th ed.). Wiley-Blackwell. p. 10. ISBN 978-1405111171.
- ^ "Haplogroup". DNA-Newbie Glossary. International Society of Genetic Genealogy. Retrieved 5 September 2012.
- ^ "The descent of man Chapter 6 – On the Affinities and Genealogy of Man". Darwin-online.org.uk. Retrieved 11 January 2011.
In each great region of the world the living mammals are closely related to the extinct species of the same region. It is, therefore, probable that Africa was formerly inhabited by extinct apes closely allied to the gorilla and chimpanzee; and as these two species are now man's nearest allies, it is somewhat more probable that our early progenitors lived on the African continent than elsewhere. But it is useless to speculate on this subject, for an ape nearly as large as a man, namely the Dryopithecus of Lartet, which was closely allied to the anthropomorphous Hylobates, existed in Europe during the Upper Miocene period; and since so remote a period the earth has certainly undergone many great revolutions, and there has been ample time for migration on the largest scale.
- ^ a b Underhill PA, Shen P, Lin AA, Jin L, Passarino G, Yang WH, et al. (November 2000). "Y chromosome sequence variation and the history of human populations". Nature Genetics. 26 (3): 358–61. doi:10.1038/81685. PMID 11062480. S2CID 12893406.
- ^ a b "New Research Proves Single Origin of Humans in Africa". Science Daily. 19 July 2007. Retrieved 5 September 2011.
- ^ Manica A, Amos W, Balloux F, Hanihara T (July 2007). "The effect of ancient population bottlenecks on human phenotypic variation". Nature. 448 (7151): 346–8. Bibcode:2007Natur.448..346M. doi:10.1038/nature05951. PMC 1978547. PMID 17637668.
- ^ Tishkoff SA, Reed FA, Friedlaender FR, Ehret C, Ranciaro A, Froment A, et al. (May 2009). "The genetic structure and history of Africans and African Americans" (PDF). Science. 324 (5930): 1035–44. Bibcode:2009Sci...324.1035T. doi:10.1126/science.1172257. PMC 2947357. PMID 19407144.
We incorporated geographic data into a Bayesian clustering analysis, assuming no admixture (TESS software) (25) and distinguished six clusters within continental Africa (Fig. 5A). The most geographically widespread cluster (orange) extends from far Western Africa (the Mandinka) through central Africa to the Bantu speakers of South Africa (the Venda and Xhosa) and corresponds to the distribution of the Niger-Kordofanian language family, possibly reflecting the spread of Bantu-speaking populations from near the Nigerian/Cameroon highlands across eastern and southern Africa within the past 5000 to 3000 years (26,27). Another inferred cluster includes the Pygmy and SAK populations (green), with a noncontiguous geographic distribution in central and southeastern Africa, consistent with the STRUCTURE (Fig. 3) and phylogenetic analyses (Fig. 1). Another geographically contiguous cluster extends across northern Africa (blue) into Mali (the Dogon), Ethiopia, and northern Kenya. With the exception of the Dogon, these populations speak an Afroasiatic language. Chadic-speaking and Nilo-Saharan–speaking populations from Nigeria, Cameroon, and central Chad, as well as several Nilo-Saharan–speaking populations from southern Sudan, constitute another cluster (red). Nilo-Saharan and Cushitic speakers from the Sudan, Kenya, and Tanzania, as well as some of the Bantu speakers from Kenya, Tanzania, and Rwanda (Hutu/Tutsi), constitute another cluster (purple), reflecting linguistic evidence for gene flow among these populations over the past ~5000 years (28,29). Finally, the Hadza are the sole constituents of a sixth cluster (yellow), consistent with their distinctive genetic structure identified by PCA and STRUCTURE.
- ^ Schlebusch CM, Jakobsson M (August 2018). "Tales of Human Migration, Admixture, and Selection in Africa". Annual Review of Genomics and Human Genetics. 19: 405–428. doi:10.1146/annurev-genom-083117-021759. PMID 29727585. S2CID 19155657. Retrieved 28 May 2018.
- ^ Zimmer, Carl (17 May 2023). "Study Offers New Twist in How the First Humans Evolved – A new genetic analysis of 290 people suggests that humans emerged at various times and places in Africa". The New York Times. Archived from the original on 17 May 2023. Retrieved 18 May 2023.
- ^ Ragsdale,vAaron P.; et al. (17 May 2023). "A weakly structured stem for human origins in Africa". Nature. 167 (7962): 755–763. Bibcode:2023Natur.617..755R. doi:10.1038/s41586-023-06055-y. PMC 10208968. PMID 37198480.
- ^ Auton A, Brooks LD, Durbin RM, Garrison EP, Kang HM, Korbel JO, et al. (1000 Genomes Project Consortium) (October 2015). "A global reference for human genetic variation". Nature. 526 (7571): 68–74. Bibcode:2015Natur.526...68T. doi:10.1038/nature15393. PMC 4750478. PMID 26432245.
- ^ Li, Hui; Cho, Kelly; Kidd, J.; Kidd, K. (2009). "Genetic landscape of Eurasia and "admixture" in Uyghurs". American Journal of Human Genetics. 85 (6): 934–937. doi:10.1016/j.ajhg.2009.10.024. PMC 2790568. PMID 20004770. S2CID 37591388.
- ^ a b Gabriel SB, Schaffner SF, Nguyen H, Moore JM, Roy J, Blumenstiel B, et al. (June 2002). "The structure of haplotype blocks in the human genome". Science. 296 (5576): 2225–9. Bibcode:2002Sci...296.2225G. doi:10.1126/science.1069424. PMID 12029063. S2CID 10069634.
- ^ a b Lewontin RC (1972). "The Apportionment of Human Diversity". In Theodosius Dobzhansky, Max K. Hecht, William C. Steere (eds.). Evolutionary Biology. Vol. 6. New York: Appleton–Century–Crofts. pp. 381–97. doi:10.1007/978-1-4684-9063-3_14. ISBN 978-1-4684-9065-7. S2CID 21095796.
- ^ Bamshad MJ, Wooding S, Watkins WS, Ostler CT, Batzer MA, Jorde LB (March 2003). "Human population genetic structure and inference of group membership". American Journal of Human Genetics. 72 (3): 578–89. doi:10.1086/368061. PMC 1180234. PMID 12557124.
- ^ 마니카, 안드레아, 윌리엄 아모스, 프랑수아 발루, 그리고 하니하라 쓰네히코."고대인구 병목현상이 인간 표현형 변이에 미치는 영향"네이처 448호, No. 7151 (2007.7): 346-48. Doi:10.1038/네이처 05951.
- ^ Jablonski NG (10 January 2014). "The Biological and Social Meaning of Skin Color". Living Color: The Biological and Social Meaning of Skin Color. University of California Press. ISBN 978-0-520-28386-2. JSTOR 10.1525/j.ctt1pn64b.
- ^ Grubert F, Zaugg JB, Kasowski M, Ursu O, Spacek DV, Martin AR, et al. (August 2015). "Genetic Control of Chromatin States in Humans Involves Local and Distal Chromosomal Interactions". Cell. 162 (5): 1051–65. doi:10.1016/j.cell.2015.07.048. PMC 4556133. PMID 26300125.
- ^ Cenik C, Cenik ES, Byeon GW, Grubert F, Candille SI, Spacek D, et al. (November 2015). "Integrative analysis of RNA, translation, and protein levels reveals distinct regulatory variation across humans". Genome Research. 25 (11): 1610–21. doi:10.1101/gr.193342.115. PMC 4617958. PMID 26297486.
- ^ Wu L, Candille SI, Choi Y, Xie D, Jiang L, Li-Pook-Than J, Tang H, Snyder M (July 2013). "Variation and genetic control of protein abundance in humans". Nature. 499 (7456): 79–82. Bibcode:2013Natur.499...79W. doi:10.1038/nature12223. PMC 3789121. PMID 23676674.
- ^ Phillips ML (9 January 2007). "Ethnicity tied to gene expression". The Scientist. Archived from the original on 8 May 2015. Retrieved 5 September 2011.
- ^ Spielman RS, Bastone LA, Burdick JT, Morley M, Ewens WJ, Cheung VG (February 2007). "Common genetic variants account for differences in gene expression among ethnic groups". Nature Genetics. 39 (2): 226–31. doi:10.1038/ng1955. PMC 3005333. PMID 17206142.
- ^ Swaminathan N (9 January 2007). "Ethnic Differences Traced to Variable Gene Expression". Scientific American. Retrieved 5 September 2011.
- ^ Check E (2007). "Genetic expression speaks as loudly as gene type". Nature News. doi:10.1038/news070101-8. S2CID 84380725.
- ^ Bell L (15 January 2007). "Variable gene expression seen in different ethnic groups". BioNews.org. Archived from the original on 26 March 2016. Retrieved 5 September 2011.
- ^ Kamrani K (28 February 2008). "Differences of gene expression between human populations". Anthropology.net. Retrieved 5 September 2011.
- ^ Storey JD, Madeoy J, Strout JL, Wurfel M, Ronald J, Akey JM (March 2007). "Gene-expression variation within and among human populations". American Journal of Human Genetics. 80 (3): 502–9. doi:10.1086/512017. PMC 1821107. PMID 17273971.
- ^ a b c Graves JL (2006). "What We Know and What We Don't Know: Human Genetic Variation and the Social Construction of Race". Is Race "Real"?. Social Science Research Council. Retrieved 22 January 2011.
- ^ Keita SO, Kittles RA, Royal CD, Bonney GE, Furbert-Harris P, Dunston GM, Rotimi CN (November 2004). "Conceptualizing human variation". Nature Genetics. 36 (11 Suppl): S17–20. doi:10.1038/ng1455. PMID 15507998.
- ^ Hawks J (2013). Significance of Neandertal and Denisovan Genomes in Human Evolution. pp. 433–49. doi:10.1146/annurev-anthro-092412-155548. ISBN 978-0-8243-1942-7.
{{cite book}}:journal=무시됨(도움말) - ^ * Wright S (1978). Evolution and the Genetics of Populations. Vol. 4, Variability Within and Among Natural Populations. Chicago, Illinois: Univ. Chicago Press. p. 438.
- ^ Long JC, Kittles RA (August 2003). "Human genetic diversity and the nonexistence of biological races". Human Biology. 75 (4): 449–71. doi:10.1353/hub.2003.0058. PMID 14655871. S2CID 26108602.
- ^ Harris, Kelley; Nielsen, Rasmus (June 2016). "The Genetic Cost of Neanderthal Introgression". Genetics. 203 (2): 881–891. doi:10.1534/genetics.116.186890. ISSN 0016-6731. PMC 4896200. PMID 27038113.
- ^ Wall, Jeffrey D.; Yang, Melinda A.; Jay, Flora; Kim, Sung K.; Durand, Eric Y.; Stevison, Laurie S.; Gignoux, Christopher; Woerner, August; Hammer, Michael F.; Slatkin, Montgomery (May 2013). "Higher Levels of Neanderthal Ancestry in East Asians than in Europeans". Genetics. 194 (1): 199–209. doi:10.1534/genetics.112.148213. ISSN 0016-6731. PMC 3632468. PMID 23410836.
- ^ a b Reich D, Green RE, Kircher M, Krause J, Patterson N, Durand EY, et al. (December 2010). "Genetic history of an archaic hominin group from Denisova Cave in Siberia". Nature. 468 (7327): 1053–60. Bibcode:2010Natur.468.1053R. doi:10.1038/nature09710. PMC 4306417. PMID 21179161.
- ^ Wall JD, Yang MA, Jay F, Kim SK, Durand EY, Stevison LS, et al. (May 2013). "Higher levels of neanderthal ancestry in East Asians than in Europeans". Genetics. 194 (1): 199–209. doi:10.1534/genetics.112.148213. PMC 3632468. PMID 23410836.
- ^ Hammer MF, Woerner AE, Mendez FL, Watkins JC, Wall JD (September 2011). "Genetic evidence for archaic admixture in Africa". Proceedings of the National Academy of Sciences of the United States of America. 108 (37): 15123–8. Bibcode:2011PNAS..10815123H. doi:10.1073/pnas.1109300108. PMC 3174671. PMID 21896735.
- ^ Durvasula A, Sankararaman S (February 2020). "Recovering signals of ghost archaic introgression in African populations". Science Advances. 6 (7): eaax5097. Bibcode:2020SciA....6.5097D. doi:10.1126/sciadv.aax5097. PMC 7015685. PMID 32095519.
- ^ Rosenberg NA, Mahajan S, Gonzalez-Quevedo C, Blum MG, Nino-Rosales L, Ninis V, et al. (December 2006). "Low levels of genetic divergence across geographically and linguistically diverse populations from India". PLOS Genetics. 2 (12): e215. doi:10.1371/journal.pgen.0020215. PMC 1713257. PMID 17194221.
- ^ Wohns, Anthony Wilder; Wong, Yan; Jeffery, Ben; Akbari, Ali; Mallick, Swapan; Pinhasi, Ron; Patterson, Nick; Reich, David; Kelleher, Jerome; McVean, Gil (15 April 2021). "A unified genealogy of modern and ancient genomes". bioRxiv 10.1101/2021.02.16.431497.
- ^ Biddanda A, Rice DP, Novembre J (2020). "A variant-centric perspective on geographic patterns of human allele frequency variation". eLife. 9. doi:10.7554/eLife.60107. PMC 7755386. PMID 33350384.
- ^ Bergström A, McCarthy SA, Hui R, Almarri MA, Ayub Q, Danecek P, et al. (2020). "Insights into human genetic variation and population history from 929 diverse genomes". Science. 367 (6484). doi:10.1126/science.aay5012. PMC 7115999. PMID 32193295.
- ^ Albers, Patrick K.; McVean, Gil (13 September 2018). "Dating genomic variants and shared ancestry in population-scale sequencing data". bioRxiv. 18 (1): 416610. doi:10.1101/416610. PMC 6992231. PMID 31951611.
- ^ Coop G, Pickrell JK, Novembre J, Kudaravalli S, Li J, Absher D, et al. (June 2009). Schierup MH (ed.). "The role of geography in human adaptation". PLOS Genetics. 5 (6): e1000500. doi:10.1371/journal.pgen.1000500. PMC 2685456. PMID 19503611. 참고 항목: .
- ^ Hancock AM, Witonsky DB, Ehler E, Alkorta-Aranburu G, Beall C, Gebremedhin A, et al. (May 2010). "Colloquium paper: human adaptations to diet, subsistence, and ecoregion are due to subtle shifts in allele frequency". Proceedings of the National Academy of Sciences of the United States of America. 107 (Suppl 2): 8924–30. Bibcode:2010PNAS..107.8924H. doi:10.1073/pnas.0914625107. PMC 3024024. PMID 20445095.
- ^ Duforet-Frebourg N, Luu K, Laval G, Bazin E, Blum MG (April 2016). "Detecting Genomic Signatures of Natural Selection with Principal Component Analysis: Application to the 1000 Genomes Data". Molecular Biology and Evolution. 33 (4): 1082–93. arXiv:1504.04543. doi:10.1093/molbev/msv334. PMC 4776707. PMID 26715629.
- ^ Cunha, Eugénia; Ubelaker, Douglas H. (23 December 2019). "Evaluation of ancestry from human skeletal remains: a concise review". Forensic Sciences Research. 5 (2): 89–97. doi:10.1080/20961790.2019.1697060. ISSN 2096-1790. PMC 7476619. PMID 32939424.
- ^ Thomas, Richard M.; Parks, Connie L.; Richard, Adam H. (July 2017). "Accuracy Rates of Ancestry Estimation by Forensic Anthropologists Using Identified Forensic Cases". Journal of Forensic Sciences. 62 (4): 971–974. doi:10.1111/1556-4029.13361. ISSN 1556-4029. PMID 28133721. S2CID 3453064.
- ^ Winkler CA, Nelson GW, Smith MW (2010). "Admixture mapping comes of age". Annual Review of Genomics and Human Genetics. 11: 65–89. doi:10.1146/annurev-genom-082509-141523. PMC 7454031. PMID 20594047.
- ^ Bryc K, Auton A, Nelson MR, Oksenberg JR, Hauser SL, Williams S, et al. (January 2010). "Genome-wide patterns of population structure and admixture in West Africans and African Americans". Proceedings of the National Academy of Sciences of the United States of America. 107 (2): 786–91. Bibcode:2010PNAS..107..786B. doi:10.1073/pnas.0909559107. PMC 2818934. PMID 20080753.
- ^ Beleza S, Campos J, Lopes J, Araújo II, Hoppfer Almada A, Correia e Silva A, et al. (2012). "The admixture structure and genetic variation of the archipelago of Cape Verde and its implications for admixture mapping studies". PLOS ONE. 7 (11): e51103. Bibcode:2012PLoSO...751103B. doi:10.1371/journal.pone.0051103. PMC 3511383. PMID 23226471.
- ^ Arrieta-Bolaños E, Madrigal JA, Shaw BE (2012). "Human leukocyte antigen profiles of Latin American populations: differential admixture and its potential impact on hematopoietic stem cell transplantation". Bone Marrow Research. 2012: 1–13. doi:10.1155/2012/136087. PMC 3506882. PMID 23213535.
- ^ a b Risch N, Burchard E, Ziv E, Tang H (July 2002). "Categorization of humans in biomedical research: genes, race and disease". Genome Biology. 3 (7): comment2007. doi:10.1186/gb-2002-3-7-comment2007. PMC 139378. PMID 12184798.
- ^ Lu YF, Goldstein DB, Angrist M, Cavalleri G (July 2014). "Personalized medicine and human genetic diversity". Cold Spring Harbor Perspectives in Medicine. 4 (9): a008581. doi:10.1101/cshperspect.a008581. PMC 4143101. PMID 25059740.
- ^ Limborska SA, Balanovsky OP, Balanovskaya EV, Slominsky PA, Schadrina MI, Livshits LA, et al. (2002). "Analysis of CCR5Delta32 geographic distribution and its correlation with some climatic and geographic factors". Human Heredity. 53 (1): 49–54. doi:10.1159/000048605. PMID 11901272. S2CID 1538974.
- ^ Tishkoff SA, Verrelli BC (2003). "Patterns of human genetic diversity: implications for human evolutionary history and disease". Annual Review of Genomics and Human Genetics. 4 (1): 293–340. doi:10.1146/annurev.genom.4.070802.110226. PMID 14527305.
- ^ Tang H, Quertermous T, Rodriguez B, Kardia SL, Zhu X, Brown A, et al. (February 2005). "Genetic structure, self-identified race/ethnicity, and confounding in case-control association studies". American Journal of Human Genetics. 76 (2): 268–75. doi:10.1086/427888. PMC 1196372. PMID 15625622.
- ^ Rosenberg NA, Pritchard JK, Weber JL, Cann HM, Kidd KK, Zhivotovsky LA, Feldman MW (December 2002). "Genetic structure of human populations". Science. 298 (5602): 2381–5. Bibcode:2002Sci...298.2381R. doi:10.1126/science.1078311. PMID 12493913. S2CID 8127224.
- ^ 다유성 점수, 다유성 적응 및 인간 표현형 차이 해석
추가열람
- Race, Ethnicity (October 2005). "The use of racial, ethnic, and ancestral categories in human genetics research". American Journal of Human Genetics. 77 (4): 519–32. doi:10.1086/491747. PMC 1275602. PMID 16175499.
- Altmüller J, Palmer LJ, Fischer G, Scherb H, Wjst M (November 2001). "Genomewide scans of complex human diseases: true linkage is hard to find". American Journal of Human Genetics. 69 (5): 936–50. doi:10.1086/324069. PMC 1274370. PMID 11565063.
- Aoki K (2002). "Sexual selection as a cause of human skin colour variation: Darwin's hypothesis revisited". Annals of Human Biology. 29 (6): 589–608. doi:10.1080/0301446021000019144. PMID 12573076. S2CID 22703861.
- Bamshad M, Wooding S, Salisbury BA, Stephens JC (August 2004). "Deconstructing the relationship between genetics and race". Nature Reviews. Genetics. 5 (8): 598–609. doi:10.1038/nrg1401. PMID 15266342. S2CID 12378279. 전재 우편의
- Bamshad M, Wooding SP (February 2003). "Signatures of natural selection in the human genome". Nature Reviews. Genetics. 4 (2): 99–111. doi:10.1038/nrg999. PMID 12560807. S2CID 13722452.
- Cann RL, Stoneking M, Wilson AC (1987). "Mitochondrial DNA and human evolution". Nature. 325 (6099): 31–36. Bibcode:1987Natur.325...31C. doi:10.1038/325031a0. PMID 3025745. S2CID 4285418.
- Cardon LR, Abecasis GR (March 2003). "Using haplotype blocks to map human complex trait loci" (PDF). Trends in Genetics. 19 (3): 135–40. doi:10.1016/S0168-9525(03)00022-2. PMID 12615007.
- Cavalli-Sforza LL, Feldman MW (March 2003). "The application of molecular genetic approaches to the study of human evolution". Nature Genetics. 33 Suppl (3s): 266–75. doi:10.1038/ng1113. PMID 12610536. S2CID 8314161.
- Collins FS (November 2004). "What we do and don't know about 'race', 'ethnicity', genetics and health at the dawn of the genome era". Nature Genetics. 36 (11 Suppl): S13–15. doi:10.1038/ng1436. PMID 15507997. S2CID 26968169.
- Collins FS, Green ED, Guttmacher AE, Guyer MS (April 2003). "A vision for the future of genomics research". Nature. 422 (6934): 835–47. Bibcode:2003Natur.422..835C. doi:10.1038/nature01626. PMID 12695777. S2CID 205209730.
- Ebersberger I, Metzler D, Schwarz C, Pääbo S (June 2002). "Genomewide comparison of DNA sequences between humans and chimpanzees". American Journal of Human Genetics. 70 (6): 1490–97. doi:10.1086/340787. PMC 379137. PMID 11992255.
- Edwards AW (August 2003). "Human genetic diversity: Lewontin's fallacy". BioEssays. 25 (8): 798–801. doi:10.1002/bies.10315. PMID 12879450.
- Foster MW, Sharp RR (October 2004). "Beyond race: towards a whole-genome perspective on human populations and genetic variation". Nature Reviews. Genetics. 5 (10): 790–96. doi:10.1038/nrg1452. PMID 15510170. S2CID 25764082.
- Foster MW, Sharp RR, Freeman WL, Chino M, Bernsten D, Carter TH (June 1999). "The role of community review in evaluating the risks of human genetic variation research". American Journal of Human Genetics. 64 (6): 1719–27. doi:10.1086/302415. PMC 1377916. PMID 10330360.
- Gabriel SB, Schaffner SF, Nguyen H, Moore JM, Roy J, Blumenstiel B, Higgins J, DeFelice M, Lochner A, Faggart M, Liu-Cordero SN, Rotimi C, Adeyemo A, Cooper R, Ward R, Lander ES, Daly MJ, Altshuler D (June 2002). "The structure of haplotype blocks in the human genome". Science. 296 (5576): 2225–29. Bibcode:2002Sci...296.2225G. doi:10.1126/science.1069424. PMID 12029063. S2CID 10069634.
- Harding RM, Healy E, Ray AJ, Ellis NS, Flanagan N, Todd C, Dixon C, Sajantila A, Jackson IJ, Birch-Machin MA, Rees JL (April 2000). "Evidence for variable selective pressures at MC1R". American Journal of Human Genetics. 66 (4): 1351–61. doi:10.1086/302863. PMC 1288200. PMID 10733465.
- Ingman M, Kaessmann H, Pääbo S, Gyllensten U (December 2000). "Mitochondrial genome variation and the origin of modern humans". Nature. 408 (6813): 708–13. Bibcode:2000Natur.408..708I. doi:10.1038/35047064. PMID 11130070. S2CID 52850476.
- The International Hapmap Consortium (December 2003). "The International HapMap Project". Nature. 426 (6968): 789–96. Bibcode:2003Natur.426..789G. doi:10.1038/nature02168. hdl:2027.42/62838. PMID 14685227. S2CID 4387110.
- The International Hapmap Consortium (June 2004). "Integrating ethics and science in the International HapMap Project". Nature Reviews. Genetics. 5 (6): 467–75. doi:10.1038/nrg1351. PMC 2271136. PMID 15153999.
- Lander ES, Linton LM, Birren B, Nusbaum C, Zody MC, Baldwin J, et al. (February 2001). "Initial sequencing and analysis of the human genome". Nature. 409 (6822): 860–921. Bibcode:2001Natur.409..860L. doi:10.1038/35057062. PMID 11237011.
- Jorde LB, Bamshad M, Rogers AR (February 1998). "Using mitochondrial and nuclear DNA markers to reconstruct human evolution" (PDF). BioEssays. 20 (2): 126–36. doi:10.1002/(SICI)1521-1878(199802)20:2<126::AID-BIES5>3.0.CO;2-R. PMID 9631658. S2CID 17203268. Archived from the original (PDF) on 28 November 2007. Retrieved 28 October 2007.
- Jorde LB, Watkins WS, Bamshad MJ, Dixon ME, Ricker CE, Seielstad MT, Batzer MA (March 2000). "The distribution of human genetic diversity: a comparison of mitochondrial, autosomal, and Y-chromosome data". American Journal of Human Genetics. 66 (3): 979–88. doi:10.1086/302825. PMC 1288178. PMID 10712212.
- Jorde LB, Watkins WS, Kere J, Nyman D, Eriksson AW (2000). "Gene mapping in isolated populations: new roles for old friends?". Human Heredity. 50 (1): 57–65. doi:10.1159/000022891. PMID 10545758. S2CID 26960216.
- Kaessmann H, Heissig F, von Haeseler A, Pääbo S (May 1999). "DNA sequence variation in a non-coding region of low recombination on the human X chromosome". Nature Genetics. 22 (1): 78–81. doi:10.1038/8785. PMID 10319866. S2CID 9153915.
- Kaessmann H, Wiebe V, Weiss G, Pääbo S (February 2001). "Great ape DNA sequences reveal a reduced diversity and an expansion in humans". Nature Genetics. 27 (2): 155–56. doi:10.1038/84773. PMID 11175781. S2CID 19384784.
- Keita SO, Kittles RA (1997). "The Persistence of Racial Thinking and the Myth of Racial Divergence". American Anthropologist. 99 (3): 534–44. doi:10.1525/aa.1997.99.3.534.
- Marks J (1995). Human Biodiversity: Genes, Race, and History. Aldine Transaction. ISBN 978-0-202-02033-4.
- Mountain JL, Risch N (November 2004). "Assessing genetic contributions to phenotypic differences among 'racial' and 'ethnic' groups". Nature Genetics. 36 (11 Suppl): S48–53. doi:10.1038/ng1456. PMID 15508003.
- Pääbo S (January 2003). "The mosaic that is our genome". Nature. 421 (6921): 409–12. Bibcode:2003Natur.421..409P. doi:10.1038/nature01400. PMID 12540910.
- Ramachandran S, Deshpande O, Roseman CC, Rosenberg NA, Feldman MW, Cavalli-Sforza LL (November 2005). "Support from the relationship of genetic and geographic distance in human populations for a serial founder effect originating in Africa". Proceedings of the National Academy of Sciences of the United States of America. 102 (44): 15942–47. Bibcode:2005PNAS..10215942R. doi:10.1073/pnas.0507611102. PMC 1276087. PMID 16243969.
- Relethford JH (August 2002). "Apportionment of global human genetic diversity based on craniometrics and skin color". American Journal of Physical Anthropology. 118 (4): 393–98. CiteSeerX 10.1.1.473.5972. doi:10.1002/ajpa.10079. PMID 12124919. S2CID 8717358.
- Sankar P, Cho MK (November 2002). "Genetics. Toward a new vocabulary of human genetic variation". Science. 298 (5597): 1337–38. doi:10.1126/science.1074447. PMC 2271140. PMID 12434037.
- Sankar P, Cho MK, Condit CM, Hunt LM, Koenig B, Marshall P, Lee SS, Spicer P (June 2004). "Genetic research and health disparities". JAMA. 291 (24): 2985–89. doi:10.1001/jama.291.24.2985. PMC 2271142. PMID 15213210.
- Serre D, Pääbo S (September 2004). "Evidence for gradients of human genetic diversity within and among continents". Genome Research. 14 (9): 1679–85. doi:10.1101/gr.2529604. PMC 515312. PMID 15342553.
- Templeton AR (1998). "Human Races: A Genetic and Evolutionary Perspective". American Anthropologist. 100 (3): 632–50. doi:10.1525/aa.1998.100.3.632.
- Weiss KM (1998). "Coming to Terms with Human Variation". Annual Review of Anthropology. 27: 273–300. doi:10.1146/annurev.anthro.27.1.273.
- Weiss KM, Terwilliger JD (October 2000). "How many diseases does it take to map a gene with SNPs?". Nature Genetics. 26 (2): 151–57. doi:10.1038/79866. PMID 11017069. S2CID 685795.
- Yu N, Jensen-Seaman MI, Chemnick L, Kidd JR, Deinard AS, Ryder O, Kidd KK, Li WH (August 2003). "Low nucleotide diversity in chimpanzees and bonobos". Genetics. 164 (4): 1511–18. doi:10.1093/genetics/164.4.1511. PMC 1462640. PMID 12930756.
- Zietkiewicz E, Yotova V, Gehl D, Wambach T, Arrieta I, Batzer M, Cole DE, Hechtman P, Kaplan F, Modiano D, Moisan JP, Michalski R, Labuda D (November 2003). "Haplotypes in the dystrophin DNA segment point to a mosaic origin of modern human diversity". American Journal of Human Genetics. 73 (5): 994–1015. doi:10.1086/378777. PMC 1180505. PMID 14513410.
- Pennisi E (December 2007). "Breakthrough of the year. Human genetic variation". Science. 318 (5858): 1842–43. doi:10.1126/science.318.5858.1842. PMID 18096770.
- Ramachandran S, Tang H, Gutenkunst RN, Bustamante CD (2010). "Genetics and Genomics of Human Population Structure". In Speicher MR, Antonarakis SE, Motulsky AG (eds.). Vogel and Motulsky's Human Genetics: Problems and Approaches (4th ed.). Springer. ISBN 978-3-540-37653-8.


