게놈 편집
Genome editing게놈 편집, 또는 게놈 공학, 또는 유전자 편집은 DNA가 살아있는 유기체의 게놈에 삽입, 삭제, 수정 또는 대체되는 유전 공학의 한 종류이다.유전자 물질을 무작위로 숙주 게놈에 삽입하는 초기 유전공학 기술과 달리 게놈 편집은 특정 부위에 [1]삽입하는 것을 목표로 한다.
역사
게놈 편집은 일반적인 핵산가수분해효소 기반 유전자 편집 플랫폼이 등장하기 전인 1990년대에 [2]처음 시작되었지만 편집 효율성이 낮았기 때문에 사용이 제한되었습니다.Nature Methods는 이러한 효소의 세 가지 주요 등급인 ZFN(아연 핑거 뉴클라아제), 전사 활성제 유사 이펙터 뉴클라아제(TALENs) 및 공학적 메가뉴클라아제를 사용한 게놈 편집을 2011년 [3]올해의 방법으로 선정했다.CRISPR-Cas 시스템은 사이언스에 의해 2015년 올해의 [4]돌파구로 선정되었다.
2015년 현재[update], 공학적 핵산 분해효소(Meganuclearase), 아연 핑거 핵산 분해효소(ZFN), 전사 활성제 유사 이펙터 기반 핵산 분해효소(TALEN) 및 클러스터화된 정기적인 인터스페이스 짧은 회문 반복(CRISPR/Cas9) 시스템 [5][6][7][8]등 4개 패밀리가 사용되었다.2017년 [9]현재[update] 9개의 게놈 편집기를 이용할 수 있다.
2018년, 이러한 편집을 위한 일반적인 방법은 "분자 가위"인 공학적 핵산 분해 효소를 사용했다.이러한 핵산 분해 효소는 게놈에서 원하는 위치에 부위 특이적 이중 가닥 절단(DSB)을 생성합니다.유도된 이중 가닥 절단은 비호몰로지 엔드 결합(NHEJ) 또는 호몰로지 재조합(HR)을 통해 복구되며, 결과적으로 표적 돌연변이('편집')가 발생한다.
2019년 5월 중국의 변호사들은 중국 과학자 He Jiankui가 최초의 유전자 편집 인간을 만들었다고 알려진 것에 비추어 CRISPR과 같은 유전자 편집 기술로 인간 게놈을 조작하는 사람은 관련된 부작용에 [10]대한 책임을 져야 한다는 규정을 입안했다고 보고했다.CRISPR 및 관련 바이오 테크놀로지의 가능한 사각지대 및 위험에 대한 주의적 관점은 세포 제어 프로세스의 확률적 특성에 초점을 맞추어 최근에 [11]논의되었다.
에딘버러 대학 로슬린 연구소는 돼지 생식 및 호흡기 증후군을 일으키는 바이러스에 내성을 가진 돼지를 개발했는데, 돼지들은 미국과 유럽 양돈업자들에게 [12]연간 26억 달러의 비용을 지불한다.
2020년 2월, 미국에서는 3명의 [13]암환자를 대상으로 한 CRISPR 유전자 편집이 안전하게 행해졌다.2020년에는 긴장을 풀어주는 아미노산을 많이 만드는 토마토인 시실리안 루즈 하이 GABA가 일본에서 [12]판매 허가를 받았다.
2021년, 영국(영국의 나머지 지역이 아님)은 유전자 편집 식물과 동물에 대한 규제를 유럽연합에 준거한 규제에서 미국과 다른 일부 국가의 규제와 더 가까운 규제로 바꾸려고 계획했다.4월, 2021년까지 유럽 위원회 보고서는 나중에 2021년에, 연구자들이고 핵산 절단 transposons에서 발견되는 obligate 모바일element–guided 활동(오메가)단백질 IscB, IsrB과 TnpB을 포함한 라벨에 의해 유도된 CRISPR 대안을 발표한 현재 규제 제도 유전자 editing[12]에 적합하지 않았다고" 뚜렷한 증거"을 발견했다.sm모든 [14][15]RNA
배경
새로운 유전 요소를 유기체에 도입하는 방법으로서의 유전 공학은 1970년대부터 있어 왔다.이 기술의 한 가지 단점은 DNA가 숙주 게놈에 삽입되는 무작위적인 특성으로, 이것은 유기체 내의 다른 유전자를 손상시키거나 바꿀 수 있다.하지만, 유기체 [2]게놈 내의 특정 부위에 삽입된 유전자를 대상으로 하는 몇 가지 방법이 발견되었다.또한 게놈 내의 특정 염기서열을 편집할 수 있을 뿐만 아니라 표적 효과를 줄일 수 있었습니다.이것은 연구 목적, 특정 유전자에 대한 돌연변이를 목표로 하여 유전자 치료에 사용될 수 있다.기능성 유전자를 유기체에 삽입하여 결함이 있는 유전자를 대체함으로써 특정 유전병을 치료할 수 있을 것이다.
유전자 타겟팅
상동재조합
유기체의 게놈 내 특정 부위에 유전자를 표적화하는 초기 방법은 상동 재조합(HR)[16]에 의존했다.표적 게놈 배열과 일치하는 템플릿을 포함하는 DNA 구조를 생성함으로써 세포 내의 HR 프로세스가 원하는 위치에 해당 구조를 삽입할 수 있습니다.배아줄기세포에 이 방법을 사용하는 것은 표적 유전자가 녹아웃된 형질전환 생쥐의 발달을 이끌었다.유전자를 복제하거나 유전자 발현 [17]패턴을 바꾸는 것도 가능했다.마리오 카페치, 마틴 에반스, 올리버 스미시스는 배아줄기세포를 통해 생쥐에게 유전자 변형을 도입하기 위해 어떻게 상동 재조합을 사용할 수 있는지에 대한 그들의 발견을 인정받아 2007년 노벨 [18]생리의학상을 수상했다.
조건부 타겟팅
만약 중요한 유전자가 제거된다면 그것은 유기체에게 치명적일 수 있다.이러한 유전자의 기능을 연구하기 위해 부위 특이적 재조합효소(SSR)가 사용되었다.가장 일반적인 두 가지 유형은 Cre-LoxP 및 Flp-FRT 시스템입니다.크레 재조합 효소는 Lox-P 부위로 알려진 결합 배열 사이의 상동 재조합에 의해 DNA를 제거하는 효소이다.FLIP-FRT 시스템은 유사한 방식으로 작동하며 FLIP 재조합 효소가 FRT 시퀀스를 인식합니다.해당 유전자 옆에 재조합효소 부위가 있는 생물과 조직특이촉진제의 제어 하에 SSR를 발현하는 생물과의 교잡에 의해 특정 세포에서만 유전자를 녹아웃 또는 켤 수 있다.이 기술들은 또한 유전자 변형 동물에서 표식 유전자를 제거하기 위해 사용되었다.이러한 시스템의 추가적인 수정은 연구자들이 특정한 조건에서만 재조합을 유도할 수 있게 하여, 유전자를 녹아웃시키거나 개발의 [17]원하는 시기나 단계에서 발현시킬 수 있게 했다.
과정
이중 가닥 절단 수리
게놈 편집의 일반적인 형태는 DNA 이중 가닥 절단(DSB) 복구 메커니즘의 개념에 의존합니다.DSB를 복구하는 주요 경로는 Non-Homologous End Joining(NHEJ; 비호몰로지 엔드 결합)과 Homology Directed Repair(HDR; 호몰로지 다이렉트 복구)입니다.NHEJ는 다양한 효소를 사용하여 DNA 말단을 직접 결합하는 반면, 보다 정확한 HDR은 중단점에서 누락된 DNA 배열의 재생을 위한 템플릿으로 상동 배열을 사용합니다.이것은 DSB의 측면 시퀀스와 상동하는 시퀀스 내에서 원하는 유전 요소를 가진 벡터를 생성함으로써 이용할 수 있다.이로 인해 DSB 사이트에 원하는 변경이 삽입됩니다.HDR 기반 유전자 편집은 상동재조합 기반 유전자 타깃팅과 유사하지만 재조합 속도는 최소 [19]3배 이상 증가한다.
인공핵산가수분해효소
게놈 편집의 핵심은 게놈 내의 특정 지점에서 DSB를 생성하는 것입니다.일반적으로 사용되는 제한 효소는 DNA를 절단하는 데 효과적이지만, 일반적으로 여러 부위에서 인식하고 절단한다.이러한 과제를 극복하고 현장 특이적 DSB를 만들기 위해, 세 가지 종류의 핵산 분해 효소가 발견되어 현재까지 생물 공학적으로 설계되었다.이것들은 아연 핑거뉴클라아제(ZFNs), 이펙터뉴클라아제(TALEN), 메가뉴클라아제(meganuclease), 클러스터화된 정기적인 인터스페이스 짧은 회문 반복(CRISPR/Cas9) 시스템이다.
메가핵산가수분해효소
1980년대 후반에 발견된 메가핵산가수분해효소는 핵산가수분해효소 계열의 효소로 큰 DNA [20]염기서열을 인식하고 절단하는 능력이 특징이다.가장 광범위하고 가장 잘 알려진 메가핵산 분해 효소는 LAGLIDADG 계열의 단백질로, 보존된 아미노산 배열에서 이름을 따왔다.
미생물 종에서 흔히 발견되는 메가핵산가수분해효소는 매우 긴 인식 염기서열(14bp 이상)을 가지고 있어 자연적으로 매우 [21][22]특이합니다.그러나 선택된 특정 DNA 배열에 작용하기 위해 필요한 정확한 메가핵산가수분해효소를 찾을 가능성은 사실상 없다.이 과제를 극복하기 위해 돌연변이 유발과 높은 스루풋 스크리닝 방법을 사용하여 고유한 [22][23]염기서열을 인식하는 메가핵산가수분해효소 변형을 만들어냈다.다른 것들은 다양한 메가핵산가수분해효소를 융합하고 새로운 [24][25]염기서열을 인식하는 하이브리드 효소를 만들어 낼 수 있었다.그러나 다른 이들은 이성적으로 설계된 메가핵산가수분해효소라는 방법으로 배열 특이적 메가핵산가수분해효소를 [26]설계하기 위해 메가핵산 상호작용을 하는 DNA를 바꾸려고 시도했다.또 다른 접근법은 변형된 메가핵산가수분해효소의 활성과 인식된 핵 [27]배열의 특이성을 가능한 한 정확하게 예측하기 위해 컴퓨터 모델을 사용하는 것을 포함한다.
수만 개의 단백질 단위를 포함하는 큰 은행이 만들어졌습니다.이러한 단위를 조합하여 대상 부위를 인식하는 키메라 메가핵산가수분해효소를 얻을 수 있으며, 이를 통해 다양한 요구(기본 연구, 보건, 농업, 산업, 에너지 등)를 충족하는 연구개발 도구를 제공할 수 있다.이것들은 인간의 XPC 유전자를 절단할 수 있는 두 개의 메가핵산 효소의 산업적인 생산을 포함한다; 이 유전자의 돌연변이는 환자들을 피부암에 걸리게 하고 그들의 피부가 자외선에 [28]노출될 때마다 화상을 입는 심각한 단성 질환인 Xeroderma 색소증을 초래한다.
한 조합 가능성들 때문에은 곳에 이익을 주지 않다 Meganucleases,[22] 하지만,sequence-specific 효소에 대한 모든 가능한 시퀀스에 대한 공사 비용과 시간이 많이 드는, 세포에서 아연은 손가락 뉴 클레아제(ZFN)과 같은 방법보다 더 엄격한 DNA서열 인식이 때문에 덜 독성을 야기시키고의 혜택을 받는다.나를 아는 일ZFN이나 TALEN 베이스의 퓨전등의 thod가 이용된다.
아연핑거핵산가수분해효소
메가핵산가수분해효소와는 달리 ZFN과 TALEN 기술의 개념은 비특이적 DNA 절단 촉매 도메인을 기반으로 하며, 이는 아연 핑거 및 전사 활성제 유사 효과자(TALE)[29]와 같은 펩타이드를 인식하는 특정 DNA 배열과 연결될 수 있다.이에 대한 첫 번째 단계는 DNA 인식 부위와 절단 부위가 서로 분리된 엔도핵산가수분해효소(Endonuclease)를 찾는 것이었는데, 이는 제한 효소 [29]중 가장 흔하지 않은 상황이었다.일단 이 효소가 발견되면, 그것의 절단 부분은 분리될 수 있었고, 그것은 인식 능력이 없기 때문에 매우 비특이적일 것이다.이 부분은 매우 높은 특이성으로 이어질 수 있는 배열 인식 펩타이드와 연결될 수 있다.
아연 손가락 모티브는 몇 가지 전사 인자에서 발생합니다.모든 인간 단백질의 8%에서 발견되는 아연 이온은 그들의 3차원 구조의 구성에 중요한 역할을 한다.전사인자에서는 단백질-DNA 상호작용 부위에 위치하여 모티브를 안정시킨다.각 손가락의 C 말단 부분은 DNA 서열의 특정 인식을 담당합니다.
인식된 염기서열은 약 3개의 염기쌍으로 구성되며, 인식부위가 특징지어진 6~8개의 아연 핑거를 조합함으로써 약 20개의 염기쌍의 염기서열에서 특정 단백질을 얻을 수 있다.따라서 특정 유전자의 발현을 제어하는 것이 가능하다.동물에서 [30]혈관신생의 과정을 촉진하기 위해 이 전략이 사용될 수 있다는 것이 증명되었다.이와 같이 구축된 단백질을 엔도핵산가수분해효소의 촉매 도메인과 융합시켜 표적 DNA 파괴를 유도하고, 따라서 이들 단백질을 게놈 공학적 [31]도구로 사용할 수도 있다.
이를 위해 일반적으로 채택되는 방법은 두 개의 DNA 결합 단백질(각각 특별히 선택된 아연 핑거 3~6개를 포함)을 이중 가닥 DNA를 절단하기 위해 이량화해야 하는 FokI 엔도핵산가수분해효소의 촉매 도메인과 연관시키는 것이다.두 단백질은 몇 개의 뉴클레오티드가 떨어져 있는 두 개의 DNA 서열을 인식합니다.두 개의 아연 핑거 단백질을 각각의 배열에 연결하면 두 개의 FokI 도메인이 서로 더 가까워집니다.FokI는 핵산가수분해효소 활성을 가지기 위해 이합체를 필요로 하며, 이는 각 핵산가수분해효소 파트너가 고유한 DNA 서열을 인식함에 따라 특이성이 급격히 증가함을 의미한다.이 효과를 높이기 위해 FokI 핵산가수분해효소는 헤테로디머로만 [32]기능할 수 있도록 설계되었다.
선택된 시퀀스에 대한 특정 아연 핑거 핵산 분해효소 설계에 몇 가지 접근방식이 사용된다.가장 널리 사용되는 것은 아연 핑거 유닛을 알려진 특이성과 결합하는 것이다(모듈러 어셈블리.박테리아, 효모 또는 포유동물 세포를 사용하여 최적의 특이성과 최고의 세포 내성을 제공하는 조합을 식별하기 위해 다양한 선택 기술이 개발되었습니다.아연 핑거 뉴클레아제 활성의 직접적인 게놈 전체 특성은 보고되지 않았지만, 세포에서 이중 가닥 DNA 파괴의 총 횟수를 측정하는 분석 결과, 24 bp 복합 인식 부위가 있는 아연 핑거 핵산 분해 효소로 처리된 세포에서 그러한 파괴가 백그라운드 위에서 1~2개만 발생하는 것으로 밝혀졌다.mer FokI 핵산가수분해효소 [32]도메인
헤테로디머 기능성 핵산가수분해효소는 원치 않는 호모디머 활성의 가능성을 피하고 따라서 DSB의 특이성을 높일 것이다.ZFN과 TALEN 구조의 핵산가수분해효소 부분은 유사한 특성을 가지지만, 이러한 공학적 핵산가수분해효소 간의 차이는 DNA 인식 펩타이드에 있다.ZFN은 Cys2-His2 아연 핑거에 의존하고 TALEN은 TALE에 의존합니다.이들 DNA 인식 펩타이드 도메인은 모두 단백질의 조합에서 자연적으로 발견되는 특성을 가지고 있다.Cys2-His2 아연 손가락은 일반적으로 3bp 간격으로 반복되며 전사인자와 같은 다양한 핵산 상호작용 단백질에서 다양한 조합으로 발견된다.아연 핑거 도메인의 각 핑거는 완전히 독립적이며 1개의 핑거의 바인딩 용량은 인접 핑거의 영향을 받습니다.한편, TAL은 아미노산과 인식된 뉴클레오티드 쌍 사이에서 일대일 인식비로 반복하여 발견된다.아연 핑거와 TALE은 모두 반복 패턴으로 발생하기 때문에 다양한 배열 특이성을 만들기 위해 [21]다른 조합을 시도할 수 있습니다.아연 핑거는 모듈러 조립체(필요한 염기서열과 상관된 아연 핑거가 일렬로 부착되어 있는 경우), OPEN(펩타이드 도메인 대 트리플렛 뉴클레오티드의 낮은 문자열 선택 후 펩타이드 조합 대 더 높은 문자열 선택)과 같은 용어와 접근법에서 더욱 확립되었다.박테리아 시스템의 최종 표적) 및 아연 핑거 라이브러리의 박테리아 1회 선별을 사용하여 부위별 핵산 분해 효소를 만들었다.
아연 핑거 핵산가수분해효소는 다양한 게놈, 특히 아연 핑거 컨소시엄의 연구실에서 이미 사용된 연구 개발 도구입니다.미국 기업인 Sangamo BioSciences는 아연 핑거 핵산가수분해효소를 사용하여 줄기세포의 유전자 공학과 치료 [33][34]목적의 면역 세포 변형 연구를 수행하고 있습니다.변형된 T림프구는 현재 뇌종양(교아종)을 치료하기 위해 1단계 임상시험을 진행 중이며 에이즈와의 [32]싸움에서 사용되고 있다.
탈렌
전사활성화유사 이펙터핵산가수분해효소(TALENs)는 33 또는 34-아미노산 반복의 배열을 특징으로 하는 특이 DNA 결합 단백질이다.TALEN은 핵산가수분해효소의 DNA 절단 도메인을 TALE 도메인으로 융합하여 설계된 인공 제한 효소이며, TALEN은 고유한 DNA 염기서열을 인식하도록 특별히 조정될 수 있다.이러한 융합 단백질은 살아있는 [35]세포에서 염기서열 삽입, 삭제, 복구 및 치환과 같은 표적 게놈 수정을 수행할 수 있는 유전자 편집 애플리케이션을 위한 쉽게 표적화 가능한 "DNA 가위" 역할을 합니다.원하는 DNA 염기서열을 결합하도록 설계될 수 있는 DNA 결합 도메인은 식물 병원성 크산토마노스 앱에 의해 배설되는 DNA 결합 단백질인 TAL 이펙터에서 유래합니다.TAL 이펙터는 34개의 아미노산의 고도로 보존된 배열을 각각 포함하는 반복 도메인으로 구성되며 표적 부위 내에서 단일 DNA 뉴클레오티드를 인식한다.핵산가수분해효소는 표적 부위에 이중 가닥 절단을 만들 수 있으며, 이는 오류 발생 가능성이 높은 비호몰로지 말단 결합(NHEJ)에 의해 복구될 수 있으며, 작은 삽입 또는 결실의 도입을 통해 유전자 교란을 일으킨다.아미노산 위치 12, 13의 이른바 반복 가변 디레지드(RVDs)를 제외하고 각 반복은 보존된다.RVD는 TALE이 결합하는 DNA 시퀀스를 결정합니다.TALE 반복과 해당 DNA 염기서열 사이의 이 간단한 일대일 대응은 새로운 DNA 염기서열을 인식하기 위해 반복배열을 조립하는 과정을 쉽게 만든다.이러한 TAL은 DNA 핵산가수분해효소 FokI로부터 촉매 도메인으로 융합되어 전사 활성제 유사 이펙터 뉴클레아제(TALEN)를 생성할 수 있다.결과 TALEN 구조는 특이성과 활성을 결합하여 사전 선택된 부위에서만 DNA 염기서열을 결합하고 분해하는 엔지니어링된 염기서열 특이 핵산 분해 효소를 효과적으로 생성한다.TALEN 표적 인식 시스템은 예측하기 쉬운 코드를 기반으로 합니다.TAL핵산가수분해효소는 부분적으로 30개 이상의 염기쌍 결합 부위의 길이로 인해 표적 특이적이다.TALEN은 전체 [36]게놈에서 단일 뉴클레오티드의 6개 염기쌍 범위 내에서 수행될 수 있다.
TALEN 구조는 설계된 아연 핑거 핵산가수분해효소와 유사한 방법으로 사용되며 표적 돌연변이 유발에 있어 (1) DNA 결합특성이 더 높고 (2) 표적 외 효과가 낮으며 (3) DNA 결합 도메인의 구축이 더 용이하다는 세 가지 장점이 있다.
크리스퍼
CRISPRs(Clustered Regularly Interspace Short Palindromic Repeats)는 박테리아가 바이러스로부터 후천성 면역의 일종으로 사용하는 유전자 요소이다.그것들은 바이러스 게놈에서 유래하여 박테리아 게놈에 통합되는 짧은 배열로 구성되어 있다.Cas(CRISPR 관련 단백질)는 이러한 염기서열을 처리하고 일치하는 바이러스 DNA 염기서열을 절단합니다.Cas 유전자를 포함한 플라스미드와 특이하게 구성된 CRISPR을 진핵세포에 도입함으로써 원하는 위치에서 [37]진핵유전체를 절단할 수 있다.
핵염기 수정에 의한 편집(BASE 편집)
핵산을 효율적으로 편집하는 가장 초기의 방법 중 하나는 핵산 가이드 배열에 의해 지시된 핵산 수정 효소를 사용하는 것으로 1990년대에 처음 기술되었고 보다 [2][38][39][40]최근에 부활을 보았다.이 방법은 게놈 DNA 가닥을 절단할 필요가 없으므로 DNA 가닥 절단과 관련된 무작위 삽입 및 결실을 피할 수 있다는 장점이 있다.단일 뉴클레오티드 변경이 필요한 정밀한 편집에만 적합하며 이러한 유형의 [40][41]편집에 매우 효율적인 것으로 밝혀졌다.
아크컷
ARCUT는 인공 제한 DNA 절단기의 약자로, 코미야마씨가 개발한 기술입니다.이 방법은 염색체 내의 분열 부위를 식별하기 위해 의사보완 펩타이드 핵산(pcPNA)을 사용한다.pcPNA가 부위를 특정하면 세륨(CE)과 EDTA(화학혼합물)에 의해 절제가 이루어지며, EDTA는 스플라이싱 기능을 수행한다.[42]
엔지니어링된 핵산가수분해효소의 정밀도 및 효율성
유전자 편집의 메가핵산가수분해효소 방법은 위에서 언급한 방법 중 가장 효율적이지 않다.DNA 결합 원소와 분해 원소의 특성으로 인해, 1,000개의 뉴클레오티드 [8]당 하나의 잠재적 표적을 인식하는 것으로 제한됩니다.ZFN은 메가핵산가수분해효소의 한계를 극복하기 위해 개발되었다.ZFN이 인식할 수 있는 가능한 목표물의 수는 140개의 뉴클레오티드 [8]중 1개로 증가했다.그러나 두 방법 모두 DNA 결합 요소가 서로 영향을 미치기 때문에 예측할 수 없습니다.그 결과, 고도의 전문 지식과 장시간의 고비용 검증 프로세스가 요구됩니다.
TALE 핵산가수분해효소는 가장 정확하고 구체적인 방법으로서 앞의 두 방법보다 더 높은 효율성을 산출한다.DNA결합요소는 TALE 서브유닛의 배열로 구성되며, 각 서브유닛은 서로 독립적으로 특정 DNA 뉴클레오티드 사슬을 인식할 수 있는 능력을 가지기 때문에 높은 정밀도로 표적부위가 많아지기 때문이다.새로운 TALE 핵산 분해 효소는 분자생물학과 단백질 [8]공학에 대한 특정한 전문 지식을 가지고 만드는 데 약 1주일과 수백 달러가 소요됩니다.
CRISPR 핵산가수분해효소는 TALE 핵산가수분해효소에 비해 정밀도가 약간 낮다.이것은 CRISPR이 유도하는 이중 가닥을 복구하기 위해 사용하는 가이드 RNA를 생산하기 위해 한쪽 끝에 특정 뉴클레오티드가 필요하기 때문에 발생합니다.200달러도 안 들고 시간도 [8]며칠 안 걸리는 가장 빠르고 저렴한 방법인 것으로 나타났습니다.CRISPR은 또한 설계가 단백질 대신 가이드 RNA에 있기 때문에 분자 생물학에 대한 최소한의 전문 지식을 필요로 한다.ZFN 및 TALEN 방법보다 CRISPR이 갖는 한 가지 주요 장점은 약 80nt CRISPR sgRNA를 사용하여 다른 DNA 염기서열을 표적화하도록 유도할 수 있는 반면 ZFN 및 TALEN 방법 모두 각 DNA [43]염기서열을 표적화하기 위해 생성된 단백질의 구축과 테스트가 필요하다는 것이다.
활성핵산가수분해효소의 표적외 활동이 유전적 및 유기적 수준에서 잠재적으로 위험한 결과를 초래하기 때문에 메가핵산가수분해효소, ZFN, CRISPR 및 TALEN 기반 융합의 정밀도는 활발한 연구 영역이었다.가변 수치가 보고되었지만, ZFN은 TALEN 방법이나 RNA 유도 핵산 분해효소보다 세포독성이 더 높은 경향이 있는 반면, TALEN과 RNA 유도 접근법은 가장 큰 효율성을 가지고 목표 외의 효과는 [44]더 적은 경향이 있다.DNA 결합과 핵산가수분해효소 활성 사이의 최대 이론 거리에 기초하여 TALEN 접근법은 가장 정확한 [8]결과를 도출한다.
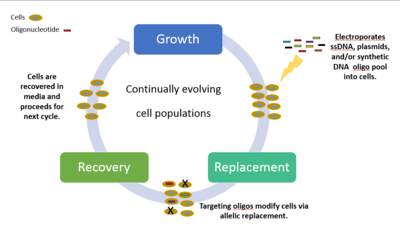
다중 자동 게놈 엔지니어링(MAGE)
게놈 다양성과 관련된 모든 표현형을 연구하고자 하는 과학자들과 연구자들을 위한 방법들은 매우 느리고, 비싸고, 비효율적이었다.이 새로운 혁명이 일어나기 전에 연구자들은 단일 유전자 조작을 하고 게놈을 한 번에 한 부분씩 조정하고 표현형을 관찰하고 다른 단일 유전자 조작으로 [45]이 과정을 다시 시작해야 했다.따라서, 하버드 대학의 와이즈 연구소의 연구원들은 생체 내 게놈 편집 과정을 개선하는 강력한 기술인 MAGE를 고안했다.게놈을 빠르고 효율적으로 조작할 수 있습니다이 모든 것은 작은 부엌 식탁 위에 놓을 수 있을 정도로 작은 기계에서 이루어집니다이 돌연변이는 세포 유사분열 중에 자연적으로 발생하는 변화와 결합되어 수십억 개의 세포 돌연변이를 일으킨다.
화학적으로 조합된 합성 단일사슬 DNA(ssDNA)와 올리고뉴클레오티드 풀이 세포의 표적 영역에 도입되어 유전자 변형을 생성한다.순환 과정은 ssDNA의 변형과 그 후 박테리오파지 상동 재조합 단백질이 그들의 게놈 표적에 대한 ssDNA의 어닐링을 매개하는 발육을 포함한다.선택적 표현형 마커를 대상으로 한 실험을 선별하고 차등 매질 위에 세포를 도금함으로써 식별한다.각 사이클은 최종적으로 2.5시간이 소요되며, 등성배양을 성장시키고 돌연변이를 특징짓는 데 추가 시간이 필요합니다.MAGE는 여러 사이트를 대상으로 하는 돌연변이 유발 ssDNA 라이브러리를 반복적으로 도입함으로써 세포 집단에서 조합적 유전적 다양성을 발생시킬 수 있다.단일 뉴클레오티드 염기쌍에서 전체 게놈 또는 유전자 네트워크에 이르기까지 최대 50개의 게놈 편집이 수일 [45]내에 동시에 이루어질 수 있습니다.
MAGE 실험은 (i) 많은 표적 부위, 단일 유전자 돌연변이, (ii) 많은 표적 부위, 많은 유전자 돌연변이, 그리고 (ii) 많은 표적 부위, 많은 유전자 [45]돌연변이의 세 가지 클래스로 나눌 수 있다.세 번째 등급의 예는 2009년에 반영되었는데, Church와 동료들은 보통 토마토 씨앗에서 발견되고 항암 특성과 관련이 있는 항산화 물질인 리코펜을 정상량의 5배나 생산하도록 대장균을 프로그래밍할 수 있었다.MAGE를 적용하여 대장균의 1-디옥시-d-자일룰로오스-5-인산(DXP) 대사 경로를 최적화하여 이소프레노이드 리코펜을 과다 생산하였다.그들은 약 3일이 걸렸고 재료비는 1,000달러가 조금 넘었다.MAGE가 게놈을 변경할 수 있는 용이성, 속도 및 비용 효율은 업계가 바이오 엔지니어링, 바이오 에너지, 생물의학 엔지니어링, 합성 생물학, 제약, 농업 및 화학 산업에서 중요한 화합물의 제조 및 생산에 접근하는 방식을 변화시킬 수 있다.
적용들
2012년 현재 효율적인 게놈 편집은 식물에서 동물에 이르는 광범위한 실험 시스템에 대해 개발되었으며 [46]임상적 관심 범위를 넘어 연구소에서 표준 실험 전략이 되고 있다.최근 생쥐, 제브라피시, 옥수수 및 담배 ZFN 매개 돌연변이 세대와 TALEN 기반 접근법의 개선은 이 방법의 중요성을 증명하고 있으며, 그 목록은 빠르게 확대되고 있다.조작된 핵산 분해 효소를 이용한 게놈 편집은 식물과 동물의 유전자 기능을 연구하는 것에서부터 인간의 유전자 치료에 이르기까지 생명과학의 많은 분야에 기여할 것이다.예를 들어, 새로운 기능을 수행하기 위해 세포와 유기체를 엔지니어링하는 것을 목표로 하는 합성생물학 분야는 유전자 요소를 추가하거나 제거하여 [46]복잡한 시스템을 만드는 공학적 핵산가수분해효소의 능력으로부터 이익을 얻을 가능성이 높다.또한 유전자 기능은 조작된 핵산가수분해효소를 가진 줄기세포를 사용하여 연구될 수 있다.
다음은 이 방법으로 수행할 수 있는 특정 작업입니다.
동물의 표적 유전자 변형
유전자 공학에서의 최근 발견, 특히 유전자 편집과 소의 생식 기술(예: 시험관내 배아 배양)의 최신 개선의 조합은 합성 고특이 엔도핵산가수분해효소를 사용하여 수정 난모세포에서 직접 게놈 편집을 가능하게 한다.RNA유도핵산가수분해효소:Cas9(CRISPR/Cas9)와 관련된 클러스터화된 규칙적인 인터스페이스 짧은 회문 반복은 사용 가능한 방법의 범위를 더욱 늘리는 새로운 도구입니다.특히 CRISPR/Cas9 공학적 엔도핵산가수분해효소는 포유동물 [47]접합체에 세포질 직접주입(CDI)을 통해 동시 녹아웃(KO)을 위한 다중 가이드 RNA를 한 단계로 사용할 수 있도록 한다.
또한 유전자 편집은 대서양 연어 등 양식 어종의 특정 어류에 적용할 수 있다.물고기의 유전자 편집은 현재 실험적이지만, 가능성에는 성장, 질병 저항성, 불임, 통제된 번식, 그리고 색이 포함됩니다.이러한 특징들을 고르는 것은 [48]물고기들에게 더 지속 가능한 환경과 더 나은 복지를 제공할 수 있다.
Aqua Advantage 연어는 AquaBounty Technologies가 개발한 유전자 변형 대서양 연어입니다.대서양 연어의 성장호르몬 조절 유전자는 태평양 치누크 연어의 성장호르몬 조절 유전자와 바다 입에서[49] 나오는 프로모터 배열로 대체된다.
단세포 전사체학, 게놈 편집 및 새로운 줄기세포 모델의 병행 개발 덕분에 우리는 이제 기능 유전학이 더 이상 동물 모델에 국한되지 않고 인간 샘플에서 직접 수행될 수 있는 과학적으로 흥미로운 시기로 접어들고 있다.단세포 유전자 발현 분석은 기능 연구를 위해 핵심 후보 유전자가 확인되고 있는 인간 발달의 전사 로드맵을 해결했다.CRISPR 기반의 게놈 편집 툴은 실험을 가이드하기 위해 전지구적 전사체 데이터를 사용하여 인간의 [50]환경에서 기능을 명확히 하기 위해 주요 유전자를 파괴하거나 제거하는 것을 가능하게 했습니다.
식물의 표적 유전자 변형
메가핵산가수분해효소,[51] ZFNs, TALEN을 사용한 게놈 편집은 식물에서 유전자 조작을 위한 새로운 전략을 제공하며 내생 유전자를 수정함으로써 원하는 식물 특성의 공학을 도울 가능성이 높다.예를 들어, 주요 작물 종에 대한 부위 특이적 유전자 첨가는 번식 [32]과정 동안 그들의 공동 분리를 보장하기 위해 여러 가지 원하는 특성이 물리적으로 연결되는 '여행 쌓기'에 사용될 수 있다.이러한 경우의 진전은 아라비도시스 탈리아나와[52][53][54] 제아메이스에서 최근 보고되었다.Arabidopsis thaliana에서는 ZFN 지원 유전자 타겟팅을 사용하여 2개의 제초제 내성 유전자(담배 아세트락테이트 합성효소 SuRA 및 SuRB)가 [52]돌연변이를 동반한 2%의 높은 변환 세포로 SuR 위치에 도입되었다.Zea mays에서는 표적 궤적의 교란이 ZFN 유도 DSBs에 의해 달성되었으며,[55] 그 결과 NHEJ. ZFN은 또한 표적 내인성 궤적 IPK1에 제초제 내성 유전자 발현 카세트(PAT)를 구동하기 위해 사용되었다.재생 식물에서 관찰된 이러한 유전자 변형은 유전될 수 있고 다음 [55]세대에 전파되었다.작물 개선에 게놈 편집 기술을 적용한 잠재적인 성공 사례는 바나나에서 찾을 수 있습니다. 이 바나나에서는 과학자들이 CRISPR/Cas9 편집을 사용하여 바나나 B 게놈(Musa spp.)의 내인성 바나나 스트릭 바이러스를 비활성화하여 바나나 [56]사육의 큰 어려움을 극복했습니다.
또한 TALEN 기반 게놈 공학은 [57]식물에서 사용하기 위해 광범위하게 테스트되고 최적화되었습니다.TALEN 퓨전 또한 콩기름[59] 제품의 품질을 개선하고 감자의 저장 가능성을[60] 높이기 위해 미국의 식재료 회사인 [58]Calyxt에 의해 사용되어 왔다.
ZFN 매개 [61]타겟팅을 사용하여 식물 게놈 편집을 개선하려면 몇 가지 최적화가 필요합니다.핵산가수분해효소의 신뢰성 있는 설계와 후속 시험, 핵산가수분해효소의 독성 부재, 표적화를 위한 식물 조직의 적절한 선택, 효소 활성의 유도 경로, 표적 외 돌연변이 유발의 부재, 그리고 돌연변이 사례의 [61]신뢰성 있는 검출이 필요하다.
식물에서 CRISPR/Cas9의 일반적인 전달 방법은 아그로박테륨 기반의 [62]변환이다.T-DNA는 T4SS 메커니즘에 의해 식물 게놈에 직접 도입된다.Cas9 및 gRNA 기반 발현 카세트는 식물 [62]적용을 위해 아그로박테륨에서 변형된 Ti 플라스미드로 변환된다.살아있는 식물에서 Cas9의 전달을 개선하기 위해 바이러스는 보다 효과적인 트랜스유전자 [62]전달을 사용하고 있다.
| 에 관한 일련의 기사의 일부 |
| 합성생물학 |
|---|
| 합성 생물 회로 |
| 게놈 편집 |
| 인공 세포 |
| 이종생물학 |
| 기타 토픽 |
조사.
유전자 치료
이상적인 유전자 치료 방법은 결함이 있는 유전자를 자연적 위치에서 정상적인 대립 유전자로 대체하는 것입니다.이것은 종종 있는 [63][64]경우처럼 유전자의 작은 부분만 바뀌어야 할 때 완전한 코드 배열과 조절 배열을 포함할 필요가 없기 때문에 바이러스 전달 유전자보다 유리하다.부분적으로 치환된 유전자의 발현 또한 바이러스 벡터에 의해 운반되는 완전한 유전자보다 정상적인 세포 생물학과 더 일치합니다.
TALEN 기반 게놈 편집의 임상적 사용은 2015년 생후 11개월 아동의 CD19+ 급성 림프아구성 백혈병 치료에서 처음 사용되었다.변형된 기증자 T세포는 백혈병 세포를 공격하고, 알렘투주맙에 내성을 가지며,[65][66] 도입 후 숙주 면역체계에 의한 검출을 회피하도록 설계되었다.
다운증후군, 척추 이피다, 무뇌증,[67] 터너와 클라인펠터 증후군과 같은 유전적 질병을 일으키는 유전자 돌연변이를 교정하기 위해 CRISPR-Cas9을 사용하는 세포와 동물에 대한 광범위한 연구가 이루어졌다.
2019년 2월 캘리포니아 리치몬드에 본사를 둔 Sangamo Therapeutics와 함께 일하는 의학자는 헌터 [68]증후군 환자의 DNA를 영구적으로 바꾸는 최초의 "인체" 인간 유전자 편집 요법을 발표했다.아연 핑거뉴클레아제(ZFN)를 이용한 유전자 편집을 포함한 상모 임상시험이 [69]진행 중이다.
질병의 근절
연구자들은 [70]CRISPR-Cas9 유전자 드라이브를 사용하여 말라리아에 대한 매개체인 A. gambiae의 불임과 관련된 유전자를 수정했다.이 기술은 황열, 뎅기열, 지카 [71]등과 같은 다른 벡터 매개 질환을 근절하는 데 더 큰 의미가 있다.
CRISPR-Cas9 시스템은 임상 유전자형 또는 역학 격리체를 대상으로 하여 세균 종의 개체 수를 조절하도록 프로그래밍할 수 있습니다.병원체를 제거함으로써 유해 세균보다 유익한 박테리아 종을 선택적으로 활성화 할 수 있어 광범위한 [45]항생제보다 유리하다.
HIV, 헤르페스, B형 간염 바이러스 등 인간 바이러스를 대상으로 한 치료법에 대한 항바이러스 응용이 연구되고 있다.CRISPR은 바이러스 세포 표면 수용체 [43]단백질을 코드하는 유전자를 교란시키기 위해 바이러스 또는 숙주를 대상으로 사용될 수 있다.2018년 11월, He Jiankui는 두 개의 인간 배아를 편집하여 HIV가 세포에 들어가는 수용체를 코드화하는 CCR5의 유전자를 비활성화하려고 시도했다고 발표했다.그는 쌍둥이 소녀인 루루와 나나가 몇 주 전에 태어났다고 말했다.그는 소녀들이 CCR5와 함께 CCR5의 기능성 복사본을 가지고 다녔으며 여전히 HIV에 취약하다고 말했다.그 작품은 비윤리적이고 위험하며 [72]시기상조라는 비난을 많이 받았다.
2019년 1월, 중국의 과학자들은 최초의 복제 원숭이인 종중과 화화와 양 돌리와 같은 복제 기술과 허젠쿠이가 최초의 유전자 변형 원숭이를 만들 때 사용한 것으로 알려진 것과 같은 유전자 편집 기술인 크리스프-카스9 기술을 사용하여 5마리의 동일한 복제 유전자 편집 원숭이가 탄생했다고 보고했다.인간 아기 루루와 나나를 낳았다.원숭이 복제는 몇 가지 의학적 [73][74]질병을 연구하기 위해 만들어졌다.
가까운 미래에 새로운 CRISPR 시스템은 또한 인간이 잘 걸리는 질병과 상태를 근절할 수 있을 것이다.이 새로운 기술로 과학자들은 인간의 정자 세포와 난자의 유전자를 채취하여 암이나 다른 이상 또는 원치 않는 결함을 활성화시키는 유전자를 대체할 수 있을 것이다.이것은 아이를 갖는 것에 대해 걱정하고 정상적인 삶을 살 수 없는 부모들의 스트레스를 덜어줄 것이다.이 과정을 한 세대만 거치면 인류의 미래 전체가 기형이나 소인 [75]상태의 문제에 대해 걱정하지 않아도 될 것이다.
전망과 한계
미래에는 조작된 핵산가수분해효소에 의한 게놈 편집 연구의 중요한 목표는 핵산가수분해효소 [76]작용의 안전성과 특이성의 향상이어야 한다.예를 들어, 목표를 벗어난 이벤트를 탐지하는 능력을 향상시키면 이벤트를 예방하는 방법을 배울 수 있는 능력을 향상시킬 수 있습니다.또한, ZFN에 사용되는 아연 핑거는 완전히 특이적이지 않고, 어떤 것들은 독성 반응을 일으킬 수 있다.그러나 독성은 [64]ZFN의 절단 영역에 가해진 변형에 의해 감소된 것으로 보고되었다.
게다가, 다나 캐럴의 유전자 변형에 대한 연구는 DNA의 기본 재조합과 복구 기계에 대한 더 나은 이해의 필요성을 보여주었다.향후 2차 표적을 식별하는 가능한 방법은 ZFN을 발현하는 세포에서 절단된 끝을 포착하고 높은 처리량 [64]염기서열을 사용하여 측면 DNA를 배열하는 것이다.
CRISPR의 사용 편의성과 비용 효율성으로 인해 현재 광범위한 연구가 진행 중입니다.CRISPR의 발견이 [43]얼마나 최근인가에도 불구하고, 현재 CRISPR에 관한 출판물은 ZFN이나 TALEN보다 많다.CRISPR과 TALEN은 모두 정밀도와 효율성으로 인해 대규모 프로덕션에서 구현될 수 있는 선택지로 선호됩니다.
유전자 편집은 인위적인 유전공학이 없는 자연스러운 과정으로도 일어난다.유전자 코드를 편집할 수 있는 매개체는 바이러스 또는 서브바이러스 RNA-에이전트입니다.
GEEN은 역유전학의 많은 다른 방법보다 효율이 높지만, 여전히 효율성이 높지 않다. 많은 경우 치료된 인구의 절반 미만이 원하는 [52]변화를 얻는다.예를 들어, 돌연변이를 만들기 위해 셀의 NHEJ를 사용할 계획일 때 셀의 HDR 시스템도 낮은 돌연변이율로 DSB를 수정하는 작업을 하게 됩니다.
전통적으로, 쥐는 질병 모델의 숙주로서 연구자들에게 가장 흔한 선택이었습니다.CRISPR은 돼지, 개, 그리고 인간이 [77][78]아닌 영장류와 같은 더 큰 동물에서 유전자 변형 질병 모델을 만들어냄으로써 이 모델과 인간 임상 실험 사이의 차이를 메우는데 도움을 줄 수 있습니다.CRISPR-Cas9 시스템을 사용하여 프로그램된 Cas9 단백질과 sgRNA는 설치류에서 유전자 변환 모델을 만들 때 원하는 유전자 수정을 달성하기 위해 수정 접합체에 직접 도입될 수 있습니다.이를 통해 트랜스제닉 라인을 생성할 때 일반적인 셀 타깃팅 단계를 우회할 수 있으며, 그 결과 생성 시간을 90% [78]단축할 수 있습니다.
CRISPR이 그 효과와 함께 가져오는 가능성 중 하나는 이종 이식의 응용이다.이전 연구 실험에서 CRISPR은 내인성 레트로바이러스를 표적화하고 제거하는 능력을 입증했으며, 이는 질병의 전염 위험을 줄이고 면역 장벽을 [43]낮췄다.이러한 문제를 제거함으로써 기증자의 장기 기능이 향상되어 이 응용 프로그램이 보다 현실로 다가갑니다.
식물에서, 게놈 편집은 생물다양성 보존에 대한 실행 가능한 해결책으로 여겨진다.유전자 구동은 비록 상당한 [79]관련 위험이 있지만 침습 종의 생식 속도를 바꿀 수 있는 잠재적인 도구이다.
인간 기능 강화
많은 트랜스휴머니스트들은 게놈 편집을 인간 [80][81][82]강화를 위한 잠재적 도구로 보고 있다.호주의 생물학자이자 유전학 교수인 데이비드 앤드류 싱클레어는 "게놈 편집 기능이 있는 새로운 기술을 통해 개인(...)이 더 건강한 아이를 가질 수 있게 될 것" 즉 디자이너의 [83]아기를 가질 수 있다고 말합니다.2016년 9월 Nuffield Council of BioEthics의 보고서에 따르면, 예를 들어 야간 시력과 [84][85]후각을 향상시키기 위해 다른 유기체의 유전자 또는 완전히 합성된 유전자를 가진 사람들을 증강시킬 수 있을 것이다.George Church는 수면의 필요성 감소, 알츠하이머 병으로부터 보호하는 인지 관련 변화, 질병 저항력 및 학습 능력 향상과 같은 유익한 특징에 대한 잠재적 유전자 변형 목록을 관련 연구 및 잠재적인 부정적인 [86][87]영향과 함께 작성했습니다.
미국 국립과학원과 국립의학원은 2017년 2월 인간 게놈 [88]편집을 지원하는 보고서를 발표했다.그들은 "엄격한 [89]감독 아래 심각한 상태에 대해서만" 안전과 효율성 문제에 대한 해답이 발견되면 언젠가 게놈 편집을 위한 임상실험이 허용될 것이라고 권고했다.
리스크
2016년 미국 정보 커뮤니티 성명에 대한 세계 위협 평가에서 미국 국가 정보 책임자 제임스 R. "서방 국가와는 다른" 규제 또는 윤리적 기준을 가진 국가들에 의해 수행되는 게놈 편집은 아마도 유해한 생물학적 물질이나 제품의 생성 위험을 증가시킨다고 말하면서 게놈 편집을 대량 살상 무기로 지목했다.성명에 따르면 이 기술의 광범위한 보급, 저비용 및 빠른 개발 속도에 따르면 의도적이거나 의도적이지 않은 오용은 광범위한 경제 및 국가 안보의 영향을 [90][91][92]미칠 수 있다.예를 들어 CRISPR과 같은 기술은 주요 작물을 [92]전멸시키는 전염병을 일으키는 "살생 모기"를 만드는 데 사용될 수 있다.
너필드 생명윤리위원회의 2016년 9월 보고서에 따르면 유전자 코드를 편집하는 도구의 단순성과 저비용은 아마추어 또는 "바이오 해커"가 그들 자신의 실험을 수행할 수 있도록 허용하고 유전자 변형 버그의 방출에 따른 잠재적 위험을 야기할 것이다.이 리뷰는 또한 개인의 게놈을 수정하고 그러한 변화를 미래 세대에 물려주는 것의 위험과 이득이 매우 복잡하기 때문에 긴급한 윤리적 정밀 조사가 필요하다는 것을 발견했다.이러한 수정은 의도하지 않은 결과를 초래할 수 있으며, 이는 변경된 유전자가 그들의 정자나 [84][85]난자에 있을 수 있기 때문에 아이뿐만 아니라 미래의 자녀들에게도 해를 끼칠 수 있다.2001년 호주의 연구원 Ronald Jackson과 Ian Ramshaw는 제공된 민감한 정보가 제조로 이어질 수 있기 때문에 불임을 야기할 수 있는 변형된 쥐에 감염시킴으로써 호주의 주요 해충인 쥐의 잠재적 통제를 탐구한 논문을 바이러스학 저널에 발표하여 비난을 받았다.천연두와 같은 [85][93]인간에게 영향을 미칠 수 있는 다른 수두 바이러스의 백신 내성 변종을 만들기 위해 지식을 사용할 수 있는 잠재적 생물 테러범들에 의한 생물 무기.게다가, 유전자 드라이브를 [85][94][95]야생 개체군에 방출하는 것의 생태학적 위험에 대한 추가적인 우려가 있다.
「 」를 참조해 주세요.
- CRISPR/Cpf1
- RNA편집
- 후생유전자 편집
- 생식기 선택 기술
- ssDNA 유도 아르고나이트핵산가수분해효소인 NgAgo
레퍼런스
- ^ Bak, Rasmus O.; Gomez-Ospina, Natalia; Porteus, Matthew H. (August 2018). "Gene Editing on Center Stage". Trends in Genetics. 34 (8): 600–611. doi:10.1016/j.tig.2018.05.004. ISSN 0168-9525. PMID 29908711. S2CID 49269023.
- ^ a b c Woolf TM (April 1998). "Therapeutic repair of mutated nucleic acid sequences". Nature Biotechnology. 16 (4): 341–4. doi:10.1038/nbt0498-341. PMID 9555723. S2CID 9210810.
- ^ "Method of the Year 2011". Nature Methods. 9 (1): 1. January 2012. doi:10.1038/nmeth.1852. PMID 22312634.
- ^ Science News Staff (17 December 2015). "Breakthrough of the Year: CRISPR makes the cut".
- ^ Esvelt KM, Wang HH (2013). "Genome-scale engineering for systems and synthetic biology". Molecular Systems Biology. 9 (1): 641. doi:10.1038/msb.2012.66. PMC 3564264. PMID 23340847.
- ^ Tan WS, Carlson DF, Walton MW, Fahrenkrug SC, Hackett PB (2012). "Precision editing of large animal genomes". Advances in Genetics Volume 80. Advances in Genetics. Vol. 80. pp. 37–97. doi:10.1016/B978-0-12-404742-6.00002-8. ISBN 9780124047426. PMC 3683964. PMID 23084873.
- ^ Puchta H, Fauser F (2013). "Gene targeting in plants: 25 years later". The International Journal of Developmental Biology. 57 (6–8): 629–37. doi:10.1387/ijdb.130194hp. PMID 24166445.
- ^ a b c d e f Boglioli E, Richard M. "Rewriting the book of life: a new era in precision genome editing" (PDF). Boston Consulting Group. Retrieved November 30, 2015.
- ^ Church G. "The future of genetic codes and BRAIN codes". YouTube. NIHvcast. Retrieved 10 February 2017.
- ^ Cyranoski D (May 2019). "China set to introduce gene-editing regulation following CRISPR-baby furore". Nature. doi:10.1038/d41586-019-01580-1. PMID 32424191. S2CID 182604140. Retrieved 20 May 2019.
- ^ Cheong KH, Koh JM, Jones MC (July 2019). "Black Swans of CRISPR: Stochasticity and Complexity of Genetic Regulation". BioEssays. 41 (7): e1900032. doi:10.1002/bies.201900032. PMID 31090950. S2CID 155086967.
- ^ a b c Stokstad, Erik (2021-05-26). "U.K. set to loosen rules for gene-edited crops and animals". Science AAAS. Retrieved 2021-05-27.
- ^ "US Trial Shows 3 Cancer Patients Had Their Genomes Altered Safely by CRISPR". ScienceAlert. Retrieved 2020-02-09.
- ^ Altae-Tran, Han; Kannan, Soumya; Demircioglu, F. Esra; Oshiro, Rachel; Nety, Suchita P.; McKay, Luke J.; Dlakić, Mensur; Inskeep, William P.; Makarova, Kira S.; Macrae, Rhiannon K.; Koonin, Eugene V. (2021-10-01). "The widespread IS200/IS605 transposon family encodes diverse programmable RNA-guided endonucleases". Science. 374 (6563): 57–65. Bibcode:2021Sci...374...57A. doi:10.1126/science.abj6856. ISSN 0036-8075. PMC 8929163. PMID 34591643. S2CID 238237947.
- ^ "New programmable gene editing proteins found outside of CRISPR systems". Broad Institute. 2021-09-09. Retrieved 2021-10-04.
- ^ Lodish H, Berk A, Zipursky SL, Matsudaira P, Baltimore D, Darnell J (2000). "Chapter 8.5: Gene Replacement and Transgenic Animals: DNA Is Transferred into Eukaryotic Cells in Various Ways". Molecular Cell Biology (4th ed.). W. H. Freeman and Company. ISBN 978-0-7167-3136-8.
- ^ a b Rocha-Martins M, Cavalheiro GR, Matos-Rodrigues GE, Martins RA (August 2015). "From Gene Targeting to Genome Editing: Transgenic animals applications and beyond". Anais da Academia Brasileira de Ciencias. 87 (2 Suppl): 1323–48. doi:10.1590/0001-3765201520140710. PMID 26397828.
- ^ "The Nobel Prize in Physiology or Medicine 2007". The Nobel Foundation. Retrieved December 15, 2008.
- ^ Jasin M (June 1996). "Genetic manipulation of genomes with rare-cutting endonucleases". Trends in Genetics. 12 (6): 224–8. doi:10.1016/0168-9525(96)10019-6. PMID 8928227.
- ^ Stoddard BL (February 2005). "Homing endonuclease structure and function". Quarterly Reviews of Biophysics. 38 (1): 49–95. doi:10.1017/s0033583505004063. PMID 16336743. S2CID 27841011.
- ^ a b de Souza N (January 2012). "Primer: genome editing with engineered nucleases". Nature Methods. 9 (1): 27. doi:10.1038/nmeth.1848. PMID 22312638. S2CID 26924628.
- ^ a b c Smith J, Grizot S, Arnould S, Duclert A, Epinat JC, Chames P, et al. (2006). "A combinatorial approach to create artificial homing endonucleases cleaving chosen sequences". Nucleic Acids Research. 34 (22): e149. doi:10.1093/nar/gkl720. PMC 1702487. PMID 17130168.
- ^ Seligman LM, Chisholm KM, Chevalier BS, Chadsey MS, Edwards ST, Savage JH, Veillet AL (September 2002). "Mutations altering the cleavage specificity of a homing endonuclease". Nucleic Acids Research. 30 (17): 3870–9. doi:10.1093/nar/gkf495. PMC 137417. PMID 12202772.
- ^ Chevalier BS, Kortemme T, Chadsey MS, Baker D, Monnat RJ, Stoddard BL (October 2002). "Design, activity, and structure of a highly specific artificial endonuclease". Molecular Cell. 10 (4): 895–905. doi:10.1016/S1097-2765(02)00690-1. PMID 12419232.
- ^ Arnould S, Chames P, Perez C, Lacroix E, Duclert A, Epinat JC, et al. (January 2006). "Engineering of large numbers of highly specific homing endonucleases that induce recombination on novel DNA targets". Journal of Molecular Biology. 355 (3): 443–58. doi:10.1016/j.jmb.2005.10.065. PMID 16310802.
- ^ Rationally-designed meganucleases with altered sequence specificity and DNA-binding affinity, 2006-10-18, retrieved 2018-08-11
- ^ Ashworth J, Taylor GK, Havranek JJ, Quadri SA, Stoddard BL, Baker D (September 2010). "Computational reprogramming of homing endonuclease specificity at multiple adjacent base pairs". Nucleic Acids Research. 38 (16): 5601–8. doi:10.1093/nar/gkq283. PMC 2938204. PMID 20435674.
- ^ Redondo P, Prieto J, Muñoz IG, Alibés A, Stricher F, Serrano L, et al. (November 2008). "Molecular basis of xeroderma pigmentosum group C DNA recognition by engineered meganucleases". Nature. 456 (7218): 107–11. Bibcode:2008Natur.456..107R. doi:10.1038/nature07343. PMID 18987743. S2CID 4300643.
- ^ a b Baker M (January 2012). "Gene-editing nucleases". Nature Methods. 9 (1): 23–6. doi:10.1038/nmeth.1807. PMID 22312637. S2CID 37050234.
- ^ Rebar EJ, Huang Y, Hickey R, Nath AK, Meoli D, Nath S, et al. (December 2002). "Induction of angiogenesis in a mouse model using engineered transcription factors". Nature Medicine. 8 (12): 1427–32. doi:10.1038/nm1202-795. PMID 12415262. S2CID 23318821.
- ^ Kim YG, Cha J, Chandrasegaran S (February 1996). "Hybrid restriction enzymes: zinc finger fusions to Fok I cleavage domain". Proceedings of the National Academy of Sciences of the United States of America. 93 (3): 1156–60. Bibcode:1996PNAS...93.1156K. doi:10.1073/pnas.93.3.1156. PMC 40048. PMID 8577732.
- ^ a b c d Urnov FD, Rebar EJ, Holmes MC, Zhang HS, Gregory PD (September 2010). "Genome editing with engineered zinc finger nucleases". Nature Reviews. Genetics. 11 (9): 636–46. doi:10.1038/nrg2842. PMID 20717154. S2CID 205484701.
- ^ Reik A, et al. (2008). "Zinc finger nucleases targeting the glucocorticoid receptor allow IL-13 zetakine transgenic CTLs to kill glioblastoma cells in vivo in the presence of immunosuppressing glucocorticoids". Mol. Ther. 16 (Supplement 1): S13–S14. doi:10.1016/S1525-0016(16)39437-0.
- ^ Holt N, Wang J, Kim K, Friedman G, Wang X, Taupin V, et al. (August 2010). "Human hematopoietic stem/progenitor cells modified by zinc-finger nucleases targeted to CCR5 control HIV-1 in vivo". Nature Biotechnology. 28 (8): 839–47. doi:10.1038/nbt.1663. PMC 3080757. PMID 20601939.
- ^ Gaj T, Gersbach CA, Barbas CF (July 2013). "ZFN, TALEN, and CRISPR/Cas-based methods for genome engineering". Trends in Biotechnology. 31 (7): 397–405. doi:10.1016/j.tibtech.2013.04.004. PMC 3694601. PMID 23664777.
- ^ Pérez-Quintero AL, Rodriguez-R LM, Dereeper A, López C, Koebnik R, Szurek B, Cunnac S (2013-07-15). "An improved method for TAL effectors DNA-binding sites prediction reveals functional convergence in TAL repertoires of Xanthomonas oryzae strains". PLOS ONE. 8 (7): e68464. Bibcode:2013PLoSO...868464P. doi:10.1371/journal.pone.0068464. PMC 3711819. PMID 23869221.
- ^ Young S (11 February 2014). "Genome Surgery". MIT Technology Review.
- ^ Woolf TM, Chase JM, Stinchcomb DT (August 1995). "Toward the therapeutic editing of mutated RNA sequences". Proceedings of the National Academy of Sciences of the United States of America. 92 (18): 8298–302. Bibcode:1995PNAS...92.8298W. doi:10.1073/pnas.92.18.8298. PMC 41144. PMID 7545300.
- ^ Woolf TM, Gurumurthy CB, Boyce F, Kmiec EB (April 2017). "To cleave or not to cleave: therapeutic gene editing with and without programmable nucleases". Nature Reviews. Drug Discovery. 16 (4): 296. doi:10.1038/nrd.2017.42. PMID 28303022.
- ^ a b Komor AC, Kim YB, Packer MS, Zuris JA, Liu DR (May 2016). "Programmable editing of a target base in genomic DNA without double-stranded DNA cleavage". Nature. 533 (7603): 420–4. Bibcode:2016Natur.533..420K. doi:10.1038/nature17946. PMC 4873371. PMID 27096365.
- ^ Cervantes-Gracia K, Gramalla-Schmitz A, Weischedel J, Chahwan R (2021). "APOBECs orchestrate genomic and epigenomic editing across health and disease". Trends Genet. 37 (11): 1028–1043. doi:10.1016/j.tig.2021.07.003. ISSN 0168-9525. PMID 34353635. S2CID 236934922.
- ^ Khan, Sikandar Hayat (2019-06-07). "Genome-Editing Technologies: Concept, Pros, and Cons of Various Genome-Editing Techniques and Bioethical Concerns for Clinical Application". Molecular Therapy. Nucleic Acids. Elsevier BV. 16: 326–334. doi:10.1016/j.omtn.2019.02.027. ISSN 2162-2531. PMC 6454098. PMID 30965277.
- ^ a b c d Barrangou R, Doudna JA (September 2016). "Applications of CRISPR technologies in research and beyond". Nature Biotechnology. 34 (9): 933–941. doi:10.1038/nbt.3659. PMID 27606440. S2CID 21543486.
- ^ Kim H, Kim JS (May 2014). "A guide to genome engineering with programmable nucleases". Nature Reviews. Genetics. 15 (5): 321–34. doi:10.1038/nrg3686. PMID 24690881. S2CID 9373606.
- ^ a b c d Gallagher RR, Li Z, Lewis AO, Isaacs FJ (October 2014). "Rapid editing and evolution of bacterial genomes using libraries of synthetic DNA". Nature Protocols. 9 (10): 2301–16. doi:10.1038/nprot.2014.082. PMID 25188632. S2CID 16447825.
- ^ a b McMahon MA, Rahdar M, Porteus M (December 2011). "Gene editing: not just for translation anymore". Nature Methods. 9 (1): 28–31. doi:10.1038/nmeth.1811. PMID 22205513. S2CID 2144013.
- ^ Daigneault BW, Rajput S, Smith GW, Ross PJ (May 2018). "Embryonic POU5F1 is Required for Expanded Bovine Blastocyst Formation". Scientific Reports. 8 (1): 7753. Bibcode:2018NatSR...8.7753D. doi:10.1038/s41598-018-25964-x. PMC 5958112. PMID 29773834.
- ^ Wargelius, Anna (2019). "Application of genome editing in aquatic farm animals". Transgenic Research. 28 (Suppl 2): 101–105. doi:10.1007/s11248-019-00163-0. PMID 31321691. S2CID 197665984. Retrieved 29 April 2021.
- ^ Yaskowiak, Edward S.; Shears, Margaret A.; Agarwal-Mawal, Alka; Fletcher, Garth L. (2006). "Characterization and multi-generational stability of the growth hormone transgene (EO-1α) responsible for enhanced growth rates in Atlantic Salmon". Transgenic Research. 15 (4): 465–480. doi:10.1007/s11248-006-0020-5. PMID 16906447. S2CID 22247493.
- ^ Ortega NM, Winblad N, Plaza Reyes A, Lanner F (October 2018). "Functional genetics of early human development". Current Opinion in Genetics & Development. 52: 1–6. doi:10.1016/j.gde.2018.04.005. PMID 29729430.
- ^ Arnould S, Delenda C, Grizot S, Desseaux C, Pâques F, Silva GH, Smith J (January 2011). "The I-CreI meganuclease and its engineered derivatives: applications from cell modification to gene therapy". Protein Engineering, Design & Selection. 24 (1–2): 27–31. doi:10.1093/protein/gzq083. PMID 21047873.
- ^ a b c Townsend JA, Wright DA, Winfrey RJ, Fu F, Maeder ML, Joung JK, Voytas DF (May 2009). "High-frequency modification of plant genes using engineered zinc-finger nucleases". Nature. 459 (7245): 442–5. Bibcode:2009Natur.459..442T. doi:10.1038/nature07845. PMC 2743854. PMID 19404258.
- ^ Zhang F, Maeder ML, Unger-Wallace E, Hoshaw JP, Reyon D, Christian M, et al. (June 2010). "High frequency targeted mutagenesis in Arabidopsis thaliana using zinc finger nucleases". Proceedings of the National Academy of Sciences of the United States of America. 107 (26): 12028–33. Bibcode:2010PNAS..10712028Z. doi:10.1073/pnas.0914991107. PMC 2900673. PMID 20508152.
- ^ Osakabe K, Osakabe Y, Toki S (June 2010). "Site-directed mutagenesis in Arabidopsis using custom-designed zinc finger nucleases". Proceedings of the National Academy of Sciences of the United States of America. 107 (26): 12034–9. Bibcode:2010PNAS..10712034O. doi:10.1073/pnas.1000234107. PMC 2900650. PMID 20508151.
- ^ a b Shukla VK, Doyon Y, Miller JC, DeKelver RC, Moehle EA, Worden SE, et al. (May 2009). "Precise genome modification in the crop species Zea mays using zinc-finger nucleases". Nature. 459 (7245): 437–41. Bibcode:2009Natur.459..437S. doi:10.1038/nature07992. PMID 19404259. S2CID 4323298.
- ^ Tripathi JN, Ntui VO, Ron M, Muiruri SK, Britt A, Tripathi L (2019-01-31). "CRISPR/Cas9 editing of endogenous banana streak virus in the B genome of Musa spp. overcomes a major challenge in banana breeding". Communications Biology. 2 (1): 46. doi:10.1038/s42003-019-0288-7. PMC 6355771. PMID 30729184.
- ^ Townson J (2017-01-01). "Recent developments in genome editing for potential use in plants". Bioscience Horizons. 10. doi:10.1093/biohorizons/hzx016.
- ^ Regalado A (19 December 2017). "These Are Not Your Father's GMOs". MIT Technology Review. Retrieved 16 April 2018.
- ^ Haun W, Coffman A, Clasen BM, Demorest ZL, Lowy A, Ray E, et al. (September 2014). "Improved soybean oil quality by targeted mutagenesis of the fatty acid desaturase 2 gene family". Plant Biotechnology Journal. 12 (7): 934–40. doi:10.1111/pbi.12201. PMID 24851712.
- ^ Clasen BM, Stoddard TJ, Luo S, Demorest ZL, Li J, Cedrone F, et al. (January 2016). "Improving cold storage and processing traits in potato through targeted gene knockout". Plant Biotechnology Journal. 14 (1): 169–76. doi:10.1111/pbi.12370. PMID 25846201.
- ^ a b Puchta H, Hohn B (June 2010). "Breaking news: plants mutate right on target". Proceedings of the National Academy of Sciences of the United States of America. 107 (26): 11657–8. Bibcode:2010PNAS..10711657P. doi:10.1073/pnas.1006364107. PMC 2900667. PMID 20554917.
- ^ a b c Paul JW, Qi Y (July 2016). "CRISPR/Cas9 for plant genome editing: accomplishments, problems and prospects". Plant Cell Reports. 35 (7): 1417–27. doi:10.1007/s00299-016-1985-z. PMID 27114166. S2CID 8035222.
- ^ Monga, Isha; Qureshi, Abid; Thakur, Nishant; Gupta, Amit Kumar; Kumar, Manoj (September 2017). "ASPsiRNA: A Resource of ASP-siRNAs Having Therapeutic Potential for Human Genetic Disorders and Algorithm for Prediction of Their Inhibitory Efficacy". G3. 7 (9): 2931–2943. doi:10.1534/g3.117.044024. PMC 5592921. PMID 28696921.
- ^ a b c Carroll D (November 2008). "Progress and prospects: zinc-finger nucleases as gene therapy agents". Gene Therapy. 15 (22): 1463–8. doi:10.1038/gt.2008.145. PMC 2747807. PMID 18784746.
- ^ Pollack A (2015-11-05). "A Cell Therapy Untested in Humans Saves a Baby With Cancer". The New York Times. ISSN 0362-4331. Retrieved 2015-11-30.
- ^ Couzin-Frankel J (November 2015). "CANCER IMMUNOTHERAPY. Baby's leukemia recedes after novel cell therapy". Science. 350 (6262): 731. doi:10.1126/science.350.6262.731. PMID 26564829.
- ^ Mentis AF (December 2016). "Epigenomic engineering for Down syndrome". Neuroscience and Biobehavioral Reviews. 71: 323–327. doi:10.1016/j.neubiorev.2016.09.012. PMID 27646312. S2CID 24192441.
- ^ Marchione M (7 February 2019). "Tests suggest scientists achieved 1st 'in body' gene editing". AP News. Retrieved 7 February 2019.
- ^ Staff (2 February 2019). "Ascending Dose Study of Genome Editing by the Zinc Finger Nuclease (ZFN) Therapeutic SB-913 in Subjects With MPS II". ClinicalTrials.gov. U.S. National Library of Medicine. Retrieved 7 February 2019.
- ^ Hammond A, Galizi R, Kyrou K, Simoni A, Siniscalchi C, Katsanos D, et al. (January 2016). "A CRISPR-Cas9 gene drive system targeting female reproduction in the malaria mosquito vector Anopheles gambiae". Nature Biotechnology. 34 (1): 78–83. doi:10.1038/nbt.3439. PMC 4913862. PMID 26641531.
- ^ Fletcher M (2018-08-11). "Mutant mosquitoes: Can gene editing kill off malaria?". The Telegraph. ISSN 0307-1235. Retrieved 2018-08-12.
- ^ Begley S (28 November 2018). "Amid uproar, Chinese scientist defends creating gene-edited babies - STAT". STAT.
- ^ Science China Press (23 January 2019). "Gene-edited disease monkeys cloned in China". EurekAlert!. Retrieved 24 January 2019.
- ^ Mandelbaum RF (23 January 2019). "China's Latest Cloned-Monkey Experiment Is an Ethical Mess". Gizmodo. Retrieved 24 January 2019.
- ^ "WHO, 인간 게놈 편집 글로벌 레지스트리 시작"PharmaBiz, 2019년 8월 31일게일 제너럴 원 파일, 2020년 4월 27일 접속
- ^ Teboul L, Herault Y, Wells S, Qasim W, Pavlovic G (June 2020). "Variability in Genome Editing Outcomes: Challenges for Research Reproducibility and Clinical Safety". Molecular Therapy. 28 (6): 1422–1431. doi:10.1016/j.ymthe.2020.03.015. PMC 7264426. PMID 32243835.
- ^ Im W, Moon J, Kim M (September 2016). "Applications of CRISPR/Cas9 for Gene Editing in Hereditary Movement Disorders". Journal of Movement Disorders. 9 (3): 136–43. doi:10.14802/jmd.16029. PMC 5035944. PMID 27667185.
- ^ a b Hsu PD, Lander ES, Zhang F (June 2014). "Development and applications of CRISPR-Cas9 for genome engineering". Cell. 157 (6): 1262–1278. doi:10.1016/j.cell.2014.05.010. PMC 4343198. PMID 24906146.
- ^ Johnson JA, Altwegg R, Evans DM, Ewen JG, Gordon IJ, Pettorelli N, Young JK (2016-04-01). "Is there a future for genome-editing technologies in conservation?". Animal Conservation. 19 (2): 97–101. doi:10.1111/acv.12273. ISSN 1469-1795. S2CID 27633404.
- ^ Pearlman A (2015-12-03). "Geneticists Are Concerned Transhumanists Will Use CRISPR on Themselves". Vice Motherboard. Retrieved 26 December 2016.
- ^ Jorgensen E. "How DIY bio-hackers are changing the conversation around genetic engineering". The Washington Post. Retrieved 26 December 2016.
- ^ "Human Enhancement". Pew Research Center. 2016-07-26. Retrieved 26 December 2016.
- ^ Regalado A. "Engineering the Perfect Baby". MIT Technology Review. Retrieved 26 December 2016.
- ^ a b Sample I (30 September 2016). "Experts warn home 'gene editing' kits pose risk to society". The Guardian. Retrieved 26 December 2016.
- ^ a b c d "Genome editing: an ethical review" (PDF). Nuffield Council on Bioethics. September 2016. Retrieved 27 December 2016.
- ^ "George Church told us why he's listing "superhuman" gene hacks". Futurism. Retrieved 25 July 2021.
- ^ "Protective alleles". arep.med.harvard.edu. Retrieved 25 July 2021.
- ^ Harmon A (2017-02-14). "Human Gene Editing Receives Science Panel's Support". The New York Times. ISSN 0362-4331. Retrieved 2017-02-17.
- ^ "Scientists OK genetically engineering babies". New York Post. Reuters. 2017-02-14. Retrieved 2017-02-17.
- ^ Clapper JR (9 February 2016). "Worldwide Threat Assessment of the US Intelligence Community" (PDF). Retrieved 26 December 2016.
- ^ Warmflash D (2016-09-06). "Genome editing: Is it a national security threat?". Retrieved 26 December 2016.
- ^ a b Regalado A. "Top U.S. Intelligence Official Calls Gene Editing a WMD Threat". MIT Technology Review. Retrieved 26 December 2016.
- ^ Jackson R, Ramshaw I (January 2010). "The mousepox experience. An interview with Ronald Jackson and Ian Ramshaw on dual-use research. Interview by Michael J. Selgelid and Lorna Weir". EMBO Reports. 11 (1): 18–24. doi:10.1038/embor.2009.270. PMC 2816623. PMID 20010799.
- ^ Broad WJ (23 January 2001). "Australians Create a Deadly Mouse Virus". The New York Times. Retrieved 27 December 2016.
- ^ Radford T (10 January 2001). "Lab creates killer virus by accident". The Guardian. Retrieved 27 December 2016.
"WHO, 인간 게놈 편집 글로벌 레지스트리 시작"PharmaBiz, 2019년 8월 31일게일 제너럴 원 파일, 2020년 4월 27일 접속
추가 정보
- "Special Issue on Human Germline Editing". Bioethics. 34. 2020.
- "Customized Human Genes: New Promises and Perils". Scientific American. Retrieved 2019-02-21.
- Connor S (25 April 2014). "Scientific split - the human genome breakthrough dividing former colleagues". The Independent. Retrieved 2016-02-11.
- "What is genome editing?". yourgenome.org. Retrieved 2018-04-13.