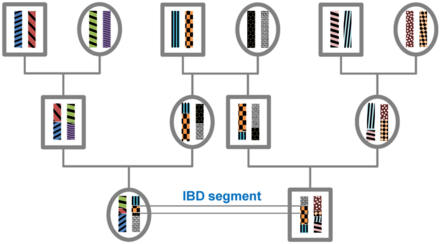
강하별 정체성
Identity by descentDNA 세그먼트는 둘 이상의 개인에서 상태(IBS)별로 동일하며, 이 세그먼트에서 뉴클레오티드 시퀀스가 동일할 경우 동일하다. IBS 세그먼트는 재조합 없이 공통의 조상으로부터 물려받은 경우, 즉 그 세그먼트가 이들 개인에서 동일한 조상 기원을 갖는 경우 둘 이상의 개인에서 강하(IBD)에 의해 동일하다. IBD인 DNA 세그먼트는 정의에 따라 IBS이지만 IBD가 아닌 세그먼트는 서로 다른 개인에서의 동일한 돌연변이나 세그먼트를 변경하지 않는 재조합으로 인해 여전히 IBS일 수 있다.
이론
유한한 모집단의 모든 개인은 충분히 오랫동안 추적되고 따라서 그들의 게놈 IBD의 세그먼트를 공유한다면 연관된다. IBD의 감수분열 부분은 재조합에 의해 분해된다. 따라서 IBD 세그먼트의 예상 길이는 세그먼트 위치의 가장 최근 공통 조상 이후 세대 수에 따라 달라진다. 과거의 공통 조상 n 세대(따라서 2n 감수분열 포함)에서 비롯되는 IBD 세그먼트의 길이는 평균 1/(2n) 모르간(M)으로 기하급수적으로 분포한다.[1] 예상 IBD 세그먼트 수는 이 위치의 공통 조상 이후 세대 수에 따라 감소한다. 특정 DNA 세그먼트의 경우, 각 감수분열에서 이 세그먼트를 전송할 확률은 1/2이기 때문에 IBD일 확률은−2n 2로 감소한다.[2]
적용들
식별된 IBD 세그먼트는 광범위한 목적을 위해 사용될 수 있다. 위에서 언급한 바와 같이 IBD 공유의 양(길이 및 수)은 시험 대상 개인 간의 가족 관계에 따라 달라진다. 따라서 IBD 세그먼트 검출의 한 가지 적용은 관련성을 정량화하는 것이다.[3][4][5][6] 관련성의 측정은 법의학 유전학에서는 사용될 수 있지만,[7] 유전적 연계 지도에[3][8] 관한 정보를 증가시키고 표준 연관 연구에서의 미등록 관계에 의한 편견을 감소시키는데 도움을 줄 수 있다.[6][9] IBD의 또 다른 적용은 유전자형 귀납과 하플라형 위상 추론이다.[10][11][12] 짧은 지역에 의해 분할된 IBD의 긴 공유 세그먼트는 단계적 오류를 나타낼 수 있다.[5][13]: SI
IBD 매핑
IBD 매핑은[3] 연결 분석과 유사하지만 관련 없는 개인들의 코호트에 대해 알려진 혈통 없이 수행될 수 있다. IBD 매핑은 복수의 희귀질환 감수성 변형이 포함된 유전자 또는 유전체 영역을 매핑하는 힘을 증가시키는 새로운 형태의 연관 분석으로 볼 수 있다.[6][14]
Browning과 Thompson은 시뮬레이션 데이터를 사용하여 유전자 내의 여러 희귀 변종이 질병 감수성에 기여할 때 IBD 매핑이 연관성 시험보다 더 높은 힘을 가지고 있다는 것을 보여주었다.[14] IBD 매핑을 통해, 표준 연관성 테스트가 실패하는 동안 고립된 모집단뿐만 아니라 다른 모집단에서도 게놈 범위의 유의한 영역이 발견되었다.[11][15] 후웬 외 IBD 공유를 사용하여 고립된 어군에서 양성 재발성 자궁내 담석증을 담당하는 유전자의 염색체 위치를 식별했다.[16] 케니 외 연구진은 또한 장에서 콜레스테롤 흡수의 대용치인 혈장 식물 스테롤(PPS) 수준의 게놈 전체 연관 연구(GWAS)에 의해 발견된 신호를 정밀 지도화하기 위해 고립된 모집단을 이용했다.[17] Francks 외 연구진은 환자-대조군 검체의 유전자형 데이터로 조현병 및 조울증 장애에 대한 잠재적 민감도 위치를 식별할 수 있었다.[18] 린 외 연구진은 다수의 경화증 환자 데이터 집합에서 게놈 범위 유의한 연계 신호를 발견했다.[19] 레투제 외 IBD 지도를 사용하여 암 검체에서 창시자의 돌연변이를 찾아냈다.[20]
인구유전학의 IBD
인간의 게놈에서 자연선택의 검출은 또한 검출된 IBD 세그먼트를 통해서도 가능하다. 선발은 보통 모집단의 개인들 사이에서 IBD 세그먼트의 수를 증가시키는 경향이 있다. IBD 공유가 초과된 지역을 스캔함으로써, 인간 게놈의 강한, 매우 최근의 선택 영역을 확인할 수 있다.[21][22]
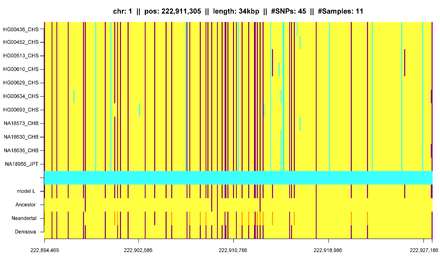
그 외에도 IBD 세그먼트는 인구구조에 대한 다른 영향을 측정하고 식별하는 데 유용할 수 있다.[6][23][24][25][26] Gusev 등은 IBD 세그먼트를 병목 현상과 혼합물을 포함한 인구통계학적 이력을 추정하기 위해 추가 모델링과 함께 사용할 수 있다는 것을 보여주었다.[24] 유사한 모델 Palamara 외 연구진 및 Carmi 외 연구진을 사용하여 Ashkenazi 유태인과 케냐 마사이 개인의 인구통계학적 역사를 재구성했다.[25][26][27] 보티구에 외 연구진은 유럽 인구들 사이의 아프리카 조상의 차이를 조사했다.[28] Ralph와 Coop은 IBD 탐지를 사용하여 서로 다른 유럽 인구의[29] 공통 조상을 정량화했으며 Gravel 등에서도 마찬가지로 아메리카 대륙 인구의 유전적 이력의 결론을 도출하려고 노력했다.[30] 링바우어 외 IBD 세그먼트의 지리적 구조를 활용하여 지난 세기 동안 동유럽 내 분산을 추정했다.[31] 1000개의 게놈 데이터를 사용하여 Hochreiter는 아프리카, 아시아, 유럽 인구와 네안데르탈인이나 데니소바와 같은 고대 게놈과 공유되는 IBD 세그먼트 사이의 IBD 공유에서 차이를 발견했다.[13]
방법 및 소프트웨어
관련 없는 개인에서 IBD 세그먼트를 탐지하는 프로그램:
- RAPID: 포지션 버로우스-휠러 변환을 사용한 바이오뱅크-스케일 코호트 내 강하 감지에 의한 초고속 ID
- Parente: 인쇄되지 않은 유전자형 데이터에서[33] 개인 쌍 사이의 IBD 세그먼트를 식별
- 비글/빠른 속도IBD: 게놈 범위 SNP 데이터에서[34] 개인 쌍 간의 IBD 세그먼트 찾기
- 비글/정품IBD: 해싱 방법을 사용하여 개인 쌍으로 IBD 세그먼트를 찾고 우도비를[35] 통해 그 중요성을 평가한다.
- IBDseq: 시퀀싱 데이터에서[36] 쌍방향 IBD 세그먼트 탐지
- 세균라인: 개인[5] 쌍으로 구성된 선형 시간 IBD 세그먼트에서 발견
- DASH: 한 쌍의 IBD 세그먼트를 기반으로 구축되어 단일 하플로타입이[15] 공유될 가능성이 있는 개인 클러스터를 유추함
- PLINK: 쌍방향 IBD 세그먼트 검출[6] 방법을 포함하여 전체 게놈 연관성 및 모집단 기반 연결 분석을 위한 도구 세트
- 관련: SNP를[3] 사용하여 특정 위치에 있는 개인 쌍 간의 IBD 확률을 추정함
- MCMC_IBDfinder: 여러 개인에서[37] IBD 세그먼트를 찾기 위한 MMC(Markov Chain Monte Carlo) 기반
- IBD-Groupon: 쌍별 IBD 관계를[38] 기반으로 그룹별 IBD 세그먼트 탐지
- HapFABIA: 여러 개인에서[13] 동시에 대규모 시퀀싱 데이터의 희귀한 변형이 특징인 매우 짧은 IBD 세그먼트를 식별
참고 항목
참조
- ^ Browning, S. R. (2008). "Estimation of Pairwise Identity by Descent from Dense Genetic Marker Data in a Population Sample of Haplotypes". Genetics. 178 (4): 2123–2132. doi:10.1534/genetics.107.084624. PMC 2323802. PMID 18430938.
- ^ Thompson, E. A. (2008). "The IBD process along four chromosomes". Theoretical Population Biology. 73 (3): 369–373. doi:10.1016/j.tpb.2007.11.011. PMC 2518088. PMID 18282591.
- ^ a b c d Albrechtsen, A.; Sand Korneliussen, T.; Moltke, I.; Van Overseem Hansen, T.; Nielsen, F. C.; Nielsen, R. (2009). "Relatedness mapping and tracts of relatedness for genome-wide data in the presence of linkage disequilibrium". Genetic Epidemiology. 33 (3): 266–274. doi:10.1002/gepi.20378. PMID 19025785. S2CID 12029712.
- ^ Browning, S. R.; Browning, B. L. (2010). "High-Resolution Detection of Identity by Descent in Unrelated Individuals". The American Journal of Human Genetics. 86 (4): 526–539. doi:10.1016/j.ajhg.2010.02.021. PMC 2850444. PMID 20303063.
- ^ a b c Gusev, A.; Lowe, J. K.; Stoffel, M.; Daly, M. J.; Altshuler, D.; Breslow, J. L.; Friedman, J. M.; Pe'Er, I. (2008). "Whole population, genome-wide mapping of hidden relatedness". Genome Research. 19 (2): 318–326. doi:10.1101/gr.081398.108. PMC 2652213. PMID 18971310.
- ^ a b c d e Purcell, S.; Neale, B.; Todd-Brown, K.; Thomas, L.; Ferreira, M. A. R.; Bender, D.; Maller, J.; Sklar, P.; De Bakker, P. I. W.; Daly, M. J.; Sham, P. C. (2007). "PLINK: A Tool Set for Whole-Genome Association and Population-Based Linkage Analyses". The American Journal of Human Genetics. 81 (3): 559–575. doi:10.1086/519795. PMC 1950838. PMID 17701901.
- ^ Ian W. Evett; Bruce S. Weir (January 1998). Interpreting DNA Evidence: Statistical Genetics for Forensic Scientists. Sinauer Associates, Incorporated. ISBN 978-0-87893-155-2.
- ^ Leutenegger, A.; Prum, B.; Genin, E.; Verny, C.; Lemainque, A.; Clergetdarpoux, F.; Thompson, E. (2003). "Estimation of the Inbreeding Coefficient through Use of Genomic Data". The American Journal of Human Genetics. 73 (3): 516–523. doi:10.1086/378207. PMC 1180677. PMID 12900793.
- ^ Voight, B. F.; Pritchard, J. K. (2005). "Confounding from Cryptic Relatedness in Case-Control Association Studies". PLOS Genetics. 1 (3): e32. doi:10.1371/journal.pgen.0010032. PMC 1200427. PMID 16151517.
- ^ Kong, A.; Masson, G.; Frigge, M. L.; Gylfason, A.; Zusmanovich, P.; Thorleifsson, G.; Olason, P. I.; Ingason, A.; Steinberg, S.; Rafnar, T.; Sulem, P.; Mouy, M.; Jonsson, F.; Thorsteinsdottir, U.; Gudbjartsson, D. F.; Stefansson, H.; Stefansson, K. (2008). "Detection of sharing by descent, long-range phasing and haplotype imputation". Nature Genetics. 40 (9): 1068–1075. doi:10.1038/ng.216. PMC 4540081. PMID 19165921.
- ^ a b Gusev, A.; Shah, M. J.; Kenny, E. E.; Ramachandran, A.; Lowe, J. K.; Salit, J.; Lee, C. C.; Levandowsky, E. C.; Weaver, T. N.; Doan, Q. C.; Peckham, H. E.; McLaughlin, S. F.; Lyons, M. R.; Sheth, V. N.; Stoffel, M.; De La Vega, F. M.; Friedman, J. M.; Breslow, J. L.; Pe'Er, I. (2011). "Low-Pass Genome-Wide Sequencing and Variant Inference Using Identity-by-Descent in an Isolated Human Population". Genetics. 190 (2): 679–689. doi:10.1534/genetics.111.134874. PMC 3276614. PMID 22135348.
- ^ Browning, B. L.; Browning, S. R. (2009). "A Unified Approach to Genotype Imputation and Haplotype-Phase Inference for Large Data Sets of Trios and Unrelated Individuals". The American Journal of Human Genetics. 84 (2): 210–223. doi:10.1016/j.ajhg.2009.01.005. PMC 2668004. PMID 19200528.
- ^ a b c Hochreiter, S. (2013). "HapFABIA: Identification of very short segments of identity by descent characterized by rare variants in large sequencing data". Nucleic Acids Research. 41 (22): e202. doi:10.1093/nar/gkt1013. PMC 3905877. PMID 24174545.
- ^ a b Browning, S. R.; Thompson, E. A. (2012). "Detecting Rare Variant Associations by Identity-by-Descent Mapping in Case-Control Studies". Genetics. 190 (4): 1521–1531. doi:10.1534/genetics.111.136937. PMC 3316661. PMID 22267498.
- ^ a b Gusev, A.; Kenny, E. E.; Lowe, J. K.; Salit, J.; Saxena, R.; Kathiresan, S.; Altshuler, D. M.; Friedman, J. M.; Breslow, J. L.; Pe'Er, I. (2011). "DASH: A Method for Identical-by-Descent Haplotype Mapping Uncovers Association with Recent Variation". The American Journal of Human Genetics. 88 (6): 706–717. doi:10.1016/j.ajhg.2011.04.023. PMC 3113343. PMID 21620352.
- ^ Houwen, R. H. J.; Baharloo, S.; Blankenship, K.; Raeymaekers, P.; Juyn, J.; Sandkuijl, L. A.; Freimer, N. B. (1994). "Genome screening by searching for shared segments: Mapping a gene for benign recurrent intrahepatic cholestasis". Nature Genetics. 8 (4): 380–386. doi:10.1038/ng1294-380. hdl:1765/55192. PMID 7894490. S2CID 8131209.
- ^ Kenny, E. E.; Gusev, A.; Riegel, K.; Lutjohann, D.; Lowe, J. K.; Salit, J.; Maller, J. B.; Stoffel, M.; Daly, M. J.; Altshuler, D. M.; Friedman, J. M.; Breslow, J. L.; Pe'Er, I.; Sehayek, E. (2009). "Systematic haplotype analysis resolves a complex plasma plant sterol locus on the Micronesian Island of Kosrae". Proceedings of the National Academy of Sciences. 106 (33): 13886–13891. Bibcode:2009PNAS..10613886K. doi:10.1073/pnas.0907336106. PMC 2728990. PMID 19667188.
- ^ Francks, C.; Tozzi, F.; Farmer, A.; Vincent, J. B.; Rujescu, D.; St Clair, D.; Muglia, P. (2008). "Population-based linkage analysis of schizophrenia and bipolar case–control cohorts identifies a potential susceptibility locus on 19q13". Molecular Psychiatry. 15 (3): 319–325. doi:10.1038/mp.2008.100. PMID 18794890.
- ^ Lin, R.; Charlesworth, J.; Stankovich, J.; Perreau, V. M.; Brown, M. A.; Anzgene, B. V.; Taylor, B. V. (2013). Toland, Amanda Ewart (ed.). "Identity-by-Descent Mapping to Detect Rare Variants Conferring Susceptibility to Multiple Sclerosis". PLOS ONE. 8 (3): e56379. Bibcode:2013PLoSO...856379L. doi:10.1371/journal.pone.0056379. PMC 3589405. PMID 23472070.
- ^ Letouzé, E.; Sow, A.; Petel, F.; Rosati, R.; Figueiredo, B. C.; Burnichon, N.; Gimenez-Roqueplo, A. P.; Lalli, E.; De Reyniès, A. L. (2012). Mailund, Thomas (ed.). "Identity by Descent Mapping of Founder Mutations in Cancer Using High-Resolution Tumor SNP Data". PLOS ONE. 7 (5): e35897. Bibcode:2012PLoSO...735897L. doi:10.1371/journal.pone.0035897. PMC 3342326. PMID 22567117.
- ^ Albrechtsen, A.; Moltke, I.; Nielsen, R. (2010). "Natural Selection and the Distribution of Identity-by-Descent in the Human Genome". Genetics. 186 (1): 295–308. doi:10.1534/genetics.110.113977. PMC 2940294. PMID 20592267.
- ^ Han, L.; Abney, M. (2011). "Identity by descent estimation with dense genome-wide genotype data". Genetic Epidemiology. 35 (6): 557–567. doi:10.1002/gepi.20606. PMC 3587128. PMID 21769932.
- ^ Cockerham, C. C.; Weir, B. S. (1983). "Variance of actual inbreeding". Theoretical Population Biology. 23 (1): 85–109. doi:10.1016/0040-5809(83)90006-0. PMID 6857551.
- ^ a b Gusev, A.; Palamara, P. F.; Aponte, G.; Zhuang, Z.; Darvasi, A.; Gregersen, P.; Pe'Er, I. (2011). "The Architecture of Long-Range Haplotypes Shared within and across Populations". Molecular Biology and Evolution. 29 (2): 473–486. doi:10.1093/molbev/msr133. PMC 3350316. PMID 21984068.
- ^ a b Palamara, P. F.; Lencz, T.; Darvasi, A.; Pe’Er, I. (2012). "Length Distributions of Identity by Descent Reveal Fine-Scale Demographic History". The American Journal of Human Genetics. 91 (5): 809–822. doi:10.1016/j.ajhg.2012.08.030. PMC 3487132. PMID 23103233.
- ^ a b Palamara, P. F.; Pe'Er, I. (2013). "Inference of historical migration rates via haplotype sharing". Bioinformatics. 29 (13): i180–i188. doi:10.1093/bioinformatics/btt239. PMC 3694674. PMID 23812983.
- ^ Carmi, S.; Palamara, P. F.; Vacic, V.; Lencz, T.; Darvasi, A.; Pe'Er, I. (2013). "The Variance of Identity-by-Descent Sharing in the Wright-Fisher Model". Genetics. 193 (3): 911–928. arXiv:1206.4745. doi:10.1534/genetics.112.147215. PMC 3584006. PMID 23267057.
- ^ Botigue, L. R.; Henn, B. M.; Gravel, S.; Maples, B. K.; Gignoux, C. R.; Corona, E.; Atzmon, G.; Burns, E.; Ostrer, H.; Flores, C.; Bertranpetit, J.; Comas, D.; Bustamante, C. D. (2013). "Gene flow from North Africa contributes to differential human genetic diversity in southern Europe". Proceedings of the National Academy of Sciences. 110 (29): 11791–11796. Bibcode:2013PNAS..11011791B. doi:10.1073/pnas.1306223110. PMC 3718088. PMID 23733930.
- ^ Ralph, P.; Coop, G. (2013). Tyler-Smith, Chris (ed.). "The Geography of Recent Genetic Ancestry across Europe". PLOS Biology. 11 (5): e1001555. doi:10.1371/journal.pbio.1001555. PMC 3646727. PMID 23667324.
- ^ Gravel, S.; Zakharia, F.; Moreno-Estrada, A.; Byrnes, J. K.; Muzzio, M.; Rodriguez-Flores, J. L.; Kenny, E. E.; Gignoux, C. R.; Maples, B. K.; Guiblet, W.; Dutil, J.; Via, M.; Sandoval, K.; Bedoya, G.; 1000 Genomes, T. K.; Oleksyk, A.; Ruiz-Linares, E. G.; Burchard, J. C.; Martinez-Cruzado, C. D.; Bustamante, C. D. (2013). Williams, Scott M (ed.). "Reconstructing Native American Migrations from Whole-Genome and Whole-Exome Data". PLOS Genetics. 9 (12): e1004023. arXiv:1306.4021. doi:10.1371/journal.pgen.1004023. PMC 3873240. PMID 24385924.
- ^ Ringbauer, Harald; Coop, Graham; Barton, Nicholas H. (2017-03-01). "Inferring Recent Demography from Isolation by Distance of Long Shared Sequence Blocks". Genetics. 205 (3): 1335–1351. doi:10.1534/genetics.116.196220. ISSN 0016-6731. PMC 5340342. PMID 28108588.
- ^ 나세리 A, 류 X, 장 S, 지 D. 포지셔날 버로우스-휠러 변환 BioRxiv 2017을 사용하는 바이오뱅크-스케일 코호트의 강하 감지별 초고속 ID
- ^ 로드리게스 JM, 바트조글루 S, 베르코비치 S. 내장된 우도 비율 검정을 통해 인쇄되지 않은 유전자 유형의 대규모 데이터 집합에서 관련성을 유추하는 정확한 방법. RECOMB 2013, LNBI 7821:212-229.
- ^ Browning, B. L.; Browning, S. R. (2011). "A Fast, Powerful Method for Detecting Identity by Descent". The American Journal of Human Genetics. 88 (2): 173–182. doi:10.1016/j.ajhg.2011.01.010. PMC 3035716. PMID 21310274.
- ^ Browning, B. L.; Browning, S. R. (2013). "Improving the Accuracy and Efficiency of Identity-by-Descent Detection in Population Data". Genetics. 194 (2): 459–471. doi:10.1534/genetics.113.150029. PMC 3664855. PMID 23535385.
- ^ Browning, B. L.; Browning, S. R. (2013). "Detecting Identity by Descent and Estimating Genotype Error Rates in Sequence Data". The American Journal of Human Genetics. 93 (5): 840–851. doi:10.1016/j.ajhg.2013.09.014. PMC 3824133. PMID 24207118.
- ^ Moltke, I.; Albrechtsen, A.; Hansen, T. V. O.; Nielsen, F. C.; Nielsen, R. (2011). "A method for detecting IBD regions simultaneously in multiple individuals--with applications to disease genetics". Genome Research. 21 (7): 1168–1180. doi:10.1101/gr.115360.110. PMC 3129259. PMID 21493780.
- ^ He, D. (2013). "IBD-Groupon: An efficient method for detecting group-wise identity-by-descent regions simultaneously in multiple individuals based on pairwise IBD relationships". Bioinformatics. 29 (13): i162–i170. doi:10.1093/bioinformatics/btt237. PMC 3694672. PMID 23812980.