이탈리아의 유전사
Genetic history of Italy| 이탈리아의 역사 |
|---|
 |
| |
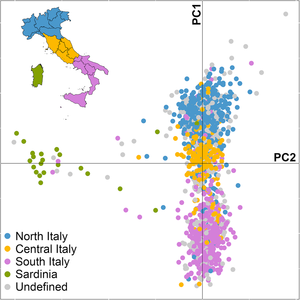
이탈리아의 유전사는 지리와 역사의 영향을 많이 받습니다. 이탈리아인들의 조상은 주로 인도유럽어를 사용하는 민족들(라틴어, 팔리스치어, 피켄테스어, 움브리아어, 삼니테스어, 오스칸어, 시켈어, 아드리아해 베네치아어, 켈트어, 이아피아어, 그리스어)과 인도유럽어 이전의 민족들(이탈리아 본토의 에트루리아어, 리구레어, 라에티아어, 카뮌니어, 시칠리아어, 사르데냐어의 누라어족)이었습니다. 로마 제국 시대에 이탈리아 반도는 남유럽, 북아프리카, 중동 등 지중해 유역의 다양한 지역에서 사람들을 끌어 모았습니다.[2] DNA 분석을 바탕으로 고대 지역 유전적 하부구조와 근대 이탈리아 내의 연속성에 대한 증거가 로마 이전과 로마 시대로 거슬러 올라갑니다.[3][4][5][6]
이탈리아인들은 혼혈 비율에서 다른 남유럽인들과 비슷하며, 이는 주로 신석기 시대 초기 유럽인 농부의 조상이며, 작지만 여전히 중요한 양의 중석기 서양 사냥꾼-채터러입니다. 청동기 시대 스텝 목회자(인도유럽어 사용자)와 찰콜리스틱 또는 청동기 시대 이란/카우카스 관련 조상.[4][7][8][9] 남부 이탈리아인들은 현대 그리스인들과 가장 가깝고,[10] 북부 이탈리아인들은 스페인인들과 남부 프랑스인들과 가장 가깝습니다.[11][12][13][14] 또한 이탈리아에는 청동기/철기 시대의 서아시아 및 중동 혼합물이 있으며, 중부 이탈리아 및 남부 이탈리아에 비해 북부 이탈리아의 발병률이 훨씬 낮습니다.[15][8] 북아프리카 혼합물은 이탈리아 남부와 주요 섬에서도 발견됩니다.[15][8][4]
개요
철기 시대와 초기 로마 공화정 시대의 라티움의 라틴 샘플은 일반적으로 현대 북부 이탈리아인과 중부 이탈리아인과 유전적으로 가장 가까운 것으로 밝혀졌습니다(6명 중 4명은 북부 이탈리아인과 중부 이탈리아인과 가장 가까운 반면, 나머지 2명은 남부 이탈리아인과 가장 가까운 것).[16] DNA 분석은 고대 그리스 식민지가 남부 이탈리아와 시칠리아(마그나 그라이키아)의 지역 유전 지형에 상당한 지속적인 영향을 미쳤다는 것을 보여주며, 그 지역의 현대인들은 상당한 그리스 혼혈을 가지고 있습니다.[17][18] 전반적으로 라틴계, 에트루리아인과 이탈리아의 이전 빌라노반 인구 사이의 유전적 분화는 미미한 것으로 밝혀졌습니다.[19] 2019년, 로마 화석에 대한 DNA 분석은 고대 말과 중세 시대에 로마 도시의 주민들에게서 중유럽과 북유럽 조상으로 상당한 유전적 조상 이동을 발견했습니다. 저자들은 이 조상의 기원을 서고트족과 롬바르드족과 잠정적으로 연관 짓습니다.[2][20] 2020년 고대 및 현대 표본의 모계 하플로그룹을 분석한 결과 이탈리아 중부의 움브리아 현대 주민과 이탈리아어를 사용하는 움브리아 문화에 속하는 지역의 고대 주민 사이에 상당한 유전적 유사성과 연속성이 있음을 알 수 있습니다.[6]
여러 DNA 연구를 통해 이탈리아의 유전자 변이는 동부에서 서부 지중해로 이어지는 임상적인 것으로 확인되었습니다. 사르데냐인은 이탈리아와 실제로 유럽에서 유전적 이상치로 예외이며, 이는 주로 신석기 시대, 인도유럽 이전 및 이탈리아가 아닌 누라기 조상에서 비롯됩니다.[21][7][8][9] 유럽과 더 넓은 지중해 유역의 역사를 반영하여 이탈리아 인구는 유럽의 중석기, 신석기 및 청동기 정착지와 비율은 다르지만 대부분 동일한 조상 구성 요소로 구성되어 있는 것으로 밝혀졌습니다.[15][12][22]
북부 이탈리아인과 남부 이탈리아인 사이의 유전적 격차는 중간 중부 이탈리아 군집에 의해 채워져 지리를 반영하는 연속적인 변이 계통을 만듭니다.[23] 유일한 예외는 슬로베니아에서 슬라브어를 사용하는 중앙유럽인들과 군집을 이루는 일부 소수 민족([24]대부분 프리울리-베네치아 줄리아 지역의 슬로베니아 소수 민족)과 이탈리아 본토와 시칠리아 본토의 인구와 분명히 구별되는 사르데냐인들입니다.[12][4] 이탈리아에 거주하는 일부 언어 및 고립된 커뮤니티에 대한 연구에 따르면 짧은 거리(0-200km)와 중간 거리(700-800km)에서 유전적 다양성이 유럽 대륙 전체에서 관찰된 것보다 더 큰 것으로 나타났습니다.[5]
북부 이탈리아인과 남부 이탈리아인 사이의 유전적 거리는 단일 유럽 국적으로는 크지만 북부 독일인과 남부 독일인 사이의 유전적 거리와 비슷합니다.[25] 북부 이탈리아인과 남부 이탈리아인은 후기 빙하기부터 갈라지기 시작했으며, 유럽 전역에서 관찰 가능한 유전적 다양성의 계통을 더 작은 규모로 요약하는 것으로 보입니다.[26]
이탈리아의 역사적 인구
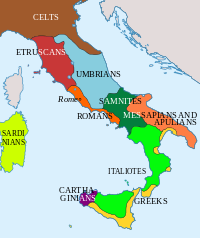
현생 인류는 상부 구석기 시대에 나타났습니다. 오리가니아 시대의 표본은 후마네 동굴에서 발견되었으며, 그 연대는 약 34,000년 전으로 거슬러 올라갑니다. 마그달레니아 시대에 피레네 산맥에서 온 최초의 인류가 사르데냐에 거주했습니다.[27]
신석기 시대에는 동쪽에서 온 사람들에 의해 농사가 소개되어 최초의 마을이 지어졌습니다. 무기는 더욱 정교해졌고 점토에 있는 최초의 물건들이 생산되었습니다. 신석기 시대 후기에 구리의 사용이 확산되어 호수 근처의 더미 위에 마을이 지어졌습니다. 사르데냐, 시칠리아 그리고 이탈리아 본토의 일부에서 비커 문화가 서유럽과 중앙 유럽으로부터 퍼져나갔습니다. 시칠리아는 미케네 시대에도 에게 해의 영향을 받았습니다.
후기 청동기 시대에 어른필드 원형 빌라노반 문화가 이탈리아 중부와 북부에서 나타났습니다. 중앙유럽에서 유래한 시체 화장 의식이 특징입니다. 철의 사용이 퍼지기 시작했습니다.[28] 사르데냐에서는 누라식 문명이 번성했습니다.
철기 시대의 여명기에 이탈리아의 많은 지역에는 라틴족, 사비니족, 삼니트족, 움브리아족과 같은 이탈리아 부족들이 살고 있었습니다. 북서쪽과 알프스 지역은 주로 에트루리아인, 리구리아인, 카뮌니인, 레이티인과 같은 인도 이전의 유럽어 사용자들에 의해 거주되었습니다. 반면, 일리리아 출신일 가능성이 있는 이아피아 부족들은 아풀리아에 거주했습니다.
기원전 8세기부터, 그리스 식민지 주민들은 남부 이탈리아 해안에 정착했고 나중에 마그나 그라이키아라고 불릴 도시들을 만들었습니다. 비슷한 시기에 페니키아 식민지 주민들은 시칠리아의 서쪽에 정착했습니다. 같은 기간 동안 에트루리아 문명은 남부 토스카나와 북부 라티움 해안에서 발전했습니다. 기원전 4세기에 갈리아는 북부 이탈리아와 중부 이탈리아의 일부에 정착했습니다.[29] 서로마 제국의 멸망과 함께 게르만계의 다양한 인구가 이탈리아를 침공했는데, 가장 중요한 것은 롬바르드족이었고,[30] 5세기 후에는 시칠리아의 노르만족이 그 뒤를 이었습니다.
Y-DNA 유전자 다양성
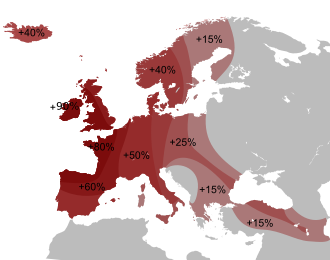
특히 북이탈리아와 중앙이탈리아의 많은 이탈리아인들은 서유럽과 중앙유럽에서 흔히 볼 수 있는 하플로그룹 R1b에 속합니다. R1b의 가장 높은 빈도는 토스카나의 가르파냐나(76.2%)와 롬바르디아의 베르가모 계곡(80.8%)에서 발견됩니다.[31][32] 이 비율은 이탈리아 남부 칼라브리아(33.2%)[31]에서 낮아집니다. 반면 사르데냐인의 39%는 중석기 유럽 하플로그룹 I2a1a에 속합니다.[33][34]
Università Cattolica del Sacro Cuore의 연구에 따르면 그리스 식민지화는 중요한 유전적 기여를 거의 남기지 않았지만 이탈리아 반도의 12개 지역을 샘플링한 데이터 분석에서는 남성 개체군 확산 모델과 중석기 거주자와의 신석기 혼합을 지원했습니다.[35] 이 결과는 남북 축을 따라 유전적 변이가 분포하는 것을 지지하고 인구 확산을 지지했습니다. 남이탈리아 표본은 남동 및 중남부 유럽 표본과 군집을 이루고 북부 그룹은 서유럽과 군집을 이루었습니다.[35][36]
Semino 등의 2004년 연구에 따르면 중북부 지역의 이탈리아인은 약 26.9%의 J2를 가지고 있으며, 아풀리아인, 칼라브리아인 및 시칠리아인은 각각 29.1%, 21.5% 및 16.7%의 J2를 가지고 있으며, 사르데냐인은 9.7%의 J2를 가지고 있습니다.[37]
Y-염색체 및 하플로그룹 계통, 그 다양성 및 817개의 대표적인 피험자를 대상으로 한 2018년 유전자 연구는 전통적인 북부-남부 인구 구분의 공로를 인정합니다. 신석기 시대의 이동으로 인해 남부 이탈리아인들은 "북쪽보다 중동 및 남부 발칸 지역의 개체군과 더 높은 유사성을 보인다. 반대로 북부 샘플은 유전적으로 북서 유럽 및 북부 발칸 지역 그룹에 더 가깝다."고 결론을 내렸습니다. 토스카나 중부에 있는 볼테라의 위치는 에트루리아인의 기원에 대한 논쟁을 열어두고 있지만, 숫자는 자율적인 주장을 강력하게 지지합니다. J2a-M67*(2.7%)의 낮은 존재는 아나톨리아인과의 해상 접촉을 시사합니다. 중앙 유럽 혈통 G2a-L497(7).1%) 상당한 빈도에서 오히려 에트루리아인의 중앙 유럽 기원을 지지합니다. 그리고 마지막으로 유럽 R1b 계통의 높은 발병률(R1b 50% 약, R1b-U15224).5%) - 특히 하플로그룹 R1b-U152 - 고고학, 인류학 및 언어학에서 이미 뒷받침된 [31]할리카르나소스의 디오니시우스 이론에 따라 이전 빌라노반 문화에서 에트루리아 문명이 형성된 과정으로 인해 자율적인 기원을 시사할 수 있습니다.[38][39][40][41] 2019년 사이언스에 발표된 스탠포드 연구에서 이탈리아 마르케 지역의 안코나 지방의 리파비안카 디 몬테라도 신석기 시대 정착지에서 채취한 두 개의 고대 샘플이 Y-DNA J-L26과 J-M304인 것으로 밝혀졌습니다.[2] 따라서 Y-DNA J-L26 및 J-M304의 하류인 Y-DNA J2a-M67은 신석기 시대 이후 이탈리아에 있을 가능성이 가장 높으며 최근 아나톨리아와의 접촉의 증거가 될 수 없습니다.
역사적 이민이 소개한 Y-DNA
라치오와 아브루초의 두 마을(카파도키아와 발레피에트라)에서 I1은 35%와 28%[42] 수준으로 기록된 가장 흔한 Y-DNA입니다. 시칠리아에서 반달족과 사라센족의 추가 이주는 시칠리아 사람들의 민족 구성에 약간의 영향을 미쳤을 뿐입니다. 그러나 구체적으로 그리스의 유전적 유산은 시칠리아에서 37%로 추정됩니다.[17]
노르만족의 이탈리아 남부 정복으로 인해 1130년에 시칠리아의 노르만 왕국이 세워지게 되었고, 최초의 노르만족 침공 이후 70년, 1091년 마지막 마을인 노토를 정복한 후 40년이 지난 후 1198년까지 지속되었습니다. 오늘날 시칠리아 중부와 서부에서 노르만 Y-DNA가 흔하며, 계통의 15%에서 20%가 하플로그룹 I에 속하며, 이 비율은 섬의 동부에서 8%로 떨어집니다. 시칠리아에 대한 북아프리카 남성의 기여는 0%에서 7.5%[43][18][44] 사이로 추정되었습니다. 전체적으로 볼 때, 시칠리아의 남발칸 반도와 서유럽 부계 기여도는 각각 약 63%와 26%입니다.[44]
2015년 라치오 동부의 6개 작은 산촌과 인근 서부 아브루초의 1개 산촌에 대한 유전자 연구에서 이들 지역사회와 주로 남성 유전자 풀에서 근동 개체군 사이에 약간의 유전적 유사성이 있음을 발견했습니다. 서아시아와 중앙아시아에서 흔히 볼 수 있는 하플로그룹 Q도 이 표본 집단에서 발견되었는데, 이는 과거에 아나톨리아에서 정착지를 유치했을 수 있음을 시사합니다.[42] 또한 이탈리아 대륙에서는 약 0.6%이지만, 시칠리아에서는 2.5%(6/236)로 상승하여 마자라 델 발로 지역에서는 16.7%(3/18)에 이르고, 라구사에서는 7.1%(2/28), 스키아카에서는 3.6%,[17] 벨베데레 마리티모에서는 3.7%에 달합니다.[45]
이탈리아 mtDNA의 유전자 구성
유럽의 다른 곳과 마찬가지로 이탈리아에서도 mtDNA 계통의 대부분은 하플로그룹 H에 속합니다. 여러 독립적인 연구에 따르면 하플로그룹 H는 아마도 25,000년 전에 서아시아에서 진화했을 것입니다. 그것은 20-25,000년 전에 유럽으로 이주하여 대륙 남서부의 인구와 함께 퍼졌습니다.[46][47] 그것의 도래는 대략 그라베티아 문화의 부상과 동시대적이었습니다. 하위 등급인 H1, H3 및 자매 하플로그룹 V의 확산은 13,000년 전 마지막 빙하 최대치 이후 프랑코-칸타브리아 지역에서 두 번째 유럽 내 확장을 반영합니다.[46][48]
아프리카 하플로그룹 L 계통은 2~3% 사이의 빈도가 발견된 라티움, 볼테라, 바실리카타 및 시칠리아를 제외하고 이탈리아 전역에서 비교적 드물게(1% 미만) 발생합니다.[49]
2012년 Brisighelli 등의 연구에 따르면 조상의 정보 표지자를 분석한 결과 이탈리아는 매우 사소한 사하라 사막 이남의 아프리카 성분을 보여주지만 지중해가 아닌 유럽보다 약간 높습니다. 아프리카 mtDNA에 대해 논의하면서, 이 연구는 이들 혈통의 상당 부분이 10,000년 이상 전에 이탈리아에 도착했을 수 있다는 것을 나타낸다고 말합니다. 따라서 이탈리아에서의 그들의 존재는 반드시 로마 제국 시대, 대서양 노예 무역 또는 현대의 이주로 거슬러 올라가는 것은 아닙니다."[22] Brisighelli 등의 이러한 mtDNA는 "아프리카 기원의 미토콘드리아 DNA 일배체형은 주로 시험된 583개의 샘플에 대해 하플로그룹 M1(0.3%), U6(0.8%) 및 L(1.2%)로 표시됩니다.[22] 하플로그룹 M1과 U6은 북아프리카 기원으로 간주될 수 있으므로 문서화된 아프리카 역사 입력을 알리는 데 사용될 수 있습니다. 하플로그룹 M1은 트라파니(서시칠리아)에서 온 2개의 보균자에서만 관찰된 반면, U6는 남아풀리아의 루세라와 반도 끝(칼라브리아)에서만 관찰되었습니다.[22]
알레시오 보아티니 등의 2013년 연구에서는 865개의 샘플 중 이탈리아 전체에서 0개의 아프리카 하플로그룹을 발견했습니다. 베르베르 M1 및 U6 하플로 그룹의 비율은 각각 0.46% 및 0.35%였습니다.[50]
Stefania Sarno et al. 의 2014년 연구에 따르면 115개의 샘플 중 남부 이탈리아 본토에서 0개의 아프리카 L 및 M1 하플로 그룹을 발견했습니다. 115개의 샘플 중 2개의 Berber U6만 발견되었으며, 1개는 Lecce에서, 1개는 Cosenza에서 발견되었습니다.[44]
아슈케나짐과 이탈리아인 사이의 유전적 유사성은 유전자 연구에서 주목되어 왔는데, 아마도 아슈케나짐 유대인들이 상당한 유럽인 혼혈(30-60%)을 가지고 있기 때문이며, 그 대부분은 남유럽인입니다. 중동 출신의 유대인 디아스포라 남성들이 로마로 이주해 유대교로 개종한 현지 여성들 사이에서 아내를 찾았을 때 이탈리아에서 온 사람들이 많았습니다.[51][52][53][54][55][13][56] 좀 더 구체적으로, 아슈케나지 유대인은 레반틴 50%와 유럽인 50%로 모델화할 수 있으며, 남유럽인의 평균 혼합률은 37.5%로 추정됩니다. 대부분(30.5%)은 이탈리아 출처에서 비롯된 것으로 보입니다.[57][58]
2010년 유대인 계보에 대한 연구에 따르면 비유대인 유럽 집단과 관련하여 아슈케나지 유대인과 가장 밀접한 관련이 있는 인구는 현대 이탈리아인이며 프랑스인과 사르디니아인이 그 뒤를 이었습니다.[59][60]
최근의 연구들은 이탈리아가 마지막 빙하기 말에 "서유럽"의 회복에 중요한 역할을 했다는 것을 보여주었습니다. 미토콘드리아 U5b3 하플로그룹에 초점을 맞춘 이 연구는 이 여성 계통이 사실 이탈리아에서 유래했으며 약 10,000년 전에 반도에서 프로방스와 발칸 반도로 확장되었다는 것을 발견했습니다. 프로방스에서는 약 9,000년에서 7,000년 전 사이에 하플로그룹 하위 분류인 U5b3a1을 생성했습니다. 이 하위 분류군 U5b3a1은 나중에 흑요석 상인을 통해 프로방스에서 사르데냐 섬으로 왔습니다. 프랑스에서 발견되는 흑요석의 80%가 사르데냐의 몬테 아르시에서 온 것으로 추정되기 때문에 이 두 지역 사이에 한때 존재했던 밀접한 관계를 반영합니다. 여전히 사르데냐의 여성 개체군의 약 4%가 이 일배체형에 속합니다.[61]
2018년 American Journal of Physical Anthology 저널에 발표된 mtDNA 연구는 선사시대, 에트루리아 시대, 로마 시대, 르네상스 및 현대 토스카나의 고대 및 현대 표본을 비교했으며 에트루리아인이 선사시대와 다른 표본의 중간에 해당하는 지역 인구로 나타난다는 결론을 내렸습니다. 신석기 시대와 로마 시대 사이의 시간적 네트워크에 위치합니다.[62]
이탈리아 중부 움브리아 지역의 고대 및 현대 표본에서 추출한 모계 하플로그룹을 2020년에 분석한 결과, 현대 움브리아인과 이 지역의 로마 이전 거주자들 사이에 상당한 유전적 유사성이 있으며, 로마 이전부터 현재까지 이 지역에서 상당한 유전적 연속성의 증거를 발견했습니다. 현대와 고대의 움브리아인 모두 mtDNA 하플로그룹 U4와 U5a의 비율이 높고 J를 과도하게 표현하는 것으로 밝혀졌습니다(약 30%). 이 연구는 또한 고대와 현대의 움브리아인들이 공유하는 "6개의 말단 가지(H1e1, J1c3, J2b1, U2e2a, U8b1b1 및 K1a4a)에 의해 국소적인 유전적 연속성이 추가로 입증된다"는 것을 발견했습니다.[6]
상염색체

남/서유럽 북유럽/동유럽 캅카스
서아시아인 남아시아인 동아시아인 북아프리카/사하라 이남 아프리카[63]
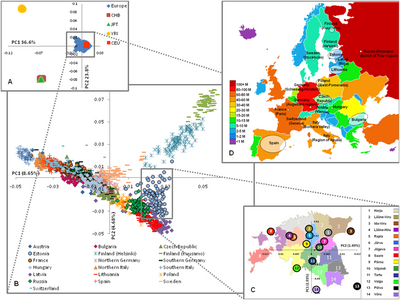
웨이드 외. (2008)은 이탈리아가 유럽에 남아있는 마지막 두 개의 유전 섬 중 하나이며, 다른 하나는 핀란드라고 밝혔습니다. 이것은 부분적으로 수백 년 동안 대규모 이주 흐름을 막아온 알파인 산맥의 존재 때문입니다.
최근의 게놈 전체 연구는 이전과는 달리 혼합물을 감지하고 정량화할 수 있었습니다. Li et al. (2008)은 600,000개 이상의 상염색체 SNP를 사용하여 유럽, 중동 및 중앙/남아시아를 포함한 7개의 글로벌 인구 클러스터를 식별합니다. 모든 이탈리아 표본은 신석기 시대의 영향을 약간 받은 중서부 그룹에 속합니다.[65]
López Herráez et al. (2009)는 100만 개에 가까운 SNP에서 동일한 샘플을 입력하고 서유라시아 맥락에서 분석하여 다수의 하위 클러스터를 식별했습니다. 이번에는 유럽 샘플 모두 약간의 혼합물을 보여줍니다. 이탈리아 사람들 중에 토스카나가 아직 가장 많고 사르데냐도 약간 있지만 더 북쪽에 있는 롬바르디아(베르가모)도 마찬가지입니다.[66]
Morjani 등의 2011년 연구에 따르면 많은 남유럽인들이 1-3%의 사하라 사막 이남의 조상을 물려받았지만, 그 비율은 '구조' 통계 모델로 재분석했을 때 더 낮았습니다(0.2-2.1%). 약 55세대/1100년 전의 평균 혼합 날짜도 "로마 제국 말기의 북아프리카 유전자 흐름과 그 이후의 아랍인 이주와 일치한다"고 계산되었습니다.[67]
Di Gaetano et al.의 2012년 연구. 지리적 범위가 넓은 1,014명의 이탈리아인을 사용했습니다. 현재 사르데냐의 개체군은 이탈리아 본토 및 시칠리아와 유전적으로 명확히 구별될 수 있으며, 현재 이탈리아 반도 개체군 내에서 어느 정도의 유전적 분화가 감지 가능하다는 것을 보여주었습니다.
저자들은 ADMIXTEXT 소프트웨어를 사용하여 K = 4에서 가장 낮은 교차 valid 오차를 얻었습니다. HapMap CEU 개인은 평균 83%의 북유럽(NE) 혈통을 보였습니다. NE 조상이 각각 70%, 56% 및 52%인 프랑스, 북부 이탈리아 및 중앙 이탈리아 인구에서도 유사한 패턴이 관찰됩니다. PCA 플롯에 따르면 ADMIXTER 분석에서도 북부 이탈리아인과 중부 이탈리아인 사이에 비교적 작은 조상 차이가 있는 반면 남부 이탈리아인은 북부 이탈리아와 중부 이탈리아보다 낮은 평균 혼합물 NE 비율(44%)과 높은 백인 조상 비율(28%)을 보였습니다. 사르데냐 샘플은 다른 유럽 개체군에서 흔히 볼 수 있는 진홍색 패턴을 보여주지만 더 높은 빈도(70%)를 나타냅니다. 현재 사르디니아 인구 내 북유럽 혈통의 평균 혼합 비율은 14.[12]3%이며, 일부 개인은 매우 낮은 북유럽 혈통을 나타냅니다(268명의 36명 중 5% 미만).
Peristera Paschou et al. 의 2013년 연구는 지중해가 초기 정착지에 이어 지리적 고립을 통한 유전자 흐름에 강력한 장벽으로 작용했음을 확인시켜줍니다. (북부) 이탈리아, 토스카나, 시칠리아 및 사르데냐의 샘플은 이베리아, 발칸 반도 및 그리스의 다른 남유럽인과 가장 가까우며, 이들은 아나톨리아의 카파도키아 샘플로 대표됩니다. 하지만 그 이후로 중동이나 북아프리카에서 이탈리아와 남유럽의 다른 지역으로 큰 혼합물이 유입되지는 않았습니다.[14]
고대 DNA 분석에 따르면 아이스맨 외치는 현대 남유럽인과 가장 가까운 이탈리아인, 특히 사르데냐 섬에서 온 사람들과 군집을 이루고 있습니다. 다른 이탈리아인들은 지리학과 일치하는 남동 유럽과 중앙 유럽 쪽으로 철수하고 일부 후기 신석기 시대의 유전자 흐름(예: 이탈리아어, 그리스어, 에트루리아어, 켈트어)이 있지만, 그것과 수세기의 역사에도 불구하고 그들은 여전히 선사 시대의 조상과 매우 유사합니다.[68]
Botigué 등의 2013년 연구는 아프리카인과 유럽인 간의 대립유전자 기반 공유를 추정하기 위해 비지도 클러스터링 알고리즘인 ADMIXTECHERT를 적용했습니다. 이탈리아인의 경우 북아프리카 혈통은 유전체의 2%를 넘지 않습니다. 평균적으로 유대인 혈통의 1%는 토스카나 합맵 인구와 이탈리아계 스위스인, 그리고 그리스인과 키프로스인에서 발견됩니다. 과거 관측과 달리, 카나리아 제도를 제외한 유럽에서는 사하라 사막 이남의 조상이 <1%로 감지됩니다.[44]
Haak et al. (2015)은 유럽과 러시아의 고대 골격 94개에 대한 게놈 전체 연구를 수행했습니다. 이 연구는 청동기 시대가 얌나 문화에서 온 목회자들이 인도유럽어족 언어를 유럽에 전파했다고 주장합니다. 자가학적 검사 결과, 얌나야족은 서로 다른 두 수렵-채집 개체군 사이의 혼합물의 결과임을 알 수 있습니다. 러시아 스텝 지역의 동부 수렵채집인들과 코카서스 수렵채집인들 또는 칼콜리트계 이란인들(매우 유사함). 볼프강 하크(Wolfgang Haak)는 현대 투스카인들의 DNA에서 얌나야족의 조상 기여도를 27%로 추정했으며, 베르가모 출신의 현대 북이탈리아인들의 DNA에서 얌나야족의 조상 기여도는 25%로 사르디니아인(7%)을 제외하고 시칠리아인(12%)[11]보다 적었습니다.
2016년 연구 사치니 등은 Di Gaetano 등(2012)과 Fiorito 등(2015)의 이전 연구 결과를 확인했지만 15개 지역의 20개 위치에서 737명의 개인이 테스트되는 등 샘플의 지리적 적용 범위가 훨씬 더 우수합니다. 이 연구에는 또한 처음으로 서유라시아 샘플을 모두 사용하여 혼합 이벤트를 추론하여 이탈리아인의 조상을 모델링하는 공식 혼합 테스트가 포함됩니다. 이 결과는 지난 몇 년 동안 나온 고대 DNA 증거에 비추어 볼 때 매우 흥미로운 것입니다.
본문에 설명된 패턴 외에도 SARD 샘플은 이동 과정의 수용체라기보다는 조사된 대부분의 개체군, 특히 이탈리아 개체군의 혼합물 공급원으로서 주요 역할을 한 것으로 보입니다. 사실, SARD를 포함한 트리오에 대한 가장 중요한 f3 점수는 반도 이탈리아인을 SARD와 이란, 코카서스 및 러시아 인구 간의 혼합의 그럴듯한 결과로 나타냈습니다. 이 시나리오는 사르데냐인들이 적어도 신석기 시대까지 유럽 전역에 상당히 널리 퍼져 있었고 그 후 현재 대부분의 유럽 인구에서 지워지거나 가려진 것으로 추정되는 조상 유전체 배경의 높은 비율을 유지하고 있다는 추가 증거로 해석될 수 있습니다.[23]
Sarno et al. (2017)은 지중해 주변의 역사적 이주가 남이탈리아와 시칠리아에 미친 유전적 영향에 초점을 맞추고, "그 결과는 오늘날 남이탈리아 인구의 유전적 변이성이 공유된 유전적 연속성을 특징으로 한다는 것을 보여주고," "이탈리아 남부는 그리스 대륙의 표본보다 키프로스 동쪽에 이르는 그리스어를 사용하는 지중해의 섬들과 더 유사한 것으로 보입니다." 이 섬들에서 덜 혼합된 형태로 생존했을 가능성이 있는 조상의 연관성을 시사합니다." 또한 "주로 신석기 시대와 유사한 구성 요소 외에도, 우리의 분석은 후기 신석기 코카서스 및 레반틴 관련 조상의 중요한 영향을 어떻게 드러냈는지 정확하게 보여줍니다."[18] 막스플랑크학회의 한 뉴스 기사는 "지중해 동부 연안의 인구는 국적을 초월한 유전적 유산을 공유하고 있다"는 내용으로 시작하면서 그 결과를 검토하고 있습니다. 또한 코카서스의 영향을 보여주면서도 폰틱-카스피안 스텝과 관련된 유전적 표지가 없기 때문에 이 연구가 유럽에서 인도유럽어족의 확산과 관련된 논쟁에서 얼마나 흥미로운지를 지적합니다. "북중동유럽과 동유럽에서 잘 대표되는 매우 특징적인 유전적 신호," 이전의 연구들이 인도유럽어족 언어들을 대륙에 도입하는 것과 관련이 있습니다."[18]
Raveane et al. (2019)은 현대 이탈리아인을 대상으로 한 게놈 전체 연구에서 주로 남부 이탈리아에서 3천년 아나톨리아 청동기 시대의 코카서스 헌터-채집자의 기여를 발견했습니다. 또한 지역별 변이 패턴은 이전 연구와 같이 남부 이탈리아, 북부 이탈리아 및 사르데냐에서 지리적 구조를 나타냈습니다. 지리와 거리, 로마 제국 말기와 그 이후의 기간 동안의 사건과 관련된 역사적 혼합으로 인해 하위 지역 군집 사이에서 훨씬 더 자세한 구조가 관찰되었습니다.[4]
안토니오 외(2019)는 라티움과 로마의 다양한 시대의 역사적 인구를 연구했습니다. 그들은 언어적 차이에도 불구하고 라틴어와 에트루리아어는 큰 유전적 차이를 보이지 않는다는 것을 발견했습니다. 그들의 상염색체 DNA는 서양 수렵-채집가(중석기), 초기 유럽 농부들(신석기), 그리고 서양 스텝 목동들(동기)의 비슷한 비율의 혼합물이었습니다.[2]
2022년 유전체 전체를 대상으로 한 연구에서는 남 지중해 지역의 700명 이상의 개체(이탈리아 남부 102명)와 인근 지역의 고대 DNA를 결합하여 남동 이탈리아인과 현대 동부 펠로폰네소스인의 높은 친화력을 발견했습니다. 그리고 고대 그리스 유전체와 남이탈리아의 특정 지역의 유전체는 현대 그리스 유전체보다 더 밀접한 관계가 있습니다.[69]
참고 항목
참고문헌
- ^ Parolo S, Lisa A, Gentilini D, Di Blasio AM, Barlera S, Nicolis EB, et al. (November 2015). "Characterization of the biological processes shaping the genetic structure of the Italian population". BMC Genetics. 16: 132. doi:10.1186/s12863-015-0293-x. PMC 4640365. PMID 26553317.
- ^ a b c d Antonio ML, Gao Z, Moots HM, Lucci M, Candilio F, Sawyer S, et al. (November 2019). "Ancient Rome: A genetic crossroads of Europe and the Mediterranean". Science. American Association for the Advancement of Science (published November 8, 2019). 366 (6466): 708–714. Bibcode:2019Sci...366..708A. doi:10.1126/science.aay6826. hdl:2318/1715466. PMC 7093155. PMID 31699931.
Interestingly, although Iron Age individuals were sampled from both Etruscan (n=3) and Latin (n=6) contexts, we did not detect any significant differences between the two groups with f4 statistics in the form of f4(RMPR_Etruscan, RMPR_Latin; test population, Onge), suggesting shared origins or extensive genetic exchange between them. ... In the Medieval and early modern periods (n = 28 individuals), we observe an ancestry shift toward central and northern Europe in PCA (Fig. 3E), as well as a further increase in the European cluster (C7) and loss of the Near Eastern and eastern Mediterranean clusters (C4 and C5) in ChromoPainter (Fig. 4C). The Medieval population is roughly centered on modern-day central Italians (Fig. 3F). It can be modeled as a two-way combination of Rome's Late Antique population and a European donor population, with potential sources including many ancient and modern populations in central and northern Europe: Lombards from Hungary, Saxons from England, and Vikings from Sweden, among others (table S26).
- ^ Ralph P, Coop G (2013). "The geography of recent genetic ancestry across Europe". PLOS Biology. 11 (5): e1001555. doi:10.1371/journal.pbio.1001555. PMC 3646727. PMID 23667324.
- ^ a b c d e Raveane A, Aneli S, Montinaro F, Athanasiadis G, Barlera S, Birolo G, et al. (September 2019). "Population structure of modern-day Italians reveals patterns of ancient and archaic ancestries in Southern Europe". Science Advances. 5 (9): eaaw3492. Bibcode:2019SciA....5.3492R. doi:10.1126/sciadv.aaw3492. PMC 6726452. PMID 31517044.
- ^ a b Capocasa M, Anagnostou P, Bachis V, Battaggia C, Bertoncini S, Biondi G, et al. (2014). "Linguistic, geographic and genetic isolation: a collaborative study of Italian populations". Journal of Anthropological Sciences. 92 (92): 201–31. doi:10.4436/JASS.92001. PMID 24607994.
- ^ a b c Modi A, Lancioni H, Cardinali I, Capodiferro MR, Rambaldi Migliore N, Hussein A, et al. (July 2020). "The mitogenome portrait of Umbria in Central Italy as depicted by contemporary inhabitants and pre-Roman remains". Scientific Reports. 10 (1): 10700. Bibcode:2020NatSR..1010700M. doi:10.1038/s41598-020-67445-0. PMC 7329865. PMID 32612271.
- ^ a b Chiang CW, Marcus JH, Sidore C, Biddanda A, Al-Asadi H, Zoledziewska M, et al. (October 2018). "Genomic history of the Sardinian population". Nature Genetics. 50 (10): 1426–1434. doi:10.1038/s41588-018-0215-8. PMC 6168346. PMID 30224645.
- ^ a b c d Marcus JH, Posth C, Ringbauer H, Lai L, Skeates R, Sidore C, et al. (February 2020). "Genetic history from the Middle Neolithic to present on the Mediterranean island of Sardinia". Nature Communications. 11 (1): 939. Bibcode:2020NatCo..11..939M. doi:10.1038/s41467-020-14523-6. PMC 7039977. PMID 32094358.
- ^ a b Fernandes DM, Mittnik A, Olalde I, Lazaridis I, Cheronet O, Rohland N, et al. (March 2020). "The spread of steppe and Iranian-related ancestry in the islands of the western Mediterranean". Nature Ecology & Evolution. 4 (3): 334–345. doi:10.1038/s41559-020-1102-0. PMC 7080320. PMID 32094539.
- ^ «시실리와 남부 이탈리아는 기원전 8-9세기에 시작하여 그리스인들에 의해 심하게 식민지화되었습니다. 남부 이탈리아에 있는 그리스 식민지의 인구학적인 발전은 놀라웠고, 고전 시대에 이 지역은 아마도 모국의 그리스 인구수를 능가했기 때문에 마그나 그레키아 (그레이트 그리스)라고 불렸습니다. »
- ^ a b Haak W, Lazaridis I, Patterson N, Rohland N, Mallick S, Llamas B, et al. (June 2015). "Massive migration from the steppe was a source for Indo-European languages in Europe". Nature. 522 (7555): 207–11. arXiv:1502.02783. Bibcode:2015Natur.522..207H. doi:10.1038/nature14317. PMC 5048219. PMID 25731166.
- ^ a b c d Di Gaetano C, Voglino F, Guarrera S, Fiorito G, Rosa F, Di Blasio AM, et al. (2012). "An overview of the genetic structure within the Italian population from genome-wide data". PLOS ONE. 7 (9): e43759. Bibcode:2012PLoSO...743759D. doi:10.1371/journal.pone.0043759. PMC 3440425. PMID 22984441.
- ^ a b Price AL, Butler J, Patterson N, Capelli C, Pascali VL, Scarnicci F, et al. (January 2008). "Discerning the ancestry of European Americans in genetic association studies". PLOS Genetics. 4 (1): e236. doi:10.1371/journal.pgen.0030236. PMC 2211542. PMID 18208327.
- ^ a b Paschou P, Drineas P, Yannaki E, Razou A, Kanaki K, Tsetsos F, et al. (June 2014). "Maritime route of colonization of Europe". Proceedings of the National Academy of Sciences of the United States of America. 111 (25): 9211–6. Bibcode:2014PNAS..111.9211P. doi:10.1073/pnas.1320811111. PMC 4078858. PMID 24927591.
- ^ a b c Fiorito G, Di Gaetano C, Guarrera S, Rosa F, Feldman MW, Piazza A, Matullo G (July 2016). "The Italian genome reflects the history of Europe and the Mediterranean basin". European Journal of Human Genetics. 24 (7): 1056–62. doi:10.1038/ejhg.2015.233. PMC 5070887. PMID 26554880.
- ^ 안토니오 외. 2019, 페이지 2.
- ^ a b c Di Gaetano C, Cerutti N, Crobu F, Robino C, Inturri S, Gino S, et al. (January 2009). "Differential Greek and northern African migrations to Sicily are supported by genetic evidence from the Y chromosome". European Journal of Human Genetics. 17 (1): 91–9. doi:10.1038/ejhg.2008.120. PMC 2985948. PMID 18685561.
The genetic contribution of Greek chromosomes to the Sicilian gene pool is estimated to be about 37% whereas the contribution of North African populations is estimated to be around 6%.
- ^ a b c d Sarno S, Boattini A, Pagani L, Sazzini M, De Fanti S, Quagliariello A, et al. (May 2017). "Ancient and recent admixture layers in Sicily and Southern Italy trace multiple migration routes along the Mediterranean". Scientific Reports. 7 (1): 1984. Bibcode:2017NatSR...7.1984S. doi:10.1038/s41598-017-01802-4. PMC 5434004. PMID 28512355.
- ^ 안토니오 외. 2019, 페이지 3.
- ^ Wade, Lizzie (2019). "Immigrants from the Middle East shaped Rome". Science. 366 (6466): 673. Bibcode:2019Sci...366..673W. doi:10.1126/science.366.6466.673. PMID 31699914. S2CID 207965960. Archived from the original on 2021-02-24. Retrieved 2021-01-25."무역로는 사람과 물건을 새로운 수도로 보냈고, 전염병과 침입으로 로마의 인구는 약 10만 명으로 줄었습니다. 침략한 야만인들은 더 많은 유럽 혈통을 가져왔습니다. 로마는 점차 동지중해와 중동과의 강한 유전적 연관성을 상실했습니다. 중세 시대에 도시 주민들은 유전적으로 다시 유럽 인구와 닮았습니다."
- ^ ..."La separazione dell Sardegna dal resto del continente, Anzi dutte altre popolazioni europe, che probamente revela laun' original pi ù 안티카 델라 수아 포폴라지오네, 인디펜덴테 다 quella delle popolazioni Italiche consendenzel Miterano Medi-Orientale." 알베르토 광장, I profili genetici degli 이탈리아니, Academia delle Scienze di Torino
- ^ a b c d Brisighelli F, Álvarez-Iglesias V, Fondevila M, Blanco-Verea A, Carracedo A, Pascali VL, et al. (2012). "Uniparental markers of contemporary Italian population reveals details on its pre-Roman heritage". PLOS ONE. 7 (12): e50794. Bibcode:2012PLoSO...750794B. doi:10.1371/journal.pone.0050794. PMC 3519480. PMID 23251386.
- ^ a b Sazzini M, Gnecchi Ruscone GA, Giuliani C, Sarno S, Quagliariello A, De Fanti S, et al. (September 2016). "Complex interplay between neutral and adaptive evolution shaped differential genomic background and disease susceptibility along the Italian peninsula". Scientific Reports. 6: 32513. Bibcode:2016NatSR...632513S. doi:10.1038/srep32513. PMC 5007512. PMID 27582244. Zendo: 165505.
- ^ Esko T, Mezzavilla M, Nelis M, Borel C, Debniak T, Jakkula E, et al. (June 2013). "Genetic characterization of northeastern Italian population isolates in the context of broader European genetic diversity". European Journal of Human Genetics. 21 (6): 659–65. doi:10.1038/ejhg.2012.229. PMC 3658181. PMID 23249956.
- ^ a b Nelis M, Esko T, Mägi R, Zimprich F, Zimprich A, Toncheva D, et al. (2009). "Genetic structure of Europeans: a view from the North-East". PLOS ONE. 4 (5): e5472. Bibcode:2009PLoSO...4.5472N. doi:10.1371/journal.pone.0005472. PMC 2675054. PMID 19424496.
- ^ Sazzini M, Abondio P, Sarno S, Gnecchi-Ruscone GA, Ragno M, Giuliani C, et al. (May 2020). "Genomic history of the Italian population recapitulates key evolutionary dynamics of both Continental and Southern Europeans". BMC Biology. 18 (1): 51. doi:10.1186/s12915-020-00778-4. PMC 7243322. PMID 32438927.
- ^ Rootsi S (December 2006). "Y-Chromosome haplogroup I prehistoric gene flow in Europe" (PDF). Documenta Praehistorica. 33: 17–20. doi:10.4312/dp.33.3. Archived from the original (PDF) on 2009-03-06.
- ^ "Culture del bronzo recente in Italia settentrionale e loro rapporti con la "cultura dei campi di urne"". Gruppo Archeologico Monteclarense. Archived from the original on 10 May 2006.
- ^ Haywood J (2014). The Celts: Bronze Age to New Age. Routledge. p. 21. ISBN 978-1-317-87017-3.
- ^ "Lombard people". Encyclopaedia Britannica.
- ^ a b c Grugni V, Raveane A, Mattioli F, Battaglia V, Sala C, Toniolo D, et al. (February 2018). "Reconstructing the genetic history of Italians: new insights from a male (Y-chromosome) perspective". Annals of Human Biology. 45 (1): 44–56. doi:10.1080/03014460.2017.1409801. PMID 29382284. S2CID 43501209.
- ^ Di Giacomo F, Luca F, Anagnou N, Ciavarella G, Corbo RM, Cresta M, et al. (September 2003). "Clinal patterns of human Y chromosomal diversity in continental Italy and Greece are dominated by drift and founder effects" (PDF). Molecular Phylogenetics and Evolution. 28 (3): 387–95. doi:10.1016/s1055-7903(03)00016-2. PMID 12927125. Archived from the original (PDF) on 2017-01-20.
- ^ Francalacci, P.; Morelli, L.; Underhill, P. A.; Lillie, A. S.; Passarino, G.; Useli, A.; Madeddu, R.; Paoli, G.; Tofanelli, S.; Calò, C. M.; Ghiani, M. E.; Varesi, L.; Memmi, M.; Vona, G.; Lin, A. A. (July 2003). "Peopling of three Mediterranean islands (Corsica, Sardinia, and Sicily) inferred by Y-chromosome biallelic variability". American Journal of Physical Anthropology. 121 (3): 270–279. doi:10.1002/ajpa.10265. ISSN 0002-9483. PMID 12772214.
- ^ Francalacci, Paolo; Morelli, Laura; Angius, Andrea; Berutti, Riccardo; Reinier, Frederic; Atzeni, Rossano; Pilu, Rosella; Busonero, Fabio; Maschio, Andrea; Zara, Ilenia; Sanna, Daria; Useli, Antonella; Urru, Maria Francesca; Marcelli, Marco; Cusano, Roberto (2013-08-02). "Low-Pass DNA Sequencing of 1200 Sardinians Reconstructs European Y-Chromosome Phylogeny". Science. 341 (6145): 565–569. Bibcode:2013Sci...341..565F. doi:10.1126/science.1237947. ISSN 0036-8075. PMC 5500864. PMID 23908240.
- ^ a b Capelli C, Brisighelli F, Scarnicci F, Arredi B, Caglia' A, Vetrugno G, et al. (July 2007). "Y chromosome genetic variation in the Italian peninsula is clinal and supports an admixture model for the Mesolithic-Neolithic encounter". Molecular Phylogenetics and Evolution. 44 (1): 228–39. doi:10.1016/j.ympev.2006.11.030. PMID 17275346.
- ^ Cavalli-Sforza L, Menozzi P, Piazza A (1994). The History and Geography of Human Genes. Princeton University Press. ISBN 978-0-691-08750-4.>: 295
- ^ Semino O, Magri C, Benuzzi G, Lin AA, Al-Zahery N, Battaglia V, et al. (May 2004). "Origin, diffusion, and differentiation of Y-chromosome haplogroups E and J: inferences on the neolithization of Europe and later migratory events in the Mediterranean area". American Journal of Human Genetics. 74 (5): 1023–34. doi:10.1086/386295. PMC 1181965. PMID 15069642.
- ^ Barker, Graeme; Rasmussen, Tom (2000). The Etruscans. The Peoples of Europe. Oxford: Blackwell Publishing. p. 44. ISBN 978-0-631-22038-1.
- ^ Turfa, Jean MacIntosh (2017). "The Etruscans". In Farney, Gary D.; Bradley, Gary (eds.). The Peoples of Ancient Italy. Berlin: De Gruyter. pp. 637–672. doi:10.1515/9781614513001. ISBN 978-1-61451-520-3.
- ^ De Grummond, Nancy T. (2014). "Ethnicity and the Etruscans". In McInerney, Jeremy (ed.). A Companion to Ethnicity in the Ancient Mediterranean. Chichester, UK: John Wiley & Sons, Inc. pp. 405–422. doi:10.1002/9781118834312. ISBN 9781444337341.
- ^ Shipley, Lucy (2017). "Where is home?". The Etruscans: Lost Civilizations. London: Reaktion Books. pp. 28–46. ISBN 9781780238623.
- ^ a b Messina F, Finocchio A, Rolfo MF, De Angelis F, Rapone C, Coletta M, et al. (2015). "Traces of forgotten historical events in mountain communities in Central Italy: A genetic insight". American Journal of Human Biology. 27 (4): 508–19. doi:10.1002/ajhb.22677. PMID 25728801. S2CID 30111156.
- ^ Capelli C, Onofri V, Brisighelli F, Boschi I, Scarnicci F, Masullo M, et al. (June 2009). "Moors and Saracens in Europe: estimating the medieval North African male legacy in southern Europe". European Journal of Human Genetics. 17 (6): 848–52. doi:10.1038/ejhg.2008.258. PMC 2947089. PMID 19156170.
- ^ a b c d Sarno S, Boattini A, Carta M, Ferri G, Alù M, Yao DY, et al. (2014). "An ancient Mediterranean melting pot: investigating the uniparental genetic structure and population history of sicily and southern Italy". PLOS ONE. 9 (4): e96074. Bibcode:2014PLoSO...996074S. doi:10.1371/journal.pone.0096074. PMC 4005757. PMID 24788788.
 This article contains quotations from this source, which is available under a Creative Commons Attribution 4.0 International (CC BY 4.0) license.
This article contains quotations from this source, which is available under a Creative Commons Attribution 4.0 International (CC BY 4.0) license. - ^ Brisighelli, F (2012). "Uniparental markers of contemporary Italian population reveals details on its pre-Roman heritage". PLOS ONE. 7 (12): e50794. Bibcode:2012PLoSO...750794B. doi:10.1371/journal.pone.0050794. PMC 3519480. PMID 23251386. 표 S4에서 #BEL50은 일배체형으로 Q-M378로 추정되지만 P*(xR1)로만 표시됩니다.
- ^ a b Pereira L, Richards M, Goios A, Alonso A, Albarrán C, Garcia O, et al. (January 2005). "High-resolution mtDNA evidence for the late-glacial resettlement of Europe from an Iberian refugium". Genome Research. 15 (1): 19–24. doi:10.1101/gr.3182305. PMC 540273. PMID 15632086.
- ^ Richards M, Macaulay V, Hickey E, Vega E, Sykes B, Guida V, et al. (November 2000). "Tracing European founder lineages in the Near Eastern mtDNA pool". American Journal of Human Genetics. 67 (5): 1251–76. doi:10.1016/S0002-9297(07)62954-1. PMC 1288566. PMID 11032788.
- ^ Achilli A, Rengo C, Magri C, Battaglia V, Olivieri A, Scozzari R, et al. (November 2004). "The molecular dissection of mtDNA haplogroup H confirms that the Franco-Cantabrian glacial refuge was a major source for the European gene pool". American Journal of Human Genetics. 75 (5): 910–8. doi:10.1086/425590. PMC 1182122. PMID 15382008.
- ^ Achilli A, Olivieri A, Pala M, Metspalu E, Fornarino S, Battaglia V, et al. (April 2007). "Mitochondrial DNA variation of modern Tuscans supports the near eastern origin of Etruscans". American Journal of Human Genetics. 80 (4): 759–68. doi:10.1086/512822. PMC 1852723. PMID 17357081.
4/138=2.9% in Latium; 3/114=2.6% in Volterra; 2/92=2.2% in Basilicata and 3/154=2.0% in Sicily
- ^ Boattini A, Martinez-Cruz B, Sarno S, Harmant C, Useli A, Sanz P, et al. (2013). "Uniparental markers in Italy reveal a sex-biased genetic structure and different historical strata". PLOS ONE. 8 (5): e65441. Bibcode:2013PLoSO...865441B. doi:10.1371/journal.pone.0065441. PMC 3666984. PMID 23734255.
- ^ Balter M (3 June 2010). "Tracing the Roots of Jewishness". Science Magazine. American Association for the Advancement of Science.
- ^ Zoossmann-Diskin A (October 2010). "The origin of Eastern European Jews revealed by autosomal, sex chromosomal and mtDNA polymorphisms". Biology Direct. 5 (57): 57. doi:10.1186/1745-6150-5-57. PMC 2964539. PMID 20925954.
- ^ Balter M (8 October 2013). "Did Modern Jews Originate in Italy?". ScienceNOW.
- ^ Yandell K (2013). "Genetic Roots of the Ashkenazi Jews". The Scientist.
- ^ Rosenberg NA, Pritchard JK, Weber JL, Cann HM, Kidd KK, Zhivotovsky LA, Feldman MW (December 2002). "Genetic structure of human populations". Science. 298 (5602): 2381–5. Bibcode:2002Sci...298.2381R. doi:10.1126/science.1078311. PMID 12493913. S2CID 8127224.
- ^ Costa MD, Pereira JB, Pala M, Fernandes V, Olivieri A, Achilli A, et al. (2013). "A substantial prehistoric European ancestry amongst Ashkenazi maternal lineages". Nature Communications. 4: 2543. Bibcode:2013NatCo...4.2543C. doi:10.1038/ncomms3543. PMC 3806353. PMID 24104924.
- ^ Banda Y, Kvale M, Hoffmann T, Hesselson S, Tang H, Ranatunga D, et al. (2013). "Admixture Estimation in a Founder Population". Am Soc Hum Genet. Archived from the original on 2019-08-11. Retrieved 2016-10-31.
- ^ Bray SM, Mulle JG, Dodd AF, Pulver AE, Wooding S, Warren ST (September 2010). "Signatures of founder effects, admixture, and selection in the Ashkenazi Jewish population". Proceedings of the National Academy of Sciences of the United States of America. 107 (37): 16222–7. Bibcode:2010PNAS..10716222B. doi:10.1073/pnas.1004381107. PMC 2941333. PMID 20798349.
- ^ Atzmon G, Hao L, Pe'er I, Velez C, Pearlman A, Palamara PF, Morrow B, Friedman E, Oddoux C, Burns E, Ostrer H (June 2010). "Abraham's children in the genome era: major Jewish diaspora populations comprise distinct genetic clusters with shared Middle Eastern Ancestry". American Journal of Human Genetics. 86 (6): 850–9. doi:10.1016/j.ajhg.2010.04.015. PMC 3032072. PMID 20560205.
- ^ "Genes Set Jews Apart, Study Finds". American Scientist. Retrieved 8 November 2013.
- "Study finds genetic links among Jewish people". EurekAlert! (Press release). 3 June 2010.
- ^ Pala M, Achilli A, Olivieri A, Hooshiar Kashani B, Perego UA, Sanna D, et al. (June 2009). "Mitochondrial haplogroup U5b3: a distant echo of the epipaleolithic in Italy and the legacy of the early Sardinians" (PDF). American Journal of Human Genetics. 84 (6): 814–21. doi:10.1016/j.ajhg.2009.05.004. PMC 2694970. PMID 19500771. Archived from the original (PDF) on 2018-07-22. Retrieved 2010-07-15.
- ^ Leonardi, Michela; Sandionigi, Anna; Conzato, Annalisa; Vai, Stefania; Lari, Martina (2018). "The female ancestor's tale: Long‐term matrilineal continuity in a nonisolated region of Tuscany". American Journal of Physical Anthropology. New York City: John Wiley & Sons (published September 6, 2018). 167 (3): 497–506. doi:10.1002/ajpa.23679. PMID 30187463. S2CID 52161000.
- ^ Kovacevic L, Tambets K, Ilumäe AM, Kushniarevich A, Yunusbayev B, Solnik A, et al. (2014). "Standing at the gateway to Europe--the genetic structure of Western balkan populations based on autosomal and haploid markers". PLOS ONE. 9 (8): e105090. Bibcode:2014PLoSO...9j5090K. doi:10.1371/journal.pone.0105090. PMC 4141785. PMID 25148043.
- ^ Wade N (13 August 2008). "Genetic Map of Europe". New York Times. Retrieved 25 February 2014.
- ^ Li JZ, Absher DM, Tang H, Southwick AM, Casto AM, Ramachandran S, Cann HM, Barsh GS, Feldman M, Cavalli-Sforza LL, Myers RM (February 2008). "Worldwide human relationships inferred from genome-wide patterns of variation" (PDF). Science. New York, N.Y. 319 (5866): 1100–4. Bibcode:2008Sci...319.1100L. doi:10.1126/science.1153717. PMID 18292342. S2CID 53541133. Archived from the original (PDF) on 2009-03-27.
- ^ López Herráez D, Bauchet M, Tang K, Theunert C, Pugach I, Li J, et al. (November 2009). "Genetic variation and recent positive selection in worldwide human populations: evidence from nearly 1 million SNPs". PLOS ONE. 4 (11): e7888. Bibcode:2009PLoSO...4.7888L. doi:10.1371/journal.pone.0007888. PMC 2775638. PMID 19924308.
- ^ Moorjani P, Patterson N, Hirschhorn JN, Keinan A, Hao L, Atzmon G, Burns E, Ostrer H, Price AL, Reich D (April 2011). "The history of African gene flow into Southern Europeans, Levantines, and Jews". PLOS Genetics. 7 (4): e1001373. doi:10.1371/journal.pgen.1001373. PMC 3080861. PMID 21533020.
- ^ Keller A, Graefen A, Ball M, Matzas M, Boisguerin V, Maixner F, et al. (February 2012). "New insights into the Tyrolean Iceman's origin and phenotype as inferred by whole-genome sequencing". Nature Communications. 3: 698. Bibcode:2012NatCo...3..698K. doi:10.1038/ncomms1701. PMID 22426219.
- ^ Raveane, Alessandro; Molinaro, Ludovica; Aneli, Serena; Capodiferro, Marco Rosario; Ongaro, Linda; Migliore, Nicola Rambaldi; Soffiati, Sara; Scarano, Teodoro; Torroni, Antonio; Achilli, Alessandro; Ventura, Mario; Pagani, Luca; Capelli, Cristian; Olivieri, Anna; Bertolini, Francesco (2022-03-01). "Assessing temporal and geographic contacts across the Adriatic Sea through the analysis of genome-wide data from Southern Italy": 2022.02.26.482072. doi:10.1101/2022.02.26.482072. S2CID 247231413.
{{cite journal}}: 저널 인용은 다음과 같습니다.journal=(도움말)


