O-GlcNAc
O-GlcNAcO-GlcNAc(O-Linked GlcNAc 또는 O-Linked β-N-acetyllucosamine의 줄임말)는 핵세포질 단백질의 세린과 세로닌 잔류물에서 발견되는 가역성 효소 후 변형이다.수정은 세린 또는 트레오닌 사이드 체인의 히드록실 그룹과 N-아세틸글루코사민(GlcNAc) 사이의 β-글리코시디드 결합으로 특징지어진다.O-GlcNAc differs from other forms of protein glycosylation: (i) O-GlcNAc is not elongated or modified to form more complex glycan structures, (ii) O-GlcNAc is almost exclusively found on nuclear and cytoplasmic proteins rather than membrane proteins and secretory proteins, and (iii) O-GlcNAc is a highly dynamic modification that turns over more ra변형되는 단백질보다 더 작고 보송보송하다.O-GlcNAc는 메타조인들에 걸쳐 보존되어 있다.[1]

O-GlcNAc의 동적 특성과 세린 및 트레오닌 잔류물에 존재하기 때문에, O-GlcNAcylation은 어떤 면에서는 단백질 인산화와 유사하다.인간에게 단백질 인산화를 조절하는 키나제 약 500개와 인산염 150개가 있는 반면 O-GlcNAc : O-GlcNAc전달효소(OGT)와 O-GlcNAcase(OGA)의 순환을 조절하는 효소는 각각 2개에 불과하다.[2]OGT는 UDP-GlcNAc를 설탕 전이를 위한 기증 설탕으로 사용한다.[3]
1984년에 처음 보고된 이 변환 후 수정은 그 후 5,000개 이상의 단백질에서 확인되었다.[4][5]O-GlcNAcylation의 수많은 기능적 역할은 세린/트레오닌 인산화와의 크로스스토킹, 단백질-단백질 상호작용 조절, 단백질 구조 또는 효소 활성 변화, 단백질 아세포 국산화 변화, 단백질 안정성과 저하 조절을 포함한다.[1]셀의 전사 기계의 수많은 구성 요소들이 O-GlcNAc에 의해 변형된 것으로 확인되었으며, 많은 연구에서 O-GlcNAc, 전사, 후생유전학 사이의 연관성이 보고되었다.[6][7]많은 다른 세포 과정들은 세포사멸, 세포주기, 스트레스 반응과 같은 O-GlcNAc의 영향을 받는다.[8]UDP-GlcNAc는 아미노산, 탄수화물, 지방산, 뉴클레오티드 대사를 통합한 헥소아민 생합성 경로의 최종 산물인 만큼 O-GlcNAc가 '영양 센서' 역할을 하며 세포의 대사 상태에 대응한다는 제안이 나왔다.[9]O-GlcNAc의 조절장애는 알츠하이머병, 암, 당뇨병, 신경퇴행장애를 포함한 많은 병리학에서 연관되어 왔다.[10]
디스커버리
1984년, 하트 연구소는 흉선세포와 림프구 표면에 있는 단자 GlcNAc 잔류물을 조사하고 있었다.단자 GlcNAc 잔류물과 반응하는 소젖 β-1,4-갈락토실전달효소는 UDP-[3H]갈락토스로 방사선을 쬐는 데 사용되었다.세린과 트레오닌 잔류물의 β-제거는 3[H]갈락토오스 대부분이 단백질 O-글리코시즘에 부착되었음을 입증하였으며, 크로마토그래피에 따르면 주요 β-제거제 제품은 Galβ1-4GlcNAcitol이었다.펩타이드 N글리코시다아제 치료제에 대한 불감증이 O연계 GlcNAc에 대한 추가 증거를 제공했다.방사선 처리 전 세제로 세포 투과 안정화는 갈β1-4GlcNAcitol에 통합된 3[H]갈락토오스 함량을 크게 증가시켜, 저자들은 O-연계 GlcNAc 단당 잔류물의 대부분이 세포 내라고 결론지었다.[11]
메커니즘
O-GlcNAc는 일반적으로 다양한 단백질을 껐다 켤 수 있는 동적 변형이다.일부 잔류물은 O-GlcNAc에 의해 구성적으로 변형된 것으로 생각된다.[12][13]O-GlcNAc 수정은 OGT에 의해 순차적 bi-bi 메커니즘에 설치되며, 기증자 설탕 UDP-GlcNAc가 먼저 OGT에 결합한 후 기질 단백질로 결합된다.[14]O-GlcNAc 수정은 수정되지 않은 단백질과 GlcNAc를 산출하기 위해 앵커리지 보조(부하 보조 촉매)를 포함하는 가수 분해 메커니즘에서 OGA에 의해 제거된다.[15]결정 구조가[14] OGT와 OGA에 대해 보고되었지만,[16][17] OGT와 OGA가 기판을 인식하는 정확한 메커니즘은 완전히 설명되지 않았다.글리코실레이션이 특정 컨센서스 시퀀스(Asn-X-Ser/Thr, 여기서 X는 Pro를 제외한 모든 아미노산)에서 발생하는 N 연계 글리코실레이션과 달리 O-GlcNAc에 대한 결정적인 컨센서스 시퀀스는 확인되지 않았다.따라서 O-GlcNAc 수정 부지를 예측하는 것은 어려운 일이며, 수정 부지를 식별하는 것은 일반적으로 질량 분석 방법이 필요하다.OGT의 경우, 초헬리틱 TPR 영역의 루멘에 있는 아스파라게이트[18] 및 아스파라긴[19] 사다리 모티브를 포함한 여러 요인에 의해 기질 인식이 조절된다는 연구 결과가 나왔다.[20][21]결정 구조에서 OGT는 그 기질이 확장된 순응에 있어야 한다는 것을 보여주었듯이, OGT는 유연한 기판을 선호한다는 것이 제안되었다.[20]단백질 기질 패널에서 OGT와 OGA 활성을 측정하는 체외 운동 실험에서 OGT에 대한 운동 매개변수는 다양한 단백질 사이에서 가변적인 반면, OGA에 대한 운동 매개변수는 다양한 단백질 사이에서 상대적으로 일정했다.이 결과는 OGT가 O-GlcNAc를 규제하는 "상위 파트너"이며 OGA는 변형된 단백질의 정체성보다는 O-GlcNAc의 존재를 통해 기판을 주로 인식한다는 것을 시사했다.[12]

탐지 및 특성화
O-GlcNAc의 존재를 감지하고 수정된 특정 잔류물을 특성화하기 위한 몇 가지 방법이 존재한다.
렉틴스
식물 렉틴인 밀 배아 아글루틴은 단자 GlcNAc 잔류물을 인식할 수 있어 O-GlcNAc 검출에 자주 사용된다.이 강의는 O-GlcNAc의 농축과 탐지를 위한 렉틴 친화도 크로마토그래피에 적용되었다.[22]
항체
변형된 단백질의 정체성과 무관하게 O-GlcNAc 수정을 인식하는 범O-GlcNAc 항체가 주로 사용된다.여기에는 O-GlcNAcylated 핵공기 복합단백질에 대해 배양된 IgG 항체 [23]RL2와 단일 세린 O-GlcNAC 수정으로 면역 유발 펩타이드에 대해 배양된 IgM 항체 [24]CTD110.6이 포함된다.다른 O-GlcNAC 특이 항체가 보고되었고 변형된 단백질의 정체성에 어느 정도 의존하고 있는 것으로 입증되었다.[25]
대사 라벨링
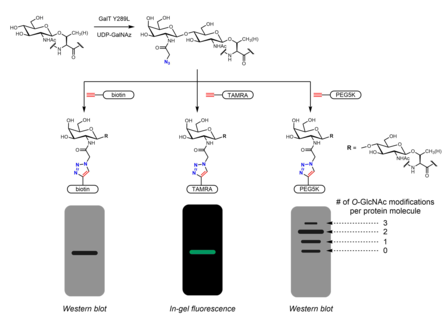
많은 대사 화학 리포터들이 O-GlcNAc를 식별하기 위해 개발되었다.대사 화학 리포터는 일반적으로 추가적인 반응성을 허용하는 추가적인 화학 물질을 함유하는 설탕 유사점이다.예를 들어, 과민성 GlcNAc(AcGlcNAz4)는 GlcNAz에 에스테라제(esterase)에 의해 세포내 분해되어 헥소사민 인양 경로에서 UDP-GlcNAz로 변환되는 세포 과민성 아지도당이다.UDP-GlcNAz는 OGT에 의해 설탕 기증자로 활용되어 O-GlcNAz 수정을 할 수 있다.[26]아지도 설탕의 존재는 아지드 알키네 사이클로어 추가 반응에서 알키네 함유 생체 직교 화학 탐침을 통해 시각화할 수 있다.이러한 탐침은 쉽게 식별할 수 있는 태그(예: Flag 펩타이드, 바이오틴, 염료 분자)를 포함할 수 있다.[26][27]폴리에틸렌 글리콜(PEG)을 기반으로 한 질량 태그도 O-GlcNAc 스토이치측정법 측정에 사용되었다.5 kDa PEG 분자의 결합은 변형된 단백질의 질량 이동으로 이어진다 - O-GlcNAcylated 단백질은 여러 개의 PEG 분자를 가지며 따라서 젤 전기영동체에서 더 천천히 이동한다.[28]아지드나 알킨(일반적으로 2개 또는 6개 포지션에서)을 함유한 다른 대사 화학 리포터가 보고되었다.[29]UDP-갈락토스-4'-에피메라아제(GALE)의 작용으로 인해 UDP-GalNAc가 세포 내 UDP-GlcNAc와 평형을 이루면서 GalNAc 아날로그를 사용할 수 있다.AcGalNAz를4 사용한 치료는 AcGlcNAz에4 비해 O-GlcNAc의 라벨링이 강화된 것으로 확인되었으며, 이는 UDP-GlcNAz-1-P에서 UDP-GlcNAz까지의 UDP-Glcna pyrophosphorylase 처리의 병목 현상으로 인한 것일 수 있다.[30]불소 라벨 UDP-GlcNAc 아날로그에 세포내 처리되는 대사화학 리포터 2AcGlcN-β-Ala-NBD-α-1-P3(Ac-SAATE)가 살아있는 세포에서 O-GlcNAC의 1단계 형광 표시를 달성한 것으로 나타났다.[31]
대사 라벨링은 또한 O-GlcNAcylated 단백질의 결합 파트너를 식별하는 데 사용될 수 있다.N-acetyl 그룹은 diazirine moiety를 포함하도록 길어질 수 있다.인산염으로 보호된 과민성 AcGlcNDAz-1-P3(Ac-SATE)2로 세포를 치료하면 O-GlcNDAz로 단백질이 변형된다.그 후 UV 조사는 O-GlcNDaz 수정을 포함하는 단백질과 상호작용하는 단백질 사이의 광합성을 유도한다.[32]
예를 들어, 여러 가지 대사 화학 리포터에게 일부 문제가 확인되었다. 예를 들어, 그 사용으로 인해 헥소아민 생합성 경로를 억제할 수 있고,[29] OGA에 의해 인식되지 않을 수 있으며, 따라서 O-GlcNAc 사이클링을 포착할 수 없거나,[33] 비밀 단백질에서 볼 수 있는 O-GlcNAc 이외의 글리코실화 변형에 통합될 수 있다.[34]아세틸 그룹이 단백질 아세틸화에 사용될 수 있는 아세틸 유사물로 가수 분해될 수 있기 때문에 N-아세틸 위치에 화학적 핸들이 있는 대사 화학 리포터도 아세틸화 단백질에 라벨을 붙일 수 있다.[35]
화학적 라벨링
화학적 라벨링은 클릭 화학에 대한 핸들을 통합하는 대체 전략을 제공한다.하이윌슨그룹이 개발해 인비트로젠이 상용화한 클릭-IT O-GlcNAc 효소표기시스템은 돌연변이 GalT Y289L 효소를 활용, 아지도갈락토오스(GalNAz)를 O-GlcNAC에 전달할 수 있다.[27][36]GalNAz(따라서 O-GlcNAc도 역시 O-GlcNAc)의 존재는 바이오틴,[36] 염색분자,[27] PEG와 같은 식별 가능한 태그를 가진 다양한 알키네 함유 탐침을 통해 검출할 수 있다.[28]
Förster 공명 에너지 전달 바이오센서
Förster 공명 에너지 전달을 이용하여 O-GlcNAc 수준의 변화를 감지할 수 있는 공학적 단백질 바이오센서가 개발되었다.이 센서는 시안 형광 단백질(CFP), O-GlcNAc 결합 도메인(단자 β-O-GlcNAc에 민감한 렉틴, 단자 β-GlcNAc에 민감한 렉틴), 알려진 OGT 기질인 CKII 펩타이드, 황색 형광 단백질(YFP) 등 4가지로 구성된다.CKII 펩타이드의 O-GlcNAcylation에 따라 GafD 도메인은 O-GlcNAc moiety를 결합하여 CFP와 YFP 도메인을 근접하게 만들고 FET 신호를 생성한다.이 신호의 생성은 되돌릴 수 있으며 다양한 치료에 대응하여 O-GlcNAC 역학을 모니터링하는 데 사용할 수 있다.이 센서는 유전적으로 암호화되어 세포에 사용될 수 있다.[37]국소화 시퀀스를 추가하면 이 O-GlcNAc 센서를 핵, 세포질 또는 플라스마 막에 조준할 수 있다.[38]
질량분석법
Western Blotting과 같은 생화학적 접근방식은 단백질이 O-GlcNAc에 의해 변형된다는 입증 증거를 제공할 수 있다; 질량분석(MS)은 O-GlcNAc의 존재에 대한 결정적인 증거를 제공할 수 있다.MS를 적용한 당단백질 연구는 O-GlcNAc에 의해 수정된 단백질의 식별에 기여했다.
O-GlcNAc는 부분하위계수학이고 수정되지 않은 펩타이드의 존재에서 이온 억제가 일어나기 때문에 보통 질량 분광 분석 전에 농축 단계를 수행한다.이것은 강의실, 항체 또는 화학적 태깅을 사용하여 수행될 수 있다.O-GlcNAc 수정은 충돌 유도 분절(CID) 및 고에너지 충돌 분절(HCD)과 같은 충돌 유도 단편화 방법 하에서 실행되기 쉬우므로 격리된 이러한 방법은 O-GlcNAc 사이트 매핑에 쉽게 적용할 수 없다.HCD는 O-GlcNAcylation 상태를 결정하는 데 사용할 수 있는 N-acetylhexosamines의 파편 이온 특성을 생성한다.[39]HCD와의 사이트 매핑을 용이하게 하기 위해, β-제거에 이어 마이클 추가에 디티오트레이톨(BEMAD)을 사용하여 실험용 O-GlcNAc 수정을 보다 안정적인 질량 태그로 변환할 수 있다.O-GlcNAc의 BEMAD 매핑의 경우, 샘플은 반드시 인산염으로 처리되어야 하며 그렇지 않으면 인산염과 같은 다른 세린/트레오닌 후 수정사항이 검출될 수 있다.[40]ETD는 O-GlcNAc와 같은 변환 후 수정은 그대로 두고 ETD는 펩타이드 백본 파열을 일으키기 때문에 현장 매핑에 사용된다.[41]
전통적인 단백질 연구는 완전 스캔 질량 스펙트럼에서 가장 풍부한 종에 대해 탠덤 MS를 수행하며, 저유량 종의 완전한 특성화를 금지한다.표적 단백질학을 위한 한 가지 현대 전략은 O-GlcNAcylated 단백질에 태그를 붙이기 위해 동위원소 라벨(예: 디브로마이드)을 사용한다.이 방법은 알고리즘 검출이 가능하며, 이는 탠덤 MS.[42] Directed tandem MS와 표적 글리코펩타이드 할당으로 배열되어 O-GlcNAcylated 펩타이드 시퀀스를 식별할 수 있다.한 가지 예시 탐침은 바이오틴 친화도 태그, 산성 제거 가능 실란, 동위원소 재코딩 모티브 및 알킨으로 구성된다.[43][44][45]세린/트레오닌 잔류물이 1개만 있는 펩타이드에 대한 모호하지 않은 사이트 매핑이 가능하다.[46]
이 동위원소 대상 당단백질학(IsoTaG) 방법에 대한 일반적인 절차는 다음과 같다.
- O-GlcNAz를 단백질에 설치하기 위해 신진대사 라벨 O-GlcNAc
- 클릭 케미컬을 사용하여 O-GlcNAz에 IsoTaG 프로브 연결
- Streptavidin 구슬을 사용하여 태그가 붙은 단백질을 풍부하게 함
- 비수정 펩타이드 방출을 위한 트립신 비드를 트립신 처리
- 경산을 사용한 비드에서 동위원소 처리된 글리코펩타이드 클리브
- 동위원소 변환 글리코펩타이드에서 전체 스캔 질량 스펙트럼 획득
- 프로브에서 고유한 동위원소 서명을 탐지하는 알고리즘 적용
- 동위원소 재코딩된 종에 탠덤 MS를 수행하여 글리코펩타이드 아미노산 시퀀스 획득
- 확인된 시퀀스에 대한 단백질 데이터베이스 검색
차동위원소 라벨링을 이용한 O-GlcNAc의 정량적 프로파일링을 위한 다른 방법론이 개발되었다.[47]예시 탐침은 일반적으로 바이오틴 친화도 태그, 분해성 링커(산소 또는 광삭제 가능), 무겁거나 가벼운 동위원소 태그 및 알킨으로 구성된다.[48][49]
O-GlcNAc 조작 전략
다양한 화학적, 유전적 전략이 단백질 전체와 특정 단백질 둘 다로 O-GlcNAc를 조작하기 위해 개발되었다.
화학적 방법
작은 분자 억제제는 세포 내 또는 생체내 기능하는 OGT와[50][51] OGA[52][53] 모두에 대해 보고되었다.OGT 억제제는 O-GlcNAc의 전지구적 감소를 초래하는 반면, OGA 억제제는 O-GlcNAc의 전지구적 증가를 초래한다. 이러한 억제제는 특정 단백질에 대해 O-GlcNAc를 변조할 수 없다.
헥소아민 생합성 경로의 억제도 O-GlcNAc 수준을 감소시킬 수 있다.예를 들어 글루타민 아날로그 아자세린과 6-diazo-5-oxo-L-Norleucine(DON)은 GFAT를 억제할 수 있지만, 이러한 분자는 다른 경로에도 비특이적으로 영향을 미칠 수 있다.[54]
단백질 합성
표현된 단백질레이저는 O-GlcNAc로 변형된 단백질을 현장 고유 방식으로 준비하기 위해 사용되어 왔다.GlcNAc 수정 세린, 트레오닌 또는 시스테인의 고체상 펩타이드 합성을 위한 방법이 존재한다.[55][56]
유전적 방법
사이트 방향 돌연변이 유발
특정 잔류물에서 O-GlcNAc의 기능을 평가하기 위해 알라닌에 대한 O-GlcNAc 수정 세린 또는 트레오닌 잔류물의 현장 유도 돌연변이 유발물을 사용할 수 있다.알라닌의 사이드 체인은 메틸 그룹이고 따라서 O-GlcNAc 사이트 역할을 할 수 없기 때문에 이 돌연변이는 특정 잔류물에서 O-GlcNAc를 효과적으로 영구적으로 제거한다.세린/트레오닌 인산염은 카르복실산 사이드 체인을 음전하한 아스파테이트 또는 글루탐산염에 의해 모델링될 수 있지만, 20개의 정합 아미노산 중 O-GlcNAc의 특성을 충분히 재현하는 것은 없다.[57]트립토판은 O-GlcNAc보다 훨씬 소수성이 강하지만, 트립토판은 O-GlcNAc의 견고한 덩어리를 모방하기 위해 사용되어 왔다.[58][59]무타게네시스는 세린이 대체적으로 인산염 또는 O-GlcNAcylated인 경우, 알라닌 무타게네시스는 인산염과 O-GlcNAcylation의 가능성을 영구적으로 제거할 수 있다.
S-GlcNAc
질량 분광 분석 결과 S-GlcNAc는 사이스틴 잔류물에서 발견된 변환 후 수정으로 확인되었다.시험관내 실험에서 OGT는 S-GlcNAc 형성을 촉진할 수 있고 OGA는 S-GlcNAc를 가수 분해할 수 없다는 것을 입증했다.[60]이전의 보고서에서 OGA가 티오글리코시드를 가수 분해할 수 있다고 제안했지만, 이것은 아릴 티오글리코시드 파라-니트로페놀-S-GlcNAc에서만 입증되었다; 파라 니트로티오페놀은 시스틴 잔류물보다 더 활성화된 이탈 그룹이다.[61]최근 연구에서는 고체상 펩타이드 합성이나 현장 지향 돌연변이 유발에 의해 통합될 수 있는 O-GlcNAc의 효소학적 안정적 구조모델로서 S-GlcNAc의 사용을 뒷받침하고 있다.[62][57][55][63]
엔지니어링된 OGT
나노바디와 TPR-tracted OGT의 융합 구조는 세포에서 근접 유도 단백질 특이 O-GlcNAcylation을 허용한다.나노바디는 표적 단백질과 융합된 단백질 태그(예: GFP)로 향하거나, 나노바디는 내생 단백질로 향할 수 있다.예를 들어, C-단자 EPEA 시퀀스를 인식하는 나노바디는 OGT 효소 활성을 α-시뉴클레인으로 유도할 수 있다.[64]
O-GlcNAc의 기능
사멸
세포사멸의 한 형태인 세포사멸은 O-GlcNAc에 의해 규제되어야 한다고 제안되어 왔다.여러 암에서 O-GlcNAc 수치가 상승하여 사멸을 억제하는 것으로 보고되었다.[65][66]Caspase-3, Caspase-8, Caspase-9는 O-GlcNAc에 의해 수정된 것으로 보고되었다.Caspase-8은 갈라짐/활성화 현장 근처에서 수정된다. O-GlcNAc 수정은 카스파제-8 갈라짐과 장력 장애에 의한 활성화를 차단할 수 있다.5S-GlcNAc를 사용한 O-GlcNAc의 약리학적 저하는 캐스파아제 활성화를 가속화하고 티아멧-G를 사용한 O-GlcNAc의 약리학적 상승은 캐스파아제 활성화를 억제했다.[59]
후생유전학
작가들과 에라져스
유전학을 조절하는 단백질은 흔히 작가, 독자, 지우개로 분류되는데, 즉 후생유전적 수정을 설치하는 효소, 이러한 변형을 인식하는 단백질, 그리고 이러한 변형을 제거하는 효소로 분류된다.[67]현재까지 O-GlcNAc는 필자와 지우개 효소에서 확인되었다.O-GlcNAc는 PRC2의 촉매 메틸전달효소 소단위인 EZH2의 여러 위치에서 발견되며, PRC2 복합형성 이전에 EZH2를 안정화시키고 di-methyl전달효소 활성 및 tri-methyltransferase 활성을 규제하는 것으로 생각된다.[68][69]이산화지질소(TET1, TET2, TET3)의 10개 일레븐 번역(TET) 계열의 3개 멤버 모두 O-GlcNAc가 수정한 것으로 알려졌다.[70]O-GlcNAc는 TET3의 핵 수출을 유발해 핵에서 고갈시켜 효소 활성을 감소시킬 것을 제안했다.[71]HDAC1의 O-GlcNAcylation은 HDAC1의 인산화 활성화가 상승하는 것과 관련이 있다.[72]
히스톤 O-GlcNAcylation
염색질의 1차 단백질 성분인 히스톤 단백질이 O-GlcNAc에 의해 변형된 것으로 알려져 있다.[7]O-GlcNAc는 모든 코어 히스톤(H2A,[7] H2B,[7] H3, [73]H4[7])에서 식별되었다.히스톤에 대한 O-GlcNAc의 존재는 아세틸화[7], 일신화 등의 다른 히스톤 마크뿐만 아니라 유전자 전사에도 영향을 미치는 것으로 제안되었다.[74]TET2는 OGT의 TPR 영역과 상호 작용하고 히스톤에 OGT의 채용을 촉진하는 것으로 보고되었다.[75]이 상호작용은 H2B S112 O-GlcNAc와 연관되어 있으며, 이는 H2B K120 모뉴퀴티션과 연관된다.[74]AMPK를 통한 OGT T444의 인산화효소는 OGT-크롬화합성을 억제하고 H2B S112 O-GlcNAc를 다운규제하는 것으로 밝혀졌다.[76]
영양감지
헥소사민 생합성 경로의 제품인 UDP-GlcNAc는 OGT가 O-GlcNAc의 첨가를 촉진하기 위해 활용한다.이 경로는 아미노산, 탄수화물, 지방산, 뉴클레오티드를 포함한 다양한 대사물의 농도에 대한 정보를 통합한다.따라서 UDP-GlcNAC 수준은 세포 대사물 수준에 민감하다.OGT 활성도는 부분적으로 UDP-GlcNAc 농도에 의해 조절되며, 세포 영양 상태와 O-GlcNAc 간의 관계를 형성한다.[77]
포도당 결핍은 UDP-GlcNAc 수치의 하락과 O-GlcNAc의 초기 감소를 야기하지만, 역직관적으로 O-GlcNAc는 나중에 크게 상향 조정된다.이후 이러한 증가는 AMPK와 p38 MAPK 활성화에 의존하는 것으로 나타났으며, 이러한 효과는 부분적으로 OGT mRNA와 단백질 수준의 증가에 기인한다.[78]칼슘과 CaMKII에 의존하는 효과도 제시됐다.[79]활성화된 p38은 신경필라멘트 H를 포함한 특정 단백질 표적에 OGT를 모집할 수 있으며, 신경필라멘트 H의 O-GlcNAc 수정은 용해성을 강화한다.[78]포도당 결핍 동안 글리코겐 싱타아제는 O-GlcNAc에 의해 수정되어 활동을 억제한다.[80]
산화 응력
산화 스트레스에 대한 세포 반응과 관련된 전사 계수인 NRF2는 O-GlcNAc에 의해 간접적으로 조절되는 것으로 밝혀졌다.Cullin 3 의존성 E3 ubiquitin ligase 복합체의 어댑터 단백질인 KEAP1은 NRF2의 저하를 매개한다; 산화적 응력은 NRF2의 저하를 억제하는 KEAP1의 순응적 변화를 이끈다.O-GlcNAc와 산화 응력을 연결하는 NRF2의 효율적인 편재 및 후속 저하를 위해 S104에서 KEAP1의 O-GlcNAc 개조가 필요하다.포도당 결핍은 O-GlcNAc의 감소로 이어지고 NRF2의 저하를 감소시킨다.KEAP1 S104A 돌연변이를 발현하는 세포는 S104 O-GlcNAc를 제거할 때 더 높은 NRF2 수준과 일치하는 에라스틴 유도 강박증에 내성이 있다.[81]
높은 O-GlcNAC 수치는 중요한 세포 항산화 물질인 간 글루타티온의 합성이 감소하는 것과 관련이 있다.아세트아미노펜 과다복용은 강한 산화 대사물 NAPQI가 간에 축적되어 글루타티온에 의해 해독된다.생쥐의 경우 OGT 녹아웃은 아세트아미노펜으로 인한 간손상에 대한 보호효과가 있는 반면 티아멧-G로 OGA 억제를 하면 아세트아미노펜으로 인한 간손상이 악화된다.[82]
단백질집합
O-GlcNAc는 단백질의 집적을 늦추는 것으로 밝혀졌지만, 이러한 현상의 일반성은 알려져 있지 않다.
고체상 펩타이드 합성은 T72에서 O-GlcNAc 수정으로 전신 α-시뉴클레인을 준비하기 위해 사용되었다.티오플라빈 T 집적 분석과 전송 전자 현미경 검사는 이 변형된 α-시뉴클레인이 쉽게 집적체를 형성하지 않는다는 것을 증명했다.[56]
OGA 억제제를 사용한 JNPL3 tau transgenic 마우스를 치료하면 미세관류 관련 단백질 tau O-GlcNAcylation이 증가하는 것으로 나타났다.뇌시스템의 면역역학화학 분석 결과 신경섬유 엉킴의 형성이 감소했다는 것이 밝혀졌다.재조합형 O-GlcNAcylated tau는 시험관내 티오플라빈 S 집계 분석에서 수정되지 않은 타우보다 느린 집계로 나타났다.재조합된 O-GlcNAcylated TAB1 구조 대 수정되지 않은 형태에 대해 유사한 결과가 도출되었다.[83]
단백질인산화
크로스스토크
알려진 많은 인산화 사이트와 O-GlcNAcylation 사이트는 서로 가까이 있거나 겹친다.[46]단백질 O-GlcNAcylation과 인산화 모두 세린과 세로닌 잔류물에서 발생하므로 이러한 변환 후 수정은 서로 조절할 수 있다.예를 들어, CKIiα에서 S347 O-GlcNAc는 T344 인산염에 대항하는 것으로 나타났다.[55]상호 억제, 즉 O-GlcNAcylation의 인산화 억제와 인산화 O-GlcNAcylation의 O-GlcNAcylation 억제, 뮤린 에스트로겐 수용체 β,[84] RNA Pol II,[85] p53,[86][87] CaMKIV,[88] p65,[89] β-catenin,[90] α-synuclein을 포함한 다른 단백질에서 관찰되었다.[56]이러한 두 변환 후 수정들 사이에서도 양의 협력성이 관찰되었다. 즉, 인산염은 O-GlcNAcylation을 유도하거나 O-GlcNAcylation을 유도한다.이것은 MeCP2와[28] HDAC1에서 입증되었다.[72]다른 단백질에서는 코필린, 인산화, O-GlcNAcylation 등이 서로 독립적으로 발생하는 것으로 보인다.[91]
어떤 경우에는 O-GlcNAcylation을 변형하여 인산화에 대한 다운스트림 효과를 가지는 치료 전략이 조사되고 있다.예를 들어 타우 O-GlcNAcylation을 상승시키면 병리학적 타우 하이퍼인산화를 억제하여 치료적 이익을 제공할 수 있다.[92]
O-GlcNAc는 인산화 외에도 리신 아세틸화[89], 단당화 등 다른 변환 후 변경에 영향을 미치는 것으로 밝혀졌다.[74]
키나세스
단백질 키나아제는 세린과 트레오닌 잔류물의 인산화 작용을 담당하는 효소다.O-GlcNAc는 100개 이상의 키노메 키나제(인간 키노메의 약 20%)에서 확인되었으며, 이러한 수정은 종종 키나제 활성 또는 키나제 기판 범위의 변경과 관련이 있다.[93]
키나아제가 O-GlcNAc에 의해 직접 규제되고 있다는 최초의 보고서는 2009년에 발표되었다.CaMKIV는 여러 사이트에서 글리코실화되지만 S189가 주요 사이트로 확인되었다.S189A 돌연변이는 CaMKIV T200 인산염에 의해 더 쉽게 활성화되었으며, S189의 O-GlcNAc가 CaMKIV 활동을 억제한다는 것을 암시한다.동질학 모델링은 S189 O-GlcNAc가 ATP 바인딩을 방해할 수 있다는 것을 보여주었다.[88]
AMPK and OGT are known to modify each other, i.e., AMPK phosphorylates OGT and OGT O-GlcNAcylates AMPK. AMPK activation by AICA ribonucleotide is associated with nuclear localization of OGT in differentiated C2C12 mouse skeletal muscle myotubes, resulting in increased nuclear O-GlcNAc.이 효과는 증식세포와 미분화 근막세포에서 관찰되지 않았다.[94]OGT T444의 AMPK 인산화효소는 염색질과의 OGT 연관성을 차단하고 H2B S112 O-GlcNAc를 감소시키는 것으로 밝혀졌다.[76]마우스 아디포스 조직에서 포도당 플럭스를 헥소사민 생합성 경로로 제어하는 효소인 GFAT의 과도한 억압이 AMPK 활성화와 다운스트림 ACC 억제 및 지방산 산화를 유발하는 것으로 나타났다.배양된 3T3L1 아디프세포에서의 글루코사민 치료도 비슷한 효과를 보였다.[95]다양한 연구는 OGA 억제 또한glucosamine 치료로 AMPK activation,[76]upregulating O-GlcNAc을 억제하고 AMPK activation,[94]송신용 간선 억제 작동을 막아 보도했다 O-GlcNAc과 AMPK 사이의 정확한 관계는 완전히 활성 AMPK,[95]과 송신용 간선 링 AMPK을 활성화시키고,[96]이 결과는 a를 제안한다 설명되지 않았습니다dditional indirAMPK 경로와 O-GlcNAc 또는 셀 유형별 효과 사이의 ect 통신.
CKIIα 기질 인식은 S347 O-GlcNAcylation에 따라 변경되는 것으로 나타났다.[55]
인산염
단백질인산효소 1 서브유닛 PP1β 및 PP1γ는 OGT와 함께 기능 복합체를 형성하는 것으로 나타났다.합성 인산염은 OGT 면역복제효소에 의해 비인산화 및 O-GlcNAcylated될 수 있었다.이 단지는 인산염 개조를 O-GlcNAc 개조로 대체해 '인양 복합체'로 일컬어 왔다.[97]
MYPT1은 OGT와 콤플렉스를 형성하고 그 자체가 O-GlcNAcylated인 또 다른 단백질 인산염 소단위다.MYPT1은 OGT를 특정 기판으로 유도하는 역할을 하는 것으로 보인다.[98]
단백질-단백질 상호작용
단백질의 O-GlcNAcylation은 그것의 상호작용을 바꿀 수 있다.O-GlcNAc는 높은 친수성을 가지고 있기 때문에, 그것의 존재는 소수성 단백질과 단백질 상호작용을 방해할 수 있다.예를 들어 O-GlcNAc는 TAF110과의II Sp1 상호작용을 방해하고 [99]O-GlcNAc는 TAF130II 및 CRTC와의 CREB 상호작용을 방해한다.[100][101]
일부 연구는 또한 O-GlcNAc에 의해 단백질과 단백질 상호작용이 유도되는 사례도 확인했다.O-GlcNAc에 의해 유도된 단백질-단백질 상호작용을 식별하기 위해 디아지린을 함유한 O-GlcNDAz를 사용한 대사 라벨링이 적용되었다.[32]O-GlcNAc, α-enolase, EBP1, 14-3-3에 대한 컨센서스 시퀀스에 따라 대략적으로 미끼 글리코펩타이드(glycopeptide)[102]를 사용하는 것이 잠재적 O-GlcNAc 판독기로 확인되었다. X-ray 결정학에서 14-3-3은 또한 인지된 인광합체 홈을 통해 O-GlcNAc를 인식했다.hsp70은 또한 O-GlcNAc를 인정하는 강연의 역할을 하도록 제안되었다.[103]O-GlcNAc는 α-catenin과 β-catenin의 상호작용에 역할을 하는 것으로 제안되었다.[90]
단백질 안정성 및 분해
동시 변환 O-GlcNAc는 Sp1과 Nup62에서 확인되었다.이 수정은 동시 전이 유비쿼터스화를 억제하여 초기 폴리펩타이드들을 단백질 분해로부터 보호한다.O-GlcNAc가 전신 Sp1에 미치는 유사한 보호 효과가 관찰되었다.이 패턴이 보편적인지 아니면 특정 단백질에만 적용되는지는 알 수 없다.[13]
단백질 인산화효소는 종종 후속 분해의 표시로 사용된다.종양 억제기 단백질 p53은 T155의 COP9 신호상 매개 인산염에 의한 단백질 분해의 대상이다.p53 S149의 O-GlcNAcylation은 T155 인산화 감소 및 p53의 분해로부터 보호와 관련이 있다.[87]β-catenin O-GlcNAcylation은 β-catenin의 분해 신호를 보내는 T41 인산염과 경쟁하여 단백질을 안정화시킨다.[90]
26S 프로테아솜의 Rpt2 ATPase 하위 유닛의 O-GlcNAcylation은 프로테아솜 활동을 억제하는 것으로 나타났다.다양한 펩타이드 시퀀스를 테스트한 결과, 이러한 변경으로 소수성 펩타이드의 단백질 분해 속도가 느려지고 친수성 펩타이드의 분해는 영향을 받지 않는 것으로 나타났다.[104]이러한 수정은 cAMP 의존성 단백질 키나아제에 의한 Rpt6 인산화 같은 프로테아솜을 활성화하는 다른 경로를 억제하는 것으로 나타났다.[105]
OGA-S는 지질 방울에 국소화되며 지질 방울 표면 단백질의 리모델링을 촉진하기 위해 프로테아솜을 국소적으로 활성화할 것을 제안했다.[106]
응력반응
다양한 세포 스트레스 자극은 O-GlcNAc의 변화와 연관되어 왔다.과산화수소 처리, 코발트()II) 염화물, UVB 빛, 에탄올, 염화나트륨, 열 쇼크, 비소나트륨 모두 O-GlcNAc가 상승한다.OGT의 녹아웃은 세포를 열응력에 민감하게 만든다.상승된 O-GlcNAc는 Hsp40 및 Hsp70의 표현과 연관되어 있다.[107]
치료 관련성
알츠하이머병
많은 연구들이 타우의 이상 인산화 현상을 알츠하이머병의 특징으로 밝혀냈다.[108]소 타우의 O-GlcNAcylation은 1996년에 처음으로 특징지어졌다.[109]2004년의 후속 보고서는 인간의 두뇌 tau도 O-GlcNAc에 의해 변형된다는 것을 증명했다.타우의 O-GlcNAcylation은 OGT가 부족한 생쥐의 뇌에서 관찰된 타우의 초인산화로 타우인산화를 조절하기 위해 입증되었으며,[110] 이는 신경세동성 엉클의 형성과 관련이 있다.뇌 시료 분석 결과 알츠하이머병에서 단백질 O-GlcNAcylation이 손상되고 쌍체 나선 조각-tau가 기존 O-GlcNAc 검출방법에 의해 인식되지 않아 병리학적 tau가 제어 뇌 시료에서 격리된 tau에 비해 O-GlcNAcylation이 손상된 것으로 나타났다.타우 O-GlcNAcylation의 상승은 타우인산화 감소를 위한 치료 전략으로 제안되었다.[86]
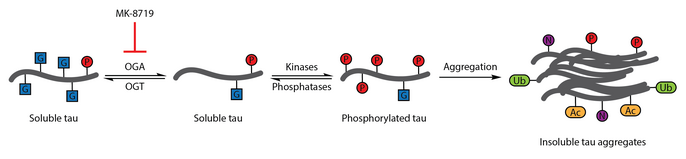
이 치료 가설을 시험하기 위해 선택적이고 혈중 뇌 장벽이 가능한 OGA 억제제인 티아멧-G가 개발되었다.티아멧-G 치료는 건강한 스프래그-다울리 쥐에서 타우 O-GlcNAcylation을 증가시키고 세포 배양과 생체내 타우인산화 억제를 할 수 있었다.[53]후속 연구는 티아멧-G 치료법이 JNPL3 tau 유전자 변형 마우스 모델에서도 타우 O-GlcNAcylation을 증가시켰다는 것을 보여주었다.이 모델에서 타우인산화술은 신경세동성 엉클의 수가 감소하고 운동 신경세포 손실의 속도가 느린 것이 관찰되었지만 티아메트-G 치료의 영향을 크게 받지 않았다.또한, 타우의 O-GlcNAcylation은 시험관내 타우 집적이 느리게 나타나는 것으로 파악되었다.[83]
MK-8719에 의한 OGA 억제제는 알츠하이머병과 진행성 초핵성 마비를 포함한 다른 치료법의 잠재적 치료 전략으로서 임상시험에서 연구되고 있다.[92][111][112]
암
O-GlcNAc의 조절장애는 암세포 증식과 종양 성장과 관련이 있다.
S529에서 PFK1 글리콜리틱 효소의 O-GlcNAcylation은 PFK1 효소 활성을 억제하여 글리콜리틱 플럭스를 감소시키고 포도당을 펜토오스 인산염 경로로 리디렉션하는 것으로 밝혀졌다.구조 모델링과 생화학적 실험은 S529의 O-GlcNAc가 과당 2,6비스포스포네이트와 올리고머를 활성 형태로 만들어 PFK1 알로스테릭 활성화를 억제할 수 있음을 시사했다.생쥐 모델에서 PFK1 S529A 돌연변이를 발현하는 세포를 주입한 생쥐는 PFK1 야생형을 발현하는 세포를 주입한 생쥐보다 종양 성장률이 낮았다.또한 OGT 과다압박은 후자 시스템에서 종양 성장을 강화했지만 돌연변이 PFK1을 사용하는 시스템에는 큰 영향을 미치지 않았다.저산소증은 PFK1 S529 O-GlcNAc를 유도하고 펜토오스 인산염 경로를 통해 플럭스를 증가시켜 글루타티온 수치를 유지하고 활성산소 종을 해독하는 NADPH를 더 많이 생성시켜 암세포에 성장우위를 부여한다.PFK1은 사람의 가슴과 폐종양 조직에서 글리코실린 처리된 것으로 밝혀졌다.[113]OGT는 또한 HIF-1α를 긍정적으로 조절하는 것으로 보고되었다. HIF-1α는 일반적으로 α-케토글루타레이트(α-케토글루타레이트)를 공동하중으로 이용하는 프롤릴 하이드록실라제(prolyl hydroxylase)에 의해 노르목성 조건에서 분해된다.OGT는 α-케토글루타레이트 레벨을 억제하여 pVHL에 의한 단백질 분해로부터 HIF-1α를 보호하고 에어로빅 글리콜리분해를 촉진한다.PFK1에 대한 이전 연구와 대조적으로, 이 연구는 두 연구가 O-GlcNAc 수준이 펜토오스 인산염 경로를 통한 플럭스와 확실히 연관되어 있다는 것을 발견하는데 일관하지만 OGT 또는 O-GlcNAc 상향 조정 PFK1을 찾는다는 것을 발견했다.이 연구는 또한 O-GlcNAc의 감소는 ER 스트레스에 의한 사멸을 통해 암세포를 선택적으로 죽였다는 것을 발견했다.[65]
인간 췌장관선상피종(PDAC) 세포 라인은 인간 췌장관 상피세포보다 O-GlcNAc 수치가 높다.PDAC 셀은 OGT 녹다운이 PDAC 셀 증식을 선택적으로 억제(OGT 녹다운이 HPDE 셀 증식에 유의한 영향을 미치지 않음)하고, 5S-GlcNAc를 이용한 OGT 억제도 동일한 결과를 보여 생존을 위해 O-GlcNAc에 어느 정도 의존한다.PDAC 세포의 하이퍼 O-GlcNAcylation은 항중독성으로 나타나 카스파제-3와 카스파제-9의 갈라짐과 활성화를 억제하였다.Numerous sites on the p65 subunit of NF-κB were found to be modified by O-GlcNAc in a dynamic manner; O-GlcNAc at p65 T305 and S319 in turn positively regulate other modifications associated with NF-κB activation such as p300-mediated K310 acetylation and IKK-mediated S536 phosphorylation.이러한 결과는 NF-162B가 췌장암에서 O-GlcNAc에 의해 구성적으로 활성화되었음을 시사했다.[66][89]
다양한 유방암 세포 라인에서 EZH2의 OGT 안정화는 종양 억제기 유전자의 발현을 억제하는 것으로 밝혀졌다.[68]간세포암 모델에서 O-GlcNAc는 HDAC1의 인산화 활성과 연관되어 있으며, 이는 세포주기 조절기 p21과Waf1/Cip1 세포운동 조절기 E-cadherin의 발현을 조절한다.[72]
OGT는 SREBP-1을 안정화하고 유방암 세포 라인의 지방유전화를 활성화하는 것으로 밝혀졌다.이러한 안정화는 프로테아솜과 AMPK에 의존했다. OGT 녹다운은 핵 SREBP-1을 감소시켰지만 MG132를 통한 프로테아솜 억제가 이 효과를 막았다.OGT 녹다운은 또한 SREBP-1과 E3 유비퀴티틴 리가제 FBW7 사이의 상호작용을 증가시켰다.AMPK는 OGT 녹다운 시 T172 인산염, AMPK 인산염 SREBP-1 S372에 의해 활성화되어 갈라짐과 성숙을 억제한다.OGT 녹다운은 AMPK-null 셀 라인의 SREBP-1 레벨에 대한 영향을 감소시켰다.마우스 모델에서 OGT 녹다운은 종양 성장을 억제했지만 SREBP-1 과다압박은 부분적으로 이 효과를 구했다.[96]이러한 결과는 OGT 녹다운/침입이 AMPK T172 인산화를 억제하고 지방 발생을 증가시켰다는 이전 연구와 대조적이다.[76]
유방과 전립선 암세포 라인에서 높은 수준의 OGT와 O-GlcNAc는 혈관신생, 침공, 전이 등과 같은 질병 진행과 관련된 과정과 체외 및 체내 모두에서 연관되어 왔다.OGT 녹다운 또는 억제가 전사인자 FoxM1을 다운규제하고 셀주기억제제Kip1 p27(E3 ubiquititin ligase 성분 Skp2의 FoxM1 의존적 표현에 의해 규제됨)을 상향조정하여 G1 세포주기억제를 유발하는 것으로 밝혀졌다.이것은 FoxM1 돌연변이의 표현이 OGT 녹다운의 효과를 구조했기 때문에 FoxM1의 단백질 분해에 의존하는 것으로 보였다.FoxM1은 O-GlcNAc에 의해 직접 수정되지 않는 것으로 밝혀져 FoxM1 규제기관의 하이퍼 O-GlcNAcylation은 FoxM1의 저하를 저해한다는 것을 시사했다.Targeting OGT also lowered levels of FoxM1-regulated proteins associated with cancer invasion and metastasis (MMP-2 & MMP-9), and angiogenesis (VEGF).[114][115] O-GlcNAc modification of cofilin S108 has also been reported to be important for breast cancer cell invasion by regulating cofilin subcellular localization in invadopodia.[91]
당뇨병
높은 O-GlcNAc는 당뇨병과 연관되어 있다.
췌장 β 세포는 혈당 수치를 조절하기 위해 인슐린을 합성하고 분비한다.한 연구는 OGA의 streptozotocin과 억제 글루코사민 치료에 이어β 세포에서 O-GlcNAc 축적과 세포에;[116]은 차후의 연구는 streptozotocin의galactose-based 아날로그 OGA을 억제할 수 없지만 아직도 세포 자살사에서 제안하다는 것을 발견은apoptotic 효과 streptozotocin an이 전무하다는OGA 억제 때문에 [117]직접 발생
O-GlcNAc는 인슐린 신호를 약화시킬 것을 제안했다.3T3-L1 아디포모세포에서 PUGNAc에 의한 OGA 억제에서는 인슐린 매개 포도당 흡수를 억제했다.PUGNAc 치료는 인슐린 자극 Akt T308 인산화 및 다운스트림 GSK3β S9 인산화도 억제했다.[118]이후 연구에서 COS-7 세포의 인슐린 자극으로 OGT가 혈장 막으로 국소화되었다.워트만닌을 사용한 PI3K의 억제는 이 효과를 역전시켜 인산염(3,4,5)-삼인산염에 대한 의존성을 시사했다.세포를 높은 포도당 조건이나 PUGNAc 치료로 O-GlcNAc 수준을 증가시킴으로써 Akt T308 및 Akt 활성의 인슐린 자극 인산화를 억제하였다.인슐린 신호 감쇠와 관련된 S307 및 S632/S635의 IRS1 인산화가 강화되었다.OGT의 아데노바이러스 전달을 가진 생쥐를 대상으로 한 후속 실험에서는 OGT 과압축이 체내 인슐린 신호를 부정적으로 조절하는 것으로 나타났다.PI3K의 β-카테닌,[118] IR-β, IRS1, Akt,[119] PDK1 및 p110α 서브유닛을 포함한 인슐린 신호 경로의 많은 성분이 O-GlcNAc에 의해 직접 수정된 것으로 밝혀졌다.인슐린 신호는 또한 OGT tyrosine phosphorylation과 OGT 활성화로 이어져 O-GlcNAc 수치가 증가하는 것으로 보고되었다.[120]
PUGNAc는 Lysosomal β-헥소사미디제(Rysosomal β-헥소사미디제)도 억제하므로 OGA 선택 억제제 NButGT는 3T3-L1 아디프세포에서 O-GlcNAc와 인슐린 신호의 관계를 더욱 심도 있게 조사하기 위해 개발되었다.이 연구는 또한 PUGNAc가 인슐린 신호의 손상을 초래했지만 NBut은GT는 Akt T308의 인산화 변화에 의해 측정되지 않았으며, PUGNAc로 관측된 효과는 OGA 억제 외에 오프타깃 효과 때문일 수 있음을 시사했다.[121]
파킨슨병
파킨슨병은 α-시뉴클레인의 집적과 관련이 있다.[122]α-시뉴클레인의 O-GlcNAc변형이 집적을 억제하는 것으로 밝혀지면서 α-시뉴클레인의 O-GlcNAc를 높이는 것이 파킨슨병을 치료하기 위한 치료전략으로 모색되고 있다.[56][123]
전염병
세균
그램 음성 박테리아 외막의 주요 성분인 LPS(LopolySaccharide)를 이용한 대식세포 처리로 세포와 마우스 모델에서 O-GlcNAc가 상승한다.감염 기간 동안 세포질 OGT는 디-S-니트로사이슬링되어 활성화되었다.DON으로 O-GlcNAc를 억제함으로써 O-GlcNAclation과 NF-κB의 핵번역을 억제하고, 유도성 질산화물 싱타아제 및 IL-1β 생산의 다운스트림 유도를 억제하였다.DON 치료는 또한 LPS 치료 중 세포 생존을 향상시켰다.[124]
바이러스
O-GlcNAc는 인플루엔자 A 바이러스(IAV)로 인한 사이토카인 폭풍에 연루됐다.구체적으로, 인터페론 규제 인자-5 (IRF5)에 대한 S430의 O-GlcNAcylation은 세포 및 마우스 모델에서 TNF 수용체 관련 인자 6 (TRAF6)과의 상호작용을 촉진하는 것으로 나타났다.TRAF6는 IRF5 활동과 후속 사이토카인 생산에 필요한 IRF5의 K63 연계 유비쿼터스화를 매개한다.임상 검체 분석 결과 IAV 감염 환자의 혈당 수치가 건강한 개인에 비해 높아진 것으로 나타났다.IAV에 감염된 환자의 경우 혈당 수치는 IL-6 및 IL-8 수준과 확실히 상관관계가 있다.IRF5의 O-GlcNAcylation은 IAV 감염 환자의 말초혈액 단핵세포에서도 비교적 높았다.[125]
기타 응용 프로그램
펩타이드 치료제 등 펩타이드 치료제는 특이성과 효능이 높아 매력적이지만 혈청 프로테아제에 의한 분해로 약동학적 프로파일이 좋지 않은 경우가 많다.[126]O-GlcNAc는 일반적으로 세포내 단백질과 관련이 있지만, O-GlcNAc에 의해 수정된 공학적 펩타이드 치료제는 마우스 모델에서 혈청 안정성을 강화했으며 각각의 변형되지 않은 펩타이드와 유사한 구조와 활동을 가지고 있는 것으로 밝혀졌다.이 방법은 GLP-1과 PTH 펩타이드 엔지니어에게 적용되었다.[127]
참고 항목
참조
- ^ a b Zeidan, Quira; Hart, Gerald W. (2010-01-01). "The intersections between O-GlcNAcylation and phosphorylation: implications for multiple signaling pathways". Journal of Cell Science. 123 (1): 13–22. doi:10.1242/jcs.053678. ISSN 0021-9533. PMC 2794709. PMID 20016062.
- ^ Dias, Wagner B.; Cheung, Win D.; Hart, Gerald W. (2012-06-01). "O-GlcNAcylation of Kinases". Biochemical and Biophysical Research Communications. 422 (2): 224–228. doi:10.1016/j.bbrc.2012.04.124. ISSN 0006-291X. PMC 3387735. PMID 22564745.
- ^ Haltiwanger, RS; Holt, GD; Hart, GW (1990-02-15). "Enzymatic Addition of O-GlcNAc to Nuclear and Cytoplasmic Proteins. Identification of a Uridine diphospho-N-acetylglucosamine:peptide beta-N-acetylglucosaminyltransferase". Journal of Biological Chemistry. 265 (5): 2563–8. doi:10.1016/S0021-9258(19)39838-2. PMID 2137449.
- ^ Wulff-Fuentes E, Berendt RR, Massman L, Danner L, Malard F, Vora J, Kahsay R, Olivier-Van Stichelen S (January 2021). "The human O-GlcNAcome database and meta-analysis". Scientific Data. 8 (1): 25. Bibcode:2021NatSD...8...25W. doi:10.1038/s41597-021-00810-4. PMC 7820439. PMID 33479245.
- ^ Ma, Junfeng; Hart, Gerald W (2014-03-05). "O-GlcNAc profiling: from proteins to proteomes". Clinical Proteomics. 11 (1): 8. doi:10.1186/1559-0275-11-8. ISSN 1542-6416. PMC 4015695. PMID 24593906.
- ^ Kelly, WG; Dahmus, ME; Hart, GW (1993-05-15). "RNA Polymerase II Is a Glycoprotein. Modification of the COOH-terminal Domain by O-GlcNAc". Journal of Biological Chemistry. 268 (14): 10416–24. doi:10.1016/S0021-9258(18)82216-5. PMID 8486697.
- ^ a b c d e f Sakabe, K; Wang, Z; Hart, GW (2010-11-16). "Beta-N-acetylglucosamine (O-GlcNAc) Is Part of the Histone Code". Proceedings of the National Academy of Sciences of the United States of America. 107 (46): 19915–20. Bibcode:2010PNAS..10719915S. doi:10.1073/pnas.1009023107. PMC 2993388. PMID 21045127.
- ^ Levine, Z; Walker, S (2016-06-02). "The Biochemistry of O-GlcNAc Transferase: Which Functions Make It Essential in Mammalian Cells?". Annual Review of Biochemistry. 85: 631–57. doi:10.1146/annurev-biochem-060713-035344. PMID 27294441.
- ^ Ong, Qunxiang; Han, Weiping; Yang, Xiaoyong (2018-10-16). "O-GlcNAc as an Integrator of Signaling Pathways". Frontiers in Endocrinology. 9: 599. doi:10.3389/fendo.2018.00599. ISSN 1664-2392. PMC 6234912. PMID 30464755.
- ^ Hart, Gerald W.; Slawson, Chad; Ramirez-Correa, Genaro; Lagerlof, Olof (2011-06-07). "Cross Talk Between O-GlcNAcylation and Phosphorylation: Roles in Signaling, Transcription, and Chronic Disease". Annual Review of Biochemistry. 80: 825–858. doi:10.1146/annurev-biochem-060608-102511. ISSN 0066-4154. PMC 3294376. PMID 21391816.
- ^ Torres, CR; Hart, GW (1984-03-10). "Topography and Polypeptide Distribution of Terminal N-acetylglucosamine Residues on the Surfaces of Intact Lymphocytes. Evidence for O-linked GlcNAc". Journal of Biological Chemistry. 259 (5): 3308–17. doi:10.1016/S0021-9258(17)43295-9. PMID 6421821.
- ^ a b Shen, David L.; Gloster, Tracey M.; Yuzwa, Scott A.; Vocadlo, David J. (2012-05-04). "Insights into O-Linked N-Acetylglucosamine (O-GlcNAc) Processing and Dynamics through Kinetic Analysis of O-GlcNAc Transferase and O-GlcNAcase Activity on Protein Substrates". Journal of Biological Chemistry. 287 (19): 15395–15408. doi:10.1074/jbc.M111.310664. ISSN 0021-9258. PMC 3346082. PMID 22311971.
- ^ a b Zhu, Y; Liu, TW; Cecioni, S; Eskandari, R; Zandberg, WF; Vocadlo, DJ (May 2015). "O-GlcNAc Occurs Cotranslationally to Stabilize Nascent Polypeptide Chains". Nature Chemical Biology. 11 (5): 319–25. doi:10.1038/nchembio.1774. PMID 25774941.
- ^ a b c Lazarus, MB; Nam, Y; Jiang, J; Sliz, P; Walker, S (2011-01-27). "Structure of Human O-GlcNAc Transferase and Its Complex With a Peptide Substrate". Nature. 469 (7331): 564–7. Bibcode:2011Natur.469..564L. doi:10.1038/nature09638. PMC 3064491. PMID 21240259.
- ^ Macauley, MS; Whitworth, GE; Debowski, AW; Chin, D; Vocadlo, DJ (2005-07-08). "O-GlcNAcase Uses Substrate-Assisted Catalysis: Kinetic Analysis and Development of Highly Selective Mechanism-Inspired Inhibitors". Journal of Biological Chemistry. 280 (27): 25313–22. doi:10.1074/jbc.M413819200. PMID 15795231.
- ^ Roth, Christian; Chan, Sherry; Offen, Wendy A; Hemsworth, Glyn R; Willems, Lianne I; King, Dustin T; Varghese, Vimal; Britton, Robert; Vocadlo, David J; Davies, Gideon J (June 2017). "Structural and functional insight into human O-GlcNAcase". Nature Chemical Biology. 13 (6): 610–612. doi:10.1038/nchembio.2358. ISSN 1552-4450. PMC 5438047. PMID 28346405.
- ^ Elsen, NL; Patel, SB; Ford, RE; Hall, DL; Hess, F; Kandula, H; Kornienko, M; Reid, J; Selnick, H (June 2017). "Insights Into Activity and Inhibition From the Crystal Structure of Human O-GlcNAcase". Nature Chemical Biology. 13 (6): 613–615. doi:10.1038/nchembio.2357. PMID 28346407.
- ^ Joiner, CM; Levine, ZG; Aonbangkhen, C; Woo, CM; Walker, S (2019-08-21). "Aspartate Residues Far From the Active Site Drive O-GlcNAc Transferase Substrate Selection". Journal of the American Chemical Society. 141 (33): 12974–12978. doi:10.1021/jacs.9b06061. PMC 6849375. PMID 31373491.
- ^ Levine, ZG; Fan, C; Melicher, MS; Orman, M; Benjamin, T; Walker, S (2018-03-14). "O-GlcNAc Transferase Recognizes Protein Substrates Using an Asparagine Ladder in the Tetratricopeptide Repeat (TPR) Superhelix". Journal of the American Chemical Society. 140 (10): 3510–3513. doi:10.1021/jacs.7b13546. PMC 5937710. PMID 29485866.
- ^ a b S, Pathak; J, Alonso; M, Schimpl; K, Rafie; De, Blair; Vs, Borodkin; O, Albarbarawi; Dmf, van Aalten (Sep 2015). "The Active Site of O-GlcNAc Transferase Imposes Constraints on Substrate Sequence". Nature Structural & Molecular Biology. 22 (9): 744–750. doi:10.1038/nsmb.3063. PMC 4979681. PMID 26237509.
- ^ Cheung, WD; Sakabe, K; Housley, MP; Dias, WB; Hart, GW (2008-12-05). "O-linked beta-N-acetylglucosaminyltransferase Substrate Specificity Is Regulated by Myosin Phosphatase Targeting and Other Interacting Proteins". Journal of Biological Chemistry. 283 (49): 33935–41. doi:10.1074/jbc.M806199200. PMC 2590692. PMID 18840611.
- ^ Zachara, Natasha E.; Vosseller, Keith; Hart, Gerald W. (November 2011). "Detection and Analysis of Proteins Modified by O-Linked N-Acetylglucosamine". Current Protocols in Protein Science. CHAPTER: Unit12.8. doi:10.1002/0471140864.ps1208s66. ISSN 1934-3655. PMC 3349994. PMID 22045558.
- ^ Snow, C. M.; Senior, A.; Gerace, L. (1987-05-01). "Monoclonal antibodies identify a group of nuclear pore complex glycoproteins". The Journal of Cell Biology. 104 (5): 1143–1156. doi:10.1083/jcb.104.5.1143. ISSN 0021-9525. PMC 2114474. PMID 2437126.
- ^ Comer, FI; Vosseller, K; Wells, L; Accavitti, MA; Hart, GW (2001-06-15). "Characterization of a Mouse Monoclonal Antibody Specific for O-linked N-acetylglucosamine". Analytical Biochemistry. 293 (2): 169–77. doi:10.1006/abio.2001.5132. PMID 11399029.
- ^ Teo, CF; Ingale, S; Wolfert, MA; Elsayed, GA; Nöt, LG; Chatham, JC; Wells, L; Boons, GJ (May 2010). "Glycopeptide-specific Monoclonal Antibodies Suggest New Roles for O-GlcNAc". Nature Chemical Biology. 6 (5): 338–43. doi:10.1038/nchembio.338. PMC 2857662. PMID 20305658.
- ^ a b DJ, Vocadlo; HC, Hang; Ej, Kim; Ja, Hanover; Cr, Bertozzi (2003-08-05). "A Chemical Approach for Identifying O-GlcNAc-modified Proteins in Cells". Proceedings of the National Academy of Sciences of the United States of America. 100 (16): 9116–21. Bibcode:2003PNAS..100.9116V. doi:10.1073/pnas.1632821100. PMC 171382. PMID 12874386.
- ^ a b c Clark, PM; Dweck, JF; Mason, DE; Hart, CR; Buck, SB; Peters, EC; Agnew, BJ; Hsieh-Wilson, LC (2008-09-03). "Direct In-Gel Fluorescence Detection and Cellular Imaging of O-GlcNAc-modified Proteins". Journal of the American Chemical Society. 130 (35): 11576–7. doi:10.1021/ja8030467. PMC 2649877. PMID 18683930.
- ^ a b c Rexach, JE; Rogers, CJ; Yu, SH; Tao, J; Sun, YE; Hsieh-Wilson, LC (September 2010). "Quantification of O-glycosylation Stoichiometry and Dynamics Using Resolvable Mass Tags". Nature Chemical Biology. 6 (9): 645–51. doi:10.1038/nchembio.412. PMC 2924450. PMID 20657584.
- ^ a b Walter, LA; Batt, AR; Darabedian, N; Zaro, BW; Pratt, MR (2018-09-17). "Azide- And Alkyne-Bearing Metabolic Chemical Reporters of Glycosylation Show Structure-Dependent Feedback Inhibition of the Hexosamine Biosynthetic Pathway". ChemBioChem. 19 (18): 1918–1921. doi:10.1002/cbic.201800280. PMC 6261355. PMID 29979493.
- ^ Boyce, M; Carrico, IS; Ganguli, AS; Yu, SH; Hangauer, MJ; Hubbard, SC; Kohler, JJ; Bertozzi, CR (2011-02-22). "Metabolic Cross-Talk Allows Labeling of O-linked beta-N-acetylglucosamine-modified Proteins via the N-acetylgalactosamine Salvage Pathway". Proceedings of the National Academy of Sciences of the United States of America. 108 (8): 3141–6. Bibcode:2011PNAS..108.3141B. doi:10.1073/pnas.1010045108. PMC 3044403. PMID 21300897.
- ^ Tan, HY; Eskandari, R; Shen, D; Zhu, Y; Liu, TW; Willems, LI; Alteen, MG; Madden, Z; Vocadlo, DJ (2018-11-14). "Direct One-Step Fluorescent Labeling of O-GlcNAc-Modified Proteins in Live Cells Using Metabolic Intermediates". Journal of the American Chemical Society. 140 (45): 15300–15308. doi:10.1021/jacs.8b08260. PMID 30296064.
- ^ a b Yu, SH; Boyce, M; Wands, AM; Bond, MR; Bertozzi, CR; Kohler, JJ (2012-03-27). "Metabolic Labeling Enables Selective Photocrosslinking of O-GlcNAc-modified Proteins to Their Binding Partners". Proceedings of the National Academy of Sciences of the United States of America. 109 (13): 4834–9. Bibcode:2012PNAS..109.4834Y. doi:10.1073/pnas.1114356109. PMC 3323966. PMID 22411826.
- ^ Rodriguez, AC; Kohler, JJ (2014-08-01). "Recognition of Diazirine-Modified O-GlcNAc by Human O-GlcNAcase". MedChemComm. 5 (8): 1227–1234. doi:10.1039/C4MD00164H. PMC 4109824. PMID 25068034.
- ^ Zaro, BW; Yang, YY; Hang, HC; Pratt, MR (2011-05-17). "Chemical Reporters for Fluorescent Detection and Identification of O-GlcNAc-modified Proteins Reveal Glycosylation of the Ubiquitin Ligase NEDD4-1". Proceedings of the National Academy of Sciences of the United States of America. 108 (20): 8146–51. Bibcode:2011PNAS..108.8146Z. doi:10.1073/pnas.1102458108. PMC 3100932. PMID 21540332.
- ^ Zaro, Balyn W.; Chuh, Kelly N.; Pratt, Matthew R. (2014-09-19). "Chemical Reporter for Visualizing Metabolic Cross-Talk between Carbohydrate Metabolism and Protein Modification". ACS Chemical Biology. 9 (9): 1991–1996. doi:10.1021/cb5005564. ISSN 1554-8929. PMC 4168799. PMID 25062036.
- ^ a b "Click-IT™ O-GlcNAc Enzymatic Labeling System". www.thermofisher.com. Retrieved 2020-05-30.
- ^ Carrillo, LD; Krishnamoorthy, L; Mahal, LK (2006-11-22). "A Cellular FRET-based Sensor for beta-O-GlcNAc, a Dynamic Carbohydrate Modification Involved in Signaling". Journal of the American Chemical Society. 128 (46): 14768–9. doi:10.1021/ja065835+. PMID 17105262.
- ^ Carrillo, Luz D.; Froemming, Joshua A.; Mahal, Lara K. (2011-02-25). "Targeted in Vivo O-GlcNAc Sensors Reveal Discrete Compartment-specific Dynamics during Signal Transduction". Journal of Biological Chemistry. 286 (8): 6650–6658. doi:10.1074/jbc.M110.191627. ISSN 0021-9258. PMC 3057821. PMID 21138847.
- ^ Ma, Junfeng; Hart, Gerald W. (2017-02-02). "Analysis of Protein O-GlcNAcylation by Mass Spectrometry". Current Protocols in Protein Science. 87: 24.10.1–24.10.16. doi:10.1002/cpps.24. ISSN 1934-3655. PMC 5300742. PMID 28150883.
- ^ Wells, L; Vosseller, K; Cole, RN; Cronshaw, JM; Matunis, MJ; Hart, GW (October 2002). "Mapping Sites of O-GlcNAc Modification Using Affinity Tags for Serine and Threonine Post-Translational Modifications". Molecular & Cellular Proteomics. 1 (10): 791–804. doi:10.1074/mcp.m200048-mcp200. PMID 12438562.
- ^ Zhao, Peng; Viner, Rosa; Teo, Chin Fen; Boons, Geert-Jan; Horn, David; Wells, Lance (2011-09-02). "Combining High-energy C-trap Dissociation and Electron Transfer Dissociation for Protein O-GlcNAc Modification Site Assignment". Journal of Proteome Research. 10 (9): 4088–4104. doi:10.1021/pr2002726. ISSN 1535-3893. PMC 3172619. PMID 21740066.
- ^ Palaniappan, Krishnan K.; Pitcher, Austin A.; Smart, Brian P.; Spiciarich, David R.; Iavarone, Anthony T.; Bertozzi, Carolyn R. (2011-08-19). "Isotopic Signature Transfer and Mass Pattern Prediction (IsoStamp): An Enabling Technique for Chemically-Directed Proteomics". ACS Chemical Biology. 6 (8): 829–836. doi:10.1021/cb100338x. ISSN 1554-8929. PMC 3220624. PMID 21604797.
- ^ Woo, CM; Iavarone, AT; Spiciarich, DR; Palaniappan, KK; Bertozzi, CR (June 2015). "Isotope-targeted Glycoproteomics (IsoTaG): A Mass-Independent Platform for Intact N- And O-glycopeptide Discovery and Analysis". Nature Methods. 12 (6): 561–7. doi:10.1038/nmeth.3366. PMC 4599779. PMID 25894945.
- ^ Woo, Christina M.; Felix, Alejandra; Byrd, William E.; Zuegel, Devon K.; Ishihara, Mayumi; Azadi, Parastoo; Iavarone, Anthony T.; Pitteri, Sharon J.; Bertozzi, Carolyn R. (2017-04-07). "Development of IsoTaG, a Chemical Glycoproteomics Technique for Profiling Intact N- and O-Glycopeptides from Whole Cell Proteomes". Journal of Proteome Research. 16 (4): 1706–1718. doi:10.1021/acs.jproteome.6b01053. ISSN 1535-3893. PMC 5507588. PMID 28244757.
- ^ Woo, Christina M.; Felix, Alejandra; Zhang, Lichao; Elias, Joshua E.; Bertozzi, Carolyn R. (January 2017). "Isotope Targeted Glycoproteomics (IsoTaG) analysis of sialylated N- and O-glycopeptides on an Orbitrap Fusion Tribrid using azido and alkynyl sugars". Analytical and Bioanalytical Chemistry. 409 (2): 579–588. doi:10.1007/s00216-016-9934-9. ISSN 1618-2642. PMC 5342897. PMID 27695962.
- ^ a b Woo, CM; Lund, PJ; Huang, AC; Davis, MM; Bertozzi, CR; Pitteri, SJ (April 2018). "Mapping and Quantification of Over 2000 O-linked Glycopeptides in Activated Human T Cells With Isotope-Targeted Glycoproteomics (Isotag)". Molecular & Cellular Proteomics. 17 (4): 764–775. doi:10.1074/mcp.RA117.000261. PMC 5880114. PMID 29351928.
- ^ Khidekel, N; Ficarro, SB; Clark, PM; Bryan, MC; Swaney, DL; Rexach, JE; Sun, YE; Coon, JJ; Peters, EC; Hsieh-Wilson, LC (June 2007). "Probing the Dynamics of O-GlcNAc Glycosylation in the Brain Using Quantitative Proteomics" (PDF). Nature Chemical Biology. 3 (6): 339–48. doi:10.1038/nchembio881. PMID 17496889.
- ^ Qin, K; Zhu, Y; Qin, W; Gao, J; Shao, X; Wang, YL; Zhou, W; Wang, C; Chen, X (2018-08-17). "Quantitative Profiling of Protein O-GlcNAcylation Sites by an Isotope-Tagged Cleavable Linker". ACS Chemical Biology. 13 (8): 1983–1989. doi:10.1021/acschembio.8b00414. PMID 30059200.
- ^ Li, J; Li, Z; Duan, X; Qin, K; Dang, L; Sun, S; Cai, L; Hsieh-Wilson, LC; Wu, L; Yi, W (2019-01-18). "An Isotope-Coded Photocleavable Probe for Quantitative Profiling of Protein O-GlcNAcylation" (PDF). ACS Chemical Biology. 14 (1): 4–10. doi:10.1021/acschembio.8b01052. PMID 30620550.
- ^ Liu, Tai-Wei; Zandberg, Wesley F.; Gloster, Tracey M.; Deng, Lehua; Murray, Kelsey D.; Shan, Xiaoyang; Vocadlo, David J. (June 25, 2018). "Metabolic Inhibitors of O-GlcNAc Transferase That Act In Vivo Implicate Decreased O-GlcNAc Levels in Leptin-Mediated Nutrient Sensing". Angewandte Chemie International Edition. 57 (26): 7644–7648. doi:10.1002/anie.201803254. ISSN 1521-3773. PMC 6055616. PMID 29756380.
- ^ Martin, Sara E. S.; Tan, Zhi-Wei; Itkonen, Harri M.; Duveau, Damien Y.; Paulo, Joao A.; Janetzko, John; Boutz, Paul L.; Törk, Lisa; Moss, Frederick A.; Thomas, Craig J.; Gygi, Steven P. (October 24, 2018). "Structure-Based Evolution of Low Nanomolar O-GlcNAc Transferase Inhibitors". Journal of the American Chemical Society. 140 (42): 13542–13545. doi:10.1021/jacs.8b07328. ISSN 1520-5126. PMC 6261342. PMID 30285435.
- ^ Dorfmueller, Helge C.; Borodkin, Vladimir S.; Schimpl, Marianne; Shepherd, Sharon M.; Shpiro, Natalia A.; van Aalten, Daan M. F. (2006-12-27). "GlcNAcstatin: a picomolar, selective O-GlcNAcase inhibitor that modulates intracellular O-glcNAcylation levels". Journal of the American Chemical Society. 128 (51): 16484–16485. doi:10.1021/ja066743n. ISSN 0002-7863. PMC 7116141. PMID 17177381.
- ^ a b Yuzwa, SA; Macauley, MS; Heinonen, JE; Shan, X; Dennis, RJ; He, Y; Whitworth, GE; Stubbs, KA; McEachern, EJ (August 2008). "A Potent Mechanism-Inspired O-GlcNAcase Inhibitor That Blocks Phosphorylation of Tau in Vivo". Nature Chemical Biology. 4 (8): 483–90. doi:10.1038/nchembio.96. PMID 18587388.
- ^ Akella, Neha M.; Ciraku, Lorela; Reginato, Mauricio J. (2019-07-04). "Fueling the fire: emerging role of the hexosamine biosynthetic pathway in cancer". BMC Biology. 17 (1): 52. doi:10.1186/s12915-019-0671-3. ISSN 1741-7007. PMC 6610925. PMID 31272438.
- ^ a b c d Tarrant, MK; Rho, HS; Xie, Z; Jiang, YL; Gross, C; Culhane, JC; Yan, G; Qian, J; Ichikawa, Y (2012-01-22). "Regulation of CK2 by Phosphorylation and O-GlcNAcylation Revealed by Semisynthesis". Nature Chemical Biology. 8 (3): 262–9. doi:10.1038/nchembio.771. PMC 3288285. PMID 22267120.
- ^ a b c d Marotta, NP; Lin, YH; Lewis, YE; Ambroso, MR; Zaro, BW; Roth, MT; Arnold, DB; Langen, R; Pratt, MR (Nov 2015). "O-GlcNAc Modification Blocks the Aggregation and Toxicity of the Protein α-Synuclein Associated With Parkinson's Disease". Nature Chemistry. 7 (11): 913–20. Bibcode:2015NatCh...7..913M. doi:10.1038/nchem.2361. PMC 4618406. PMID 26492012.
- ^ a b Gorelik, A; Bartual, SG; Borodkin, VS; Varghese, J; Ferenbach, AT; van Aalten, DMF (November 2019). "Genetic Recoding to Dissect the Roles of Site-Specific Protein O-GlcNAcylation". Nature Structural & Molecular Biology. 26 (11): 1071–1077. doi:10.1038/s41594-019-0325-8. PMC 6858883. PMID 31695185.
- ^ Lewis, YE; Galesic, A; Levine, PM; De Leon, CA; Lamiri, N; Brennan, CK; Pratt, MR (2017-04-21). "O-GlcNAcylation of α-Synuclein at Serine 87 Reduces Aggregation Without Affecting Membrane Binding". ACS Chemical Biology. 12 (4): 1020–1027. doi:10.1021/acschembio.7b00113. PMC 5607117. PMID 28195695.
- ^ a b Chuh, Kelly N.; Batt, Anna R.; Zaro, Balyn W.; Darabedian, Narek; Marotta, Nicholas P.; Brennan, Caroline K.; Amirhekmat, Arya; Pratt, Matthew R. (2017-06-14). "The New Chemical Reporter 6-Alkynyl-6-deoxy-GlcNAc Reveals O-GlcNAc Modification of the Apoptotic Caspases That Can Block the Cleavage/Activation of Caspase-8". Journal of the American Chemical Society. 139 (23): 7872–7885. doi:10.1021/jacs.7b02213. ISSN 0002-7863. PMC 6225779. PMID 28528544.
- ^ Maynard, JC; Burlingame, AL; Medzihradszky, KF (November 2016). "Cysteine S-linked N-acetylglucosamine (S-GlcNAcylation), A New Post-translational Modification in Mammals". Molecular & Cellular Proteomics. 15 (11): 3405–3411. doi:10.1074/mcp.M116.061549. PMC 5098038. PMID 27558639.
- ^ Macauley, MS; Stubbs, KA; Vocadlo, DJ (2005-12-14). "O-GlcNAcase Catalyzes Cleavage of Thioglycosides Without General Acid Catalysis". Journal of the American Chemical Society. 127 (49): 17202–3. doi:10.1021/ja0567687. PMID 16332065.
- ^ Mehta, AY; Veeraiah, RKH; Dutta, S; Goth, CK; Hanes, MS; Gao, C; Stavenhagen, K; Kardish, R; Matsumoto, Y; Heimburg-Molinaro, J; Boyce, M; Pohl, NLB; Cummings, RD (17 September 2020). "Parallel Glyco-SPOT Synthesis of Glycopeptide Libraries". Cell Chemical Biology. 27 (9): 1207–1219.e9. doi:10.1016/j.chembiol.2020.06.007. PMC 7556346. PMID 32610041.
- ^ De Leon, CA; Levine, PM; Craven, TW; Pratt, MR (2017-07-11). "The Sulfur-Linked Analogue of O-GlcNAc (S-GlcNAc) Is an Enzymatically Stable and Reasonable Structural Surrogate for O-GlcNAc at the Peptide and Protein Levels". Biochemistry. 56 (27): 3507–3517. doi:10.1021/acs.biochem.7b00268. PMC 5598463. PMID 28627871.
- ^ Ramirez, DH; Aonbangkhen, C; Wu, HY; Naftaly, JA; Tang, S; O'Meara, TR; Woo, CM (2020-04-17). "Engineering a Proximity-Directed O-GlcNAc Transferase for Selective Protein O-GlcNAcylation in Cells". ACS Chemical Biology. 15 (4): 1059–1066. doi:10.1021/acschembio.0c00074. PMC 7296736. PMID 32119511.
- ^ a b Ferrer, Christina M.; Lynch, Thomas P.; Sodi, Valerie L.; Falcone, John N.; Schwab, Luciana P.; Peacock, Danielle L.; Vocadlo, David J.; Seagroves, Tiffany N.; Reginato, Mauricio J. (2014-06-05). "O-GlcNAcylation regulates cancer metabolism and survival stress signaling via regulation of the HIF-1 pathway". Molecular Cell. 54 (5): 820–831. doi:10.1016/j.molcel.2014.04.026. ISSN 1097-4164. PMC 4104413. PMID 24857547.
- ^ a b Ma, Z; Vocadlo, DJ; Vosseller, K (2013-05-24). "Hyper-O-GlcNAcylation Is Anti-Apoptotic and Maintains Constitutive NF-κB Activity in Pancreatic Cancer Cells". The Journal of Biological Chemistry. 288 (21): 15121–30. doi:10.1074/jbc.M113.470047. PMC 3663532. PMID 23592772.
- ^ Torres, IO; Fujimori, DG (December 2015). "Functional Coupling Between Writers, Erasers and Readers of Histone and DNA Methylation". Current Opinion in Structural Biology. 35: 68–75. doi:10.1016/j.sbi.2015.09.007. PMC 4688207. PMID 26496625.
- ^ a b Chu, CS; Lo, PW; Yeh, YH; Hsu, PH; Peng, SH; Teng, YC; Kang, ML; Wong, CH; Juan, LJ (2014-01-28). "O-GlcNAcylation Regulates EZH2 Protein Stability and Function". Proceedings of the National Academy of Sciences of the United States of America. 111 (4): 1355–60. Bibcode:2014PNAS..111.1355C. doi:10.1073/pnas.1323226111. PMC 3910655. PMID 24474760.
- ^ Lo, PW; Shie, JJ; ChChen, CH; Wu, CY; Hsu, TL; Wong, CH (2018-07-10). "O-GlcNAcylation Regulates the Stability and Enzymatic Activity of the Histone Methyltransferase EZH2". Proceedings of the National Academy of Sciences of the United States of America. 115 (28): 7302–7307. doi:10.1073/pnas.1801850115. PMC 6048490. PMID 29941599.
- ^ Zhang, Q; Liu, X; Gao, W; Li, P; Hou, J; Li, J; Wong, J (2014-02-28). "Differential Regulation of the Ten-Eleven Translocation (TET) Family of Dioxygenases by O-linked β-N-acetylglucosamine Transferase (OGT)". Journal of Biological Chemistry. 289 (9): 5986–96. doi:10.1074/jbc.M113.524140. PMC 3937666. PMID 24394411.
- ^ Zhang, Qiao; Liu, Xiaoguang; Gao, Wenqi; Li, Pishun; Hou, Jingli; Li, Jiwen; Wong, Jiemin (2014-02-28). "Differential Regulation of the Ten-Eleven Translocation (TET) Family of Dioxygenases by O-Linked β-N-Acetylglucosamine Transferase (OGT)". Journal of Biological Chemistry. 289 (9): 5986–5996. doi:10.1074/jbc.M113.524140. ISSN 0021-9258. PMC 3937666. PMID 24394411.
- ^ a b c Zhu, Guizhou; Tao, Tao; Zhang, Dongmei; Liu, Xiaojuan; Qiu, Huiyuan; Han, LiJian; Xu, Zhiwei; Xiao, Ying; Cheng, Chun; Shen, Aiguo (Aug 2016). "O-GlcNAcylation of histone deacetylases 1 in hepatocellular carcinoma promotes cancer progression". Glycobiology. 26 (8): 820–833. doi:10.1093/glycob/cww025. ISSN 1460-2423. PMID 27060025.
- ^ Fong, Jerry J.; Nguyen, Brenda L.; Bridger, Robert; Medrano, Estela E.; Wells, Lance; Pan, Shujuan; Sifers, Richard N. (2012-04-06). "β-N-Acetylglucosamine (O-GlcNAc) Is a Novel Regulator of Mitosis-specific Phosphorylations on Histone H3". Journal of Biological Chemistry. 287 (15): 12195–12203. doi:10.1074/jbc.M111.315804. ISSN 0021-9258. PMC 3320971. PMID 22371497.
- ^ a b c Fujiki, R; Hashiba, W; Sekine, H; Yokoyama, A; Chikanishi, T; Ito, S; Imai, Y; Kim, J; He, HH (2011-11-27). "GlcNAcylation of Histone H2B Facilitates Its Monoubiquitination". Nature. 480 (7378): 557–60. Bibcode:2011Natur.480..557F. doi:10.1038/nature10656. PMC 7289526. PMID 22121020.
- ^ Chen, Q; Chen, Y; Bian, C; Fujiki, R; Yu, X (2013-01-24). "TET2 Promotes Histone O-GlcNAcylation During Gene Transcription". Nature. 493 (7433): 561–4. Bibcode:2013Natur.493..561C. doi:10.1038/nature11742. PMC 3684361. PMID 23222540.
- ^ a b c d Xu, Qiuran; Yang, Caihong; Du, Yu; Chen, Yali; Liu, Hailong; Deng, Min; Zhang, Haoxing; Zhang, Lei; Liu, Tongzheng; Liu, Qingguang; Wang, Liewei (2014-05-01). "AMPK regulates histone H2B O-GlcNAcylation". Nucleic Acids Research. 42 (9): 5594–5604. doi:10.1093/nar/gku236. ISSN 0305-1048. PMC 4027166. PMID 24692660.
- ^ Kreppel, L. K.; Hart, G. W. (1999-11-05). "Regulation of a cytosolic and nuclear O-GlcNAc transferase. Role of the tetratricopeptide repeats". Journal of Biological Chemistry. 274 (45): 32015–32022. doi:10.1074/jbc.274.45.32015. ISSN 0021-9258. PMID 10542233.
- ^ a b Cheung, Win D.; Hart, Gerald W. (2008-05-09). "AMP-activated Protein Kinase and p38 MAPK Activate O-GlcNAcylation of Neuronal Proteins during Glucose Deprivation". Journal of Biological Chemistry. 283 (19): 13009–13020. doi:10.1074/jbc.M801222200. ISSN 0021-9258. PMC 2435304. PMID 18353774.
- ^ Zou, Luyun; Zhu-Mauldin, Xiaoyuan; Marchase, Richard B.; Paterson, Andrew J.; Liu, Jian; Yang, Qinglin; Chatham, John C. (2012-10-05). "Glucose deprivation-induced increase in protein O-GlcNAcylation in cardiomyocytes is calcium-dependent". The Journal of Biological Chemistry. 287 (41): 34419–34431. doi:10.1074/jbc.M112.393207. ISSN 1083-351X. PMC 3464547. PMID 22908225.
- ^ Taylor, Rodrick P.; Parker, Glendon J.; Hazel, Mark W.; Soesanto, Yudi; Fuller, William; Yazzie, Marla J.; McClain, Donald A. (2008-03-07). "Glucose deprivation stimulates O-GlcNAc modification of proteins through up-regulation of O-linked N-acetylglucosaminyltransferase". Journal of Biological Chemistry. 283 (10): 6050–6057. doi:10.1074/jbc.M707328200. ISSN 0021-9258. PMID 18174169.
- ^ Chen, PH; Smith, TJ; Wu, J; Siesser, PJ; Bisnett, BJ; Khan, F; Hogue, M; Soderblom, E; Tang, F; Marks, JR; Major, MB; Swarts, BM; Boyce, M; Chi, Jen-Tsan (2017-08-01). "Glycosylation of KEAP1 Links Nutrient Sensing to Redox Stress Signaling". The EMBO Journal. 36 (15): 2233–2250. doi:10.15252/embj.201696113. PMC 5538768. PMID 28663241.
- ^ McGreal, SR; Bhushan, B; Walesky, C; McGill, MR; Lebofsky, M; Kandel, SE; Winefield, RD; Jaeschke, H; Zachara, NE; Zhang, Z; Tan, EP; Slawson, C; Apte, U (2018-04-01). "Modulation of O-GlcNAc Levels in the Liver Impacts Acetaminophen-Induced Liver Injury by Affecting Protein Adduct Formation and Glutathione Synthesis". Toxicological Sciences. 162 (2): 599–610. doi:10.1093/toxsci/kfy002. PMC 6012490. PMID 29325178.
- ^ a b Yuzwa, SA; Shan, X; Macauley, MS; Clark, T; Skorobogatko, Y; Vosseller, K; Vocadlo, DJ (2012-02-26). "Increasing O-GlcNAc Slows Neurodegeneration and Stabilizes Tau Against Aggregation". Nature Chemical Biology. 8 (4): 393–9. doi:10.1038/nchembio.797. PMID 22366723.
- ^ Cheng, X.; Cole, R. N.; Zaia, J.; Hart, G. W. (2000-09-26). "Alternative O-glycosylation/O-phosphorylation of the murine estrogen receptor beta". Biochemistry. 39 (38): 11609–11620. doi:10.1021/bi000755i. ISSN 0006-2960. PMID 10995228.
- ^ Comer, F. I.; Hart, G. W. (2001-07-03). "Reciprocity between O-GlcNAc and O-phosphate on the carboxyl terminal domain of RNA polymerase II". Biochemistry. 40 (26): 7845–7852. doi:10.1021/bi0027480. ISSN 0006-2960. PMID 11425311.
- ^ a b Liu, Fei; Iqbal, Khalid; Grundke-Iqbal, Inge; Hart, Gerald W.; Gong, Cheng-Xin (2004-07-20). "O-GlcNAcylation regulates phosphorylation of tau: A mechanism involved in Alzheimer's disease". Proceedings of the National Academy of Sciences of the United States of America. 101 (29): 10804–10809. Bibcode:2004PNAS..10110804L. doi:10.1073/pnas.0400348101. ISSN 0027-8424. PMC 490015. PMID 15249677.
- ^ a b Yang, WH; Kim, JE; Nam, HW; Ju, JW; Kim, HS; Kim, YS; Cho, JW (Oct 2006). "Modification of p53 With O-linked N-acetylglucosamine Regulates p53 Activity and Stability". Nature Cell Biology. 8 (10): 1074–83. doi:10.1038/ncb1470. PMID 16964247. S2CID 12326082.
- ^ a b Dias, WB; Cheung, WD; Wang, Z; Hart, GW (2009-08-07). "Regulation of Calcium/Calmodulin-Dependent Kinase IV by O-GlcNAc Modification". Journal of Biological Chemistry. 284 (32): 21327–37. doi:10.1074/jbc.M109.007310. PMC 2755857. PMID 19506079.
- ^ a b c Ma, Z; Chalkley, RJ; Vosseller, K (2017-06-02). "Hyper- O-GlcNAcylation Activates Nuclear Factor κ-Light-Chain-Enhancer of Activated B Cells (NF-κB) Signaling Through Interplay With Phosphorylation and Acetylation". The Journal of Biological Chemistry. 292 (22): 9150–9163. doi:10.1074/jbc.M116.766568. PMC 5454098. PMID 28416608.
- ^ a b c Olivier-Van Stichelen, Stéphanie; Dehennaut, Vanessa; Buzy, Armelle; Zachayus, Jean-Luc; Guinez, Céline; Mir, Anne-Marie; El Yazidi-Belkoura, Ikram; Copin, Marie-Christine; Boureme, Didier; Loyaux, Denis; Ferrara, Pascual (Aug 2014). "O-GlcNAcylation stabilizes β-catenin through direct competition with phosphorylation at threonine 41". FASEB Journal. 28 (8): 3325–3338. doi:10.1096/fj.13-243535. ISSN 1530-6860. PMC 4101651. PMID 24744147.
- ^ a b Huang, Xun; Pan, Qiuming; Sun, Danni; Chen, Wei; Shen, Aijun; Huang, Min; Ding, Jian; Geng, Meiyu (2013-12-20). "O-GlcNAcylation of Cofilin Promotes Breast Cancer Cell Invasion". Journal of Biological Chemistry. 288 (51): 36418–36425. doi:10.1074/jbc.M113.495713. ISSN 0021-9258. PMC 3868755. PMID 24214978.
- ^ a b c Selnick, Harold G.; Hess, J. Fred; Tang, Cuyue; Liu, Kun; Schachter, Joel B.; Ballard, Jeanine E.; Marcus, Jacob; Klein, Daniel J.; Wang, Xiaohai; Pearson, Michelle; Savage, Mary J.; Kaul, Ramesh; Li, Tong-Shuang; Vocadlo, David J.; Zhou, Yuanxi; Zhu, Yongbao; Mu, Changwei; Wang, Yaode; Wei, Zhongyong; Bai, Chang; Duffy, Joseph L.; McEachern, Ernest J. (Nov 2019). "Discovery of MK-8719, a Potent O-GlcNAcase Inhibitor as a Potential Treatment for Tauopathies". Journal of Medicinal Chemistry. 62 (22): 10062–10097. doi:10.1021/acs.jmedchem.9b01090. ISSN 1520-4804. PMID 31487175.
- ^ Schwein, Paul A; Woo, Christina M (2020-03-20). "The O-GlcNAc Modification on Kinases". ACS Chemical Biology. 15 (3): 602–617. doi:10.1021/acschembio.9b01015. PMC 7253032. PMID 32155042.
- ^ a b Bullen, JW; Balsbaugh, JL; Chanda, D; Shabanowitz, J; Hunt, DF; Neumann, D; Hart, GW (2014-04-11). "Cross-talk Between Two Essential Nutrient-Sensitive Enzymes: O-GlcNAc Transferase (OGT) and AMP-activated Protein Kinase (AMPK)". Journal of Biological Chemistry. 289 (15): 10592–606. doi:10.1074/jbc.M113.523068. PMC 4036179. PMID 24563466.
- ^ a b Luo, Bai; Parker, Glendon J.; Cooksey, Robert C.; Soesanto, Yudi; Evans, Mark; Jones, Deborah; McClain, Donald A. (2007-03-09). "Chronic hexosamine flux stimulates fatty acid oxidation by activating AMP-activated protein kinase in adipocytes". Journal of Biological Chemistry. 282 (10): 7172–7180. doi:10.1074/jbc.M607362200. ISSN 0021-9258. PMID 17227772.
- ^ a b Sodi, VL; Bacigalupa, ZA; Ferrer, CM; Lee, JV; Gocal, WA; Mukhopadhyay, D; Wellen, KE; Ivan, M; Reginato, MJ (2018-02-15). "Nutrient Sensor O-GlcNAc Transferase Controls Cancer Lipid Metabolism via SREBP-1 Regulation". Oncogene. 37 (7): 924–934. doi:10.1038/onc.2017.395. PMC 5814337. PMID 29059153.
- ^ Wells, Lance; Kreppel, Lisa K.; Comer, Frank I.; Wadzinski, Brian E.; Hart, Gerald W. (2004-09-10). "O-GlcNAc transferase is in a functional complex with protein phosphatase 1 catalytic subunits". The Journal of Biological Chemistry. 279 (37): 38466–38470. doi:10.1074/jbc.M406481200. ISSN 0021-9258. PMID 15247246.
- ^ Cheung, Win D.; Sakabe, Kaoru; Housley, Michael P.; Dias, Wagner B.; Hart, Gerald W. (2008-12-05). "O-linked beta-N-acetylglucosaminyltransferase substrate specificity is regulated by myosin phosphatase targeting and other interacting proteins". The Journal of Biological Chemistry. 283 (49): 33935–33941. doi:10.1074/jbc.M806199200. ISSN 0021-9258. PMC 2590692. PMID 18840611.
- ^ Yang, X.; Su, K.; Roos, M. D.; Chang, Q.; Paterson, A. J.; Kudlow, J. E. (2001-06-05). "O-linkage of N-acetylglucosamine to Sp1 activation domain inhibits its transcriptional capability". Proceedings of the National Academy of Sciences of the United States of America. 98 (12): 6611–6616. Bibcode:2001PNAS...98.6611Y. doi:10.1073/pnas.111099998. ISSN 0027-8424. PMC 34401. PMID 11371615.
- ^ Lamarre-Vincent, Nathan; Hsieh-Wilson, Linda C. (2003-06-04). "Dynamic glycosylation of the transcription factor CREB: a potential role in gene regulation" (PDF). Journal of the American Chemical Society. 125 (22): 6612–6613. doi:10.1021/ja028200t. ISSN 0002-7863. PMID 12769553.
- ^ Rexach, Jessica E.; Clark, Peter M.; Mason, Daniel E.; Neve, Rachael L.; Peters, Eric C.; Hsieh-Wilson, Linda C. (2012-01-22). "Dynamic O-GlcNAc modification regulates CREB-mediated gene expression and memory formation". Nature Chemical Biology. 8 (3): 253–261. doi:10.1038/nchembio.770. ISSN 1552-4469. PMC 3288555. PMID 22267118.
- ^ Toleman, Clifford A.; Schumacher, Maria A.; Yu, Seok-Ho; Zeng, Wenjie; Cox, Nathan J.; Smith, Timothy J.; Soderblom, Erik J.; Wands, Amberlyn M.; Kohler, Jennifer J.; Boyce, Michael (2018-06-05). "Structural basis of O-GlcNAc recognition by mammalian 14-3-3 proteins". Proceedings of the National Academy of Sciences of the United States of America. 115 (23): 5956–5961. doi:10.1073/pnas.1722437115. ISSN 0027-8424. PMC 6003352. PMID 29784830.
- ^ Guinez, Céline; Lemoine, Jérôme; Michalski, Jean-Claude; Lefebvre, Tony (2004-06-18). "70-kDa-heat shock protein presents an adjustable lectinic activity towards O-linked N-acetylglucosamine". Biochemical and Biophysical Research Communications. 319 (1): 21–26. doi:10.1016/j.bbrc.2004.04.144. ISSN 0006-291X. PMID 15158436.
- ^ Zhang, F; Su, K; Yang, X; Bowe, DB; Paterson, AJ; Kudlow, JE (2003-12-12). "O-GlcNAc Modification Is an Endogenous Inhibitor of the Proteasome". Cell. 115 (6): 715–25. doi:10.1016/s0092-8674(03)00974-7. PMID 14675536. S2CID 8221476.
- ^ Zhang, Fengxue; Hu, Yong; Huang, Ping; Toleman, Clifford A.; Paterson, Andrew J.; Kudlow, Jeffrey E. (2007-08-03). "Proteasome function is regulated by cyclic AMP-dependent protein kinase through phosphorylation of Rpt6". The Journal of Biological Chemistry. 282 (31): 22460–22471. doi:10.1074/jbc.M702439200. ISSN 0021-9258. PMID 17565987.
- ^ Keembiyehetty, Chithra N.; Krzeslak, Anna; Love, Dona C.; Hanover, John A. (2011-08-15). "A lipid-droplet-targeted O-GlcNAcase isoform is a key regulator of the proteasome". Journal of Cell Science. 124 (Pt 16): 2851–2860. doi:10.1242/jcs.083287. ISSN 1477-9137. PMC 3148132. PMID 21807949.
- ^ Zachara, Natasha E.; O'Donnell, Niall; Cheung, Win D.; Mercer, Jessica J.; Marth, Jamey D.; Hart, Gerald W. (2004-07-16). "Dynamic O-GlcNAc modification of nucleocytoplasmic proteins in response to stress. A survival response of mammalian cells". The Journal of Biological Chemistry. 279 (29): 30133–30142. doi:10.1074/jbc.M403773200. ISSN 0021-9258. PMID 15138254.
- ^ Iqbal, Khalid; Liu, Fei; Gong, Cheng-Xin; Grundke-Iqbal, Inge (December 2010). "Tau in Alzheimer Disease and Related Tauopathies". Current Alzheimer Research. 7 (8): 656–664. doi:10.2174/156720510793611592. ISSN 1567-2050. PMC 3090074. PMID 20678074.
- ^ Arnold, CS; Johnson, GV; Cole, RN; Dong, DL; Lee, M; Hart, GW (1996-11-15). "The Microtubule-Associated Protein Tau Is Extensively Modified With O-linked N-acetylglucosamine". Journal of Biological Chemistry. 271 (46): 28741–4. doi:10.1074/jbc.271.46.28741. PMID 8910513.
- ^ O'Donnell, N, Zachara, N, Hart, GW, Marth JD. (2004)Ogt 의존성 X-크롬 연계 단백질 글리코실화는 체세포 기능과 배아 생존성의 필수 변형이다.분자 및 세포 생물학.24: 1680-1690.디올.
- ^ Sandhu, Punam; Lee, Junghoon; Ballard, Jeanine; Walker, Brittany; Ellis, Joan; Marcus, Jacob; Toolan, Dawn; Dreyer, Daniel; McAvoy, Thomas; Duffy, Joseph; Michener, Maria (July 2016). "P4-036: Pharmacokinetics and Pharmacodynamics to Support Clinical Studies of MK-8719: an O-GlcNAcase Inhibitor for Progressive Supranuclear Palsy". Alzheimer's & Dementia. 12 (7S_Part_21): P1028. doi:10.1016/j.jalz.2016.06.2125. S2CID 54229492.
- ^ Medina, Miguel (2018-04-11). "An Overview on the Clinical Development of Tau-Based Therapeutics". International Journal of Molecular Sciences. 19 (4): 1160. doi:10.3390/ijms19041160. ISSN 1422-0067. PMC 5979300. PMID 29641484.
- ^ Yi, Wen; Clark, Peter M.; Mason, Daniel E.; Keenan, Marie C.; Hill, Collin; Goddard, William A.; Peters, Eric C.; Driggers, Edward M.; Hsieh-Wilson, Linda C. (2012-08-24). "PFK1 Glycosylation Is a Key Regulator of Cancer Cell Growth and Central Metabolic Pathways". Science. 337 (6097): 975–980. doi:10.1126/science.1222278. ISSN 0036-8075. PMC 3534962. PMID 22923583.
- ^ Caldwell, SA; Jackson, SR; Shahriari, KS; Lynch, TP; Sethi, G; Walker, S; Vosseller, K; Reginato, MJ (2010-05-13). "Nutrient Sensor O-GlcNAc Transferase Regulates Breast Cancer Tumorigenesis Through Targeting of the Oncogenic Transcription Factor FoxM1". Oncogene. 29 (19): 2831–42. doi:10.1038/onc.2010.41. PMID 20190804. S2CID 25957261.
- ^ Lynch, TP; Ferrer, CM; Jackson, SR; Shahriari, KS; Vosseller, K; Reginato, MJ (2012-03-30). "Critical Role of O-Linked β-N-acetylglucosamine Transferase in Prostate Cancer Invasion, Angiogenesis, and Metastasis". The Journal of Biological Chemistry. 287 (14): 11070–81. doi:10.1074/jbc.M111.302547. PMC 3322861. PMID 22275356.
- ^ Liu, K; Paterson, AJ; Chin, E; Kudlow, JE (2000-03-14). "Glucose Stimulates Protein Modification by O-linked GlcNAc in Pancreatic Beta Cells: Linkage of O-linked GlcNAc to Beta Cell Death". Proceedings of the National Academy of Sciences of the United States of America. 97 (6): 2820–5. Bibcode:2000PNAS...97.2820L. doi:10.1073/pnas.97.6.2820. PMC 16013. PMID 10717000.
- ^ Pathak, Shalini; Dorfmueller, Helge C.; Borodkin, Vladimir S.; van Aalten, Daan M.F. (2008-08-25). "Chemical Dissection of the Link between Streptozotocin, O-GlcNAc, and Pancreatic Cell Death". Chemistry & Biology. 15 (8): 799–807. doi:10.1016/j.chembiol.2008.06.010. ISSN 1074-5521. PMC 2568864. PMID 18721751.
- ^ a b Vosseller, K; Wells, L; Lane, MD; Hart, GW (2002-04-16). "Elevated Nucleocytoplasmic Glycosylation by O-GlcNAc Results in Insulin Resistance Associated With Defects in Akt Activation in 3T3-L1 Adipocytes". Proceedings of the National Academy of Sciences of the United States of America. 99 (8): 5313–8. Bibcode:2002PNAS...99.5313V. doi:10.1073/pnas.072072399. PMC 122766. PMID 11959983.
- ^ Yang, X; Ongusaha, PP; Miles, PD; Havstad, JC; Zhang, F; So, WV; Kudlow, JE; Michell, RH; Olefsky, HM; Field, SJ; Evans, RMdate=2008-02-21 (2008). "Phosphoinositide Signalling Links O-GlcNAc Transferase to Insulin Resistance". Nature. 451 (7181): 964–9. Bibcode:2008Natur.451..964Y. doi:10.1038/nature06668. PMID 18288188. S2CID 18459576.
- ^ Whelan, Stephen A.; Lane, M. Daniel; Hart, Gerald W. (2008-08-01). "Regulation of the O-Linked β-N-Acetylglucosamine Transferase by Insulin Signaling". Journal of Biological Chemistry. 283 (31): 21411–21417. doi:10.1074/jbc.M800677200. ISSN 0021-9258. PMC 2490780. PMID 18519567.
- ^ Macauley, MS; Bubb, AK; Martinez-Fleites, C; Davies, GJ; Vocadlo, DJ (2008-12-12). "Elevation of Global O-GlcNAc Levels in 3T3-L1 Adipocytes by Selective Inhibition of O-GlcNAcase Does Not Induce Insulin Resistance". The Journal of Biological Chemistry. 283 (50): 34687–95. doi:10.1074/jbc.M804525200. PMC 3259902. PMID 18842583.
- ^ Stefanis, Leonidas (Feb 2012). "α-Synuclein in Parkinson's Disease". Cold Spring Harbor Perspectives in Medicine. 2 (2): a009399. doi:10.1101/cshperspect.a009399. ISSN 2157-1422. PMC 3281589. PMID 22355802.
- ^ "Glycosylation as an Inhibitor of Alpha-synuclein Aggregation". The Michael J. Fox Foundation for Parkinson's Research Parkinson's Disease. Retrieved 2020-06-05.
- ^ Ryu, In-Hyun; Do, Su-Il (2011-04-29). "Denitrosylation of S-nitrosylated OGT is triggered in LPS-stimulated innate immune response". Biochemical and Biophysical Research Communications. 408 (1): 52–57. doi:10.1016/j.bbrc.2011.03.115. ISSN 1090-2104. PMID 21453677.
- ^ Wang, Qiming; Fang, Peining; He, Rui; Li, Mengqi; Yu, Haisheng; Zhou, Li; Yi, Yu; Wang, Fubing; Rong, Yuan; Zhang, Yi; Chen, Aidong (2020-04-15). "O-GlcNAc transferase promotes influenza A virus–induced cytokine storm by targeting interferon regulatory factor–5". Science Advances. 6 (16): eaaz7086. Bibcode:2020SciA....6.7086W. doi:10.1126/sciadv.aaz7086. ISSN 2375-2548. PMC 7159909. PMID 32494619.
- ^ Kaspar, AA; Reichert, JM (Sep 2013). "Future Directions for Peptide Therapeutics Development". Drug Discovery Today. 18 (17–18): 807–17. doi:10.1016/j.drudis.2013.05.011. PMID 23726889.
- ^ Levine, PM; Balana, AT; Sturchler, E; Koole, C; Noda, H; Zarzycka, B; Daley, EJ; Truong, TT; Katritch, V; Gardella, TJ; Wootten, D; Sexton, PM; McDonald, P; Pratt, MR (2019-09-11). "O-GlcNAc Engineering of GPCR Peptide-Agonists Improves Their Stability and in Vivo Activity". Journal of the American Chemical Society. 141 (36): 14210–14219. doi:10.1021/jacs.9b05365. PMC 6860926. PMID 31418572.
추가 읽기
- Zachara, Natasha; Akimoto, Yoshihiro; Hart, Gerald W. (2015), Varki, Ajit; Cummings, Richard D.; Esko, Jeffrey D.; Stanley, Pamela (eds.), "The O-GlcNAc Modification", Essentials of Glycobiology (3rd ed.), Cold Spring Harbor Laboratory Press, PMID 28876858.