DNA합성
DNA synthesisDNA 합성은 디옥시리보핵산(DNA) 분자의 자연적 또는 인위적인 생성이다.DNA는 반복구조에서 공유결합과 수소결합으로 연결된 뉴클레오티드 단위로 이루어진 고분자이다.DNA 합성은 이러한 뉴클레오티드 단위들이 DNA를 형성하기 위해 결합될 때 일어난다; 이것은 인공적으로 또는 자연적으로 일어날 수 있다.뉴클레오티드 단위는 질소 염기(시토신, 구아닌, 아데닌 또는 티민), 펜토오스 당(디옥시리보스) 및 인산기로 구성됩니다.각 단위는 그 인산기와 다음 뉴클레오티드의 펜토스당 사이에 공유결합이 형성될 때 결합되어 당-인산 골격을 형성한다.DNA는 특정 염기쌍(아데닌과 티민, 구아닌과 시토신)이 뉴클레오티드 염기 사이에 수소 결합이 형성될 때 자연적으로 발생하기 때문에 상보적인 이중 가닥 구조이다.
DNA 합성에 대한 몇 가지 다른 정의가 있다: DNA 복제 - DNA 생합성(체외 DNA 증폭), 중합효소 연쇄 반응 - 효소적 DNA 합성(체외 DNA 증폭) 또는 유전자 합성이 물리적으로 인공 유전자 서열을 생성하는 것을 나타낼 수 있다.각 합성 유형은 매우 다르지만 몇 가지 특징을 공유합니다.폴리뉴클레오티드를 형성하기 위해 결합된 뉴클레오티드는 DNA 합성의 한 형태인 PCR이 발생하기 위한 DNA 템플릿 역할을 할 수 있다.DNA 복제는 또한 DNA 템플릿을 사용하여 작동하며, DNA 이중 나선은 복제하는 동안 풀리고, 새로운 뉴클레오티드를 위한 짝을 이루지 않은 염기를 수소 결합에 노출시킵니다.그러나 유전자 합성은 DNA 템플릿이 필요하지 않으며 유전자는 새로 조립된다.
DNA 합성은 일부 바이러스뿐만 아니라 모든 진핵 생물과 원핵 생물에서 일어난다.DNA에 대한 돌연변이를 피하기 위해서는 DNA의 정확한 합성이 중요하다.인간의 경우 돌연변이는 암과 같은 질병으로 이어질 수 있기 때문에 DNA 합성과 관련된 생체 내 기계들은 수십 년 동안 광범위하게 연구되어 왔다.미래에는 이러한 연구가 데이터 저장에 사용될 DNA 합성과 관련된 기술을 개발하는 데 사용될 수 있다.
DNA복제
자연에서, DNA 분자는 DNA 복제 과정을 통해 모든 살아있는 세포에 의해 합성된다.이것은 일반적으로 세포 분열의 일부로 발생합니다.DNA 복제가 일어나므로 세포 분열 중에 각 딸 세포는 세포의 유전 물질에 대한 정확한 복사본을 가지고 있다.생체내 DNA 합성(DNA 복제)은 세포주기의 S상 동안 조화롭게 작용하도록 진화한 복잡한 효소 세트에 의존한다.진핵생물과 원핵생물 모두에서, DNA 복제는 특정 토포이소머라아제, 헬리케이스, 자이라아제(복제 개시 단백질)가 이중 가닥 DNA를 풀어서 질소 [1]염기를 노출시킬 때 일어난다.이 효소들은 보조 단백질과 함께 DNA 배열의 정확한 복제를 보장하는 고분자 기계를 형성합니다.상보적인 염기쌍이 일어나 새로운 이중사슬 DNA 분자를 형성합니다.이것은 새로운 DNA 분자의 한 가닥이 '부모' 가닥에서 나온 것이기 때문에 반 보존적 복제로 알려져 있다.
지속적으로, 진핵생물 효소는 DNA 복제를 교란시킬 수 있는 DNA 손상과 마주친다.이 손상은 자연적으로 발생하거나 DNA 손상 물질에 의해 발생하는 DNA 병변의 형태입니다.따라서 [2]손상을 입었을 때 붕괴를 방지하기 위해 DNA 복제기구를 고도로 제어한다.DNA 복제 시스템의 제어는 게놈이 주기당 한 번만 복제되도록 보장합니다. 과잉 복제는 DNA 손상을 유발합니다.DNA 복제의 규제 완화는 암 [3]발생 시 게놈 불안정성의 핵심 요인이다.
이것은 생체 내 DNA 합성 기계의 특이성을 강조한다.자연적으로 발생하는 DNA의 복제를 인위적으로 자극하거나 인공 유전자 서열을 만들기 위한 다양한 수단이 존재한다.그러나 시험관 내 DNA 합성은 오류가 발생하기 쉬운 과정일 수 있다.
DNA복구합성
손상된 DNA는 여러 가지 효소적 복구 프로세스에 의해 복구되며, 각 개별 프로세스는 특정 유형의 손상을 복구하는 데 특화되어 있습니다.인간의 DNA는 여러 자연적 원천에 의해 손상되기 쉬우며, 부족한 복구는 질병과 조기 [4]노화와 관련이 있다.대부분의 DNA 수복 과정은 수복의 중간 단계 동안 DNA에 단일 가닥 간극을 형성하며, 이러한 간극은 수복 [4]합성에 의해 채워진다.DNA 합성에 의한 갭 메우기가 필요한 특정 복구 프로세스에는 뉴클레오티드 절제 복구, 염기 절제 복구, 불일치 복구, 상동 재조합 복구, 비상동 말단 결합 및 마이크로 호몰로지 매개 말단 결합이 포함된다.
역문자 변환
역전사는 레트로바이러스를 포함한 특정 바이러스군의 복제 주기의 일부입니다.그것은 역전사효소 효소를 사용하여 RNA를 이중가닥보완DNA(cDNA)로 복사하는 것을 포함한다.레트로바이러스에서는 바이러스 RNA가 숙주세포핵에 삽입된다.여기서, 바이러스 역전사효소 효소는 RNA 배열에 DNA 뉴클레오티드를 첨가하여, 바이러스 단백질을 [5]코드하는 효소 인테그레이스에 의해 숙주 세포 게놈에 삽입되는 cDNA를 생성한다.
중합효소 연쇄반응
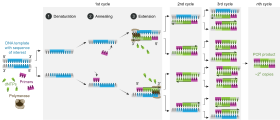
중합효소 연쇄반응은 DNA 용융 및 DNA의 효소적 복제를 위해 반응의 반복적인 가열 및 냉각 사이클을 사용하여 실험실에서 효소적 DNA 합성의 한 형태이다.
PCR 중의 DNA 합성은 살아있는 세포와 매우 유사하지만 매우 특정한 시약과 조건을 가지고 있다.PCR 중에 숙주 샤페론 단백질로부터 화학적으로 DNA를 추출한 후 가열하여 DNA 가닥의 열해리를 일으킨다.두 개의 새로운 cDNA 가닥이 원래 가닥에서 생성되며, 이러한 가닥은 다시 분할되어 향후 PCR 제품의 템플릿 역할을 할 수 있습니다.원래 DNA는 여러 차례의 [1]PCR을 통해 증식된다.원본 DNA 가닥의 10억 개 이상의 복사본이 만들어질 수 있다.
무작위 돌연변이 유발
구조 및 진화 연구와 같은 많은 실험을 위해, 과학자들은 특정한 DNA 배열의 변형에 대한 큰 라이브러리를 생산할 필요가 있다.무작위 돌연변이 유발은 체외에서 일어나며, 저충실도 DNA 중합효소에 의한 돌연변이 유발 복제가 선택적 PCR 증폭과 결합되어 돌연변이 [6]DNA의 많은 복사를 생성한다.
RT-PCR
RT-PCR은 템플릿 DNA가 아닌 mRNA에서 cDNA를 합성한다는 점에서 기존 PCR과 다르다.이 기술은 RNA 배열이 효소인 역전사효소의 템플릿 역할을 하기 때문에 역전사 반응을 PCR 기반 증폭과 결합합니다.RT-PCR은 다양한 발달 단계에서 특정 조직이나 세포 유형에서 유전자 발현을 테스트하거나 유전적 [7]장애를 테스트하기 위해 종종 사용된다.
유전자 합성
인공 유전자 합성은 초기 템플릿 DNA 샘플 없이 체외에서 유전자를 합성하는 과정이다.2010년 J. Craig Venter와 그의 팀은 마이코플라스마 [8]라보토리엄이라고 불리는 자기 복제 미생물을 만들기 위해 완전히 합성된 DNA를 최초로 사용했다.
올리고뉴클레오티드합성
올리고뉴클레오티드 합성은 핵산 배열의 화학적 합성이다.대부분의 생물 연구와 생물 공학은 올리고뉴클레오티드, 합성 유전자, 또는 심지어 염색체를 포함할 수 있는 합성 DNA를 포함한다.오늘날, 모든 합성 DNA는 마빈 H. Caruthers의 인광 아미다이트 방법을 사용하여 맞춤 제작되었습니다.올리고는 천연 염기를 복제하는 블록으로부터 합성된다.이 과정은 1970년대 후반부터 자동화되었으며 바람직한 유전자 염기서열을 형성하고 의학 및 분자생물학에서 다른 용도로 사용될 수 있다.그러나 화학적으로 시퀀스를 작성하는 것은 200~300개의 염기를 넘어서는 비현실적이며 환경적으로 위험한 프로세스입니다.약 200개의 염기로 이루어진 이 올리고들은 더 큰 DNA [9]분자를 만들면서 DNA 조립 방법을 사용하여 연결될 수 있습니다.
일부 연구는 템플릿이 필요 없는 DNA 중합효소인 말단 디옥시뉴클레오티딜전달효소(TdT)를 사용하여 효소 합성의 가능성을 탐구했다.그러나 이 방법은 아직 화학 합성만큼 효과적이지 않으며 상업적으로도 [10]이용되지 않습니다.
인공 DNA 합성이 발전함에 따라, DNA 데이터 저장의 가능성이 탐색되고 있다.초고밀도 스토리지와 장기적인 안정성을 갖춘 합성 DNA는 대량의 데이터를 저장할 수 있는 흥미로운 옵션입니다.차세대 염기서열 분석 기술을 통해 DNA에서 매우 빠르게 정보를 검색할 수 있지만 DNA의 새로운 합성은 이 과정에서 주요 병목 현상이다.한 사이클에 하나의 뉴클레오티드만 첨가될 수 있고, 각각의 사이클은 몇 초가 걸리기 때문에, 전체적인 합성은 매우 시간이 걸릴 뿐만 아니라 오류가 발생하기 쉽다.그러나 생명공학이 발전하면 언젠가는 합성 DNA가 데이터 [11]저장에 사용될 수 있다.
염기쌍 합성
A-T(아데닌 - 티민), G-C(구아닌 - 시토신)와 더불어 새로운 핵염기쌍을 합성할 수 있는 것으로 보고되었다.합성 뉴클레오티드는 유전자 알파벳을 확장하고 DNA 부위의 특정 변형을 허용하기 위해 사용될 수 있다.심지어 세 번째 염기쌍만 있어도 DNA로 암호화될 수 있는 아미노산의 수가 기존의 20개의 아미노산에서 [8]172개로 늘어날 것이다.하치모지 DNA는 8개의 뉴클레오티드 문자로 만들어져 4개의 가능한 염기쌍을 형성한다.그러므로 그것은 자연 DNA의 정보 밀도를 두 배로 높인다.연구에서 RNA는 하치모지 DNA에서 만들어지기도 했다.이 기술은 [12]DNA에 데이터를 저장하는 데도 사용될 수 있습니다.
레퍼런스
- ^ a b Pelt-Verkuil, Evan (2008). "A Brief Comparison Between In Vivo DNA Replication and In Vitro PCR Amplification". Principles and Technical Aspects of PCR Amplification (PDF). Rotterdam: Springer Netherlands. pp. 9–15. doi:10.1007/978-1-4020-6241-4_2. ISBN 978-1-4020-6240-7. S2CID 215257488.
- ^ Patel, Darshil R.; Weiss, Robert S. (2018). "A tough row to hoe: when replication forks encounter DNA damage". Biochem Soc Trans. 46 (6): 1643–1651. doi:10.1042/BST20180308. PMC 6487187. PMID 30514768.
- ^ Reusswig, Karl-Uwe; Pfander, Boris (2019). "Control of Eukaryotic DNA replication Initiation - Mechanisms to Ensure Smooth Transitions". Genes (Basel). 10 (2): 99. doi:10.3390/genes10020099. PMC 6409694. PMID 30700044.
- ^ a b 티와리 5번가, 윌슨 DM 3번가질병과 조기 노화의 DNA 손상 및 관련 DNA 복구 결함.Am J Hum Genet. 2019년 8월 1일;105 (2):237-257.doi:10.1016/j.ajhg.2019.06.005.검토.PMID 31374202
- ^ Hughes, Stephen H (2015). "Reverse Transcription of Retroviruses and LTR Retrotransposons". Microbiology Spectrum. 3 (2): 1051–1077. doi:10.1128/microbiolspec.MDNA3-0027-2014. ISBN 9781555819200. PMC 6775776. PMID 26104704.
- ^ Forloni, M (2018). "Random Mutagenesis Using Error-prone DNA Polymerases". Cold Spring Harbor Protocols. 2018 (3): pdb.prot097741. doi:10.1101/pdb.prot097741. PMID 29496818.
- ^ Bachman, Julia (2013). "Chapter Two - Reverse-Transcription PCR (RT-PCR)". Methods in Enzymology. 530: 67–74. doi:10.1016/B978-0-12-420037-1.00002-6. PMID 24034314.
- ^ a b Fikes, Bradley J. (May 8, 2014). "Life engineered with expanded genetic code". San Diego Union Tribune. Archived from the original on 9 May 2014. Retrieved 8 May 2014.
- ^ Palluk, Sebastian; Arlow, Daniel H; et al. (2018). "De novo DNA synthesis using polymerase-nucleotide conjugates". Nature Biotechnology. 36 (7): 645–650. doi:10.1038/nbt.4173. OSTI 1461176. PMID 29912208. S2CID 49271982.
- ^ Perkel, Jeffrey M. (2019). "The race for enzymatic DNA synthesis heats up". Nature. 566 (7745): 565. doi:10.1038/d41586-019-00682-0. PMID 30804572.
- ^ Tabatabaei, S. Kasra (2020). "DNA punch cards for storing data on native DNA sequences via enzymatic nicking". Nature Communications. 11 (1): 1742. doi:10.1038/s41467-020-15588-z. PMC 7142088. PMID 32269230.
- ^ Hoshika, Shuichi (2020). "Hachimoji DNA and RNA. A Genetic System with Eight Building Blocks". Science. 363 (6429): 884–887. doi:10.1126/science.aat0971. PMC 6413494. PMID 30792304.