조형
Formylation생화학에서 포밀 기능 그룹의 추가는 포밀화라고 불린다. 포밀 기능성 그룹은 수소에 접합된 카보닐로 구성된다. R 그룹에 붙으면 포밀 그룹을 알데히드라고 부른다.
형성은 몇 가지 중요한 생물학적 과정에서 확인되었다. 메티오닌은 1964년[1] 마르커와 생거에 의해 대장균에서 분무된 것이 처음 발견되었고, 이후 박테리아와 오르가넬에서 단백질 합성을 시작하는 데 관여하는 것으로 확인되었다.[2] N-포르밀메티오닌의 형성은 메티오닐-tRNA transformylase 효소에 의해 촉매된다.[3] 또한, 두 가지 포뮬레이션 반응은 청개의 디노보 생합성에서 발생한다. 이러한 반응은 글리시나미드 리보뉴클레오티드(GAR) transformylase와 5-aminoimidazole-4-carboxyamide 리보타이드(AICAR) transformyelase에 의해 촉매로 작용한다.[4] 더 최근에, 포밀화는 유전자 발현을 변형시킬 수 있는 히스톤 변형인 것으로 밝혀졌다.
일반 포뮬레이션 반응
Formylation은 화합물이 Formyl 그룹(-CH=O)으로 기능하는 모든 화학적 과정을 말한다. 유기 화학에서 이 용어는 방향족 화합물과 관련하여 가장 일반적으로 사용된다(예: 개터만-코흐 반응에서 벤츠알데히드로 벤젠 변환). 생화학에서는 포밀전달효소와 같은 효소에 의해 반응이 촉매된다.
생물학에서의 형성 반응
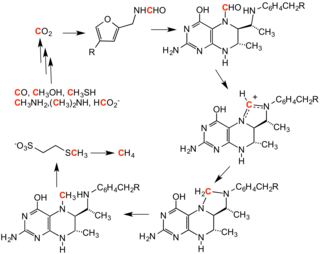
메타노제시스
메탄노푸란의 형성은 메탄노제시스 사이클을 개시한다. 포밀 그룹은 이산화탄소에서 파생되어 메탄으로 변환된다.
단백질 합성에서의 형성
박테리아와 오르가넬에서 단백질 합성의 시작은 formyl-methionyl-tRNA(tRNA)의 형성에 의해 신호를 받는다.fMet 이 반응은 10-포밀테트라하이드로폴리스와 메티오닐-tRNA 포밀전달효소에 의존한다.[3] 이 반응은 eukaryotes나 Acrocia에 의해 사용되지 않는데, 이는 비 박테리아 세포에 tRNA의 존재를 침입 물질로 칭하고 신속하게 제거하기 때문이다. tRNA는 생산 후 단백질 합성을 시작하기 위해 리보솜의 30S 서브유닛에 전달된다. fMet은 메티오닌과 같은 코돈 배열을 가지고 있다. 그러나 fMet은 단백질 합성을 시작하는데만 사용되므로 단백질의 N단말에서만 발견된다. 메티오닌은 나머지 번역에 사용된다. 대장균에서 tRNA는 포밀 그룹이 메티오닌의 N-terminus에서 펩타이드 결합 형성을 차단하기 때문에 시작 인자 IF-2에 의해 특별히 인식된다.[3]
단백질 합성이 완료되면 메티오닌의 포밀군을 펩타이드 변형효소에 의해 제거할 수 있다. 메티오닌 잔류물은 메티오닌아미노펩티아제 효소에 의해 추가로 제거될 수 있다.
푸린 생합성에서의 거푸집중
푸린 리보뉴클레오티드 AMP와 GMP의 전구체인 이노신 모노포스인산염(IMP)의 11단계 합성에 두 가지 포뮬레이션 반응이 필요하다. 글리시나미드 리보뉴클레오티드(GAR) 변환효소는 GAR의 포뮬레이션을 촉진하여 경로의 네 번째 반응에서 포뮬글리시나미딘 리보타이드(FGAR)를 형성한다. de novo purine 생합성성의 penultimate 단계에서 5-aminoimidazole-4-carboxyamide 리보타이드(AICAR)는 AICAR transformyzilase에 의해 5-formaminoimidazole-4-carboxamide 리보타이드(FAICAR)로 공식화된다.[4]
GAR transformylase
PurN GAR transformylase는 eukaryotes와 prokaryotes에서 발견된다. 그러나 두 번째 GAR 변환효소인 PurT GAR 변환효소는 대장균에서 확인되었다. 두 효소는 시퀀스 보존이 없고 서로 다른 포밀 기증자를 필요로 하지만, 특정 활동과 Km for GAR은 PurT와 PurN GAR transformylase 모두에서 동일하다.
PurN GAR transformylase
PurN GAR transformylase 1CDE는 코엔자임 N10-formyltetrahydrofolate(N10-formyl-THF)를 Formyl 기증자로 사용하여 GAR의 α-아미노 그룹을 형성한다. eukaryotes에서 PurN GAR transformylase는 큰 다기능성 단백질의 일부지만 원핵생물에서 단일 단백질로 발견된다.[6]
메커니즘
포밀화 반응은 GAR 핵물질의 아민 그룹이 N10-포밀-THF를 공격하여 사면체 중간을 생성하는 직접 전달 반응을 통해 발생하도록 제안된다.[4] GAR의 α-아미노 그룹은 비교적 반응성이 높기 때문에 용제에 의해 핵소화 현상이 발생하는 것을 제안한다. 활성 사이트에서는 Asn 106, His 108 및 Asp 144가 포밀 이송을 지원하기 위해 배치된다.[6] 그러나 돌연변이 유발 연구에서는 두 개 이상의 잔류물의 돌연변이만이 효소를 억제하기 때문에 이러한 잔류물이 개별적으로 촉매분석에 필수적이지는 않다는 것을 밝혀냈다. 음전하를 띤 아스프144는 His108의 pKa를 증가시켜 His108의 양성자 이미다졸륨 그룹이 N10-포밀-THF 포밀 그룹의 전기영양성을 향상시킬 수 있다고 생각된다. 또한 His108과 Asn106은 전환 상태에서 형성되는 옥시아니온을 안정시키는 것으로 여겨진다.[7]
PurT GAR transformylase
PurT GAR transformylase는 Formyl 기증자로서 포메이테이트와 ATP를 필요로 한다. PurT GAR transformylase는 대장균에서 GAR 포뮬레이션의 14-50%를 수행하는 것으로 추정되었다. 이 효소는 ATP-그래프 단백질의 슈퍼 패밀리의 일원이다.[8]
메커니즘
PurT GAR transformylase에 대해 순차적 메커니즘이 제안되었으며, 여기서 단명 포밀 인산염 중간이 첫 번째 형태에 제안되었다. 이 포밀 인산염 중간은 포밀 그룹의 전달을 위해 GAR 아민의 핵포밀 공격을 받는다. 돌연변이 PurT GAR 수혈밀라아제가 형성에 약한 친화력을 보인 돌연변이 유발 실험에서 포밀 인산염 중간이 검출됐다.[6] Formyl phosphate, ADP, GAR로 PurT GAR transformyase를 배양하면 ATP와 FGAR가 모두 생성된다. 이는 효소의 포밀화 반응을 수행하는 것이 운동학적으로나 화학적으로 유능하기 때문에 포밀인산염은 중간일 수 있음을 더욱 나타낸다.[9] 포밀인산염 매개체 앞에 있는 효소 인산염 매개체도 위치 동위원소 교환 연구에 기초하여 형성하도록 제안되었다.[9] 그러나 구조 데이터는 포밀인산 중간을 형성하기 위해 효소의 활성 부위에서 ATP의 of-인산염에 대한 직접 공격을 위해 포메이트가 위치할 수 있음을 나타낸다.[8]
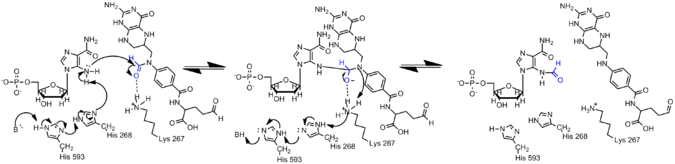
AICAR transformylase
AICAR transformylase는 FAICAR에 대한 AICAR의 포뮬레이션을 위해 포밀 공여자로서 코엔자임 N10-포밀테트라하이드로폴(N10-포밀-THF)을 요구한다. 단, AICAR transformylase와 GAR transformylase는 높은 시퀀스 유사성 또는 구조 호몰로지를 공유하지 않는다.[7]
메커니즘
AICAR의 아민은 결합을 통한 AICAR의 전자의 소산화로 인해 GAR의 아민보다 훨씬 적은 핵소성 물질이다. 따라서 형성 반응이 일어나려면 Aircar의 N5 핵분열체가 활성화되어야 한다. 히스티딘 268과 리신 267은 카탈루션에 필수적인 것으로 밝혀졌으며 모든 AICAR transformylase에 보존되어 있다. 히스티딘 268은 AICAR의 N5 핵소포체의 감응에 관여하는 반면, 리신 267은 사면중간의 안정화를 위해 제안된다.[7]
히스톤 단백질의 형성
ε-형식화는 히스톤 단백질에서 일어나는 많은 변환 후 수정 중 하나로, 염색질 순응과 유전자 활성화를 변조하는 것으로 나타났다.
히스톤과 단백질의 라이신 잔류물의 Nε에서 포뮬레이션이 확인되었다. 이 수정은 링커 히스톤과 높은 이동성 그룹 단백질에서 관찰되었으며, 매우 풍부하며, 염색질 기능의 후생유전학에서 역할을 하는 것으로 생각된다. 성형된 라이신은 DNA 결합에 역할을 하는 것으로 밝혀졌다. 또한 아세틸화 및 메틸화라고도 알려진 히스톤 라이신에서도 포밀화가 검출되었다. 따라서 공식화는 다른 변환 후 변경을 차단할 수 있다.[10] 히스톤 H1의 19개 수정 사이트에서 포뮬레이션이 가장 자주 검출된다. 세포의 유전적 발현이 형성됨으로써 매우 교란되어 암과 같은 질병을 일으킬 수 있다. 이러한 변조의 발달은 산화적 스트레스 때문일 수 있다.[10]
히스톤 단백질에서 리신은 일반적으로 히스톤 아세틸-전송(HATs)과 히스톤 디아세틸라제(HDAC 또는 KDAC)에 의해 수정된다. 리신의 아세틸화는 특정 유전자의 조절과 발현에 기본이다. 산화 스트레스는 포밀인산염종의 높은 반응성으로 인해 포밀-리신 형성에 의해 아세틸-리신이 빠르게 경쟁할 수 있는 상당히 다른 환경을 만들어낸다. 이 상황은 현재 산화 DNA 손상에 의한 것으로 추정된다. 산화적으로 손상된 DNA에 크게 의존하며 주로 세포 내 급진적인 화학에 의해 구동되는 포밀인산염 형성을 위한 메커니즘이 제안되었다.[11] 생산되는 포밀인산염은 리신을 형성하는 데 사용될 수 있다. 산화 응력은 단백질 표면에서 리신 잔류물의 가용성과 형성 가능성에 영향을 미치는 것으로 생각된다.
의학의 형성
약물 대상으로서의 포뮬레이션
푸린 생합성에 관여하는 효소의 억제는 화학요법의 잠재적인 약물 표적으로 이용되어 왔다.
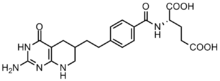
암세포는 분열을[6] 용이하게 하기 위해 고농도의 청바지를 필요로 하며, 뉴클레오티드 인양 경로보다는 드 노보 합성에 의존하는 경향이 있다.[7][12] GAR transformylase와 AICAR transformylase에 의한 형성 반응을 억제하기 위해 여러 엽산 기반 억제제가 개발되었다.[13] 최초의 GAR 변환효소 억제제 로메트렉솔[(6R)5,10-dideazatetrahydrofolate]은 Eli Lilly와 학술 연구소의 협력을 통해 1980년대에 개발되었다.[14]
구조는 N10-포밀-THF와 유사하지만, 로메트렉솔은 탄소 전달 반응을 한 번 수행할 수 없다.[13] 또한 GAR transformylase의 여러 GAR 기반 억제제도 합성되었다.[13] 억제제 역시 효소 친화력을 높이기 위해 세포에 들어간 후 단글루타민산 엽산 및 항이폴레이트에 ut-글루타민산을 첨가하는 효소 폴리글루타마이트 싱타제를 억제제로서 억제제 개발이 특히 어려운 것으로 밝혀졌다. 이러한 친화력이 증가하면 고엽제 저항으로 이어질 수 있다.[12]
레이 신드롬
레이 증후군은 효소 형성 반응의 결함과 관련이 있는 신경퇴행성 질환이다. 레이 증후군은 일반적으로 미토콘드리아에서 발생하는 산화 인산화 결함과 관련이 있다.[15] Exome 시퀀싱은 레이 증후군 환자의 미토콘드리아 메티오닐-tRNA 포밀전달효소(MTFMT)에 대한 유전자 코딩의 돌연변이를 식별하기 위해 사용되어 왔다. 리 증후군의 MTFMT에서 확인된 c.626C>T 돌연변이는 엑손 스플라이싱을 변화시켜 프레임스위트 돌연변이와 조기 정지 코돈으로 이어지는 것으로 생각된다. MTFMT c.626C>T 돌연변이를 가진 개인은 fMet-tRNAMet 수준을 감소시키고 미토콘드릭하게 번역된 COX1의 형성 수준의 변화를 감소시킨 것으로 밝혀졌다. 이 링크는 특정 미토콘드리아 유전자에 대한 발현을 시작할 때 합성 메티오닌의 필요성에 대한 증거를 제공한다.[16]
참조
- ^ Marcker, K; Sanger, F. (1964). "N-formyl-methionyl-S-RNA". J. Mol. Biol. 8 (6): 835–840. doi:10.1016/S0022-2836(64)80164-9. PMID 14187409.
- ^ Adams, J.M.; Capecchi, M.R. (1966). "N-Formylmethionyl-sRNA as the initiator of protein synthesis". PNAS. 55 (1): 147–155. Bibcode:1966PNAS...55..147A. doi:10.1073/pnas.55.1.147. PMC 285768. PMID 5328638.
- ^ a b c Kozak, M (1983). "Comparison of Initiation of Protein synthesis in Procaryotes, Eucaryotes, and Organelles". Microbiological Reviews. 47 (1): 1–45. doi:10.1128/MMBR.47.1.1-45.1983. PMC 281560. PMID 6343825.
- ^ a b c Voet and Voet (2008). Fundamentals of Biochemistry 3rd edition. New York: Wiley.
- ^ Thauer, R. K. (1998). "Biochemistry of Methanogenesis: a Tribute to Marjory Stephenson". Microbiology. 144: 2377–2406. doi:10.1099/00221287-144-9-2377. PMID 9782487.
- ^ a b c d Warren, M.S.; K.M. Mattia; A.E. Marolewski; S.J. Benkovic (1996). "The transformylase enzymes of de novo purine biosynthesis" (PDF). Pure Appl. Chem. 68 (11): 2029–2036. doi:10.1351/pac199668112029. S2CID 39555269. Retrieved 24 February 2013.
- ^ a b c d Wolan, D; Greasley, S.E.; Beardsley, P.; Wilson, I.A. (2002). "Structural Insights into the Avian AICAR Transformylase Mechanism". Biochemistry. 41 (52): 15505–15513. doi:10.1021/bi020505x. PMID 12501179.
- ^ a b Thoden, J.B.; Firestine, S.; Nixon, A.; Benkovic, S.J.; Holden, H.M (2000). "Molecular Structure of Escherichia coli PurT-Encoded Glycinamide Ribonucleotide Transformylase". Biochemistry. 39 (30): 8791–8802. doi:10.1021/bi000926j. PMID 10913290.
- ^ a b Marolewski, A.E.; Mattia, K.M.; Warren, M.S.; Benkovic, S.J. (1997). "Formyl phosphate: a proposed intermediate in the reaction catalyzed by Escherichia coli PurT GAR transformylase". Biochemistry. 36 (22): 6709–6716. doi:10.1021/bi962961p. PMID 9184151.
- ^ a b Wisniewski, J.R.; Zougman, A.; Mann, M. (2002). "N-Formylation of lysine is a widespread post-translational modification of nuclear proteins occurring at residues involved in regulation of chromatin function". Nucleic Acids Research. 36 (2): 570–577. doi:10.1093/nar/gkm1057. PMC 2241850. PMID 18056081.
- ^ Jiang, T; Zhou, X.; Taghizadeh, K.; Dong, M.; Dedon, PC. (2007). "N-formylation of lysine in histone proteins as a secondary modification arising from oxidative DNA damage". PNAS. 104 (1): 60–65. Bibcode:2007PNAS..104...60J. doi:10.1073/pnas.0606775103. PMC 1765477. PMID 17190813.
- ^ a b DeMartino, J.K.; Hwang, I.; Xu, L.; Wilson, I.A.; Boger, D.L. (2006). "Discovery of a Potent, Nonpolyglutamatable Inhibitor of Glycinamide Ribonucleotide Transformylase". Journal of Medicinal Chemistry. 49 (10): 2998–3002. doi:10.1021/jm0601147. PMC 2531195. PMID 16686541.
- ^ a b c Christopherson, R.I.; Lyons, S.D.; Wilson, P.K (2002). "Inhibitors of de Novo Nucleotide Biosynthesis as Drugs". Acc. Chem. Res. 35 (11): 961–971. doi:10.1021/ar0000509. PMID 12437321.
- ^ Wang, L; Desmoulin, S.K.; Cherian, C.; Polin, L.; White, K.; Kushner, J.; Fulterer, A.; Chang, M.; Mitchell, S.; Stout, M.; Romero, M.F.; Hou, Z.; Matherly, L.H.; Gangjee, A (2011). "Synthesis, biological and antitumor activity of a highly potent 6-substituted pyrrolo[2,3-d]pyrimidine thienoyl antifolate inhibitor with proton-coupled folate transporter and folate receptor selectivity over the reduced folate carrier that inhibits β-glycinamide ribonucleotide formyltransferase". Journal of Medicinal Chemistry. 54 (20): 7150–7164. doi:10.1021/jm200739e. PMC 3209708. PMID 21879757.
- ^ "Leigh Syndrome". Online Mendelian Inheritance in Man. Retrieved 24 February 2013.
- ^ Tucker EJ, Hershman SG, Köhrer C, Belcher-Timme CA, Patel J, Goldberger OA, Christodoulou J, Silberstein JM, McKenzie M, Ryan MT, Compton AG, Jaffe JD, Carr SA, Calvo SE, RajBhandary UL, Thorburn DR, Mootha VK (2011). "Mutations in MTFMT underlie a human disorder of formylation causing impaired mitochondrial translation". Cell Metab. 14 (3): 428–434. doi:10.1016/j.cmet.2011.07.010. PMC 3486727. PMID 21907147.