LIG-I형 수용체
RIG-I-like receptorRIG형 수용체(Retinoic acid-induced gene-I형 수용체, RLRs)는 선천적인 면역체계에 의한 바이러스 인식에 관여하는 세포내 패턴인식 수용체의 일종이다.[1][2]RLR(Retino-Acid indurable gene 또는 DDX58)은 RIG-I like receptor(RLR) 계열 내에서 가장 잘 특성화된 수용체다.MDA5(멜라노마 분화 관련 5), LGP2(유전학과 생리학2의 실험실)와 함께 이 세포질 패턴인식 수용체(PRRs) 계열은 바이러스 감염의 산물인 세포내 바이러스 RNA를 위한 송신소다.RLR 수용체는 대부분의 조직에서 바이러스 감염에 대한 최전방 방어 기능을 제공한다.
RLR 리간즈
RIG-I 수용체는 5' 트라이인산 및 폴리-우리딘 리치 RNA 모티브와 같은 추가 모티브를 가진 짧은 (<2000 bp) 단일 또는 이중 가닥 RNA를 결합하는 것을 선호한다.[3][4]RIG-I는 파라믹소바이러스(예: 홍역), 횡두바이루스(예: 배시성 구내염 바이러스), 정형외과바이러스(예: 인플루엔자 A) 등 다양한 제품군의 RNA 바이러스에 대한 면역 반응을 유발한다.[5][6][7][8][9]MDA5 리간드는 특성이 좋지 않지만 피코나바이러스 감염 세포에서 발견되는 피코나바이러스 RNA의 복제 형태 등 긴 이중 가닥 RNA(>2000 bp)를 선호한다.[10][11]LGP2는 가변 길이의 무뚝뚝한 이중 가닥 RNA와 결합하고,[12][13] 또한 필라멘트 형성을 조절하기 위해 RNA 결합 MDA5에도 결합한다.[14]후자는 MDA5에 따른 피코르나비루스(예: 뇌심막염 바이러스)를 LGP2가 인정한 것과 관련이 있다.[15]
구조 특성
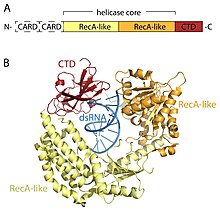
RLR 수용체는 DEAD-box(SF2) 헬리캐아제 계열(가족의 DEAD 모티브 특성이 아닌 DExD/H 모티브를 포함함에도 불구하고)의 구성원으로 공통 도메인 아키텍처를 공유한다.모두 두 개의 RecA와 유사한 영역으로 구성된 촉매 헬리코아제 코어를 포함하고 있다.The catalytic helicase core contains at least 9 highly conserved sequence motifs that coordinate ATP and RNA binding and the hydrolysis of ATP to unwind RNA. A C-terminal domain (CTD; InterPro: IPR021673) follows the helicase core and this domain also binds viral RNA. Distinct RNA-binding loops within the CTD of the three RLRs dictate the type of그들이 묶을 수 있는 RNA.[16]헬리카아제 코어 및 CTD 외에도, LIG-I와 MDA5에는 다운스트림 신호의 시작에 필수적인 2개의 N-터미널 CARD(Caspase 활성채용 도메인)가 있다.LGP2는 CARD 신호 도메인이 없고 대신 RIG-I와 MDA5의 양성 및 음성 조절기 역할을 하기 때문에 RIG-I와 MDA5 모두와 다르다.[14][17][18][19][20][15][21]
신호 활성화
바이러스성 RNA RIG-I가 없는 비감염 세포에는 CTD와의 상호 작용으로 인해 CARD 도메인이 마스크되는 비활성 순응으로 존재한다.[22]RNA를 결합하면, RIG-I는 CARD 도메인이 노출되고 신호에 '사용 가능한' 순응으로 변한다.반대로, MDA5 CARD는 바이러스 RNA가 없을 때 숨김이 없다.[23] RLR 활성화를 위한 안전장치로서 노출된 RG-I와 MDA5 CARD는 다운스트림 신호를 긍정적이거나 부정적으로 조절하는 변환 후 수정(예: 편재, 인산화)을 겪을 수 있다.
RIG-I 항바이러스 신호
활성 상태에서 노출된 RIG-I CARD 도메인은 미토콘드리아의 외부 표면에 위치한 MAVS(Mitochondrial 항바이러스 신호 단백질, IPS-1, VISA 또는 Cardif라고도 한다)의 CARD 도메인과 상호작용한다.이 결합 이벤트는 MAVS가 TRAF3(TNF 수용체 관련 인자 3)와 그에 따라 IKKε/TBK1(I-kinapa-B kinase-엡실론/KANK-binding kinase 1) 복합체를 모집하는 대형 기능 골재를 형성하게 하기 때문에 신호에 필수적이다.IKKε/TBK1 복합체는 IRF3(Interferon Regulation Factor 3) 및 IRF7의 전사로 이어져 IRF3(IFNα 및 IFNβ 포함)과 III형 인터페론(IFN)을 유도한다.IFN형식은 IFN 수용체를 생산한 세포의 표면에 IFN 수용체를 결합하고 수용체를 표현하는 다른 세포형태를 결합하여 JAK-STAT(Janus kinase/신호 변환기와 전사 활성제) 신호를 활성화한다.이를 통해 IFN 반응을 증폭시키는 수백 개의 인터페론 자극 유전자(ISG)가 유도된다.이것은 전체적으로 감염된 세포의 사망, 주변 세포의 보호, 항원 특유의 항바이러스 면역반응의 활성화를 야기한다.전체적으로 이 조정된 항바이러스 면역 반응은 바이러스 감염을 통제한다.
규정
IFN 생산이 장기화되는 것은 인간 질병과 연관되므로 RLR 신호는 엄격하게 규제되어야 한다.이를 달성하는 다양한 방법 중 하나는 인산염(인산염) 또는 유비퀴틴(유비쿼티션)으로 RLR 신호 단백질을 변환 후 수정하거나 태깅하는 것이다.이 태그들은 또한 제거될 수 있으며, 이것은 RLR 신호에 추가적인 규제 계층을 추가한다.이러한 변환 후 수정 및 제거는 RLR 신호에 널리 적용되며 심지어 LIG-I 수용체 자체를 조절한다.가장 유명한 RIG-I CARD 영역은 신호를 부정적으로 조절하기 위해 휴식 상태에서 단백질 키나제 C-α(PKC-α)와 PKC-β에 의해 인산염화된다.[24][25][26]바이러스 감염 시 RLR-I는 PP1α 및 PP1γ에 의해 인산 제거되며,[27] E3 ligase TRIM25에 의한 RIG-I CARD 도메인의 편재화가 허용되어 RLR 매개 항바이러스 면역반응이 활성화된다.[28]변환 후 수정은 RLR 신호의 활성화와 매우 관련성이 있으므로, 그것들이 각각 인플루엔자 A와[29] 홍역 같은 바이러스의 표적이 되어 신호를 억제한다는 것은 놀라운 일이 아니다.[30]
RLR 신호의 바이러스 납치
바이러스는 생존을 강화하기 위해 RLR 신호를 전복시키는 방법을 발전시켜 왔다.예를 들어 인플루엔자 A 바이러스와 웨스트 나일 바이러스(WNV)는 NS1(비구조 단백질 1) 단백질을 사용하여 TRIM25에 의한 LIG-I 편재화를 차단하거나, 각각 LIG-I 열화를 유발하여 IFN 생성을 억제한다.[29][31]이 결과는 또한 MAVS의 일부를 절개하여 [32]C형 간염(HCV) NS3/4A 단백질과 LGP2를 절개하는 구제역 바이러스(FMDV) 리더 프로테아제(Lpro)에 의해 달성된다.[33]마찬가지로 뎅기바이러스(DENV)는 자신의 NS2B3, NS2A, NS4B 단백질을 사용하여 IKK bind을 바인딩하고 IRF3 인산화[34][35] 및 지카 바이러스에 따라 NS4A 단백질을 바인딩하여 MAVS를 바인딩하여 RLR 수용체 바인딩을 차단한다.[36][37]또 다른 두드러진 예는 MDA5, LGP2, STAT을 포함한 다양한 RLR 또는 다운스트림 신호 단백질을 직접 결합하는 파라믹소바이러스 V 단백질이나 [38][39][40]RLR 신호를 부정적으로 조절하는 PP1α, PP1γ[30] 등의 단백질이다.
참고 항목
참조
![]() 본 기사는 CC BY 4.0 라이센스(2019년)에 따라 다음과 같은 출처에서 개작되었다. Natalie Borg (2019). "RIG-I like receptors" (PDF). WikiJournal of Science. 2 (1): 1. doi:10.15347/WJS/2019.001. ISSN 2470-6345. Wikidata Q62604415.
본 기사는 CC BY 4.0 라이센스(2019년)에 따라 다음과 같은 출처에서 개작되었다. Natalie Borg (2019). "RIG-I like receptors" (PDF). WikiJournal of Science. 2 (1): 1. doi:10.15347/WJS/2019.001. ISSN 2470-6345. Wikidata Q62604415.
- ^ Mahla RS, Reddy MC, Prasad DV, Kumar H (September 2013). "Sweeten PAMPs: Role of Sugar Complexed PAMPs in Innate Immunity and Vaccine Biology". Frontiers in Immunology. 4: 248. doi:10.3389/fimmu.2013.00248. PMC 3759294. PMID 24032031.
- ^ Offermanns S, Rosenthal W (2008-08-14). Encyclopedia of Molecular Pharmacology, Volume 1. Springer. ISBN 9783540389163. Retrieved 30 August 2011.
The other two families of PRRs, the NOD-like receptors (NLRs) and the RIG-like helicases (RLHs) are soluble receptors present in the cytosol and act as sensors to detect a variety of viral and bacterial products.
- ^ Kato H, Takeuchi O, Mikamo-Satoh E, Hirai R, Kawai T, Matsushita K, et al. (July 2008). "Length-dependent recognition of double-stranded ribonucleic acids by retinoic acid-inducible gene-I and melanoma differentiation-associated gene 5". The Journal of Experimental Medicine. 205 (7): 1601–10. doi:10.1084/jem.20080091. PMC 2442638. PMID 18591409.
- ^ Saito T, Owen DM, Jiang F, Marcotrigiano J, Gale M (July 2008). "Innate immunity induced by composition-dependent RIG-I recognition of hepatitis C virus RNA". Nature. 454 (7203): 523–7. Bibcode:2008Natur.454..523S. doi:10.1038/nature07106. PMC 2856441. PMID 18548002.
- ^ Baum A, Sachidanandam R, García-Sastre A (September 2010). "Preference of RIG-I for short viral RNA molecules in infected cells revealed by next-generation sequencing". Proceedings of the National Academy of Sciences of the United States of America. 107 (37): 16303–8. Bibcode:2010PNAS..10716303B. doi:10.1073/pnas.1005077107. PMC 2941304. PMID 20805493.
- ^ Gitlin L, Barchet W, Gilfillan S, Cella M, Beutler B, Flavell RA, et al. (May 2006). "Essential role of mda-5 in type I IFN responses to polyriboinosinic:polyribocytidylic acid and encephalomyocarditis picornavirus". Proceedings of the National Academy of Sciences of the United States of America. 103 (22): 8459–64. Bibcode:2006PNAS..103.8459G. doi:10.1073/pnas.0603082103. PMC 1464000. PMID 16714379.
- ^ Hornung V, Ellegast J, Kim S, Brzózka K, Jung A, Kato H, et al. (November 2006). "5'-Triphosphate RNA is the ligand for RIG-I". Science. 314 (5801): 994–7. Bibcode:2006Sci...314..964H. doi:10.1126/science.1132505. PMID 17038590. S2CID 22436759.
- ^ Wang H, Ryu WS (July 2010). "Hepatitis B virus polymerase blocks pattern recognition receptor signaling via interaction with DDX3: implications for immune evasion". PLOS Pathogens. 6 (7): e1000986. doi:10.1371/journal.ppat.1000986. PMC 2904777. PMID 20657822.
- ^ Schlee M, Roth A, Hornung V, Hagmann CA, Wimmenauer V, Barchet W, et al. (July 2009). "Recognition of 5' triphosphate by RIG-I helicase requires short blunt double-stranded RNA as contained in panhandle of negative-strand virus". Immunity. 31 (1): 25–34. doi:10.1016/j.immuni.2009.05.008. PMC 2824854. PMID 19576794.
- ^ Feng Q, Hato SV, Langereis MA, Zoll J, Virgen-Slane R, Peisley A, et al. (November 2012). "MDA5 detects the double-stranded RNA replicative form in picornavirus-infected cells". Cell Reports. 2 (5): 1187–96. doi:10.1016/j.celrep.2012.10.005. PMC 7103987. PMID 23142662.
- ^ Kato H, Takeuchi O, Sato S, Yoneyama M, Yamamoto M, Matsui K, et al. (May 2006). "Differential roles of MDA5 and RIG-I helicases in the recognition of RNA viruses". Nature. 441 (7089): 101–5. Bibcode:2006Natur.441..101K. doi:10.1038/nature04734. PMID 16625202. S2CID 2270879.
- ^ Li X, Ranjith-Kumar CT, Brooks MT, Dharmaiah S, Herr AB, Kao C, Li P (May 2009). "The RIG-I-like receptor LGP2 recognizes the termini of double-stranded RNA". The Journal of Biological Chemistry. 284 (20): 13881–91. doi:10.1074/jbc.m900818200. PMC 2679488. PMID 19278996.
- ^ Murali A, Li X, Ranjith-Kumar CT, Bhardwaj K, Holzenburg A, Li P, Kao CC (June 2008). "Structure and function of LGP2, a DEX(D/H) helicase that regulates the innate immunity response". The Journal of Biological Chemistry. 283 (23): 15825–33. doi:10.1074/jbc.m800542200. PMC 2414291. PMID 18411269.
- ^ a b Bruns AM, Leser GP, Lamb RA, Horvath CM (September 2014). "The innate immune sensor LGP2 activates antiviral signaling by regulating MDA5-RNA interaction and filament assembly". Molecular Cell. 55 (5): 771–81. doi:10.1016/j.molcel.2014.07.003. PMC 4156907. PMID 25127512.
- ^ a b Satoh T, Kato H, Kumagai Y, Yoneyama M, Sato S, Matsushita K, et al. (January 2010). "LGP2 is a positive regulator of RIG-I- and MDA5-mediated antiviral responses". Proceedings of the National Academy of Sciences of the United States of America. 107 (4): 1512–7. Bibcode:2010PNAS..107.1512S. doi:10.1073/pnas.0912986107. PMC 2824407. PMID 20080593.
- ^ Takahasi K, Kumeta H, Tsuduki N, Narita R, Shigemoto T, Hirai R, et al. (June 2009). "Solution structures of cytosolic RNA sensor MDA5 and LGP2 C-terminal domains: identification of the RNA recognition loop in RIG-I-like receptors". The Journal of Biological Chemistry. 284 (26): 17465–74. doi:10.1074/jbc.m109.007179. PMC 2719387. PMID 19380577.
- ^ Childs KS, Randall RE, Goodbourn S (2013-05-09). "LGP2 plays a critical role in sensitizing mda-5 to activation by double-stranded RNA". PLOS ONE. 8 (5): e64202. Bibcode:2013PLoSO...864202C. doi:10.1371/journal.pone.0064202. PMC 3650065. PMID 23671710.
- ^ Komuro A, Horvath CM (December 2006). "RNA- and virus-independent inhibition of antiviral signaling by RNA helicase LGP2". Journal of Virology. 80 (24): 12332–42. doi:10.1128/jvi.01325-06. PMC 1676302. PMID 17020950.
- ^ Parisien JP, Lenoir JJ, Mandhana R, Rodriguez KR, Qian K, Bruns AM, Horvath CM (June 2018). "RNA sensor LGP2 inhibits TRAF ubiquitin ligase to negatively regulate innate immune signaling". EMBO Reports. 19 (6): e45176. doi:10.15252/embr.201745176. PMC 5989757. PMID 29661858.
- ^ Saito T, Hirai R, Loo YM, Owen D, Johnson CL, Sinha SC, et al. (January 2007). "Regulation of innate antiviral defenses through a shared repressor domain in RIG-I and LGP2". Proceedings of the National Academy of Sciences of the United States of America. 104 (2): 582–7. Bibcode:2007PNAS..104..582S. doi:10.1073/pnas.0606699104. PMC 1766428. PMID 17190814.
- ^ Uchikawa E, Lethier M, Malet H, Brunel J, Gerlier D, Cusack S (May 2016). "Structural Analysis of dsRNA Binding to Anti-viral Pattern Recognition Receptors LGP2 and MDA5". Molecular Cell. 62 (4): 586–602. doi:10.1016/j.molcel.2016.04.021. PMC 4885022. PMID 27203181.
- ^ Luo D, Ding SC, Vela A, Kohlway A, Lindenbach BD, Pyle AM (October 2011). "Structural insights into RNA recognition by RIG-I". Cell. 147 (2): 409–22. doi:10.1016/j.cell.2011.09.023. PMC 3222294. PMID 22000018.
- ^ Berke IC, Modis Y (April 2012). "MDA5 cooperatively forms dimers and ATP-sensitive filaments upon binding double-stranded RNA". The EMBO Journal. 31 (7): 1714–26. doi:10.1038/emboj.2012.19. PMC 3321199. PMID 22314235.
- ^ Gack MU, Nistal-Villán E, Inn KS, García-Sastre A, Jung JU (April 2010). "Phosphorylation-mediated negative regulation of RIG-I antiviral activity". Journal of Virology. 84 (7): 3220–9. doi:10.1128/jvi.02241-09. PMC 2838087. PMID 20071582.
- ^ Maharaj NP, Wies E, Stoll A, Gack MU (February 2012). "Conventional protein kinase C-α (PKC-α) and PKC-β negatively regulate RIG-I antiviral signal transduction". Journal of Virology. 86 (3): 1358–71. doi:10.1128/jvi.06543-11. PMC 3264329. PMID 22114345.
- ^ Nistal-Villán E, Gack MU, Martínez-Delgado G, Maharaj NP, Inn KS, Yang H, et al. (June 2010). "Negative role of RIG-I serine 8 phosphorylation in the regulation of interferon-beta production". The Journal of Biological Chemistry. 285 (26): 20252–61. doi:10.1074/jbc.m109.089912. PMC 2888438. PMID 20406818.
- ^ Wies E, Wang MK, Maharaj NP, Chen K, Zhou S, Finberg RW, Gack MU (March 2013). "Dephosphorylation of the RNA sensors RIG-I and MDA5 by the phosphatase PP1 is essential for innate immune signaling". Immunity. 38 (3): 437–49. doi:10.1016/j.immuni.2012.11.018. PMC 3616631. PMID 23499489.
- ^ Gack MU, Shin YC, Joo CH, Urano T, Liang C, Sun L, et al. (April 2007). "TRIM25 RING-finger E3 ubiquitin ligase is essential for RIG-I-mediated antiviral activity". Nature. 446 (7138): 916–920. Bibcode:2007Natur.446..916G. doi:10.1038/nature05732. PMID 17392790. S2CID 4404114.
- ^ a b Gack MU, Albrecht RA, Urano T, Inn KS, Huang IC, Carnero E, et al. (May 2009). "Influenza A virus NS1 targets the ubiquitin ligase TRIM25 to evade recognition by the host viral RNA sensor RIG-I". Cell Host & Microbe. 5 (5): 439–49. doi:10.1016/j.chom.2009.04.006. PMC 2737813. PMID 19454348.
- ^ a b Davis ME, Wang MK, Rennick LJ, Full F, Gableske S, Mesman AW, et al. (July 2014). "Antagonism of the phosphatase PP1 by the measles virus V protein is required for innate immune escape of MDA5". Cell Host & Microbe. 16 (1): 19–30. doi:10.1016/j.chom.2014.06.007. PMC 4120867. PMID 25011105.
- ^ Zhang HL, Ye HQ, Liu SQ, Deng CL, Li XD, Shi PY, Zhang B (September 2017). "West Nile Virus NS1 Antagonizes Interferon Beta Production by Targeting RIG-I and MDA5". Journal of Virology. 91 (18). doi:10.1128/jvi.02396-16. PMC 5571242. PMID 28659477.
- ^ Li XD, Sun L, Seth RB, Pineda G, Chen ZJ (December 2005). "Hepatitis C virus protease NS3/4A cleaves mitochondrial antiviral signaling protein off the mitochondria to evade innate immunity". Proceedings of the National Academy of Sciences of the United States of America. 102 (49): 17717–22. doi:10.1073/pnas.0508531102. PMC 1308909. PMID 16301520.
- ^ Rodríguez Pulido M, Sánchez-Aparicio MT, Martínez-Salas E, García-Sastre A, Sobrino F, Sáiz M (June 2018). "Innate immune sensor LGP2 is cleaved by the Leader protease of foot-and-mouth disease virus". PLOS Pathogens. 14 (6): e1007135. doi:10.1371/journal.ppat.1007135. PMC 6042790. PMID 29958302.
- ^ Angleró-Rodríguez YI, Pantoja P, Sariol CA (January 2014). "Dengue virus subverts the interferon induction pathway via NS2B/3 protease-IκB kinase epsilon interaction". Clinical and Vaccine Immunology. 21 (1): 29–38. doi:10.1128/cvi.00500-13. PMC 3910921. PMID 24173023.
- ^ Dalrymple NA, Cimica V, Mackow ER (May 2015). "Dengue Virus NS Proteins Inhibit RIG-I/MAVS Signaling by Blocking TBK1/IRF3 Phosphorylation: Dengue Virus Serotype 1 NS4A Is a Unique Interferon-Regulating Virulence Determinant". mBio. 6 (3): e00553-15. doi:10.1128/mbio.00553-15. PMC 4436066. PMID 25968648.
- ^ He Z, Zhu X, Wen W, Yuan J, Hu Y, Chen J, et al. (August 2016). "Dengue Virus Subverts Host Innate Immunity by Targeting Adaptor Protein MAVS". Journal of Virology. 90 (16): 7219–7230. doi:10.1128/jvi.00221-16. PMC 4984625. PMID 27252539.
- ^ Ma J, Ketkar H, Geng T, Lo E, Wang L, Xi J, et al. (2018-06-25). "Zika Virus Non-structural Protein 4A Blocks the RLR-MAVS Signaling". Frontiers in Microbiology. 9: 1350. doi:10.3389/fmicb.2018.01350. PMC 6026624. PMID 29988497.
- ^ Andrejeva J, Childs KS, Young DF, Carlos TS, Stock N, Goodbourn S, Randall RE (December 2004). "The V proteins of paramyxoviruses bind the IFN-inducible RNA helicase, mda-5, and inhibit its activation of the IFN-beta promoter". Proceedings of the National Academy of Sciences of the United States of America. 101 (49): 17264–9. Bibcode:2004PNAS..10117264A. doi:10.1073/pnas.0407639101. PMC 535396. PMID 15563593.
- ^ Childs K, Stock N, Ross C, Andrejeva J, Hilton L, Skinner M, et al. (March 2007). "mda-5, but not RIG-I, is a common target for paramyxovirus V proteins". Virology. 359 (1): 190–200. doi:10.1016/j.virol.2006.09.023. PMID 17049367.
- ^ Rodriguez KR, Horvath CM (July 2014). "Paramyxovirus V protein interaction with the antiviral sensor LGP2 disrupts MDA5 signaling enhancement but is not relevant to LGP2-mediated RLR signaling inhibition". Journal of Virology. 88 (14): 8180–8. doi:10.1128/jvi.00737-14. PMC 4097766. PMID 24829334.



